对DNA片段进行标记的单分子标签
文献发布时间:2023-06-19 12:13:22
本申请是申请日为
技术领域
本发明涉及分子生物学领域,尤其涉及一种对DNA片段进行标记的单分子标签。
背景技术
DNA测序作为一种非常重要的实验技术,在生物学研究领域中有着广泛应用,特别是在基因检测行业。从最初的末端终止法,到目前比较成熟的第二代测序技术(Next-Generation Sequencing)。目前的二代测序较一代测序而言,费用更低,通量更高,速度更快。
Illumina测序的基本原理是边合成边测序。在文库构建过程中,需要将基因组DNA随机片段化,并在片段化后的基因组两端添加特定测序接头(Adaptor),然后进行文库PCR扩增。由于最后的PCR扩增和片段化的随机性,使得最终文库测序的每个读长存在一个问题,即不确定所得读长最初是由哪个模版扩增而来。CN106834503A提供了一种引物标签结合深度测序法从外周血游离DNA扩增癌症基因的引物组、引物标签设计方法及应用,该发明通过给每个起始样品DNA分子模板两边加标签的方式,给模板中的每个DNA分子都贴上分子标签,在经过PCR和测序以后,可以通过分子标签将样本中原始的DNA分子都鉴别分类,进而可以通过标签去除或降低NGS测序中PCR扩增以及测序阶段引入的系统误差。CN107254514A公开了一种检测异源cfDNA的SNP分子标记,基于SNP分子标记设计的检测探针和芯片,以及异源cfDNA的检测装置、试剂盒和检测方法;SNP分子标记中包括药物代谢基因的SNP位点,为肾移植排斥患者提供用药基因的突变信息,实现个体化用药;该发明还提供了一种单分子标记接头,单分子标记接头用于构建测序文库有效去除重复数据和测序、PCR过程中随机引入的错误,降低测序背景噪音,提高了检测的准确性。但上述现有技术均无法对测序读长进行有效溯源的目标,且操作步骤复杂,对设备要求高,难以应用推广。
综上,研发一种单分子标签及其应用,对每条最初的DNA片段进行标记,从而在最终测序结果中呈现每条读长的具体来源,具有广阔的应用前景和巨大的市场价值。
发明内容
针对现有技术的不足及实际的需求,本发明提供一种单分子标签及其应用,通过设计特异性的标签,并选择与标签相适配的引入方法,在构建文库前期对DNA样品进行处理,以实现对每条最初的DNA片段进行标记,从而在最终测序结果中呈现每条读长的具体来源,
为达此目的,本发明采用以下技术方案:
第一方面,本发明提供一种单分子标签,所述标签为一条oligo退火形成的茎环结构、两条oligo退火形成两端互补配对的结构或两条oligo退火形成一端互补配对的结构中的任意一种,其中,所述标签的3’端突出,5’端磷酸化,在未互补配对的环状结构上,有尿嘧啶修饰。
优选地,所述标签的尿嘧啶修饰的个数为至少1个。
优选地,所述标签的3’端突出为悬突胸腺嘧啶。
在本发明中,发明人在深入研究DNA测序技术的基础上,分析Illumina测序的优缺点,发现由于PCR扩增和片段化的随机性,导致无法得知最终测序文库的所得读长的来源模板,因此,为实现对测序样本的单分子编码测序,发明人通过大量实验并结合创造性思路,研发了一种特异性标签,最终实现对每条DNA片段进行标记,从而在最终的DNA文库测序结果中呈现出每条读长的具体来源。
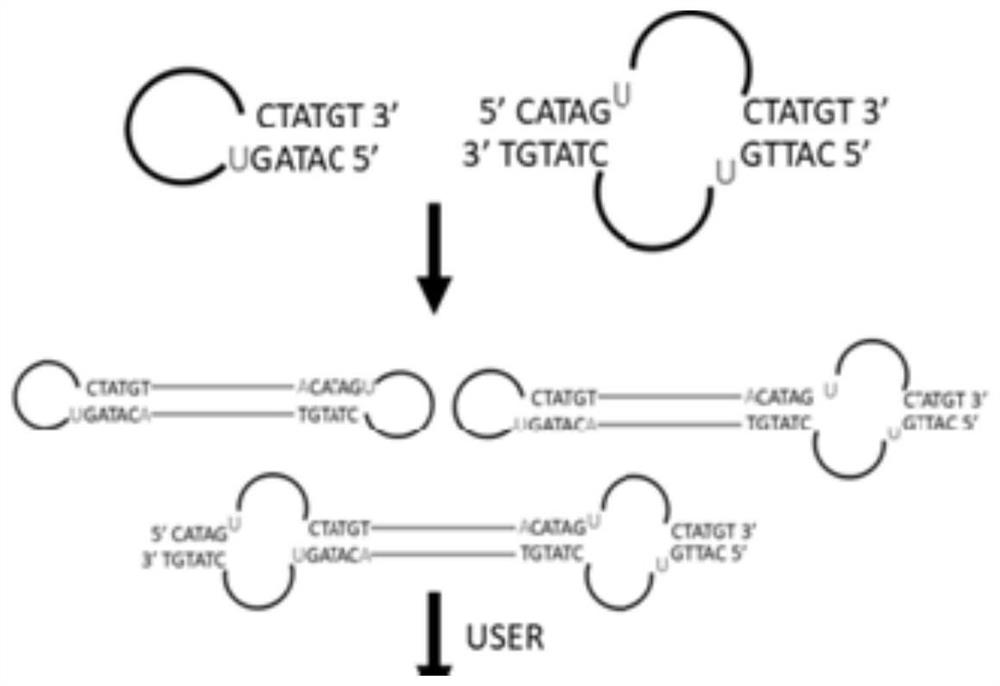
本发明中,所述标签存在三种情况,当一条链退火形成茎环结构;第二种情况:当两条链发生两端互补配对时形成结构;第三种情况:当两条链发生一端互补配对形成结构,三种情况示意图如图1。
第二方面,本发明提供一种标签引入方法,包括如下步骤:
(1)将第一方面所述标签连接至DNA样本上;
(2)将步骤(1)得到的产物进行处理形成5’端突出的结构;
(3)将步骤(2)得到的产物的DNA末端去磷酸化,形成末端羟基的结构;
(4)将步骤(3)得到的产物进行延伸反应形成两端为平末端且带标签的DNA片段。
在本发明中,若步骤(1)所述DNA样本为gDNA,所述方法还包括首先对DNA样品进行片段化处理的步骤,若被处理样本为cfDNA,可以直接用于后续实验过程。
优选地,所述片段化的步骤如下:
(1’)采用片段化酶将gDNA进行片段化处理;
(2’)对步骤(1’)的产物进行筛选。
优选地,所述筛选的方法包括磁珠双筛或切胶。
优选地,所述筛选的片段长度为100-250bp。
优选地,步骤(1)所述DNA样品在连接标签前需要进行末端修复和3’端加腺嘌呤的处理。
优选地,步骤(2)所述处理采用的酶包括尿嘧啶-特异性切除酶,优选为USER酶(NEB M5505 V/S/L)。
优选地,步骤(3)所述去磷酸化的酶包括磷酸酶,优选为重组虾碱性磷酸酶(rSAPNEB M0371V/S/L)。
优选地,所述方法还包括将步骤(4)得到的平末端且带标签的DNA片段进行DNA末端修复,3’末端加A的处理,为后续接头连接做准备。。
第三方面,本发明还提供一种试剂盒,所述试剂盒包含第一方面所述的标签。
第四方面,本发明还提供一种如第一方面所述标签和/或第三方面所述试剂盒用于构建基因测序文库、用于DNA双端单分子编码标记或用于定位测序读长的有效来源,后续用于测序结果的分析解读。
与现有技术相比,本发明具有如下有益效果:
(1)本发明提供的标签构思巧妙,结构精简,功能显著,通过选择特定结构和修饰后的标签,实现对DNA双端单分子编码标记,更为有效的定位测序读长的最初来源,解决了现有技术由于PCR扩增和片段化的随机性,无法确定所得读长最初是由哪个模板扩增而来的问题。
(2)本发明提供的标签引入方法简洁高效,与特定标签相适配,便于操作,辅助发挥并实现了标签的标记功能,节省人力物力,具有推广应用的前景和价值。
附图说明
图1为本发明的标签的三种情况示意图;
图2为本发明的随机标签(Barcode)引入DNA片段两端的示意图。
具体实施方式
为更进一步阐述本发明所采取的技术手段及其效果,以下结合附图并通过具体实施方式来进一步说明本发明的技术方案,但本发明并非局限在实施例范围内。
实施例中,相关术语解释:
(1)甲基化修饰引物:引物序列中,胞嘧啶(C)被甲基化处理为5-甲基胞嘧啶(5-mdC)。
(2)Barcode:随机标签。
(3)纯化:如无特殊标注,实施例中的“纯化”指的是磁珠纯化。
(4)退火:两条或一条全部或部分反向互补的DNA oligo在一定反应条件下,结合为双链DNA。
(5)P5/P7:Illumina二代文库构建过程终文库扩增的通用引物。
(6)加A:在DNA片段3’末端加上一个腺嘌呤碱基(A)。
实施例1
(1)一种标签,所述标签的核苷酸序列如SEQ ID NO.1所示:
SEQ ID NO.1即Oligo:
Oligo的5’端磷酸化修饰(
(2)样本处理:
若被处理样本为gDNA,利用NEB公司的片段化酶将2μg样本gDNA片段化处理,通过磁珠双筛或者切胶的方法选择100-250bp的DNA片段,片段化酶处理条件:37℃,43min,将片段选择后的DNA用30μL去离子水溶解;
若被处理样本为cfDNA,可以直接用于后续实验过程。
(3)片段化DNA末端修复,3’端加A:
利用试剂盒NEBNext Ultra II DNA Library Prep Kit for Illumina进行DNA末端修复,3’端加A。
(4)标签Barcode连接:上述产物不需经过纯化,按表1所示体系进行反应,随机标签(Barcode)引入DNA片段两端的示意图如图2所示:
表1
(5)在上述产物中加入3μL的USER酶和3μL的重组虾碱性磷酸酶(rSAP),37℃反应30min,见表2;
表2
(6)标签延伸:将上述产物纯化后按表3所示体系进行反应:
表3
PCR循环条件如表4所示:
表4
(7)对上述产物进行纯化,利用试剂盒NEBNext Ultra II DNA Library Prep Kitfor Illumina进行DNA末端修复,3’端加A,接头连接,纯化。
(8)终文库扩增:将上述纯化产物进行如表5所示反应:
表5
PCR循环条件如表6所示:
表6
(11)对上述产物进行纯化,-20℃保存。
实施例2
(1)一种标签,所述标签的核苷酸序列如SEQ ID NO.1所示:
SEQ ID NO.1即Oligo:
Oligo的5’端磷酸化修饰(
(2)样本处理:
若被处理样本为gDNA,利用NEB公司的片段化酶将2μg样本gDNA片段化处理,通过磁珠双筛或者切胶的方法选择100-250bp的DNA片段,片段化酶处理条件:37℃,43min,将片段选择后的DNA用30μL去离子水溶解;
若被处理样本为cfDNA,可以直接用于后续实验过程。
(3)片段化DNA末端修复,3’端加A:
利用试剂盒NEBNext Ultra II DNA Library Prep Kit for Illumina进行DNA末端修复,3’端加A。
(4)标签Barcode连接:上述产物不需经过纯化,按表7所示体系进行反应:
表7
(5)在上述产物中加入3μL的USER酶和3μL的重组虾碱性磷酸酶(rSAP),37℃反应30min,见表8;
表8
(6)标签延伸:将上述产物纯化后按表9所示体系进行反应:
表9
PCR循环条件如表10所示:
表10
(8)对上述产物进行纯化,利用试剂盒NEBNext Ultra II DNA Library Prep Kitfor Illumina进行DNA末端修复,3’端加A,甲基化接头连接。
(9)对上述产物进行纯化,纯化后按照ZYMO EZ DNA Methylation Gold Kit进行重亚硫酸盐处理、柱子纯化。
(10)终文库扩增加INDEX:将上述纯化产物进行如表11所示反应:
表11
PCR循环条件如表12所示:
表12
(10)对上述产物进行纯化,-20℃保存。
综上所述,本发明提供一种标签及其应用,通过选择特定结构和修饰后的标签,实现对DNA双端单分子编码标记,更为有效的定位测序读长的最初来源,解决了现有技术由于PCR扩增和片段化的随机性,无法确定所得读长最初是由哪个模板扩增而来的问题;提供的标签引入方法简洁高效,与特定标签相适配,便于操作,辅助发挥并实现了标签的标记功能,节省人力物力,具有推广应用的前景和价值。
申请人声明,本发明通过上述实施例来说明本发明的详细方法,但本发明并不局限于上述详细方法,即不意味着本发明必须依赖上述详细方法才能实施。所属技术领域的技术人员应该明了,对本发明的任何改进,对本发明产品各原料的等效替换及辅助成分的添加、具体方式的选择等,均落在本发明的保护范围和公开范围之内。
SEQUENCE LISTING
<110> 深圳海普洛斯医学检验实验室
<120> 一种对DNA片段进行标记的单分子标签
<160> 1
<170> PatentIn version 3.5
<210> 1
<211> 21
<212> DNA
<213> 人工序列
<220>
<221> misc_feature
<222> (7)..(14)
<223> n is a, c, g, or t
<400> 1
catagdunnn nnnnnctatg t 21
