RNA快速建库方法及其应用
文献发布时间:2023-06-19 12:24:27
技术领域
本发明专利涉及一种RNA快速建库方法,属于生物技术领域。
背景技术
RNA二代测序技术(RNA Next-generation sequencing,RNA-seq)是一种高通量大规模的RNA并行测序技术,可以同时对几十万乃至几百万个RNA分子进行序列测定,用于未知病原的鉴定、生物遗传进化分析、基因表达差异分析和RNA合成及加工分析等。因此,RNA-seq广泛用于科学研究和疾病诊断等领域,并取得了许多突破性的结果。RNA NGS文库构建是指通过逆转录和接头连接等过程将RNA转化为二代测序仪可识别的双链DNA的过程,是RNA-seq的关键步骤。传统的RNA建库方法需要的RNA投入量较大(1ng以上),因此建库需要的细胞量较多(每个人类细胞中的总RNA量低于10pg)。此外,传统的RNA建库技术获得的RNANGS文库无法区分RNA的具体细胞来源,RNA-seq获得的数据只能分析样本中RNA丰度的平均值,而不能计算出样本中每个细胞的RNA表达丰度。这对于异质性较大的样本如血液样本、肿瘤组织样本、羊水样本、胚胎样本和小型动植物个体等的RNA种类及丰度的检测是极为不利的,在很大程度上影响了疾病的诊断和生长发育进程的分析。
单细胞转录组测序技术(Single cell RNA sequencing,scRNA-seq)的出现,为解决样本异质性问题提供了有效的方案。通过激光捕获显微切割技术、流式细胞仪技术、微滴技术和微流体技术等单细胞分离技术获取独立的单个细胞,并通过逆转录反应为每个细胞的cDNA加上具有独立标识DNA序列(Cell barcode),在测序数据分析过程中通过这个独立标识的DNA序列鉴别每条RNA序列的细胞来源,从而分析样本中每个细胞中的RNA丰度。scRNA-seq技术大大提高了我们对生物系统和细胞病变的了解,使研究者能够以前所未有的分辨率研究基因表达图谱。scRNA-seq为分析和诊断疾病进程、血液细胞分型、产前诊断和生物生长发育提供了更加高效准确的工具与方案,因此受到科学研究和疾病诊治领域的广泛关注。但是,scRNA-seq需要对单个细胞的RNA进行文库构建,这极大地提高了RNA建库的难度。传统的RNA建库技术操作复杂、损失严重,因此无法应用在scRNA-seq的文库构建上。为解决这一问题,研究者们开发了多种单细胞RNA文库构建方法,来提高RNA文库构建的效率,减少建库过程的损失,如Smart-seq、CEL-seq、Quartz-seq、SUPeR-seq和MATQ-seq等。但这些scRNA-seq技术由于建库设计的原因,存在基因检出率低、RNA信息完整度差和操作复杂等缺陷,严重阻碍了scRNA-seq技术的发展和应用。因此,开发一种更加高效的单细胞转录组测序技术,对于疾病诊断和生物生长发育等研究,具有重要的意义。
本发明提供一种高效快速的RNA建库技术,称为Easy-seq(Easy RNAsequencing)。相较于传统的RNA建库方法,Easy-seq具有耗时短、操作简单和损失小等优点,因此可以应用于单细胞转录组测序(scEasy-seq)。相较于已有的scRNA-seq技术,scEasy-seq具有基因检出率高、RNA信息完整和建库产量高等优点,是一种高效灵敏的单细胞转录组测序技术,可以广泛应用于科学研究和疾病诊断等领域。
发明内容
本发明的目的是提供一种RNA快速建库方法,具有耗时短、操作简单和损失小的优点。
本发明采取的技术方案为:一种RNA快速建库方法,其特征在于其步骤包括:
(1)提取样本中的total RNA,采用逆转录阻碍探针法去除rRNA,同时逆转录获得一端带有P7序列的cDNA;
(2)对多余的随机引物进行消解处理;
(3)对cDNA进行多聚腺苷酸化,在cDNA的3’端加上polyA,采用带有3’突出末端的双链DNA接头,在cDNA的另一端接上P5序列;
(4)文库扩增及回收。
优选的,步骤(1)中使用的逆转录引物的5’端带有P7序列和识别细胞的独立标识DNA序列。
优选的,步骤(3)中双链DNA接头的3’突出末端为一段多聚胸腺嘧啶的单链DNA序列,与cDNA的多聚腺苷酸配对。
优选的,步骤(3)中多聚胸腺嘧啶的数量为10-15个。
优选的,步骤(2)中消解处理使用的酶为T4 DNA聚合酶、Exonuclease I、Exonuclease T的一种或多种。
优选的,步骤(2)中消解处理的温度为37℃,时间为10-60分钟。
优选的,步骤(3)中多聚腺苷酸化和接头连接同时进行,采用的酶混合物包括TdT,还包括T4 DNA连接酶、Taq DNA连接酶或E.coli DNA连接酶的一种或多种。
优选的,步骤(1)中逆转录引物序列为:ACACTCTTTCCCTACACGACGCTCTTCCGATCT+细胞识别序列+NNNNNNNN;
步骤(3)中的接头序列为:F:/5Phos/分子识别标志+ACACTCTTTCCCTACACGACGCTCTTCCGATCT/NH2C6/;
R:AGATCGGAAGAGCGTCGTGTAGGGAAAGAGTGT+分子识别标志+poly(dT)/NH2C6/,其中poly(dT)的数量为10-15个。
其中细胞识别序列可用于区分细胞来源,分子识别标志用于区分原始的DNA分子拷贝。
优选的,步骤(3)中反应温度为37℃,时间为10-30分钟。
本发明公开的上述的RNA快速建库方法可应用于单细胞转录组测序。
本发明目的在于提供一种高效快速的RNA建库技术,称为(Easy RNAsequencing)。相较于传统的RNA建库方法,Easy-seq具有耗时短、操作简单和损失小等优点,因此可以应用于单细胞转录组测序(scEasy-seq)。相较于已有的scRNA-seq技术,scEasy-seq具有基因检出率高、RNA信息完整和建库产量高等优点,是一种高效灵敏的单细胞转录组测序技术,可以广泛应用于科学研究和疾病诊断等领域。
附图说明
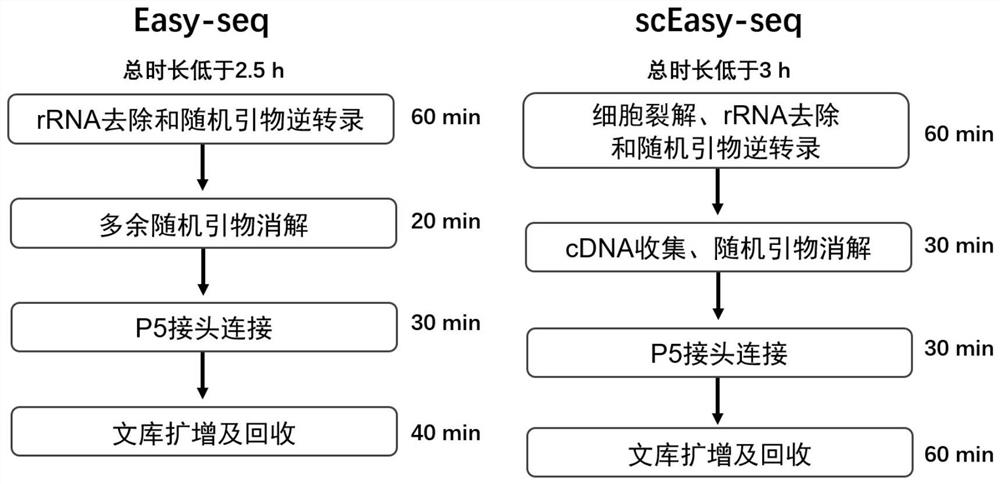
图1Easy-seq和scEasy-seq流程。
图2Easy-seq和scEasy-seq建库原理示意图。
图3Easy-seq与传统RNA-seq在文库产量上对比。
图4Easy-seq与传统RNA-seq的文库大小对比。
图5Easy-seq对不同RNA投入量建库的文库大小。
图6scEasy-seq与其他三种scRNA-seq的原理比较。
图7scEasy-seq与其他三种scRNA-seq的特点比较。
图8scEasy-seq与其他三种scRNA-seq的文库大小分布比较。
图9scEasy-seq与其他三种scRNA-seq的文库产量比较。
图10scEasy-seq与其他三种scRNA-seq的转录组比对率比较。
图11scEasy-seq与其他三种scRNA-seq的rRNA残留率比较。
图12scEasy-seq与其他三种scRNA-seq的基因检出数比较。
图13scEasy-seq与其他三种scRNA-seq的基因区域覆盖均一性比较。
图14scEasy-seq结合微流控进行scRNA-seq流程示意图。
具体实施方式
下面结合附图对本发明的具体实施方式做进一步说明。
本实施例所使用的探针和引物序列及修饰如表1所示,N为随机碱基,即A、T、C、G中任意一种碱基。
表1探针及引物序列
实施例1:Easy-seq流程的建立
在本实施例中,我们组建了Easy-seq的流程,流程和原理示意图见图1。具体方式如下:
1)rRNA去除、逆转录和随机引物消解:
表2
94℃ 5min,75℃ 1min,55℃ 1min,4℃保存。
表3
25℃ 10min,42℃ 30min,85℃ 5min,4℃保存。加入1μL T4 DNA聚合酶(5U/μL,ThermoFisher),37℃ 30min,85℃ 10min,立即置于冰上。
2)P5接头连接
P5接头退火:将100μM P5-F与100μM P5-R1溶解于1×Annealing buffer(10mMTris-HCl,50mM NaCl,1mM EDTA,pH 7.9)中,吸取等体积混匀,94℃ 5min,94-15℃ 0.1℃/min,15℃保存。退火完成后将接头稀释成10μM终浓度。
表4
10×P5连接缓冲液:85mM MgCl2,0.26mM NAD+,pH 7.9。
37℃ 60min,95℃ 2min,保存于4℃。
3)文库扩增及回收
表5
98℃ 3min,7cycles(98℃ 10s,60℃ 30s,72℃ 30s),72℃ 3min,保存于4℃。
反应结束后,加入45μL Agencourt AMPure XP beads(Beckman,A63881)混匀后,室温静置孵育5min。将PCR管置于磁力架上,待溶液澄清后,吸去上清;加入200μL新鲜配制的80%乙醇静置30s,吸去上清;加入200μL新鲜配制的80%乙醇静置30s,吸干净乙醇。室温静置3min,晾干乙醇,加入20μL无核酸酶水悬浮磁珠。室温静置5min,将PCR管置于磁力架上,待溶液澄清后,吸出上清。
实施例2:不同外切酶消化对Easy-seq文库产量的影响。
在本实施例中,我们验证了3种不同外切酶消化方法对Easy-seq文库产量的影响。具体方式如下:
1)rRNA去除、逆转录和随机引物消解:
表6
94℃ 5min,75℃ 1min,55℃ 1min,4℃保存。
表7
25℃ 10min,42℃ 30min,85℃ 5min,4℃保存。
外切酶条件1:加入1μL T4 DNA聚合酶(5U/μL,ThermoFisher),37℃ 30min,85℃10min,立即置于冰上。
外切酶条件2:加入0.25μL Exonuclease I(20U/μL,NEB),37℃ 30min,85℃10min,立即置于冰上。
外切酶条件3:加入1μL Exonuclease T(5U/μL,ThermoFisher),37℃ 30min,85℃10min,立即置于冰上。
按照实施例1进行P5接头连接和文库扩增及回收。结果如下表所示:
表8三种外切酶消解方式对Easy-seq文库产量的影响
结果如表8所示,三种外切酶的消化都具有良好的文库产量,其中T4 DNA聚合酶消化未参与逆转录的引物,Easy-seq的文库产量最高。
实施例3:不同T4 DNA聚合酶消化时间对Easy-seq文库产量的影响。
在本实施例中,我们验证了不同T4 DNA聚合酶消化时间对Easy-seq文库产量的影响。
具体方式如下:
1)rRNA去除、逆转录和随机引物消解:
表9
94℃ 5min,75℃ 1min,55℃ 1min,4℃保存。
表10
25℃ 10min,42℃ 30min,85℃ 5min,4℃保存。
加入1μL T4 DNA聚合酶(5U/μL,ThermoFisher),37℃ x min,85℃ 10min,立即置于冰上。(x为10、20、30、40、50、60)
按照实施例1进行P5接头连接和文库扩增及回收。结果如下表所示:
表11不同T4 DNA聚合酶消化时间对Easy-seq文库产量的影响
结果如表11所示,T4 DNA聚合酶的消化时间在10-20min时,Easy-seq的文库产量最高。为保证逆转录引物消解充分,选定T4 DNA聚合酶的消化时间为20min。
实施例4:不同连接酶对Easy-seq文库产量的影响。
在本实施例中,我们验证了不同T4 DNA聚合酶消化时间对Easy-seq文库产量的影响。
具体方式如下:
1)rRNA去除、逆转录和随机引物消解:见实施例3。
2)P5接头连接
P5接头退火:将100μM P5-F分别与100μM P5-R1溶解于1×Annealing buffer(10mM Tris-HCl,50mM NaCl,1mM EDTA,pH 7.9)中,吸取等体积混匀,94℃ 5min,94-15℃0.1℃/min,15℃保存。退火完成后将接头稀释成10μM终浓度。
表12
37℃ 60min,95℃ 2min,保存于4℃。
连接酶1:E.coli DNA ligase(60U/μl,TaKaRa)
连接酶2:T4 DNA ligase(400U/μl,NEB)
连接酶3:Taq DNA ligase(40U/μl,NEB)
按照实施例1进行文库扩增及回收。结果如下表所示:
表13不同DNA连接酶对Easy-seq文库产量的影响
结果如表13所示,使用E.coli DNA ligase时,Easy-seq的文库产量最高。
实施例5:DNA接头寡聚胸腺苷酸长度对Easy-seq文库产量的影响。
在本实施例中,我们验证了DNA接头寡聚胸腺苷酸长度对Easy-seq文库产量的影响。
具体方式如下:
1)rRNA去除、逆转录和随机引物消解:见实施例3。
2)P5接头连接
P5接头退火:将100μM P5-F与100μM P5-R(1-6)溶解于1×Annealing buffer(10mM Tris-HCl,50mM NaCl,1mM EDTA,pH 7.9)中,吸取等体积混匀,94℃ 5min,94-15℃0.1℃/min,15℃保存。退火完成后将接头稀释成10μM终浓度。
表14
37℃ 60min,95℃ 2min,保存于4℃。
按照实施例1进行文库扩增及回收。结果如下表所示:
表15 DNA接头寡聚胸腺苷酸长度对Easy-seq文库产量的影响
结果如表15所示,当DNA接头寡聚胸腺苷酸长度为12nt(P5-R2)时,Easy-seq的文库产量最高。
实施例6:DNA接头连接时间对Easy-seq文库产量的影响。
在本实施例中,我们验证了DNA接头连接时间对Easy-seq文库产量的影响。具体方式如下:
1)rRNA去除、逆转录和随机引物消解:见实施例3。
2)P5接头连接
P5接头退火:将100μM P5-F与100μM P5-R2溶解于1×Annealing buffer(10mMTris-HCl,50mM NaCl,1mM EDTA,pH 7.9)中,吸取等体积混匀,94℃ 5min,94-15℃ 0.1℃/min,15℃保存。退火完成后将接头稀释成10μM终浓度。
表16
37℃ x min,95℃ 2min,保存于4℃。(x为10,15,20,30,40,60)
按照实施例1进行文库扩增及回收。结果如下表所示:
表17 DNA接头连接时间对Easy-seq文库产量的影响
结果如表17所示,当DNA接头连接时间为15min时,Easy-seq的文库产量最高。
实施例7:Easy-seq与传统RNA建立方法的比较
根据实施例1-6,我们组建出最佳的Easy-seq流程。流程和原理示意图见图1和图2,具体流程如下:
1)rRNA去除、逆转录和随机引物消解:
表18
94℃ 5min,75℃ 1min,55℃ 1min,4℃保存。
表19
25℃ 10min,42℃ 30min,85℃ 5min,4℃保存。加入1μL T4 DNA聚合酶(5U/μL,ThermoFisher),37℃ 20min,85℃ 10min,立即置于冰上。
2)P5接头连接
P5接头退火:将100μM P5-F与100μM P5-R2溶解于1×Annealing buffer(10mMTris-HCl,50mM NaCl,1mM EDTA,pH 7.9)中,吸取等体积混匀,94℃ 5min,94-15℃ 0.1℃/min,15℃保存。退火完成后将接头稀释成10μM终浓度。
表20
10×P5连接缓冲液:85mM MgCl2,0.26mM NAD+,pH 7.9。
37℃ 15min,95℃ 2min,保存于4℃。
3)文库扩增及回收
表21
98℃ 3min,7cycles(98℃ 10s,60℃ 30s,72℃ 30s),72℃ 3min,保存于4℃。
反应结束后,加入45μL Agencourt AMPure XP beads(Beckman,A63881)混匀后,室温静置孵育5min。将PCR管置于磁力架上,待溶液澄清后,吸去上清;加入200μL新鲜配制的80%乙醇静置30s,吸去上清;加入200μL新鲜配制的80%乙醇静置30s,吸干净乙醇。室温静置3min,晾干乙醇,加入20μL无核酸酶水悬浮磁珠。室温静置5min,将PCR管置于磁力架上,待溶液澄清后,吸出上清。
在本实施例中,我们将优化好的Easy-seq与传统的RNA建库方法进行了对比。传统RNA建库方法使用
使用Qubit对构建好的文库进行定量,Qsep 100验证文库大小分布。构建好的文库在Illumina NovaSeq 6000平台上进行测序并分析。结果见表22、图3-图4。
表22 1000ng RNA投入量的文库产量
结果如表22和图3、图4所示,相较于传统RNA-seq方法,Easy-seq能够显著提高文库的产量。在比对率和rRNA去除效率上,Easy-seq和传统RNA-seq都能够高效去除rRNA,并具有很高的比对率。在基因检出数上,Easy-seq要比传统RNA-seq略高。这些结果表明,Easy-seq比传统RNA-seq具有更高的建库效率和产量。
实施例8:Easy-seq对不同RNA投入量的建库效果
在本实施例中,我们验证了Easy-seq在HEK293F 0.01ng-1000ng RNA投入量条件下的建库效果,实验流程见实施例1,PCR循环数和文库产量见表23,文库大小分布见图5。
表23
结果如表23和图5所示,Easy-seq可以对投入量为0.01-1000ng的RNA进行转录组建库,且具有较高的文库产量和基因检出率。这说明Easy-seq可以应用于单个细胞RNA起始量(10pg左右)的文库构建。
实施例9:Easy-seq在单细胞转录组建库过程的应用
在本实施例中,我们验证了Easy-seq对单个细胞来源样本的转录组建库效果,流程示意图见图1。具体方式如下:
1)细胞裂解,rRNA去除、逆转录和随机引物消解:
使用流式细胞分离技术分离出单个HEK293F细胞,置于5.5μL细胞裂解液中。
表24
94℃ 5min,75℃ 1min,55℃ 1min,4℃保存。
表25
25℃ 10min,42℃ 30min,85℃ 5min,4℃保存。加入1μL T4 DNA聚合酶(5U/μL,ThermoFisher),37℃ 30min,85℃ 10min,立即置于冰上。
2)P5接头连接
P5接头退火:将100μM P5-F与100μM P5-R2溶解于1×Annealing buffer(10mMTris-HCl,50mM NaCl,1mM EDTA,pH 7.9)中,吸取等体积混匀,94℃ 5min,94-15℃ 0.1℃/min,15℃保存。退火完成后将接头稀释成10μM终浓度。
表26
10×P5连接缓冲液:85mM MgCl2,0.26mM NAD+,pH 7.9。
37℃ 15min,95℃ 2min,保存于4℃。
3)文库扩增及回收
表27
98℃ 3min,25cycles(98℃ 10s,60℃ 30s,72℃ 30s),72℃ 3min,保存于4℃。
反应结束后,加入45μL Agencourt AMPure XP beads(Beckman,A63881)混匀后,室温静置孵育5min。将PCR管置于磁力架上,待溶液澄清后,吸去上清;加入200μL新鲜配制的80%乙醇静置30s,吸去上清;加入200μL新鲜配制的80%乙醇静置30s,吸干净乙醇。室温静置3min,晾干乙醇,加入20μL无核酸酶水悬浮磁珠。室温静置5min,将PCR管置于磁力架上,待溶液澄清后,吸出上清。
Smart-seq使用TaKaRa的
MATQ-seq按照Nature Methods(Sheng et al.,2017)的流程进行操作。
SUPeR-seq按照Genome Biology(Fan X.et al.,2015)的流程进行操作。
使用Qubit对构建好的文库进行定量,Qsep 100验证文库大小分布。构建好的文库在Illumina NovaSeq 6000平台上进行测序并分析。各类单细胞测序技术示意图和特点见图6和图7。结果见表28和图8-13。
表28
如表28和图8-13所示,相较于其他三种单细胞转录组测序技术,scEasy-seq具有文库产量高、比对率高、rRNA残留少、基因检出数高和覆盖均一性好等优点,能够显著单细胞转录组测序文库和数据的质量,简化单细胞转录组测序的操作和降低单细胞转录组测序的成本。
综上,本发明提供一种高效快速的RNA建库技术,称为(Easy RNA sequencing)。相较于传统的RNA建库方法,Easy-seq具有耗时短、操作简单和损失小等优点,因此可以应用于单细胞转录组测序(scEasy-seq,如图14)。相较于已有的scRNA-seq技术,scEasy-seq具有基因检出率高、RNA信息完整和建库产量高等优点,是一种高效灵敏的单细胞转录组测序技术,可以广泛应用于科学研究和疾病诊断等领域。
