一种多模态阿尔兹海默症医学图像识别分类方法和系统
文献发布时间:2023-06-19 11:11:32
技术领域
本申请涉及医学图像分析技术领域,尤其涉及一种多模态阿尔兹海默症医学图像识别分类方法和系统。
背景技术
阿尔茨海默病(AD)是一种起病隐匿的进行性发展的神经系统退行性疾病。临床上以记忆障碍、失语、失用、失认、视空间技能损害、执行功能障碍以及人格和行为改变等全面性痴呆表现为特征。65岁以前发病者,称早老性痴呆;65岁以后发病者称老年性痴呆。
对阿尔兹海默症的不同阶段人群进行人群分类,有利于识别阿尔兹海默症早期人群,同时获取有效的基因信息,可以为早期阿尔兹海默症患者的预防和诊断提供辅助。传统的阿尔兹海默症的医学诊断中图像数据与遗传数据多模态融合效果不佳,主要体现在:目前预处理后的医学图像所提取的特征存在诸多对人群分类没有效果的特征,影响分类的准确度;现今在SNP(单核苷酸多态性)数据用于阿尔兹海默症诊断方面一般人工选取与病症相关的基因的SNP数据,然而人工选取的SNP数据有可能在挑选过程中出现遗漏,且许多与疾病相关的SNP未被记录,SNP数据的计算复杂程度高。因此,提高阿尔兹海默症的医学诊断中图像数据与遗传数据多模态融合效果,进一步提升阿尔兹海默症的不同阶段人群识别与分类准确度,仍然是本领域技术人员亟待解决的技术问题。
发明内容
本申请提供了一种多模态阿尔兹海默症医学图像识别分类方法和系统,用于解决现有的阿尔兹海默症的医学诊断中图像数据与遗传数据多模态融合效果不佳,影响阿尔兹海默症的不同阶段人群识别与分类准确度的技术问题。
有鉴于此,本申请第一方面提供了一种多模态阿尔兹海默症医学图像识别分类方法,包括:
构建阿尔兹海默症不同人群的医学数据库,所述医学数据库包括冠状位MRI图像数据和基因SNP数据;
对所述MRI图像数据进行图像预处理后,使用CNN构造分类器,挑选最优的至少三个分类器作为优质MRI基分类器;
使用GWAS全基因组关联分析法对所述基因SNP数据进行预处理,得到编码的SNP位点数据集;
以决策树为基分类器使用随机森林分类器、Bagging分类器和XGBoost分类器三种集成策略构建分类器,得到三种SNP基分类器;
基于改进的概率权重集成学习模式,对所有所述优质MRI基分类器和所述SNP基分类器进行集成学习,得到最终的强化版分类器;
使用所述强化版分类器进行多模态阿尔兹海默症医学图像识别分类。
可选地,所述使用GWAS全基因组关联分析法对所述基因SNP数据进行预处理,得到编码的SNP位点数据集,包括:
使用PLINK软件对所述基因SNP数据进行GWAS全基因组关联分析,包括:根据位点缺失率筛选基因SNP数据、根据位点信息缺失率筛选基因SNP数据、根据Hardy-Weinberg平衡筛选基因SNP数据、根据连锁不平衡性筛选基因SNP数据、根据个体独立性筛选基因SNP数据,使用Logistic回归模型进行分析,得到每个SNP与表型的相关显著性p值,根据p值挑选出相关性高的SNP进行编码,构成编码的SNP位点数据集。
可选地,对所述MRI图像数据进行图像预处理包括:
对所述MRI图像数据进行去头骨和配准处理;
对所述MRI图像数据进行平滑处理;
对所述MRI图像数据进行灰度归一化;
对所述MRI图像数据进行二维切片。
可选地,使用SPM12软件对所述MRI图像数据进行图像预处理。
可选地,基于改进的概率权重集成学习模式为:
p(x)=sigmoid(w1)p(x|h1)+sigmoid(w2)p(x|h2)+···sigmoid(wn)p(x|hn)
其中,n为分类器的数量,sigmoid()为激活函数,w为分类器的性能指标,p为当前分类器的概率,h为网络层数。
本申请第二方面提供了一种多模态阿尔兹海默症医学图像识别分类系统,包括:
数据模块,用于构建阿尔兹海默症不同人群的医学数据库,所述医学数据库包括冠状位MRI图像数据和基因SNP数据;
MRI图像处理模块,用于对所述MRI图像数据进行图像预处理后,使用CNN构造分类器,挑选最优的至少三个分类器作为优质MRI基分类器;
第一基因数据处理模块,用于使用GWAS全基因组关联分析法对所述基因SNP数据进行预处理,得到编码的SNP位点数据集;
第二基因数据处理模块,用于以决策树为基分类器使用随机森林分类器、Bagging分类器和XGBoost分类器三种集成策略构建分类器,得到三种SNP基分类器;
集成学习强化模块,用于基于改进的概率权重集成学习模式,对所有所述优质MRI基分类器和所述SNP基分类器进行集成学习,得到最终的强化版分类器;
识别分类模块,用于使用所述强化版分类器进行多模态阿尔兹海默症医学图像识别分类。
可选地,所述第一基因数据处理模块具体用于:
使用PLINK软件对所述基因SNP数据进行GWAS全基因组关联分析,包括:根据位点缺失率筛选基因SNP数据、根据位点信息缺失率筛选基因SNP数据、根据Hardy-Weinberg平衡筛选基因SNP数据、根据连锁不平衡性筛选基因SNP数据、根据个体独立性筛选基因SNP数据,使用Logistic回归模型进行分析,得到每个SNP与表型的相关显著性p值,根据p值挑选出相关性高的SNP进行编码,构成编码的SNP位点数据集。
可选地,对所述MRI图像数据进行图像预处理包括:
对所述MRI图像数据进行去头骨和配准处理;
对所述MRI图像数据进行平滑处理;
对所述MRI图像数据进行灰度归一化;
对所述MRI图像数据进行二维切片。
可选地,使用SPM12软件对所述MRI图像数据进行图像预处理。
可选地,基于改进的概率权重集成学习模式为:
p(x)=sigmoid(w1)p(x|h1)+sigmoid(w2)p(x|h2)+···sigmoid(wn)p(x|hn)
其中,n为分类器的数量,sigmoid()为激活函数,w为分类器的性能指标,p为当前分类器的概率,h为网络层数。
从以上技术方案可以看出,本申请实施例具有以下优点:
本申请中提供了一种多模态阿尔兹海默症医学图像识别分类方法,包括:构建阿尔兹海默症不同人群的医学数据库,所述医学数据库包括冠状位MRI图像数据和基因SNP数据;对所述MRI图像数据进行图像预处理后,使用CNN构造分类器,挑选最优的至少三个分类器作为优质MRI基分类器;使用GWAS全基因组关联分析法对所述基因SNP数据进行预处理,得到编码的SNP位点数据集;以决策树为基分类器使用随机森林分类器、Bagging分类器和XGBoost分类器三种集成策略构建分类器,得到三种SNP基分类器;基于改进的概率权重集成学习模式,对所有所述优质MRI基分类器和所述SNP基分类器进行集成学习,得到最终的强化版分类器;使用所述强化版分类器进行多模态阿尔兹海默症医学图像识别分类。
针对二维空间上的每个MRI图像使用深度卷积神经网络训练一组基分类器,之后选取出对疾病人群有分类效果的前至少三个图像切片分类器当做集成时图像分类器的基分类器,确保选取的切片跟该疾病的临床表现具有一定的吻合性,同时也使集成时图像分类器具有多样性,集成学习的性能不止与分类器的性能有关,还与分类器的多样性有关,多个图像分类器用于集成比起单个图像分类器用于集成效果要好。
本申请中,基因组数据的预处理使用的是GWAS(全基因组关联分析),GWAS分析的是SNP位点与表型的关联,从而筛选出与表型相关的SNP,表型的选择有两种方式,第一种是线性的表型,如果身高、体重、智力等;第二种是二元的表型,比如患病和未患病,即通常所说的case和control,患病为0,未患病为1。使用GWAS分析降低SNP数据的维度,大大降低了计算的复杂程度,减少了冗余信息所造成的识别误差,提高了识别的精度;除此之外,为了提高SNP数据分类的性能,本发明使用多种集成策略构建SNP分类器,一方面提高分类性能,另一方面提高SNP分类器的多样性。
将医学影像和基因组学两种模态数据相结合,通过对图像数据的解读,结合基因组关联分析,利用图像与基因数据的结合使得阿尔兹海默症的诊断更具准确性和可靠性,解决了现有的阿尔兹海默症的医学诊断中图像数据与遗传数据多模态融合效果不佳,影响阿尔兹海默症的不同阶段人群识别与分类准确度的技术问题。
附图说明
为了更清楚的说明本发明实施例或现有技术中的技术方案,下面将对实施例或现有技术描述中所需要使用的附图作简单的介绍,显而易见地,下面描述中的附图仅仅是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他相关的附图。
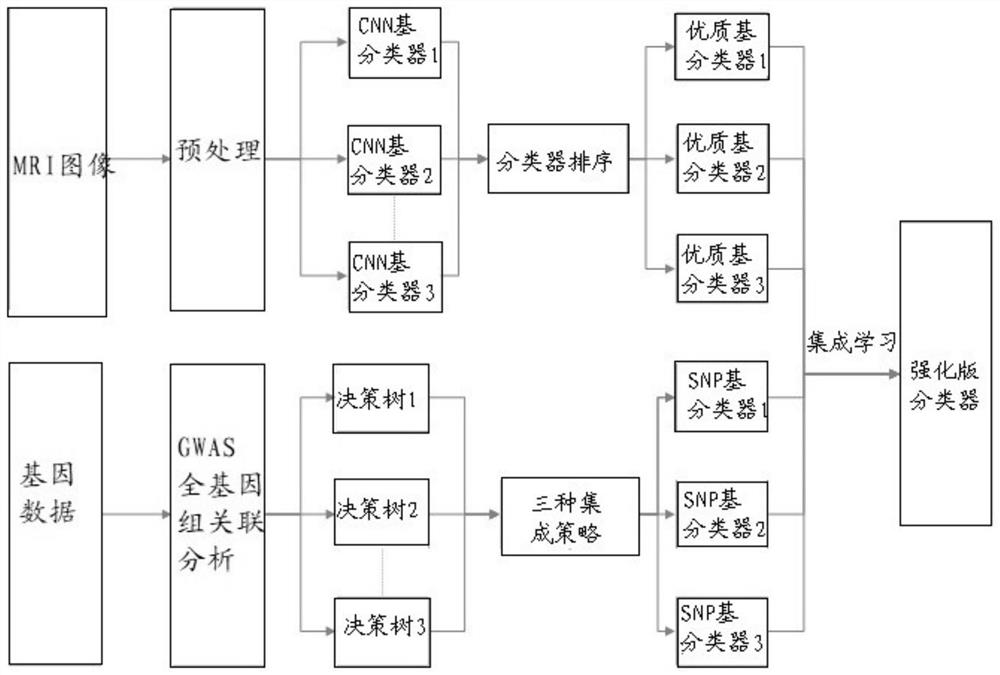
图1为本申请实施例提供的一种多模态阿尔兹海默症医学图像识别分类方法的流程框架示意图;
图2为本申请实施例中的MRI图像数据预处理过程示意图;
图3为本申请实施例中的使用CNN构造分类器的流程示意图;
图4为本申请实施例中的基因数据的预处理过程示意图;
图5为本申请实施例中的SNP分类器模型构建分类器过程示意图;
图6为本申请实施例中的集成学习过程示意图。
具体实施方式
为了使本技术领域的人员更好地理解本申请方案,下面将结合本申请实施例中的附图,对本申请实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅是本申请一部分实施例,而不是全部的实施例。基于本申请中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本申请保护的范围。
实施例1
为了便于理解,请参阅图1,本申请提供了一种多模态阿尔兹海默症医学图像识别分类方法的实施例,包括:
步骤101、构建阿尔兹海默症不同人群的医学数据库,医学数据库包括冠状位MRI图像数据和基因SNP数据。
本发明涉及多模态集成学习,需要将医学影像和基因组学两种模态数据相结合,因此需要预先构建包含冠状位MRI图像数据和基因SNP数据的阿尔兹海默症不同人群的稳定可靠的医学图像以及SNP数据的数据库。
步骤102、对MRI图像数据进行图像预处理后,使用CNN构造分类器,挑选最优的至少三个分类器作为优质MRI基分类器。
从医学数据库中获取冠状位MRI图像数据,如图2所示对冠状位MRI图像数据进行预处理,预处理过程可以用SPM12软件进行,旨在将原始图像规范化以及适当的降低噪声处理,便于后续医学图像分类的实现,包括:
1.首先通过头动校正,剥头骨等操作去除噪声和非脑组织结构的影响,然后所有被试的结构图像经过空间标准化将不同被试的MRI图像配准到统一的坐标空间下以此来消除个体间的差异。
2.随后对得到的结果图像用高斯平滑,去除噪声对图像的影响,使得数据更接近似于正太分布,以此来增加参数检验的有效性。
3.将图像进行灰度归一化。
4.进行二维上切片。
对MRI图像数据进行图像预处理后,使用CNN构造分类器,挑选最优的至少三个分类器作为优质MRI基分类器,如图3所示,采用的CNN模型结构由6个卷积层(图3中简写为conv),3个池化层(图3中简写为pool)和3个全连接层(图3中简写为FC)组成,最后一个全连接层只有两个节点,采用softmax函数实现二分类。每个CNN基分类器训练40个epoch,经过测试,40个epoch足够使基分类器收敛,使基分类器在训练集原始切片上分类准确率达到100%。所有卷积层激活函数采用ReLU函数,梯度更新算法采用Adam,学习率设为0.0001,每批次输入切片张数(batchsize)设为200。
步骤103、使用GWAS全基因组关联分析法对基因SNP数据进行预处理,得到编码的SNP位点数据集。
如图4所示,图4为基因数据的预处理过程,可以使用PLINK软件进行GWAS全基因组关联分析的方法进行预处理,得到的SNP位点数据进行编码成0,1,2(AA-0,Aa-1,aa-2)。其过程如下:
(1)根据杂合率筛选
在基因型数据中,每两个字符表示一个SNP的基因型,如GGGCAATA这串基因型数据包含了四个SNP的基因型,分别为GG、GC、AA、TA,其中GG、AA这类属于纯合型,GC、TA这类属于杂合型。根据遗传学规律,在自然群体中,不同样本的杂合基因频率是相近的。可以根据此规律剔除不符合该规律的异常被试数据。
(2)根据位点缺失率筛选
样本SNP缺失率是反映样本基因型数据质量的一个重要指标,如果一个样本的位点缺失率太高,则表明该样本数据质量差,需要剔除以免影响后续分析。
(3)根据位点信息缺失率筛选
位点信息缺失率是指,在所有的被试中,某个SNP的信息缺失率。如果某个SNP的信息缺失率太高,则说明该SNP的数据质量差,不适于用于后续分析,需删除该SNP信息。
(4)根据Hardy–Weinberg平衡筛选
Hardy–Weinberg平衡定律也称遗传平衡定律,是群体遗传中的重要法则,分别在1908年和1909年由英国数学家G·H·哈迪(Godfrey Harold Hardy)和德国医生威廉·温伯格(Wilhelm Weinberg)独立证明。其主要内容为:一个群体在理想情况(不受特定的干扰因素影响,如非随机交配、天择、族群迁移、突变或群体大小有限),经过多个世代,基因频率与基因型频率会保持恒定并处于稳定的平衡状态。
(5)根据连锁不平衡性筛选
连锁不平衡(Linkage Disequilibrium,LD)是指分属两个或两个以上基因座或等位基因上存在非随机的组合。简单地说,如果某两个基因在遗传过程中不是完全独立遗传的,那他们之间就存在连锁不平衡现象。实际中,常用r2表示SNP的连锁不平衡强度,r2越大,连锁不平衡现象越强,相关SNP的独立性越弱。在进行GWAS分析时希望最终找出的SNP是独立性较强的SNP,因此通过连锁不平衡删除连锁现象较强的SNP(每组连锁的SNP中会留下一个SNP作为代表)。
(6)根据个体独立性筛选
需要尽量保持数据的独立性,样本间如果具有较近的亲缘关系或数据采集时同一个样本的数据被采用了多次,将会导致SNP的分布并非自然状态,使分析结果产生偏差。亲缘系数又称血缘系数,将群体中个体之间基因组成的相似程度用数值来表示即为血缘系数,它可以反映两个个体间的遗传相关程度。
(7)关联分析
在GWAS中,表型的选择有两种方式,第一种是线性的表型,如果身高、体重、智力等;第二种是二元的表型,比如患病和未患病,即通常所说的case和control,患病为0,未患病为1。当待分析的表型是二元性状时,通常使用Logistic回归模型进行分析;当待分析的表型是线性性状,通常使用普通线性回归模型。本发明使用Logistic回归模型进行分析得到每个SNP与表型的相关显著性p值,根据p值挑选出相关性高的SNP进行编码成0,1,2构成数据集。
步骤104、以决策树为基分类器使用随机森林分类器、Bagging分类器和XGBoost分类器三种集成策略构建分类器,得到三种SNP基分类器。
如图5所示,图5为SNP分类器模型,以决策树为基分类器,使用随机森林分类器、Bagging分类器和XGBoost分类器三种集成方式构建分类器。
步骤105、基于改进的概率权重集成学习模式,对所有优质MRI基分类器和所述SNP基分类器进行集成学习,得到最终的强化版分类器。
步骤106、使用强化版分类器进行多模态阿尔兹海默症医学图像识别分类。
如图6所示,构造好MRI分类器和SNP分类器后,接着使用基于改进的概率权重集成的学习模式,最终得到一个强化版的分类器。使用的基于改进的概率权重加权的集成方式:
p(x)=sigmoid(w1)p(x|h1)+sigmoid(w2)p(x|h2)+···sigmoid(wn)p(x|hn)
其中,n为分类器的数量,sigmoid()为激活函数,w为分类器的性能指标,由验证集的概率组成,p为当前分类器的概率,h为网络层数。能够有效解决分类器之间权重不平衡的程度,使其形成一个高效的强化版分类器。使用强化版分类器进行多模态阿尔兹海默症医学图像识别分类。
集成学习结果不止与单个分类器性能有关,还有所集成分类器之间的多样性有关。MRI根据每个切片分类器性能挑选最终要集成的基分类器,确保选取的切片跟该疾病的临床表现具有一定的吻合性,同时也使集成时图像分类器具有多样性;根据卷积网络的实践可以证明,卷积神经网络有利于降低过拟合风险,同时学习到图像的深层特征。
使用GWAS分析降低SNP数据的维度,大大降低了计算的复杂程度,减少了冗余信息所造成的识别误差,提高了识别的精度;SNP分类器以决策树为基分类器使用多种集成方式构造分类器,一方面提高SNP分类器的性能,另一方面也是提高SNP分类器的多样性。
将医学影像和基因组学两种模态数据相结合,通过对图像数据的解读,结合基因组关联分析,利用图像与基因数据的结合使得阿尔兹海默症的诊断更具准确性和可靠性,解决了现有的阿尔兹海默症的医学诊断中图像数据与遗传数据多模态融合效果不佳,影响阿尔兹海默症的不同阶段人群识别与分类准确度的技术问题。
本申请中还提供了一种多模态阿尔兹海默症医学图像识别分类系统的实施例,包括:
数据模块,用于构建阿尔兹海默症不同人群的医学数据库,医学数据库包括冠状位MRI图像数据和基因SNP数据;
MRI图像处理模块,用于对所述MRI图像数据进行图像预处理后,使用CNN构造分类器,挑选最优的至少三个分类器作为优质MRI基分类器;
第一基因数据处理模块,用于使用GWAS全基因组关联分析法对基因SNP数据进行预处理,得到编码的SNP位点数据集;
第二基因数据处理模块,用于以决策树为基分类器使用随机森林分类器、Bagging分类器和XGBoost分类器三种集成策略构建分类器,得到三种SNP基分类器;
集成学习强化模块,用于基于改进的概率权重集成学习模式,对所有优质MRI基分类器和SNP基分类器进行集成学习,得到最终的强化版分类器;
识别分类模块,用于使用强化版分类器进行多模态阿尔兹海默症医学图像识别分类。
第一基因数据处理模块具体用于:
使用PLINK软件对基因SNP数据进行GWAS全基因组关联分析,包括:根据位点缺失率筛选基因SNP数据、根据位点信息缺失率筛选基因SNP数据、根据Hardy-Weinberg平衡筛选基因SNP数据、根据连锁不平衡性筛选基因SNP数据、根据个体独立性筛选基因SNP数据,使用Logistic回归模型进行分析,得到每个SNP与表型的相关显著性p值,根据p值挑选出相关性高的SNP进行编码,构成编码的SNP位点数据集。
对MRI图像数据进行图像预处理包括:
对MRI图像数据进行去头骨和配准处理;
对MRI图像数据进行平滑处理;
对MRI图像数据进行灰度归一化;
对MRI图像数据进行二维切片。
使用SPM12软件对MRI图像数据进行图像预处理。
基于改进的概率权重集成学习模式为:
p(x)=sigmoid(w1)p(x|h1)+sigmoid(w2)p(x|h2)+···sigmoid(wn)p(x|hn)
其中,n为分类器的数量,sigmoid()为激活函数,w为分类器的性能指标,p为当前分类器的概率,h为网络层数。
集成学习结果不止与单个分类器性能有关,还有所集成分类器之间的多样性有关。MRI根据每个切片分类器性能挑选最终要集成的基分类器,确保选取的切片跟该疾病的临床表现具有一定的吻合性,同时也使集成时图像分类器具有多样性;根据卷积网络的实践可以证明,卷积神经网络有利于降低过拟合风险,同时学习到图像的深层特征。
使用GWAS分析降低SNP数据的维度,大大降低了计算的复杂程度,减少了冗余信息所造成的识别误差,提高了识别的精度;SNP分类器以决策树为基分类器使用多种集成方式构造分类器,一方面提高SNP分类器的性能,另一方面也是提高SNP分类器的多样性。
将医学影像和基因组学两种模态数据相结合,通过对图像数据的解读,结合基因组关联分析,利用图像与基因数据的结合使得阿尔兹海默症的诊断更具准确性和可靠性,解决了现有的阿尔兹海默症的医学诊断中图像数据与遗传数据多模态融合效果不佳,影响阿尔兹海默症的不同阶段人群识别与分类准确度的技术问题。
以上所述,以上实施例仅用以说明本申请的技术方案,而非对其限制;尽管参照前述实施例对本申请进行了详细的说明,本领域的普通技术人员应当理解:其依然可以对前述各实施例所记载的技术方案进行修改,或者对其中部分技术特征进行等同替换;而这些修改或者替换,并不使相应技术方案的本质脱离本申请各实施例技术方案的精神和范围。
- 一种多模态阿尔兹海默症医学图像识别分类方法和系统
- 一种基于多模态融合的医学图像识别系统及方法
