一种基因与表型关联知识库、构建方法及其应用
文献发布时间:2023-06-19 10:41:48
技术领域
本发明属于生物信息学领域,具体涉及一种基因与表型关联知识库及其构建方法和应用。
背景技术
孟德尔遗传定律由奥地利帝国遗传学家格里哥·孟德尔在1865年发表,包括分离定律和自由组合定律。符合孟德尔遗传定律的表型或性状又可称为单基因性状,广泛存在于生物各界中,例如动物毛色、水稻籽粒的有芒无芒、人类单双眼皮。通常,因单基因上一个或少数几个突变即造成表型差异。
具体的,以人类孟德尔遗传病为例,现已发现约有7000种孟德尔遗传病,其中仅有约5000种具有相关遗传分子机制信息。在实际应用中,为了根据表型确定致病的候选基因,通常需要利用测序手段获得个体的序列信息、从大量变异位点中确定与表型密切相关的变异位点、与数据库信息或科技文献进行比较分析,这是一个耗费大量财力、人力及时间的过程。
随着计算机技术的发展,生物信息学突飞猛进,各种生物数据库的涌现为基于计算机技术实现基因、表型的关联提供了可能,但目前构建基因与表型关联信息的方法还未见报道。
发明内容
本发明的目的之一是提供一种基因与表型关联知识库的构建方法。
本发明的目的之二是提供一种基因与表型关联知识库。
本发明的目的之三是提供一种量化基因与表型关联关系的方法。
具体的,本发明的技术方案如下:
本发明第一个方面公开了一种构建基因与表型关联知识库的方法,包括以下步骤:
S1:获取文献实体;
S2:判断文献类型并标识;
S3:抽取文献实体中基因词条、表型词条,获得文献语料库;
S4:存储基因与表型关联关系,获得基因与表型关联知识库。
应该理解,本发明不限于上述步骤,还可以包含其他的步骤,例如在步骤S1之前、步骤S1和S2之间、步骤S2和S3之间、步骤S3和S4之间、步骤S4之后,还包含其他额外的步骤,而不超出本发明的保护范围。
在一些优选的实施方案中,步骤S2包括:
S21:利用机器学习训练得到文献类型判断模型;
S22:使用所述文献类型判断模型判断文献类型。
应该理解,本发明不限于上述步骤,还可以包含其他的步骤,例如在步骤S21之前、步骤S21和S22之间、步骤S22之后,还包含其他额外的步骤,而不超出本发明的保护范围。
在S21中,将文献分为单基因型、非单基因型;机器学习训练是基于现有技术中已知的文献类型,例如OMIM数据库(https://omim.org/),对已知的人单基因疾病进行了标识。因仅已知部分文献的类型,所以需利用S21获得的文献类型判断模型对S1获取的所有文献实体进行判断。
进一步的,所述文献类型判断模型为LogisticRegression;更进一步的,LogisticRegression特征为TF-IDF。
在一些优选的实施方案中,在步骤S21前,还包括步骤S20:去除文献实体中的停用词,所述停用词为不反应实体实质内容的词。去除停用词后,可以大幅度的降低判断文献类型的计算量。
在一些优选的实施方案中,步骤S2是根据文献实体标题、摘要信息进行文献类型判断。仅根据文献实体标题、摘要信息进行文献类型判断,而非文献实体全文,可以大幅度的降低判断文献类型的计算量。
在本发明的一具体实施例中,使用PubMed数据库(https://www.ncbi.nlm.nih.gov/pubmed/)收集文献的标题和摘要信息。
在本发明的一具体实施例中,利用OMIM数据库资源(https://omim.org/),根据该数据库对PubMed数据库的引用信息,如在OMIM中有引用就将此文献标记为单基因遗传疾病相关文献,反之亦然。
在一些优选的实施方案中,步骤S3是根据标准语料库抽取文献实体中的词条,所述标准语料库包括基因标准词条和表型标准词条。
标准词条来源于已有数据库,例如,基因标准词条来自于基因数据库HGNC(https://www.genenames.org/)和蛋白数据库UniProt(https://www.uniprot.org/),表型标准词条来自于数据库Hpo(https://hpo.jax.org/)。对基因标准词条及抽取的基因词条,按其固有格式形式存储。对表型标准词条及抽取的表型词条,以小写形式存储。
在一些优选的实施方案中,对步骤S3获得的文献语料库,根据词条的前缀、词根或后缀,构建次级文献语料库。构建次级文献语料库后,在查找、提取目标表型和目标基因的信息时,可以大幅度提高效率。
在一些优选的实施方案中,步骤S3和S4之间还包括步骤:
a1:基于文献主题和抽取的词条,利用机器学习训练得到主题关联判断模型;
a2:使用所述主题关联判断模型对词条进行判断,保留与主题关联的词条。
机器学习训练是基于现有技术中已知的文献主题,例如,OMIM数据库(https://omim.org/)对收录的文献进行了主题标识。过滤掉偏离主题的词条,有助于得到准确的基因与表型关联关系。
进一步的,所述主题关联判断模型为LogisticRegression。优选的,LogisticRegression特征为N-gram;更优选的,N-gram中N为2-4。
在一些优选的实施方案中,所述词条为文献实体中抽取的基因词条。仅对基因词条进行主题关联判断,在准确判断的前提下可减少计算量。其原因在于,文献中提及的表型描述通常比较贴合主题,基因描述偏离主题的可能性远远大于表型描述。
在一些优选的实施方案中,还包括基因与表型关联知识库的自动更新。
本发明第二个方面公开了一种基因与表型关联知识库,包括文献获取单元、文献类型判断单元、词条抽取单元和存储单元。
所述文献获取单元用于获取文献实体。所述文献类型判断单元用于判断文献类型并标识。词条抽取单元用于抽取文献实体中基因词条、表型词条,获得文献语料库。存储单元用于存储基因与表型关联关系,获得基因与表型关联知识库。
优选的,所述基因与表型关联知识库通过上述方法构建得到。
优选的,所述基因与表型关联知识库仅适用于单基因的情况。在文献类型判断后,仅选择标识为单基因型的文献进行后续操作。
本发明第三个方面公开了一种量化基因与表型关联关系的方法,包括以下步骤:
(1)根据基因与表型关联知识库提取目标表型和目标基因的关联信息;优选的,所述基因与表型关联知识库为如上所述的基因与表型关联知识库;
(2)分别计算各表型、各基因的信息量,得到基因与表型关联关系的量化信息。
在一些优选的实施方案中,利用公式
利用公式
进一步的,步骤(2)中还包括将基因信息量与表型信息量进行迭代计算。
在符合本领域常识的基础上,上述各优选条件,可任意组合,而不超出本发明的构思与保护范围。
本发明相对于现有技术具有如下的显著优点及效果:
本发明首次公开了一种基因与表型关联知识库,该知识库整合、提取并存储了大量的基因与对应表型之间的关联关系,并可实现自动更新。
该知识库与量化算法相配合,可实现基因与表型关系的自动量化。所有分析人员均使用相同的系统,避免了人为偏差,保证了可重复性。与传统方式相比,可靠、通用、灵活、高效,节约了人力物力且实用性高。
附图说明
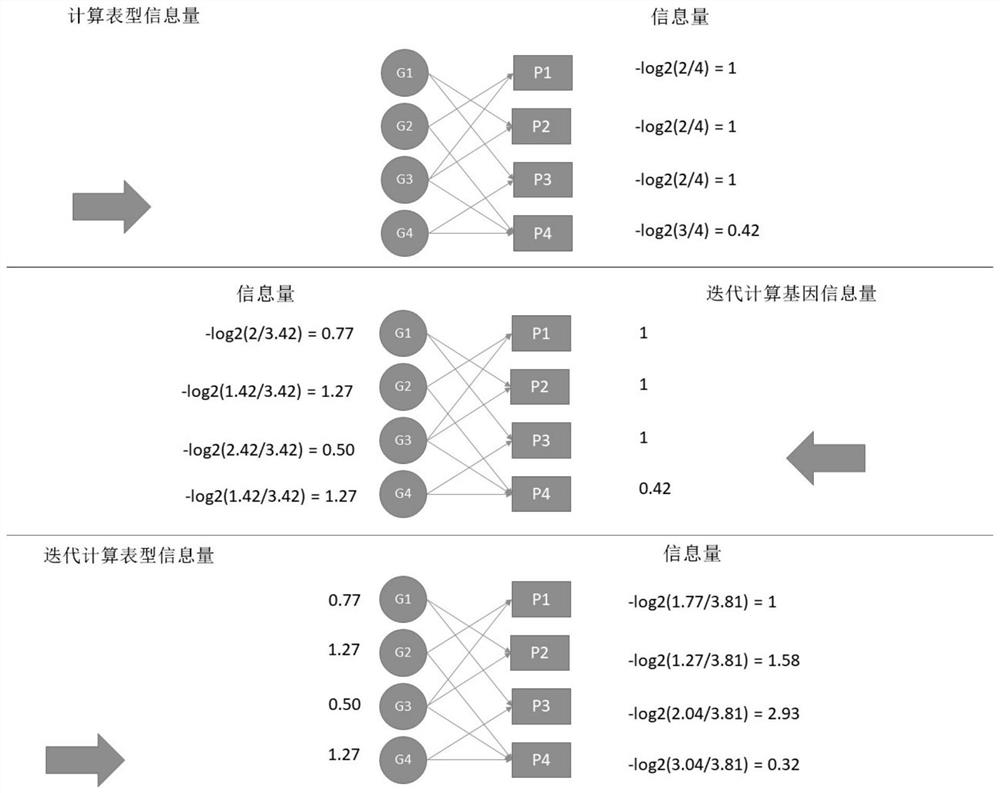
图1为本发明实施例中迭代计算的示意图。
具体实施方式
下面以人类单基因病为例,结合附图和实施例对本发明的技术方案进行详细描述,但并不因此将本发明限制在所述的实施例范围之中。
需特别指出,本申请不限制物种,文中列举的各数据库仅为示例而非限制范围。
实施例1
本实施例公开了一种构建基因与表型关联知识库的方法,包括以下步骤:
S1:获取文献实体;
S2:判断文献类型并标识;
S3:抽取文献实体中基因词条、表型词条,获得文献语料库;
S4:存储基因与表型关联关系,获得基因与表型关联知识库。
具体步骤如下:
S1:获取文献实体
使用PubMed数据库(https://www.ncbi.nlm.nih.gov/pubmed/)收集文献标题和摘要信息,相比文献全文,其信息量更小,分析的效率更高。截至2018.7总共获取27,853,513篇。
S2:判断文献类型
其中判断文献类型之前还包括过滤步骤,即:去除文献实体中的停用词,停用词是指不反应文献实体实质内容的词。过滤后,实现保留更少数据量、更体现文献内容的信息。
以下表1中6篇文献为例:
表1
仅考虑标题中各单词的词频,词频前四的单词如表2所示:
表2
明显的,of、and、in这类单词,对分类的贡献明显不如disease、genetic。为了避免这些高频、低贡献词条的影响,将这些词确定为停用词并进行过滤。
其中,步骤S2包括:
S21:利用机器学习训练得到文献类型判断模型;
S22:使用所述文献类型判断模型判断文献类型。
步骤S21具体如下:
利用OMIM数据库(https://omim.org/),根据该数据库对PubMed数据库的引用信息,如在OMIM数据库中有引用就将此文献标记为单基因型,反之亦然。总共获得单基因型文献128797篇。
在标记的单基因型文献、非单基因型文献中随机各抽取出5000篇,合计10000篇,过滤掉标题和摘要同时缺失的文献,最终得到9612篇。各抽取单基因型文献、非单基因型文献的3/4,合计7209篇,作为样本集。
再一次在标记的单基因型文献、非单基因型文献中随机各抽取出5000篇,合计10000篇,过滤标题和摘要同时缺失的文献,最终得到9578篇,作为测试集。
利用不同机器学习模型进行训练,以TF-IDF为模型训练特征。其中TF(TermFrequency)指词频;IDF(Inverse Document Frequency)指逆文本频率指数,用以评估某词条在对应文献的重要程度,该词条的重要性随着它在相关文献中出现的次数成正比增加,但同时会随着它在非相关文献中出现的频率成反比下降。结果如表3所示:
表3
其中,MLP的模型参数为1000×100×2;准确性=模型判断的某类型文献数/该类型实际文献数。
综合准确性及耗时,确定Logistic Regression为文献类型判断模型。
步骤S22具体如下:
用Logistic Regression模型对总共27,837,552篇文献进行预测,总共判定了2,218,850篇为单基因型文献。
步骤S3具体如下:
建立标准语料库,根据标准语料库抽取文献实体中基因词条和表型词条,获得文献语料库。标准语料库包括基因标准词条和表型标准词条,基因标准词条来自于基因数据库HGNC(https://www.genenames.org/)和蛋白数据库UniProt(https://www.uniprot.org/),表型标准词条来自于数据库Hpo(https://hpo.jax.org/)。
为了实现对基因标准词条的准确识别,保留描述中的固有格式(例如大小写)。例如:基因UGT1A1是一个基因名称,文献Marcuello,E.,et al."UGT1A1 gene variationsand irinotecan treatment in patients with metastatic colorectal cancer."British journal of cancer 91.4(2004):678.中,UGT1A1出现的时候,保持特定的大小写,因此可以确定这个单词是代表UGT1A1基因。又如:文献Lee,Jong Woo,et al."Inactivating mutations of proapoptotic Bad gene in human colon cancers."Carcinogenesis 25.8(2004):1371-1376.中,标题中Bad是指特定基因,需保留首字母大写。文献Di Fede,Giuseppe,et al."Good gene,bad gene:new APP variant may beboth."Progress in neurobiology 99.3(2012):281-292.中,bad是与good相对的一个描述,用作一般的描述,均为小写。
为了实现对表型标准词条的准确识别,将目标语句和表型标准词条统一转化为小写,提高识别效率。在人类表型数据库HPO中收集的表型描述,第一个单词首字母都是大写。例如:Hemiclonic seizures偏瘫发作这个表型描述,第一个单词H大写,而在文献
因为文献语料库比较大,进行搜索、提取时若按每一个词条依次进行则效率低,建立次级文献语料库,能够显著提高效率。例如,存在很多具有相同前缀的基因词条,alphakinase1、alpha kinase 2、alpha kinase 3、alpha tocopherol transfer protein、alphatocopherol transfer protein like,他们具有相同的“alpha”开头,把所有以“alpha”开头的基因标准词条集合为二级文献语料库。搜索时,在在多个二级文献语料库中搜索,找到alpha前缀的二级文献语料库,然后在该二级文献语料库中搜索具体的基因词条,可以明显提高搜索效率。通过这种处理,原语料库大小为201068,处理后大小为19726,搜索范围下降至不到10%。
在步骤S3和S4之间,还包括步骤:
a1:基于文献主题和抽取的词条,利用机器学习训练得到主题关联判断模型;
a2:使用所述主题关联判断模型对词条进行判断,保留与主题关联的词条。
由于文献中可能会列举一些与文献主题并不密切相关的基因(例如,在背景描述中),这些基因如果被提取到会影响基因与表型关联关系的准确性。因此,通过机器学习的方法进一步对提取的每一个基因词条是否贴切文献主题进行判断及过滤。文献主题通过OMIM的引用信息进行确定。
从步骤S2标记的单基因型文献中,随机抽取5000篇。抽取4/5约4000篇为训练集,剩余1/5约1000篇为测试集。对初步提取出的基因词条,设置N-gram进行提取,根据其出现频率判断是否属于文献主题,N设置为2-4。例如,当N=2时,语句“A B C D gene1 E F G Hgene2 I J K”中,对基因词条“gene1”提取“CD gene1 E F”,对基因词条“gene2”提取“G Hgene2 I J”。
利用不同机器学习模型进行训练,进行5-fold交叉验证,结果如表4所示:
表4
确定LogisticRegression为最佳主题关联判断模型。
将全部单基因型文献中的基因和表型的关联关系提取出来,该文献也可以作为该关联关系的支持,获得基因与表型关联知识库。
实施例2
本实施例公开了一种基因与表型关联知识库。所述基因与表型关联知识库包括文献获取单元、文献类型判断单元、词条抽取单元和存储单元。
所述文献获取单元用于获取文献实体。所述文献类型判断单元用于判断文献类型并标识。词条抽取单元用于抽取文献实体中基因词条、表型词条,获得文献语料库。存储单元用于存储基因与表型关联关系,获得基因与表型关联知识库。
实施例3
本实施例公开了利用实施例1所述方法构建得到的基因与表型关联知识库、或者利用实施例2所述的基因与表型关联知识库对基因与表型关联关系进行量化的方法,包括以下步骤:
(1)提取目标表型和目标基因的关联信息
(2)分别计算各表型、各基因的信息量
利用公式
利用公式
将基因信息量与表型信息量进行迭代计算,直至稳定。根据P
图1为简化的迭代示例,其中表型P1至P4均为第一级表型(即均无父表型)。从图中可清楚的看出迭代后信息量的变化。
实施例4
某病人出现的表型如表5所示:
表5
某医生根据其经验,推测该病人可能是以下30个基因存在异常,采用如实施例3所述的方法,但不进行基因信息量与表型信息量的迭代计算,即仅计算一轮。根据计算结果,对各基因进行排序,KMT2A得分最高为最相关基因。具体结果如表6所示,其中Not Found为在基因与表型关联知识库中未找到关联关系:
表6
采用如实施例3所述的方法,经迭代计算。根据计算结果,对各基因进行排序,FANCD2得分从第四上升至第一,即FANCD2为最相关基因。具体结果如表7所示,其中NotFound为在基因与表型关联知识库中未找到关联关系:
表7
经人工核查,发现KMT2A基因与HP:0000365癫痫发作、HP:0000252全面发育迟缓、HP:0001250身材矮小三个表型相关,FANCD2与HP:0001263小头畸形、HP:0004322听力障碍、HP:0001250身材矮小三个表型相关。其中,HP:0000252全面发育迟缓是一个综合性的表型,它的父表型有多个,即在各类病症中均有表征。因此,FANCD2基因比KMT2A基因更能解释该患者的情况。由此可见,迭代计算的结果与实际情况更为吻合。
上述实施例为本发明较佳的实施方式,但本发明的实施方式并不受上述实施例的限制,其他的任何未背离本发明的精神实质与原理下所作的改变、修饰、替代、组合、简化,均应为等效的置换方式,都包含在本发明的保护范围之内。
- 一种基因与表型关联知识库、构建方法及其应用
- 保健食品关联知识库构建方法和装置
