神经退行性疾病检测和治疗的新靶标
文献发布时间:2023-06-19 11:59:12
技术领域
本发明属于分子生物学、神经科学、和临床神经内科学领域,具体地,本发明提供了一种神经退行性疾病检测和治疗的新的分子靶标,以及相应的诊断、治疗方法和相应的试剂盒。
背景技术
随着世界人口老龄化加快,神经系统退行性疾病患者数量呈现快速增长趋势,例如痴呆、帕金森病等疾病。罹患前者可能出现记忆障碍、失语、失用、失认、视空间技能损害、执行功能障碍以及人格和行为改变;后者则导致运动障碍,如震颤麻痹,行动迟缓,肌肉异常强直,姿势步态障碍等病情特征。此类疾病病因和发病机制往往不甚明确,治疗困难,并且严重影响患者寿命和生存质量。
PGRN即颗粒蛋白前体(progranulin),在中枢神经系统及周围组织中均有表达。PGRN的缺乏与前额叶痴呆(FTD)
越来越多的证据显示,PGRN的水平可能受一些基因突变的调节
然而,目前尚无GWAS研究去探索脑脊液PGRN的调节因素。与外周血PGRN相比,脑脊液PGRN可能存在不同的调节机制,也避免了PGRN在外周组织中引起肿瘤和肥胖的副作用。于是,我们针对脑脊液PGRN 的遗传调节因素开展了GWAS,实验对象为1362例非痴呆成年人,并成功筛选出位于FAM171A2基因上与 PGRN水平变化具有较强相关性的独立位点。我们还用了生物信息学的手段注释了该突变的功能,进一步确定了它与神经退行性疾病的关系。
发明概述
本发明的目的在于提供一种辅助诊断和治疗神经系统退行性疾病的方法。具体地,本发明通过检测FAM171A2基因的表达水平,或检测单核苷酸多态性位点rs708384的基因型,判断PGRN的表达水平是否处于正常,以辅助诊断神经系统退行性疾病。本发明的目的还在于提供一种通过上调脑内PGRN的水平以治疗神经系统退行性疾病的方法。
在本发明的一个实施方案中,开展了一项两阶段的全基因组关联研究(GWAS),旨在筛选脑脊液PGRN 水平的调节因素。ADNI数据库中的数据显示,位于FAM171A2基因上的突变位点rs708384和脑脊液PGRN 水平降低有关(p=3.95×10
在本发明的一个实施方案中,本发明提供一种预测或确定个体颗粒蛋白前体(PGRN)水平的方法,所述方法包括确定一个或多个得自所述个体的样本中一种或多种生物标志物的水平,其中所述一种或多种生物标志物包含FAM171A2。该方法还可包括确定一个或多个得自个体的样本中以下生物标志物的水平:ITGA2B。
在本发明的一个实施方案中,本发明提供一种确定个体是否具有颗粒蛋白前体(PGRN)水平失调的风险的方法,所述方法包括确定一个或多个得自所述个体的样本中一种或多种生物标志物的水平,其中所述一种或多种生物标志物包含FAM171A2。该方法还可包括确定一个或多个得自个体的样本中以下生物标志物的水平:ITGA2B。
在本发明的一个实施方案中,本发明提供一种确定个体是否处于发展成神经退行性疾病的风险中的方法,所述方法包括确定一个或多个得自个体的样本中一种或多种生物标志物的水平,其中所述一种或多种生物标志物包含FAM171A2。该方法还可包括确定一个或多个得自个体的样本中以下生物标志物的水平: ITGA2B。
在一个实施方案中,通过确定单核苷酸多态性位点rs708384的核苷酸类型来确定FAM171A2的表达水平。当该位点为胞嘧啶(野生型)时,FAM171A2的表达水平为正常。当该位点为腺嘌呤(突变型)时,FAM171A2 的表达水平明显上调。
在一个实施方案中,将一种或多种生物标志物的水平与一个或多个参考值进行比较。在此情况下,优选地使用相同的分析方法确定每个样本中的每种生物标志物水平和对应的参考值。参考值可基于例如先前已被确定为具有正常或失调的PGRN水平的个体群体中一种或多种生物标志物的值(例如,平均值)。
相应地,在本发明的一个实施方案中,提供了特异性检测FAM171A2的试剂和/或特异性检测ITGA2B 的试剂,在制备预测或确定个体PGRN水平的试剂盒中的用途。本发明还提供了这样的试剂盒,其包括特异性检测FAM171A2的试剂和/或特异性检测ITGA2B的试剂。优选地,这样的试剂是引物和/或探针和/或向导RNA,相应的方法是普通PCR、RT-PCR、荧光实时定量PCR、等温扩增、DNA/RNA印记、基因芯片、核酸酶切割等。或者优选地,这样的试剂是抗体和/或特异性结合蛋白,相应的方法是免疫印迹、ELISA、原位杂交、免疫组化等。
相应地,在本发明的另一个实施方案中,提供了特异性检测FAM171A2的试剂和/或特异性检测ITGA2B 的试剂,在制备确定个体是否具有颗粒蛋白前体(PGRN)水平失调的风险的试剂盒中的用途。本发明还提供了这样的试剂盒,其包括特异性检测FAM171A2的试剂和/或特异性检测ITGA2B的试剂。优选地,这样的试剂是引物和/或探针和/或向导RNA,相应的方法是普通PCR、RT-PCR、荧光实时定量PCR、等温扩增、DNA/RNA 印记、基因芯片、核酸酶切割等。或者优选地,这样的试剂是抗体和/或特异性结合蛋白,相应的方法是免疫印迹、ELISA、原位杂交、免疫组化等。
相应地,在本发明的另一个实施方案中,提供了特异性检测FAM171A2的试剂和/或特异性检测ITGA2B 的试剂,在制备确定个体是否处于发展成神经退行性疾病的风险中的试剂盒中的用途。本发明还提供了这样的试剂盒,其包括特异性检测FAM171A2的试剂和/或特异性检测ITGA2B的试剂。优选地,这样的试剂是引物和/或探针和/或向导RNA,相应的方法是普通PCR、RT-PCR、荧光实时定量PCR、等温扩增、DNA/RNA 印记、基因芯片、核酸酶切割等。或者优选地,这样的试剂是抗体和/或特异性结合蛋白,相应的方法是免疫印迹、ELISA、原位杂交、免疫组化等。
在本发明的一个具体的实施方案中,特异性检测FAM171A2的试剂是能够在PCR反应中特异性扩增 FAM171A2基因的引物对。
进而,在本发明的一个实施方案中,通过在中枢神经系统超表达FAM171A2,降低了PGRN水平。在本发明的另一个实施方案中,通过在中枢神经系统抑制FAM171A2的表达,升高了PGRN水平,并上调小胶质细胞中的溶酶体酶活性,增强小胶质细胞吞噬、清除、降解异常沉积蛋白的能力。因此,在一个实施方案中,本发明提供了一种治疗神经系统退行性疾病的方法,其特征为在中枢神经系统抑制FAM171A2的表达。在一个具体的实施方案中,通过基因治疗手段以降低FAM171A2的表达。在另一个更具体的实施方案中,基因治疗手段是体内转入siRNA,优选地转入裸siRNA,也可以优选地通过载体转入siRNA,或shRNA,或 miRNA,或反义寡核苷酸(ASO),或者可被转录以生产这些的核酸构建物,或其他可行的核酸形式;载体例如质粒,病毒,或其他常用的载体形式。在另一个更具体的实施方案中,siRNA的序列是5'-GCAAUGGCACUGGUGUAAUTT-3',或5'-AUUACACCAGUGCCAUUGCTT-3'。在另一个更具体的实施方案中,病毒载体由能够有效地将小RNA转导入靶细胞的任何载体组成,优选地,所述病毒载体是慢病毒载体,或优选地,该病毒载体是腺相关病毒载体,或优选地,该病毒载体是腺病毒载体。在其他一个更具体的实施方案中,基因治疗手段是基因编辑,优选地,基因编辑可通过锌指核酸酶(ZFN)、TALE-效应子(TALEN)、CRISPR/Cas系统或NgAgo系统优选地是CRISPR/Cas9系统。在一些实施方案中,基因治疗手段修饰内源性 FAM171A2基因可包括引入如下突变:其降低内源性FAM171A2基因的表达或导致FAM171A2转录产物mRNA 的降解(例如,通过无义介导的衰变)。
在一个具体的实施方案中,降低FAM171A2的体内蛋白含量。在一个更具体的实施方案中,通过拮抗性特异性抗体,结合FAM171A2以降低其水平。在另一个更具体的实施方案中,通过非抗体的特异性结合蛋白/多肽,结合FAM171A2以促进其降解,从而降低其体内水平。
在另一个具体的实施方案中,向中枢神经系统转入FAM171A2基因上游强启动子的反义核酸,或相应的转录因子的反义核酸或抗体,或其他能够达到类似效果的、已知的其他手段或方案,实际达到FAM171A2 分子表达下降的效果。在一些实施方案中,对FAM171A2基因上游强启动子进行基因治疗或基因修饰,使其由野生型变为突变型,或将点突变纠正为野生型序列,最终达到下调下游FAM171A2表达量的效果。
进而,本发明提供了一种通过恢复中枢神经系统单核-巨噬细胞系统(如小胶质细胞)对Aβ等异常沉积蛋白的吞噬能力以治疗神经退行性疾病的方法。在一个具体的实施方案中,向机体内引入能够降低 FAM171A2基因表达水平的转录调控物质。
本发明的另一个方面,提供了降低FAM171A2的表达,或抑制FAM171A2体内水平的物质,在制备用于治疗神经系统退行性疾病,或用于预防或降低神经退行性疾病发病风险,或用于升高PGRN水平或治疗或预防PGRN水平失调所致的疾病,的药物组合物中的用途。优选地,所述降低FAM171A2的表达,或抑制 FAM171A2体内水平的物质,是前文中提及的基因治疗手段以降低FAM171A2的表达的物质,或降低FAM171A2 的体内蛋白含量的物质,或FAM171A2基因上游强启动子的反义核酸,或相应的转录因子的反义核酸或抗体。更优选地,所述物质是siRNA。更加优选地,所述siRNA的序列为5'-GCAAUGGCACUGGUGUAAUTT-3',或5'-AUUACACCAGUGCCAUUGCTT-3'。
本发明首先发现了FAM171A2的表达量对于小胶质细胞吞噬Aβ等异常累积蛋白的促进作用。过多 FAM171A2所致的下调的PGRN表达直接导致小胶质细胞吞噬能力下降,即使在某些异常累积蛋白的生成速度并未加快的生理情形下,仍然不能清除后者,使得其在中枢神经系统中大量累积,进而对神经产生毒性作用,进展到一定程度即发生神经退行性疾病。虽然此类疾病的病因是非常复杂的,难以用单一原因或诱因概而论之,但是如果检出中枢神经系统,如脑脊液中,PGRN表达量显著下调,这一情形可能与神经退行性疾病有很强的关联。而调节FAM171A2的表达量和/或上调PGRN的表达则可能预防或治疗此类神经退行性疾病。
附图说明
图1.GWAS结果和与CSF PGRN水平关联的区域图。a.曼哈顿图(显示单个单核苷酸多态性的log 10(p 值))和qq图。b排列测试后的关联结果。EMP1=经验p值;EMP2=基于置换的校正后的经验家族错误率。c GRN FAM171A2 ITGA2B区域的区域关联结果。d控制rs708384后的区域关联结果。
图2.rs708384的次要等位基因(A等位基因,MAF=0.41)与脑脊液PGRN水平呈剂量依赖性显著相关。
图3.脑脊液PGRN水平的变异性由遗传变异解释的程度。17号染色体解释了约17.4%的脑脊液PGRN水平变异。b最重要的基因座多数与rs708384连锁不平衡。c GRN区中的SNP解释了脑脊液PGRN中的大部分但不是全部变异。对FAM171A2和ITGA2B区域的分析表明,这两个区域分别解释了脑脊液PGRN水平变异的9.1%和5.6%。已显示rs708384可以解释9.1%的变异性。d在CEU人群中rs5848仅与rs708384 有低至中度LD中(r
图4.重复队列中脑脊液PGRN水平与rs708384基因型的关系。
在930名非痴呆中国人参与的更大规模独立队列中,比较了rs708384的AA,AC和CC基因型的脑脊液PGRN 水平,以验证初始观察到的最显著信号。观察脑脊液PGRN水平降低与等位基因A(rs708384突变)剂量有显著相关,而与年龄,性别,教育程度,APOE4基因型,基线MMSE评分,CV和rs5848基因型无关。
图5.GO和通路分析。
a有显著差异的通路类别(p<0.001)主要与神经系统发育,分子运输,信号转导,细胞粘附和对刺激的反应。b基因网络中形成了四个集簇。最重要的基因(FAM171A2和GRN)聚集在一起。c FAM171A2和GRN 获得了最高的z分数。GADO使用Z评分对候选基因进行优先排序:z评分较高的基因更有可能解释该表型。
图6.FAM171A2在脑血管内皮上大量表达。
a b小鼠皮质和海马体上FAM171A2的IHC染色。箭头标记了大脑血管周围和周围的DAB着色。c FAM171A2 和CD31在小鼠皮质上的IF染色。它们的共同定位以星号标记。d小鼠原代脑血管内皮细胞(VEC)上 FAM171A2和CD31的IF染色。
图7.rs708384促进FAM171A2的表达,并随后抑制GRN/PGRN水平。
a萤火虫荧光素酶报道质粒的结构。包含rs708384的序列用红色方块标记。b带有或不带有rs708384 的质粒(c a),包括空对照,在转染到HEK293细胞中后,萤火虫荧光素酶的表达水平不同。用海肾荧光素酶校准发光强度,并以萤火虫/海藻荧光素酶之比表示结果。c,d在FAM171A2过表达后观察到细胞内 GRN的显着降低。e FAM171A2过表达后,上清液PGRN显着下降。
图8.亚组和敏感性分析。
a在亚组分析中,根据性别(男性,p=2.67×10-6;女性,p=1.76×10-6)和基线诊断(HC,p=0.01742; MCI,p=1.36×10-11)划分的不同分层人群中,rs708384的次要等位基因(C)与脑脊液PGRN水平较高相关。值得注意的是,HC人群的效应大小急剧增加(HC的beta=-100.9,MCI的beta=-8.77×10-5)b 进行了进一步调整这些混杂因素的敏感性分析。调整年龄和基线诊断并没有显着改变结果(rs708384的p =3.51×10-12)。在对rs5848进行调整后,我们还发现了rs708384与脑脊液PGRN水平虽然稍降低但仍显著的关联(p=8.28×10-5)。c在排除了自基线以来的三年内发展为AD的73个人之后,rs708384仍然是最显著的SNP(n=359,p=4.73×10-10)。
图9.GRN,FAM171A2和ITGA2B的差异表达分析。
所有结果均根据性别和性别进行了调整。内嗅皮层数据集:GSE26927,GSE26972,GSE48350,GSE5281;海马的数据集:GSE28146,GSE29378,GSE36980,GSE48350,GSE5281;时间皮层数据集:GSE29652,GSE36980, GSE37263,GSE5281;额叶皮质数据集:GSE12685,GSE36980,GSE5281,GSE53890,GSE66333
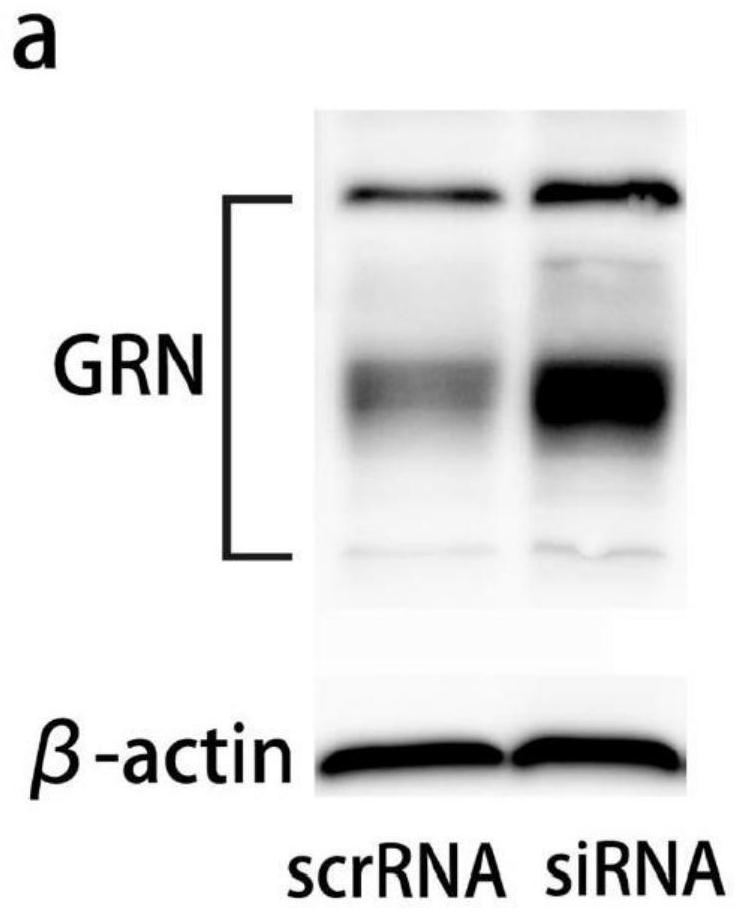
图10.敲低FAM171A2促进细胞内GRN表达。
a,b GRN的细胞内表达。c FAM171A2 siRNA和过表达(OE)的效率。
图11.rs708384基因型与整体认知和脑脊液tau水平的关联。rs708384的“AA”基因型与HC人群中较高的脑脊液tau水平(a)和总体认知水平较差(b&c)相关。
图12.ADNI非西班牙裔白种人样本的MDS图。
使用成对的全基因组通过比例下降的比例同一性估计,检查了样品之间的意外重复和隐秘相关性。除了基于第二个MDS分量的两个离群值之外(在图的底部;IID=024_S_4084和024_S_2239),样本似乎通常形成紧密的簇,表明潜在的种群亚结构。
发明详述
将通过非限制性示例来描述本发明的各优选特征和实施方案。
除非另外指明,本发明的实践将采用常规化学、生物化学、分子生物学、微生物学和免疫学技术,这些技术均在本领域普通技术人员的能力范围之类。此类技术在文献中有所阐述。参见:例如 Sambrook,J.,Fritsch,E.F.和Maniatis,T.(1989)“MolecularCloning:A Laboratory Manual”,第二版,Cold Spring Harbor Laboratory Press;Ausubel,F.M.等人(1995and periodic supplements)Current Protocols in MolecularBiology,Ch.9,13and 16,John Wiley&Sons;Roe,B.、Crabtree、J.和Kahn,A., 1996年,“DNA Isolation and Sequencing:Essential Techniques”,John Wiley&Sons;Polak,J.M. 和McGee,J.O’D.,1990年,“In Situ Hybridization:Principles and Practice”,Oxford University Press;Gait,M.J.(1984)Oligonucleotide Synthesis:A PracticalApproach,IRL Press;以及 Lilley,D.M.And和Dahlberg,J.E.(1992)Methods inEnzymology:DNA Structures Part A:Synthesis and Physical Analysis of DNA,Academic Press。这些一般性文本中的每一个以引用的方式并入本文。
定义
即颗粒蛋白前体(progranulin)。人类的PGRN基因位于17q21.32,是一种由593个氨基酸残基组成的分泌性糖蛋白,非糖基化的PGRN相对分子质量约为6.8×10
神经退行性疾病是指以下病症,包括:前额叶痴呆,阿尔茨海默病,帕金森病,神经元脂褐质沉积症,多发性硬化和肌萎缩侧索硬化。还可以指以下病症,包括:齿状核红核苍白球路易体萎缩症(DRPLA)、神经元核内透明包涵体病(NIHID)、路易体痴呆、唐恩综合征、哈勒沃登-施帕茨病、朊病毒疾病、嗜银颗粒痴呆、皮层基底退化、拳击员痴呆、弥散性神经原纤维缠结、GSS氏病、哈勒沃登-施帕茨病、克-雅二氏病、尼曼-皮克病3型、进行性核上性麻痹、亚急性硬化性全脑炎、脊髓小脑共济失调、亨廷顿病、皮克病和齿状核红核苍白球路易体萎缩症。神经退行性病症的通常特征在于脑中的神经原纤维缠结和/或淀粉样肽的沉积。
神经退行性疾病或病症的“诊断”是指预测与这些病症相关的症状,例如受损的认知功能、累积的A β沉积和神经元细胞死亡。
阿尔茨海默病由大脑区域萎缩所致。虽然人们不知道是什么引发萎缩,但研究发现在阿尔茨海默病患者的大脑中存在淀粉样斑块、神经纤维缠结和乙酰胆碱失衡。大脑中可能损伤健康神经元的血管损伤在阿尔茨海默病患者中也是常见的。
阿尔茨海默病是影响多个脑功能的渐进式病症。疾病的早期迹象通常包括较小的记忆问题,例如忘记最近的活动或位置或物体的名字。随着疾病的发展,记忆问题变得越来越严重并且可引起另外的症状,诸如混淆、定向障碍、难以做出决定、言语障碍以及人格改变。
血管性痴呆由流向大脑的血液减少所致,血液减少将损伤脑细胞。减少的血流可由多种原因引起,包括脑血管变窄(皮层下血管性痴呆)、中风(单发梗塞性痴呆)以及多次小中风(多发梗塞性痴呆)。另外,减少的血流还可能由阿尔茨海默病所致,阿尔茨海默病是被称为混合性痴呆的组合症状。
血管性痴呆的早期症状包括思维缓慢、规划困难、语言困难、注意力和专注度问题以及行为改变。在数月或数年的稳定期之后,症状通常逐步恶化。
帕金森病是黑质中的神经细胞渐进损伤的病症。大脑该区域中的神经细胞产生多巴胺,多巴胺充当大脑部分与控制身体运动的神经系统之间的信使。这些神经细胞损伤导致大脑中所产生的多巴胺量减少,产生的影响是减弱大脑中控制运动的部分的功能。
帕金森病的症状包括震颤、动作迟缓以及肌肉僵硬和不灵活。帕金森病患者还可能经历另外的症状,包括抑郁、便秘、失眠、嗅觉缺失和记忆问题。
肌萎缩性脊髓侧索硬化症(ALS)会影响脑和脊髓中的运动神经元,从而导致这种细胞群退化和死亡。 ALS患者最终会丧失说话、行动和呼吸的能力。虽然ALS的因果关系尚且未知,但特定突变与疾病发生有关,并且是一个广泛研究的领域。
多发性硬化是由脑和脊髓中神经元周围髓鞘上的免疫系统攻击所引起的。这种病会导致神经元之间的通信不佳以及轴突弱化。MS表现为体能退化,并且还会导致视力、疼痛处理和语言能力的改变。虽然性别、种族和环境是MS发生的风险因素,但确切原因尚且未知。
如本文所用,“预防”是指通过施用根据本发明的药物遏制或推迟神经退行性疾病发生的所有行动,并且“治疗”是指通过施用药物使患有或疑似患有神经退行性疾病的对象的症状已经有所转好或者已经被有利地改变的所有行动。
利用常规分子生物学手段对于某个编码蛋白的基因进行表达水平检测,一般可以分为两大类。第一类是针对该基因的mRNA水平进行检测。第二类是针对该基因表达后得到的产物蛋白质进行检测。
过表达或高表达指,相对于正常细胞而言,在细胞中以可检测的更高的水平转录或翻译的蛋白或核酸。该术语包括由于转录、转录后加工、翻译、翻译后加工、细胞定位(例如细胞器、细胞质、细胞核、细胞表面),以及RNA和蛋白质稳定性(相对于正常细胞而言)而引起的过表达。可以使用检测mRNA的常规技术(即RT-PCR、PCR、杂交)或检测蛋白质的常规技术(即ELISA、免疫组化技术)来检测过表达。过表达可以是比正常细胞高10%、20%、30%、40%、50%、60%、70%、80%、90%或更高。在一些实例中,过表达是相对于正常细胞而言高1倍、2倍、3倍、4倍或更高的转录或翻译水平。
低表达是指,相对于正常细胞而言,细胞中以可检测的较低的水平转录或翻译的蛋白或核酸。该术语包括由于转录、转录后加工、翻译、翻译后加工、细胞定位(例如细胞器、细胞质、细胞核、细胞表面),以及RNA和蛋白质稳定性(相对于对照而言)而引起的低表达。可以使用检测mRNA的常规技术(即RT-PCR、PCR、杂交)或检测蛋白质的常规技术(即ELISA、免疫组化技术)来检测低表达。低表达可以是对照低10%、20%、30%、40%、50%、60%、70%、80%、90%或更低。在一些实例中,低表达是相对于对照而言低1倍、2倍、3倍、4倍或更低的转录或翻译水平。
差别化表达或差异化表达一般是指相对于至少一种其它样品而言,在一种样品中的蛋白或核酸是过表达(上调)或低表达(下调)。为了本发明的目的,通常将来自于怀疑患有神经退行性病症的个体的样品与来自于已知患有该病症(阳性对照)或已知对该病症为阴性(阴性对照)的个体的样品进行比较。
“标记物”或“可检测部分”是可以通过分光光度法、光化学法、生物化学法、免疫化学法、化学法或其它物理方法检测的成分。例如,有用的标记物包括32P、荧光染料、电子密度高的试剂、酶(例如 ELISA中通常使用的酶)、生物素、异羟基洋地黄毒苷(digoxigenin)或半抗原和可以被制备成可检测的蛋白(例如通过向肽中掺入放射性标志物)或用于检测与肽特异性反应的抗体的蛋白。标记物可以结合至靶向分子,例如抗体或用于特异性检测靶标化合物的核苷酸序列。
“抑制剂”是例如结合、部分阻断或完全阻断活性、降低、阻止、延迟激活、灭活、去敏、或下调神经退化生物标志物的活性或表达的化合物。抑制剂、激活剂或调节剂还包括经过遗传修饰的神经退化生物标志物,例如具有改变的活性的形式,以及天然存在的和合成的配体、拮抗剂、激动剂、抗体、肽、环肽、核酸、反义分子、核酶、RNAi和siRNA分子,有机小分子等。
“核酸”、“核苷酸”或“多核苷酸”是指脱氧核糖核苷酸或核糖核苷酸,以及它们的单链或双链形式的聚合物,及其互补物。该术语包括含有已知的核苷酸类似物或修饰的骨架残基或键的核酸,其为合成的、天然存在的和非天然存在的,其与参考核酸具有相似的结合特征,并且其以类似于参考核苷酸的方式代谢。此种类似物的实例包括但不限于,硫代磷酸酯、氨基磷酸酯、甲基膦酸酯、手性甲基膦酸酯、 2-O-甲基核糖核苷酸、肽核酸(PNA)。
小RNA是指非编码RNA,其长度通常小于约200个核苷酸或更少并且具有沉默或干扰功能。在其他实施方式中,小RNA长度为约175个核苷酸或更短,约150个核苷酸或更短,约125个核苷酸或更短,约 100个核苷酸或更短,或约75个核苷酸或更短。此类RNA包括微小RNA(miRNA)、小干扰RNA(siRNA)、双链RNA(dsRNA)和短发夹RNA(shRNA)。本公开的“小RNA”应能够抑制或敲减靶基因的基因表达,通常通过会导致靶基因mRNA破坏的途径。
“多肽”、“肽”和“蛋白”在本文中相互替换地使用,其是指氨基酸残基的聚合物。该术语适用于这样的氨基酸聚合物:其中一个或多个氨基酸残基是相应的天然存在的氨基酸的人造化学模拟物,还适用于天然存在的氨基酸聚合物和非天然存在的氨基酸聚合物。
氨基酸是指天然存在的和合成的氨基酸,以及以类似于天然存在的氨基酸的方式作用的氨基酸类似物和氨基酸模拟物。天然存在的氨基酸是由遗传密码编码的氨基酸,以及后来经修饰的氨基酸,例如羟基脯氨酸、γ-羧基谷氨酸和O-磷酸丝氨酸。氨基酸类似物是指与天然存在的氨基酸具有相同的基本化学结构的化合物,即与氢、羧基、氨基和R基团结合的碳,例如高丝氨酸、正亮氨酸、蛋氨酸亚砜、蛋氨酸甲基锍。此种类似物具有修饰的R基团(例如正亮氨酸)或修饰的肽骨架,但保持与天然存在的氨基酸相同的基本化学结构。氨基酸模拟物是指结构不同于氨基酸的一般化学结构、但是以类似于天然存在的氨基酸的方式作用的化学化合物。
抗体是指这样的多肽,其包含来自特异性识别并结合抗原的免疫球蛋白基因的框架区或其片段。识别的免疫球蛋白基因包括κ、λ、α、γ、δ、ε、和μ恒定区基因,以及众多的免疫球蛋白可变区基因。轻链被分类为κ或λ。重链被分类为γ、μ、α、δ或ε,其继而又定义了免疫球蛋白的类别,分别是IgG、IgM、IgA、IgD和IgE。通常,抗体的抗原结合区对于结合的特异性和亲和性是最重要的。抗体可以是多克隆的或单克隆的,衍生自血清、杂交瘤的或重组克隆的,还可以是嵌合的、灵长源化的或人源化的。
示例性的免疫球蛋白(抗体)结构单元包括四聚体。每个四聚体由两对相同的多肽链组成,每对具有一条“轻链”(大约25kDa)和一条“重链”(大约50-70kDa)。每条链的N-末端定义了大约100至110或更多的氨基酸的可变区,主要负责抗原识别。术语轻链可变区(VL)和重链可变区(VH)分别指这些轻链和重链。
抗体以例如完整免疫球蛋白的形式存在,或以通过各种肽酶消化而产生的多种清楚表征的片段的形式存在。因此,例如,胃蛋白酶在铰链区的二硫键下消化抗体,从而产生F(ab)’2,其为Fab的二聚体, Fab本身是通过二硫键与VH-CH1连接的轻链。F(ab)’2可以在温和条件下被还原以破坏铰链区中的二硫键,从而将F(ab)’2二聚体转化为Fab’单体。Fab’单体基本上是具有部分铰链区的Fab(参见Fundamental Immunology(Paul ed.,3ded.1993)。虽然根据完整抗体的消化定义了多种抗体片段,但是本领域技术人员将认识到可以通过化学方式或通过使用重组DNA方法从新(de novo)合成此类片段。因此,本文使用的术语抗体还包括通过对整个抗体进行修饰产生的抗体片段,或那些使用重组DNA方法从新合成的抗体片段(例如单链Fv)或那些使用噬菌体显示文库鉴定的抗体片段(参见,例如McCafferty等人,Nature 348:552-554(1990))。
本文使用的“抗体”还可以指任何功能性VH和VL对(即能够特异性结合表位),其各自以多种构型连接至可以执行多种功能的其它多肽,例如受体、受体抑制剂或VH-VL复合物的稳定剂。
当指称蛋白、核酸、抗体或小分子化合物时,词语“特异性(或选择性)结合”是指结合反应,其通常测定在蛋白或核酸及其它生物制剂的异质群体中是否存在蛋白或核酸(例如STAT3、TYK2或其修饰形式)。在抗体的情况下,在指定的免疫测定条件下,指定抗体与特定蛋白的结合可以是背景的至少2倍,更通常是背景的10倍以上至100倍。在这样的条件下与抗体的特异性结合需要那些就其针对特定蛋白的选择性而被选择的抗体。例如,可以筛选多克隆抗体以只获得那些与所选抗原具有特异性免疫反应而与其它蛋白没有特异性免疫反应的多克隆抗体。这种筛选可以通过减掉与其它分子有交叉反应的抗体来实现。可以使用多种免疫测定形式来选择与特定蛋白具有特异性免疫反应的抗体。例如,固相ELISA免疫测定被常规地用于选择与蛋白具有特异性免疫反应的抗体(参见例如Harlow&Lane,Antibodies,A Laboratory Manual(1988),其描述了可用于测定特异性免疫反应性的免疫测定的形式和条件)。
在一些实施方式中,使用来自生物样品的RNA通过实时或定量PCR来检测FAM171A2和/或ITGA2B的表达。可以通过本领域技术人员已知的任何方法来提取RNA,例如使用Trizol和RNeasy。可以通过本领域技术人员已知的任何方法进行实时PCR,例如使用Applied Biosystem测定法的Taqman实时PCR。相对于汇集的正常肺RNA计算基因表达,并将该表达针对持家基因进行校正。合适的寡核苷酸引物由本领域技术人员选择。
在一个实施方式中,使用核酸结合分子例如探针、寡核苷酸、寡核苷酸阵列和引物来检测RNA生物标志物,以检测患者样品中的差别化的RNA表达。在一个实施方式中,根据本领域已知的标准方法使用 RT-PCR。在另一个实施方式中,可以使用定量PCR测定法(例如可以从例如Applied Biosystems获得的测定法)来检测核酸及其变体。在其它实施方式中,可以使用核酸微阵列来检测核酸。可以使用常规技术例如northern分析来实现核酸的分析,或者,任何其它基于与核酸序列杂交(所述核酸序列互补于标志物编码序列的一部分)的方法(例如条形斑点杂交)也包括在本发明的范围内。可以根据本领域技术人员已知的方法制备与选择的核酸生物标志物结合的试剂,或者从商业渠道购买。
可应用的PCR扩增技术描述于例如Ausubel等人和Innis等人。一般性的核酸杂交方法描述于 Anderson,“Nucleic Acid Hybridization,”BIOS Scientific Publishers,1999。多个核酸序列(例如基因组DNA、mRNA或cDNA)的扩增或杂交还可以从排列在微阵列中的mRNA或cDNA序列进行。微阵列方法的一般性描述见Hardiman,“Microarrays Methodsand Applications:Nuts&Bolts,”DNA Press, 2003;和Baldi等人,“DNA Microarrays andGene Expression:From Experiments to Data Analysis and Modeling,”CambridgeUniversity Press,2002。
在另一个实施方式中,可以使用本领域技术人员已知的多种免疫测定法中的任意方法,在测定中使用抗体试剂来检测生物样品中本发明的神经退化生物标志物的表达和/或活性水平。免疫测定技术和程序的一般性描述见Price and Newman,“Principles andPractice of Immunoassay,”2nd Edition,Grove′ s Dictionaries,1997;和Gosling,“Immunoassays:A Practical Approach,”Oxford University Press, 2000。可以使用多种免疫测定技术,包括竞争性和非竞争性免疫测定。参见,例如Self等人,Curr.Opin.Biotechnol.,7:60-65(1996)。术语“免疫测定”涵盖的技术包括但不限于,酶免疫测定(EIA),例如酶放大免疫测定技术(EMIT)、酶联免疫吸附测定(ELISA)、IgM抗体捕获ELISA(MAC ELISA)和微颗粒酶免疫测定(MEIA);毛细管电泳免疫测定(CEIA);放射免疫测定(RIA);免疫放射性测定(IRMA);荧光极化免疫测定(FPIA);和化学发光测定(CL)。如果需要的话,此类免疫测定法可以进行自动化。免疫测定还可以与激光诱导的荧光联合使用。参见,例如Schmalzing等人,Electrophoresis,18:2184-93(1997); Bao,J.Chromatogr.B.Biomed.Sci.,699:463-80(1997)。脂质体免疫测定,例如流注射脂质体免疫测定和脂质体免疫传感器也适合用于本发明。参见,例如Rongen等人,J.Immunol.Methods,204: 105-133(1997)。此外,比浊法测定也适合用于本发明的方法中,其中,蛋白质/抗体复合物的形成引起增加的光散射,其被转化为峰速信号作为标志物浓度的函数。比浊法测定可通过商业渠道从Beckman Coulter(Brea,CA;Kit#449430)获得,并且可以使用Behring Nephelometer Analyzer(Fink等人, J.Clin.Chem.Clin.Biochem.,27:261-276(1989))来进行。
可以在本文描述的测定法中使用可检测的部分以进行直接或间接检测。可以使用多种可检测部分,标记物的选择取决于所需的灵敏性、与抗体缀合的容易程度、稳定性需求和可用的仪器和处理规定 (disposal provision)。合适的可检测部分包括但不限于,放射性核苷酸、荧光染料(例如荧光素、异硫氰酸荧光素(FITC)、Oregon GreenTM、若丹明、Texasred、异硫氰酸四甲基若丹明(TRITC)、Cy3、Cy5 等)、荧光标志物(例如,绿色荧光蛋白(GFP)、藻红蛋白等)、由肿瘤相关的蛋白酶激活的自动猝灭荧光化合物、酶(例如,荧光素酶、辣根过氧化物酶、碱性磷酸酶等)、纳米颗粒、生物素、异羟基洋地黄毒苷、金属等。
使用核酸特异性的化学发光抗体的化学发光测定法适合于进行蛋白水平的灵敏性、非放射活性检测。以荧光团标记的抗体也是适合的。荧光团的例子包括但不限于DAPI、荧光素、Hoechst 33258、R-藻青蛋白、B-藻红蛋白、R-藻红蛋白、若丹明、Texas red和丽丝胺(lissamine)。间接的标记物包括各种本领域熟知的酶,例如辣根过氧化物酶(HRP)、碱性磷酸酶(AP)、β-半乳糖苷酶、脲酶等。辣根过氧化物酶检测系统可以与例如发色底物四甲基联苯胺(TMB)一起使用,其在存在过氧化氢的情况下产生可以在 450nm检出的可溶性产物。碱性磷酸酶检测系统可以与例如发色底物对硝基苯磷酸盐一起使用,其产生可在450nm容易地检出的可溶性产物。类似地,β-半乳糖苷酶检测系统可以与发色底物邻硝基苯-β-D-吡喃半乳糖苷(ONPG)一起使用,其产生可在410nm检出的可溶性产物。脲酶检测系统可以与底物例如脲 -溴甲酚紫(Sigma Immunochemicals;St.Louis,MO)一起使用。
来自直接或间接标记物的信号可以这样分析,例如:使用分光光度计检测来自发光底物的颜色;使用放射计数器检测放射,例如使用γ计数器检测125I;或者使用荧光计在存在一定波长的光的情况下检测荧光。对于酶联抗体的检测,可以使用分光光度计例如EMAX Microplate Reader(Molecular Devices; Menlo Park,CA)根据生产商的说明书进行定量分析。如果需要的话,本发明的测定可以自动化或由机器人操作,并且可以同时检测来自多个样品的信号。
可以将抗体固定在多种固体载体上,例如磁性或色谱基质颗粒、测定板的表面(例如微量滴定孔)、多片固体基底物质或膜(例如塑料、尼龙、纸)等。可以通过将抗体或多个抗体包被在固体载体上的阵列上来制备检测条(assay strip)。然后可以将这种条浸没在测试样品中并通过洗涤和检测步骤迅速处理以产生可测定的信号,例如有色斑点。
有用的物理形式包括,具有多个不连续、可寻址的位置的表面,以检测多个不同的标志物。此类形式包括微阵列和一些毛细设备。参见,例如Ng等人,J.Cell Mol.Med.,6:329-340(2002)。在这些实施方式中,每个不连续表面位置可以包含抗体以固定一种或多种标志物,以便在每个位置进行检测。或者,表面可以包含一个或多个不连续颗粒(例如微颗粒或纳米颗粒),它们固定在表面的不连续位置上,其中所述微颗粒包含抗体以固定一种或多种标志物以进行检测。
可以通过多种物理形式进行分析。例如,微量滴定板或自动化设备可用于促进大量测试样品的处理。或者,可以开发单一样品形式以促进以实时的方式进行诊断或预后。
或者,本发明的抗体或核酸探针可应用于固定在显微镜玻片上的患者活组织检查样品的切片上。可以使用本领域已知的多种光学或荧光显微镜方法中的任一种来显示产生的抗体染色或原位杂交模式。
在另一种形式中,本发明的各种标志物还提供了用于体内成像的试剂,例如标记试剂的图像,其检测本发明的生物标志物的核酸或编码的蛋白。
与外周血PGRN相比,脑脊液PGRN可能存在不同的调节机制,也避免了PGRN在外周组织中引起肿瘤
在过去的研究中已有报道,位于GRN基因外的的基因座可以调节血液中PGRN水平
目前,已有一些互相独立的研究显示,已确定的基因组范围内的基因座是真实特异的信号,而非I类错误。首先,rs708384呈现出相当显著的p值,并已通过基于置换的经验矫正。而且,这些基因座是被直接确定基因型,减少了人工输入错误的可能。第二,已发现的相关性可以在更大规模的独立样本中重复和验证。第三,生物信息学和体外实验的证据都显示rs708384可调节GRN和FAM171A2基因的表达。第四, FAM171A2和GRN基因紧密相连,有可能在脑内共同发挥作用。最后,rs708384的基因型同神经退行性疾病的发病风险及其脑脊液生物标记具有相关性。这些发现和意象最近的研究相一致,该研究称PGRN可通过影响tau而非Aβ病变而增加AD的发病风险
最近,有报道称在AD患者中,PGRN的表达被上调
GRN和FAM171A2共同参与一些常见的分子通路,这些通路可能参与脑脊液PGRN水平的调节。其中最显著的通路为L1CAM相互作用。L1CAM是轴突糖蛋白,对神经的发生与存活有重要作用
本研究有几点不足。首先,评价队列是基于中国汉族人医院队列,代表性有限。未来尚需在其他人种的社区队列中进行验证。第二,尽管rs5848和rs708384在CEU人群中只具有轻中度的LD,但作为GWAS 阶段重要的协变量,也纳入研究,但是未检测基因型。为了排除输入错误带来的潜在偏倚,我们明确了rs5949 的基因型,并在重复样本的分析中将其添加为协变量。结果显示rs708384对脑脊液PGRN的影响不完全取决于rs5484。第三,我们的体外实验证实了rs708384对FAM171A2的影响,也验证了FAM171A2和GRN 的表达密切相关,但是具体的机制仍有待研究。在接下来的研究中,我们计划进一步研究FAM171A2调节 PGRN的机制。我们也会在多种神经退行性疾病的模型中研究FAM171A2的作用。
具体实施方式
现将通过非限制性示例来描述本发明的各优选特征和实施方案。
实施例1 本发明所采用的实验材料与方法
GWAS纳入标准
在发现性队列中,我们从ADNI-GO/2数据库中纳入了432例(健康对照157例,轻度认知障碍275 例)非西班牙裔非痴呆的白人个体。表1总结了这些样本的基本特征。进行GWAS的数据来源于ADNI 数据库(adni.loni.usc.edu)。包含脑脊液PGRN和GWAS数据的原始队列共有508例纳入对象。我们将分析局限于非西班牙裔白人(n=434),避免由于人群分层产生偏倚的风险。我们使用PLINK软件(beta6.4)
该研究得到所有参与者所在机构审查委员会的批准,并获得所有参与者或授权代表的书面知情同意。
表1.GWAS样本的基本特征
表1注:两个不同诊断组之间差异的显著性的检测由两样本t检验或曼·惠特尼U检验(适用于连续变量) 和带有连续性校正的Pearson卡方检验(适用于分类变量)完成。结果显示年龄(p=1.38×10
性别(p=0.2171),PGRN水平(p=0.9304)以及PGRN测量的变异系数(p=0.2003)在组间无显著性差异。
脑脊液PGRN水平测量
脑脊液PGRN通过质谱检测平台(MSD)
基因型检测及输入
ADNI-GO/2样本基因型是通过Human OmniExpress BeadChip(Illumina,Inc,SanDiego,CA)确定。在相关性研究之前,所有的样品和基因型都经过严格的质量控制,标准如下:SNPs应答率>95%,个体应答率>95%,最小等位基因频率>0.2并且哈代-温伯格平衡检测p>0.001。原始数据库包含710618个基因型编译,包括rs7412和rs429358,这些基因型是单独使用APOE基因检测试剂盒测定的,目的是明确 APOEε2/ε3/ε4亚型
统计学方法
由于脑脊液PGRN数值为偏态分布(shapiro-wilk检验p<0.05)。我们使用R语言工具包进行数据变换,得到呈现正态分布的数据。使用附加了基因模型的PLINK v1.9软件中的线性回归模型来分析脑脊液 PGRN水平和基因多态性的相关性。分步的线性回归分析被用来检验脑脊液PGRN水平是否受诊断、年龄、性别、受教育程度、APOEε4等位基因以及PGRN测量的变异系数等因素的影响。那些p<0.2的因素被纳入为协变量。为了纠正可能导致种群分层的遗传预测的混淆,将成对个体之间的遗传关系矩阵的前三个主要成分进一步作为协变量包括在内。鉴于30%的非痴呆老年人符合AD的生物标志物标准
通过探索与最显著信号(p值最小的SNP)关联的特异性,以检查该关联是否受到与神经退行性疾病有关的其他脑脊液蛋白的影响,包括Aβ,p-tau181(P-tau),总tau(T-tau)
Bonferroni校正后的统计显著性定义为p<5×10
重复性队列
作为独立的重复性队列,非痴呆的北方汉族老年人来自中国阿尔茨海默病生物标志物和生活方式 (CABLE)研究,该研究已得到青岛市医院机构审查委员会的批准。自2017年以来,CABLE是一项正在进行的大规模研究,主要研究中国汉族人群AD的危险因素和生物标志物。CABLE旨在确定AD生物标志物的遗传和环境修饰因素及其在早期诊断中的效用。研究个体在中国山东省青岛市立医院招募。
所有参与者均为年龄在40至90岁之间的汉族。排除标准包括(1)中枢神经系统感染,头部外伤,癫痫,多发性硬化症或其他主要神经系统疾病;(2)主要心理疾病;(3)严重系统疾病(例如恶性肿瘤); (4)家族史遗传疾病:所有参与者均经过临床和神经心理学评估,生化测试以及血液和脑脊液样本采集,并通过结构化问卷和电子病历系统收集了人口统计学信息和病史。
在微孔板读数器(Thermo ScientificTM MultiskanTM MK3)上,使用人Progranulin ELISA试剂盒 (Biovendor Laboratornimedicinaa.s。Czech Republic)确定CABLE中的脑脊液PGRN水平。将样品稀释十倍并一式两份运行。最重要基因座的关联(选择发现阶段中最显著的SNP进行验证。选择了几个位点进行限制性片段长度多态性(RFLP)技术进行基因分型,包括rs708384(用于验证的最显著SNP),rs5848 (GRN中的重要混杂因素),与APOE 4状态(rs7412和rs429358)相关的两个基因座和与先前GWAS的血液PGRN相关的三个基因座(rs660240,rs4747197和rs646776)。直到2019年10月,对930名非痴呆症患者进行了脑脊液PGRN和基因分型的合格测量。数据分析通过R语言进行线性回归,调整了年龄,性别,受教育程度,APOE4状态,基线MMSE和PGRN测量值的变异系数。敏感性分析是通过将rs5848纳入协变量进行的。使用Stata/SE 12.0软件中的metap进行Meta分析以汇总两个阶段发现的p值。此外,我们还检查了与血液PGRN相关的基因座是否也影响了其在脑脊液的水平。
生物信息学分析
使用单核苷酸多态性的NCBI数据库(dbSNP,GRCh37/hg19程序集,105版) http://www.ncbi.nlm.nih.gov/SNP/)
通过HaploReg v4.1进行了增强子富集分析,以评估标记变体在哪些细胞类型中得到了显着富集。使用路线图表观基因组学数据通过四种不同方法定义增强子(包括15-状态核心模型,合并推定表观基因组的25-状态模型,H3K4me1/H3K4me3峰和H3K27ac/H3K9ac峰)
为了验证增强子富集分析的结果并表征这些关联,进行了表达分析,以确定:1)确定的重要基因座所在的基因的表达水平是否与AD病例对照状态相关。通过使用NCBI网络工具GEO2R分析来自GEO的基因表达谱,研究了AD血液
Gene Ontology(GO)的表征分析和路径分析
协会列表GO注释器(ALIGATOR)方法[51]用于搜索Gene Ontology(GO)术语和KEGG通路。如果SNP在1Mb之内且处于高LD(r
使用代表进化统计的蛋白质分析(PANTHER)代表统计学统计测试v9.0[52](www.go.pantherdb.org/org/)和代表基因集共识路径数据库(CPDB)进行了代表生物学之上的基因本体分析analysis Release 33[53](http://cpdb.molgen.mpg.de/)。两者都使用了来自基因本体联盟的数据 (http://www.geneontology.org),并计算了相对于不同背景(CPDB为18,043个基因,PANTHER为20,814 个基因)的候选基因。在分析的34个基因中,CPDB识别了全部基因,而PANTHER将至少一个GO属于分配给29个基因。使用CPDB中的FDR方法校正生成的p值以进行多次测试(p<0.05)。至于PANTHER,使用Bonferroni调整(六个独立测试)后的p值(p<8.33×10
此外,我们使用了多种途径集的来源,包括《京都议定书》中的基因和基因组百科全书(2018年8月 1日,https://www.genome.jp/kegg/
体外实验
1)荧光素酶报告质粒及抗体
该质粒购自中国上海的吉玛制药有限公司。简要地说,我们克隆了含有SNPrs708384或野生型位点的FAM171A2内含子1的约500bp区域,并将克隆的片段插入到pGL3启动子载体中,该载体在萤火虫荧光素酶(luc)报告基因上游包含SV40启动子。转移空的pGL3启动子载体作为系统对照。抗FAM171A2抗体(ab121614,IHC及IF稀释比1:20)。抗FAM171A2抗体(Proteintech, 208361 AP,1:500进行蛋白质印迹)。抗CD31抗体(ab24590,IF稀释比1:250)。抗GRN抗体(ab108608,蛋白印迹稀释比为1:1000)。抗β-actin抗体(Affinity,AF7018,蛋白印迹稀释比为1:1000)。
2)FAM171A2基因沉默及过表达
体外FAM171A2的沉默是通过siRNA(购自GenePharma Company Ltd,中国上海)实现的。序列:5’GCAAUGGCACUGGUGUAAUTT 3',5’AUUACACCAGUGCCAUUGCTT 3'。FAM171A2过表达质粒购自GenePharma。pEX-3载体用于质粒构建。FAM171A2 siRNA和质粒都通过lipofectamine2000 转移到人脐静脉内皮细胞中。转染48小时后证实沉默和过表达效果。
3)细胞培养
我们使用的人脐静脉内皮细胞购自中国上海的iCell公司。该细胞系在2018年通过慢病毒转化使人原代脐静脉内皮细胞永生化,已通过CD31 IF染色证实了细胞纯度。该细胞在ECM+5%FBS +1%内皮生长因子+1%青霉素/链霉素(P/S)中培养。HEK293细胞购自中国科学院细胞研究所。在DMEM+FBS+1%P/S中培养细胞。保温箱保持37℃和5%CO 2的环境。将细胞以1:3传代培养,直至达到80%的汇合度。
4)双荧光素酶报告实验
根据Promega Dual
5)免疫组织化学
通过戊巴比妥钠腹腔注射使小鼠达深度麻醉,并用生理盐水灌注,然后注入4%w/v多聚甲醛(PFF)。使用冷冻切片机对海马和皮层进行冠状位切片,并收集30μm连续切片。将切片在封闭缓冲液(0.01%TX 100,PBS中的20%正常山羊血清,pH 7.4)7.4中孵育1h。随后,将切片在兔抗FAM171A2一抗中于4℃孵育过夜,然后在室温下于生物素化二抗中孵育2h。洗涤3次后,将切片在抗生物素蛋白生物素复合物(ABC Standard,VectorLaboratories)中温育,并通过使用0.025% 3,3'二氨基联苯胺和0.1%H2O2显色。然后将切片粘在微玻片上,并浸入苏木精溶液中1分钟,然后用去离子水洗涤。用乙醇和二甲基苯处理后,切片用中性树胶封片。使用Olympus相机(DP72; Olympus)拍摄图像。
6)免疫荧光染色
该过程类似于免疫组织化学。将脑切片与兔抗FAM 171A2抗体和小鼠抗CD31抗体一起孵育,并摇动过夜。洗涤后,将切片浸入山羊抗兔Alexflour 488和山羊抗小鼠Alexflour 647中2小时,然后洗涤并用Olympus FV10激光共聚焦显微镜观察。
7)蛋白质印迹
将30μg蛋白上样到10%SDS PAGE凝胶泳道中。转膜(PVDF)条件:250mA的恒定电流持续 90分钟。将膜用5%BSA封闭1小时,并在兔抗GRN抗体中孵育过夜。洗涤后,将膜与HRP偶联的二抗孵育2小时。使用Super Signal West Pico化学发光底物(Thermo FisherScientific Inc.)观察印迹。灰度是通过Image J软件测量的。所有实验均设置3次重复。最终数据表示为目标蛋白质相对光密度(ROD)与β-actin ROD之比。
8)酶联免疫吸附实验
我们使用了R&D progranulin quantikine ELISA试剂盒来检测上清液中PGRN的含量。根据ELISA 试剂盒的手册进行实验。简而言之,每孔加入50μl细胞上清,并在室温下孵育2小时。洗涤4次后,每孔加入200μl人PGRN结合物,每孔洗涤量为400μl,在室温下孵育2小时。随后将200ul 底物溶液加入到每个孔中,并在室温下孵育30分钟。最后,通过每孔50μl终止溶液终止反应。在 30分钟内,在450nm处检测每个样品OD,并通过540nm进行校正。根据标准曲线由OD计算出 PGRN含量。
9)统计学
数据以平均值±标准差表示。使用GraphPad Prism 8软件(8.0.2版,GraphPad软件,Inc.,美国加利福尼亚州拉荷亚)通过单因素方差分析,Turkey事后分析和非配对t检验来测试差异。P<0.01 被认为具有统计学意义。
最显著信号与神经退行性疾病之间的相关性
使用ADNI进一步探讨了最显著SNP对感兴趣的大脑区域(海马,海马旁,后扣带回,前胎,楔形,内嗅皮层和中颞叶区域)的认知,大脑Aβ沉积,FDG和体积/厚度的影响。在最初三年内发展为AD的患者被排除在外。进行了线性回归模型,以确定与上述指标在基线时的关联。进行了基于诊断的亚组分析(HC vs MCI)。R包“lm”用于执行上述分析。另外,还搜索了AD
数据和材料可用性:
所有ADNI数据均可通过LONI图像和数据获得档案馆(IDA)和感兴趣的科学家可以在ADNI网站上申请访问(https://ida.loni.usc.edu/collaboration/)
实施例2 两阶段的GWAS筛选
本申请首先在ADNI数据库中分析了432例非痴呆健康个体的遗传信息及其脑脊液PGRN水平。结果如下。
2.1脑脊液PGRN水平受性别、受教育程度及APOE4水平影响。
在GWAS队列中,性别可显著影响脑脊液PGRN水平(p=3.87×10
2.2位于FAM171A2的一些常见突变与脑脊液PGRN水平变化相关。
GWAS研究共纳入了432例非痴呆个体(157例健康对照以及275例轻度认知障碍个体)。年龄(p=1.38 ×10
表2.与脑脊液PGRN水平显著相关(p<10
表2注:SNP=单核苷酸多态性;NA=无数据;
除了位于FAM171A2的位点,ITGA2B和GRN基因上的13个单核苷酸多态性也和脑脊液PGRN水平相关(经验性p=0.0002基于置换的经验家族错误率控制在<0.005)。值得注意的是,8个位点和rs708384 呈现连锁不匹配(r
表e-1:可能于脑脊液PGRN水平相关的基因座
亚组及敏感性分析
亚组分析显示,rs708384的次要等位基因(A)在依据性别和初始诊断划分的阶层内均与脑脊液PGRN 水平降低相关(男性,p=2.67×10
rs708384与PGRN关联的特异性
我们通过检测发现,其他与神经退行性疾病相关的蛋白不影响目前观察到的二者之间的相关性。首先,将脑脊液Aβ42,T-tau,P-tau,α-SYN,NFL或sTREM2纳入协变量后,显著性无明显变化。其次,并未发现Aβ42或P-tau等生物标记与rs708384有明确的关联(r
表e-2.与PGRN水平相关性的特异度
脑脊液PGRN水平变异中可由基因突变解释的部分。
我们也使用了基因组划分分析来估计CSF中PGRN水平的变异中可由染色体及重要基因、位点解释的部分。17号染色体所有的基因型及囊括的突变可以解释脑脊液中PGRN水平变异的17.4%(p<0.05)(图 3a)。值得注意的时,大部分的显著位点和rs708384具有较强的LD(r
因此,我们合理地推断出目前已观察到的相关性显著的基因座更倾向于受rs708384的影响。
独立队列验证及meta分析
我们认为rs708384是和脑脊液PGRN水平最相关的SNP,为了验证真实性,我们在另一更大型的独立队列中再次测量了脑脊液PGRN水平和rs708384基因型。该队列包含了930例非痴呆的中国北方汉族人(表3)。在这一队列中,收集脑脊液时,纳入对象年龄范围是40到88岁(平均年龄62.6岁,标准差10.4 岁)。性别分布为男性380人,女性550人,具有代表性。脑脊液平均PGRN水平为1739pg/ml(标准差 372pg/ml)。平均批内变异系数为2.95%,平均批间变异系数为3.82%。通过验证,我们得到了和之前一样的结论,即:在包含了年龄、性别、受教育程度、APOE4基因型、基线MMSE评分以及变异系数等参数的线性回归模型中,rs708384的A等位基因和脑脊液PGRN水平降低显著相关。(p=7.47×10
表3.CABLE样本的基本特征总结
为了排除GRN基因上rs5848的潜在影响,我们也检测了这一基因型并将其纳入协变量。单因素回归分析显示rs5848和脑脊液PGRN水平显著相关(p<0.005)。然而,将rs708384纳入协变量后,rs5848不再具有显著性(p=0.88)。而控制rs5848后,rs708384和脑脊液PGRN水平的相关性依然具有显著性(p=0.1)。这可能是因为在汉族人群中rs5848和rs708384具有较高但不完全的LD(r
表e-3.在重复性队列中的相关性结果。
表e-3注:所有分析均已矫正年龄、性别、受教育程度、APOE4突变以及基线MMSE评分。
FAM171A2基因中最强信号的功能注释。
强化富集分析显示rs708384标记的突变在一些脑区由显著的富集(例如:海马中部,下颞叶和前额叶)(表e-4),说明这些突变可能和这些脑区的基因调节相关。另外,我们发现FAM171A2在AD患者内页皮层有显著的差异表达(p=0.004,图9)。此外,eQTL分析显示rs708384和它标记的一些突变可以潜在影响血液(表e-5)和一些脑区中(前循环皮质和前额叶皮质)的GRN表达(表e-6)。除了GRN,rs708384 还在多种组织中和FAM171A2、ITGA2B的表达相关,如前额叶(表e-7)。上述证据均显示,位于转录因子结合区的rs708384位点可调节GRN及FAM171A2基因的表达和功能。更有趣的是,我们发现GRN基因和FAM171A2基因的表达(p=1.1×10
表e-4.增强子的富集分析
表e-4注:P<0.05视为有显著性差异。O,观察(observed);E,期望(expected);BLD,血液(blood);BRN,脑 (brain)。
表e-5.在外周血中被标记的变异和基因表达
表e-5注:P<0.05视为差异显著。NA=不详,z评分=beta/标准误,beta是基于有效等位基因的回归系数。Beta>或<0分别代表该等位基因上调或下调所在的基因表达。数据集为[1] http://www.genenetwork.nl/bloodeqtlbrowser/[2]http://cnsgenomics.com/shiny/CAGE/[3]https://preview.n cbi.nlm.nih.gov/gap/eqtl/studies/
表e-6.脑组织中被标记的变异和基因表达
表e-6注:CER=小脑(cerebellum)TX=顶叶皮质(temporal cortex)
表e-7.Rs708384和rs5848对FAM171A2-GRN-ITGA2B表达的影响
FAM171A2同GRN基因可共同出现在一些常见通路中。
二者可产生很多富集的类别:PANTHER分析中由96个类别,CPDB分析中由107个类别。共有34 个显著类别在两种分析中都出现(表e-8)。这些分类一般参与调节神经系统发育,分子转运,信号转导,细胞间黏附和应激反应(图5a)。更重要的是,基因网络分析揭示出31个最显著(p<0.001)的类别,组成4个基因簇。像预期的一样,最显著的基因(GRN,FAM171A2和ITGA2B)富集在一起(图5b)。除了上述分类(p<0.001)之外,这一特殊集簇的GO条目还包括两个新分类:血压调节(p=3.84×10
在具有统计学差异(p<0.05)的16条通路中(12条来自REACTOME,4来自KEGG),L1细胞黏附分子(L1CAM)间的相互租用是差异最显著的通路(REACTOME,p=2.49×10
表e-8.在PANTHER和CPDB分析中都有显著性差异的GO类别
表e-9.通路分析
实施例3 FAM171A2表达研究
在研究该基因功能之前,明确其在脑内的定位是很必要的。因此我们用小鼠脑切片进行了免疫组化和免疫荧光染色。在皮层和海马,沿着血管走行或血管周围的区域我们观察到了显著的DAB着色(图6a,6b)。在相应脑区,FAM171A2的免疫荧光染色与CD31(血管内皮细胞标志物)共定位(图6c)。我们在小鼠原代脑血管内皮细胞中也观察到了FAM171A2的表达。上述结果说明FAM171A2在血管内皮细胞上有比较丰富的表达。
实施例4 rs708384可以调节FAM171A2和GRN基因的表达水平
我们用HEK293细胞进行了双荧光素酶报告实验,来明确rs708384对FAM171A2表达的调节。和野生型相比,我们观察到C突变成A后,报告基因萤火虫荧光素酶与海肾荧光素酶发光亮度之比明显增加,说明上述突变显著增加了FAM171A2的表达(图7b)。
接着,我们检验了FAM171A2表达增加对PGRN水平的影响。在人脐静脉内皮细胞中过表达 FAM171A2后,我们观察到细胞内GRN蛋白表达以及细胞上清中PGRN水平均有明显下降(图7c d e),这一结论同临床数据相符。同时,FAM171A2沉默后,我们观察到细胞内GRN表达上调(图10a,b)。这些结果显示,FAM171A2表达增加可以降低PGRN水平,而沉默可带来相反的效应。
实施例5 rs708384对神经退行性疾病表型的影响
Aβ是在脑脊液中存在的与神经损伤,认知功能,脑结构/代谢以及神经退行性疾病(AD,PD,ALS和 FTD等)风险相关的标志物,因此,我们研究了rs708384是否和Aβ的病理表现相关。Rs708384的AA 基因型和脑脊液中tau水平升高(图11a)以及总体认知功能下降(图11b,c)有关。Rs708384未发现与A β病理表现,脑脊液p-tau水平以及感兴趣脑区的结构和代谢特点有关。目前发现rs708384可以标记10 个r
表e-10:以显著SNP标签和神经退行性疾病易感性标记的变体
表e-10注:p<0.05的显著关联以粗体显示。Beta,效应等位基因的总体估计效应大小;SE,效果大小估算的总体标准误差;p值,荟萃分析使用回归系数(β和标准误差)得出p值。Beta是基于加成模型的效应等位基因的回归系数。17号染色体位置(GRCh37);Beta>0和Beta<0表示此效应等位基因分别增加并降低了AD风险。这些变异均未在1+2GWAS阶段被鉴定出来。*IGAP第一阶段GWAS;在第二阶段GWAS中找不到上述基因座。#先前GWAS中用于PD,ALS和FTD的meta p值;(参考文献:Human Molecular Genetics,2008,Vol.17,No.2336313642,doi:10.1093/hmg/ddn257)
本申请部分参考文献如下,其完整内容通过引用方式并入本申请:
1.X.Zhou,L.Sun,O.A.Brady,K.A.Murphy,F.Hu,Elevated TMEM106B levelsexaggerate lipofuscin accumulation and lysosomal dysfunction in aged micewith progranulin deficiency.Acta neuropathologica communications5,9(2017).
2.S.S.Minami,S.W.Min,G.Krabbe,C.Wang,Y.Zhou,R.Asgarov,Y.Li,L.H.Martens,L.P.Elia,M.E. Ward,L.Mucke,R.V.Farese,Jr.,L.Gan,Progranulinprotects against amyloid beta deposition and toxicity in Alzheimer's diseasemouse models.Nature medicine20,1157-1164(2014).
3.J.M.Van Kampen,D.Baranowski,D.G.Kay,Progranulin gene deliveryprotects dopaminergic neurons in a mouse model of Parkinson's disease.PloSone9,e97032(2014).
4.L.H.Martens,J.Zhang,S.J.Barmada,P.Zhou,S.Kamiya,B.Sun,S.W.Min,L.Gan,S.Finkbeiner,E.J. Huang,R.V.Farese,Jr.,Progranulin deficiency promotesneuroinflammation and neuron loss following toxin-induced injury.The Journalof clinical investigation122,3955-3959(2012).
5.M.C.Chang,K.Srinivasan,B.A.Friedman,E.Suto,Z.Modrusan,W.P.Lee,J.S.Kaminker,D.V.Hansen, M.Sheng,Progranulin deficiency causes impairment ofautophagy and TDP-43accumulation.The Journal of experimental medicine214,2611-2628(2017).
6.J.M.Van Kampen,D.G.Kay,Progranulin gene delivery reduces plaqueburden and synaptic atrophy in a mouse model of Alzheimer's disease.PloSone12,e0182896(2017).
7.M.Suárez-Calvet.,A.Capell.,M.
8.M.M.Carrasquillo,A.M.Nicholson,N.Finch,J.R.Gibbs,M.Baker,N.J.Rutherford,T.A.Hunter,M. DeJesus-Hernandez,G.D.Bisceglio,I.R.Mackenzie,A.Singleton,M.R.Cookson,J.E.Crook,A.Dillman,D. Hernandez,R.C.Petersen,N.R.Graff-Radford,S.G.Younkin,R.Rademakers,Genome-wide screen identifiesrs646776 near sortilin as a regulator of progranulin levels in humanplasma.Am J Hum Genet87,890-897(2010).
9.A.Tonjes,M.Scholz,J.Kruger,K.Krause,D.Schleinitz,H.Kirsten,C.Gebhardt,C.Marzi,H.Grallert,C. Ladenvall,H.Heyne,E.Laurila,J.Kriebel,C.Meisinger,W.Rathmann,C.Gieger,L.Groop,I.Prokopenko,B. Isomaa,F.Beutner,J.Kratzsch,A.Fischer-Rosinsky,A.Pfeiffer,K.Krohn,J.Spranger,J.Thiery,M.Bluher,M. Stumvoll,P.Kovacs,Genome-wide meta-analysis identifies noveldeterminants of circulating serum progranulin. Human molecular genetics27,546-558(2018).
10.L.H.Meeter,H.Patzke,G.Loewen,E.G.Dopper,Y.A.Pijnenburg,R.vanMinkelen,J.C.van Swieten, Progranulin Levels in Plasma andCerebrospinal Fluidin Granulin Mutation Carriers.Dementia and geriatric cognitive disordersextra6,330-340(2016).
11.A.M.Nicholson,N.A.Finch,C.S.Thomas,A.Wojtas,N.J.Rutherford,M.M.Mielke,R.O.Roberts,B.F. Boeve,D.S.Knopman,R.C.Petersen,R.Rademakers,Progranulin protein levels are differently regulated in plasma andCSF.Neurology82,1871-1878(2014).
12.Y.Lu,L.Zheng,W.Zhang,T.Feng,J.Liu,X.Wang,Y.Yu,M.Qi,W.Zhao,X.Yu,W.Tang,Growth factor progranulin contributes to cervical cancer cellproliferation and transformation in vivo and in vitro.Gynecol Oncol134,364-371(2014).
13.F.Arechavaleta-Velasco,C.E.Perez-Juarez,G.L.Gerton,L.Diaz-Cueto,Progranulin and its biological effects in cancer.Medical oncology34,194(2017).
14.H.Qu,H.Deng,Z.Hu,Plasma progranulin concentrations are increasedin patients with type 2 diabetes and obesity and correlated with insulinresistance.Mediators of inflammation2013,360190(2013).
15.J.C.Lambert,C.A.Ibrahim-Verbaas,D.Harold,A.C.Naj,R.Sims,C.Bellenguez,A.L.DeStafano,J.C.Bis, G.W.Beecham,B.Grenier-Boley,G.Russo,T.A.Thorton-Wells,N.Jones,A.V.Smith,V.Chouraki,C. Thomas,M.A.Ikram,D.Zelenika,B.N.Vardarajan,Y.Kamatani,C.F.Lin,A.Gerrish,H.Schmidt,B.Kunkle,M.L.Dunstan,A.Ruiz,M.T.Bihoreau,S.H.Choi,C.Reitz,F.Pasquier,C.Cruchaga,D.Craig,N.Amin,C. Berr,O.L.Lopez,P.L.De Jager,V.Deramecourt,J.A.Johnston,D.Evans,S.Lovestone,L.Letenneur,F.J. Moron,D.C.Rubinsztein,G.Eiriksdottir,K.Sleegers,A.M.Goate,N.Fievet,M.W.Huentelman,M.Gill,K. Brown,M.I.Kamboh,L.Keller,P.Barberger-Gateau,B.McGuiness,E.B.Larson,R.Green,A.J.Myers,C.Dufouil,S.Todd,D.Wallon,S.Love,E.Rogaeva,J.Gallacher,P.St George-Hyslop,J.Clarimon,A.Lleo,A. Bayer,D.W.Tsuang,L.Yu,M.Tsolaki,P.Bossu,G.Spalletta,P.Proitsi,J.Collinge,S.Sorbi,F. Sanchez-Garcia,N.C.Fox,J.Hardy,M.C.DenizNaranjo,P.Bosco,R.Clarke,C.Brayne,D.Galimberti,M. Mancuso,F.Matthews,I.European Alzheimer's Disease,Genetic,D.Environmental Risk in Alzheimer's,C.Alzheimer's Disease Genetic,H.Cohorts for,E.Aging Research in Genomic,S.Moebus,P.Mecocci,M.Del Zompo,W.Maier,H.Hampel,A.Pilotto,M.Bullido,F.Panza,P.Caffarra,B.Nacmias,J.R.Gilbert,M. Mayhaus,L.Lannefelt,H.Hakonarson,S.Pichler,M.M.Carrasquillo,M.Ingelsson,D.Beekly,V.Alvarez,F. Zou,O.Valladares,S.G.Younkin,E.Coto,K.L.Hamilton-Nelson,W.Gu,C.Razquin,P.Pastor,I.Mateo,M.J. Owen,K.M.Faber,P.V.Jonsson,O.Combarros,M.C.O'Donovan,L.B.Cantwell,H.Soininen,D.Blacker,S. Mead,T.H.Mosley,Jr.,D.A.Bennett,T.B.Harris,L.Fratiglioni,C.Holmes,R.F.de Bruijn,P.Passmore,T.J. Montine,K.Bettens,J.I.Rotter,A.Brice,K.Morgan,T.M.Foroud,W.A.Kukull,D.Hannequin,J.F.Powell, M.A.Nalls,K.Ritchie,K.L.Lunetta,J.S.Kauwe,E.Boerwinkle,M.Riemenschneider,M.Boada,M.Hiltuenen, E.R.Martin,R.Schmidt,D.Rujescu,L.S.Wang,J.F.Dartigues,R.Mayeux,C.Tzourio,A.Hofman,M.M. Nothen,C.Graff,B.M.Psaty,L.Jones,J.L.Haines,P.A.Holmans,M.Lathrop,M.A.Pericak-Vance,L.J.Launer,L.A.Farrer,C.M.van Duijn,C.Van Broeckhoven,V.Moskvina,S.Seshadri,J.Williams,G.D. Schellenberg,P.Amouyel,Meta-analysis of 74,046 individualsidentifies 11 new susceptibility loci for Alzheimer's disease.Nat Genet45,1452-1458(2013).
16.C.M.Lill,J.T.Roehr,M.B.McQueen,F.K.Kavvoura,S.Bagade,B.M.Schjeide,L.M.Schjeide,E. Meissner,U.Zauft,N.C.Allen,T.Liu,M.Schilling,K.J.Anderson,G.Beecham,D.Berg,J.M.Biernacka,A. Brice,A.L.DeStefano,C.B.Do,N.Eriksson,S.A.Factor,M.J.Farrer,T.Foroud,T.Gasser,T.Hamza,J.A. Hardy,P.Heutink,E.M.Hill-Burns,C.Klein,J.C.Latourelle,D.M.Maraganore,E.R.Martin,M.Martinez,R.H.Myers,M.A.Nalls,N.Pankratz,H.Payami,W.Satake,W.K.Scott,M.Sharma,A.B.Singleton,K. Stefansson,T.Toda,J.Y.Tung,J.Vance,N.W.Wood,C.P.Zabetian,C.
17.R.Rademakers,J.L.Eriksen,M.Baker,T.Robinson,Z.Ahmed,S.J.Lincoln,N.Finch,N.J.Rutherford,R.J. Crook,K.A.Josephs,B.F.Boeve,D.S.Knopman,R.C.Petersen,J.E.Parisi,R.J.Caselli,Z.K.Wszolek,R.J. Uitti,H.Feldman,M.L.Hutton,I.R.Mackenzie,N.R.Graff-Radford,D.W.Dickson,Common variation inthe miR-659 binding-site of GRN is a major risk factor for TDP43-positivefrontotemporal dementia.Human molecular genetics17,3631-3642(2008).
18.H.Takahashi,Z.A.Klein,S.M.Bhagat,A.C.Kaufman,M.A.Kostylev,T.Ikezu,S.M.Strittmatter,I. Alzheimer's Disease Neuroimaging,Opposing effects ofprogranulin deficiency on amyloid and tau pathologies via microglial TYROBPnetwork.Acta Neuropathol133,785-807(2017).
19.Y.A.Cooper,D.Nachun,D.Dokuru,Z.Yang,A.M.Karydas,G.Serrero,B.Yue,I.Alzheimer's Disease Neuroimaging,A.L.Boxer,B.L.Miller,G.Coppola,Progranulinlevels in blood in Alzheimer's disease and mild cognitive impairment.Annalsof clinical and translational neurology5,616-629(2018).
20.C.R.Jack,Jr.,D.A.Bennett,K.Blennow,M.C.Carrillo,B.Dunn,S.B.Haeberlein,D.M.Holtzman,W. Jagust,F.Jessen,J.Karlawish,E.Liu,J.L.Molinuevo,T.Montine,C.Phelps,K.P.Rankin,C.C.Rowe,P. Scheltens,E.Siemers,H.M.Snyder,R.Sperling,Contributors,NIA-AA Research Framework:Toward abiological definition of Alzheimer's disease.Alzheimer's&dementia:the journalof the Alzheimer's Association14,535-562(2018).
21.D.Lutz,G.Loers,R.Kleene,I.Oezen,H.Kataria,N.Katagihallimath,I.Braren,G.Harauz,M.Schachner, Myelin basic protein cleaves cell adhesionmolecule L1 and promotes neuritogenesis and cell survival.The Journal ofbiological chemistry289,13503-13518(2014).
22.N.Djogo,I.Jakovcevski,C.Muller,H.J.Lee,J.C.Xu,M.Jakovcevski,S.Kugler,G.Loers,M.Schachner, Adhesion molecule L1 binds to amyloid beta andreduces Alzheimer's disease pathology in mice.Neurobiology of disease56,104-115(2013).
23.C.C.Chang,C.C.Chow,L.C.Tellier,S.Vattikuti,S.M.Purcell,J.J.Lee,Second-generation PLINK:rising to the challenge of larger and richerdatasets.Gigascience4,7(2015).
24.A.L.Price,N.J.Patterson,R.M.Plenge,M.E.Weinblatt,N.A.Shadick,D.Reich,Principal components analysis corrects for stratification in genome-wide association studies.Nat Genet38,904-909(2006).
25.A.Capell,S.Liebscher,K.Fellerer,N.Brouwers,M.Willem,S.Lammich,I.Gijselinck,T.Bittner,A.M. Carlson,F.Sasse,B.Kunze,H.Steinmetz,R.Jansen,D.Dormann,K.Sleegers,M.Cruts,J.Herms,C.Van Broeckhoven,C.Haass,Rescue ofprogranulin deficiency associated with frontotemporal lobar degeneration byalkalizing reagents and inhibition of vacuolar ATPase.J Neurosci31,1885-1894(2011).
26.S.Kim,S.Swaminathan,L.Shen,S.L.Risacher,K.Nho,T.Foroud,L.M.Shaw,J.Q.Trojanowski,S.G. Potkin,M.J.Huentelman,D.W.Craig,B.M.DeChairo,P.S.Aisen,R.C.Petersen,M.W.Weiner,A.J.Saykin, I.Alzheimer's Disease Neuroimaging,Genome-wide association study of CSF biomarkers Abeta1-42,t-tau,and p-tau181pin the ADNI cohort.Neurology76,69-79(2011).
27.J.A.Schneider,N.T.Aggarwal,L.Barnes,P.Boyle,D.A.Bennett,Theneuropathology of older persons with and without dementia from communityversus clinic cohorts.Journal of Alzheimer's disease:JAD18,691-701 (2009).
28.L.M.Shaw,H.Vanderstichele,M.Knapik-Czajka,C.M.Clark,P.S.Aisen,R.C.Petersen,K.Blennow,H. Soares,A.Simon,P.Lewczuk,R.Dean,E.Siemers,W.Potter,V.M.Lee,J.Q.Trojanowski,I.Alzheimer's Disease Neuroimaging,Cerebrospinalfluid biomarker signature in Alzheimer's disease neuroimaging initiativesubjects.Annals of neurology65,403-413(2009).
29.A.Olsson,H.Vanderstichele,N.Andreasen,G.De Meyer,A.Wallin,B.Holmberg,L.Rosengren,E. Vanmechelen,K.Blennow,Simultaneous measurement ofbeta-amyloid(1-42),total tau,and phosphorylated tau (Thr181)in cerebrospinalfluid by the xMAP technology.Clinical chemistry51,336-345(2005).
30.Z.Hong,M.Shi,K.A.Chung,J.F.Quinn,E.R.Peskind,D.Galasko,J.Jankovic,C.P.Zabetian,J.B. Leverenz,G.Baird,T.J.Montine,A.M.Hancock,H.Hwang,C.Pan,J.Bradner,U.J.Kang,P.H.Jensen,J. Zhang,DJ-1 and alpha-synuclein in humancerebrospinal fluid as biomarkers of Parkinson's disease.Brain:a journal ofneurology133,713-726(2010).
31.A.Nordlund,S.Rolstad,P.Hellstrom,M.Sjogren,S.Hansen,A.Wallin,TheGoteborg MCI study:mild cognitive impairment isa heterogeneouscondition.Journal of neurology,neurosurgery,and psychiatry76, 1485-1490(2005).
32.M.Suarez-Calvet,G.Kleinberger,M.A.Araque Caballero,M.Brendel,A.Rominger,D.Alcolea,J.Fortea,A. Lleo,R.Blesa,J.D.Gispert,R.Sanchez-Valle,A.Antonell,L.Rami,J.L.Molinuevo,F.Brosseron,A. Traschutz,M.T.Heneka,H.Struyfs,S.Engelborghs,K.Sleegers,C.Van Broeckhoven,H.Zetterberg,B.Nellgard,K.Blennow,A.Crispin,M.Ewers,C.Haass,sTREM2 cerebrospinal fluidlevels are a potential biomarker for microglia activity in early-stageAlzheimer's disease and associate with neuronal injury markers. EMBO MolMed8,466-476(2016).
33.J.Yang,S.H.Lee,M.E.Goddard,P.M.Visscher,GCTA:a tool for genome-wide complex trait analysis.Am J Hum Genet88,76-82(2011).
34.R.J.Pruim,R.P.Welch,S.Sanna,T.M.Teslovich,P.S.Chines,T.P.Gliedt,M.Boehnke,G.R.Abecasis,C. J.Willer,LocusZoom:regional visualization ofgenome-wide association scan results.Bioinformatics26, 2336-2337(2010).
35.J.Yang,T.A.Manolio,L.R.Pasquale,E.Boerwinkle,N.Caporaso,J.M.Cunningham,M.de Andrade,B. Feenstra,E.Feingold,M.G.Hayes,W.G.Hill,M.T.Landi,A.Alonso,G.Lettre,P.Lin,H.Ling,W.Lowe,R. A.Mathias,M.Melbye,E.Pugh,M.C.Cornelis,B.S.Weir,M.E.Goddard,P.M.Visscher,Genome partitioning of geneticvariation for complex traits using common SNPs.Nat Genet43,519-525(2011).
36.S.T.Sherry,M.H.Ward,M.Kholodov,J.Baker,L.Phan,E.M.Smigielski,K.Sirotkin,dbSNP:the NCBI database of genetic variation.Nucleic Acids Res29,308-311(2001).
37.A.Z.Dayem Ullah,J.Oscanoa,J.Wang,A.Nagano,N.R.Lemoine,C.Chelala,SNPnexus:assessing the functional relevance of genetic variation tofacilitate the promise of precision 31 medicine.Nucleic Acids Res46,W109-W113(2018).
38.L.D.Ward,M.Kellis,HaploReg:a resource for exploring chromatinstates,conservation,and regulatory motif alterations within sets ofgenetically linked variants.Nucleic Acids Res40,D930-934(2012).
39.A.P.Boyle,E.L.Hong,M.Hariharan,Y.Cheng,M.A.Schaub,M.Kasowski,K.J.Karczewski,J.Park,B.C. Hitz,S.Weng,J.M.Cherry,M.Snyder,Annotation offunctional variation in personal genomes using RegulomeDB.Genome research22,1790-1797(2012).
40.L.D.Ward,M.Kellis,HaploReg v4:systematic mining of putative causalvariants,cell types,regulators and target genes for human complex traits anddisease.Nucleic Acids Res44,D877-881(2016).
41.T.Barrett,S.E.Wilhite,P.Ledoux,C.Evangelista,I.F.Kim,M.Tomashevsky,K.A.Marshall,K.H. Phillippy,P.M.Sherman,M.Holko,A.Yefanov,H.Lee,N.Zhang,C.L.Robertson,N.Serova,S.Davis,A. Soboleva,NCBI GEO:archive forfunctional genomics data sets--update.Nucleic Acids Res41,D991-995(2013).
42.A.J.Myers,J.R.Gibbs,J.A.Webster,K.Rohrer,A.Zhao,L.Marlowe,M.Kaleem,D.Leung,L.Bryden,P. Nath,V.L.Zismann,K.Joshipura,M.J.Huentelman,D.Hu-Lince,K.D.Coon,D.W.Craig,J.V.Pearson,P. Holmans,C.B.Heward,E.M.Reiman,D.Stephan,J.Hardy,A survey of genetic human cortical gene expression. NatGenet39,1494-1499(2007).
43.M.Xu,D.F.Zhang,R.Luo,Y.Wu,H.Zhou,L.L.Kong,R.Bi,Y.G.Yao,Asystematic integrated analysis of brain expression profiles reveals YAP1 andother prioritized hub genes as important upstream regulators in Alzheimer'sdisease.Alzheimer's&dementia:the journal of the Alzheimer's Association14,215-229(2018).
44.Y.Benjamini,R.Heller,Screening for partial conjunctionhypotheses.Biometrics64,1215-1222(2008).
45.F.Zou,H.S.Chai,C.S.Younkin,M.Allen,J.Crook,V.S.Pankratz,M.M.Carrasquillo,C.N.Rowley,A.A. Nair,S.Middha,S.Maharjan,T.Nguyen,L.Ma,K.G.Malphrus,R.Palusak,S.Lincoln,G.Bisceglio,C. Georgescu,N.Kouri,C.P.Kolbert,J.Jen,J.L.Haines,R.Mayeux,M.A.Pericak-Vance,L.A.Farrer,G.D.Schellenberg,C.Alzheimer's Disease Genetics,R.C.Petersen,N.R.Graff-Radford,D.W.Dickson,S.G. Younkin,N.Ertekin-Taner,Brain expression genome-wideassociation study(eGWAS)identifies human disease-associated variants.PLoSGenet8,e1002707(2012).
46.G.T.Consortium,Human genomics.The Genotype-Tissue Expression(GTEx)pilot analysis:multitissue gene regulation in humans.Science348,648-660(2015).
47.H.J.Westra,M.J.Peters,T.Esko,H.Yaghootkar,C.Schurmann,J.Kettunen,M.W.Christiansen,B.P. Fairfax,K.Schramm,J.E.Powell,A.Zhernakova,D.V.Zhernakova,J.H.Veldink,L.H.Van den Berg,J. Karjalainen,S.Withoff,A.G.Uitterlinden,A.Hofman,F.Rivadeneira,P.A.C.Hoen,E.Reinmaa,K.Fischer,M.Nelis,L.Milani,D.Melzer,L.Ferrucci,A.B.Singleton,D.G.Hernandez,M.A.Nalls,G.Homuth,M.Nauck,D. Radke,U.Volker,M.Perola,V.Salomaa,J.Brody,A.Suchy-Dicey,S.A.Gharib,D.A.Enquobahrie,T.Lumley, G.W.Montgomery,S.Makino,H.Prokisch,C.Herder,M.Roden,H.Grallert,T.Meitinger,K.Strauch,Y.Li,R. C.Jansen,P.M.Visscher,J.C.Knight,B.M.Psaty,S.Ripatti,A.Teumer,T.M.Frayling,A.Metspalu,J.B.J. van Meurs,L.Franke,Systematic identification of trans eQTLs asputative drivers of known disease associations. Nat Genet45,1238-1243(2013).
48.L.R.Lloyd-Jones,A.Holloway,A.McRae,J.Yang,K.Small,J.Zhao,B.Zeng,A.Bakshi,A.Metspalu,M. Dermitzakis,G.Gibson,T.Spector,G.Montgomery,T.Esko,P.M.Visscher,J.E.Powell,The Genetic Architecture of Gene Expression inPeripheral Blood.Am J Hum Genet100,228-237(2017).
49.R.Joehanes,X.Zhang,T.Huan,C.Yao,S.X.Ying,Q.T.Nguyen,C.Y.Demirkale,M.L.Feolo,N.R. Sharopova,A.Sturcke,A.A.Schaffer,N.Heard-Costa,H.Chen,P.C.Liu,R.Wang,K.A.Woodhouse,K. Tanriverdi,J.E.Freedman,N.Raghavachari,J.Dupuis,A.D.Johnson,C.J.O'Donnell,D.Levy,P.J.Munson, Integrated genome-wide analysisof expression quantitative trait loci aids interpretation of genomicassociation studies.Genome Biol18,16(2017).
50.Z.Tang,C.Li,B.Kang,G.Gao,C.Li,Z.Zhang,GEPIA:a web server forcancer and normal gene expression profiling and interactive analyses.NucleicAcids Res45,W98-W102(2017).
51.P.Holmans,E.K.Green,J.S.Pahwa,M.A.Ferreira,S.M.Purcell,P.Sklar,C.Wellcome Trust Case-Control, M.J.Owen,M.C.O'Donovan,N.Craddock,Geneontology analysis of GWA study data sets provides insights into the biologyof bipolar disorder.Am J Hum Genet85,13-24(2009).
52.H.Mi,A.Muruganujan,P.D.Thomas,PANTHER in 2013:modeling theevolution of gene function,and other gene attributes,in the context ofphylogenetic trees.Nucleic Acids Res41,D377-386(2013).
53.A.Kamburov,K.Pentchev,H.Galicka,C.Wierling,H.Lehrach,R.Herwig,ConsensusPathDB:toward a more complete picture of cell biology.Nucleic AcidsRes39,D712-717(2011).
54.M.Kanehisa,S.Goto,Y.Sato,M.Furumichi,M.Tanabe,KEGG for integrationand interpretation of large-scale molecular data sets.Nucleic Acids Res40,D109-114(2012).
55.D.Croft,A.F.Mundo,R.Haw,M.Milacic,J.Weiser,G.Wu,M.Caudy,P.Garapati,M.Gillespie,M.R. Kamdar,B.Jassal,S.Jupe,L.Matthews,B.May,S.Palatnik,K.Rothfels,V.Shamovsky,H.Song,M.Williams, E.Birney,H.Hermjakob,L.Stein,P.D'Eustachio,The Reactome pathway knowledgebase.Nucleic Acids Res42,D472-477(2014).
56.L.Zhou,F.Zhao,Prioritization and functional assessment ofnoncoding variants associated with complex diseases.Genome Med10,53(2018).
57.C.M.Lill,O.Abel,L.Bertram,A.Al-Chalabi,Keeping up with geneticdiscoveries in amyotrophic lateral sclerosis:the ALSoD and ALSGenedatabases.Amyotrophic lateral sclerosis:official publication of the WorldFederation of Neurology Research Group on Motor Neuron Diseases12,238-249(2011).
58.R.Ferrari,M.Grassi,E.Salvi,B.Borroni,F.Palluzzi,D.Pepe,F.D'Avila,A.Padovani,S.Archetti,I. Rainero,E.Rubino,L.Pinessi,L.Benussi,G.Binetti,R.Ghidoni,D.Galimberti,E.Scarpini,M.Serpente,G. Rossi,G.Giaccone,F.Tagliavini,B.Nacmias,I.Piaceri,S.Bagnoli,A.C.Bruni,R.G.Maletta,L.Bernardi,A. Postiglione,G.Milan,M.Franceschi,A.A.Puca,V.Novelli,C.Barlassina,N.Glorioso,P.Manunta,A. Singleton,D.Cusi,J.Hardy,P.Momeni,A genome-widescreening and SNPs-to-genes approach to identify novel genetic risk factorsassociated with frontotemporal dementia.Neurobiology of aging36,2904 e2913-2926(2015).
59.R.Ferrari,D.G.Hernandez,M.A.Nalls,J.D.Rohrer,A.Ramasamy,J.B.Kwok,C.Dobson-Stone,W.S. Brooks,P.R.Schofield,G.M.Halliday,J.R.Hodges,O.Piguet,L.Bartley,E.Thompson,E.Haan,I.Hernandez, A.Ruiz,M.Boada,B.Borroni,A.Padovani,C.Cruchaga,N.J.Cairns,L.Benussi,G.Binetti,R.Ghidoni,G. Forloni,D.Galimberti,C.Fenoglio,M.Serpente,E.Scarpini,J.Clarimon,A.Lleo,R.Blesa,M.L.Waldo,K.Nilsson,C.Nilsson,I.R.Mackenzie,G.Y.Hsiung,D.M.Mann,J.Grafman,C.M.Morris,J.Attems,T.D. Griffiths,I.G.McKeith,A.J.Thomas,P.Pietrini,E.D.Huey,E.M.Wassermann,A.Baborie,E.Jaros,M.C. Tierney,P.Pastor,C.Razquin,S.Ortega-Cubero,E.Alonso,R.Perneczky,J.Diehl-Schmid,P.Alexopoulos,A. Kurz,I.Rainero,E.Rubino,L.Pinessi,E.Rogaeva,P.St George-Hyslop,G.Rossi,F.Tagliavini,G.Giaccone,J. B.Rowe,J.C.Schlachetzki,J.Uphill,J.Collinge,S.Mead,A.Danek,V.M.Van Deerlin,M.Grossman,J.Q. Trojanowski,J.van der Zee,W.Deschamps,T.VanLangenhove,M.Cruts,C.Van Broeckhoven,S.F.Cappa,I. Le Ber,D.Hannequin,V.Golfier,M.Vercelletto,A.Brice,B.Nacmias,S.Sorbi,S.Bagnoli,I.Piaceri,J.E.Nielsen,L.E.Hjermind,M.Riemenschneider,M.Mayhaus,B.Ibach,G.Gasparoni,S.Pichler,W.Gu,M.N. Rossor,N.C.Fox,J.D.Warren,M.G.Spillantini,H.R.Morris,P.Rizzu,P.Heutink,J.S.Snowden,S. Rollinson,A.Richardson,A.Gerhard,A.C.Bruni,R.Maletta,F.Frangipane,C.Cupidi,L.Bernardi,M.Anfossi, M.Gallo,M.E.Conidi,N.Smirne,R.Rademakers,M.Baker,D.W.Dickson,N.R.Graff-Radford,R.C. Petersen,D.Knopman,K.A.Josephs,B.F.Boeve,J.E.Parisi,W.W.Seeley,B.L.Miller,A.M.Karydas,H. Rosen,J.C.van Swieten,E.G.Dopper,H.Seelaar,Y.A.Pijnenburg,P.Scheltens,G.Logroscino,R.Capozzo,V. Novelli,A.A.Puca,M.Franceschi,A.Postiglione,G.Milan,P.Sorrentino,M.Kristiansen,H.H.Chiang,C. Graff,F.Pasquier,A.Rollin,V.Deramecourt,F.Lebert,D.Kapogiannis,L.Ferrucci,S.Pickering-Brown,A.B.Singleton,J.Hardy,P.Momeni,Frontotemporal dementia and its subtypes:a genome-wide association study.The Lancet.Neurology13,686-699(2014).
60.I.Broce,C.M.Karch,N.Wen,C.C.Fan,Y.Wang,C.H.Tan,N.Kouri,O.A.Ross,G.U.Hoglinger,U. Muller,J.Hardy,F.T.D.G.C.International,P.Momeni,C.P.Hess,W.P.Dillon,Z.A.Miller,L.W.Bonham, G.D.Rabinovici,H.J.Rosen,G.D.Schellenberg,A.Franke,T.H.Karlsen,J.H.Veldink,R.Ferrari,J.S. Yokoyama,B.L.Miller,O.A.Andreassen,A.M.Dale,R.S.Desikan,L.P.Sugrue,Immune-related geneticenrichment in frontotemporal dementia:An analysis of genome-wide associationstudies.PLoS medicine15, e1002487(2018).
61.A.Mishra,R.Ferrari,P.Heutink,J.Hardy,Y.Pijnenburg,D.Posthuma,F.T.D.G.C.International, Gene-based association studies report genetic linksfor clinical subtypes of frontotemporal dementia.Brain:a journal ofneurology140,1437-1446(2017) 。
- 神经退行性疾病检测和治疗的新靶标
- 用于治疗神经退行性疾病的伊格美新
