一种茄子抗青枯病性状紧密连锁的SNP分子标记及其应用
文献发布时间:2023-06-19 18:53:06
技术领域
本发明属于植物分子标记辅助育种技术领域,具体涉及一种茄子抗青枯病性状紧密连锁的SNP分子标记及其应用。
背景技术
由茄科雷尔氏菌(Ralstonia solanacearum)侵染茄子引起的青枯病是茄子生产的毁灭性病害,危害严重时经济损失高达50-60%,严重阻碍了茄子产业的发展。
优良的种质资源是品种选育的基础,虽然国内外学者利用苗期接种鉴定和自然病圃对茄子种质资源开展抗青枯病筛选和评价研究发现多数茄子野生种及近缘野生种对青枯病具有较强的抗性,但这些茄子具有生长势强旺、茎叶多刺、果小等特点,因而无法在育种中直接利用。常年高温多雨的热带和亚热带气候是青枯病频发的主要因素,也使得拥有该自然条件的地区具有较为丰富的抗病资源,但由于茄子栽培类型和果实性状各异,所收集鉴评的抗源与各地域的育种目标差距较大,且材料创新难度大、周期长。因此,提高茄子抗青枯病育种的效率至关重要,申请人在茄子育种实践中筛选获得具有优良商品性和丰产性的高抗青枯病自交系(R06112)和高感青枯病自交系(S51193)(李涛,孙保娟,李植良,黎振兴,罗少波,徐小万,衡周,宫超,游倩.茄子对青枯病的抗性机制研究现状与展望[J].广东农业科学,2021,48(09):40-50.DOI:10.16768/j.issn.1004-874X.2021.09.005.)。在此基础上,开展茄子抗青枯病基因的分子标记开发和精细定位,通过精确的抗病遗传分析和紧密的连锁标记的获得,可以更好地服务于茄子抗青枯病的分子育种过程。
针对茄子重要性状进行遗传研究,挖掘其调控基因,进而开发连锁分子标记应用于分子辅助育种,对于提高育种效率有重要意义。近年来,基于高通量测序进行混合样本分组分析(Bulked segregant analysis,BSA),能够实现快速定位目标基因并已得到广泛应用。随着基于荧光信号的高通量SNP分型技术(如PARMS、KASP、TaqMan等)的发展成熟,SNP标记在主要农作物中得到广泛应用。因此,利用高通量测序,挖掘调控茄子抗青枯病性状的相关基因,并开发连锁SNP标记应用于育种实践,将在很大程度上提高茄子抗青枯病育种的效率。
发明内容
为了克服上述现有技术的不足,本发明公开了一种茄子抗青枯病性状紧密连锁的SNP分子标记,可用于茄子抗青枯病种质资源的预测和筛选,为茄子抗青枯病育种提供新的途径。
为了实现上述目的,本发明所采用的技术方案是:
本发明第一方面提供了茄子抗青枯病性状紧密连锁的SNP分子标记SNP910在茄子分子辅助育种中的应用,所述应用包括茄子青枯病抗性早期预测和选育茄子抗青枯病品种,所述SNP分子标记SNP910具有如SEQ NO.1所示的核苷酸,SEQ NO.1所示序列的第22个碱基存在SNP变异位点C/A。
本发明第二方面提供了第一方面所述的茄子抗青枯病性状紧密连锁的SNP分子标记SNP910的扩增引物在茄子分子辅助育种中的应用,所述应用包括茄子青枯病抗性早期预测和选育茄子抗青枯病品种。
本发明采用分子标记BSA-seq方法进行SNP变异位点查找,发现与茄子青枯病抗性相关的SNP位点位于10号染色体上第91075951bp碱基,且核苷酸多态性为C/A,并将该SNP分子标记命名为SNP910。同时,根据找到的与茄子青枯病抗性相关的SNP分子标记,进一步设计了检测SNP位点的KASP引物。本发明首次公开控制茄子抗青枯病性状的基因座位及与其紧密连锁的SNP分子标记,该分子标记能够准确、快速地鉴别出抗青枯病茄子品种和个体,所述SNP分子标记的CC或CA基因型对应的茄子青枯病为抗病,所述的SNP分子标记的AA基因型对应的茄子青枯病为感病,可用于茄子抗青枯病种质资源的预测和筛选,也可以用于通过分子辅助育种创制茄子抗青枯病种质资源。
优选地,所述扩增引物包括两条分别与C和A结合的正向引物以及一条反向引物。
更优选地,所述扩增引物中,两条正向引物的5’端分别含有HEX基团和FAM基团。
更优选地,所述扩增引物包括如SEQ NO.2和SEQ NO.3所示的两条正向引物以及如SEQNO.4所示的反向引物。
本发明第三方面提供了一种鉴定茄子青枯病抗性的方法,该方法包括以下步骤:
S1、提取待测茄子的基因组DNA;
S2、以待测茄子提取的DNA为模板,利用茄子抗青枯病性状紧密连锁的SNP分子标记SNP910的扩增引物进行PCR扩增反应,所述扩增引物包括两条分别与C和A结合的正向引物以及一条反向引物,且两条正向引物的5’端分别含有HEX基团和FAM基团;
S3、采用荧光检测平台对步骤S2的产物进行分型,若产物的荧光信号为HEX(对应碱基为C或杂合型碱基C/A)则该样品为抗病,若产物的荧光信号为FAM(对应碱基为A)则该样品为感病。
优选地,茄子抗青枯病性状紧密连锁的SNP分子标记SNP910的扩增引物与第二方面所述的引物相同。更优选如SEQ NO.2和SEQ NO.3所示的两条正向引物以及如SEQ NO.4所示的反向引物。
优选地,PCR扩增反应的体系为10μL,其中包含2×KASP master mix 5μL,反向引物0.4μL,正向引物各0.15μL,10-100ng基因组DNA 1μL,ddH
优选地,PCR扩增反应的程序为94℃、15min;94℃、20s,65-57℃Touch-down,-0.88℃/cycles,10个循环;94℃、20s,57℃、1min,30个循环。
优选地,步骤S3的分型方法为:使用TECAN infinite M1000酶标仪读取荧光信号,并利用在线软件snpdecoder解析转换荧光信号,得到清晰直观的分型图。
与现有技术相比,本发明的有益效果是:
本发明首次公开了控制茄子抗青枯病性状的基因座位及与其紧密连锁的SNP分子标记,所述SNP分子标记为SNP910,且核苷酸多态性为C/A。该分子标记能够准确、快速地鉴别出抗青枯病茄子品种和个体,可用于茄子抗青枯病种质资源的预测和筛选,也可以用于分子辅助育种创制抗青枯病茄子品种,具有重要的应用价值。
附图说明
图1为伤根灌根法实施后单株的发病分级图;
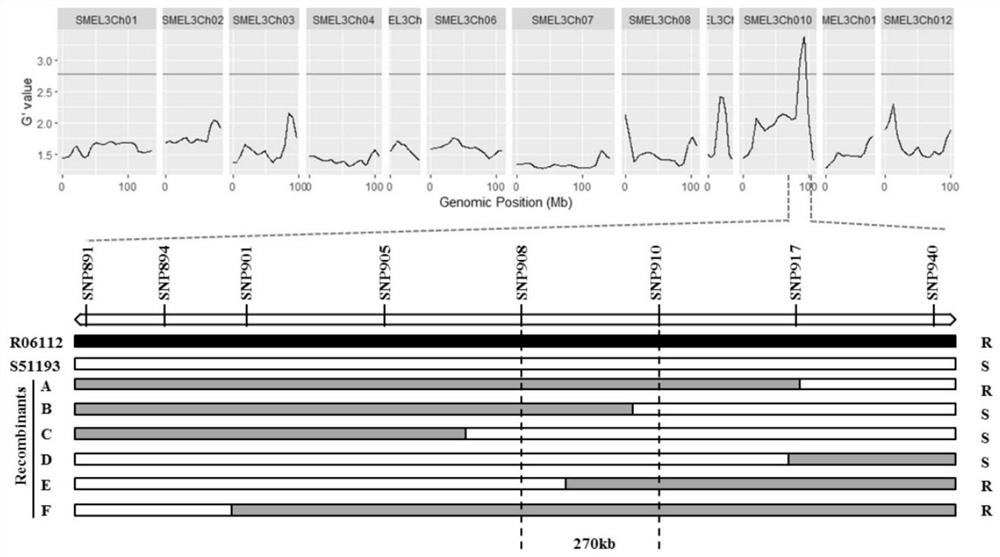
图2为通过F2群体抗青枯病个体混合池和感青枯病个体混合池进行BSA将抗青枯病基因定位于茄子第10染色体;
图3为分子标记SNP910在100个创新材料中的基因分型图,其中绿色为抗病纯化,红色为抗病杂合,蓝色为感病纯合,灰色为空白对照。
具体实施方式
下面对本发明的具体实施方式作进一步说明。在此需要说明的是,对于这些实施方式的说明用于帮助理解本发明,但并不构成对本发明的限定。此外,下面所描述的本发明各个实施方式中所涉及的技术特征只要彼此之间未构成冲突就可以相互组合。
下述实施例中的实验方法,如无特殊说明,均为常规方法,下述实施例中所用的试验材料,如无特殊说明,均为可通过常规的商业途径购买得到。
以下实施例中所采用的常见分子生物学实验技术,比如DNA提取、PCR扩增等,若无特殊说明,通常按照常规方法操作,具体参见《分子克隆实验指南》(第三版)(Sambrook J&Russell DW,Molecular Cloning:a Laboratory Manual,2001),或按照制造厂商说明书建议的条件实施。
实施例1茄子抗青枯病遗传群体构建与遗传分析
(1)试验材料
以广东省农业科学院蔬菜研究所选育保存的具有优良商品性和丰产性的高抗青枯病自交系(R06112)和高感青枯病自交系(S51193)为亲本,杂交获得F1,F1自交获得320株F2分离群体。
(2)茄子抗青枯病遗传分析
对F2群体采用伤根灌根法(菌株采用广东省农业科学院蔬菜研究所保存的演化Ⅰ型GMI1000、SG-2、LZ-3、DG-2青枯菌,操作方法参照文献“李涛,李植良,夏碧波,等.茄子创新资源的耐热及抗青枯病评价研究[J].中国农学通报,2017,33(36):72-77.”)处理5片叶的茄子植株(图1A),于接种后21d按照1-5级分级调查发病情况,并统计发病率(图1B-F),其中接种21d后抗病自交系‘06112’的发病率为3.94%,感病自交系的发病率为96.35%,F1的发病率为43.91%,F2的发病率为29.69%,结合BC1Pr(抗病亲本回交F1所得后代,发病率48.44%)和BC1Ps(感病亲本回交F1所得后代,发病率42.19%)的发病结果,表明以‘06112’为抗源的青枯病抗性由1对主效基因控制。
实施例2茄子抗青枯病基因定位与KASP引物开发
(1)基因初步定位
利用F2群体中的抗病单株和感病单株各30株,提取DNA,构建两个DNA池并进行混池测序(BSA-seq)。筛选出子代与双亲纯合且有差异的SNP位点,并计算出显性池与隐性池的SNP-index值。利用QTLseq-R计算两个混合池的G’value,并以1Mb为窗口、1kb为步长进行滑窗分析,将茄子抗青枯病基因定位于10号染色体上的89.16Mb-94.12Mb区间;根据亲本的序列变异,在区间内开发多态性分子标记,对320个F2单株进行基因分型,结合青枯病发病表型数据进一步缩小定位区间。将茄子抗青枯病基因定位到270kb区间内。根据两个亲本的全基因组重测序数据,查找该定位区间内的SNP变异位点,在91075951bp位置查找到一个SNP位点变异C/A(图2)。
(2)设计KASP引物
根据91075951bp位置SNP位点上下游100bp的序列,利用primer premier5.0软件设计KASP引物。
针对该SNP,设计等位基因特异性引物进行扩增和检测,获得分子标记SNP910。该分子标记的预测扩增片段长度为49个碱基,序列如SEQ NO.1所示,C/A SNP位于第22个碱基;扩增的正向引物为两个分别与C和A结合的特异性引物,序列分别为SEQ NO.2和SEQNO.3,反向引物为SEQ NO.4。在两条特异性正向引物的5’端添加荧光报告基团,其中SEQNO.2的5’端含有HEX基团(对应碱基为C),而SEQ NO.3的5’端含有FAM基团(对应碱基为A),从而可以通过兼容的荧光检测平台进行分型。
SNP910的预测扩增片段(SEQ ID NO.1):
TGAGGAAGGATTTGTGCTCAC[C/A]TGTGTTGCCTATCCAAACTCTCATGTT。
SNP910的正向引物F1(SEQ ID NO.2):
GAAGGTGACCAAGTTCATGCTGAGGAAGGATTTGTGCTCACC。
SNP910的正向引物F2(SEQ ID NO.3):
GAAGGTCGGAGTCAACGGATTTGAGGAAGGATTTGTGCTCACA。
SNP910的反向引物R(SEQ ID NO.4):AACATGAGAGTTTGGATAGGCAAC。
实施例3与茄子抗青枯病性状连锁SNP标记的应用
通过F2群体(320个单株)及100株茄子创新材料(以抗病自交系R06112为父本杂交创新的自交系)验证SNP910标记的应用效果。取F2群体样本叶片,通过CTAB法提取茄子基因组DNA,利用上述引物(SEQ ID NO.2、3、4)进行PCR扩增,PCR反应体系为10μL,其中包含2×KASP master mix 5μL,反向引物0.4μL,正向引物F1为0.15μL,正向引物F2为0.15μL,基因组DNA(10-100ng)1μL,ddH
以上对本发明的实施方式作了详细说明,但本发明不限于所描述的实施方式。对于本领域的技术人员而言,在不脱离本发明原理和精神的情况下,对这些实施方式进行多种变化、修改、替换和变型,仍落入本发明的保护范围内。
- 一种梅花垂枝性状紧密连锁的SNP分子标记及其检测方法与应用
- 与黄瓜抗白粉病紧密连锁的两个SNP标记及其应用
- 与花生抗青枯病主效QTL位点连锁的SNP分子标记及其应用
- 与花生抗青枯病主效QTL位点连锁的SNP分子标记及其应用
