一种利用重组微生物全细胞催化合成L-苯乳酸的方法
文献发布时间:2023-06-19 11:37:30
技术领域
本发明涉及一种利用重组微生物全细胞催化合成L-苯乳酸的方法,属于酶工程和基因工程技术领域。
背景技术
苯乳酸(phenyllactic acid,PLA),即2-羟基-3-苯基丙酸,是一种小分子天然有机酸,广泛存在于干酪、天然蜂蜜中。作为一种天然抑菌物质,对多种病原性微生物都具有光谱抑制作用,但对人和动物细胞均无毒性。其溶解性良好,可均匀溶解于水性溶剂中。此外,苯乳酸具有耐酸、耐碱、耐高温等特性,并能有效的抑制食腐性微生物的生长,在食品工业中具有广阔的应用前景。
L-苯乳酸作为苯乳酸两种对映异构体中的一种,是合成许多药物制剂的前体,此外,作为一种重要化工合成的原材料,可以用于合成非蛋白氨基酸、抗HIV试剂和降血糖剂等。
目前国内外合成L-苯乳酸主要是化学合成法、微生物发酵和生物催化合成。化学合成法技术路线复杂,反应条件苛刻,使用大量有机溶剂易于产生环境污染,副产物多,后续分离困难,不易纯化。微生物发酵法主要指利用筛选或改造的具有特殊性能的菌体,在合适的发酵条件下,通过微生物分解代谢与合成代谢生产出苯乳酸。目前报道以苯丙酮酸为底物合成苯乳酸的文献较多,关于苯丙氨酸一步合成苯乳酸的报道较少,且苯丙酮酸价格偏高,挖掘能够成本低廉并高效转化的生物催化合成苯乳酸方法是未来研究亟待解决的问题。
目前,以苯丙氨酸为底物转化合成苯乳酸,并实现辅因子NAD
全细胞转化相比分离酶具有如下优势:全细胞生物催化剂更容易制备,成本低;比分离酶更稳定,不易受环境温度、pH等因素影响,使用方便;转化过程中不产生有毒害产品,不产生其他副产物。有望实现低能耗、高效率、高纯度、无污染的工业化苯乳酸生产。
因此,提供构建一种操作简便、成本低、转化率高、适合工业化生产的多酶级联催化反应体系,实现苯丙氨酸到L-苯乳酸的一步反应,对于工业上制备L-苯乳酸有重要的应用价值。
发明内容
技术问题:
本发明要解决的技术问题是:提供一种能够转化苯丙氨酸生成L-苯乳酸的基因工程菌,并且构建一种操作简便、成本低、转化率高、适合工业化生产的多酶级联催化反应体系,实现苯丙氨酸到L-苯乳酸的一步反应,以解决工业化苯乳酸生产高成本、底得率、污染严重的问题。
技术方案:
为了解决上述技术问题,本发明提供了一种以苯丙氨酸为底物生产L-苯乳酸的基因工程菌,该基因工程菌共表达L-氨基酸脱氨酶、苯丙酮酸还原酶和葡萄糖脱氢酶。
在本发明的一种实施方式中,以细菌或真菌为宿主细胞。
在本发明的一种实施方式中,以pET-28a为表达载体。
在本发明的一种实施方式中,所述L-氨基酸脱氨酶来源于普通变形杆菌Proteusvulgaris。
在本发明的一种实施方式中,所述苯丙酮酸还原酶来源于乳酸杆菌Lactobacillus sp.CGMCC 9967。
在本发明的一种实施方式中,所述葡萄糖脱氢酶来源于枯草芽孢杆菌Bacillussubtilis。
在本发明的一种实施方式中,所述基因工程菌以大肠杆菌为宿主细胞。
在本发明的一种实施方式中,所述大肠杆菌为E.coli BL21(DE3)。
在本发明的一种实施方式中,所述L-氨基酸脱氨酶的氨基酸序列如SEQ ID NO.1所示。
在本发明的一种实施方式中,所述苯丙酮酸还原酶的氨基酸序列如SEQ ID NO.2所示。
在本发明的一种实施方式中,所述葡萄糖脱氢酶的氨基酸序列如SEQ ID NO.3所示。
在本发明的一种实施方式中,编码所述L-氨基酸脱氨酶的基因如SEQ ID NO.4所示。
在本发明的一种实施方式中,编码所述苯丙酮酸还原酶的基因如SEQ ID NO.5所示。
在本发明的一种实施方式中,编码所述葡萄糖脱氢酶的基因如SEQ ID NO.6所示。
本发明还提供了一种以苯丙氨酸为底物生产L-苯乳酸方法,将上述基因工程菌添加至含有底物苯丙氨酸的反应体系中,反应制备得到L-苯乳酸。
在本发明的一种实施方式中,所述反应体系中还含有辅底物葡萄糖。
在本发明的一种实施方式中,底物苯丙氨酸与辅底物葡萄糖按照摩尔比为(1:1)~(5:1)的比例进行添加。
在本发明的一种实施方式中,将上述基因工程菌按照菌体OD
在本发明的一种实施方式中,将上述基因工程菌按照菌体OD
在本发明的一种实施方式中,所述底物苯丙氨酸的终浓度至少为10g/L。
在本发明的一种实施方式中,所述底物苯丙氨酸的终浓度为10~50g/L。
在本发明的一种实施方式中,以温度为25~45℃,初始pH 6.0~10.0。
在本发明的一种实施方式中,所述方法是以30g/L终浓度的苯丙氨酸为底物,加入OD
在本发明的一种实施方式中,全细胞催化剂是在培养基后培养获得的菌液或离心获得的菌体细胞。
在本发明的一种实施方式中,反应条件为:反应温度30℃,初始pH 8.0,底物苯丙氨酸终浓度30g/L,辅底物葡萄糖按照与底物苯丙氨酸照摩尔比为1:1的比例进行添加,菌体OD
本发明还提供了上述基因工程菌在制备含有L-苯乳酸的产品方面的应用。
有益效果
(1)本发明首次将L-氨基酸脱氨酶和苯丙酮酸还原酶用于催化苯丙氨酸合成L-苯乳酸,成功实现了L-氨基酸脱氨酶,苯丙酮酸还原酶和葡萄糖脱氢酶共表达菌株的构建,采用本发明的技术方案,最终L-苯乳酸的产量达到21.39g/L,摩尔转化率为71.33%。
(2)本发明通过对辅酶再生酶的筛选,制备全细胞催化剂转化合成L-苯乳酸,解决了还原性辅酶NADH价格昂贵,稳定性差的问题。本研究所构建的重组大肠杆菌全细胞催化方法及其相关研究结果,为L-苯乳酸工业化生产及应用奠定了理论和实践基础。
附图说明
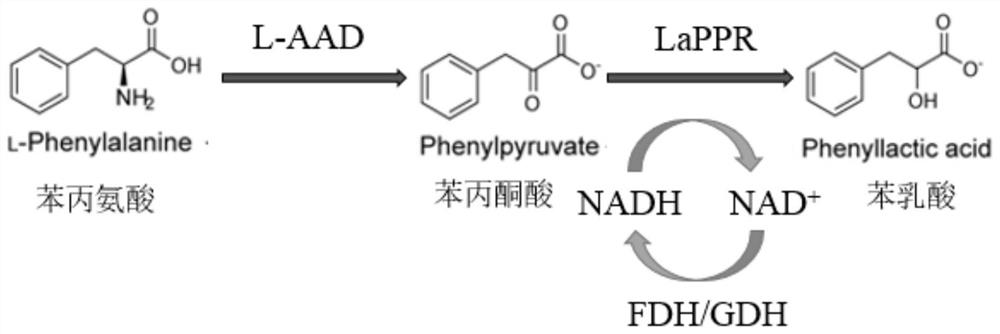
图1:苯丙氨酸合成L-苯乳酸途径示意图。
图2:L-氨基酸脱氨酶、苯丙酮酸还原酶和葡萄糖脱氢酶基因的克隆时电泳图,其中,图中M:10000marker,1-2:葡萄糖脱氢酶基因基因gdh,3-4:苯丙酮酸还原酶基因lappr,5-7:L-氨基酸脱氨酶基因laad。
图3:重组菌株E.coli BL21/pET28a-lappr-gdh-laad产生的粗酶液的SDS-PAGE蛋白电泳图;其中,1:E.coil BL21/pET-28a产生的粗酶液;2-4:E.coil BL21/pET-28a-laad-lappr-gdh产生的粗酶液。
图4:不同的反应条件对对重组菌E.coli BL21/pET28a-laad-lappr-gdh全细胞转化合成L-苯乳酸的产量的影响;其中,A:温度对全细胞转化合成L-苯乳酸的产量的影响,B:温度对全细胞转化合成L-苯乳酸的产量的影响,C:温度对全细胞转化合成L-苯乳酸的产量的影响,D:温度对全细胞转化合成L-苯乳酸的产量的影响,E:温度对全细胞转化合成L-苯乳酸的产量的影响。
具体实施方式
下面结合具体实施例,对本发明进行进一步的阐述。
下述实施例中涉及的大肠杆菌E.coli BL21(DE3)购自北纳生物,pET-28a(+)质粒购自Novagen公司。(上述菌株大肠杆菌E.coli BL21(DE3)可以购买得到,不需要进行用于专利程序的保藏)HindⅢ、NotⅠ、XhoⅠ等限制性核酸内切酶与DNA聚合酶购于TaKaRa公司;小量质粒提取试剂盒、同源重组试剂盒和胶回收试剂盒购于南京诺维赞生物科技有限公司;L-苯丙氨酸、L-苯丙酮酸、L-苯乳酸购于国药集团;酵母提取物和胰蛋白胨购于英国Oxoid公司;所有实验试剂如果无特殊说明均为分析纯。
下述实施例中涉及的培养基及所需溶液如下:
LB液体培养基:蛋白胨10g/L、酵母膏5g/L、NaCl 10g/L。
LB固体培养基:蛋白胨10g/L、酵母膏5g/L、NaCl 10g/L、琼脂粉2%(m/v)。
TB培养基:蛋白胨12g/L、酵母提取物24g/L、甘油4g/L、KH
下述实施例中涉及的检测方法如下:
L-氨基酸脱氨酶酶活的检测方法:L-氨基酸脱氨酶酶活检测方法:将15mL浓度约为30g/L的L-苯丙氨酸溶液30℃预热,分别称取0.5g左右的湿菌体于250mL锥形瓶中,加入14.5mL预热的pH7.5的缓冲液,再加入预热的L-苯丙氨酸转化液中,30℃、200rpm的摇床中反应30min。反应完成后,取适量反应液快速离心稀释进行液相检测。
L-氨基酸脱氨酶酶活定义:1min内转化生成1umol PPA所需的酶量。
L-氨基酸脱氨酶比酶活定义为每毫克蛋白每分钟从苯丙氨酸生产多少微摩尔的苯丙酮酸。
苯丙酮酸还原酶酶活的检测方法:反应体系为200μL,由10μL NADH(10mM),10μL苯丙酮酸(20mM),10μL苯丙酮酸还原酶在170μL PB缓冲液(pH 7.0,100mM)中组成。每隔30s测定340nm吸光度的值并记录数据,根据反应液在340nm吸光值增量,计算出NAD
苯丙酮酸还原酶酶活定义为在上述反应条件下每分钟催化1μmol NADH氧化所需要的酶量。
葡萄糖脱氢酶酶活的检测方法:反应体系由10μL NAD
葡萄糖脱氢酶酶活定义:每分钟产生1μmol NADH所需的酶量为1个单位U。
甲酸脱氢酶酶活的检测方法:反应体系中含有167mM甲酸钠溶液(PB缓冲液,pH7.0,100mM)和1.67mM NAD
甲酸脱氢酶酶活定义:每分钟产生1μmol NADH所需的酶量为1个单位U。
其中,苯丙酮酸还原酶及葡萄糖脱氢酶酶活(U/mL)=吸光度变化值×反应总体系(μL)/(酶液量(μL)×摩尔吸光度值(6.22×10
测定纯化后的苯丙酮酸还原酶及葡萄糖脱氢酶酶活(U/mL),并且,采用Bradford法测定纯化后的苯丙酮酸还原酶及葡萄糖脱氢酶酶活的蛋白含量(mg/mL),以计算苯丙酮酸还原酶及葡萄糖脱氢酶酶的比酶活;
其中,苯丙酮酸还原酶及葡萄糖脱氢酶酶比酶活的计算公式如下:
苯丙酮酸还原酶及葡萄糖脱氢酶酶比酶活(U/mg)=纯化后的苯丙酮酸还原酶及葡萄糖脱氢酶酶的酶活(U/mL)/纯化后的苯丙酮酸还原酶及葡萄糖脱氢酶酶的蛋白含量(mg/mL)。(Bradford法记载于参考文献“Bradford,M.M.1976.A rapid and sensitivemethod for the quantitation of microgram quantities of protein utilizing theprinciple of protein-dyebinding.Anal.Biochem.72:248–254.”中)。
苯乳酸含量的检测方法:HPLC分析:用C18柱反相高效液相色谱测定;洗针:10%甲醇;洗针后进行自动进样,柱温::30℃,进样量:10.0μL,流速:1.0mL/min,流动相:A相为体积分数0.05%三氟乙酸甲醇溶液,B相为体积分数0.05%三氟乙酸溶液,洗脱程序:0-20min为A:B由10%线性变化为100%,20-23min为100%A相,23-25min保持A:B为10%,检测波长:210nm。
下述实施例中所涉及的PCR扩增程序为:预变性95℃,10min,变性95℃,10min,复性58℃,30s,延伸72℃,1min,终延伸10min,保存4℃;PCR扩增得到的目的基因的SDS-PAGE蛋白电泳图如图2所示。
实施例1:苯丙酮酸还原酶表达载体的构建
具体步骤如下:
以乳酸杆菌Lactobacillus sp.CGMCC 9967基因组为模板,扩增得到如SEQ IDNO.5所示的苯丙酮酸还原酶基因lappr,将其与pET-28a(+)表达质粒经限制性内切酶HindⅢ酶切后进行连接,得到重组质粒pET28a-lappr。
构建表达载体所用的引物序列如下:
上游引物(含HindⅢ酶切位点):AATTCGAGCTCCGTCGAC
下游引物(含HindⅢ酶切位点):GGTGCTCGAGTGCGGCCGC
实施例2:葡萄糖脱氢酶表达载体的构建
具体步骤如下:
以枯草芽孢杆菌Bacillus subtilis基因组为模板,扩增得到如SEQ ID NO.6所示的葡萄糖脱氢酶基因,将其与实施例1中构建得到的pET28a-lappr表达质粒经限制性内切酶NcoⅠ酶切后进行连接,得到重组质粒pET28a-lappr-gdh。
构建表达载体所用的引物序列如下:
上游引物(含NcoⅠ酶切位点):ACTTTAAGAAGGAGATATA
下游引物(含NcoⅠ酶切位点):TGATGATGATGATGATGGCTGCTGC
实施例3:L-氨基酸脱氨酶表达载体的构建
具体步骤如下:
以普通变形杆菌Proteus vulgaris基因组为模板,扩增得到如SEQ ID NO.4所示的L-氨基酸脱氨酶基因,将其与实施例2中构建得到的pET28a-lappr-gdh表达质粒经限制性内切酶XhoⅠ酶切后进行连接,得到重组质粒pET28a-lappr-gdh-laad。
构建表达载体所用的引物序列如下:
上游引物(含XhoⅠ酶切位点):AAGCTTGCGGCCGCA
下游引物(含XhoⅠ酶切位点):GGTGGTGGTGGTG
实施例4:E.coli BL21/pET28a-laad-lappr-gdh重组菌体的构建和表达
具体步骤如下:
(1)将实施例3所得重组质粒转化大肠杆菌BL21,获得重组菌E.coli BL21/pET28a-laad-lappr-gdh。
(2)将步骤(1)制备得到的重组菌株E.coli BL21/pET28a-laad-lappr-gdh的菌落分别接种至10mL LB液体培养基中,于温度37℃、120-180rpm的条件下培养8-12h,得到种子液。
(3)将种子液以1%(v/v)的接种量接种至500mL LB液体培养基中,于温度16-37℃、180r/min的条件下培养12h,得到重组菌E.coli BL21/pET28a-laad-lappr-gdh的培养液。
(4)将培养液经细胞破碎仪破碎后于8000-10000rpm、4℃的条件下离心5-10min,得到培养重组菌E.coli BL21/pET28a-laad-lappr-gdh的粗酶液,用于SDS-PAGE分析及酶活测定。
SDS-PAGE分析结果显示(如图3所示),在52.05kDa、37kDa、28.8Kda有明显的蛋白条带,证明各基因成功在大肠杆菌中表达。
经检测,粗酶液中L-氨基酸脱氨酶、苯丙酮酸还原酶、葡萄糖脱氢酶酶活分别为13.56U/mL、12.89U/mL、11.79U/mL。
实施例5:E.coli BL21/pET28a-laad-lappr-gdh全细胞催化剂的制备L-苯乳酸
具体步骤如下(合成过程如图1所示):
(1)制备全细胞催化剂:将实施例4中制备得到的重组菌株E.coli BL21/pET28a-laad-lappr-gdh接种LB液体培养基(卡那霉素50mg/L),在37℃,220r/min进行培养,当培养至OD
(2)制备L-苯乳酸:
向含有20mM PB缓冲液(pH 8.0)的溶液中,添加终浓度为30g/L的苯丙氨酸、OD
结果显示:重组菌株E.coli BL21/pET28a-laad-lappr-gdh全细胞转化苯丙氨酸生产L-苯乳酸的转化率为71.33%,此时的苯乳酸的产量为21.39g/L,时空产量为1.78g/L/h。
实施例6:E.coli BL21/pET28a-laad-lappr-gdh全细胞催化剂的制备苯乳酸反应条件的优化
具体步骤如下:
(1)具体实施方式同实施例5,区别在于调整反应温度,温度范围为25、30、35、40、45℃,结果为温度为30℃时,L-苯乳酸产量达到最高为20.24g/L,转化率为67.49%(如图4A所示)。
(2)具体实施方式同实施例5,区别在于调整反应的初始pH,范围为6.0、7.0、8.0、9.0、10.0,结果为初始pH 8.0时达到最高21.02g/L,转化率为70.09%(如图4B所示)。
(3)具体实施方式同实施例5,区别在于调整反应的菌体量,范围为10、20、30、40、50,结果为菌体OD
(4)具体实施方式同实施例5,区别在于调整反应的底物浓度,范围为10、20、30、40、50g/L,结果为底物浓度为30g/L时,产量达到最大值,21.36g/L,转化率为71.22%(如图4D所示)。
(5)具体实施方式同实施例5,区别在于调整反应的辅底物葡萄糖添加量,范围为与底物相比,0、0.5、1、1.5、2、2.5倍摩尔当量(即:辅底物葡萄糖添加量浓度分别为0g/L、15g/L、30g/L、45g/L、60g/L、75g/L),结果为葡萄糖添加量为1倍摩尔当量(即:辅底物葡萄糖添加量浓度分别为30g/L),苯乳酸产量达到最高,21.39g/L,转化率为71.33%(如图4E所示)。
对比例1
具体实施方式同实施例1~5,区别在于,调整实施例2中的葡萄糖脱氢酶为枯草芽孢杆菌Bacillus subtilis来源,以及甲酸脱氢酶为博伊丁假丝酵母Candida boidinii来源,所涉及的引物序列如下:
上游引物FDH-F(含XbaⅠ酶切位点):ATAACAATTCCCCTCTAGAAAGGAGATGAAGATCGTTTTAGTCTTATACGATTGTGGTAA AC(SEQ ID NO.13);
下游引物FDH-R(含XbaⅠ酶切位点):AAAGTTAAACAAAATTATTTCTAGATTATTTCTTATCGTGCTTACCGTAAGCTTTGG(SEQ ID NO.14)。
结果显示,构建得到了共表达了L-氨基酸脱氨酶、苯丙酮酸还原酶和甲酸脱氢酶的基因工程菌E.coli BL21/pET28a-lappr-fdh-laad,采用该基因工程菌生产制备苯乳酸的产量为11.78g/L。
虽然本发明已以较佳实施例公开如上,但其并非用以限定本发明,任何熟悉此技术的人,在不脱离本发明的精神和范围内,都可做各种的改动与修饰,因此本发明的保护范围应该以权利要求书所界定的为准。
SEQUENCE LISTING
<110> 江南大学
<120> 一种利用重组微生物全细胞催化合成L-苯乳酸的方法
<130> BAA210043A
<160> 14
<170> PatentIn version 3.3
<210> 1
<211> 471
<212> PRT
<213> 人工序列
<400> 1
Met Ala Ile Ser Arg Arg Lys Phe Ile Ile Gly Gly Thr Val Val Ala
1 5 10 15
Val Ala Ala Gly Ala Gly Ile Leu Thr Pro Met Leu Thr Arg Glu Gly
20 25 30
Arg Phe Val Pro Gly Thr Pro Arg His Gly Phe Val Glu Gly Thr Glu
35 40 45
Gly Ala Leu Pro Lys Gln Ala Asp Val Val Val Val Gly Ala Gly Ile
50 55 60
Leu Gly Ile Met Thr Ala Ile Asn Leu Val Glu Arg Gly Leu Ser Val
65 70 75 80
Val Ile Val Glu Lys Gly Asn Ile Ala Gly Glu Gln Ser Ser Arg Phe
85 90 95
Tyr Gly Gln Ala Ile Ser Tyr Lys Met Pro Asp Glu Thr Phe Leu Leu
100 105 110
His His Leu Gly Lys His Arg Trp Arg Glu Met Asn Ala Lys Val Gly
115 120 125
Ile Asp Thr Thr Tyr Arg Thr Gln Gly Arg Val Glu Val Pro Leu Asp
130 135 140
Glu Glu Asp Leu Val Asn Val Arg Lys Trp Ile Asp Glu Arg Ser Lys
145 150 155 160
Asn Val Gly Ser Asp Ile Pro Phe Lys Thr Arg Ile Ile Glu Gly Ala
165 170 175
Glu Leu Asn Gln Arg Leu Arg Gly Ala Thr Thr Asp Trp Lys Ile Ala
180 185 190
Gly Phe Glu Glu Asp Ser Gly Ser Phe Asp Pro Glu Val Ala Thr Phe
195 200 205
Val Met Ala Glu Tyr Ala Lys Lys Met Gly Val Arg Ile Tyr Thr Gln
210 215 220
Cys Ala Ala Arg Gly Leu Glu Thr Gln Ala Gly Val Ile Ser Asp Val
225 230 235 240
Val Thr Glu Lys Gly Ala Ile Lys Thr Ser Gln Val Val Val Ala Gly
245 250 255
Gly Val Trp Ser Arg Leu Phe Met Gln Asn Leu Asn Val Asp Val Pro
260 265 270
Thr Leu Pro Ala Tyr Gln Ser Gln Gln Leu Ile Ser Gly Ser Pro Thr
275 280 285
Ala Pro Gly Gly Asn Val Ala Leu Pro Gly Gly Ile Phe Phe Arg Glu
290 295 300
Gln Ala Asp Gly Thr Tyr Ala Thr Ser Pro Arg Val Ile Val Ala Pro
305 310 315 320
Val Val Lys Glu Ser Phe Thr Tyr Gly Tyr Lys Tyr Leu Pro Leu Leu
325 330 335
Ala Leu Pro Asp Phe Pro Val His Ile Ser Leu Asn Glu Gln Leu Ile
340 345 350
Asn Ser Phe Met Gln Ser Thr His Trp Asn Leu Asp Glu Val Ser Pro
355 360 365
Phe Glu Gln Phe Arg Asn Met Thr Ala Leu Pro Asp Leu Pro Glu Leu
370 375 380
Asn Ala Ser Leu Glu Lys Leu Lys Ala Glu Phe Pro Ala Phe Lys Glu
385 390 395 400
Ser Lys Leu Ile Asp Gln Trp Ser Gly Ala Met Ala Ile Ala Pro Asp
405 410 415
Glu Asn Pro Ile Ile Ser Glu Val Lys Glu Tyr Pro Gly Leu Val Ile
420 425 430
Asn Thr Ala Thr Gly Trp Gly Met Thr Glu Ser Pro Val Ser Ala Glu
435 440 445
Leu Thr Ala Asp Leu Leu Leu Gly Lys Lys Pro Val Leu Asp Pro Lys
450 455 460
Pro Phe Ser Leu Tyr Arg Phe
465 470
<210> 2
<211> 348
<212> PRT
<213> 人工序列
<400> 2
Met Gly Ser Ser His His His His His His Ser Ser Gly Leu Val Pro
1 5 10 15
Arg Gly Ser His Met Ala Ser Met Thr Gly Gly Gln Gln Met Gly Arg
20 25 30
Gly Ser Glu Phe Met Met Lys Ile Leu Asn Ser Phe Ser Leu Lys Pro
35 40 45
Glu Gln Arg Gln Thr Leu Glu Ala Ala Gly His Thr Val Ile Asp Ala
50 55 60
Asp Lys Leu Asp Asp Ala Thr Ala Gln Gln Ile Asp Val Val Tyr Gly
65 70 75 80
Trp Asn Ala Ala Ala Thr Arg Val Asn Phe Asp Arg Leu Gln Phe Val
85 90 95
Gln Ala Met Ser Ala Gly Val Asp Tyr Leu Pro Leu Ala Glu Leu Ala
100 105 110
Lys His His Val Leu Leu Ala Asn Thr Ser Gly Ile His Ala Glu Pro
115 120 125
Ile Ala Glu Tyr Val Leu Gly Ala Leu Phe Thr Ile Ser Arg Gly Ile
130 135 140
Leu Pro Ala Ile Arg Ala Asp Arg Asp Met Trp Thr Leu Arg Gln Glu
145 150 155 160
Arg Pro Pro Met Thr Leu Leu Lys Gly Lys Thr Ala Val Ile Phe Gly
165 170 175
Thr Gly His Ile Gly Ser Thr Ile Ala Thr Lys Leu Gln Ala Leu Gly
180 185 190
Leu His Thr Ile Gly Val Ser Ala His Gly Arg Pro Ala Ala Gly Phe
195 200 205
Asp Gln Val Met Thr Asp Val Ala Thr His Glu Ala Ala Gly Arg Ala
210 215 220
Asp Val Val Ile Asn Ala Leu Pro Leu Thr Pro Asp Thr Lys His Phe
225 230 235 240
Tyr Asp Glu Ala Phe Phe Ala Ala Ala Ser Lys Gln Pro Leu Phe Ile
245 250 255
Asn Ile Gly Arg Gly Pro Ser Val Asp Met Ala Ala Leu Thr Gln Ala
260 265 270
Leu Lys Asn Lys Gln Ile Ser Ala Ala Ala Leu Asp Val Val Asp Pro
275 280 285
Glu Pro Leu Pro Gln Asp Ser Pro Leu Trp Gly Met Thr Asn Val Leu
290 295 300
Leu Thr Pro His Ile Ser Gly Thr Val Pro Gln Leu Arg Asp Lys Val
305 310 315 320
Phe Lys Ile Phe Asn Asp Asn Leu Lys Thr Leu Ile Ser Ser Gly Gln
325 330 335
Leu Ala Ser His Gln Val Asp Leu Thr Arg Gly Tyr
340 345
<210> 3
<211> 262
<212> PRT
<213> 人工序列
<400> 3
Met Gly Tyr Pro Asp Leu Lys Gly Lys Val Val Ala Ile Thr Gly Ala
1 5 10 15
Ser Ser Gly Leu Gly Arg Ala Met Ala Ile Arg Phe Gly Gln Glu Gln
20 25 30
Ala Lys Val Val Ile Asn Tyr Tyr Ser Asn Glu Lys Glu Ala Gln Thr
35 40 45
Val Lys Glu Glu Val Gln Lys Ala Gly Gly Glu Ala Val Ile Ile Gln
50 55 60
Gly Asp Val Thr Lys Glu Glu Asp Val Lys Asn Ile Val Gln Thr Ala
65 70 75 80
Val Lys Glu Phe Gly Thr Leu Asp Ile Met Ile Asn Asn Ala Gly Met
85 90 95
Glu Asn Pro Val Glu Ser His Lys Met Pro Leu Lys Asp Trp Asn Lys
100 105 110
Val Ile Asn Thr Asn Leu Thr Gly Ala Phe Leu Gly Cys Arg Glu Ala
115 120 125
Ile Lys Tyr Tyr Val Glu Asn Asp Ile Gln Gly Asn Val Ile Asn Met
130 135 140
Ser Ser Val His Glu Met Ile Pro Trp Pro Leu Phe Val His Tyr Ala
145 150 155 160
Ala Ser Lys Gly Gly Ile Lys Leu Met Thr Glu Thr Leu Ala Leu Glu
165 170 175
Tyr Ala Pro Lys Arg Ile Arg Val Asn Asn Ile Gly Pro Gly Ala Ile
180 185 190
Asn Thr Pro Ile Asn Ala Glu Lys Phe Ala Asp Pro Val Gln Lys Lys
195 200 205
Asp Val Glu Ser Met Ile Pro Met Gly Tyr Ile Gly Glu Pro Glu Glu
210 215 220
Ile Ala Ala Val Ala Val Trp Leu Ala Ser Lys Glu Ser Ser Tyr Val
225 230 235 240
Thr Gly Ile Thr Leu Phe Ala Asp Gly Gly Met Thr Gln Tyr Pro Ser
245 250 255
Phe Gln Ala Gly Arg Gly
260
<210> 4
<211> 1416
<212> DNA
<213> 人工序列
<400> 4
atggccatta gccgccgcaa attcattatt ggcggcaccg tggtggcagt ggcagccggt 60
gctggtattc tgacaccgat gttaacccgt gagggccgct ttgttccggg tacccctcgt 120
cacggcttcg tggaaggcac cgaaggtgct ttacctaaac aagccgatgt tgttgtggtg 180
ggtgccggta ttttaggtat catgaccgca atcaatttag tggaacgtgg tttaagcgtg 240
gtgattgtgg aaaaaggtaa tattgccggc gaacagagca gtcgcttcta cggccaagct 300
atcagctaca aaatgccgga cgaaaccttt ttactgcatc atttaggtaa acatcgttgg 360
cgtgagatga acgccaaggt gggtattgat accacctacc gtacacaagg tcgtgttgaa 420
gtgccgctgg atgaagagga tctggtgaac gtgcgcaaat ggattgacga gcgcagcaaa 480
aacgtgggca gcgacatccc gttcaagaca cgcatcatcg aaggtgcaga gctgaaccag 540
cgtctgcgtg gtgccaccac cgattggaaa atcgccggtt tcgaagaaga tagcggtagc 600
tttgatccgg aagtggcaac cttcgtgatg gccgagtatg ccaagaagat gggtgtgcgc 660
atctacaccc agtgcgcagc acgcggtctg gaaacccaag ctggtgtgat cagcgatgtg 720
gtgaccgaaa aaggtgccat caaaaccagc caagttgtgg tggctggtgg tgtttggagt 780
cgtttattca tgcagaattt aaatgtggat gtgccgacac tgccggccta tcagagccaa 840
cagctgatta gcggtagccc gacagcaccc ggtggcaatg ttgctttacc gggcggcatt 900
ttctttcgcg aacaagctga cggtacatat gccaccagtc ctcgcgttat cgtggccccg 960
gtggtgaaag agagcttcac atacggctac aagtatttac cgctgctggc tttaccggat 1020
ttcccggttc atatcagtct gaacgagcag ctgattaata gttttatgca gagcacacac 1080
tggaatttag atgaggttag cccgttcgaa cagttccgta acatgaccgc actgcccgat 1140
ctgccggaac tgaatgcctc tttagaaaag ctgaaagccg agtttccggc ctttaaggag 1200
agcaagctga ttgaccagtg gagcggtgca atggccatcg cccccgatga gaacccgatc 1260
atcagcgaag tgaaggagta tccgggttta gtgattaata ccgcaaccgg ctggggtatg 1320
acagaaagtc cggttagtgc cgagctgacc gcagatttac tgctgggcaa gaaaccggtt 1380
ctggacccga agccgttttc tttatatcgt ttttaa 1416
<210> 5
<211> 1047
<212> DNA
<213> 人工序列
<400> 5
atgggcagca gccatcatca tcatcatcac agcagcggcc tggtgccgcg cggcagccat 60
atggctagca tgactggtgg acagcaaatg ggtcgcggat ccgaattcat gatgaaaatt 120
ttaaacagct tttctttaaa accggaacag cgtcagacct tagaagccgc cggccatacc 180
gtgattgacg ccgacaaact ggatgacgcc accgcccagc agatcgatgt tgtgtatggc 240
tggaatgccg ccgcaacccg cgtgaacttc gatcgtctgc agttcgtgca agctatgagc 300
gccggcgtgg attatctgcc gctggcagaa ctggccaaac atcatgtgct gctggccaat 360
accagcggca ttcatgccga gccgattgcc gaatatgtgc tgggcgcttt atttaccatt 420
agccgcggca ttctgccggc aattcgtgcc gaccgcgata tgtggacttt acgtcaagaa 480
cgccctccga tgactttact gaaaggtaaa accgccgtta tcttcggcac cggtcatatt 540
ggcagcacca ttgccaccaa actgcaagca ctgggtctgc ataccattgg cgttagcgca 600
catggccgtc ccgctgctgg ttttgaccaa gttatgaccg atgtggccac acacgaagca 660
gccggtcgtg ccgatgtggt tatcaatgca ctgccgctga ccccggacac caagcacttc 720
tatgacgaag ccttcttcgc cgcagcaagc aaacagccgc tgttcatcaa cattggccgc 780
ggtcctagcg tggatatggc agctttaaca caagctttaa agaataagca gattagcgcc 840
gcagccttag acgtggttga tccggaaccg ctgcctcaag atagtccgct gtggggcatg 900
acaaacgtgc tgctgacccc gcatattagc ggtaccgttc cgcagctgcg cgataaggtg 960
ttcaaaattt ttaatgataa tttaaaaact ttaattagca gcggtcagct ggcaagccat 1020
caagttgatc tgacccgcgg ctattaa 1047
<210> 6
<211> 789
<212> DNA
<213> 人工序列
<400> 6
atgggttatc cggatttaaa aggaaaagtc gtcgccatta caggagcatc atcaggatta 60
ggaagagcga tggcgatccg cttcgggcag gagcaggcga aagtcgtgat taactactac 120
agtaatgaaa aagaggctca aaccgtaaaa gaagaagttc aaaaagcggg cggcgaagcg 180
gtcattattc aaggtgacgt tacaaaagaa gaggatgtca aaaacattgt gcagaccgcg 240
gtcaaggaat tcggcacatt agatatcatg atcaacaacg ccggcatgga aaatccggtc 300
gagtcgcata aaatgccgct aaaagactgg aacaaagtca tcaacaccaa cctgaccggc 360
gcttttctgg gatgccgcga agccattaaa tattacgtag agaatgatat tcaaggaaac 420
gtcattaaca tgtcgagcgt acatgaaatg attccgtggc cgctgtttgt ccactatgcg 480
gcaagtaaag gcggcattaa attaatgacg gaaacattgg cgcttgagta cgcgccgaag 540
cgcatccgtg ttaacaatat cgggccgggc gccatcaata cgccgatcaa tgcggaaaag 600
tttgcggatc ccgttcagaa aaaagatgtg gaaagcatga ttccgatggg gtatatcggt 660
gagccggaag aaatcgcggc tgtcgccgtc tggcttgctt caaaggaatc aagctacgtg 720
accggcatta cgctgtttgc tgacggcgga atgacacaat atccgtcatt ccaggcaggc 780
cgcggttaa 789
<210> 7
<211> 42
<212> DNA
<213> 人工序列
<400> 7
aattcgagct ccgtcgacaa gcttgatggg cagcagccat ca 42
<210> 8
<211> 45
<212> DNA
<213> 人工序列
<400> 8
ggtgctcgag tgcggccgca agcttttaat agccgcgggt cagat 45
<210> 9
<211> 53
<212> DNA
<213> 人工序列
<400> 9
actttaagaa ggagatatac catggatggg ttatccggat ttaaaaggaa aag 53
<210> 10
<211> 42
<212> DNA
<213> 人工序列
<400> 10
tgatgatgat gatgatggct gctgcccatg gttaaccgcg gc 42
<210> 11
<211> 51
<212> DNA
<213> 人工序列
<400> 11
aagcttgcgg ccgcactcga gtaatacgac tcactatagg ggaattgtga g 51
<210> 12
<211> 50
<212> DNA
<213> 人工序列
<400> 12
ggtggtggtg gtgctcgagt taaaaacgat ataaagaaaa cggcttcggg 50
<210> 13
<211> 62
<212> DNA
<213> 人工序列
<400> 13
ataacaattc ccctctagaa aggagatgaa gatcgtttta gtcttatacg attgtggtaa 60
ac 62
<210> 14
<211> 57
<212> DNA
<213> 人工序列
<400> 14
aaagttaaac aaaattattt ctagattatt tcttatcgtg cttaccgtaa gctttgg 57
- 一种利用重组微生物全细胞催化合成L-苯乳酸的方法
- 一种在生物反应器内利用半疏水晶胶基全细胞催化剂合成苯乳酸的方法
