一种诺卡氏菌核酸保守双管家基因、引物和应用
文献发布时间:2024-01-17 01:16:56
技术领域
本发明属于诺卡氏菌检测技术领域,具体涉及一种诺卡氏菌核酸保守双管家基因、用于扩增所述诺卡氏菌核酸保守双管家基因的引物和应用。
背景技术
“生鱼”可指乌鳢,俗称黑鱼。生鱼因个体大、生长快、产量和经济价值较高等特点,在我国南方地区,生鱼一向被视为病后康复和体虚者的滋补珍品。近年,随着国内养殖业的发展和国外贸易需要,生鱼渐成为产销两旺的特种养殖品种,生鱼的养殖技术已引起人们的广泛关注和重视。随着生鱼养殖业的发展,据不完全统计,2021年,全国生鱼养殖总产量约70万吨,广东约占比60%,其中,顺德、中山、江门、清远等珠三角地区养殖量就达40万吨。众所周知,苗种培育是水产动物人工繁殖的关键阶段,对于大多数鱼类而言,仔鱼期被认为是整个养殖周期中最关键的阶段,生鱼苗种培育尤其如此。即使在养殖水平最高的顺德区域,苗种培育成活率平均仅为2成。其中,水体病原增多、疾病爆发等是造成生鱼苗存活率低的主要限制因素之一,严重制约了生鱼苗种繁育产业的发展。
诺卡氏菌病是一种典型危害严重的慢性细菌病,近年来在我国南方如广东、福建和浙江等省有大范围蔓延趋势,危害日益严重,多发于大乌醴、口黑鲈、斑醴等大宗养殖品种。有人认为此病的流行与养殖大环境、养殖密度、投喂饵料、鱼种质量、滥用药物均有很大关系。目前暂无较好药物控制,水产病害防治单位及养殖户应引起重视。早期诊断,及早发现鱼苗期的诺卡氏菌隐性感染,实施早期的卫生管理和药物预防是该病防治的关键。
管家基因,是指所有细胞中均要稳定表达的一类基因,其产物是对维持细胞基本生命活动所必需的。因此,利用管家基因进行菌种鉴定已被广泛应用于生物鉴定领域,并展现出良好的应用前景。目前,在菌种鉴定领域中常见的管家基因为16S rDNA,但16S rRNA序列非常保守,一般仅能在种以上水平区分,种下水平很难区分,即便是亚种也较难区分。因此,利用16S rRNA进行菌种鉴定时,需要结合菌落形态观察和伯杰士手册生理生化特征来进行最终的菌种确定,且常出现分类错误的现象发生,为后续的疾病的合理预防、科学的处理提供了错误的信息参考,错过了最佳的处置时机,导致严重的后果。
发明内容
本发明目的在于提供一种诺卡氏菌核酸保守双管家基因,该双管家基因具有种内和种间双重鉴定的特点。
本发明的目的还在于提供一种用于扩增诺卡氏菌核酸保守双管家基因的引物,该引物具有高度的结合性和特异性,能够与诺卡氏菌核酸管家基因特异性结合,可先后有效区分诺卡氏菌种间和种内的差异,有效缩短中间鉴定步骤,并大大提高了鉴定的准确率。
本发明的目的还在于提供上述引物在制备具有检测诺卡氏菌病效果的药物中的应用。
为实现上述第一目的,本发明采用以下技术方案:一种诺卡氏菌核酸保守双管家基因,所述双管家基因为16S rDNA和topA管家基因,所述topA管家基因包括topA全长基因topAQ和topA核心鉴定片段topAH,所述16S rDNA的核苷酸序列如SEQ ID NO:7所示,所述topA全长基因topAQ的核苷酸序列如SEQ ID NO:8所示,所述topA核心鉴定片段topAH的核苷酸序列如SEQ ID NO:9所示。
为实现上述第二目的,本发明采用以下技术方案:一种用于扩增上述诺卡氏菌核酸保守双管家基因的引物,所述引物包括用于扩增16S rDNA管家基因的16S rDNA引物、用于扩增topA全长基因topAQ的topAQ引物和用于扩增topA核心鉴定片段topAH的topAH引物,其中所述16S rDNA引物、topAQ引物和topAH引物的上、下游序列依次如SEQ ID NO:1~6所示。
具体的:
16S rDNA引物:
16S-F:TCAACGGAGAGTTTGATCCTGG(SEQ ID NO:1所示);
16S-R:AAAGGAGGTGATCCAGCCG(SEQ ID NO:2所示);
topAQ引物:
topAQ-F:GTGATTGTCGAGTCGCCGACGAAGG(SEQ ID NO:3所示);
topAQ-R:TCAGGACTTGGCCGCGGCGGCGGTC(SEQ ID NO:4所示)。
topAH引物:
topAH-F:TGGTGGGCGAACGGCTCGACGGCAT(SEQ ID NO:5所示);
topAH-R:TCAGGACTTGGCCGCGGCGGCGGTC(SEQ ID NO:6所示)。
本发明通过体外筛选技术从16S rDNA、merB、rctB、rpoD、topA和topR基因中筛选获得诺卡氏菌核酸保守双管家基因16S rDNA和topA,并根据诺卡氏菌核酸保守双管家基因16S rDNA和topA设计并筛选获得了上述特异性引物。
本发明中的双管家基因具有种内和种间双重鉴定的特点,特异性引物具有高度的结合性和特异性,能够与诺卡氏菌核酸管家基因特异性结合,具有快速、简单、便捷、准确、快速、低成本等特点。
本发明还提供了一种用于检测诺卡氏菌的试剂盒,含有上述引物。
为实现上述第三目的,本发明采用以下技术方案:上述引物或试剂盒在制备具有检测诺卡氏菌病效果的药物中的应用。
与现有技术相比,本发明具有以下优点:
(1)本发明筛选的两组管家基因可先后有效区分种间和种内的差异,相较于现有方法,无需再利用形态学观察和伯杰士手册生理生化特性鉴定便可直接鉴定到种,有效缩短了中间鉴定步骤,并大大提高了鉴定的准确率;
(2)本发明具有快速、简单、便捷、准确、成本低等特点,在对病原微生物的检测及鉴定中能够发挥出巨大的应用作用,具有巨大的应用潜力。
附图说明
图1是实施例1中获得的可有效区分种内和种间的保守管家基因扩增序列;
图2是实施例1中扩增产物的0.8%的琼脂糖凝胶电泳分析结果;
图3是实施例2中16S rDNA序列比对结果显示;
图4是实施例2中通过topAQ序列比对结果显示;
图5实施例2中通过通过topAH序列比对结果显示。
具体实施方式
下面结合具体实施例详细说明本发明的技术方案,以便本领域技术人员更好理解和实施本发明的技术方案。实施例中所用试剂或材料,如未特别说明,均来源于商业渠道。
实施例1
本实施例提供的用于扩增诺卡氏菌核酸保守双管家基因的引物,通过以下方法获得:
(1)核酸匹配管家基因的筛选,包括如下步骤:
管家基因的筛选遵循以下原则,ANI(average nucleotide identity)分析,即平均核苷酸相似性分析,在全基因组层面进行比较分析,尤其适用于鉴定新的菌株和物种,即获得管家基因扩增序列信息,在管家基因的核酸序列层面上与多株菌的所有基因序列进行相互比较,即进行同源基因序列分析,通过一定的筛选条件如相似度大小和覆盖率等找到core genome genes,在此基础上,选择核心基因,构成基因集,通过种内和中间序列进行比对分析,获得其交集部分即为匹配结果。
管家基因确定之后,设计的引物包括中间的随机序列和两端的引物序列,双管家基因部分序列及组合引物,具有放大种内间差异,调和种间差异的同源率,且相较于传统的鉴定方法具有操作简单、周期短、成本低、易于操作、准确率高等优点。
具体包括:
(1)对发病生鱼鱼苗病患处进行剪取,将组织简单研磨后进行平板涂布,待培养48h后,进行菌落形态观察。
(2)挑取单菌落,接种于含100mL营养肉汤培养基的250mL三角瓶中进行液体培养,培养48h后进行离心机集菌,并利用天根细菌基因组DNA提取试剂盒进行基因组DNA的提取,具体操作步骤参考天根细菌-DNA提取试剂盒说明书,DNA样品保存于-80℃备用。
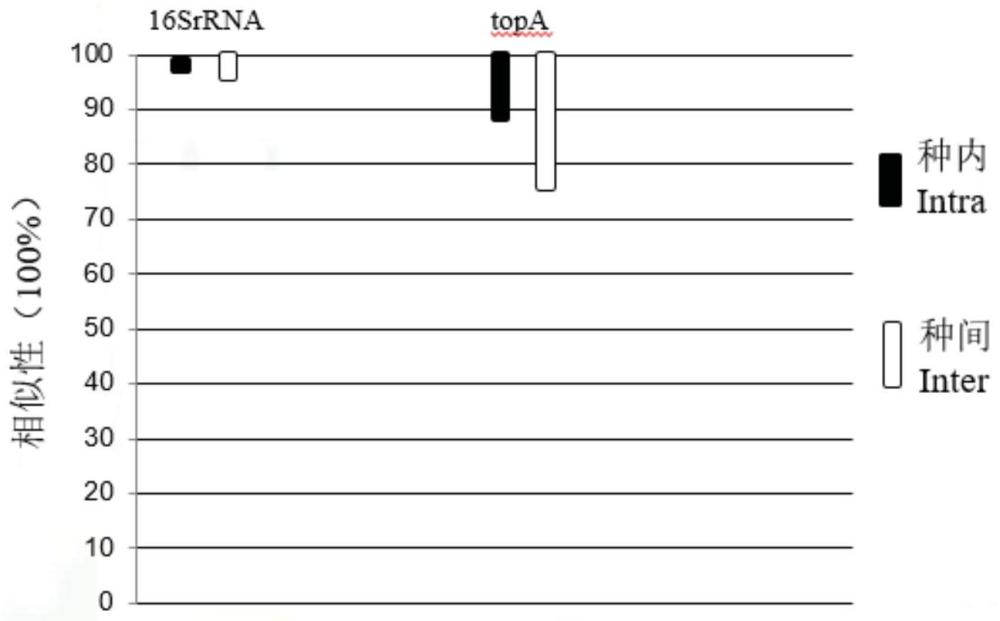
(3)利用NCBI中BLAST软件对GeneBank中的诺卡氏菌的管家基因进行分析,对16SrDNA和topA管家基因扩增序列与Nucleotidecollection(nr/nt)序列进行比对、分析,获得可用于鉴定的管家基因分别为:16S rDNA、merB、rctB、rpoD、topR和topA,并对管家基因进行比对分析。找出在有效交集内的序列可有效区分种内和种间的保守管家基因,即两个管家基因之间种内和种间匹配相似率应分别高于95%和低于90%,其中高于95%的管家可基本确定至属名,然后种内和种间相似率低于90%的管家基因可将种名确定。
筛选原则根据图1进行,即管家基因topA包含16S,且16S管家基因种内和种间匹配率相似率大于95%,topA管家基因匹配率的种内和种间的相似率低于90%,遵循此原则可以先鉴定到属名,再确定到种名。
(4)利用NCBI中BLAST软件对筛选获得16S rDNA(SEQ ID NO:7所示)和topA管家基因序列(SEQ ID NO:8所示)进行比对,通过特异性序列分析,找出差异序列及差异核心序列,以此为扩增核心片段,筛选出最优扩增、鉴定片段。同时,因为PCR快速扩增片段若超过2000bp易出现碱基突变现象,且不易被扩增。而如果低于1000bp则会出现扩增片段较短,后续匹配易出现比对差异等错误结果,故设计扩增鉴定片段大小以1000-2000的易变区作为核心片段进行扩增。通过比对筛选出topA管家基因的核心区域(topAH,SEQ ID NO:9所示),并设计特异性引物,并优化扩增产物片段长度,以便实现PCR扩增反应,最终设计扩增片段长度为1067bp。
针对特异性保守片段和基因序列两端分别设计引物,并确定引物的正确性。
获得的引物如下:
16S rDNA引物:
16S-F:TCAACGGAGAGTTTGATCCTGG(SEQ ID NO:1所示);
16S-R:AAAGGAGGTGATCCAGCCG(SEQ ID NO:2所示);
topAQ引物:
topAQ-F:GTGATTGTCGAGTCGCCGACGAAGG(SEQ ID NO:3所示);
topAQ-R:TCAGGACTTGGCCGCGGCGGCGGTC(SEQ ID NO:4所示)。
topAH引物:
topAH-F:TGGTGGGCGAACGGCTCGACGGCAT(SEQ ID NO:5所示);
topAH-R:TCAGGACTTGGCCGCGGCGGCGGTC(SEQ ID NO:6所示);
扩增序列如下:
16S rDNA管家基因扩增序列(SEQ ID NO:7所示):
TCAACGGAGAGTTTGATCCTGGCTCAGGACGAACGCTGGCGGCGTGCTTAACACATGCAAGTCGAGCGGTAAGGCCCTTCGGGGTACACGAGCGGCGAACGGGTGAGTAACACGTGGGTGATCTGCCTCGCACTCTGGGATAAGCCTGGGAAACTGGGTCTAATACCGGATATGACCACGGATCGCATGGTTTGTGGTGGAAAGATTTATCGGTGCGAGATGGGCCCGCGGCCTATCAGCTTGTTGGTGGGGTAATGGCCTACCAAGGCGACGACGGGTAGCCGACCTGAGAGGGTGACCGGCCACACTGGGACTGAGACACGGCCCAGACTCCTACGGGAGGCAGCAGTGGGGAATATTGCACAATGGGCGAAAGCCTGATGCAGCGACGCCGCGTGAGGGATGACGGCCTTCGGGTTGTAAACCTCTTTCGACAGGGACGAAGCGCAAGTGACGGTACCTGTAGAAGAAGCACCGGCCAACTACGTGCCAGCAGCCGCGGTAATACGTAGGGTGCGAGCGTTGTCCGGAATTACTGGGCGTAAAGAGCTTGTAGGCGGTTCGTCGCGTCGATTGTGAAAACTTGCAGCTCAACTGCAAGCTTGCAGTCGATACGGGCGGACTAGAGTACTTCAGGGGAGACTGGAATTCCTGGTGTAGCGGTGAAATGCGCAGATATCAGGAGGAACACCGGTGGCGAAGGCGGGTCTCTGGGAAGTAACTGACGCTGAGAAGCGAAAGCGTGGGTAGCGAACAGGATTAGATACCCTGGTAGTCCACGCCGTAAACGGTGGGTACTAGGTGTGGGTTTCCTTCCACGGGATCCGTGCCGTAGCTAACGCATTAAGTACCCCGCCTGGGGAGTACGGCCGCAAGGCTAAAACTCAAAGGAATTGACGGGGGCCCGCACAAGCGGCGGAGCATGTGGATTAATTCGATGCAACGCGAAGAACCTTACCTGGGTTTGACATACACCAGAAAGCCGTAGAGATACGGCCCCCCTTGTGGTTGGTGTACAGGTGGTGCATGGCTGTCGTCAGCTCGTGTCGTGAGATGTTGGGTTAAGTCCCGCAACGAGCGCAACCCTTATCTTATGTTGCCAGCGCGTAATGGCGGGGACTCGTGAGAGACTGCCGGGGTCAACTCGGAGGAAGGTGGGGACGACGTCAAGTCATCATGCCCCTTATGTCCAGGGCTTCACACATGCTACAATGGCCGGTACAGAGGGCTGCGATACCGTGAGGTGGAGCGAATCCCTTAAAGCCGGTCTCAGTTCGGATCGGGGTCTGCAACTCGACCCCGTGAAGTTGGAGTCGCTAGTAATCGCAGATCAGCAACGCTGCGGTGAATACGTTCCCGGGCCTTGTACACACCGCCCGTCACGTCATGAAAGTCGGTAACACCCGAAGCCGGTGGCCTAACCCTTGTGGAGGGAGCCGTCGAAGGTGGGATTGGCGATTGGGACGAAGTCGTAACAAGGTAGCCGTACCGGAAGGTGCGGCTGGATCACCTCCTTT 1517;
topA管家基因扩增全长序列(topAQ,SEQ ID NO:8所示):
GTGATTGTCGAGTCGCCGACGAAGGCCAAGAAGATCGCGCCCTACCTGGGCCGCGGCTATGTGGTGGAGGCTTCGGTCGGCCACATTCGGGACCTGCCGCGCGGCGCGGCGGATGTGCCCGCCAAATACAAGGGACAGCAGTGGGCGCGCCTCGGCGTCGACGTCGATCACGATTTCGAGCCCATCTATGTGGTCAGCCCCGAAAAGAAGGGGAAGGTCAGCGAGCTGAAGAGCCTGCTGGCCGACGCCGACGAGCTCTACCTCGCCACCGACCCCGACCGCGAGGGCGAGGCCATCGCCTGGCACCTGCTCGAGACGCTCAAGCCCAAGGTGCCGGTGCGGCGCATGGTCTTCCACGAGATCACCGAGTCGGCCATTCGGGCCGCGGCTGCCGACACCCGGGAGCTGGACCAGGACCTGGTCGACGCGCAGGAGACGCGGCGCATCCTGGACCGGCTCTACGGCTACGAGGTCTCGCCCGTGCTGTGGAAGAAGGTCATGCCGAGGTTGTCGGCGGGCCGGGTGCAGTCGGTGGCGACGCGGGTCATCGTGCAGCGCGAGCGCGAGCGCATGGCGTTCCGGTCGGCGGAGTACTGGGATATCGCGGCCAAGCTGGATGCGGGCGGCAATGAAGAGGGCGGGAATCCGCGGGTCTTCGGTGCGCGGCTGGTCAGTGTCGAGGGTGCGCGCGTGGCGACCGGTCGTGATTTCGGATCCGACGGGCAGCTGAAATCGCAGGGCGTCGTCGTGCTCGATGCCGGTTATGCGCAGCGGCTGGCGGAATCGCTGCACGGGGTGGATCTCACGGTCACCTCGGTGGAATCCAAGCCGTACACGCGAAAGCCGTACGCGCCGTTCATGACTTCGACGCTGCAGCAGGAGGCCGCGCGCAAGCTGCGTTTCACCTCCGAGCGCACCATGCGGGTGGCGCAGCGGCTGTACGAAAACGGTTACATCACCTATATGCGTACCGACTCGACGACCCTGTCGCAGTCGGCGATCAATGCGGCGCGGGCACAGGCCACGCAGCTGTACGGCGCGGAATACGTGAGTCCCACACCGCGGCAGTACAACCGCAAGGTCAAGAACGCGCAGGAGGCGCACGAGGCGATTCGCCCCTCGGGTGACGTCTTCGCCACGCCGGGGCAGCTGCACAACAAGATCGACAATGACGAATTCCGTCTGTACGAGCTGATCTGGCAGCGCACGGTGGCCTCGCAGATGCAGGACGCGCGGGGCACCACGCTGACGGTGCGCATTACCGGTGTCGCGAATAGCGGTGAGGAATGCGTGTTTTCGGCATCGGGCCGGACCATTACCTTCGCGGGCTTCCTCAAGGCGTACGTGGAGAGTGTCGACGAGGAGGCGGGCGGTCAGTCCGACGACGCCGAATCCCGGCTGCCCGCGCTGACCGAGGGCCAGGGCGTCACGGCCGTGGAGCTGGCGCCGGACGGGCACACCACCAATCCGCCCGCGCGTTTCACCGAAGCCTCGCTGATCAAGGCGCTGGAAGACCTCGGCATCGGCCGTCCGTCCACCTACTCCTCGATCATCAAGACCATTCTCGACCGCGGGTACGTCTACAAGCGCGGTAGTGCGCTGGTGCCGTCGTGGGTGGCGTTCGCGGTGATCGGGCTGCTGGAGGAGCACTTCGGGCGGCTGGTGGACTTCGACTTCACCGCGGCCATGGAGGATGATCTCGACGCCATCGCCGGTGGGCGCGAGCAGCGCGGAAACTGGTTGTCCTCCTTCTATTTCGGCGGCGATCACGGTGCGCAGGGATCGGTGGCGCGCTCGGGCGGCCTGAAAAAGATGGTGGGCGAACGGCTCGACGGCATCGACGCGCGAGAAGTCAACTCCATCAAGCTGTTCGCCGACGACGAGGGTCGCGACGTGGTGGTGCGGGTCGGCAAGTTCGGGCCGTACCTGGAGCGCATGATCGCCAATCCCGATGACCCGGACGGGAATCCGGTATCGCAGCGGGCCAACCTGCCCGACGATCTGCCGCCGGACGAGCTGACGCCGGAGGTCGCGGAGAAGCTTTTCGCGACGCCGCAGGAGGGTCGCTCGCTGGGTACCGATCCGGCGACCGGGCACGAAATCGTCGCCAAGGAAGGGCGTTTCGGGCCGTACGTCACCGAGGTGCTGCCGGAGCCGGCGGAGGACATCGACACCGGCGCGAAGAAGACCGCGAAGAAGGCGGCCGACAAGCCGAAGCCGCGCACCGGGTCGCTGTTCAAGTCCATGAGCGTGGAGACGGTGACGCTGGACGAGGCGCTGAAACTGCTCTCGCTGCCGCGCGTGGTGGGTGTGGATCCGGCCTCCGGCGACGAGATCACCGCGCAGAACGGGCGGTACGGGCCGTATCTGAAGAAGGGCACGGACTCCCGCTCGCTCACCGGTGAGGATCAGATCTTCTCGGTGACGCTCGAGGAGGCGCTCAAGATCTATGCCGAGCCCAAACGGCGCGGCGGGCAGTCGGCCAGTGCGCCGCCGCTGCGCGAGCTGGGCGTGGACCCGGTGTCGGAGAAGCCGATGGTGATCAAGGACGGGCGGTTCGGCCCGTATGTCACCGACGGTGAGACCAATGCCAGCCTGCGCAAGGGCGACGAGGTCGAGTCCATCACCGATGCGCGGGCCTCGGAACTGCTGGCGGATCGGCGGGCGCGCGGGCCGGTGAAGAAGGCCGCGAAGAAGACTGCCAAGAAGGCGGCGGCGAAGAAGACCGCGGTTGCCAAGACCGTGGTCAAGAAGGCGGCCGCGAAGCAGGCGGCGGCCAAGACGGCCGACGGCGTGACCACCAAGAAGGCCGCGGCCGCGAAGAAGACCCCGGTCAAGAAGGCGGCCGCGAAGAAGACCGCCGCCGCGGCCAAGTCCTGA2880:
topA管家基因扩增核心序列(topAH,SEQ ID NO:9所示):
TGGTGGGCGAACGGCTCGACGGCATCGACGCGCGAGAAGTCAACTCCATCAAGCTGTTCGCCGACGACGAGGGTCGCGACGTGGTGGTGCGGGTCGGCAAGTTCGGGCCGTACCTGGAGCGCATGATCGCCAATCCCGATGACCCGGACGGGAATCCGGTATCGCAGCGGGCCAACCTGCCCGACGATCTGCCGCCGGACGAGCTGACGCCGGAGGTCGCGGAGAAGCTTTTCGCGACGCCGCAGGAGGGTCGCTCGCTGGGTACCGATCCGGCGACCGGGCACGAAATCGTCGCCAAGGAAGGGCGTTTCGGGCCGTACGTCACCGAGGTGCTGCCGGAGCCGGCGGAGGACATCGACACCGGCGCGAAGAAGACCGCGAAGAAGGCGGCCGACAAGCCGAAGCCGCGCACCGGGTCGCTGTTCAAGTCCATGAGCGTGGAGACGGTGACGCTGGACGAGGCGCTGAAACTGCTCTCGCTGCCGCGCGTGGTGGGTGTGGATCCGGCCTCCGGCGACGAGATCACCGCGCAGAACGGGCGGTACGGGCCGTATCTGAAGAAGGGCACGGACTCCCGCTCGCTCACCGGTGAGGATCAGATCTTCTCGGTGACGCTCGAGGAGGCGCTCAAGATCTATGCCGAGCCCAAACGGCGCGGCGGGCAGTCGGCCAGTGCGCCGCCGCTGCGCGAGCTGGGCGTGGACCCGGTGTCGGAGAAGCCGATGGTGATCAAGGACGGGCGGTTCGGCCCGTATGTCACCGACGGTGAGACCAATGCCAGCCTGCGCAAGGGCGACGAGGTCGAGTCCATCACCGATGCGCGGGCCTCGGAACTGCTGGCGGATCGGCGGGCGCGCGGGCCGGTGAAGAAGGCCGCGAAGAAGACTGCCAAGAAGGCGGCGGCGAAGAAGACCGCGGTTGCCAAGACCGTGGTCAAGAAGGCGGCCGCGAAGCAGGCGGCGGCCAAGACGGCCGACGGCGTGACCACCAAGAAGGCCGCGGCCGCGAAGAAGACCCCGGTCAAGAAGGCGGCCGCGAAGAAGACCGCCGCCGCGGCCAAGTCCTGA1067。
还可以将这些引物设计成试剂盒,便于使用。
实施例2
(1)对珠海市源润水产科技有限公司繁育的发病生鱼鱼苗进行组织剪取,经剪碎后进行营养肉汤固体平板涂布,培养48h:
(2)挑取单菌落接种于含lOOmL营养肉汤培养基的250mL三角瓶中进行液体培养,培养48h:
(3)对培养的液体菌液进行集菌,然后利用天根细菌基因组DNA提取试剂盒进行基因组DNA的提取,具体操作步骤参考天根细菌-DNA提取试剂盒说明书。
(4)以提取的细菌基因组DNA作为模板,通过实施例1中设计的16S和topAH引物对基因组DNA进行扩增。
扩增体系分别采用50μLPCR扩增反应体系:
16S-F:TCAACGGAGAGTTTGATCCTGG;
16S-R:AAAGGAGGTGATCCAGCCG;
topAQ-F:GTGATTGTCGAGTCGCCGACGAAGG;
topAQ-R:TCAGGACTTGGCCGCGGCGGCGGTC;
topAH-F:TGGTGGGCGAACGGCTCGACGGCAT;
topAH-R:TCAGGACTTGGCCGCGGCGGCGGTC(10μmol/L)各5μL,模板DNA 3μL,2×PCRbuffer 25μL,加灭菌双蒸水至50μL。
16S rDNA、topAQ、topAH扩增程序分别为:
1)16SrDNA扩增程序:95℃预变性5min;95℃变性30s,60℃退火30s,72℃延伸100s,30个循环;72℃延伸5min。
2)topAQ扩增程序:95℃预变性5min;95℃变性30s,58℃退火30s,72℃延伸100s,30个循环;72℃延伸5min。
3)topAH扩增程序:95℃预变性5min;95℃变性30s,58℃退火30s,72℃延伸100s,30个循环;72℃延伸5min。
(5)以步骤(2)中提取得到的基因组DNA为模版,利用实施例1步骤(4)中设计的引物对DNA模板进行扩增,将扩增产物进行0.8%的琼脂糖凝胶电泳分析(见图2),后送PCR扩增产物至天一辉远生物科技有限公司进行测序、分析。
(6)将获得的扩增序列先通过NCBI-BLAST-Nucleotide BLAST程序进行16S rDNA比对,筛选出种间关系。然后通过topAH序列比对区别出种内关系。
比对结果见图:
(1)经鉴定后,16S rDNA序列比对结果显示(图3),匹配前4均为Nocardia nova,匹配率均为99.8%以上。但同时,在前100的对比结果中,分别出现Nocardia sp(99.66%),Nocardia aurantia(97.96%),Nocardia vinacea(98.26%),Nocardia niigatensis(98.32%),Nocardia crassostreae(97.88%),Nocardia cyriacigeorgica(97.82%),Nocardia pseudosporangifera(99.8%),Nocardia albiluteola(98.81%),Nocardiaotitidiscaviarum(98.55%),Nocardia aobensis(98.54%),Nocardia macrotermitis(98.54%),Nocardia coffeae(98.79%),Nocardia seFiolae(97.96%),Nocardiacyriacigeorgica(97.96%),Nocardia huaxiensis(98.22%),Nocardia terpenica(97.83%)。通过此步骤可确定该菌株为Nocardia属,已将该菌株范围确定至Nocardia属(诺卡氏菌)内,但是具体的的菌种名还不能确定。
(2)经过(1)确定种属范围后,通过topAQ序列比对结果显示(图4),匹配前5均为Nocardia seriolae,匹配率均为99.97%以上。排第6位的为Nocardia yunnanensis,匹配率为92.43%,与前5的同源性差异较大。故最终确定菌种名为Nocardia seriolae(鰤鱼诺卡氏菌)。
(3)因topAQ扩增产物全长2880bp,而常规PCR扩增超过2000bp易出现碱基突变现象,且不易被扩增。故,通过比对筛选出topA管家基因的核心区域(topAH),并设计特异性引物,扩增片段长度为1067bp。通过topAH序列比对结果显示(图5),匹配前5均为Nocardiaseriolae,匹配率均为99.91%以上。排第6位的为Nocardia yunnanensis,匹配率为92.60%,与前5的同源性差异较大,其结果与扩增全长的匹配率非常相近,结果一致。故最终确定菌种名为Nocardia seriolae(鰤鱼诺卡氏菌),且鉴定仅用16S rDNA和topAH及相对应引物便可进行菌种的快速鉴定。
因此,传统的菌种鉴定方法包含:
(1)16S rDNA测序、比对:确定属名(此过程仅仅能确定至属);
(2)菌落形态观察加染色:此过程能鉴定出革兰氏阳性或阴性,是否产气,是否分泌胞外产物,是否失水,是否规则等,对于菌种鉴定的辅助作用很小;
(3)伯杰士手册生理生化特性鉴定:此步骤,即便是同种细菌的不同菌株之间通常也会有较大差异,且特性的阴性、阳性等,很难界定。传统的菌种鉴定方法需要消耗大量的人力、物力、时间、资金等成本,且不能获得准确的菌种名称,现在的菌种鉴定及最终确定的手段主要还是依赖于基因组的测定、比对。
传统的菌种鉴定方法和基因组测序对于学术研究具有重要的参考意义,但是对于企业或是菌种筛选等,存在重大的弊端。而本发明所阐述的方法可以很好的解决以上的问题,具有快速、简单、便捷、准确、成本低等特点,为菌种的筛选、病原菌的快速鉴定、微生物检测等提供了重要的鉴定手段,具有极大的应用市场及潜力。
以上实施例仅用于阐述本发明,而本发明的保护范围并非仅仅局限于以上实施例。所属技术领域的普通技术人员依据以上本发明公开的内容均可实现本发明的目的,任何基于本发明构思基础上做出的改进和变形,均落入本发明的保护范围之内,具体保护范围以权利要求书记载的为。
