光遗传控制下的遗传编码荧光指示剂及其应用
文献发布时间:2023-06-19 12:07:15
发明领域
本发明涉及与细胞生理学的无伪影全光学表征有关的方法和组合物,其可用于药物筛选、毒性测试和细胞功能评估。
钙离子(Ca
遗传编码的钙离子指示剂(GECIs)的应用已被证明是研究体内和体外细胞钙离子信号时空动态的一个不可或缺的工具。它们已被作为慢性探针用于同时监测从体外分离的神经元到行为动物的大脑活动等环境中的神经元活动。在其他细胞类型,如心肌细胞和胰腺β细胞中对钙瞬变进行成像以研究钙离子介导的信号转导的众多实例也可以在文献中找到。
光遗传学是遗传学和光学方法的结合,其可以对细胞培养物、细胞器、活组织的特定细胞和行为动物进行高时空精度的控制。将光遗传致动器与遗传编码的荧光指示剂结合使用,可以同时操纵和监测细胞生理学、信号转导和神经元活动的动态变化。在这方面,使用具有红移荧光光谱的指示剂应该是与常用的基于视蛋白的致动器(例如光敏感通道蛋白)配对使用的更好选择,因为它们的激发不会干扰这种蓝光激活的光遗传学的激活。此外,该光谱范围内的指示剂具有许多固有的优点,例如对组织散射、光毒性和背景荧光的降低,从而有利于更深入的成像。
然而,众所周知,与优化程度最高的绿色GECIs(即GCaMP6)相比,红色GECIs目前存在许多局限性。这些局限性包括对RCaMP变体的敏感性降低、复杂的光物理性和R-GECO变体的溶酶体积累,尤其是不良蓝光激活光开关行为的遗传特性,该特性也存在于设计其的DsRed衍生模板(mApple)中。这种伪影限制了其与蓝光激活的光遗传致动器结合用于全光刺激和观察的用途。现有的具有更长激发和发射波长的指示器在近红外(NIR)光学窗口(~650nm至900nm)范围内,虽然其由于组织的散射和吸收最少而成为体内成像的理想选择,但其遇到的关键问题是荧光微弱,无法进行合格的成像采集。
提供该背景信息只是为了便于理解本文所描述的发明,而不是承认任何特定技术是现有技术或相关技术。
发明概要
本发明整体涉及遗传编码的Ca
本发明的各方面可包括将红移GECIs与光遗传致动器结合用于无伪影全光学刺激和观察细胞中钙动力学的用途。本发明的实施方案基于光遗传致动器的使用,所述光遗传致动器由波长显著不同于用于激发GECI的光的波长的光激活。
因此,在一个方面,本发明可包括一种用于检查细胞功能的方法,所述方法包括:
(a)在细胞中表达红移遗传编码的Ca
(b)响应刺激,从红移GECI获得光信号。
在另一方面,本发明可包括一种用于检查细胞功能的方法,该方法包括:
(a)在所述细胞中表达由约400nm至约570nm波长范围内的光激活的光学致动器,和红移遗传编码的Ca
(b)响应于由光学致动器暴露于激活光而引起的刺激,从红移GECI获得光信号。
本发明的特征可以在以下阐述有实施例的详细说明中描述,所述实施例中利用了本发明的原理,并且随之附图:
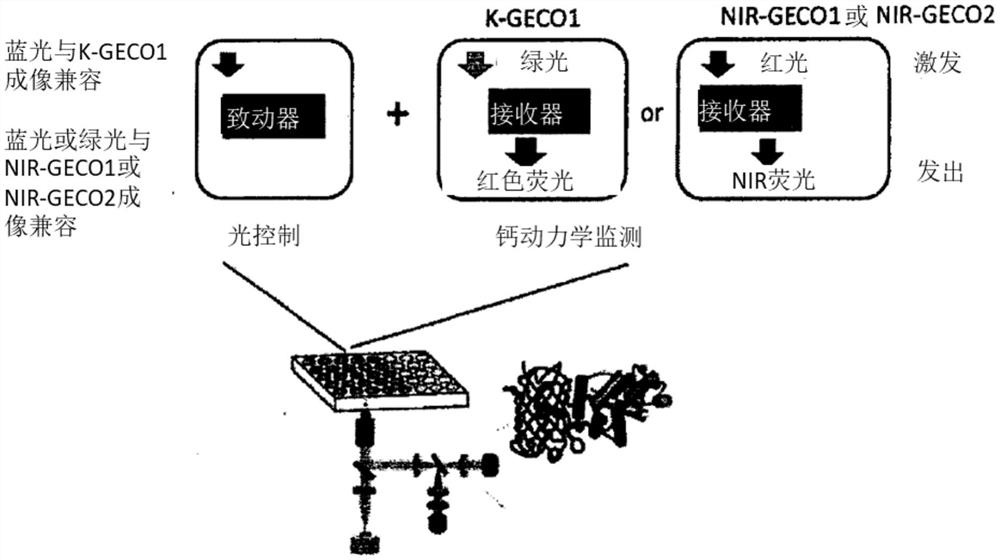
图1显示了光遗传致动器和红移GECI联合用于全光学控制和观察细胞Ca
图2显示了在存在和不存在Ca
图3显示了在人诱导多能干细胞衍生心肌细胞(iPSC CMs)中表达的K-GECO1的性能。(A)用K-GECO1成像的iPSC-CMs中Ca
图4显示了经典神经细胞模型PC12细胞系中Ca
图5显示了NIT-1胰腺β细胞中K-GECO1与ChR2结合的性能。(A)NIT-1胰腺β细胞共表达K-GECO1(红色显示)和ChR2增强的黄色荧光蛋白(EYFP)(绿色显示)。(B)NIT-1胰腺β细胞中联合使用K-GECO1和ChR2进行全光学控制和观察Ca
图6显示了HeLa细胞中K-GECO1与Opto-CRAC致动器联合使用。在蓝光刺激下可以观察到钙瞬变的增加。灰色条表示在脉冲持续时间为1s的CRAC通道上470nm蓝光刺激。
图7.iPSC CMs中表达的NIR-GECO1和NIR-GECO2的光激活效应的检测。(A)用NIR-GECO1成像的iPSC CMs中Ca
图8显示了在HL-1细胞(A)和iPSC CMs(B)中表达的NIR-GECO1的性能,以及在iPSCCMs(C)中表达的NIR-GECO2的性能。受咖啡因(10mm)药理刺激影响的Ca
图9显示了在iPSC CMs中使用NIR-GECO1(A)和NIR-GECO2(B)结合ChR2致动器成像的Ca
图10显示了NIT-1胰腺β细胞中Ca
图11显示了HeLa细胞中被Opto-CRAC激活的NIR-GECO1(A)和NIR-GECO2(B)成像的激发的Ca
说明书中对“一个实施方案”、“实施方案”等的引用,说明所描述的实施方案可以包括特定的方面、特征、结构或特性,但并非每个实施方案都必须包括该方面、特征、结构或特性。此外,这些短语可以(但不一定)指在说明书的其他部分中提及的相同实施方案。此外,当结合实施方案描述特定模块、方面、特征、结构或特征时,无论是否明确描述,在其他实施方案中该模块、方面、特征、结构或特的影响或结合都在本领域技术人员的知识范围内。换言之,任何模块、元件或特征可以在不同的实施方案中与任何其他元件或特征组合,除非存在明显的或固有的不相容或者明确地排除。
本发明提供一种使用表达红移GECI的完全遗传编码系统对Ca
如本文所用,“红移GECI”是指激发波长大于500nm且发射光谱的峰值大于550nm的GECI。“光遗传致动器”是指遗传编码的、光学激活的致动器,其体现了遗传学和光学方法的组合,并且能够在活组织和行为动物的细胞培养物、细胞器、特定细胞中以高空间和时间精度的方式施加光控制(双向控制,即激活或抑制信号通路)。
图1示意性地描绘了本发明的全遗传编码系统的一个实施方案。所述系统包括一个蓝光或绿光激活的光遗传致动器和一个用于全光学控制和记录细胞内Ca
K-GECO1、NIR-GECO1和NIR-GECO2的体外性能
指示剂K-GECO1是基于红色荧光蛋白(RFP)eqFP578组织的GECIs谱系的原型。其具有高灵敏度和快速动力学,类似或优于目前最先进的指标,且其在培养的细胞中具有减弱的溶酶体积累和可忽略的蓝光光激活。K-GECO1是基于RFP eqFP578组织的单体红色荧光指示剂。在Ca
NIR-GECO1是一种近红外指示剂,由天然单体红外荧光蛋白(mIFP)构建而成。NIR-GECO1在678nm和704nm处分别具有吸收峰和发射峰,并且在Ca
本发明的实施方案包含K-GECO1、NIR-GECO1或NIR-GECO2多肽或其组合、以及其实质类似变体的用途。所述多肽和实质类似变体可以由本文公开的多核苷酸序列和实质类似变体进行遗传编码。
K-GECO1、NIR-GECO1和NIR-GECO2的氨基酸序列在下面的实施例7中示出。
无伪影干扰的光遗传致动器精确控制下对细胞Ca
Ca
通常,细胞或细胞器中Ca
用于精确控制Ca
理论上,红移GECIs与用于同时刺激和成像的光遗传致动器最为兼容,因为它们的激发不会干扰常用蓝光激活的光遗传蛋白的激活。然而,目前大多数红色荧光蛋白(RFPs)还存在缺陷,包括灵敏度低、光激活干扰严重和溶酶体积累等。尤其是,蓝光激活的光开关行为阻碍了它们与用于全光学控制和监测钙动力学的光遗传致动器结合的实际应用。关于近红外范围内的遗传编码报告基因,目前可用的近红外GECIs在获得合格成像时存在荧光暗淡的问题。
在一些实施方案中,K-GECO1、NIR-GECO1和/或NIR-GECO2以及A型光遗传学工具的联合使用,能够在不存在或存在外部刺激(例如候选药物)的情况下,对体内或体外兴奋性细胞中Ca
在其它实施方案中,K-GECO1、NIR-GECO1和/或NIR-GECO2以及上述B型光遗传学工具的联合使用,也能够在不存在或存在外部刺激(例如候选药物)的情况下,在体内或体外细胞培养物(不限于仅兴奋性细胞)中提供对Ca
K-GECO1在兴奋性细胞中的性能
在一个方面,K-GECO1可以提供心肌细胞、神经元和胰腺β细胞中胞内Ca
如图3A所示,使用K-GECO1可以实现对iPSC-CMs中自发Ca
当iPSC CMs与K-GECO1和ChR2共转染时,蓝光刺激可靠地诱导Ca
本发明的用于在细胞培养物中全光操纵和可视化Ca
电压依赖性L型钙通道抑制剂,维拉帕米(具有非靶向hERG抑制)和硝苯地平的应用在临床相关浓度下进行了测试,结果如图3E所示。正如预期,两种钙通道抑制剂都能抑制K-GECO1成像所触发的强度变化。
在其它实施方案中,将K-GECO1和ChR2的联合使用应用于经典神经细胞模型,PC12细胞系。
蓝光刺激在表达K-GECO1和ChR2的PC12细胞系中可靠地诱导Ca
至于其他类型的兴奋性细胞,例如,胰腺β细胞也用于本发明中的测试。由葡萄糖触发的电压变化产生的Ca
在本发明的一个实施方案中,NIT-1胰腺β细胞可用作说明K-GECO1/NIR-GECO1与ChR2组合的相容性和性能的模型。在图5A中,在显微镜下可以清楚地看到同时表达K-GECO1(红色显示)和ChR2-EYFP(绿色显示)的NIT-1胰腺β细胞。
如图5B所示,在不同起搏频率下,在10mM葡萄糖存在下,蓝光刺激诱导Ca
可以使用K-GECO1与ChR2结合,对响应于葡萄糖浓度的外部刺激的钙离子动态变化进行成像。如图5C所示,在存在10mM葡萄糖的情况下,如F/F0所示,记录到较大的钙流入。
K-GECO1在非兴奋性细胞中的作用
在表达Opto-CRAC-EYFP(B型光遗传学工具)和K-GECO1的HeLa细胞中记录了持续时间长达2分钟的大量钙瞬变,如图6所示,在蓝光刺激后使用K-GECO1成像。
NIR-GECO1和NIR-GECO2在兴奋性细胞中的作用
如图7A和7B所示,NIR-GECO1和NIR-GECO2分别在iPSC-CMs中表达用于光激活测试。在光照下(用由灰色条指示470nm的0.19W/cm
然后对NIR-GECO1和NIR-GECO2在心血管研究中的性能进行了测试。在本发明的一个方面中,使用源自小鼠心房心肌细胞的稳定永生化细胞系(称为HL-1细胞系)的细胞培养物作为模型。
参考图8,检测HL-1(A)和iPSC CMs(B)中表达的NIR-GECO1和iPSC CMs(C)中表达的NIR-GECO2的药理刺激作用。添加10mM咖啡因后,荧光强度显著降低,表明使用NIR-GECO1或NIR-GECO2成像的HL-1和iPSC CMs的细胞溶质Ca
参考图9,当NIR-GECO1(A)/NIR-GECO2(B)和ChR2在iPSC CMs中共表达时,可以通过蓝光刺激来控制Ca
参考图10,对NIR-GECO1结合ChR2在NIT-1胰腺β细胞中的性能也进行了评估。蓝光刺激的应用可靠地诱导了Ca
NIR-GECO1和NIR-GECO2在非兴奋性细胞中的作用
参考图11,对于表达Opto-CRAC EYFP(一种B型光遗传学工具)和NIR-GECO1(a)和NIR-GECO2(B)的HeLa细胞,在470nm LED光以1s的脉冲持续时间刺激CRAC通道后,使用NIR-GECO1/NIR-GECO2成像观察到大量钙流入。灰色条表示蓝光刺激。
实施例
现在参考以下实施例描述本发明的实施方案。这些实施例仅供说明之用。
实施例1:用于哺乳动物细胞成像和病毒产生的质粒
通过2A肽与K-GECO1连接的ChR2-EYFP被扩增并亚克隆到pshuttle骨架中,用于商业腺病毒生产(Vector Biolabs,Malvern,PA,US)。实施例7中提供了所有序列。
实施例2:体外表征
为了纯化K-GECO变体以进行体外表征,编码该变体的pBAD/hisb质粒用于转化电转感受态E.coli DH10B细胞,然后接种在含有氨苄青霉素(400μg/mL)的LB琼脂平板上。取单个菌落接种于添加了100g/mL氨苄青霉素的5ml LB培养基中。细菌传代培养物在37℃培养过夜。然后,将5ml的细菌传代培养物添加到500ml含有100μg/mL氨苄青霉素的LB培养基中。培养物在37℃下培养直到OD值为0.6。添加L-阿拉伯糖至其最终浓度为0.02%(wt/vol)诱导,然后将培养物在20℃下培养过夜。在4000g下离心10分钟收菌,然后用30mM Tris-HCl缓冲液(pH 7.4)重悬,使用弗氏压碎器(French press)裂解,然后在13,000g下离心30分钟使其澄清。通过Ni-NTA亲和层析(MCLAB)从无细胞提取物中纯化蛋白质。
将纯化蛋白的缓冲液交换成10mM MOPS,100mM KCl,pH 7.2。吸收光谱记录在DU-800紫外可见分光光度计(Beckman)上,荧光光谱记录在Safire2荧光平板阅读器(Tecan)上。为了确定量子产率,使用荧光蛋白mCherry作为标准品。详细的操作说明已经描述在Campbell et al.An expanded palette of genetically encoded Ca
对于Ca
为了纯化NIR-GECO变体以进行体外表征,从pBAD载体表达编码在C末端带有多组氨酸标签的目标变体的基因。用细胞破碎器(Constant Systems Ltd)裂解细菌,然后以15000g离心30分钟,然后通过Ni-NTA亲和层析(Agarose Bead Technologies)纯化蛋白。通常用离心浓缩器(GE Healthcare Life Sciences)将缓冲液交换为10mM MOPS,100mM KCl(pH 7.2)。
通过比较678nm处的吸光度值和391nm处的吸光度值并假设消光系数为39900M
使用EGTA缓冲Ca
实施例3A:HL-1细胞系的细胞培养条件
为了培养HL-1细胞系,将烧瓶用明胶/纤连蛋白预包被在37℃下过夜。细胞在补充Claycomb培养基(含有10%胎牛血清(Sigma-Aldrich 12103C(批号8A0177)、1U/ml青霉素/链霉素、0.1mM去甲肾上腺素和2mM L-谷氨酰胺的Claycomb培养基)中培养,并在达到融合时按1:3分离。
实施例3B:人iPSC衍生心肌细胞的细胞培养条件
人iPSC衍生心肌细胞(人iPSC心肌细胞-男性|ax2505)购自Axol Bioscience。将细胞置于6孔板的两个孔中并在Axol心肌细胞维持培养基(Axol's CardiomyocyteMaintenance Medium)中培养8天,达到80-90%的融合率。然后将细胞重新置于纤连蛋白/明胶(0.5%/0.1%)包被的玻璃底皿上,用Tyrode缓冲液进行最终观察。
实施例3C:NIT-1胰腺β-细胞的细胞培养条件
NIT-1胰腺β-细胞在含2mM L-谷氨酰胺和2.5g/L碳酸氢钠的Ham F12K培养基中培养。pH值为7.2。每次更换培养基时,在Ham F12K培养基中加入10%热灭活FBS和青霉素/链霉素(全部为Invitrogen)。细胞接种于35mm的玻璃底皿上。
实施例3D:PC12细胞的细胞培养条件
PC12细胞于自制的35mm玻璃底培养皿中,在含有10%胎牛血清(Invitrogen)和10%马血清(BioWest)的Dulbecco改良Eagle培养基(Sigma–Aldrich)中培养。
实施例3E:HeLa细胞的细胞培养条件
HeLa细胞于自制的35mm玻璃底培养皿中,在含有10%胎牛血清(Invitrogen)的Dulbecco改良Eagle培养基(Sigma–Aldrich)中培养。
实施例4:转染和转导
用Lipofetamine 2000(Invitrogen)转染试剂将CMV-R-GECO1、pcDNA3.1-K-GECO1或pcDNA3.1-NIR-GECO1或pcDNA3.1-NIR-GECO2和pcDNA3.1-hChR2-EYFP转染至HL-1细胞、iPSC-CMs和NIT-12胰腺β细胞(图3A-D、图5、7-10)。对于PC12细胞(图4)和iPSC CMs(图3E),在成像前2天,在MOI为5的情况下进行24小时的病毒转染。HeLa细胞用Lipofetamine 2000(Invitrogen)转染试剂与Opto-CRAC EYFP和pcDNA3.1-K-GECO1或pcDNA3.1-NIR-GECO1或pcDNA3.1-NIR-GECO2共转染(图6和11)。
实施例5:使用K-GECO1和R-GECO1的光学控制和钙成像
使用配有63倍物镜(NA 1.4,Zeiss)和多波长LED光源(pE-4000,CoolLED)的倒置显微镜(D1,Zeiss)。蓝色(470nm)和绿色(550nm)激发分别用于照亮ChR2-EYFP或Opto-CRAC-EYFP,以及K-GECO1或R-GECO1。使用GFP滤光片组(BP 470-490、T495lpxr二向色镜和HQ525/50发射滤光片)和RFP滤光片组(HQ 545/30x、Q565lp二向色镜和HQ620/60发射滤光片)确认ChR2 EYFP和K-GECO1或R-GECO1在iPSC CMs中的表达。所述滤光片组(Q565lp二向色镜和HQ620/60发射滤光片)用于刺激ChR2并获取K-GECO1或R-GECO1的荧光成像。采用功率密度为1.9mW/mm
实施例6:使用NIR-GECO1和NIR-GECO2的光学控制和钙成像
使用配有63倍物镜(NA 1.4,Zeiss)和多波长LED光源(pE-4000,CoolLED)的倒置显微镜(D1,Zeiss)。蓝色(470nm)和红色(635nm)激发分别用于照亮ChR2-EYFP或Opto-CRAC-EYFP,以及NIR-GECO1。使用GFP滤光片组(BP 470-490、T495lpxr二向色镜和HQ525/50发射滤光片)和NIR滤光片组(ET 650/45x、T685lpxr二向色镜和ET720/60发射滤光片)确认ChR2 EYFP和NIR-GECO1在iPSC CMs中的表达。所述滤光片组(T685lpxr二向色镜和ET720/60发射滤光片)用于刺激ChR2并获取NIR-GECO1的荧光成像。采用功率密度为1.9mW/mm
实施例7:序列
编码K-GECO1的核苷酸序列(SEQ ID NO:1)
ATGGGCAGCGTGAAGCTGATCCCCAGCCTGACCACCGTGATCCTCGTGAAGTCCATGCTGCGGAAGCGGAGCTTCGGCAACCCCTTCAAGTATAATACGGAGACCCTGTACCCCGCTGACGGCGGCCTGGAAGGCGCATGTGACATGGCCCTGAAGCTCGTGGGCGGGGGCCACCTGAACTGCAGCCTTGAGACCACATACAGATCCAAGAAACCCGCTACGAACCTCAAGATGCCCGGTGTCTACAACGTGGACCACAGACTGGAACGAATCAAAGAGGCCGACGATGAGACCTACGTCGAGCTGCACGAGGTGGCTGTGGCCAGATACGTGGGCCTGGGTGGTGGCGGAGGTACAGGCGGGAGTGTGAGCGAGCTGATTAAGGAGAACATGCCAATGAAGCTGTACATGGAGGGCACCGTGAACAACCACCACTTCAAGTGCACATCCGAGGGCGAAGGCAAGCCCTACGAGGGCACCCAGACCATGAGAATCAAGGTCGTCGAGGGCGGCCCTCTCCCCTTCGCCTTCGACATCCTGGCTACCAGCTTCATGTACGGCAGCAGAACCTTCATCAAGCACCCTCCTGGCATCCCCGACTTCTTTAAGCAGTCCTTCCCTGAGGGCTTCACATGGGAGAGAGTCACCACATACGAAGACGGGGGCGTGCTGACCGCTACCCAGGACACCAGCCTCCAGGACGGCTGCCTCATCTACAACGTCAAGGTTAGAGGGATGAACTTCCCAGCCAACGGCCCTGTGATGCAGAAGAAAACACTCGGCTGGGAGGCCAGTAATGGCCAACTGACTGAAGAGCAGATCGCAGAATTTAAAGAGGCTTTCTCCCTATTTGACAAGGACGGGGATGGGACGATAACAACCAAGGAGCTGGGGACGGTGATGCGGTCTCTGGGGCAGAACCCCACAGAAGCGGAGCTGCAGGACATGATCAATGAAGTAGATGCCGACGGTGACGGCACATTCGACTTCCCTGAGTTCCTGACGATGATGGCAAGAAAAATGAGTTATAGAGTCACTGAAGAGGAAATTAGAGAAGCGTTCCGCGTGTTTGATAAGGACGGCAATGGCTACATCGGCGCAGCAGAGCTTCGCCACGTGATGACAGACCTTGGAGAGAAGTTAACAGATGAGGAGGTTGATGAAATGATCAGGGTAGCAGACATCGATGGGGATGGTCAGGTAAACTACGAAGAGTTTGTACAAATGATGACAGCGAAGTAG
编码ChR2-EYFP-2AP-K-GECO1的核苷酸序列(SEQ ID NO:2)
(灰色高亮部分为K-GECO1)
编码NIR-GECO1的核苷酸序列(SEQ ID NO:3)
ATGTCGGTACCGCTGACTACCTCAGCATTCGGCCACGCGTTTCTGGCTAACTGTGAACGTGAGCAGATCCACCTGGCGGGCTCCATTCAGCCGCACGGTATCCTGCTGGCTGTTAAAGAGCCTGACAACGTGGTGATCCAGGCTTCTATTAACGCTGCGGAGTTCCTGAACACCAACTTTGTTGTTGGCCGTCCGCTGCGTGACCTGGGCGGCGATCTGCCTTTGCAGATCCTGCCGCACCTGAACGGCCCGCTGCACCTGGCTCCGATGACCCTGCGTTGTACTGTGGGTTCTCCGCCGCGTCGTGTGGACTGTACCATTCATCGTCCGTCTAACGGCGGCCTGATCGTAGAACTGGAACCAGCAACCAAGGCCACTAACATTGCGCCGGCTCTGGTCGGTGCGCTTCATCGTATCACTTCTTCATCCTCCCTGATGGGCCTGTGTGACGAAACCGCGACTATTTTCCGTGAGATTACTGGCTTCGACCGTGTGATGGTAATGCGTCTCGGCGCGCTTGACGATCTGACTGAAGAGCAGATCGCAGAGATTAAAGAGGCTTTCTCCCTATTTGACAAGGACGGGGACGGGACGATAACAACCAAGGAGCTGGGGACGGTGTTCCGGTCTCTGGGGCAGAACCCCACAGAAGCAGAGCTGCAGGACATGATCAATGAAGTAGATGCCGATGGCGACGGCACATTCGACTTCCCTGAGTTCCTGACGATGATGGCAAGGAAAATGAATGACTCAGACAGTGAAGAGGAAATTAGAGAAGCGTTCCGCGTGTTTGATAAGGACGGCAATGGCTACATCGGCGCAGCAGAGCTTCGCCACGTGATGACAGACCTTGGTGAGAAGTTAACTGATGAGGAGGTTGATGAAATGATCAGGGTAGCAGACAACGATGGGGATGGTCAGGTAAACTACGAAGAGTTTGTACAAATGATGACAGCGAAGGGTGGCGGAGGTTCTGTAGATTCATCACGTCGTAAGTGGAATAAGGCAGGTCACGCAGTCAGAGCTATAGGTCGGCTGAGCTCGCGTAGGCATGATTTGCTGTCCGAATGTCGTCGTGCGGACCTGGAAGCGTTCCTGGGTAACCGCTACCCGGCGTCTACTATTCCGCAGATCGCTCGTCGCCTGTACGAACTTAACCGTGTTCGCCTGCTGGTAGATGTGAACTATACTCCGGTTCCGCTACAGCCGCGCATCAGCCCGCTGAACGGTCGTGATCTGGATATGTCCCTGTCTTGCCTGCGCTCTATGTCCCCGATCCACCAGAAATACATGCAGGACATGGGCGTTGGCGCAACCCTGGTTTGCTCTCTGATGGTGTCTGGTCGTCTGTGGGGTCTGATCGTTTGCCACCACTACGAACCGCGCTTCGTTCCGTCCCACATTCGCGCTGCTGGCGAAGCGCTGGCGGAAACTTGTGCGAACCGCATCGCGACGCTGGAGAGCTTTGCACAGTCTCAGTCCAAATGA
编码NIR-GECO2的核苷酸序列(SEQ ID NO:4)
ATGTCGGTACCGCTGACTACCTCAGCATTCGGCCACGCGTTTCTGGCTAACTGTGAACGTGAGCAGATCCACCTGGCGGGCTCCATTCAGCCGCACGGTATCCTGCTGGCTGTTAAAGAGCCTGACAACGTGGTGATCCAGGCTTCTATTAACGCTGCGGAGTTCCTGAACACCAACTTTGTTGTTGGCCGTCCGCTGCGTGACCTGGGCGGCGATCTGCCTTTGCAGATCCTGCCGCACCTGAACGGCCCGCTGCACCTGGCTCCGATGACCCTGCGTTGTACTGTGGGTTCTCCGCCGCGTCGTGTGGACTGTACCATTCATCGACCGTCTAACGGCGGCCTGATCGTAGAACTGGAACCAGCAACCAAGGCCACTAACATTGCGCCGGCTCTGGTCGGTGCGCTTCATCGTATCACTTCTTCATCCTCCCTGATGGGCCTGTGTGACGAAACCGCGACTATTTTCCGTGAGATTACTGGTTTCGACCGTGTGATGGTAATGCGTCTCGGCGCGCTTGACGATCTGACTGAAGAGCAGATCGCAGAGATTAAAGAGGCTTTCTCCCTATTTGACAAGGACGGGGACGGGACGATAACAACCAAGGAGCTGGGGACGGTGTTCCGGTCTCTGGGGCAGAACCCCACAGAAGCAGAGCTGCAGGACATGATCAATGAAGTAGATGCCGATGGCGACGGCATATTCGACTTCCCTGAGTTCCTGACGATGATGGCAAGGAAAATGAATGACTCAGACAGTGAAGAGGAAATTAGAGGAGCGTTCCGCGTGTTTGATAAGGACGGCAATGGCTACATCGGCGCAGCAGAGCTTCGCCACGTGATGACAGACCTTGGTGAGAAGTTAACTGATGAGGAGGTTGATGAAATGATCAGGGTAGCAGACAACGATGGGGATGGTCAGGTAAACTACGAAGAGTTTGTACAAATGATGACAGCGAAGGGTGGCGGAGGTTCTGTAGATTCATCACGTCGTAAGTGGAATAAGGCAGGTCACGCAGTCAGAGCTATAGGTCGGCTGGGCTCGCGTAGGCATGATTTGCTGTCCGAATGTCGTCGTGCGGACCTGGAAGCGTTCCTAGGTAACCGCTACCCGGCGTCTACTATTCCGCAGATCGCTCGTCGCCTGTACGAACTCAACCGTGTTCGCCTGCTGGTAGATGTGAACTATACTCCGGTTCCGCTAGAGCCGCGCATCAGCCCGCTGAACGGTCGTGATCTGGATATGTCCCTGTCTTGCCTGCGCTCTATGTCCCCGATCCACCAGAAATACATGCAGGACATGGGCGTTGGCGCAACCCTGGTTTGCTCTCTGATGGTGTCTGGTCGTCTGTGGGGTCTGATCGTTTGCCACCACTACGAACCGCGCTACGTTCCGTCCCACATTCGCGCTGCTGGCGAAGCGCTGGCGGAAGCATGTGCGAACCGCATCGCGACGCTGGAGAGCTTTGCACAGTCTCAGTCCAAA
K-GECO1的氨基酸序列(SEQ ID NO:5)
MGSVKLIPSLTTVILVKSMLRKRSFGNPFKYNTETLYPADGGLEGACDMA LKLVGGGHLNCSLETTYRSKKPATNLKMPGVYNVDHRLERIKEADDETYVELHEVAVARYVGLGGGGGTGGSVSELIKENMPMKLYMEGTVNNHHFKCTSEGEGKPYEGTQTMRIKVVEGGPLPFAFDILATSFMYGSRTFIKHPPGIPDFFKQSFPEGFTWERVTTYEDGGVLTATQDTSLQDGCLIYNVKVRGMNFPA NGPVMQKKTLGWEASNGQLTEEQIAEFKEAFSLFDKDGDGTITTKELGTVMRSLGQNPTEAELQDMINEVDADGDGTFDFPEFLTMMARKMSYRVTEEEIREAFRVFDKDGNGYIGAAELRHVMTDLGEKLTDEEVDEMIRVADIDGDGQVNYEEFVQMMTAK
NIR-GECO1的氨基酸序列(SEQ ID NO:6)
MSVPLTTSAFGHAFLANCEREQIHLAGSIQPHGILLAVKEPDNVVIQASINAAEFLNTNFVVGRPLRDLGGDLPLQILPHLNGPLHLAPMTLRCTVGSPPRRVDCTIHRPSNGGLIVELEPATKATNIAPALVGALHRITSSSSLMGLCDETATIFREITGFDRVMVMRLGALDDLTEEQIAEIKEAFSLFDKDGDGTITTKELGTVFRSLGQNPTEAELQDMINEVDADGDGTFDFPEFLTMMARKMNDSDSEEEIREAFRVFDKDGNGYIGAAELRHVMTDLGEKLTDEEVDEMIRVADNDGDGQVNYEEFVQMMTAKGGGGSVDSSRRKWNKAGHAVRAIGRLSSRRHDLLSECRRADLEAFLGNRYPASTIPQIARRLYELNRVRLLVDVNYTPVPLQPRISPLNGRDLDMSLSCLRSMSPIHQKYMQDMGVGATLVCSLMVSGRLWGLIVCHHYEPRFVPSHIRAAGEALAETCANRIATLESFAQSQSK
NIR-GECO2的氨基酸序列(SEQ ID NO:7)
MSVPLTTSAFGHAFLANCEREQIHLAGSIQPHGILLAVKEPDNVVIQASINAAEFLNTNFVVGRPLRDLGGDLPLQILPHLNGPLHLAPMTLRCTVGSPPRRVDCTIHRPSNGGLIVELEPATKATNIAPALVGALHRITSSSSLMGLCDETATIFREITGFDRVMVMRLGALDDLTEEQIAEIKEAFSLFDKDGDGTITTKELGTVFRSLGQNPTEAELQDMINEVDADGDGIFDFPEFLTMMARKMNDSDSEEEIRGAFRVFDKDGNGYIGAAELRHVMTDLGEKLTDEEVDEMIRVADNDGDGQVNYEEFVQMMTAKGGGGSVDSSRRKWNKAGHAVRAIGRLGSRRHDLLSECRRADLEAFLGNRYPASTIPQIARRLYELNRVRLLVDVNYTPVPLEPRISPLNGRDLDMSLSCLRSMSPIHQKYMQDMGVGATLVCSLMVSGRLWGLIVCHHYEPRYVPSHIRAAGEALAEACANRIATLESFAQSQSK
本文使用的标准重组DNA和分子克隆技术为本领域公知,并且在Sambrook,J.,Fritsch,E.F.and Maniatis,T.Molecular Cloning:A Laboratory Manual;Cold SpringHarbor Laboratory:Cold Spring Harbor,N.Y.(1989)中有更全面的描述。转化方法为本领域技术人员公知,并在下文有所描述。
如本文所用,术语“表达”是指功能性终产物(例如,mRNA或蛋白质[前体或成熟])的产生。
如本文所用,“核酸”是指多核苷酸以及包括脱氧核糖核苷酸或核糖核苷酸碱基的单链或双链聚合物。核酸也可以包括片段和修饰的核苷酸。因此,术语“多核苷酸”、“核酸序列”、“核苷酸序列”或“核酸片段”可互换使用,并且为单链或双链的RNA或DNA的聚合物,任选地包含合成的、非天然的或经改变的核苷酸碱基。核苷酸(通常以其5’-单磷酸形式存在)通过其单字母名称表示如下:“A”表示腺苷酸或脱氧腺苷酸(分别表示RNA或DNA),“C”表示胞苷酸或脱氧胞苷酸,“G”表示鸟苷酸或脱氧鸟苷酸,“U”表示尿苷酸,“T”表示脱氧胸苷酸,“R”表示嘌呤(A或G),“Y”表示嘧啶(C或T),“K”表示G或T,“H”表示A或C或T,“I”表示肌苷,“N”表示任何核苷酸。
术语“功能等效的亚片段”和“功能等同的子片段”在本文中互换使用。这些术语指分离的核酸片段的一部分或子序列,其中无论该片段或亚片段是否编码活性蛋白,都保留了改变基因表达或产生特定表型的能力。例如,所述片段或亚片段可用于设计嵌合基因,以在转化的细胞中产生所需表型。嵌合基因可被设计通过以相对于启动子序列的正义或反义方向连接核酸片段或其亚片段来用于抑制,无论其是否编码活性酶。
术语“实质相似”是指其中一个或多个核苷酸碱基的变化不影响核酸介导基因表达或产生特定表型的能力的核酸。这些术语还指本发明的核酸的修饰,例如一个或多个核苷酸的缺失或插入,其相对于初始未修饰的核酸不会实质性地改变所得到的核酸的功能特性。因此,如本领域技术人员所理解,本发明包含的不仅仅是特定的示例性序列。
此外,本领域技术人员认识到,本发明所涵盖的实质相似的核酸序列也由其与本文所列举的序列或与本文公开的核苷酸序列的任何部分的杂交能力来定义(在中等严格的条件下,例如,0.5×SSC、0.1%SDS、60℃),并且在功能上与本文公开的任何核酸序列等同。严格的条件可以调整,以筛选适度相似的片段(如来自远缘生物的同源序列)到高度相似的片段(如从密切相关生物的功能酶复制的基因)。杂交后洗涤决定严格性条件。
术语“选择性杂交”包括在严格的杂交条件下,将核酸序列与指定的核酸靶序列杂交至可检测的比其与非靶核酸序列和实质排除的非靶核酸杂交更高的程度(例如,至少为背景的2倍)。选择性杂交序列通常彼此之间具有大约至少80%的序列同一性或90%的序列同一性,达到并包括100%的序列同一性(即,完全互补)。
术语“严格条件”或“严格的杂交条件”包括探针将选择性地与其靶序列杂交的条件。严格条件取决于序列,且在不同的情况下会有所不同。通过控制杂交和/或洗涤条件的严格性,与探针(同源探针)100%互补的靶序列能够被识别。或者,可以调整严格条件以允许序列中的一些不匹配,从而检测到较低程度的相似性(异源探测)。一般而言,探针长度小于约1000个核苷酸,可选择小于500个核苷酸。通常,严格条件是其中盐浓度小于约1.5M Na离子,尤其是约0.01至1.0M Na离子的浓度(或其它盐),pH为7.0至8.3,且对于短探针(例如,10到50个核苷酸)温度至少约30℃;对于长探针(例如大于50个核苷酸)温度至少约60℃。严格的条件也可以通过添加去稳定剂如甲酰胺来实现。示例性的低严格条件包括:在37℃下缓冲溶液为30%至35%甲酰胺、1M NaCl、1%SDS(十二烷基硫酸钠)的条件杂交,50至55℃下在1×至2×SSC(20×SSC=3.0M NaCl/0.3M柠檬酸三钠)中洗涤。示例性的中等严格条件包括37℃下在40%至45%甲酰胺、1M NaCl、1%SDS中杂交,55至60℃下在0.5×至1×SSC中洗涤。示例性的高严格条件包括37℃下在50%甲酰胺、1M NaCl、1%SDS中杂交,60至65℃下在0.1×SSC中洗涤。特异性通常是杂交后洗涤的功能,关键因素是最终洗涤液的离子强度和温度。对于DNA-DNA杂交,T
目前所公开的多肽和实质类似变体之间的差异是有限的,因此前者和后者的序列总体上是非常相似的,并且在许多区域中是相同的。它们可能通过一个或多个替换、添加、缺失、融合和截短而在氨基酸序列上不同,这些替换、添加、缺失、融合和截短可能以任何组合形式存在。这些差异可能产生沉默的变化,并导致功能等效的多肽。因此,本领域技术人员将容易理解,各种多核苷酸序列中的任何一种都能够编码本发明的多肽和变体多肽。多肽序列变体可具有“保守”变化,其中取代氨基酸具有相似的结构或化学性质。因此,可以基于残基的极性、电荷、溶解度、疏水性、亲水性和/或两亲性性质的相似性来进行有意的氨基酸替换,只要保留了多肽的大量功能性或生物活性。例如,带负电的氨基酸可包括天冬氨酸和谷氨酸,带正电的氨基酸可包括赖氨酸和精氨酸,具有类似亲水性值的不带电荷的极性头基的氨基酸可包括亮氨酸、异亮氨酸和缬氨酸;甘氨酸和丙氨酸;天冬酰胺和谷氨酰胺;丝氨酸和苏氨酸;苯丙氨酸和酪氨酸。更为罕见的是,变体可能具有“非保守”变化,例如用色氨酸替换甘氨酸。类似的微小变化也可能包括氨基酸的缺失或插入,或两者兼而有之。相关多肽可包括例如一个或多个N-连接或O-连接糖基化位点的添加和/或删除,或一个或多个半胱氨酸残基的添加和/或删除。使用本领域众所周知的计算机程序,例如DNASTAR软件(参见美国专利No.5840544),可以找到在不消除功能性或生物活性的情况下确定哪些氨基酸残基和多少氨基酸残基可以被取代、插入或删除的指南。
在核酸或多肽序列的上下文中,“序列同一性”或“同一性”是指两个序列中的核酸碱基或氨基酸残基,当在指定的比较窗口上比对以获得最大对应性时,它们是相同的。因此,“序列同一性百分比”是指通过在比较窗口中比较两个最佳排列的序列而确定的值,其中,与参考序列(不包括添加或缺失)以最佳排列方式相比,比较窗口中的多核苷酸或多肽序列的部分可包括添加或缺失(即缺口)。百分比的计算是通过确定两个序列中具有相同的核酸碱基或氨基酸残基出现的位置的数,从而得到匹配位置的数量,将匹配的位置数除以比较窗口中的位置总数,然后将结果乘以100,得到序列一致性的百分比。百分比序列同一性的有用示例包括但不限于50%、55%、60%、65%、70%、75%、80%、85%、90%或95%,或50%到100%的任何整数百分比。可以使用本文描述的任何程序来确定这些同一性。
可以使用设计用于检测同源序列的多种比较方法来确定序列比对和同一性或相似性百分比,包括但不限于LASERGENE生物信息学计算套件(DNASTAR Inc.,Madison,Wis)的MegAlign
本领域技术人员清楚地理解,许多水平的序列同一性可用于识别多肽变体,其中所述多肽变体具有相同或相似的功能或活性。同一性百分比的有用示例包括但不限于50%、55%、60%、65%、70%、75%、80%、85%、90%或95%,或50%到100%之间的任何整数百分比。实际上,从50%到100%的任何整数氨基酸同一性可用于描述本发明,例如90%、91%、92%、93%、94%、95%、96%、97%、98%或99%。
本说明书所附权利要求书中所有手段或步骤的相应结构、材料、动作和等效物加上功能元件旨在包括用于与具体要求保护的其他权利要求元件组合执行功能的任何结构、材料或动作。
应当注意的是,起草权利要求书时可以排除任何可选元素。因此,本声明旨在作为在叙述权利要求要素或使用“否定”限制时使用专有术语(例如“单独”、“仅”等)的先行基础。术语“优选地”、“优选的”、“优选”、“可选地”、“可以”和类似术语用于指示的所提及的项目、条件或步骤为本发明的可选(非必需)特征。
单数形式“a”、“an”和“The”包括复数指代,除非上下文另有明确规定。术语“和/或”是指与本术语相关的任何一个项目、项目的任何组合或所有项目。本领域技术人员容易理解短语“一个或多个”,特别是当结合其用法上下文阅读时。
除非上下文另外明确指出,否则单数形式“一个”,“一种”和“该”包括复数形式。术语“和/或”是指与该术语相关联的项目中的任何一个、任何组合或全部。短语“一个或多个”是本领域的技术人员容易理解的,特别是在阅读应用其的上下文时。
术语“约”可以指所指定值的±5%、±10%、±20%或±25%的变化。例如,在一些实施方案中,“约50%”可以携带从45%到55%的变化。对于整数范围,术语“约”可以在该范围的每一端包括大于和/或小于所列举的整数的一个或两个整数。除非本文另外指出,否则术语“约”旨在包括与所述范围接近的值和范围,所述值和范围就组合物或实施方案的功能而言是等效的。
如本领域技术人员将理解的,出于任何目的和所有目的,特别是在提供书面描述方面,本文列举的所有范围还涵盖任何和所有可能的子范围及其子范围的组合,以及组成范围的各个值,特别是整数值。列举的范围包括该范围内的每个特定值、整数、小数或同一性。任何列出的范围都可以很容易地识别为充分描述,并且可以将相同范围分解为至少相等的一半、三分之一、四分之一、五分之一或十分之一。作为非限制性实施方案,可以容易地将本文讨论的每个范围分解为下三分之一、中三分之一和上三分之一等。
如本领域技术人员还将理解的,诸如“最多”、“至少”、“大于”、“小于”、“多于”、或“更多”等的所有语言都包括所列举的数字,并且这些术语是指随后细分为上述子范围的范围。以相同的方式,本文列举的所有比率还包括落入更宽比率内的所有子比率。
SEQUENCE LISTING
<110> 昱星生物科技股份有限公司
艾伯塔大学校董
<120> 光遗传控制下的遗传编码荧光指示剂及其应用
<130> P21113905WP
<150> 62/753,121
<151> 2018-10-31
<160> 7
<170> PatentIn version 3.5
<210> 1
<211> 1242
<212> DNA
<213> Artificial Sequence
<220>
<223> Artificial sequence based on bacterial sequence
<400> 1
atgggcagcg tgaagctgat ccccagcctg accaccgtga tcctcgtgaa gtccatgctg 60
cggaagcgga gcttcggcaa ccccttcaag tataatacgg agaccctgta ccccgctgac 120
ggcggcctgg aaggcgcatg tgacatggcc ctgaagctcg tgggcggggg ccacctgaac 180
tgcagccttg agaccacata cagatccaag aaacccgcta cgaacctcaa gatgcccggt 240
gtctacaacg tggaccacag actggaacga atcaaagagg ccgacgatga gacctacgtc 300
gagctgcacg aggtggctgt ggccagatac gtgggcctgg gtggtggcgg aggtacaggc 360
gggagtgtga gcgagctgat taaggagaac atgccaatga agctgtacat ggagggcacc 420
gtgaacaacc accacttcaa gtgcacatcc gagggcgaag gcaagcccta cgagggcacc 480
cagaccatga gaatcaaggt cgtcgagggc ggccctctcc ccttcgcctt cgacatcctg 540
gctaccagct tcatgtacgg cagcagaacc ttcatcaagc accctcctgg catccccgac 600
ttctttaagc agtccttccc tgagggcttc acatgggaga gagtcaccac atacgaagac 660
gggggcgtgc tgaccgctac ccaggacacc agcctccagg acggctgcct catctacaac 720
gtcaaggtta gagggatgaa cttcccagcc aacggccctg tgatgcagaa gaaaacactc 780
ggctgggagg ccagtaatgg ccaactgact gaagagcaga tcgcagaatt taaagaggct 840
ttctccctat ttgacaagga cggggatggg acgataacaa ccaaggagct ggggacggtg 900
atgcggtctc tggggcagaa ccccacagaa gcggagctgc aggacatgat caatgaagta 960
gatgccgacg gtgacggcac attcgacttc cctgagttcc tgacgatgat ggcaagaaaa 1020
atgagttata gagtcactga agaggaaatt agagaagcgt tccgcgtgtt tgataaggac 1080
ggcaatggct acatcggcgc agcagagctt cgccacgtga tgacagacct tggagagaag 1140
ttaacagatg aggaggttga tgaaatgatc agggtagcag acatcgatgg ggatggtcag 1200
gtaaactacg aagagtttgt acaaatgatg acagcgaagt ag 1242
<210> 2
<211> 2973
<212> DNA
<213> Artificial Sequence
<220>
<223> Artificial sequence based on bacterial sequence
<400> 2
atggactatg gcggcgcttt gtctgccgtc ggacgcgaac ttttgttcgt tactaatcct 60
gtggtggtga acgggtccgt cctggtccct gaggatcaat gttactgtgc cggatggatt 120
gaatctcgcg gcacgaacgg cgctcagacc gcgtcaaatg tcctgcagtg gcttgcagca 180
ggattcagca ttttgctgct gatgttctat gcctaccaaa cctggaaatc tacatgcggc 240
tgggaggaga tctatgtgtg cgccattgaa atggttaagg tgattctcga gttctttttt 300
gagtttaaga atccctctat gctctacctt gccacaggac accgggtgca gtggctgcgc 360
tatgcagagt ggctgctcac ttgtcctgtc atccttatcc acctgagcaa cctcaccggc 420
ctgagcaacg actacagcag gagaaccatg ggactccttg tctcagacat cgggactatc 480
gtgtgggggg ctaccagcgc catggcaacc ggctatgtta aagtcatctt cttttgtctt 540
ggattgtgct atggcgcgaa cacatttttt cacgccgcca aagcatatat cgagggttat 600
catactgtgc caaagggtcg gtgccgccag gtcgtgaccg gcatggcatg gctgtttttc 660
gtgagctggg gtatgttccc aattctcttc attttggggc ccgaaggttt tggcgtcctg 720
agcgtctatg gctccaccgt aggtcacacg attattgatc tgatgagtaa aaattgttgg 780
gggttgttgg gacactacct gcgcgtcctg atccacgagc acatattgat tcacggagat 840
atccgcaaaa ccaccaaact gaacatcggc ggaacggaga tcgaggtcga gactctcgtc 900
gaagacgaag ccgaggccgg agccgtgcca gcggccgcca ccatggtgag caagggcgag 960
gagctgttca ccggggtggt gcccatcctg gtcgagctgg acggcgacgt aaacggccac 1020
aagttcagcg tgtccggcga gggcgagggc gatgccacct acggcaagct gaccctgaag 1080
ttcatctgca ccaccggcaa gctgcccgtg ccctggccca ccctcgtgac caccttcggc 1140
tacggcctgc agtgcttcgc ccgctacccc gaccacatga agcagcacga cttcttcaag 1200
tccgccatgc ccgaaggcta cgtccaggag cgcaccatct tcttcaagga cgacggcaac 1260
tacaagaccc gcgccgaggt gaagttcgag ggcgacaccc tggtgaaccg catcgagctg 1320
aagggcatcg acttcaagga ggacggcaac atcctggggc acaagctgga gtacaactac 1380
aacagccaca acgtctatat catggccgac aagcagaaga acggcatcaa ggtgaacttc 1440
aagatccgcc acaacatcga ggacggcagc gtgcagctcg ccgaccacta ccagcagaac 1500
acccccatcg gcgacggccc cgtgctgctg cccgacaacc actacctgag ctaccagtcc 1560
gccctgagca aagaccccaa cgagaagcgc gatcacatgg tcctgctgga gttcgtgacc 1620
gccgccggga tcactctcgg catggacgag ctgtacaagg gatccggagc tactaacttc 1680
agcctgctga agcaggctgg agacgtggag gagaaccctg gacctactag tatgggcagc 1740
gtgaagctga tccccagcct gaccaccgtg atcctcgtga agtccatgct gcggaagcgg 1800
agcttcggca accccttcaa gtataatacg gagaccctgt accccgctga cggcggcctg 1860
gaaggcgcat gtgacatggc cctgaagctc gtgggcgggg gccacctgaa ctgcagcctt 1920
gagaccacat acagatccaa gaaacccgct acgaacctca agatgcccgg tgtctacaac 1980
gtggaccaca gactggaacg aatcaaagag gccgacgatg agacctacgt cgagctgcac 2040
gaggtggctg tggccagata cgtgggcctg ggtggtggcg gaggtacagg cgggagtgtg 2100
agcgagctga ttaaggagaa catgccaatg aagctgtaca tggagggcac cgtgaacaac 2160
caccacttca agtgcacatc cgagggcgaa ggcaagccct acgagggcac ccagaccatg 2220
agaatcaagg tcgtcgaggg cggccctctc cccttcgcct tcgacatcct ggctaccagc 2280
ttcatgtacg gcagcagaac cttcatcaag caccctcctg gcatccccga cttctttaag 2340
cagtccttcc ctgagggctt cacatgggag agagtcacca catacgaaga cgggggcgtg 2400
ctgaccgcta cccaggacac cagcctccag gacggctgcc tcatctacaa cgtcaaggtt 2460
agagggatga acttcccagc caacggccct gtgatgcaga agaaaacact cggctgggag 2520
gccagtaatg gccaactgac tgaagagcag atcgcagaat ttaaagaggc tttctcccta 2580
tttgacaagg acggggatgg gacgataaca accaaggagc tggggacggt gatgcggtct 2640
ctggggcaga accccacaga agcggagctg caggacatga tcaatgaagt agatgccgac 2700
ggtgacggca cattcgactt ccctgagttc ctgacgatga tggcaagaaa aatgagttat 2760
agagtcactg aagaggaaat tagagaagcg ttccgcgtgt ttgataagga cggcaatggc 2820
tacatcggcg cagcagagct tcgccacgtg atgacagacc ttggagagaa gttaacagat 2880
gaggaggttg atgaaatgat cagggtagca gacatcgatg gggatggtca ggtaaactac 2940
gaagagtttg tacaaatgat gacagcgaag tag 2973
<210> 3
<211> 1488
<212> DNA
<213> Artificial Sequence
<220>
<223> Artificial sequence based on bacterial sequence
<400> 3
atgtcggtac cgctgactac ctcagcattc ggccacgcgt ttctggctaa ctgtgaacgt 60
gagcagatcc acctggcggg ctccattcag ccgcacggta tcctgctggc tgttaaagag 120
cctgacaacg tggtgatcca ggcttctatt aacgctgcgg agttcctgaa caccaacttt 180
gttgttggcc gtccgctgcg tgacctgggc ggcgatctgc ctttgcagat cctgccgcac 240
ctgaacggcc cgctgcacct ggctccgatg accctgcgtt gtactgtggg ttctccgccg 300
cgtcgtgtgg actgtaccat tcatcgtccg tctaacggcg gcctgatcgt agaactggaa 360
ccagcaacca aggccactaa cattgcgccg gctctggtcg gtgcgcttca tcgtatcact 420
tcttcatcct ccctgatggg cctgtgtgac gaaaccgcga ctattttccg tgagattact 480
ggcttcgacc gtgtgatggt aatgcgtctc ggcgcgcttg acgatctgac tgaagagcag 540
atcgcagaga ttaaagaggc tttctcccta tttgacaagg acggggacgg gacgataaca 600
accaaggagc tggggacggt gttccggtct ctggggcaga accccacaga agcagagctg 660
caggacatga tcaatgaagt agatgccgat ggcgacggca cattcgactt ccctgagttc 720
ctgacgatga tggcaaggaa aatgaatgac tcagacagtg aagaggaaat tagagaagcg 780
ttccgcgtgt ttgataagga cggcaatggc tacatcggcg cagcagagct tcgccacgtg 840
atgacagacc ttggtgagaa gttaactgat gaggaggttg atgaaatgat cagggtagca 900
gacaacgatg gggatggtca ggtaaactac gaagagtttg tacaaatgat gacagcgaag 960
ggtggcggag gttctgtaga ttcatcacgt cgtaagtgga ataaggcagg tcacgcagtc 1020
agagctatag gtcggctgag ctcgcgtagg catgatttgc tgtccgaatg tcgtcgtgcg 1080
gacctggaag cgttcctggg taaccgctac ccggcgtcta ctattccgca gatcgctcgt 1140
cgcctgtacg aacttaaccg tgttcgcctg ctggtagatg tgaactatac tccggttccg 1200
ctacagccgc gcatcagccc gctgaacggt cgtgatctgg atatgtccct gtcttgcctg 1260
cgctctatgt ccccgatcca ccagaaatac atgcaggaca tgggcgttgg cgcaaccctg 1320
gtttgctctc tgatggtgtc tggtcgtctg tggggtctga tcgtttgcca ccactacgaa 1380
ccgcgcttcg ttccgtccca cattcgcgct gctggcgaag cgctggcgga aacttgtgcg 1440
aaccgcatcg cgacgctgga gagctttgca cagtctcagt ccaaatga 1488
<210> 4
<211> 1485
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 4
atgtcggtac cgctgactac ctcagcattc ggccacgcgt ttctggctaa ctgtgaacgt 60
gagcagatcc acctggcggg ctccattcag ccgcacggta tcctgctggc tgttaaagag 120
cctgacaacg tggtgatcca ggcttctatt aacgctgcgg agttcctgaa caccaacttt 180
gttgttggcc gtccgctgcg tgacctgggc ggcgatctgc ctttgcagat cctgccgcac 240
ctgaacggcc cgctgcacct ggctccgatg accctgcgtt gtactgtggg ttctccgccg 300
cgtcgtgtgg actgtaccat tcatcgaccg tctaacggcg gcctgatcgt agaactggaa 360
ccagcaacca aggccactaa cattgcgccg gctctggtcg gtgcgcttca tcgtatcact 420
tcttcatcct ccctgatggg cctgtgtgac gaaaccgcga ctattttccg tgagattact 480
ggtttcgacc gtgtgatggt aatgcgtctc ggcgcgcttg acgatctgac tgaagagcag 540
atcgcagaga ttaaagaggc tttctcccta tttgacaagg acggggacgg gacgataaca 600
accaaggagc tggggacggt gttccggtct ctggggcaga accccacaga agcagagctg 660
caggacatga tcaatgaagt agatgccgat ggcgacggca tattcgactt ccctgagttc 720
ctgacgatga tggcaaggaa aatgaatgac tcagacagtg aagaggaaat tagaggagcg 780
ttccgcgtgt ttgataagga cggcaatggc tacatcggcg cagcagagct tcgccacgtg 840
atgacagacc ttggtgagaa gttaactgat gaggaggttg atgaaatgat cagggtagca 900
gacaacgatg gggatggtca ggtaaactac gaagagtttg tacaaatgat gacagcgaag 960
ggtggcggag gttctgtaga ttcatcacgt cgtaagtgga ataaggcagg tcacgcagtc 1020
agagctatag gtcggctggg ctcgcgtagg catgatttgc tgtccgaatg tcgtcgtgcg 1080
gacctggaag cgttcctagg taaccgctac ccggcgtcta ctattccgca gatcgctcgt 1140
cgcctgtacg aactcaaccg tgttcgcctg ctggtagatg tgaactatac tccggttccg 1200
ctagagccgc gcatcagccc gctgaacggt cgtgatctgg atatgtccct gtcttgcctg 1260
cgctctatgt ccccgatcca ccagaaatac atgcaggaca tgggcgttgg cgcaaccctg 1320
gtttgctctc tgatggtgtc tggtcgtctg tggggtctga tcgtttgcca ccactacgaa 1380
ccgcgctacg ttccgtccca cattcgcgct gctggcgaag cgctggcgga agcatgtgcg 1440
aaccgcatcg cgacgctgga gagctttgca cagtctcagt ccaaa 1485
<210> 5
<211> 413
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial sequence based on bacterial sequence
<400> 5
Met Gly Ser Val Lys Leu Ile Pro Ser Leu Thr Thr Val Ile Leu Val
1 5 10 15
Lys Ser Met Leu Arg Lys Arg Ser Phe Gly Asn Pro Phe Lys Tyr Asn
20 25 30
Thr Glu Thr Leu Tyr Pro Ala Asp Gly Gly Leu Glu Gly Ala Cys Asp
35 40 45
Met Ala Leu Lys Leu Val Gly Gly Gly His Leu Asn Cys Ser Leu Glu
50 55 60
Thr Thr Tyr Arg Ser Lys Lys Pro Ala Thr Asn Leu Lys Met Pro Gly
65 70 75 80
Val Tyr Asn Val Asp His Arg Leu Glu Arg Ile Lys Glu Ala Asp Asp
85 90 95
Glu Thr Tyr Val Glu Leu His Glu Val Ala Val Ala Arg Tyr Val Gly
100 105 110
Leu Gly Gly Gly Gly Gly Thr Gly Gly Ser Val Ser Glu Leu Ile Lys
115 120 125
Glu Asn Met Pro Met Lys Leu Tyr Met Glu Gly Thr Val Asn Asn His
130 135 140
His Phe Lys Cys Thr Ser Glu Gly Glu Gly Lys Pro Tyr Glu Gly Thr
145 150 155 160
Gln Thr Met Arg Ile Lys Val Val Glu Gly Gly Pro Leu Pro Phe Ala
165 170 175
Phe Asp Ile Leu Ala Thr Ser Phe Met Tyr Gly Ser Arg Thr Phe Ile
180 185 190
Lys His Pro Pro Gly Ile Pro Asp Phe Phe Lys Gln Ser Phe Pro Glu
195 200 205
Gly Phe Thr Trp Glu Arg Val Thr Thr Tyr Glu Asp Gly Gly Val Leu
210 215 220
Thr Ala Thr Gln Asp Thr Ser Leu Gln Asp Gly Cys Leu Ile Tyr Asn
225 230 235 240
Val Lys Val Arg Gly Met Asn Phe Pro Ala Asn Gly Pro Val Met Gln
245 250 255
Lys Lys Thr Leu Gly Trp Glu Ala Ser Asn Gly Gln Leu Thr Glu Glu
260 265 270
Gln Ile Ala Glu Phe Lys Glu Ala Phe Ser Leu Phe Asp Lys Asp Gly
275 280 285
Asp Gly Thr Ile Thr Thr Lys Glu Leu Gly Thr Val Met Arg Ser Leu
290 295 300
Gly Gln Asn Pro Thr Glu Ala Glu Leu Gln Asp Met Ile Asn Glu Val
305 310 315 320
Asp Ala Asp Gly Asp Gly Thr Phe Asp Phe Pro Glu Phe Leu Thr Met
325 330 335
Met Ala Arg Lys Met Ser Tyr Arg Val Thr Glu Glu Glu Ile Arg Glu
340 345 350
Ala Phe Arg Val Phe Asp Lys Asp Gly Asn Gly Tyr Ile Gly Ala Ala
355 360 365
Glu Leu Arg His Val Met Thr Asp Leu Gly Glu Lys Leu Thr Asp Glu
370 375 380
Glu Val Asp Glu Met Ile Arg Val Ala Asp Ile Asp Gly Asp Gly Gln
385 390 395 400
Val Asn Tyr Glu Glu Phe Val Gln Met Met Thr Ala Lys
405 410
<210> 6
<211> 495
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial sequence based on bacterial sequence
<400> 6
Met Ser Val Pro Leu Thr Thr Ser Ala Phe Gly His Ala Phe Leu Ala
1 5 10 15
Asn Cys Glu Arg Glu Gln Ile His Leu Ala Gly Ser Ile Gln Pro His
20 25 30
Gly Ile Leu Leu Ala Val Lys Glu Pro Asp Asn Val Val Ile Gln Ala
35 40 45
Ser Ile Asn Ala Ala Glu Phe Leu Asn Thr Asn Phe Val Val Gly Arg
50 55 60
Pro Leu Arg Asp Leu Gly Gly Asp Leu Pro Leu Gln Ile Leu Pro His
65 70 75 80
Leu Asn Gly Pro Leu His Leu Ala Pro Met Thr Leu Arg Cys Thr Val
85 90 95
Gly Ser Pro Pro Arg Arg Val Asp Cys Thr Ile His Arg Pro Ser Asn
100 105 110
Gly Gly Leu Ile Val Glu Leu Glu Pro Ala Thr Lys Ala Thr Asn Ile
115 120 125
Ala Pro Ala Leu Val Gly Ala Leu His Arg Ile Thr Ser Ser Ser Ser
130 135 140
Leu Met Gly Leu Cys Asp Glu Thr Ala Thr Ile Phe Arg Glu Ile Thr
145 150 155 160
Gly Phe Asp Arg Val Met Val Met Arg Leu Gly Ala Leu Asp Asp Leu
165 170 175
Thr Glu Glu Gln Ile Ala Glu Ile Lys Glu Ala Phe Ser Leu Phe Asp
180 185 190
Lys Asp Gly Asp Gly Thr Ile Thr Thr Lys Glu Leu Gly Thr Val Phe
195 200 205
Arg Ser Leu Gly Gln Asn Pro Thr Glu Ala Glu Leu Gln Asp Met Ile
210 215 220
Asn Glu Val Asp Ala Asp Gly Asp Gly Thr Phe Asp Phe Pro Glu Phe
225 230 235 240
Leu Thr Met Met Ala Arg Lys Met Asn Asp Ser Asp Ser Glu Glu Glu
245 250 255
Ile Arg Glu Ala Phe Arg Val Phe Asp Lys Asp Gly Asn Gly Tyr Ile
260 265 270
Gly Ala Ala Glu Leu Arg His Val Met Thr Asp Leu Gly Glu Lys Leu
275 280 285
Thr Asp Glu Glu Val Asp Glu Met Ile Arg Val Ala Asp Asn Asp Gly
290 295 300
Asp Gly Gln Val Asn Tyr Glu Glu Phe Val Gln Met Met Thr Ala Lys
305 310 315 320
Gly Gly Gly Gly Ser Val Asp Ser Ser Arg Arg Lys Trp Asn Lys Ala
325 330 335
Gly His Ala Val Arg Ala Ile Gly Arg Leu Ser Ser Arg Arg His Asp
340 345 350
Leu Leu Ser Glu Cys Arg Arg Ala Asp Leu Glu Ala Phe Leu Gly Asn
355 360 365
Arg Tyr Pro Ala Ser Thr Ile Pro Gln Ile Ala Arg Arg Leu Tyr Glu
370 375 380
Leu Asn Arg Val Arg Leu Leu Val Asp Val Asn Tyr Thr Pro Val Pro
385 390 395 400
Leu Gln Pro Arg Ile Ser Pro Leu Asn Gly Arg Asp Leu Asp Met Ser
405 410 415
Leu Ser Cys Leu Arg Ser Met Ser Pro Ile His Gln Lys Tyr Met Gln
420 425 430
Asp Met Gly Val Gly Ala Thr Leu Val Cys Ser Leu Met Val Ser Gly
435 440 445
Arg Leu Trp Gly Leu Ile Val Cys His His Tyr Glu Pro Arg Phe Val
450 455 460
Pro Ser His Ile Arg Ala Ala Gly Glu Ala Leu Ala Glu Thr Cys Ala
465 470 475 480
Asn Arg Ile Ala Thr Leu Glu Ser Phe Ala Gln Ser Gln Ser Lys
485 490 495
<210> 7
<211> 495
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial sequence based on bacterial sequence
<400> 7
Met Ser Val Pro Leu Thr Thr Ser Ala Phe Gly His Ala Phe Leu Ala
1 5 10 15
Asn Cys Glu Arg Glu Gln Ile His Leu Ala Gly Ser Ile Gln Pro His
20 25 30
Gly Ile Leu Leu Ala Val Lys Glu Pro Asp Asn Val Val Ile Gln Ala
35 40 45
Ser Ile Asn Ala Ala Glu Phe Leu Asn Thr Asn Phe Val Val Gly Arg
50 55 60
Pro Leu Arg Asp Leu Gly Gly Asp Leu Pro Leu Gln Ile Leu Pro His
65 70 75 80
Leu Asn Gly Pro Leu His Leu Ala Pro Met Thr Leu Arg Cys Thr Val
85 90 95
Gly Ser Pro Pro Arg Arg Val Asp Cys Thr Ile His Arg Pro Ser Asn
100 105 110
Gly Gly Leu Ile Val Glu Leu Glu Pro Ala Thr Lys Ala Thr Asn Ile
115 120 125
Ala Pro Ala Leu Val Gly Ala Leu His Arg Ile Thr Ser Ser Ser Ser
130 135 140
Leu Met Gly Leu Cys Asp Glu Thr Ala Thr Ile Phe Arg Glu Ile Thr
145 150 155 160
Gly Phe Asp Arg Val Met Val Met Arg Leu Gly Ala Leu Asp Asp Leu
165 170 175
Thr Glu Glu Gln Ile Ala Glu Ile Lys Glu Ala Phe Ser Leu Phe Asp
180 185 190
Lys Asp Gly Asp Gly Thr Ile Thr Thr Lys Glu Leu Gly Thr Val Phe
195 200 205
Arg Ser Leu Gly Gln Asn Pro Thr Glu Ala Glu Leu Gln Asp Met Ile
210 215 220
Asn Glu Val Asp Ala Asp Gly Asp Gly Ile Phe Asp Phe Pro Glu Phe
225 230 235 240
Leu Thr Met Met Ala Arg Lys Met Asn Asp Ser Asp Ser Glu Glu Glu
245 250 255
Ile Arg Gly Ala Phe Arg Val Phe Asp Lys Asp Gly Asn Gly Tyr Ile
260 265 270
Gly Ala Ala Glu Leu Arg His Val Met Thr Asp Leu Gly Glu Lys Leu
275 280 285
Thr Asp Glu Glu Val Asp Glu Met Ile Arg Val Ala Asp Asn Asp Gly
290 295 300
Asp Gly Gln Val Asn Tyr Glu Glu Phe Val Gln Met Met Thr Ala Lys
305 310 315 320
Gly Gly Gly Gly Ser Val Asp Ser Ser Arg Arg Lys Trp Asn Lys Ala
325 330 335
Gly His Ala Val Arg Ala Ile Gly Arg Leu Gly Ser Arg Arg His Asp
340 345 350
Leu Leu Ser Glu Cys Arg Arg Ala Asp Leu Glu Ala Phe Leu Gly Asn
355 360 365
Arg Tyr Pro Ala Ser Thr Ile Pro Gln Ile Ala Arg Arg Leu Tyr Glu
370 375 380
Leu Asn Arg Val Arg Leu Leu Val Asp Val Asn Tyr Thr Pro Val Pro
385 390 395 400
Leu Glu Pro Arg Ile Ser Pro Leu Asn Gly Arg Asp Leu Asp Met Ser
405 410 415
Leu Ser Cys Leu Arg Ser Met Ser Pro Ile His Gln Lys Tyr Met Gln
420 425 430
Asp Met Gly Val Gly Ala Thr Leu Val Cys Ser Leu Met Val Ser Gly
435 440 445
Arg Leu Trp Gly Leu Ile Val Cys His His Tyr Glu Pro Arg Tyr Val
450 455 460
Pro Ser His Ile Arg Ala Ala Gly Glu Ala Leu Ala Glu Ala Cys Ala
465 470 475 480
Asn Arg Ile Ala Thr Leu Glu Ser Phe Ala Gln Ser Gln Ser Lys
485 490 495
- 光遗传控制下的遗传编码荧光指示剂及其应用
- 半滑舌鳎性逆转遗传控制位点、包含其的试剂盒及其应用
