一种SNP分子标记组合及其在玉米种质鉴定和育种中的应用
文献发布时间:2023-06-19 18:32:25
技术领域
本发明涉及生物信息学技术领域,尤其涉及一种SNP分子标记组合及其在玉米种质鉴定和育种中的应用。
背景技术
DNA芯片,又称基因芯片,是生物芯片的一种,通过将多种探针分子固定在基底上,根据A-T和C-G的碱基互补配对的原理,与检测的样品的基因进行杂交后检测杂交信号的强度即可得到样品的序列和数量信息。
DNA芯片目前广泛应用与疾病的诊断和治疗,农作物的鉴定和筛选和食品卫生监督等领域中,DNA芯片的出现有效提高了农作物的育种效率。目前关于玉米基因芯片的开发,最初基于GoldenGate平台,玉米上开发的一款芯片包含了1536个SNP标记。随后,基于Illumina的检测平台,利用美国玉米巢式关联作图群体27个遗传多样性自交系的合成测序鉴定出的约2百万个SNP标记,设计了一款包含49585个SNP标记的50K芯片,该芯片已被广泛应用于遗传研究和育种改良,然而大多数位点是来自于热带材料,不能更好地应用于鉴定温带材料基因型;此外,不同研究者也开发了其它密度的芯片,例如3K芯片。基于Affymetrix的检测平台,利用30个欧洲代表性温带自交系的重测序结果,开发了600K芯片,此外现有技术还研究开发了55K芯片、6H-60K芯片,然而这些芯片多态性位点均代表欧洲材料,不能有效解决我国温带种质的基因型。近期,基于靶向测序基因型检测的液相芯片被广泛利用,在先前芯片基础上,开发了10K、5K以及1K芯片等。尽管先前已开发了高密度、中等密度、低密度的玉米芯片,然而芯片设计的标记仅来源于少数美国或欧洲玉米种质资源(几十份或几百份)的重测序或其它测序结果,并且一些材料的重测序深度偏低且不一致,在鉴定现有的玉米育种材料时可能导致筛选出的SNP标记遗传多样性低、代表性差。此外,现有的芯片存在多态性位点代表性差、功能标记少、多样性低等问题。
发明内容
为了解决现有技术中存在的技术问题,本发明提供一种SNP分子标记组合及其在玉米种质鉴定和育种中的应用。
第一方面,本发明一种SNP分子标记组合,本发明通过2004份种质资源测序结果得到20M的SNP位点信息,后通过进一步筛选得到1050个SNP位点信息,这1050个SNP位点具备较低的个体缺失率(Miss值)和较高的最小等位基因频率(MAF值),具体信息如表1所示,SNP的位置信息在表1中染色体和位置一栏表示,位点的多态性在表1中的位点一栏表示,位点前后50bp的核苷酸序列信息如序列一栏的[]前后所示。
本发明进一步提供所述SNP分子标记,或所述探针组合,或所述DNA芯片在玉米分子标记辅助选育中的应用。
本发明进一步提供所述SNP分子标记,或所述探针组合,或所述DNA芯片在玉米类群分析或遗传转化中的应用。
本发明进一步提供所述SNP分子标记,或所述探针组合,或所述DNA芯片在玉米品种鉴定中的应用。
本发明具备如下有益效果:
本发明基于2004份具有遗传多样性丰富、不同育种阶段利用的全球温带玉米种质资源重测序结果,获得约2千万个高质量SNP标记,之后经过两大步骤的筛选得到1050个SNP分子标记,其中包含330个的花期性状、产量及产量相关性状、株型性状等全基因组关联获得的功能标记及育种选择位点,和272个杂种优势类群保守区段的特异性位点,经大群体验证的稳定检出的位点。这些SNP分子标记具备较低的个体缺失率和较高的最小等位基因频率。以本发明提供的SNP分子标记作为检测对象制备的DNA芯片之后,可以应用于玉米品种鉴定、杂种优势类群分析,具有通量高、重复性好、实用性广和准确性高的优点,在玉米种质资源鉴定评价、优良玉米新品种培育等方面具有重要的应用价值。
附图说明
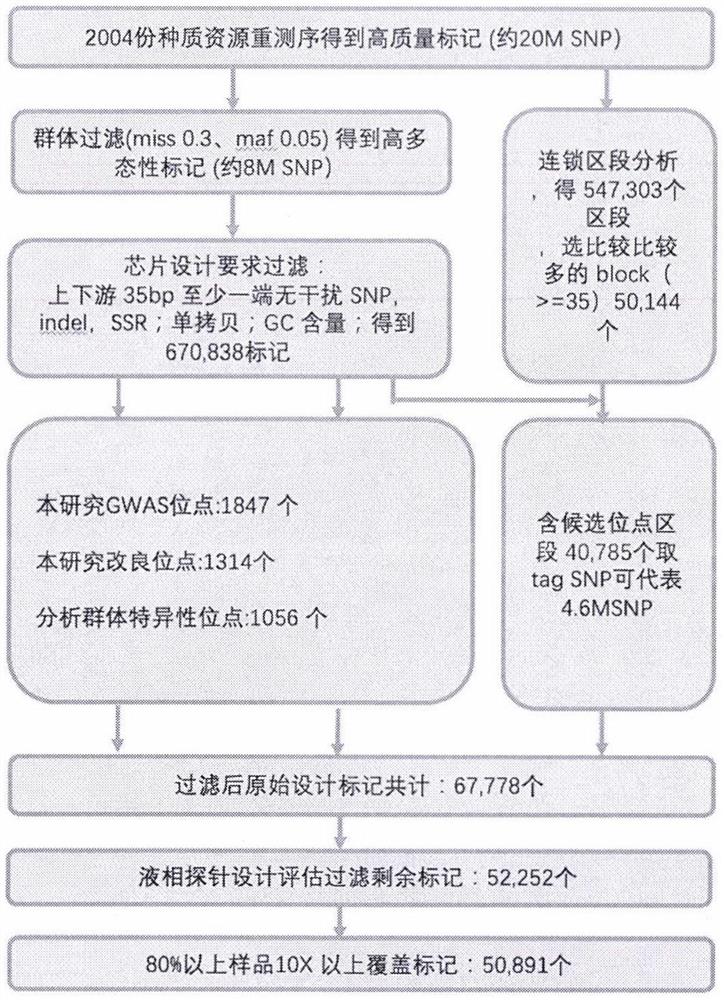
图1为本发明实施例1提供的筛选分子标记的流程图。
图2为本发明实施例1提供的1050个SNP分子标记在染色体上的分布结果示意图。
图3为本发明实施例1提供的1050个SNP分子标记的个体缺失率(miss)和最小等位基因频率(maf)的统计结果示意图,其中左图为个体缺失率的统计结果,右图为最小等位基因频率的统计结果示意图。
图4为本发明实施例2提供的类群分析结果示意图。
图5为本发明实施例2提供的类群划分结果示意图。
图6为本发明实施例2提供的STRUCTURE群体分析结果示意图群体分析结果示意图。
图7为本发明实施例2提供的STRUCTURE群体分析结果示意图群体分析结果示意图。
图8为本发明实施例2提供的相似度分析结果示意图。
图9为本发明实施例2提供的指纹图谱的分析结果示意图。
具体实施方式
以下实施例用于说明本发明,但不用来限制本发明的范围。
实施例1
本发明采用两千多份全球玉米种质资源材料的重测序vcf数据(使用maize v4版基因组)为基础,通过如下流程进行分子标记的筛选:
第一步如图1所示:首先,过滤掉群体标记多态性(MAF)低于0.05的位点;其次,筛选上下游共101bp的序列GC含量在30%-70%的标记;同时进行20M标记的LD的分析。选取block区段内标记数量大于35个的区段中代表性的标记(优先计划分:非同义突变>外显子变异>基因上下游及剪切区域;MAF值高于0.2(内含子>基因间区))共得到52k标记,进行液相探针捕获开发设计。第二步:使用设计的探针试剂盒进行2700份地方品种和145份代表系的检测,进行变异检测分析(BWA,GATK),选取在本次实验中表现优秀的位点,对于性状关联位点及育种改良等重要位点,作为第一优先级选取其中检出率和多态性最好的位点,按照1M保留一个标记的原则,进行最后的取舍;然后按照2M划区段,筛选未被最优先标记覆盖的区段的位点,尽量选择block代表位点中MAF>0.3;miss<0.1。经过以上两步的标记过滤、筛选获得1050个高质量代表性标记(如图2所示)。
本发明针对这1050个SNP位点进行统计,其个体缺失率(miss)和最小等位基因频率(maf)的统计结果如图3所示,可以看出个体缺失率在0.2以下,且普遍分布在0.05左右;而最小等位基因频率普遍分布在0.3~0.5的区间,说明本发明筛选得到的1050个SNP位点具备高质量、高检测率等特点。
实施例2
本发明采用这1050个分子标记进行类群分析,流程如下:
本发明通过液相捕获测序的技术进行二代测序,测序后的结果进行标准的变异检测分析流程。
二代测序流程如下:
1、提取基因组DNA后,利用酶切将基因组DNA进行片段化并且加上测序接头;
2、将带有生物素标记的RNA探针与已经带上接头序列的DNA片段进行结合;
3、链霉亲和素包裹的磁珠会和带有生物素标记的RNA探针与DNA相结合的双链复合物相结合(探针过量);
4、清洗(Washing)得到目标区域的DNA,其目的是去除非特异性杂交提高捕获效率;
5、对洗脱下来的DNA产物进行PCR扩增,构建Illumina测序文库进行测序;
变异检测信息分析的主要步骤如下:
1、对下机得到的Raw data进行质控得到Clean data
2、将Clean data比对到参考基因组上
3、根据比对结果,进行群体SNP的检测及过滤
利用GATK的HaplotypeCaller模型先进行gvcf的检测,得到的数据用GATK官网推荐的基础过滤参数进行过滤,选择使用vcftools对标记进行进一步的过滤,过滤后得到在2845样品中高多态性的标记。
然后分别使用群体分析的主流软件GCTA,Treebest和admixture分别进行群体的PCA,TREE和structure分析,并且使用R软件包进行相似度的分析。得到如图4所示的结果,结果显示,本发明采用的1050个分子标记针对2845个样本(样本来源为145份来自我国玉米育种不同时期骨干亲本和明星杂交种亲本,涵盖了玉米育种的主要杂种优势类群中的骨干系;2700份地方品种来自于我国不同生态区历史上种植的重要农家种,具有丰富的遗传多样性。)进行类群分析,可以准确地将多个类群分离开。得到如图5-图7所示的结果,图5、图6和图7的结果表明本发明的1050个标记可成功准确将这些样本进行类群划分,其中,图4为TREE,即群体进化树的分析结果;图5为PCA的分析结果;图6和图7为structure分析的结果。
相似度分析结果如图8所示,结果显示本发明提供的1050个标记可以准确地进行种质资源或品种的差异分析和品种鉴定。
指纹图谱构建结果如图9所示,结果显示:用不同颜色展示A、T、C、G和杂合基因型情况,每个个体的基因型就可以形象化的展示出来,并且具有唯一性。
虽然上文中已经用一般性说明及具体实施方案对本发明作了详尽的描述,但在本发明基础上,可以对之作一些修改或改进,这对本领域技术人员而言是显而易见的。因此,在不偏离本发明精神的基础上所做的这些修改或改进,均属于本发明要求保护的范围。
