一种基于多组学数据预测癌症患者预后风险的深度学习方法
文献发布时间:2023-06-19 11:02:01
技术领域
本发明涉及癌症患者生存分析技术领域,更具体地,涉及一种基于多组学数据预测癌症患者预后风险的深度学习方法。
背景技术
近年来癌症的高发生率促进了医学辅助技术的发展,预后风险分析是一种关键的医学辅助技术,它可以根据不同病人预后的潜在风险来辅助选择不同的治疗方案。
大多数针对癌症预后预测的方法都是通过分析单一组学的表达数据来实现,比如使用基因mRNA表达数据,甲基化数据,或miRNA数据等,然而患者的预后受到不同层次多种分子的共同调控,而且不同层次分子之间存在强烈的互补效应和相互作用,所以单组学数据分析结果往往只能提供片面的信息。此外,融合不同组学不同模态的数据分析,通过误差抵消,可缓解单组学方法对噪音过于敏感的问题。因此近年来融合多种数据进行癌症分析已经成为一种强有力的手段。
融合多组学数据最大的难点在于如何利用小样本的癌症数据,优化高维组学数据的降维效果。2018年,李鑫等人(李鑫,魏锣沛,吕章艳,等基于多组学数据构建肺腺癌预后相关风险预测模型[J].南京医科大学学报(自然科学版),2018,38(12);1820-1825)使用传统的L1正则化的Cox方法构建了基于多组学数据的肺癌预后相关风险预测模型,通过整合肺腺癌临床信息组、基因组和转录组的多组学信息,构建预后相关风险预测模型,但该方法不够稳健,无法解决在高维小样本癌症数据中表现不佳的缺陷,且预测准确率不高。之后有研究人员将深度学习应用于这一领域,利用自编码器提取肝癌的高维多组学特征(包括mRNA,miRNA和甲基化数据),之后将压缩后的特征用于识别其患者不同的临床亚型。在此基础上,研究人员又融合了拷贝数变异的相关数据,用于区分高危神经母细胞瘤的两种预后亚型。除此之外此方法还衍生出一些基于其他自编码器方法的变体。然而,此框架最大的问题在于,它把特征降维和患者风险预测拆分为两个模型来进行,方法不够稳健。2019年研究人员将比例风险模型的损失函数和深度神经网络结合,利用多组学数据直接预测患者的生存风险。此方法的问题在于,深度神经网络直接对风险预测的损失函数进行优化,并不能保证网络中多层压缩后的重建特征依然保持初始特征的空间分布特征,因此限制了此方法的性能。
发明内容
本发明为克服上述现有中预后风险预测准确率不高,无法解决目标数据集小的缺陷,提供一种基于多组学数据预测癌症患者预后风险的深度学习方法。
本发明的首要目的是为解决上述技术问题,本发明的技术方案如下:
一种基于多组学数据预测癌症患者预后风险的深度学习方法,用于预测癌症患者的预后风险,包括以下步骤:
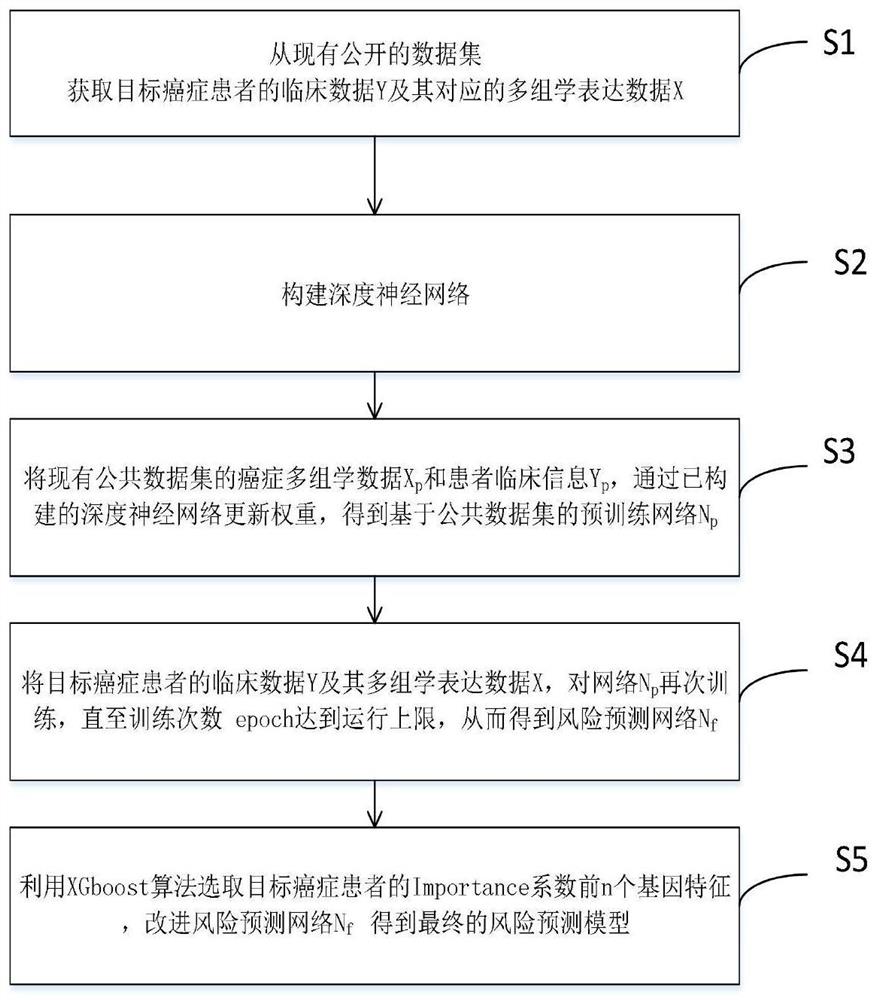
S1:从现有的公开数据集获取目标癌症患者的临床数据Y及其对应的多组学表达数据X;
S2:构建深度神经网络;
S3:将现有公共数据集的癌症多组学数据X
S4:将目标癌症患者的临床数据Y及其多组学表达数据X,对网络N
S5:利用XGboost算法选取目标癌症患者的Importance系数前n个基因特征,改进风险预测网络N
进一步的,步骤S2构建深度神经网络具体过程为:
S201:对多组学表达数据X进行编码生成压缩特征z=E(X),将压缩特征解码后产生新的特征X’,并计算解码后的数据恢复损失Lr;
S202:定义生存风险函数,所述生存风险函数表示癌症患者在时间设定时间t之前的存活率;
S203:利用生存风险函数构建比例风险函数;
S204:利用比例风险函数构建最大似然函数,通过最大似然函数得出初步预后风险预测损失函数;
S205:将数据恢复损失Lr加入初步预后风险预测损失函数构建最终损失函数。
进一步的,所述损失函数表达式为:
进一步的,所述生存风险函数表示为:S(t)=Pr(T>t)
其中,T是收集到患者的生存时间;
t时刻的生存风险函数:
进一步的,所述比例风险函数为:
λ(t|x)=λ
进一步的,所述最大似然函数可以表示为:
进一步的,所述初步预后风险预测损失函数则可表示为:
进一步的,最终损失函数表示为:l
进一步的,步骤S5所述的最终的风险预测模型表示:
其中,X
进一步的,步骤S5所述的Importance系数前n个基因特征,其中n的取值为200。
与现有技术相比,本发明技术方案的有益效果是:
本发明在对深度神经网络学习时通过公开数据集获取更多的先验知识,提升了预测模型的稳健性,同时引入了数据恢复损失函数和风险预测损失函数,更准确地利用多组学数据预测癌症患者的预后风险。
附图说明
图1为本发明方法流程图。
图2为本发明实施例中不同患者预后风险预测方法在模拟数据中的表现图。
图3为本发明预测的风险识别影响膀胱癌预后的靶向基因和通路示意图。
具体实施方式
为了能够更清楚地理解本发明的上述目的、特征和优点,下面结合附图和具体实施方式对本发明进行进一步的详细描述。需要说明的是,在不冲突的情况下,本申请的实施例及实施例中的特征可以相互组合。
在下面的描述中阐述了很多具体细节以便于充分理解本发明,但是,本发明还可以采用其他不同于在此描述的其他方式来实施,因此,本发明的保护范围并不受下面公开的具体实施例的限制。
实施例1
如图1所示,一种基于多组学数据预测癌症患者预后风险的深度学习方法,用于预测癌症患者的预后风险,包括以下步骤:
S1:从现有的公开数据集(例如TCGA,GEO))获取目标癌症患者的临床数据Y及其对应的多组学表达数据X;
在一个具体的实施例,使用14个TCGA数据集(BRCA,CESC,COAD,ESCA,HNSC,KIRC,LGG,LIHC,LUAD,LUSC,MESO,PAAD,SRAC和SKCM)进行预训练,而膀胱癌(BLCA)数据作为目标癌症。
其中,多组学数据包括了膀胱癌患者的mRNA表达,miRNA表达,DNA甲基化信息及拷贝数变异信息。mRNA数据是由UNC Illumina HiSeq_RNASeq V2生成的RNA测序数据。miRNA是由BCGSC Illumina HiSeq miRNASeq获得的miRNA测序数据。DNA甲基化数据由USCHumanMethylation450生成;CNV数据是由BROAD-MIT全基因组SNP_6生成的。所有这些数据均来自TCGA lv3级数据。我们计算了每个基因在CpG位点的DNA甲基化的平均值作为甲基化表达。通过平均一个基因上所有CNV变异的拷贝数来提取CNV特征。
S2:构建深度神经网络;
本发明中构建深度神经网络的步骤包括:
S201:对多组学表达数据X进行编码生成压缩特征z=E(X),
将压缩特征解码后产生新的特征X’,并计算解码后的数据恢复损失Lr,所述损失函数表达式为:
S202:定义生存风险函数,所述生存风险函数表示癌症患者在时间设定时间t之前的存活率;
所述生存风险函数表示为:S(t)=Pr(T>t)
其中,T是收集到患者的生存时间;
t时刻的生存风险函数:
S203:利用生存风险函数构建比例风险函数,所述比例风险函数为:
λ(t|x)=λ
S204:利用比例风险函数构建最大似然函数,通过最大似然函数得出初步预后风险预测损失函数,
所述最大似然函数可以表示为:
所述初步预后风险预测损失函数则可表示为:
S205:将数据恢复损失Lr加入初步预后风险预测损失函数构建最终损失函数,最终损失函数表示为:l
在本发明中利用TCGA癌症公共数据集获得公开的癌症多组学数据Xp和患者临床信息Yp对神经网络进行与训练,其中患者临床信息Yp包括患者的生存时间t及其状态st,st=1表示患者在此时间点已经死亡,st=0表示患者在此时间尚未死亡。
在进行与训练之前先对TCGA癌症公共数据集的数据进行预处理,即缺失值超过20%的基因和样本都被删除,之后其余缺失值根据中位数法进行填补。
S3:将预处理后的现有公共数据集的癌症多组学数据X
具体过程为:
在深度神经网络中编码产生的压缩特征为z,z=E(X),数据解码后的产生的新特征X’,则可表示为:x′=D(E(x)),并计算解码后的数据恢复损失Lr,所述损失函数表达式为:
计算深度神经网络中的预测风险的损失:
构建最终损失函数:l
通过随机梯度下降算法优化模型,更新深度神经网络权重θ,得到基于公共数据集的预训练网络Np。
S4:将目标癌症患者的临床数据Y及其多组学表达数据X,对网络N
如图2所示,在仿真实验中,我们测试了不同改进机制对肿瘤预后预测性能的提升效果:无迁移学习的Cox神经网络(Deep_surv),结合两种损失函数的深度Cox网络(Deep_Cox),使用预训练数据集的transfer-Cox神经网络(trans_Cox)和我们提出的方法TRCN。不同数量的训练数据得到的C-index值如图2所示。从图2中可以看出,Deep_surv在每个数据集中C-index的值都是最低的,而使用综合损失函数的Deep_Cox的性能比Cox好,但比其他方法差。与Deep_surv相比,Deep_Cox提高了平均3.7%的C-index指数,但不如trans_Cox_all(13.8%)和TRCN(17.9%)明显。与trans_Cox相比,TRCN得到的三种模拟数据的C-index指数分别提高了3.3%、4.2%、2.9%。这些结果表明,整合损失是一种有效的提高预测性能的方法,并且预先训练的模型可以为学习任务带来更多有用的信息。
表1不同方法预测膀胱癌预后风险的C-index值
在表1中,本实施例比较了通过现有不同方法预测膀胱癌(真实数据)预后风险的准确性,其中包括四种传统方法和四种基于深度学习的方法。在这些传统方法中,简单Cox方法的C-index最低(0.525)表现最差,而具有弹性网络正则化(Cox-elastic net)的Cox的C-index最高值为0.561。这些通过传统方法获得的C-index值比通过基于深度学习的方法获得的C-index值小得多。在基于深度学习的方法中,使用自动编码器(AE-Cox)重建的功能的Cox模型的性能要优于具有深度神经网络(Deep_surv)的Cox模型。没有迁移学习机制的TRCN的C-index值高于Deep_surv和AE-Cox,这证明了本发明中提出的组合损失函数的机制可以带来准确性上的提升。而TRCN在这些方法中获得的最高C-index值表明,迁移学习有助于提高模型学习的性能。
本实施例还进行了基于多组学数据预测患者风险的消融研究,来探讨不同组学数据对预测准确性的贡献如表2所示。
表2不同组学数据在预测膀胱癌预后风险的贡献
结果表明,当使用单一类型的组学数据时,mRNA的C-index表现最好,为0.624,miRNA的C-index最低,为0.552。CNV和DNA甲基化分别排在第二位和第三位。而当我们试图从TRCN的四种组学数据中剔除一种类型时,剔除mRNA导致C-index值从0.643下降到0.599,降幅最大。排除miRNA后,C-index下降幅度最小,为0.09。这些结果表明,在膀胱癌预后预测中,mRNA数据起着最重要的作用,而miRNA的贡献最小。
S5:利用XGboost算法选取目标癌症患者的Importance系数前200个基因特征,改进风险预测网络N
步骤S5所述的最终的风险预测模型表示:
其中,X
下面对本实施例建立的最终的风险预测模型进行验证分析:
本实施例中下载了GEO中的四个膀胱癌数据集作为独立测试,验证基于XGboost方法构建的模型的稳健性:GSE13507包含忠北国立大学医院收集的165位原发性膀胱癌患者的RNA-seq数据和生存信息。Dana-Farber癌症研究所在GSE31684中共享了93位膀胱癌患者的数据。GSE32894包含来自瑞典隆德大学SCIBLU基因组学中心的224位膀胱癌患者的信息。GSE42876包含了辅仁大学收集的有关43例膀胱癌患者的信息。
表3展示了独立验证的结果,可以看出,这四组数据的C-index值都大于0.6,验证了我们模型预测患者风险的准确性,而不同风险组间的p值都小于0.05,说明了不同风险组之间存在显著差异。这些结果证明了用XGboost算法构建的预测模型在这四个数据集上取得了良好的效果。
表3基于的XGboost风险预测模型在4个GEO数据集上的独立检验结果
根据预测预患者风险的中位值可将患者分为高危组和低危组。之后根据不同风险组进行差异表达分析,寻找影响预后的差异表达基因。根据结果一共鉴定出244个基因,其中下调基因90个,上调基因154个(图3A)。其中相关系数最高的前20个差异基因在图3A中额外标注。根据这些差异基因的表达绘制的热图如图3B所示。经文献回顾,已有104个基因被证实与膀胱癌相关。除了已知的癌症基因外,我们的结果还揭示了140个尚未被充分研究的影响膀胱癌预后的潜在基因。
利用这244个基因,我们进行了KEGG通路分析,以查明差异表达基因的富集通路。总共发现38个KEGG通路(2个下调通路和36个上调通路)与膀胱癌的预后相关。考虑到上调的途径比下调的途径要多得多,我们仅在图3(c)中显示基因数量>4的通路。代谢通路是癌症中的常见途径之一,因此它所包含的差异基因最多(n=12)。而在这些通路中,PI3K-Akt信号通路的p值最低。PI3K-Akt信号传导通路是在调节细胞周期中重要的细胞内信号传导通路,通过调节PI3K-Akt信号传导途径可以抑制人膀胱癌细胞的生长。此外,我们还发现了MAPK信号通路,Ras信号通路,PPAR信号通路,蛋白聚糖和癌症通路等。MAPK信号通路也被证明可以影响膀胱癌患者的治疗。这些结果进一步证明TRCN预测的癌症结局具有生物学意义。
显然,本发明的上述实施例仅仅是为清楚地说明本发明所作的举例,而并非是对本发明的实施方式的限定。对于所属领域的普通技术人员来说,在上述说明的基础上还可以做出其它不同形式的变化或变动。这里无需也无法对所有的实施方式予以穷举。凡在本发明的精神和原则之内所作的任何修改、等同替换和改进等,均应包含在本发明权利要求的保护范围之内。
- 一种基于多组学数据预测癌症患者预后风险的深度学习方法
- 一种基于大数据深度学习方法的低阻层精准预测方法与装置
