禾谷缢管蚜实时荧光定量PCR检测的内参基因及其扩增引物和应用
文献发布时间:2023-06-19 10:52:42
技术领域
本发明涉及蚜科昆虫内参基因的筛选,尤其涉及禾谷缢管蚜内参基因的筛选,本发明进一步涉及它们作为内参基因应用于禾谷缢管蚜不同发育阶段、不同地理种群、不同身体部位、不同翅型以及不同处理条件下基因表达的实时荧光定量PCR检测中的用途,属于禾谷缢管蚜内参基因的筛选及应用领域。
背景技术
禾谷缢管蚜Rhopalosiphum padi(L.)属半翅目(Hemiptera)蚜科(Aphididae),是重要的小麦害虫之一,其广泛分布于世界各小麦种植区。禾谷缢管蚜在中国各麦区均有分布,1980年以来是黄淮麦区和长江中下游麦区的重要害虫,但近些年来,表现出成灾区向北方扩散且逐渐加重的趋势。目前,中国禾谷缢管蚜的防治仍以化学防治为主,但该虫为典型的R-对策害虫,具有个体小、内禀增长率高、世代周期短、繁殖率高、环境适应性强等多样的适应机制,以及无性和有性生殖共存的繁殖方式,能够快速的适应复杂多变的环境,即使遭遇恶劣的外界环境变化,如低温、暴雨、农药使用等导致其种群数量降低,但当环境条件适宜时,种群数量则能够在短时间得以迅速恢复。随着长年杀虫剂的大量、不合理使用,已造成禾谷缢管蚜对多种、不同类型杀虫剂敏感性下降,其抗药性水平逐年上升,使得防治难度不断增加。由于防治问题的存在,人们对麦蚜抗药性问题日益关注,禾谷缢管蚜抗药性的研究也日益深入。
在昆虫基因转录水平的研究中,实时荧光定量PCR技术(qRT-PCR)是一种检测特定基因mRNA表达水平和转录分析的有效手段。qRT-PCR技术具有实时检测、特异性强、快速灵敏、准确定量的优点,实现了传统PCR从定性到精准定量的飞跃。进行qRT-PCR试验时,内参基因必须在给定的实验条件下稳定表达。为促进基因表达研究,获得更准确的表达量数据,筛选相对稳定的内参基因是必要的基础工作。理想的内参基因要求不受外界任何环境因素影响,在不同实验条件下均能稳定表达。但是,大量研究表明,不存在绝对稳定表达的基因(Zhu,X.,Yuan,M.,Shakeel,M.,Zhang,Y.,Wang,S.,Wang,X.,Zhan,S.,Kang,T.and Li,J.(2014)Selection and evaluation of reference genes for expression analysisusing qRT-PCR in the beet armyworm Spodoptera exigua(Hübner)(Lepidoptera:Noctuidae).PlOS ONE,9,e84730.)。
在昆虫样本体内,同一基因在不同种类昆虫中的功能、表达水平存在差异。同样,候选内参基因的表达情况也会因为不同昆虫物种或不同实验条件而存在差异。随着qRT-PCR技术的广泛应用,研究发现,昆虫样品种类和实验条件的不同,持家基因的表达量在某些特定条件下会发生较大的变化,如果盲目地借鉴,可能影响最终定量结果的准确性。
发明内容
本发明的目的之一是提供禾谷缢管蚜的内参基因,尤其是提供禾谷缢管蚜不同发育阶段,不同地理种群,不同身体部位,不同翅型以及不同处理条件下能够进行稳定表达的内参基因。
本发明的目的之二提供扩增所述内参基因的特异性定量引物。
本发明的主要目的之三将应用所述的禾谷缢管蚜的内参基因以及特异性定量引物采用实时荧光定量PCR方法对于禾谷缢管蚜不同发育阶段,不同地理种群,不同身体部位稳定,不同翅型或不同处理条件下基因表达进行实时荧光定量PCR分析。
本发明的上述目的主要是通过以下技术方案来实现的:
本发明收集不同发育阶段、不同地理种群、不同身体部位、不同翅型以及经抗生素、杀虫剂、饥饿、温度等处理的禾谷缢管蚜样品,提取供试材料的总RNA,反转录合成cDNA,然后以各处理为材料进行荧光定量PCR验证,采用ΔCt、geNorm、Normfinder和BestKeeper四种数据分析软件,结合网页工具RefFinder(https://www.heartcure.com.au/reffinder)评估内参基因的稳定性,从而筛选出禾谷缢管蚜在上述条件下最稳定表达的内参基因,结果发现通过实时荧光定量PCR检测表明,禾谷缢管蚜的持家基因EF-1α、β-actin、AK、TATA、GAPDH、GST、RPL13、RPS6、RPS18、18S、28S在某一种或几种试验条件下能够稳定表达,其中,β-actin或GAPDH基因片段适宜作为内参基因用于禾谷缢管蚜功能基因或不同发育阶段或杀虫剂处理下基因表达的实时荧光定量PCR检测;AK或TATA基因片段适宜作为内参基因用于禾谷缢管蚜功能基因或不同地理种群基因表达的定量PCR检测;RPS18或RPL13基因片段适宜作为内参基因用于禾谷缢管蚜功能基因或不同身体部位基因表达的实时荧光定量PCR检测;TATA或GAPDH基因片段适宜作为内参基因用于禾谷缢管蚜功能基因或不同翅型基因表达的实时荧光定量PCR检测;EF-1α或RPS6基因片段适宜作为内参基因用于禾谷缢管蚜功能基因或抗生素处理条件下基因表达的实时荧光定量PCR检测;GAPDH、TATA或RPS18基因片段适宜作为内参基因用于禾谷缢管蚜功能基因或饥饿处理条件下基因表达的实时荧光定量PCR检测;TATA、RPS6或AK基因片段适宜作为内参基因用于禾谷缢管蚜功能基因或不同温度处理条件下基因表达的实时荧光定量PCR检测。
本发明进一步提供了EF-1α、β-actin、AK、TATA、GAPDH、GST、RPL13、RPS6、RPS18、18S或28S基因作为内参基因采用实时荧光定量PCR检测时能够特异性扩增的定量引物,其中,用于扩增EF-1α基因的定量引物的核苷酸序列为SEQ ID No.1和SEQ ID No.2所示;用于扩增β-actin基因的定量引物的核苷酸序列为SEQ ID No.3和SEQ ID No.4所示;用于扩增AK基因的定量引物的核苷酸序列为SEQ ID No.5和SEQ ID No.6所示;用于扩增TATA基因的定量引物的核苷酸序列为SEQ ID No.7和SEQ ID No.8所示;用于扩增GAPDH基因的定量引物的核苷酸序列为SEQ ID No.9和SEQ ID No.10所示;用于扩增GST基因的定量引物的核苷酸序列为SEQ ID No.11和SEQ ID No.12所示;用于扩增RPL13基因的定量引物的核苷酸序列为SEQ ID No.13和SEQ ID No.14所示;用于扩增RPS6基因的定量引物的核苷酸序列为SEQ ID No.15和SEQ ID No.16所示;用于扩增RPS18基因的定量引物的核苷酸序列为SEQID No.17和SEQ ID No.18所示;用于扩增18S基因的定量引物的核苷酸序列为SEQ IDNo.19和SEQ ID No.20所示;用于扩增28S基因的定量引物的核苷酸序列为SEQ ID No.21和SEQ ID No.22所示。
本发明在设计上述EF-1α(SEQ ID No.23)、β-actin(SEQ ID No.24)、AK(SEQ IDNo.25)、TATA(SEQ ID No.26)、GAPDH(SEQ ID No.27)、GST(SEQ ID No.28)、RPL13(SEQ IDNo.29)、RPS6(SEQ ID No.30)、RPS18(SEQ ID No.31)、18S(SEQ ID No.32)或28S(SEQ IDNo.33)基因的特异性定量引物的过程中,是在对这些基因序列进行充分分析研究的基础上,避免颈环结构和非特异扩增,并保证引物的扩增效率和检测的灵敏度。
本发明进一步提供了禾谷缢管蚜在不同发育阶段、不同地理种群、不同身体部位、不同翅型、经不同处理(抗生素、杀虫剂、饥饿、温度)条件下的禾谷缢管蚜样品中基因表达的荧光定量PCR扩增方法,包括:
一种禾谷缢管蚜不同发育阶段或杀虫剂处理下的基因表达的实时荧光定量PCR检测方法,包括:提取禾谷缢管蚜基因组总RNA,进行RT反应反应得到产物第一链cDNA,以产物第一链cDNA作为模板进行实时荧光定量PCR扩增,荧光染料为SYBR Green,其中实时荧光定量PCR扩增的定量引物为用于扩增β-actin基因的定量引物,其核苷酸序列为SEQ ID No.3和SEQ ID No.4所示;或者为用于扩增GAPDH基因的定量引物,其核苷酸序列为SEQ ID No.9和SEQ ID No.10所示。
一种禾谷缢管蚜不同地理种群的基因表达的实时荧光定量PCR检测方法,包括:提取禾谷缢管蚜基因组总RNA,进行RT反应得到产物第一链cDNA,以产物第一链cDNA作为模板进行实时荧光定量PCR扩增,荧光染料为SYBR Green;其中,实时荧光定量PCR扩增的定量引物为用于扩增AK基因的定量引物,其核苷酸序列为SEQ ID No.5和SEQ ID No.6所示;或者为用于扩增TATA基因的定量引物,其核苷酸序列为SEQ ID No.7和SEQ ID No.8所示。
一种禾谷缢管蚜不同身体部位的基因表达的实时荧光定量PCR检测方法,包括:提取禾谷缢管蚜基因组总RNA,进行RT反应反应得到产物第一链cDNA,以产物第一链cDNA作为模板进行实时荧光定量PCR扩增,荧光染料为SYBR Green,其中,实时荧光定量PCR扩增的定量引物为用于扩增RPS18基因的定量引物,其核苷酸序列为SEQ ID No.17和SEQ ID No.18所示;或者为用于扩增RPL13基因的定量引物,其核苷酸序列为SEQ ID No.13和SEQ IDNo.14所示。
一种禾谷缢管蚜不同翅型的基因表达的实时荧光定量PCR检测方法,包括:提取禾谷缢管蚜基因组总RNA,进行RT反应反应得到产物第一链cDNA,以产物第一链cDNA作为模板进行实时荧光定量PCR扩增,荧光染料为SYBR Green,其中,实时荧光定量PCR扩增的定量引物为用于扩增TATA基因的定量引物,其核苷酸序列为SEQ ID No.7和SEQ ID No.8所示;或者为用于扩增GAPDH基因的定量引物,其核苷酸序列为SEQ ID No.9和SEQ ID No.10所示。
一种禾谷缢管蚜抗生素处理条件下的基因表达的实时荧光定量PCR检测方法,包括:提取禾谷缢管蚜基因组总RNA,进行RT反应反应得到产物第一链cDNA,以产物第一链cDNA作为模板进行实时荧光定量PCR扩增,荧光染料为SYBR Green,其中,实时荧光定量PCR扩增的定量引物为用于扩增EF-1α基因的定量引物,其核苷酸序列为SEQ ID No.1和SEQ IDNo.2所示;或者为用于扩增RPS6基因的定量引物,其核苷酸序列为SEQ ID No.15和SEQ IDNo.16所示。
一种禾谷缢管蚜饥饿处理条件下的基因表达的实时荧光定量PCR检测方法,包括:提取禾谷缢管蚜基因组总RNA,进行RT反应反应得到产物第一链cDNA,以产物第一链cDNA作为模板进行实时荧光定量PCR扩增,荧光染料为SYBR Green,其中,实时荧光定量PCR扩增的定量引物为用于扩增GAPDH基因的定量引物,其核苷酸序列为SEQ ID No.9和SEQ ID No.10所示;或者为用于扩增TATA基因的定量引物,其核苷酸序列为SEQ ID No.7和SEQ ID No.8所示;或者为用于扩增RPS18基因的定量引物,其核苷酸序列为SEQ ID No.17和SEQ IDNo.18所示。
一种禾谷缢管蚜温度处理条件下的基因表达的实时荧光定量PCR检测方法,包括:提取禾谷缢管蚜基因组总RNA,进行RT反应反应得到产物第一链cDNA,以产物第一链cDNA作为模板进行实时荧光定量PCR扩增,荧光染料为SYBR Green,其中,实时荧光定量PCR扩增的定量引物为用于扩增TATA基因的定量引物,其核苷酸序列为SEQ ID No.7和SEQ ID No.8所示;或者为用于扩增RPS6基因的定量引物,其核苷酸序列为SEQ ID No.15和SEQ ID No.16所示;或者为用于扩增AK基因的定量引物,其核苷酸序列为SEQ ID No.5和SEQ ID No.6所示。
所述实时荧光定量PCR扩增的反应条件优选为:95℃预变性3min后;先运行40个循环的95℃变性5sec,60℃退火15sec,再运行溶解曲线阶段的60-95℃。
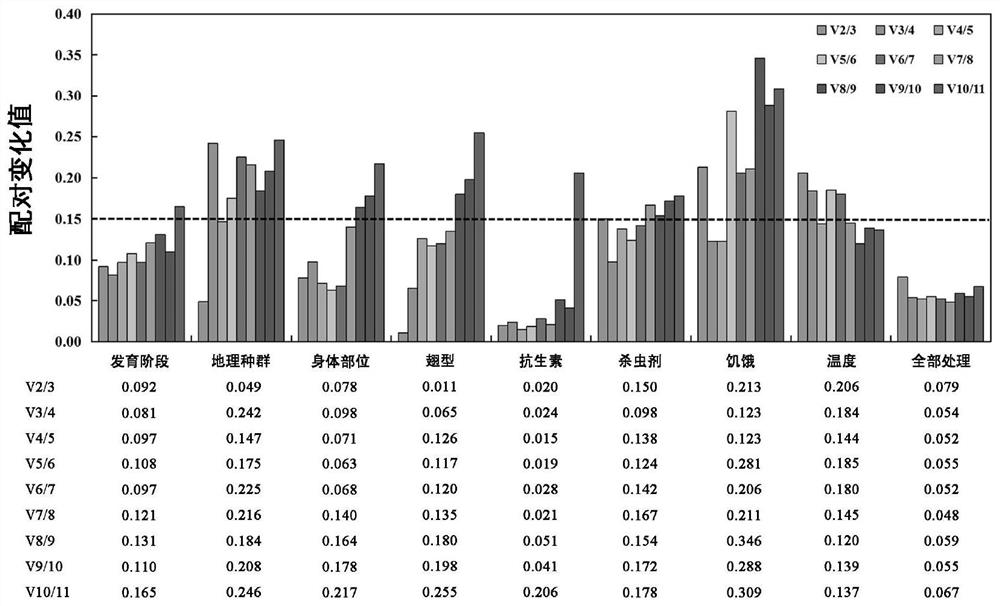
本发明利用荧光定量PCR技术,建立了禾谷缢管蚜在不同试验条件下(发育阶段、地理种群、身体部位、翅型、以及抗生素、杀虫剂、饥饿、温度处理)内参基因的筛选方法;各候选内参基因定量PCR数据输入ΔCt、BestKeeper、Normfinder和geNorm软件以及RefFinder网站进行分析。荧光定量PCR获得的Ct值直接用于BestKeeper分析,其根据标准差值和变异系数值评估候选内参基因的稳定性。Normfinder和geNorm分析时需要的Ct值是通过公式2
本发明进一步设计相应的特异性的定量检测引物,本发明所提供的定量检测引物特异性强,优化了PCR扩增程序,大大的提高了检测效率、缩短了检测时间,提高了检测结果的可信度。本发明进一步建立基于SYBR Green I染料技术的RT-PCR方法,从而为禾谷缢管蚜的EF-1α、β-actin、AK、TATA、GAPDH、GST、RPL13、RPS6、RPS18、18S和28S作为内参基因,在利用qPCR对禾谷缢管蚜功能基因或不同发育时期、地理种群、身体部位、翅型基因表达,或作为抗生素处理,或作为杀虫剂处理,或作为饥饿处理,或作为不同温度处理下基因表达研究中提供有用的方法。
附图说明
图1禾谷缢管蚜11个候选内参基因EF-1α、β-actin、AK、TATA、GAPDH、GST、RPL13、RPS6、RPS18、18S和28S的熔解曲线。
图2禾谷缢管蚜11个候选内参基因在不同试验条件下的表达水平;各候选内参基因表达水平由该试验条件下的原始Ct值表示;箱型上下线分别为上四分位数和下四分位数,中线为中位数,中垂线的上下限分别为最大值和最小值;(A)禾谷缢管蚜11个候选内参基因在不同发育阶段条件下的表达水平;(B)禾谷缢管蚜11个候选内参基因在不同地理种群条件下的表达水平;(C)禾谷缢管蚜11个候选内参基因在不同身体部位条件下的表达水平;(D)禾谷缢管蚜11个候选内参基因在有翅或无翅条件下的表达水平;(E)禾谷缢管蚜11个候选内参基因在抗生素条件下的表达水平;(F)禾谷缢管蚜11个候选内参基因在杀虫剂处理下的表达水平;(G)禾谷缢管蚜11个候选内参基因在饥饿处理条件下的表达水平;(H)禾谷缢管蚜11个候选内参基因在不同温度条件下的表达水平。
图3基于geNorm计算方法确定禾谷缢管蚜不同试验条件下内参基因的最佳数目。根据每个柱形图上面的V值来确定试验所需的最适内参基因数目,判定标准为当Vn/Vn+1<0.15时,则最合适内参基因的数量是n个;而如果Vn/Vn+1>0.15时,则最合适内参基因的数量是n+1个。
图4基于RefFinder方法对在不同试验条件下11个内参基因稳定性排序;其中,横坐标为内参基因名称,从左至右稳定性排序,越靠近坐标轴右侧稳定性越差;纵坐标代表RefFinder计算出的内参基因的稳定值;(A)禾谷缢管蚜11个候选内参基因在不同发育阶段条件下的稳定性排序;(B)禾谷缢管蚜11个候选内参基因在不同地理种群条件下的稳定性排序;(C)禾谷缢管蚜11个候选内参基因在不同身体部位条件下的稳定性排序;(D)禾谷缢管蚜11个候选内参基因在有翅或无翅条件下的稳定性排序;(E)禾谷缢管蚜11个候选内参基因在抗生素条件下的稳定性排序;(F)禾谷缢管蚜11个候选内参基因在杀虫剂处理下的稳定性排序;(G)禾谷缢管蚜11个候选内参基因在饥饿处理条件下的稳定性排序;(H)禾谷缢管蚜11个候选内参基因在不同温度条件下的稳定性排序。
具体实施方式
下面结合具体实施例来进一步描述本发明,本发明的优点和特点将会随着描述而更为清楚。但是应理解所述实施例仅是范例性的,不对本发明的范围构成任何限制。本领域技术人员应该理解的是,在不偏离本发明的精神和范围下可以对本发明技术方案的细节和形式进行修改或替换,但这些修改或替换均落入本发明的保护范围。
实施例1禾谷缢管蚜候选内参基因的筛选和确定
1.禾谷缢管蚜总RNA提取
取无翅成蚜6头样品放入干净1.5ml无RNase离心管中,在液氮中研磨至细粉状,加入1ml Trizol液;立即用振荡器振荡1min,使之快速溶解于裂解液中;室温10min期间不断震荡,让虫体充分裂解;加入200ul氯仿,快速震荡使溶液充分混匀至乳白色,静置3min,4℃,12,000rpm,离心15min;取400ul上清至含1ml无水乙醇的离心管中,静置5min,于-80℃冻存,冷冻4-5小时;将有RNA沉淀的离心管从冰柜中取出,4℃,12 000rpm,离心10min;小心弃上清,加入1ml的75%乙醇,用移液枪轻轻吹起沉淀,4℃,7 500rpm,离心10min;弃上清,4℃,7500rpm,空管离心2min,超净台晾干;加入30ul RNase free水溶解,晃动离心管,使之溶解;利用超微量紫外分光光度计TGem spectrophotometer Plus检测所提总RNA浓度,3个生物学重复中只保留1.9﹤A260/280﹤2.1的样品进行下一步反转录。
2.反转录
使用PrimeScript
(1)在200μL RNase-free的PCR管中按照表1的体系加入试剂:
表1第一步反应体系
(2)轻柔混匀后置于PCR仪42℃孵育2min。
(3)在第一步的反应液中按照表2的体系加入试剂:
表2第二步反应体系
(4)混匀离心后置于PCR仪37℃15min,85℃5s。合成的cDNA于-20℃保存备用。
3.荧光定量PCR扩增引物合成
11个候选内参基因,其中EF-1α、18S和28S序列信息来自GeneBank数据库(http://www.ncbi.nlm.nih.gov/genbank/);β-actin和GAPDH序列信息来自文献(王康.细胞色素P450和羧酸酯酶在禾谷缢管蚜抗药性中的作用[D].西北农林科技大学,2019.);AK、TATA、GST、RPL13、RPS6、RPS18来自禾谷缢管蚜转录组测序信息,利用Primer 5.0软件设计持家基因引物,oligo7软件分析引物结构特征。各基因的荧光定量PCR的引物序列见表3。
表3 11个候选内参基因的PCR扩增的引物序列
4.荧光定量PCR
以RT得到的第一链cDNA为模板进行荧光定量PCR检测。基于SYBR Green I染料的qPCR的反应体系为:2×Talent qPCR PreMix 10μL、上、下游引物(10.0μM)各0.6μL,cDNA0.6μL,ddH
5.引物的扩增效率
使用禾谷缢管蚜无翅成虫cDNA进行一系列5倍梯度,稀释后作为模板,进行实时荧光定量PCR,制作引物的扩增效率标准曲线。引物扩增效率计算方式如下:扩增效率E=(10
6.实验结果
本发明按照定量PCR反应程序将禾谷缢管蚜的11个候选基因EF-1α、β-actin、AK、TATA、GAPDH、GST、RPL13、RPS6、RPS18、18S或28S进行PCR扩增,凝胶电泳验证,荧光定量PCR扩增,获得熔解曲线(melting curve)(图1)判断各引物的特异性。
以5倍梯度稀释cDNA模板,进行实时荧光定量PCR检测,获得一系列模板的Ct值,以Ct值为纵坐标,以稀释倍数为横坐标绘制禾谷缢管蚜11个候选基因的拟合曲线,将拟合曲线的斜率带入引物扩增效率计算方程式:扩增效率E=(10
表4候选内参基因引物的扩增效率
所用11个候选基因引物的扩增效率在100%左右,说明该引物的扩增效率很高,结合熔解曲线判断扩增产物的特异性,理想的熔解曲线应该为单峰曲线。从本实验的引物熔解曲线分析得出,所选禾谷缢管蚜的11个候选基因检测显现单峰曲线,无引物二聚体等非特异性扩增产生,说明本发明涉及的荧光定量PCR扩增引物扩增条带单一,特异性强,没有非特异性扩增出现。综上说明所选择的EF-1α、β-actin、AK、TATA、GAPDH、GST、RPL13、RPS6、RPS18、18S以及28S这11个基因符合作为禾谷缢管蚜的候选内参基因。
实施例2不同发育阶段禾谷缢管蚜内参基因表达定量的PCR检测
1.禾谷缢管蚜总RNA提取
所选试虫为宁夏石嘴山禾谷缢管蚜,饲养条件为20±1℃,湿度60%~80%,光照:黑暗=16h:8h。每管收集无翅禾谷缢管蚜约40头1龄若虫,30头2龄若虫,20头3龄若虫,20头4龄若虫,20头无翅成蚜,每一发育时期均收集3管,编号后放入液氮中速冻,于-80℃冰箱保存。利用Trizol(TIANGEN,Beijing,China)提取麦长管蚜的总RNA,具体方法同实施例1。
2.反转录
使用PrimeScript
3.荧光定量PCR扩增引物合成
荧光定量PCR扩增引物合成同实施例1。
4.荧光定量PCR
荧光定量PCR同实施例1。
5.实验结果
本发明按照定量PCR反应程序将禾谷缢管蚜的不同发育阶段的基因组RNA样品进行荧光定量PCR扩增,得出Ct值(图2(A)),然后将Ct值输入ΔCt、BestKeeper、Normfinder和geNorm软件以及RefFinder网站进行分析,对内参基因进行稳定性排序。
利用ΔCt软件计算11个候选内参基因的Ct值平均标准偏差(SD),SD越小,表明基因稳定性越好。将计算结果按稳定性由高到低进行排序,依次为:β-actin>GAPDH>RPS18>TATA>RPL13>AK>EF-1α>RPS6>18S>28S>GST(表5),即在ΔCt软件的算法中,β-actin的稳定性最好。
将qRT-PCR反应获得的Ct值导入BestKeeper软件中,根据各候选基因标准偏差值(SD)来确定候选基因的相对稳定性,SD值越小,表明基因稳定性越好。将计算结果按稳定性由高到低进行排序,依次为:28S>18S>AK>TATA>β-actin>GAPDH>RPS18>RPS6>EF-1α>RPL13>GST(表5),即在BestKeeper软件的算法中,28S的稳定性最好。
表5不同发育阶段下内参基因的稳定性
按NormFinder软件要求,将处理好的数据导入软件进行计算,得出各候选内参基因的稳定值M,M值越大说明基因的稳定性越差;反之则说明基因的表达稳定性越好。经计算获得11个候选内参基因的M值,按稳定性由高到低依次为:β-actin>GAPDH>RPS18>TATA>RPL13>AK>RPS6>EF-1α>18S>28S>GST(表5),即在NormFinder软件的算法中,β-actin的稳定性最好。处理好的数据导入geNorm软件计算各候选内参基因的稳定值(M值),M值越小,表示稳定性越好。计算结果表明,11个候选内参基因稳定性从高到低依次为:β-actin=GAPDH>RPS18>TATA>EF-1α>RPL13>RPS6>AK>18S>28S>GST(表5),即在geNorm软件的算法中,β-actin与GAPDH的稳定性最好。GeNorm软件分析表明(图3),除了V10/11大于0.15外,其余Vn/Vn+1均小于0.15,最适内参基因个数为2个。RefFinder对11个候选基因综合稳定性排序结果显示(请参见图4(A)),稳定性从大到小依次是:β-actin、GAPDH、RPS18、TATA、AK、28S、18S、RPL13、EF-1α、RPS6、GST;其中,横坐标为内参基因名称,从左至右稳定性排序,越靠近坐标轴右侧稳定性越差。纵坐标代表RefFinder计算出的内参基因的稳定值。所以,根据上述分析结果,最终确定β-actin和GAPDH基因片段可作为内参基因能够应用于不同发育阶段禾谷缢管蚜基因表达定量PCR的检测。
实施例3不同地理种群禾谷缢管蚜内参基因表达定量的PCR检测
1.禾谷缢管蚜总RNA提取
采集宁夏石嘴山(39°01'58.81”N,106°37'75.60'E),河北廊坊(39°8'9.8”N,116°10'4.05”E)两个不同地理种群禾谷缢管蚜无翅成虫,液氮速冻,于-80℃冰箱保存。利用TRizol(TIANGEN,Beijing,China)提取麦长管蚜的总RNA,具体方法同实施例1。
2.反转录
使用PrimeScript
3.荧光定量PCR扩增引物
荧光定量PCR扩增引物同实施例1。
4.荧光定量PCR
荧光定量PCR同实施例1。
5.实验结果
本发明按照定量PCR反应程序将禾谷缢管蚜的不同地理种群的基因组RNA样品进行荧光定量PCR扩增,得出Ct值(图2(B)),然后将Ct值输入ΔCt、BestKeeper、Normfinder和geNorm软件以及RefFinder网站进行分析,对内参基因进行稳定性排序。
表6不同地理种群下内参基因的稳定性
利用ΔCt软件计算11个候选内参基因的Ct值平均标准偏差(SD),SD越小,表明基因稳定性越好。将计算结果按稳定性由高到低进行排序,依次为:AK>TATA>RPS18>EF-1α>18S>GAPDH>RPS6>GST>RPL13>28S>β-actin(表6),即在ΔCt软件的算法中,AK的稳定性最好。
将qRT-PCR反应获得的Ct值导入BestKeeper软件中,根据各候选基因标准偏差值(SD)来确定候选基因的相对稳定性,SD值越小,表明基因稳定性越好。将计算结果按稳定性由高到低进行排序,依次为:18S>RPS6>RPS18>TATA>AK>28S>RPL13>EF-1α>GST>GAPDH>β-actin(表6),即在BestKeeper软件的算法中,18S的稳定性最好。
按NormFinder软件要求,将处理好的数据导入软件进行计算,得出各候选内参基因的稳定值M,M值越大说明基因的稳定性越差;反之则说明基因的表达稳定性越好。经计算获得11个候选内参基因的M值,按稳定性由高到低依次为:AK>RPS18>TATA>EF-1α>18S>GAPDH>GST>RPS6>RPL13>28S>β-actin(表6),即在NormFinder软件的算法中,AK的稳定性最好。处理好的数据导入geNorm软件计算各候选内参基因的稳定值(M值),M值越小,表示稳定性越好。计算结果表明,11个候选内参基因稳定性从高到低依次为:AK=TATA>RPS18>EF-1α>18S>RPS6>RPL13>GAPDH>GST>28S>β-actin(表6),即在geNorm软件的算法中,AK与TATA的稳定性最好。GeNorm软件分析表明(图3),V2/3和V4/V5小于0.15,其余Vn/Vn+1均小于0.15,最适内参基因个数为2个。RefFinder对11个候选基因综合稳定性排序结果显示(请参见图4(B)),稳定性从大到小依次是:AK、TATA、RPS18、18S、EF-1α、RPS6、GAPDH、RPL13、GST、28S、β-actin;其中,横坐标为内参基因名称,从左至右稳定性排序,越靠近坐标轴右侧稳定性越差。纵坐标代表RefFinder计算出的内参基因的稳定值。所以,根据上述分析结果,最终确定AK和TATA基因片段可作为内参基因,应于不同地理种群禾谷缢管蚜基因表达定量PCR的检测。
实施例4不同身体部位禾谷缢管蚜内参基因表达定量的PCR检测
1.禾谷缢管蚜总RNA提取
所选试虫为宁夏石嘴山禾谷缢管蚜无翅成虫,饲养条件为20±1℃,湿度60%-~80%,光照:黑暗=16h:8h。在4℃冷冻2h后置于干净的一次性无菌平皿中,平皿中倒入一定量琼脂糖,凝固后放入冰箱冷冻,作为解剖平面。在超净台中操作,点燃酒精灯,一手持解剖针,一手持镊子,用镊子的尖端分离出头部、胸部和腹部,分别放入装有1ml Trizol试剂的干净1.5ml无RNase离心管中,每一部位收集6管,液氮速冻,于-80℃冰箱保存。利用TRizol(TIANGEN,Beijing,China)提取禾谷缢管蚜的总RNA,具体方法同实施例1。
2.反转录
使用PrimeScript
3.荧光定量PCR扩增引物合成
荧光定量PCR扩增引物合成同实施例1。
4.荧光定量PCR
荧光定量PCR同实施例1。
5.实验结果
本发明按照定量PCR反应程序将禾谷缢管蚜的不同身体部位的基因组RNA样品进行荧光定量PCR扩增,得出Ct值(图2(C)),然后将Ct值输入ΔCt、BestKeeper、Normfinder和geNorm软件以及RefFinder网站进行分析,对内参基因进行稳定性排序。
利用ΔCt软件计算11个候选内参基因的Ct值平均标准偏差(SD),SD越小,表明基因稳定性越好。将计算结果按稳定性由高到低进行排序,依次为:RPL13>GAPDH>RPS18>β-actin>EF-1α>TATA>RPS6>28S>GST=AK>18S(表7),即在ΔCt软件的算法中,RPL13的稳定性最好。
将qRT-PCR反应获得的Ct值导入BestKeeper软件中,根据各候选基因标准偏差值(SD)来确定候选基因的相对稳定性,SD值越小,表明基因稳定性越好。将计算结果按稳定性由高到低进行排序,依次为:28S>18S>GAPDH>RPS18=β-actin>EF-1α=RPS6>RPL13=TATA>GST>AK(表7),即在BestKeeper软件的算法中,28S的稳定性最好。
表7不同身体部位下内参基因的稳定性
按NormFinder软件要求,将处理好的数据导入软件进行计算,得出各候选内参基因的稳定值M,M值越大说明基因的稳定性越差;反之则说明基因的表达稳定性越好。经计算获得11个候选内参基因的M值,按稳定性由高到低依次为:RPS18=RPL13>GAPDH>β-actin>TATA>EF-1α>RPS6>28S>AK>GST>18S(表7),即在NormFinder软件的算法中,RPS18和RPL13的稳定性最好。处理好的数据导入geNorm软件计算各候选内参基因的稳定值(M值),M值越小,表示稳定性越好。计算结果表明,11个候选内参基因稳定性从高到低依次为:RPS18=EF-1α>RPL13>GAPDH>β-actin>TATA>RPS6>GST>AK>28S>18S(表7),即在geNorm软件的算法中,RPS18与EF-1α的稳定性最好。GeNorm软件分析表明(图3),V8/9至V10/11大于0.15,其余Vn/Vn+1均小于0.15,最适内参基因个数为2个。RefFinder对11个候选基因综合稳定性排序结果显示(请参见图4(C)),稳定性从大到小依次是:RPS18、RPL13、GAPDH、EF-1α、β-actin、28S、TATA、RPS6、18S、GST、AK;其中,横坐标为内参基因名称,从左至右稳定性排序,越靠近坐标轴右侧稳定性越差。纵坐标代表RefFinder计算出的内参基因的稳定值。所以,根据上述分析结果,最终确定RPS18和RPL13基因片段可作为内参基因,应于不同身体部位禾谷缢管蚜基因表达定量PCR的检测。
实施例5有翅或无翅禾谷缢管蚜内参基因表达定量的PCR检测
1.禾谷缢管蚜总RNA提取
室内饲养宁夏石嘴山禾谷缢管蚜,饲养条件为20±1℃,湿度60%-~80%,光照:黑暗=16h:8h,挑选成虫有翅类型30头,无翅类型30头,分别装入干净1.5ml无RNase离心管中,编号后放入液氮中速冻,于-80℃冰箱短期保存。利用TRizol(TIANGEN,Beijing,China)提取禾谷缢管蚜的总RNA,具体方法同实施例1。
2.反转录
使用PrimeScript
3.荧光定量PCR扩增引物合成
荧光定量PCR扩增引物合成同实施例1。
4.荧光定量PCR
荧光定量PCR同实施例1。
5.实验结果
本发明按照定量PCR反应程序将有翅或无翅禾谷缢管蚜的基因组RNA样品进行荧光定量PCR扩增,得出Ct值(图2(D)),然后将Ct值输入ΔCt、BestKeeper、Normfinder和geNorm软件以及RefFinder网站进行分析,对内参基因进行稳定性排序。
利用ΔCt软件计算11个候选内参基因的Ct值平均标准偏差(SD),SD越小,表明基因稳定性越好。将计算结果按稳定性由高到低进行排序,依次为:GAPDH>TATA>RPS6>RPL13>RPS18>β-actin>EF-1α>AK>GST>28S>18S(表8),即在ΔCt软件的算法中,GAPDH的稳定性最好。
表8不同翅型下内参基因的稳定性
将qRT-PCR反应获得的Ct值导入BestKeeper软件中,根据各候选基因标准偏差值(SD)来确定候选基因的相对稳定性,SD值越小,表明基因稳定性越好。将计算结果按稳定性由高到低进行排序,依次为:28S>β-actin>RPS18>TATA>RPS6>GAPDH>18S>RPL13>EF-1α>AK>GST(表8),即在BestKeeper软件的算法中,28S的稳定性最好。
按NormFinder软件要求,将处理好的数据导入软件进行计算,得出各候选内参基因的稳定值M,M值越大说明基因的稳定性越差;反之则说明基因的表达稳定性越好。经计算获得11个候选内参基因的M值,按稳定性由高到低依次为:TATA>GAPDH>RPS6>RPL13>RPS18>β-actin>EF-1α>AK>28S>GST>18S(表8),即在NormFinder软件的算法中,TATA的稳定性最好。处理好的数据导入geNorm软件计算各候选内参基因的稳定值(M值),M值越小,表示稳定性越好。计算结果表明,11个候选内参基因稳定性从高到低依次为:TATA=GAPDH>RPS18>β-actin>RPS6>RPL13>EF-1α>AK>GST>28S>18S(表8),即在geNorm软件的算法中,TATA与GAPDH的稳定性最好。GeNorm软件分析表明(图3),V8/9至V10/11大于0.15,其余Vn/Vn+1均小于0.15,最适内参基因个数为2个。RefFinder对11个候选基因综合稳定性排序结果显示(请参见图4(D)),稳定性从大到小依次是:TATA、GAPDH、RPS6、RPS18、β-actin、RPL13、28S、EF-1α、AK、GST、18S;其中,横坐标为内参基因名称,从左至右稳定性排序,越靠近坐标轴右侧稳定性越差。纵坐标代表RefFinder计算出的内参基因的稳定值。所以,根据上述分析结果,最终确定TATA或GAPDH基因片段可作为内参基因,应用于有翅或无翅禾谷缢管蚜基因表达定量PCR的检测。
实施例6抗生素处理禾谷缢管蚜内参基因表达定量的PCR检测
1.禾谷缢管蚜总RNA提取
所选试虫为宁夏石嘴山禾谷缢管蚜无翅成虫,将抗生素利福平溶解在蔗糖(30%)水中,配置浓度为50ug/mL利福平溶液,以双通玻璃管(高33mm,直径22mm)作为饲养器,一端用2层PF膜封口膜封住,含有抗生素的蔗糖(30%)水作为食物夹在两层封口膜之间,另一端朝下,放置于铺有湿润滤纸的培养皿中,每管饲养20头,无抗生素的蔗糖(30%)水作为对照,饲养条件为20±1℃,湿度60%-~80%,光周期为光照:黑暗=16:8h,处理48h后收集样品,放入液氮中速冻,于-80℃冰箱保存。利用TRizol(TIANGEN,Beijing,China)提取禾谷缢管蚜的总RNA,具体方法同实施例1。
2.反转录
使用PrimeScript
3.荧光定量PCR扩增引物合成
荧光定量PCR扩增引物合成同实施例1。
4.荧光定量PCR
荧光定量PCR同实施例1。
5.实验结果
本发明按照定量PCR反应程序将抗生素处理禾谷缢管蚜的基因组RNA样品进行荧光定量PCR扩增,得出Ct值(图2(E)),然后将Ct值ΔCt、BestKeeper、Normfinder和geNorm软件以及RefFinder网站进行分析,对内参基因进行稳定性排序。
利用ΔCt软件计算11个候选内参基因的Ct值平均标准偏差(SD),SD越小,表明基因稳定性越好。将计算结果按稳定性由高到低进行排序,依次为:RPS6>EF-1α>RPS18=RPL13=GAPDH>TATA>β-actin>28S>AK>18S>GST(表9),即在ΔCt软件的算法中,RPS6的稳定性最好。
表9抗生素处理下内参基因的稳定性
将qRT-PCR反应获得的Ct值导入BestKeeper软件中,根据各候选基因标准偏差值(SD)来确定候选基因的相对稳定性,SD值越小,表明基因稳定性越好。将计算结果按稳定性由高到低进行排序,依次为:28S>18S>β-actin>AK>RPS6>GAPDH>TATA>RPL13>EF-1α>RPS18>GST(表9),即在BestKeeper软件的算法中,28S的稳定性最好。
按NormFinder软件要求,将处理好的数据导入软件进行计算,得出各候选内参基因的稳定值M,M值越大说明基因的稳定性越差;反之则说明基因的表达稳定性越好。经计算获得11个候选内参基因的M值,按稳定性由高到低依次为:EF-1α>RPS6>RPL13>RPS18>TATA>GAPDH>β-actin>AK>28S>18S>GST(表9),即在NormFinder软件的算法中,EF-1α的稳定性最好。处理好的数据导入geNorm软件计算各候选内参基因的稳定值(M值),M值越小,表示稳定性越好。计算结果表明,11个候选内参基因稳定性从高到低依次为:EF-1α=RPS18>RPL13>RPS6>GAPDH>TATA>β-actin>28S>18S>AK>GST(表9),即在geNorm软件的算法中,EF-1α与RPS18的稳定性最好。GeNorm软件分析表明(图3),除V10/11大于0.15,其余Vn/Vn+1均小于0.15,最适内参基因个数为2个。RefFinder对11个候选基因综合稳定性排序结果显示(请参见图4(E)),稳定性从大到小依次是:EF-1α、RPS6、RPS18、RPL13、28S、GAPDH、β-actin、TATA、18S、AK、GST;其中,横坐标为内参基因名称,从左至右稳定性排序,越靠近坐标轴右侧稳定性越差。纵坐标代表RefFinder计算出的内参基因的稳定值。所以,根据上述分析结果,最终确定EF-1α或RPS6基因片段可作为内参基因,应用于抗生素处理禾谷缢管蚜基因表达定量PCR的检测。
实施例7杀虫剂处理禾谷缢管蚜内参基因表达定量的PCR检测
1.禾谷缢管蚜总RNA提取
所选杀虫剂包括高效氯氰菊酯、吡虫啉和氟啶虫胺腈,采用浸苗法,将麦苗全被浸泡在供试药剂中,停留5秒后取出室温干燥10-15min,放入含有湿润滤纸的玻璃培养皿中,接入试虫,处理24h。所选试虫为宁夏石嘴山禾谷缢管蚜无翅成虫,饲养条件为20±1℃,湿度60%~80%,光照:黑暗=16h:8h。各药剂浓度为生测所得的LC
2.反转录
使用PrimeScriptTM RT Reagent Kit with gDNA Eraser反转录试剂盒(Takara公司)进行反转录,具体操作步骤同实施例1中的反转录。
3.荧光定量PCR扩增引物合成
荧光定量PCR扩增引物合成同实施例1。
4.荧光定量PCR
荧光定量PCR同实施例1。
5.实验结果
本发明按照定量PCR反应程序将杀虫剂处理禾谷缢管蚜的基因组RNA样品进行荧光定量PCR扩增,得出Ct值(图2(F)),然后将Ct值输入ΔCt、BestKeeper、Normfinder和geNorm软件以及RefFinder网站进行分析,对内参基因进行稳定性排序。
利用ΔCt软件计算11个候选内参基因的Ct值平均标准偏差(SD),SD越小,表明基因稳定性越好。将计算结果按稳定性由高到低进行排序,依次为:GAPDH>β-actin=TATA>28S>EF-1α>RPL13>AK>18S>RPS18>RPS6>GST(表10),即在ΔCt软件的算法中,GAPDH的稳定性最好。
将qRT-PCR反应获得的Ct值导入BestKeeper软件中,根据各候选基因标准偏差值(SD)来确定候选基因的相对稳定性,SD值越小,表明基因稳定性越好。将计算结果按稳定性由高到低进行排序,依次为:18S>β-actin>28S>RPS18>TATA=GAPDH>EF-1α>RPL13>AK>RPS6>GST(表10),即在BestKeeper软件的算法中,18S的稳定性最好。
按NormFinder软件要求,将处理好的数据导入软件进行计算,得出各候选内参基因的稳定值M,M值越大说明基因的稳定性越差;反之则说明基因的表达稳定性越好。经计算获得11个候选内参基因的M值,按稳定性由高到低依次为:GAPDH>β-actin>TATA>28S>EF-1α>RPL13>AK>18S>RPS18>RPS6>GST(表10),即在NormFinder软件的算法中,GAPDH的稳定性最好。
表10杀虫剂处理下内参基因的稳定性
处理好的数据导入geNorm软件计算各候选内参基因的稳定值(M值),M值越小,表示稳定性越好。计算结果表明,11个候选内参基因稳定性从高到低依次为:GAPDH=TATA>β-actin>28S>18S>RPS18>EF-1α>RPL13>AK>RPS6>GST(表10),即在geNorm软件的算法中,GAPDH与TATA的稳定性最好。GeNorm软件分析表明(图3),V2/V3等于0.15,最适内参基因个数为2个。RefFinder对11个候选基因综合稳定性排序结果显示(请参见图4(F)),稳定性从大到小依次是:GAPDH、β-actin、TATA、28S、18S、EF-1α、RPS18、RPL13、AK、RPS6、GST;其中,横坐标为内参基因名称,从左至右稳定性排序,越靠近坐标轴右侧稳定性越差。纵坐标代表RefFinder计算出的内参基因的稳定值。所以,根据上述的分析结果,最终确定GAPDH或β-actin基因片段可作为内参基因,应用于杀虫剂处理禾谷缢管蚜基因表达定量PCR的检测。实施例8饥饿处理禾谷缢管蚜内参基因表达定量的PCR检测
1.禾谷缢管蚜总RNA提取
所选试虫为宁夏石嘴山禾谷缢管蚜无翅成虫,接到培养皿中,皿中不放入任何食物源,置于智能人工气候箱中,培养条件为20±1℃,湿度60%-~80%,光照:黑暗=16h:8h,32h后观察,死亡率约为10%,收集剩余活虫,对照组的培养皿中放入小麦麦苗作为食物源,样品收集后放入液氮中速冻,于-80℃冰箱保存。利用TRizol(TIANGEN,Beijing,China)提取禾谷缢管蚜的总RNA,具体方法同实施例1。
2.反转录
使用PrimeScript
3.荧光定量PCR扩增引物合成
荧光定量PCR扩增引物合成同实施例1。
4.荧光定量PCR
荧光定量PCR同实施例1。
5.实验结果
本发明按照定量PCR反应程序将饥饿处理禾谷缢管蚜的基因组RNA样品进行荧光定量PCR扩增,得出Ct值(图2(G)),然后将Ct值输入ΔCt、BestKeeper、Normfinder和geNorm软件以及RefFinder网站进行分析,对内参基因进行稳定性排序。
利用ΔCt软件计算11个候选内参基因的Ct值平均标准偏差(SD),SD越小,表明基因稳定性越好。将计算结果按稳定性由高到低进行排序,依次为:GAPDH>TATA>β-actin>RPS18>EF-1α>RPS6>RPL13>AK>GST>28S>18S(表11),即在ΔCt软件的算法中,GAPDH的稳定性最好。
表11饥饿处理下内参基因的稳定性
将qRT-PCR反应获得的Ct值导入BestKeeper软件中,根据各候选基因标准偏差值(SD)来确定候选基因的相对稳定性,SD值越小,表明基因稳定性越好。将计算结果按稳定性由高到低进行排序,依次为:28S>18S>GST>RPS18>β-actin>GAPDH>TATA>EF-1α>RPS6>RPL13>AK(表11),即在BestKeeper软件的算法中,28S的稳定性最好。
按NormFinder软件要求,将处理好的数据导入软件进行计算,得出各候选内参基因的稳定值M,M值越大说明基因的稳定性越差;反之则说明基因的表达稳定性越好。经计算获得11个候选内参基因的M值,按稳定性由高到低依次为:GAPDH>RPS18>β-actin>TATA>EF-1α>RPS6>RPL13>GST>AK>28S>18S(表11),即在NormFinder软件的算法中,GAPDH的稳定性最好。处理好的数据导入geNorm软件计算各候选内参基因的稳定值(M值),M值越小,表示稳定性越好。计算结果表明,11个候选内参基因稳定性从高到低依次为:GAPDH=TATA>β-actin>RPS18>EF-1α>RPS6>RPL13>AK>GST>28S>18S(表11),即在geNorm软件的算法中,GAPDH与TATA的稳定性最好。GeNorm软件分析表明(图3),除V3/4和V4/5小于0.15,其余Vn/Vn+1均大于0.15,最适内参基因个数为3个。RefFinder对11个候选基因综合稳定性排序结果显示(请参见图4(G)),稳定性从大到小依次是:GAPDH、TATA、RPS18、β-actin、EF-1α、28S、RPS6、GST、18S、RPL13、AK;其中,横坐标为内参基因名称,从左至右稳定性排序,越靠近坐标轴右侧稳定性越差。纵坐标代表RefFinder计算出的内参基因的稳定值。所以,根据上述的分析结果,最终确定GAPDH、TATA和RPS18基因片段可作为内参基因,应用于饥饿处理禾谷缢管蚜基因表达定量PCR的检测。
实施例9不同温度处理禾谷缢管蚜内参基因表达定量的PCR检测
1.禾谷缢管蚜总RNA提取
取宁夏石嘴山禾谷缢管蚜无翅成虫分为5组,分别置于不同温度的智能人工气候箱中(4℃、10℃、15℃、20℃、30℃),处理24h,湿度60%-~80%,光照:黑暗=16h:8h,之后取出观察,每种温度下均无试虫死亡。每一温度处理收集6管,每管约20头,放入液氮中速冻,于-80℃冰箱保存。利用TRizol(TIANGEN,Beijing,China)提取禾谷缢管蚜的总RNA,具体方法同实施例1。
2.反转录
使用PrimeScript
3.荧光定量PCR扩增引物合成
荧光定量PCR扩增引物合成同实施例1。
4.荧光定量PCR
荧光定量PCR同实施例1。
5.实验结果
本发明按照定量PCR反应程序将不同温度处理禾谷缢管蚜的基因组RNA样品进行荧光定量PCR扩增,得出Ct值(图2(H)),然后将Ct值输入ΔCt、BestKeeper、Normfinder和geNorm软件以及RefFinder网站进行分析,对内参基因进行稳定性排序。
利用ΔCt软件计算11个候选内参基因的Ct值平均标准偏差(SD),SD越小,表明基因稳定性越好。将计算结果按稳定性由高到低进行排序,依次为:TATA>RPS6>AK>GAPDH>β-actin>28S>18S>RPL13>RPS18>>GST>EF-1α(表12),即在ΔCt软件的算法中,TATA的稳定性最好。
将qRT-PCR反应获得的Ct值导入BestKeeper软件中,根据各候选基因标准偏差值(SD)来确定候选基因的相对稳定性,SD值越小,表明基因稳定性越好。将计算结果按稳定性由高到低进行排序,依次为:18S>TATA>RPS6>28S>RPS18>GAPDH>AK>β-actin>RPL13>EF-1α>GST(表12),即在BestKeeper软件的算法中,18S的稳定性最好。
表12不同温度处理下内参基因的稳定性
按NormFinder软件要求,将处理好的数据导入软件进行计算,得出各候选内参基因的稳定值M,M值越大说明基因的稳定性越差;反之则说明基因的表达稳定性越好。经计算获得11个候选内参基因的M值,按稳定性由高到低依次为:TATA>AK>RPS6>GAPDH>β-actin>28S>18S>RPL13>RPS18>GST>EF-1α(表12),即在NormFinder软件的算法中,TATA的稳定性最好。处理好的数据导入geNorm软件计算各候选内参基因的稳定值(M值),M值越小,表示稳定性越好。计算结果表明,11个候选内参基因稳定性从高到低依次为:TATA=RPS6>AK>28S>18S>GAPDH>RPS18>β-actin>RPL13>GST>EF-1α(表12),即在geNorm软件的算法中,TATA与RPS6的稳定性最好。GeNorm软件分析表明(图3),V2/V3大于0.15,最适内参基因个数为3个。RefFinder对11个候选基因综合稳定性排序结果显示(请参见图4(H)),稳定性从大到小依次是:TATA、RPS6、AK、18S、GAPDH、28S、β-actin、RPS18、RPL13、GST、EF-1α;其中,横坐标为内参基因名称,从左至右稳定性排序,越靠近坐标轴右侧稳定性越差。纵坐标代表RefFinder计算出的内参基因的稳定值。所以,根据上述的分析结果,最终确定TATA、RPS6和AK基因片段可作为内参基因,应用于不同温度处理禾谷缢管蚜基因表达定量PCR的检测。
SEQUENCE LISTING
<110> 中国农业科学院植物保护研究所
<120> 禾谷缢管蚜实时荧光定量PCR检测的内参基因及其扩增引物和应用
<130> BJ-2012-201204A
<160> 33
<170> PatentIn version 3.5
<210> 1
<211> 16
<212> DNA
<213> Artifical sequence
<400> 1
ctgttgcttt cgttcc 16
<210> 2
<211> 18
<212> DNA
<213> Artifical sequence
<400> 2
gactgttcca atacctcc 18
<210> 3
<211> 20
<212> DNA
<213> Artifical sequence
<400> 3
tgagacattc aacacccctg 20
<210> 4
<211> 21
<212> DNA
<213> Artifical sequence
<400> 4
ccttcataga ttgggacagt g 21
<210> 5
<211> 16
<212> DNA
<213> Artifical sequence
<400> 5
ggaagaaggg tggtgt 16
<210> 6
<211> 16
<212> DNA
<213> Artifical sequence
<400> 6
cagcgtcagg agcata 16
<210> 7
<211> 16
<212> DNA
<213> Artifical sequence
<400> 7
tgtcggcttg acctaa 16
<210> 8
<211> 16
<212> DNA
<213> Artifical sequence
<400> 8
acaactgcca accatg 16
<210> 9
<211> 22
<212> DNA
<213> Artifical sequence
<400> 9
gctccattag ccaaggttat tc 22
<210> 10
<211> 21
<212> DNA
<213> Artifical sequence
<400> 10
cagcacctct accatctctc c 21
<210> 11
<211> 16
<212> DNA
<213> Artifical sequence
<400> 11
atgacggtta ttttgt 16
<210> 12
<211> 16
<212> DNA
<213> Artifical sequence
<400> 12
caggtctttt tgcttg 16
<210> 13
<211> 15
<212> DNA
<213> Artifical sequence
<400> 13
caaagactgg caacg 15
<210> 14
<211> 15
<212> DNA
<213> Artifical sequence
<400> 14
ccaatggtac gagca 15
<210> 15
<211> 16
<212> DNA
<213> Artifical sequence
<400> 15
actcggtgat gaatgg 16
<210> 16
<211> 16
<212> DNA
<213> Artifical sequence
<400> 16
gggcgataac aagaat 16
<210> 17
<211> 17
<212> DNA
<213> Artifical sequence
<400> 17
cacatcttgc gtatcct 17
<210> 18
<211> 17
<212> DNA
<213> Artifical sequence
<400> 18
tacattctcc agccctc 17
<210> 19
<211> 20
<212> DNA
<213> Artifical sequence
<400> 19
acgcatcttt caaatgtctg 20
<210> 20
<211> 18
<212> DNA
<213> Artifical sequence
<400> 20
tgtggtagcc gtttctca 18
<210> 21
<211> 16
<212> DNA
<213> Artifical sequence
<400> 21
aacaaccgtg attccc 16
<210> 22
<211> 16
<212> DNA
<213> Artifical sequence
<400> 22
cgccacaact cccata 16
<210> 23
<211> 227
<212> DNA
<213> Rhopalosiphum padi(L.)
<400> 23
ctgttgcttt cgttcccatc tctggatgga atggagacaa catgttggaa gtttccgaca 60
agatgtcatg gttcaaagga tggaatgttg aacgtaaaga aggaaaggct gatggtaaat 120
gtctgattga agctttagac gctatcctac cccccagtcg cccaactgac aaggctcttc 180
gtctcccact ccaggatgtc tacaaaattg gaggtattgg aacagtc 227
<210> 24
<211> 132
<212> DNA
<213> Rhopalosiphum padi(L.)
<400> 24
tgagacattc aacacccctg ctatgtacgt tgctatccaa gccgtacttt cattgtacgc 60
ttctggtcgt accactggta tcgtcttgga ttctggtgat ggtgtatctc acactgtccc 120
aatctatgaa gg 132
<210> 25
<211> 178
<212> DNA
<213> Rhopalosiphum padi(L.)
<400> 25
gaagggtggt gttcgctata ctcttctgat agcaaatcat tgctaaagaa atattgacaa 60
aagaggtact agatgactta aagaacaaaa agacaaaatt ccggatccac actttttgga 120
ttgtgtgcaa tctggatttg aaaaccatga ttctggagtt ggaatttatg ctcctgac 178
<210> 26
<211> 240
<212> DNA
<213> Rhopalosiphum padi(L.)
<400> 26
tgtcggcttg ccctaaaaac cattgcttta catgcacgta atgctgaatt taatccaaaa 60
cgatttgctg ctgtcataat gagaatacgt gaaccacgta ctacagcttt aatctttagt 120
tctggtaaaa tggtatgtac tggtgcaaaa agtgaagaag attctagact ggcggcaagg 180
aaatatgcta ggatcattca aaagctcagc tttccagcaa agtttttaga ctttaaaatt 240
<210> 27
<211> 136
<212> DNA
<213> Rhopalosiphum padi(L.)
<400> 27
gctccattag ccaaggttat tcatgacaac tttggaatta ttgagggtct tatgactact 60
gttcacgcta ccaccgccac tcaaaaaact gttgatggac cgtctggaaa attgtggaga 120
gatggtagag gtgctg 136
<210> 28
<211> 186
<212> DNA
<213> Rhopalosiphum padi(L.)
<400> 28
atgacggtta ttttgtaaat ggaaagttgt cctgggctga tatatacttt gtagcttgtt 60
tagatttttt gaactatatg gcaaaagtgg tccttttaga gagccgtcca aaattacaag 120
cgcataaaag aaaaatattg gaaatacctc aaattaaatc gtgggtcggc caagcaaaaa 180
gacctg 186
<210> 29
<211> 240
<212> DNA
<213> Rhopalosiphum padi(L.)
<400> 29
caaagactgg caacgttaca ttaagacctg gttcaatcag ccagctcgta aatacaggcg 60
ccaccaaaac cgtatcaaga aggccaaagc gttggcacct aaaccagcgg caggtccttt 120
gcgaccggtt gtacactgcc ccacgttaag atatcacact aaagttcgtg ctggtcgagg 180
ttttactctg caagagctta agggtgctgg tttaaataaa cattatgctc gtaccattgg 240
<210> 30
<211> 138
<212> DNA
<213> Rhopalosiphum padi(L.)
<400> 30
actcggtgat gaatggaaag gttatgtgct taaaatatct ggaggcaatg acaaacaagg 60
gtttccaatg aaacaaggtg ttctctctaa tgtccgagtg cgcctattat tatctaaggg 120
acattcttgt tatcgccc 138
<210> 31
<211> 148
<212> DNA
<213> Rhopalosiphum padi(L.)
<400> 31
cacatcttgc gtatcctcag taccaacatc gatggcaaac gtaaagtcat gttcgccatg 60
accgccatca aaggtattgg tagacggttt tccaacattg tgttgaaaaa ggccgatgtc 120
gacctaaaca agagggctgg agaatgta 148
<210> 32
<211> 125
<212> DNA
<213> Rhopalosiphum padi(L.)
<400> 32
acgcatcttt caaatgtctg ccttatcaac tgtcgacggt aggttccatg cctaccgtgg 60
tggtaacggg taacggggaa tcagggttcg attccggaga gggagcctga gaaacggcta 120
ccaca 125
<210> 33
<211> 101
<212> DNA
<213> Rhopalosiphum padi(L.)
<400> 33
aacaaccgtg attcccccag tagcggcgag cgaacaggga aaagcccatc gccgatcccg 60
ccgcgcactt ttgtgtcgcg gcacgtatgg gagttgtggc g 101
- 禾谷缢管蚜实时荧光定量PCR检测的内参基因及其扩增引物和应用
- 禾谷缢管蚜ACT1内参基因部分序列、克隆方法及应用
