无细胞DNA中的微卫星不稳定性检测
文献发布时间:2023-06-19 11:17:41
相关申请的交叉引用
本申请要求2018年8月31日提交的美国临时专利申请第62/726,182号、2019年3月25日提交的美国临时专利申请第62/823,578号和2019年6月4日提交的美国临时专利申请第62/857,048号的权益,并且依赖于这些美国临时专利申请的申请日期,这些美国临时专利申请的全部公开内容通过引用并入本文。
背景
重复核酸元件是在整个真核生物基因组和原核生物基因组中以多个拷贝出现的核苷酸(DNA或RNA)模式。这样的重复元件的实例包括微卫星(microsatellite)、短串联重复序列(STR)和小卫星(minisatellite)等等。微卫星通常包括少于10个碱基对的重复单元。STR通常包括在给定的核DNA的链段中通常重复数百次的2个至13个核苷酸的重复单元。STR分析是法医学分析中常用的工具。小卫星是通常具有约10个至60个碱基对的重复单元的重复元件。
特别地,微卫星是高度多态性的DNA重复区域。微卫星不稳定性(microsatelliteinstability,MSI)是一种经指南推荐的生物标志物,其用于评估预后和治疗选择,包括最近被批准用于治疗具有MSI高(MSI-H)状态的癌症的检查点抑制剂。基于血浆的下一代DNA测序(NGS)测试越来越多地用于癌症的全面基因组谱分析,然而,根据无细胞DNA(cfDNA)数据检测MSI状态的方法开发不足。此外,先前没有评估过可变肿瘤脱落对MSI检测的影响。
仍然需要可用于评估各种样品(尤其是cfDNA样品)中的重复元件不稳定性状态(包括MSI)的方法和相关方面。
概述
本申请公开了可用于确定来自患者的无细胞DNA(cfDNA)样品的微卫星和/或其他重复DNA不稳定性状态并且有助于指导疾病预后和治疗决策的方法、计算机可读介质和系统。通常,本文公开的方法的至少一部分是计算机实现的,并且获得的结果与使用更常规的基于聚合酶链式反应(PCR)的MSI评估方法所获得的结果高度一致。
从以下详细描述中,本公开内容的另外的方面和优点对本领域技术人员而言将变得明显,详细描述中仅示出和描述了本公开内容的说明性实施方案。如将意识到的,本公开内容能够具有其他且不同的实施方案,并且其若干细节能够在各种明显的方面进行修改,所有这些都不偏离本公开内容。相应地,附图和描述被认为本质上是说明性的,而不是限制性的。
在一方面,本公开内容提供了一种确定核酸样品的重复核酸不稳定性状态的方法。该方法包括(a)根据序列信息定量多于一个重复核酸基因座的每一个处存在的不同重复序列长度的数目,以产生多于一个重复核酸基因座的每一个的位点评分。序列信息来自核酸样品中重复核酸基因座的群体。该方法还包括(b)当给定的重复核酸基因座的位点评分超过给定的重复核酸基因座的位点特异性训练阈值时,将给定的重复核酸基因座识别(call)为不稳定,以产生重复核酸不稳定性评分,所述重复核酸不稳定性评分包括来自多于一个重复核酸基因座的不稳定的重复核酸基因座的数目。此外,该方法还包括(c)当重复核酸不稳定性评分超过核酸样品中重复核酸基因座的群体的群体训练阈值时,将核酸样品的重复核酸不稳定性状态分类为不稳定,从而确定核酸样品的重复核酸不稳定性状态。
在另一方面,本公开内容提供了一种确定样品(例如无细胞DNA(cfDNA)样品)的重复DNA不稳定性状态的方法。该方法包括(a)根据序列信息定量多于一个重复DNA基因座的每一个处存在的不同重复序列长度的数目,以产生多于一个重复DNA基因座的每一个的位点评分。序列信息来自样品中重复DNA基因座的群体。该方法还包括(b)对于多于一个重复DNA基因座的每一个,将给定的重复DNA基因座的位点评分与给定的重复DNA基因座的位点特异性训练阈值进行比较。该方法还包括(c)当给定的重复DNA基因座的位点评分超过给定的重复DNA基因座的位点特异性训练阈值时,将给定的重复DNA基因座识别为不稳定,以产生重复DNA不稳定性评分,所述重复DNA不稳定性评分包括来自多于一个重复DNA基因座的不稳定的重复DNA基因座的数目。此外,该方法还包括(d)当重复DNA不稳定性评分超过样品中重复DNA基因座的群体的群体训练阈值时,将样品的重复DNA不稳定性状态分类为不稳定,从而确定样品的重复DNA不稳定性状态。本文公开的方法通常至少部分是计算机实现的。
在另一方面,本公开内容提供了一种确定样品的微卫星不稳定性(MSI)状态的方法。该方法包括(a)根据序列信息定量多于一个微卫星基因座的每一个处存在的不同重复序列长度的数目,以产生多于一个微卫星基因座的每一个的位点评分,其中序列信息来自样品中微卫星基因座的群体。该方法还包括(b)对于多于一个微卫星基因座的每一个,将给定的微卫星基因座的位点评分与给定的微卫星基因座的位点特异性训练阈值进行比较。该方法还包括(c)当给定的微卫星基因座的位点评分超过给定的微卫星基因座的位点特异性训练阈值时,将给定的微卫星基因座识别为不稳定,以产生微卫星不稳定性评分,所述微卫星不稳定性评分包括来自多于一个微卫星基因座的不稳定的微卫星基因座的数目。此外,该方法还包括(d)当微卫星不稳定性评分超过样品中微卫星基因座的群体的群体训练阈值时,将样品的MSI状态分类为不稳定,从而确定样品的MSI状态。
在另一方面,本公开内容提供了一种确定样品的微卫星不稳定性(MSI)状态的方法。该方法包括(a)接收来自样品中微卫星基因座的群体的序列信息,和(b)根据序列信息定量多于一个微卫星基因座的每一个处存在的不同重复序列长度的数目,以产生多于一个微卫星基因座的每一个的位点评分。该方法还包括(c)对于多于一个微卫星基因座的每一个,将给定的微卫星基因座的位点评分与给定的微卫星基因座的位点特异性训练阈值进行比较。该方法还包括(d)当给定的微卫星基因座的位点评分超过给定的微卫星基因座的位点特异性训练阈值时,将给定的微卫星基因座识别为不稳定,以产生微卫星不稳定性评分,所述微卫星不稳定性评分包括来自多于一个微卫星基因座的不稳定的微卫星基因座的数目。此外,该方法还包括(e)当微卫星不稳定性评分超过样品中微卫星基因座的群体的群体训练阈值时,将样品的MSI状态分类为不稳定,从而确定样品的MSI状态。
在另一方面,本公开内容提供了一种鉴定用于治疗受试者的疾病的一种或更多种定制疗法的方法。该方法包括(a)根据序列信息定量多于一个微卫星基因座的每一个处存在的不同重复序列长度的数目,以产生多于一个微卫星基因座的每一个的位点评分,其中序列信息来自样品中微卫星基因座的群体。该方法还包括(b)对于多于一个微卫星基因座的每一个,将给定的微卫星基因座的位点评分与给定的微卫星基因座的位点特异性训练阈值进行比较。该方法还包括(c)当给定的微卫星基因座的位点评分超过给定的微卫星基因座的位点特异性训练阈值时,将给定的微卫星基因座识别为不稳定,以产生微卫星不稳定性评分,所述微卫星不稳定性评分包括来自多于一个微卫星基因座的不稳定的微卫星基因座的数目。该方法还包括(d)当微卫星不稳定性评分超过样品中微卫星基因座的群体的群体训练阈值时,将样品的MSI状态分类为不稳定,以鉴定不稳定的样品。此外,该方法还包括(e)将样品的微卫星不稳定性状态与用一种或更多种疗法加索引的(indexed)一种或更多种比较用结果(comparator result)进行比较,以鉴定用于治疗受试者的疾病的一种或更多种定制疗法。
在另一方面,本公开内容提供了一种治疗受试者的疾病的方法。该方法包括(a)根据序列信息定量多于一个微卫星基因座的每一个处存在的不同重复序列长度的数目,以产生多于一个微卫星基因座的每一个的位点评分,其中序列信息来自样品中微卫星基因座的群体。该方法还包括(b)对于多于一个微卫星基因座的每一个,将给定的微卫星基因座的位点评分与给定的微卫星基因座的位点特异性训练阈值进行比较。该方法还包括(c)当给定的微卫星基因座的位点评分超过给定的微卫星基因座的位点特异性训练阈值时,将给定的微卫星基因座识别为不稳定,以产生微卫星不稳定性评分,所述微卫星不稳定性评分包括来自多于一个微卫星基因座的不稳定的微卫星基因座的数目。该方法还包括(d)当微卫星不稳定性评分超过样品中微卫星基因座的群体的群体训练阈值时,将样品的MSI状态分类为不稳定,以鉴定不稳定的样品。该方法还包括(e)将样品的微卫星不稳定性状态与用一种或更多种疗法加索引的一种或更多种比较用结果进行比较,以鉴定用于治疗受试者的疾病的一种或更多种定制疗法。此外,该方法还包括(f)当样品的微卫星不稳定性状态和比较用结果之间存在基本匹配时,向受试者施用至少一种经鉴定的定制疗法,从而治疗受试者的疾病。
在另一方面,本公开内容提供了一种治疗受试者的疾病的方法。该方法包括向受试者施用一种或更多种定制疗法,从而治疗受试者的疾病,其中定制疗法已经通过以下被鉴定:(a)根据序列信息定量多于一个微卫星基因座的每一个处存在的不同重复序列长度的数目,以产生多于一个微卫星基因座的每一个的位点评分,其中序列信息来自样品中微卫星基因座的群体。该方法还包括(b)对于多于一个微卫星基因座的每一个,将给定的微卫星基因座的位点评分与给定的微卫星基因座的位点特异性训练阈值进行比较。该方法还包括(c)当给定的微卫星基因座的位点评分超过给定的微卫星基因座的位点特异性训练阈值时,将给定的微卫星基因座识别为不稳定,以产生微卫星不稳定性评分,所述微卫星不稳定性评分包括来自多于一个微卫星基因座的不稳定的微卫星基因座的数目。该方法还包括(d)当微卫星不稳定性评分超过样品中微卫星基因座的群体的群体训练阈值时,将样品的MSI状态分类为不稳定,以鉴定不稳定的样品。该方法还包括(e)将样品的微卫星不稳定性状态与用一种或更多种疗法加索引的一种或更多种比较用结果进行比较。此外,该方法还包括(f)当样品的微卫星不稳定性状态和比较用结果之间存在基本匹配时,鉴定用于治疗受试者的疾病的一种或更多种定制疗法。
在一些实施方案中,多于一个微卫星基因座的位点评分包括似然性评分(likelihood score)。在这些实施方案中的某些实施方案中,似然性评分包括基于概率对数似然性的评分,所述基于概率对数似然性的评分将从样品中许多体细胞来源的核酸片段(在一些实施方案中-cfDNA片段)获得的生物信号与由样品中样品收集后伪象产生的噪声区分开。在一些实施方案中,方法包括使用至少两个参数确定来自样品的序列信息中的个体微卫星基因座的基于概率对数似然性的评分,其中至少第一参数包括等位基因频率,并且至少第二参数包括至少一种错误模式。通常,等位基因频率包括来自样品的序列信息中包括不同重复序列长度的核酸的频率。在一些实施方案中,至少一种错误模式包括随机错误模式和链特异性错误模式。在某些实施方案中,多于一个微卫星基因座的位点评分包括以下(a)和(b)之间的差异或比率:(a)度量所观察到的序列支持给定的微卫星基因座是稳定的零假设的评分,和(b)度量所观察到的序列支持给定的微卫星基因座是不稳定的备择假设(alternate hypothesis)的评分。在一些实施方案中,多于一个微卫星基因座的位点评分使用以下中的一种或更多种产生:似然准则、对数似然准则、后验概率准则、赤池信息量准则(Akaike information criterion,AIC)、贝叶斯信息准则(Bayesian informationcriterion)和/或类似物。
在一些实施方案中,多于一个微卫星基因座的位点评分包括基于赤池信息量准则(AIC)的位点评分,所述基于赤池信息量准则(AIC)的位点评分测试多于一个微卫星基因座处体细胞得失位(indel)的存在。在这些实施方案中的某些实施方案中,给定的基于AIC的位点评分使用以下公式计算:
AIC=k-对数似然性,
其中k是模型中使用的参数的数目。任选地,方法包括使用最大似然估计(MLE)来估计模型的参数。在这些实施方案中的一些实施方案中,方法包括使用Nelder-Mead算法来确定MLE。在某些实施方案中,方法包括使用以下公式计算模型的零假设评分(例如,度量所观察到的序列支持给定的微卫星基因座是稳定的零假设的评分):
AIC
其中AIC
AIC
其中AIC
ΔAIC=AIC
在这些实施方案中的一些实施方案中,γ包括:(a)在测序读段中观察到的微卫星长度比起始核酸分子链的预期微卫星长度长一个重复单元的读段水平错误率;和/或(b)在测序读段中观察到的微卫星长度比起始核酸分子链的预期微卫星长度短一个重复单元的读段水平错误率。在这些实施方案中的某些实施方案中,β包括:(a)有义链的预期微卫星长度比核酸起始分子的预期微卫星长度长一个重复单元的链水平错误率;(b)反义链的预期微卫星长度比核酸起始分子的预期微卫星长度长一个重复单元的链水平错误率;(c)有义链的预期微卫星长度比核酸起始分子的预期微卫星长度短一个重复单元的链水平错误率;和/或(d)反义链的预期微卫星长度比核酸起始分子的预期微卫星长度短一个重复单元的链水平错误率。通常,方法包括当给定的微卫星基因座的位点评分在统计学上超过给定的微卫星基因座的位点特异性训练阈值时,将给定的微卫星基因座识别为不稳定。
在一些实施方案中,基于AIC的位点评分使用以下公式来计算:
AIC=2(k-对数似然性),
其中k是模型中使用的参数的数目。在这些实施方案中,AIC
为了清楚的目的,在使用公式AIC=2(k-对数似然性)确定基于AIC的评分的实施方案中,用于将位点分类为不稳定的位点特异性阈值会是先前实施方案中使用的位点特异性阈值的两倍,在先前的实施方案中,基于AIC的评分使用以下公式确定:AIC=k-对数似然性。
通常,样品(例如,cfDNA样品)的突变等位基因分数(MAF)被估计。在这些实施方案中的一些实施方案中,样品(例如,cfDNA样品)的肿瘤分数被估计。在某些实施方案中,肿瘤分数包括样品(例如,cfDNA样品)中的核酸中经鉴定的所有体细胞突变的最大突变等位基因分数(MAF)。在一些实施方案中,肿瘤分数低于样品(例如,cfDNA样品)中所有核酸的约0.05%、约0.1%、约0.2%、约0.5%、约1%、约2%、约3%、约4%、约5%、约6%、约7%、约8%、约9%、约10%、约11%、约12%、约13%、约14%或约15%。在一些实施方案中,多于一个微卫星基因座包括微卫星基因座的群体的全部,而在其他实施方案中,多于一个微卫星基因座包括微卫星基因座的群体的子集。在某些实施方案中,方法包括根据来自一个或更多个训练DNA样品中的微卫星基因座的群体的序列信息确定位点特异性训练阈值和/或群体训练阈值。在这些实施方案中的一些实施方案中,训练DNA样品包括非肿瘤cfDNA训练样品和/或来自一种或更多种肿瘤类型的DNA。
在一些实施方案中,方法包括在样品中核酸的约0.1%-0.4%肿瘤分数的检测限(LOD)处的至少约94%的灵敏度。在一些实施方案中,方法包括对样品中非肿瘤DNA的至少约99%的分析特异性。在某些实施方案中,跨越约1%至约15%的肿瘤分数范围,样品的所确定的MSI状态包括与使用基于PCR的MSI评估技术确定的样品的相应MSI状态至少约90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的一致性。在这些实施方案中的一些实施方案中,一致性是100%。在一些实施方案中,方法包括当微卫星不稳定性评分是大于约1个、约2个、约3个、约4个、约5个、约6个、约7个、约8个、约9个、约10个、约15个、约20个、约30个、约40个、约50个或更多个来自多于一个微卫星基因座的不稳定的微卫星基因座时,将样品的MSI状态分类为MSI-高(MSI-H)。在某些实施方案中,方法包括当不稳定的微卫星基因座的数目构成多于一个微卫星基因座的约0.1%、约1%、约2%、约3%、约4%、约5%、约10%、约15%、约20%或约25%时,将样品的MSI状态分类为MSI-高(MSI-H)。在一些实施方案中,不同重复序列长度的数目包括多于一个微卫星基因座的每一个处存在的每个不同重复序列长度的频率。
在各种实施方案中,本公开内容包括选择用于治疗受试者的疾病的定制疗法的方法,和/或治疗受试者的疾病的方法。在这些实施方案中的一些实施方案中,疾病包括癌症,所述癌症包括选自由以下组成的组但不限于以下的至少一种肿瘤类型:胆道癌、膀胱癌、移行细胞癌、尿路上皮癌、脑癌、神经胶质瘤、星形细胞瘤、乳腺癌、化生癌、宫颈癌、宫颈鳞状细胞癌、直肠癌、结肠直肠癌、结肠癌、遗传性非息肉性结肠直肠癌、结肠直肠腺癌、胃肠间质瘤(GIST)、子宫内膜癌、子宫内膜间质肉瘤、食管癌、食管鳞状细胞癌、食管腺癌、眼黑素瘤、葡萄膜黑素瘤、胆囊癌、胆囊腺癌、肾细胞癌、透明细胞肾细胞癌(clear cell renalcell carcinoma)、移行细胞癌、尿路上皮癌、肾母细胞瘤、白血病、急性淋巴细胞白血病(ALL)、急性髓性白血病(AML)、慢性淋巴细胞白血病(CLL)、慢性髓性白血病(CML)、慢性粒单核细胞白血病(CMML)、肝癌(liver cancer)、肝上皮癌(liver carcinoma)、肝细胞瘤、肝细胞癌、胆管癌、肝母细胞瘤、肺癌、非小细胞肺癌(NSCLC)、间皮瘤、B细胞淋巴瘤、非霍奇金淋巴瘤、弥漫性大B细胞淋巴瘤、套细胞淋巴瘤、T细胞淋巴瘤、非霍奇金淋巴瘤、前体T淋巴母细胞淋巴瘤/白血病、外周T细胞淋巴瘤、多发性骨髓瘤、鼻咽癌(NPC)、神经母细胞瘤、口咽癌、口腔鳞状细胞癌、骨肉瘤、卵巢癌、胰腺癌、胰腺导管腺癌、假乳头状肿瘤、腺泡细胞癌、前列腺癌、前列腺腺癌、皮肤癌、黑素瘤、恶性黑素瘤、皮肤黑素瘤、小肠癌、胃癌(stomach cancer)、胃上皮癌(gastric carcinoma)、胃肠间质瘤(GIST)、子宫癌和子宫肉瘤。
在一些实施方案中,疗法包括至少一种免疫疗法(例如,检查点抑制剂抗体、自体细胞毒性T细胞、个性化癌症疫苗等)。在某些实施方案中,例如,免疫疗法包括针对以下的抗体:PD-1、PD-2、PD-L1、PD-L2、CTLA-4、OX40、B7.1、B7He、LAG3、CD137、KIR、CCR5、CD27、CD40或CD47。在一些实施方案中,免疫疗法包括施用针对至少一种肿瘤类型的促炎细胞因子。任选地,免疫疗法包括施用针对至少一种肿瘤类型的T细胞。
在一些实施方案中,方法包括从受试者获得样品。基本上任何样品类型任选地被使用。在某些实施方案中,例如,样品是组织、血液、血浆、血清、痰、尿液、精液、阴道液、粪便、滑液、脊髓液、唾液和/或类似物。通常,受试者是哺乳动物受试者(例如,人类受试者)。在一些实施方案中,样品是血液。在一些实施方案中,样品是血浆。在一些实施方案中,样品是血清。在一些实施方案中,样品包括无细胞DNA(即,cfDNA样品)。在一些实施方案中,cfDNA样品包括循环肿瘤核酸。
在某些实施方案中,方法包括接收从样品产生的序列信息,其中序列信息包括来自样品中微卫星基因座的群体的测序读段。在一些实施方案中,方法包括扩增样品中的核酸的一个或更多个区段以产生至少一个经扩增的核酸。在某些实施方案中,方法包括对来自样品的核酸进行测序以产生序列信息。在一些实施方案中,样品可以是cfDNA样品。在这些实施方案中,序列信息包括来自cfDNA样品中微卫星基因座的群体的cfDNA测序读段。在一些实施方案中,序列信息从样品中核酸的靶向区段获得,其中靶向区段通过在测序之前选择性地富集样品中核酸的一个或更多个区域来获得。在这些实施方案中的一些实施方案中,方法包括在测序之前扩增所获得的靶向区段。在这些实施方案中,方法通常包括在扩增之前将一个或更多个包含分子条形码的衔接子附接到核酸。在一些实施方案中,方法包括在测序之前经由扩增来附接一个或更多个样品索引。基本上任何核酸测序技术都任选地用于或适用于执行本文公开的方法。例如,测序任选地选自靶向测序、内含子测序、外显子组测序、全基因组测序和/或类似测序。在一些实施方案中,测序是靶向测序。在一些实施方案中,方法包括对样品的核酸中至少约50个、约100个、约150个、约200个、约250个、约500个、约750个、约1,000个、约1,500个、约2,000个或更多个靶向基因组区域进行测序,以产生序列信息。
在另一方面,本公开内容提供了一种系统,该系统包括控制器,所述控制器包括或能够访问包含非暂时性计算机可执行指令的计算机可读介质,该非暂时性计算机可执行指令当被至少一个电子处理器执行时,执行至少以下:(a)接收来自样品中微卫星基因座的群体的序列信息;(b)根据序列信息定量多于一个微卫星基因座的每一个处存在的不同重复序列长度的数目,以产生多于一个微卫星基因座的每一个的位点评分;(c)对于多于一个微卫星基因座的每一个,将给定的微卫星基因座的位点评分与给定的微卫星基因座的位点特异性训练阈值进行比较;(d)当给定的微卫星基因座的位点评分超过给定的微卫星基因座的位点特异性训练阈值时,将给定的微卫星基因座识别为不稳定,以产生微卫星不稳定性评分,所述微卫星不稳定性评分包括来自多于一个微卫星基因座的不稳定的微卫星基因座的数目;和(e)当微卫星不稳定性评分超过样品中微卫星基因座的群体的群体训练阈值时,将样品的MSI状态分类为不稳定,从而确定样品的MSI状态。
在一些实施方案中,系统包括可操作地连接到控制器的核酸测序仪,该核酸测序仪被配置成提供来自样品中微卫星基因座的群体的序列信息。在这些实施方案中的一些实施方案中,核酸测序仪被配置成对核酸进行焦磷酸测序、单分子测序、纳米孔测序、半导体测序、合成测序(sequencing-by-synthesis)、连接测序(sequencing-by-ligation)或杂交测序(sequencing-by-hybridization),以产生测序读段。在某些实施方案中,系统包括可操作地连接到控制器的样品制备部件,该样品制备部件被配置成制备待由核酸测序仪进行测序的样品(在一些情况下,cfDNA样品)。在这些实施方案中的一些实施方案中,样品制备部件被配置成选择性地富集来自样品中核酸的区域。在某些实施方案中,样品制备部件被配置成将一个或更多个包含分子条形码的衔接子附接到核酸。在一些实施方案中,该系统包括可操作地连接到控制器的核酸扩增部件,该核酸扩增部件被配置成扩增DNA(在一些情况下,cfDNA)。在这些实施方案中的某些实施方案中,核酸扩增部件被配置成扩增选择性富集的来自样品中核酸的区域。
在某些实施方案中,系统包括可操作地连接到控制器的材料转移部件,该材料转移部件被配置成在核酸测序仪和样品制备部件之间转移一种或更多种材料。在一些实施方案中,系统包括可操作地连接到控制器的数据库,该数据库包括用一种或更多种疗法加索引的一个或更多个比较用结果,并且其中电子处理器还执行至少以下:(f)将样品的微卫星不稳定性状态与一个或更多个比较用结果进行比较,其中微卫星不稳定性评分和比较用结果之间的基本匹配指示预测的受试者对疗法的应答。
在又另一方面,本公开内容提供了包含非暂时性计算机可执行指令的计算机可读介质,该非暂时性计算机可执行指令当被至少一个电子处理器执行时,执行至少以下:(a)接收来自样品中微卫星基因座的群体的序列信息;(b)根据序列信息定量多于一个微卫星基因座的每一个处存在的不同重复序列长度的数目,以产生多于一个微卫星基因座的每一个的位点评分;(c)对于多于一个微卫星基因座的每一个,将给定的微卫星基因座的位点评分与给定的微卫星基因座的位点特异性训练阈值进行比较;(d)当给定的微卫星基因座的位点评分超过给定的微卫星基因座的位点特异性训练阈值时,将给定的微卫星基因座识别为不稳定,以产生微卫星不稳定性评分,所述微卫星不稳定性评分包括来自多于一个微卫星基因座的不稳定的微卫星基因座的数目;和(e)当微卫星不稳定性评分超过样品中微卫星基因座群体的群体训练阈值时,将样品的MSI状态分类为不稳定,从而确定样品的MSI状态。
本文公开的系统和计算机可读介质包括各种实施方案。在一些实施方案中,例如,多于一个微卫星基因座的位点评分包括似然性评分。在这些实施方案中的某些实施方案中,似然性评分包括基于概率对数似然性的评分,所述基于概率对数似然性的评分将从样品中许多体细胞来源的核酸片段(在一些实施方案中-cfDNA片段)获得的生物信号与由样品中样品收集后伪象产生的噪声区分开。通常使用至少两个参数确定来自样品的序列信息中的个体微卫星基因座的基于概率对数似然性的评分,其中至少第一参数包括等位基因频率,并且至少第二参数包括至少一种错误模式。等位基因频率包括来自样品的序列信息中包含不同重复序列长度的核酸的频率。至少一种错误模式通常包括随机错误模式和链特异性错误模式。在一些实施方案中,多于一个微卫星基因座的位点评分包括以下(a)和(b)之间的差异或比率:(a)度量所观察到的序列支持给定的微卫星基因座是稳定的零假设的评分,和(b)度量所观察到的序列支持给定的微卫星基因座是不稳定的备择假设的评分。在一些实施方案中,多于一个微卫星基因座的位点评分使用一个或更多个统计模型选择准则产生,所述统计模型选择准则诸如似然准则、对数似然准则、后验概率准则、赤池信息量准则(AIC)、贝叶斯信息准则和/或类似物。
在系统或计算机可读介质的一些实施方案中,多于一个微卫星基因座的位点评分包括基于赤池信息量准则(AIC)的位点评分,所述基于赤池信息量准则(AIC)的位点评分测试多于一个微卫星基因座处体细胞得失位的存在。在某些实施方案中,例如,给定的基于AIC的位点评分使用以下公式计算:
AIC=k-对数似然性,
其中k是模型中使用的参数的数目。任选地,使用最大似然估计(MLE)估计模型的参数。在这些实施方案中的一些实施方案中,使用Nelder-Mead算法确定MLE。在某些实施方案中,使用以下公式计算模型的零假设评分:
AIC
其中AIC
AIC
其中AIC
ΔAIC=AIC
在一些实施方案中,γ包括:(a)在测序读段中观察到的微卫星长度比起始核酸分子链的预期微卫星长度长一个重复单元的读段水平错误率;和/或(b)在测序读段中观察到的微卫星长度比起始核酸分子链的预期微卫星长度短一个重复单元的读段水平错误率。在某些实施方案中,β包括:(a)有义链的预期微卫星长度比核酸起始分子的预期微卫星长度长一个重复单元的链水平错误率;(b)反义链的预期微卫星长度比核酸起始分子的预期微卫星长度长一个重复单元的链水平错误率;(c)有义链的预期微卫星长度比核酸起始分子的预期微卫星长度短一个重复单元的链水平错误率;和/或(d)反义链的预期微卫星长度比核酸起始分子的预期微卫星长度短一个重复单元的链水平错误率。
在系统或计算机可读介质的某些实施方案中,当给定的微卫星基因座的位点评分在统计学上超过给定的微卫星基因座的位点特异性训练阈值时,将给定的微卫星基因座识别为不稳定。通常,肿瘤分数被估计,所述肿瘤分数包括在样品中的核酸中经鉴定的所有体细胞突变的最大突变等位基因分数(MAF)。在某些实施方案中,肿瘤分数低于样品中所有核酸的约0.05%、约0.1%、约0.2%、约0.5%、约1%、约2%、约3%、约4%、约5%、约6%、约7%、约8%、约9%、约10%、约11%、约12%、约13%、约14%或约15%。在一些实施方案中,多于一个微卫星基因座包括微卫星基因座的群体的全部,而在其他实施方案中,多于一个微卫星基因座包括微卫星基因座的群体的子集。在某些实施方案中,根据来自一个或更多个训练DNA样品中的微卫星基因座的群体的序列信息确定位点特异性训练阈值和/或群体训练阈值。任选地,当微卫星不稳定性评分是大于约1个、约2个、约3个、约4个、约5个、约6个、约7个、约8个、约9个、约10个、约15个、约16个、约17个、约18个、约19个、约20个、约30个、约40个、约50个或更多个来自多于一个微卫星基因座的不稳定的微卫星基因座时,将样品的MSI状态分类为MSI-高(MSI-H)。在一些实施方案中,当不稳定的微卫星基因座的数目构成多于一个微卫星基因座的约0.1%、约1%、约2%、约3%、约4%、约5%、约10%、约15%、约20%或约25%时,将样品的MSI状态分类为MSI-高(MSI-H)。
在又另一方面,本公开内容提供了一种系统,该系统包括通信接口,该通信接口通过通信网络获得来自受试者的样品中的一个或更多个核酸的测序信息;以及与通信接口通信的计算机,其中计算机包括至少一个计算机处理器和包含机器可执行代码的计算机可读介质,所述机器可执行代码在被至少一个计算机处理器执行时实现一种方法,该方法包括:(a)接收来自样品中微卫星基因座的群体的序列信息;(b)根据序列信息定量多于一个微卫星基因座的每一个处存在的不同重复序列长度的数目,以产生多于一个微卫星基因座的每一个的位点评分;(c)对于多于一个微卫星基因座的每一个,将给定的微卫星基因座的位点评分与给定的微卫星基因座的位点特异性训练阈值进行比较;(d)当给定的微卫星基因座的位点评分超过给定的微卫星基因座的位点特异性训练阈值时,将给定的微卫星基因座识别为不稳定,以产生微卫星不稳定性评分,所述微卫星不稳定性评分包括来自多于一个微卫星基因座的不稳定的微卫星基因座的数目;和(e)当微卫星不稳定性评分超过样品中微卫星基因座的群体的群体训练阈值时,将样品的MSI状态分类为不稳定,从而确定样品的MSI状态。
在一些实施方案中,序列信息由核酸测序仪提供。通常,核酸测序仪对核酸进行焦磷酸测序、单分子测序、纳米孔测序、半导体测序、合成测序、连接测序、杂交测序和/或另一种测序技术,以产生测序读段。在一些实施方案中,核酸测序仪使用源自测序文库的克隆单分子阵列来产生测序读段。在某些实施方案中,核酸测序仪包括具有微孔阵列的芯片以便对测序文库进行测序以产生测序读段。
本文公开的系统的计算机可读介质通常包括存储器、硬盘驱动器或计算机服务器。在一些实施方案中,通信网络包括能够进行分布式计算的一个或更多个计算机服务器。在一些实施方案中,分布式计算是云计算。在一些实施方案中,计算机位于远离核酸测序仪定位的计算机服务器上。在一些实施方案中,本文公开的系统包括通过网络与计算机通信的电子显示器,其中电子显示器包括用于在实施(i)-(iv)后显示结果的用户界面。在这些实施方案中的一些实施方案中,用户界面是图形用户界面(GUI)或基于网络的用户界面。在一些实施方案中,电子显示器在个人计算机中。在某些实施方案中,电子显示器在启用互联网的计算机(internet enabled computer)中。在这些实施方案中的一些实施方案中,启用互联网的计算机位于远离计算机的位置。通常,计算机可读介质包括存储器、硬盘驱动器或计算机服务器。在一些实施方案中,通信网络包括电信网络、互联网、外联网或内联网。
在一些实施方案中,本文公开的系统和方法的结果被用作生成报告的输入。报告可以呈纸质格式或电子格式。例如,通过本文公开的方法和系统获得的MSI评分和/或MSI状态可以被直接显示在这样的报告中。可选择地或另外地,基于MSI状态的诊断信息或一种或更多种定制疗法可以被包括在报告中。在一些实施方案中,报告被传送至受试者(例如,患者)或卫生保健提供者。
在一些实施方案中,方法、系统或计算机可读介质还包括如果重复核酸不稳定性评分低于核酸样品中重复核酸基因座的群体的群体训练阈值或处于所述群体训练阈值,则将核酸样品的重复核酸不稳定性状态分类为稳定。
在一些实施方案中,方法、系统或计算机可读介质还包括如果重复DNA不稳定性评分低于样品中重复DNA基因座的群体的群体训练阈值或处于所述群体训练阈值,则将样品的重复DNA不稳定性状态分类为稳定。
在一些实施方案中,方法、系统或计算机可读介质还包括如果微卫星不稳定性评分低于样品中微卫星基因座的群体的群体训练阈值或处于所述群体训练阈值,则将样品的微卫星不稳定性状态分类为稳定。
本文公开的方法的各个步骤,或通过本文公开的系统实施的步骤,可以在相同或不同的时间、在相同或不同的地理位置(例如国家)以及由相同或不同的人来实施。
附图简述
附图图示了某些实施方案,并且与书面描述一起用来解释本文公开的方法、计算机可读介质和系统的某些原理,所述附图并入本说明书并构成本说明书的一部分。当结合附图阅读时,本文提供的描述被更好地理解,附图以实例的方式而非限制的方式被包括。将理解,除非上下文另外指示,否则在整个附图中,类似的参考数字标识类似的部件。还将理解,一些或所有附图可以是出于说明目的的示意性表示,并且不必然描绘所示的元件的实际相对尺寸或位置。
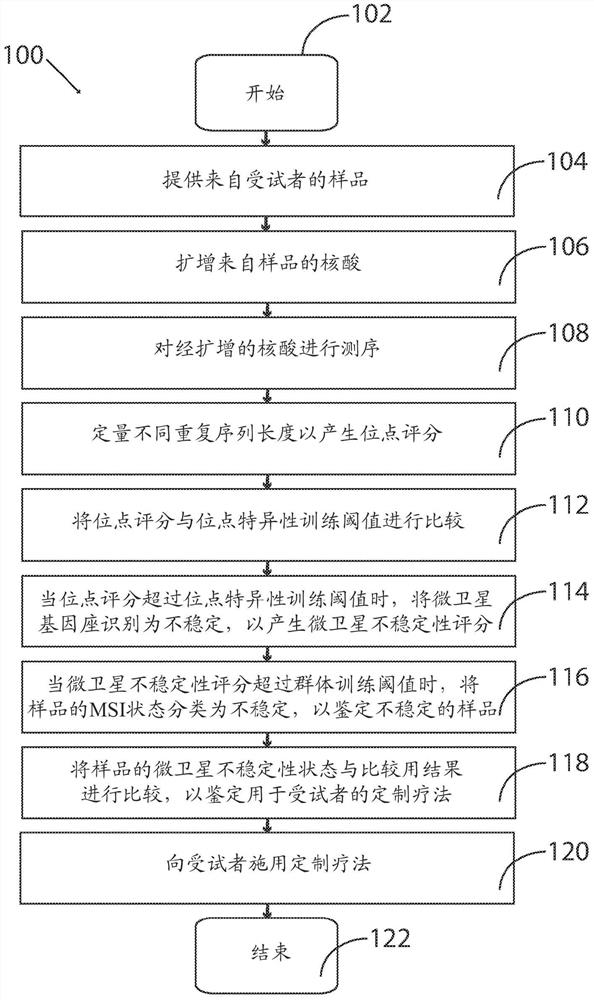
图1是示意性地描绘根据本发明的一些实施方案的确定微卫星不稳定性(MSI)状态的示例性方法步骤的流程图。
图2是适合用于本发明的某些实施方案的示例性系统的示意图。
图3是关于模拟样品的检测限(LoD)的图(检测概率(y轴);突变等位基因分数(MAF)(x轴))。
图4A(MSI评分(y轴);流动池(x轴))和图4B(MSI评分(y轴);参考样品(x轴))是来自重复性和再现性分析的数据图。
图5A(体细胞最大MAF(y轴);MSI评分(x轴))和图5B(体细胞最大MAF(y轴);MSI评分(x轴))是示出肿瘤分数不与MSI评分相关的图。
图6A和图6B是示出微卫星检测的技术特征的图。图6A是示出来自84名健康供体的cfDNA测序结果的99个候选微卫星基因座的赤池信息量准则评分的层次聚类的图。具有差的独特分子覆盖度的基因座以黑色示出,而具有过多技术伪象的基因座以深灰色示出。定义提供信息的位点(informative site)的微卫星重复序列长度的稳健但一致的测量值以浅灰色示出。箭头指示本研究中包括的三个Bethesda基因座。图6B是示出所观察到的与数字测序的每个组成部分相关的错误率降低的图。
图7A-图7C是示出ctDNA MSI检测的分析验证的图。所观察到的MSI检测率通过滴定水平(灰点)绘制,并且使用概率单位回归来确定5ng(图7A)和30ng(图7B)cfDNA输入的95%检测限。图7C是示出在499次单独的测序运行中运行的两个微卫星稳定(MSS)和两个MSI-H人为材料的499个独立重复的样品水平MSI评分的图。虚线指示MSI检测的样品水平阈值。
图8是示出精度(precision)研究的图,该精度研究使用人为样品以三个输入水平(5ng、10ng和30ng)在运行内和运行之间以一式三份处理。每个灰度阴影代表不同的运行。
图9是示出临床验证队列中有代表性的肿瘤类型的图。特别地,仅示出了具有至少5个代表性样品的肿瘤类型。所有其他肿瘤类型(n=25种不同的肿瘤类型)被分组在“其他”类别中。
图10A-图10C示出了ctDNA MSI状态与组织测试的一致性数据。图10A是示出按组织测试结果和观察到的肿瘤分数进行分类的1137个cfDNA样品的样品水平MSI评分的图。虚线指示MSI检测的样品水平阈值。图10B是示出按组织测试方法进行分类的一致性结果的图。图10C是示出可评估的独特患者队列的描述性统计的表格。
图11A-图11C是示出28,459个临床样品的ctDNA MSI景观(landscape)的图。图11A是示出了正轴报告了在样品集中16种最盛行的肿瘤类型中ctDNA MSI盛行率(prevalence)的图。负轴报告了基于Hause等人(52)的所述样品集中16种最盛行的肿瘤类型中的组织MSI盛行率。报告了样品的总数目,各自在括号内具有MSI-H样品的数目。图11B是对于具有≥5个MSI-H样品的肿瘤类型,按肿瘤类型示出样品水平MSI评分的图。虚线指示MSI检测的样品水平阈值。图11C是示出对于具有≥5个MSI-H样品的肿瘤类型,按肿瘤类型示出对MSI-H样品作出贡献的个体微卫星基因座的频率的图。UCEC,子宫体子宫内膜癌(uterine corpusendometrial carcinoma);STAD,胃腺癌;COAD,结肠腺癌;PRAD,前列腺腺癌;COUP,未知原发性癌;BLCA,膀胱癌;CHCA,胆管癌;HNSC,头颈部鳞状细胞癌;LUSC,肺鳞状细胞癌;BRST,乳腺癌;PANC,胰腺腺癌;LUNG,非特殊型肺癌(lung cancer,not otherwise specified);LIHC,肝细胞癌;KIRC,肾细胞癌;OV,卵巢癌;LUAD,肺腺癌。
图12A和图12B是按MSI状态示出肿瘤突变负荷的图。跨越278个MSI-H和28,181个MSS样品,按MSI状态进行分类的每个样品检测到的单核苷酸变异(SNV)(图12A)和得失位(图12B)的数目。
图13A-图13E示出了在ctDNA MSI-H患者中免疫检查点阻断(ICB)疗法的临床结果数据。图13A是按月计的派姆单抗(pembrolizumab)疗法的持续时间的游泳图(swimmerplot)。患者2的基线(图13B和图13C)和疗法后(图13D和图13E)的CT(图13B和图13D)和胃镜检查(图13C和图13E)。
定义
为了更容易地理解本公开内容,在下文中首先定义某些术语。以下术语和其他术语的另外的定义可以通过说明书来阐述。如果下文阐述的术语的定义与通过引用并入的申请或专利中的定义不一致,则本申请中阐述的定义应当用于理解该术语的含义。
如本说明书和所附权利要求书中使用的,除非上下文中另外清楚地指定,否则单数形式“一(a)”、“一(an)”和“该(the)”包括复数指示物。因此,例如,提及“一种方法”包括本文描述的和/或对于本领域普通技术人员而言在阅读本公开内容后将变得明显的一种或更多种方法,和/或类型的步骤,等等。
还应理解,本文使用的术语仅用于描述特定实施方案的目的,并且不意图进行限制。此外,除非另外定义,否则本文使用的所有技术术语和科学术语具有与由本公开内容所属的领域的普通技术人员通常理解的相同的含义。在描述和要求保护方法、计算机可读介质和系统时,将根据下文阐述的定义使用以下术语及其语法变形。
约:如本文使用的,“约(about)”或“约(approximately)”在被应用于一个或更多个感兴趣的值或要素时,是指与所陈述的参考值或要素类似的值或要素。在某些实施方案中,术语“约(about)”或“约(approximately)”是指值或要素的范围,所述范围以任一方向(大于或小于)落入所陈述的参考值或要素的25%、20%、19%、18%、17%、16%、15%、14%、13%、12%、11%、10%、9%、8%、7%、6%、5%、4%、3%、2%、1%或更小百分比内,除非另外说明或另外从上下文是明显的(这样的数字将超过可能的值或要素的100%时除外)。
衔接子:如本文使用的,“衔接子”是指短核酸(例如,长度小于约500个核苷酸、小于约100个核苷酸或小于约50个核苷酸),其通常至少部分为双链,并且被用于连接给定样品核酸分子的任一个末端或两个末端。衔接子可以包括核酸引物结合位点和/或测序引物结合位点,所述核酸引物结合位点允许扩增两个末端处侧翼有衔接子的核酸分子,和/或所述测序引物结合位点包括用于测序应用诸如各种下一代测序(NGS)应用的引物结合位点。衔接子还可以包括用于捕获探针诸如附接到流动池支持物的寡核苷酸等的结合位点。衔接子还可以包括如本文描述的核酸标签。核酸标签通常相对于扩增引物和测序引物的结合位点被定位,使得核酸标签被包含在给定核酸分子的扩增子和测序读段中。相同或不同的衔接子可以被连接到核酸分子的相应末端。在一些实施方案中,除了核酸标签不同的相同序列的衔接子被连接到核酸分子的相应末端。在一些实施方案中,衔接子是Y形衔接子,其中一个末端如本文描述的是平端的或加尾的,用于连接核酸分子,所述核酸分子也是平端的或用一个或更多个互补核苷酸加尾的。在仍其他的示例性实施方案中,衔接子是钟形衔接子,其包含用于连接至待分析的核酸分子的平端或加尾末端。衔接子的其他实例包括T加尾和C加尾的衔接子。
施用:如本文使用的,向受试者“施用(administer)”或“施用(administering)”治疗剂(例如,免疫治疗剂)意指给予受试者组合物、将组合物应用于受试者或使组合物与受试者接触。施用可以通过许多途径中的任何途径来完成,包括例如局部、口服、皮下、肌内、腹膜内、静脉内、鞘内和皮内。
赤池信息量准则:如本文使用的,“赤池信息量准则”或“AIC”是指用于从有限的一组模型中选择统计模型的准则,并且包括用于模型中的参数数目的罚分项。在一些实施方案中,选择具有最低AIC的模型。
等位基因频率:如本文使用的,“等位基因频率”是指在群体或给定受试者中特定基因座处的等位基因的相对频率。等位基因频率通常被表示为分数或百分比。
扩增:如本文使用的,在核酸的上下文中的“扩增(amplify)”或“扩增(amplification)”是指通常从少量的多核苷酸(例如,单个多核苷酸分子)开始产生多个拷贝的该多核苷酸或该多核苷酸的一部分,其中扩增产物或扩增子通常是可检测的。多核苷酸的扩增涵盖各种化学和酶促过程。
条形码:如本文使用的,在核酸的上下文中的“条形码”或“分子条形码”是指包含可以用作分子标识符的序列的核酸分子。例如,在下一代测序(NGS)文库制备期间,单独的“条形码”序列通常被添加到每个DNA片段,使得在最终数据分析之前可以鉴定和分选每个测序读段。
癌症类型:如本文使用的,“癌症”、“癌症类型”或“肿瘤类型”是指例如通过组织病理学定义的癌症的类型或亚型。癌症类型可以通过任何常规标准来定义,诸如基于在给定组织中的发生(例如,血癌、中枢神经系统(CNS)癌、脑癌、肺癌(小细胞和非小细胞)、皮肤癌、鼻癌、喉癌、肝癌、骨癌、淋巴瘤、胰腺癌、肠癌、直肠癌、甲状腺癌、膀胱癌、肾癌、口癌、胃癌、乳腺癌、前列腺癌、卵巢癌、肺癌、小肠癌、软组织癌、神经内分泌癌、胃食管癌、头颈癌、妇科癌症、结肠直肠癌、尿路上皮癌、实体状态癌(solid state cancers)、异质性癌症(heterogeneous cancer)、同质性癌症(homogeneous cancer))、未知的原发性来源等,和/或具有相同细胞谱系(例如,癌、肉瘤、淋巴瘤、胆管癌、白血病、间皮瘤、黑素瘤或胶质母细胞瘤)和/或显示出癌症标志物诸如Her2、CA15-3、CA19-9、CA-125、CEA、AFP、PSA、HCG、激素受体和NMP-22的癌症。癌症也可以通过分期(例如,1期、2期、3期或4期)以及是否为原发性来源或继发性来源来分类。
无细胞核酸:如本文使用的,“无细胞核酸”是指不包含在细胞内或不以其他方式与细胞结合的核酸,或者在一些实施方案中,是指在去除完整细胞后天然保留在样品中的核酸。无细胞核酸可以包括,例如,来源于来自受试者的体液(例如,血液、血浆、血清、尿液、脑脊液(CSF)等)的所有未被包封的核酸。无细胞核酸包括DNA(cfDNA)、RNA(cfRNA),以及它们的混杂物(hybrids),包括基因组DNA、线粒体DNA、循环DNA、siRNA、miRNA、循环RNA(cRNA)、tRNA、rRNA、小核仁RNA(snoRNA)、Piwi相互作用RNA(piRNA)、长非编码RNA(长ncRNA),和/或这些中的任一种的片段。无细胞核酸可以是双链的、单链的,或它们的混杂物。无细胞核酸可以通过分泌或细胞死亡过程,例如细胞坏死、凋亡等,被释放到体液中。一些无细胞核酸从癌细胞释放到体液中,例如,循环肿瘤DNA(ctDNA)。其他的从健康细胞释放。ctDNA可以是未被包封的肿瘤来源的片段化DNA。无细胞核酸的另一个实例是在母体血流中自由循环的胎儿DNA,也称为无细胞胎儿DNA(cffDNA)。无细胞核酸可以具有一种或更多种表观遗传修饰,例如,无细胞核酸可以被乙酰化、5-甲基化、泛素化、磷酸化、sumo化(sumoylated)、核糖基化和/或瓜氨酸化(citrullinated)。
比较用结果:如本文使用的,“比较用结果”意指一个结果或一组结果,可以将给定的测试样品或测试结果与所述一个结果或一组结果进行比较,以鉴定测试样品或结果的一个或更多个可能的性质、和/或一种或更多种可能的预后结果、和/或一种或更多种用于受试者的定制疗法,所述测试样品从受试者获取或以其他方式获得。比较用结果通常从一组参考样品(例如,从与测试受试者具有相同疾病或癌症类型的受试者和/或从接受或已经接受与测试受试者相同的疗法的受试者)获得。在某些实施方案中,例如,将样品(例如不稳定的cfDNA样品)的微卫星不稳定性状态与比较用结果进行比较,以鉴定cfDNA测试样品的微卫星不稳定性状态和针对一组参考样品确定的微卫星不稳定性状态之间的基本匹配。针对该组参考样品确定的微卫星不稳定性评分通常用一种或更多种定制疗法加索引。因此,当鉴定出基本匹配时,相应的定制疗法因此也被鉴定为用于从其获取测试样品的受试者的潜在治疗途径。
对照样品:如本文使用的,“对照样品”或“对照DNA样品”是指具有已知组成和/或具有已知性质和/或已知参数(例如,已知肿瘤分数、已知覆盖度、已知微卫星不稳定性评分和/或类似参数)的样品,其与测试样品一起被分析或与测试样品进行比较,以评估分析程序的准确性。
覆盖度:如本文使用的,“覆盖度”是指代表特定碱基位置的核酸分子的数目。
定制疗法:如本文使用的,“定制疗法”是指与基于给定标准选择的受试者或受试者群体的期望治疗结果相关的疗法,所述给定标准例如具有给定的微卫星不稳定性状态或在微卫星不稳定性评分的定义范围内。
脱氧核糖核酸或核糖核酸:如本文使用的,“脱氧核糖核酸”或“DNA”是指在糖部分的2'-位置处具有氢基团的天然或修饰的核苷酸。DNA通常包括包含以下四种类型的核苷酸碱基的核苷酸链:腺嘌呤(A)、胸腺嘧啶(T)、胞嘧啶(C)和鸟嘌呤(G)。如本文使用的,“核糖核酸”或“RNA”是指在糖部分的2'-位置处具有羟基基团的天然或修饰的核苷酸。RNA通常包括包含以下四种类型的核苷酸的核苷酸链:A、尿嘧啶(U)、G和C。如本文使用的,术语“核苷酸”是指天然核苷酸或修饰的核苷酸。某些核苷酸对以互补方式彼此特异性结合(被称为互补碱基配对)。在DNA中,腺嘌呤(A)与胸腺嘧啶(T)配对并且胞嘧啶(C)与鸟嘌呤(G)配对。在RNA中,腺嘌呤(A)与尿嘧啶(U)配对并且胞嘧啶(C)与鸟嘌呤(G)配对。当第一条核酸链结合由与第一条链中的那些核苷酸互补的核苷酸构成的第二条核酸链时,两条链结合形成双链。如本文使用的,“核酸测序数据”、“核酸测序信息”、“序列信息”、“核酸序列”、“核苷酸序列”、“基因组序列”、“基因序列”,或“片段序列”,或“核酸测序读段”表示指示核酸诸如DNA或RNA的分子(例如,全基因组、全转录组、外显子组、寡核苷酸、多核苷酸,或片段)中核苷酸碱基(例如,腺嘌呤、鸟嘌呤、胞嘧啶,和胸腺嘧啶或尿嘧啶)的顺序和身份的任何信息或数据。应当理解,本教导设想了使用所有可用的各种技术(technique)、平台或技术(technology),包括但不限于以下获得的序列信息:毛细管电泳、微阵列、基于连接的系统、基于聚合酶的系统、基于杂交的系统、直接或间接的核苷酸鉴定系统、焦磷酸测序、基于离子或pH的检测系统以及基于电子签名的系统(electronic signature-based system)。
免疫疗法:如本文使用的,“免疫疗法”是指用一种或更多种剂进行的治疗,所述一种或更多种剂用于刺激免疫系统以杀死或至少抑制癌细胞的生长,并且优选地用于减少癌症的进一步生长、减小癌症的大小和/或消除癌症。一些这样的剂结合癌细胞上存在的靶;一些结合免疫细胞上存在的靶而不是癌细胞上存在的靶;一些结合癌细胞和免疫细胞两者上存在的靶。这样剂包括但不限于检查点抑制剂和/或抗体。检查点抑制剂是免疫系统途径的抑制剂,其维持自身耐受性并且调节外周组织中生理免疫应答的持续时间和幅度,以使附带组织损伤最小化(参见,例如Pardoll,Nature Reviews Cancer 12,252-264(2012))。示例性的剂包括针对以下中的任一种的抗体:PD-1、PD-2、PD-L1、PD-L2、CTLA-4、OX40、B7.1、B7He、LAG3、CD137、KIR、CCR5、CD27、CD40或CD47。其他示例性的剂包括促炎细胞因子,诸如IL-1β、IL-6和TNF-α。其他示例性的剂是针对肿瘤活化的T细胞,诸如通过表达嵌合抗原而活化的T细胞,该嵌合抗原靶向由T细胞识别的肿瘤抗原。
得失位(Indel):如本文使用的,“得失位”是指涉及受试者的基因组中一个或更多个核苷酸的插入或缺失的突变。
加索引的:如本文使用的,“加索引的”是指第一要素(例如,微卫星不稳定性评分)与第二要素(例如,给定的疗法)相关。
不稳定性状态:如本文使用的,在重复核酸的上下文中的“不稳定性状态”或“不稳定性评分”(例如,重复核酸/重复DNA不稳定性状态或评分、微卫星不稳定性状态或评分)是指对一个或更多个核酸样品中给定的重复核酸基因座或重复核酸基因座的群体是否表现出高于、处于或低于针对该基因座或基因座的群体确定的阈值水平的突变水平或程度(例如,可变重复序列长度等)的测量或确定。为了清楚起见,不稳定性状态和不稳定性评分不可互换,而是相关的概念。不稳定性状态基于不稳定性评分。例如,如果样品的不稳定性评分低于群体训练阈值或处于群体训练阈值,则样品被分类为稳定的样品(例如,MSI-MSS或MSI-低),并且如果样品的不稳定性评分高于群体训练阈值,则样品被分类为不稳定的样品(例如,MSI-MSI-高)。
检测限(LoD):如本文使用的,“检测限”或“LoD”意指样品中物质(例如核酸)可以通过给定的测定或分析方法来测量的最小量。
最大MAF:如本文使用的,“最大MAF(maximum MAF)”或“最大MAF(max MAF)”是指样品中所有体细胞变异的最大MAF。
微卫星:如本文使用的,“微卫星”是指具有长度小于约10个碱基对或核苷酸的重复单元的重复核酸。
小卫星:如本文使用的,“小卫星”是指具有长度为约10个至约60个碱基对或核苷酸的重复单元的重复核酸。
突变等位基因分数:如本文使用的,“突变等位基因分数”、“突变剂量”或“MAF”是指在给定基因组位置处携带等位基因改变或突变的核酸分子的分数。MAF通常被表示为分数或百分比。例如,MAF通常小于给定基因座处存在的所有体细胞变异或等位基因的约0.5、0.1、0.05或0.01(即,小于约50%、10%、5%或1%)。
突变:如本文使用的,“突变”是指与已知参考序列的变异,并且包括突变,诸如例如单核苷酸变异(SNV)、拷贝数变体或变异(copy number variants or variations)(CNV)/畸变、插入或缺失(得失位)、基因融合、颠换、易位、移码、复制、重复序列扩增和表观遗传变异。突变可以是种系突变或体细胞突变。在一些实施方案中,用于比较目的的参考序列是提供测试样品的受试者的物种的野生型基因组序列,通常是人类基因组。
赘生物:如本文使用的,术语“赘生物”和“肿瘤”可互换地使用。它们是指受试者中细胞的异常生长。赘生物或肿瘤可以是良性的、潜在恶性的,或恶性的。恶性肿瘤是指癌症或癌性肿瘤。
下一代测序:如本文使用的,“下一代测序”或“NGS”是指与基于传统的Sanger和毛细管电泳的方法相比具有增加的通量的测序技术,例如,具有一次产生数十万个相对小的序列读段的能力的测序技术。下一代测序技术的一些实例包括但不限于合成测序、连接测序和杂交测序。
核酸标签:如本文使用的,“核酸标签”是指短核酸(例如,长度小于约500个核苷酸、约100个核苷酸、约50个核苷酸或约10个核苷酸),用于区分来自不同样品的核酸(例如,代表样品索引),或同一样品中不同类型的或经历不同处理的不同核酸分子(例如,代表分子条形码)。核酸标签包含预定的、固定的、非随机的、随机的或半随机的寡核苷酸序列。这样的核酸标签可以用于标记不同的核酸分子或不同的核酸样品或子样品。核酸标签可以是单链、双链或至少部分双链的。核酸标签任选地具有相同的长度或不同的长度。核酸标签还可以包括具有一个或更多个平端的双链分子,包括5'或3'单链区域(例如,突出端),和/或包括在给定分子内的其他位置处的一个或更多个其他单链区域。核酸标签可以被附接到其他核酸(例如,待扩增和/或测序的样品核酸)的一个末端或两个末端。核酸标签可以被解码以揭示诸如给定核酸的样品来源、形式或对给定核酸进行的处理的信息。例如,核酸标签也可以用于实现汇集和/或并行处理包含带有不同分子条形码和/或样品索引的核酸的多个样品,其中核酸随后通过检测(例如,读取)核酸标签被解卷积(deconvolved)。核酸标签也可以被称为标识符(例如分子标识符、样品标识符)。另外地或可选择地,核酸标签可以用作分子条形码(例如,以区分同一样品或子样品中的不同分子或不同亲本分子的扩增子)。这包括,例如,对给定样品中的不同的核酸分子独特地加标签,或对这样的分子非独特地加标签。在非独特加标签应用的情况下,可以使用有限数目的标签(例如,分子条形码)对核酸分子加标签,使得不同分子可以基于其内源序列信息(例如,其映射至所选择的参考基因组的起始位置和/或终止位置、序列的一个末端或两个末端的子序列,和/或序列的长度)与至少一个分子条形码的组合而被区分。通常,使用足够数目的不同的分子条形码,使得以下情况的概率低(例如,小于约10%、小于约5%、小于约1%,或小于约0.1%的概率):任何两个分子可能具有相同的内源序列信息(例如,起始位置和/或终止位置、序列的一个末端或两个末端的子序列,和/或长度)并且还具有相同的分子条形码。
多核苷酸:如本文使用的,“多核苷酸”、“核酸”、“核酸分子”或“寡核苷酸”是指通过核苷间连接进行连接的核苷(包括脱氧核糖核苷、核糖核苷或它们的类似物)的线性聚合物。通常,多核苷酸包含至少三个核苷。寡核苷酸的尺寸范围通常从几个单体单元(例如3-4个)到几百个单体单元。每当多核苷酸以一串字母诸如“ATGCCTG”表示时,将理解,这些核苷酸从左到右是5’→3'的顺序,并且在DNA的情况下,“A”表示脱氧腺苷,“C”表示脱氧胞苷,“G”表示脱氧鸟苷,并且“T”表示脱氧胸苷,除非另外说明。如本领域标准的,字母A、C、G和T可以用于指碱基本身、核苷或包含这些碱基的核苷酸。
群体训练阈值(population trained threshold):如本文使用的,在重复核酸的上下文中的“群体训练阈值”是指单独确定的在包括不稳定的重复核酸基因座的训练DNA样品(例如,非肿瘤样品、肿瘤样品等)中预期观察到的所述不稳定的重复核酸基因座的合计最大数目(aggregate maximum number)(例如,不稳定的微卫星基因座的数目)。群体训练阈值通常用于表征特定样品的经实验确定的重复核酸不稳定性评分。
处理:如本文使用的,术语“处理”、“计算”和“比较”可以可互换地使用。在某些应用中,该术语是指确定差异,例如数目或序列的差异。例如,可以处理重复DNA不稳定性评分(例如,微卫星不稳定性评分)、基因表达、拷贝数变异(CNV)、得失位和/或单核苷酸变异(SNV)值或序列。
参考序列:如本文使用的,“参考序列”是指用于与经实验确定的序列进行比较的目的的已知序列。例如,已知序列可以是整个基因组、染色体,或它们的任何区段。参考序列通常包括至少约20个、至少约50个、至少约100个、至少约200个、至少约250个、至少约300个、至少约350个、至少约400个、至少约450个、至少约500个、至少约1000个,或更多个核苷酸。参考序列可以与基因组或染色体的单个连续序列对齐,或者可以包括与基因组或染色体的不同区域对齐的非连续区段。示例性参考序列包括,例如,人类基因组,诸如,hG19和hG38。
重复序列长度:如本文使用的,在重复核酸的上下文中的“重复序列长度”是指在给定的重复核酸基因座处存在的重复单元的数目。为了说明,以下单链核酸链具有8的重复序列长度:
重复单元:如本文使用的,在重复核酸的上下文中的“重复单元”是指在给定的重复核酸基因组处重复的个体核苷酸模式或基序(例如,同聚物或异聚物)。为了说明,以下单链核酸链具有“ATT”的重复单元:
重复核酸:如本文使用的,“重复核酸”或“重复元件”是指在整个给定的基因组和/或基因组的群体中以多个拷贝存在的重复出现的核苷酸模式。重复核酸包括重复DNA和重复RNA。重复核酸的非限制性实例包括微卫星、末端重复序列、串联重复序列、小卫星、卫星DNA、散在重复序列、转座元件(例如DNA转座子、逆转录转座子(例如LTR-逆转录转座子(HERV)和LTR-逆转录转座子(HERV))等)、成簇规律间隔短回文重复(CRISPR)、直接重复序列、反向重复序列、镜像重复序列(mirror repeats)和翻转重复序列(everted repeats)。
重复核酸不稳定性评分:如本文使用的,在重复核酸的上下文中的“重复核酸不稳定性评分”(例如,重复DNA不稳定性评分、微卫星不稳定性评分等)是指来自给定样品中被识别为或以其他方式确定为不稳定的重复核酸基因座的群体的重复核酸基因座的合计数目。该重复核酸不稳定性评分是样品水平的评分(或样品评分),并且不同于位点评分,所述位点评分是特定于基因座的。
样品:如本文使用的,“样品”意指能够通过本文公开的方法和/或系统分析的任何事物。
灵敏度:如本文使用的,“灵敏度”意指以给定的MAF和覆盖度检测到突变存在的概率。
测序:如本文使用的,“测序”是指用于确定生物分子例如核酸诸如DNA或RNA的序列(例如,单体单元的身份和顺序)的许多技术中的任一种。示例性的测序方法包括但不限于靶向测序、单分子实时测序、外显子或外显子组测序、内含子测序、基于电子显微术的测序、组测序(panel sequencing)、晶体管介导的测序、直接测序、随机鸟枪法测序、Sanger双脱氧终止测序、全基因组测序、杂交测序、焦磷酸测序、毛细管电泳、双链体测序、循环测序、单碱基延伸测序、固相测序、高通量测序、大规模平行签名测序(massively parallelsignature sequencing)、乳液PCR、低变性温度共扩增PCR(COLD-PCR)、多重PCR、可逆染料终止子测序、成对末端测序、近末端测序(near-term sequencing)、核酸外切酶测序、连接测序、短读段测序、单分子测序、合成测序、实时测序、反向终止子测序、纳米孔测序、454测序、Solexa基因组分析仪测序、SOLiD
序列信息:如本文使用的,在核酸聚合物的上下文中的“序列信息”意指该聚合物中单体单元(例如,核苷酸等)的顺序和身份。
位点评分:如本文使用的,“位点评分”是指在样品中给定的重复核酸基因座处除种系重复序列长度外,存在另外的重复序列长度的可能性的量度。在某些实施方案中,通过计算基因座的Δ赤池信息量准则(AIC),确定给定基因座的位点评分。
位点特异性训练阈值(site specific trained threshold):如本文使用的,“位点特异性训练阈值”是指对于给定的重复核酸基因座(例如,给定的微卫星基因座)单独确定的使得该基因座稳定的位点评分的最大值。
体细胞突变:如本文使用的,“体细胞突变”意指在受孕之后发生的基因组突变。体细胞突变可以发生在除生殖细胞外的任何身体细胞中,并且因此不会传给子代。
特异性:如本文使用的,在诊断分析或测定的上下文中的“特异性”是指分析或测定检测预期的靶分析物而排除给定样品的其他组分的程度。
基本匹配:如本文使用的,“基本匹配”意指至少第一值或要素至少约等于至少第二值或要素。例如,在某些实施方案中,当微卫星不稳定性评分和比较用结果之间存在至少基本匹配或近似匹配时,定制疗法被鉴定。
受试者:如本文使用的,“受试者”是指动物,诸如哺乳动物物种(例如,人类),或禽类(例如,鸟类)物种,或其他生物体,诸如植物。更具体地,受试者可以是脊椎动物,例如,哺乳动物,诸如小鼠、灵长类动物、猿或人类。动物包括农场动物(例如,生产用牛(productioncattle)、奶牛、家禽、马、猪等)、运动动物和伴侣动物(例如,宠物或支持动物)。受试者可以是健康的个体,患有或疑似患有疾病或有患该疾病的倾向的个体,或需要治疗或疑似需要治疗的个体。术语“个体”或“患者”意在与“受试者”可互换。
例如,受试者可以是已经被诊断为患有癌症、将要接受癌症治疗和/或已经接受至少一种癌症治疗的个体。受试者可以处于癌症缓解中。作为另一个实例,受试者可以是被诊断为患有自身免疫性疾病的个体。作为另一个实例,受试者可以是妊娠或计划妊娠的雌性个体,其可能已经被诊断为患有或被疑似患有疾病,例如癌症、自身免疫性疾病。
阈值:如本文使用的,“阈值”是指用于表征或分类经实验确定的值的单独确定的值。
训练DNA样品:如本文使用的,“训练DNA样品”是指用于估计位点特异性训练阈值和群体训练阈值的DNA样品。训练DNA样品数据集包括一个或更多个训练DNA样品。训练DNA样品包括一个或更多个正常DNA样品和/或肿瘤DNA样品。在一些实施方案中,训练DNA样品包括具有MSI-高和/或MSI-低/MSS状态的一个或更多个样品。
肿瘤分数:如本文使用的,“肿瘤分数”是指对给定样品中源自肿瘤的核酸分子的分数的估计。例如,样品的肿瘤分数可以是从样品的最大MAF或样品的覆盖度或样品中的cfDNA片段的长度或样品的任何其他所选特征得到的量度。在一些实施方案中,样品的肿瘤分数等于样品的最大MAF。
不稳定:如本文使用的,在重复核酸的上下文中的“不稳定”或“不稳定性”是指在核酸样品(例如,cfDNA样品)中给定的重复核酸基因座处或给定的重复核酸基因座群体中观察到的超过阈值(例如,位点特异性训练阈值-基因座水平;群体训练阈值-样品水平;或类似物)的突变(例如,得失位或类似物)的水平。
详述
引言
癌症包括一大组遗传疾病,其共同特征是异常细胞生长以及转移到身体内起源细胞部位以外的潜力。该疾病的潜在分子基础是导致细胞表型转化的突变和/或表观遗传变化,无论这些有害变化是通过遗传获得的还是具有体细胞基础。更复杂的是,这些分子变化通常不仅在具有相同类型的癌症的患者中不同,而且甚至在给定的患者的自身肿瘤内不同。
鉴于在大多数癌症中观察到的突变变异性,癌症护理的挑战之一是鉴于患者的个体化癌症类型,鉴定患者将最可能对其做出应答的疗法。各种生物标志物被用于将癌症患者与适当的治疗(包括癌症免疫疗法)相匹配。应答的一个生物标志物是微卫星不稳定性(MSI),微卫星不稳定性(MSI)是由受损的DNA错配修复(MMR)机制引起的基因超突变的状况或倾向。具有被分类为高的微卫星不稳定性(MSI-H或MSI-高)的癌症患者经常表现出肿瘤细胞中体细胞突变的积累,这导致一系列分子和生物学变化,包括高肿瘤突变负荷、增加的新抗原(neoantigens)表达和丰富的肿瘤浸润性淋巴细胞。Chang等人.“MicrosatelliteInstability:A Predictive Biomarker for Cancer Immunotherapy,”ApplImmunohistochem Mol Morphol,26(2):e15-e21(2018)。这些变化与对检查点抑制剂药物的敏感性增加有关,所述检查点抑制剂药物诸如派姆单抗
本公开内容提供了可用于确定和分析患者样品(尤其是无细胞DNA(cfDNA)样品)中的MSI的方法、计算机可读介质和系统。使用这些方法和相关方面确定的MSI状态有助于指导疾病预后和治疗决策。用本文公开的方法和相关方面实现的结果通常与例如使用更常规的基于PCR的MSI评估方法获得的那些结果具有高度的一致性。
确定微卫星不稳定性状态的方法
本申请公开了准确地确定样品(尤其是无细胞DNA(cfDNA)样品)的微卫星不稳定性(MSI)状态和/或其他重复DNA不稳定性状态的各种方法。在某些实施方案中,评估MSI状态的方法包括cfDNA的靶向测序,例如,使用来自Guardant Health,Inc.(Redwood City,CA,USA)的数字测序平台,允许简单重复序列的广泛覆盖度,其中微卫星不稳定性可以跨越广泛的癌症类型发生。数字测序平台是一个癌症相关基因的NGS组套检测(NGS panel),其利用从简单、非侵入性血液抽取物分离的无细胞DNA(其可以包括循环肿瘤DNA)的高质量测序来进行。数字测序采用测序前制备的单独加标签的cfDNA分子的数字文库,结合测序后生物信息学重建,以消除几乎所有的假阳性。为了说明,图1提供了示意性地描绘根据本发明的一些实施方案的确定MSI状态的示例性方法步骤的流程图。如所示的,方法100包括在步骤110,根据序列信息定量多于一个微卫星基因座的每一个处存在的不同重复序列长度的数目,以产生多于一个微卫星基因座的每一个的位点评分。序列信息通常从cfDNA样品中微卫星基因座的群体获得。如本文另外描述的,在一些实施方案中,使用基于概率对数似然性的位点评分来定量给定微卫星基因座处存在的不同重复序列长度的数目。还如本文另外描述的,还任选地使用其他定量方法,只要它们同样准确地将从相对少量的体细胞来源的cfDNA片段获得的生物信号与例如由样品中的技术或样品收集后伪象(例如扩增伪象、测序伪象和类似物)产生的噪声区分开。
方法100还包括在步骤112,将给定微卫星基因座的位点评分与该特定微卫星基因座的位点特异性训练阈值进行比较。对于多于一个微卫星基因座的每一个,通常将特定基因座的经实验确定的位点评分与其相应的位点特异性训练阈值进行比较。给定基因座的位点特异性训练阈值通常是源自训练DNA样品的群体(诸如正常cfDNA样品或非肿瘤cfDNA样品的队列)的特定基因座的预定值。如所示的,方法100还包括在步骤114,当给定微卫星基因座的位点评分(例如,似然性评分等)超过(例如,在统计学上大于)该给定微卫星基因座的位点特异性训练阈值时,将该给定微卫星基因座识别为不稳定。基于这些比较,产生微卫星不稳定性评分,所述微卫星不稳定性评分包括来自多于一个微卫星基因座的被识别为不稳定的微卫星基因座的数目(例如,是样品的总体MSI评分或合计MSI评分)。此外,方法100还包括在步骤116,当微卫星不稳定性评分超过cfDNA样品中微卫星基因座的群体的群体训练阈值时,将cfDNA样品的MSI状态分类为不稳定,从而鉴定不稳定的cfDNA样品(例如,将样品评分或预测为MSI-高)。换句话说,在某些实施方案中,样品的MSI状态由最少数目的不稳定的微卫星基因座的存在决定。群体训练阈值通常是从训练DNA样品的群体获得的预定值,诸如从正常cfDNA样品或非肿瘤cfDNA样品的队列获得的预定值。
在一些实施方案中,从至少一个训练DNA样品数据集确定或以其他方式获得阈值(例如,位点特异性训练阈值、群体训练阈值等)。训练DNA样品数据集通常包括至少约25个至至少约30,000个或更多个训练样品。在一些实施方案中,训练DNA样品数据集包括约50个、75个、100个、150个、200个、300个、400个、500个、600个、700个、800个、900个、1,000个、2,500个、5,000个、7,500个、10,000个、15,000个、20,000个、25,000个、50,000个、100,000个、1,000,000个或更多个训练DNA样品。
在某些实施方案中,方法100包括另外的上游和/或下游步骤。在一些实施方案中,例如,方法100从步骤102开始,在步骤104提供来自受试者的样品(例如,提供从受试者获取的血液样品)。在这些实施方案中,方法100的工作流程通常还包括在步骤106,扩增样品中的核酸以产生经扩增的核酸,并且在步骤108,对经扩增的核酸进行测序以产生序列信息,然后在步骤110,根据序列信息定量多于一个微卫星基因座的每一个处存在的不同重复序列长度的数目。本文还描述了核酸扩增(包括相关的样品制备)、核酸测序和相关数据分析。
在一些实施方案中,方法100包括在步骤116,鉴定不稳定的cfDNA样品的下游的各个步骤。这些步骤中的一些实例包括在步骤118,将cfDNA样品的微卫星不稳定性状态与用疗法加索引的比较用结果进行比较,以鉴定用于治疗受试者的疾病(例如,癌症或另一种基于遗传的疾病、病症或状况)的定制疗法。在其他示例性实施方案中,方法100还包括在步骤122结束之前,在步骤120,当样品的微卫星不稳定性状态和比较用结果之间存在基本匹配时向受试者施用至少一种经鉴定的定制疗法(例如,以治疗受试者的癌症或另一种疾病、病症或状况)。
本文描述的方法包括各种可选择的实施方案。例如,微卫星基因座的位点评分任选地包括似然性评分。在一些实施方案中,似然性评分包括基于概率对数似然性的评分。在这些实施方案中的一些实施方案中,方法包括使用各个参数,诸如等位基因频率和一种或更多种错误模式(例如,随机错误模式、链特异性错误模式和/或类似错误模式),确定从样品获得的序列信息中的个体微卫星基因座的基于概率对数似然性的评分。等位基因频率通常包括在从样品获得的序列信息中的给定微卫星基因座处具有不同重复序列长度的核酸的所观察到的频率。在一些实施方案中,特定微卫星基因座的位点评分包括以下(a)和(b)之间的差异或比率:(a)度量所观察到的核酸序列支持给定的微卫星基因座是稳定的零假设的评分,和(b)度量所观察到的核酸序列支持给定的微卫星基因座是不稳定的备择假设的评分。零假设是假定位点是稳定的所有假设中具有最小AIC评分的假设,并且备择假设是假定位点是不稳定的所有假设中具有最小AIC评分的假设。通常,使用模型准确度的各种量度来产生位点评分,所述各种量度诸如似然准则、对数似然准则、后验概率准则、赤池信息量准则(AIC)、贝叶斯信息准则和/或类似物。关于统计建模的另外的细节,包括统计模型准确度的量度,任选地适用于执行本文公开的方法,在例如Bruce,Practical Statisticsfor Data Scientists:50Essential Concepts,第1版,O'Reilly Media(2017);Freedman等人,Statistics,第4版,W.W.Norton&Company(2007);James等人,An Introduction toStatistical Learning:with Applications in R,第1版,Springer(2013)和Hastie等人,The Elements of Statistical Learning:Data Mining,Inference,and Prediction,第2版,Springer(2016)中提供,这些文献各自通过引用以其整体并入本文。
为了进一步说明,给定的微卫星基因座的位点评分任选地包括基于AIC的位点评分,所述基于AIC的位点评分测试该微卫星基因座处体细胞得失位的存在。在这些实施方案中的一些实施方案中,给定的基于AIC的位点评分使用以下公式计算:
AIC=k-对数似然性,
其中k是模型中使用的参数的数目。在一些实施方案中,方法包括使用最大似然估计(MLE)(例如,使用Nelder-Mead算法或另一种单纯形搜索算法)估计模型的参数。方法任选地包括使用以下公式计算模型的零假设:
AIC
其中AIC
AIC
其中AIC
ΔAIC=AIC
在某些实施方案中,参数γ包括:(a)在测序读段中观察到的微卫星长度比起始核酸分子链的预期微卫星长度长一个重复单元的读段水平错误率;和/或(b)在测序读段中观察到的微卫星长度比起始核酸分子链的预期微卫星长度短一个重复单元的读段水平错误率。在一些实施方案中,参数β包括:(a)有义链的预期微卫星长度比核酸起始分子的预期微卫星长度长一个重复单元的链水平错误率;(b)反义链的预期微卫星长度比核酸起始分子的预期微卫星长度长一个重复单元的链水平错误率;(c)有义链的预期微卫星长度比核酸起始分子的预期微卫星长度短一个重复单元的链水平错误率;和/或(d)反义链的预期微卫星长度比核酸起始分子的预期微卫星长度短一个重复单元的链水平错误率。
在一些实施方案中,基于AIC的位点评分使用以下公式计算:
AIC=2(k-对数似然性),
其中k是模型中使用的参数的数目。在这些实施方案中,AIC
为了清楚的目的,在使用公式AIC=2(k-对数似然性)确定基于AIC的评分的实施方案中,用于将位点分类为不稳定的位点特异性阈值会是先前实施方案中使用的位点特异性阈值的两倍,在先前的实施方案中,基于AIC的评分使用以下公式确定:AIC=k-对数似然性。
使用本文描述的方法分析的样品通常包括各种突变等位基因分数(MAF)(例如,表现出特定微卫星基因座的不同重复序列长度或其他等位基因改变的样品分数)。此外,在一些实施方案中,样品包括肿瘤分数。在某些实施方案中,最大MAF(max MAF)用作给定样品中肿瘤分数的近似值。肿瘤分数通常低于样品中所有核酸的约0.05%、约0.1%、约0.2%、约0.5%、约1%、约2%、约3%、约4%、约5%、约6%、约7%、约8%、约9%、约10%、约11%、约12%、约13%、约14%或约15%。
在一些实施方案中,本文公开的方法通常包括给定样品中核酸的约0.2%肿瘤分数的检测限(LOD)处的至少约94%的灵敏度。方法通常还具有对样品中非肿瘤DNA的至少约99%的特异性。跨越约1.4%至约15%的肿瘤分数范围,样品的所确定的MSI状态通常与使用标准的基于PCR的MSI评估技术确定的样品的相应MSI状态具有至少约95%、96%、97%、98%或99%的一致性。在一些实施方案中,该一致性是100%。
在某些实施方案中,当样品的微卫星不稳定性评分是该样品中大于约1个、约2个、约3个、约4个、约5个、约6个、约7个、约8个、约9个、约10个、约11个、约12个、约13个、约14个、约15个、约16个、约17个、约18个、约19个、约20个、约30个、约40个、约50个、约60个、约70个、约80个、约90个、约100个或多于100个不稳定的微卫星基因座时,将特定样品的MSI状态分类为MSI-高(MSI-H)。在某些实施方案中,用于确定样品的不稳定性状态(例如,MSI状态)的群体训练阈值为约1个、约2个、约3个、约4个、约5个、约6个、约7个、约8个、约9个、约10个、约11个、约12个、约13个、约14个、约15个、约16个、约17个、约18个、约19个、约20个、约30个、约40个、约50个、约60个、约70个、约80个、约90个、约100个或多于100个不稳定的重复核酸(例如,微卫星)基因座。在一些实施方案中,样品的群体训练阈值为约5个不稳定的微卫星基因座。在一些实施方案中,样品的群体训练阈值为约6个不稳定的重复核酸基因座。在一些实施方案中,样品的群体训练阈值为约10个不稳定的重复核酸基因座。在一些实施方案中,样品的群体训练阈值为约15个不稳定的重复核酸基因座。在一些实施方案中,样品的群体训练阈值为约16个不稳定的重复核酸基因座。在一些实施方案中,样品的群体训练阈值为约20个不稳定的重复核酸基因座。在一些实施方案中,样品的群体训练阈值为约25个不稳定的重复核酸基因座。在一些实施方案中,样品的群体训练阈值为约26个不稳定的重复核酸基因座。在一些实施方案中,样品的群体训练阈值为约30个不稳定的重复核酸基因座。在一些实施方案中,样品的群体训练阈值为约35个不稳定的重复核酸基因座。在一些实施方案中,样品的群体训练阈值为约36个不稳定的重复核酸基因座。在一些实施方案中,样品的群体训练阈值为约40个不稳定的重复核酸基因座。在一些实施方案中,样品的群体训练阈值为约45个不稳定的重复核酸基因座。在一些实施方案中,样品的群体训练阈值为约46个不稳定的重复核酸基因座。在一些实施方案中,样品的群体训练阈值为约50个不稳定的重复核酸基因座。在一些实施方案中,重复核酸基因座可以是微卫星基因座。在一些实施方案中,当不稳定的微卫星基因座的数目构成该样品中估计的所有微卫星基因座的约0.1%、约1%、约2%、约3%、约4%、约5%、约10%、约15%、约20%或约25%时,将给定样品的MSI状态分类为MSI-H。在一些实施方案中,约50个、约60个、约70个、约80个、约90个、约100个、约200个、约300个、约400个、约500个、约600个、约700个、约800个、约900个、约1000个、约1100个、约1200个、约1300个、约1400个、约1500个、约1600个、约1700个、约1800个、约1900个、约2000个或多于2000个重复核酸(例如,微卫星)基因座用于确定给定样品的重复核酸不稳定性(例如,MSI)状态。在一些实施方案中,约50个重复核酸基因座用于确定给定样品的重复核酸不稳定性(例如,MSI)状态。在一些实施方案中,约60个重复核酸基因座用于确定给定样品的重复核酸不稳定性(例如,MSI)状态。在一些实施方案中,约70个重复核酸基因座用于确定给定样品的重复核酸不稳定性(例如,MSI)状态。在一些实施方案中,约80个重复核酸基因座用于确定给定样品的重复核酸不稳定性(例如,MSI)状态。在一些实施方案中,约90个重复核酸基因座用于确定给定样品的重复核酸不稳定性(例如,MSI)状态。在一些实施方案中,约100个重复核酸基因座用于确定给定样品的重复核酸不稳定性(例如,MSI)状态。在一些实施方案中,约200个重复核酸基因座用于确定给定样品的重复核酸不稳定性(例如,MSI)状态。在一些实施方案中,约300个重复核酸基因座用于确定给定样品的重复核酸不稳定性(例如,MSI)状态。在一些实施方案中,约400个重复核酸基因座用于确定给定样品的重复核酸不稳定性(例如,MSI)状态。在一些实施方案中,约500个重复核酸基因座用于确定给定样品的重复核酸不稳定性(例如,MSI)状态。在一些实施方案中,约1000个重复核酸基因座用于确定给定样品的重复核酸不稳定性(例如,MSI)状态。在一些实施方案中,约1100个重复核酸基因座用于确定给定样品的重复核酸不稳定性(例如,MSI)状态。在一些实施方案中,约1200个重复核酸基因座用于确定给定样品的重复核酸不稳定性(例如,MSI)状态。在一些实施方案中,约1300个重复核酸基因座用于确定给定样品的重复核酸不稳定性(例如,MSI)状态。在一些实施方案中,约1400个重复核酸基因座用于确定给定样品的重复核酸不稳定性(例如,MSI)状态。在一些实施方案中,至少1500个重复核酸基因座用于确定给定样品的重复核酸不稳定性(例如,MSI)状态。在一些实施方案中,约1600个重复核酸基因座用于确定给定样品的重复核酸不稳定性(例如,MSI)状态。在一些实施方案中,至少1700个重复核酸基因座用于确定给定样品的重复核酸不稳定性(例如,MSI)状态。在一些实施方案中,至少1800个重复核酸基因座用于确定给定样品的重复核酸不稳定性(例如,MSI)状态。在一些实施方案中,至少1900个重复核酸基因座用于确定给定样品的重复核酸不稳定性(例如,MSI)状态。在一些实施方案中,至少2000个重复核酸基因座用于确定给定样品的重复核酸不稳定性(例如,MSI)状态。在一些实施方案中,重复核酸基因座可以是微卫星基因座。在一些实施方案中,重复核酸不稳定性状态可以是MSI状态。
在一些实施方案中,方法包括从受试者获得样品。任选地使用基本上任何样品类型。在某些实施方案中,例如,样品是组织、血液、血浆、血清、痰、尿液、精液、阴道液、粪便、滑液、脊髓液、唾液和/或类似物。本文还描述了任选地使用的另外的示例性样品类型。通常,受试者是哺乳动物受试者(例如,人类受试者)。基本上任何类型的核酸(例如,DNA和/或RNA)可以根据本申请中公开的方法来评估。一些实例包括无细胞核酸(例如,肿瘤来源、胎儿来源、母体来源和/或类似来源的cfDNA)、细胞核酸,包括循环肿瘤细胞(例如,通过裂解样品中的完整细胞获得)、循环肿瘤核酸等。在一些实施方案中,样品包括无细胞DNA(cfDNA样品)。在一些实施方案中,cfDNA样品包括循环肿瘤核酸。
本申请中公开的方法通常包括从取自受试者的样品中的核酸获得序列信息。在某些实施方案中,序列信息从核酸的靶向区段获得。基本上任何数目的基因组区域任选地被靶向。靶向区段可以包括至少10个、至少50个、至少100个、至少500个、至少1000个、至少2000个、至少5000个、至少10,000个、至少20,000个或至少50,000个(例如,25个、50个、75个、100个、200个、300个、400个、500个、600个、700个、800个、900个、1,000个、2,000个、3,000个、4,000个、5,000个、6,000个、7,000个、8,000个、9,000个、10,000个、15,000个、25,000个、30,000个、35,000个、40,000个、45,000个)不同或重叠的基因组区域。在一些实施方案中,靶向区段包括至少50个、至少60个、至少70个、至少80个、至少90个、至少100个、至少200个、至少300个、至少400个、至少500个、至少600个或至少700个基因的所选区域。在一些实施方案中,靶向区段包括至少70个基因的所选区域。在一些实施方案中,靶向区段包括至少500个基因的区域。
在这些实施方案中,方法通常还包括各个样品或文库制备步骤,以制备用于测序的核酸。本领域技术人员熟知许多不同的样品制备技术。这些技术中的基本上任何技术被用于或适用于执行本文描述的方法。例如,除了将核酸与给定样品中的其他组分分离的各个纯化步骤之外,制备用于测序的核酸的典型步骤包括用分子标识符或条形码对核酸加标签、添加衔接子(例如,其可以包括条形码)、将核酸扩增一次或更多次、富集核酸的靶向区段(例如,使用各种靶向捕获策略等),和/或类似步骤。本文还描述了示例性的文库制备过程。关于核酸样品/文库制备的另外的细节也在例如van Dijk等人,Library preparationmethods for next-generation sequencing:Tone down the bias,Experimental CellResearch,322(1):12-20(2014),Micic(Ed.),Sample Preparation Techniques forSoil,Plant,and Animal Samples(Springer Protocols Handbooks),第1版,HumanaPress(2016),和Chiu,Next-Generation Sequencing and Sequence Data Analysis,Bentham Science Publishers(2018)中描述,这些文献各自通过引用以其整体并入本文。
通过本文公开的方法确定的微卫星和/或其他重复核酸的不稳定性状态任选地用于诊断受试者中疾病或状况,特别是癌症的存在,以表征这样的疾病或状况(例如,对给定的癌症进行分期,确定癌症的异质性等),监测对治疗的应答,评估发展给定的疾病或状况的潜在风险,和/或评估疾病或状况的预后。微卫星和/或其他重复核酸的不稳定性状态还任选地用于表征特定形式的癌症。由于癌症在组成和分期两者方面通常是异质性的,微卫星和/或其他重复核酸的不稳定性状态数据可以允许表征癌症的具体亚型,从而帮助诊断和治疗选择。该信息还可以为受试者或卫生保健从业者提供关于特定类型的癌症的预后的线索,并且使受试者和/或卫生保健从业者能够根据疾病的进展来调整治疗选项。随着癌症进展,一些癌症变得更具侵袭性并且遗传上不稳定。其他肿瘤保持为良性的、非活动的或休眠的。
微卫星和/或其他重复核酸的不稳定性状态还可以用于确定疾病进展和/或监测复发。在某些情况下,例如,成功的治疗最初可能随着癌细胞死亡和脱落核酸的数目增加而增加观察到的微卫星和/或其他重复核酸的不稳定性。在这些情况下,随着疗法进展,微卫星和/或其他重复核酸的不稳定性随后通常会随着肿瘤尺寸的继续减小而降低。在其他情况下,成功的治疗还可以降低微卫星和/或其他重复核酸的不稳定性,而不具有这样的不稳定性的最初增加。另外,如果观察到癌症在治疗之后缓解,则微卫星和/或其他重复核酸的不稳定性状态可以用于监测患者中余留的疾病或疾病的复发。
样品
样品可以是从受试者分离的任何生物样品。样品可以包括身体组织、全血、血小板、血清、血浆、粪便、红细胞、白细胞(white blood cell)或白细胞(leucocyte)、内皮细胞、组织活组织检查(例如,来自已知或疑似的实体瘤的活组织检查)、脑脊液、滑液、淋巴液、腹水、组织间液或细胞外液(例如,来自细胞间隙的流体)、齿龈液、龈沟液、骨髓、胸腔积液、脑脊液、唾液、黏液、痰、精液、汗液、尿液。样品优选地是体液,特别是血液及其级分,以及尿液。这样的样品包括从肿瘤脱落的核酸。核酸可以包括DNA和RNA,并且可以呈双链形式和单链形式。样品可以呈最初从受试者分离的形式,或者可以经受进一步处理以去除或添加组分,诸如细胞,相对于另一种组分富集一种组分,或者将一种形式的核酸转化为另一种,诸如将RNA转化为DNA或将单链核酸转化为双链的。因此,例如,用于分析的体液样品是含有无细胞核酸例如无细胞DNA(cfDNA)的血浆或血清。
在一些实施方案中,取自受试者的体液的样品体积取决于测序区域的期望读段深度。示例性体积为约0.4ml-40ml、约5ml-20ml、约10ml-20ml。例如,体积可以为约0.5ml、约1ml、约5ml、约10ml、约20ml、约30ml、约40ml或更多毫升。取样的血浆的体积通常在约5ml至约20ml之间。
样品可以包含各种量的核酸。通常,给定样品中核酸的量等同于多个基因组当量。例如,约30ng DNA的样品可以包含约10,000(10
在一些实施方案中,样品包括来自不同来源,例如,来自细胞来源和来自无细胞来源(例如,血液样品等)的核酸。通常,样品包括携带突变的核酸。例如,样品任选地包括携带种系突变和/或体细胞突变的DNA。通常,样品包括携带癌症相关突变(例如,癌症相关的体细胞突变)的DNA。
扩增之前样品中无细胞核酸的示例性量的范围通常为约1飞克(fg)至约1微克(μg),例如,约1皮克(pg)至约200纳克(ng)、约1ng至约100ng、约10ng至约1000ng。在一些实施方案中,样品包括高达约600ng、高达约500ng、高达约400ng、高达约300ng、高达约200ng、高达约100ng、高达约50ng或高达约20ng的无细胞核酸分子。任选地,该量为至少约1fg、至少约10fg、至少约100fg、至少约1pg、至少约10pg、至少约100pg、至少约1ng、至少约10ng、至少约100ng、至少约150ng或至少约200ng的无细胞核酸分子。在某些实施方案中,该量为高达约1fg、约10fg、约100fg、约1pg、约10pg、约100pg、约1ng、约10ng、约100ng、约150ng或约200ng的无细胞核酸分子。在某些实施方案中,方法包括从样品获得约5ng至约30ng之间的无细胞核酸分子。在某些实施方案中,方法包括从样品获得约5ng至约100ng之间的无细胞核酸分子。在某些实施方案中,方法包括从样品获得约5ng至约150ng之间的无细胞核酸分子。在某些实施方案中,方法包括从样品获得约5ng至约200ng之间的无细胞核酸分子。在一些实施方案中,该量为来自样品的高达约100ng的无细胞核酸分子。在一些实施方案中,该量为来自样品的高达约150ng的无细胞核酸分子。在一些实施方案中,该量为来自样品的高达约200ng的无细胞核酸分子。在一些实施方案中,该量为来自样品的高达约250ng的无细胞核酸分子。在一些实施方案中,该量为来自样品的高达约300ng的无细胞核酸分子。在一些实施方案中,方法包括从样品获得约1fg至约200ng之间的无细胞核酸分子。
无细胞核酸通常具有长度约100个核苷酸和长度约500个核苷酸之间的尺寸分布,其中长度约110个核苷酸至长度约230个核苷酸之间的分子代表样品中约90%的分子,其中众数为长度约168个核苷酸,并且第二次要峰在长度为约240个核苷酸至约440个核苷酸之间的范围内。在某些实施方案中,无细胞核酸的长度为约160个核苷酸至约180个核苷酸,或长度为约320个核苷酸至约360个核苷酸,或长度为约440个核苷酸至约480个核苷酸。
在一些实施方案中,通过分配步骤(partitioning step)从体液分离无细胞核酸,在该分配步骤中,将在溶液中发现的无细胞核酸与体液中的完整细胞和其他不可溶组分分离。在这些实施方案中的一些实施方案中,分配包括诸如离心或过滤的技术。可选择地,体液中的细胞被裂解,并且无细胞核酸和细胞核酸被一起处理。通常,在添加缓冲液和洗涤步骤之后,用例如醇来使无细胞核酸沉淀。在某些实施方案中,使用另外的清理(clean up)步骤诸如基于二氧化硅的柱来去除污染物或盐。例如,任选地在整个反应中添加非特异性批量载体核酸(non-specific bulk carrier nucleic acids)以对示例性程序的某些方面(诸如收率)进行优化。在这样的处理之后,样品通常包含各种形式的核酸,包括双链DNA、单链DNA和/或单链RNA。任选地,单链DNA和/或单链RNA被转化成双链形式,使得它们被包括在随后的处理和分析步骤中。
核酸标签
在一些实施方案中,可以用样品索引和/或分子条形码(通常被称为“标签”)对(来自于多核苷酸的样品的)核酸分子加标签。标签可以通过化学合成、连接(例如,平端连接或粘端连接)或重叠延伸聚合酶链式反应(PCR)以及其他方法掺入到衔接子中或以其他方式连接到衔接子。这样的衔接子最终可以被连接到靶核酸分子。在其他实施方案中,通常应用一轮或更多轮的扩增循环(例如,PCR扩增)来使用常规核酸扩增方法将样品索引引入核酸分子。扩增可以在一种或更多种反应混合物(例如,阵列中的多于一个微孔)中进行。分子条形码和/或样品索引可以被同时引入或以任何顺序引入。在一些实施方案中,分子条形码和/或样品索引在进行序列捕获步骤之前和/或之后引入。在一些实施方案中,在探针捕获之前仅引入分子条形码,并且在进行序列捕获步骤之后引入样品索引。在一些实施方案中,分子条形码和样品索引两者均在进行基于探针的捕获步骤之前引入。在一些实施方案中,样品索引在进行序列捕获步骤之后引入。在一些实施方案中,分子条形码通过衔接子经由连接(例如,平端连接或粘端连接)掺入样品中的核酸分子(例如,cfDNA分子)。在一些实施方案中,样品索引通过重叠延伸聚合酶链式反应(PCR)掺入样品中的核酸分子(例如cfDNA分子)。通常,序列捕获方案涉及引入与被靶向的核酸序列互补的单链核酸分子,所述被靶向的核酸序列例如基因组区域的编码序列,并且这样的区域的突变与癌症类型相关。
在一些实施方案中,标签可以位于样品核酸分子的一个末端或两个末端处。在一些实施方案中,标签是预定的或随机的或半随机的序列寡核苷酸。在一些实施方案中,标签的长度可以小于约500个、200个、100个、50个、20个、10个、9个、8个、7个、6个、5个、4个、3个、2个或1个核苷酸。标签可以被随机地或非随机地连接到样品核酸。
在一些实施方案中,每个样品被样品索引或样品索引的组合独特地加标签。在一些实施方案中,样品或子样品的每个核酸分子被分子条形码或分子条形码的组合独特地加标签。在其他实施方案中,可以使用多于一个分子条形码,使得分子条形码在所述多于一个分子条形码中彼此不一定是独特的(例如,非独特分子条形码)。在这些实施方案中,分子条形码通常被附接(例如,通过连接)到个体分子,使得分子条形码和其可以附接到的序列的组合产生可以被单独地追踪的独特序列。非独特地加标签的分子条形码与内源序列信息(例如,对应于样品中原始核酸分子序列的开始(起始)和/或结束(终止)部分,在一个末端或两个末端处的序列读段的子序列,序列读段的长度和/或样品中原始核酸分子的长度)的组合的检测,通常允许为特定分子分配独特的身份。个体序列读段的长度或碱基对数目也任选地用于为给定分子分配独特的身份。如本文描述的,来自已经被分配了独特身份的核酸的单链的片段可以从而允许对来自亲本链和/或互补链的片段的随后鉴定。
在一些实施方案中,分子条形码以预期的标识符的集合(例如,独特分子条形码或非独特分子条形码的组合)与样品中的分子的比率被引入。一种示例形式使用约2个至约1,000,000个不同的分子条形码,或约5个至约150个不同的分子条形码,或约20个至约50个不同的分子条形码。可选择地,可以使用约25个至约1,000,000个不同的分子条形码。分子条形码可以被连接到靶分子的两个末端。例如,可以使用20-50个×20-50个分子条形码。在一些实施方案中,可以使用20-50个不同的分子条形码。在一些实施方案中,可以使用5-100个不同的分子条形码。在一些实施方案中,可以使用5-150个分子条形码。在一些实施方案中,可以使用5-200个不同的分子条形码。这样的标识符数目通常足以使具有相同起点和终点的不同分子具有接收不同标识符组合的高概率(例如,至少94%、99.5%、99.99%或99.999%)。在一些实施方案中,约80%、约90%、约95%或约99%的分子具有相同的分子条形码组合。
在一些实施方案中,反应中独特或非独特的分子条形码的分配使用例如美国专利申请第20010053519、第20030152490号和第20110160078号、以及美国专利第6,582,908号、第7,537,898号、第9,598,731号和第9,902,992号中描述的方法和系统来进行,这些文献中的每一个在此通过引用以其整体并入本文。可选择地,在一些实施方案中,可以仅使用内源序列信息(例如,起始位置和/或终止位置,序列的一个末端或两个末端的子序列,和/或长度)来鉴定样品的不同核酸分子。
核酸扩增
侧翼有衔接子的样品核酸通常使用核酸引物通过PCR和其他扩增方法来扩增,所述核酸引物与待扩增的DNA分子侧翼的衔接子中的引物结合位点结合。在一些实施方案中,扩增方法涉及由热循环产生的延伸、变性和退火的循环,或者可以是等温的,例如,在转录介导的扩增中。任选地使用的其他示例性的扩增方法包括连接酶链式反应、链置换扩增、基于核酸序列的扩增、和基于自主持续序列的复制以及其他方法。
通常应用一轮或更多轮扩增循环来使用常规核酸扩增方法将样品索引引入核酸分子。扩增通常在一种或更多种反应混合物中进行。分子标签和样品索引/标签任选地被同时引入或以任何顺序引入。在一些实施方案中,在进行核酸分子捕获步骤(即,核酸富集)之前和/或之后引入分子标签和样品索引/标签。在一些实施方案中,在探针捕获之前仅引入分子标签,并且在进行序列捕获步骤之后引入样品索引/标签。在某些实施方案中,分子标签和样品索引/标签两者均在进行基于探针的捕获步骤之前引入。在一些实施方案中,样品索引/标签在进行序列捕获步骤之后引入。通常,序列捕获方案涉及引入与被靶向的核酸序列互补的单链核酸分子,所述被靶向的核酸序列例如基因组区域的编码序列,并且这样的区域的突变与癌症类型相关。通常,扩增反应产生具有分子标签和样品索引/标签的多于一个非独特或独特地加标签的核酸扩增子,所述分子标签和样品索引/标签的尺寸范围为约200个核苷酸(nt)至约700nt、250nt至约350nt,或约320nt至约550nt。在一些实施方案中,扩增子具有约300nt的尺寸。在一些实施方案中,扩增子具有约500nt的尺寸。
核酸富集
在一些实施方案中,在对核酸测序之前富集序列。对特定靶区域(“靶序列”)任选地进行富集。在一些实施方案中,感兴趣的靶区域可以用针对一个或更多个诱饵集组(baitset panels)选择的核酸捕获探针(“诱饵”)使用差异性平铺(differential tiling)和捕获方案来富集。差异性平铺和捕获方案通常使用不同相对浓度的诱饵组以在跨越与诱饵相关的基因组区域中差异性平铺(例如,以不同的“分辨率”),经受一组限制(例如,测序仪限制,诸如测序载量、每种诱饵的效用等),并以下游测序所期望的水平捕获被靶向的核酸。这些感兴趣的被靶向的基因组区域任选地包括核酸构建体的天然核苷酸序列或合成核苷酸序列。在一些实施方案中,具有针对一个或更多个感兴趣的区域的探针的生物素标记的珠可以用于捕获靶序列,并且任选地随后扩增这些区域以富集感兴趣的区域。
序列捕获通常涉及使用与靶核酸序列杂交的寡核苷酸探针。在某些实施方案中,探针组策略涉及将探针平铺在感兴趣的区域上。这样的探针的长度可以是例如约60个至约120个核苷酸。该组可以具有约2x、3x、4x、5x、6x、8x、9x、10x、15x、20x、30x、40x、50x或多于50x的深度。序列捕获的有效性通常部分地取决于靶分子中与探针的序列互补(或几乎互补)的序列的长度。
核酸测序
任选地侧翼有衔接子、有或没有预先扩增的样品核酸通常经受测序。任选地使用的测序方法或商购可得形式包括,例如,Sanger测序、高通量测序、焦磷酸测序、合成测序、单分子测序、基于纳米孔的测序、半导体测序、连接测序、杂交测序、RNA-Seq(Illumina)、数字基因表达(Helicos)、下一代测序(NGS)、单分子合成测序(SMSS)(Helicos)、大规模平行测序、克隆单分子阵列(Solexa)、鸟枪法测序、Ion Torrent、Oxford纳米孔、Roche Genia、Maxim-Gilbert测序、引物步移、使用PacBio、SOLiD、Ion Torrent或纳米孔平台的测序。测序反应可以在许多种样品处理单元中进行,所述样品处理单元可以包括多泳道(multiplelane)、多通道、多孔或基本上同时处理多个样品组的其他装置。样品处理单元还可以包括多个样品室,以实现同时处理多个运行。
可以对已知包含癌症或其他疾病的标志物(例如,微卫星和/或其他重复核酸元件)的一个或更多个核酸片段类型或区域进行测序反应。也可以对样品中存在的任何核酸片段进行测序反应。序列反应可以提供基因组的至少约5%、10%、15%、20%、25%、30%、40%、50%、60%、70%、80%、90%、95%、99%、99.9%或100%的基因组的序列覆盖度。在其他情况下,基因组的序列覆盖度可以是基因组的小于约5%、10%、15%、20%、25%、30%、40%、50%、60%、70%、80%、90%、95%、99%、99.9%或100%。在一些实施方案中,基因组的序列覆盖度可以是基因组的小于约0.01%、0.02%、0.05%、0.1%、0.2%、0.5%、1%、2%或5%。在一些实施方案中,基因组的序列覆盖度可以是基因组的小于约0.01%。在一些实施方案中,基因组的序列覆盖度可以是基因组的小于约0.02%。在一些实施方案中,基因组的序列覆盖度可以是基因组的小于约0.05%。在一些实施方案中,基因组的序列覆盖度可以是基因组的小于约0.1%。在一些实施方案中,基因组的序列覆盖度可以是基因组的小于约0.2%。在一些实施方案中,基因组的序列覆盖度可以是基因组的小于约0.5%。在一些实施方案中,基因组的序列覆盖度可以是基因组的小于约1%。在一些实施方案中,基因组的序列覆盖度可以是基因组的小于约2%。在一些实施方案中,基因组的序列覆盖度可以是基因组的小于约5%。在一些实施方案中,基因组的序列覆盖度可以是基因组的至少约5%。在一些实施方案中,基因组的序列覆盖度可以是基因组的至少约10%。在一些实施方案中,基因组的序列覆盖度可以是基因组的至少约20%。
可以使用多重测序技术进行同时的测序反应。在一些实施方案中,用至少约1000个、2000个、3000个、4000个、5000个、6000个、7000个、8000个、9000个、10000个、50000个或100,000个测序反应对无细胞多核苷酸进行测序。在其他实施方案中,用少于约1000个、2000个、3000个、4000个、5000个、6000个、7000个、8000个、9000个、10000个、50000个或100,000个测序反应对无细胞多核苷酸进行测序。测序反应通常顺序进行或同时进行。通常对全部或一部分的测序反应进行随后的数据分析。在一些实施方案中,对至少约1000个、2000个、3000个、4000个、5000个、6000个、7000个、8000个、9000个、10000个、50000个或100,000个测序反应进行数据分析。在其他实施方案中,可以对少于约1000个、2000个、3000个、4000个、5000个、6000个、7000个、8000个、9000个、10000个、50000个或100,000个测序反应进行数据分析。示例性读段深度是约1000个至约50000个读段/基因座(碱基位置)或>50,000个读段/基因座。
在一些实施方案中,通过在一个末端或两个末端处具有单链突出端的双链核酸上酶促形成平端来制备用于测序的核酸群体。在这些实施方案中,在呈dNTP形式的核苷酸(例如,A、C、G和T或U)存在的情况下,通常用具有5'-3'DNA聚合酶活性和3'-5'核酸外切酶活性的酶处理该群体。任选地使用的示例性酶或其催化片段包括Klenow大片段和T4聚合酶。在5'突出端处,酶通常使相对链上凹陷的3’末端延伸,直到它与5’末端齐平以产生平端。在3'突出端处,酶通常从3’末端消化,达到相对链的5’末端并且有时超过相对链的5’末端。如果该消化行进超过了相对链的5'末端,则空位可以通过具有与对5'突出端使用的具有相同聚合酶活性的酶填充。双链核酸上平端的形成有利于例如衔接子的附接和随后的扩增。
在一些实施方案中,核酸群体经受另外的处理,诸如将单链核酸转化为双链核酸和/或将RNA转化为DNA。这些形式的核酸还任选地与衔接子连接并扩增。
在有或没有预先扩增的情况下,经受上文描述的形成平端的处理的核酸以及任选地样品中的其他核酸,可以被测序以产生经测序的核酸。经测序的核酸可以指核酸的序列(即,序列信息)或其序列已被确定的核酸。可以进行测序,以便从样品中的个体核酸分子的扩增产物的共有序列直接地或间接地提供样品中的个体核酸分子的序列数据。
在一些实施方案中,样品中具有单链突出端的双链核酸在平端形成后,与包含条形码的衔接子在两个末端处连接,并且测序确定了核酸序列以及通过衔接子引入的内联(in-line)条形码。平端DNA分子任选地与至少部分双链的衔接子(例如,Y形衔接子或钟形衔接子)进行平端连接。可选择地,样品核酸和衔接子的平端可以用互补核苷酸加尾以有利于连接(例如,粘端连接)。
通常使核酸样品与足够数目的衔接子接触,使得任何两个相同核酸从连接在两个末端处的衔接子接收相同衔接子条形码组合的概率低(例如,小于<1%或<0.1%)。以这种方式使用衔接子允许鉴定具有参考核酸上相同的起点和终点并且被连接到相同条形码组合的核酸序列家族。这样的家族代表扩增之前的样品中的核酸的扩增产物序列。可以对家族成员的序列进行汇编,以获得原始样品中的核酸分子的一个或更多个共有核苷酸或完整的共有序列,所述核酸分子通过平端形成和衔接子附接被修饰。换句话说,占据样品中核酸的特定位置的核苷酸被确定为占据家族成员序列中对应位置的核苷酸的共有核苷酸。家族可以包括双链核酸的一条链或两条链的序列。如果家族的成员包括来自双链核酸的两条链的序列,为了对所有序列汇编以获得一个或更多个共有核苷酸或序列的目的,一条链的序列可以被转化为它们的互补序列。一些家族仅包含单个成员序列。在该情况下,该序列可以被视为扩增之前样品中的核酸的序列。可选择地,仅有单个成员序列的家族可以从随后的分析中排除。
通过将经测序的核酸与参考序列进行比较,可以确定经测序的核酸中的核苷酸变异。参考序列通常是已知序列,例如,来自受试者的已知的全部或一部分的基因组序列(例如,人类受试者的全基因组序列)。参考序列可以是例如hG19或hG38。如上文描述的,经测序的核酸可以代表直接确定的样品中的核酸的序列或这样的核酸的扩增产物的共有序列。可以在参考序列上的一个或更多个指定位置处进行比较。当相应的序列被最大程度地比对时,可以鉴定经测序的核酸的子集,该子集包括与参考序列的指定位置对应的位置。在这样的子集中,可以确定哪些(如果有的话)经测序的核酸在指定位置处包括核苷酸变异,以及任选地哪些(如果有的话)包括参考核苷酸(即,与参考序列中的相同)。如果子集中包括核苷酸变异的经测序的核酸的数目超过所选阈值,则变异核苷酸可以在指定位置处被识别。阈值可以是简单的数字,诸如子集中包括核苷酸变异的至少1个、2个、3个、4个、5个、6个、7个、9个或10个经测序的核酸,或者阈值可以是子集中包括核苷酸变异的经测序的核酸的比率,诸如至少0.5、1、2、3、4、5、10、15或20,以及其他可能性。可以对参考序列中感兴趣的任何指定位置进行重复比较。有时可以对占据参考序列上至少约20个、100个、200个或300个连续位置,例如,约20个-500个或约50个-300个连续位置的指定位置进行比较。
关于核酸测序的另外的细节,包括本文描述的形式和应用,还提供在以下文献中:例如,Levy等人,Annual Review of Genomics and Human Genetics,17:95-115(2016);Liu等人,J.of Biomedicine and Biotechnology,2012期,文章ID 251364:1-11(2012);Voelkerding等人,Clinical Chem.,55:641-658(2009);MacLean等人,NatureRev.Microbiol.,7:287-296(2009);Astier等人,J Am Chem Soc.,128(5):1705-10(2006);美国专利第6,210,891号、美国专利第6,258,568号、美国专利第6,833,246号、美国专利第7,115,400号、美国专利第6,969,488号、美国专利第5,912,148号、美国专利第6,130,073号、美国专利第7,169,560号、美国专利第7,282,337号、美国专利第7,482,120号、美国专利第7,501,245号、美国专利第6,818,395号、美国专利第6,911,345号、美国专利第7,501,245号、美国专利第7,329,492号、美国专利第7,170,050号、美国专利第7,302,146号、美国专利第7,313,308号和美国专利第7,476,503号,这些文献各自通过引用以其整体并入本文。
比较用结果
根据本申请中公开的方法确定的给定受试者的微卫星和/或其他重复核酸的不稳定性状态,通常与来自参考群体的比较用结果的数据库进行比较,以鉴定用于该受试者的定制疗法或靶向疗法。在一些实施方案中,测试受试者的微卫星和/或其他重复核酸的不稳定性状态和比较用结果是跨越例如整个基因组或整个外显子组测量的,而在其他实施方案中,这些标志物是基于例如基因组或外显子组的子集或靶向区域测量的,其任选地被外推以确定例如全基因组或全外显子组的微卫星不稳定性。通常,参考群体包括与测试受试者具有相同癌症类型的患者和/或正在接受或已经接受与测试受试者相同的疗法的患者。在一些实施方案中,测试受试者的微卫星和/或其他重复核酸的不稳定性状态以及比较用的微卫星和/或其他重复核酸的不稳定性状态通过确定预定或所选的基因或基因组区域的集合中的突变计数或载量来测量。基本上任何基因(例如,癌基因)任选地被选择用于这样的分析。在这些实施方案中的某些实施方案中,选择的基因或基因组区域包括至少约50个、100个、200个、300个、400个、500个、600个、700个、800个、900个、1000个、1,500个、2,000个或更多个选择的基因或基因组区域。在这些实施方案中的一些实施方案中,选择的基因或基因组区域任选地包括表1中列出的一个或更多个基因。
表1
癌症和其他疾病
在某些实施方案中,本文公开的方法和系统用于鉴定定制疗法,以治疗患者中的给定疾病、病症或状况。通常,被考虑的疾病是一种类型的癌症。这样的癌症的非限制性实例包括胆道癌、膀胱癌、移行细胞癌、尿路上皮癌、脑癌、神经胶质瘤、星形细胞瘤、乳腺癌、化生癌、宫颈癌、宫颈鳞状细胞癌、直肠癌、结肠直肠癌、结肠癌、遗传性非息肉性结肠直肠癌、结肠直肠腺癌、胃肠间质瘤(GIST)、子宫内膜癌、子宫内膜间质肉瘤、食管癌、食管鳞状细胞癌、食管腺癌、眼黑素瘤、葡萄膜黑素瘤、胆囊癌、胆囊腺癌、肾细胞癌、透明细胞肾细胞癌(clear cell renal cell carcinoma)、移行细胞癌、尿路上皮癌、肾母细胞瘤、白血病、急性淋巴细胞白血病(ALL)、急性髓性白血病(AML)、慢性淋巴细胞白血病(CLL)、慢性髓性白血病(CML)、慢性粒单核细胞白血病(CMML)、肝癌(liver cancer)、肝上皮癌(livercarcinoma)、肝细胞瘤、肝细胞癌、胆管癌、肝母细胞瘤、肺癌、非小细胞肺癌(NSCLC)、间皮瘤、B细胞淋巴瘤、非霍奇金淋巴瘤、弥漫性大B细胞淋巴瘤、套细胞淋巴瘤、T细胞淋巴瘤、非霍奇金淋巴瘤、前体T淋巴母细胞淋巴瘤/白血病、外周T细胞淋巴瘤、多发性骨髓瘤、鼻咽癌(NPC)、神经母细胞瘤、口咽癌、口腔鳞状细胞癌、骨肉瘤、卵巢癌、胰腺癌、胰腺导管腺癌、假乳头状肿瘤、腺泡细胞癌、前列腺癌、前列腺腺癌、皮肤癌、黑素瘤、恶性黑素瘤、皮肤黑素瘤、小肠癌、胃癌(stomach cancer)、胃上皮癌(gastric carcinoma)、胃肠间质瘤(GIST)、子宫癌或子宫肉瘤。
使用本文公开的方法和系统任选地评估的其他基于遗传的疾病、病症或状况的非限制性实例包括软骨发育不全、α-1抗胰蛋白酶缺乏症、抗磷脂综合征、孤独症、常染色体显性多囊肾病、沙尔科-马里-图思病(Charcot-Marie-Tooth,CMT)、猫叫综合征、克罗恩病、囊性纤维化、德卡姆病(Dercum disease)、唐氏综合征、Duane综合征、杜兴氏肌营养不良症、因子V Leiden易栓症、家族性高胆固醇血症、家族性地中海热、脆性X综合征、戈谢病、血色素沉着症、血友病、前脑无裂畸形(holoprosencephaly)、亨廷顿病、克兰费尔特综合征、马方综合征、强直性肌营养不良、神经纤维瘤病、努南综合征、成骨不全、帕金森病、苯丙酮尿症、Poland异常、卟啉症、早老症、视网膜色素变性、重症联合免疫缺陷(scid)、镰状细胞病、脊髓性肌萎缩症、Tay-Sachs、地中海贫血、三甲基胺尿症、特纳综合征、颚心脸综合征(velocardiofacial syndrome)、WAGR综合征、威尔逊病等。
定制疗法和相关施用
在一些实施方案中,本文公开的方法涉及鉴定定制疗法和向具有给定微卫星和/或其他重复核酸的不稳定性状态的患者施用定制疗法。基本上任何癌症疗法(例如,外科疗法、放射疗法、化疗和/或类似疗法)被纳入作为这些方法的一部分。通常,定制疗法包括至少一种免疫疗法(或免疫治疗剂)。免疫疗法通常是指增强针对给定癌症类型的免疫应答的方法。在某些实施方案中,免疫疗法是指增强针对肿瘤或癌症的T细胞应答的方法。
在一些实施方案中,免疫疗法或免疫治疗剂靶向免疫检查点分子。某些肿瘤能够通过招安(co-opting)免疫检查点途径来逃避免疫系统。因此,靶向免疫检查点已经成为用于对抗肿瘤逃避免疫系统的能力和活化针对某些癌症的抗肿瘤免疫的有效方法。Pardoll,Nature Reviews Cancer,2012,12:252-264。
在某些实施方案中,免疫检查点分子是减少T细胞对抗原的应答中涉及的信号的抑制性分子。例如,CTLA4在T细胞上表达,并且通过结合抗原呈递细胞上的CD80(又称B7.1)或CD86(又称B7.2)在下调T细胞活化中发挥作用。PD-1是另一种在T细胞上表达的抑制性检查点分子。PD-1在炎性应答期间限制外周组织中T细胞的活性。此外,PD-1的配体(PD-L1或PD-L2)通常在许多不同肿瘤的表面上被上调,导致肿瘤微环境中抗肿瘤免疫应答的下调。在某些实施方案中,抑制性免疫检查点分子是CTLA4或PD-1。在其他实施方案中,抑制性免疫检查点分子是PD-1的配体,诸如PD-L1或PD-L2。在其他实施方案中,抑制性免疫检查点分子是CTLA4的配体,诸如CD80或CD86。在其他实施方案中,抑制性免疫检查点分子是淋巴细胞活化基因3(LAG3)、杀伤细胞免疫球蛋白样受体(KIR)、T细胞膜蛋白3(TIM3)、半乳凝素9(GAL9)或腺苷A2a受体(A2aR)。
靶向这些免疫检查点分子的拮抗剂可以用于增强针对某些癌症的抗原特异性T细胞应答。因此,在某些实施方案中,免疫疗法或免疫治疗剂是抑制性免疫检查点分子的拮抗剂。在某些实施方案中,抑制性免疫检查点分子是PD-1。在某些实施方案中,抑制性免疫检查点分子是PD-L1。在某些实施方案中,抑制性免疫检查点分子的拮抗剂是抗体(例如,单克隆抗体)。在某些实施方案中,抗体或单克隆抗体是抗CTLA4抗体、抗PD-1抗体、抗PD-L1抗体或抗PD-L2抗体。在某些实施方案中,抗体是单克隆抗PD-1抗体。在一些实施方案中,抗体是单克隆抗PD-L1抗体。在某些实施方案中,单克隆抗体是抗CTLA4抗体和抗PD-1抗体的组合、抗CTLA4抗体和抗PD-L1抗体的组合,或抗PD-L1抗体和抗PD-1抗体的组合。在某些实施方案中,抗PD-1抗体是派姆单抗
在某些实施方案中,免疫疗法或免疫治疗剂是针对CD80、CD86、LAG3、KIR、TIM3、GAL9或A2aR的拮抗剂(例如抗体)。在其他实施方案中,拮抗剂是抑制性免疫检查点分子的可溶性形式,诸如包含抑制性免疫检查点分子的细胞外结构域和抗体的Fc结构域的可溶性融合蛋白。在某些实施方案中,可溶性融合蛋白包含CTLA4、PD-1、PD-L1或PD-L2的细胞外结构域。在一些实施方案中,可溶性融合蛋白包含CD80、CD86、LAG3、KIR、TIM3、GAL9或A2aR的细胞外结构域。在一种实施方案中,可溶性融合蛋白包含PD-L2或LAG3的细胞外结构域。
在某些实施方案中,免疫检查点分子是放大T细胞对抗原的应答中涉及的信号的共刺激分子。例如,CD28是一种在T细胞上表达的共刺激受体。当T细胞通过其T细胞受体与抗原结合时,CD28与抗原呈递细胞上的CD80(又称B7.1)或CD86(又称B7.2)结合,以放大T细胞受体信号传导并促进T细胞活化。因为CD28与CTLA4结合相同的配体(CD80和CD86),所以CTLA4能够抵消或调节由CD28介导的共刺激信号传导。在某些实施方案中,免疫检查点分子是选自CD28、诱导型T细胞共刺激因子(ICOS)、CD137、OX40或CD27的共刺激分子。在其他实施方案中,免疫检查点分子是共刺激分子的配体,包括例如CD80、CD86、B7RP1、B7-H3、B7-H4、CD137L、OX40L或CD70。
靶向这些共刺激检查点分子的激动剂可以用于增强针对某些癌症的抗原特异性T细胞应答。因此,在某些实施方案中,免疫疗法或免疫治疗剂是共刺激检查点分子的激动剂。在某些实施方案中,共刺激检查点分子的激动剂是激动剂抗体,并且优选地是单克隆抗体。在某些实施方案中,激动剂抗体或单克隆抗体是抗CD28抗体。在其他实施方案中,激动剂抗体或单克隆抗体是抗ICOS抗体、抗CD137抗体、抗OX40抗体或抗CD27抗体。在其他实施方案中,激动剂抗体或单克隆抗体是抗CD80抗体、抗CD86抗体、抗B7RP1抗体、抗B7-H3抗体、抗B7-H4抗体、抗CD137L抗体、抗OX40L抗体或抗CD70抗体。
用于治疗除了癌症之外的特定的基于遗传的疾病、病症或状况的治疗选项通常是本领域普通技术人员熟知的,并且鉴于所考虑的特定疾病、病症或状况将是明显的。
在某些实施方案中,本文描述的定制疗法通常被肠胃外(例如,静脉内或皮下)施用。包含免疫治疗剂的药物组合物通常被静脉内施用。某些治疗剂被口服施用。然而,定制疗法(例如,免疫治疗剂等)还可以通过本领域已知的任何方法施用,包括例如含服、舌下、直肠、阴道、尿道内、局部、眼内、鼻内和/或耳内,该施用可以包括片剂、胶囊、颗粒、水性悬浮液、凝胶、喷雾剂、栓剂、药膏、软膏或类似物。
系统和计算机可读介质
本公开内容还提供了各种系统和计算机程序产品或机器可读介质。在一些实施方案中,例如,本文描述的方法任选地至少部分地使用系统、分布式计算硬件和应用(例如,云计算服务器)、电子通信网络、通信接口、计算机程序产品、机器可读介质、电子存储介质、软件(例如,机器可执行代码或逻辑指令)等来执行或促进。为了说明,图2提供了适合用于实现至少本申请中公开的方法的方面的示例性系统的示意图。如所示的,系统200包括至少一个控制器或计算机,例如服务器202(例如,搜索引擎服务器),服务器202包括处理器204和存储器、存储设备或存储器部件206;以及一个或更多个其他通信设备214和216(例如客户端计算机终端、电话、平板电脑、膝上型电脑、其他移动设备等),所述一个或更多个其他通信设备214和216远离远程服务器202定位并通过电子通信网络212(诸如因特网或其他互联网络)与远程服务器202通信。通信设备214和216通常包括通过网络212与例如服务器202计算机通信的电子显示器(例如,启用互联网的计算机或类似物),其中电子显示器包括用于在实现本文描述的方法后显示结果的用户界面(例如,图形用户界面(GUI)、基于网络的用户界面,和/或类似界面)。在某些实施方案中,通信网络还包括例如使用硬盘驱动器、拇指驱动器或其他数据存储机制将数据从一个位置物理传输到另一个位置。系统200还包括存储在计算机或机器可读介质上的程序产品208,诸如例如一个或更多个各种类型的存储器,诸如服务器202的存储器206,储存器206可由服务器202读取,以便于例如引导搜索应用或由一个或更多个其他通信设备执行的其他应用,所述通信设备诸如214(示意性地示出为台式计算机或个人计算机)和216(示意性地示出为平板计算机)。在一些实施方案中,系统200任选地还包括至少一个数据库服务器,诸如例如,与在线网站相关联的服务器210,该在线网站具有存储在其上的可直接地或通过搜索引擎服务器202搜索的数据(例如,对照样品或比较用结果数据、加索引的定制疗法等)。系统200任选地还包括一个或更多个远离服务器202定位的其他服务器,每个所述其他服务器任选地与一个或更多个数据库服务器210相关联,该数据库服务器210远离其他服务器中的每一个定位或与其他服务器中的每一个定位在本地。其他服务器可以有益地向地理上远程的用户提供服务,并且增强地理分布式操作。
如本领域普通技术人员理解的,服务器202的存储器206任选地包括易失性存储器和/或非易失性存储器,包括例如RAM、ROM和磁盘或光盘以及其他。本领域普通技术人员还应当理解,尽管被图示为单个服务器,但是所图示的服务器202的配置仅通过实例的方式给出,并且也可以使用根据各种其他方法或架构配置的其他类型的服务器或计算机。图2中示意性地示出的服务器202代表服务器或服务器集群或服务器场,并且不限于任何个体物理服务器。服务器站点可以被部署为由服务器托管提供商管理的服务器场或服务器集群。服务器的数目及其架构和配置可以基于系统200的使用、需求和容量需求而增加。如本领域普通技术人员还理解的,这些实施方案中的其他用户通信设备214和216例如可以是膝上型计算机、台式计算机、平板计算机、个人数字助理(PDA)、手机、服务器或其他类型的计算机。如本领域普通技术人员已知和理解的,网络212可以包括因特网、内联网、电信网络、外联网或通过通信网络与一个或更多个其他计算机通信的多于一个计算机/服务器的万维网,和/或局域网或其他区域网络的一部分。
如本领域普通技术人员进一步理解的,示例性的程序产品或机器可读介质208任选地是提供一个或更多个组的有序操作的微码、程序、云计算格式、例程和/或符号语言的形式,这些有序操作控制硬件的功能发挥并指导其操作。根据示例性实施方案,程序产品208也不需要全部驻留在易失性存储器中,而是可以根据本领域普通技术人员已知和理解的各种方法,根据需要选择性地加载。
如本领域普通技术人员进一步理解的,术语“计算机可读介质”或“机器可读介质”是指参与向处理器提供指令以用于执行的任何介质。为了说明,术语“计算机可读介质”或“机器可读介质”包括分布式介质、云计算格式、中间存储介质、计算机的执行存储器以及能够存储实现本公开内容的各种实施方案的功能或方法的程序产品208的任何其他介质或设备,例如用于由计算机读取。“计算机可读介质”或“机器可读介质”可以采用许多形式,包括但不限于非易失性介质、易失性介质和传输介质。非易失性介质包括例如光盘或磁盘。易失性介质包括动态存储器,诸如给定系统的主存储器。传输介质包括同轴电缆、铜线和光纤,包括构成总线的导线。传输介质也可以采用声波或光波的形式,诸如在无线电波和红外数据通信等期间产生的那些。计算机可读介质的示例性形式包括软盘(floppy disk)、软磁盘(flexible disk)、硬盘、磁带、闪存驱动器,或任何其他磁介质、CD-ROM、任何其他光学介质、穿孔卡片、纸带、具有孔图案的任何其他物理介质、RAM、PROM和EPROM、FLASH-EPROM、任何其他存储器芯片或盒、载波,或者计算机可以从其读取的任何其他介质。
程序产品208任选地从计算机可读介质拷贝到硬盘或类似的中间存储介质。当要运行程序产品208或其部分时,它任选地从其分布式介质、其中间存储介质等加载到一个或更多个计算机的执行存储器中,配置一个或更多个计算机以根据各种实施方案的功能或方法来发挥作用。所有这样的操作是例如计算机系统领域的普通技术人员熟知的。
为了进一步说明,在某些实施方案中,本申请提供了包括一个或更多个处理器以及与处理器通信的一个或更多个存储器部件的系统。存储器部件通常包括一个或更多个指令,所述指令当被执行时使得处理器提供信息,该信息使得序列信息、微卫星和/或其他重复核酸的不稳定性状态、比较用结果、定制疗法和/或类似物被显示(例如,经由通信设备214、216或类似设备)和/或从其他系统部件和/或从系统用户接收信息(例如,经由通信设备214、216或类似设备)。
在一些实施方案中,程序产品208包括非暂时性计算机可执行指令,该非暂时性计算机可执行指令当被电子处理器204执行时,执行至少以下:(i)接收来自样品中微卫星基因座的群体的序列信息;(ii)根据序列信息定量多于一个微卫星基因座的每一个处存在的不同重复序列长度的数目,以产生多于一个微卫星基因座的每一个的位点评分;(iii)对于多于一个微卫星基因座的每一个,将给定的微卫星基因座的位点评分与给定的微卫星基因座的位点特异性训练阈值进行比较;(iv)当给定的微卫星基因座的位点评分超过给定的微卫星基因座的位点特异性训练阈值时,将给定的微卫星基因座识别为不稳定,以产生微卫星不稳定性评分,所述微卫星不稳定性评分包括来自多于一个微卫星基因座的不稳定的微卫星基因座的数目;(v)当微卫星不稳定性评分超过样品中微卫星基因座的群体的群体训练阈值时,将样品的MSI状态分类为不稳定,以鉴定不稳定的样品;以及任选地(vi)将不稳定的样品的微卫星不稳定性评分与一个或更多个比较用结果进行比较,其中不稳定的样品的微卫星不稳定性评分和比较用结果之间的基本匹配指示预测的受试者对疗法的应答。
系统200通常还包括被配置成执行本文描述的方法的各个方面的另外的系统部件。在这些实施方案中的一些实施方案中,这些另外的系统部件中的一个或更多个远离远程服务器202定位,并且通过电子通信网络212与远程服务器202通信,而在其他实施例中,这些另外的系统部件中的一个或更多个位于本地,并且与服务器202通信(即,在不存在电子通信网络212的情况下),或者直接与例如台式计算机214通信。
在一些实施方案中,例如,包括样品制备部件218的另外的系统部件可操作地连接(直接地或间接地(例如,经由电子通信网络212))到控制器202。样品制备部件218被配置成制备样品中的核酸(例如,制备核酸的文库),以通过核酸扩增部件(例如,热循环仪等)和/或核酸测序仪来扩增和/或测序。在这些实施方案中的某些实施方案中,样品制备部件218被配置成将核酸与样品中的其他组分分离,将一个或更多个包括条形码的衔接子附接到如本文描述的核酸,在测序之前选择性地富集基因组或转录组的一个或更多个区域,和/或类似操作。
在某些实施方案中,系统200还包括可操作地连接(直接地或间接地(例如,经由电子通信网络212))到控制器202的核酸扩增部件220(例如,热循环仪等)。核酸扩增部件220被配置成扩增来自受试者的样品中的核酸。例如,核酸扩增部件220任选地被配置成扩增来自如本文描述的样品中的基因组或转录组的选择性富集的区域。
系统200通常还包括可操作地连接(直接地或间接地(例如,经由电子通信网络212))到控制器202的至少一个核酸测序仪222。核酸测序仪222被配置成提供来自受试者的样品中的核酸(例如,经扩增的核酸)的序列信息。基本上任何类型的核酸测序仪可以适用于这些系统。例如,核酸测序仪222任选地被配置成对核酸执行焦磷酸测序、单分子测序、纳米孔测序、半导体测序、合成测序、连接测序、杂交测序或其他技术以产生测序读段。任选地,核酸测序仪222被配置成将序列读段分组到序列读段的家族中,每个家族包括从给定样品中的核酸产生的序列读段。在一些实施方案中,核酸测序仪222使用源自测序文库的克隆单分子阵列来产生测序读段。在某些实施方案中,核酸测序仪222包括具有微孔阵列的至少一个芯片以用于对测序文库进行测序,以产生测序读段。
为了便于完全或部分系统自动化,系统200通常还包括可操作地连接(直接地或间接地(例如,经由电子通信网络212))到控制器202的材料转移部件224。材料转移部件224被配置成将一种或更多种材料(例如,核酸样品、扩增子、试剂和/或类似物)转移到核酸测序仪222、样品制备部件218和核酸扩增部件220,和/或从核酸测序仪222、样品制备部件218和核酸扩增部件220转移一种或更多种材料。
关于计算机系统和网络、数据库以及计算机程序产品的另外的细节还提供在以下文献中:例如,Peterson,Computer Networks:A Systems Approach,Morgan Kaufmann,第5版(2011);Kurose,Computer Networking:A Top-Down Approach,Pearson,第7版(2016);Elmasri,Fundamentals of Database Systems,Addison Wesley,第6版(2010);Coronel,Database Systems:Design,Implementation,&Management,Cengage Learning,第11版(2014);Tucker,Programming Languages,McGraw-Hill Science/Engineering/Math,第2版(2006);和Rhoton,Cloud Computing Architected:Solution Design Handbook,Recursive Press(2011),这些文献各自通过引用以其整体并入本文。
实施例
实施例1
使用非肿瘤样品作为背景,用可变肿瘤分数和不稳定位点的数目来计算地模拟MSI高(MSI-H)样品。将在3000个不同癌症类型的样品的队列中观察到的分布用作不稳定位点的数目的先验。该分析证明0.2%的肿瘤含量的检测限(LoD)处的灵敏度为94%。根据本文描述的实施方案确定MSI状态的方法对非肿瘤供体样品的预期特异性为99.999%。这些结果与标准或常规的基于PCR的MSI评估的比较示出了跨越1.4%-15%的肿瘤含量范围的100%一致性。此外,对来自三种癌症类型的155个临床样品测试该分析的性能,对于所述样品,标准的基于PCR的MSI状态评估是可得的(10个MSI-H,145个微卫星稳定(MSS))。根据本文描述的实施方案产生的MSI识别示出了与标准的基于PCR的MSI评估的100%一致性。
实施例2
使用数字测序临床平台(Guardant Health,Inc.,Redwood City,CA,USA)对82个样品的MSI状态进行评估。数字测序平台是一个癌症相关基因的NGS组套检测,其利用从简单、非侵入性血液抽取物分离的无细胞DNA(其可以包括循环肿瘤DNA)的高质量测序来进行。数字测序采用测序前制备单独加标签的cfDNA分子的数字文库,结合测序后的生物信息学重建,以消除几乎所有的假阳性。使用样品中的cfDNA的靶向测序来获得序列信息。确定了每个样品中61个提供最多信息的微卫星基因座的位点评分(ΔAIC)。样品的肿瘤分数的范围为0.5%至15%。将每个样品的位点评分与相应的位点特异性训练阈值进行比较,以鉴定每个样品中不稳定的微卫星基因座的数目。在给定样品中鉴定出的不稳定的微卫星基因座的数目用作该特定样品的微卫星不稳定性评分(即MSI样品评分)。确定样品中61个微卫星基因座的群体训练阈值,其中大于或等于5的微卫星不稳定性评分被预测将样品分类为MSI-高(MSI-H),而小于或等于4的微卫星不稳定性评分被预测将样品分类为微卫星稳定(MSS)。82个样品中的9个样品被分类为MSI-H。其余73个样品被分类为MSS。在所有82个样品中,预测的稳定性状态与预期的稳定性状态相匹配。基于正交验证确认MSI状态。
实施例3
引言
微卫星不稳定性(MSI)是一种经指南推荐的生物标志物,对许多种肿瘤类型具有预后意义,以及对用免疫检查点抑制剂的治疗也具有预测意义。传统上,微卫星不稳定性检测依赖于通过PCR或免疫组织化学测试肿瘤组织。最近,已经开发了下一代测序(NGS)方法,该方法也依赖于肿瘤组织的可得性。相比之下,基于血浆的MSI检测方法可以提供MSI状态的非侵入性实时评估。Guardant Health的大的组套检测(panel)无细胞DNA(cfDNA)NGS测定评估了500个癌症相关基因,以鉴定基因组改变和肿瘤突变负荷(TMB)。除了单核苷酸变异(SNV)、得失位、拷贝数扩增(CNA)、融合和TMB之外,该组套检测可以基于>1,000个MSI位点中的体细胞变化检测微卫星不稳定性高(MSI-高)状态。本实施例中呈现的分析验证具有用于确定500个癌症相关基因cfDNA NGS测定用于MSI-高检测的性能的四个主要组成部分:准确度、检测限(LoD)、精度和空白限(Limit of Blank,LoB)。
方法
准确度分析使用来自3个来源的258个样品,其MSI状态由500个癌症相关基因cfDNA NGS测定进行预测,与基于用正交方法确定的组织MSI状态的真实性进行比较。使用具有组织MSI状态的36个合作者样品(真实:组织MSI状态)、121个健康供体(真实:微卫星稳定,MSS),以及通过500个癌症相关基因cfDNA NGS测定(大的组套检测测定)和73个癌症相关基因cfDNA NGS测定(小的组套检测测定,MSI状态为真实)进行测序的101个样品。再现性和重复性分析使用2组重复(总计56个重复)。比较了运行内和运行间的MSI状态和MSI评分。通过模拟获得两者的LoD。使用健康供体样品和已知的MSS样品计算LoB。
结果
1.准确度分析
在基于组织MSI状态的13个MSI高样品中,12个被大的组套检测测定识别为MSI-高。所有MSS/MSI-低(MSI-L)样品均被正确地检测到(表2)。当限于通过小的组套检测测定覆盖的微卫星区域(~90个位点)时,由大的组套检测测定检测为MSI-高的12个样品也满足被识别为MSI-高的小的组套检测测定阈值。
表2
2.LoD分析
在用于跨越0.05%至1%的范围的5个肿瘤分数检测MSI的>1,000个位点上的模拟指示了0.1%的LoD(图3)。
3.使用500个癌症相关基因cfDNA NGS测定的MSI-高检测是可复现的和可重复的
在两次运行中测试了24个MSI-高的重复。用500个癌症相关基因cfDNA NGS测定将所有重复检测为MSI-高。在运行内和运行间的MSI数值评分为±4(图4A)。10个MSS/MSI低样品具有在同一流动池中测试的2-3个重复(总共32个重复)。用500个癌症相关基因cfDNANGS测定将所有重复检测为MSS/MSI低。每个样品内的MSI评分为±3(图4B)。
4.LoB分析
在LoB分析中使用121个健康供体和25个具有已知MSS状态的合作者样品。用大的组套检测测定将所有146个样品正确地分类为MSS/MSI低,示出0%的假阳性率。
5.肿瘤分数与MSI评分
将多于2,000个大的组套检测测定样品中的MSI评分对体细胞识别(somaticcalls)的最大突变等位基因分数(MAF)制图(图5A和图5B),示出了肿瘤分数(如通过MAF测量的)与MSI状态不相关。
结论
用500个癌症相关基因cfDNA NGS测定的MSI高检测示出了高灵敏度(>90%)和特异性(100%)。运行内和运行间的重复性和再现性是高的。MSI高检测的LoD为0.1%MAF。LoB研究示出了0%的假阳性率。500个癌症相关基因cfDNA NGS测定提供了用cfDNA进行的MSI高状态的可靠预测而无需组织样品,这将给予医师治疗价值。
实施例4
引言
由于微卫星不稳定性(MSI)作为对免疫检查点阻断(ICB)应答的预测性生物标志物的重要性(如由派姆单抗的泛癌批准所例示的(10,11)),微卫星不稳定性(MSI)是美国国家综合癌症网络(National Comprehensive Cancer Network,NCCN)临床实践指南推荐的至少以下九种癌症类型中的生物标志物:宫颈癌、胆管癌、结肠直肠癌、子宫内膜癌、食管和食管胃癌、胃癌、卵巢癌、胰腺癌和前列腺癌(1-9)。患有晚期癌症的患者中的MSI的检测也可以提醒临床医师评估患者的无症状家庭成员的遗传性癌症风险。
MSI是缺陷性DNA错配修复(dMMR)的原型表现,它导致整个基因组的突变率显著增加,包括被称为微卫星区(microsatellite tracts)的重复基序中核苷酸的增加和/或损失,该实体由微卫星区而得名。MSI在子宫内膜癌、结肠直肠癌和胃食管癌中最为盛行,其中它可以是MMR相关基因中散发性突变的后遗症或林奇综合征的表现,林奇综合征是一种最常见地由MLH1、MSH2、MSH6、PMS2或EPCAM中的种系突变引起的遗传性癌症易感性综合征(12)。然而,尽管在这些癌症类型中的盛行率增加,但景观分析已经示出,MSI在大多数其他实体瘤中也以不可忽略的比率发生,所述实体瘤包括常见的肿瘤类型,诸如肺癌、前列腺癌和乳腺癌(13)。
最近的研究已经示出,MSI预测了使用PD-1/PD-L1抑制剂的ICB的临床益处,这导致在若干适应症(MSI存在时)中批准了这些剂,包括用于MSI-高(MSI-H,MSI阳性)转移性结肠直肠癌的纳武单抗±伊匹单抗和用于在先前批准的疗法上进展之后不可切除的或转移性MSI-H实体瘤的派姆单抗。除了作为ICB益处的预测性生物标志物的价值之外,MSI还具有预后意义,最明显地是在结肠直肠癌(CRC)中具有预后意义,在临床实践指南中建议对所有患者进行测试(3,15)。
目前,MSI测试最常见地经由肿瘤组织样本的聚合酶链式反应(PCR)和/或免疫组织化学(IHC)分析来进行。前者评估最初由Bethesda组套检测(16,17)推荐的五个典型微卫星基因座,并且相对于在匹配的非肿瘤DNA中评估的种系基因型比较它们在肿瘤DNA中的长度;每个微卫星区的长度的不稳定性被用作MSI的直接证据。然而,这种有限的微卫星组套检测主要是针对CRC开发的,并且在其他癌症类型中具有更有限的灵敏度(18)。相比之下,IHC方法评估四种MMR蛋白的水平,其中一种或更多种(缺陷性MMR,dMMR)的表达的缺乏与MSI状态强烈相关。然而,约5%至11%的MSI-H病例展示出完整的MMR染色和定位(完整MMR(proficient MMR),pMMR),因为保留了另外的非功能性蛋白质的抗原性和细胞内移行(19)。最近的出版物(20,21)已经证明,下一代测序(NGS)也可以准确地表征肿瘤的MSI状态,允许经由单个NGS测试对可靶向的基因组生物标志物以及MSI状态进行综合谱分析。
尽管在NCCN指南和相关的经FDA批准的治疗选项中存在对许多癌症类型的建议,但目前在CRC和胃食管癌之外的MSI测试率仍然非常低(22)。即使在CRC中,自2005年就具有MSI测试建议的情况下(17,23),不到50%的患者进行了测试(24),这导致错过了ICB治疗机会,并且未能识别出其家庭成员可能处于增加的癌症风险的患者。虽然是多因素的,但这样的MSI基因分型不足(undergenotyping)通常是由于与组织获取和复杂的测试建议/算法相关的障碍。例如,存档诊断样本(archival diagnostic specimens)的测试可能导致与定位和获得该材料相关的显著延迟,并且由于肿瘤进化和/或异质性而导致对MSI状态的不准确评估。类似地,对新获得的组织样本的测试也可能导致与活组织检查的时间安排相关的显著延迟和失败,并且另外还与由于手术并发症的风险和成本相关。因此,侵入性组织获取程序在许多经大量预治疗的和/或虚弱的患者中是禁忌的。此外,快速增长的生物标志物数目和测试选项的多样化给已经负担过重的医师带来令人生畏的复杂性。
无细胞循环肿瘤DNA(ctDNA)测定(“液体活组织检查”)通过实现同时期肿瘤DNA的微创谱分析,成功地解决了许多基因分型适应症中的这样的障碍。因此,通过鉴定其肿瘤携带由于组织取样的限制而原本无法被鉴定的感兴趣的生物标志物的患者,液体活组织检查扩展了患者对标准护理靶向疗法(包括ICB)的可获得性(access)并且比典型的组织测试更快(25)。此外,综合的液体活组织检查可以在单次测试中为所有成人实体瘤提供所有经指南推荐的体细胞基因组生物标志物信息。在本研究中,试图通过加入MSI检测来提高先前验证的基于ctDNA的基因分型测试的效用。此处描述了该平台上MSI评估的设计和验证,报告了其在又进行描述的最大的ctDNA-组织MSI验证队列(n=1145)中的性能,并且评估了用ICB治疗的16例晚期胃癌患者的应答预测。此处还报告了超过28,000名连续实体瘤患者的MSI-H景观,这些患者在经美国临床实验室改进修正案(Clinical LaboratoryImprovement Amendment,CLIA)认证的美国病理学家协会(College of AmericanPathologists,CAP)认可的纽约州卫生局(New York State Department of Health)批准的实验室中进行测试。
材料和方法
1.微卫星基因座选择
Guardant Health的小的组套检测无细胞DNA(cfDNA)NGS测定是一个74个基因的组套检测,先前在所有经指南推荐的晚期实体瘤的适应症中被验证为用于检测SNV、得失位、CNA和融合(26,27)。该测定最初纳入了由长度为7或更长的短串联重复序列(STR)组成的99个推定微卫星基因座,这些基因座被选择为包括对跨越多种肿瘤类型的不稳定性易感的位点,包括五个Bethesda组套检测位点中的三个(BAT-25、BAT-26和NR-21)。不包括剩余的两个Bethesda位点(NR-24和MONO-27),因为所述区域的可映射性极低。这些位点的覆盖度和噪声谱使用来自84个健康供体样品的集合的测序数据进行评估,以将未提供信息的位点从最终MSI检测算法排除。
2.模型描述
MSI检测基于将观察到的读段序列与分子条形码信息整合到单一的概率模型中,该模型将PCR和测序噪声假设情况下观察到的数据的似然性与体细胞MSI不稳定性假设情况下的数据的似然性进行比较。每个个体位点使用赤池信息量准则(AIC)来独立地评分(28)。AIC模型产生基因座评分(范围从0到无穷大),反映了在任何给定微卫星基因座处观察到的可变性是由于生物不稳定性相对于噪声的似然性,并且如果基因座评分(即位点评分)高于位点特异性训练阈值,则基因座被认为是不稳定的。跨越最后的90个位点计算受影响的基因座的数目,并且如果不稳定基因座的数目(“MSI评分”)高于群体训练阈值(n=6),则样品被识别为阳性。个体基因座的阈值和每个样品的总MSI评分使用基于排列(permutation)的模拟用来自健康供体样品的数据和个体基因座的错误参数以及模拟样品中不稳定基因座的总数目来建立,所述健康供体样品中具有不同重复序列长度的分子的频率不同。通过该方法,在此处使用模拟以便询问微卫星长度和不稳定基因座数目的100,000个组合,这允许评估情景的不同景观,其中一些可能在非模拟数据集中未表现出。该算法未区分微卫星稳定(MSS)和MSI-低(通过使用PCR方法观察单个不稳定Bethesda基因座定义的一个类别),将它们分组到单个类别中。这是基于先前的报告,该报告即MSI-L状态不是明显的表型而是测试少量微卫星的伪象,使得当测试大量微卫星基因座时,先前表征的MSI-L样品在总体MSI负荷中拟似MSS表型。
3.样品
MSI算法开发和训练使用模拟数据以及84个健康供体样品的集合进行。临床验证研究包括如先前描述的(26)在Guardant Health CLIA实验室中作为常规护理临床测试标准的一部分被收集和处理的1145份存档样品(余留血浆和/或无细胞DNA),或者包括收集在EDTA血液收集管中的存档患者血浆样品。20个健康供体样品也被用于分析特异性研究。分析验证研究中使用的人为样品包括从细胞系上清液和健康供体血浆提取的cfDNA汇集物。使用从来自以下细胞系的培养物上清液制备的无细胞DNA(ATCC,Inc.):KM12、NCI-H660、HCC1419、NCI-H2228、NCI-H1650、NCI-H1648、NCI-H1975、NCI-H1993、NCIH596、HCC78、GM12878、MCF-7。从细胞系培养物上清液中分离的cfDNA模拟片段尺寸和细胞外释放的机制(29)、文库转化和患者来源的cfDNA的测序性质,同时还提供了足够数量的明确定义的材料的可再生来源,以支持研究的高材料需求,诸如检测限和精度。
4.样品处理和生物信息学分析
从血浆样品或细胞系上清液提取无细胞DNA(QIAmp循环核酸试剂盒,Qiagen,Inc.),并且用非随机寡核苷酸条形码(IDT,Inc.)标记最多30ng的经提取的cfDNA,然后进行文库制备、杂交捕获富集(Agilent Technologies,Inc.),并通过成对末端合成(NextSeq500/550或HiSeq 2500,Illumina,Inc.)进行测序,如先前描述的(26)。进行生物信息学分析和变体检测,如先前描述的(26)。
5.分析验证方法
为了分析验证而进行的研究基于已建立的CLIA、Nex-StoCT工作组和美国分子病理学协会(Association of Molecular Pathologists)/CAP关于性能特征和验证原则的指导。为了确定针对MSI状态的测定的灵敏度,将来自MSI-H细胞系(KM12)(29)的细胞系上清液的cfDNA用来自微卫星稳定(MSS)细胞系(NCI-H660)(30,31)的cfDNA稀释,并且在标准(30ng)cfDNA输入和低(5ng)cfDNA输入两者时进行测试。对于5ng输入,稀释系列靶向0.03%-2%的最大突变等位基因分数(最大MAF),并且对于30ng输入,稀释系列靶向0.01%-1%的最大突变等位基因分数(最大MAF)。使用滴定剂和稀释剂材料独特的已知种系变异来验证靶向肿瘤分数。重复性(运行内精度)和再现性(运行间精度)的评估基于临床和人为模型样品。一定精度的六个临床样品(三个MSI-H和三个MSS)被选择为具有1%-2%的最大MAF值,代表5ng时预测的LoD的~2-3x。MSI分析特异性通过分析20个健康供体样品和245个已知MSS人为样品来确定。
6.临床验证方法
使用ctDNA MSI算法对来自患者的临床样品的存档血浆或cfDNA进行测试,这些样品具有来自标准护理基于组织的MSI测试的可用结果(n=1145)。基于组织的MSI状态源自IHC、PCR,或不太常见地源自NGS。临床结果数据从患者医疗记录中提取,并且由治疗医师进行去标识化(deidentified)。
7.来自28,459个晚期癌症患者样品的血浆MSI状态的景观分析
该队列包括28459个连续晚期癌症患者样品,在他们的临床护理过程中使用73个癌症相关基因cfDNA NGS测定(小的组套检测测定)进行测试。所有的分析都是用去标识化的数据进行的,并且根据经IRB批准的方案进行。评估了该队列中MSI-H跨越16种原发性肿瘤类型的盛行率:膀胱癌、乳腺癌、胆管癌、结肠腺癌、未知原发性癌症、头颈部鳞状细胞癌、肝细胞癌、肺腺癌、非特殊型肺癌、肺鳞状细胞癌、“其他”癌症诊断、胰腺腺癌、前列腺癌、胃腺癌和子宫内膜癌。
8.统计学
使用学生t检验进行统计分析,以分析每个样品的变异数目,并且使用Fisher精确检验进行统计分析以进行比例比较。二项式比例的95%置信区间(CI)的下限和上限使用带有连续性校正的Wilson评分区间计算。
9.伦理学
本研究根据群体机构审查委员会(Quorum Institutional Review Board)批准的方案、利用去标识化的数据进行。
结果
1.MSI算法开发
使用NGS进行ctDNA基因分型的传统挑战包括由于低输入和循环中低肿瘤分数的有效分子捕获(26,27)以及测序和其他技术伪象的纠正。MSI检测提出了另外的挑战,因为需要1)准确反映MSI状态的有效分子捕获、测序和重复基因组区域的映射;2)重复区域内的错误纠正和变异检测;和3)由于MSI引起的信号与非MSI体细胞变异引起的信号的区别,以及在通常受到体细胞不稳定性影响的位点处的强PCR滑移伪象(PCR slippageartifacts)。事实上,技术性PCR错误通常比均聚物位点中的典型测序错误率高至少一个数量级,这就需要迭代位点选择和分子条形码的优化使用以跨越大量候选微卫星基因座实现相关的信噪检测比。
虽然组织测序检测组套通常仅由于大的检测组套尺寸和较长的DNA片段长度而包含足够的提供信息的微卫星基因座(13,32),但此处使用的中等尺寸的ctDNA检测组套和短的无细胞DNA(cfDNA)片段长度需要有目的的微卫星选择和纳入。为了实现这一点,使用文献和组织测序汇编(compendia)提供的迭代方法来评估候选位点,以提供具有最小背景噪声的泛癌MSI检测。候选基因座的清单基于上文使用健康供体cfDNA参考的性能准则进行进一步细化。
基于训练健康供体样品的性能评估,提供信息的基因座被定义为那些被有效捕获、测序和映射的基因座,并且与MSS样品中的微小变异相关(在图6A中以浅灰色示出)。未提供信息的基因座未能捕获、测序或映射,导致不充分的分子呈现(在图6A中以黑色示出),或者在MSS样品中展示出显著的变异,导致过多的伪象信号(在图6A中以深灰色示出)。有趣地,在传统的MSI组织测试(16,17)和一些ctDNA组套检测(33)中使用的BAT-25、BAT-26和NR-21Bethesda基因座相对于其他候选位点表现不佳,并且被排除在最终标志物集合之外(在图6A中以箭头指示)。
使用该方法,选择90个微卫星基因座用于纳入到最终测试形式中:89个单核苷酸重复序列和单个三核苷酸重复序列,所有这些都包括7或大于7的重复序列长度。对独特分子覆盖度分布的评估示出,这些基因座中的65%的基因座具有的覆盖度高于0.5X中值样品覆盖度。
除了有效分子捕获和映射,MSI检测还需要高度准确地区分癌症相关信号和背景噪声,所述背景噪声由于ctDNA被通常发现处于的非常低的等位基因分数时的测序和聚合酶错误(26,27,34)。重要地,使微卫星候选物由于体内细胞复制期间的聚合酶滑移而为MSI检测提供信息的相同重复基因组背景也使它们在体外文库制备和测序期间特别易受相同聚合酶滑移的影响,导致高水平的技术噪声。为了解决这个问题,使用数字测序错误纠正来以高保真度定义微卫星基因座处的真正生物插入-缺失事件,如先前描述的(26,27)。数字测序平台是一个癌症相关基因的NGS组套检测,其利用从简单、非侵入性血液抽取物分离的无细胞DNA(其可以包括循环肿瘤DNA)的高质量测序来进行。数字测序采用测序前制备单独加标签的cfDNA分子的数字文库,结合测序后生物信息学重建,以消除几乎所有的假阳性。
在这些高背景错误重复序列中,相对于标准测序方法,数字测序与每分子测序错误减少100倍相关(图6B),允许有效和准确地重建原始患者血液样品中存在的单独的独特分子的微卫星序列。然后,使用基于排列的健康供体样品的阈值模拟来建立位点特异性MSI状态确定阈值和合计样品水平MSI状态确定阈值。当将这些每位点阈值和每样品阈值与数字测序纠正的效果相结合时,每样品假阳性率估计为~10-7.3。此外,针对临床输入的分布调整的滴定模拟将稳健的MSI检测预测为~0.2%的肿瘤分数,其中此后检测效率显著下降。因此,具有<0.2%的循环肿瘤分数(如由最大体细胞变异等位基因分数定义的)的样品被认为不可评估MSI状态。
2.分析验证研究
为了评估MSI检测的分析灵敏度,将源自MSI-H细胞系KM12的上清液的cfDNA稀释到MSS cfDNA中,靶向五个滴定点,包括15个独立处理的重复,囊括通过上文描述的计算机模拟预测的检测限(LoD)。每个滴定系列都以5ng(可接受的最小cfDNA输入)和30ng(最大且最常见的cfDNA输入)进行分析。使用概率单位分析,95%LoD(LOD95)在5ng输入时被计算为0.4%(图7A),并且在30ng输入时被计算为0.1%(图7B)。
为了评估分析中间精度,分析了四种不同人为材料(两种MSS和两种MSI-H)的重复(图7C)。跨越499个重复,MSI状态的分类一致性为100%(499/499,95%CI 99-100%)),其中MSI-H样品的定量MSI评分的变异系数范围为6.3%-7.2%(表3)。重复性和输入稳健性也通过在5ng、10ng和30ng cfDNA输入时对MSS和MSI-H人为材料的重复测试来评估,这类似地展示出100%的一致性(27/27,95%CI 85-100%,图8和表4)。在72个独立的患者样品重复中确认了临床精度,代表了跨越三个独立批次、天数、操作员和试剂批次处理的MSI评分和肿瘤分数的范围,证明了100%的定性一致性(72/72,95%CI 94-100%),其中基本定量MSI评分的变异系数为2.0-15.2%(表5)。
表3
表4
表5
为了评估分析特异性,对健康供体血浆样品(不同于用于训练的那些样品)、MSS人为材料和MSS患者样品进行分析,以用于伪MSI-H识别。跨越健康供体样品(20/20,95%CI83-100%)、人为材料(245/245,95%CI 98-100%)和患者样品(48/48,95%CI 92-100%)的分析特异性为100%。
3.临床验证研究
由于没有正交的基于cfDNA的方法可用作比较物,临床准确度通过将ctDNA MSI评估与来自医疗记录的MSI状态进行比较来确定,所述MSI状态使用标准护理组织测试(IHC、PCR和NGS方法的混合)对包括40种不同癌症类型的1145个样品来确定,其中15种癌症类型至少具有5个代表性样品(图9)。在949名独特的可评估患者中,ctDNA检测到87%的患者被报告为MSI-H(71/82,95%CI 77-93%)和99.5%的患者被报告为MSS/MSI-L(863/867,95%CI 98.7-99.8%),总体准确度为98.4%(934/949,95%CI 97.3-99.1%),其中阳性预测值(PPV)为95%(71/75,95%CI 86-98%)(图10C,表8)。与计算机模拟建模研究一致,由于低的肿瘤分数,MSI-H检测在被分类为不可评估的样品中是罕见的(0/19)(图10A),这解释了在总的独特患者样品组中的57%(16/28)的观察到的ctDNA-组织不一致性(表6-表9)。对于具有高于1%的肿瘤分数的样品,ctDNA PPA上升至93%(54/58,95%CI 82-98%,表9)。
表6
A.所有样品,无论最大VAF如何
表7
B.排除未检测的肿瘤
表8
C.最大VAF≥0.2%
表9
D.最大VAF≥1%
有趣地,尽管文献(23,35)中报道了IHC和PCR组织测试之间的高度相关性,但在这里,ctDNA和组织MSI状态之间的一致性因组织测试方法而不同(PCR为97.4%(450/462),NGS为98.0%(239/244),并且IHC为83.0%(93/112),图10B和表10和表11)。在进一步研究中,注意到这种不一致性是由于增加的组织IHC阳性ctDNA阴性群体(PCR为2.4%,NGS为2.0%,并且IHC为12.5%,对于IHC-PCR和IHC-NGS,Fisher精确检验p<0.001)和增加的组织IHC阴性ctDNA阳性群体(PCR为0.2%,NGS为0%,并且IHC为4.5%,两个比较的Fisher精确检验p<0.01)两者。这些差异促使我们去研究IHC限制是否可能导致观察到的IHC-ctDNA不一致性,而不是受损的ctDNA准确度。在IHC和另一个组织测试结果可得的25个样品中,12个样品展示出IHC-ctDNA不一致性。重要地,在12个不一致性的5个不一致性中,PCR和/或NGS组织测试支持ctDNA NGS结果,而不是组织IHC。总之,这些数据支持先前的报道,即IHC在MSI确定中可能不如PCR诊断原型可靠(36)。
表10
A.所有样品
表11
B.可评估的
4. 28,459名连续晚期癌症患者中的ctDNA MSI状态
虽然许多研究已经评估了组织中MSI跨越不同肿瘤类型的盛行率(13,32,37),但迄今为止,还没有已公开的跨越癌症类型的对ctDNA MSI状态的景观分析。为此,上文描述的MSI算法被应用于在Guardant Health临床实验室中测试的28,459个连续晚期癌症患者的临床样品。在该队列中,包括16种不同肿瘤类型的278个样品(肿瘤分数中值为6.55%,范围为0.09%-89%)通过ctDNA被鉴定为MSI-H,这对应于~1%的总体泛癌盛行率,与先前关于组织所报告的(13,32,37)相似。类似地,MSI-H在肿瘤类型中的盛行率也密切地反映了在基于组织的分析中所观察到的(图11A);如预期的,MSI-H在子宫内膜癌、结肠直肠癌和胃癌中最盛行,而其他肿瘤诸如肺癌、膀胱癌和头颈癌展示出较低的盛行率。先前MSI-H盛行率估计的具体例外包括子宫内膜癌、结肠直肠癌和胃癌中的盛行率略低,并且前列腺癌中的盛行率略高。
鉴于ctDNA预期使用群体的泛实体瘤性质和批准用于MSI-H肿瘤的免疫疗法,该组微卫星基因座被有意选择为对跨越所有实体瘤类型的MSI状态提供信息。与该设计意图一致,对样品水平和基因座水平的MSI评分分布的分析—即分别为MSI评分和位点评分(图11B和图11C)展示出跨越肿瘤类型的一致表现,其中MSI-H样品展示出显著高于阈值的信号。此外,除了通常测试MSI的肿瘤类型之外,MSI评估的诊断结果是可观的;超过一半的经鉴定的病例(143/278)发生在MSI测试非常罕见的肿瘤类型中,并且因此经鉴定的患者可能从未被测试过。
与组织中所报告的(38)一致,相对于那些被表征为具有MSS状态的样品,MSI-H样品中的得失位和SNV(包括非同义变异和同义变异)的数目显著增加(图12)。具体地,MSI-H样品中SNV的中位数为6.3,对比MSS中的1.4(卡方p<0.0001),并且MSI-H样品中得失位的中位数为2.6,对比MSS中的0.4(卡方p<0.0001)。
5.ctDNA MSI状态预测免疫疗法应答
当今MSI状态的最显著的效用是其选择患者进行免疫疗法的能力。尽管如此以及在许多患者中获得组织的障碍,ctDNA MSI状态预测免疫疗法应答的能力还没有被报道。为了建立针对该生物标志物的临床有效性,我们呈现了在胃癌的II期派姆单抗试验(NCT#02589496)中标准护理化疗失败后使用派姆单抗(n=15)或纳武单抗(n=1)治疗的16名ctDNA MSI-H转移性胃癌患者的临床结果。在预治疗的样品中进行的cfDNA和组织PCR MSI评估对于MSI-H是100%一致的(16/16,95%CI 76-100%)。16名患者中的10名达到按RECIST 1.1准则的经研究人员评估的完全(n=3)客观应答或经研究人员评估的部分(n=7)客观应答,其中另外3名患者具有稳定的疾病(图13A),客观应答率为63%(10/16,95%CI36-84%),并且疾病控制率为81%(13/16,95%CI 54-95%),与先前关于由组织测试定义的MSI-H患者报道的应答(39)相似。重要地,即使在这种预治疗的群体中,这些应答也是持久的,具有39周的平均治疗持续时间。事实上,例如,患者21在氟嘧啶/铂化疗失败之后,在派姆单抗治疗后经历疾病的完全消退,并且在完成35个周期的疗法后超过6个月仍然是无疾病的(图13B-图13E)。
讨论
一种新的基于cfDNA的靶向NGS方法被验证用于MSI检测—通过使用大的微卫星组,该方法相对于基于组织的方法实现了高灵敏度,同时保持非常高的特异性。血浆检测的MSI-H跨越16种常见实体瘤的盛行率与已发表的基于组织的汇编相似,证明了用MSI检测算法设计所预期的泛肿瘤性能。此外,通过示出如通过cfDNA检测的MSI-H患者以与关于组织定义的群体所报道的(39)类似方式从ICB疗法中获益展示了临床效用,将MSI检测的可用性扩展到所有患者,而不管组织可得性如何或是否需要经历侵入性组织获取程序。
本实施例展示了MSI检测对ctDNA检测组套的稳健分析性能,该ctDNA检测组套先前在所有经指南推荐的适应症中对其他四种变异类型的检测进行了验证(26)。特别地,人为样品中MSI检测的分析灵敏度展示出可重复检测达0.1%,与先前关于得失位和SNV的相似灵敏度的报道(26)一致。重要地,本实施例评估了ctDNA MSI测试在具有正交组织MSI的1145个样品中的性能,所述样品构成了又进行描述的最大的ctDNA-组织MSI一致性队列。相对于用于相同患者的标准护理组织MSI测试,ctDNA MSI评估展示出高PPV(95%),这与针对基于局部组织对比基于中心组织的MSI评估报道的90%-92%的经报道的PPV(36)媲美,并且在可评估群体中展示出高PPA(87%),这与先前检查其他变异类型的血浆和组织基因分型的一致性的研究(25,26,45,46)一致。可导致不完全一致性的因素可能包括肿瘤异质性、原发性对比转移性病变的不同脱落、组织和血浆收集的时间不一致性以及一些肿瘤的低肿瘤脱落(40,44,47-49)。有趣地,在本报告中,通过血浆和五重PCR鉴定为MSI-H的胃癌患者先前被报告为包括MSS和MSI-H疾病的离散肿瘤群体,如通过对组织进行的IHC和PCR两者评估的(40)。同一项研究发现,在同一名患者的成对组织活组织检查之间的MSI-H的9%不一致性(40),这突出了肿瘤内异质性对MSI状态中的不一致性的潜在贡献。此外,在本报告中观察到的PCR和IHC组织方法之间的有意义的不一致性突出了准确MSI测试的重要性,这已被报道为ICB失败的主要来源(36)。与晚期实体瘤中组织基因分型呈现的挑战一致,一项对转移性非小细胞肺癌(NSCLC)的研究已经示出,相对于组织,基于血浆的测试增加了具有成功的肿瘤基因分型结果的患者数目,以及检测可靶向突变的频率,同时比典型的组织基因分型结果(25,45)至少快一周返回结果。
本实施例呈现了MSI在大的晚期泛癌队列中的首次基于ctDNA的景观分析。总的来说,在>28,000个连续临床样品的集合中,跨肿瘤类型的相对盛行率与已经关于组织所报道的盛行率(13,32,37)一致,仅有微小的差异。例如,CRC和子宫内膜癌中的盛行率低于关于组织所报道的盛行率(13),这很可能反映了基于组织的景观分析包括大量早期MSI-H肿瘤的事实,这些早期MSI-H肿瘤具有较好的预后(15)并且不太可能是用ctDNA测试的晚期癌症群体的一部分。另一方面,MSI-H前列腺癌的高于预期的盛行率可归因于晚期患者中增加的MSI-H疾病的表现;最近两项集中于晚期前列腺癌中MSI状态的研究已经示出在该患者群体中3.1%和3.8%的MSI-H盛行率(50,51),这与本研究中观察到的2.6%相似。不出意料地,鉴于泛癌MSI检测的设计意图,景观分析没有揭示肿瘤类型特异性微卫星不稳定性模式。然而,这并不排除这样的可能性,即在血浆中,类似于在组织中所示出的(37,52),肿瘤类型特异性模式可以随着大量微卫星基因座和大量代表性MSI-H样品的评估而出现。
此处报告的临床结果限于胃癌;然而,观察到的客观应答率与来自基于组织的研究的预期是一致的,表明基于cfDNA MSI结果的ICB治疗应达到跨越实体瘤类型的预期结果。此外,种系dMMR数据的缺乏阻碍了关于cfDNA检测的MSI的家族意义的结论。最后,大多数具有cfDNA-/组织+不一致性的患者的治疗数据不可得,但预期至少一些患者已经接受基于肿瘤结果的ICB疗法,这可能抑制MSI-H疾病并且导致缺乏通过cfDNA的MSI检测。因此,相对于组织,MSI-H检测的87%灵敏度可能在未经治疗或未接受疗法的患者中更高。未来的研究应致力于解决这些问题。
总之,已经开发并验证了基于cfDNA的靶向NGS组套检测,其准确地评估MSI状态,同时还提供全面的肿瘤基因分型,允许泛实体瘤指南-以高灵敏度、特异性和精度从单次外周血液抽取物完成测试。使用与组织测试的比较、群体水平的患病率分析以及首次报告的用ICB疗法治疗的cfDNA MSI-H患者的结果两者进行的临床验证支持了这种方法的临床准确性和相关性。这样的从简单的外周血液抽取物同时表征MSI状态和肿瘤基因型具有将靶向疗法和免疫疗法两者扩展到所有晚期癌症患者的潜力,所述晚期癌症患者包括目前基于组织的测试模式对其是不充分的那些患者。
参考文献
1.Koh W-J,Abu-Rustum NR,Bean S,Bradley K,Campos SM,Cho KR,etal.Cervical Cancer,Version 3.2019,NCCN Clinical Practice Guidelines inOncology.J Natl Compr Canc Netw.2019;17:64–84.
2.Benson,Al B.,D’Angelica,Michael I.,Abbott,Daniel E.,Abrams,ThomasA.,Alberts,Steven R.,Anaya,Daniel A.,et al.Hepatobiliary Cancers,Version4.2018:Featured Updates to the NCCN Guidelines.National Comprehensive CancerNetwork Clinical Practice Guidelines in Oncology[Internet].2018;Availablefrom:https://www.nccn.org/professionals/physician_gls/pdf/hepatobiliary.pdf.
3.Benson,Al B.,Venook,Alan P.,Bekaii-Saab,Tanios,Chan,Emily,Chen,Yi-Jen,Cooper,Harry S.,et al.Colon Cancer Version 1.2016,NCCN PracticeGuidelines in Oncology.2015;Available from:https://www.nccn.org/professionals/physician_gls/pdf/colorectal.pdf.
4.Koh W-J,Abu-Rustum NR,Bean S,Bradley K,Campos SM,Cho KR,etal.Uterine Neoplasms,Version 1.2018,NCCN Clinical Practice Guidelines inOncology.J Natl Compr Canc Netw.2018;16:170–99.
5.Ajani,Jaffer A.,D’Amico,Thomas A.,Baggstrom,Maria,Bentrem,David J.,Chao,Joseph,Das,Prajnan,et al.Esophageal and Esophagogastric Junction CancersVersion 4.2017.2017;Available from:https://www.nccn.org/professionals/physician_gls/pdf/esophageal.pdf.
6.Ajani,Jaffer A.,D’Amico,Thomas A.,Baggstrom,Maria,Bentrem,David J.,Chao,Joseph,Das,Prajnan,et al.Gastric Cancer,Version 5.2017,NCCN ClinicalPractice Guidelines in Oncology.2017;Available from:https://www.nccn.org/professionals/physician_gls/pdf/gastric.pdf.
7.Armstrong,Deborah K.,Alvarez,Ronald D.,Bakkum-Gamez,Jamie N.,Barroilhet,Lisa,Behbakht,Kian,Berchuck,Andrew,et al.NCCN Guidelines Version1.2019 Ovarian Cancer.2019;Available from:https://www.nccn.org/professionals/physician_gls/pdf/ovarian.pdf.
8.Tempero,Margaret A.,Malafa,Mokenge P.,Al-Hawary,Mahmoud,Asbun,Horacio,Bain,Andrew,Behrman,Stephen W.,et al.Pancreatic AdenocarcinomaVersion 2.2018,NCCN Clinical Practice Guidelines in Oncology.2018;Availablefrom:httpshttps://www.nccn.org/professionals/physician_gls/pdf/pancreatic.pdf.
9.Mohler JL,Lee RJ,Antonarakis ES,Armstrong AJ,D’Amico AV,Davis BJ,etal.NCCN Guidelines Version 1.2018 Prostate Cancer[Internet].NCCN ClinicalPractice Guidelines in Oncology(NCCN Guidelines).2018.Available from:https://www.nccn.org.
10.Diaz LA,Le DT.PD-1 Blockade in Tumors with Mismatch-RepairDeficiency.N Engl J Med.2015;373:1979.
11.Le DT,Durham JN,Smith KN,Wang H,Bartlett BR,Aulakh LK,etal.Mismatch-repair deficiency predicts response of solid tumors to PD-1blockade.Science.2017.
12.Buza N,Ziai J,Hui P.Mismatch repair deficiency testing in clinicalpractice.Expert Rev Mol Diagn.2016;16:591–604.
13.Bonneville R,Krook MA,Kautto EA,Miya J,Wing MR,Chen H-Z,etal.Landscape of Microsatellite Instability Across 39 Cancer Types.JCO PrecisOncol.2017.
14.Marcus L,Lemery SJ,Keegan P,Pazdur R.FDA Approval Summary:Pembrolizumab for the treatment of microsatellite instability-high solidtumors.Clin Cancer Res.2019.
15.Benson AB,Arnoletti JP,Bekaii-Saab T,Chan E,Chen Y-J,Choti MA,etal.Colon cancer.J Natl Compr Canc Netw.2011;9:1238–90.
16.Boland CR,Thibodeau SN,Hamilton SR,Sidransky D,Eshleman JR,BurtRW,et al.A National Cancer Institute Workshop on Microsatellite Instabilityfor cancer detection and familial predisposition:development of internationalcriteria for the determination of microsatellite instability in colorectalcancer.Cancer Res.1998;58:5248–57.
17.Umar A,Boland CR,Terdiman JP,Syngal S,de la Chapelle A,Rüschoff J,et al.Revised Bethesda Guidelines for hereditary nonpolyposis colorectalcancer(Lynch syndrome)and microsatellite instability.J Natl Cancer Inst.2004;96:261–8.
18.Lu Y,Soong TD,Elemento O.A novel approach for characterizingmicrosatellite instability in cancer cells.PLoS ONE.2013;8:e63056.
19.Dudley JC,Lin M-T,Le DT,Eshleman JR.Microsatellite Instability asa Biomarker for PD-1 Blockade.Clin Cancer Res.2016;22:813–20.
20.Salipante SJ,Scroggins SM,Hampel HL,Turner EH,PritchardCC.Microsatellite instability detection by next generation sequencing.ClinChem.2014;60:1192–9.
21.Latham A,Srinivasan P,Kemel Y,Shia J,Bandlamudi C,Mandelker D,etal.Microsatellite Instability Is Associated With the Presence of LynchSyndrome Pan-Cancer.J Clin Oncol.2019;37:286–95.
22.Guardant Health,Inc.Tissue Findings Submitted with Guardant360Test Requisitions–Data on File.Redwood City,California;2019.
23.Hampel H,Frankel WL,Martin E,Arnold M,Khanduja K,Kuebler P,etal.Screening for the Lynch syndrome(hereditary nonpolyposis colorectalcancer).N Engl J Med.2005;352:1851–60.
24.Shaikh T,Handorf EA,Meyer JE,Hall MJ,Esnaola NF.Mismatch RepairDeficiency Testing in Patients With Colorectal Cancer and Nonadherence toTestingGuidelines in Young Adults.JAMAOncol.2017;e173580.
25.Leighl NB,Page RD,Raymond VM,Daniel DB,Divers SG,Reckamp KL,etal.Clinical Utility of Comprehensive Cell-Free DNA Analysis to IdentifyGenomic Biomarkers in Patients with Newly Diagnosed Metastatic Non-Small CellLung Cancer.Clinical Cancer Research.2019;clincanres.0624.2019.
26.Odegaard JI,Vincent JJ,Mortimer S,Vowles JV,Ulrich BC,Banks KC,etal.Validation of a Plasma-Based Comprehensive Cancer Genotyping AssayUtilizing Orthogonal Tissue-and Plasma-Based Methodologies.Clin CancerRes.2018;24:3539–49.
27.Lanman RB,Mortimer SA,Zill OA,Sebisanovic D,Lopez R,Blau S,etal.Analytical and Clinical Validation of a Digital Sequencing Panel forQuantitative,Highly Accurate Evaluation of Cell-Free Circulating TumorDNA.PLoS ONE.2015;10:e0140712.
28.Akaike H.Information Theory and an Extension of the MaximumLikelihood Principle.In:Petrov BN,Csaki F,editors.Proceedings of the 2ndInternational Symposium on Information Theory(pp 267-281)Budapest:AkademiaiKiado.1973.
29.Berg KCG,Eide PW,Eilertsen IA,Johannessen B,Bruun J,Danielsen SA,et al.Multi-omics of 34 colorectal cancer cell lines-a resource forbiomedical studies.Mol Cancer.2017;16:116.
30.Cosmic.COSMIC-Catalogue of Somatic Mutations in Cancer[Internet].[cited 2019 Apr 17].Available from:https://cancer.sanger.ac.uk/cosmic.
31.Forbes SA,Beare D,Boutselakis H,Bamford S,Bindal N,Tate J,etal.COSMIC:somatic cancer genetics at high-resolution.Nucleic AcidsResearch.2017;45:D777–83.
32.Hause RJ,Pritchard CC,Shendure J,Salipante SJ.Classification andcharacterization of microsatellite instability across 18 cancer types.NatMed.2016;22:1342–50.
33.Georgiadis A,Wood D,Murphy D,Parpart-Li S,Riley D,Sengamalay N,etal.Abstract 1286:Analytical validation of an integrated next-generationsequencing pan-cancer liquid biopsy approach for detection of microsatelliteinstability.Cancer Res.2018;78:1286.
34.Zill OA,Banks KC,Fairclough SR,Mortimer SA,Vowles JV,Mokhtari R,etal.The Landscape of Actionable Genomic Alterations in Cell-Free CirculatingTumor DNA from 21,807 Advanced Cancer Patients.Clin Cancer Res.2018;24:3528–38.
35.Lindor NM,Burgart LJ,Leontovich O,Goldberg RM,Cunningham JM,Sargent DJ,et al.Immunohistochemistry versus microsatellite instabilitytesting in phenotyping colorectal tumors.J Clin Oncol.2002;20:1043–8.
36.Cohen R,Hain E,Buhard O,Guilloux A,Bardier A,Kaci R,etal.Association of Primary Resistance to Immune Checkpoint Inhibitors inMetastatic Colorectal Cancer With Misdiagnosis of Microsatellite Instabilityor Mismatch Repair Deficiency Status.JAMA Oncology.2019;5:551.
37.Vanderwalde A,Spetzler D,Xiao N,Gatalica Z,MarshallJ.Microsatellite instability status determined by next-generation sequencingand compared with PD-L1 and tumor mutational burden in 11,348 patients.CancerMedicine.2018;7:746–56.
38.Bonneville R,Krook MA,Kautto EA,Miya J,Wing MR,Chen H-Z,etal.Landscape of Microsatellite Instability Across 39 Cancer Types.JCO PrecisOncol.2017;2017.
39.Le DT,Uram JN,Wang H,Bartlett BR,Kemberling H,Eyring AD,et al.PD-1Blockade in Tumors with Mismatch-Repair Deficiency.New England Journal ofMedicine.2015;372:2509–20.
40.Kim ST,Cristescu R,Bass AJ,Kim K-M,Odegaard JI,Kim K,etal.Comprehensive molecular characterization of clinical responses to PD-1inhibition in metastatic gastric cancer.Nat Med.2018;24:1449–58.
41.Accordino MK,Wright JD,Buono D,Neugut AI,Hershman DL.Trends in useand safety of image-guided transthoracic needle biopsies in patients withcancer.J Oncol Pract.2015;11:e351-359.
42.Lokhandwala T,Bittoni MA,Dann RA,D’Souza AO,Johnson M,Nagy RJ,etal.Costs of Diagnostic Assessment for Lung Cancer:A Medicare ClaimsAnalysis.Clin Lung Cancer.2017;18:e27–34.
43.De Mattos-Arruda L,Weigelt B,Cortes J,Won HH,Ng CKY,Nuciforo P,etal.Capturing intratumor genetic heterogeneity by de novo mutation profilingof circulating cell-free tumor DNA:a proof-of-principle.Ann Oncol.2014;25:1729–35.
44.Goyal L,Saha SK,Liu LY,Siravegna G,Leshchiner I,Ahronian LG,etal.Polyclonal Secondary FGFR2 Mutations Drive Acquired Resistance to FGFRInhibition in Patients with FGFR2 Fusion-Positive Cholangiocarcinoma.CancerDiscov.2017;7:1–12.
45.Aggarwal C,Thompson JC,Black TA,Katz SI,Fan R,Yee SS,etal.Clinical Implications of Plasma-Based Genotyping With the Delivery ofPersonalized Therapy in Metastatic Non-Small Cell Lung Cancer.JAMAOncol.2018.
46.Siravegna G,Lazzari L,Crisafulli G,Sartore-Bianchi A,Mussolin B,Cassingena A,et al.Radiologic and Genomic Evolution of Individual Metastasesduring HER2 Blockade in Colorectal Cancer.Cancer Cell.2018;34:148-162.e7.
47.Pectasides E,Stachler MD,Derks S,Liu Y,Maron S,Islam M,etal.Genomic Heterogeneity as a Barrier to Precision Medicine inGastroesophageal Adenocarcinoma.Cancer Discov.2018;8:37–48.
48.Thompson JC,Yee SS,Troxel AB,Savitch SL,Fan R,Balli D,etal.Detection of Therapeutically Targetable Driver and Resistance Mutations inLung Cancer Patients by Next-Generation Sequencing of Cell-Free CirculatingTumor DNA.Clin Cancer Res.2016;22:5772–82.
49.Sacher AG,Komatsubara KM,Oxnard GR.Application of plasmagenotyping technologies in non-small cell lung cancer:a practical review.JThorac Oncol.2017.
50.Mayrhofer M,De Laere B,Whitington T,Van Oyen P,Ghysel C,Ampe J,etal.Cell-free DNA profiling of metastatic prostate cancer revealsmicrosatellite instability,structural rearrangements and clonalhematopoiesis.Genome Med.2018;10:85.
51.Abida W,Armenia J,Gopalan A,Brennan R,Walsh M,Barron D,etal.Prospective Genomic Profiling of Prostate Cancer Across Disease StatesReveals Germline and Somatic Alterations That May Affect Clinical DecisionMaking.JCO Precision Oncology.2017;1–16.
52.Hause RJ,Pritchard CC,Shendure J,Salipante SJ.Classification andcharacterization of microsatellite instability across 18cancer types.NatureMedicine.2016;22:1342–50.
虽然为了清楚与理解的目的,已经通过说明和实例的方式对前述公开内容进行了一些详细描述,但是本领域普通技术人员通过阅读本公开内容将会清楚,在不偏离本公开内容的实际范围的情况下,可以进行形式和细节上的各种改变,并且可以在所附权利要求的范围内实践。例如,所有方法、系统、计算机可读介质和/或组件特征、步骤、元件或其他方面都可以以各种组合来使用。
本文引用的所有专利、专利申请、网站、其他出版物或文件、登录号等都为了所有目的通过引用以其整体并入,其程度如同每个单独的项目都被具体且单独地指示通过引用如此并入一样。如果一个序列的不同版本在不同时间与一个登录号相关联,则意指在本申请的有效申请日期与该登录号相关联的版本。如果适用的话,有效申请日期意指真实申请日期或提及该登录号的优先权申请的申请日期中较早的一个。同样,如果出版物、网站等的不同版本在不同时间发布,则意指在本申请的有效申请日期最近发布的版本,除非另有指示。
- 无细胞DNA中的微卫星不稳定性检测
- 用于检测癌症中的微卫星不稳定性和DNA碱基切除修复途径抑制的合成致死性的新标记
