一种生物化学法制备索马鲁肽的方法
文献发布时间:2023-06-19 12:18:04
技术领域
本发明属于重组多肽的制备领域,特别涉及利用化学和生物结合的方法制备重组多肽的技术领域,更为具体的说是涉及生物化学法制备索马鲁肽的方法。
背景技术
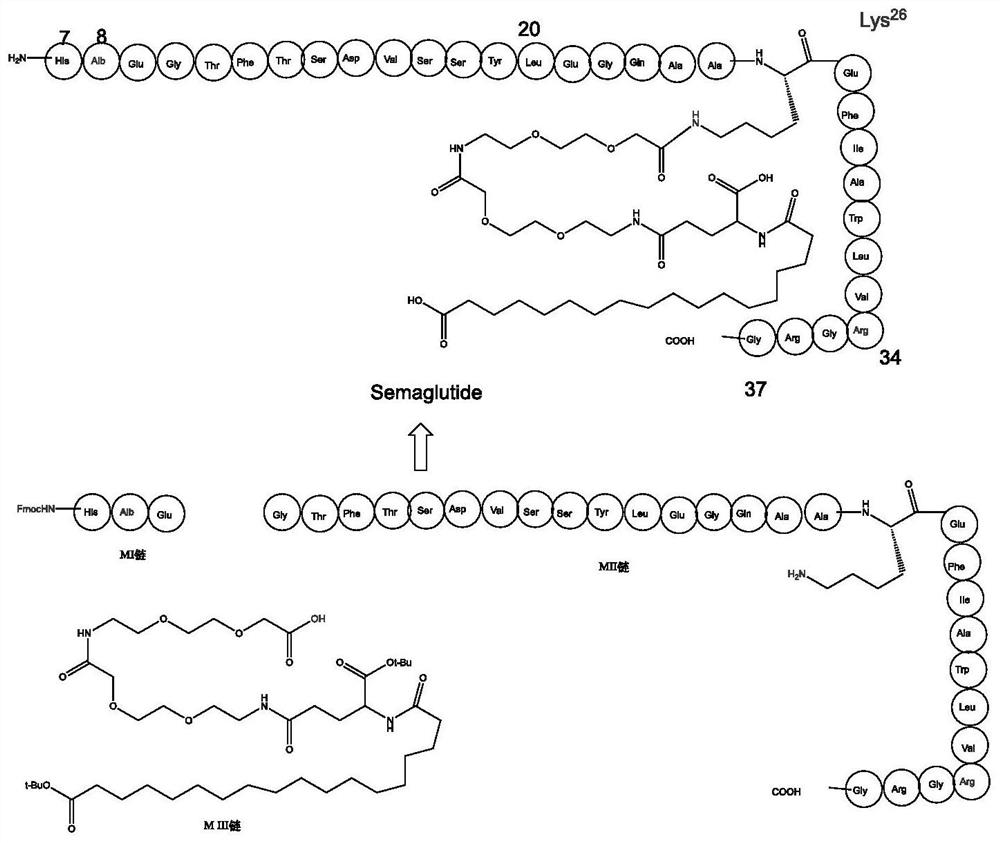
索马鲁肽是一种GLP-1类似物,分子量约4113.57754(CAS#910463-68-2)。由含有31位氨基酸的多肽链与一条化学侧链组成,其化学结构式如下所示:
GLP-1是由机体肠道细胞分泌的一种具有葡萄糖依赖性降糖作用的活性多肽,由37 个氨基酸构成,L细胞分泌的GLP-1有两种生物活性形式分别为GLP-1(7-37)和 GLP-1(7-36)酰胺,这两者仅有一个氨基酸序列不同。两类多肽通过作用于相同受体发挥相同的活性。完整的GLP-1活性多肽,由于DPP-4的作用,在体内很快代谢,因此,要将GLP-1应用于糖尿病的治疗,首先需要解决的是其半衰期过短的问题。
索马鲁肽正是基于这样的需求而产生的化合物。将索马鲁肽的结构与GLP-1的结构进行比对,可以看到,在索马鲁肽中,其8位氨基酸进行了化学修饰,26位氨基酸Lys 增加了化学侧链,34位氨基酸为Arg。经过验证,修饰后的GLP-1即索马鲁肽应用在糖尿病治疗中时可以大幅度延长半衰期至一周仅一次用药。
索马鲁肽的原研公司是诺和诺德,作为全球第七个获批的GLP-1受体激动剂,在与礼来的度拉糖肽的对头试验中,索马鲁肽在降糖和减重两项指标中双双胜出,不良反应也较轻。该药于2017年12月获FDA批准上市,被誉为史上最好的降糖药。
索马鲁肽原研专利(WO2006097537)制备方法是采用纯化学合成的方法,在微波辅助合成仪上,采用标准的Fmoc策略、在树脂上通过氨基酸顺序对接来进行固相肽 [Aib8,Arg34]GLP-1-(7-37)-肽合成,并通过制备级HPLC纯化;连接侧链,17-((S)-1- 叔丁氧基羰基-3-{2-[2-({2-[2-(2,5-二氧吡咯烷-1-基氧基羰基甲氧基)乙氧基]乙基氨基甲酰}丙基氨基甲酰)十七烷酸叔丁酯与主链多肽链连接后获得索马鲁肽。由于在制备过程中多肽主链的合成主要采用固相法,因此需要使用大量有机溶剂,对环境不友好。同时,这种化学合成的方法在长链多肽的制备中也存在产率低、成本高等问题
发明内容
本发明所要解决的技术问题是提供一种新的索马鲁肽中间体的制备方法,从而符合绿色生产要求,降低对环境的影响,同时该方法还必须能够满足生产成本低,适合工业化推广使用的要求。
为了解决上述技术问题,本发明公开了生物化学法制备索马鲁肽的方法,所述方法的合成路线如下:
其中MI肽链通过化学合成法合成;MII肽链通过大肠杆菌发酵制备而成;MIII肽链通过化学合成法合成;
MI肽链、MII肽链、MII肽链经化学法连接形成索马鲁肽;
其中MII肽链为Lys
不同于现有技术中的化学合成法或者生物酵母制备法,本发明所使用的是生物制备体系,并且是大肠杆菌生物制备体系。相较于化学法,大肠杆菌生物制备体系具有条件温和、对环境友好、产物中杂质少等优势,相较于生物酵母制备法,本发明所公开的大肠杆菌体系具有快速表达、生产效率高、培养条件更简单等优势。
进一步优选地,所述MII肽链通过融合蛋白表达,所述融合蛋白的DNA序列包含含有His-tag基因序列的TrxA DNA序列和如SEQ ID NO.3所示的MII链DNA序列。
本发明发明人首次将大肠杆菌生物制备体系用于索马鲁肽的合成中,但是在具体的应用中发现,由于目的多肽链MII链在水中极难溶解(溶解度<1mg/mL),且肽链片段过短,直接合成后分离纯化难度较高,因此,在本发明中通过在N端添加融合蛋白从而达到促进溶解表达的技术效果。本发明通过添加TrxA融合蛋白及His-tag,在促进溶解表达的同时,进一步还提供了标签纯化位点,有效降低了目的肽链的生产成本。
进一步优选地,所述融合蛋白中还添加有酶切位点,优选地该酶切位点为TEV酶切位点。改进后优选的融合蛋白的DNA序列如SEQ ID NO.1所示,其氨基酸序列如SEQ ID NO.5所示。
通过TEV酶酶切位点去融合标签具有肠激酶等常规酶无法比拟的快速、高效、专一性强等优点,但是遗憾的是,TEV酶酶切会在目的多肽蛋白上残留一个氨基酸Gly,如果再纯化去掉该残留氨基酸将极大的增加生产成本。但是在本发明中发明人发现通过对索马鲁肽合成路线合理的设计,可以利用该残留氨基酸作为MII肽链起始氨基酸,从而无需后续的纯化流程,大大节约生产成本和生产时间。
本发明还公开了一种重组表达载体,该重组表达载体含有编码融合蛋白的编码基因 SEQ ID NO.1。
进一步,本发明还公开了所述的重组表达载体为环状载体,其启动子为T7promoter,操纵子为乳糖操纵子,该载体序列如SEQ ID NO.2所示,其质粒构建图如图1中所示。该质粒的构建方式能够使工程菌细胞在被IPTG诱导的情况下高效表达,从而进一步提高生物制备效率。
进一步优选地,本发明所公开的大肠杆菌优选为E.coli BL21(DE3)。
同时,在本发明中还公开了一种索马鲁肽中间体Lys
S1:合成包含含有His-tag基因的TrxADNA序列和TEV酶切位点序列和M II链序列的编码基因,
S2:将编码基因连接到表达载体中,
S3:将带有编码基因的重组表达载体转化入大肠杆菌,构建重组工程菌;
S4:将重组工程菌接种、培养,诱导融合蛋白表达;
S5:收集包涵体;
S6:酶切去标签获得具有MII肽链的多肽粗品。
优选地,在步骤S2与S3之间还包含有融合蛋白目的基因确认步骤。进一步优选的,该确认采用PCR确认方法,具体为:设计上下游引物F: 5'-TAATACGACTCACTATAGGG-3'R:5'-GCTAGTTATTGCTCAGCGG-3'。PCR条件为:98℃5min,98℃30s,55℃90s、72℃90s,36个循环;PCR扩增体系:模板1.5μL,上下游引物各1.5μL,灭菌的双蒸水20.5μL,PrimerSTAR Mix 25μL;PCR产物在琼脂糖凝胶制作的胶板上电泳确认后,送检测序,确认融合蛋白DNA 序列是否无误。
作为一种优选的技术方案,S3中将带有编码基因的重组表达载体转化入大肠杆菌,构建重组工程菌的方法具体为:将构建好的载体通过电击1S,转入表达细胞E.coliBL21(DE3)中,涂布在含卡那霉素的LB平板中,放入37℃培养箱中过夜,将长出来的单菌落进行质粒提取和测序,最终获得含该酯酶基因的重组工程菌确定阳性工程菌;
进一步优选地,S4中将重组工程菌接种、培养,诱导融合蛋白表达的具体方法为:重组阳性工程菌菌株种子液按照1~5%的接种量转接于LB培养基中,37℃培养至 OD600=0.6时,加入50μl 0.5mol/L的IPTG,15~37℃诱导4~16h;
作为一种优选的技术方案,发酵后离心收集菌株,每1g湿菌体重悬于10mL裂解缓冲液(50mM Tris-Hcl,50mM NaCl,5%glycerol,pH 8.0,17μl(10mg/mL PMSF)),超声波破碎离心获得上清液与沉淀,收集上清液;
进一步优选地,还包括有融合蛋白的纯化步骤。
具体的,所述纯化的具体方法为:将该上清液与镍柱吸附缓冲液体积比1:1~5体积比(10mM咪唑,50mM NaCl,50mMTris-HCl pH8.0)混匀,镍柱吸附,两倍柱体积吸附缓冲液冲洗,20mM咪唑缓冲液(20mM咪唑,50mM NaCl,50mMTris-HCl pH8.0) 再一次冲洗去杂蛋白,洗脱缓冲液进行目的融合蛋白洗脱(250mM咪唑,50mM NaCl,50mMTris-HCl pH8.0);
作为一种优选的技术方案还包括有浓缩融合蛋白步骤,具体为融合蛋白蛋白MW约为20.62KDa,使用Amicon超滤管10KDa和30KDa超滤离心,进一步去除>30KDa 和<10KDa杂蛋白,并浓缩融合蛋白;
作为一种优选的技术方案,在S6中酶切去标签获得具有MII肽链的多肽粗品的具体方法为:将上述融合蛋白进行BCA定量后透析至酶切缓冲液(50mM NaCl,50mMTris-HCl,5mM EDTA,and 1mM DTT pH8.0)进行酶切,酶切条件如下: 3μg蛋白加入1单位TEV酶,25~35℃酶切1~8h,纯化去标签,冻干获得索马鲁肽中间体28位氨基酸多肽粗品。
这里的纯化方法可以参考前述纯化方法,也可以使用Akta纯化仪或液相法。
作为一种进一步优选的技术方案,还包括有融合蛋白包涵体的再利用步骤。具体的,该再利用的方法为,向细胞破碎离心后的沉淀中添加2M尿素50mM pH8.0的Tris-HCl 缓冲液进行杂蛋白洗脱,离心收集沉淀,加入含有8M尿素50mM pH8.0的Tris-HCl缓冲液进行变性溶解,然后按照前述纯化、浓缩方法处理后,进一步酶切、去标签,冻干得到MII肽链粗品。
在本发明中还公开了在合成索马鲁肽产物的过程中MII肽链先与MII肽链经化学法连接后再与MI肽链连接形成索马鲁肽。该合成顺序是一种较为优选的合成方式,通过该合成方式可以提高合成的有效性和产物稳定性,提高产物得率。
本发明首次提出并实现了以大肠杆菌生物制备体系为基础的索马鲁肽生物化学制备方法。通过创造性地将索马鲁肽主肽链31个氨基酸拆分为3位氨基酸多肽链(MI链) 和28位氨基酸多肽链(MII链)从而实现了大肠杆菌制备体系下索马鲁肽制备的可能性。采用本发明公开的技术方案后,能够快速、高效、高专一性地获得索马鲁肽产品,同时由于无需使用大量化学试剂,因此是一种绿色的,环境友好型制备方法。
附图说明
图1是质粒图谱示意图。
图2是索马鲁肽生物—化学法路线示意图。
图3是BCA标准曲线图。
图4是实施例3中和实施例4中细胞破碎获得的沉淀进行蛋白电泳实验结果示意图。
图5是M II链纯品液相图谱。
图6是M II链纯品质谱。
具体实施方式
为了更好的理解本发明,下面结合具体的实施例和附图对本发明进行进一步的阐述。
实施例1融合蛋白表达质粒的构建
设计表达载体序列(全序列如SEQ ID No.2所示),外送进行质粒的DNA序列合成,质粒中包含编码基因,所述编码基因包含TrxA基因和TEV酶切位点序列和M II 链序列;合成质粒后,通过PCR进行确认融合蛋白目的基因合成无误,设计上下游引物F:5'-TAATACGACTCACTATAGGG-3'R:5'-GCTAGTTATTGCTCAGCGG-3'。PCR 条件为:98℃5min,98℃30s,56℃90s、72℃90s,36个循环;PCR扩增体系:模板1.5μL,上下游引物各1.5μL,灭菌的双蒸水20.5μL,PrimerSTAR Mix 25 μL;PCR产物跑胶确认后,送检测序,确认融合蛋白DNA序列无误,进行工程菌构建。
实施例2表达工程菌制备及阳性验证
将构建好的载体通过电击1S,转入表达细胞E.coli BL21(DE3)中,涂布在含卡那霉素的LB平板中,放入37℃培养箱中过夜,将长出来的单菌落进行质粒提取和测序,最终获得含该酯酶基因的重组工程菌确定阳性工程菌。
实施例3融合蛋白的表达与纯化
将实施例2重组阳性工程菌菌株种子液按照2%的接种量转接于LB培养基中,37℃培养至OD600=0.6时,加入50μl 0.5mol/L的IPTG,15℃诱导16h,离心收集菌株,每1g湿菌体重悬于10mL裂解缓冲液(50mM Tris-HCl,50mM NaCl,5%glycerol,pH 8.0,17μl(10mg/mL PMSF)),超声波破碎离心获得上清液与沉淀。收集上清液,1:1 与镍柱吸附缓冲液(10mM咪唑,50mM NaCl,50mMTris-HCl pH8.0)(混匀),镍柱吸附(预装柱,市购),两倍柱体积吸附缓冲液冲洗,20mM咪唑缓冲液(20mM咪唑, 50mM NaCl,50mMTris-HCl pH8.0)再一次冲洗去杂蛋白。洗脱缓冲液进行目的融合蛋白洗脱(250mM咪唑,50mM NaCl,50mMTris-HClpH8.0)。将收集的洗脱液与缓冲液 (150mM NaCl,20mMTris-HCl,pH8.0)混匀稀释后,使用Amicon超滤管(10KD) 5000rpm离心1h,收集上清液加入Amicon超滤管(30KD)5000rpm离心1h,收集下层溶液,经BCA法检测,浓缩蛋白含量约为943.069μg/mL(BCA标曲见附图3)。
实施例4融合蛋白的表达与纯化
将实施例2重组阳性工程菌菌株种子液按照2%的接种量转接于LB培养基中,37℃培养至OD600=0.6时,加入50μl 0.5mol/L的IPTG,37℃诱导4h,离心收集菌株,每 1g湿菌体重悬于10mL裂解缓冲液(50mM Tris-HCl,50mM NaCl,5%glycerol,pH 8.0,17μl(10mg/mL PMSF)),超声波破碎离心获得上清液与沉淀。收集上清液,1:1 与镍柱吸附缓冲液(10mM咪唑,50mM NaCl,50mMTris-HCl pH8.0)(混匀),镍柱吸附(预装柱,市购),两倍柱体积吸附缓冲液冲洗,20mM咪唑缓冲液(20mM咪唑, 50mM NaCl 50mMTris-HCl pH8.0)再一次冲洗去杂蛋白。洗脱缓冲液进行目的融合蛋白洗脱(250mM咪唑,50mM NaCl,50mMTris-HClpH8.0)。将收集的洗脱液与缓冲液 (150mM NaCl,20mMTris-HCl,pH8.0)混匀稀释后,使用Amicon超滤管(10KD) 5000rpm离心1h,收集上清液加入Amicon超滤管(30KD)5000rpm离心1h,收集下层溶液,经BCA法检测,蛋白含量约为171.782μg/mL(BCA标曲见附图3)。
实施例5融合蛋白酶切
将实施例3获得的融合蛋白通过透析袋透析至酶切缓冲液中,酶切反应缓冲液体系: 50mM NaCl,50mMTris-HCl,5mM EDTA,1mM DTT,5%Glycerol pH8.0),酶切条件如下:3μg蛋白加入1单位TEV酶(市购),25℃酶切1h,上柱去标签,获得的目的蛋白索马鲁肽中间体Lys26Arg34GLP-1(10-37)(M II链)粗品,送检液相,经液相检测,酶切转化效率约为57%。
实施例6融合蛋白酶切
将实施例3获得的融合蛋白通过透析袋透析至酶切缓冲液中,酶切反应缓冲液体系: 50mM NaCl,50mMTris-HCl,5mM EDTA,1mM DTT,5%Glycerol pH8.0,酶切条件如下: 3μg蛋白加入1单位TEV酶(市购),30℃酶切4h,上柱去标签,获得的目的蛋白索马鲁肽中间体Lys26Arg34GLP-1(10-37)(M II链)粗品,送检液相,经液相检测,酶切转化效率约为91%。
实施例7融合蛋白包涵体再利用
索马鲁肽中间体MII链过纯品进行溶解性实验,发现其水中溶解度<1mg/mL,极难溶解,将实施例3中和实施例4中细胞破碎获得的沉淀进行蛋白电泳实验(如图4所示),发现沉淀中还残留有大量未能溶解形成包涵体的融合蛋白浪费。在该包涵体中添加1M 尿素50mM pH8.0的Tris-HCl缓冲液进行杂蛋白洗脱,离心收集沉淀,加入含有8M尿素50mMpH8.0的Tris-HCl缓冲液进行变性溶解后,参考实施例3进行上镍柱纯化,将获得的纯化变性蛋白1:14的比例向含有融合蛋白的变性液中加入复性液(50mM Tris-HCl,5mM EDTA,10%甘油,pH9.5),4℃下温和搅拌10-12h,4000rpm、4℃离心10min,获得上清。参考实施例3进行纯化并浓缩,将获得的融合蛋白通过透析袋透析至酶切缓冲液中,酶切反应缓冲液体系:50mM NaCl,50mMTris-HCl,5mM EDTA,1 mM DTT,5%Glycerol pH8.0,酶切条件如下:3μg蛋白加入1单位TEV酶(市购), 30℃酶切4h,上柱去标签,获得的目的蛋白索马鲁肽中间体Lys26Arg34GLP-1(10-37) (M II链)粗品,送检液相,经液相检测,酶切转化效率约93%。MII链粗品通过流动相A:0.1%三氟乙酸水溶液;流动相B:0.1%乙腈溶液,制备液相分离精制,冻干后提纯。
实施例8制备索马鲁肽
将发酵得到的Lys26Arg34GLP-1(10-37)精制(精制后产物的液相谱图及质谱谱图如图5和图6所示),并取8.94g加入反应瓶中,先加入355ml水,再加入16ml DIPEA。搅拌混合物并不断监测pH,至pH为10.6时,停止搅拌。然后在10分钟内,滴加溶有固相制备所得的MIII肽链的乙腈溶液(3.66g/270ml)。搅拌反应混合物1小时后,真空除去乙腈,将溶液冻干,得到中间产物。
将所得残余物(即中间产物)溶于NMP(355ml)中,然后加DIPEA(8ml)和固相制备所得的MI肽链(含保护基)4.6g。搅拌反应混合物4小时后,再加入一份固相制备所得的MI链(含保护基)2.24g,搅拌混合物过夜(≥16小时)。
加入预先冷至0℃的乙醚7L,形成白色沉淀。通过离心分离沉淀,沉淀用3.5L乙醚洗涤后在室温下干燥(或采用鼓风干燥机,在室温下干燥),得到粗制中间体。
将粗制中间体溶于TFA-三异丙基甲硅烷-水(95:2.5:2.5,3.5L)并搅拌3小时,然后将溶液真空浓缩至约350ml。加入预先冷至0℃的乙醚7L,有沉淀形成,离心分离,沉淀用乙醚洗涤后干燥,得到粗品索马鲁肽。
将粗品用乙酸-水(3:7,3.5L)洗涤,经HPLC纯化后冻干得到索马鲁肽精制产物,即为本实施例中的目标产品。
以上所述是本发明的具体实施方式。应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也视为本发明的保护范围。
序列表
<110> 南京林业大学,南京欧信医药技术有限公司
<120> 一种生物化学法制备索马鲁肽的方法
<130> 202110030
<160> 5
<170> SIPOSequenceListing 1.0
<210> 1
<211> 579
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 1
atgagcgata aaattattca cctgactgac gacagttttg acacggatgt actcaaagcg 60
gacggggcga tcctcgtcga tttctgggca gagtggtgcg gtccgtgcaa aatgatcgcc 120
ccgattctgg atgaaatcgc tgacgaatat cagggcaaac tgaccgttgc aaaactgaac 180
atcgatcaaa accctggcac tgcgccgaaa tatggcatcc gtggtatccc gactctgctg 240
ctgttcaaaa acggtgaagt ggcggcaacc aaagtgggtg cactgtctaa aggtcagttg 300
aaagagttcc tcgacgctaa cctggccggt tctggttctg gccatatgca ccatcatcat 360
catcattctt ctggtctggt gccacgcggt tctggtatga aagaaaccgc tgctgctaaa 420
ttcgaacgcc agcacatgga cagcccagat ctgggtaccg gtggtggttc cggtgaaaac 480
ttgtatttcc aaggaacctt taccagtgat gtgtctagct atctggaagg tcaggcagcc 540
aaagaattta ttgcatggtt agttcgcggt cgtggctaa 579
<210> 2
<211> 5940
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 2
tggcgaatgg gacgcgccct gtagcggcgc attaagcgcg gcgggtgtgg tggttacgcg 60
cagcgtgacc gctacacttg ccagcgccct agcgcccgct cctttcgctt tcttcccttc 120
ctttctcgcc acgttcgccg gctttccccg tcaagctcta aatcgggggc tccctttagg 180
gttccgattt agtgctttac ggcacctcga ccccaaaaaa cttgattagg gtgatggttc 240
acgtagtggg ccatcgccct gatagacggt ttttcgccct ttgacgttgg agtccacgtt 300
ctttaatagt ggactcttgt tccaaactgg aacaacactc aaccctatct cggtctattc 360
ttttgattta taagggattt tgccgatttc ggcctattgg ttaaaaaatg agctgattta 420
acaaaaattt aacgcgaatt ttaacaaaat attaacgttt acaatttcag gtggcacttt 480
tcggggaaat gtgcgcggaa cccctatttg tttatttttc taaatacatt caaatatgta 540
tccgctcatg agacaataac cctgataaat gcttcaataa tattgaaaaa ggaagagtat 600
gagtattcaa catttccgtg tcgcccttat tccctttttt gcggcatttt gccttcctgt 660
ttttgctcac ccagaaacgc tggtgaaagt aaaagatgct gaagatcagt tgggtgcacg 720
agtgggttac atcgaactgg atctcaacag cggtaagatc cttgagagtt ttcgccccga 780
agaacgtttt ccaatgatga gcacttttaa agttctgcta tgtggcgcgg tattatcccg 840
tattgacgcc gggcaagagc aactcggtcg ccgcatacac tattctcaga atgacttggt 900
tgagtactca ccagtcacag aaaagcatct tacggatggc atgacagtaa gagaattatg 960
cagtgctgcc ataaccatga gtgataacac tgcggccaac ttacttctga caacgatcgg 1020
aggaccgaag gagctaaccg cttttttgca caacatgggg gatcatgtaa ctcgccttga 1080
tcgttgggaa ccggagctga atgaagccat accaaacgac gagcgtgaca ccacgatgcc 1140
tgcagcaatg gcaacaacgt tgcgcaaact attaactggc gaactactta ctctagcttc 1200
ccggcaacaa ttaatagact ggatggaggc ggataaagtt gcaggaccac ttctgcgctc 1260
ggcccttccg gctggctggt ttattgctga taaatctgga gccggtgagc gtgggtctcg 1320
cggtatcatt gcagcactgg ggccagatgg taagccctcc cgtatcgtag ttatctacac 1380
gacggggagt caggcaacta tggatgaacg aaatagacag atcgctgaga taggtgcctc 1440
actgattaag cattggtaac tgtcagacca agtttactca tatatacttt agattgattt 1500
aaaacttcat ttttaattta aaaggatcta ggtgaagatc ctttttgata atctcatgac 1560
caaaatccct taacgtgagt tttcgttcca ctgagcgtca gaccccgtag aaaagatcaa 1620
aggatcttct tgagatcctt tttttctgcg cgtaatctgc tgcttgcaaa caaaaaaacc 1680
accgctacca gcggtggttt gtttgccgga tcaagagcta ccaactcttt ttccgaaggt 1740
aactggcttc agcagagcgc agataccaaa tactgtcctt ctagtgtagc cgtagttagg 1800
ccaccacttc aagaactctg tagcaccgcc tacatacctc gctctgctaa tcctgttacc 1860
agctgccagt ggcgataagt cgtgtcttac cgggttggac tcaagacgat agttaccgga 1920
taaggcgcag cggtcgggct gaacgggggg ttcgtgcaca cagcccagct tggagcgaac 1980
gacctacacc gaactgagat acctacagcg tgagctatga gaaagcgcca cgcttcccga 2040
agggagaaag gcggacaggt atccggtaag cggcagggtc ggaacaggag agcgcacgag 2100
ggagcttcca gggggaaacg cctggtatct ttatagtcct gtcgggtttc gccacctctg 2160
acttgagcgt cgatttttgt gatgctcgtc aggggggcgg agcctatgga aaaacgccag 2220
caacgcggcc tttttacggt tcctggcctt ttgctggcct tttgctcaca tgttctttcc 2280
tgcgttatcc cctgattctg tggataaccg tattaccgcc tttgagtgag ctgataccgc 2340
tcgccgcagc cgaacgaccg agcgcagcga gtcagtgagc gaggaagcgg aagagcgcct 2400
gatgcggtat tttctcctta cgcatctgtg cggtatttca caccgcatat atggtgcact 2460
ctcagtacaa tctgctctga tgccgcatag ttaagccagt atacactccg ctatcgctac 2520
gtgactgggt catggctgcg ccccgacacc cgccaacacc cgctgacgcg ccctgacggg 2580
cttgtctgct cccggcatcc gcttacagac aagctgtgac cgtctccggg agctgcatgt 2640
gtcagaggtt ttcaccgtca tcaccgaaac gcgcgaggca gctgcggtaa agctcatcag 2700
cgtggtcgtg aagcgattca cagatgtctg cctgttcatc cgcgtccagc tcgttgagtt 2760
tctccagaag cgttaatgtc tggcttctga taaagcgggc catgttaagg gcggtttttt 2820
cctgtttggt cactgatgcc tccgtgtaag ggggatttct gttcatgggg gtaatgatac 2880
cgatgaaacg agagaggatg ctcacgatac gggttactga tgatgaacat gcccggttac 2940
tggaacgttg tgagggtaaa caactggcgg tatggatgcg gcgggaccag agaaaaatca 3000
ctcagggtca atgccagcgc ttcgttaata cagatgtagg tgttccacag ggtagccagc 3060
agcatcctgc gatgcagatc cggaacataa tggtgcaggg cgctgacttc cgcgtttcca 3120
gactttacga aacacggaaa ccgaagacca ttcatgttgt tgctcaggtc gcagacgttt 3180
tgcagcagca gtcgcttcac gttcgctcgc gtatcggtga ttcattctgc taaccagtaa 3240
ggcaaccccg ccagcctagc cgggtcctca acgacaggag cacgatcatg cgcacccgtg 3300
gggccgccat gccggcgata atggcctgct tctcgccgaa acgtttggtg gcgggaccag 3360
tgacgaaggc ttgagcgagg gcgtgcaaga ttccgaatac cgcaagcgac aggccgatca 3420
tcgtcgcgct ccagcgaaag cggtcctcgc cgaaaatgac ccagagcgct gccggcacct 3480
gtcctacgag ttgcatgata aagaagacag tcataagtgc ggcgacgata gtcatgcccc 3540
gcgcccaccg gaaggagctg actgggttga aggctctcaa gggcatcggt cgagatcccg 3600
gtgcctaatg agtgagctaa cttacattaa ttgcgttgcg ctcactgccc gctttccagt 3660
cgggaaacct gtcgtgccag ctgcattaat gaatcggcca acgcgcgggg agaggcggtt 3720
tgcgtattgg gcgccagggt ggtttttctt ttcaccagtg agacgggcaa cagctgattg 3780
cccttcaccg cctggccctg agagagttgc agcaagcggt ccacgctggt ttgccccagc 3840
aggcgaaaat cctgtttgat ggtggttaac ggcgggatat aacatgagct gtcttcggta 3900
tcgtcgtatc ccactaccga gatatccgca ccaacgcgca gcccggactc ggtaatggcg 3960
cgcattgcgc ccagcgccat ctgatcgttg gcaaccagca tcgcagtggg aacgatgccc 4020
tcattcagca tttgcatggt ttgttgaaaa ccggacatgg cactccagtc gccttcccgt 4080
tccgctatcg gctgaatttg attgcgagtg agatatttat gccagccagc cagacgcaga 4140
cgcgccgaga cagaacttaa tgggcccgct aacagcgcga tttgctggtg acccaatgcg 4200
accagatgct ccacgcccag tcgcgtaccg tcttcatggg agaaaataat actgttgatg 4260
ggtgtctggt cagagacatc aagaaataac gccggaacat tagtgcaggc agcttccaca 4320
gcaatggcat cctggtcatc cagcggatag ttaatgatca gcccactgac gcgttgcgcg 4380
agaagattgt gcaccgccgc tttacaggct tcgacgccgc ttcgttctac catcgacacc 4440
accacgctgg cacccagttg atcggcgcga gatttaatcg ccgcgacaat ttgcgacggc 4500
gcgtgcaggg ccagactgga ggtggcaacg ccaatcagca acgactgttt gcccgccagt 4560
tgttgtgcca cgcggttggg aatgtaattc agctccgcca tcgccgcttc cactttttcc 4620
cgcgttttcg cagaaacgtg gctggcctgg ttcaccacgc gggaaacggt ctgataagag 4680
acaccggcat actctgcgac atcgtataac gttactggtt tcacattcac caccctgaat 4740
tgactctctt ccgggcgcta tcatgccata ccgcgaaagg ttttgcgcca ttcgatggtg 4800
tccgggatct cgacgctctc ccttatgcga ctcctgcatt aggaagcagc ccagtagtag 4860
gttgaggccg ttgagcaccg ccgccgcaag gaatggtgca tgcaaggaga tggcgcccaa 4920
cagtcccccg gccacggggc ctgccaccat acccacgccg aaacaagcgc tcatgagccc 4980
gaagtggcga gcccgatctt ccccatcggt gatgtcggcg atataggcgc cagcaaccgc 5040
acctgtggcg ccggtgatgc cggccacgat gcgtccggcg tagaggatcg agatctcgat 5100
cccgcgaaat taatacgact cactataggg gaattgtgag cggataacaa ttcccctcta 5160
gaaataattt tgtttaactt taagaaggag atatacatat gagcgataaa attattcacc 5220
tgactgacga cagttttgac acggatgtac tcaaagcgga cggggcgatc ctcgtcgatt 5280
tctgggcaga gtggtgcggt ccgtgcaaaa tgatcgcccc gattctggat gaaatcgctg 5340
acgaatatca gggcaaactg accgttgcaa aactgaacat cgatcaaaac cctggcactg 5400
cgccgaaata tggcatccgt ggtatcccga ctctgctgct gttcaaaaac ggtgaagtgg 5460
cggcaaccaa agtgggtgca ctgtctaaag gtcagttgaa agagttcctc gacgctaacc 5520
tggccggttc tggttctggc catatgcacc atcatcatca tcattcttct ggtctggtgc 5580
cacgcggttc tggtatgaaa gaaaccgctg ctgctaaatt cgaacgccag cacatggaca 5640
gcccagatct gggtaccggt ggtggttccg gtgaaaactt gtatttccaa ggaaccttta 5700
ccagtgatgt gtctagctat ctggaaggtc aggcagccaa agaatttatt gcatggttag 5760
ttcgcggtcg tggctaactc gagcaccacc accaccacca ctgagatccg gctgctaaca 5820
aagcccgaaa ggaagctgag ttggctgctg ccaccgctga gcaataacta gcataacccc 5880
ttggggcctc taaacgggtc ttgaggggtt ttttgctgaa aggaggaact atatccggat 5940
<210> 3
<211> 87
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 3
ggaaccttta ccagtgatgt gtctagctat ctggaaggtc aggcagccaa agaatttatt 60
gcatggttag ttcgcggtcg tggctaa 87
<210> 4
<211> 28
<212> PRT
<213> 人工序列(Artificial Sequence)
<400> 4
Gly Thr Phe Thr Ser Asp Val Ser Ser Tyr Leu Glu Gly Gln Ala Ala
1 5 10 15
Lys Glu Phe Ile Ala Trp Leu Val Arg Gly Arg Gly
20 25
<210> 5
<211> 192
<212> PRT
<213> 人工序列(Artificial Sequence)
<400> 5
Met Ser Asp Lys Ile Ile His Leu Thr Asp Asp Ser Phe Asp Thr Asp
1 5 10 15
Val Leu Lys Ala Asp Gly Ala Ile Leu Val Asp Phe Trp Ala Glu Trp
20 25 30
Cys Gly Pro Cys Lys Met Ile Ala Pro Ile Leu Asp Glu Ile Ala Asp
35 40 45
Glu Tyr Gln Gly Lys Leu Thr Val Ala Lys Leu Asn Ile Asp Gln Asn
50 55 60
Pro Gly Thr Ala Pro Lys Tyr Gly Ile Arg Gly Ile Pro Thr Leu Leu
65 70 75 80
Leu Phe Lys Asn Gly Glu Val Ala Ala Thr Lys Val Gly Ala Leu Ser
85 90 95
Lys Gly Gln Leu Lys Glu Phe Leu Asp Ala Asn Leu Ala Gly Ser Gly
100 105 110
Ser Gly His Met His His His His His His Ser Ser Gly Leu Val Pro
115 120 125
Arg Gly Ser Gly Met Lys Glu Thr Ala Ala Ala Lys Phe Glu Arg Gln
130 135 140
His Met Asp Ser Pro Asp Leu Gly Thr Gly Gly Gly Ser Gly Glu Asn
145 150 155 160
Leu Tyr Phe Gln Gly Thr Phe Thr Ser Asp Val Ser Ser Tyr Leu Glu
165 170 175
Gly Gln Ala Ala Lys Glu Phe Ile Ala Trp Leu Val Arg Gly Arg Gly
180 185 190
- 一种生物化学法制备索马鲁肽的方法
- 一种基于可溶性疏水标记载体的液相法制备索马鲁肽的方法
