免疫检测方法
文献发布时间:2024-01-17 01:27:33
本申请为2021年12月21日提交的、申请号为202111572524.4的中国专利申请的分案申请,在此通过引用将其全文并入本文。
技术领域
本发明涉及蛋白A变体,尤其是具有高耐碱性的蛋白A变体。本发明还涉及该蛋白A变体的应用。
背景技术
近年来,抗体药物发展迅速。2020年,全球销售额前10位的药物中有一半是抗体药物。抗体药物生产工艺中的纯化步骤通常采用经典的三步或两步层析法。金黄色葡萄球菌蛋白A是上世纪70年代发现的能与多物种抗体Fc端进行结合的菌体蛋白,自发现以来就一直作为重要的抗体纯化层析配体进行研究,目前也是商业化较完善、应用较为广泛的亲和配基。研究发现,蛋白A配基上多个区域对抗体Fc段独特的专一吸附能力,可用作抗体的特异性捕获,一步工艺即可去除细胞发酵液中大部分的杂质,可达98%以上的纯度。随着抗体药物的发展,用作抗体捕获的亲和层析介质也取得了快速发展。
亲和层析介质的选择与产品的质量息息相关。例如早期蛋白A(Protein A)亲和层析配基脱落严重,不易清洗,若这些工艺相关杂质不能在后续工艺中很好地去除,则存在引起免疫反应的风险,进而对药品质量提出很大挑战。因此,外国填料生产商很早就开始致力于增加介质的配基稳定性、结合载量和碱耐受性。例如目前主流蛋白A亲和层析介质如Cytiva公司的MabSelect SuRe,为耐碱层析介质,可耐0.1-0.5M NaOH清洗,从而高效、低成本地除去附着在填料上的蛋白沉淀物、疏水性蛋白、核酸、内毒素和病毒等。配基稳定性也得到了增强,配基脱落<100ppm。动态结合载量达到30g/L以上。目前国内抗体药物生产企业大都采用进口蛋白A亲和层析介质。而这些进口蛋白A亲和层析介质通常价格昂贵,约占抗体药物下游纯化工艺中层析介质总成本的85%,并且存在各种可改进的不足之处。例如,MabSelect SuRe的基架微球为高交联的琼脂糖,优点是亲水性极佳,生物相容性好,非常适合抗体类生物大分子的分离纯化;但缺点是:其一,结构偏软,粒径分布较宽,在实际使用过程中存在装柱重复性差,机械强度低等问题;其二,Mabselect Sure在生物制药中一般使用0.1M NaOH清洗,其耐碱性能还有差距;其三,MabSelect Sure的动态载量>30g/L,对于大规模高表达量抗体生产所需的载量还有一定差距。又如,Millipore公司的Prosep UltraPlus也是一种使用较为广泛的蛋白A亲和层析介质,基质为孔道可控的玻璃珠结构,物理稳定好,载量很高。但是,玻璃珠结构有一致命缺陷,即不可耐受高浓度NaOH清洗,因而限制其在大规模生物样品中应用。
正因为进口填料昂贵的价格以及各种不足,因此有必要开发国产高效低成本的蛋白A亲和填料。
发明内容
本文提供了一种免疫检测方法,包括使用耐碱蛋白A变体,所述耐碱蛋白A变体包括
1)SEQ ID NO:1-3、5-10中任一个所示的氨基酸序列;或
2)与SEQ ID NO:1-3、5-10中任一个所示的氨基酸序列有至少90%序列同一性的氨基酸序列。
在一些实施方案中,所述耐碱蛋白A变体与IgG抗体结合的KD值不高于1×10
在一些实施方案中,所述IgG抗体为IgG1分子。
在一些实施方案中,所述耐碱蛋白A变体的氨基酸序列如SEQ ID NO:1所示。
在一些实施方案中,所述耐碱蛋白A变体的氨基酸序列如SEQ ID NO:3所示。
在一些实施方案中,所述耐碱蛋白A变体的氨基酸序列如SEQ ID NO:10所示。
在一些实施方案中,所述耐碱蛋白A变体为融合蛋白形式,所述融合蛋白包括串联连接的2个、3个、4个、5个或更多个所述耐碱蛋白A变体。
在一些实施方案中,所述耐碱蛋白A变体偶联有示踪物。
在一些实施方案中,所述示踪物选自荧光素、酶、胶体金和铁蛋白。
附图说明
图1为蛋白A结构示意图。其中S为信号序列、E、D、B、A、C为五个IgG结合结构域,X和M序列与细胞壁附着有关。图中还显示了Z结构域,其为B结构域的突变版本,相对于B结构域包括G29A突变。
图2为本发明蛋白A变体定向优化方法的流程图。
图3为本发明获得的蛋白A变体目标序列与初始序列间的同一性矩阵图。
图4为用于蛋白A变体的表达的pET28a(+)载体图谱。
图5为纯化后蛋白A变体的电泳图。
图6显示了本发明蛋白A变体动态载量的测定结果。
具体实施方式
除非另有说明,本文使用的所有技术和科学术语具有本领域普通技术人员所通常理解的含义。
蛋白A(protein A,SpA)为一种来自金黄色葡萄球菌(Staphylococcus aureus)的细胞壁蛋白。天然蛋白A含有五个高度同源的结构域,分别为E、D、A、B和C结构域(图1)。每个结构域都能够与人或其他哺乳动物如小鼠、猪、狗、牛等的免疫球蛋白G(IgG)的Fc段结合,这种结合通常不影响IgG分子中的Fab片段与抗原特异性结合的能力。天然蛋白A在与微球(如琼脂糖)偶联后或以其他方式固定后被广泛用于抗体分子的纯化。由于天然蛋白A的五个结构域与相同抗体分子的结合强度会稍有差异,导致洗脱条件不一,经常需要在不同pH或变性剂浓度条件下对纯化产物进行洗脱,导致操作复杂或抗体分子因经受不了这样的条件而失活。因此,有研究者例如采用基因工程技术对蛋白A基因进行改造,仅使用某一种或几种特定的结构域进行串联,使获得的重组蛋白A具有期望的结合亲和力或其他特性(例如耐碱性)。
“结构域(domain)”指较大的蛋白分子中在空间上可明显区分的、相对独立的区域性结构。对于较小的蛋白分子来说,结构域常常等同于其三级结构。结构域可以具有特定的功能,例如与配体结合(例如蛋白A的B结构域与IgG结合),或者酶学活性等。在本文中,除非另有说明,结构域也可指多肽片段或仅包括一个结构域的蛋白本身。
“蛋白A变体”通常指具有IgG结合能力而在氨基酸序列上不同于天然蛋白A或其特定结构域的蛋白或蛋白结构域,例如可相对天然蛋白A包括一个或更多个氨基酸突变(例如替换、缺失、插入等)。在本文中,“蛋白A变体”指发明人在蛋白A和其他蛋白基础上进行定向优化后获得的目标蛋白(或多肽片段),其在氨基酸序列上明显不同于天然蛋白A或其结构域,例如氨基酸序列比对后同一性不高于85%;另外,其具有耐碱性,并且可以具有IgG结合能力,或也可以不具有IgG结合能力。
“耐碱蛋白A变体”指能够耐受碱处理的蛋白A变体。这里的“耐碱”指蛋白A变体接触碱性溶液(例如0.1、0.2、0.3、0.4或0.5M NaOH溶液)一段时间或一定次数后仍保持其结合IgG分子的能力。优选地,耐碱蛋白A变体在接触碱性溶液(例如0.1或0.5M NaOH溶液)10次(例如50次、100次、150次或甚至180次)后仍保持其结合IgG分子的亲和力的50%以上、60%以上、70%以上、80%以上或甚至90%以上。
“Z结构域”为天然蛋白A的B结构域经改造后获得的也具有IgG结合能力的结构域。Z结构域相对于结构域B包括G29A突变。该突变使得Z结构域相对于B结构域对某些化学试剂(例如,CNBr、羟胺等)的稳定性,并且提高了与IgG结合的能力。关于Z结构域的更多信息,可参见Nilsson B等的描述(A Synthetic IgG-Binding Domain Based on StaphylococcalProtein A.Protein Eng.1987,1(2):107–113,在此通过引用将其全文并入本文。
提及氨基酸或核苷酸序列时,术语“序列同一性(Sequence identity)”(也称为“序列一致性”)指两氨基酸或核苷酸序列(例如查询序列和参照序列)之间一致性程度的量,一般以百分比表示。通常,在计算两氨基酸或核苷酸序列之间的一致性百分比之前,先进行序列比对(alignment)并引入缺口(gap)(如果有的话)。如果在某个比对位置,两序列中的氨基酸残基或碱基相同,则认为两序列在该位置一致或匹配;两序列中的氨基酸残基或碱基不同,则认为在该位置不一致或错配。在一些算法中,用匹配位置数除以比对窗口中的位置总数以获得序列一致性。在另一些算法中,还将缺口数量和/或缺口长度考虑在内。出于本发明的目的,可以采用公开的比对软件BLAST(可在网页ncbi.nlm.nih.gov找到),通过使用缺省设置来获得最佳序列比对并计算出两氨基酸或核苷酸序列之间的序列一致性。
在一些实施方案中,本文提供的蛋白A变体可包括与SEQ ID NO:1-3、5-10所示序列中任一个有至少90%序列同一性(例如,至少95%、至少98%、至少99%或甚至100%序列一致性)的氨基酸序列。
在一些实施方案中,本文提供的蛋白A变体的编码DNA序列可包括与SEQ ID NO:11-13、15-20所示序列中任一个有至少90%序列同一性(例如,至少95%、至少98%、至少99%或甚至100%序列一致性)的核苷酸序列。
本领域技术人员可以理解的是,在本文提供的具体氨基酸序列基础上,可以通过对少数氨基酸进行替换(例如保守性替换)、删除、添加并验证或筛选所得产物与IgG结合能力和/或耐碱能力,从而获得本文提供的蛋白A变体的变体,这些变体也应包括在本发明的范围内。类似地,在本文提供的具体核苷酸序列基础上,本领域技术人员可以通过对少数碱基进行替换(例如同义突变)、删除、添加,并验证或筛选其表达目的产物以及目的产物与IgG的结合能力和/或耐碱能力,从而获得在功能上与本文提供的核苷酸序列基本上相同的其他核苷酸序列,它们也应该包括在本发明的范围内。
在本文提供的蛋白A变体可作为其他蛋白的组成部分。在一个实施方案中,本文提供了包括一个或更多个蛋白A变体(例如1、2、3、4、5、6或甚至更多个)的融合蛋白。该融合蛋白中蛋白A变体之间可通过接头分子(例如氨基酸短序列)串联连接。优选地,该接头分子也具有耐碱性,这样的接头分子例如在PCT公开WO03080655中有描述。在另一实施方案中,本文提供了包括一个或更多个蛋白A变体(例如1、2、3、4、5、6或甚至更多个)以及至少一个也具有IgG结合能力的其他结构域的融合蛋白。该其他结构域例如为蛋白A的结构域E、D、A、B和C,或者上文描述的“Z结构域”。另外,还预期本文提供的蛋白A变体可连接有信号肽或方便制备的纯化标签,例如组氨酸标签、Strep II标签等。在本文提供了蛋白A变体的氨基酸和核苷酸序列基础上,本领域技术人员可方便的通过基因工程技术获得这些融合蛋白。
本文提供的蛋白A变体基本上都具有IgG(例如IgG1)结合能力(除#4),甚至部分蛋白A变体(#1、#3和#10)具有比用作对照的Z结构域更高的IgG结合亲和力(参见实施例2)。相应地,在将这些蛋白A变体用于从含IgG的细胞组分(如细胞培养上清)中分离IgG时,可具有更高的动态载量或纯化能力。
同时,本文提供的部分蛋白A变体(#1、#3和#10)具有良好的耐碱性,其中蛋白A变体#10的耐碱性能甚至优于Mabselect SuRe(参见实施例4)。这些蛋白A变体可耐受高至0.5M的NaOH清洗,可满足制药工艺中对设备的反复除菌、除热源等要求。
另外,本文提供的部分经密码子优化的蛋白A变体编码序列(#1、#2、#3和#10)可提高蛋白A变体在宿主细胞(如大肠杆菌BL21)的表达量,例如高于用作对照的Z结构域的编码序列的表达量30%。
如前文所述,本文提供的蛋白A变体(以及包括该蛋白A变体的融合蛋白)可用于IgG纯化。另外,在一些情况下,可用于从某些样品(例如血液)中除去IgG分子,以获得不含IgG分子的样品。还预期本文提供的蛋白A变体(以及包括该蛋白A变体的融合蛋白)可用于免疫检测,例如蛋白A变体可用示踪物(如荧光素、酶、胶体金、铁蛋白)标记后替代第二抗体用于抗原检测。
以下通过具体实施例来进一步描述本发明。
实施例1耐碱蛋白A变体的定向优化方法
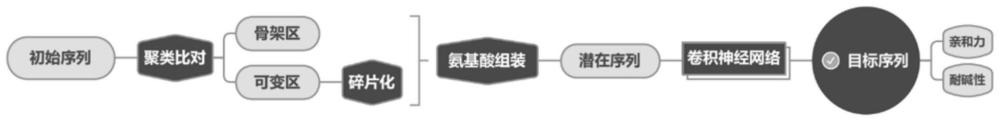
针对提高耐碱性和提高与IgG的结合能力的蛋白功能优化目标,本发明通过生物信息学和卷积神经网络来获得定向优化的蛋白A变体。
该方法的具体技术方案可包括以下步骤(参见图2):
步骤1:将蛋白A的E、D、C、B和A结构域以及Z结构域序列作为用于定向优化的初始序列,并从RCSB-PDB数据库中得到相应序列的晶体结构,其中A结构域的结构通过同源模建方法获得。
步骤2:将初始序列进行交叉比对并对氨基酸残基进行结构叠合聚类分析。
步骤3:根据序列同一性和氨基酸残基结构匹配的结果,将序列分为骨架区和可变区,骨架区作为保守序列,可变区作为碎片化序列,然后将可变区的序列拆分为包含单氨基酸、双氨基酸、三氨基酸的三类短序列。
骨架区序列基础氨基酸包含:
Q/AFY/L/PNL/QRN/FIQSL/DPS/S/L/EA/KLN/QAPK。
可变区序列基础氨基酸包含:
QAVI/QDA/NA/NKQ/FH/ND/KE/DEA/QH/SNA/EQ/IV/HN/LM/TN/EA/EDA/AG/KR/D H/VQ/LAKT/EN/ILV/GAS/KQ/DE/AS。
步骤4:通过随机序列生长和组合的方式,在骨架区基础上组合装配碎片区的短序列,建立一个包含随机序列的潜在序列库,该库中的序列长度为58个氨基酸,并与蛋白A的E、D、C、B和A结构域以及Z结构域序列具有满足一定条件的同一性(50%<同一性<85%)。
步骤5:通过文献调研获取具有耐碱性和IgG结合能力的Protein A序列并计算一级结构分子描述符,再经过归一化处理后作为训练集,对包含输入层、隐藏层和输出层的卷积神经网络进行预训练,隐藏层包含4个卷积层和2个池化层,输出层为密集连接层。
步骤6:对潜在序列库中的序列计算一级结构分子描述符并做归一化处理,使用前步训练好的模型进行预测,根据预测值选出兼具高耐碱性和高IgG结合能力的前10条序列进行进一步实验验证。
本方法获得的目标序列如下:
>T_SEQAA-1(SEQ ID NO:1)
IDNKFNEEQQAAFYEVLHMPNLNAEQRNGFIQSLKDDPSQSTNLLAEAQKLNEAQAPK
>T_SEQAA-2(SEQ ID NO:2)
QDNQFNKEQQNAFYQILHLPNLNAEQRNAFIQSLRHDPSQSLNLLGEAQKLNDSQAPK
>T_SEQAA-3(SEQ ID NO:3)
AQNKFDKEQQNAFYQILHMPNLTADQRNGFIQSLKDDPSQSANVLAEAQKLNDAQAPK
>T_SEQAA-4(SEQ ID NO:4)
AQNKHNKEHQNAFYQILHLPNLNEEQRNGFIQSLKDDPSVSANILGEAKKLNESQAPK
>T_SEQAA-5(SEQ ID NO:5)
QQNKHDEAQQSAFYEVLHMPNLTEEQRNGFIQSLKDDPSQSLELLGEAQKLNDSQAPK
>T_SEQAA-6(SEQ ID NO:6)
AAAQFNEEQQNAFYEILHMPNLTEAQRNAFIQSLKDDPSQSTNVLGEAQKLNDSQAPK
>T_SEQAA-7(SEQ ID NO:7)
QDNKFDEDQQSAFYQILHMPNLTEDQRNGFIQSLKHDPSVSANLLSEAQKLNESQAPK
>T_SEQAA-8(SEQ ID NO:8)
QDANFDKAHQSAFYEVLHLPNLNEEQRNAFIQSLKDDPSQSKNVLAEAQKLNDAQAPK
>T_SEQAA-9(SEQ ID NO:9)
IDNKFNKAQQNAFYEVLNMPNLTAAQRNGFIQSLRDDPSVSTELLGEAKKLNESQAPK
>T_SEQAA-10(SEQ ID NO:10)
QDNQHDEAQQAAFYEILNLPNLNEEQRNGFIQSLRHDPSQSAEILSEAKKLNESQAPK
本方法初始序列如下:
>SEQAA_Parental(Z结构域)(SEQ ID NO:21)
VDNKFNKEQQNAFYEILHLPNLNEEQRNAFIQSLKDDPSQSANLLAEAKKLNDAQA PK
本方法获得的目标序列与初始序列间的同一性矩阵如图3所示。
实施例2候选蛋白A变体的表达和纯化
将上述T_SEQAA-1至T_SEQAA-10共十个氨基酸序列以及Z结构域序列,利用大肠杆菌优势密码子进行优化,得到如下对应DNA编码序列。
T-SEQDNA-1(SEQ ID NO:11)
ATTGATAACAAATTTAACGAAGAACAGCAGGCGGCGTTTTATGAAGTGCTGCAT
ATGCCGAACCTGAACGCGGAACAGCGTAACGGCTTTATTCAGAGCCTGAAAGAT
GATCCGAGCCAGAGCACCAACCTGCTGGCGGAAGCGCAGAAACTGAACGAAGC
GCAGGCGCCGAAA
T_SEQDNA-2(SEQ ID NO:12)
CAGGATAACCAGTTTAACAAAGAACAGCAGAACGCGTTTTATCAGATTCTGCAT
CTGCCGAACCTGAACGCGGAACAGCGTAACGCGTTTATTCAGAGCCTGCGTCAT
GATCCGAGCCAGAGCCTGAACCTGCTGGGCGAAGCGCAGAAACTGAACGATAG
CCAGGCGCCGAAA
T_SEQDNA-3(SEQ ID NO:13)
GCGCAGAACAAATTTGATAAAGAACAGCAGAACGCGTTTTATCAGATTCTGCAT
ATGCCGAACCTGACCGCGGATCAGCGTAACGGCTTTATTCAGAGCCTGAAAGAT
GATCCGAGCCAGAGCGCGAACGTGCTGGCGGAAGCGCAGAAACTGAACGATGC
GCAGGCGCCGAAA
T_SEQDNA-4(SEQ ID NO:14)
GCGCAGAACAAACATAACAAAGAACATCAGAACGCGTTTTATCAGATTCTGCAT
CTGCCGAACCTGAACGAAGAACAGCGTAACGGCTTTATTCAGAGCCTGAAAGAT
GATCCGAGCGTGAGCGCGAACATTCTGGGCGAAGCGAAAAAACTGAACGAAAG
CCAGGCGCCGAAA
T_SEQDNA-5(SEQ ID NO:15)
CAGCAGAACAAACATGATGAAGCGCAGCAGAGCGCGTTTTATGAAGTGCTGCAT
ATGCCGAACCTGACCGAAGAACAGCGTAACGGCTTTATTCAGAGCCTGAAAGAT
GATCCGAGCCAGAGCCTGGAACTGCTGGGCGAAGCGCAGAAACTGAACGATAG
CCAGGCGCCGAAA
T_SEQDNA-6(SEQ ID NO:16)
GCGGCGGCGCAGTTTAACGAAGAACAGCAGAACGCGTTTTATGAAATTCTGCAT
ATGCCGAACCTGACCGAAGCGCAGCGTAACGCGTTTATTCAGAGCCTGAAAGAT
GATCCGAGCCAGAGCACCAACGTGCTGGGCGAAGCGCAGAAACTGAACGATAG
CCAGGCGCCGAAA
T_SEQDNA-7(SEQ ID NO:17)
CAGGATAACAAATTTGATGAAGATCAGCAGAGCGCGTTTTATCAGATTCTGCAT
ATGCCGAACCTGACCGAAGATCAGCGTAACGGCTTTATTCAGAGCCTGAAACAT
GATCCGAGCGTGAGCGCGAACCTGCTGAGCGAAGCGCAGAAACTGAACGAAAG
CCAGGCGCCGAAA
T_SEQDNA-8(SEQ ID NO:18)
CAGGATGCGAACTTTGATAAAGCGCATCAGAGCGCGTTTTATGAAGTGCTGCAT
CTGCCGAACCTGAACGAAGAACAGCGTAACGCGTTTATTCAGAGCCTGAAAGAT
GATCCGAGCCAGAGCAAAAACGTGCTGGCGGAAGCGCAGAAACTGAACGATGC
GCAGGCGCCGAAA
T_SEQDNA-9(SEQ ID NO:19)
ATTGATAACAAATTTAACAAAGCGCAGCAGAACGCGTTTTATGAAGTGCTGAAC
ATGCCGAACCTGACCGCGGCGCAGCGTAACGGCTTTATTCAGAGCCTGCGTGAT
GATCCGAGCGTGAGCACCGAACTGCTGGGCGAAGCGAAAAAACTGAACGAAAG
CCAGGCGCCGAAA
T_SEQDNA-10(SEQ ID NO:20)
CAGGATAACCAGCATGATGAAGCGCAGCAGGCGGCGTTTTATGAAATTCTGAAC
CTGCCGAACCTGAACGAAGAACAGCGTAACGGCTTTATTCAGAGCCTGCGTCAT
GATCCGAGCCAGAGCGCGGAAATTCTGAGCGAAGCGAAAAAACTGAACGAAAG
CCAGGCGCCGAAA
SEQDNA-Parental(Z结构域)(SEQ ID NO:22)
GTGGATAACAAATTTAACAAAGAACAGCAGAACGCGTTTTATGAAATTCTGCAT
CTGCCGAACCTGAACGAAGAACAGCGTAACGCGTTTATTCAGAGCCTGAAAGAT
GATCCGAGCCAGAGCGCGAACCTGCTGGCGGAAGCGAAAAAACTGAACGATGC
GCAGGCGCCGAAA
利用5'和3'端的BamHI和EcoRI将上述10条DNA序列以及Z结构域的DNA序列分别插入至pET28a(+)载体中(图4),随后利用感受态细菌热激转化入BL21表达菌中,接种至LB平板过夜培养后,挑单菌落于3mL LB培养基中37℃摇床培养18小时。3mL菌液进一步扩增培养至20mL(OD600<0.4)。取出10mL菌液加入甘油至30%(v/v)冻存保种。剩余10mL加入1L LB培养基中(1:100)扩大培养至OD600=0.6-0.8(~2小时),加入IPTG至终浓度0.5mM,降温至16℃过夜诱导蛋白表达。过夜培养24小时后,离心收集菌体。细胞沉淀重悬于裂解缓冲液(50mM NaHPO4,pH=8,0.3NaCl,Dnase I,Protease inhibitor)。超声破菌后,加入MgCl
表1蛋白A变体和Z结构域对照与IgG1结合的亲和力检测结果
ND:未检出N/A:不适用
从表1可看出,除#4外,其他9种蛋白A变体都对IgG1抗体具有可检测的结合亲和力,其中#1、#3、#10与IgG1抗体结合亲和力甚至优于对照Z结构域。
实施例3候选蛋白A变体和微球的偶联
本实施例选用粒径均一度高、机械强度好的琼脂糖微球为基架,对亲和力与Z结构域相近的候选蛋白A变体(1#、3#和10#)进行偶联。
简要偶联步骤如下。
步骤1:量取5mL环氧活化好的微球,使用10mL 0.1M PB,pH 8.6缓冲液清洗2遍;
步骤2:每毫升填料加入3mg候选蛋白A变体;
步骤3:30℃±1℃反应1小时;
步骤4:每次使用20ml水洗清洗凝胶,共清洗2次,收集清洗液测蛋白浓度(OD280),计算偶联上的蛋白A变体含量;
步骤5:使用20ml水洗清洗凝胶,再清洗5次;
步骤6:用5ml 20%乙醇洗2次;
步骤7:20%乙醇3mL,2-8℃保存。
实施例4亲和层析介质耐碱性和动态载量的测定
用上述制备的蛋白A亲和层析介质装填Hitrip 1mL柱,用平衡液冲洗。以Mabselect SuRe(Cytiva)作为参考品,测试待测突变体偶联的亲和介质的耐碱性和动态结合载量。AKTA purifier蛋白纯化仪在使用前,用纯化水冲洗平衡,平衡后基线归零。20mMPBS,150mM NaCl,pH7.0缓冲液和50mM Gly,pH3.0缓冲液分别作为平衡相及洗脱相,系统泵用平衡相平衡。将小柱连接至AKTA purifier,注意连接过程中防止气泡进入,连接时泵以一定流速运行,接头部分用流动相充分润湿,小柱上端也用流动相充满,然后连接。小柱依次用50mM Gly,pH3.0和20mM PBS,150mM NaCl,pH7.0各10CV冲洗至基线平衡。开始以0.25mL/min进抗体细胞培养液上清(~1mg/ml),约50ml每个层析小柱,并用洗脱相洗脱抗体,通过分析抗体洗脱总量计算层析柱动态结合载量。随后层析柱分别用0.1M NaOH和0.5MNaOH进行CIP。重复上述步骤180个循环。检测和分析动态载量变化情况。结果如图6所示。三种变体(#1、#3和#10)都显示出超强的耐碱能力,能够耐受浓度高至0.5M的NaOH的多次CIP处理。它们在循环使用100次时基本上都还能够达到初始载量的80%以上,并且在循环使用180次时也没有表现出动态载量的迅速下降。尤其是,无论是0.1M还是0.5M NaOH处理,#10变体在整个测试过程中都表现出比Mabselect SuRe更高的动态载量。
Mabselect SuRe对照目前在对IgG亲和力和耐碱性方面都表现优异,而本发明提供的新蛋白A变体在亲和力和耐碱性方面与其相仿,或者更优,可以为使用者提供更多选择。考虑到蛋白A变体#1、#2、#3和#10在表达量方面与野生型Z结构域相比有很大优势(高30%以上),通过本文提供的编码DNA序列来表达这些蛋白A变体还具有一定成本优势。
- C肽化学发光免疫检测试剂盒及其制备方法和检测方法
- Ⅳ型胶原化学发光免疫检测试剂盒及其制备方法和检测方法
- 一检多项一步免疫检测方法与半定量一步免疫检测方法
- 一种微流控免疫检测芯片及免疫检测方法
