预测结直肠癌预后风险的模型构建方法
文献发布时间:2024-04-18 19:58:26
技术领域
本发明属于数据分析技术领域,具体涉及一种预测结直肠癌预后风险的模型构建方法。
背景技术
结直肠癌(Colorectal cancer,CRC)是指发生于结肠或者是直肠的癌变,可发生于结直肠的任何部位,但尤以直肠、乙状结肠最为常见。CRC给医疗系统造成了沉重负担,严重威胁着人们的身体健康。目前临床上治疗结CRC的方法包括手术、放化疗、靶向治疗和免疫治疗,提高了CRC癌患者5年生存率,但是异质性依然影响着CRC的诊疗效果。CRC预后不仅可以帮助临床医师制定合理的治疗方案,实现最优治疗效果,还可以帮助患者了解病情,更好配合医师诊疗。因此,构建CRC预后模型在临床上具有重要意义。
发明内容
本发明的目的是提供一种预测结直肠癌预后风险的模型构建方法。
为了解决上述技术问题,本发明公开了一种预测结直肠癌预后风险的模型构建方法,包括如下步骤:
a.数据下载与处理:从TCGA数据库下载包含临床和RNAseq数据的结直肠癌数据集CRC_TCGA,提取TPM格式的数据,用log2方法处理数据;从GEO数据库下载结直肠癌数据集GSE39582和GSE17536,经过id转换和标准化处理;
b.筛选训练集中预后相关基因:采用训练集GSE39582作为处理对象,使用R软件包“survival”,整合生存时间、生存状态和基因表达数据,利用cox方法评估每个基因的预后显著性;
c.利用训练集构建预后标签PSCRC及其预后评估:
c1.Lasso-cox分析
预后lasso变量轨迹的建立:使用R软件包的glmnet对清洗过后的GSE39582数据进行分析得到变量系数值、λ对数值、L1正则化值,并对数据进行可视化;预后lasso系数筛选:使用glmnet包对清洗过后的GSE39582数据进行分析得到变量λ值、最大似然数或C指数并对数据进行可视化;
c2.PSCRC预后效果评估
根据PSCRC风险分数计算公式计算预后标签风险评分,并据此将GSE39582数据集中患者分为两组,使用R包“survival”的“survfit”函数分析两组患者预后差异,并使用logrank检验方法评估差异显著性;随后,利用R包“pROC”的“ROC”函数在1年、3年和5年三个时间点进行ROC分析,并使用“ci”函数评估曲线下面积和置信区间,获得最终AUC结果;最后,分析PSCRC风险评分与患者随访时间、结局以及每个基因表达变化之间的关系;
d.外部数据集验证PSCRC
d1.根据PSCRC风险分数计算公式计算两个外部数据集GSE17536和CRC_TCGA中PSCRC的风险评分,验证PSCRC的预后效果;
d2.Kaplan-Meier生存曲线绘制:首先根据50%的百分位数将患者分成两组,进一步使用R软件包“surviva1”的“survfit”函数分析两组的预后差异,利用logrank test方法评估了不同组样本之间的预后差异显著性;
d3.ROC分析:利用R软件包“pROC”进行了ROC分析以获得AUC;
d4.风险热图绘制:利用R包“ggplot2”分析不同风险分数与患者的随访时间、事件及各个基因的表达变化的关系;
e.多因素预后模型绘制与预后效果评估;
e1.绘制预后模型和校准曲线:使用R软件包“survival”包进行比例风险假设检验,并进行Cox回归分析,使用“rms”包构建nomogram相关模型,Calibration分析,并进行可视化;
e2.决策曲线分析评估预后效果:通过“survival”包拟合预后模型,使用“stdca.R”文件进行决策曲线分析。
优选地,c2中,PSCRC风险分数计算公式为:
PSCRC风险分数=(0.34)×(ZEB1-AS1)+(0.08)×(PTPN14)+(-0.11)×(MYB)+(0.05)×(LINC00973)+(0.03)×(GDI1)+(0.04)×(SLC2A3)+(0.01)×(SIX4)+(-0.08)×(ACAT2)+(0.04)×(KRT6A)+(-0.18)×(ZNF552)+(0.06)×(SEMA4C)+(0.29)×(KIF7)+(0.09)×(GABRG2)+(-0.09)×(TNFRSF14)+(0.09)×(LINC00638)+(-0.14)×(OIT3)+(0.25)×(HCN4)+(0.73)×(OFCC1)。
本发明的预测结直肠癌预后风险的模型构建方法,在传统的结直肠癌临床指标的基础上,整合新发现的标签PSCRC,通过构建结直肠癌预后标签PSCRC、PSCRC风险分数计算公式和多因素预后模型,提高了预后效果,可以及早发现疾病的严重程度,从而尽早采取相应的治疗措施,帮助临床医师制定合理的治疗方案,实现最优治疗效果,对提高患者的生存率和生活质量有着重要的指导和参考意义。
附图说明
图1示出了单因素生存分析筛选的部分预后相关基因。
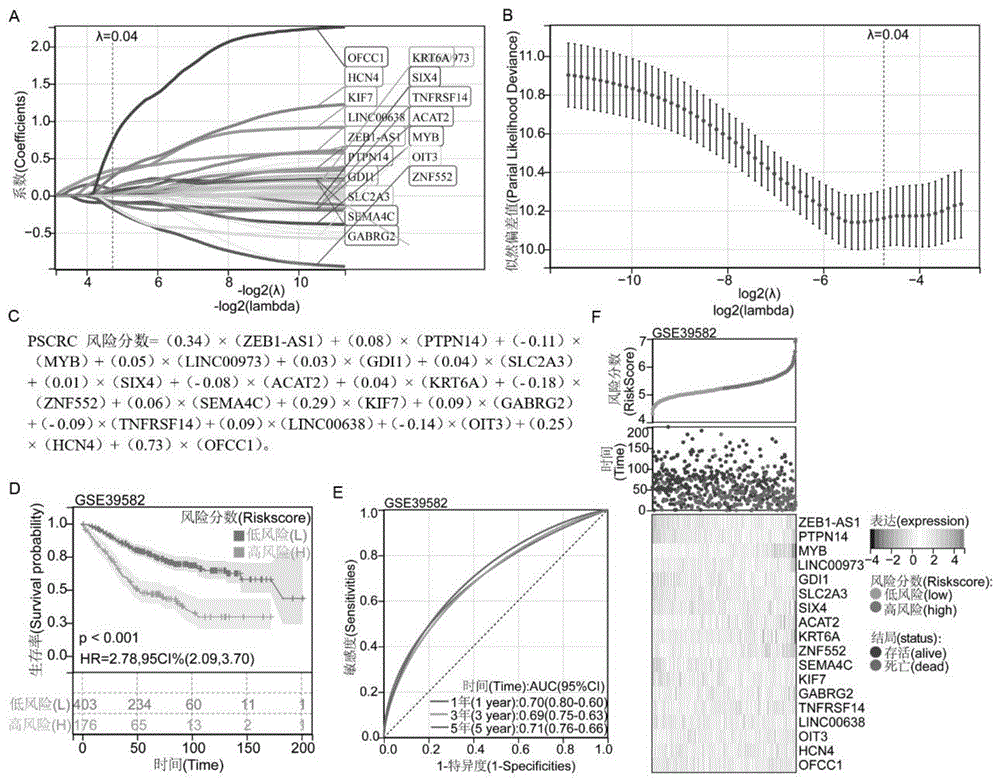
图2示出了训练集GSE39582中拟合预后基因形成的结直肠癌预后标签PSCRC,其中A为LASSO回归系数路径图;B为LASSO回归交叉验证图;C为PSCRC风险分数计算公式;D为生存曲线;E为时间依赖ROC曲线;F为预后风险因子热图。
图3示出了2个外部数据集验证PSCRC预后效果,其中,A-C为GSE17536队列中基于PSCRC风险分数绘制的生存曲线、时间依赖、ROC曲线和预后风险因子热图;D-F为CRC_TCGA队列中基于PSCRC风险分数绘制的生存曲线、时间依赖ROC曲线和预后风险因子热图。
图4示出了CRC多因素预后模型与预后评估,其中,A为预后模型;B为预后校准曲线;C为预后DCA(decision curve analysis,DCA)。
具体实施方式
下面通过实施例对本发明做进一步的详细说明,以令本领域技术人员参照说明书文字能够据以实施。
应当理解,本文所使用的诸如“具有”,“包含”以及“包括”术语并不排除一个或多个其它元件或其组合的存在或添加。
通过结合附图对本发明示例性实施方式进行更详细的描述,本发明的上述以及其它目的、特征和优势将变得更加明显。
实施例1
本实施例用于说明构建结直肠癌预后模型所需数据类型、下载与处理。
1、数据下载与处理:
表1.结直肠癌表达谱和临床随访数据队列
从TCGA数据库(https://portal.gdc.cancer.gov)下载CRC_TCGA临床和RNAseq数据,提取TPM格式的数据,用log2(表达值+1)方法处理数据。从GEO(https://www.ncbi.nlm.nih.gov/gds)数据库下载结直肠癌数据集GSE39582和GSE17536,经过id转换和标准化处理。
实施例2
本实施例用于说明筛选训练集中预后相关基因,用于构建预后标签PSCRC。
如图1,示出了GSE39582队列中部分预后相关基因。
1、训练集GSE39582中预后基因筛选:
使用R(4.2.1版本)软件包“survival”[3.3.1],整合生存时间、生存状态和基因表达数据,利用cox方法评估每个基因的预后显著性。
实施例3
本实施例用于说明利用训练集构建预后标签PSCRC及其预后评估。
如图2所示,展示了LASSO-cox回归分析构建预后标签PSCRC,其中,(A)LASSO回归系数路径图;(B)LASSO回归交叉验证图;(C)PSCRC风险分数计算公式;(D)生存曲线;(E)时间依赖ROC曲线;(F)预后风险因子热图。
1、Lasso-cox分析
预后lasso(Construction of Prognostic Signature With least absoluteshrinkage and selection operator)变量轨迹:使用R(4.2.1版本)R包:glmnet[4.1.4]对清洗过后的GSE39582数据进行分析得到变量系数值、λ对数值、L1正则化值,并对数据进行可视化。预后lasso系数筛选:使用glmnet包对清洗过后的数据进行分析得到变量λ值、最大似然数或C指数并对数据进行可视化。
2、PSCRC预后效果评估
根据预后标签(Prognostic signature of colorectal cancer,PSCRC)风险评分百分位数(50%)将患者分为两组,使用R包“survival”的“survfit”函数分析两组患者预后差异,并使用logrank检验方法评估差异显著性。随后,利用R包“pROC”的“ROC”函数在1年、3年和5年三个时间点进行ROC(Receiver operating characteristic curve,ROC)分析,并使用“ci”函数评估曲线下面积(Area Under The Curve,AUC)和置信区间,获得最终AUC结果。最后,分析PSCRC风险评分与患者随访时间、结局以及每个基因表达变化之间的关系。
实施例4
本实施例用于说明利用2个外部数据集验证预后标签PSCRC。
如图3,示出了外部数据集验证预后标签PSCRC。其中,(A-C)GSE17536队列中基于PSCRC风险分数绘制的生存曲线、时间依赖、ROC曲线和预后风险因子热图;(D-F)CRC_TCGA队列中基于PSCRC风险分数绘制的生存曲线、时间依赖ROC曲线和预后风险因子热图。
1、根据风险分数公式计算两个外部队列GSE17536和CRC_TCGA中PSCRC的风险评分,验证PSCRC的预后效果。
2、Kaplan-Meier生存曲线绘制
首先根据百分位数(50%)将患者分成两组,进一步使用R软件包“survival”的“survfit”函数分析两组的预后差异,利用logrank test方法评估了不同组样本之间的预后差异显著性。
3、ROC分析
利用R软件包“pROC”(version 1.17.0.1)进行了ROC分析以获得AUC。
4、风险热图绘制
利用R包“ggplot2”(3.3.3版本)分析不同风险分数与患者的随访时间、事件及各个基因的表达变化的关系。
实施例5
本实施例用于说明多因素预后模型的绘制与预后效果评估。
如图4所示,示出了一种CRC预后模型与预后评估。其中,(A)预后模型;(B)预后校准曲线;(C)预后DCA(decision curve analysis,DCA)。
本实施例用于说明预后模型绘制与预后效果评估;
1、绘制预后模型和校准曲线
使用R(4.2.1版本)包“survival”[3.3.1]包进行比例风险假设检验,并进行Cox回归分析,使用“rms”包构建nomogram相关模型,Calibration分析,并进行可视化。
2、决策曲线分析(DCA)评估预后效果
通过“survival”包拟合预后模型,使用“stdca.R”文件进行决策曲线分析(decision curve analysis,DCA)。
- 一种用于预测结直肠癌预后风险的试剂盒及其预测装置和预测模型的训练方法
- 数据收集装置
