一种检测卵巢癌的引物组合、试剂盒及文库构建方法
文献发布时间:2023-06-19 19:30:30
技术领域
本发明涉及基因检测领域,具体涉及一种检测卵巢癌的引物组合、试剂盒及文库构建方法。
背景技术
卵巢恶性肿瘤是妇科常见的恶性肿瘤。卵巢组织成分复杂,是全身各脏器原发肿瘤类型最多的器官,不同类型卵巢肿瘤的组织结构和生物学行为及好发年龄都存在较大差异,组织类型繁多。卵巢上皮癌是其中最多见的组织病理分型,占到卵巢恶性肿瘤的85%-90%。因其早期诊断困难,治疗效果不佳,其病死率居妇科恶性肿瘤首位,甚至超过宫颈癌和子宫内膜癌的总和,严重威胁妇女的健康。卵巢癌是我国女性病死率最高的妇科肿瘤,据中国癌症年报统计,2015年约有5.21万新患病患者,同年约有2.25万人死于卵巢癌。
在过去的几十年里,因卵巢癌造成的死亡率不断攀升,卵巢癌患者通常需要手术切除附件,甚至是子宫和输卵管,对于年轻患者来说,这意味着她们失去了生育能力,同时,失去雌激素也会加速衰老。卵巢癌临床期别不同,预后明显不同。早期发现卵巢癌是高度可治愈的。随着我国人口老龄化日益进展,卵巢癌的发病率逐年提升,并且发病年龄有年轻化的趋势。卵巢癌发病早期几乎没有特殊症状,很少被发现,同时早期卵巢癌可能会发生腹腔内转移。多数卵巢癌患者直到晚期才出现临床症状,因而卵巢癌确诊时75%的患者已属晚期,仅有25%的患者在Ⅰ期被发现。晚期卵巢癌(Ⅲ~Ⅳ期)患者5年生存率仅在20%~40%,但早期卵巢癌(Ⅰ~Ⅱ期)患者5年生存率却在70%以上。因此,高效、精确、敏感的早期筛查手段是治疗卵巢癌的最重要一步,有效的卵巢癌早期筛查能帮助实现对卵巢癌的早期诊断,从而大幅度地提高卵巢癌患者的生存率。因此对于卵巢癌更需要及时预防、及早发现。
目前,针对高危人群每年通过检查盆腔、经阴道超声,以及CA125生物标记检测来筛查卵巢癌。然而临床实践表明,以上方法存在极大的局限性。B超诊断卵巢癌的准确性较低,难以发现微小肿瘤,且易受检查操作医生的主观经验影响。而血清癌胚抗原CA125检查特异性及敏感性较低,通常在早期卵巢癌中无法检出,除卵巢恶性肿瘤外,许多其他疾病,如子宫内膜异位症、早期妊娠、炎症、子宫肌瘤、消化道恶性肿瘤、慢性肝炎、肝硬化等血清CA125亦有高表达,并易受月经周期和妊娠的影响,且CA125在20%的卵巢癌患者血清中很少或几乎没有。联合应用CA125与超声检测,也仅能提高20%的阳性检测率。
目前科学界公认所有癌症都被认为是由体细胞突变(机体中除生殖细胞以外的任何细胞的突变)造成的,虽然迄今为止科学家们对所涉及的突变过程知之甚少。癌症从本质上来说是一种基因病,所有的癌症均来源于基因突变,但是并非所有的基因突变都来源于父母的遗传,病毒感染、化学物质、射线等多种环境因素都可能使基因突变,从而导致癌症的发生。研究表明,卵巢癌存在多种基因突变,在许多不同的基因中发现了驱动突变,包括ERBB2、ERBB3、FGFR2、HRA、KRAS、BRAF、PIK3CA、PIK3R1、ARHGAP35、RRAS2、NF1、PPP2R1A、PTEN、ZFHX3、FOXA2、KMT2D、ARID5B和FBXW7等,已经有多个研究报道基因突检测变可能用于卵巢癌的筛查和诊断,此外卵巢癌的单一的病理分型已不能满足临床需要,需从基因水平明确癌组织的突变状况,为靶向药物的临床使用和疗效预测提供分子基础。鉴于此,临床上对于卵巢癌基因突变的精准检测需求不断增多,有必要开发卵巢癌相关的多基因突变检测方法。
目前,检测基因突变的常见方法有荧光定量PCR法、基因芯片法、Sanger法、高通量测序法等。荧光定量PCR法、基因芯片法只能针对一个或数个已知突变的位点检测,Sanger测序法测序每次只能对一个样品的某一段区域进行测序,检测的共同缺点是无法用于检测未知突变位点,检测效率低,结果不易判读、重复性差、假阳性和假阴性多等。
发明内容
根据第一方面,一种实施例中提供一种引物组合,所述引物组合用于靶向扩增如下至少一种基因中的位点:PTEN、TP53、PIK3CA、PIK3R1、KRAS、CTNNB1、FGFR2、RNF43、POLE、PPP2R1A、FBXW7、AKT1、APC、BRAF、CDKN2A、EGFR、NRAS、MAPK1。
根据第二方面,一种实施例中提供一种试剂盒,包含第一方面的引物组合。
根据第三方面,一种实施例中提供一种文库构建方法,包括:
接头连接步骤,包括将带分子标签的接头连接至核酸样本,获得连接有接头的样本;
巢式PCR第一轮扩增步骤,包括使用外侧引物对所述连接有接头的样本进行扩增,获得第一轮扩增产物;
巢式PCR第二轮扩增步骤,包括使用内侧引物对所述连接有接头的样本进行扩增,获得第二轮扩增产物。
依据上述实施例的一种检测卵巢癌的引物组合、试剂盒及文库构建方法,本发明能够同时对18个卵巢癌致癌基因的多个区域中的基因突变位点进行检测,直接反映相关基因的具体突变位点。
在一实施例中,本发明采用的分子标签加巢式多重PCR能够有效消除PCR过程中带来的假阳性,提高检测低频突变样本的检测限,可以检测到0.1%的低频率变异。能够高效灵敏地检测出卵巢癌致癌基因的突变情况,同时降低了成本、简化了操作步骤。可用于卵巢癌筛查、早期诊断、辅助诊断、癌症愈后监测和靶向用药筛选等。
附图说明
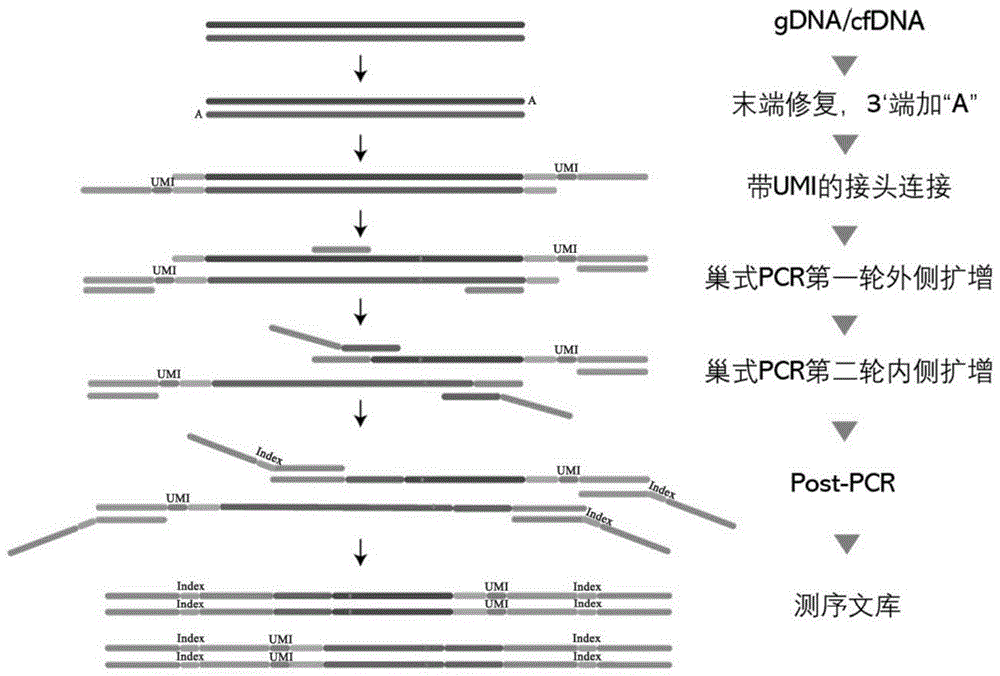
图1为一种实施例中的文库构建流程图。
具体实施方式
下面通过具体实施方式结合附图对本发明作进一步详细说明。在以下的实施方式中,很多细节描述是为了使得本申请能被更好的理解。然而,本领域技术人员可以毫不费力的认识到,其中部分特征在不同情况下是可以省略的,或者可以由其他材料、方法所替代。在某些情况下,本申请相关的一些操作并没有在说明书中显示或者描述,这是为了避免本申请的核心部分被过多的描述所淹没,而对于本领域技术人员而言,详细描述这些相关操作并不是必要的,他们根据说明书中的描述以及本领域的一般技术知识即可完整了解相关操作。
另外,说明书中所描述的特点、操作或者特征可以以任意适当的方式结合形成各种实施方式。同时,方法描述中的各步骤或者动作也可以按照本领域技术人员所能显而易见的方式进行顺序调换或调整。因此,说明书和附图中的各种顺序只是为了清楚描述某一个实施例,并不意味着是必须的顺序,除非另有说明其中某个顺序是必须遵循的。
本文中为部件所编序号本身,例如“第一”、“第二”等,仅用于区分所描述的对象,不具有任何顺序或技术含义。
高通量测序技术NGS是一项国际先进的新型测序技术,能一次并行对几十万到几百万条DNA分子进行序列测定,大大提高了测序的通量。同时,随着生物信息学的快速发展,通过多基因、多位点的集成式检测分析,可一次性检测分析与肿瘤密切相关的基因位点,检出患者的基因突变信息。
主流高通量测序技术主要有两种:一种是大而全的全基因组/全转录组建库测序,另一种是小而精的靶向建库测序。全基因组和全外显子测序方法虽能在通量上满足临床需要,但由于价格昂贵,不能在临床应用中普及。因此,靶向建库测序凭借成本低、数据利用率高、组合灵活等优势,在临床的疾病辅助诊断、治疗以及肿瘤基因检测中发挥出积极的作用。
靶向建库测序技术中的文库构建方法又分为:杂交捕获法建库和多重PCR法建库。其中靶向多重PCR建库无需对样本DNA进行打断,仅通过加入多种引物,同时扩增出目标序列,减少了建库流程,因此大大缩短了建库时间。多重PCR是在一个反应体系中加入多对引物,同时扩增同一份DNA样品不同基因片段的PCR技术。通过分析不同基因片段的序列,判断基因中是否存在缺失、插入或点突变。
根据第一方面,在一实施例中,提供一种引物组合,所述引物组合用于靶向扩增如下至少一种基因中的位点:PTEN、TP53、PIK3CA、PIK3R1、KRAS、CTNNB1、FGFR2、RNF43、POLE、PPP2R1A、FBXW7、AKT1、APC、BRAF、CDKN2A、EGFR、NRAS、MAPK1。
在一实施例中,所述引物组合包含外侧引物、内侧引物。
在一实施例中,所述外侧引物包含SEQ ID No.1~300所示的核苷酸序列中,序列编号为2n的序列中的至少一种,n为≥0的整数。
在一实施例中,所述内侧引物包含SEQ ID No.1~300所示的核苷酸序列中,序列编号为2n+1的序列中的至少一种,n为≥0的整数。
在一实施例中,所述引物组合用于检测卵巢癌。
根据第二方面,在一实施例中,提供一种试剂盒,包含第一方面所述引物组合。
在一实施例中,还包含带分子标签的接头、文库前处理试剂、靶向扩增试剂、文库扩增试剂、I5标签、I7标签、纯化试剂中的至少一种。
在一实施例中,所述文库前处理试剂包含末端加“A”试剂、接头连接试剂中的至少一种。
在一实施例中,所述纯化试剂包含磁珠,包括但不限于IGT
根据第三方面,在一实施例中,提供一种文库构建方法,包括:
接头连接步骤,包括将带分子标签的接头连接至核酸样本,获得连接有接头的样本。
巢式PCR第一轮扩增步骤,包括使用外侧引物对所述连接有接头的样本进行扩增,获得第一轮扩增产物。
巢式PCR第二轮扩增步骤,包括使用内侧引物对所述连接有接头的样本进行扩增,获得第二轮扩增产物。
在一实施例中,所述外侧引物包含SEQ ID No.1~300所示的核苷酸序列中,序列编号为2n的序列中的至少一种,n为≥0的整数。
在一实施例中,所述内侧引物包含SEQ ID No.1~300所示的核苷酸序列中,序列编号为2n+1的序列中的至少一种,n为≥0的整数。
在一实施例中,所述巢式PCR第一轮扩增步骤中,反应体系中含有与所述带分子标签的接头中的至少部分序列特异性反向互补配对的引物。
在一实施例中,所述巢式PCR第一轮扩增步骤中,反应体系中还含有靶向扩增试剂。
在一实施例中,所述巢式PCR第二轮扩增步骤中,反应体系中含有与所述带分子标签的接头中的至少部分序列特异性反向互补配对的引物。
在一实施例中,所述巢式PCR第一轮扩增步骤中,反应体系中还含有靶向扩增试剂。
在一实施例中,还包括纯化步骤,包括对所述第二轮扩增产物进行磁珠纯化,获得纯化产物。
在一实施例中,还包括文库扩增步骤,包括对所述纯化产物进行扩增,获得文库扩增产物。
在一实施例中,所述文库扩增步骤中,反应体系中含有文库扩增试剂、I5标签、I7标签中的至少一种。
在一实施例中,还包括再次纯化步骤,包括对所述文库扩增产物进行磁珠纯化,获得可用于上机测序的文库。
在一实施例中,接头连接步骤中,所述核酸样本为经过末端修复、加“A”反应后的样本。
根据第四方面,在一实施例中,提供第三方面所述的文库构建方法制备得到的文库。
在一实施例中,本发明在多重PCR过程中加入分子标签UMI(Unique molecularidentifer)可以帮助纠正扩增、测序过程中产生的错误,对于UMI标记的reads,可以在数据分析过程中,将具有相同UMI的测序reads成为一个群组,通过同一群组的数据矫正可以得到最终的单链共识序列(SSCS,根据UMI序列和比对到基因组上位置去除重复序列,并形成单链共识序列),得到真实的突变结果,降低背景噪音。
在一实施例中,本发明提供一种基于靶向高通量测序技术检测卵巢癌多基因突变的多重PCR特异性引物、试剂盒和检测方法,能够同时检测多个基因、多个位点突变,且有效提高检测效率、准确率,同时降低成本、简化操作步骤等。
在一实施例中,本发明提供一种基于分子标签加巢式多重PCR的靶向高通量测序检测卵巢癌多基因突变的PCR引物对、试剂盒和方法。采用的分子标签加巢式多重PCR技术能够有效消除PCR过程中带来的假阳性,提高了检测灵敏度、准确率。
在一实施例中,本发明提供一种基于分子标签加巢式多重PCR的高通量靶向测序检测卵巢癌多基因的引物对,所述2轮巢式多重PCR引物的核苷酸序列如SEQ ID No.1至SEQID No.300所示,扩增的目的片段在染色体上的区域起始位置清单如表1所示,巢式PCR第一轮扩增使用的是外侧引物,巢式PCR第二轮扩增使用的是内侧引物:所述的致癌基因为PTEN、TP53、PIK3CA、PIK3R1、KRAS、CTNNB1、FGFR2、RNF43、POLE、PPP2R1A、FBXW7、AKT1、APC、BRAF、CDKN2A、EGFR、NRAS、MAPK1。所述突变可以是一个或多个碱基的置换、插入和/或缺失。
外侧引物具体为SEQ ID No.1~300所示的核苷酸序列中,序列编号为2n的序列,n为≥0的整数。例如SEQ ID No.2、SEQ ID No.4、SEQ ID No.6、SEQ ID No.8所示的核苷酸序列,再例如SEQ ID No.152、SEQ ID No.154、SEQ ID No.156、SEQ ID No.158所示的核苷酸序列等等。
外侧引物中的上游引物为SEQ ID No.1~150所示的核苷酸序列中,序列编号为2n的序列,n为≥1的整数,例如SEQ ID No.2、SEQ ID No.4、SEQ ID No.6、SEQ ID No.8所示的核苷酸序列等等。
外侧引物中的下游引物为SEQ ID No.151~300所示的核苷酸序列中,序列编号为2n的序列,n为≥1的整数,例如SEQ ID No.152、SEQ ID No.154、SEQ ID No.156SEQ IDNo.158所示的核苷酸序列等等。
内侧引物具体为SEQ ID No.1~300所示的核苷酸序列中,序列编号为2n+1的序列,n为≥0的整数。例如SEQ ID No.1、SEQ ID No.3、SEQ ID No.5、SEQ ID No.7所示的核苷酸序列,再例如SEQ ID No.151、SEQ ID No.153、SEQ ID No.155、SEQ ID No.157所示的核苷酸序列等等。
内侧引物中的上游引物为SEQ ID No.1~150所示的核苷酸序列中,序列编号为2n+1的序列,n为≥0的整数,例如SEQ ID No.1、SEQ ID No.3、SEQ ID No.5、SEQ ID No.7所示的核苷酸序列等等。
内侧引物中的下游引物为SEQ ID No.151~300所示的核苷酸序列中,序列编号为2n+1的序列,n为≥0的整数,例如SEQ ID No.151、SEQ ID No.153、SEQ ID No.155、SEQ IDNo.157所示的核苷酸序列等等。
在一实施例中,本发明的SEQ ID No.1至SEQ ID No.300所示的特异性引物是针对18个卵巢癌致癌基因特定设计的序列,具体可以扩增PTEN、TP53、PIK3CA、PIK3R1、KRAS、CTNNB1、FGFR2、RNF43、POLE、PPP2R1A、FBXW7、AKT1、APC、BRAF、CDKN2A、EGFR、NRAS、MAPK1等18个致癌基因的突变位点区域,扩增产物经高通量测序获得其序列信息,从而检测出样品中这些基因的突变状况。由于检测的都是基因目的区域片段,为了保证每个区域检测的特异性并增加稳定性,在多重PCR扩增建库方法原理基础上,采用2轮巢式多重扩增。本发明所用SEQ ID No.1至SEQ ID No.300所示的引物,可使用2轮巢式多重PCR扩增技术,第一轮多重PCR反应在1管中同时有效的扩增目标序列,经过PCR产物纯化,第二轮多重PCR反应在1管中进一步同时有效的扩增目标序列,经过PCR产物纯化,经过post-PCR反应及再次纯化,所得PCR产物经用于制备测序文库和上机进行高通量测序,所构建的测序文库经Qubit 3.0检测合格后适用于华大和Illumina等等多种高通量测序平台。
引物具体如下:
表1
/>
/>
/>
/>
/>
在一实施例中,基于分子标签加巢式多重PCR的高通量靶向测序检测卵巢癌多基因的引物对序列和扩增的目的片段在染色体上的区域也在本发明的保护范围之内。
在一实施例中,本发明提供了一种基于分子标签加巢式多重PCR的高通量靶向测序检测卵巢癌多基因的试剂盒,包括2轮巢式所用的多重PCR引物(巢式PCR第一轮扩增使用的是外侧引物,巢式PCR第二轮扩增使用的是内侧引物)、文库前处理试剂、靶向扩增试剂、文库扩增试剂、I5标签、I7标签、纯化磁珠。
在一实施例中,文库前处理试剂包括末端加“A”试剂、接头连接试剂中的至少一种。
在一实施例中,所述末端加“A”试剂包括末端加“A”缓冲液、末端加“A”酶。
在一实施例中,所述接头连接试剂包括带UMI的接头、DNA连接酶、DNA连接缓冲液。
在一实施例中,所述靶向扩增试剂包括高保真DNA聚合酶、PCR缓冲液、dNTP混合物。
在一实施例中,试剂盒还包含与带UMI的接头的特异性互补的引物AnchorSeq
在一实施例中,所述文库扩增试剂包括高保真DNA聚合酶、PCR缓冲液、dNTP混合物、AnchorSeq
在一实施例中,所述纯化磁珠是用于从靶向扩增产物与文库扩增产物的中纯化目的大小范围DNA。
实施例1
建库流程如图1所示,具体步骤如下:
1.对已知突变的基因组DNA标准品检测4次,每次使用25ng(浓度为2.5ng/μL),基因组DNA打断后使用商品化试剂盒进行末端修复,3'端加“A”。
配制反应体系:
表2
设置PCR仪参数如下:
表3
2.将AnchorSeq
配制反应体系:
表4
设置PCR仪参数如下:
表5
3.使用IGT
4.使用外侧引物对进行巢式PCR第一轮扩增,向反应体系中加入前述外侧混合引物对、靶向扩增试剂、与带UMI的接头的特异性互补的引物AnchorSeq
外侧引物对具体为SEQ ID No.1~300所示的核苷酸序列中,序列编号为2n的序列,n为≥0的整数。
配制如下反应体系:
表6
/>
设置PCR仪参数如下:
表7
5.使用IGT
6.使用内侧引物对进行巢式PCR第二轮扩增,向反应体系中加入前述外侧混合引物对、靶向扩增试剂、与带UMI的接头的特异性互补的引物AnchorSeq
内侧引物对为SEQ ID No.1~300所示的核苷酸序列中,序列编号为2n+1的序列,n为≥0的整数
配制如下反应体系:
表8
设置PCR仪参数如下
表9
7.使用IGT
8.在步骤7中获得的扩增子液体中加入文库扩增缓冲液、I5标签、I7标签和双蒸水,进行PCR反应使扩增子两侧带上测序接头序列。
配制如下反应体系:
表10
设置PCR仪参数如下:
表11
9.使用IGT
10.检测合格后,华大MGISEQ-2000上机测序。
已知突变标准品按照技术流程的检测数据显示,各引物的覆盖率(coveragerate,%)均为100%。
检测结果见表12。
表12
续表12
续表12
续表12
续表12
续表12为表12的右侧数据。
表12中各指标的含义如下:
测序数据量统计表:
(1)Raw reads(M):原始数据总序列数,单位为M;
(2)Raw bases(Mb):原始数据总碱基数,单位为Mb;
(3)UMI reads(M):提取分子标签后总序列数,单位为M;
(4)UMI bases(Mb):提取分子标签后总碱基数,单位为Mb;
(5)Pollution rate(%):过滤比率,1-UMI bases(Mb)/Raw bases(Mb)
(6)Clean reads(M):提取分子标签后数据过滤后剩余总序列数,单位为Mb;
(7)Clean bases(Mb):提取分子标签后数据过滤后剩余总碱基数,单位为Mb;
(8)QC rate(%):clean比率,Clean bases(Mb)/UMI bases(Mb)
文库质量统计表:
(1)Sample:样本名称,一般填写的是公司编号;
(2)Average read length:测序平均读长(经过质控后平均读长会比原始数据读长略短);
(3)Average base quality:测序碱基平均质量值,一般情况下>=30;
(4)Average insert size:平均建库长度,读长为150时,平均建库长度在200以上比较适合;
(5)Align rate:比对率,比对到参考基因组上的read/clean reads
(6)Target size:需要捕获的目标区域大小,以碱基为单位;
(7)Target covered size:实际覆盖到的的目标区域大小;
(8)Coverage rate(%):覆盖率,揭示目标区域是否覆盖良好的指标,Targetcovered size/Target size;
(9)Total effffective bases(Mb):总的有效碱基总数;
(10)Target effffective bases(Mb):目标区域覆盖到的碱基总数;
(11)Bases capture rate(%):碱基捕获率,揭示该样本数据利用率的指标,Target effff ectivebases/Accurate mapped bases;
(12)Target average depth:目标区域的平均深度,Target effffective bases/Target size;
(13)1000X coverage rate(%):深度>=1000X的目标区域比率,1000X覆盖度;
(14)2000X coverage rate(%):深度>=2000X的目标区域比率,2000X覆盖度;
(15)5000X coverage rate(%):深度>=5000X的目标区域比率,5000X覆盖度;
(16)10000X coverage rate(%):深度>=10000X的目标区域比率,10000X覆盖度;
(17)5%X mean depth coverage rate(%):深度>=平均深度5%X的目标区域比率(假设平均深度20000层,就是大于等于1000层的比率);
(18)10%X mean depth coverage rate(%):深度>=平均深度10%X的目标区域比率;
(19)20%X mean depth coverage rate(%):深度>=平均深度20%X的目标区域比率;
(20)30%X mean depth coverage rate(%):深度>=平均深度30%X的目标区域比率;
(21)40%X mean depth coverage rate(%):深度>=平均深度40%X的目标区域比率;
(22)50%X mean depth coverage rate(%):深度>=平均深度50%X的目标区域比率;
(23)Flank 10%X mean depth coverage rate(%):平均深度两侧10%深度的目标区域比率(假设平均深度10000层,就是9000-11000层的比率);
(24)Average tag family size:平均tag家族大小,当一个片段的起始终止位置以及标签一致时,认为是一个家族;
(25)SSCS effffective reads(M):单端校正前有效read数目;
(26)SSCS after reads(M):单端校正后有效read数目;
(27)SSCS effffective rate(%):单端校正比;
(28)SSCS Target average depth:单端校正后目标区域的平均深度;
(29)Again Align rate(%):双端校正后的再次比对率;
(30)DCS Target average depth:双端校正后目标区域的平均深度;
(31)Panel name:目标区域名称。
已知突变位点的数据如下:
表13
实施例2
从湘雅三院采集得到子宫内膜脱落细胞,使用天根血液/细胞/组织因组试剂盒(货号DP304)进行基因组DNA提取,Quibt检测基因组DNA浓度,按照实施例1的操作方法进行样本处理和上机检测。
样本的检测数据见表14。
表14
续表14
/>
续表14
续表14
续表14为表14的右侧数据。
可见,Align rate比对率是指比对到参考基因组上的read/clean reads,超过98%的短序列能够比对上基因组,覆盖率(Coverage rate,%)均为100%,揭示目标区域覆盖良好,证明了多重PCR序列富集的高度特异性。并且目标区域平均测序深度达1万X左右。
实施例3
以至少3个基因举例说明,巢式PCR的内侧/外侧引物所对应的检测基因片段在染色体上的具体位置。
本发明为提高突变检出的准确率,根据扩增片段的起始位点设计了2对巢式PCR引物,如下是其中3个片段的示例。
表15
实施例4
本发明从方法学上进行了优化,卵巢突变覆盖了18基因9330位点,每个扩增的基因片段2轮巢式方案中每轮设计了3对引物对,设计了3种配对方案,多重PCR步骤中进行了F端和R端的匹配,选择覆盖率(coverage rate,%)达到最优(100%)的引物,确定为优选的引物对。以下是关于优化方案的3个以上gene实施例的示例。
表16
/>
/>
在一实施例中,本发明的方法是一种高效、可靠的高通量测序序列富集方法。本发明的方法对卵巢癌18个基因的9330个突变位点具有检测通量高、特异性强、样本来源广泛、不易发生污染且安全性高等众多优点,检测结果具有很好的重复性和准确性。
在一实施例中,本发明提供了一种基于分子标签加巢式多重PCR的高通量靶向测序检测卵巢癌多基因突变的引物对、试剂盒和方法。本发明具体公开了如SEQ ID No.1至SEQ ID No.308所示的多重PCR引物、含有上述引物的试剂盒,及使用上述引物或试剂盒,结合2轮巢式PCR扩增反应,靶向富集卵巢癌致癌基因片段检测基因突变位点的方法,该方法具体包括样本DNA提取、末端加A、单端含UMI分子标签的接头序列连接、2轮巢式PCR扩增富集目的基因片段、文库质控和高通量测序。
在一实施例中,本发明的方法是一种高效、可靠的方法,可实现对突变特点的扩增富集。本发明的多重PCR引物、试剂盒和检测方法,能够同时对18个卵巢癌致癌基因的多个区域中的基因突变位点进行检测,直接反映相关基因的具体突变位点,采用的分子标签加巢式多重PCR能够有效消除PCR过程中带来的假阳性,提高检测低频突变样本的检测限,可以检测到0.1%的低频率变异。能够高效灵敏地检测出卵巢癌致癌基因的突变情况,同时降低了成本、简化了操作步骤。可用于卵巢癌筛查、早期诊断、辅助诊断、癌症愈后监测和靶向用药筛选等。
在一实施例中,本发明提供了一种基于分子标签加巢式多重PCR的高通量靶向测序检测卵巢癌多基因的引物对、试剂盒及方法。
以上应用了具体个例对本发明进行阐述,只是用于帮助理解本发明,并不用以限制本发明。对于本发明所属技术领域的技术人员,依据本发明的思想,还可以做出若干简单推演、变形或替换。
