基于高斯混合模型聚类算法的分层级数据可视化方法
文献发布时间:2023-06-19 11:32:36
技术领域
本发明属于数据可视化领域,具体涉及一种基于高斯混合模型聚类算法的分层级数据可视化方法。
背景技术
在生物信息学中分析、收集遗传等数据后,大多都需进行数据的可视化。随着B/S架构的飞速发展以及多数使用者的认可,使得将数据通过前端页面进行展示的方法也广泛应用于生物信息方面。在生物信息中基因与基因之间进行节点关系可视化中,多用Echarts的关系图以及其他开源的可视化库,引用到前端页面,实现B/S架构为基础的数据可视化的过程。
但是该种数据可视化技术还存在以下问题:
1.信息分析后产生的数据量较为庞大,在利用一些开源可视化库的“力导向图”模块或“关系图”模块的过程中,会因为数据量过大导致网页渲染极其缓慢,最后导致浏览器崩溃,网页无响应。具体的在前端页面中显示“力导向图”时,当导入的数据量大于一个阈值时(对于不同的硬件设备运行,阈值也有所不同;此处说明的是市面上普遍的、适中的硬件设备运行),Html页面需要渲染,由于数据过多导致加载过慢,从而使网页无响应,最后数据可视化失败。
2.市面上可使用的开源可视化工具中的“力导向图”模块或“关系图”模块,展示的效果在一些需要查看节点与节点之间“链接线”的长度时并不是很好,再加上大部分的线条会重叠,类似于“毛球”,想要通过人工精准找到两个节点之间的关系链接线条难度极大,给人们在这方面的可观察性大大降低。另外若针对某一个节点,有很多数据节点链接,由于不同数据节点之间的联系程度(节点距离)不同,再考虑到一些必要的参数的设置,例如节点与节点间斥力参数的设定等,则会在一个正常的显示页面中显示内容不全,如图6(此处为导入500个数据节点显示效果)。
3.当需要数据量达到一定程度,并传给前端进行可视化的时候,大多数工程师会采用“懒加载(懒渲染)”的方法。即每次渲染一部分数据,并监听窗口的滚动,当某个设定的元素展示后,将加载下一部分数据。这种方式的弊端在于,当用户滑动窗口次数过多,由于页面将加载数据而没有数据释放,页面会愈加卡顿,直到某批数据的加入依旧会引起页面崩溃。
4.虚拟滚动技术在大数据量的数据可视化中也有广泛的应用。首先,虚拟滚动技术并不适合“力导向图”或“关系图”此类数据展示方式;其次,虚拟滚动技术是一次性加载全部数据,然后对可见范围内的数据进行渲染。但由于大数据分析等方向所传输的数据量极大,导致当一次性加载全部数据时也会引起页面的一些延时问题。
在生物信息的这个方向中,当利用机器学习等计算机算法得出一些数据后,需进行数据展示,从而进行人为的数据利用与筛选。但到目前为止,市面上还没有一个较好的、可利用的、大数据量的数据可视化方法。
发明内容
本发明的目的就在于为了解决上述问题而提供一种较好的、可利用的、大数据量的一种基于高斯混合模型聚类算法的分层级数据可视化方法。
本发明通过以下技术方案来实现上述目的:
一种基于高斯混合模型聚类算法的分层级数据可视化方法,包括以下步骤:
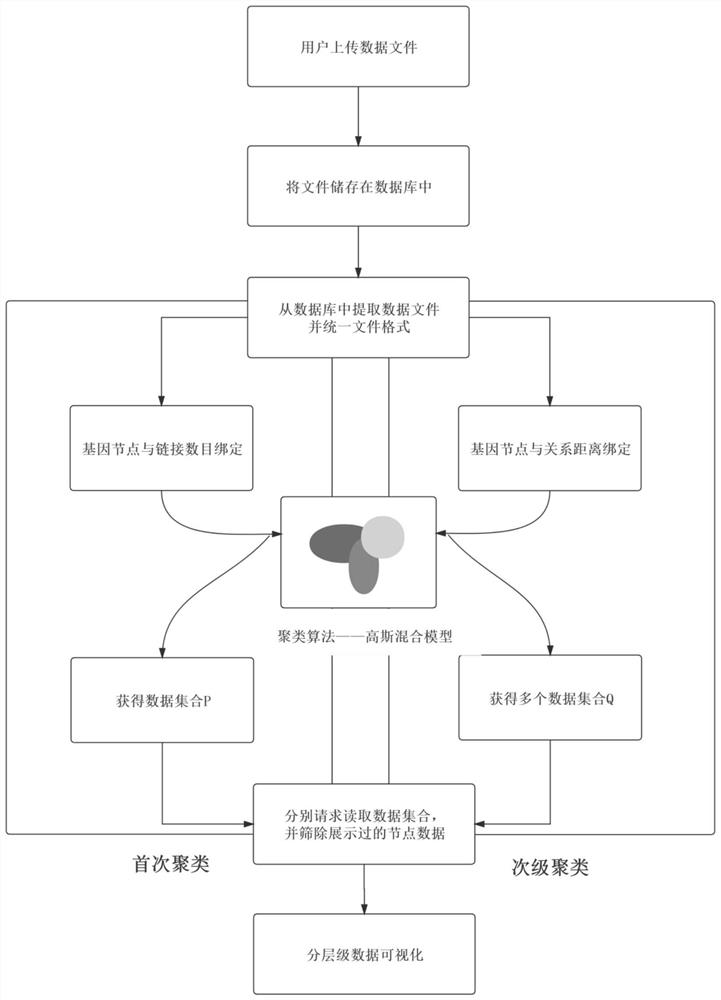
S1、在网页中接收用户上传的数据文件储存至后台数据库,并统一文件格式;
S2、对统一文件格式后的数据进行分析、清洗预处理,得到待聚类数据;
S3、利用高斯混合模型将待聚类数据进行首次聚类和次级聚类,得到待展示数据;
S4、建立前端页面,利用虚拟滚动技术将待展示数据进行分层级可视化展示。
作为本发明的进一步优化方案,步骤S1中所述的数据文件包括source、target、value三类数据,source、target为两类基因数据,value值表示source中基因与target中基因之间的关系距离,两类基因数据中的每个基因数据均包括多个基因节点和多个基因节点对应的链接数量。
作为本发明的进一步优化方案,步骤S1中,在数据文件储存至后台数据库后,由于在数据库中提取文件为.NET文件,为了后续对于数据的使用与分析,将需要批量更改文件格式,将所提取的数据文件统一保存在相同文件夹目录下,在存有数据文件的文件夹目录下新加入bat文件,并对bat文件进行编写,实现文件格式统一,并对文件夹中的数据资源文件进行迭代,更改文件格式为.csv文件。
作为本发明的进一步优化方案,步骤S2中,统一文件格式的数据分析、清洗预处理步骤包括:利用计算机编译语言,查看数据信息,对于数据缺失情况,进行消灭空值操作,并将本该与其关联的数据清除;检查文件数据字段格式,并对数据进行格式一致化;检测是否有数据同时包含数值与字符串,并对value列进行删除字符串,保留数值操作;另外两类source、target基因数据将保留字符串,删除数值。
作为本发明的进一步优化方案,步骤S3中,利用高斯混合模型将待聚类数据首次聚类的步骤为:从后台数据库中获取文件数据,将基因节点与其对应的链接数量绑定,用序号标记基因节点并以基因节点序号作为X坐标,以统计好的链接数量作为y坐标,建立二维平面坐标系,将坐标系中的坐标值带入高斯混合模型聚类算法中进行首次聚类,聚类后将每类基因数据的坐标y值相加求和,总和最大的类的基因数据标记为数据集合P;
其中,首次聚类时,以链接数量为基础条件,对基因节点进行升序排列,再将坐标值数据导入高斯混合模型聚类算法中进行聚类;
聚类后以链接数量总和为基础条件,进行类的排序,将链接数量最大的类标记为数据集合P。
作为本发明的进一步优化方案,步骤S3中,利用高斯混合模型将待聚类数据次级聚类的步骤为:从后台数据库中获取文件数据,将基因节点与其对应的节点链接关系距离总和进行绑定,用序号重新标记基因节点并以基因节点序号作为X坐标,以基因节点间的关系距离总和为y坐标,进行次级聚类并将每类文件数据的坐标y值相加求和,根据总和排序后将每类文件数据标记为数据集合Q;
其中,次级聚类时,以基因节点间的关系距离总和为基础条件,对基因节点进行升序排列,将坐标值数据导入高斯混合模型聚类算法中进行聚类;
聚类后将每类文件数据中包含的所有基因节点的链接关系距离相加并与其他类文件数据进行比较,以链接关系距离总和为基础条件,进行类的排序,由距离量从高到低依次将类中数据保存于数据集Q
作为本发明的进一步优化方案,步骤S3中首次聚类和次级聚类均采用序号1,2,3,……,n标记基因节点,以标记的基因节点序号作为X坐标。
作为本发明的进一步优化方案,步骤S4中利用虚拟滚动技术进行分层级可视化展示的步骤如下:
S410、加载数据可视化页面,前端请求读取首次聚类结果数据中的数据集合P,以图形的形式展现数据集合P中的基因节点关系;
S420、对页面进行放大操作,将数据集合P中的基因节点关系显示图隐藏,并释放数据,与此同时前端请求读取次级聚类结果数据中的数据集合Q
S430、用户每次对页面执行放大操作,将会重复步骤S420中的类似操作,依次读取次级聚类结果中的数据集合Q
作为本发明的进一步优化方案,步骤S410中以关系图/力导向图的形式展现数据集合P中的基因节点关系。
本发明的有益效果在于:
1)本发明利用序号标记基因节点的想法,设计了一种对于关系型基因数据聚类的处理方法,采用分层级显示数据,并且只加载当前层级数据进行显示,根据聚类算法将数据划分成了许多类,这样大大减少一个页面显示的数据量,方便人们观察;
2)本发明利用浏览器为载体进行数据可视化,有别于C/S架构体系,获取数据信息方便、快捷,后台算法可升级,开发及使用性价比较高;
3)本发明经过实验了解浏览器显示良好的数据量的区间,导入的数据量应在此区间内再进行数据可视化,解决浏览器对大数据量的关系图进行可视化时,由于加载时间过长导致无响应的问题;
4)本发明改善力导向图显示多个数据时关系线条过多,导致观察节点之间关系效果较差的情况,节省大量的人力、物力,并提高了观察的精细化程度;
5)本发明在保证数据的精准下,采用高斯聚类技术对基因节点进行分类,保证了数据分类的准确性;
6)本发明根据节点的重要性进行分层级展示节点,直观的展现了节点的相对重要程度。
附图说明
图1是本发明的整体流程图。
图2是本发明的首次聚类时基因节点坐标在坐标系中显示的二维平面图。
图3是本发明的首次聚类结果以散点图形式进行展示的平面图。
图4是本发明的次级聚类时基因节点坐标在坐标系中显示的二维平面图。
图5是本发明的次级聚类结果以散点图形式进行展示的平面图。
图6是现有技术中500个数据节点显示效果图。
具体实施方式
下面结合附图对本申请作进一步详细描述,有必要在此指出的是,以下具体实施方式只用于对本申请进行进一步的说明,不能理解为对本申请保护范围的限制,该领域的技术人员可以根据上述申请内容对本申请作出一些非本质的改进和调整。
实施例1
如图1所示,一种基于高斯混合模型聚类算法的分层级数据可视化方法,包括以下步骤:
步骤一:在网页中接收用户上传的数据文件并储存至后台数据库,在后台数据库中获取数据,并统一文件格式;
在浏览器网页中接收用户上传的数据文件并储存至后台数据库,将所提取的数据文件统一保存在相同文件夹目录下,由于在数据库中提取文件为.NET文件,为了后续对于数据的使用与分析,将需要批量更改文件格式。在存有数据文件的文件夹目录下新加入bat文件,并对bat文件进行编写,实现文件格式统一,并对文件夹中的数据资源文件进行迭代,更改文件格式为.csv文件。
其中,数据文件中包含source,target,value三类文件数据,source、target为两类基因数据,value值表示source中基因与target中基因之间的关系距离数据。两类基因数据中的每个基因数据均包括多个基因节点和多个基因节点对应的链接数量。
部分数据(25行):
步骤二:对统一文件格式后的数据进行分析、清洗预处理,得到待聚类数据;
利用计算机编译语言,对数据信息进行宏观查看,了解数据集所包含总数据量,对于数据缺失情况,进行消灭空值操作,并将本该与其关联的数据清除;
检查文件数据字段格式,并对数据进行格式一致化;
检测是否有数据同时包含数值与字符串,并对两类基因间的链接关系距离数据进行删除字符串,保留数值操作;另外两类基因数据将保留字符串,删除数值。
步骤三:利用高斯混合模型将待聚类数据进行首次聚类和次级聚类,得到待展示数据;
首次聚类;
1.为首次聚类储存数据,将source、target两类基因数据合并,从首个基因进行迭代,统计每个基因节点的链接数量,将基因节点与其对应的链接数目进行绑定;再使统计结果输出到一个新的文件中进行储存。为后续数据分析与聚类的研究处理提供更优秀的数据支撑。
2.对数据文件进行遍历,将基因节点分别用有序序号标记1,2,3,……,n。设序号作为x坐标值,节点链接数量作为y坐标值,从而确定每个基因在平面直角坐标系中的坐标。将基因节点坐标在坐标系中显示如图2所示。
3.对处理后的数据采取高斯聚类进行分类(簇的数量随着数据总量的变化而变化),并将聚类结果储存,方便后续前端显示调用;
为保证聚类结果理想,我们将以链接数量为基础条件,对基因节点进行升序排列。将坐标数据导入算法中进行聚类,经过多次更换聚类算法进行比较,最终将采取高斯聚类。并且经过测试可知,在浏览器中显示此方法的可视化效果时,渲染500左右个数据节点时较为适中,所以我们将类的数量参数调为5。聚类结果以散点图形式进行展示,效果如图3所示。
聚类后将每类基因数据的坐标y值相加并与不同类基因数据之间进行比较,以链接数量总和为基础条件,进行类的排序,将链接数量总和最大的类的基因数据标记为数据集合P,并将数据集合P暂时保存,等待前端页面请求显示数据。
次级聚类;
1.次级聚类数据储存,对用户上传的数据文件进行重新读取,读取数据方式与首次相同,从首对基因进行迭代。然后统计每个基因与其他相连基因的链接关系距离数据,将基因节点与其对应的节点链接关系距离总和进行绑定,再使统计结果输出到一个新的文件中进行储存;
2.对数据文件进行遍历,舍弃首次聚类时绑定的基因节点序号,将基因节点重新用有序序号标记1,2,3,……,n,并设序号作为x坐标值,以基因节点间的关系距离总和为y坐标,从而确定每个基因在平面直角坐标系中的坐标;将基因节点坐标在坐标系中显示,如图4所示。
3.对处理后的次级聚类数据采取同样的高斯聚类进行分类(簇的数量随着数据总量的变化而变化),并将聚类结果储存,在进行数据可视化时调用;
为保证聚类结果理想,我们将以基因节点间的关系距离总和为基础条件,对基因节点进行升序排列,将坐标值数据导入高斯混合模型聚类算法中进行聚类,此处我们依旧采取高斯算法进行聚类。基础数据集没有更改,此处我们为保证浏览器的正常显示,依旧将采取聚类数量为5。聚类结果以散点图形式进行展示,效果如图5所示。
聚类后将每类文件数据中包含的所有基因节点的链接关系距离相加并与其他类文件数据进行比较,以链接关系距离总和为基础条件,进行类的排序,由距离量从高到低依次将类中数据保存于数据集Q
步骤四:建立前端页面,利用虚拟滚动技术将待展示数据进行分层级可视化展示:
1.加载数据可视化页面,前端请求读取首次聚类结果数据中的数据集合P,以关系图/力导向图的形式展现数据集合P中的基因节点关系;
2.对页面进行放大操作,将数据集合P中的基因节点关系显示图隐藏,并释放数据,与此同时前端请求读取次级聚类结果数据中的数据集合Q
3.用户每次对页面执行放大操作,将会重复步骤S420中的类似操作,依次读取次级聚类结果中的数据集合Q
所述虚拟滚动技术实现数据分级显示:
用户进入前端页面显示首次聚类的数据(关系图),然后用户对页面进行放大,首次聚类的数据消失不显示,并将其释放掉(缩小再显示回来,只不过这时候是重新加载这批数据),显示一批新的数据,即次级聚类的数据,后期继续放大页面操作传来的数据都是用次级聚类的方法处理。页面展示中不显示的数据都先不加载,只加载显示的局部,能够大大减小浏览器的负荷,避免卡顿。
当大数据量在网页中进行数据可视化时,因为数据量巨大,在用户导入项目数据后,浏览器对数据进行分析与渲染,可能因为上传时间过长而导致停止响应。而本发明方法采用分层级显示数据,并且只加载当前层级数据进行显示,当用户进行放大或缩小操作时会切换数据可视化的层级,对于不显示的数据web端将会将其释放,再次请求时重新加载读取与渲染。
本发明方法所显示的“力导向图”/“关系图”根据聚类算法将数据划分成了许多类,这样大大减少一个页面显示的数据量,方便人们观察。
以上所述实施例仅表达了本发明的几种实施方式,其描述较为具体和详细,但并不能因此而理解为对本发明专利范围的限制。应当指出的是,对于本领域的普通技术人员来说,在不脱离本发明构思的前提下,还可以做出若干变形和改进,这些都属于本发明的保护范围。
- 基于高斯混合模型聚类算法的分层级数据可视化方法
- 基于高斯混合模型聚类算法的分层级数据可视化方法
