靶向5/B/6金属β-内酰胺酶的2’-全修饰XNA适配体及其获取方法
文献发布时间:2024-04-18 19:58:30
技术领域
本发明涉及生物技术领域,具体为利用定向进化后的SFM4-6聚合酶在SELEX每一轮筛选当中转录DNA文库得到2’-全修饰的非天然核酸适配体文库,并用该文库筛选靶向5/B/6金属β-内酰胺酶的2’-全修饰非天然核酸适配体,最终得到三条70nt的靶向5/B/6金属β-内酰胺酶的2’-全修饰非天然核酸适配体。
背景技术
β-内酰胺环类抗生素药物如青霉素,头孢等是生活中最常见的抗菌类药物之一,随着这类抗生素的广泛使用,细菌体内进化出了相应的耐药机制。其中,细菌产生耐药性最常见的机制是细菌体内产生的β-内酰胺酶对β-内酰胺环类抗生素进行水解,β-内酰胺环发生开环,从而使β-内酰胺环类抗生素失活(如图1所示为β-内酰胺酶使抗生素开环失活机制)。β-内酰胺酶大致可以分为A、B、C、D四类,其中A、C、D类均以丝氨酸残基作为活性部位,而B类为金属β-内酰胺酶MBLs(Metallo-β-lactamases,MBLs),该类β-内酰胺酶需要以一个或两个金属锌离子作为活性中心。据文献报道,产生MBLs的细菌数量正在增加,并在耐药菌中占了很大的比例。
适配体是基于SELEX技术(即指数富集的配体系统进化技术)从随机寡核苷酸文库中筛选获得的对靶物质具有很高特异性与亲和力的单链核苷酸序列,相比化学小分子药物,适配体药物具极高的亲和性和特异性识别作用。与抗体相比,核酸适配体具有无免疫原性、不需要使用动物产生、可以直接化学合成、筛选周期短、可反复变性、易于标记等优点。在过去的三十年,核酸适配体越来越受欢迎,被视为可用作亲和试剂,分子成像,诊断,甚至是治疗药物的潜在分子。但是,由于ssDNA/RNA的不稳定性,在适配体进入细菌体内后很快被核酸酶降解而失去活性,因此增强适配体的稳定性成为热点研究方向。大量的研究证明:RNA核糖2’位的羟基被甲氧基(2’-OMe)或氟(2’-F)修饰取代的核酸链可以显著提高核酸分子的稳定性。因此,如果可以建立一个2’-F,2’-OMe修饰的单链核酸文库,再从中筛选出可以靶向靶标蛋白的全修饰核酸适配体,就可以得到既对核酸酶稳定又具有结合活性的核酸适配体。
XNA(这里主要指以2’-F或2’-OMe修饰的核苷酸为底物,通过酶法或者化学合成的核酸链)适配体是由非天然核苷酸单体通过3’-5’磷酸二酯键连接,在一定的缓冲液下,通过自身卷曲和折叠,形成特定的空间二级结构,并对靶标分子具有高亲和力和高特异性的核酸单链。与天然的DNA或RNA适配体相比,XNA适配体具有更强的稳定性以及耐核酸酶降解能力,这使得XNA核酸适配体替代天然的DNA或RNA适配体成为可能。
由于聚合酶对底物的特异性,天然的聚合酶难以识别修饰的核苷酸。但是随着聚合酶定向进化技术的发展,使得聚合酶识别非天然核苷酸底物成为可能,实验室酶法合成非天然核酸序列变得更加容易。本专利通过使用定向进化的Taq DNA聚合酶Stoffel片段Stoffel fragment(SF)突变株——SFM4-6转录出糖环2’-C不同修饰的XNA文库,利用SELEX技术对得到的文库与固定好的靶标蛋白蜡状芽孢杆菌5/B/6中的金属β-内酰胺酶进行孵育结合。通过不同的洗脱条件,将未结合的XNA序列除掉,最后用变性条件洗脱得到结合靶标蛋白的序列。然后再通过反转录,扩增,转录构建后续筛选的文库。经过多轮筛选,最终得到靶向蜡状芽孢杆菌5/B/6中的金属β-内酰胺酶的XNA适配体。最后通过EMSA测试XNA适配体对金属β-内酰胺酶5/B/6的结合活性,证明筛选到的适配体均具有较高的结合5/B/6中的金属β-内酰胺酶的活性,未来有开发为检测分子的潜在应用。
发明内容
本发明包括2’-全修饰非天然核酸适配体文库的制备方法,其具体操作是采用四种不同组合(包括2’-F修饰的尿嘧啶核苷酸,2’-F修饰的胞嘧啶核苷酸,2’-OMe修饰的腺嘌呤核苷酸以及2’-OMe修饰的鸟嘌呤核苷酸;2’-F修饰的胞嘧啶核苷酸,2’-OMe修饰的尿嘧啶核苷酸,2’-OMe修饰的腺嘌呤核苷酸以及2’-OMe修饰的鸟嘌呤核苷酸;2’-F修饰的尿嘧啶核苷酸,2’-F修饰的鸟嘌核苷酸,2’-OMe修饰的腺嘌呤核苷酸以及2’-OMe修饰的胞嘧啶核苷酸;2’-F修饰的腺嘌呤核苷酸,2’-F修饰的鸟嘌核苷酸,2’-OMe修饰的尿嘧啶核苷酸以及2’-OMe修饰的胞嘧啶核苷酸)的非天然核苷酸为元件,在SFM4-6聚合酶的作用下,以L90-N30文库为模板,直接转录合成DNA/XNA杂合体产物,经TURBO DNase降解后,获取到2’-全修饰的XNA文库。
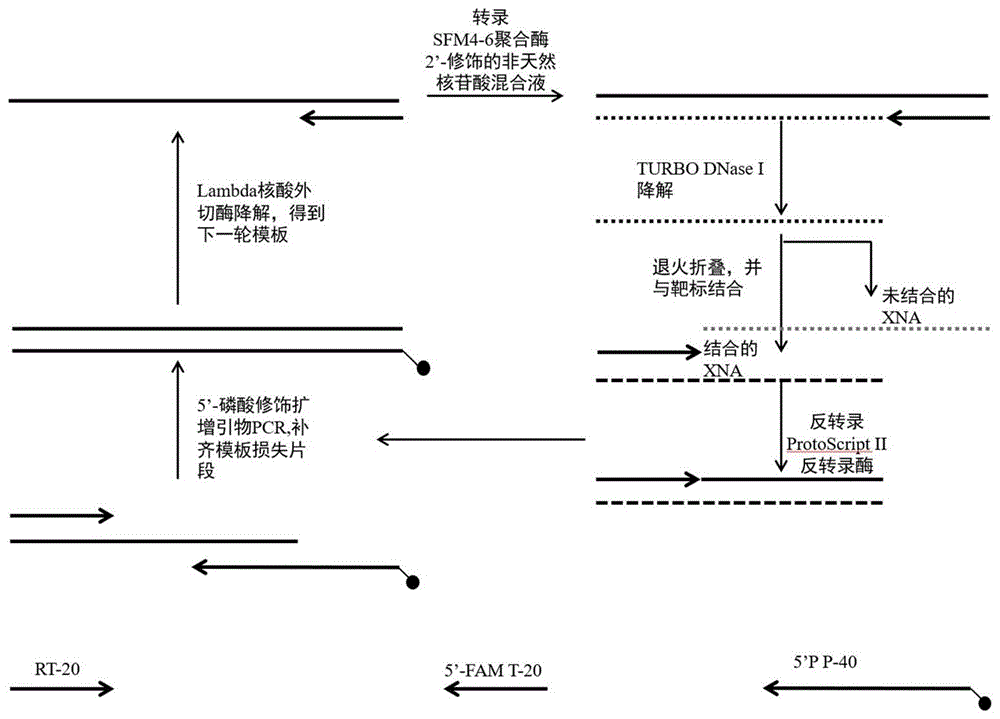
本发明包括筛选靶向蜡状芽孢杆菌5/B/6中的金属β-内酰胺酶全修饰非天然核酸适配体的技术。筛选靶向5/B/6金属β-内酰胺酶非天然核酸适配体的过程中,第二轮以后的转录文库获取方法主要采用5’-磷酸化扩增引物对筛选到的XNA序列反转录产物做PCR扩增,该引物与模板3’-端互补40nt,包含与转录产物5’-端互补的20nt以及转录引物部分。扩增产物用Lambda核酸外切酶降解,通过控制降解时间,能最大效率降解掉磷酸化的序列以获取单链DNA文库,然后再使用SFM4-6聚合酶转录获取下一轮的XNA文库(筛选流程如图2所示)。在筛选靶向5/B/6金属β-内酰胺酶非天然核酸适配体的过程中,每轮筛选所用的盐离子浓度、洗脱时间、洗脱次数会有所不同,随着轮数的增加,逐渐增加选择压力。
本发明采用高通量测序的方法,通过对不同修饰的文库添加不同的识别碱基进行合并测序,共测得有效序列为1,946,602条,其中2’-F-C,2’-F-U,2’-OMe-A,2’-OMe-G的文库共测序193,889条,2’-F-C,2’-OMe-U,2’-OMe-A,2’-OMe-G的文库共测序360,992条,2’-F-A,2’-F-G,2’-OMe-C,2’-OMe-U,的文库共测序62,886条,2’-F-G,2’-F-U,2’-OMe-A,2’-OMe-C共测序1,328,440条。
本发明共使用了四种不同修饰的XNA文库进行SELEX,且四种不同修饰的文库在筛选之后均富集到以下三条相同的序列,利用RNA-fold预测其可能的二级结构如图2所示;
5/B/6-XAp-1:
5’-AUCUCUAUGCUAUCAAUGCGCUCUCGUUUCAUCGGUAUCAUUACCCCCAUCGGUUUCACACAGUCCCAGU-3’
5/B/6-XAp-2:
5’-AUCUCUAUGCUAUCAAUGCGUGGCUACAUGGACUCGUUUAUCCAAUCGGACGGUUUCACACAGUCCCAGU-3’
5/B/6-XAp-3:
5’-AUCUCUAUGCUAUCAAUGCGCGGCUAAAAAUCGUACCAAUGUUUCCGAGCGGUUUCACACAGTCCCAGU-3’
其中AUCG均为糖环2’-C修饰的非天然核苷酸。
序列5/B/6-XAp-1在2’-F-C,2’-F-U,2’-OMe-A,2’-OMe-G的文库中富集到11.2910%,在2’-F-C,2’-OMe-U,2’-OMe-A,2’-OMe-G的文库中富集到25.4191%,在2’-F-A,2’-F-G,2’-OMe-C,2’-OMe-U的文库中富集到36.8270%,在2’-F-G,2’-F-U,2’-OMe-A,2’-OMe-C的文库中富集到12.6144%。
序列5/B/6-XAp-2在2’-F-C,2’-F-U,2’-OMe-A,2’-OMe-G的文库中富集到3.8315%,在2’-F-C,2’-OMe-U,2’-OMe-A,2’-OMe-G的文库中富集到2.8463%,在2’-F-A,2’-F-G,2’-OMe-C,2’-OMe-U的文库中富集到0.4198%,在2’-F-G,2’-F-U,2’-OMe-A,2’-OMe-C的文库中富集到1.5169%。
序列5/B/6-XAp-3在2’-F-C,2’-F-U,2’-OMe-A,2’-OMe-G的文库中富集到2.2647%,在2’-F-C,2’-OMe-U,2’-OMe-A,2’-OMe-G的文库中富集到2.3895%,在2’-F-A,2’-F-G,2’-OMe-C,2’-OMe-U的文库中富集到0.29577%,在2’-F-G,2’-F-U,2’-OMe-A,2’-OMe-C的文库中富集到0.9630%。
与现有技术相比,本发明的优势在于:
相较于固相合成来说,固相合成全修饰XNA难度非常大,通常超过30nt的XNA序列的合成成本就非常高,且成功率低。并且固相合成无法完成SELEX整个筛选过程的循环,即使能合成第一轮的全修饰文库,也无法进行下一轮的扩增-转录-反转录的循环。本发明利用定向进化后的SFM4-6聚合酶转录合成XNA适配体文库,不仅合成效率高,合成的文库序列更长,并且在整个SELEX过程获取每一轮的全修饰XNA文库中起到了决定性的作用,是整个筛选流程得以循环的关键。与现今常用的利用其他聚合酶突变株转录得到XNA序列相比,定向进化后的SFM4-6聚合酶能够耐高温,通过高温转录得到的文库多样性更大,并且转录效率更高,在适宜的条件下转录得到的全长产物更多,操作流程更加简便。
附图说明
图1为β-内酰胺酶使β-内酰胺环类抗生素失活的机制图;
图2为靶向5/B/6金属β-内酰胺酶2’-全修饰的非天然核酸适配体的筛选流程图;
图3a为2’-全修饰XNA文库的转录图,其中,泳道M:Low molecular weightladder;泳道1:2’-F-C,U,2’-OMe-A,G非天然核酸适配体的转录;泳道2:2’-F-C,2’-OMe-A,U,G非天然核酸适配体的转录;泳道3:2’-F-A,G,2’-OMe-C,U非天然核酸适配体的转录;泳道4:2’-F-U,G,2’-OMe-A,C非天然核酸适配体的转录。
图3b为2’修饰XNA文库转录后降解的结果。其中,泳道M:Low molecular weightladder;泳道1:2’-F-C,U,2’-OMe-A,G非天然核酸适配体转录后降解;泳道2:2’-F-C,2’-OMe-A,U,G非天然核酸适配体转录后降解;泳道3:2’-F-A,G,2’-OMe-C,U非天然核酸适配体转录后降解;泳道4:2’-F-U,G,2’-OMe-A,C非天然核酸适配体转录后降解。
图3c表示2’-F-C,U,2’-OMe-A,G非天然核酸适配体的纯度测试结果,黑色实心圆点的qPCR曲线的模板是转录后未降解的2’-F-C,U,2’-OMe-A,G非天然核酸适配体,使用RT-20和P-40进行qPCR验证,起峰循环数为5。黑色空心圆点qPCR曲线的模板是转录后经降解的2’-F-C,U,2’-OMe-A,G非天然核酸适配体,使用RT-20和P-40进行qPCR验证,起峰循环数为20。两者的起峰相差15个循环,可以判定降解后的2’-F-C,U,2’-OMe-A,G非天然核酸适配体较纯。
图4a为2’-F-C,U,2’-OMe-A,G非天然核酸适配体第一轮筛选的qPCR结果。其中,黑色实心圆点的qPCR曲线的模板为未与5/B/6金属β-内酰胺酶结合的非天然核酸适配体。随后的qPCR曲线的模板为第1、5、8、10次洗脱的洗脱液。黑色实心三角形的qPCR曲线的模板为95%甲酰胺95℃洗脱的洗脱液纯化产物。可以看出最终洗脱曲线的起峰循环早于第十次洗涤的起峰循环,证明靶向5/B/6金属β-内酰胺酶2’-F-C,U,2’-OMe-A,G非天然核酸适配体在第一轮筛选中存在富集。
图4b为2’-F-C,U,2’-OMe-A,G非天然核酸适配体第二轮筛选的qPCR结果。其中,黑色实心圆点的qPCR曲线的模板为未与5/B/6金属β-内酰胺酶结合的非天然核酸适配体。随后的qPCR曲线的模板为第1、5、8、10次洗脱的洗脱液。黑色实心三角形的qPCR曲线的模板为95%甲酰胺95℃洗脱的洗脱液纯化产物。可以看出最终洗脱曲线的起峰循环早于第十次洗涤的起峰循环,证明靶向5/B/6金属β-内酰胺酶2’-F-C,U,2’-OMe-A,G非天然核酸适配体在第二轮筛选中存在富集。
图4c为2’-F-C,U,2’-OMe-A,G非天然核酸适配体第四轮筛选的qPCR结果。其中,黑色实心圆点的qPCR曲线的模板为未与5/B/6金属β-内酰胺酶结合的非天然核酸适配体。随后的qPCR曲线的模板为第1、5、8、10次洗脱的洗脱液。黑色实心三角形的qPCR曲线的模板为95%甲酰胺95℃洗脱的洗脱液纯化产物。可以看出最终洗脱曲线的起峰循环早于第十次洗涤的起峰循环,证明靶向5/B/6金属β-内酰胺酶2’-F-C,U,2’-OMe-A,G非天然核酸适配体在第四轮筛选中存在富集。
图4d为2’-F-C,U,2’-OMe-A,G非天然核酸适配体第五轮筛选的qPCR结果。其中,黑色实心圆点的qPCR曲线的模板为未与5/B/6金属β-内酰胺酶结合的非天然核酸适配体。随后的qPCR曲线的模板为第1、5、8、10次洗脱的洗脱液。黑色实心三角形的qPCR曲线的模板为95%甲酰胺95℃洗脱的洗脱液纯化产物。可以看出elution曲线的起峰循环数几乎与未结合曲线的起峰循环重合,证明靶向5/B/6金属β-内酰胺酶2’-F-C,U,2’-OMe-A,G非天然核酸适配体在第五轮筛选中存在大量富集。
图5a为序列5/B/6-XAp-1的二级结构预测图;
图5b为序列5/B/6-XAp-2的二级结构预测图;
图5c为序列5/B/6-XAp-3的二级结构预测图;
图6为三条70nt的全修饰非天然核酸适配体与5/B/6金属β-内酰胺酶结合验证。泳道1为marker。泳道2为适配体序列一,泳道3为适配体序列一与5/B/6金属β-内酰胺酶孵育;泳道4为适配体序列二,泳道5为适配体序列二与5/B/6金属β-内酰胺酶孵育;泳道6为适配体序列三,泳道7为适配体序列三与5/B/6金属β-内酰胺酶孵育。
图7a为适配体序列一分别与5/B/6金属β-内酰胺酶以及569金属β-内酰胺酶的特异性结合验证。泳道1为marker。泳道2为适配体序列一,泳道3为适配体序列一与5/B/6金属β-内酰胺酶孵育结果;泳道4为适配体序列一与569金属β-内酰胺酶孵育结果。
图7b适配体序列二分别与5/B/6金属β-内酰胺酶以及569金属β-内酰胺酶的特异性结合验证。泳道1为marker。泳道2为适配体序列二,泳道3为适配体序列二与5/B/6金属β-内酰胺酶孵育结果;泳道4为适配体序列二与569金属β-内酰胺酶孵育结果。
图7c为适配体序列三分别与5/B/6金属β-内酰胺酶以及569金属β-内酰胺酶的特异性结合验证。泳道1为marker。泳道2为适配体序列三,泳道3为适配体序列三与5/B/6金属β-内酰胺酶孵育结果;泳道4为适配体序列三与569金属β-内酰胺酶孵育结果。
具体实施方式
下面结合说明书附图和具体实施例对本发明作出进一步地详细阐述,所述实施例只用于解释本发明,并非用于限定本发明的范围。
本实施例中的靶向5/B/6金属β-内酰胺酶的2’-全修饰XNA适配体的制备方法,步骤为:利用定向进化后的SFM4-6聚合酶转录得到2’-全修饰的非天然核酸适配体文库,并用该文库筛选靶向5/B/6金属β-内酰胺酶的2’-全修饰非天然核酸适配体,最终得到三条70nt的靶向5/B/6金属β-内酰胺酶的2’-全修饰非天然核酸适配体。
所述全修饰的非天然核酸适配体文库制备方法为采用四种不同组合的非天然核苷酸为元件,在SFM4-6聚合酶的作用下,以L90-N30文库为模板,直接转录合成DNA/XNA杂合体产物,经TURBO DNase降解后,制备得到2’-全修饰的XNA文库。
所述四种不同组合包括2’-F修饰的尿嘧啶核苷酸,2’-F修饰的胞嘧啶核苷酸,2’-OMe修饰的腺嘌呤核苷酸以及2’-OMe修饰的鸟嘌呤核苷酸;2’-F修饰的胞嘧啶核苷酸,2’-OMe修饰的尿嘧啶核苷酸,2’-OMe修饰的腺嘌呤核苷酸以及2’-OMe修饰的鸟嘌呤核苷酸;2’-F修饰的尿嘧啶核苷酸,2’-F修饰的鸟嘌核苷酸,2’-OMe修饰的腺嘌呤核苷酸以及2’-OMe修饰的胞嘧啶核苷酸;或2’-F修饰的腺嘌呤核苷酸,2’-F修饰的鸟嘌核苷酸,2’-OMe修饰的尿嘧啶核苷酸以及2’-OMe修饰的胞嘧啶核苷酸。
具体方法为:利用固相合成的随机并且5’-端和3’-端不对称的DNA文库,设计与其互补的转录引物部分以及降解后补齐原始文库序列的扩增引物部分,不仅使得整个筛选流程简单化,而且使酶法进行非天然核酸适配体的SELEX得以循环。
三条靶向5/B/6金属β-内酰胺酶的2’-全修饰XNA适配体,其特征在于,所述三条靶向5/B/6金属β-内酰胺酶的2’-全修饰XNA适配体的序列分别为:
5/B/6-XAp-1:
5’-AUCUCUAUGCUAUCAAUGCGCUCUCGUUUCAUCGGUAUCAUUACCCCCAUCGGUUUCACACAGUCCCAGU-3’
其中AUCG均为糖环2’-C修饰的非天然核苷酸。
5/B/6-XAp-2:
AUCUCUAUGCUAUCAAUGCGUGGCUACAUGGACUCGUUUAUCCAAUCGGACGGUUUCACACAGUCCCAGU
其中AUCG均为糖环2’-C修饰的非天然核苷酸。
5/B/6-XAp-3:
AUCUCUAUGCUAUCAAUGCGCGGCUAAAAAUCGUACCAAUGUUUCCGAGCGGUUUCACACAGTCCCAGU
其中AUCG均为糖环2’-C修饰的非天然核苷酸。
序列5/B/6-XAp-1在2’-F-C,2’-F-U,2’-OMe-A,2’-OMe-G的文库中富集到11.2910%,在2’-F-C,2’-OMe-U,2’-OMe-A,2’-OMe-G的文库中富集到25.4191%,在2’-F-A,2’-F-G,2’-OMe-C,2’-OMe-U的文库中富集到36.8270%,在2’-F-G,2’-F-U,2’-OMe-A,2’-OMe-C的文库中富集到12.6144%。
序列5/B/6-XAp-2在2’-F-C,2’-F-U,2’-OMe-A,2’-OMe-G的文库中富集到3.8315%,在2’-F-C,2’-OMe-U,2’-OMe-A,2’-OMe-G的文库中富集到2.8463%,在2’-F-A,2’-F-G,2’-OMe-C,2’-OMe-U的文库中富集到0.4198%,在2’-F-G,2’-F-U,2’-OMe-A,2’-OMe-C的文库中富集到1.5169%。
序列5/B/6-XAp-3在2’-F-C,2’-F-U,2’-OMe-A,2’-OMe-G的文库中富集到2.2647%,在2’-F-C,2’-OMe-U,2’-OMe-A,2’-OMe-G的文库中富集到2.3895%,在2’-F-A,2’-F-G,2’-OMe-C,2’-OMe-U的文库中富集到0.29577%,在2’-F-G,2’-F-U,2’-OMe-A,2’-OMe-C的文库中富集到0.9630%。
通过改变筛选得到的三条2’-全修饰XNA适配体序列的修饰类型,其结合活性不变,不同的修饰类型对适配体结合活性无明显影响,但对其抗核酸酶降解的稳定性有显著提高。XNA适配体对靶标蛋白5/B/6金属β-内酰胺酶具有很高的特异性,对5/B/6金属β-内酰胺酶同源度大于90%的569金属β-内酰胺酶无结合活性。
实施例1:高容量单链寡核苷酸文库和引物的设计与合成
构建了长度为90nt的单链寡核苷酸文库,5’端和3’端分别为20nt和40nt的固定序列区(结合引物所必需的序列)组成,中间为30nt的随机序列。
L90-N30:5’-ACTGGGACTGTGTGAAACCG-30N-CGCATTGATAGCATAGAGATCCGTCATAAAGTGTAAAGCC-3’
5’-FAM-T-20:5’-FAM-GGCTTTACACTTTATGACGG-3’
T-20:5’-GGCTTTACACTTTATGACGG-3’
RT-20:5’-ACTGGGACTGTGTGAAACCG-3’
P-40:5’-GGCTTTACACTTTATGACGGATCTCTATGCTATCAATGCG-3’
实施例2:四种全修饰非天然单链寡核苷酸文库的构建
分别制备40管不同搭配的全修饰文库,转录体系如表1-1、1-2、1-3、1-4所示,将模板与引物、聚合酶缓冲溶液以及ddH
表1-1 转录体系
表1-2 转录体系
表1-3 转录体系
表1-4 转录体系
将上述转录产物用Zymo试剂盒纯化后,通过TURBO DNase酶降解除去体系中的模板和引物DNA(如表2),反应于37℃孵育16h。将上述降解产物用Zymo试剂盒纯化后得到70nt的2’-全修饰非天然文库。如图3a所示:泳道1-4得到产物大小为70nt的四种不同2’修饰的XNA文库;如图3b泳道1-4无明显模板和引物条带,说明模板L90-N30和转录引物5’-FAM-T-20已经基本完全降解了。
表2 TURBO DNase酶降解体系
为验证非天然核酸文库的制备纯度,降解前和降解后分别取1μl稀释100倍为扩增模板进行qPCR扩增(如表3),反应条件为94℃ 2min,40个循环:94℃ 30s,60℃ 30s,70℃1min,70℃ 5min。从图3c可以看出,降解前产物与降解后产物先后起峰,这两者Ct值相差10个以上的循环,说明获得了纯度较高的2’全修饰非天然文库。
表3 文库验证qPCR体系
实施例3:5/B/6金属β-内酰胺酶的筛选流程
5/B/6金属β-内酰胺酶的筛选流程如图2所示。向全修饰非天然文库加入结合缓冲溶液(20mM HEPES pH7.5,150mM NaCl,6mM KCl,2mM MgCl
为排除与酶标板结合的适配体,先将候选适配体文库加入空白的酶标板中37℃孵育2h,后再将未与酶标板结合的适配体文库转移到过夜孵育上5/B/6金属β-内酰胺酶的酶标板中,37℃孵育2h后,用200μl结合缓冲溶液清洗孔10次,保留未结合的候选适配体以及10次清洗的结合缓冲溶液。
用95℃预热的95%甲酰胺洗脱酶标板3次,使其终体积为200μl,保留含有竞争性结合的候选适配体文库的洗脱液溶液。
为了提高全修饰非天然核酸适配体的特异性识别能力,第二轮筛选时,候选适配体文库加入孵育上5/B/6金属β-内酰胺酶的酶标板之前,先加入到用BSA孵育的酶标板中,37℃孵育2h后,再将未与BSA结合的适配体文库加入到孵育上5/B/6金属β-内酰胺酶的酶标板中。
取等体积的上述保留的未结合的候选适配体,8次清洗的结合缓冲溶液,洗脱溶液进行反转录得到相对应的cDNA链,体系如表4所示,将上述样品与反转录引物RT-20、反转录酶缓冲液、ddH
取反转录体系体积的1/20为扩增模板进行qPCR扩增,体系如表5所示,反应条件为94℃ 2min,30个循环:94℃ 30s,60 ℃ 30s,70℃ 1min,70℃ 5min。从图4a-图4d可以看出,黑色实心圆点的qPCR曲线的模板为未与5/B/6金属β-内酰胺酶结合的非天然核酸适配体。随后的qPCR曲线的模板为第1、5、8、10次洗脱的洗脱液。黑色实心三角形的qPCR曲线的模板为95%甲酰胺95℃洗脱的洗脱液纯化产物。可以看出最终洗脱曲线的起峰循环均早于第十次洗涤的起峰循环,证明靶向5/B/6金属β-内酰胺酶2’-F-C,U,2’-OMe-A,G非天然核酸适配体在每一轮的筛选中均存在富集。特别是第五轮的qPCR结果(图4d)可以看出elution曲线的起峰循环数几乎与未结合曲线的起峰循环重合,证明靶向5/B/6金属β-内酰胺酶2’-F-C,U,2’-OMe-A,G非天然核酸适配体在第五轮筛选中存在大量富集,因此我们停止了筛选。
表4 反转录体系
表5 qPCR体系
实施例4:PCR制备下一轮转录模板
将上述洗脱液用Zymo试剂盒纯化后进行反转录得到适配体文库相对应的cDNA文库,体系如表6所示,将纯化后的产物与反转录引物、反转录酶缓冲液、ddH
表6 反转录体系
以结合的核酸适配体反转录合成的cDNA为模板,扩增得到下一轮转录的模板,体系如表7所示,为了便于后续得到下一轮转录的单链模板,体系所用的是5’磷酸化修饰的扩增产物,反应条件为94℃ 2min,30个循环:94℃ 30s,60℃ 30s,70℃ 1min,70℃ 5min。将上述扩增产物用大量DNA纯化试剂盒纯化后,用Lambda核酸外切酶于37℃降解16h,体系如表8所示,降解产物用Zymo试剂盒纯化除去未降解完全的双链得到下一轮转录的单链模板。
表7 扩增体系
表8 Lambda核酸外切酶降解体系
为了提高不同修饰的XNA适配体对5/B/6金属β-内酰胺酶的亲和力,当每轮筛选后,发现文库的富集已达最大化后,可以通过提高结合缓冲液中NaCl的浓度以及增加表面活性剂浓度来增加不同修饰的XNA适配体对5/B/6金属β-内酰胺酶结合的难度,筛选过程使用的结合缓冲液离子浓度如表9-1所示。
表9-1 SELEX基础结合缓冲液
表9-2 不同轮数缓冲液改变量
实施例5:高通量测序
以最后一轮与5/B/6金属β-内酰胺酶结合的候选适配体文库对应的cDNA文库为模板,用四条带有不同条形码序列的引物分别扩增,使四个文库一端带上不同的碱基序列。(如表10-1至10-4)。反应条件为94℃ 2min,30个循环:94℃ 30s,60℃ 30s,70℃ 1min,70℃ 5min。将扩增产物用大量DNA纯化试剂盒纯化后,跑变性胶,如图5b所示。切下相对应的目标条带,去掉扩增中非特异性产生的其他dsDNA。用聚丙烯酰胺DNA回收试剂盒纯化得到高纯度的文库,将最终得到的文库送到上海生工生物科技有限公司进行扩增子测序,测序平台为Illumina HiSeq250,运行模式为PE150。
比对测序结果,将所有序列按照出现的频率进行由高到低的排序。重复出现频率最高的几条序列即为可能靶向5/B/6金属β-内酰胺酶的候选适配体序列,我们从上海生工订购得到重复率排名前三的序列的互补序列用建库的转录方法进行转录得到富集后的全修饰XNA适配体序列,并进行进一步的活性表征实验。
表10-1 2’-F-CU,2’-OMe-AG文库测序扩增体系
表10-2 2’-F-C,2’-OMe-AUG文库测序扩增体系
表10-3 2’-F-AG,2’-OMe-CU文库测序扩增体系
表10-4 2’-F-UG,2’-OMe-AC文库测序扩增体系
Seq-FC:5’-TAATACGACTCACTATAGGCATACTGGGACTGTGTGAAACCG-3’
Seq-FAG:5’-TAATACGACTCACTATAGGTTCACTGGGACTGTGTGAAACCG-3’
Seq-FUG:5’-TAATACGACTCACTATAGGGATACTGGGACTGTGTGAAACCG-3’
实施例6:适配体结合活性验证
根据测序的结果,合成富集频率前三的三条序列的互补序列,运用5’-FAM-T-20转录出不同修饰的XNA序列,并将转录降解得到的不同修饰的XNA序列在反应缓冲液中进行95℃ 5min处理,并缓慢降至室温,然后在冰上放置10分钟使其折叠成二级结构,然后与5/B/6金属β-内酰胺酶在基础缓冲液中孵育3h,然后通过跑PAGE胶验证XNA适配体与5/B/6金属β-内酰胺酶的结合情况,结合实验数据见图6。
实施例7:适配体与靶标蛋白特异性结合活性验证
根据测序的结果,合成富集频率前三的三条序列的互补序列,运用5’-FAM-T-20转录出不同修饰的XNA序列,并将转录降解得到的不同修饰的XNA序列在反应缓冲液中进行95℃ 5min处理,并缓慢降至室温,然后在冰上放置10分钟使其折叠成二级结构,然后分别与5/B/6金属β-内酰胺酶以及569金属β-内酰胺酶在基础缓冲液中孵育3h,然后通过跑PAGE胶验证XNA适配体对5/B/6金属β-内酰胺酶的特异性结合情况,结合实验数据见图7a、图7b和图7c。
序列表
<110> 华南理工大学
<120> 靶向5/B/6金属β-内酰胺酶的2’-全修饰XNA适配体及其获取方法
<160> 13
<170> SIPOSequenceListing 1.0
<210> 2
<211> 90
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 2
actgggactg tgtgaaaccg nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn cgcattgata 60
gcatagagat ccgtcataaa gtgtaaagcc 90
<210> 2
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 2
ggctttacac tttatgacgg 20
<210> 3
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 3
ggctttacac tttatgacgg 20
<210> 4
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 4
actgggactg tgtgaaaccg 20
<210> 5
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 5
ggctttacac tttatgacgg atctctatgc tatcaatgcg 40
<210> 6
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 6
ggctttacac tttatgacgg atctctatgc tatcaatgcg 40
<210> 7
<211> 42
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 7
taatacgact cactatagga tgactgggac tgtgtgaaac cg 42
<210> 8
<211> 42
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 8
taatacgact cactataggc atactgggac tgtgtgaaac cg 42
<210> 9
<211> 42
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 9
taatacgact cactataggt tcactgggac tgtgtgaaac cg 42
<210> 10
<211> 42
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 10
taatacgact cactataggg atactgggac tgtgtgaaac cg 42
<210> 14
<211> 47
<212> DNA/RNA
<213> 人工序列(Artificial Sequence)
<400> 14
accagcacaa gcgcccgcac ggacaacccc cacggcacac agcccag 47
<210> 14
<211> 70
<212> DNA/RNA
<213> 人工序列(Artificial Sequence)
<400> 14
aucucuaugc uaucaaugcg uggcuacaug gacucguuua uccaaucgga cgguuucaca 60
cagucccagu 70
<210> 14
<211> 68
<212> DNA/RNA
<213> 人工序列(Artificial Sequence)
<400> 14
aucucuaugc uaucaaugcg cggcuaaaaa ucguaccaau guuuccgagc gguuucacac 60
agcccagu 68
- 一种胆固醇氧化酶修饰的杂化金属有机框架肿瘤靶向纳米制剂的制备方法
- 一种胆固醇氧化酶修饰的杂化金属有机框架肿瘤靶向纳米制剂的制备方法
