一种橡胶树品种DNA指纹库的构建方法
文献发布时间:2024-04-18 20:01:30
技术领域
本发明属于橡胶树品种分子生物技术领域,尤其涉及一种橡胶树品种DNA指纹库的构建方法。
背景技术
橡胶树自1904年引种至中国以来,迄今国内保存的橡胶树栽培品种、野生种质、以及同属种质材料5个种、1个变种共计6165份种质材料。长期以来,橡胶树品种资源鉴定一直以形态性状鉴定为主要依据,这种经典的、常规鉴定方法一般要在橡胶树开花后才能完成其鉴定过程。然而,橡胶树属多年生异花授粉高大乔木,一般在定植后5-6年才能开花结果,具有有性世代周期长,品种高度杂合等不利因素,导致其性状鉴定过程中不仅鉴定周期较长,而且其农艺性状易受环境条件和栽培管理措施的影响。此外,传统的鉴定方法对于一些性状差异较小、亲缘关系较近品种的鉴定十分困难,给橡胶树种质资源和品种鉴定带来了诸多不便。因此,亟需准确的分子标记方法来对橡胶树种质资源和品种进行鉴定。
分子标记分析方法均以DNA的多态性为基础,如RFLP、RAPD、AFLP、ISSR、SSR、以及SNP标记等,它们均能产生具有高度个体特异性的DNA指纹图谱。早期RFLP、RAPD、AFLP分子标记技术曾被应用到多个物种的DNA指纹图谱建库上,但以上几个标记技术在使用中经常出现稳定性差、多态性低、以及数据整合困难等问题未能被广泛应用。因此,国际新品种保护联盟建议SSR和SNP作为首选的DNA指纹标记技术。由于SNP标记具有覆盖全基因组、高通量、位点特异、共显性遗传、误检率低、开发和检测成本急剧降低、数据易整合等优点,将成为未来基因型鉴定的主要标记类型。
随着二代测序技术的发展,测序通量呈现指数增长趋势,而测序成本则急剧降低,为全基因组水平SNP标记的检测提供了强有力的技术支持。每个物种上存在丰富的SNP,如何根据不同鉴定目标,制定SNP位点筛选原则、位点数目、位点组合、判定阈值等成为筛选SNP构建指纹图谱的关键。本发明基于橡胶树重测序获得的SNP,通过SNP测序质量的控制、SNP位点质量控制、SNP筛选的判定阈值设定等,以获得标准化的SNP构建指纹图谱的流程,用来建立橡胶树的分子身份证。目前,还没有建立起完整的橡胶树品种指纹图谱库从而鉴别品种的报道。
发明内容
本发明的目的在于针对现有技术的不足,提供一种橡胶树品种DNA指纹库的构建方法。以解决现有技术中使用RAPD、ISSR等共显性DNA标记,重复性差、多态性低的问题;本发明的另一目的是提供橡胶树核基因组SNP分子标记的多态性引物在橡胶树品种鉴定、遗传结构和资源遗传多样性分析中的应用,从而为从分子水平鉴定橡胶树种质资源和栽培品种提供依据。
本发明的第一个方面是提供一种用于构建橡胶树DNA指纹图谱的SNP分子标记组合,所述SNP分子标记组合包括LG01_54721919、和/或LG01_96036745、和/或LG02_214754、和/或LG02_8025843、和/或LG02_57849162、和/或LG03_40084932、和/或LG03_57340517、和/或LG03_96006922、和/或LG03_104230492、和/或LG04_224562、和/或LG04_16174898、和/或LG04_33465133、和/或LG04_71146652、和/或LG04_80306944、和/或LG04_96013598、和/或LG05_56475、和/或LG05_16004126、和/或LG05_88010730、和/或LG06_32377524、和/或LG07_8072524、和/或LG07_32411839、和/或LG07_57540924、和/或LG07_72003724、和/或LG07_80004402、和/或LG08_8000585、和/或LG08_28146509、和/或LG08_35232681、和/或LG08_48608369、和/或LG09_8012416、和/或LG09_16001432、和/或LG09_46132314、和/或LG09_50708986、和/或LG09_64555870、和/或LG10_34723841、和/或LG11_54415、和/或LG11_8139610、和/或LG11_34802586、和/或LG12_284283、和/或LG12_32322540、和/或LG12_40522222、和/或LG13_32072502、和/或LG13_40000353、和/或LG14_605466、和/或LG14_33209388、和/或LG14_56176655、和/或LG15_32001166、和/或LG15_24017141、和/或LG17_24096219、和/或LG17_32001525、和/或LG18_10766;
上表中,“Sequence”为SNP分子标记的核苷酸序列;“[]”标注的为SNP分子标记位点位置,其中的碱基为该位点的多态性。
应当理解的是,在实际应用时,本领域技术人员可根据实际情况来选择合适所述的SNP分子标记进行应用。比如,实际已知待测橡胶树为海垦1或RRIM623,那么可用于鉴定的SNP分子标记可选择LG01_96036745、LG03_104230492、LG04_16174898、LG04_33465133、LG04_71146652、LG04_96013598、LG07_57540924、LG07_72003724、LG09_50708986、LG09_64555870、LG11_54415、LG11_34802586、LG14_33209388、LG18_10766等(参照下文表5)中的一种即可,也可以选择两种或两种以上进行多重验证鉴定。又比如,实际已知待测橡胶树为海垦1、RRIM623或大丰95,也可以只采用一种SNP分子标记即可,如LG03_104230492、LG04_33465133、LG11_34802586、LG15_32001166等(参照下文表5),当然也可以采用两种以上的SNP分子标记。当已知待测橡胶树为四种或四种以上橡胶树中的一种时,本领域技术人员也可以参照下文中的表5,选择适当的SNP分子标记来进行鉴定。当待测橡胶树完全未知时,本领域技术人员也可以将上述SNP分子标记分批次来进行鉴定。
优选地,所述SNP分子标记组合包括LG01_54721919、LG01_96036745、LG02_214754、LG02_8025843、LG02_57849162、LG03_40084932、LG03_57340517、LG03_96006922、LG03_104230492、LG04_224562、LG04_16174898、LG04_33465133、LG04_71146652、LG04_80306944、LG04_96013598、LG05_56475、LG05_16004126、LG05_88010730、LG06_32377524、LG07_8072524、LG07_32411839、LG07_57540924、LG07_72003724、LG07_80004402、LG08_8000585、LG08_28146509、LG08_35232681、LG08_48608369、LG09_8012416、LG09_16001432、LG09_46132314、LG09_50708986、LG09_64555870、LG10_34723841、LG11_54415、LG11_8139610、LG11_34802586、LG12_284283、LG12_32322540、LG12_40522222、LG13_32072502、LG13_40000353、LG14_605466、LG14_33209388、LG14_56176655、LG15_32001166、LG15_24017141、LG17_24096219、LG17_32001525和LG18_10766中的至少两种。
优选地,所述SNP分子标记组合至少包括LG03_104230492、LG02_8025843、LG01_54721919、LG15_32001166、LG07_72003724、LG05_88010730和LG02_214754。
优选地,所述SNP分子标记组合包括LG01_54721919、LG01_96036745、LG02_214754、LG02_8025843、LG02_57849162、LG03_40084932、LG03_57340517、LG03_96006922、LG03_104230492、LG04_224562、LG04_16174898、LG04_33465133、LG04_71146652、LG04_80306944、LG04_96013598、LG05_56475、LG05_16004126、LG05_88010730、LG06_32377524、LG07_8072524、LG07_32411839、LG07_57540924、LG07_72003724、LG07_80004402、LG08_8000585、LG08_28146509、LG08_35232681、LG08_48608369、LG09_8012416、LG09_16001432、LG09_46132314、LG09_50708986、LG09_64555870、LG10_34723841、LG11_54415、LG11_8139610、LG11_34802586、LG12_284283、LG12_32322540、LG12_40522222、LG13_32072502、LG13_40000353、LG14_605466、LG14_33209388、LG14_56176655、LG15_32001166、LG15_24017141、LG17_24096219、LG17_32001525和LG18_10766。
本发明的第二个方面是提供用于扩增如本发明第一个方面所述的SNP分子标记组合的引物组。
优选地,各个SNP分子标记的引物对如下文中表3所示。
本发明的第三个方面是提供含有本发明第二个方面所述的引物组的试剂盒或基因芯片。
本发明的第四个方面是提供本发明第一个方面所述的SNP分子标记组合、或者本发明第二个方面所述的引物组、或者本发明第三个方面所述的试剂盒或基因芯片在橡胶树品种群体遗传结构、和/或橡胶树资源遗传多样性分析、和/或橡胶树品种鉴定、和/或构建橡胶树DNA指纹图谱中的应用。
本发明的第五个方面是提供一种橡胶树品种DNA指纹库的构建方法:提取各个橡胶树品种的总DNA;采用本发明第二个方面所述的SNP分子标记的引物组进行扩增;利用本发明第一个方面所述的SNP分子标记组合进行SNP位点多态性检测,根据SNP位点的检测结果对各个橡胶树品种进行基因分型,从而构建橡胶树品种DNA指纹库;其中,所述橡胶树品种为下文表2所述的橡胶树品种。
优选地,应用SNaPshot方法进行SNP位点多态性检测,进行橡胶树品种鉴定、群体遗传结构和遗传多样性分析。
优选地,所述SNP分子标记组合至少包括'LG03_104230492、LG02_8025843、LG01_54721919、LG15_32001166、LG07_72003724、LG05_88010730和LG02_214754。
优选地,所述SNP分子标记组合包括LG01_54721919、LG01_96036745、LG02_214754、LG02_8025843、LG02_57849162、LG03_40084932、LG03_57340517、LG03_96006922、LG03_104230492、LG04_224562、LG04_16174898、LG04_33465133、LG04_71146652、LG04_80306944、LG04_96013598、LG05_56475、LG05_16004126、LG05_88010730、LG06_32377524、LG07_8072524、LG07_32411839、LG07_57540924、LG07_72003724、LG07_80004402、LG08_8000585、LG08_28146509、LG08_35232681、LG08_48608369、LG09_8012416、LG09_16001432、LG09_46132314、LG09_50708986、LG09_64555870、LG10_34723841、LG11_54415、LG11_8139610、LG11_34802586、LG12_284283、LG12_32322540、LG12_40522222、LG13_32072502、LG13_40000353、LG14_605466、LG14_33209388、LG14_56176655、LG15_32001166、LG15_24017141、LG17_24096219、LG17_32001525和LG18_10766。
本发明的第六个方面是提供采用本发明第五个方面所述的构建方法所构建的橡胶树品种DNA指纹库。
本发明的第七个方面是提供一种橡胶树品种的鉴定方法,其特征在于,采用本发明第二个方面所述的引物组对待测样品的总DNA进行PCR扩增,然后进行SNP位点多态性检测,根据SNP位点的检测结果确定SNP分子标记的基因分型,再与本发明第六个方面所述的橡胶树品种DNA指纹库中的基因分型进行比对。
优选地,应用SNaPshot方法进行SNP位点多态性检测。
本发明的有益效果:
本发明筛选的50个SNP分子标记特异性、灵敏度和分辨力高,该SNP分子标记不受环境条件影响,且检测结果准确、重复性和稳定性好,能够全面揭示橡胶树遗传多样性真实水平利用;利用该套SNP标记组合建立DNA指纹图谱库,可以对橡胶树品种进行聚类分析和品种鉴定,使得检测结果更精准、高效,为橡胶树资源保护利用提供理论依据,首次有效区分出橡胶树66个品种,本发明对育成品种的区分率可达100%。利用本发明的单核苷酸标记位点进行检测具有简单、快速、成本低的优点,能够实现生产中大规模的应用。
附图说明
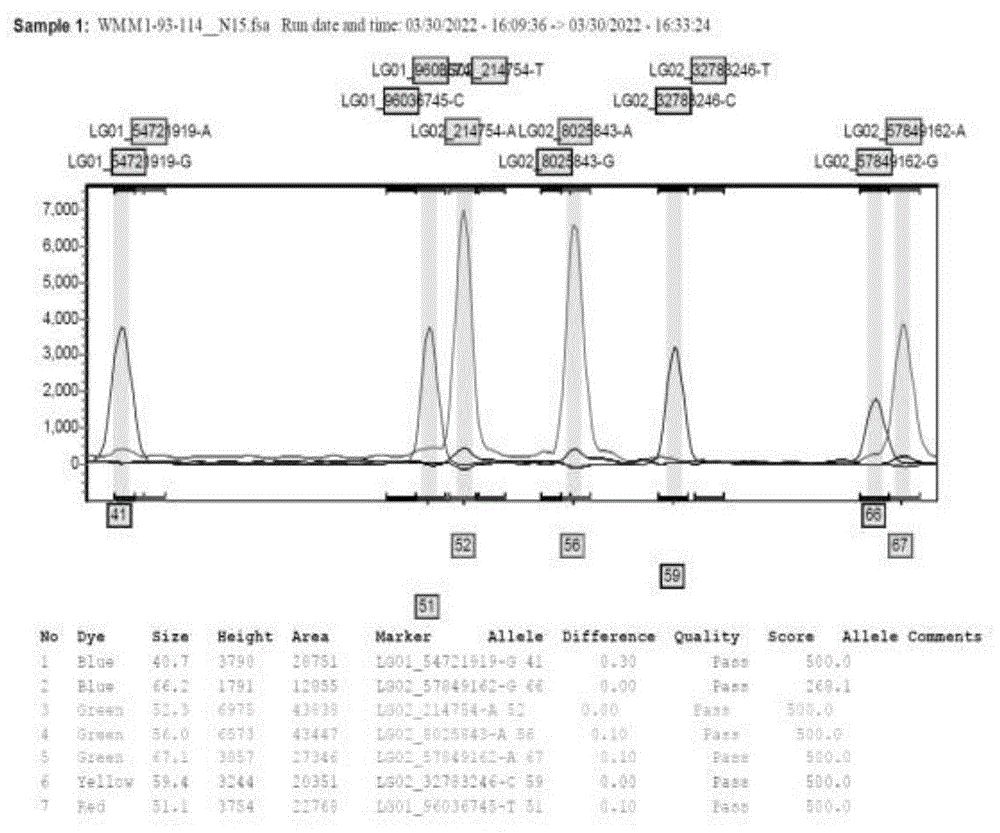
图1为本发明实施例2中部分样品扩增产物的分型检测结果示例图;
图2为本发明实施例2基于SNP的橡胶树品种的UPGAM法聚类分析图
图3为本发明实施例2核心SNP对橡胶树品种的识别率。
具体实施方式
下面参照附图,结合具体的实施例对本发明作进一步的说明,以更好地理解本发明。实施例中未注明具体技术或条件者,按照本领域内的文献所描述的技术或条件或者按照产品说明书进行。所用试剂或仪器未注明生产厂商者,均为可以通过市购获得的常规产品。
实施例1:橡胶树SNP位点的筛选与确定
以9个橡胶树品种PR107、GT1、IAN873、RRIM600、93-114、云研77-4、热研7-33-97、热研8-79、热垦628为样本,进行基因组重测序。具体操作参照“Du,H.,Yang,J.,Chen,B.etal.Target sequencing reveals genetic diversity,population structure,core-SNPmarkers,and fruit shape-associated lociin pepper varieties.BMC Plant Biol 19,578(2019)”。
基于橡胶树基因组重测序数据,与橡胶树参考基因组(https://www.ncbi.nlm.nih.gov/genome/?term=rubber+tree)比对,挖掘SNP位点,根据以下原则进行SNP标记候选位点的筛选分析:(a)最小等位基因频率MAF>0.4;(b)缺失率<0.2;(c)杂合度<0.2;(d)上下游200bp无序列变异;(e)两等位基因位点;(f)染色体分布情况清晰;(g)在基因组上均匀分布。
从上述9个代表品种的基因组重测序数据中,最终筛选出50个高质量、单拷贝、高多态性的SNP标记,用于不同来源的橡胶树品种鉴定、群体遗传结构和橡胶树资源遗传多样性分析。50个SNP分子标记的序列信息如表1所示。
表1:
/>
/>
/>
/>
/>
上表中,“Sequence”为SNP分子标记所在的核苷酸序列;“[]”标注的为SNP分子标记位点位置,其中的碱基为该位点的多态性。
实施例2:橡胶树SNP标记的测序验证与应用
1供试材料
供试材料来源于中国热带农业科学院橡胶研究所国家橡胶树种质资源圃,选取66个橡胶树品种(表2),每个品种分别采集3株不同植株的叶片,经液氮速冻于-80℃冰箱保存,用于提取橡胶树叶片DNA。
表2 66个橡胶树品种的信息
/>
2橡胶树总DNA提取与检测
参照CTAB法提取所述各个橡胶树品种的叶片总DNA,后用琼脂糖凝胶电泳和分光光度计检测各品种的总DNA质量,以确保所提取的各个橡胶树品种总DNA完整性好,且A260/A280比值在1.6-2.0之间。
3SNP引物设计与筛选
根据上述筛选的50个SNP分子标记位点序列信息,使用引物设计软件PrimerPlex2设计PCR反应引物,设计了167对SNP引物,经筛选后共得到50对特异性较好且多态性较高的SNP扩增引物,具体见表3。并使用引物设计软件Primer3plus设计单碱基延伸引物,具体见表4。
表3扩增引物
表4延伸引物
/>
/>
表中,延伸引物中,大写是特异序列,与SNP区域进行结合;小写是加长序列,保证一个反应中的多个分型能够准确判读;方向代表测序方向。
4分型实验
4.1PCR扩增反应
以前述提取获得的66个橡胶树品种的基因组DNA为模版,利用表3的引物,扩增出含SNP位点的核苷酸片段:在1.5mLEP管中配置PCR master mix,并振荡低速离心。选用8道移液器,在384孔板的每个加样孔中加入9μl PCR master mix,最后加入1μl模板DNA(20ng/uL)混匀,小心盖上384孔封板膜,并压牢每个孔,防止PCR程序时出现蒸发。1000rpm离心1min。
PCR扩增体系为:2×Taq PCR Master Mix 5.0μl、基因组DNA(~20ng)1.0μl、上下游引物的混合物(浓度10pmol/uL,上下游引物的比例为1:1)1.0μl、dd H2O 3.0μl,体系的总体积为10μL。
PCR扩增程序为:94℃预变性5min;94℃变性20s,60℃退火30s,72℃延伸30s,共35个循环;72℃终延伸3min;16℃1min;4℃保存。
4.2产物碱性磷酸酶处理
在PCR反应结束后,将PCR产物用SAP(shrimp alkaline phosphatase,虾碱性磷酸酶)处理,以去除体系中游离的dNTPs。在新1.5mLEP管中配制碱性磷酸酶处理反应液,SAPMix反应组分如下:ddH
4.3单碱基延伸反应
在碱性磷酸酶处理结束后,进行单碱基延伸反应,反应体系为:SNaPshot Mix 0.5μl,Pooled PCR Products3.0μl,Pooled Primers(表4)1.0μl,ddH
4.4上机
取2uL SNaPshot反应产物加入到8μl含有0.8%LIZ120的去离子甲酰胺中,95℃变性5分钟,然后放-20℃骤冷,然后上3730XL测序。
5引物优化调整
从ABI 3730xl仪器上导出.fsa格式原始数据,按检测反应进行分类归档后,分别导入到GeneMarker分析软件中,进行基因型数据的读取。根据各位点信号值与出峰情况,对各位点引物比例进行调整,保证各位点信号值与出峰情况基本一致。
6结果质控与分析
对正式实验结果进行分析,按样本、位点情况进行统计,得到样本各位点的分型结果,导出各反应结果的PDF图。部分样品扩增产物的分型检测结果示例如图1所示。根据结果信息进行参数分析,得出详细的分型结果,66个橡胶树品种的50个SNP位点的基因型如表5-9所示。
表5 66个橡胶树品种在50个SNP位点的基因型(1)
/>
表666个橡胶树品种在50个SNP位点的基因型(2)
/>
表766个橡胶树品种在50个SNP位点的基因型(3)
/>
表8 66个橡胶树品种在50个SNP位点的基因型(4)
/>
/>
表9 66个橡胶树品种在50个SNP位点的基因型(5)
/>
根据样本间Nei氏遗传距离,使用phangorn R包进行非加权组平均法构建系统发育树。对66个橡胶树品种进行UPGAM法聚类分析,结果显示一些亲缘关系较近的品系聚为一簇(如图3所示),如高产品系热研879、热研8-333、热研87-4-26聚为一簇;抗寒品系云研77-2、云研77-4、GT1聚为一簇,表明50个SNP标记能实现对66份橡胶树品种的区分。
本发明首次区分了66个橡胶树品种,对育成品种的区分率可达100%。这说明,利用本发明的单核苷酸标记位点进行检测具有简单、快速、成本低的优点,能够实现生产中大规模的应用。
为了进一步筛选核心SNP分子标记位点,从上述50个SNP分子标记的位点中选择不同数量、不同组合的位点对66个品种进行区分,统计不同数量、不同组合的位点对于66个橡胶树品种的区分率,以位点数量为横轴,以该位点数量下,不同位点组合的最大的区分率为纵轴作图,如图3所示。从图3可以看出,位点数量为7的时候,对66个品种的区分率可以达到100%。达到100%区分率的SNP分子标记组合为:LG03_104230492、LG02_8025843、LG01_54721919、LG15_32001166、LG07_72003724、LG05_88010730和LG02_214754。
以上对本发明的具体实施例进行了详细描述,但其只是作为范例,本发明并不限制于以上描述的具体实施例。对于本领域技术人员而言,任何对本发明进行的等同修改和替代也都在本发明的范畴之中。因此,在不脱离本发明的精神和范围下所作的均等变换和修改,都应涵盖在本发明的范围内。
