一种利用胚乳特异性自杀获得非转基因的定向基因突变植株的方法及其应用
文献发布时间:2023-06-19 11:16:08
技术领域
本发明属于植物基因工程领域,具体涉及一种利用胚乳特异性自杀获得非转基因的定向基因突变植株的方法及其应用。
背景技术
近年来,随着生物技术的发展,基因组定点突变技术被逐步建立起来,其中主要依赖于一些序列特异核酸酶(Sequence-specific nucleases,SSNs)的功能解析及应用。其中主要包括三种SSN:锌指核酸酶(Zinc finger nucleases,ZFN)、转录激活样效应因子核酸酶(Transcription activator-like effector nucleases,TALEN)和成簇规律间隔短回文重复序列及其相关系统(Clustered regularly interspaced short palindromicrepeats/CRISPR associated proteins,CRISPR/Cas system)。这几种SSN的共同特征是能够切割特定的DNA序列,诱导DNA双链断裂(Double-stranded breaks,DSBs)。随后生物体内的自我修复机制会自我启动,将断裂的DNA修复,根据修复方式的不同可以分为非同源重组连接(Nonhomologous end joining,NHEJ)和同源重组修复(homology-directed repair,HDR)(Symington LS,Gautier J.Double-strand break end resection and repairpathway choice.Annu Rev Genet,2011,45:247-271)。NHEJ修复方式主要是通过直接将断裂位置的染色体连接起来,但是这个连接修复不能保证非常精确的修复,使得在断裂位置出现核苷酸的缺失或者插入从而导致基因发生突变。HDR修复主要发生在有同源序列存在的情况下,生物体在修复DSBs的时候可以以同源序列为模板来完成断裂位置的修复,这种方式因为有模板的存在,会产生精确的修复,如果在模板中设计一些人为的突变则会将这些突变精确的引入生物体的基因组中。
这几种基于SSN的的基因定点突变技术中,因为CRISPR/CAS9技术操作简单,成本低廉,仅仅通过改变一小段RNA序列即可达到识别不同靶位点,所以CRISPR/CAS9技术被广泛应用于几乎任何可转化的生物体中高效地靶向编辑DNA,为农业改良提供前所未有的工具。自第一次在真核生物中报道CRISPR/Cas9-meidated编辑事件(Cong L,Ran FA,Cox D,Lin S,Barretto R,Habib N,Hsu PD,Wu X,Jiang W,Marraffini LA,Zhang F.Multiplexgenome engineering using CRISPR/Cas systems.Science,2013,339(6121):819-823)以来,CRISPR基因编辑技术在植物中也得到了广泛的应用。从编辑的植物中及时清除转基因片段是评估CRISPR编辑的植物的遗传性和表型稳定性的关键步骤,对于作物改良至关重要。首先,在育种领域,如果在农作物品种中存在转基因片段,那么从政府管理机构获得商业种植的批准就非常困难,转基因的清除是CRISPR编辑作物获取监管批准进行商业应用的先决条件。其次,在研究领域,转基因片段上的基因编辑元件的存在大大增加了脱靶效应的风险,使得表型稳定性成为一个问题。此外,对于遗传学研究来说,转基因片段上的基因编辑元件的存在使得难以确定检测到的突变是从上一代遗传还是由当代新产生的。
植物的有性生殖需要经历孢子体和配子体的世代交替,孢子体(2n)的孢子母细胞(2n)经过减数分裂产生单倍体的配子体(n);雌雄配子体通过受精作用结合产生合子(2n),合子继续生长发育长成孢子体(2n)。转基因植物的孢子母细胞会经过减数分裂产生携带有转基因构建体的配子体和不带有转基因构建体的配子体,雌雄胚子体随着受精作用会自由组合产生合子。根据孟德尔的自由组合定律,当转基因构建体的拷贝数为1时,产生的合子中75%(理论值)的植株是带有转基因构建体,当转基因构建体的拷贝数大于1时,产生的合子中带有转基因构建体的数目会大于75%,并且随着拷贝数的增加这个比例还会继续增加。以上情况增加了获取不含有转基因构建体的难度。目前,从基因编辑体系中获得不带有转基因片段的方法主要有如下几种:
1)使用多代自交或者回交,通过遗传分离等传统方法鉴定无转基因,这种方法需要对植物进行多代种植或者杂交,非常费力和费时。2)使用在种子中特异性表达的mCherry荧光报告基因或者颜色报告基因作为转基因片段存在的标记。这种方法目前主要在拟南芥中被使用,尽管荧光标记辅助选择无转基因的植物减少75%左右的筛选鉴定无转基因突变植株的工作量,但该策略仍然比较费时费力(Gao X,Chen J,Dai X,Zhang D,Zhao Y.AnEffective Strategy for Reliably Isolating Heritable and Cas9-Free ArabidopsisMutants Generated by CRISPR/Cas9-Mediated Genome Editing.Plant Physiol,2016,171(3):1794-1800;He Y,Zhu M,Wu J,Ouyang L,Wang R,Sun H,Yan L,Wang L,Xu M,ZhanH,Zhao Y.Repurposing of Anthocyanin Biosynthesis for Plant Transformation andGenome Editing.Front Genome Ed,2020,2(19))。此外,该方法不会在T2代中富集或增加无转基因植物的比例。3)使用化学筛选标记基因作为转基因片段存在的标志,通过特殊化学物质筛选获取无转基因的基因编辑植株。比如将CRISPR构建体与靶向除草剂抗性P450酶的RNA干扰元件偶联。使得可以通过特定的除草剂筛选不含转基因片段的植物(Lu HP,LiuSM,Xu SL,Chen WY,Zhou X,Tan YY,Huang JZ,Shu QY.CRISPR-S:an activeinterference element for a rapid and inexpensive selection of genome-edited,transgene-free rice plants.Plant Biotechnol J,2017,15(11):1371-1373)。但是,这种策略仍然没有丰富无转基因植物,同时需要对后代进行种植和筛选,增加了劳动力的投入。4)使用瞬时转化的方法直接将Cas9蛋白和gRNA导入到植物细胞内,避免DNA的整合。比如使用基因枪、PEG、农杆菌、纳米材料介导的瞬时转化(He Y,Zhao Y.Technologicalbreakthroughs in generating transgene-free and genetically stable CRISPR-edited plants.aBIOTECH,2020,1(1):88-96)。由于瞬时转化无法进行有效筛选,所以许多未发生突变的植株将掺杂到分化出的幼苗中。Liang的研究显示仅有4%左右的植株发生了单个基因的编辑(Liang Z,Chen K,Li T,Zhang Y,Wang Y,Zhao Q,Liu J,Zhang H,Liu C,Ran Y,Gao C.Efficient DNA-free genome editing of bread wheat using CRISPR/Cas9 ribonucleoprotein complexes.Nat Commun,2017,8:14261)。虽说后代可以通过PCR的鉴定来筛选有突变的植株,但是这也是一项劳动强度比较大的工作,尤其是对于系统性研究多个基因时,获取可用材料的时间和人力成本将成为研发的限速因素。5)使用TKC转基因自杀技术。该方法使用水稻早期胚胎发育基因REG2的启动子(Sun JL,Nakagawa H,Karita S,Ohmiya K,Hattori T.Rice embryo globulins:amino-terminal amino acidsequences,cDNA cloning and expression.Plant Cell Physiol,1996,37(5):612-620)驱动枯草芽孢杆菌核糖核酸酶BARNASE(Hartley RW.Barnase and barstar.Expressionof its cloned inhibitor permits expression of a cloned ribonuclease.J MolBiol,1988,202(4):913-915),同时将水稻的细胞质不育基因CMS2利用35S启动子驱动。BARNASE可以通过降解细胞中的RNA来杀死细胞(Lannenpaa M,Hassinen M,Ranki A,Holtta-Vuori M,Lemmetyinen J,Keinonen K,Sopanen T.Prevention of flowerdevelopment in birch and other plants using a BpFULL1::BARNASEconstruct.Plant Cell Rep,2005,24(2):69-78),而CMS2则可以通过阻碍花粉发育过程中线粒体的功能而导致花粉败育(Wang Z,Zou Y,Li X,Zhang Q,Chen L,Wu H,Su D,Chen Y,Guo J,Luo D,Long Y,Zhong Y,Liu YG.Cytoplasmic male sterility of rice withboro II cytoplasm is caused by a cytotoxic peptide and is restored by tworelated PPR motif genes via distinct modes of mRNA silencing.Plant Cell,2006,18(3):676-687)。在将两个表达盒与正常的CRISPR/Cas9基因编辑元件整合成一个连锁的载体。该技术通过特异的杀死携带有转基因片段的花粉和胚胎,使得转基因T0植株只能长出非转基因的种子,其中包括DNA被编辑的后代。但是该方法存在一定的局限性,不适用于使用幼胚转化的一些植物。
发明内容
本研究的目的在于开发一种特异性破坏胚乳的新型转基因自消除基因编辑系统,本技术方案将基因编辑元件与特异性杀死胚乳的元件组合成一个连锁的系统,将其命名为TEKE(Transgene Endosperm Killer Editing:转基因胚乳杀手基因编辑)系统。TEKE系统中的基因编辑元件可以行使基因编辑的功能,特异性杀死胚乳的元件可以破坏携带有转基因构建体的胚乳,达到转基因种子无法正常生长而死亡的效果。这样就能达到转基因植株自主消除自己的转基因后代,自主筛选出不带有转基因构建体的定点突变后代。该系统能够在基因编辑发生之后,种子生长阶段自主破坏携带有转基因构建体的胚乳,达到转基因种子无法正常生长而死亡的效果,为通过基因编辑培育无转基因植株提供了一种简单有效、省时省力的方法。TEKE系统将两个关键元件人为连锁在一个T-DNA上。1)CRISPR/Cas9基因编辑元件,包括Cas9蛋白表达盒和gRNA转录盒。2)胚乳特异性致死元件,使用胚乳特异启动子OsEP驱动毒蛋白BARNASE,REG2启动子非常特异地在种子的胚乳发育过程中发挥功能,将BARNASE基因置于水稻OsEP启动子的控制之下时,BARNASE毒蛋白不在愈伤组织或营养生长期间产生,只在种子的胚乳发育过程中产生,因此利用OsEP启动子驱动的BARNASE将确保含有转基因元件的种子胚乳被破坏,继而导致水稻种子发育出现缺陷。将OsEP-BARNASE导入带有Cas9基因的质粒pCUCas9中,从而完成TEKE系统的载体构建。
具体地,本发明通过以下技术方案实现:
申请人提供了一种方法,利用胚乳特异性自杀获得非转基因的定向基因突变植株,该方法用于创制定向基因突变的非转基因植株(简称TEKE系统),步骤如下所述:
a)构建用于对植物进行遗传转化的构建体,所述构建体含有第一核酸分子和第二核酸分子,所述第一核酸分子为基因编辑元件;所述第二核酸分子为胚乳致死或停止发育元件;
b)将需要处理的植株通过引入构建体外源核酸分子实施转基因。
进一步的,可应用于上述步骤a)和b)的植物为种子植物;包括禾本科、豆科、十字花科、菊科、茄科等可通过农杆菌浸染获取转基因材料的植物;优选水稻、玉米、高粱、大麦、燕麦、小麦、粟、二穗短柄草、大刍草、甘蔗、大豆、油菜、拟南芥、红花、番茄、烟草、苜蓿、马铃薯、甘薯、向日葵和棉花。
进一步的,所述第一核酸分子为能够编辑核酸的基因元件。
进一步的,所述述编辑核酸的基因元件选自任意一种基因编辑系统的基因元件构成的组。
进一步的,所述基因编辑系统优选:ZFN基因编辑系统、TALEN基因编辑系统、CRISPR/CAS9基因编辑系统或CRISPR/CPF1基因编辑系统。
进一步的,所述CRISPR/CAS9基因编辑系统的基因元件包括CAS9基因(CAS9基因的核苷酸序列如SEQ ID NO:1所示的核苷酸序列)和gRNA基因(gRNA基因的核心骨架的核苷酸序列如SEQ ID NO:2所示)。
进一步的,所述第二核酸分子为胚乳致死或停止发育的基因元件。
进一步的,所述第二核酸分子的核苷酸序列如SEQ ID NO:3所示的核苷酸序列。
具体实施步骤可以详见实施例。
本发明还提供上述方法在植物育种中的应用。
进一步的,所述植物为种子植物门所属植物。
序列表SEQ ID NO:1是CAS9基因的核苷酸序列,序列长度为4131bp。
序列表SEQ ID NO:2是gRNA基因的核心骨架核苷酸序列,序列长度为76bp。
序列表SEQ ID NO:3是第二核酸分子的核苷酸序列,序列长度为4522bp。
序列表SEQ ID NO:4是豌豆rbcS-E9基因终止子的核苷酸序列,序列长度为635bp。
序列表SEQ ID NO:5是BARNASE基因的核苷酸序列,序列长度为854bp。
序列表SEQ ID NO:6是OsEP启动子的核苷酸序列,序列长度为3018bp。
序列表SEQ ID NO:7是OsU6P DNA的核苷酸序列,序列长度为457bp。
本发明的有益效果体现在:
本发明的方法可以快速获得不含转基因片段的定点突变水稻,也可以用于快速高效构建聚合多种基因突变的非转基因植株。该方法有助于加快水稻基因的功能研究,同时便于进行复杂的多基因互作研究,此外有助于加速改造和聚合水稻中优良基因的育种进度。
本发明可以保证转基因当代高效基因编辑的基础上,使转基因植物结出的转基因种子发育出现缺陷,让转基因植物自主消除含有转基因构建体的子代。本发明极大地减少了分离无转基因片段的基因编辑植物所需的时间和劳力,极大地加速了获取无转基因突变体的进度,并且本发明可以防止由种子的飘散而引起的转基因漂移,为作物改良提供了非常有用的工具。
附图说明
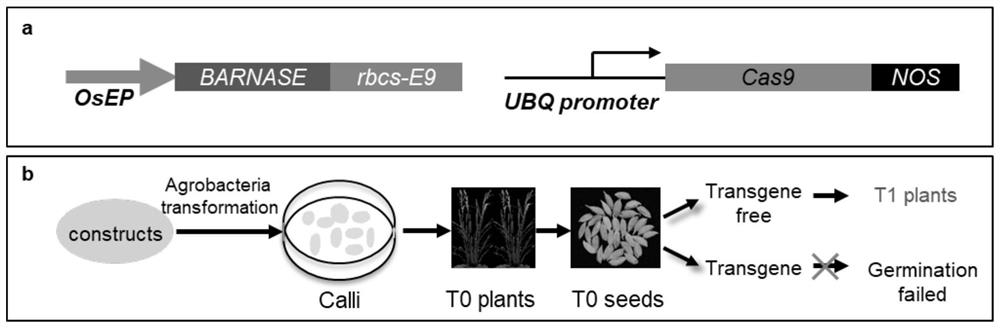
图1为本发明构建的TEKE质粒图。
图2为编辑靶基因后,自杀转基因介导的CRISPR/Cas9构建体的自我消除示意图。
附图标记说明a)TEKE(转基因杀手CRISPR)质粒的两个关键组成部分的示意图。NOS是指来自根癌农杆菌的胭脂氨酸合酶基因的终止子。OsEP是胚乳特异启动子,被用于驱动BARNASE基因,该基因编码一种有毒的酶来植物细胞。rbcs-E9终止子最初是从豌豆rbcS-E9基因中克隆。密码子优化的Cas9被置于玉米的泛素启动子UBQ的控制之下。b)TEKE介导的分离无转基因和CRISPR/Cas9编辑的水稻植物的流程图。
图3为TEKE-Gn1a产生的T1植物的转基因片段检测。
图4为由TEKE-Gn1a产生的T1植物中的突变和无转基因植株的分离模式示意图。
附图标记说明:a)Cas9切割所需的PAM站点“CCT”以浅色标记。WT指野生型植物水稻品种中花11(简称ZH11)。“-”是指删除一个碱基对。浅色和上标中的“a”和“t”分别表示插入“A”和“T”。大写的浅色“A”表示碱基发生替换。
具体实施方式
现以第一核酸分子以CRISPR/CAS9基因编辑系统的基因元件为例,说明本发明技术方案可行性验证基因以水稻Gn1a基因(GeneBank编号XM_015773930)为例。转基因方法以农杆菌介导的水稻稳定转化方法为例(水稻遗传转化现已为水稻转基因领域的普遍操作方法,其中详细的转化步骤及使用的各种培养基配方参见文献:Hiei Y,Ohta S,Komari T,Kumashiro T.Efficient transformation of rice(Oryza sativa L.)mediated byAgrobacterium and sequence analysis of the boundaries of the T-DNA.Plant J,1994,6(2):271-282)。
实施例1构建带有Cas蛋白表达盒的载体pCUCas9
本实施例将Cas蛋白表达盒,即能编码Cas蛋白的Cas9基因连入pCXUN载体获得载体pCUCas9。具体构建步骤:
(1)通过PCR扩增获得Cas9基因DNA。以来源于水稻密码子优化的Cas9基因,序列如SEQ ID NO.1所示,为模板DNA。以引物对:
UCas9-F:
5’-CCCGGGGGATCCCCAATACTATGGCCCCAAAGAAGAAGCGCAAGG-3’
UCas9-R:
5’-GAAATTCGGATCCCCAATACTTCAATCGCCGCCGAGTTGTGAGAGG-3’进行PCR扩增。
PCR反应体系:
PCR扩增程序:
PCR产物为:Cas9基因DNA,大小为4172bp,其中括SEQ ID NO.1的Cas9基因序列,由于PCR所用的引物加上了额外的用于Gibson连接的接头序列,所以PCR产物略大于SEQ IDNO.1所示Cas9基因的序列长度。
(2)构建出pCUCas9载体:使用Xcm I酶切pCXUN载体质粒,将本实施例步骤(1)获得的Cas9基因DNA的PCR产物通过Gibson连接的方式连入pCXUN载体(GenBank编号FJ905215)的Xcm I处,获得pCUCas9载体。
实施例2构建TEKE载体
pCUCas9将第二核酸分子添加到pCUCas9载体上来验证本发明的TEKE系统。其中第二核酸分子以OsEP-BARNASE基因表达盒(其核苷酸序列见SEQ ID NO:3)为例。
具体构建步骤如下:
1)扩增豌豆rbcS-E9基因的终止子,序列如SEQ ID NO.4所示,为模板,以引物对:full-rE9T-F
(5’-CTGCAGGAATTCGATATCATTTAAATAGAGCTTTCGTTCGTATCATCGGTT-3’)和full-rE9T-R
(5’-GTAAAACGACGGCCAGTGCCAAGCTTGTTGTCAATCAATTGGCAAGTCAT-3’)为引物扩增出rbcS-E9基因的终止子DNA,然后将其切胶回收后连入被Hind III酶切过的pCUCas9载体,得到pCXR9T。
2)扩增BARNASE基因DNA,序列如SEQ ID NO.5所示,为模板,以引物对:BAR-F
(5’-CTGCAGGAATTCGATATCATTTAAATATGGCACAGGTTATCAACACG-3’)和BAR-R
(5’-ACCGATGATACGAACGAAAGCTCTTTTAATTTTAAGAAAGTATGATGGTGATGTCGCAG-3’)为引物,扩增出BARNASE DNA;将该DNA切胶回收后连入被Swa I酶切过的pCXR9T载体,得到BARNASE-pCXR9T载体,
3)扩增OsEP启动子DNA,序列如SEQ ID NO.6所示,为模板,以引物对:OsEPF
(5’-CACTGCAGGAATTCGATATCATTTGTGGAAGTGTTACTACATGGCAGGTACAC-3’)和OsEPR
(5’-GTTGATAACCTGTGCCATATTTAAATGCTCTCTCAAGTCTCAATGACCTGTACT-3’)为引物扩增出OsEP启动子DNA,将其切胶回收后连入被Swa I酶切过的BARNASE-pCXR9T载体,得到OsEP-BARNASE-pCXR9T载体即TEKE质粒(图1)。
实施例3将能靶向目标DNA的gRNA转录盒构建到TEKE载体上。
为了检验本发明技术方案的有效性,申请人将Gn1a基因作为靶基因设计了特异性的gRNA,靶序列为5’-GGGCTCGGTCCACCTGAACCAGG-3’。将实施例2中所得到的终载体TEKE(见图1),用Pme I消化成线性DNA,以重叠PCR(为常规方法)扩增的方法将能转录gRNA的DNA引入。本实施例采用OsU6启动子作为gRNA转录单元的启动子,具体步骤如下:
以本实验室已经构建的带有OsU6P DNA(序列如SEQ ID NO.7所示)、gRNA骨架DNA(序列如SEQ ID NO.2所示)的载体gRNA为模板DNA,分别以OsU6PF
(5’-GTCGTTTCCCGCCTTCAGTTTATGTACAGCATTACGTAGG-3’)和Gn1a-U6R
(5’-GCAGGTACTCCGGGCGCGACAACCTGAGCCTCAGCGCAGC-3’)引物对,和OsU6TR
(5’-CTGTCAAACACTGATAGTTTAAACGATGGTGCTTACTGTTTAG-3’)和Gn1a-U6F
(5’-GTCGCGCCCGGAGTACCTGCGTTTTAGAGCTAGAAATAGCAAGTTA-3’)引物对为模板扩增出两种DNA,将上述两种DNA切胶回收后混合作为模板,以OsU6PF和OsU6TR为引物扩增出完整的gRNA转录单元DNA,然后将该DNA切胶回收后连入经过Pme I酶切过的TEKE载体,得到TEKE-Gn1a。
实施例4将TEKE-Gn1a载体转化水稻愈伤组织
将测序后的阳性质粒TEKE-Gn1a电转化到农杆菌(EHA105)中,并侵染水稻愈伤组织。转化品种为水稻“中花11”(又称ZH11,来自中国农业科学院作物科学研究所),(水稻遗传转化现已为水稻转基因领域的普遍操作方法,其中详细的转化步骤及使用的各种培养基配方参见文献:Hiei Y,Ohta S,Komari T,Kumashiro T.Efficient transformation ofrice(Oryza sativa L.)mediated by Agrobacterium and sequence analysis of theboundaries of the T-DNA.Plant J,1994,6(2):271-282)。
如图2所示,通过农杆菌介导的转化将TEKE质粒转化到水稻愈伤组织中。在愈伤生长和营养生长期间,BARNASE基因不被表达,并且可能由Cas9编辑靶基因。然而在产生子代时,含有Cas9的种子因为胚乳被BARNASE杀死而无法萌发。因此,只有来自T0植物的非转基因种子才能正常发育。
实施例5对TEKE-Gn1a转化的水稻T0材料进行阳性检测
使用常规CTAB法(He Y,Yan L,Ge C,Yao XF,Han X,Wang R,Xiong L,Jiang L,Liu CM,Zhao Y.PINOID Is Required for Formation of the Stigma and Style inRice.Plant Physiol,2019,180(2):926-936)对不同愈伤组织分别抽提DNA。
通过使用引物对:水稻基因组DNA质量检测F:5’-CTCAACCCCAAGGCTAACAG-3’(SEQID NO.28)+水稻基因组DNA质量检测R:5’-ACCTCAGGGCATCGGAAC-3’(SEQ ID NO.29)进行PCR扩增,PCR产物大小为526bp,用来检测此次样品DNA质量。
使用C9-F:5’-CCCTGCCTTCATACGCTATTT-3’(SEQ ID NO.30)+C9-R:5’-GACTTGAAGTTCGGGGTGAG-3’(SEQ ID NO.31)为引物对进行PCR扩增检测基因C9是否转化到水稻中,PCR产物大小为887bp。
PCR反应体系:
PCR扩增程序:
通过电泳检测发现两次转化事件的电泳检测胶图中下面一条带表示使用能扩增水稻基因组上一段特异DNA片段的引物进行PCR扩增的结果,扩增的结果反映提取的样品DNA质粒均较好。上面一条带表示使用能扩增基因C9的特异引物进行PCR扩增的结果。
转基因阳性统计结果见表1。
表1 T0转基因阳性检测结果
实施例6对TEKE-Gn1a转化的水稻T1材料进行阳性检测
从每个单独的T0植物收获种子,并分析来自4个独立的阳性T0植物的后代(T1代)。
具体步骤如下:
使用常规CTAB法(He Y,Yan L,Ge C,Yao XF,Han X,Wang R,Xiong L,Jiang L,Liu CM,Zhao Y.PINOID Is Required for Formation of the Stigma and Style inRice.Plant Physiol,2019,180(2):926-936)对不同愈伤组织分别抽提DNA。
通过使用引物对:水稻基因组DNA质量检测F:5’-CTCAACCCCAAGGCTAACAG-3’(SEQID NO.28)+水稻基因组DNA质量检测R:5’-ACCTCAGGGCATCGGAAC-3’(SEQ ID NO.29)进行PCR扩增,PCR产物大小为526bp,用来检测此次样品DNA质量。
使用C9-F:5’-CCCTGCCTTCATACGCTATTT-3’(SEQ ID NO.30)+C9-R:5’-GACTTGAAGTTCGGGGTGAG-3’(SEQ ID NO.31)为引物对进行PCR扩增检测基因C9是否转化到水稻中,PCR产物大小为887bp。
PCR反应体系:
PCR扩增程序:
通过电泳检测发现两次转化事件的电泳检测胶图中下面一条带表示使用能扩增水稻基因组上一段特异DNA片段的引物进行PCR扩增的结果,扩增的结果反映提取的样品DNA质粒均较好。上面一条带表示使用能扩增基因C9的特异引物进行PCR扩增的结果。
另外使用可以扩增T-DNA上另外一个位置的引物重复进行阳性检测。
BAR-377F:5’-AATTCAGACCGGATTCTTTACTCA-3’,BAR-377R:5’-GTCGCTGATACTTCTGATTTGTTC-3’;
扩增产物大小为377bp。
PCR反应体系:
PCR扩增程序:
使用Hyg-280F(5’-ACGGTGTCGTCCATCACAGTTTGCC-3’)和Hyg-280R(5’-TTCCGGAAGTGCTTGACATTGGGGA-3’)引物对普通CRISPR载体pCUCas9转化的T1代植株的基因组DNA进行PCR扩增来鉴定其阳性转基因植株。
随机选取4个T0代阳性植株(编号分别为402-8、402-10、402-12和402-13)收获种子后发芽获取T1代苗子后进行转基因片段检测。结果显示这4个T1家系中除402-8的14株植株中有4株苗子含有转基因片段之外,其它51株苗子均为转基因阴性植株。TEKE的T1代植株检测结果见图3。转基因阳性统计结果见表2。
表2 T1转基因阳性检测结果
由表2,当使用常规的CRISPR/Cas9构建体时,至少75%的T1代转基因植物具有CRISPR/Cas9构建体;当时用TEKE质粒的技术方案时,来自3个独立T0代转基因植物中所有T1代转基因植物(总共55株)不含有CRISPR构建体,有一个编号的T1植株中含有28.6%的转基因植株,也远低于常规方法的75%的。表明本发明的TEKE质粒技术方案在消除转基因方面非常有效。
实施例7对TEKE-Gn1a转化的水稻T1材料进行靶向突变检测
使用Gn1a-GT1(5’-AGCTGCCTTCCATCGTCAGCACACA-3’)和Gn1a-GT2
(5’-GCGTGCGTAAGCGTAAGCCCCAGG-3’)作为基因型检测引物,分别对4个独立的T1代家系中所有植株进行PCR扩增。然后,对PCR产物测序,通过Dsdecode网站(http://dsdecode.scgene.com/)对测序结果进行分析,鉴别出各个T1代植株的突变形式。
通过对4个独立的T0代转基因后代的55个T1代植物进行测序后发现:所有的转基因植物都在目标位点含有突变,突变效率为100%,具体突变形式和突变效率见图4。表明本发明的TEKE系统既能高效率自主清除转基因T1代植株转基因构建体又能保证后代发生高效的定点突变。本发明可大大减少分离无转基因定点DNA编辑的水稻所需的时间和人力,是对我们前期开发的第一版TKE技术的有意义的升级,对一些依赖幼胚转化的作物提供了潜在可用的高效清除转基因元件的基因编辑工具。
序列表
<110> 南京农业大学
<120> 一种利用胚乳特异性自杀获得非转基因的定向基因突变植株的方法及其应用
<160> 7
<170> SIPOSequenceListing 1.0
<210> 1
<211> 4131
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 1
atggccccaa agaagaagcg caaggtcgac aagaagtact ccatcggcct cgacatcggc 60
accaattctg ttggctgggc cgtgatcacc gacgagtaca aggtgccgtc caagaagttc 120
aaggtcctcg gcaacaccga ccgccactcc atcaagaaga atctcatcgg cgccctgctg 180
ttcgactctg gcgagacagc cgaggctaca aggctcaaga ggaccgctag acgcaggtac 240
accaggcgca agaaccgcat ctgctacctc caagagatct tctccaacga gatggccaag 300
gtggacgaca gcttcttcca caggctcgag gagagcttcc tcgtcgagga ggacaagaag 360
cacgagcgcc atccgatctt cggcaacatc gtggatgagg tggcctacca cgagaagtac 420
ccgaccatct accacctccg caagaagctc gtcgactcca ccgataaggc cgacctcagg 480
ctcatctacc tcgccctcgc ccacatgatc aagttcaggg gccacttcct catcgagggc 540
gacctcaacc cggacaactc cgatgtggac aagctgttca tccagctcgt gcagacctac 600
aaccagctgt tcgaggagaa cccgatcaac gcctctggcg ttgacgccaa ggctattctc 660
tctgccaggc tctctaagtc ccgcaggctc gagaatctga tcgcccaact tccgggcgag 720
aagaagaatg gcctcttcgg caacctgatc gccctctctc ttggcctcac cccgaacttc 780
aagtccaact tcgacctcgc cgaggacgcc aagctccagc tttccaagga cacctacgac 840
gacgacctcg acaatctcct cgcccagatt ggcgatcagt acgccgatct gttcctcgcc 900
gccaagaatc tctccgacgc catcctcctc agcgacatcc tcagggtgaa caccgagatc 960
accaaggccc cactctccgc ctccatgatc aagaggtacg acgagcacca ccaggacctc 1020
acactcctca aggccctcgt gagacagcag ctcccagaga agtacaagga gatcttcttc 1080
gaccagtcca agaacggcta cgccggctac atcgatggcg gcgcttctca agaggagttc 1140
tacaagttca tcaagccgat cctcgagaag atggacggca ccgaggagct gctcgtgaag 1200
ctcaatagag aggacctcct ccgcaagcag cgcaccttcg ataatggctc catcccgcac 1260
cagatccacc tcggcgagct tcatgctatc ctccgcaggc aagaggactt ctacccgttc 1320
ctcaaggaca accgcgagaa gattgagaag atcctcacct tccgcatccc gtactacgtg 1380
ggcccgctcg ccaggggcaa ctccaggttc gcctggatga ccagaaagtc cgaggagaca 1440
atcaccccct ggaacttcga ggaggtggtg gataagggcg cctctgccca gtctttcatc 1500
gagcgcatga ccaacttcga caagaacctc ccgaacgaga aggtgctccc gaagcactca 1560
ctcctctacg agtacttcac cgtgtacaac gagctgacca aggtgaagta cgtgaccgag 1620
gggatgagga agccagcttt ccttagcggc gagcaaaaga aggccatcgt cgacctgctg 1680
ttcaagacca accgcaaggt gaccgtgaag cagctcaagg aggactactt caagaaaatc 1740
gagtgcttcg actccgtcga gatctccggc gtcgaggata ggttcaatgc ctccctcggg 1800
acctaccacg acctcctcaa gattatcaag gacaaggact tcctcgacaa cgaggagaac 1860
gaggacatcc tcgaggacat cgtgctcacc ctcaccctct tcgaggaccg cgagatgatc 1920
gaggagcgcc tcaagacata cgcccacctc ttcgacgaca aggtgatgaa gcagctgaag 1980
cgcaggcgct ataccggctg gggcaggctc tctaggaagc tcatcaacgg catccgcgac 2040
aagcagtccg gcaagacgat cctcgacttc ctcaagtccg acggcttcgc caaccgcaac 2100
ttcatgcagc tcatccacga cgactccctc accttcaagg aggacatcca aaaggcccag 2160
gtgtccggcc aaggcgattc cctccatgag catatcgcca atctcgccgg ctccccggct 2220
atcaagaagg gcattctcca gaccgtgaag gtggtggacg agctggtgaa ggtgatgggc 2280
aggcacaagc cagagaacat cgtgatcgag atggcccgcg agaaccagac cacacagaag 2340
ggccaaaaga actcccgcga gcgcatgaag aggatcgagg agggcattaa ggagctgggc 2400
tcccagatcc tcaaggagca cccagtcgag aacacccagc tccagaacga gaagctctac 2460
ctctactacc tccagaacgg ccgcgacatg tacgtggacc aagagctgga catcaaccgc 2520
ctctccgact acgacgtgga ccatattgtg ccgcagtcct tcctgaagga cgactccatc 2580
gacaacaagg tgctcacccg ctccgacaag aacaggggca agtccgataa cgtgccgtcc 2640
gaagaggtcg tcaagaagat gaagaactac tggcgccagc tcctcaacgc caagctcatc 2700
acccagagga agttcgacaa cctcaccaag gccgagagag gcggcctttc cgagcttgat 2760
aaggccggct tcatcaagcg ccagctcgtc gagacacgcc agatcacaaa gcacgtggcc 2820
cagatcctcg actcccgcat gaacaccaag tacgacgaga acgacaagct catccgcgag 2880
gtgaaggtca tcaccctcaa gtccaagctc gtgtccgact tccgcaagga cttccagttc 2940
tacaaggtgc gcgagatcaa caactaccac cacgcccacg acgcctacct caatgccgtg 3000
gtgggcacag ccctcatcaa gaagtaccca aagctcgagt ccgagttcgt gtacggcgac 3060
tacaaggtgt acgacgtgcg caagatgatc gccaagtccg agcaagagat cggcaaggcg 3120
accgccaagt acttcttcta ctccaacatc atgaatttct tcaagaccga gatcacgctc 3180
gccaacggcg agattaggaa gaggccgctc atcgagacaa acggcgagac aggcgagatc 3240
gtgtgggaca agggcaggga tttcgccaca gtgcgcaagg tgctctccat gccgcaagtg 3300
aacatcgtga agaagaccga ggttcagacc ggcggcttct ccaaggagtc catcctccca 3360
aagcgcaact ccgacaagct gatcgcccgc aagaaggact gggacccgaa gaagtatggc 3420
ggcttcgatt ctccgaccgt ggcctactct gtgctcgtgg ttgccaaggt cgagaagggc 3480
aagagcaaga agctcaagtc cgtcaaggag ctgctgggca tcacgatcat ggagcgcagc 3540
agcttcgaga agaacccaat cgacttcctc gaggccaagg gctacaagga ggtgaagaag 3600
gacctcatca tcaagctccc gaagtacagc ctcttcgagc ttgagaacgg ccgcaagaga 3660
atgctcgcct ctgctggcga gcttcagaag ggcaacgagc ttgctctccc gtccaagtac 3720
gtgaacttcc tctacctcgc ctcccactac gagaagctca agggctcccc agaggacaac 3780
gagcaaaagc agctgttcgt cgagcagcac aagcactacc tcgacgagat catcgagcag 3840
atctccgagt tctccaagcg cgtgatcctc gccgatgcca acctcgataa ggtgctcagc 3900
gcctacaaca agcaccgcga taagccaatt cgcgagcagg ccgagaacat catccacctc 3960
ttcaccctca ccaacctcgg cgctccagcc gccttcaagt acttcgacac caccatcgac 4020
cgcaagcgct acacctctac caaggaggtt ctcgacgcca ccctcatcca ccagtctatc 4080
acaggcctct acgagacacg catcgacctc tcacaactcg gcggcgattg a 4131
<210> 2
<211> 76
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 2
gttttagagc tagaaatagc aagttaaaat aaggctagtc cgttatcaac ttgaaaaagt 60
ggcaccgagt cggtgc 76
<210> 3
<211> 4522
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 3
gtggaagtgt tactacatgg caggtacacg ttaccacatg tttatacagt aacactattc 60
ataaattgca gtaagacgca ttgttactac aaaacttatg gtttttacta cacacttaca 120
gtaacgtcgc gatgggattg tagtaacgca taacagatag tactaacaag tagtaatgct 180
atagtattgc tacttgtgga gagttaagat caaaaaaccc aagcttcgaa taacaatatc 240
tctcatgtga tgtatgattt tttaaaaccg tttgcaccat gatgttagaa aaaaatgctt 300
atcaaaaata gatccgtcat gactatattt caactaaatt tgaatgttgt tactgcacgt 360
aagacacagt gttactgcac gatggctgtc gctttactgc actgtttcgc aagacgagag 420
ctgagaagag gtgggtggtg ggggagttgg taggcagttt gaccgggttt tgaccgacgt 480
cgggggtgct ttgactgacc tttgactgag gtgggaggtg ggtttggccc ttgggaggga 540
gggggggata tagaatagct attctatacc cgggtgtaga atagtttgtg tatgtgtgtg 600
tgtgtgtata tatatatata tataaaatca aagtgtggga tattatattt catgtacaat 660
ctcataaaga gtctactaaa tatacttctc tctgaatata gtttgacaaa ttagttttgt 720
gtgttgctaa taactaatat aattttttac ttttagctcg attattactt aggaatttcc 780
gcagcaaagt gcggggaatc acctagttaa ttaaagaatt gagtcctcat aaaatacctt 840
ttcaatatgg cctgaatcat tgtttggttt tttggccttc tgttctattt ggtccaaaag 900
gccatctttc taagttggac tgggcccatg gtttcgttat gggccaaaaa caacaatagc 960
attttgtttt attctcccct tttgaaaaca gtatgcaggg gtgatgcttg ttcattataa 1020
gaggcaaagg gtagcatatg atatataagt ttataattta ttatagatga caacctcaaa 1080
ggtaactatc gttgacatat gtaaattatg ctacattaca aaagaaataa tgtgaacatt 1140
gacattggta catacgtgga cattatctat aattatagac tgacatctac attccctatg 1200
gttatcaatc ggaatgtcgg ctcttgtagc cttgtgaaca tacgtactct ctcgcactat 1260
atgtgcattg atgtgaatcc tccaatttgc tataggaatg tcgtaggttt gagtagccta 1320
tgtattcatt gtcagcaaat gcgcaccgtg aactctttac gtcagaggat ctacgtcagt 1380
ccgctagtac gctcatgaca tattccccct gtttttgaag atgcggttag ttactccttc 1440
tgtacactcc ataaaatgtg ctatcctaca atacattccc cgcttcaatg tatgagatat 1500
ttttccccat tttgttagaa aatacttaag caatatgttc caaatataat gttatattgg 1560
gttgatgttg tgttaaaatg gagtggttaa gcatgagcct tcatttccct cataactgtt 1620
tttttagaac tcctcgcaac tgttgcactc aacaatttca ttttccttcc tgttattcaa 1680
ttaaggttct ccttaaaaat aagtctttca tgaataattt agtattttga aggtactatt 1740
gaaagaatta tttggagatg tctaatgctt gaatgataag taatttggaa ggcatctact 1800
cctacaaagt tgaatcttct aaatactcct tcgcttttaa atatacggca ttgttaactt 1860
ttgaatatga caatatttaa caatttatct tgtacaaaga tttagtgcaa atatgcacaa 1920
tgcgtaaagt acctttaatg taaaacaagc cacaacaaaa caaatgatac ttagagcatt 1980
ttttaataag ccgattggtc gcatcatgtt taaaagtcaa ccatgtcata tataaaaaaa 2040
gggagatagt ttcatacata gctctaatat attttgaagg tactaaattt taaaaattgt 2100
tgttgcagat tgtaagtctt gcatataatt tattgtttct tttacttagc ttattagctc 2160
taattaatca caccctatct ctcctatatt tatataataa aaacatcccg acacaccaac 2220
gtacattttc tcatctattt ctaaggtatg gatggtattg tcatgtcaac ttatcttctt 2280
tctagttatg caccctcctt aattattgta ccaacttaaa ataagccata tattttgaaa 2340
ttatcatacc aactcacaat atgccatata ttttaaaatt attatgtcaa ctcacaatat 2400
gctatatatt ttgatattat cgtgccaact cacaatatac catatatttt gaaatgaagg 2460
tcatatttaa gatgtagagt aaatgtatga aatcctccca tccatatcct cattattaat 2520
tgcttccacc taccccaagc atttatagtt agagtagaac agttgtgctc ttgcaccgaa 2580
gggatgacat gtaggagcca tgcctacgtc ccctggccac ccaagcccgt tgaattaaca 2640
tggtccctca acagaaacat ccatttttga tggtgtgctt ccttcttggc gaccttactg 2700
gtaggtgaca tgctaacaaa aactgcacag tcatgcaagg caggggccgg tcaaaattga 2760
agtccctcct accttgattg taacgctaat actatggctc accgcctctg cggcaatctg 2820
gcagccactt ctcctcctct tggtgccaca gctgcacctt tgacgtggct ggctgcttgc 2880
atttgtccaa gccggcgcag ccctgatgta agtcctttgg tataaatact gcccaagcat 2940
agagcgagag agagccatca caatccaata caagttcaca acataagcat agtacaggtc 3000
attgagactt gagagagcat ttaaatatgg cacaggttat caacacgttt gacggggttg 3060
cggattatct tcagacatat cataagctac ctgataatta cattacaaaa tcagaagcac 3120
aagccctcgg ctgggtggca tcaaaaggga accttgcaga cgtcgctccg gggaaaagca 3180
tcggcggaga catcttctca aacagggaag gcaaactccc gggcaaaagc ggacgaacat 3240
ggcgtgaagc ggatattaac tatacatcag gcttcagaaa ttcagaccgg attctttact 3300
caagcgactg gctgatttac aaaacaacgg accattatca gacctttaca aaaatcagat 3360
aacgaaaaaa acggcttccc tgcgggaggc cgtttttttc agctttacat aaagtgtgta 3420
ataaattttt cttcaaactc tgatcggtca atttcacttt ccggctctag agctctagag 3480
tccggtccaa tctgcagccg tccgagacag gaggacatcg tccagctgaa accggggcag 3540
aatccggcca tttctgaaga gaaaaatggt aaactgatag aataaaatca taagaaagga 3600
gccgcacatg aaaaaagcag tcattaacgg ggaacaaatc agaagtatca gcgacctcca 3660
ccagacattg aaaaaggagc ttgcccttcc ggaatactac ggtgaaaacc tggacgcttt 3720
atgggattgt ctgaccggat gggtggagta cccgctcgtt ttggaatgga ggcagtttga 3780
acaaagcaag cagctgactg aaaatggcgc cgagagtgtg cttcaggttt tccgtgaagc 3840
gaaagcggaa ggctgcgaca tcaccatcat actttcttaa aattaaaaga gctttcgttc 3900
gtatcatcgg tttcgacaac gttcgtcaag ttcaatgcat cagtttcatt gcgcacacac 3960
cagaatccta ctgagtttga gtattatggc attgggaaaa ctgtttttct tgtaccattt 4020
gttgtgcttg taatttactg tgttttttat tcggttttcg ctatcgaact gtgaaatgga 4080
aatggatgga gaagagttaa tgaatgatat ggtccttttg ttcattctca aattaatatt 4140
atttgttttt tctcttattt gttgtgtgtt gaatttgaaa ttataagaga tatgcaaaca 4200
ttttgttttg agtaaaaatg tgtcaaatcg tggcctctaa tgaccgaagt taatatgagg 4260
agtaaaacac ttgtagttgt accattatgc ttattcacta ggcaacaaat atattttcag 4320
acctagaaaa gctgcaaatg ttactgaata caagtatgtc ctcttgtgtt ttagacattt 4380
atgaactttc ctttatgtaa ttttccagaa tccttgtcag attctaatca ttgctttata 4440
attatagtta tactcatgga tttgtagttg agtatgaaaa tattttttaa tgcattttat 4500
gacttgccaa ttgattgaca ac 4522
<210> 4
<211> 635
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 4
agagctttcg ttcgtatcat cggtttcgac aacgttcgtc aagttcaatg catcagtttc 60
attgcgcaca caccagaatc ctactgagtt tgagtattat ggcattggga aaactgtttt 120
tcttgtacca tttgttgtgc ttgtaattta ctgtgttttt tattcggttt tcgctatcga 180
actgtgaaat ggaaatggat ggagaagagt taatgaatga tatggtcctt ttgttcattc 240
tcaaattaat attatttgtt ttttctctta tttgttgtgt gttgaatttg aaattataag 300
agatatgcaa acattttgtt ttgagtaaaa atgtgtcaaa tcgtggcctc taatgaccga 360
agttaatatg aggagtaaaa cacttgtagt tgtaccatta tgcttattca ctaggcaaca 420
aatatatttt cagacctaga aaagctgcaa atgttactga atacaagtat gtcctcttgt 480
gttttagaca tttatgaact ttcctttatg taattttcca gaatccttgt cagattctaa 540
tcattgcttt ataattatag ttatactcat ggatttgtag ttgagtatga aaatattttt 600
taatgcattt tatgacttgc caattgattg acaac 635
<210> 5
<211> 854
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 5
atggcacagg ttatcaacac gtttgacggg gttgcggatt atcttcagac atatcataag 60
ctacctgata attacattac aaaatcagaa gcacaagccc tcggctgggt ggcatcaaaa 120
gggaaccttg cagacgtcgc tccggggaaa agcatcggcg gagacatctt ctcaaacagg 180
gaaggcaaac tcccgggcaa aagcggacga acatggcgtg aagcggatat taactataca 240
tcaggcttca gaaattcaga ccggattctt tactcaagcg actggctgat ttacaaaaca 300
acggaccatt atcagacctt tacaaaaatc agataacgaa aaaaacggct tccctgcggg 360
aggccgtttt tttcagcttt acataaagtg tgtaataaat ttttcttcaa actctgatcg 420
gtcaatttca ctttccggct ctagagctct agagtccggt ccaatctgca gccgtccgag 480
acaggaggac atcgtccagc tgaaaccggg gcagaatccg gccatttctg aagagaaaaa 540
tggtaaactg atagaataaa atcataagaa aggagccgca catgaaaaaa gcagtcatta 600
acggggaaca aatcagaagt atcagcgacc tccaccagac attgaaaaag gagcttgccc 660
ttccggaata ctacggtgaa aacctggacg ctttatggga ttgtctgacc ggatgggtgg 720
agtacccgct cgttttggaa tggaggcagt ttgaacaaag caagcagctg actgaaaatg 780
gcgccgagag tgtgcttcag gttttccgtg aagcgaaagc ggaaggctgc gacatcacca 840
tcatactttc ttaa 854
<210> 6
<211> 3018
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 6
gtggaagtgt tactacatgg caggtacacg ttaccacatg tttatacagt aacactattc 60
ataaattgca gtaagacgca ttgttactac aaaacttatg gtttttacta cacacttaca 120
gtaacgtcgc gatgggattg tagtaacgca taacagatag tactaacaag tagtaatgct 180
atagtattgc tacttgtgga gagttaagat caaaaaaccc aagcttcgaa taacaatatc 240
tctcatgtga tgtatgattt tttaaaaccg tttgcaccat gatgttagaa aaaaatgctt 300
atcaaaaata gatccgtcat gactatattt caactaaatt tgaatgttgt tactgcacgt 360
aagacacagt gttactgcac gatggctgtc gctttactgc actgtttcgc aagacgagag 420
ctgagaagag gtgggtggtg ggggagttgg taggcagttt gaccgggttt tgaccgacgt 480
cgggggtgct ttgactgacc tttgactgag gtgggaggtg ggtttggccc ttgggaggga 540
gggggggata tagaatagct attctatacc cgggtgtaga atagtttgtg tatgtgtgtg 600
tgtgtgtata tatatatata tataaaatca aagtgtggga tattatattt catgtacaat 660
ctcataaaga gtctactaaa tatacttctc tctgaatata gtttgacaaa ttagttttgt 720
gtgttgctaa taactaatat aattttttac ttttagctcg attattactt aggaatttcc 780
gcagcaaagt gcggggaatc acctagttaa ttaaagaatt gagtcctcat aaaatacctt 840
ttcaatatgg cctgaatcat tgtttggttt tttggccttc tgttctattt ggtccaaaag 900
gccatctttc taagttggac tgggcccatg gtttcgttat gggccaaaaa caacaatagc 960
attttgtttt attctcccct tttgaaaaca gtatgcaggg gtgatgcttg ttcattataa 1020
gaggcaaagg gtagcatatg atatataagt ttataattta ttatagatga caacctcaaa 1080
ggtaactatc gttgacatat gtaaattatg ctacattaca aaagaaataa tgtgaacatt 1140
gacattggta catacgtgga cattatctat aattatagac tgacatctac attccctatg 1200
gttatcaatc ggaatgtcgg ctcttgtagc cttgtgaaca tacgtactct ctcgcactat 1260
atgtgcattg atgtgaatcc tccaatttgc tataggaatg tcgtaggttt gagtagccta 1320
tgtattcatt gtcagcaaat gcgcaccgtg aactctttac gtcagaggat ctacgtcagt 1380
ccgctagtac gctcatgaca tattccccct gtttttgaag atgcggttag ttactccttc 1440
tgtacactcc ataaaatgtg ctatcctaca atacattccc cgcttcaatg tatgagatat 1500
ttttccccat tttgttagaa aatacttaag caatatgttc caaatataat gttatattgg 1560
gttgatgttg tgttaaaatg gagtggttaa gcatgagcct tcatttccct cataactgtt 1620
tttttagaac tcctcgcaac tgttgcactc aacaatttca ttttccttcc tgttattcaa 1680
ttaaggttct ccttaaaaat aagtctttca tgaataattt agtattttga aggtactatt 1740
gaaagaatta tttggagatg tctaatgctt gaatgataag taatttggaa ggcatctact 1800
cctacaaagt tgaatcttct aaatactcct tcgcttttaa atatacggca ttgttaactt 1860
ttgaatatga caatatttaa caatttatct tgtacaaaga tttagtgcaa atatgcacaa 1920
tgcgtaaagt acctttaatg taaaacaagc cacaacaaaa caaatgatac ttagagcatt 1980
ttttaataag ccgattggtc gcatcatgtt taaaagtcaa ccatgtcata tataaaaaaa 2040
gggagatagt ttcatacata gctctaatat attttgaagg tactaaattt taaaaattgt 2100
tgttgcagat tgtaagtctt gcatataatt tattgtttct tttacttagc ttattagctc 2160
taattaatca caccctatct ctcctatatt tatataataa aaacatcccg acacaccaac 2220
gtacattttc tcatctattt ctaaggtatg gatggtattg tcatgtcaac ttatcttctt 2280
tctagttatg caccctcctt aattattgta ccaacttaaa ataagccata tattttgaaa 2340
ttatcatacc aactcacaat atgccatata ttttaaaatt attatgtcaa ctcacaatat 2400
gctatatatt ttgatattat cgtgccaact cacaatatac catatatttt gaaatgaagg 2460
tcatatttaa gatgtagagt aaatgtatga aatcctccca tccatatcct cattattaat 2520
tgcttccacc taccccaagc atttatagtt agagtagaac agttgtgctc ttgcaccgaa 2580
gggatgacat gtaggagcca tgcctacgtc ccctggccac ccaagcccgt tgaattaaca 2640
tggtccctca acagaaacat ccatttttga tggtgtgctt ccttcttggc gaccttactg 2700
gtaggtgaca tgctaacaaa aactgcacag tcatgcaagg caggggccgg tcaaaattga 2760
agtccctcct accttgattg taacgctaat actatggctc accgcctctg cggcaatctg 2820
gcagccactt ctcctcctct tggtgccaca gctgcacctt tgacgtggct ggctgcttgc 2880
atttgtccaa gccggcgcag ccctgatgta agtcctttgg tataaatact gcccaagcat 2940
agagcgagag agagccatca caatccaata caagttcaca acataagcat agtacaggtc 3000
attgagactt gagagagc 3018
<210> 7
<211> 457
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 7
tatgtacagc attacgtagg tacgttttct ttttcttccc ggagagatga tacgataatc 60
atgtaaaccc agaatttaaa aaatattctt tactataaaa attttaatta gggaacgtat 120
tattttttac atgacacctt ttgagaaaga gggacttgta atatgggaca aatgaacaat 180
ttctaagaaa tgggcatatg actctcagta caatggacca aattccctcc agtcggccca 240
gcaatacaaa gggaaagaaa tgagggggcc cacaggccac ggcccacttt tctccgtggt 300
ggggagatcc agctagaggt ccggcccaca agtggccctt gccccgtggg acggtgggat 360
tgcagagcgc gtgggcggaa acaacagttt agtaccacct cgctcacgca acgacgcgac 420
cacttgctta taagctgctg cgctgaggct caggttg 457
- 一种利用胚乳特异性自杀获得非转基因的定向基因突变植株的方法及其应用
- 一种快速高效获得非转基因的定向基因突变植物的方法和应用
