海水青鳉母源效应基因org及其sgRNA片段
文献发布时间:2024-01-17 01:27:33
技术领域
本发明属于分子生物学技术领域,设计了一种海水青鳉母源效应基因org及其sgRNA片段。
背景技术
母源效应基因(MEGs)在卵母细胞生长发育过程中转录母源因子并积累在卵母细胞细胞质中,这些母源因子参与动物卵母细胞减数分裂恢复,受精后雌雄原核融合以及早期胚胎合子基因激活等过程。这些母源效应基因在不同的过程中发挥特异性的功能,保证生命体活动的正常进行。近年来,研究人员在哺乳动物中鉴定了许多母源效应基因,包括小鼠中Hsf1、Bnc1、Figla和Nlrp5基因,然而在鱼类中母源效应基因的挖掘还不够深入,特别是海水青鳉的母源效应基因功能研究较少。海水青鳉已被公认为亚洲地区生态毒理学和环境研究的模式鱼类,具有繁殖周期短(3-4个月),每日产卵,小尺寸(3-4厘米),透明胚胎,以及易于在实验室中进行大规模培养等特征,这些特点使海水青鳉成为体内基因功能试验的模式生物。
近几年,CRISPR/Cas9基因编辑技术的发展取得了革命性的成功,在模式生物青鳉中的应用也越来越成熟。如研究人员通过CRISPR/Cas9技术获得了白化的青鳉,为研究色素沉着提供了重要的平台。此外,通过非同源末端连接的方法将含有热休克启动子和报告基因的供体质粒转入青鳉胚胎中,高效生成了敲入型转基因青鳉鱼,并促进了基因功能分析。这些为我们利用CRISPR/Cas9技术以海水青鳉为实验对象研究基因功能提供了理论和实验基础。
发明内容
本发明通过CRISPR/Cas9技术鉴定了海水青鳉母源效应基因org及其sgRNA片段。发现海水青鳉org基因与其他母源效应基因一致,它的缺失将导致胚胎无法完成早期合子基因组的激活和母本向合子转变的遗传控制过程,造成早期胚胎发育停滞并最终死亡。此外,我们发现org基因与其他母源效应基因在早期胚胎发育中的表达模式一致。
本发明是通过如下技术方案来实现的:
本发明的第一方面提供一种海水青鳉母源效应基因org,所述基因的核苷酸序列为SEQ ID NO.1。
本发明的第二方面提供org基因的sgRNA,SEQ ID NO.2:GGTGGGTCCAGAGCGCTCTG和SEQ ID NO.3:CTCAGCGTTTCCACCCAGGA。
本发明与现有技术相比的有益效果:本发明首次克隆了海水青鳉卵子发生相关基因org,设计并合成了org基因的sgRNA,检测了sgRNA的有效性。我们利用CRISPR/Cas9技术成功敲除了海水青鳉早期胚胎中的org基因,实验组中org基因的缺失导致胚胎无法完成早期合子基因组的激活以及母本向合子转变的遗传控制过程,造成早期胚胎发育停滞并最终死亡,这与其他母源效应基因敲除后胚胎发育一致。此外,我们研究了org基因在早期胚胎发育中的表达模式,与其他母源效应基因一致,在合子激活期表达量十分丰富,并在完成母本向合子转变的遗传控制过程后降解。证明了org作为母源效应基因在早期胚胎发育中的重要作用。
本发明提供org基因的核苷酸序列,核苷酸序列为SEQ ID NO.1;提供org基因靶位点序列及设计合成的sgRNA序列SEQ ID NO.2-3,提供用于扩增靶位点片段的引物,所述引物序列分别为org-gRNAF12、org-gRNAF3和org-gRNAR。此外,本发明提供海水青鳉养殖及胚胎显微注射的具体步骤,sgRNA设计及合成的具体步骤;海水青鳉单个胚胎DNA提取的步骤以及用于RT-qPCR的引物:O.melastigma-qF、O.melastigma-qR、O.melastigma-actin-qF、O.melastigma-actin-qR。
附图说明
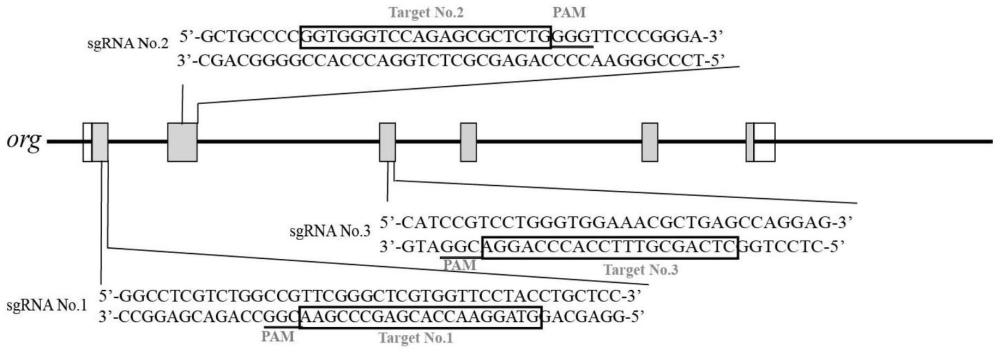
图1为本发明实施例3中的org基因靶位点序列图。
图2为本发明实施例3中的sgRNA合成纯化后的电泳结果图。
图3为本发明实施例4中的野生组胚胎及实验组胚胎发育的表型图。
图4为本发明实施例5中的SEQ ID NO.2工作效率检测峰图。
图5为本发明实施例5中的SEQ ID NO.3工作效率检测峰图。
图6为本发明实施例6中的org基因在早期胚胎发育中的表达模式图。
具体实施方式
下面将结合实例中的附图,对本发明技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术工作人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
实施例1:海水青鳉养殖及受精卵收集包括以下步骤:
1、海水青鳉及胚胎养殖
海水青鳉循环养殖系统购于上海海圣生物实验设备有限公司。循环系统水温26℃,PH范围维持7.0—8.0,盐度范围32-34,溶解氧5—8mg/L,光周期为14小时(8:00—22:00)光照和10小时(22:00—8:00)黑暗。根据海水青鳉交配产卵特性,每个鱼缸中雌鱼与雄鱼比例为3:2,养殖密度一般5—10尾/L,每日进食新鲜的蠕虫2-3次,保证海水青鳉的生长和繁殖。成年海水青鳉每条每日产卵30-50颗,在其产卵后及时取卵,放在培养皿中进行培养,且每日更换培养皿中的海水,保证海水中充足的含氧量,维持胚胎的正常发育。
2、海水青鳉受精卵收集
在显微注射前一晚,将性成熟海水青鳉雌雄分缸,每个鱼缸中雌鱼6-8尾,雄6尾,注射当天系统亮灯前一小时将雌雄青鳉合为一缸,雌鱼开始产卵,雄鱼在系统亮灯之后去追尾并完成受精。根据雌鱼产卵的数量和后期实验的安排可以实时调整实验中雌雄鱼的数量。
受精后约半小时,受精卵发育到单细胞时期,将挂在生殖孔周围的胚胎及时取下,用循环系统水清洗2-3遍,显微镜下观察胚胎的发育时期,用吸管吸取单细胞时期胚胎并排列在制备好的胚胎卡槽上,显微镜视野中用调整胚胎位置,使其动物极与显微注射针在一条直线上。剩余胚胎置于冰上或4℃冰箱,减慢分裂速度。二细胞时期之后的胚胎不再适用显微注射。
实施例2:海水青鳉胚胎显微注射包括以下步骤:
1、显微注射胚胎卡槽的制备
取2%的琼脂溶解于50mL无菌水,微波炉加热直至沸腾倒入70mm无菌培养皿中,厚度约2/3,将排卵模具带有棱纹的一面放入培养皿的琼脂溶液上层,放入4℃冰箱加速凝固,待凝固后将模具从琼脂中取出即可完成胚胎拉槽的制备,使用前在卡槽中加入26℃培养箱预热,使用后在卡槽中加入无菌水放入4℃冰箱中保存,可反复利用。
2、显微注射针准备
显微注射针由毛细玻璃管在水平拉针仪(Sutter Instaument,P-1000)中按照青鳉胚胎的参数拉制,毛细玻璃管购于WPI(World Precision Instruments)公司,外径0.8mm。青鳉胚胎的拉针参数为:拉力PULL:55,温度HEAT:550,压力Pressure:300,速率VEL:60。将毛细玻璃管固定在拉针仪的加热丝中,利用加热丝的温度以及玻璃管两端的拉力将玻璃管烧断,此时呈封闭状态。在显微注射前,将显微注射针(内含有0.05%酚红指示剂溶液)固定在注射器中,在显微镜载物台放置载玻片,调整显微注射针角度,与载玻片夹角约40-50度,调整显微注射针在视野中位置,使其置于视野中央,缓慢调节螺母直到针头刚接触载玻片,有酚红指示剂溶液流出后立刻抬起显微注射针,完成断针。
3、海水青鳉胚胎显微注射
(1)先开氮气罐的大阀,控制在12-13兆帕,再开减压阀,0.3兆帕以内,打开注射仪,调节注射仪注射压16psi以下,hold压在zero范围内。Hold压处的开关调到vent,打开此处通路,Eject压处的开关调到hold,防止倒吸
(2)上样装针。用RNase free枪头吸取RNP复合物试剂2μL于显微注射针顶端,注意不要产生气泡。
(3)调整显微注射剂量。将显微注射针头置于视野中心,先使hold压力,慢慢增大,使RNA复合物形成液滴聚在显微注射针头,再微调hold压,使液滴不再增大也不滴落。
(4)注射。将单细胞时期胚胎置于体视显微镜下,调整视野清晰度,调整胚胎位置,使显微注射针头与胚胎动物极细胞在同一水平线,旋转螺旋使注射针的针尖进入胚胎单细胞期动物极,踩动脚踏板,动物极出现红色液滴(RNP复合物)且胚胎未破损为注射成功。
(5)注射结束后,先关氮气罐大阀,操作系统完全排气后,再关减压阀,使用后的显微注射针放入废弃医疗盒。
(6)注射完成后,将鱼卵放入盛有海水的培养皿中,并在26℃的培养箱中培养。每天观察鱼卵状态,吸出死卵并换水。
实施例3:sgRNA设计及合成纯化包括以下步骤:
1、sgRNA设计
在NCBI数据库中查询青鳉org基因序列信息,提取青鳉卵巢RNA并反转成cDNA,具体步骤如下:
采用Total RNA Purification Kit(Norgen Biotek,Canada)进行总RNA提取,具体步骤如下:
(1)选取雌性性成熟海水青鳉冰水麻醉,使用灭菌的镊子手术刀等工具解剖海水请将并取约20mg的卵巢组织放进1.5mL RNase-free的离心管中,加入300μL的Buffer RL,用组织研磨器进行研磨至溶液透亮。
(2)向充分研磨的溶液中加入300μL的Buffer RL补充至600μL,再加入600μL70%的酒精溶液,涡旋混合。
(3)将混合液转入RNA Binding Column中,13000rpm,离心1min,弃掉滤液。
(4)重复步骤3。
(5)RNA Binding Column放入新的RNase-free的1.5mL的离心管中,并加入50μL的Elution Solution A,200rpm离心2min,14000rpm离心1min。
(6)取溶解在Elution Solution A中的总RNA 1μL,使用NanoDrop2000分光光度计(Thermo,Waltham,MA,USA)进行RNA浓度和纯度的检测,另取1μL的RNA溶液用1%琼脂糖凝胶电泳检测RNA的完整性,其余放入-80℃的超低温冰箱中进行保存。
用DNA-free
(1)取3μg质量合格的RNA放入RNase-free的200μL离心管中,并置于冰上,加入10×DNase I Buffer和1μL rDNase I,混合均匀。
(2)将离心管放入PCR仪中,37℃孵育30min。
(3)孵育完成后将离心管取出,置于冰上,加入DNase Inactivation Reagent并混合均匀。
(4)室温放置2min,中间吹打1-2次。
(5)10000rpm离心1.5min,将RNA转移至新的RNase-free离心管中。
将擦除DNA之后的RNA使用Takara的PrimeScript
(1)取擦除DNA后的RNA1μg放到RNase-free的200μL离心管中,并置于冰上,加入1μL Oligo dT Primer,1μL dNTP Mixture,加入RNase Free水使总体系为10μL,混合均匀并瞬时离心。
(2)将离心管放入PCR仪中,65℃孵育5min。终止后立即置于冰上快速终止反应。
(3)取新的离心管,加入4μL 5×PrimeScript II Buffer、0.5μL RNaseInhibitor、1μL PrimeScript II RTase、4.5μL RNase Free水。并将步骤1中的产物转移到新的离心管中,混合均匀并瞬时离心。
(4)将混合后的样品放入PCR仪中,42℃反应60min,95℃反应5min得到cDNA,并将产物放在-20℃冰箱,供后续使用。
根据org基因序列设计引物以海水青鳉卵巢cDNA为模板进行序列验证,海水青鳉org基因CDS区序列(SEQ ID NO.1)为:ATGGCGTCTGGACGGCGCGTGGACGCAGCGGAGCGGGGAGGAGCCTCTGAGGAGGCGGAGGGAGCCGTGGCCCCGCGGGACGGCGGGCTCCGCGCGCTGCTCCGCGGCCTCGTCTGGCCGTTCGGGCTCGTGGTTCGAGTCTGCTCCAGAGCCTGGCAGCTGCTCGGCTTCCAGGAACCGCACGCGGTCACCTCGGCTTCCGGCCCCCCGCGTCCCAGCCGTCGGAAGCGCCTGCACCGTGTCACGCGCGCGCTGCTGTCCGTGCTGCCCCGGTGGGTCCAGAGCGCTCTGGGGTTCCCGGGACCCGCCAGCATCGGGGTCTCCCTGTCCCCAGAAATCCGGAGTTCTCCCACCAAACCTCACGGGAAAGGCAGCAAGCGCAAACAGGACGACCTGGATGACGAGGAGGAGGAGGAGCATCCGTCCTGGGTGGAAACGCTGAGCCAGGAGCTGGCAGACGACGACGGCCCGGCTGAAGATCCGGACTATGAGCCCAGCTCCGTGGAGACGGACACGGAGGAGTACGCCTCCCACAACAACAGGAGAGCGACCTGGAGGCCCCAGGAGGAGGTGTGCTGATCCGGGACGTCCAGACGACACGCCCACTCTGTAACCGACCTGGACTCGTTCTACTCGGAGCTGCTTTGTTTTCATACTTTGATGGAAATCTTGCTGGAATTGAAGTCTTTTAG。
对org基因进行sgRNA序列设计,设计的原则是:首先超过4个胸腺苷酸(5‘-TTTT-3’)的sgRNA被排除,因为它们被RNA聚合酶III识别为转录终止信号。其次为了gRNA的效率,靶位点位置应尽量在第一外显子上,靶位点序列长度在20bp,不可以是3的倍数,GC含量一般在60%左右。针对青鳉org基因序列设计了三条sgRNA:
SEQ ID NO.2:GGTGGGTCCAGAGCGCTCTG;
SEQ ID NO.3:CTCAGCGTTTCCACCCAGGA;
SEQ ID NO.4:ACTCGAACCACGAGCCCGAA。此外,通过NCBI中的BLAST工具,在海水青鳉整个基因组上面输入靶位点碱基序列,检查靶位点序列的特异性。
如图1是根据sgRNA设计原则选取的三个靶位点。方框内序列表示sgRNAs的目标序列,划线序列表示NGG原间隔子相邻基序序列。
2、sgRNA合成及纯化
以质粒PGL3-U6-sgRNA-PGK-puromycin(Addgene,USA)为模板,正向引物为T7启动子引物序列+提高T7聚合酶效率的碱基GGG+靶位点序列+gRNA骨架上游的20个碱基;反向引物为gRNA骨架下游的反向互补序列。上下游引物分别为:org-gRNAF1:TGTAATACGACTCACTATAGGAGCTCACCTGAAGAAGCGCGGTTTT AGAGCTAGAAAT;org-gRNAF2:TGTAATACGACTCACTATAGGTGGACGGCGCGTGGACGCAGGTTTT AGAGCTAGAAAT;org-gRNAF3:TGTAATACGACTCACTATAGGGGAGCGGGGAGGAGCCTCTGGTTTT AGAGCTAGAAAT;org-gRNAR:AAAAGCACCGACTCGGTGCC。将PCR产物通过琼脂糖凝胶电泳进行验证并纯化。纯化后的产物作为转录模板,使用T7 HighYieldTranscriptionKit(Thermo Fisher)进行转录反应,0.5μg的模板投入量可以产生150-200μg的RNA。
转录后的RNA按照下述步骤进行纯化:
(1)加入160μL RNase-free ddH
(2)加入20μL,3M的醋酸钠(pH 5.2)到稀释后的产物中,充分混匀。
(3)加入200μL的酚/氯仿混合液(1:1)进行抽提,室温12,000rpm离心5min,将上层溶液转移至新的RNase-free EP管中。
(4)加入与水相等体积的氯仿抽提2次,收集上层水相。
(5)加入2倍体积的无水乙醇,混匀,-20℃孵育30min,4℃12,000rpm离心15min。
(6)弃上清,加入500μL预冷的70%乙醇洗涤RNA沉淀,4℃12,000rpm离心,弃上清。
(7)开盖干燥2min,加入20μL RNase-free ddH
如图2是纯化之后的sgRNA电泳图。orggRNA的条带大小为120bp左右。org sgRNA浓度分别为org SEQ ID NO.2:903.76ng/μL,org SEQ ID NO.3:872.05ng/μL,org SEQ IDNO.4:956.63ng/μL。
实施例4:显微注射组胚胎的发育过程
将三个sgRNA分别用0.05%的无菌无酶追踪指示剂酚红溶液稀释至100ng/μL,cas9mRNA稀释至400ng/μL,将sgRNA分别于cas9mRNA室温孵育5-10min,形成RNP复合物。向青鳉胚胎动物极的单细胞期每次注射2nL RNP复合物,在二细胞期之前可注射受精卵约100枚,野生型受精卵作为对照。实验组及野生组的胚胎均放在26℃的恒温培养箱中,及时清除死卵,防止污染其他胚胎。根据不同时期胚胎发育的不同阶段观察其发育情况,如动物极细胞分裂速度。如图3所示,我们发现在受精后3小时内,野生组和实验组胚胎发育并没有明显差异。而在受精3小时后,实验组胚胎开始出现发育缓慢的现象。在受精后约6小时,野生组和实验组的胚胎发育速度出现明显的差异,实验组的动物极细胞数量相比于受精后3小时的胚胎并没有增加,并且细胞形态出现异常。受精后约10小时,实验组的胚胎动物极细胞凋亡,卵黄膜颜色发黑。到受精后约22小时,实验组胚胎卵黄膜皱缩并死亡。相比之下,野生型胚胎正常发育。根据海水青鳉胚胎发育过程结合我们根据时间记录的胚胎发育过程,可以总结出:实验组胚胎在桑葚胚期之前发育已经停滞,囊胚晚期开始死亡。受精后24小时约54%的胚胎完全死亡,剩下约56%的胚胎与野生组相比发育稍微迟缓。
实施例5:靶位点工作效率检测
收集实验组和野生组胚胎,用裂解法提取青鳉胚胎基因组DNA。
方法如下:
(1)将清洗后的单个胚胎放入200μL小离心管中,用RNase Free的枪头或牙签(经过高温灭菌)捅破胚胎。
(2)离心管中加入8μL DNA裂解液(含20mg/ml的蛋白酶k),充分混匀并瞬时离心。
(3)将离心管放入PCR仪,65℃反应30min,95℃反应10min。12000rpm离心5min,取上清溶液DNA,-20℃保存。
设计引物以基因组DNA为模板对org基因扩增,上下游引物(sgRNA1-2F:AGCGGAGCGGGGAGGAGC;sgRNA1-2R:CCCCGCCGCATTTTGGTT;sgRNA-3F:TGCGTCCCACAGAAATCC;sgRNA-3R:CCGTGTTGTTGTGGGAGG)在靶位点上下游各400-500bp设计,产物长度应在800bp以内,保证测序的正确性。PCR体系为1μLDNA模板,1μL正反向引物,10μL Rapid酶,7μLddH
如图4、5所示:野生型和实验组未死亡胚胎的org基因序列峰图均为单一峰,而实验组死亡胚胎中有23枚发生碱基突变,分别在靶位点1和靶位点2出现连续套峰,其中靶位点1的突变发生在反义链上,靶位点2的突变发生在正义链上。靶位点3并没有出现突变,说明sgRNA1和sgRNA2具有引导cas9蛋白发挥切割的作用。经过统计,实验组胚胎发生碱基突变的共有23枚,占实验组死亡胚胎(63枚)的36.5%,占整个实验组(112枚)的20.5%。本实验中org基因敲除的胚胎发育停滞并最终死亡,org基因未敲除的胚胎出现死亡或发育迟缓的原因可能是胚胎受到物理损伤。
实施例6:org基因在早期胚胎发育中的表达模式:
O.melastigma-qF:CCCTGTCCCCAGAAATCC;
我们根据青鳉org基因序列设计引物:
O.melastigma-qR:CTCCTGGCTCAGCGTTTC;
O.melastigma-actin-qF:CTGAGGATTGCTGAGCCTTACAT;
O.melastigma-actin-qR:CTCAACACAGATGATGCCGTATT。
通过RT-qPCR检测了海水青鳉早期胚胎发育过程中org基因的表达水平。如图6所示,org基因在单细胞期到囊胚期间的表达水平小幅度上升,并在囊胚期达到最高值,之后orgmRNA的表达水平迅速下降,在器官形成期几乎检测不到org基因的表达。本实施例中,我们探究了org基因在海水青鳉早期胚胎中的表达模式,母源效应基因nmp2表达模式一致,在早期合子基因激活阶段表达丰富,并在母体向合子遗传控制的转变过程中迅速降解。
本发明中org基因敲除胚胎发育的过程与母源效应基因nmp2、foxr1敲除后发育表型一致,并且,org基因与母源效应基因nmp2在海水青鳉早期胚胎发育过程中的表达模式一致证明org作为母源效应基因,通过参与早期胚胎合子基因的激活过程保证胚胎的正常发育。org基因的敲除使早期合子基因组的激活失败,org基因可能参与了早期胚胎中特定母体mRNA的翻译激活,敲除org基因后,母源产物在囊胚期之前不能积累,导致胚胎没有能够从母体控制过渡到合子控制,胚胎的全能型遭到破坏,从而导致胚胎死亡。
本发明并不局限于上述特定的实施方案范围内,上述实施方案只是为了能够对本发明的使用过程进行详细的说明,而且有相同功能的生产方法和技术细节也属于本发明的一部分。事实上,本领域技术人员根据前文的描述,就能够根据需要找到不同的调整方案,这些调整也在本文所附的权利要求书的范围内。
- 用于转基因青鳉鱼的基因片段
- 用于转基因青鳉鱼的基因片段
