一种基于PIK3R1基因鉴定鸡饲料利用率性状的分子标记及其鉴定方法和应用
文献发布时间:2023-06-19 13:27:45
技术领域
本发明涉及分子标记技术领域,具体涉及一种基于PIK3R1基因鉴定鸡饲料利用率性状的分子标记及其鉴定方法和应用。
背景技术
饲料效率性状是畜禽生产中的重要经济性状之一,其中饲料利用率是评估饲料效率重要方法之一。饲料利用率指的是家禽每日增加的重量,受测家禽在测定期中的平均饲料利用率,体现了家禽的生长情况。其计算公式为饲料利用率等于试验期内耗料量除以体增重。近年来,剩余采食量(residual feed intake,RFI)作为衡量饲料利用率的重要指标,广泛应用于家禽育种工作中。RFI指动物实际采食量与用于维持和生产的期望采食量之差。RFI是一个负向选择性状,RFI值越小,饲料利用率越高。影响饲料利用率的因素众多,包括遗传因素。研究人员发现传统的遗传选育对家禽育种的进展十分缓慢。近10年来,分子遗传和分子生物技术发展突飞猛进,尤其是分子标记技术。通过检测个体遗传物质变异,实现对个体进行分子标记选育。
PCR-SSCP可以实现对不同个体基因突变进行快速检测。SSCP是单链构象多态性,一种基于DNA构象差别来检测点突变的方法。相同长度的单链DNA,如果碱基序列不同,形成的构象就不同,这样就形成了单链构象多态性。单链DNA片段除其碱基数量不同引起长度的不同外,最主要的是复杂的空间折叠构象的不同,这种立体空间折叠构象是由其内部碱基配对和分子内相互作用力来维持的,当有碱基发生改变时,会影响其空间构象,使构象发生改变,空间构象有差异的单链DNA分子在非变性丙烯酰胺凝胶中受排阻大小不同。因此通过非变性丙烯酰胺凝胶电泳,可以非常敏锐地将构象上有差异的分子分离开。
PIK3R1基因(phosphoinositide-3-kinase,regulatory subunit 1),中文名磷酸肌醇3激酶调控亚基1,分布于细胞内的磷酸肌醇3激酶复合体,是一种脂质激酶,可以与胰岛素受体、胰岛素样生长因子受体、磷脂酰肌醇、激酶等结合,参与细胞内信号转导级联、胰岛素受体信号转导途径、磷酸肌醇磷酸化、胰岛素样生长因子受体信号转导途径等,参与生物体的多种生命活动和代谢途径,在细胞增殖、生长、代谢、葡萄糖稳态中发挥重要作用。研究发现该激酶在下丘脑瘦素与食物摄入减少的信号通路中起关键作用。Jing等对去势的纯种约克夏猪饲料效率的研究发现,参与骨骼肌分化增殖的PIK3R1上调。而骨骼肌在能量代谢中占据重要地位,减少能量代谢和增加肌肉可以提高饲料利用率。Chen等对莱芜猪的研究发现,PIK3R1基因参与食物摄入和能量消耗两组神经元的活动,是维持食物平衡的中枢调控因子。
皖南三黄鸡是我省重要的地方优良鸡种,提高其饲料利用率可以从根本上较少成本的支出,增加养鸡业的收入。早期研究人员常使用常规选育但效率并不高,随着技术发展,分子标记辅助选育映入眼帘,其可以从遗传上根本改良性状,从而加速遗传进展。基于上述内容,提出一种基于PIK3R1基因鉴定鸡饲料利用率性状的分子标记及其鉴定方法和应用。
发明内容
本发明的目的在于克服现有技术的不足,提供了一种基于PIK3R1基因鉴定鸡饲料利用率性状的分子标记及其鉴定方法和应用,针对与鸡的饲料利用率性状相关的候选基因的SNP(单核苷酸多态性)分子标记,以解决常规表型育种选育进展缓慢,实现早期鉴定饲料利用率性状这一难题。
本发明通过以下技术方案来实现上述目的:
本发明提供了一种基于PIK3R1基因鉴定鸡饲料利用率性状的分子标记,所述分子标记为G或T,所述分子标记位于PIK3R1基因的第579位。
进一步改进在于,所述PIK3R1基因具有如SEQ ID NO.1所示的核苷酸序列。
本发明还提供了一种上述基于PIK3R1基因鉴定鸡饲料利用率性状的分子标记在鉴定鸡饲料利用率性状中的应用。
本发明还提供了一种利用上述分子标记鉴定鸡饲料利用率性状的鉴定方法,包括以下步骤:
(1)提取鸡翅静脉血液总DNA;
(2)以所述分子标记所在位点及其上下游碱基组成的序列为目标序列设计特异性扩增引物,以所述总DNA为模板,利用特异性扩增引物进行PCR扩增,获得扩增产物;
(3)对扩增产物进行基因分型检测和测序,获得待测鸡的分子标记类型;
(4)根据分子标记类型判断鸡饲料利用率性状性状。
进一步改进在于,所述分子标记所在位点的上下游扩增产物长度大于200bp。
进一步改进在于,所述特异性扩增引物序列为:
SEQ ID NO.2:F1:TTCAGCGATAAACTCCGGGC
SEQ ID NO.3:R1:TGTCACCAGACCACTCCTGT。
进一步改进在于,所述基因分型检测方法为将PCR扩增产物进行非变性聚丙烯酰胺凝胶电泳和银染获得图像,根据图像进行基因分型:
(1)包含1条条带,则为GG型;
(2)包含2条条带,则为TT型;
(3)包含3条条带,则为TG型。
进一步改进在于,所述步骤(4)中根据分子标记类型判断肉鸡饲料利用率性状的具体步骤为:
(1)若待测鸡分子标记类型为GG型,该鸡饲料利用率性状最好;
(2)若待测鸡分子标记类型为TT型,该鸡饲料利用率性状较差;
(3)若待测鸡分子标记类型为TG型,该鸡饲料利用率性状中等。
本发明的有益效果在于:本发明提供了一种基于PIK3R1基因鉴定鸡饲料利用率性状的分子标记及其鉴定方法和应用,利用PCR-SSCP的方法来检测PIK3R1基因的突变,根据基因型对鸡的饲料利用率性状进行选择,建立了一种家禽饲料利用率早期选择的育种方法,该法简单、快速、低成本、不需要特殊的仪器,适合实验的需要。
附图说明
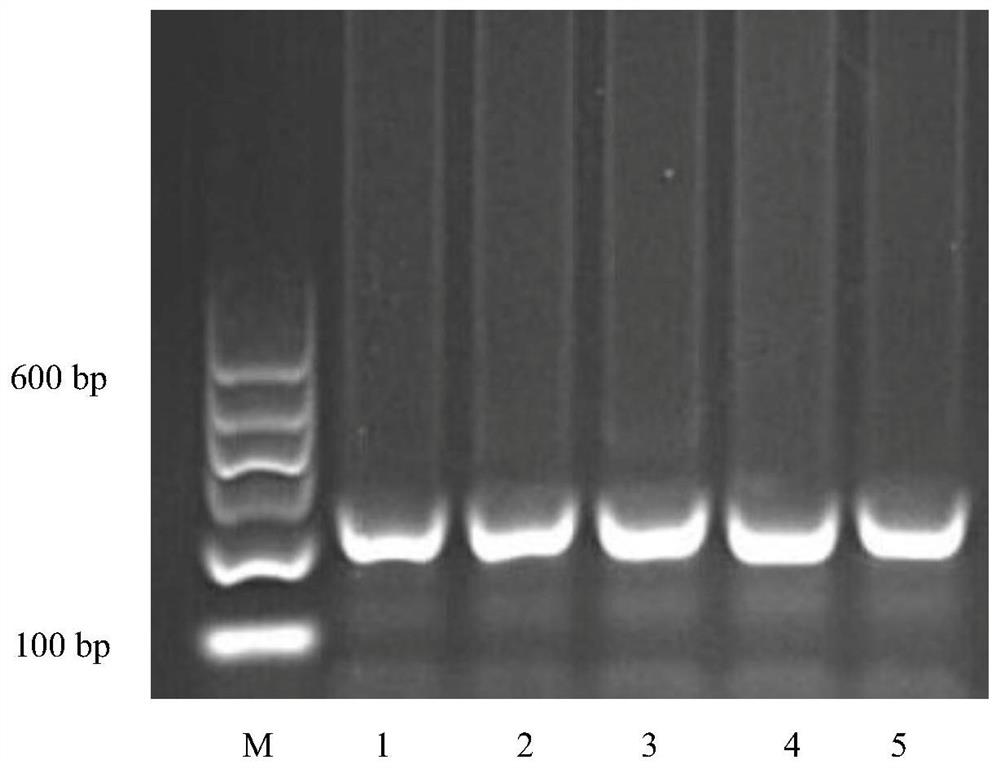
图1为部分样品PCR扩增产物的琼脂糖凝胶电泳图(M为DNA marker;1-5为PCR扩增产物琼脂糖凝胶电泳图);
图2为部分样品PCR扩增产物的聚丙烯酰胺凝胶电泳图;
图3为鸡PIK3R1基因组第579位不同基因型个体的测序图。
具体实施方式
下面结合附图对本申请作进一步详细描述,有必要在此指出的是,以下具体实施方式只用于对本申请进行进一步的说明,不能理解为对本申请保护范围的限制,该领域的技术人员可以根据上述申请内容对本申请作出一些非本质的改进和调整。
1、材料
本实施例所用方法如无特别说明均为本领域的技术人员所知晓的常规方法,所用的试剂等材料,如无特别说明,均为市售购买产品。
2、方法
2.1获得鸡PIK3R1基因多态位点
2.1.1基因组DNA提取与检测
选取449只皖南三黄鸡为试验材料,进行鸡翅静脉采血,利用生工生物公司生产的血液DNA提取试剂盒提取鸡翅静脉血样中总DNA,具体参照试剂盒使用说明书进行。
用NanoDrop2000测量DNA浓度及OD值。用1.5%琼脂糖凝胶电泳检测DNA,结果如图1所示,提取的基因组DNA质量较好,主带单一、清晰。
2.1.2引物设计
从鸡基因组数据库中找到SEQ ID NO.1所示的基因PIK3R1对应的DNA序列,以SEQID NO.1所示基因PIK3R1的部分DNA序列外显子为模板,在引物设计的过程中注意尽量将SNP位点设于中间位置,避免发夹结构、引物二聚体和错配等情况的发生,使引物序列最优化,引物序列如下所示:
F1:TTCAGCGATAAACTCCGGGC(SEQ ID NO.2)
R1:TGTCACCAGACCACTCCTGT(SEQ ID NO.3)
该引物的扩增区域的长度为235bp序列,其中包含第579位的分子标记位点。
2.1.3 PCR扩增
利用已合成的测序特异性引物对PIK3R1基因的目的片段进行PCR扩增反应,PCR扩增体系为:
PCR反应条件为:94℃预变性5min;94℃变性30s,52℃复性30s,72℃延伸30s,共34个循环;然后72℃延伸10min;最后4℃保存。
2.1.4 PCR扩增产物检测
利用1.5%质量比的琼脂糖凝胶电泳检测PCR扩增产物,在凝胶成像仪成像后获得一条长度略大于200bp的条带,如图1所示,与预测的长度一致,说明获得了目的片段。将PCR产物送到上海生工生物工程测序,与预测结果一致。
2.1.5 PCR产物变性及SSCP检测
对PCR扩增后的产物首先变性,然后进行聚丙烯酰胺凝胶检测,最终根据不同的带型结果判定其突变,具体步骤如下:
(1)根据说明配置非变性聚丙烯酰胺胶体,非变性丙烯酰胺凝胶体系如下:
(2)TEMED最后加入,加入后立即倒入模具中(浇灌前要确保模具封闭,防止灌胶时漏胶,以及胶条和孔梳的尺寸要一致),倾斜约45°角,胶液从垂直板中间(可有效避免气泡产生)缓慢倒入,当浇灌至约离模具上沿时停止灌胶,插入事先准备好的梳子,室温聚合40分钟,多余的丙烯酰胺4℃保存,随时观察玻璃板凝胶聚合情况,并且补加适量非变性丙烯酰胺凝胶体系混合液。
(3)在凝胶聚合等待过程时准备电泳卡槽,等聚合完成后安装好玻璃板向电泳卡槽中加入1×TBE,致TBE溶液超过加样孔约3cm,并赶走其中气泡。
(4)取3μL的PCR扩增产物置于PCR管中,加入7μL变性试剂,短暂离心混匀,98℃变性10min,迅速取出放入20℃冰盒10min,用10μL移液管点样。
(5)打开电源,先220伏电泳10min,然后将电压调制120V,电泳21h。
(6)电泳结束后关上电泳仪,拿出玻璃板,小心取出凝胶,放入盛有清水的白瓷盘中清洗1-2次。
(7)将凝胶放入染色液中避光轻摇15min。染色液:由硝酸银和纯水组成,硝酸银浓度为0.2%。
(8)染色结束,回收AgNO
(9)显色液显色,使条带清晰,背景淡黄,染色时间使得条带清晰可见为止,显色后立即倒掉显色液。显色液:500mL,其中2%NaOH+0.04%Na
(10)拍照,保存。
2.1.6基因分型
PCR-SSCP图片如图2所示,不同的带型表示不同的基因型,从图2中可以看出PIK3R1基因第579位存在三种基因型,即TT、GG、TG,具体如下:
(1)包含1条条带,则为GG型;
(2)包含2条条带,则为TT型;
(3)包含3条条带,则为TG型。
2.1.7 DNA验证测序
将显色的基因分型胶图进行统计,获得TT、GG、TG三种分型,对这三种分型分别挑选一个个体进行测序比对,测序比对图如图3所示;测序结果中G突变成T,箭头标出了突变位置,与PCR-SSCP结果一致。
2.2 PIK3R1基因G579T突变位点与皖南三黄鸡的鸡饲料利用率性状的关联分析
2.2.1基因分型
为确定鸡PIK3R1基因外显子579位的G/T多态性与鸡重要表型性状关联性,以
2.1.1中的449只皖南三黄鸡为试验材料,测统计56-98日龄采食量(ADFI)、平均日增重(ADG)、代谢体增重(MBW
采用2.1.5的基因分型方法,对449只皖南三黄鸡,进行基因分型,结果如表1。
表1不同表型个体基因型检测结果
卡方检验结果显示,试验鸡群体基因型处于Hardy-Weinberg平衡(P>0.05)。
2.2.2统计分析
用SAS9.4软件中最小二乘分析法分析三种基因型与鸡饲料利用率性状之间的关联性,不同基因型与各性状间的关联分析结果见如表2所示:
表2鸡PIK3R1基因型与鸡饲料利用率性状关联分析
注:同行不同小写字母表示差异显著(P<0.05)。
表2中可以看出,通过不同基因型个体饲料效率性状比较得出,在RFI方面,GG和TG个体显著低于TT个体,而GG个体与TG个体差异不显著;在ADFI方面,GG和TG个体显著低于TT个体,GG个体与TG个体差异不显著;在FCR方面,GG和TG个体显著低于TT个体,TG个体略高于GG个体,但GG与TG个体差异不显著。得出结论,GG基因型个体饲料利用率性状最好,TG基因型个体饲料利用率性状中等,TT基因型个体饲料利用率性状较差。
以上所述实施例仅表达了本发明的几种实施方式,其描述较为具体和详细,但并不能因此而理解为对本发明专利范围的限制。应当指出的是,对于本领域的普通技术人员来说,在不脱离本发明构思的前提下,还可以做出若干变形和改进,这些都属于本发明的保护范围。
序列表
<110> 安徽农业大学
<120> 一种基于PIK3R1基因鉴定鸡饲料利用率性状的分子标记及其鉴定方法和应用
<141> 2021-10-11
<160> 3
<170> SIPOSequenceListing 1.0
<210> 1
<211> 1610
<212> DNA
<213> 鸡(chicken)
<400> 1
ccctttgttg tggttatttt cggtagctgg ttttctgctg ccgttgagga tagtgaggat 60
gaagcattcg gattgctttc cacagcactc tgctctggaa tcaatagcct gtcctgcgtg 120
tgtttcccaa agaggtctga cagagcagca gtccatgatg ggggcttagg aagaatgttt 180
ctgagcacca ggagtacacg ggaggcttaa agtcaagttc ggaagaaaat gctgacaagg 240
actcttgctt ttaaaaacag ataacttggt gaatatctca gtgagaacag ggtaaagaaa 300
cggtgtgaaa aggtaaactc aatcctgctg tatcctgacg gactggcatt ttgtattcag 360
cagaatcatt tttcaagatc ttaactcagc tttttacttt cttccatagt gtaggagagc 420
ctggtgaatg caaggttgca gtattcgatg cttaggtttt gaaatgagtt agtccggatc 480
agttggaatt gtattttatt atgaagtgat agcgggggga ggcatctatt gcagtacgtc 540
cactttctat agtaattcta ggagcatcag gtctgtgtgc ccattaaaat acagaaatct 600
gtacagtttg aaaacctaat ccatatttca ttatgtggat acatacatgc ctaagtacgt 660
gtgtgtgtaa cggccacata acggatacgg acacacacaa acacgcatgc agagtcatgc 720
tattattccc ttcctcatca ttaacacgta cagaaaccct ccaaagcctt ttcagcgata 780
aactccgggc atctgagtag gaaaatgaat ctgagaagtg acctacctat ccataaaggg 840
agctgcgctg tgtgtgagcg tgcatgtctt gtgtgcgcgc gtgtgtgaga gctacgtgct 900
gcagcatatc agccttttgt catcctgcca gccttggact gtaccatgct gcacagtcag 960
gaaaatgaaa tggagacaag ggtgtgcgag gagagcccgc tcggtctttt tgcttgtaat 1020
accacgttta tattgcagtt ctgctaaatg caggctcttt ctgaaactga aattgtttta 1080
agattaatgt agagcaaaat gatgaaacag gagtggtctg gtgacattat tttgacatga 1140
ttggctgagg attatgatgt aataggttac agtgcagccc cttataggtt ttaaaatgaa 1200
ttccaagaca ccattacaaa gagagcctga ctctttcctt gtatctgagc ttactcagtg 1260
aaactcatac aaatgtacaa tactgtttgg aatatggaag aactggatat agaatatcct 1320
aagacagata taaactgtgg cacagacttg atgttttaca tagaaatgga tccaccaggt 1380
aaaactgcag tcttattatg gaagacttaa ttctttttgg tctgtatggt agcagctgaa 1440
aaattcttag tggatatgta ggatttattt acattgtcat tgatgcaaat actttttcct 1500
ttttttcctt aataataatt aagttgtgtt ttagtgcatg gttatagtaa ctagaatcat 1560
aacctgtaga aatacctgcc tgcttactta attgattttt ttcggaattg 1610
<210> 2
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 2
ttcagcgata aactccgggc 20
<210> 3
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 3
tgtcaccaga ccactcctgt 20
- 一种基于PIK3R1基因鉴定鸡饲料利用率性状的分子标记及其鉴定方法和应用
- 一种基于MyoD基因鉴定鸭腿肌性状的分子标记及其方法和应用
