FLO19基因在提高稻米品质中的应用
文献发布时间:2023-06-19 12:08:44
技术领域
本发明涉及水稻分子育种领域,涉及FLO19基因在调节和筛选稻米品质中的应用,尤其是Hap2这种单倍型在调节和筛选稻米品质上的应用。
背景技术
水稻是世界三大粮食作物之一,产量一直是育种者关注的最重要的性状。随着生活水平的提高,消费者越来越关注大米的品质,特别是蒸煮食味品质(CEQ)和外观品质。然而,目前为止,只有非常少的水稻蒸煮食味品质和外观品质自然变异的基因被克隆。因此,通过各种方法挖掘更多能够同时提高大米蒸煮食味品质和外观品质的基因资源对优良水稻品种的选育和改良具有重要意义。
粉质胚乳是指胚乳淀粉颗粒排列疏松,籽粒外观呈现不透明的一种表型,主要包括部分粉质化和全部粉质化两大类。粉质胚乳往往导致大米外观品质不佳,因此,有研究者对导致胚乳粉质化的相关基因进行研究和鉴定,以期应用于育种。
发明内容
本发明的发明人通过对533个不同栽培稻品种的粉质胚乳表型进行全基因组关联分析,检测到了一批跟稻米粉质胚乳表型相关的候选基因,并从中筛选出基因FLO19,该基因存在11个单倍型,其中单倍型Hap2在四种主要的单倍型(Hap1-4)中具有最低的垩白率、直链淀粉含量和糊化温度(以碱消值表示),以及最高的胶稠度和食味值。我们通过基因编辑的方法将FLO19等位基因Hap2突变,得到两个突变株(flo19-1和2)。结果发现,两个突变株均产生了外观劣化的表型。相反地,我们对其他单倍型的稻种进行编辑,使其突变成FLO19单倍型Hap2,结果显示,突变后的水稻品种的籽粒中垩白率显著降低。此外,我们还意外地发现,flo19-2在食味品质方面发生了显著提高。这提示我们,可通过这种基因编辑方法在不考虑外观的情况下获得更好的食味值。
基于以上研究,本发明提供了FLO19基因单倍型Hap2在提高稻米品质中的应用,所述FLO19基因单倍型Hap2的基因序列如SEQ ID NO:3所示。
在一个具体实施方案中,所述稻米品质包括蒸煮食味品质和/或外观品质。
本发明还提供了一种提高稻米品质的水稻育种方法,包括对生产所述稻米的稻种导入FLO19基因单倍型Hap2的步骤,所述FLO19基因单倍型Hap2的基因序列如SEQ ID NO:3所示。
在一个具体实施方案中,通过杂交的方法向所述稻种中导入所述FLO19基因单倍型Hap2。目前国家暂时未开放转基因水稻品种商业化,因此,可通过杂交的方法来将FLO19基因单倍型Hap2与其他优势性状聚合,得到具有更好外观和蒸煮食味品质的水稻品种。
在一个具体实施方案中,通过基因编辑的方法向所述稻种中导入所述FLO19基因单倍型Hap2。通过基因编辑的方法,可将其他单倍型的FLO19基因突变成Hap2,简单直接,育种时间短。
本发明还提供一种提高稻米食味值的水稻育种方法,包括对生产所述稻米的稻种的FLO19基因进行基因编辑的步骤,使所述FLO19基因突变成FLO19-2,所述FLO19-2编码的氨基酸序列如SEQ ID NO:7所示。
在一个具体实施方案中,所述FLO19-2的核酸序列如SEQ ID NO:8所示。
在一个具体实施方案中,所述稻种中的原始FLO19基因为FLO19基因单倍型Hap2。
将FLO19基因单倍型Hap2突变成氨基酸序列如SEQ ID NO:7的180个氨基酸的基因,得到的突变株尽管在外观上发生了劣化,垩白率升高,但是在食味值等蒸煮品质方法出乎意料地显著提高了。通过该方法可培育出垩白率等外观参数不高,但是蒸煮食味品质显著提高的水稻品种。
本发明提供了一种新的基因可影响和调节水稻籽粒的外观性状和食味品质性状,为水稻育种提供了新的方法和路径。
附图说明
图1为533份栽培稻的全基因组关联分析结果;
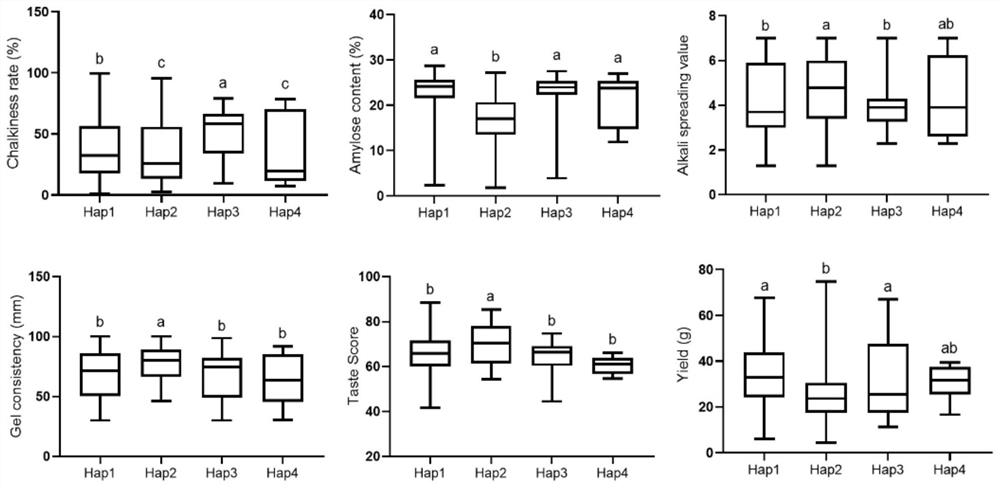
图2为Hap1-4稻种籽粒的垩白率、直链淀粉、碱消值、胶稠度、食味值和产量的统计图;
图3为Hap1-4稻种籽粒的蛋白含量、抽穗期、千粒重、每穂颖花数、结实率的统计图;
图4为ZH11野生型和突变株的籽粒的外观照片(A)、横截面照片(B)、胚乳外层显微照片(C)和胚乳细胞中淀粉体的透射电镜照片(D);
图5为ZH11野生型和突变株的株高统计图;
图6为ZH11野生型和突变株的籽粒的相关性状,其中,A为灌浆开始后不同时期的籽粒照片;B为灌浆开始后不同时期的千粒糙米干重统计图;C千粒重统计图;D为粒长、粒宽和粒厚统计图;
图7为ZH11野生型和突变株的籽粒中直链淀粉含量统计图(A)、糖类含量统计图(B)和储藏蛋白统计图(C);
图8为ZH11野生型和突变株的籽粒中胶稠度统计图(A)、黏度统计图(B)和碱消值统计图(C)。
具体实施方式
以下结合附图对本发明的原理和特征进行描述,所举实例只用于解释本发明,并非用于限定本发明的范围。
1、样品来源
本发明使用的水稻包括533个栽培稻品种构成的自然群体,其中,533个栽培稻品种包括50个aus品种(aus)、305个籼稻品种(indica)和178个粳稻品种(japonica)。
中花11(ZH11)是由中国农业科学院作物科学研究所选育的粳型常规水稻。flo19-1和flo19-2是以中花11为受体采用农杆菌侵染法构建的FLO19位点的敲除材料。
所有材料均在武汉或者海南的华中农业大学的试验田中栽植生长,成熟收获后的稻谷晾干后室温储藏至少三个月,然后用于实验。
2、粉质胚乳表型的全基因组关联分析
群体结构和全基因组LD分析的基因型数据是从全基因组随机选取的100万个出现在所有品种中SNPs,我们利用贝叶斯聚类软件fastSTRUCTURE来计算533份核心种质材料不同K(1-10)取值的群体结构。然后利用该软件配套的python脚本K.py来选择最佳的K值。群体机构的可视化是利用配套脚本distruct.py来实现的。全基因组LD的计算利用PLINK软件来计算SNPs间的点配对r
分别在籼稻亚群,粳稻亚群和总533份核心种质进行全基因组关联分析。简单线性模型和混合线性模型都是利用软件FaST-LMM进行分析,多位点的混合线性模型(MLMM)用到的R包来自Segura。关联分析中的显著性阈值基于Bonferroni修正(ref),将籼稻亚群,粳稻亚群和总核心种质群体的阈值分别设定为8.7×10
考察包含533份水稻种质材料的自然群体中的粉质胚乳表型,结果显示,籼稻亚群中有31个粉质胚乳品种,粳稻亚群中有22个粉质胚乳品种。对相关数据进行粉质胚乳表型的全基因组关联分析,得到343个显著性位点,分布在12条染色体上(图1)。
对上述位点进行逐一敲除验证,结果我们发现在三号染色体上存在一个编码谷氨酰胺转移酶的基因(LOC_Os03g48060)(编码293个氨基酸,氨基酸序列如SEQ ID NO:1所示,编码氨基酸的核酸序列如SEQ ID NO:2所示),该基因的敲除导致水稻籽粒不透明,以及胚乳外层粉质化,将该基因其命名为FLO19。
3、大米相关参数的测定
测定方法如下:
1)蛋白质含量的测定:糙米的蛋白质含量的测定采用凯氏定氮法,具体步骤如下:首先,在干燥的消化管中依次加入0.5±0.005g精米粉、8.5g催化剂(硫酸钾和五水硫酸铜质量比为9:1)和12ml浓硫酸;然后,将消化管置于通风橱中消化炉FOSS DT208上420℃消化1.5h,冷却至无浓烟冒出;最后,将消化管置于FOSS Kjeltec 8400上进行自动分析。每个单株样品重复测量3次,其平均值即为最终的蛋白质含量。1892S/RH003 RIL群体、FLO19和Ghd8的NIL以及转基因材料均采用此法测定蛋白质含量。
2)四种储藏蛋白的提取和测定:
提取顺序:清蛋白—球蛋白—醇溶蛋白—谷蛋白(每个材料3次重复测量)。
称取0.1g(脱脂)米粉(过0.05mm筛子,270目),脱脂。
加入清蛋白提取溶液(1:10,w/v)1mL,震荡混匀,室温摇床2h。4℃条件下,12000rpm(/g)离心15min。吸上清为清蛋白。重复3次测量,混合3次上清液即为清蛋白总含量。
加入球蛋白提取溶液(1:10,w/v)1mL,震荡混匀,室温摇床2h。4℃条件下,12000rpm离心15min。吸上清为球蛋白。吸上清为清蛋白。重复3次测量,混合3次上清液即为球蛋白总含量。
加入醇溶蛋白提取溶液(1:10,w/v)1mL,震荡混匀,室温摇床2h。4℃条件下,12000rpm离心15min。吸上清为醇溶蛋白。吸上清为清蛋白。重复2次测量,混合2次上清液即为醇溶蛋白总含量。
加入谷蛋白提取溶液(1:10,w/v)1mL,震荡混匀,室温(50℃摇效果更好!)摇床2h。4℃条件下,12 000rpm离心15min。吸上清为谷蛋白。吸上清为谷蛋白。重复4~5次测量,混合4~5次上清液即为谷蛋白总含量。
上述得到的四种上清蛋白质溶液于-20℃短期保存以备测定,长期保存为-70℃。
3)直链淀粉含量的测定:
精米粉直链淀粉含量的测定参考国标NY/T2639-2014并做了简单的调整,具体步骤如下:首先,在干燥的15ml玻璃管中依次加入10±0.5mg的精米粉、0.1ml 95%的乙醇和0.9ml 1M的氢氧化钠,依次混匀后拧上盖子,沸水浴10min后冷却至室温,加入9ml单蒸水稀释;然后,将0.5ml稀释液加入一个新的15ml玻璃管,依次加入9.25ml单蒸水、0.2ml 1M乙酸和0.15ml0.2%碘-碘化钾溶液,拧上盖子,上下颠倒充分混匀后静置20min;最后,将0.2ml上述混合液和四个直链淀粉含量的标准样品(0.4%,10.6%,16.2%和26.5%)的相同处理混合液分别加入透明的ELISA平板,利用Tecan Infinite M200型多功能酶标仪在620nm波长处测定吸光度,根据标准样品的吸光度和直链淀粉含量的线性方程计算出每个样品的直链淀粉含量。每个单株样品重复测量3次,其平均值即为最终的直链淀粉含量。
4)垩白率的测定:
使用万深SC-E型大米外观品质检测分析仪,对每个单株的200-300的精米粒扫描得到图像,并对图像分析得到垩白率的数值。
5)碱消值(糊化温度)的测定:
水稻种子成熟收获后晒干或者烘干,然后将种子进行脱粒处理。种子在室温保存3个月后利用稻谷出糙机上脱壳得到糙米。糙米在精米机上经过除去胚和糊粉层的处理,即为精米。挑选6粒成熟并且饱满的整精米放置于方盒内,加入10.0ml 1.70%的氢氧化钾(KOH)溶液。用枪头或者玻璃棒将盒内米粒排布整齐、均匀,然后加盖。将方盒平稳移至30±2℃的恒温箱内,保温约23小时左右。把方盒平稳地取出,逐粒观察米粒胚乳的分解情况,按下表1进行分级记录。
表1.碱消值(糊化温度)分级标准
稻米样品的碱消值计算:碱消值=∑(G·N)/6,其中G表示每粒米的级别,N表示同一级的米粒数。
在测定每批样品糊化温度的同时,用已知糊化温度的标准样品一套(包括高、中、低三种糊化温度)作为内标样品一起进行测定。
6)蒸煮食味品质相关参数的测定:
每种基因型的材料至少准备150g以上的精米,使用米粒食味计测定颗粒状态下的食味品质。每个样品设置10个生物学重复。
7)胶稠度的测定:
水稻种子成熟收获后晒干或者烘干,然后将种子进行脱粒处理。种子在室温保存3个月后利用稻谷出糙机上脱壳得到糙米。糙米在精米机上经过除去胚和糊粉层的处理,即为精米。将糙米或者精米在旋风式粉碎机上粉碎成米粉。称取通过200目筛的米粉试样4份,每份100mg(按含水量12%计)于试管中。加入0.2mL 0.025%百里香酚蓝溶液(0.125g溶于95%乙醇中),并轻轻摇动试管,使米粉充分分散,再加2.0mL 0.2M KOH溶液,并摇动试管,置于涡旋振荡器上使米粉充分混合均匀,紧接着迅速把试管放入沸水浴中(最好是边震边放),用玻璃弹子球盖好试管口,加热8min,控制试管内米胶溶液面在加热过程中维持在试管高度的三分之一至二分之一(在煮的时间段内要注意使用电吹风对锅中的试管吹冷风降温,防止管内米胶溶液溢出)。取出试管,拿去玻璃弹子球,室温静置冷却5min后,再将试管放在0℃左右冰水浴中冷却20min取出(控制好时间)。立即水平放置在铺有坐标纸(坐标纸上方最好铺上一大块透明玻璃),事先调好水平的操作台上,在室温25±2℃下静置1h,即时测量米胶在试管内流动的长度(mm),双试验结果允许差不超过7mm,取其平均值,即为检验结果。
8)RVA谱数据:
精米粉的RVA谱性状由快速粘度分析仪(Rapid Visco Analyser)测量。根据AACC61-02标准方法,首先称量3.0g精米粉于专用铝罐中,加入25g蒸馏水后搅拌均匀,放置于调平并预热半小时以上的RVA仪进行分析测定。从RVA谱可以直接得到四个性状,分别是最高黏度(peak viscosity,PV),热浆黏度(hot paste viscosity,HPV),冷浆黏度(coolpaste viscosity,CPV)和最大黏度时间(Peak time,PT)。同时还可以根据计算获得3个衍生性状:崩解值(breakdown,BD=PV-HPV),消减值(setback,SB=CPV-PV)和起浆温度(Pasting temp,PaT)。
4、FLO19单倍型分析
我们利用533份核心种质材料FLO19基因CDS区的变异信息对该基因进行了自然变异分析,并与每个水稻品种的大米相关参数进行关联分析。
结果显示,533份水稻品种总共可以划分为11种不同的单倍型,以Hap1-4四种单倍型为主。其中,含有单倍型Hap2(核酸序列如SEQ ID NO:3所示,编码的氨基酸序列如SEQ IDNO:4)的水稻籽粒在四种单倍型中拥有最低的垩白率、直链淀粉含量和糊化温度(消减值),而它的胶稠度和食味值却是四种单倍型中最高的(图2)。四种单倍型之间的蛋白质含量和抽穗期没有显著差异,Hap2的千粒重最高,不过产量性状每穗颖花数和结实率较低(图3)。
由此可见,FLO19基因单倍型Hap2是一种能够同时提高稻米外观品质和蒸煮食味品质的优异单倍型,可在优质稻培育和品质改良方面具有极大的应用价值。
例如,可将其他单倍型的FLO19基因突变成Hap2,从而提高相应水稻中的籽粒的外观性状和食味值,我们已经对水稻品种9311(单倍型Hap1)进行了这样的突变,突变株获得的更好的外观性状和食味值。
此外,我们还可以通过杂交的方法,将单倍型Hap2的FLO19基因导入到不含该单倍型的水稻中,以聚合两者的性状。
5、flo19基因及含有该基因突变体的突变株
我们使用CRISPR-Cas9技术对ZH11(FLO19基因的单倍型为Hap2)的FLO19基因进行编辑,根据靶点的不同得到了两个突变株flo19-1和flo19-2。其中,flo19-1中在编辑位点产生了1个碱基的缺失,导致了移码突变和提前终止,最终编码产物长度为150个氨基酸(氨基酸序列如SEQ ID NO:5所示,核酸序列如SEQ ID NO:6所示);flo19-2中在编辑位点产生了2个碱基的缺失,导致了移码突变和提前终止,最终编码产物长度为180个氨基酸(氨基酸序列如SEQ ID NO:7所示,编码核酸序列如SEQ ID NO:8所示)。
突变株flo19-1和flo19-2具有相同的籽粒粉质特征,如图4所示,ZH11外胚乳细胞中淀粉颗粒排列致密而规则(图4C)。与之相比,突变株的籽粒中,胚乳外层粉质化(图4A、B),外胚乳细胞被松散排列的复合淀粉颗粒包裹(图4C)。使用透射电子显微镜的观察灌浆15天的籽粒胚乳,结果显示,ZH11的胚乳细胞中淀粉体排列紧凑而规则,而突变体胚乳细胞中淀粉体呈现支离破碎的状态,淀粉颗粒散乱分布于淀粉体之间的基质中(图4D)。由此可见,flo19的突变会劣化其外观性状。6、突变株的其他性状比较
6.1、农艺性状
对两个突变株的株高进行统计,结果如图5所示,两个突变株的株高与ZH11相比均明显降低。
采集突变株和野生型ZH11不同灌浆时期的胚乳进行对比观察,结果如图6所示,从灌浆第7天开始,两个flo19突变体的灌浆速率均显著低于ZH11,在灌浆第23天达到最大值(图6A、B)。另外,我们发现,从灌浆第11天开始两个突变体的胚乳就要显得比对照更加扁平,并且这种充实度上的差异随着时间的推移变得越来越明显(图6A),并最终体现在成熟籽粒千粒重和粒厚的显著降低上(图6C、D)。
我们还考察了突变体的分蘖数和每穗粒数,这两个性状相比对照并没有太大的区别。TTC染色实验结果显示,flo19-1的种子活力比起ZH11显著降低,而flo19-2没有明显变化。
6.2、突变株的稻米品质
我们对突变体和ZH11中的淀粉和蛋白质含量进行了分析。结果如图7所示,flo19-1的直链淀粉含量没有明显变化,而flo19-2的直链淀粉含量显著降低(图7A)。淀粉的合成起始于蔗糖的分解,所以我们也测定了灌浆7天胚乳中三大糖类的含量,flo19-1胚乳中的蔗糖、葡萄糖和果糖全显著减少,而flo19-2中蔗糖和葡萄糖没有变化,果糖含量显著增加(图7B)。flo19-1的总蛋白和谷蛋白增加,醇溶蛋白和清蛋白减少,球蛋白没有变化;flo19-2的总蛋白和谷蛋白比起对照没有统计学差异,醇溶蛋白、清蛋白和球蛋白减少(图7C)。
胚乳中储藏物质的变化很可能会引起蒸煮食味品质的变化,所以除了前面已经测定过的直链淀粉含量以外,我们还对突变体的糊化特性进行了分析。结果如图8所示,flo19-1的胶稠度相比ZH11并没有明显变化,而flo19-2的胶稠度有显著的增加(图8A)。与此结果相一致的是,flo19-2具有更高的崩解值,而flo19-1没有太大差异(图8B)。另外,两个突变体的糊化温度均没有很明显的变化(图8C)。
通过以上数据,flo19-1和flo19-2突变株在外观性状的劣化方面均产生了预期的表型。然而,出乎所有人意料的,flo19-2突变株虽然外观性状劣化,但是蒸煮食味品质却显著提高。
针对上述差异,我们进行进一步研究。结果显示,造成两个突变株之间的稻米品质差异的原因主要是因为flo19-2中的FLO19突变体蛋白比flo19-1多出30个氨基酸,这30个氨基使得在C端形成了一个LCR结构域,该LCR结构域与FLO19蛋白本身的LCR结构域不大一样,虽然不能修复外观的劣化,但是对直链淀粉含量、胶稠度和崩解值有产生影响,提高了食味值。
基于上述实验,我们可以通过将含有Hap2的水稻品种进行以flo19-2突变株的方式进行编辑,用以提高食味值。
以上所述仅为本发明的较佳实施例,并不用以限制本发明,凡在本发明的精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
序列表
<110> 华中农业大学
<120> FLO19基因在提高稻米品质中的应用
<160> 8
<170> SIPOSequenceListing 1.0
<210> 1
<211> 293
<212> PRT
<213> Oryza sativa
<400> 1
Met Gly Met Gly Pro Leu Glu Asn Ala Val Val Asp Ala Ala Ala Glu
1 5 10 15
Ala Val Ala Ala Lys Ala Ala Val Gly Gly Val Gly Val Gly Gly Ser
20 25 30
Tyr Ala Val Leu Gln Cys Gly Glu Asp Ser Glu Tyr Val Arg Lys Ala
35 40 45
Tyr Gly Gly Tyr Phe Glu Val Phe Arg Ala Leu Leu Ala Glu Asp Gly
50 55 60
Glu Arg Trp Arg Val Tyr Arg Ala Val Arg Gly Glu Leu Pro Gly Glu
65 70 75 80
Glu Glu Ala Ala Gly Ile Asp Gly Phe Val Ile Ser Gly Ser Cys Ser
85 90 95
Asp Ala His Ala Asp Asp Pro Trp Ile Val Ala Leu Val Asp Leu Ile
100 105 110
Arg Arg Gln Asn Ala Ala Gly Lys Arg Ile Leu Gly Val Cys Phe Gly
115 120 125
His Gln Val Leu Cys Arg Ala Leu Gly Gly Lys Thr Gly Arg Ser Lys
130 135 140
Lys Gly Trp Asp Ile Gly Val Asn Cys Ile His Pro Thr Ala Ala Met
145 150 155 160
Ala Arg Leu Phe Ser Pro Ile Lys Leu Pro Val His Met Pro Ile Ile
165 170 175
Glu Phe His Gln Asp Glu Val Trp Glu Leu Pro Pro Gln Ala Glu Val
180 185 190
Leu Ala Arg Ser Asp Met Thr Gly Val Glu Met Phe Arg Leu Gly Asp
195 200 205
Arg Ala Met Gly Val Gln Gly His Pro Glu Tyr Ser Lys Asp Ile Leu
210 215 220
Met Ser Ile Ala Asp Arg Leu Leu Arg Asn Asp Leu Ile Leu Asp His
225 230 235 240
Gln Val Asp Lys Ala Lys Ala Ser Phe Asp Leu Arg Gln Pro Asp Lys
245 250 255
Asp Leu Trp Lys Lys Val Cys Arg Gly Phe Leu Lys Gly Arg Leu Gln
260 265 270
Ser Ser Gln Gln Pro Gln Gln Gln Gln His Gln Lys Gln Gln Lys Ala
275 280 285
Ala Gln Leu Val Leu
290
<210> 2
<211> 882
<212> DNA
<213> Oryza sativa
<400> 2
atggggatgg ggccactaga gaacgccgtg gtggacgcgg cggcggaggc cgtggcggcg 60
aaggccgcgg tcggcggcgt gggcgtgggg ggttcgtacg cggtgctgca gtgcggggag 120
gactcggagt acgtgaggaa ggcgtacggg ggctacttcg aggtgttcag ggcgctgctg 180
gcggaggacg gcgagcggtg gcgggtgtac cgcgccgtgc gcggggagct ccccggggag 240
gaggaggccg cggggatcga cgggttcgtc atctccggga gctgcagcga cgcccacgcc 300
gacgacccgt ggatcgtcgc cctcgtcgac ctcatccgcc gccagaacgc cgccggcaag 360
cgcatcctcg gcgtctgctt cggccaccag gtcctgtgcc gagcgctggg cggcaagacg 420
ggccgatcga agaagggctg ggacatcggc gtgaactgca tccaccccac cgcggccatg 480
gcgaggctct tttcgccgat caagctcccc gtccacatgc ccatcatcga gttccaccaa 540
gatgaggtgt gggagctgcc tcctcaggcc gaggtgctgg cgcggtcgga catgaccggc 600
gtcgagatgt tccgtctcgg cgaccgcgcc atgggcgtcc agggccaccc ggagtacagc 660
aaggacatcc tcatgagcat cgccgaccgt ctcctccgga acgatctcat cctggatcac 720
caggtggata aggcgaaggc gagcttcgac ttgcggcaac cagacaagga tctgtggaag 780
aaggtgtgca ggggttttct gaaggggagg cttcagtcat cgcagcagcc gcagcagcag 840
caacatcaga agcagcagaa ggcggcgcag ctggtgctat ag 882
<210> 3
<211> 882
<212> DNA
<213> Oryza sativa
<400> 3
atggggatgg ggccactaga gaacgccgtg gtggacgcgg cggcggaggc cgtggcggcg 60
aaggccgcgg tcggcggcgt gggcgtgggg ggttcgtacg cggtgctgca gtgcggggag 120
gactcggagt acgtgaggaa ggcgtacggg ggctacttcg aggtgttcag ggcgctgctg 180
gcggaggacg gcgagcggtg gcgggtgtac cgcgccgtgc gcggggagct ccccggggag 240
gaggaggccg cggggatcga cgggttcgtc atctccggga gctgcagcga cgcccacgcc 300
gacgacccgt ggatcgtcgc cctcgtcgac ctcatccgcc gccagaacgc cgccggcaag 360
cgcatcctcg gcgtctgctt cggccaccag gtcctgtgcc gagcgctggg cggcaagacg 420
ggccgatcga agaagggctg ggacatcggc gtgaactgca tccaccccac cgcggccatg 480
gcgaggctct tttcgccgat caagctcccc gtccacatgc ccatcatcga gttccaccaa 540
gatgaggtgt gggagctgcc tcctcaggcc gaggtgctgg cgcggtcgga catgaccggc 600
gtcgagatgt tccgtctcgg cgaccgcgcc atgggcgtcc agggccaccc ggagtacagc 660
aaggacatcc tcatgagcat cgccgaccgt ctcctccgga acgatctcat cctggatcac 720
caggtggata aggcgaaggc gagcttcgac ttgcggcaac cagacaagga tctgtggaag 780
aaggtgtgca ggggttttct gaaggggagg cttcagtcat cgcagcagcc gcagcagcag 840
caacatcaga agcagcagaa ggcggcgcag ctggtgctat ag 882
<210> 4
<211> 293
<212> PRT
<213> Oryza sativa
<400> 4
Met Gly Met Gly Pro Leu Glu Asn Ala Val Val Asp Ala Ala Ala Glu
1 5 10 15
Ala Val Ala Ala Lys Ala Ala Val Gly Gly Val Gly Val Gly Gly Ser
20 25 30
Tyr Ala Val Leu Gln Cys Gly Glu Asp Ser Glu Tyr Val Arg Lys Ala
35 40 45
Tyr Gly Gly Tyr Phe Glu Val Phe Arg Ala Leu Leu Ala Glu Asp Gly
50 55 60
Glu Arg Trp Arg Val Tyr Arg Ala Val Arg Gly Glu Leu Pro Gly Glu
65 70 75 80
Glu Glu Ala Ala Gly Ile Asp Gly Phe Val Ile Ser Gly Ser Cys Ser
85 90 95
Asp Ala His Ala Asp Asp Pro Trp Ile Val Ala Leu Val Asp Leu Ile
100 105 110
Arg Arg Gln Asn Ala Ala Gly Lys Arg Ile Leu Gly Val Cys Phe Gly
115 120 125
His Gln Val Leu Cys Arg Ala Leu Gly Gly Lys Thr Gly Arg Ser Lys
130 135 140
Lys Gly Trp Asp Ile Gly Val Asn Cys Ile His Pro Thr Ala Ala Met
145 150 155 160
Ala Arg Leu Phe Ser Pro Ile Lys Leu Pro Val His Met Pro Ile Ile
165 170 175
Glu Phe His Gln Asp Glu Val Trp Glu Leu Pro Pro Gln Ala Glu Val
180 185 190
Leu Ala Arg Ser Asp Met Thr Gly Val Glu Met Phe Arg Leu Gly Asp
195 200 205
Arg Ala Met Gly Val Gln Gly His Pro Glu Tyr Ser Lys Asp Ile Leu
210 215 220
Met Ser Ile Ala Asp Arg Leu Leu Arg Asn Asp Leu Ile Leu Asp His
225 230 235 240
Gln Val Asp Lys Ala Lys Ala Ser Phe Asp Leu Arg Gln Pro Asp Lys
245 250 255
Asp Leu Trp Lys Lys Val Cys Arg Gly Phe Leu Lys Gly Arg Leu Gln
260 265 270
Ser Ser Gln Gln Pro Gln Gln Gln Gln His Gln Lys Gln Gln Lys Ala
275 280 285
Ala Gln Leu Val Leu
290
<210> 5
<211> 150
<212> PRT
<213> Oryza sativa
<400> 5
Met Gly Met Gly Pro Leu Glu Asn Ala Val Val Asp Ala Ala Ala Glu
1 5 10 15
Ala Val Ala Ala Lys Ala Ala Val Gly Gly Val Gly Val Gly Gly Ser
20 25 30
Tyr Ala Val Leu Gln Cys Gly Glu Asp Ser Glu Tyr Val Arg Lys Ala
35 40 45
Tyr Gly Gly Tyr Phe Glu Val Phe Arg Ala Leu Leu Ala Glu Asp Gly
50 55 60
Glu Arg Trp Arg Val Tyr Arg Ala Val Arg Gly Glu Leu Pro Gly Glu
65 70 75 80
Glu Glu Ala Ala Gly Ile Asp Gly Phe Val Ile Ser Gly Ser Cys Ser
85 90 95
Asp Ala His Ala Asp Asp Pro Trp Ile Val Ala Leu Val Asp Leu Ile
100 105 110
Arg Arg Gln Asn Ala Ala Gly Lys Arg Ile Leu Gly Val Cys Phe Gly
115 120 125
His Gln Val Leu Cys Arg Ala Leu Gly Gly Lys Thr Gly Arg Ser Lys
130 135 140
Lys Ala Gly Thr Ser Ala
145 150
<210> 6
<211> 453
<212> DNA
<213> Oryza sativa
<400> 6
atggggatgg ggccactaga gaacgccgtg gtggacgcgg cggcggaggc cgtggcggcg 60
aaggccgcgg tcggcggcgt gggcgtgggg ggttcgtacg cggtgctgca gtgcggggag 120
gactcggagt acgtgaggaa ggcgtacggg ggctacttcg aggtgttcag ggcgctgctg 180
gcggaggacg gcgagcggtg gcgggtgtac cgcgccgtgc gcggggagct ccccggggag 240
gaggaggccg cggggatcga cgggttcgtc atctccggga gctgcagcga cgcccacgcc 300
gacgacccgt ggatcgtcgc cctcgtcgac ctcatccgcc gccagaacgc cgccggcaag 360
cgcatcctcg gcgtctgctt cggccaccag gtcctgtgcc gagcgctggg cggcaagacg 420
ggccgatcga agaaggctgg gacatcggcg tga 453
<210> 7
<211> 180
<212> PRT
<213> Oryza sativa
<400> 7
Met Gly Met Gly Pro Leu Glu Asn Ala Val Val Asp Ala Ala Ala Glu
1 5 10 15
Ala Val Ala Ala Lys Ala Ala Val Gly Gly Val Gly Val Gly Gly Ser
20 25 30
Tyr Ala Val Leu Gln Cys Gly Glu Asp Ser Glu Tyr Val Arg Lys Ala
35 40 45
Tyr Gly Gly Tyr Phe Glu Val Phe Arg Ala Leu Leu Ala Glu Asp Gly
50 55 60
Glu Arg Trp Arg Val Tyr Arg Ala Val Arg Gly Glu Leu Pro Gly Glu
65 70 75 80
Glu Glu Ala Ala Gly Ile Asp Gly Phe Val Ile Ser Gly Ser Cys Ser
85 90 95
Asp Ala His Ala Asp Asp Pro Trp Ile Val Ala Leu Val Asp Leu Ile
100 105 110
Arg Arg Gln Asn Ala Ala Gly Lys Arg Ile Leu Gly Val Cys Phe Gly
115 120 125
His Gln Val Leu Cys Arg Ala Leu Gly Gly Lys Thr Gly Arg Ser Lys
130 135 140
Lys Leu Gly His Arg Arg Glu Leu His Pro Pro His Arg Gly His Gly
145 150 155 160
Glu Ala Leu Phe Ala Asp Gln Ala Pro Arg Pro His Ala His His Arg
165 170 175
Val Pro Pro Arg
180
<210> 8
<211> 543
<212> DNA
<213> Oryza sativa
<400> 8
atggggatgg ggccactaga gaacgccgtg gtggacgcgg cggcggaggc cgtggcggcg 60
aaggccgcgg tcggcggcgt gggcgtgggg ggttcgtacg cggtgctgca gtgcggggag 120
gactcggagt acgtgaggaa ggcgtacggg ggctacttcg aggtgttcag ggcgctgctg 180
gcggaggacg gcgagcggtg gcgggtgtac cgcgccgtgc gcggggagct ccccggggag 240
gaggaggccg cggggatcga cgggttcgtc atctccggga gctgcagcga cgcccacgcc 300
gacgacccgt ggatcgtcgc cctcgtcgac ctcatccgcc gccagaacgc cgccggcaag 360
cgcatcctcg gcgtctgctt cggccaccag gtcctgtgcc gagcgctggg cggcaagacg 420
ggccgatcga agaagctggg acatcggcgt gaactgcatc caccccaccg cggccatggc 480
gaggctcttt tcgccgatca agctccccgt ccacatgccc atcatcgagt tccaccaaga 540
tga 543
- FLO19基因在提高稻米品质中的应用
- 稻米品质指标在鉴别再生稻头季稻米和再生季稻米中的应用
