一种急性低氧不耐的分子标志物及遗传风险评估模型的构建方法
文献发布时间:2023-06-19 19:30:30
技术领域
本发明属于体外诊断技术领域,具体涉及一种急性低氧不耐的分子标志物及遗传风险评估模型的构建方法。
背景技术
低氧敏感症或低氧不耐症是低氧敏感人员进入低氧环境后出现的一种病理性反应,表现头晕痛、胸闷、气短、心悸、恶心、呕吐,甚至肺动脉压升高、肺水肿、脑水肿等症状。低氧敏感也被称为“高山病”,分为急性(acute mountain sickness,AMS)、亚急性(subacute mountain sickness)和慢性(Chronic Mountain Sickness,CMS)三种,亚急性和慢性通常又合称为CMS。Waeber(2015)报道,平原人快速进入海拔3000m以上地区,AMS发生率中位值高达60%
遗传因素是低氧敏感的重要风险因素。Azad等(2017)总结了低氧耐受的遗传基础和分子机制,认为该过程涉及的基因超过1000个,其中参与血管生成的43个基因、调解血液循环系统的56个基因以及维持红细胞稳态的14个基因与低氧耐受或低氧敏感关系最密切
尽管如此,这些关联SNPs的调控作用以及关联基因的变化规律仍不清楚。并且到目前为止并没有直接利用SNP预测低氧敏感风险的技术方案。因此,建立一个低氧不耐的遗传风险评价模型对于高原作业人员选拔提高相关岗位适应性具有重要意义。
参考文献
[1]WaeberB,KayserB,Dumout L,etal.Impact ofstudy design on reportedincidences ofacute mountain sickness:a systematic review.High Alt Med Biol,2015,16(3):204-215.
[2]Azad P,Stobdan T,Zhou D,et al.High-altitude adaptation in humans:from genomics to integrative physiology.J Mol Med(Berl).2017Dec;95(12):1269-1282.
[3]Stobdan T1,AkbariA2,Azad P1,et al.New Insights into the GeneticBasis ofMonge's Disease andAdaptationto High-Altitude.Mol Biol Evol.2017Dec1;34(12):3154-3168.
发明内容
有鉴于此,本发明的目的在于提供一种急性低氧不耐的分子标志物及其应用,通过将特异性23个突变位点进行组合能够准确表征急性低氧不耐的表型,为快速诊断急性低氧不耐人群提供了有力工具。
本发明的目的还在于提供一种基于所述分子标记物建立遗传风险评估模型的方法,建立的评估模型具有检测准确性高、特异性强的特点。
本发明提供了一种急性低氧不耐的分子标志物,包括以下hg19基因组版本中突变位点:
rs113459798、rs9662128、rs113147548、rs9874278、rs9814174、rs10512804、rs2974528、rs6926895、rs3739429、rs4634694、rs72805322、rs74910065、rs1471459475、rs1286147、rs189264、rs8038249、rs9935018、rs3809923、rs438703、rs3752194、rs1077806、rs10626088和rs11367927。
优选的,根据hg19基因组版本,所述rs113459798位于人第1号染色体,第40149875位碱基由C突变为T;
所述rs9662128位于人第1号染色体,第40150156位碱基由G突变为A
所述rs113147548位于人第2号染色体,第184176384位碱基由A突变为G;
所述rs9874278位于人第3号染色体,第43591134位碱基由C突变为A;
所述rs9814174位于人第3号染色体,第160588505位碱基由G突变为T;
所述rs10512804位于人第5号染色体,第5689218位碱基由T突变为C;
所述rs2974528位于人第5号染色体,第114462630位碱基由T突变为C;
所述rs6926895位于人第6号染色体,第89858851位碱基由G突变为A;
所述rs3739429位于人第8号染色体,第135524797位碱基由G突变为T;
所述rs4634694位于人第8号染色体,第139151375位碱基由C突变为T;
所述rs72805322位于人第10号染色体,第76857800位碱基由C突变为T;
所述rs74910065位于人第10号染色体,第99588179位碱基由T突变为C;
所述rs1471459475位于人第14号染色体,第42361506-42361510位碱基由ACACA突变为ACA;
所述rs1286147位于人第14号染色体,第91467567位碱基由A突变为C;
所述rs189264位于人第15号染色体,第86205478位碱基由G突变为A;
所述rs8038249位于人第15号染色体,第86253911位碱基由T突变为G;
所述rs9935018位于人第16号染色体,第8010933位碱基由T突变为C;
所述rs3809923位于人第18号染色体,第46447817位碱基由G突变为C;
所述rs438703位于人第19号染色体,第6692790位碱基由A突变为T;
所述rs3752194位于人第19号染色体,第14951889位碱基由A突变为G;
所述rs1077806位于人第19号染色体,第56114045位碱基由C突变为T;
所述rs10626088位于人第20号染色体,第15360664位碱基由C突变为CT;
所述rs11367927位于人第20号染色体,第55171020位碱基G缺失。
本发明提供了所述分子标志物在制备预测低氧不耐遗传风险的产品中的应用。
本发明提供了所述分子标志物在构建低氧不耐遗传风险评估模型中的应用。
本发明提供了一种低氧不耐遗传风险评估模型的构建方法,包括以下步骤:
对待测样本进行靶向高通量测序,得到所述分子标志物的突变位点信息;
以部分待检样本的低氧耐受情况和所述分子标志物的突变位点信息作为训练数据输入随机森林模型中,得到低氧不耐遗传风险评估模型。
优选的,在所述输入随机森林模型中后,还包括进行交叉验证。
优选的,所述交叉验证的方法为以剩余的待测样本的分子标志物的突变位点信息作为测试数据,进行多次交叉验证,得到受试者工作特征曲线;
从所述受试者工作特征曲线分析得到模型评估指数AUC,当模型评估指数AUC为0.7以上时,说明低氧不耐遗传风险评估模型成立。
本发明提供了一种基于所述构建方法得到的低氧不耐遗传风险评估模型对人群的低氧耐受性的评估方法,包括以下步骤:
测定待测样本的所述分子标志物的突变位点信息,将得到的分子标志物的突变位点信息输入低氧不耐遗传风险评估模型中,计算风险系数,根据所述分险系数判断待测样本的低氧耐受性:
所述风险系数低于0.33时,判断为低风险个体;
所述风险系数为0.33~0.67时,判断为中风险个体;
所述风险系数高于0.67时,判断为高风险个体。
优选的,所述低氧的氧分压为模拟海拔4000米以上高原的氧分压。
优选的,所述待测样本为血液。
本发明提供了一种急性低氧不耐的分子标志物,包括以下hg19基因组版本中突变位点:rs113459798、rs9662128、rs113147548、rs9874278、rs9814174、rs10512804、rs2974528、rs6926895、rs3739429、rs4634694、rs72805322、rs74910065、rs1471459475、rs1286147、rs189264、rs8038249、rs9935018、rs3809923、rs438703、rs3752194、rs1077806、rs10626088和rs11367927。本发明提供的分子标记物是通过对低氧耐受人群和低氧不耐人群进行高通量测序,筛选与低氧不耐相关联的突变位点得到的。基于所述分子标志物对人群的低氧耐受情况进行分析,得具有较高的准确性。实施例结果表明,检测的125个低风险个体中,115个是低氧耐受的个体,准确性达到92%,30个高风险个体中,28个是低氧不耐的个体,准确性达到93.33%。
附图说明
图1为曼哈顿图显示关联的遗传位点;
图2为AUC曲线评估模型评价低氧不耐风险的灵敏性和特异性;
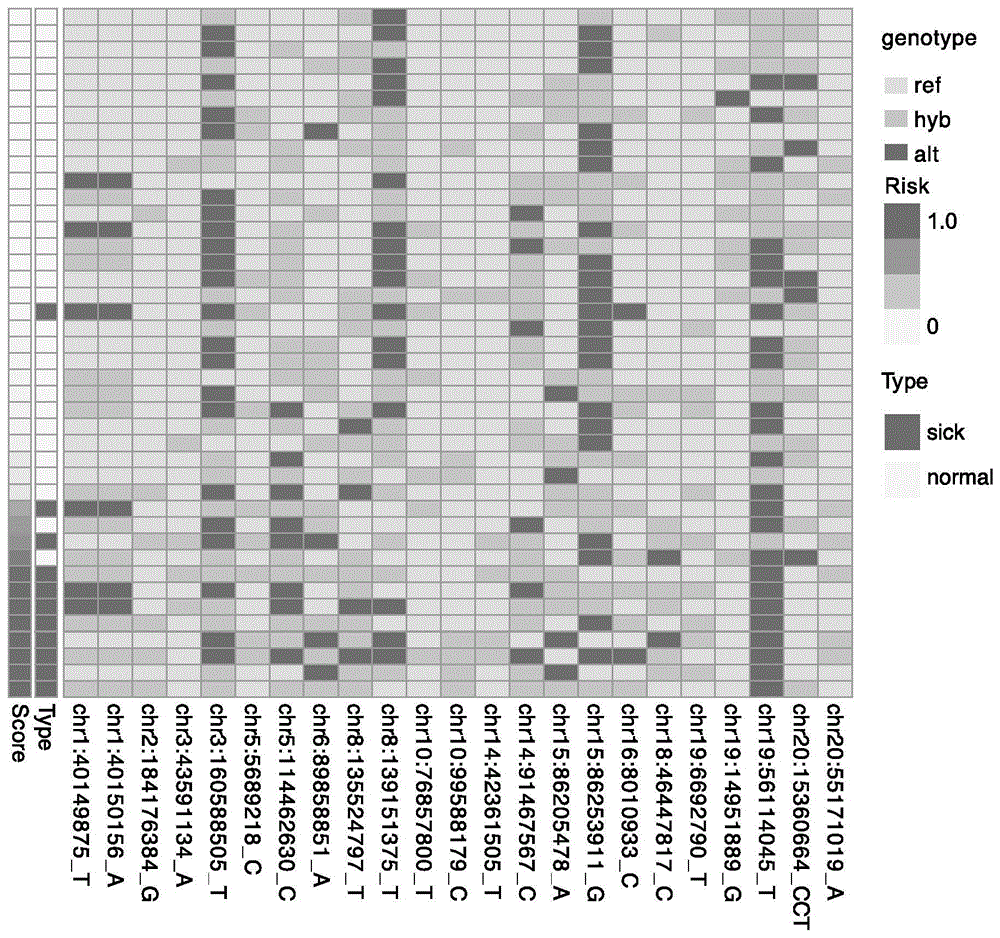
图3为23个突变验证165份个体风险图。
具体实施方式
本发明提供了一种急性低氧不耐的分子标志物,包括以下hg19基因组版本中突变位点:
rs113459798、rs9662128、rs113147548、rs9874278、rs9814174、rs10512804、rs2974528、rs6926895、rs3739429、rs4634694、rs72805322、rs74910065、rs1471459475、rs1286147、rs189264、rs8038249、rs9935018、rs3809923、rs438703、rs3752194、rs1077806、rs10626088和rs11367927。
在本发明中,根据hg19基因组版本,所述rs113459798位于人第1号染色体,第40149875位碱基由C突变为T;所述rs9662128位于人第1号染色体,第40150156位碱基由G突变为A;所述rs113147548位于人第2号染色体,第184176384位碱基由A突变为G;所述rs9874278位于人第3号染色体,第43591134位碱基由C突变为A;所述rs9814174位于人第3号染色体,第160588505位碱基由G突变为T;所述rs10512804位于人第5号染色体,第5689218位碱基由T突变为C;所述rs2974528位于人第5号染色体,第114462630位碱基由T突变为C;所述rs6926895位于人第6号染色体,第89858851位碱基由G突变为A;所述rs3739429位于人第8号染色体,第135524797位碱基由G突变为T;所述rs4634694位于人第8号染色体,第139151375位碱基由C突变为T;所述rs72805322位于人第10号染色体,第76857800位碱基由C突变为T;所述rs74910065位于人第10号染色体,第99588179位碱基由T突变为C;所述rs1471459475位于人第14号染色体,42361506-42361510位碱基由ACACA突变为ACA;所述rs1286147位于人第14号染色体,第91467567位碱基由A突变为C;所述rs189264位于人第15号染色体,第86205478位碱基由G突变为A;所述rs8038249位于人第15号染色体,第86253911位碱基由T突变为G;所述rs9935018位于人第16号染色体,第8010933位碱基由T突变为C;所述rs3809923位于人第18号染色体,第46447817位碱基由G突变为C;所述rs438703位于人第19号染色体,第6692790位碱基由A突变为T;所述rs3752194位于人第19号染色体,第14951889位碱基由A突变为G;所述rs1077806位于人第19号染色体,第56114045位碱基由C突变为T;所述rs10626088位于人第20号染色体,第15360664位碱基由C突变为CT;所述rs11367927位于人第20号染色体,第55171020位碱基G缺失。
在本发明中,所述分子标志物的筛选方法,优选如下:收集病例对照样本,依据中华人民共和国军用标准GJB1092-91,对一定数量人群进行低压氧舱模拟低氧,检测个体的血压、心率变化,并进行不耐易感性量表的筛查,通过心率和血压变化,筛选出易患或不易患低氧不耐的个体,收集血液样本,提取血液DNA;对样本进行全外显子测序和关联分析,对上述获得全外显子数据建立基因组测序文库,用全外显子捕获探针对测序文库进行靶向富集,高通量测序,检测每个样本的变异位点,关联分析鉴定与低氧不耐易感性关联的遗传变异,筛选得到分子标志物。
本发明提供了所述分子标志物在制备预测低氧不耐遗传风险的产品中的应用。
在本发明中,所述分子标志物作为检测靶点预测低氧不耐遗传风险。所述产品包括检测分子标志物的试剂。所述试剂包括靶向高通量测序分子标志物的试剂或扩增分子标志物的引物。
本发明提供了所述分子标志物在构建低氧不耐遗传风险评估模型中的应用。
本发明提供了一种低氧不耐遗传风险评估模型的构建方法,包括以下步骤:
对待测样本进行靶向高通量测序,得到所述分子标志物的突变位点信息;
以部分待检样本的低氧耐受情况和所述分子标志物的突变位点信息作为训练数据输入随机森林模型中,得到低氧不耐遗传风险评估模型。
在本发明中,在所述输入随机森林模型中后,优选还包括进行交叉验证。
在本发明中,所述交叉验证的方法优选为以剩余的待测样本的分子标志物的突变位点信息作为测试数据,进行多次交叉验证,得到受试者工作特征曲线;从所述受试者工作特征曲线分析得到模型评估指数AUC,当模型评估指数AUC为0.7以上时,说明低氧不耐遗传风险评估模型成立。所述训练数据和测试数据的数量比优选为7:3。
本发明实施例结果表明,模型评估指数AUC为0.968,表明本发明的评估模型具有准确率高的特点。可见本发明本发明构建的评估模型对低氧不耐遗传风险评估具有准确性高的特点,可为低氧不耐风险评估提供更加全面、准确、个体化的科学依据。
本发明提供了一种基于所述构建方法得到的低氧不耐遗传风险评估模型对人群的低氧耐受性的评估方法,包括以下步骤:
测定待测样本的所述分子标志物的突变位点信息,将得到的分子标志物的突变位点信息输入低氧不耐遗传风险评估模型中,计算风险系数,根据所述分险系数判断待测样本的低氧耐受性:
所述风险系数低于0.33时,判断为低风险个体;
所述风险系数为0.33~0.67时,判断为中风险个体;
所述风险系数高于0.67时,判断为高风险个体。
在本发明中,所述低氧是相当于海拔4000米以上高度的氧分压。所述低氧的氧分压优选为模拟海拔4000米以上高原的氧分压,即海平面氧分压60%以下。所述待测样本优选为血液。
本发明实施例结果表明,风险系数低于0.33的125个低风险个体中,115个是低氧耐受的个体,准确性达到92%,风险系数高于0.67的30个高风险个体中,28个是低氧不耐的个体,准确性达到93.33%。
本发明评估结果可用于指导高原作业人员选拔,提高高原工作岗位的适应性,具体所述高风险个体不建议进行高原作业。
下面结合实施例对本发明提供的一种急性低氧不耐的分子标志物及其应用和遗传风险评估模型的构建方法进行详细的说明,但是不能把它们理解为对本发明保护范围的限定。
实施例1
一种急性低氧不耐的分子标志物的筛选方法
1、收集病例对照样本
(1)在知情同意基础上,进行低氧不耐低压氧仓模拟实验和易感性量表的筛查。
收集病例对照样本,对一定数量人群进行低压氧舱模拟低氧,依据中华人民共和国军用标准GJB1092-91,检测个体的血压、心率变化,并进行不耐易感性量表的筛查,筛选出易患或不易患低氧不耐的个体,收集血液样本,总共收集165份样本。其中低氧不耐的41人,低氧耐受共124人作为对照组。
(2)提取血液基因组DNA,并进行基本DNA质控,包括DNA纯度、浓度等。
(3)对其中低氧不耐的41人,低氧耐受共124人进行靶向高通量测序。
2、对病例对照样本进行靶向高通量测序和关联分析
(1)对上述DNA样本建立基因组测序文库,并以全外显子靶向捕获探针对测序文库进行靶向富集。
(2)高通量测序并进行基本的数据质控。
具体采用艾吉泰康靶向全外显子测序的测序方法和服务完成,并对数据进行质控。具体步骤如下:每个样本的原始测序量平均为1335Mbp,去除低质量读数后的有效数据量平均为1172Mbp,平均测序质量Q30达到87.8%,平均测序读长135bp,平均插入片段大小为187bp,平均测序读数8.66M,有效比对率超过99.4%,其中靶向区域的平均测序读数为4.56M,占总测序读数的55.1%,靶向区域的平均有效测序量为219Mbp,测序覆盖度超过99.8%,平均测序深度达到4063,达到30X覆盖度的区域超过全部靶向区域的98.9%。
通过GATK软件整合所有样本的变异位点信息,运行shell脚本(参考文献Elgart,M.,Lyons,G.,Romero-Brufau,S.,Kurniansyah,N.,Brody,J.A.,Guo,X.,Lin,H.J.,Raffield,L.,Gao,Y.,Chen,H.,et al.(2022).Non-linear machine learning modelsincorporating SNPs and PRS improve polygenic prediction in diverse humanpopulations.Commun Biol 5,856.10.1038/s42003-022-03812-z.)并利用plink软件抽提只存在两个等位基因型的变异位点,整理后得到916880个变异位点。
对得到的变异位点进行质控,首先对缺失数据进行筛选,删除在样本中缺失率大于5%的变异位点,同时删除变异位点缺失率大于5%的样本,由于等位基因频率过低的变异位点可能引入假阳性,对MAF值小于0.05的变异位点进行过滤。同时,根据Hardy-Weinberg遗传平衡吻合度检验方法,删除p值小于0.05的变异位点。整理后得到165个有效样本,198058个可用于后续关联分析的变异位点。
(5)通过逻辑回归分析,筛选得到117个p值小于5e-4的变异位点,如图1所示。
3、关联遗传位点的确定
(1)遗传位点的确定
选择关联的遗传变异位点,构造基于Gradient Boosting的随机森林模型,通过贝叶斯方法优化系统参数。
参考现有文献(ME,Langaas M.Anew method for exploring gene-gene andgene-environment interactions in GWAS with tree ensemble methods and SHAPvalues.BMC Bioinformatics.2021;22(1):230.)建立基于R语言的计算程序脚本,输入之前得到的117个变异位点,及所有测序样本的变异位点信息,通过机器学习模型,计算得到117个突变的显著程度,进行特征工程,保留重要性指数大于0.01的突变共23个。具体结果见表2。
表123个突变信息
/>
实施例2
一种低氧不耐遗传风险评估模型的构建方法和验证方法对病例样本血液进行靶向高通量测序,得到病例样本的23个突变位点信息;建立基于R语言的计算程序脚本,输入这23个突变位点,及所有测序样本的突变位点的具体信息,建立随机森林模型,运行shell脚本,生成vcf文件,利用plink软件,对所述vcf文件运行命令行脚本,得到风险系数(参考文献Elgart,M.,Lyons,G.,Romero-Brufau,S.,Kurniansyah,N.,Brody,J.A.,Guo,
X.,Lin,H.J.,Raffield,L.,Gao,Y.,Chen,H.,et al.(2022).Non-linearmachine learning models incorporating SNPs and PRS improve polygenicprediction in diverse human populations.Commun Biol 5,856.10.1038/s42003-022-03812-z.),对病例样本的低氧不耐风险进行评估。
当风险系数低于0.33时,为低风险个体;当风险系数高于0.67时,为高风险个体。
以7:3的比例将样本(低氧不耐人群41例,低氧耐受人群124例)划分为训练数据和测试数据,进行多次交叉验证,受试者工作特征曲线(ROC曲线)如图2所示,模型评估指数AUC为0.968,表明本发明的评估模型具有准确率高的特点。
实施例3
独立招募另外42位志愿者参与模拟高原低氧环境的低压氧舱实验,所有人员在模拟4000米海拔高度保持30分钟,以模拟处于相似的氧环境。低氧氧仓实验参数同实施例1。采集该42位人员的血液样本,进行靶向高通量测序,得到实施例1中筛选的23个突变位点,将这些样本的23个突变位点具体信息输入实施例2建立的评估模型中,计算风险系数进行评估,并和真实的低氧耐受症状比较。
具体结果见图3。实验结果表明,风险系数低于0.33的30个低风险个体中,29个是低氧耐受的个体,准确性达到96.7%,风险系数高于0.67的10个高风险个体中,9个是低氧不耐的个体,准确性达到90%。
以上所述仅是本发明的优选实施方式,应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。
