一种ARMS-TaqMan Blocker体系的Blocker设计和筛选方法
文献发布时间:2023-06-19 09:57:26
技术领域
本发明涉及肿瘤突变检测技术领域,具体是一种ARMS-TaqMan Blocker体系的Blocker设计和筛选方法。
背景技术
近年来,探针的非放射性标记受到广泛重视,同时得到迅速发展,广泛应用于核酸序列测定、基因检测以及疾病诊断等。ARMS技术是常用的荧光探针法,其基本原理是,如果引物的3’端碱基与模板碱基不互补,则用一般耐热DNA聚合酶无法延伸。因此根据已知点突变设计3条引物,其3’端碱基分别与突变和正常的模板碱基互补,从而将有某种点突变的模板与正常模板区分开来。有时候为了提高特异性,会在体系中加入野生型模板扩增阻断剂(Blocker),用以压制野生型模板的扩增,虽然这种方法在一定程度上可以压制野生,但是由于缺乏准确的Blocker设计软件和有效的筛选规则,导致压制效果不理想;另外,也可在ARMS引物的3’端倒数第二或第三个碱基处引入错配碱基,但是引入错配碱基通常会牺牲灵敏度。因此,常规ARMS-TaqMan Blocker体系一般很难同时做到灵敏度、特异性兼顾,常需要复杂繁琐的优化工作。
传统的竞争性Blocker设计原则为:①将Blocker设计在突变位点处,覆盖突变位点,与ARMS引物部分重叠,起到位置竞争作用,且尽量使突变位点位于Blocker的中间位置,使得Blocker熔点差较大(依据:突变位点位于中间时的熔点差大于突变位点位于两边时的熔点差);②Blocker与野生型模板完全匹配,Tm值比ARMS引物与野生型模板匹配的Tm值高约5℃,使Blocker与野生型模板优先结合,而且有时为了增大熔点差,会加入LNA、MGB等修饰;③Blocker与突变型模板不完全匹配,Tm值比ARMS引物与突变型模板匹配的Tm值低,使ARMS引物与突变型模板优先结合;④Blocker的3’末端需加上封闭修饰,如硫代修饰、氨基修饰、磷酸化修饰等,以防止延伸。
传统Blocker设计一般是利用Tm值预测软件进行Tm值预测,不同的预测软件预测出的Tm值悬殊很大,同时PCR组分也会对Tm值产生一定的影响,因而无法准确评估Blocker的实际Tm值,导致Blocker的压制效果不好。按照一般设计原则设计得到的Blocker并不符合最优条件,表现在Blocker与突变型模板不完全匹配,Tm值均比ARMS引物与突变型模板匹配的Tm值低,且软件预测的Tm值也仅供参考,实际Tm值可能与预测接近,也可能相差很大,所以通常设计多条Blocker进行筛选,但同时满足②③条件的Blocker本身就很少,且鉴于修饰成本高,一般情况下仅设计1-3条供筛选。如果通过软件进行Tm值预测,很可能所有Blocker的低熔点均比ARMS引物与突变型模板匹配的Tm值低,能找到最优Blocker的几率极小,只有当设计的Blocker里面恰巧有一条Blocker的低熔点实际上与ARMS引物与突变型模板匹配的Tm值一致或极为接近,才可以起到良好的压制效果。
发明内容
针对现有ARMS-TaqMan Blocker体系的Blocker设计存在的技术缺陷,本发明提供一种ARMS-TaqMan Blocker体系的Blocker设计和筛选方法。
本发明保护一种ARMS-TaqMan Blocker体系的Blocker设计方法,将Blocker设计在突变位点处,覆盖突变位点即可;Blocker与野生型模板完全匹配,完全匹配的Tm值高于ARMS引物与野生型模板匹配的Tm值,允许熔点差低于5℃;Blocker与突变型模板不完全匹配,不完全匹配的Tm值与ARMS引物跟突变型模板的Tm值一致或接近;Blocker的3’末端添加多个与模板不匹配的无关碱基。
本发明的Blocker设计原则,不严格要求突变位点位于Blocker的中间位置,也不需要满足熔点差5℃的要求,只要存在熔点差即可,相对来说更容易设计。与此同时,由于没有严格要求5℃左右的熔点差,所以不需要引入LNA修饰,不存在LNA合成难度及额外的成本费用。
本发明的Blocker设计原则,保证Blocker与突变型模板不完全匹配的Tm值与ARMS引物与突变型模板匹配的Tm值一致或接近,因为只有在这个Tm值下,体系中的Blocker才能不会产生冗余,起到最大的压制效果。这一点与传统的竞争性Blocker设计原则是完全不同的。
本发明的Blocker设计原则,Blocker的3’末端不需要加特殊的封闭修饰,而是通过在3’末端添加个别无关碱基,这些无关碱基与模板不匹配,从而无法在Taq DNA聚合酶作用下发生延伸,因此不必加特殊修饰,节约了近几十倍的成本。
本发明还保护一种ARMS-TaqMan Blocker体系的Blocker筛选方法,按照上述Blocker设计原则设计3-10条Blocker,这些Blocker的低熔点都在ARMS引物跟突变型模板匹配的Tm值附近,有比之高的,也有比之低的,不同Blocker之间可以相差1碱基,使这些Blocker与突变型模板匹配的Tm值在ARMS引物跟突变型模板匹配的Tm值附近呈现逐步升高的梯度,这样Tm值范围广而密,不会错失最佳Blocker;然后经实时PCR验证梯度Blocker对体系的灵敏度和特异性的影响,筛选出对灵敏度无影响且特异性最好的Blocker。
传统的Blocker设计原则设计成本高,对于多位点检测体系来说,成本费用更是不可低估。本发明的Blocker设计和筛选原则,具有明确的筛选方向,一次性就能筛出最佳Blocker,不需要反复修改,而且Blocker与普通引物一样合成,不需要特殊修饰和纯化,成本低廉。
本发明还保护上述ARMS-TaqMan Blocker体系的Blocker筛选方法在肿瘤突变检测中的应用。
附图说明
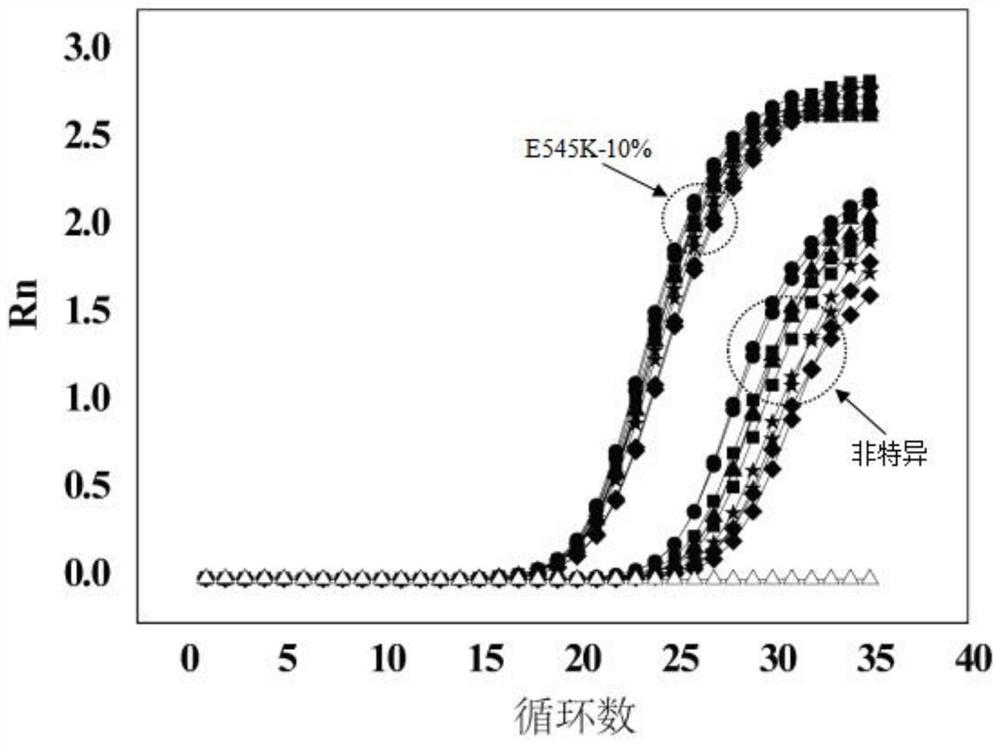
图1为针对E545K位点按照本发明设计原则设计的Blocker梯度筛选及传统设计原则设计的Blocker比较;
图2为针对braf位点按照本发明设计原则设计的Blocker梯度筛选及传统设计原则设计的Blocker比较;
图3为针对G719位点按照本发明设计原则设计的Blocker梯度筛选及传统设计原则设计的Blocker比较。
具体实施方式
下面结合附图和具体实施方式对本发明作进一步详细的说明。本发明的实施例是为了示例和描述起见而给出的,而并不是无遗漏的或者将本发明限于所公开的形式。很多修改和变化对于本领域的普通技术人员而言是显而易见的。选择和描述实施例是为了更好说明本发明的原理和实际应用,并且使本领域的普通技术人员能够理解本发明从而设计适于特定用途的带有各种修改的各种实施例。
说明:本发明使用的引物、探针、Blocker通过软件TM Utility v1.3进行熔点预测,各引物、探针、Blocker的序列信息见下表1,其中F/R表示引物、P表示探针、B表示Blocker、FAM表示荧光基团、BHQ1/MGB表示淬灭基团、PHO表示磷酸化修饰、方框表示LNA修饰、小写字母表示人为添加的碱基、Tm1表示引物/探针/Blocker与靶序列完全匹配时的熔点、Tm2表示引物/Blocker与靶序列不完全匹配时的熔点。
表1
实施例1E545K位点按照本发明设计原则设计的Blocker梯度筛选及与传统设计原则设计的Blocker比较
一、引物、探针、Blocker设计
引物按照ARMS引物设计原则设计,引物长度15-38nt,GC含量40-60%,引物自身3’端避免发卡结构,引物之间避免4个碱基的匹配,碱基分布均匀,避免连续的GC或AT。上游ARMS引物的3’端与突变位点一致,下游引物为通用引物,可同时扩增野生和突变型模板。上游引物E545K-F4序列如表1中的SEQ NO.01,下游引物E545K-R1序列如表1中的SEQ NO.02。
探针按照TaqMan探针设计原则设计,长度为14-35nt,GC含量60-70%,5’端第一个碱基避免为G,探针E545K-P序列如表1中的SEQ NO.03。
按照本发明设计原则设计3个Blocker,分别为E545K-B13、E545K-B18和E545K-B19,序列分别如表1中的SEQ NO.05-07。
按照传统设计原则设计1个Blocker E545K-B-1,序列如表1中的SEQ NO.04。
引物探针由生工生物合成,本发明设计的Blocker均使用PAGE纯化,用Tris-HCL(PH8.0)溶解至100μM。
二、PCR反应体系配制
按照下表2进行PCR反应液配制,补充去离子水至25μl。第一组反应液只加纯水,不加Blocker;第二组反应液Blocker加E545-B-1;第三组反应液Blocker加E545-B13;第四组反应液Blocker加E545-B18;第五组反应液Blocker加E545-B19。
表2
三、样本准备
将E545K质粒10^3copies/μl与10ng/μl野生型基因组混合,得到突变率为50%的标准品,将50%的标准品用10ng/μl野生型基因组进行稀释,得到10%的标准品,准备10ng/μl野生型基因组,备用。
四、加样,上机
以上五组反应液均加入5μl的10%的E545K质粒标准品和10ng/μl野生型基因组,每组各2个平行管并设置无模板对照(NTC)。上机程序为:95℃2min,1个循环;95℃5s,56℃30s(不采集荧光),72℃15s,10个循环;93℃3s,56℃30s(采集荧光),60℃30s,35个循环。仪器使用SLAN96荧光PCR仪。
五、实验结果分析
参照图1,第一组不加Blocker,扩增曲线用黑色圆圈表示;第二组加E545-B-1,扩增曲线用黑色正三角表示;第三组加E545-B13,扩增曲线用黑色正方形表示;第四组加E545-B18,扩增曲线用黑色五角星表示;第五组加E545-B19,扩增曲线用黑色斜方型表示,NTC用白色正三角表示。
从图1可以看出,第五组加E545-B19(黑色斜方型)的会影响10%的灵敏度,按照本发明设计原则设计并筛选出的E545K-B18(黑色五角星)比按照传统原则设计的E545K-B-1(黑色正三角)的特异性好,其10ng/μl野生型样本的CT值更靠后,说明本发明的Blocker设计和筛选原则更优,能更大程度地压制野生型模板,提高了体系的特异性。
实施例2braf位点按照本发明设计原则设计的Blocker梯度筛选及与传统设计原则设计的Blocker比较
一、引物、探针、Blocker设计
引物按照ARMS引物设计原则设计,引物长度15-38nt,GC含量40-60%,引物自身3’端避免发卡结构,引物之间避免4个碱基的匹配,碱基分布均匀,避免连续的GC或AT。上游ARMS引物的3’端与突变位点一致,下游引物为通用引物,可同时扩增野生和突变型模板。上游引物braf-F序列如表1中SEQ NO.08,下游引物braf-R序列如表1中SEQ NO.09。
探针按照TaqMan探针设计原则设计,长度为14-35nt,GC含量60-70%,5’端第一个碱基避免为G,探针braf-P序列如表1中SEQ NO.10。
按照本发明设计原则设计4个Blocker,分别为braf-B3、braf-B4、braf-B5、braf-B6,序列分别如表1中SEQ NO.12-15。
按照传统设计原则设计1个Blocker,braf-B2,序列如表1中SEQ NO.11。
引物探针由生工生物合成,本发明设计的Blocker均使用PAGE纯化,用Tris-HCL(PH 8.0)溶解至100μM。
二、PCR反应体系配制
按照下表3进行PCR反应液配制,补充去离子水至25μl。第一组反应液只加纯水,不加Blocker;第二组反应液Blocker加braf-B2,第三组反应液Blocker加braf-B3;第四组反应液Blocker加braf-B4;第五组反应液Blocker加braf-B5;第六组反应液Blocker加braf-B6。
表3
三、样本准备
将braf质粒10^3copies/μl与10ng/μl野生型基因组混合,得到突变率为50%的标准品,将50%的标准品用10ng/μl野生型基因组进行稀释,得到10%的标准品,准备20ng/μl野生型基因组,备用。
四、加样,上机
以上六组反应液均加入5μl的10%的标准品和20ng/μl野生型基因组,各组2个平行管并设置无模板对照(NTC)。上机程序为:95℃2min,1个循环;95℃5s,56℃30s(不采集荧光),72℃15s,10个循环;93℃3s,56℃30s(采集荧光),60℃30s,35个循环。仪器使用SLAN96荧光PCR仪。
五、实验结果分析
参照图2,第一组只加纯水,扩增曲线用黑色圆圈表示;第二组加braf-B2,扩增曲线用黑色正三角表示;第三组加braf-B3,扩增曲线用黑色方形表示;第四组加braf-B4,扩增曲线用黑色五角星表示;第五组加braf-B5,扩增曲线用黑色倒三角表示;第六组加braf-B6,扩增曲线用黑色斜方形表示,NTC用白色正三角型表示。
从图2可以看出,按照本发明设计原则设计并筛选出的braf-B4(黑色五角星)比按照传统设计原则设计的braf-B2(黑色正三角)的特异性好,20ng/μl野生型样本无CT值,说明本发明的Blocker设计原则更优,能更大程度上压制野生型模板,提高了体系的特异性。
实施例3 G719位点按照本发明设计原则设计的Blocker梯度筛选及与传统设计原则设计的Blocker比较
一、引物、探针、Blocker设计
引物按照ARMS引物设计原则设计,引物长度15-38nt,GC含量40-60%,引物自身3’端避免发卡结构,引物之间避免4个碱基的匹配,碱基分布均匀,避免连续的GC或AT。上游ARMS引物的3’端与突变位点一致,下游引物为通用引物,可同时扩增野生和突变型模板。上游引物G719-F序列如表1中SEQ NO.16,下游引物G719-R序列如表1中SEQ NO.17。
探针按照TaqMan探针设计原则设计,长度为14-35nt,GC含量60-70%,5’端第一个碱基避免为G,探针G719-P序列如表1中SEQ NO.18。
按照本发明设计原则设计4个Blocker,分别为G719-B1、G719-B2、G719-B9、G719-B10,序列分别如表1中SEQ NO.19-22。
按照传统设计原则设计的1个Blocker,G719-B11,序列如表1中SEQ NO.23。
引物探针由生工生物合成,本发明设计的Blocker均使用PAGE纯化,用Tris-HCL(PH 8.0)溶解至100μM。
二、PCR反应体系配制
按照下表4进行PCR反应液配制,补充去离子水至25μl。第一组反应液只加纯水,不加Blocker;第二组反应液Blocker加G719-B10;第三组反应液Blocker加G719-B9;第四组反应液Blocker加G719-B1;第五组反应液Blocker加G719-B2;第六组反应液Blocker加G719-B11。
表3
三、样本准备
将G719质粒10^3copies/μl与10ng/μl野生型基因组混合,得到突变率为50%的标准品,将50%的标准品用10ng/μl野生型基因组进行稀释,得到10%的标准品,准备50ng/μl野生型基因组,备用。
四、加样,上机
以上六组反应液均加入5μl的10%的标准品和50ng/μl野生型基因组,各组2个平行管并设置无模板对照(NTC)。上机程序为:95℃2min,1个循环;95℃5s,56℃30s(不采集荧光),72℃15s,10个循环;93℃3s,56℃30s(采集荧光),60℃30s,35个循环。仪器使用SLAN96荧光PCR仪。
五、实验结果分析
参照图2,第一组只加纯,扩增曲线用黑色圆圈表示;第二组加G719-B10,扩增曲线用黑色正三角表示;第三组加G719-B9,扩增曲线用黑色方形表示;第四组加G719-B1,扩增曲线用黑色五角星表示;第五组加G719-B2,扩增曲线用黑色倒三角表示;第六组加G719-B11,扩增曲线用黑色斜方形表示,NTC用白色正三角型表示。
从图3可以看出,按照本发明设计原则设计并筛选出的G719-B1(黑色五角星)比按照传统设计原则设计的G719-B11(黑色斜方形)的特异性好,50ng/μl野生型样本无CT值。
显然,所描述的实施例仅仅是本发明的一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域及相关领域的普通技术人员在没有作出创造性劳动的前提下所获得的所有其他实施例,都应属于本发明保护的范围。
- 一种ARMS-TaqMan Blocker体系的Blocker设计和筛选方法
- 一种ARMS-TaqMan Blocker体系的Blocker双链设计方法
