肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)的生物标志物
文献发布时间:2023-06-19 11:03:41
技术领域
本发明涉及疾病的诊断领域,特别是涉及肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)的诊断领域。
背景技术
肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)是以显著疲劳、劳作后的疲惫、睡眠障碍、认知功能障碍、直立不耐受为核心症状,并伴有疼痛、自律神经障碍、对光、声音、食品、化学物质的过敏症等各种症状的疾病。也有在感染之后发病的情况,病状尚不清楚,没有识别发病的生物标志物,所以有各种诊断标准,成为诊断、研究的障碍。另外,有效的治疗方法也尚未被确立。
发明内容
用于解决课题的方案
本发明提供以B细胞受体(BCR)库作为肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)的指标的方法。与健康对照相比,ME/CFS患者中BCR的IgGH链可变区中的基因的使用频率有变化,这已被本说明书证明,使用该信息,可以进行ME/CFS的诊断。除了BCR的IgGH链可变区中的基因之外,也可以使用对象的免疫细胞亚群(B细胞、调节性T细胞等)的数量作为指标。基因的使用频率可以通过包括大规模高效率BCR库分析的方法来确定。
本发明的实施方式的例子如以下的项目所示。
(项目1)一种方法,其以对象的B细胞受体(BCR)库作为该对象的肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)的指标。
(项目2)根据上述项目所述的方法,其中,以包含对象的BCR的IgGH链可变区中的1个以上的基因的使用频率在内的1个以上的变量,作为该对象的肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)的指标。
(项目2A)根据上述项目中的任意一项所述的方法,其中,所述1个以上的变量是所述对象罹患ME/CFS而不是其他疾病的指标。
(项目3)根据上述项目中任意一项所述的方法,其中,所述1个以上的基因包含从由IGHV1-2、IGHV1-3、IGHV1-8、IGHV1-18、IGHV1-24、IGHV1-38-4、IGHV1-45、IGHV1-46、IGHV1-58、IGHV1-69、IGHV1-69-2、IGHV1-69D、IGHV1/OR15-1、IGHV1/OR15-5、IGHV1/OR15-9、IGHV1/OR21-1、IGHV2-5、IGHV2-26、IGHV2-70、IGHV2-70D、IGHV2/OR16-5、IGHV3-7、IGHV3-9、IGHV3-11、IGHV3-13、IGHV3-15、IGHV3-16、IGHV3-20、IGHV3-21、IGHV3-23、IGHV3-23D、IGHV3-25、IGHV3-30、IGHV3-30-3、IGHV3-30-5、IGHV3-33、IGHV3-35、IGHV3-38、IGHV3-38-3、IGHV3-43、IGHV3-43D、IGHV3-48、IGHV3-49、IGHV3-53、IGHV3-64、IGHV3-64D、IGHV3-66、IGHV3-72、IGHV3-73、IGHV3-74、IGHV3-NL1、IGHV3/OR15-7、IGHV3/OR16-6、IGHV3/OR16-8、IGHV3/OR16-9、IGHV3/OR16-10、IGHV3/OR16-12、IGHV3/OR16-13、IGHV4-4、IGHV4-28、IGHV4-30-2、IGHV4-30-4、IGHV4-31、IGHV4-34、IGHV4-38-2、IGHV4-39、IGHV4-59、IGHV4-61、IGHV4/OR15-8、IGHV5-10-1、IGHV5-51、IGHV6-1、IGHV7-4-1、IGHV7-81、IGHD1-1、IGHD1-7、IGHD1-14、IGHD1-20、IGHD1-26、IGHD1/OR15-1a/b、IGHD2-2、IGHD2-8、IGHD2-15、IGHD2-21、IGHD2/OR15-2a/b、IGHD3-3、IGHD3-9、IGHD3-10、IGHD3-16、IGHD3-22、IGHD3/OR15-3a/b、IGHD4-4、IGHD4-11、IGHD4-17、IGHD4-23、IGHD4/OR15-4a/b、IGHD5-5、IGHD5-12、IGHD5-18、IGHD5-24、IGHD5/OR15-5a/b、IGHD6-6、IGHD6-13、IGHD6-19、IGHD6-25、IGHD7-27、IGHJ1、IGHJ2、IGHJ3、IGHJ4、IGHJ5、IGHJ6、IGHG1、IGHG2、IGHG3、IGHG4、以及IGHGP构成的组中选择的至少1个基因。
(项目3-1)根据上述项目中任意一项所述的方法,其中,所述1个以上的基因包含从由IGHV3-73、IGHV1-69-2、IGHV5-51、IGHV4-31、IGHV3-23D、IGHV1/OR15-9、IGHV4-39、IGHD5-12、IGHV3-43D、IGHD4-17、IGHV5-10-1、IGHD4/OR15-4a/b、IGHG4、IGHV1/OR15-5、IGHV3/OR16-9、IGHD1-7、IGHV3-21、IGHD6-6、IGHV3-33、IGHD4-23、IGHV3-30-5、IGHV3-23、IGHD6-13、IGHV3-64D、IGHV3-48、IGHV3-64、IGHG1、IGHV3-49、IGHV3-30-3、IGHD1-26、IGHJ6、IGHV3-30、IGHGP、IGHV1-3以及IGHD3-22构成的组中选择的至少1个基因。
(项目3-2)根据上述项目中任意一项所述的方法,其中,所述1个以上的基因包含从由IGHD6-6、IGHV3-33、IGHD4-23、IGHV3-30-5、IGHV3-23、IGHD6-13、IGHV3-64D、IGHV3-48、IGHV3-64、IGHG1、IGHV3-49、IGHV3-30-3、IGHD1-26、IGHJ6、IGHV3-30、IGHGP、IGHV1-3以及IGHD3-22构成的组中选择的至少1个基因。
(项目3-3)根据上述项目中任意一项所述的方法,其中,所述1个以上的基因包含从由IGHV3-49、IGHV3-30-3、IGHD1-26、IGHJ6、IGHV3-30、IGHGP、IGHV1-3以及IGHD3-22构成的组中选择的至少1个基因。
(项目3-4)根据上述项目中任意一项所述的方法,其中,所述1个以上的基因包含从由IGHV1-3、IGHV3-30、IGHV3-30-3、IGHV3-49、IGHD1-26、以及IGHJ6构成的组中选择的至少1个基因。
(项目4-1)根据上述项目中任意一项所述的方法,其中,所述1个以上的变量进一步含有1个以上的免疫细胞亚群的数量。
(项目4-2)根据上述项目中任意一项所述的方法,其中,所述1个以上的变量在用于判别正常对照和ME/CFS的基于回归分析的ROC曲线中显示AUC≥0.7。
(项目4-3)根据上述项目中任意一项所述的方法,其中,所述1个以上的变量在用于判别正常对照和ME/CFS的基于回归分析的ROC曲线中显示AUC≥0.8。
(项目4-4)根据上述项目中任意一项所述的方法,其中,所述免疫细胞亚群的数量从B细胞的数量、幼稚B细胞的数量、记忆B细胞的数量、浆母细胞的数量、活化幼稚B细胞的数量、过渡性B细胞的数量、调节性T细胞的数量、记忆T细胞的数量、滤泡辅助性T细胞的数量、Tfh1细胞的数量、Tfh2细胞的数量、Tfh17细胞的数量、Th1细胞的数量、Th2细胞的数量以及Th17细胞的数量中选择。
(项目5-1)根据上述项目中任意一项所述的方法,其中,所述1个以上的变量包含所述对象的BCR的IgGH链可变区中的2个以上的基因的使用频率。
(项目5-2)根据上述项目中任意一项所述的方法,其中,所述1个以上的变量在用于判别正常对照和ME/CFS的基于回归分析的ROC曲线中显示AUC≥0.7。
(项目5-2)根据上述项目中任意一项所述的方法,其中,所述1个以上的变量在用于判别正常对照和ME/CFS的基于回归分析的ROC曲线中显示AUC≥0.8。
(项目5-2)根据上述项目中任意一项所述的方法,其中,所述1个以上的变量在用于判别正常对照和ME/CFS的基于回归分析的ROC曲线中显示AUC≥0.9。
(项目6-1)根据上述项目中任意一项所述的方法,其中,所述1个以上的变量包含从由所述对象的BCR的IgGH链可变区中的1个以上的基因的使用频率、所述对象的BCR多样性指数、以及所述对象的1个以上的免疫细胞亚群的数量构成的组中选择的2个以上的变量的组合,该1个以上的变量在用于判别正常对照和ME/CFS的基于回归分析的ROC曲线中显示AUC≥0.7。
(项目6-2)根据上述项目中任意一项所述的方法,其中,所述1个以上的变量包含从所述组中选择的3个以上的变量。
(项目6-3)根据上述项目中任意一项所述的方法,其中,所述1个以上的变量包含从所述组中选择的4个以上的变量。
(项目6-4)根据上述项目中任意一项所述的方法,其中,所述1个以上的变量包含从所述组中选择的5个以上的变量。
(项目6-5)根据上述项目中任意一项所述的方法,其中,所述1个以上的变量包含从所述组中选择的6个以上的变量。
(项目6-7)根据上述项目中任意一项所述的方法,其中,所述1个以上的变量在用于判别正常对照和ME/CFS的基于回归分析的ROC曲线中显示AUC≥0.8。
(项目6-3)根据上述项目中任意一项所述的方法,其中,所述1个以上的变量在用于判别正常对照和ME/CFS的基于回归分析的ROC曲线中显示AUC≥0.9。
(项目7)根据上述项目中任意一项所述的方法,其中,所述1个以上的基因包含IGHV1-3、IGHV3-30、IGHV3-30-3、IGHV3-49、IGHD1-26、以及IGHJ6。
(项目8)根据上述项目中任意一项所述的方法,其中,所述1个以上的变量包含所述对象的B细胞的数量。
(项目9)根据上述项目中任意一项所述的方法,其中,所述1个以上的变量包含所述对象的调节性T细胞(Treg)的数量。
(项目10)根据上述项目中任意一项所述的方法,其中,所述1个以上的基因的使用频率由包含大规模高效率BCR库分析的方法来确定。
(项目11)根据上述项目中任意一项所述的方法,其中,所述1个以上的变量包含从由IGHV3-49、IGHV3-30-3、IGHD1-26、IGHV3-30、IGHJ6、IGHGP、IGHV4-31、IGHV3-64、IGHD3-22、IGHV3-33、IGHV3-73、IGHV5-10-1以及IGHV4-34构成的组中选择的至少1个基因的使用频率。
(项目12)根据上述项目中任意一项所述的方法,其中,包括:
(a)以所述1个以上的变量中的一部分作为所述对象的ME/CFS的指标,
(b)以所述1个以上的变量中的一部分作为所述对象是ME/CFS而不是其他疾病的指标。
(项目13)根据上述项目中任意一项所述的方法,其中,对于多种其他疾病多次进行(b)。
(项目14)根据上述项目中任意一项所述的方法,其中,所述其他疾病包含多发性硬化症(MS)。
(项目15)一种方法,其中,以包含对象的BCR的IgGH链可变区中的1个以上的基因的使用频率在内的1个以上的变量,作为该对象罹患肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)而不是多发性硬化症(MS)的指标。
(项目15A)根据上述项目所述的方法,其中,具有上述项目中的1个或多个所述的特征。
(项目16)根据上述项目中任意一项所述的方法,其中,所述1个以上的基因包含从由IGHV1-2、IGHV1-3、IGHV1-8、IGHV1-18、IGHV1-24、IGHV1-38-4、IGHV1-45、IGHV1-46、IGHV1-58、IGHV1-69、IGHV1-69-2、IGHV1-69D、IGHV1/OR15-1、IGHV1/OR15-5、IGHV1/OR15-9、IGHV1/OR21-1、IGHV2-5、IGHV2-26、IGHV2-70、IGHV2-70D、IGHV2/OR16-5、IGHV3-7、IGHV3-9、IGHV3-11、IGHV3-13、IGHV3-15、IGHV3-16、IGHV3-20、IGHV3-21、IGHV3-23、IGHV3-23D、IGHV3-25、IGHV3-30、IGHV3-30-3、IGHV3-30-5、IGHV3-33、IGHV3-35、IGHV3-38、IGHV3-38-3、IGHV3-43、IGHV3-43D、IGHV3-48、IGHV3-49、IGHV3-53、IGHV3-64、IGHV3-64D、IGHV3-66、IGHV3-72、IGHV3-73、IGHV3-74、IGHV3-NL1、IGHV3/OR15-7、IGHV3/OR16-6、IGHV3/OR16-8、IGHV3/OR16-9、IGHV3/OR16-10、IGHV3/OR16-12、IGHV3/OR16-13、IGHV4-4、IGHV4-28、IGHV4-30-2、IGHV4-30-4、IGHV4-31、IGHV4-34、IGHV4-38-2、IGHV4-39、IGHV4-59、IGHV4-61、IGHV4/OR15-8、IGHV5-10-1、IGHV5-51、IGHV6-1、IGHV7-4-1、IGHV7-81、IGHD1-1、IGHD1-7、IGHD1-14、IGHD1-20、IGHD1-26、IGHD1/OR15-1a/b、IGHD2-2、IGHD2-8、IGHD2-15、IGHD2-21、IGHD2/OR15-2a/b、IGHD3-3、IGHD3-9、IGHD3-10、IGHD3-16、IGHD3-22、IGHD3/OR15-3a/b、IGHD4-4、IGHD4-11、IGHD4-17、IGHD4-23、IGHD4/OR15-4a/b、IGHD5-5、IGHD5-12、IGHD5-18、IGHD5-24、IGHD5/OR15-5a/b、IGHD6-6、IGHD6-13、IGHD6-19、IGHD6-25、IGHD7-27、IGHJ1、IGHJ2、IGHJ3、IGHJ4、IGHJ5、IGHJ6、IGHG1、IGHG2、IGHG3、IGHG4、以及IGHGP构成的组中选择的至少1个基因。
(项目17)根据上述项目中任意一项所述的方法,其中,所述1个以上的基因包含从由IGHV1-3、IGHV3-30、IGHV3-30-3、IGHD1-26、IGHV3-49以及IGHJ6构成的组中选择的至少1个基因。
(项目18)根据上述项目中任意一项所述的方法,其中,所述1个以上的变量包含所述对象的BCR的IgGH链可变区中的2个以上的基因的使用频率。
(项目19)根据上述项目中任意一项所述的方法,其中,所述1个以上的变量在用于判别MS和ME/CFS的基于回归分析的ROC曲线中显示AUC≥0.7。
(项目20)根据上述项目中任意一项所述的方法,其中,所述1个以上的变量在用于判别MS和ME/CFS的基于回归分析的ROC曲线中显示AUC≥0.8。
(项目21)根据上述项目中任意一项所述的方法,其中,所述1个以上的变量在用于判别MS和ME/CFS的基于回归分析的ROC曲线中显示AUC≥0.9。
(项目22)根据上述项目中任意一项所述的方法,其中,所述1个以上的变量包含所述对象的BCR的IgGH链可变区中的3个以上的基因的使用频率。
(项目23)根据上述项目中任意一项所述的方法,其中,所述1个以上的变量在用于判别MS和ME/CFS的基于回归分析的ROC曲线中显示AUC≥0.7。
(项目24)根据上述项目中任意一项所述的方法,其中,所述1个以上的变量在用于判别MS和ME/CFS的基于回归分析的ROC曲线中显示AUC≥0.8。
(项目25)根据上述项目中任意一项所述的方法,其中,所述1个以上的变量在用于判别MS和ME/CFS的基于回归分析的ROC曲线中显示AUC≥0.9。
(项目26)根据上述项目中任意一项所述的方法,其中,所述(b)通过上述项目中任意一项所述的方法来进行。
(项目A1)一种方法,其是对对象罹患肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)进行诊断的方法,其中,包括:对对象的B细胞受体(BCR)库进行测定;根据该BCR库,对该对象罹患肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)进行诊断。
(项目A1-1)根据上述项目所述的方法,其中,具有上述项目中的1个或多个所述的特征。
(项目A2)一种方法,其是对肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)进行处置的方法,其中,包括:对对象的B细胞受体(BCR)库进行测定;根据该BCR库对该对象罹患肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)进行诊断;对该对象施以治疗。
(项目A2-1)根据上述项目所述的方法,其中,具有上述项目中的1个或多个所述的特征。
(项目A3)根据上述项目中任意一项所述的方法,其中,根据包含所述对象的BCR的IgGH链可变区中的1个以上的基因的使用频率在内的1个以上的变量,对该对象罹患肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)进行诊断。
(项目A4)根据上述项目中任意一项所述的方法,其中,对所述对象计算包含所述变量的式子的值,将该值与阈值进行比较,由此对该对象罹患肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)进行诊断。
(项目A5)根据上述项目中任意一项所述的方法,其中,包括:
(A)关于肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS),提供包含BCR的IgGH链可变区中的1个以上的基因的使用频率在内的多个变量;
(B)提供对该变量进行多变量分析而生成的判别式;
(C)将对象的该变量的值代入该判别式来计算罹患ME/CFS的概率;和
(D)在该罹患ME/CFS的概率高于规定值的情况下,确定为该对象罹患ME/CFS。
(项目A6)根据上述项目中任意一项所述的方法,其中,包括:
(A)关于肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS),提供包含BCR的IgGH链可变区中的1个以上的基因的使用频率在内的多个变量;
(B)提供对该变量进行多变量分析而生成的判别式,(B)包括:
(B-1)对该变量以患者/健康人的区分作为目标变量进行单变量或多变量逻辑回归,
(B-2)从在该逻辑回归中生成的Logit模型公式(logit model formula)的常数以及偏回归系数计算判别式的常数以及系数,和
(B-3)基于在B-2的处理中得到的常数以及系数生成判别式;
(C)将对象的该变量的值代入该判别式来计算罹患ME/CFS的概率;和
(D)在该罹患ME/CFS的概率高于规定值的情况下,确定为该对象罹患ME/CFS。
(项目A6-A)根据上述项目中任意一项所述的方法,其中,包括:
(A)确定变量的组合,(A)包括:
(A-1)针对包括肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)的患者以及健康人的被检者,对该健康人和该ME/CFS的患者进行比较,提供包含检测到显著性差异的BCR的IgGH链可变区中的1个以上的基因的使用频率在内的多个变量,和/或
(A-2)将该BCR的IgGH链可变区中的1个基因作为独立变量进行单变量逻辑分析,或将该BCR的IgGH链可变区中的2个以上的基因作为独立变量实施多变量逻辑分析,得到逻辑回归模型方程式,实施对该逻辑回归模型方程式的拟合度进行测量的ROC分析,选择显示规定值以上的AUC值的基因作为判别式用的变量;
(B)提供对该判别式用的变量进行多变量分析而生成的判别式,(B)包括:
(B-1)对该判别式用的变量以患者/健康人的区分作为目标变量进行单变量或多变量逻辑回归,
(B-2)从在B-1的该逻辑回归中生成的Logit模型方程式的常数以及偏回归系数算出判别式的常数以及系数,和
(B-3)基于在B-2的处理中得到的常数以及系数来生成判别式;
(C)将对象的该变量的值代入该判别式来计算罹患ME/CFS的概率;和
(D)在该罹患ME/CFS的概率高于规定值的情况下,确定为该对象罹患ME/CFS。
(项目A7)根据上述项目中任意一项所述的方法,其中,包括:
(A)关于肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS),提供包含BCR的IgGH链可变区中的1个以上的基因的使用频率在内的多个变量;
(B)提供对该变量进行多变量分析而生成的判别式;
(C)将对象的该变量的值代入该判别式来计算罹患ME/CFS的概率;
(AA)提供包含肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)以外的疾病的BCR的IgGH链可变区中的1个以上的基因的使用频率在内的多个变量;
(BB)提供对该变量进行多变量分析而生成的判别式;
(CC)将对象的该变量的值代入该判别式来计算罹患ME/CFS以外的疾病的概率;和
(D)在该罹患ME/CFS的概率高于规定值的情况且在该罹患ME/CFS以外的疾病的概率低于规定值的情况下,确定为该对象罹患ME/CFS。
(项目A8)一种方法,其是对对象罹患肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)进行诊断的体外方法,其中,包括:根据对象的B细胞受体(BCR)库,对该对象罹患肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)进行诊断。
(项目A8-1)根据上述项目所述的方法,其中,具有上述项目中的1个或多个所述的特征。
(项目B1)一种程序,其中,包含下述指令:在由1个以上的处理器进行执行时,使处理器获得包含与对象的BCR库相关联的变量在内的1个以上的变量,根据该1个以上的变量,判断该对象是否为ME/CFS。
(项目B1-1)根据上述项目所述的程序,其中,具有上述项目中的1个或多个所述的特征。
(项目B2)一种存储介质,其中,记录有含有下述指令的程序,所述指令是在由1个以上的处理器进行执行时,使处理器获得包含与对象的BCR库相关联的变量在内的1个以上的变量,根据该1个以上的变量,判断该对象是否为ME/CFS。
(项目B2-1)根据上述项目所述的存储介质,其中,具有上述项目中的1个或多个所述的特征。
(项目B2-2)根据上述项目中任意一项所述的存储介质,其是非临时存储介质。
(项目C1)一种系统,其中,具备:
记录部,被构成为对包含与对象的BCR库相关联的变量在内的1个以上的变量的信息进行记录;和
判断部,被构成为获得该信息并判断该对象是否为ME/CFS。
(项目C1-1)根据上述项目所述的系统,其中,具有上述项目中的1个或多个所述的特征。
发明效果
根据本发明,可以确切地诊断肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)。另外,本发明也可以活用于ME/CFS的发病预测、预后诊断,也可以用作开发ME/CFS的治疗药时的标志物。
附图说明
图1是表示从ME/CFS疾病患者以及健康对照者样品得到的、BCR的各IGHV基因(IGHV家族)的使用频率的比较结果的图。纵轴为各基因的使用频率(%),棒表示标准误差。HC:健康对照者,ME/CFS:ME/CFS疾病患者。
图2是表示从ME/CFS疾病患者以及健康对照者样品得到的、BCR的各IGHD以及IGHJ家族的使用频率的比较结果的图。纵轴为各基因的使用频率(%),棒表示标准误差。HC:健康对照者,ME/CFS:ME/CFS疾病患者。
图3A是ME/CFS疾病患者以及健康对照者样品的、所记载的IGHV基因的使用频率的点图(dot plot)。HC:健康对照者,ME/CFS:ME/CFS疾病患者。
图3A是ME/CFS疾病患者以及健康对照者样品的、所记载的IGHV基因的使用频率的点图。HC:健康对照者,ME/CFS:ME/CFS疾病患者。
图4是ME/CFS疾病患者以及健康对照者样品的、所记载的B细胞群的数量的点图。HC:健康对照者,ME/CFS:ME/CFS疾病患者。
图5是ME/CFS疾病患者以及健康对照者样品的、调节性T细胞群的数量的点图。HC:健康对照者,ME/CFS:ME/CFS疾病患者。
图6是ME/CFS疾病患者以及健康对照者样品的、所记载的T细胞群的数量的点图。HC:健康对照者,ME/CFS:ME/CFS疾病患者。
图7是表示将IGHV1-3、IGHV3-30、IGHV3-30-3、IGHV3-49、IGHD1-26、以及IGHJ6的使用频率、和B细胞量(%)用作变量时的、基于回归分析的ROC曲线的图。
图8是表示将IGHV1-3、IGHV3-30、IGHV3-30-3、IGHV3-49、IGHD1-26、以及IGHJ6的使用频率、和Treg量(%)用作变量时的、基于回归分析的ROC曲线的图。
图9是表示ME/CFS患者(n=37)和MS患者(n=10)之间的IGH基因使用频率的差异的图。ME/CFS:ME/CFS疾病患者,MS:MS疾病患者。
图10是表示ME/CFS患者(n=37)和MS患者(n=10)之间的多样性指数的差异的图。ME/CFS:ME/CFS疾病患者,MS:MS疾病患者。针对任意多样性指数,均未在ME/CFS患者(n=37)和MS患者(n=10)之间检测到显著性差异。
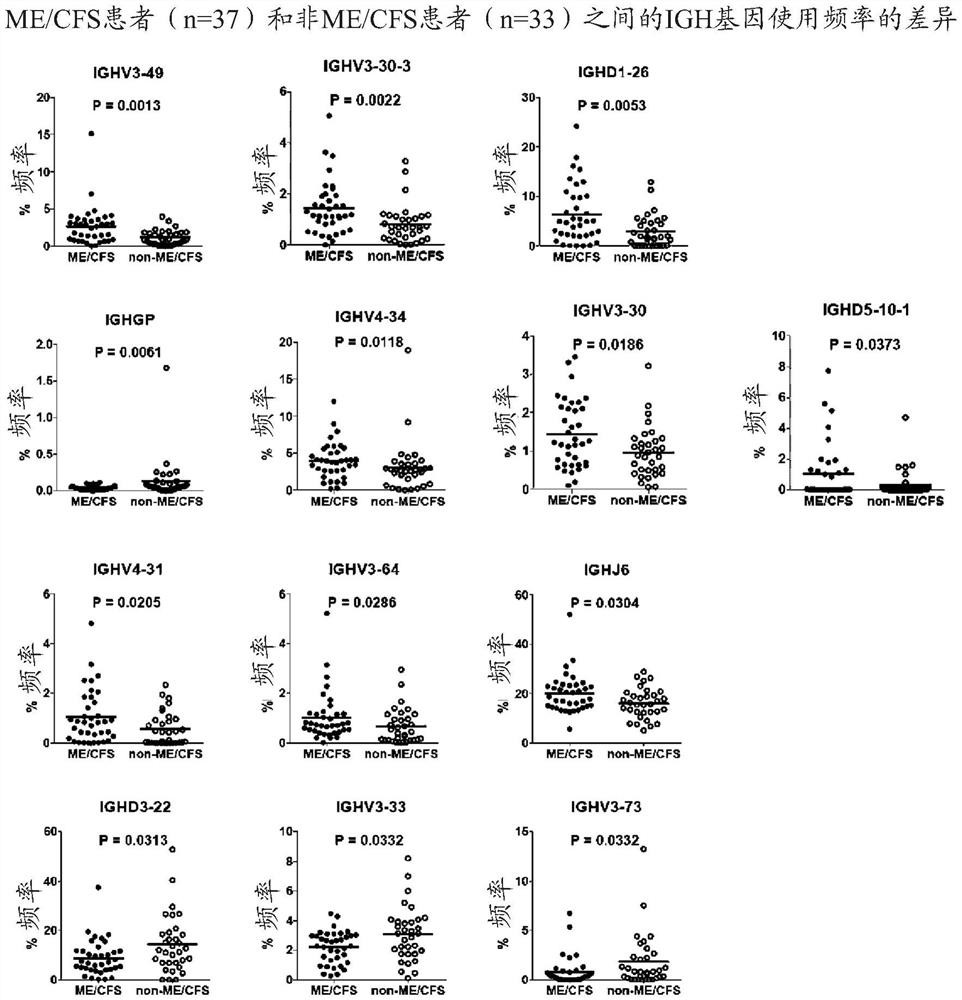
图11是表示ME/CFS患者(n=37)和非ME/CFS患者(n=33)的IGH基因使用频率的差异的图。ME/CFS:ME/CFS疾病患者,非ME/CFS:健康对照+MS疾病患者。
具体实施方式
以下,边示出最佳实施方式边对本发明进行说明。就整个本说明书而言,关于单数形式的表达,只要没有特别说明,则应该理解为也包括其复数形式的概念。因此,关于单数形式的冠词(例如,在英语的情况下,“a、an、the”等),只要没有特别说明,则应该理解为也包括其复数形式的概念。另外,关于本说明书中使用的用语,只要没有特别说明,则应该理解为以该领域中通常使用的含义来使用。因此,只要没有其他定义,本说明书中所使用的全部专业用语以及科学技术用语的含义与由本发明所属领域的本领域技术人员通常理解的含义相同。在矛盾的情况下,本说明书(包括定义在内)优先。
以下适当说明本说明书中特别使用的用语的定义和/或基本的技术内容。
(ME/CFS)
肌痛性脑脊髓炎/慢性疲劳综合症(myalgic encephalomyelitis/chronicfatigue syndrome:ME/CFS)是以持续6个月以上的难以说明的重度疲劳、劳作后的极度疲惫、睡眠障碍、认知功能障碍为核心症状并且时常伴随直立不耐受、疼痛、消化系统症状、对光、声音、气味、化学物质的过敏症等的严重慢性疾病,推测日本国内患者数量为10万人以上,但是病状尚不明确,并且没有疾病特异性的检査法、有效的治疗方法,因此现状是没受到适当的医学治疗。
近年来挪威报道了基于利妥昔单抗(rituximab)的B细胞去除疗法是有效的,ME/CFS的免疫病状受到全世界关注。获得性免疫系统中B细胞统管抗体产生,为了应对多样性抗原,基于基因重排得到的B细胞受体极为多样。在全身性红斑狼疮(systemic lupuserythematosus)之类的自身免疫疾病、B细胞系的血液肿瘤中,特定的B细胞受体(克隆)增加,可作为个体的多样性B细胞受体的总体(库)中的偏差被检出。近年来,能够通过使用新一代测序仪来排除偏差的方法进行库分析,有可能对于检测ME/CFS的B细胞系的异常是有用的。
ME/CFS的诊断是根据症状、问诊等并通过福田标准(Fukuda criteria)、加拿大标准(Canadian criteria)以及国际共识标准(International consensus criteria)等标准进行的。就ME/CFS而言,表示疾病的生物标志物截至今日尚未被开发出来,有通过组合症状进行诊断的多种诊断标准存在。福田标准是最早(1994年)作成的,接下来作成加拿大标准(2003年),随后作成国际标准(2011年),在此期间,专家针对疾病的基本症状(核心症状)已达成共识,目前也正在研究作成新的诊断标准。认为诊断标准包括旧标准并存的理由在于需要以下两个标准:1)用于推进研究并促进疾病的病状理解、治疗法开发的详细/严密的标准(研究标准);2)用于使普通内科医生作出诊断以能够推进面向患者的医疗/社会性干预的简易标准(诊疗用标准)。具体而言,福田标准作为研究标准仍被认为是当下研究用途必需的,而新标准着眼于诊疗。这种双重标准在其他疾病中通常是不被认可的,但在ME/CFS中例外地被专家们认可/接受。日本厚生劳动省作成的诊断标准是以这些海外标准为基础并结合了日本的实际情况的诊断标准(PS的设定等)。在本发明中,ME/CFS可以指由作为研究标准的福田标准规定的疾病,但也可以根据需要参考由其他的诊疗用标准规定的疾病。在本发明中,可以通过本领域中所接受的任意诊断标准来确诊ME/CFS。
就本发明而言,对于表现为ME/CFS的对象、表现有可能为ME/CFS的对象、表现有可能ME/CFS发病的对象、或表现罹患预后不良的ME/CFS的对象,可以进行适当地处置。作为ME/CFS的处置,可以使用免疫调节剂(利妥昔单抗等)、非类固醇系抗炎药、抗抑郁药、以及抗焦虑药等。
(BCR库)
就一个实施方式而言,本发明提供以B细胞受体(BCR)库作为对象的肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)的指标的方法。在本说明书中,“B细胞受体(BCR)”也被称为B细胞受体、B细胞抗原受体(receptor)、B细胞抗原受体,是指由与膜结合型免疫球蛋白(mIg)分子缔合的Igα/Igβ(CD79a/CD79b)异二聚体(α/β)构成。mIg亚基(subunit)与抗原结合,引起受体的凝集,另一方面,α/β亚基向细胞内传递信号。如果BCR凝集,则可以说与酪氨酸激酶的Syk以及Btk同样地使Src家族激酶的Lyn、Blk、以及Fyn快速活化。由于BCR信号传递的复杂性而产生很多不同的结果,其中,包括存活、耐受性(无反应性(anergy);缺乏对抗原的过敏反应)或细胞凋亡(apoptosis)、细胞分裂、向抗体产生细胞或记忆B细胞的分化等。TCR的可变区的序列不同的T细胞也会生成几亿种,另外,BCR(或抗体)的可变区的序列不同的B细胞也会生成几亿种。TCR和BCR各自的序列因基因组序列的重排、突变导入而不同,因此,关于T细胞、B细胞的抗原特异性,可以通过确定TCR·BCR的基因组序列或mRNA(cDNA)的序列来获得线索。
在本说明书中,“V区”是指TCR链或BCR链的可变区的可变部(V)区域。
在本说明书中,“D区”是指TCR链或BCR链的可变区的D区域。
在本说明书中,“J区”是指TCR链或BCR链的可变区的J区域。
在本说明书中,“C区”是指TCR链或BCR链的恒定部(C)区域。
在本说明书中,“可变区的库(repertoire)”是指由TCR或BCR通过基因重排所任意构建的V(D)J区的集合。以TCR库、BCR库等惯用语来使用,它们有时也被称作例如T细胞库、B细胞库等。库可以说具有就整体而言多样性如何的信息和各个基因以何种程度的频率使用的信息这两个方面。就一个实施方式而言,提供如下方法:将包含对象的BCR的IgGH链可变区中的1个以上的基因的使用频率在内的1个以上的变量,作为对象的肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)的指标。BCR的IgGH链可变区中的1个以上的基因的使用频率可以如下所示导出。
作为BCR库的确定方法,一个方法是:对于样品中的B细胞如何使用各个V链而言,使用特定的Vβ链特异性抗体,用流式细胞仪(flow cytometry)对表达各个Vβ链的T细胞的比例进行分析(FACS分析)。另外,可以基于从人基因组序列获得的TCR基因的信息来设计基于分子生物学方法的TCR库分析。一种方法是从细胞样品提取RNA,合成互补DNA后,对TCR基因进行PCR扩增而进行定量。
核酸从细胞样品的提取可以使用总RNA小提试剂盒(RNeasy Plus UniversalMini Kit(QIAGEN))等本技术领域中公知的工具进行。可以使用总RNA小提试剂盒(QIAGEN)从已溶解于TRIzolLS试剂的细胞进行总RNA的提取以及纯化。由提取的RNA向互补DNA的合成可以使用Superscript III
BCR基因的PCR扩增可以由本领域技术人员使用本技术领域中公知的任意聚合酶来适当进行。但是,就BCR基因之类的变动大的基因的扩增而言,如果可以“无偏差地”进行扩增,则可以说有对于准确测定有利的效果。
作为用于PCR扩增的引物,使用设计多种各个BCR V链特异性引物并分别用实时PCR法等进行定量的方法、或将这些特异性引物同时扩增的方法(Multiple PCR:多重PCR)。但是,即便在对各V链使用内源性对照进行定量的情况下,如果利用的引物多,则不能进行准确的分析。进而,在多重PCR法中,存在引物间扩增效率的差异导致PCR扩增时的偏差的缺点。
在本发明的优选实施方式中,利用如WO2015/075939(Repertoire Genesis Inc.)所记载那样的、含有1种正向引物和1种反向引物的1组引物,对含有全部的同型(isotype)、亚型(subtype)基因的BCR基因在不改变存在频率的情况下进行扩增,确定BCR多样性。以下所述的引物设计对于无偏差地扩增是有利的。
着眼于BCR基因的基因结构,不对具有高度多样性的V区设定引物,通过在其5’末端附加衔接子序列(adapter sequence),对含有全部V区的基因进行扩增。该衔接子是碱基序列上任意的长度和序列,大约20个碱基对是最佳的,也可以使用10个碱基~100个碱基的序列。在3’末端附加的衔接子被限制酶(restriction enzyme)去除,通过序列与20个碱基对的衔接子相同的衔接子引物和对作为共有序列(consensus sequence)的C区具有特异性的反向引物进行扩增,从而对全部的BCR基因进行扩增。
由BCR基因信使RNA通过逆转录酶合成互补链DNA,接着合成双链互补DNA。通过逆转录反应、双链合成反应而合成含有不同长度V区的双链互补DNA,通过DNA连接酶反应向这些基因的5’末端部附加包含20个碱基对和10个碱基对的衔接子。
关于BCR,可以对μ链、α链、δ链、γ链、ε链的重链、κ链、λ链的轻链的C区设定反向引物来对这些基因进行扩增。对C区设定的反向引物与BCR的各Cμ、Cα、Cδ、Cγ、Cε、Cκ、Cλ的序列匹配,并且设定具有不启动其他C区序列的程度的错配(mismatch)的引物。关于C区的反向引物,为了能够与衔接子引物进行扩增,考虑碱基序列、碱基组成、DNA融解温度(Tm)、有无自身互补序列而最佳地制备。通过对C区序列中的等位基因(allele)序列间不同的碱基序列以外的区域设定引物,可以对全部的等位基因进行均匀扩增。为了提高扩增反应的特异性,进行多步嵌套PCR(nested PCR)。
针对任一引物均不包含等位基因序列间不同的序列的序列,引物候选序列的长度(碱基数量)没有特别限制,但为10~100的碱基数,优选为15~50的碱基数,更优选为20~30的碱基数。使用这样的无偏差地扩增对于低频率(1/10000~1/100000或其以下)的基因的鉴定是有利的,故优选。
通过对如上所述扩增的BCR基因进行测序,可以从得到的读段(read)数据确定BCR库。
测序的方法只要能确定核酸样品的序列,就没有特别限定,可以利用本技术领域公知的任意方法,但优选使用新一代测序(NGS)。作为新一代测序,可以举出焦磷酸测序、基于合成的测序(边合成边测序:Sequencing by synthesis)、基于连接反应(ligation)的测序、离子半导体测序等,但不限于这些测序。
将得到的读段数据与包含V、D、J基因的参考序列进行比对,由此可以导出独特读段(unique read)数而确定BCR库。
在一个实施方式中,所使用的参考数据库针对V、D、J、C基因区域分别来准备。典型而言,使用由IMGT公开的每个区域、每个等位基因的核酸序列数据集,但不限于此,只要是各序列被分配了唯一ID的数据集,就可以利用。
将得到的读段数据(包括根据需要进行修剪(trimming)等适当处理的数据)作为输入序列集,对每个基因区域的参考数据库进行同源性检索,记录与最接近的参考等位基因及其序列的对齐方式。在此,同源性检索除了C还使用错配容许性高的算法。例如在使用通常的BLAST作为同源性检索程序的情况下,针对每个区域进行缩短窗口尺寸、减少错配罚分、减少空位罚分这样的设置。就最接近的参考等位基因的选择而言,以同源性得分、对齐长度、核心(kernel)长度(连续且匹配的碱基列的长度)、匹配碱基数作为指标,这些指标按照规定的优先顺序进行应用。关于本发明中使用的V以及J所确定的输入序列,将参考V上的CDR3开头以及参考J上的CDR3末尾作为标记,提取CDR3序列。通过将其翻译成氨基酸序列,从而用于D区的分类。在可以准备D区的参考数据库的情况下,将同源性检索结果和氨基酸序列翻译结果的组合作为分类结果。
基于上述情况,对输入集中的各序列分配V、D、J、C的各等位基因。接着,算出在整个输入集中V、D、J、C的各出现频率、或者其组合的出现频率,由此导出BCR库。根据对分类所要求的精确度,按等位基因单位或者基因名单位计算出现频率。后者可以通过将各等位基因翻译成基因名来实现。
在向读段数据分配V区、D区、J区、C区之后,统计匹配的读段,按独特读段(与其他不具有相同序列的读段)可以计算出样品中检测到的读段数以及在总读段数中所占的比例(频率)。
此外,可以使用样本数、读段种类、读段数等数据、并且使用ESTIMATES或R(vegan)等的统计分析软件而算出多样性指数或相似性指数。在优选实施方式中,使用TCR库分析软件(Repertoire Genesis Inc.)。
在本说明书中,“BCR多样性”是指某被检者的B细胞受体的库(repertoire)的多样性,本领域技术人员可以使用该领域公知的各种方法进行测定。将表示BCR多样性的指数称为“BCR多样性指数”。作为BCR多样性指数,有该领域中公知的任意指数,例如可以举出将香农-韦弗指数(Shannon-Weaver index)、辛普森指数(Simpson index)、逆辛普森指数(Inverse Simpson index)、皮洛均匀度指数(Pielou's species evenness index)、标准化香农-韦弗指数(Normalized Shan non-Weaver index)、DE指数(例如DE50指数、DE30指数、DE80指数)或Unique指数(例如Unique 50指数、Unique 30指数、Unique 80指数)等多样性指数应用于BCR后的指数。
(大规模高效率BCR库分析)
在本发明的优选实施方式中,使用大规模高效率BCR库分析,对BCR库进行测定。就本说明书而言,“大规模高效率库分析”记载于WO2015/075939(该文献公开的全部内容根据需要作为参考援用于本说明书中),在对象为BCR的情况下称为“大规模高效率BCR库分析”。就大规模高效率库分析而言,是使用数据库对被检者的库(Repertoire)(T细胞受体(TCR)或B细胞受体(BCR)的可变区)进行定量分析的方法,该方法是使用数据库对被检者的T细胞受体(TCR)或B细胞受体(BCR)的可变区的库(repertoire)进行定量分析的方法,该方法包括(1)提供包含从该被检者无偏差地扩增的T细胞受体(TCR)或B细胞受体(BCR)的核酸序列的核酸样品的工序、(2)确定该核酸样品中所含的该核酸序列的工序、以及(3)根据所确定的该核酸序列算出各基因的出现频率或其组合、并导出该被检者的TCR或者BCR库的工序,该核酸样品包含多种T细胞受体(TCR)或B细胞受体(BCR)的核酸序列,该工序(2)可以通过如下方法来实现,即,通过使用通用衔接子引物确定单一序列,从而确定所述核酸序列的方法;优选的是:该方法包括(1)提供包含从该被检者无偏差地扩增的T细胞受体(TCR)或B细胞受体(BCR)的核酸序列的核酸样品的工序、(2)确定该核酸样品中所含的该核酸序列的工序、以及(3)根据所确定的该核酸序列算出各基因的出现频率或其组合并导出该被检者的库的工序,所述(1)包括以下的工序:(1-1)以源自靶细胞的RNA样品作为模板来合成互补DNA的工序;(1-2)以该互补DNA为模板来合成双链互补DNA的工序;(1-3)对该双链互补DNA附加通用衔接子引物序列来合成附加衔接子的双链互补DNA合成的工序;(1-4)使用该附加衔接子的双链互补DNA、包含该通用衔接子引物序列的通用衔接子引物、和第一TCR或BCR的C区特异性引物,进行第一PCR扩增反应的工序,在该工序中,该第一TCR或BCR的C区特异性引物被设计成含有对该TCR或BCR的目标C区具有充分特异性且与其他基因序列无同源性的序列、且在被扩增的情况下在下游且在亚型间含有不匹配碱基;(1-5)使用(1-4)的PCR扩增产物、该通用衔接子引物、和第二TCR或BCR的C区特异性引物,进行第二PCR扩增反应的工序,在该工序中,该第二TCR或BCR的C区特异性引物被设计成在该第一TCR的C区特异性引物的序列的下游的序列中具有与该TCR或BCR的C区完全匹配的序列、但是还包含与其他基因序列无同源性的序列、且在被扩增的情况下在下游且在亚型间含有不匹配碱基;以及(1-6)使用(1-5)的PCR扩增产物、在该通用衔接子引物的核酸序列中含有第一追加衔接子核酸序列的附加通用衔接子引物、和对第三TCR或BCR的C区特异性序列附加有第二追加衔接子核酸序列以及分子鉴定(MID Tag)序列的带有衔接子的第三TCR的C区特异性引物,进行第三PCR扩增反应的工序,在该工序中,该第三TCR的C区特异性引物被设计成在该第二TCR或BCR的C区特异性引物的序列的下游的序列中具有与该TCR或BCR的C区完全匹配的序列、但是还包含与其他基因序列无同源性的序列、并且在被扩增的情况下在下游且在亚型间含有不匹配碱基,该第一追加衔接子核酸序列是适合于与DNA捕获珠结合以及适合于emPCR反应的序列,该第二追加衔接子核酸序列是适合于emPCR反应的序列,该分子鉴定(MID Tag)序列是用于赋予独特性以能够对扩增产物进行鉴定的序列。该方法的具体详细内容记载于WO2015/075939中,本领域技术人员可以适当参考该文献以及本说明书的实施例等来实施分析。
(诊断)
就本发明的一个实施方式而言,提供以对象的B细胞受体(BCR)库作为对象的肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)的指标的方法。该方法除了包括对象有ME/CFS发病这样的诊断之外,还可以包括提供ME/CFS的发病预测或预后诊断、或者ME/CFS的治疗药开发时的指标。方法可以是体外或计算机模拟(in silico)进行。
方法可以包括使用包含与BCR库有关的变量的1个以上的变量。在使用1个以上的变量作为ME/CFS的指标的情况下,例如,可以使用含有该1个以上的变量的合适公式,对某对象算出该公式的值,并且将该值与适当的标准进行比较等来进行。公式可以通过逻辑回归等求出。作为使用的变量,可以举出从对象的BCR的IgGH链可变区中的1个以上的基因的使用频率、对象的BCR多样性指数、以及对象的1个以上的免疫细胞亚群的数量中选择的1个以上的变量、或2个以上的变量的组合,但不限于这些变量。
就本发明的实施方式而言,优选可以使用包含对象的BCR的1个以上的IGH基因的使用频率在内的1个以上的变量作为ME/CFS的指标。由此,变得可以将该1个以上的变量用作对象罹患ME/CFS而不是其他疾病的指标,(即鉴别诊断)。虽然不希望拘泥于理论,但例如,免疫细胞亚群的数量以及BCR库多样性指数可能因对整个免疫状态产生影响的其他疾病而发生变动,因此可以暗示对象的ME/CFS的存在,但有可能不能排除是其他疾病。另一方面,认为IGH基因的使用频率反映某疾病的机制而发生变化,因此可以成为对象罹患ME/CFS而不是其他疾病的指标,认为在实际临床的诊断中非常有用。
作为相对于ME/CFS的“其他疾病”,除了出现与作为ME/CFS的症状的“以持续6个月以上的难以说明的重度疲劳、劳作后的极度疲惫、睡眠障碍、认知功能障碍作为核心症状并且伴有直立不耐受、疼痛、消化系统症状、对光、声音、气味、化学物质的过敏症等”类似的症状的所有疾病之外,还可以举出对免疫状态可能有影响的任意疾病。例如可以举出精神疾病(例如抑郁症、适应不良(maladjustment)、躯体形式障碍(somatoform disorder))、原发性睡眠障碍(例如睡眠呼吸暂停(sleep apnea)、发作性睡病(narcolepsy))、内分泌疾病(例如垂体功能减退(hypopituitarism)、甲状腺疾病)、感染性疾病(例如AIDS、B型肝炎、C型肝炎等慢性感染性疾病)、自身免疫疾病(例如类风湿性关节炎(rheumatoidarthritis)、全身性红斑狼疮、口眼干燥综合征(Sjogren's syndrome)等)、炎症性疾病(例如炎症性肠病、慢性胰腺炎等慢性炎症性疾病)、神经系疾病(例如多发性硬化症(MS)、自身免疫性脑炎)。就本发明的实施方式而言,可以提供在对象中区分ME/CFS与其他疾病的方法。作为其他疾病的一例,可以举出多发性硬化症(MS)。就本说明书中的区分ME/CFS与其他疾病的方法的实施而言,可以根据需要,应用有关以对象的B细胞受体(BCR)库作为对象的肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)的指标的方法所记载的事项。
ME/CFS的诊断通过“临床症状的组合”来进行,但此时,其他疾病、其他原因的排除是非常重要的程序。会有因生活习惯(不规律、过劳等)而满足“临床症状的组合的标准”的情况,也可能因其他原因例如恶性肿瘤、代谢性疾病以及脑神经疾病等而满足临床症状的标准。因此,如本说明书的实施例所示,认为反映疾病原因的B细胞库或其中的各基因的使用频率被认为在临床诊断中非常有用。例如,认为临床的诊断基准上辅以本说明书所记载的使用BCR库(IGH基因使用频率)的鉴别法,可以进行更准确的鉴别诊断。或者,就本发明的方法而言,可以根据需要组合临床(临床观察、MRI影像观察、脑脊液观察等)信息而使用。在一个实施方式中,对于出现包含ME/CFS的全身症状的患者,可以通过使用IGH基因的使用频率的判别模型或判别式分别判断罹患各疾病的可能性,最终确定1个疾病。
在本说明书中,“灵敏度”是指将应该判断为阳性的情况准确判断为阳性的概率,存在灵敏度高则假阴性降低的关系。如果灵敏度高,则对于排除诊断(rule out)是有用的。
在本说明书中,“特异度”是指将阴性的情况准确判断为阴性的概率,存在特异度高则假阳性降低的关系。如果特异度高,则对于确诊是有用的。
就本说明书而言,“ROC曲线”是指使基于标志物的回归方程得到的临界(cut off)值作为中介变量发生变化时的、对(灵敏度)以及(1-特异度)进行绘制得到的曲线。关于ROC曲线,本领域技术人员可以适当求出AUC(曲线下面积)。认为ROC曲线的AUC示出预测模型的性能。就本说明书的实施例而言,在基于回归分析的ROC曲线中显示AUC≥0.7、AUC≥0.8、以及AUC≥0.9的高预测性能的变量或变量的组合已被验证多个。如果AUC为7左右,则认为有作为诊断模型使用的可能性。
在本发明中,通过使用就对象而言用作ME/CFS的指标的、在基于回归分析的ROC曲线中显示AUC≥0.7的1个以上的变量(根据需要可以是变量的组合),可以对迄今为止仍难诊断的ME/CFS进行诊断。
方法可以包括获得就对象而言用作ME/CFS的指标的1个以上的变量的工序。就一个实施方式而言,获得变量的工序可以包括对对象的样品进行分析的工序,例如,可以包括对对象的BCR的库进行测定的工序、和/或对对象的细胞亚群的数量进行测定的工序。对BCR的库进行测定的工序可以包括确定对象的BCR多样性的工序、和/或确定对象的BCR的IgGH链可变区中的1个以上的基因的使用频率的工序。对象的细胞亚群的数量可以通过包括流式细胞计量术在内的本领域技术人员公知的任意方法进行测定。另外,作为变量,也可以获得与以前对对象所确定的值有关的数据。
方法可以包括根据本说明书记载的1个以上的变量判断对象是否为ME/CFS的工序。判断的工序可以通过将该包含1个以上的变量的函数的因变量的值与适当的阈值进行比较来进行。
就一个实施方式而言,本说明书记载的方法可以通过计算机模拟来进行。方法包括:获得包含与对象的BCR库相关联的变量在内的1个以上的变量的工序、和根据该1个以上的变量来判断该对象是否为ME/CFS的工序。实现本说明书记载的任意方法的程序、记录程序的存储介质、或系统也属于本发明的范围内。
就一个实施方式而言,提供包含下述指令的程序、或记录该程序的存储介质,所述指令在由1个以上的处理器执行时,使处理器获得包含与对象的BCR库相关联的变量在内的1个以上的变量,根据该1个以上的变量,判断该对象是否为ME/CFS。也可以提供一种系统,该系统具备:记录部,被构成为对包含与对象的BCR库相关联的变量在内的1个以上的变量的信息进行记录;和判断部,被构成为获得该信息并判断该对象是否为ME/CFS。系统根据需要可以是计算机系统,可以包含在本说明书中记载的程序或记录该程序的存储介质。
(指标)
(IGH基因)
本发明的一个实施方式提供以包含对象的BCR的IgGH链可变区中的1个以上的基因(IGH基因)的使用频率在内的1个以上的变量作为对象的ME/CFS的指标的方法。作为IGH基因,可以使用从由IGHV1-2、IGHV1-3、IGHV1-8、IGHV1-18、IGHV1-24、IGHV1-38-4、IGHV1-45、IGHV1-46、IGHV1-58、IGHV1-69、IGHV1-69-2、IGHV1-69D、IGHV1/OR15-1、IGHV1/OR15-5、IGHV1/OR15-9、IGHV1/OR21-1、IGHV2-5、IGHV2-26、IGHV2-70、IGHV2-70D、IGHV2/OR16-5、IGHV3-7、IGHV3-9、IGHV3-11、IGHV3-13、IGHV3-15、IGHV3-16、IGHV3-20、IGHV3-21、IGHV3-23、IGHV3-23D、IGHV3-25、IGHV3-30、IGHV3-30-3、IGHV3-30-5、IGHV3-33、IGHV3-35、IGHV3-38、IGHV3-38-3、IGHV3-43、IGHV3-43D、IGHV3-48、IGHV3-49、IGHV3-53、IGHV3-64、IGHV3-64D、IGHV3-66、IGHV3-72、IGHV3-73、IGHV3-74、IGHV3-NL1、IGHV3/OR15-7、IGHV3/OR16-6、IGHV3/OR16-8、IGHV3/OR16-9、IGHV3/OR16-10、IGHV3/OR16-12、IGHV3/OR16-13、IGHV4-4、IGHV4-28、IGHV4-30-2、IGHV4-30-4、IGHV4-31、IGHV4-34、IGHV4-38-2、IGHV4-39、IGHV4-59、IGHV4-61、IGHV4/OR15-8、IGHV5-10-1、IGHV5-51、IGHV6-1、IGHV7-4-1、IGHV7-81、IGHD1-1、IGHD1-7、IGHD1-14、IGHD1-20、IGHD1-26、IGHD1/OR15-1a/b、IGHD2-2、IGHD2-8、IGHD2-15、IGHD2-21、IGHD2/OR15-2a/b、IGHD3-3、IGHD3-9、IGHD3-10、IGHD3-16、IGHD3-22、IGHD3/OR15-3a/b、IGHD4-4、IGHD4-11、IGHD4-17、IGHD4-23、IGHD4/OR15-4a/b、IGHD5-5、IGHD5-12、IGHD5-18、IGHD5-24、IGHD5/OR15-5a/b、IGHD6-6、IGHD6-13、IGHD6-19、IGHD6-25、IGHD7-27、IGHJ1、IGHJ2、IGHJ3、IGHJ4、IGHJ5、IGHJ6、IGHG1、IGHG2、IGHG3、IGHG4及IGHGP、以及它们的任意组合构成的组中选择的至少1个基因。
在本说明书的实施例中显示各种IGH基因的使用频率可以成为ME/CFS的指标。就本发明而言,所使用的至少1个IGH基因的数量没有特别限制,可以使用1~117个基因的任意基因数。本发明中用作指标的1个以上的变量可以包含约1、约2、约3、约4、约5、约6、约7、约8、约9、约10、约15、约20、约30、约40、约50、约60、约70、约80、约90、约100、约110、或超出这些数的IGH基因的使用频率。在适当的情况下,1个以上的变量也可以是1个变量。
在一个优选实施方式中,1个以上的IGH基因包含从由IGHV3-73、IGHV1-69-2、IGHV5-51、IGHV4-31、IGHV3-23D、IGHV1/OR15-9、IGHV4-39、IGHD5-12、IGHV3-43D、IGHD4-17、IGHV5-10-1、IGHD4/OR15-4a/b、IGHG4、IGHV1/OR15-5、IGHV3/OR16-9、IGHD1-7、IGHV3-21、IGHD6-6、IGHV3-33、IGHD4-23、IGHV3-30-5、IGHV3-23、IGHD6-13、IGHV3-64D、IGHV3-48、IGHV3-64、IGHG1、IGHV3-49、IGHV3-30-3、IGHD1-26、IGHJ6、IGHV3-30、IGHGP、IGHV1-3以及IGHD3-22构成的组中选择的至少1个基因。更优选的是:1个以上的IGH基因包含从由IGHD6-6、IGHV3-33、IGHD4-23、IGHV3-30-5、IGHV3-23、IGHD6-13、IGHV3-64D、IGHV3-48、IGHV3-64、IGHG1、IGHV3-49、IGHV3-30-3、IGHD1-26、IGHJ6、IGHV3-30、IGHGP、IGHV1-3以及IGHD3-22构成的组中选择的至少1个基因。进一步更优选的是:1个以上的IGH基因包含从由IGHV3-49、IGHV3-30-3、IGHD1-26、IGHJ6、IGHV3-30、IGHGP、IGHV1-3以及IGHD3-22构成的组中选择的至少1个基因。在另一实施方式中,1个以上的基因包含从由IGHV1-3、IGHV3-30、IGHV3-30-3、IGHV3-49、IGHD1-26、以及IGHJ6构成的组中选择的至少1个基因。在本说明书的实施例中暗示上述的基因可以单独用于预测。
在本发明的另一实施方式中,提供使用包含对象的BCR的IgGH链可变区中的2个以上的基因的使用频率在内的变量作为1个以上的变量的方法。认为:通过使用2个以上的基因的使用频率,可以进一步提高方法的精度。本领域技术人员可以鉴于本说明书的记载而选择使用适当的2个以上的基因的使用频率的组合。1个以上的变量可以是按照使在基于回归分析的ROC曲线中显示AUC≥0.7、AUC≥0.8、或AUC≥0.9的方式进行选择的变量。作为基因的组合的一例,可以举出IGHV1-3、IGHV3-30、IGHV3-30-3、IGHV3-49、IGHD1-26、以及IGHJ6。本领域技术人员可以按照本申请说明书记载的方法适当算出某基因的组合在基于回归分析的ROC曲线中的AUC,可以判断某基因的组合是否显示所希望的AUC。回归分析例如可以用于判别正常对照和ME/CFS。
在本说明书的实施例中,关于2种IGH基因的组合,117个基因的任意基因在与合适的其他IGH基因组合的情况下都能够实现在基于回归分析的ROC曲线中显示AUC≥0.7的组合。
(多样性指数)
就本发明而言,在对象的肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)的诊断中,可以代替其他变量或者在其他变量的基础上而使用对象的BCR多样性指数作为变量。
作为BCR多样性指数,可以利用在该领域公知的任意指数,例如可以举出香农-韦弗指数(Shannon-Weaver index)、辛普森指数(Simpson index)、逆辛普森指数(InverseSimpson index)、皮洛均匀度指数(Pielou's species evenness index)、标准化香农-韦弗指数(Normalized Shannon-Weaver index)、DE指数(例如DE50指数、DE30指数、DE80指数)等。
在本说明书的实施例3-2、表7中,在单回归分析中提取了具有0.1≤P<0.2的显著性的若干多样性指数。另外,在本说明书的实施例6中发现:在多个变量的组合中,包含多样性指数的若干变量的组合在ROC分析中显示AUC值0.8以上(表18)。
(细胞亚群)
就本发明而言,在对象的肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)的诊断中,可以代替其他变量或者在其他变量的基础上而使用细胞亚群的数量作为变量。作为细胞亚群数量,例如可以使用免疫细胞亚群的数量。就本说明书而言,“细胞亚群”是指包含多样特性的细胞的细胞群中的具有某些共有特征的任意细胞的集合。关于特定名称已在本技术领域被公知的细胞亚群,也可以使用该用语来表述特定的细胞亚群,也可以记载任意性质(例如细胞表面标志物的表达)来表述特定的细胞亚群。
作为细胞亚群的例子,例如可以举出B细胞、幼稚B细胞、记忆B细胞、浆母细胞、活化幼稚B细胞、过渡性B细胞、调节性T细胞、记忆T细胞、滤泡辅助性T细胞、Tfh1细胞、Tfh2细胞、Tfh17细胞、Th1细胞、Th2细胞、以及Th17细胞,但不限于这些细胞亚群。
细胞亚群的数量可以由本领域技术人员通过例如流式细胞仪来确定。例如,采集样本后,用溶血法或比重离心法除去红血球,之后,使其与荧光标记抗体(针对目标抗原的抗体和其对照抗体)发生反应,充分清洗后,可以使用流式细胞仪进行观察。检测到的散射光、荧光被转换成电信号,通过计算机进行分析。其结果为:FSC的强度表示细胞的大小,SSC的强度表示细胞内结构,由此可以区分淋巴细胞、单核细胞、粒细胞。之后,根据需要对目标细胞群设门,对这些细胞中的抗原表达样式进行研究。就本发明的方法的实施而言,本领域技术人员可以适当识别所示出的细胞的表面标志物来对细胞进行区分或计数。
作为特别有用的细胞亚群,可以举出B细胞以及调节性T细胞(Treg)。就本发明而言,可以在其他变量的基础上或者代替其他变量而使用包含B细胞的数量和/或Treg的数量在内的1个以上的变量作为ME/CFS的指标。
细胞亚群的数量可以使用相对于合适的基准而言的比例。B细胞的数量例如是末梢血单核细胞中的B细胞的频率(%)。Treg的数量例如是全部CD4阳性T细胞中的Treg的频率(%)。
在本说明书的实施例3-2、表7中,在单回归分析中提取了具有0.1≤P<0.2的显著性的若干细胞亚群变量。另外,在本说明书的实施例6中发现:在多个变量的组合中,包含细胞亚群变量的若干变量的组合在ROC分析中显示AUC值0.8以上(表18)。
(组合)
如本说明书所示,可以使用包含对象的BCR的IgGH链可变区中的1个以上的基因的使用频率在内的1个以上的变量作为对象的ME/CFS的指标。1个以上的变量可以是本说明书记载的变量的任意组合。优选包括从由对象的BCR的IgGH链可变区中的1个以上的基因的使用频率、对象的BCR多样性指数、以及对象的1个以上的免疫细胞亚群的数量构成的组中选择的2个以上的变量的组合。2个以上的变量可以是3个以上、4个以上、5个以上、6个以上或超出这些数的任意数量的变量。
在本说明书的实施例中已经验证了多个变量的组合在用于判别正常对照和ME/CFS的基于回归分析的ROC曲线中显示高AUC,本领域技术人员可以适当组合变量并使用显示特定的AUC的组合来实施。在一个实施方式中,变量的组合在基于回归分析的ROC曲线中可显示AUC≥0.7、AUC≥0.8、AUC≥0.85、AUC≥0.9、AUC≥0.95、或AUC≥0.99。本领域技术人员可以按照本申请说明书记载的方法适当算出某变量的组合在基于回归分析的ROC曲线中的AUC,可以判断某变量的组合是否显示所希望的AUC。作为变量的组合的一例,可以举出对象的IGHV1-3、IGHV3-30、IGHV3-30-3、IGHV3-49、IGHD1-26以及IGHJ6的使用频率与B细胞的数量的组合、或者对象的IGHV1-3、IGHV3-30、IGHV3-30-3、IGHV3-49、IGHD1-26以及IGHJ6的使用频率与Treg的数量的组合等,但不限于这些组合。
虽然并非必须,但进一步组合通过单回归分析显示了预测性能的变量可能关系到更高的预测性能。在本说明书的实施例中,关于单回归分析中患者组和对照组之间显示出P<0.2的显著性差异的变量彼此的组合,就2变量的组合而言,组合中有46%在ROC分析中显示AUC≥0.7,在组合3个变量的情况下,组合中有81%在ROC分析中显示AUC≥0.7,在组合4变量的情况下,组合中有96%在ROC分析中显示AUC≥0.7。
在本发明中,对于对象罹患肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)的诊断,例如可以通过以下所述的工序进行。关于肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS),可以提供包含BCR的IgGH链可变区中的1个以上的基因的使用频率在内的多个变量。接着,可以提供对该变量进行多变量分析而生成的判别式。判别式的提供可包括例如如下步骤:对于变量,以患者/健康人的区分作为目标变量进行单变量或多变量逻辑回归;从在逻辑回归中生成的Logit模型方程式的常数以及偏回归系数算出判别式的常数以及系数;和基于在该处理中得到的常数以及系数生成判别式。接着,将对象的变量的值代入判别式,能够计算罹患ME/CFS的概率。在罹患ME/CFS的概率高于规定值的情况下,可以确定为对象罹患ME/CFS。
更详细而言,在本发明中,对于对象罹患肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)的诊断,例如可以通过以下所述的工序进行。在方法中,确定变量的组合。变量的组合的确定可以包括如下步骤:(1)针对包括肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)的患者以及健康人的被检者,对该健康人和该ME/CFS的患者进行比较,提供包含检测到显著性差异的BCR的IgGH链可变区中的1个以上的基因的使用频率在内的多个变量;和/或(2)将该BCR的IgGH链可变区中的1个基因作为独立变量进行单变量逻辑分析,或将该BCR的IgGH链可变区中的2个以上的基因作为独立变量实施多变量逻辑分析,得到逻辑回归模型方程式,实施对该逻辑回归模型方程式的拟合度进行测量的ROC分析,选择显示更高AUC值的基因作为判别式用的变量。
接着,可以提供对判别式用的变量进行多变量分析而生成的判别式。判别式的提供可以包括例如如下步骤:对于变量,以患者/健康人的区分作为目标变量进行单变量或多变量逻辑回归;从在逻辑回归中生成的Logit模型方程式的常数以及偏回归系数算出判别式的常数以及系数;和基于在该处理中得到的常数以及系数生成判别式。接着,将对象的变量的值代入判别式,能够计算罹患ME/CFS的概率。在罹患ME/CFS的概率高于规定值的情况下,可以确定为对象罹患ME/CFS。
(鉴别诊断)
就本发明的实施方式而言,提供在对象中区分ME/CFS与其他疾病的方法。在本发明的一个实施方式中,通过使用包含在含有正常对照以及其他疾病的患者的非ME/CFS组、和ME/CFS组之间产生差异的指标(变量)的公式,从而进行鉴别诊断。在该情况下,例如,可以将健康人+MS疾病设为非ME/CFS组,选择对于与ME/CFS组的鉴别有效的IGH基因,并使用包含该所选基因的1个判别式。可以举出根据本说明书的实施例中的ME/CFS组和非ME/CFS组之间的显著性差异检验而有显著性差异的IGH基因,作为1个以上的变量,可以使用包含从由IGHV3-49、IGHV3-30-3、IGHD1-26、IGHV3-30、IGHJ6、IGHGP、IGHV4-31、IGHV3-64、IGHD3-22、IGHV3-33、IGHV3-73、IGHV5-10-1以及IGHV4-34构成的组中选择的至少1个基因的使用频率在内的变量。
或者,就其他实施方式而言,设想如下形态:在用区分正常对照和ME/CFS患者的判别式进行判别之后,分别代入判别其他疾病(例如MS)的判别式来否定是其他疾病的可能性。此时,进行将本说明书记载的ME/CFS与正常对照区分的判别,之后(或者之前或同时),进一步使用其他判别式,进行与其他疾病的鉴别。即,可以提供包括如下步骤的方法:(a)将1个以上的变量中的一部分作为所述对象的ME/CFS的指标、(b)将1个以上的变量中的一部分作为所述对象是ME/CFS而不是其他疾病的指标。其他疾病可以包括多发性硬化症(MS)。对于多种其他疾病,可以多次进行(b)。
本发明的一个实施方式是将包含对象的BCR的IgGH链可变区中的1个以上的基因的使用频率在内的1个以上的变量作为对象罹患肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)而不是其他疾病(例如多发性硬化症、MS)的指标的方法。可以将这样的方法根据需要与本说明书记载的将ME/CFS和正常对照区分的判别组合进行。在这里,在期望进行与MS的区分的情况下,1个以上的基因可以包含从由IGHV1-3、IGHV3-30、IGHV3-30-3、IGHD1-26、IGHV3-49以及IGHJ6构成的组中选择的至少1个基因。另外,作为上述1个以上的基因,可以是按照在用于判别MS和ME/CFS的基于回归分析的ROC曲线中显示AUC≥0.7、AUC≥0.8、或AUC≥0.9的方式选择的基因。就本发明而言,用于判别MS和ME/CFS的至少1个IGH基因的数量没有特别限制,可以使用1~117个基因的任意基因数。在本发明中用作指标的1个以上的变量可以包含约1、约2、约3、约4、约5、约6、约7、约8、约9、约10、约15、约20、约30、约40、约50、约60、约70、约80、约90、约100、约110、或超出这些数的数量的IGH基因的使用频率。在合适的情况下,1个以上的变量也可以为1个变量。
在本说明书中,“对象”是指成为本发明的诊断或检测等的对象的任意生物。优选的是对象为人。
在本说明书中,“样品”是指从对象得到的任意物质,例如包括末梢血、组织活检样品、细胞样品、淋巴液、唾液、尿等。本领域技术人员可以基于本说明书的记载适当选择优选的样品。
在本说明书中,“或”在能够采用文章中列举的事项的“至少1个以上”时使用。“或者”也同样。在本说明书中,在明确记为“2个值”的“范围内”的情况下,其范围也包括2个值本身。
在本说明书中引用的科学文献、专利、专利申请等参考文献,其整体与分别具体记载的内容同等程度地作为参考被援引于本说明书中。
以上,为了容易理解而示出优选实施方式对本发明进行说明。以下,基于实施例来说明本发明,但上述的说明以及以下的实施例仅仅是以例示的目的来提供,并不是以限定本发明的目的来提供。因此,本发明的范围并不限于本说明书具体记载的实施方式和实施例,而仅受到权利要求书的限定。
实施例
在本说明书的以下记载的实施例中,适当使用以下的缩写。
[表A]
[实施例1]
使用了ME/CFS疾病患者末梢血单核细胞的新一代B细胞受体库分析
1.材料和方法
1.1.末梢血单核细胞分离和RNA提取
从表1所示的ME/CFS疾病患者(37例)以及健康对照者(23例)采集10mL的全血至含肝素的采血管中,通过Ficoll-Paque PLUS密度梯度离心分离对末梢血单核细胞(Peripheral Blood Mononuclear Cells、PBMCs)进行分离。使用总RNA小提试剂盒(Qiagen,Germany)从分离的PBMC中提取全部RNA并纯化。RNA使用Agilent 2100生物分析仪(Agilent)进行定量。
[表1]
表1 ME/CFS患者
体能状况是以0~9的10个阶段评价重症度,9为最严重。关于p值,年龄利用曼-惠特尼U检验(Mann-Whitney U-test)进行计算,性别利用费希尔精确检验(Fisher′s exacttest)进行计算,将p<0.05设为有意义。
1.2.互补DNA以及双链互补DNA的合成
BCR基因的扩增使用衔接子连接PCR(adaptor-ligation PCR)法来实施。通过含限制酶消化部位的寡聚胸腺嘧啶(Oligo dT)引物(BSL-18E:表2)和逆转录酶来合成互补链DNA(cDNA)。接着,使用大肠杆菌DNA连接酶(E.coli DNA Ligase)(Invitrogen)、DNA聚合酶I(DNA polymerase I)(Invitrogen)以及核糖核酸酶H(RNase H)(Invitrogen)进行双链互补链DNA(ds-cDNA)合成。之后,进行基于T4 DNA聚合酶的5’末端平滑化反应,用限制酶NotI切断末端。使用反应产物纯化试剂盒(MinElute Reaction Cleanup Kit(QIAGEN))进行柱纯化之后,通过基于T4连接酶(T4 Ligase)的连接反应附加P20EA/P10EA衔接子,附加有衔接子的ds-cDNA被Not I限制酶消化。
1.3.PCR
为了特异性扩增B细胞受体(B cell receptor、BCR)的免疫球蛋白γ链(IgG)的重链(IGH)基因,使用热循环仪T100(Bio Rad)利用高保真热启动聚合酶预混液(KAPA HiFiHS Ready Mix(日本Genetics))进行三次嵌套PCR。使用P20EA和CG1进行第一次PCR反应,接着使用P20EA和CG2进行第二次PCR反应。进而,通过P22EA-ST1-R以及CG-ST1-R附加测序所需的Tag序列。使用Agencourt AMPure珠除去所带入的残存引物后,利用Nextera XTIndexKit v2Set A(Illumina),附加index。
[表2]
表2引物序列
(自上而下对应于序列号1~7)
1.4.新一代测序分析
扩增产物使用Qubit(注册商标)3.0荧光计(Thermo Fisher Scientific)进行浓度测定,稀释成4nM后,混合一部分PhiX Control v3(Illumina),制成最终制备检体。使用illumina公司Miseq测序仪对Miseq Reagent Kit v3(600Cycle、Illumina)以及最终制备检体进行双端(paired-end)测序。
1.5.基于库分析软件的分析
使用通过Miseq测序获得的1对Fastq碱基序列数据集,通过库分析软件Repertoire Genesis,确定IGH基因V区序列(IGHV)、D区序列(IGHD)、J区序列(IGHJ)与C区序列(IGHC)的对照、以及CDR3序列。将具有相同的IGHV、IGHD、IGHJ、IGHC以及CDR3氨基酸序列的读段作为独特读段,对各样本中的独特读段的复制数进行计数。根据独特读段的统计结果,算出各样本的IGHV、IGHD、IGHJ、IGHC使用频率、以及多样性指数。
2.结果
从ME/CFS疾病患者以及健康对照者样品获得20万~30万个读段的测序数据(表3)。全部读段数、分配读段、框内读段数以及独特读段数在ME/CFS疾病患者和健康对照者之间未见差异。关于从读段数据算出的IGHV、IGHD、IGHJ、IGHC使用频率、以及多样性指数,在ME/CFS疾病患者和健康对照者之间进行了比较。就IGHV而言的IGHV1-3、IGHV3-30、IGHV3-30-3以及IGHV3-49的使用频率在ME/CFS疾病患者中显著高于健康对照者(图1,曼-惠特尼检验的显著性水平:P<0.05、P<0.05、P<0.001以及P<0.001)。就IGHD而言的IGHD1-26、就IGHJ而言的IGHJ6在ME/CFS疾病患者中显示出显著高于健康对照者的频率(图2,P<0.01以及P<0.05)。将点图示于图3A以及图3B中。这些结果暗示:通过新一代BCR库分析,可以将IGHV1-3、IGHV3-30、IGHV3-30-3、IGHD1-26、IGHV3-49以及IGHJ6的使用频率的测定利用于无有效的生物标志物的ME/CFS疾病的鉴别(表4)。
[表3]
表3通过NGS测序获得的读段数
[表4]
表4鉴别ME/CFS疾病的生物标志物候选I
[实施例2]
ME/CFS疾病患者的淋巴细胞的基于流式细胞计量术的比较研究
1.方法
通过[实施例1]所示的方法分离末梢血单核细胞(Peripheral BloodMononuclear Cells、PBMCs)。之后,利用各种荧光色素标记单克隆抗体进行染色,利用流式细胞仪(FACS Canto II以及FACS Aria II flow cytometer(BD Biosciences))算出以下的淋巴细胞亚类(subclas)的频率。(%)B细胞:CD19+细胞/PBMC;幼稚B细胞(nB):CD19+CD27-/CD19+细胞;记忆B细胞(mBs):CD19+CD27+CD180+/CD19+;浆母细胞(plasmablasts)(PBs):CD19+CD27+CD180-CD38high/CD19+;过渡性B细胞(TrB):CD19+CD27-CD24+Mitotracker green high/CD19+;记忆CD4T细胞(mCD4T):CD3+CD4+CD127+CD45RA-/CD3+CD4+;滤胞辅助性T细胞(Tfh):CD3+CD4+CD127+CD45RA-CXCR5+/CD3+CD4+;辅助性T细胞1(Th1细胞):CD3+CD4+CD127+CD45RA-CXCR5-CXCR3+CCR6-/CD3+CD4+CD127+CD45RA-;辅助性T细胞2(Th2细胞):CD3+CD4+CD127+CD45RA-CXCR5-CXCR3-CCR6-/CD3+CD4+CD127+CD45RA-;辅助性T细胞17(Th17细胞):CD3+CD4+CD127+CD45RA-CXCR5-CXCR3-CCR6+/CD3+CD4+CD127+CD45RA-;滤胞辅助性T细胞1(Tfh1细胞):CD3+CD4+CD127+CD45RA-CXCR5+CXCR3+CCR6-/CD3+CD4+CD127+CD45RA-CXCR5+;滤胞辅助性T细胞2(Tfh2细胞):CD3+CD4+CD127+CD45RA-CXCR5+CXCR3-CCR6-/CD3+CD4+CD127+CD45RA-CXCR5+;滤胞辅助性T细胞17(Tfh17细胞):CD3+CD4+CD127+CD45RA-CXCR5+CXCR3-CCR6+/CD3+CD4+CD127+CD45RA-CXCR5+;调节性T细胞(Treg):CD3+CD4+CD45RA-CD127-CD25++/CD3+CD4+。关于得到的频率,在ME/CFS疾病患者和健康人对象者之间进行曼-惠特尼U检验,对2组间的显著性差异进行了检验。将p<0.05设为统计学上有意义。
2.结果
如图4、图5以及图6所示,在疾病组中(%)B细胞显著较高,在疾病组中调节性T细胞(Treg)的频率显著较低,在疾病组中滤胞辅助性T细胞17(Tfh17)的频率显著较高。
[实施例3-1]
利用在2组间观察到显著性差异的IGH基因和细胞亚群频率数据的ME/CFS的预测鉴别
1.方法
添加在ME/CFS疾病患者(37例)以及健康对照者(23例)的2组间检测到显著性差异的6个IGH基因(表5)以及作为细胞亚群的频率数据的(%)B细胞、或(%)调节性T细胞,进行是否可以预测ME/CFS的研究。使用SPSS软件(IBM)实施多变量逻辑分析,使用回归方程的因变量的预测值,制作接收者操作特性(Receiver Operating Characteristic、ROC)曲线。进而,作为这些变量的预测判断的性能评价值,求出ROC曲线下面积(Area Under the Curve、AUC)值。
2.结果
在IGH基因6变量和(%)B细胞中得到AUC值为0.946的良好评价(图7)。另外,在基于使用了IGH基因6变量和(%)调节性T细胞的回归方程的分析中得到AUC值为0.957的极为良好的评价(图8)。
[表5]
表5鉴别ME/CFS疾病的生物标志物候选II
[实施例3-2]
基于单回归分析的ME/CFS疾病的预测性能高的IGH基因以及细胞亚群变量的提取
1.方法
实施将74种IGHV、32种IGHD、6种IGHJ、5种IGHC、4种多样性指数、15种细胞亚群频率数据中的1种作为独立变量并将ME/CFS疾病和健康人对照的二值变量作为因变量的单回归分析。使用的变量示于表6。提取满足显著性水平P<0.05、0.05≤P<0.1、以及0.1≤P<0.2的变量。单回归以及逻辑回归分析使用R的glm(),ROC分析使用R程序包(R package)的pROC。
[表6]
表6用于回归分析以及ROC分析的变量
2.结果
从74种IGHV、32种IGHD、6种IGHJ、5种IGHC、4种多样性指数、15种细胞亚群数据共计136种变量,提取8种IGH基因、2种细胞亚群变量共计8种作为满足显著性水平P<0.05的变量(表7)。满足0.05≤P<0.1的变量是10种IGH基因、4种细胞亚群频率数据。满足0.1≤P<0.2的变量是16种IGH基因、3种多样性指数、2种细胞亚群变量。暗示了单独一种满足显著性水平P<0.2的变量对于预测ME/CFS疾病有效。尤其暗示了单独一种满足显著性水平P<0.05的变量也是对于预测ME/CFS疾病非常有效(表8)。满足显著性水平P<0.2的变量用于基于多变量逻辑回归分析的分析中。
需要说明的是,对若干变量单独进行ROC分析,结果分别得到IGHV3-49:0.764、IGHV3-30-3:0.759、IGHD1-26:0.731、IGHJ6:0.672、IGHV3-30:0.693、IGHV1-3:0.659、Bcell:0.771、Treg:0.817、Shannon:0.617、Inverse:0.636的AUC值。
[表7]
表7在单回归分析中满足了显著性水平的变量
[表8]
表8鉴别ME/CFS疾病的生物标志物候选III
[实施例4]
使用了2种IGH基因的ME/CFS疾病的预测鉴别
1.方法
针对来自74种IGHV、32种IGHD、6种IGHJ以及5种IGHC共计117种IGH基因中的任意2种IGH基因的组合,实施多变量逻辑回归分析,使用各回归方程的因变量的预测值,作成ROC曲线。对于这些ROC曲线,算出表示ME/CFS疾病的预测性能的AUC值,提取AUC值显示0.7或0.8以上的IGH基因的组合、以及用于这些组合的IGH基因列表。逻辑回归分析使用R的glm(),ROC分析使用R程序包的pROC。
2.结果
在使用了任意2种IGH基因的ROC分析中,AUC值显示0.8、0.7以上的IGH基因的组合分别为30组、637组。将通过二变量逻辑回归以及ROC分析而显示AUC≥0.7的变量的组合示于表11中。另外,关于这些组合所使用的IGH基因,AUC值为0.8以上的基因有26个,AUC值为0.7以上的基因有117个(表9)。另外,AUC值显示0.8以上的30组基因组合中有10组(33%)是在单回归分析中显示显著性的变量彼此的组合,有20组(67%)是单方在单回归分析中显示显著性的IGH(表10)。这些结果显示:通过任意组合IGH基因,能够进行ME/CFS患者的预测鉴别。
[表9]
表9能够活用在ME/CFS疾病的预测鉴别中的IGH基因
[表10]
表10基于任意2种IGH基因组合进行的ME/CFS患者的预测
[表11-1]
通过二变量逻辑回归以及ROC分析而显示AUC≥0.7的变量的组合
[表11-2]
[表11-3]
[表11-4]
[表11-5]
[表11-6]
[表11-7]
[表11-8]
[表11-9]
[表11-10]
[表11-11]
[实施例5]
使用多种IGH基因的多变量逻辑回归分析
1.方法
针对来自74种IGHV、32种IGHD、6种IGHJ以及5种IGHC共计117种IGH基因中的任意2种以上的IGH基因的组合,以循环方式实施多变量逻辑回归分析。使用所得到的回归方程的因变量的预测值,作成ROC曲线,算出AUC值。单回归以及逻辑回归分析使用R的glm(),ROC分析使用R程序包的pROC。
2.结果
在使用来自117种IGH的任意2变量的多变量逻辑回归分析以及ROC分析中,总计6786组的组合总数中有30组(0.44%)的AUC值显示0.8以上(表12)。该组合使用的IGH基因是26个基因(表13)。在使用任意3变量的情况下,在260130组中有4469组(1.7%)的AUC值显示0.8以上,有349组(0.13%)的AUC值显示0.85以上,有4组(0.0015%)的AUC值显示0.9以上(表12)。另外,这些组合所使用的IGH基因分别是117个基因(100%)、102个基因(87%)、以及6个基因(5.1%)(表13)。在使用4变量的情况下,有3264组(0.044%)的AUC值显示0.9以上,有99458组(1.4%)的AUC值显示0.85以上,有489529组(6.6%)的AUC值显示0.8以上。在使用4变量的情况下,AUC显示0.8以上的组合中使用全部117个基因之中的至少一个。可活用在ME/CFS疾病的预测鉴别中的IGH基因示于表14中。被认为显示高预测性能的AUC值为0.9以上的基因的组合示于表15以及表16中。
[表12]
表12显示AUC高值的IGH基因的组合数和频率
[表13]
表13能够用于显示AUC高值的组合中的IGH基因的数量和频率
[表14-1]
表14能够活用在ME/CFS疾病的预测鉴别中的IGH基因
[表14-2]
[表15]
表15显示高预测性能(AUC值为0.9以上)的IGH基因的组合(3种)
[表16-1]
表16显示高预测性能(AUC值为0.9以上)的IGH基因的组合(4种)(AUC值的前100位)
[表16-2]
[表16-3]
[实施例6]
基于变量的组合进行的多变量逻辑回归分析
1.方法
使用通过单回归分析而满足显著性水平P<0.2的46种变量(35种IGH、8种细胞亚群以及3种多样性指数),基于2变量、3变量以及4变量的组合实施多变量逻辑回归分析。使用所得到的回归方程的因变量的预测值,作成ROC曲线,算出AUC值。单回归以及逻辑回归分析使用R的glm(),ROC分析使用R程序包的pROC。
2.结果
根据使用了来自所挑选出的46种变量的任意2变量的多变量逻辑回归分析以及ROC分析,有102组(9.9%)的AUC值为0.8以上,有382组(36.9%)的AUC值为0.7以上且小于0.8(表17)。另外,在使用3变量的情况下,有85组(0.6%)的AUC值为0.9以上,有3472组(22.9%)的AUC值为0.8以上且小于0.9,有8826组(58.1%)的AUC值为0.7以上且小于0.8。在使用4变量的情况下,有5330组(3.3%)的AUC值为0.9以上,有63981组(39.2%)的AUC值为0.8以上且小于0.9,有87291组(53.5%)的AUC值为0.7以上且小于0.8。认为:通过组合多种变量,可以提高ME/CFS疾病的预测性能。将2变量且AUC值显示0.8以上的组合示于表18中,将3变量且AUC值为0.9以上的组合示于表19中。与Treg的组合占优势,推测通过在变量中使用Treg而使预测性能变高。
[表17]
表17来自所挑选出的46种变量的显示AUC高值的组合的数量
[表18-1]
表18基于来自46种变量的任意2变量的组合进行的ME/CFS患者的预测性能(AUC≥0.8)
[表18-2]
[表18-3]
[表18-4]
[表19-1]
表19基于来自46种变量的任意3变量的组合进行的ME/CFS患者的预测(AUC≥0.9)
[表19-2]
[表19-3]
需要说明的是,在细胞亚群变量彼此的组合中,AUC最高值为0.87(Treg、Tfh17),但在组合细胞亚群变量和IGH基因的情况下,就Treg+IGHV1-3、Treg+IGHV3-23、Treg+IGHGP而言,得到高于0.87的AUC值。另外,就B细胞量而言,同与Treg组合时的最大值AUC=0.84相比,与IGHV3-49、IGHV3-30-3、IGHD1-26组合时得到较高的AUC值。通过对细胞亚群变量附加使用特定的IGH基因,可以得到比通过细胞亚群变量彼此的组合得到的预测性能更高的预测性能。
[实施例7]
ME/CFS疾病患者与其他疾病患者的鉴别
1.方法
除了在实施例1中实施的37例肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)疾病患者的样品之外,还重新从10例多发性硬化症(MS)患者采集全血。按照实施例1的1.材料和方法记载的方法,进行PBMC的分离和RNA的提取。接着,进行互补链DNA以及双链互补DNA的合成、PCR以及新一代测序分析,之后,通过库分析软件进行分析。根据各样本的读段数据确定IGH基因V区序列(IGHV)、D区序列(IGHD)、J区序列(IGHJ)与C区序列(IGHC)的对照、以及CDR3序列。统计各样本的独特读段数,并对74种IGHV、32种IGHD、6种IGHJ以及5种IGHC的使用频率进行计算。对于读段数据、IGHV、IGHD、IGHJ以及IGHC使用频率,在ME/CFS和MS组间用曼-惠特尼法进行显著性差异检验。使用ME/CFS患者和MS患者的使用频率数据,单回归以及逻辑回归分析利用R的glm,ROC分析利用R程序包的pROC。就ROC分析而言,使用回归方程的因变量的预测值,作出接收者操作特性(ROC)曲线。作为这些变量的预测判断的性能评价值,求出ROC曲线下面积(AUC)值。
2.结果
2-1.ME/CFS患者和MS患者间的IGH基因使用频率的差异
从10例MS患者样品获得14万~28万读段的测序数据(表20)。全部读段数、分配读段、框内读段数以及独特读段数在ME/CFS患者和MS患者之间未见显著性差异。其结果:就IGHV3-7**、IGHV3-33*、IGHV3-73*、IGHV3-NL1*、IGHV4-28*、IGHV4-39*、IGHD4-17*、IGHD5-5*、以及IGHD5-18*而言,与ME/CFS患者相比,MS患者在IGH基因使用频率方面显著较高(曼-惠特尼检验:*P<0.05、**P<0.01)。另一方面,就IGHV3-23**以及IGHV3-23D*而言,与ME/CFS患者相比,MS患者显示显著较低的使用频率(图9)。同样地,对于作为IGH基因以外的变量的多样性指数(香农指数、逆辛普森指数、皮洛指数、DE50指数)在ME/CFS患者和MS患者之间是否存在差异进行了调查(图10)。任一多样性指数均在ME/CFS患者和MS患者之间未见显著性差异。根据这些结果显示:多样性指数在ME/CFS患者和MS患者的鉴别中没有用,另一方面,IGH基因的使用频率反映疾病特异性,对于鉴别有效。
[表20]
表20 MS患者中的测序读段数
2-2.使用了多变量逻辑分析的ME/CFS患者和MS患者间的鉴别
接着,针对在ME/CFS患者和MS患者之间检测到显著性差异的上述11种IGH基因,使用多变量逻辑分析,验证了是否能够鉴别ME/CFS患者和MS患者。利用11种IGH基因中任意2种IGH基因、或任意3种IGH基因的使用频率,进行逻辑回归分析,算出AUC值。将AUC为高值的组合作为预测性能优异的IGH基因的组合列入下表(表21以及表22)中。
[表21]
表21基于在ME/CFS患者和MS患者两组间观察到显著性差异的IGH基因的任意2变量的组合进行的鉴别(AUC≥0.8)
[表22]
表22基于在ME/CFS患者和MS患者两组间观察到显著性差异的IGH基因的任意3变量的组合进行的鉴别(AUC≥0.85)
使用显示能够鉴别健康人和ME/CFS患者的IGH基因的任意2变量以及3变量,通过多变量逻辑分析,调查了是否能够鉴别ME/CFS患者和MS患者(表23~表30)。其结果可知:获得大量显示多变量逻辑模型性能高的AUC高值的组合,能够以高特异性和灵敏度预测ME/CFS患者和MS患者。在使用在健康人和ME/CFS患者之间显示p<0.2的显著性差异的35种IGH基因的任意2变量的情况下,有48个组合显示AUC≥0.8,在使用任意3变量的情况下,有21个组合显示AUC≥0.9。另外,在使用在健康人和ME/CFS患者之间显示p<0.1的显著性差异的18种IGH基因的任意2变量以及3变量的情况下,分别有17个组合(AUC≥0.8)以及21个组合(AUC≥0.875)显示高值。在使用在健康人和ME/CFS患者之间显示p<0.05的显著性差异的8种IGH基因的任意2变量以及3变量的情况下,分别有7个组合(AUC≥0.8)以及22个组合(AUC≥0.8)显示高值。在使用实施例3-1中所试验的6种IGH基因的任意2变量以及3变量的情况下,分别有1个组合(AUC≥0.7)以及11个组合(AUC≥0.7)显示高值。根据以上的结果可以明确:通过使用单独一种或多种的IGH基因使用频率,能够高精度地鉴别ME/CFS患者和MS患者。
[表23-1]
表23基于来自在健康人和ME/CFS患者之间显示p<0.2的显著性差异的IGH中的任意2变量的组合进行的ME/CFS和MS的鉴别(AUC≥0.8)
[表23-2]
[表24]
表24基于来自在健康人和ME/CFS患者之间显示p<0.2的显著性差异的IGH中的任意3变量的组合进行的ME/CFS和MS的鉴别(AUC≥0.9)
[表25]
表25基于来自在健康人和ME/CFS患者之间显示p<0.1的显著性差异的IGH中的任意2变量的组合进行的ME/CFS和MS的鉴别(AUC≥0.8)
[表26]
表26基于来自在健康人和ME/CFS患者之间显示p<0.1的显著性差异的IGH中的任意3变量的组合进行的ME/CFS和MS的鉴别(AUC≥0.85)
[表27]
表27基于来自在健康人和ME/CFS患者之间显示p<0.05的显著性差异的IGH基因中的任意2变量的组合进行的ME/CFS和MS的鉴别(AUC≥0.8)
[表28]
表28基于来自在健康人和ME/CFS患者之间显示p<0.05的显著性差异的IGH中的任意3变量的组合进行的ME/CFS和MS的鉴别(AUC≥0.8)
[表29]
表29基于来自在实施例3-1中所试验的6种IGH中的任意2变量的组合进行的ME/CFS和MS的鉴别(AUC≥0.7)
[表30]
表30基于来自在实施例3-1中所试验的6种IGH中的任意3变量的组合进行的ME/CFS和MS的鉴别(AUC≥0.7)
2-3.基于使用了任意IGH基因的多变量逻辑分析进行的ME/CFS患者和MS患者之间的鉴别
使用来自74种IGHV、32种IGHD、6种IGHJ、5种IGHC共计117种IGH基因的任意2种IGH的组合(组合总数为6786组),实施多项逻辑分析,挑选显示高预测性能的AUC高值的IGH(表31)。显示AUC≥0.9、AUC≥0.8以及AUC≥0.7的组合分别为4组、252组以及1879组。IGHD3-3以及IGHGP这2变量显示的AUC最高,为0.92。接着,使用来自117种IGH基因的任意3种IGH的组合(组合总数为260130组),实施多项逻辑分析,挑选显示高预测性能的AUC高值的IGH(表32)。
[表31-1]
表31基于任意2个IGH基因的组合进行的ME/CFS和MS的鉴别(前50名)
[表31-2]
[表32-1]
表32基于任意3个IGH基因的组合进行的ME/CFS和MS的鉴别(前50名)
[表32-2]
2-4.ME/CFS患者和非ME/CFS患者的比较
为了调查从非ME/CFS患者中鉴别ME/CFS患者的IGH基因,23例健康人加上10例MS患者作为非ME/CFS组,与37例ME/CFS患者组比较IGH基因的指标频率。根据2组间的曼-惠特尼检验,可以明确:就IGHV3-49(P=0.0013)、IGHV3-30-3(P=0.0022)、IGHD1-26(P=0.0053)、IGHV4-34(P=0.0118)、IGHV3-30(P=0.186)、IGHV4-31(P=0.0205)、IGHV3-64(P=0.0286)、IGHJ6(P=0.0304)、以及IGHD5-10-1(P=0.0373)这9种而言,ME/CFS患者比非ME/CFS患者高。另一方面,就IGHGP(P=0.0061)、IGHD3-22(P=0.0313)、IGHV3-33(P=0.0332)、以及IGHV3-73(P=0.0332)而言,非ME/CFS患者组显著较高(表33、图11)。
[表33]
表33在ME/CFS组和非ME/CFS组这两组之间检测到显著性差异的IGH基因
*曼-惠特尼检验
2-5.基于使用任意IGH基因的多变量逻辑分析进行的ME/CFS患者和非ME/CFS患者之间的鉴别
使用来自74种IGHV、32种IGHD、6种IGHJ、5种IGHC共计117种IGH基因的任意IGH的2变量(组合总数为6786组),进行多项逻辑回归分析,挑选对ME/CFS患者和非ME/CFS患者之间的鉴别显示高预测性能的AUC高值的IGH(表34)。另外,也同样挑选使用3变量(组合总数为260130组)时显示AUC高值的IGH(表35)。就任意2变量而言,显示AUC≥0.8以及AUC≥0.7的组合分别为5组以及469组。在使用任意3变量的情况下,显示AUC≥0.85以及AUC≥0.8的组合分别为37组以及1164组。就2变量而言,IGHGP以及IGH3-30-3的组合显示的AUC最高,为AUC=0.820,就任意3变量而言,IGHGP、IGHV3-30以及IGHV3-49的组合显示的AUC最高,为AUC=0.889。
[表34-1]
表34基于任意2变量的组合进行的ME/CFS和非ME/CFS的鉴别(前50名)
[表34-2]
[表35-1]
表35基于任意3变量的组合进行的ME/CFS和非ME/CFS的鉴别(前50名)
[表35-2]
3.用于ME/CFS鉴别诊断的预测判别模型
在诊断时,使用根据BCR库分析得到的IGH基因的使用频率数据,作出用于鉴别ME/CFS患者的预测模型方程式。使用2组间的显著性差异检验或多项逻辑分析中的AUC高值的IGH基因的组合,作成基于逻辑回归方程式的判别模型。鉴别诊断的例子如以下所示。
变量的组合可以例如如下所示地进行确定。
(1)对健康人和ME/CFS患者进行比较,选择检测到显著性差异的变量(例如,BCR的IgGH链可变区中的基因的使用频率);或者
(2)将1个变量(例如BCR的IgGH链可变区中的1个基因)作为独立变量来实施单变量逻辑分析,或将2个以上的变量(例如2个以上的基因)作为独立变量来实施多变量逻辑分析,得到逻辑回归模型方程式。实施对该回归模型的拟合度进行测量的ROC分析,选择显示更高AUC值的变量。
在提供变量的组合的情况下,针对各变量(例如基因的频率数据)(x1,x2,x3,…),将作为ME/CFS患者的(y=1)、或作为健康人的(y=0)设为目标变量,进行单变量或多变量逻辑回归分析。在(2)的情况下,使用多个变量以循环的方式进行逻辑分析。
逻辑回归分析可以利用作为R程序包的广义线性模型(generalized linearmodel)用函数的glm,分析不限于该程序包。在逻辑回归分析中,将作为ME/CFS的概率设为π,得到以下的Logit模型方程式,同时求出b0的常数以及相当于b1~bp的偏回归系数。即,可以使用区分开的患者/健康人和变量(基因频率等)的数据集来确定Logit模型方程式的系数。
[数学式1]
π:作为ME/CFS的概率,b0:常数,b1~bp:偏回归系数
就作为ME/CFS的概率π而言,如果常数以及偏回归系数被确定,则可以通过以下的公式求出。在新获得频率数据的情况下,可以通过输入其值来进行判别预测(将0.5以上作为ME/CFS进行判别预测等)。
[数学式2]
可以进行如下两个步骤的预测方法:用对ME/CFS和健康人进行区分的判别式来预测ME/CFS,进而用对ME/CFS和MS进行区分的判别式来排除MS患者。通过向基本上与上述相同的Logit模型方程式输入频率数据,可以对其概率进行预测。
3-1.一步预测模型
利用1个判别式来预测ME/CFS和非ME/CFS(健康人以及MS患者)。通过下述的任意例或其他预测变量的组合,以一个步骤来进行判别。
[数学式3]
(使用在2组间观察到显著性差异的13种IGH时的例子)
预测变量:IGHV3-49、IGHV3-30-3、IGHD1-26、IGHGP、IGHV4-34、IGHV3-30、IGHV4-31、IGHV3-64、IGHJ6、IGHD3-22、IGHV3-33、IGHV3-73、IGHV5-10-1
判别式:
π:作为ME/CFS的概率,π/(1-π):让步比(odds ratio)
(使用AUC高值2变量的IGH时的例子)
预测变量:IGHGP、IGHV3-30-3
判别式:
(使用AUC高值3变量的IGH时)
预测变量:IGHGP、IGHV3-30、IGHV3-49
判别式:
3-2.两步预测模型
利用判别式1对ME/CFS和健康人进行判别,接着,利用判别式2排除MS等其他疾病患者,由此预测ME/CFS患者。通过下述的任意例或其他预测变量的组合,以两个步骤来进行判别。
1)ME/CFS患者和健康人的判别(判别式1)
[数学式4]
(使用在2组间观察到显著性差异的6种IGH时的例子)
预测变量:IGHV1-3、IGHV3-30、IGHV3-30-3、IGHD1-26、IGHV3-49、IGHJ6
判别式:
(使用AUC高值2变量的IGH时的例子)
预测变量:IGHGP、IGHV3-30-3
(使用AUC高值3变量的IGH时)
预测变量:IGHGP、IGHV3-30-3、IGHV3-49
2)从ME/CFS患者排除其他疾病(MS患者)的排除判别(判别式2)
[数学式5]
预测变量:IGHV3-7、1GHV3-23、IGHV3-23D、IGHV3-33、1GHV3-73、IGHV3-NL1、IGHV4-28、IGHV4-39、IGHD4-17、IGHD5-5、IGHD5-18
判别式:
将2种IGH用作预测变量时的判别式
预测变量:IGHGP、IGHV3-3
将3种IGH用作预测变量时的判别式
预测变量:IGHGP、IGHV3-3、IGHD1-14
(注释)
如上所述,使用本发明的优选实施方式对本发明进行了例示,但可理解为本发明应该仅通过权利要求书来解释其范围。可理解为本说明书中引用的专利、专利申请以及文献的内容本身与被本说明书具体记载的内容同样、并且该内容作为对本说明书的参考而援引于此。
(相关申请)
本申请主张2018年8月22日申请的日本特愿2018-155380号以及2019年3月12日申请的日本特愿2019-44885号的优先权。为了所有目的而将这些申请的全部内容作为参考援引于本说明书中。
产业上的可利用性
本发明可以利用于肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)的诊断和诊断药中。
序列表自由文本
序列号1:BSL18E引物
序列号2:P20EA引物
序列号3:P10EA引物
序列号4:CG1引物
序列号5:CG2引物
序列号6:P22EA-ST1-R引物
序列号7:CG-ST1-R引物
序列表
<110> 国立研究开发法人国立精神·神经医疗研究中心
组库创世纪株式会社
<120> 肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)的生物标志物
<130> EC008PCT
<150> JP 2018-155380
<151> 2018-08-22
<160> 7
<170> PatentIn version 3.5
<210> 1
<211> 35
<212> DNA
<213> 人工序列
<220>
<223> BSL18E 引物
<220>
<221> misc_feature
<222> (35)..(35)
<223> n is a, c, g, t or u
<400> 1
aaagcggccg catgcttttt tttttttttt tttvn 35
<210> 2
<211> 10
<212> DNA
<213> 人工序列
<220>
<223> P20EA 引物
<400> 2
gggaattcgg 10
<210> 3
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> P10EA 引物
<400> 3
taatacgact ccgaattccc 20
<210> 4
<211> 21
<212> DNA
<213> 人工序列
<220>
<223> CG1 引物
<400> 4
caccttggtg ttgctgggct t 21
<210> 5
<211> 21
<212> DNA
<213> 人工序列
<220>
<223> CG2 引物
<400> 5
tcctgaggac tgtaggacag c 21
<210> 6
<211> 55
<212> DNA
<213> 人工序列
<220>
<223> P22EA-ST1-R 引物
<400> 6
gtctcgtggg ctcggagatg tgtataagag acagctaata cgactccgaa ttccc 55
<210> 7
<211> 54
<212> DNA
<213> 人工序列
<220>
<223> CG-ST1-R 引物
<400> 7
tcgtcggcag cgtcagatgt gtataagaga cagtgagttc cacgacaccg tcac 54
- 肌痛性脑脊髓炎/慢性疲劳综合症(ME/CFS)的生物标志物
- 用于治疗或预防纤维肌痛或慢性疲劳综合症的含有二胺氧化酶的组合物
