用于治疗癌症的AMUC_1100和免疫检查点调节剂的组合疗法
文献发布时间:2024-04-18 19:58:53
技术领域
本公开大体上涉及癌症治疗和免疫性提高的领域。
背景技术
免疫检查点疗法为癌症治疗中的一种突破性疗法。免疫检查点包含维持自身耐受性和帮助免疫反应的抑制路径和刺激路径。目前,存在若干经批准用于治疗癌症患者的免疫检查点抑制剂药物,并且更多所述种类的治疗剂正在开发中(Andrews,L.P.等人,《自然·免疫学(Nat Immunol)》,2019.20(11):第1425-1434页)。此外,正在研究刺激检查点路径(如OX40、ICOS、GITR、4-1BB、CD40)的激动剂或靶向肿瘤微环境组分(如IDO或TLR)的分子(Julian A.Marin-Acevedo等人,《血液学和肿瘤学杂志(Journal of Hematology&Oncology)》,11,39(2018))。然而,免疫检查点调节剂药物的功效在各种癌症类型当中不同。举例来说,对抗PD-1抗体的反应率在一些癌症(黑色素瘤、高微卫星不稳定性(microsatellite instability,MSI)肿瘤)中在40%到70%范围内,而反应率在大多数其它癌症中限于10%到25%,或甚至在其它癌症(三阴性乳癌、微卫星稳定(microsatellitestable,MSS)结肠直肠癌等)中限于<10%(Schoenfeld,A.J.等人,《癌细胞(CancerCell)》,2020.37(4):第443-455页;Ciardiello,D.等人,《癌症治疗综述(Cancer TreatRev)》,2019.76:第22-32页;Adams,S.等人,《美国医学会肿瘤学杂志(JAMA Oncol)》,2019)。此外,疾病可以最终在最初对癌症免疫疗法起反应的患者中发生进展。总的来说,仅小部分癌症患者群体可以长期受益于癌症免疫疗法。尽管已有超过一千个临床研究探索用于癌症中的经过批准或开发中的免疫检查点调节剂的组合疗法策略,但尚不存在证实高度有效的策略。确切地说,开发免疫检查点抑制剂的高效和协同组合疗法以实现癌症中的长期和持久反应是一个巨大难题(Xin Yu,J.等人,《自然评论药物发现(Nat Rev DrugDiscov)》,2020.19(3):第163-164页)。
近期的证据表明,肠道微生物群组合物,尤其某些细菌菌株可以预测或增加癌症免疫疗法的功效。将嗜粘蛋白阿克曼氏菌(Akkermansia muciniphila)接种到经抗生素处理的无特定病原体的小鼠中显著增强了抗PD-1治疗黑色素瘤的治疗功效(Routy,B.等人,《科学(Science)》,2018.359(6371):第91-97页)。然而,不清楚是肠道微生物群组合物的哪一种或多种组分发挥增强抗PD-1治疗功效的功能。
仍需要治疗癌症的新颖疗法。
发明内容
在一个方面,本公开提供一种治疗或预防有需要的个体的癌症的方法,其包括:a)向个体施用有效量的能够调节肠道免疫性的肠道免疫性调节剂;以及b)向个体施用有效量的免疫检查点调节剂。
在另一方面,本公开提供一种改善接受免疫检查点调节剂的治疗的个体中癌症的治疗反应的方法,其包括向个体施用有效量的肠道免疫性调节剂,任选地与有效量的免疫检查点调节剂一起施用。
在另一方面,本公开提供一种在个体中诱导或改善肠道免疫性的方法,其包括向个体施用有效量的肠道免疫性调节剂以及有效量的免疫检查点调节剂。
在另一方面,本公开提供一种在接受抗癌治疗并且表现出肠道免疫性降低的个体中改善癌症治疗反应的方法,其包括向个体施用有效量的肠道免疫性调节剂。
在另一方面,本公开提供一种治疗或预防有需要的个体的癌症的方法,其包括向个体施用有效量的Amuc_1100或表达Amuc_1100的经基因工程改造的宿主细胞,以及有效量的免疫检查点调节剂。
在另一方面,本公开提供一种改善接受免疫检查点调节剂的治疗的个体中癌症的治疗反应的方法,其包括向个体施用有效量的Amuc_1100或表达Amuc_1100的经基因工程改造的宿主细胞,任选地与有效量的免疫检查点调节剂一起施用。
在另一方面,本公开提供一种在个体中诱导或改善肠道免疫性的方法,其包括向个体施用有效量的Amuc_1100或表达Amuc_1100的经基因工程改造的宿主细胞以及有效量的免疫检查点调节剂。
在另一方面,本公开提供一种在接受抗癌治疗并且表现出肠道免疫性降低的个体中改善癌症治疗反应的方法,其包括向个体施用有效量的Amuc_1100或表达Amuc_1100的经基因工程改造的宿主细胞。
在某些实施例中,所述Amuc_1100为天然存在的Amuc_1100或其功能等效物。在某些实施例中,所述功能等效物保持至少部分调节肠道免疫性和/或活化toll样受体2(TLR2)的活性。在某些实施例中,所述功能等效物包含所述天然存在的Amuc_1100的突变体、片段、融合物、衍生物、其稳定性提高的等效物或其任意组合。在某些实施例中,所述Amuc_1100包含选自由SEQ ID NO:1、SEQ ID NO:2和SEQ ID NO:3组成的群组的氨基酸序列,或具有与前述氨基酸序列的至少80%序列一致性且仍保持调节肠道免疫性和/或活化toll样受体2(TLR2)的实质活性的氨基酸序列。
在某些实施例中,所述Amuc_1100在选自由以下组成的群组的位置处包含一个或多个突变:R36、S37、L39、D40、K41、K42、I43、K48、E49、K51、S52、R62、S63、K70、E71、L72、N73、R74、Y75、A76、K77、A78、Y86、K87、P88、F89、L90、A91、F103、Q104、K108、T109、F110、R111、D112、K119、K120、K121、N122、L124、I125、W131、L132、G133、F134、Q135、Y137、S138、L150、G151、F152、E153、L154、K155、A156、L160、V161、K163、L164、A165、L169、S170、K171、F172、I173、K174、V175、Y176、R177、W200、T201、L205、E206、F209、Q210、R213、E214、L217、K218、A219、M220、N221、Y229、L230、R237、I238、R242、M243、M244、P245、K255、P256、L268、T269、K287、P288、Y289、M290、K292、E293、F296、V297、F306、N307、K310和A311,其中编号相对于SEQ ID NO:1。在某些实施例中,所述Amuc_1100在选自由以下组成的群组的位置处包含一个或多个突变:S37、V175、Y289和F296,其中编号相对于SEQ ID NO:1。在某些实施例中,Amuc_1100包含Y289A突变,其中编号相对于SEQ ID NO:1。
在某些实施例中,所述免疫检查点调节剂为针对免疫检查点分子的抗体或其抗原结合片段或化合物。在某些实施例中,所述免疫检查点调节剂包含一个或多个选自由以下组成的群组的免疫刺激检查点的活化剂:CD2、CD3、CD7、CD16、CD27、CD30、CD70、CD83、CD28、CD80(B7-1)、CD86(B7-2)、CD40、CD40L(CD154)、CD47、CD122、CD137、CD137L、OX40(CD134)、OX40L(CD252)、NKG2C、4-1BB、LIGHT、PVRIG、SLAMF7、HVEM、BAFFR、ICAM-1、2B4、LFA-1、GITR、ICOS(CD278)、ICOSLG(CD275)和其任何组合。在某些实施例中,所述免疫检查点调节剂包含一个或多个选自由以下组成的群组的免疫抑制检查点的抑制剂:LAG3(CD223)、A2AR、B7-H3(CD276)、B7-H4(VTCN1)、BTLA(CD272)、BTLA、CD160、CTLA-4(CD152)、IDO1、IDO2、TDO、KIR、LAIR-1、NOX2、PD-1、PD-L1、PD-L2、TIM-3、VISTA、SIGLEC-7(CD328)、TIGIT、PVR(CD155)、TGFβ、SIGLEC9(CD329)和其任何组合。在某些实施例中,所述免疫检查点调节剂为一个或多个免疫抑制检查点的抑制剂,优选地抑制或减少PD-1与其配体中的任一种之间的相互作用的抗体或其抗原结合片段或化合物。在某些实施例中,所述免疫检查点调节剂为针对PD-L1、PD-L2或PD-1的抗体或其抗原结合片段或化合物。
在某些实施例中,所述个体已表现出对所述免疫检查点调节剂的不良反应或耐药性。在某些实施例中,所述个体经测定对所述免疫检查点调节剂具有原发性(de novo)耐药或获得性耐药。
在某些实施例中,所述个体是癌症病人,其中所述癌症选自由以下组成的群组:结肠直肠癌、乳癌、卵巢癌、胰腺癌、胃癌、前列腺癌、肾癌、子宫颈癌、骨髓瘤、淋巴瘤、白血病、甲状腺癌、子宫内膜癌、子宫癌、膀胱癌、神经内分泌癌、头颈癌、肝癌、鼻咽癌、睾丸癌、小细胞肺癌、非小细胞肺癌、黑色素瘤、基细胞癌、皮肤癌、鳞状细胞皮肤癌、隆凸性皮肤纤维肉瘤、梅克尔细胞癌(Merkel cell carcinoma)、成胶质细胞瘤、神经胶质瘤、肉瘤和间皮瘤。
在某些实施例中,所述Amuc_1100或表达所述Amuc_1100的经基因工程改造的宿主细胞的施用是通过口服施用。在某些实施例中,所述Amuc_1100或表达所述Amuc_1100的经基因工程改造的宿主细胞的施用在所述免疫检查点调节剂之前、之后或与其同时进行。
在某些实施例中,所述宿主细胞包含益生微生物体或非病原性微生物体。在某些实施例中,所述宿主细胞包含外源性表达盒,所述外源性表达盒包含编码可操作地连接于信号肽的Amuc_1100的核苷酸序列,任选地,其中所述经基因工程改造的宿主细胞从1×10
在另一方面,本公开提供一种包含外源性表达盒的经基因工程改造的宿主细胞,所述外源性表达盒包含编码可操作地连接于信号肽的Amuc_1100的核苷酸序列,任选地,其中所述经基因工程改造的宿主细胞从1×10
在另一方面,本公开提供一种重组表达盒,其包含编码可操作地连接于信号肽的Amuc_1100的核苷酸序列和一个或多个调节元件。
在某些实施例中,所述宿主细胞包含益生微生物体或非病原性微生物体。在某些实施例中,所述外源性表达盒整合于所述经基因工程改造的宿主细胞的质粒中。在某些实施例中,所述外源性表达盒整合于所述经基因工程改造的宿主细胞的基因组中。
在某些实施例中,所述外源性表达盒包含编码可操作地连接于信号肽的Amuc_1100的核苷酸序列,其中所述信号肽可操作地连接于所述Amuc_1100的N端。在某些实施例中,所述信号肽包含选自由SEQ ID NO:76到82和其具有至少80%序列一致性的同源序列组成的群组的氨基酸序列。在某些实施例中,信号肽是Usp45信号肽。在某些实施例中,所述信号肽可以通过存在于所述宿主细胞中的分泌系统加工。在某些实施例中,所述分泌系统对于所述宿主细胞是原生或非原生的系统。在某些实施例中,所述宿主细胞为益生细菌。在某些实施例中,所述分泌系统经工程改造和/或优化以使得至少一种外膜蛋白编码基因被删除、失活或抑制。在某些实施例中,所述外膜蛋白选自由以下组成的群组:OmpC、OmpA、OmpF、OmpT、pldA、pagP、tolA、Pal、To1B、degS、mrcA和lpp。在某些实施例中,所述分泌系统经工程改造以使得至少一种伴侣蛋白编码基因被扩增、过表达或活化。在某些实施例中,所述伴侣蛋白选自由以下组成的群组:dsbA、dsbC、dnaK、dnaJ、grpE、groES、groEL、tig、fkpA、surA、skp、PpiD和DegP。
在某些实施例中,所述表达盒进一步包含一个或多个调节元件,所述调节元件包含一个或多个选自由以下组成的群组的元件:启动子、核糖体结合位点(RBS)、顺反子、终止子和其任何组合。在某些实施例中,所述启动子为组成型启动子或诱导型启动子。
在某些实施例中,所述启动子为内源启动子或外源启动子。在某些实施例中,所述组成型启动子包含选自由SEQ ID NO:14到54和其具有至少80%序列一致性的同源序列组成的群组的核苷酸序列。在某些实施例中,所述组成型启动子包含SEQ ID NO:15。在某些实施例中,所述诱导型启动子包含选自由SEQ ID NO:55到58和其具有至少80%序列一致性的同源序列组成的群组的核苷酸序列。在某些实施例中,所述诱导型启动子包含SEQ ID NO:58。
在某些实施例中,所述RBS包含选自由SEQ ID NO:70到73和其具有至少80%序列一致性的同源序列组成的群组的核苷酸序列。在某些实施例中,所述顺反子包含选自由SEQID NO:66到69和其具有至少80%序列一致性的同源序列组成的群组的核苷酸序列。在某些实施例中,所述终止子为T7终止子。在某些实施例中,启动子是rrnB_T1_T7Te终止子,在某些实施例中,启动子包含如SEQ ID NO:74所示的核苷酸序列。
在某些实施例中,本公开的经基因工程改造的宿主细胞进一步包含营养缺陷相关基因中的至少一个失活或缺失。在某些实施例中,所述营养缺陷相关基因选自由以下组成的群组:thyA、cysE、glnA、ilvD、leuB、lysA、serA、metA、glyA、hisB、ilvA、pheA、proA、thrC、trpC、tyrA、uraA、dapF、flhD、metB、metC、proAB、yhbV、yagG、hemB、secD、secF、ribD、ribE、thiL、dxs、ispA、dnaX、adk、hemH、IpxH、cysS、fold、rplT、infC、thrS、nadE、gapA、yeaZ、aspS、argS、pgsA、yeflA、metG、folE、yejM、gyrA、nrdA、nrdB、folC、accD、fabB、gltX、ligA、zipA、dapE、dapA、der、hisS、ispG、suhB、tadA、acpS、era、rnc、fisB、eno、pyrG、chpR、Igt、ft>aA、pgk、yqgD、metK、yqgF、plsC、ygiT、pare、ribB、cca、ygjD、tdcF、yraL、yihA、ftsN、murl、murB、birA、secE、nusG、rplJ、rplL、rpoB、rpoC、ubiA、plsB、lexA、dnaB、ssb、alsK、groS、psd、orn、yjeE、rpsR、chpS、ppa、valS、yjgP、yjgQ、dnaC、ribF、IspA、ispH、dapB、folA、imp、yabQ、flsL、flsl、murE、murF、mraY、murD、ftsW、murG、murC、ftsQ、ftsA、ftsZ、IpxC、secM、secA、can、folK、hemL、yadR、dapD、map、rpsB、in/B、nusA、ftsH、obgE、rpmA、rplU、ispB、murA、yrbB、yrbK、yhbN、rpsl、rplM、degS、mreD、mreC、mreB、accB、accC、yrdC、def、fint、rplQ、rpoA、rpsD、rpsK、rpsM、entD、mrdB、mrdA、nadD、hlepB、rpoE、pssA、yfiO、rplS、trmD、rpsP、ffh、grpE、yfjB、csrA、ispF、ispD、rplW、rplD、rplC、rpsJ、fusA、rpsG、rpsL、trpS、yr/F、asd、rpoH、ftsX、ftsE、ftsY、frr、dxr、ispU、rfaK、kdtA、coaD、rpmB、djp、dut、gmk、spot、gyrB、dnaN、dnaA、rpmH、rnpA、yidC、tnaB、glmS、glmU、wzyE、hemD、hemC、yigP、ubiB、ubiD、hemG、secY、rplO、rpmD、rpsE、rplR、rplF、rpsH、rpsN、rplE、rplX、rplN、rpsQ、rpmC、rplP、rpsC、rplV、rpsS、rplB、cdsA、yaeL、yaeT、lpxD、fabZ、IpxA、IpxB、dnaE、accA、tilS、proS、yafF、tsf、pyrH、olA、rlpB、leuS、Int、glnS、fldA、cydA、in/A、cydC、ftsK、lolA、serS、rpsA、msbA、IpxK、kdsB、mukF、mukE、mukB、asnS、fabA、mviN、rne、yceQ、fabD、fabG、acpP、tmk、holB、lolC、lolD、lolE、purB、ymflC、minE、mind、pth、rsA、ispE、lolB、hemA、prfA、prmC、kdsA、topA、ribA、fabi、racR、dicA、yd B、tyrS、ribC、ydiL、pheT、pheS、yhhQ、bcsB、glyQ、yibJ和gpsA。
在某些实施例中,本公开的宿主细胞是一种或多种选自由以下组成的群组的物质的营养缺陷体:尿嘧啶、亮氨酸、组氨酸、色氨酸、赖氨酸、甲硫氨酸、腺嘌呤和非天然存在的氨基酸。在某些实施例中,所述非天然存在的氨基酸选自由以下组成的群组:l-4,4′-联苯丙氨酸、对乙酰基-l-苯丙氨酸、对碘-l-苯丙氨酸和对叠氮基-l-苯丙氨酸。
在某些实施例中,本公开的宿主细胞包含变构调节的转录因子,所述转录因子能够检测调节所述转录因子的活性的环境中的信号,其中不存在所述信号将引起细胞死亡。
在某些实施例中,本公开的益生微生物体为益生细菌或益生酵母。在某些实施例中,所述益生细菌选自由以下组成的群组:拟杆菌(Bacteroides)、双歧杆菌(Bifidobacterium)、梭菌(Clostridium)、埃希氏菌(Escherichia)、乳杆菌(Lactobacillus)和乳球菌(Lactococcus),任选地,所述益生细菌属于埃希氏菌属,并且任选地,所述益生细菌属于物种大肠杆菌(Escherichia coli)的菌株Nissle 1917(EcN)。在某些实施例中,所述益生酵母选自由以下组成的群组:酿酒酵母(Saccharomycescerevisiae)、产朊假丝酵母(Candida utilis)、乳酸克鲁维酵母(Kluyveromyces lactis)和卡氏酵母(Saccharomyces carlsbergensis)。
在另一方面,本公开提供一种组合物,其包含本公开的经基因工程改造的宿主细胞和生理学上可接受的载剂。
在另一方面,本公开提供一种组合物,其包含:a)本公开的经基因工程改造的宿主细胞或分离的Amuc_1100;和b)免疫检查点抑制剂。
在某些实施例中,本公开的组合物是可食用的。在某些实施例中,所述组合物是益生组合物。
在另一方面,本公开提供一种试剂盒,其包含:a)包含本公开的经基因工程改造的宿主细胞或包含分离的Amuc_1100的第一组合物;和b)包含免疫检查点抑制剂的第二组合物。
在某些实施例中,所述第一组合物是可食用组合物,和/或所述第二组合物进一步包含药学上可接受的载剂。在某些实施例中,所述第一组合物是食品增补剂。在某些实施例中,所述第二组合物适于口服施用或肠胃外施用。
在另一方面,本公开提供本公开的经基因工程改造的宿主细胞,其用于与免疫检查点调节剂组合使用。
在另一方面,本公开提供一种包含本公开的重组表达盒的核酸运载体,其用于与免疫检查点调节剂组合使用。
在另一方面,本公开提供一种包含本公开中任一项所述的经基因工程改造的宿主细胞或分离的Amuc_1100的口服组合物,其用于与包含免疫检查点调节剂的制剂组合使用。
在某些实施例中,所述包含免疫检查点调节剂的制剂是肠胃外制剂。在某些实施例中,所述包含免疫检查点调节剂的制剂是口服制剂。
在另一方面,本公开提供一种非天然存在的Amuc_1100蛋白,所述Amuc_1100蛋白包含Y289A突变,其中编号相对于SEQ ID NO:1。
在另一方面,本公开提供一种编码Amuc_1100蛋白的核酸分子。
在另一方面,本公开提供Usp45信号肽在构建包含编码目标多肽的表达载体中的应用,其中所述表达载体适合于在大肠杆菌中表达,从而允许目标多肽在大肠杆菌中表达并分泌到大肠杆菌外。
在另一方面,本公开提供Usp45信号肽,用于构建包含编码目标多肽的表达载体中,其中所述表达载体适合于在大肠杆菌中表达,从而允许目标多肽在大肠杆菌中表达并分泌到大肠杆菌外。
在另一方面,本公开提供一种表达载体,其包含编码Usp45信号肽以及与其可操作连接的目标多肽的多核苷酸序列,其中所述表达载体适合于在大肠杆菌中表达,从而允许目标多肽在大肠杆菌中表达并分泌到大肠杆菌外。
在某些实施例中,所述Usp45信号肽包含如SEQ ID NO:59所示的序列。
在某些实施例中,所述大肠杆菌是大肠杆菌的益生株。
在某些实施例中,所述益生株是大肠杆菌Nissle 1917菌株。
在某些实施例中,所述目标多肽包括Amuc_1100。
在某些实施例中,所述Amuc_1100是天然存在的Amuc_1100或其功能等效物。
在某些实施例中,所述目标多肽不包括Amuc_1100。
在另一方面,本公开提供一种经基因工程改造的大肠杆菌,包含外源表达盒,所述外源表达盒包含编码Usp45信号肽以及与其可操作连接的目标多肽的多核苷酸序列。
在某些实施例中,所述经基因工程改造的大肠杆菌,能够表达和分泌目标多肽。
附图说明
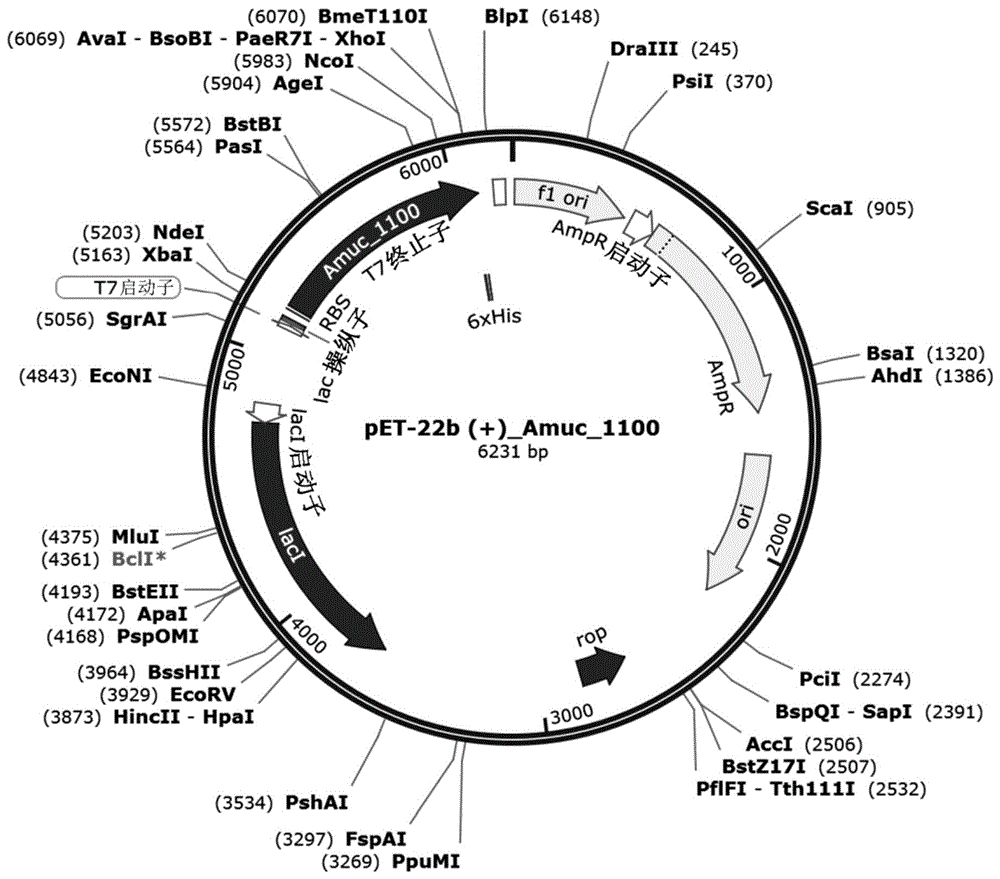
图1展示质粒pET-22b(+)_Amuc_1100的质粒概况。
图2展示SEQ ID NO:145-147的序列。
图3A展示了Amuc_1100(31-317,Y289A)突变体蛋白的稳定性检测结果。利用胰蛋白酶(0.01mg/ml)以及α-胰凝乳蛋白酶(α-chymotrypsin)(0.01mg/ml)在25℃下消化处理重组野生型(WT)Amuc_1100蛋白和突变体蛋白30分钟,随后进行SDS-PAGE凝胶电泳和考马斯亮蓝染色。A泳道:未经蛋白酶处理的Amuc_1100(31-317,Y289A);B泳道:经蛋白酶处理的Amuc_1100(31-317,Y289A);C泳道:未经蛋白酶处理的Amuc_1100(31-317,WT);D泳道:经过蛋白酶处理的Amuc_1100(31-317,WT),MW:分子量标记(KD)。
图3B展示Amuc_1100(31-317)、Amuc_1100(31-317,Y289A)和Pam3CSK4(阳性对照组)的细胞存活率分析结果。
图4展示用于测定分别通过PBS(阴性对照组)、Amuc_1100(31-317)、Amuc_1100(31-317,Y289A)、抗PD-1抗体、Amuc_1100(31-317)和抗PD-1抗体的组合、Amuc_1100(31-317,Y289A)和抗PD-1抗体的组合处理的组织中的肿瘤细胞的H&E染色结果。
图5展示分别通过PBS(阴性对照组)、Amuc_1100(31-317)、Amuc_1100(31-317,Y289A)、抗PD-1抗体、Amuc_1100(31-317)和抗PD-1抗体的组合、Amuc_1100(31-317,Y289A)和抗PD-1抗体的组合处理的每只小鼠的肿瘤生长速率。
图6展示质粒pCBT003_agaI/rsmI的质粒概况。
图7展示质粒pUC57_Amuc_1100的质粒概况。
图8展示质粒pCBT001的质粒概况。
图9展示说明使用CRISPR-Cas9将Amuc_1100(31-317)表达盒的PCR片段整合到EcN的agaI/rsmI位点中的示意图。
图10展示质粒pCBT003_agaI/rsmI_sgRNA的质粒概况。
图11展示说明将一个或多个顺反子插入到Amuc_1100(31-317)表达盒中的示意图。
图12A展示说明将一个或多个自转运蛋白结构域插入到Amuc_1100表达盒中的示意图。
图12B展示在转染了质粒载体的细菌中,携带不同信号肽的Amuc_1100分泌情况的western blot结果。
图13A展示了工程菌中Amuc_1100的分泌情况,其中,A泳道和B泳道:CBT1101,其包含单拷贝受启动子BBA_J23101调控的Amuc_1100(Y289A);C泳道:CBT1102,其包含单拷贝受启动子BBA_J23110调控的Amuc_1100(Y289A);D泳道:CBT1103,其包含双拷贝受启动子BBA_J23110调控的Amuc_1100(Y289A);E泳道:CBT1104,其包含三拷贝受启动子BBA_J23101调控的Amuc_1100(Y289A);F泳道:CBT1105,其包含两拷贝受启动子BBA_J23101调控的Amuc_1100(Y289A)以及一拷贝受启动子BBA_J23110调控的Amuc_1100(Y289A);LANE G:CBT1106,其包含三拷贝受启动子BBA_J23110调控的Amuc_1100(Y289A)。
图13B展示包含一个拷贝厌氧启动子的菌株CBT1107在有氧和无氧条件下Amuc_1100的分泌情况。
图13C展示包含两个拷贝厌氧启动子的菌株CBT1108在有氧和无氧条件下Amuc_1100的分泌情况。
图13D展示包含不同启动子组合的工程菌株在有氧条件下Amuc_1100的分泌情况。
图13E展示包含不同启动子组合的工程菌株在无氧条件下Amuc_1100的分泌情况
图13F展示包含不同拷贝数厌氧启动子的工程菌株在有氧转无氧培养条件下的生长情况。
图13G展示Lpp基因敲除对Amuc_1100蛋白分泌的影响。
图13H展示ompT基因敲除对Amuc_1100蛋白分泌的影响。
图13I展示工程菌株CBT1116或CBT1103的Amuc_1100表达情况。
图14展示说明将Amuc_1100表达盒整合到EcN基因组的适合位点(例如agaI/rsmI)中的示意图。
图15展示整合位点地图。
图16展示一个编码Amuc_1100基因盒的经基因工程改造和优化的EcN的实施例。
图17展示经基因工程改造的EcN分泌的Amuc_1100(31-317)的TLR2报告基因活性结果。
图18展示分别通过未基因改造的EcN(阴性对照组)、编码两个Amuc_1100基因盒的基因工程菌、抗PD-1抗体、编码两个Amuc_1100基因盒的基因工程菌和抗PD-1抗体的组合处理的每只小鼠的肿瘤生长速率。
图19展示pCBT003的核酸序列。
图20展示pCBT001的核酸序列。
具体实施方式
在整个本公开中,冠词“一(a/an)”和“所述”在本文中用于指一个(种)或超过一个(种)(即,至少一个(种))所述冠词的语法对象。举例来说,“一抗体”意指一种抗体或多于一种抗体。
以下对本公开的描述仅打算说明本公开的各种实施例。因此,所论述的具体修改不应被解释为对本公开范围的限制。所属领域的技术人员将显而易见,可在不脱离本公开的范围的情况下得到各种等效物、进行改变和修改,且应理解所述等效实施例将包含在本文中。本文引用的所有参考文献,包含出版物、专利和专利申请,均以全文引用的方式并入本文中。
I.
如本文所用,术语“氨基酸”是指含有胺(-NH
如本文所用,术语“抗体”包含与特异性抗原结合的任何免疫球蛋白、单克隆抗体、多克隆抗体、多价抗体、二价抗体、单价抗体、多特异性抗体或双特异性抗体。原生完整抗体包含两条重(H)链和两条轻(L)链。哺乳动物重链分类为α、δ、ε、γ和μ,每条重链由可变区(VH)以及第一、第二、第三和任选的第四恒定区(分别为CH1、CH2、CH3、CH4)组成;哺乳动物轻链分类为λ或κ,而每条轻链由可变区(VL)和恒定区组成。抗体呈“Y”形,其中Y的主干由通过二硫键结合在一起的两条重链的第二和第三恒定区组成。Y的每个臂包含与单条轻链的可变区和恒定区结合的单条重链的可变区和第一恒定区。轻链和重链的可变区负责抗原结合。两条链的可变区一般含有三个高度可变的环,称为互补决定区(CDR)(轻链CDR包含LCDR1、LCDR2和LCDR3,重链CDR包含HCDR1、HCDR2、HCDR3)。本文中所公开的抗体和抗原结合片段的CDR边界可通过Kabat、IMGT、Chothia或Al-Lazikani惯例来定义或识别(Al-Lazikani,B.,Chothia,C.,Lesk,A.M.,《分子生物学杂志(J.Mol.Biol.)》,273(4),927(1997);Chothia,C.等人,《分子生物学杂志》12月5日;186(3):651-63(1985);Chothia,C.和Lesk,A.M.,《分子生物学杂志》,196,901(1987);Chothia,C.等人,《自然(Nature)》.12月21-28日;342(6252):877-83(1989);Kabat E.A.等人,《免疫学感兴趣的蛋白质的序列(Sequences of Proteins of Immunological Interest)》,第5版,美国国家卫生研究院公共卫生服务部(Public Health Service,National Institutes of Health),马里兰州贝塞斯达(Bethesda,Md.)(1991);Marie-Paule Lefranc等人,《发育与比较免疫学(Developmental and Comparative Immunology)》,27:55-77(2003);Marie-PauleLefranc等人,《免疫组学研究(Immunome Research)》,1(3),(2005);Marie-PauleLefranc,《B细胞的分子生物学(第二版)(Molecular Biology of Bcells(secondedition))》,第26章,481-514,(2015))。三个CDR插在被称为构架区(FR)(轻链FR包含LFR1、LFR2、LFR3和LFR4,重链FR包含HFR1、HFR2、HFR3和HFR4)的侧接链段(stretch)之间,所述侧接链段比CDR更高度保守且形成支撑高度可变环的骨架。重链和轻链的恒定区不涉及抗原结合,但展现出各种效应功能。基于抗体重链恒定区的氨基酸序列来对抗体分类。抗体的五个主要类别或同型是IgA、IgD、IgE、IgG和IgM,分别以α、δ、ε、γ和μ重链的存在为特征。将数种主要抗体类别划分为亚类,如IgG1(γ1重链)、IgG2(γ2重链)、IgG3(γ3重链)、IgG4(γ4重链)、IgA1(α1重链)或IgA2(α2重链)。
在某些实施例中,本文提供的抗体涵盖任何其抗原结合片段。如本文所用,术语“抗原结合片段”是指由抗体的一部分形成的包含一个或多个CDR的抗体片段,或与抗原结合但不包含完整原生抗体结构的任何其它抗体片段。抗原结合片段的实例包含但不限于双功能抗体、Fab、Fab'、F(ab')
与氨基酸序列有关的“保守取代”是指氨基酸残基被含具有类似物理化学特性的侧链的不同氨基酸残基置换。举例来说,可在具有疏水性侧链的氨基酸残基(例如Met、Ala、Val、Leu和Ile)间、具有中性亲水性侧链的氨基酸残基(例如Cys、Ser、Thr、Asn和Gln)间、具有酸性侧链的氨基酸残基(例如Asp、Glu)间、具有碱性侧链的氨基酸残基(例如His、Lys和Arg)间、或具有芳香族侧链的氨基酸残基(例如Trp、Tyr和Phe)间进行保守取代。如所属领域中已知,保守取代通常不会引起蛋白质构象结构的显著变化,并且因此可保留蛋白质的生物活性。
如本文所用,术语“有效量”或“药学上有效量”是指产生所需结果所必需的一种或多种药剂的量和/或剂量和/或剂量方案,例如足以在个体中缓和与个体正在接受疗法的疾病病状相关的一种或多种症状的量,或足以减轻个体中疾病病状的严重程度或延缓其进展的量(例如治疗有效量),足以降低个体中疾病病状的风险或延缓其发作,和/或降低其最终严重程度的量(例如预防有效量)。
如本文所用,术语“编码(encoded/encoding)”是指能够转录成mRNA和/或翻译成肽或蛋白质。术语“编码序列”或“基因”是指编码肽或蛋白质的多核苷酸序列。这两个术语可以在本公开中互换使用。在一些实施例中,编码序列是由信使RNA(mRNA)逆转录的互补DNA(cDNA)序列。在一些实施例中,编码序列是mRNA。
如本文所用,术语“同源”是指在最好比对时,与另一序列具有至少60%(例如至少65%、70%、75%、80%、85%、88%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%)序列一致性的核酸序列(或其互补股)或氨基酸序列。
“分离”的物质已通过人工方式自天然状态改变。如果“分离”的组合物或物质存在于自然界中,则所述组合物或物质已经从其原始环境改变或从其原始环境移出,或这两种情况都有。例如,天然地存在于活动物体内的多核苷酸或多肽不是“分离”的,但如果相同多核苷酸或多肽与其天然状态的共存材料充分地分离,由此以实质上纯的状态存在,那么所述多核苷酸或多肽是“分离”的。“分离”的核酸序列是指分离的核酸分子的序列。“分离”的多肽或其片段、变异体或衍生物是指不处于其天然环境中的多肽。不需要特定的纯化水平。出于本发明的目的,认为在宿主细胞(包含但不限于细菌或哺乳动物细胞)中表达的以重组方式产生的多肽和蛋白质,以及分离、分级或部分或实质上通过任何适合技术纯化的原生或重组多肽是分离的。重组肽、多肽或蛋白质是指通过重组DNA技术产生(即由通过编码肽、多肽或蛋白质的外源性重组DNA表达构建体转化的细胞、微生物或哺乳动物产生)的肽、多肽或蛋白质。在大多数细菌培养物中表达的蛋白质或肽通常将不含聚糖。前述肽、多肽、蛋白质的片段、衍生物、类似物或变异体和其任何组合也包含为肽、多肽和蛋白质。
术语“可操作地连接(operably link/operably linked)”是指两个或更多个所关注的生物序列在存在或不存在间隔子或连接子的情况下的并接,使得它们处于允许它们以预期方式起作用的关系中。当关于多肽使用时,其意指多肽序列以允许连接的产物具有预期的生物学功能的方式连接。例如,抗体可变区可以可操作地连接于恒定区,以提供具有抗原结合活性的稳定产物。所述术语还可以关于多核苷酸使用。举例来说,当编码多肽的多核苷酸可操作地连接于调节序列(例如启动子、增强子、沉默子序列等)时,其意指多核苷酸序列以允许调节多肽由多核苷酸的表达的方式连接。
术语“多肽”、“肽”和“蛋白质”在本文中可互换地用以指氨基酸残基的聚合物。所述术语还适用于其中一个或多个氨基酸残基是对应天然存在的氨基酸的人工化学模拟物的氨基酸聚合物,并且适用于天然存在的氨基酸聚合物和非天然存在的氨基酸聚合物。
如本文所用,术语“核苷酸序列”、“核酸”或“多核苷酸”包含寡核苷酸(即短多核苷酸)。其还指合成的和/或非天然存在的核酸分子(例如,包含核苷酸类似物或经修饰的主链残基或键)。所述术语还指单股或双股形式的脱氧核糖核苷酸或核糖核苷酸寡核苷酸。所述术语涵盖含有天然核苷酸类似物的核酸。所述术语还涵盖具有合成主链的核酸样结构。除非另外指示,否则特定多核苷酸序列还隐含地涵盖其保守修饰变异体(例如简并密码子取代)、等位基因、直系同源物、SNP和互补序列,以及明确指示的序列。具体来说,可以通过产生其中一个或多个选定(或所有)密码子的第三位置被混合碱基和/或脱氧肌苷残基取代的序列实现简并密码子取代(参见Batzer等人,《核酸研究(Nucleic Acid Res.)》19:5081(1991);Ohtsuka等人,《生物化学杂志(J.Biol.Chem.)》260:2605-2608(1985);和Rossolini等人,《分子细胞探针(Mol.Cell.Probes)》8:91-98(1994))。
关于氨基酸序列(或核酸序列)的术语“序列一致性百分比(%)”定义为在比对序列并在必要时引入空位以实现最大数目的相同氨基酸(或核酸)后,候选序列中与参考序列中的氨基酸(或核酸)残基相同的氨基酸(或核酸)残基的百分比。换句话说,氨基酸序列(或核酸序列)的序列一致性百分比(%)可以通过将相对于其比较的参考序列相同的氨基酸残基(或碱基)的数目除以候选序列或参考序列中氨基酸残基(或碱基)的总数(以较短者为准)来计算。氨基酸残基的保守取代可视为或可不视为相同残基。出于确定氨基酸(或核酸)序列一致性百分比的目的进行的比对可例如使用以下实现:可公开获得的工具,如BLASTN、BLASTp(可见于美国国家生物技术信息中心(U.S.National Center for BiotechnologyInformation;NCBI)的网站,另外参见AltschulS.F.等人,《分子生物学杂志》,215:403-410(1990);Stephen F.等人,《核酸研究(Nucleic Acids Res.)》,25:3389-3402(1997))、ClustalW2(可见于欧洲生物信息研究所(European Bioinformatics Institute)网站,另外参见Higgins D.G.等人,《酶学方法(Methods in Enzymology)》,266:383-402(1996);Larkin M.A.等人,《生物信息学(Bioinformatics)》(英格兰牛津(Oxford,England)),23(21):2947-8(2007))和ALIGN或Megalign(DNASTAR)软件。所属领域的技术人员可使用所述工具所提供的默认参数,或可视需要例如通过选择适合算法来自定义比对的参数。
如本文所用,术语“益生菌”是指微生物细胞制备物(如活微生物细胞),其当以有效量施用时,对个体的健康或康乐提供有益作用。按照定义,所有益生菌都具有经证实的非病原性特征。在一个实施例中,这些健康益处与改善人类或动物微生物群的平衡和/或恢复正常微生物群相关。
如本文所用,术语“协同(synergistic/synergistically)”是指提供大于两种或更多种作为单独疗法提供的药剂的治疗效果的总和的治疗效果的两种或更多种药剂。
如本文所用,术语“个体”包含人类和非人类动物。非人类动物包含所有脊椎动物,例如哺乳动物和非哺乳动物,如非人类灵长类动物、小鼠、大鼠、猫、兔、羊、狗、牛、鸡、两栖动物和爬行动物。除指出的以外,术语“患者”或“个体”在本文中可互换使用。
如本文所用,疾病、病症或病状的“治疗(treating/treatment)”包含预防或缓解疾病、病症或病状,减缓疾病、病症或病状的发作或发展速率,降低罹患疾病、病症或病状的风险,预防或延缓与疾病、病症或病状相关的症状的发展,减少或结束与疾病、病症或病状相关的症状,使疾病、病症或病状完全或部分消退,治愈疾病、病症或病状,或其某一组合。
如本文所用,术语“载体”是指一种运载体,可将遗传元件可操作地插入其中,以实现所述遗传元件的表达,从而产生由所述遗传元件编码的蛋白质、RNA或DNA,或复制所述遗传元件。载体可用于转化、转导或转染宿主细胞,以使其携带的遗传元件在宿主细胞内表达。不同载体可适合于不同宿主细胞。载体的实例包含质粒;噬菌粒;粘粒;人工染色体,如酵母人工染色体(YAC)、细菌人工染色体(BAC)或P1衍生的人工染色体(PAC);噬菌体,如λ噬菌体或M13噬菌体;和动物病毒。载体可以含有多种用于控制表达的元件,包含启动子序列、转录起始序列、增强子序列、可选元件和报告基因。另外,载体可以含有复制起点。载体还可以包含有助于其进入细胞的材料,包含但不限于病毒粒子、脂质体或蛋白质包衣。载体可以是表达载体或克隆载体。
II.
所属领域中众所周知,肿瘤在其起始和进展期间演变以避免被免疫系统破坏。虽然最近使用免疫检查点调节剂逆转这一耐药性已展现一些成功,但仅小部分癌症患者群体可以长期受益于癌症免疫疗法。
在一个方面,本公开提供一种治疗或预防有需要的个体的癌症的方法,其包括:a)向个体施用有效量的能够调节肠道免疫性的肠道免疫性调节剂;以及b)向个体施用有效量的免疫检查点调节剂。
在另一方面,本公开提供一种改善接受免疫检查点调节剂的治疗的个体中癌症的治疗反应的方法,其包括向个体施用有效量的肠道免疫性调节剂,任选地与有效量的免疫检查点调节剂一起施用。
在另一方面,本公开提供一种在个体中诱导或改善肠道免疫性的方法,其包括向个体施用有效量的肠道免疫性调节剂以及有效量的免疫检查点调节剂。
在另一方面,本公开提供一种在接受抗癌治疗并且表现出肠道免疫性降低的个体中改善癌症治疗反应的方法,其包括向个体施用有效量的肠道免疫性调节剂。
如本文所用,术语“肠道免疫性调节剂”是指可以调节和/或促进肠道免疫系统功能,和/或维持、恢复或增加哺乳动物(例如人类)中的肠粘膜屏障的物理完整性的生物物质。在一些实施例中,肠道免疫性调节剂能够调节toll样受体2(TLR2)的活性。在一些实施例中,肠道免疫性调节剂能够活化TLR2。
TLR2在检测来自细菌、真菌和寄生虫的多样范围的微生物病原体相关分子模式中起重要作用。TLR2与肠上皮细胞和免疫细胞中的细胞表面上的TLR1和TLR6形成杂二聚体。TLR2二聚体的活化触发信号级联以引发宿主中的多样先天性和后天性免疫反应。此外,TLR2活化增加肠上皮细胞间连接并且因此增强肠道屏障。
在某些实施例中,肠道免疫性调节剂包含Amuc_1100。
另外,本发明人意外地发现Amuc_1100和其功能等效物可与一种或多种免疫检查点调节剂协同作用,使得一种或多种免疫检查点调节剂的治疗作用可得到提高或增强,和/或对一种或多种免疫检查点调节剂的耐药性可得到减少、延迟或预防。实际上,本发明人证实Amuc_1100(例如纯化的重组Amuc_1100蛋白质或表达Amuc_1100的经基因工程改造的宿主细胞)和免疫检查点调节剂(例如抗PD-1抗体)的组合展现多于用对应单疗法观察到的结果的协同抗肿瘤效果(例如用于抑制肿瘤生长)。
在一个方面,本公开提供一种治疗或预防有需要的个体的癌症的方法,其包括向个体施用有效量的Amuc_1100或表达Amuc_1100的经基因工程改造的宿主细胞,以及有效量的免疫检查点调节剂。
在另一方面,本公开提供一种改善接受免疫检查点调节剂的治疗的个体中癌症的治疗反应的方法,其包括向个体施用有效量的Amuc_1100或表达Amuc_1100的经基因工程改造的宿主细胞,任选地与有效量的免疫检查点调节剂一起施用。
在另一方面,本公开提供一种在个体中诱导或改善肠道免疫性的方法,其包括向个体施用有效量的Amuc_1100或表达Amuc_1100的经基因工程改造的宿主细胞以及有效量的免疫检查点调节剂。
在另一方面,本公开提供一种在接受抗癌治疗并且表现出肠道免疫性降低的个体中改善癌症治疗反应的方法,其包括向个体施用有效量的Amuc_1100或表达Amuc_1100的经基因工程改造的宿主细胞。
A.
Amuc_1100已知为嗜粘蛋白阿克曼氏菌(“A.muciniphila”)细菌种中保存的外膜蛋白。连同两个其它成员Amuc_1099和Amuc_1101,Amuc_1100位于涉及IV型菌毛样形成的基因簇内(Ottman等人,公共科学图书馆·综合(PLoS One),2017)。Amuc_1100的示范性氨基酸序列公开于GenBank:ACD04926.1中。在本公开中,术语“Amuc_1100”和“AMUC_1100”可互换地使用。
如本文所用,术语“Amuc_1100”广泛涵盖Amuc_1100多肽,以及Amuc_1100多核苷酸,例如编码Amuc_1100多肽的DNA或RNA序列。如本文所用,术语“Amuc_1100”进一步涵盖天然存在的Amuc_1100的所有功能等效物。如本文中关于Amuc_1100所用的术语“功能等效物”意思是尽管氨基酸序列或多核苷酸序列或化学结构具有差异,但仍至少部分保留天然存在的Amuc_1100的一种或多种生物功能的任何Amuc_1100变异体。天然存在的Amuc_1100的生物功能包含但不限于a)调节和/或促进哺乳动物的肠道免疫系统功能,b)维持、恢复和/或增加哺乳动物的肠粘膜屏障的物理完整性,c)激活TLR2,d)提高哺乳动物中对癌症免疫疗法(例如免疫检查点调节剂)的免疫反应,和e)降低、延迟和/或预防哺乳动物中对一种或多种免疫检查点调节剂的耐药性。
在某些实施例中,Amuc_1100的功能等效物保持母体分子的实质生物活性。在某些实施例中,本文所述的Amuc_1100的功能等效物仍保持与天然存在的Amuc_1100实质上类似的功能,例如,Amuc_1100的功能等效物可保持天然存在的Amuc_1100的至少部分(例如,至少80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%)例如调节肠道免疫性和/或活化TLR2的活性。
如本文中关于Amuc_1100所用的术语“变异体”涵盖所有种类的不同形式的Amuc_1100,包含但不限于天然存在的Amuc_1100的片段、突变体、融合物、衍生物、模拟物或其任何组合。
如本文所用,术语“突变体”是指与亲本序列具有至少70%序列一致性的多肽或多核苷酸。突变体可以与亲本序列相差一个或多个氨基酸残基或一个或多个核苷酸。举例来说,突变体可具有亲本序列的一个或多个氨基酸残基或一个或多个核苷酸的取代(包含但不限于保守取代)、添加、缺失、插入或截短或其任何组合。在某些实施例中,突变Amuc_1100与天然存在的对应物具有至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或更多序列一致性。
如本文所用,术语“片段”是指任何长度的亲本多肽或亲本多核苷酸的部分序列。片段仍可以保持亲本序列的至少部分功能。
当关于氨基酸序列(例如肽、多肽或蛋白质)使用时,术语“融合(fusion/fused)”是指例如通过化学键结或重组方式将两个或更多个氨基酸序列组合成非天然存在的单一氨基酸序列。融合氨基酸序列可以通过两个编码多核苷酸序列的遗传重组产生,并且可以通过将含有重组多核苷酸的构建体引入到宿主细胞中的方法表达。
如本文中关于多肽或多核苷酸所用的术语“衍生物”是指化学修饰的多肽或多核苷酸,其中一个或多个明确定义数目的取代基已经共价连接于多肽的一个或多个特定氨基酸残基或多核苷酸的一个或多个特定核苷酸。对多肽的示范性化学修饰可以是例如一个或多个氨基酸的烷基化、酰化、酯化、酰胺化、磷酸化、糖基化、标记、甲基化或与一个或多个部分结合。对多核苷酸的示范性化学修饰可以是(a)末端修饰,例如5'端修饰或3'端修饰,(b)核碱基(或“碱基”)修饰,包含碱基的置换或去除,(c)糖修饰,包含在2'、3'和/或4'位置处的修饰,和(d)主链修饰,包含磷酸二酯键的修饰或置换。
如本文关于Amuc_1100所用的术语“天然存在”意思是Amuc_1100多肽或多核苷酸的序列与自然界中发现的序列或那些序列相同。天然存在的Amuc_1100可为原生或野生型的Amuc_1100序列或其片段,即使所述片段本身可能未于自然界中发现。天然存在的Amuc_1100还可包含天然存在的变异体,如在不同细菌菌株中发现的突变体或同功异构物或不同原生序列。天然存在的全长Amuc_1100多肽具有317个氨基酸残基的长度。天然存在的Amuc_1100的示范性氨基酸序列包含但不限于Amuc_1100(1-317)(SEQ ID NO:1)、Amuc_1100(31-317)(SEQ ID NO:2)和Amuc_1100(81-317)(SEQ ID NO:3)。尽管不希望受任何理论束缚,但某些天然存在的Amuc_1100片段(例如Amuc_1100(31-317)(SEQ ID NO:2)和Amuc_1100(81-317)(SEQ ID NO:3))可以比全长对应物更高的亲和力结合和活化TLR2。
在某些实施例中,Amuc_1100包含Amuc_1100多肽。在一些实施例中,Amuc_1100多肽包含或为天然存在的Amuc_1100。在一些实施例中,天然存在的Amuc_1100具有SEQ IDNO:1、SEQ ID NO:2或SEQ ID NO:3的氨基酸序列。
在一些实施例中,Amuc_1100多肽包含天然存在的Amuc_1100多肽的功能等效物。
Amuc_1100多肽的功能等效物可包含天然存在的Amuc_1100的突变体、片段、融合物、衍生物、其稳定性提高的等效物或其任何组合。Amuc_1100的功能等效物可包含天然存在的Amuc_1100的人工产生的变异体,如通过重组方法或化学合成获得的人工多肽序列。Amuc_1100的功能等效物可在活性不受影响的前提下包含非天然存在的氨基酸残基。适合的非天然氨基酸包含例如β-氟丙氨酸、1-甲基组氨酸、γ-亚甲基谷氨酸、α-甲基亮氨酸、4,5-脱氢赖氨酸、羟基脯氨酸、3-氟苯丙氨酸、3-氨基酪氨酸、4-甲基色氨酸等。
在某些实施例中,Amuc_1100多肽在选自由以下组成的群组的位置处包含一个或多个(例如2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25个或更多个)突变:R36、S37、L39、D40、K41、K42、I43、K48、E49、K51、S52、R62、S63、K70、E71、L72、N73、R74、Y75、A76、K77、A78、Y86、K87、P88、F89、L90、A91、F103、Q104、K108、T109、F110、R111、D112、K119、K120、K121、N122、L124、I125、W131、L132、G133、F134、Q135、Y137、S138、L150、G151、F152、E153、L154、K155、A156、L160、V161、K163、L164、A165、L169、S170、K171、F172、I173、K174、V175、Y176、R177、W200、T201、L205、E206、F209、Q210、R213、E214、L217、K218、A219、M220、N221、Y229、L230、R237、I238、R242、M243、M244、P245、K255、P256、L268、T269、K287、P288、Y289、M290、K292、E293、F296、V297、F306、N307、K310和A311,其中编号相对于SEQ ID NO:1。不希望受任何理论所束缚,相信突变可用于提高Amuc_1100多肽的稳定性,使得与天然存在的对应物相比,其较不易于被酶消化。
在某些实施例中,Amuc_1100多肽在选自由以下组成的群组的位置处包含一个或多个(例如2、3或4个)突变:S37、V175、Y289和F296,其中编号相对于SEQ ID NO:1。在某些实施例中,Amuc_1100多肽包含Y289A突变,其中编号相对于SEQ ID NO:1。在某些实施例中,Amuc_1100多肽包含SEQ ID NO:4或SEQ ID NO:5的氨基酸序列。
在某些实施例中,Amuc_1100多肽进一步在N端或C端处包含标签或氨基酸延伸。标签可用于Amuc_1100多肽的纯化。氨基酸延伸可用于提高稳定性或减少清除率。可以使用任何适合的标签或延伸,例如His-标签(例如6×His标签)、蛋白质A、lacZ(β-gal)、麦芽糖结合蛋白质(MBP)、钙调蛋白结合肽(CBP)、内含肽-甲壳质结合结构域(内含肽-CBD)标签、基于链霉亲和素/生物素的标签、串联亲和纯化(TAP)标签、表位标签、报告基因标签等。
在某些实施例中,Amuc_1100多肽进一步包含将标签与Amuc_1100多肽的其余部分分隔开的酶消化位点。酶消化位点可用于在需要时去除标签。适合酶消化位点的实例包含肠激酶识别位点(例如DDDDK)、因子Xa识别位点、吉能酶(genenase)I识别位点、弗林蛋白酶(furin)识别位点等。
如下文表1中所示,Amuc_1100多肽以及包含标签和/或酶消化位点的Amuc_1100多肽的示范性氨基酸序列提供于SEQ ID NO:1-13中。
表1.示范性Amuc_1100的氨基酸序列
/>
/>
/>
/>
本领域技术人员能够理解,表1所列的Amuc_110的氨基酸序列N端的甲硫氨酸(M)由起始密码子编码,但在蛋白成熟过程中会被酶切掉。在某些实施方式中,上述Amuc_1100的氨基酸序列可以不包括N端的甲硫氨酸(M)。但是,编码甲硫氨酸(M)的密码子在翻译过程中是必要的,因此,在关于基因工程菌的实施方式中,Amuc_1100的编码序列包括编码甲硫氨酸(M)的密码子。
本文所提供的Amuc_1100多肽可通过所属领域中的各种众所周知方法产生,例如从天然材料分离和纯化、重组表达、化学合成等。所产生的Amuc_1100多肽可通过所属领域中众所周知的纯化方法进一步纯化。
Amuc_1100多核苷酸可包含编码本文所提供的Amuc_1100多肽中的任一种多肽的多核苷酸。
B.
在某些实施例中,本文所提供的方法包含向个体施用有效量的表达本文所提供的Amuc_1100的经基因工程改造的宿主细胞。本公开的另一方面为提供一种包含外源性表达盒的经基因工程改造的宿主细胞,所述外源性表达盒包含编码可操作地连接于信号肽的Amuc_1100的核苷酸序列,其中经基因工程改造的宿主细胞从1×10
在某些实施例中,经基因工程改造的宿主细胞包含外源性表达盒,所述外源性表达盒包含编码可操作地连接于信号肽的Amuc_1100的核苷酸序列,其中经基因工程改造的宿主细胞从1×10
经基因工程改造的宿主细胞(例如经基因工程改造的细菌)为在药物开发探索中的新型治疗剂。在一些实施例中,将一种或多种疾病治愈基因整合到细菌基因组中,且活经工程改造的细菌充当递送系统,以在调节病原性信号的身体部位(例如小肠或结肠)处产生一种或多种基因药物。其被称为细菌载体基因疗法或重组活生物治疗产品(LBP)。其为开发用于人类疾病的新颖类别的药剂的人类微生物群和合成生物学两个领域中的新方法。本发明人意外地发现,本发明中使用的经基因工程改造的宿主细胞实现与自然界中嗜粘蛋白阿克曼氏菌产生的Amuc_1100的量相比,显著增强的Amuc_1100的表达和分泌。在某些实施方式中,本发明中使用的经基因工程改造的宿主细胞产生的Amuc_1100的表达量与自然界中嗜粘蛋白阿克曼氏菌产生的Amuc_1100相比高至少10%、15%、20%、30%、40%、50%、60%、70%、80%、90%、100%。此外,一些已有的研究数据表明,阿克曼氏菌对宿主机体生理条件具有正反两面作用。尽管有研究支持使用活的或者灭活的阿克曼氏菌能提高癌症对PD-1抗体的应答率,Amuc_1100蛋白或者表达Amuc_1100的基因工程菌的增强作用应该更加安全和更加有特异的针对性。
尽管术语“基因工程改造”常常用于描述大量用于生物体的遗传信息的人工修饰的技术,但在整个说明书和权利要求书中,所述术语仅关于从供体微生物体和适合的载体形成重组DNA,基于所需遗传信息选择,并且将所选择的DNA引入到适合的微生物体(宿主微生物体)中,由此所需(外来)遗传信息成为宿主的遗传互补序列的一部分的体外技术而采用。如在整个说明书和权利要求书中所用,术语“经基因工程改造的宿主细胞”意指通过此技术制备的宿主细胞。
如本文所用,术语“宿主细胞”是指其中以允许编码Amuc_1100的多核苷酸表达的方式引入外源性表达盒(包含含有表达盒的载体)的细胞。宿主细胞还包含本发明宿主细胞或其衍生物的任何后代。应理解,所有后代可能与母细胞不同,因为可能存在在复制期间发生的突变。然而,当使用术语“宿主细胞”时,包含所述后代。可用于本发明的宿主细胞包含细菌细胞、真菌细胞(例如酵母细胞)、植物细胞或动物细胞。在某一实施例中,宿主细胞为低级真核细胞,如酵母细胞,或宿主细胞可为原核细胞,如细菌细胞。将表达盒引入到宿主细胞中可以通过磷酸钙转染、DEAE-葡聚糖介导的转染或电穿孔实现(Davis,L.、Dibner,M.、Battey,I.,《基本分子生物学方法(Basic Methods in Molecular Biology)》(1986))。
在一些实施例中,本公开的宿主细胞包含益生微生物体。如本文所用,术语“微生物体”包含细菌、古细菌、酵母、藻类和丝状真菌。在一些实施例中,本发明中使用的微生物体是非病原性微生物体。“非病原性微生物体”是指不能在宿主中引起疾病或有害反应的微生物体。在一些实施例中,非病原性微生物体不含脂多糖(LPS)。在一些实施例中,非病原性微生物体为共生细菌。在一些实施例中,非病原性微生物体为减毒病原性细菌。
在一些实施例中,经基因工程改造的宿主细胞(例如细菌)为革兰氏阴性细菌(Gram-negative bacteria)。经基因工程改造的宿主细胞(例如细菌)为革兰氏阳性细菌(Gram-positive bacteria)。示范性细菌包含但不限于芽孢杆菌(Bacillus)(例如凝结芽孢杆菌(Bacillus coagulans)、枯草芽孢杆菌(Bacillus subtilis))、拟杆菌(Bacteroides)(例如脆弱拟杆菌(Bacteroides fragilis)、枯草拟杆菌(Bacteroidessubtilis)、多形拟杆菌(Bacteroides thetaiotaomicron))、双歧杆菌(Bifidobacterium)(例如青春双歧杆菌(Bifidobacterium adolescentis)、两歧双歧杆菌(Bifidobacteriumbifidum)、短双歧杆菌(Bifidobacterium breve)UCC2003、婴儿双歧杆菌(Bifidobacterium infantis)、乳酸双歧杆菌(Bifidobacterium lactis)、长双歧杆菌(Bifidobacterium longum))、短杆菌(Brevibacteria)、柄杆菌(Caulobacter)、梭菌(Clostridium)(例如丙酮丁醇梭菌(Clostridium acetobutylicum)、丁酸梭菌(Clostridium butyricum)、丁酸梭菌M-55、宫入丁酸梭菌(Clostridium butyricummiyairi)、匙形梭菌(Clostridium cochlearum)、费尔西纳梭菌(Clostridiumfelsineum)、溶组织梭菌(Clostridium histolyticum)、多酶梭菌(Clostridiummultifermentans)、诺维氏梭菌(Clostridium novyi-Ν′Y)、类腐败梭菌(Clostridiumparaputrificum)、巴氏梭菌(Clostridium pasteureanum)、蚀果胶梭菌(Clostridiumpectinovorum)、产气荚膜梭菌(Clostridium perfringens)、红色梭菌(Clostridiumroseum)、生孢梭菌(Clostridium sporogenes)、第三梭菌(Clostridium tertium)、破伤风梭菌(Clostridium tetani)、酪丁酸梭菌(Clostridium tyrobutyricum))、短小棒状杆茵(Corynebacterium parvum)、肠球菌(Enterococcus)、大肠杆菌(Escherichia coli)(例如大肠杆菌MG1655、大肠杆菌Nissle 1917)、乳杆菌(Lactobacillus)、乳球菌(Lactococcus)、李斯特菌(Listeria)、分支杆菌(Mycobacterium)(例如牛分支杆菌(Mycobacterium bovis))、酵母(Saccharomyces)、沙门氏菌(Salmonella)(猪霍乱沙门氏菌(Salmonella choleraesuis)、鼠伤寒沙门氏菌(Salmonella typhimurium))、葡萄球菌(Staphylococcus)、链球菌(Streptococcus)、弧菌(Vibrio)(例如霍乱弧菌(Vibriocholera))。在某些实施例中,经基因工程改造的细菌选自由以下组成的群组:屎肠球菌(Enterococcus faecium)、嗜酸乳杆菌(Lactobacillus acidophilus)、保加利亚乳杆菌(Lactobacillus bulgaricus)、干酪乳杆菌(Lactobacillus casei)、约氏乳杆菌(Lactobacillus johnsonii)、副干酪乳杆菌(Lactobacillus paracasei)、胚芽乳杆菌(Lactobacillus plantarum)、洛德乳杆菌(Lactobacillus reuteri)、鼠李糖乳杆菌(Lactobacillus rhamnosus)、雷特氏乳球菌(Lactococcus lactis)和布拉迪酵母(Saccharomyces boulardii)。在一些实施例中,经基因工程改造的细菌为大肠杆菌。
“益生微生物体”是指在摄食时可提供健康益处的微生物体。益生微生物体(也称为“益生菌”)通常作为具有特别添加的活性活培养物(如酸奶、大豆酸奶中)的发酵食品的一部分或作为膳食增补剂被摄食。一般来说,益生菌帮助肠道微生物群保持(或重新找到)其平衡、完整性和多样性。益生菌的作用可以是菌株依赖性的。因此在本发明的上下文中,“益生组合物”不限于食品或食品增补剂,但其通常指示包含有益于患者的微生物体的任何细菌组合物。所述益生组合物可因此为药剂或药物。
在一些实施例中,益生微生物体为益生细菌或益生酵母。在一些实施例中,益生细菌选自由以下组成的群组:拟杆菌、双歧杆菌(例如两歧双歧杆菌)、梭菌、埃希氏菌、乳杆菌(例如嗜酸乳杆菌、保加利亚乳杆菌、副干酪乳杆菌、胚芽乳杆菌)和乳球菌,任选地,所述益生细菌属于埃希氏菌属,并且任选地,所述益生细菌属于物种大肠杆菌的菌株Nissle 1917(EcN)。在一些实施例中,益生酵母选自由以下组成的群组:酿酒酵母、产朊假丝酵母、乳酸克鲁维酵母和卡氏酵母。在一些实施例中,益生微生物体为大肠杆菌的菌株Nissle 1917(EcN)。
在一些实施例中,外源性表达盒整合于经基因工程改造的宿主细胞的质粒中。“质粒”意指与宿主细胞的一种或多种染色体不同并且与一种或多种染色体分开复制的实质上小于染色体的环状或线性DNA分子。“质粒”可以约每细胞一个拷贝或每细胞超过一个拷贝形式存在。一般来说,宿主细胞内质粒的维持需要抗生素选择,但也可以使用营养缺陷体互补。
在一些实施例中,外源性表达盒整合于经基因工程改造的宿主细胞的基因组中。在一些实施例中,外源性表达盒通过CRISPR-Cas基因组编辑系统整合于经基因工程改造的宿主细胞的基因组中。测试所选择的基因组位点并且将其用于整合Amuc_1100基因盒。将对基因盒和细菌基因组进行一系列修饰以确保异源基因将以一定量表达并且将对底盘(chassis)宿主细胞的生物化学和生理活性没有重大负面影响。
i.表达盒
本文所提供的表达Amuc_1100的经基因工程改造的宿主细胞包含外源性表达盒,所述外源性表达盒包含编码任选地可操作地连接于信号肽的本文所提供的Amuc_1100多肽的多核苷酸序列,和一个或多个调节元件。本公开的另一方面为提供所述外源性表达盒,其包含编码本文所提供的Amuc_1100多肽的多核苷酸序列。
如本文所用,术语“表达盒”是指能够引导特定核苷酸序列在适当的宿主细胞中表达的DNA序列,包含可操作地连接于所关注的核苷酸序列的启动子,所述所关注的核苷酸序列可操作地连接于终止信号。其还通常包含核苷酸序列恰当翻译所需的序列。编码区通常编码所关注蛋白质,但也可以在反义方向的意义上编码所关注的功能RNA,例如反义RNA或非翻译RNA。包含所关注的核苷酸序列的表达盒可以是嵌合的,这意味着其组件中的至少一个相对于其其它组件中的至少一个是异源的。
表达盒适合于在本文所提供的宿主细胞中表达Amuc_1100多肽。表达盒可以裸核酸构建体的形式使用。或者,表达盒可以作为核酸载体(例如,如上文所描述的表达载体)的一部分引入。适合于宿主细胞(如细菌细胞、真菌细胞(例如酵母细胞)、植物细胞或动物细胞)的载体可以包含质粒和病毒载体。载体可以包含侧接表达盒的序列,所述序列包含与真核基因组序列,例如哺乳动物基因组序列或病毒基因组序列同源的序列。这将允许通过同源重组将表达盒引入真核细胞或病毒的基因组中。
如本文所用,术语“重组”是指在体外合成或以其它方式操纵的多核苷酸(例如,“重组多核苷酸”或“重组表达盒”),指使用重组多核苷酸或重组表达盒在细胞或其它生物系统中产生产物的方法,或指由重组多核苷酸编码的多肽(“重组蛋白”)。重组多核苷酸涵盖接合到表达盒或载体中的来自不同来源的核酸分子,以表达例如融合蛋白;或通过诱导性或组成性表达多肽产生的蛋白质(例如,本发明的表达盒或载体可操作地连接于异源多核苷酸,例如Amuc_1100编码序列)。重组表达盒涵盖可操作地连接于一个或多个调节元件的重组多核苷酸。
在一些实施例中,表达盒包含一个或多个调节元件,所述调节元件包含一个或多个选自由以下组成的群组的元件:启动子、核糖体结合位点(RBS)、顺反子、终止子和其任何组合。
如本文所用,术语“启动子”是指含有RNA聚合酶的结合位点并且起始DNA转录的通常在编码区上游的非翻译DNA序列。启动子区还可包含充当基因表达的调控因子的其它元件。在一些实施例中,启动子适合于起始经基因工程改造的宿主细胞中的编码Amuc_1100多肽的多核苷酸。在一些实施例中,启动子为组成型启动子或诱导型启动子。
术语“组成型启动子”是指能够促进在其控制下和/或与其可操作地连接的编码序列或基因的连续转录的启动子。组成型启动子和变异体是所属领域中众所周知的,并且包含但不限于BBa_J23119、BBa_J23101、BBa_J23102、BBa_J23103、BBa_J23109、BBa_J23110、BBa_J23114、BBa_J23117、USP45_启动子、Amuc_1102_启动子、OmpA_启动子、BBa_J23100、BBa_J23104、BBa_J23105、BBa_I14018、BBa_J45992、BBa_J23118、BBa_J23116、BBa_J23115、BBa_J23113、BBa_J23112、BBa_J23111、BBa_J23108、BBa_J23107、BBa_J23106、BBa_I14033、BBa_K256002、BBa_K1330002、BBa_J44002、BBa_J23150、BBa_I14034、Oxb19、oxb20、BBa_K088007、Ptet、Ptrc、PlacUV5、BBa_K292001、BBa_K292000、BBa_K137031和BBa_K137029。示范性组成型启动子的核苷酸序列包含选自由以下组成的群组的核苷酸序列:如表2中所示的SEQ ID NO:14-54,和其具有至少80%(例如至少85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%)序列一致性的同源序列。在一些实施例中,组成型启动子包含SEQ ID NO:15的核苷酸序列。在一些实施例中,所述启动子在体外,例如在培养、扩增和/或制造条件下具有活性。在一些实施例中,所述启动子在体内,例如在体内环境,例如肠道和/或肿瘤微环境中存在的条件下具有活性。
如本文所用,术语“诱导型启动子”是指可以通过外部刺激,如化学、光、激素、应激或病原体,在一种或多种细胞类型中启用的受调控的启动子。诱导型启动子和变异体为所属领域中众所周知的,并且包含但不限于PLteto1、galP1、PLlacO1、Pfnrs。在一些实施例中,示范性诱导型启动子的核苷酸序列包含选自由以下组成的群组的核苷酸序列:如表2中所示的SEQ ID NO:55-58,和其具有至少80%(例如至少85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%)序列一致性的同源序列。在一些实施例中,诱导型启动子包含SEQ ID NO:58的核苷酸序列。
在一些实施例中,启动子为内源启动子或外源启动子。如本文所用,“外源启动子”是指与编码区可操作组合的启动子,其中所述启动子不是生物体基因组中与所述编码区天然相关的启动子。基因组中与编码区天然相关或相连的启动子被称为所述编码区的“内源启动子”。
如本文所用,术语“核糖体结合位点”(“RBS”)是指起始蛋白质翻译时核糖体结合于mRNA上的序列。RBS为大约35个核苷酸长,并且含有三个离散结构域:(1)Shine-Dalgarno(SD)序列,(2)间隔区,和(3)编码序列(CDS)的前五到六个密码子。RBS和变异体在所属领域中众所周知,并且包含但不限于USP45、合成型(Synthesized)、Amuc_1102、OmpA。示范性RBS的核苷酸序列包含选自由以下组成的群组的核苷酸序列:如表2中所示的SEQ ID NO:70-73,和其具有至少80%(例如至少85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%)序列一致性的同源序列。
如本文所用,术语“顺反子”是指经转录且编码多肽的核酸序列的区段。顺反子和变异体在所属领域中众所周知,并且包含但不限于GFP、BCD2、荧光素酶、MBP。示范性顺反子的核苷酸序列包含选自由以下组成的群组的核苷酸序列:如表2中所示的SEQ ID NO:66-69,和其具有至少80%(例如至少85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%)序列一致性的同源序列。
如本文所用,术语“终止子”是指可与酶结合的核苷酸,其防止核苷酸进一步合成到所生成的多核苷酸链中,并且由此中断聚合酶介导的延伸。在一些实施例中,本公开中使用的终止子为T7终止子。在一些实施例中,终止子包含选自由以下组成的群组的核苷酸序列:如表2中所示的SEQ ID NO:74-75,和其具有至少80%(例如至少85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%)序列一致性的同源序列。
在某些实施例中,在本文所提供的外源性表达盒中,编码Amuc_1100的多核苷酸序列可操作地连接于信号肽。
如本文所用,术语“信号肽”或“信号序列”是指可以用于将异源多肽分泌到经培养细菌的周质或培养基中或将Amuc_1100多肽分泌到周质中的肽。异源多肽的信号可以与细菌同源,或其可以是异源的,包含对于细菌中所产生的多肽原生的信号。对于Amuc_1100,信号序列通常为细菌细胞内源性的,但其不必是内源性的,只要其对于其目的有效即可。分泌肽的非限制性实例包含OppA(ECOLIN_07295)、OmpA、OmpF、cvaC、TorA、fdnG、dmsA、PelB、HlyA、粘附素(ECOLIN_19880)、DsbA(ECOLIN_21525)、Gltl(ECOLIN_03430)、GspD(ECOLIN_16495)、HdeB(ECOLIN_19410)、MalE(ECOLIN_22540)、PhoA(ECOLIN_02255)、PpiA(ECOLIN_18620)、TolB、tort、mglB和lamB。
在某些实施例中,本文提供的用于Amuc_1100分泌的信号肽包含Usp45、OmpA、DsbA、pelB、celCD、sat或Amuc_1100的内源信号肽。示范性信号肽的核苷酸序列包含选自由以下组成的群组的核苷酸序列:如表2中所示的SEQ ID NO:59-65,和其具有至少80%(例如至少85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%)序列一致性的同源序列。在一些实施例中,信号肽包含选自由以下组成的群组的氨基酸序列:如表3中所示的SEQID NO:76-82,和其具有至少80%(例如至少85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%)序列一致性的同源序列。在一些实施例中,信号肽可操作地连接于Amuc_1100的N端。
在一些实施例中,表达盒包含编码可操作地连接于信号肽和自转运蛋白结构域的Amuc_1100的核苷酸序列,其中信号肽和自转运蛋白结构域可操作地连接于Amuc_1100的相对端,例如信号肽可操作地连接于Amunc_1100的N端,并且自转运蛋白结构域可操作地连接于Amunc_1100的C端。所述构建体可用于V型自分泌介导的分泌,其中在通过原生分泌系统(如Sec系统)将前体蛋白质从细胞质易位到周质隔室中时,去除N端信号肽,并且另外,一旦自分泌物易位跨越外膜,可以通过自催化或蛋白酶催化的例如OmpT裂解去除C端自转运蛋白结构域,从而将成熟蛋白质(例如Amuc_1100多肽)释放到细胞外环境中。
表2.示范性启动子、RBS、顺反子、信号肽和终止子的核苷酸序列
/>
/>
/>
/>
表3.示范性信号肽的氨基酸序列
/>
ii.宿主细胞中的分泌系统和/或输出路径
在一些实施例中,信号肽可以通过存在于经基因工程改造的宿主细胞(例如经基因工程改造的细菌)中的输出路径和/或分泌系统加工。信号肽通常在输出的前体蛋白质的N端,并且可以将前体蛋白质引导到细菌细胞质膜中的输出路径。分泌系统能够在从经工程改造的宿主细胞(如细菌)分泌成熟蛋白质之前从前体蛋白质去除信号肽。
如本文所用,术语“分泌系统”在本文中与“输出系统”和“输出路径”可互换地使用,是指能够从微生物(例如细菌细胞质)分泌或输出所表达的多肽产物(例如Amuc_1100多肽)的原生或非原生分泌机制。如大肠杆菌的革兰氏阴性细菌中从外向内,存在外膜(OM)、肽聚糖细胞壁、周质、内膜(IM)和细胞质隔室。OM为极有效且选择性的渗透性屏障。OM为由内叶处的磷脂和外叶处的糖脂以及脂蛋白和β桶形蛋白组成的脂质双层。OM通过称为Lpp的脂蛋白固定到底层肽聚糖。周质密集地填充有蛋白质,且其比细胞质更粘稠。IM为磷脂双层并且主要位置容纳在能量产生、脂质生物合成、蛋白质分泌和转运中起作用的膜蛋白。
在细菌中,存在两种常见蛋白质输出路径,包含普遍存在的一般分泌(或Sec)路径和双精氨酸易位(或Tat)路径。通常,Sec路径处理未折叠状态的较高分子量前体蛋白质,其中信号肽将底物蛋白质靶向到膜结合的Sec移位酶。前体目标蛋白质递送到移位酶并且通过SecA、SecD和SecF穿过SecYEG孔。伴侣蛋白SecB、GroEL-GroES、DnaK-DnaJ-GrpE辅助目标蛋白运输。Tat路径通常运输完全折叠或甚至寡聚状态的蛋白质,且革兰氏阴性和阳性细菌中都由组分TatA、TatB和TatC组成。在膜易位之后,通过信号肽酶去除信号肽并且分泌成熟蛋白质。
对于革兰氏阴性细菌,其具有专用单步分泌系统,并且分泌系统的非限制性实例包含Sat分泌系统、I型分泌系统(T1SS)、II型分泌系统(T2SS)、III型分泌系统(T3SS)、IV型分泌系统(T4SS)、V型分泌系统(T5SS)、VI型分泌系统(T6SS)和多药物流出泵的耐药性-结瘤-分裂(resistance-nodulation-division,RND)家族、各种单膜分泌系统。
在一些实施例中,分泌系统对于经基因工程改造的宿主细胞是原生或非原生的系统。对于宿主细胞“原生”意思是分泌系统通常存在于宿主细胞中,而对于宿主细胞“非原生”意思是分泌系统不通常存在于宿主细胞中,例如额外分泌系统,如来自细菌或病毒的不同物种、菌株或亚菌株的分泌系统,或与来自同一亚型的宿主细胞的未经修饰的分泌系统相比经修饰和/或突变的分泌系统。
在一些实施例中,分泌系统经工程改造和/或优化以使得经基因工程改造的宿主细胞中的至少一种外膜蛋白编码基因被删除、失活或抑制。在一些实施例中,所述外膜蛋白选自由以下组成的群组:OmpC、OmpA、OmpF、OmpT、pldA、pagP、tolA、Pal、To1B、degS、mrcA和lpp。
为了制造能够分泌所需治疗性蛋白质或肽的革兰氏阴性细菌(例如EcN),需要基因工程改造细菌宿主并且使其具有“漏”或去稳定化的外膜,但不影响宿主细菌化学物理活性。基因,例如lpp、ompC、ompA、ompF、ompT、pldA、pagP、tolA、to1B、pal、degS、degP和nlpI可以被删除或诱发突变以产生“漏”的外膜。Lpp为细菌细胞中最丰富的多肽,且充当细菌细胞壁与肽聚糖的主要“订书针”。lpp的缺失对细菌生长具有极小影响,但增加Amuc_1100的分泌,例如增加至少一倍。诱导型启动子可以用于替换所选择的一种或多种基因的内源启动子以使对细胞存活率的负面影响最小化。
基因或编码区的“删除/缺失”或“失活”或“抑制”意指使由所述基因或编码区编码的酶或蛋白质不产生,或以非活性形式产生于宿主细胞中,或以低于在相同或类似生长条件下在宿主细胞的野生型形式中发现的含量的含量在宿主细胞中产生。这可以通过例如以下方法中的一种或多种实现:(1)同源重组,(2)基于RNA干扰的技术,(3)ZFN和TALEN,(4)CRISPR/Cas系统。
在一些实施例中,分泌系统经工程改造以使得至少一种伴侣蛋白编码基因被扩增、过表达或活化。
伴侣蛋白涉及许多重要的生物过程,如寡聚蛋白复合物的蛋白质折叠和聚集,使蛋白质前体维持在未折叠状态以促进蛋白质跨膜运输,并使变性的蛋白质能够解聚和修复。其主要是辅助其它肽维持正常构象,以形成正确的寡聚结构,从而发挥正常的生理功能。各种伴侣蛋白是所属领域中众所周知的。在一些实施例中,伴侣蛋白选自由以下组成的群组:dsbA、dsbC、dnaK、dnaJ、grpE、groES、groEL、tig、fkpA、surA、skp、PpiD和DegP。在一些实施例中,伴侣蛋白选自由以下组成的群组:来自70kDa热休克蛋白(Hsp70)的胞质SSA亚家族的Ssa1p、Ssa2p、Ssa3p和Ssa4p,BiP、Kar2、Lhs1、Sil1、Sec63、蛋白二硫键异构酶Pdi1p。
基因或编码区的“过表达(overexpressed/overexpression)”意指使由所述基因或编码区编码的酶或蛋白质以高于在相同或类似生长条件下在宿主细胞的野生型形式中发现的含量的含量在宿主细胞中产生。这可以通过例如以下方法中的一种或多种实现:(1)安设更强的启动子,(2)安设更强的核糖体结合位点,如5'-AGGAGG的DNA序列,位于翻译起始密码子上游约四到十个碱基处,(3)安设终止子或更强的终止子,(4)改进在编码区中的一个或多个位点处的密码子的选择,(5)提高mRNA稳定性,和(6)通过在染色体中引入多个拷贝或将盒置于多拷贝质粒上来增加基因的拷贝数。由过表达的基因产生的酶或蛋白质称为“过产生”的。“过表达”的基因或“过产生”的蛋白质可以对于宿主细胞是原生的,或其可以通过基因工程改造方法从不同生物体移植到宿主细胞中,在后一种情况下,酶或蛋白质和编码酶或蛋白质的基因或编码区称为“外来”或“异源”的。外来或异源基因和蛋白质为过表达和过产生的,因为其不存在于未工程改造的宿主细胞中。
对于酵母,分泌系统主要包含初生蛋白质易位、内质网(ER)中的蛋白质折叠、糖基化、蛋白质分选和运输。翻译期间信号肽一从核糖体出现,新合成的膜或分泌蛋白质就由信号识别颗粒(SRP)识别,随后靶向到穿过Sec61或Ssh1易位子孔的翻译后分泌。已实施各种方法以改进酵母中的异源蛋白质产生:(1)工程改造信号肽并且提高启动子强度和质粒拷贝数;(2)工程改造宿主菌株的翻译后路径,例如过表达ER折叠伴侣蛋白和囊泡运输组件,和减少细胞间和细胞外蛋白水解。异源蛋白质的分泌的示范性酵母分泌信号肽包含酿酒酵母α-交配因子(α-Mating Factor,α-MF)肽原前体(pre pro-peptide)、来自马克斯克鲁维酵母(Kluyveromyces marxianus)的菊粉酶的信号肽、来自普通菜豆凝集素(Phaseolusvulgaris agglutinin)的PHAE的信号肽、病毒毒素原前体(prepro toxin)的信号肽、米根霉(Rhizopus oryzae)淀粉酶或来自真菌里氏木霉(Trichoderma reesei)的疏水蛋白信号序列的信号肽。
iii.基因组整合位点
在一些实施例中,外源性表达盒整合于经基因工程改造的宿主细胞的基因组中。在一些实施例中,外源性表达盒通过CRISPR-Cas基因组编辑系统整合于经基因工程改造的宿主细胞的基因组中。本文提供的任何适合的宿主细胞都可以经工程改造,以使得外源性表达盒整合到基因组中。
在一些实施例中,经基因工程改造的宿主细胞为大肠杆菌的菌株Nissle1917(EcN),并且外源性表达盒整合到EcN基因组中的位点中。EcN基因组中的适合整合位点可为表4中列出的任何位点(图14中展示,agaI/rsmI作为实例展示)。在一些实施例中,EcN基因组中的适合整合位点为整合位点agaI/rsmI。
用于本发明中的Amuc_1100整合的EcN基因组的基因组位点包含表4中列出的任何位点。不希望受任何理论束缚,但认为表4中列出的EcN的基因组位点对于插入Amuc_1100的表达盒,通过以下特征中的至少一个是有利的:(1)所述位点的工程改造所影响的一种或多种细菌基因不是EcN生长所必需的,并且不改变宿主细菌的生物化学和生理活性,(2)所述位点可以容易地被编辑,和(3)所述位点中的Amuc_1100基因盒可以被转录。整合位点地图展示于图15中。用于编辑EcN中的对应基因组位点的sgRNA序列展示于下表4中。
表4.用于编辑EcN中的对应基因组位点的sgRNA序列.
/>
iv.营养缺陷体
在一些实施例中,本公开的宿主细胞进一步包含营养缺陷相关基因中的至少一个失活或缺失。
为了产生环境友好型细菌,可以通过诱发突变使细菌细胞生存所必需的一些必需基因被删除或失活,使经工程改造的细菌成为营养缺陷体。如本文所用,术语“营养缺陷体”是指宿主细胞(例如微生物体菌株)的生长需要特定代谢物的外部来源,所述代谢物由于后天基因缺陷而不能合成。如本文所用,术语“营养缺陷相关基因”是指宿主细胞(例如微生物体,如细菌)生存所需的基因。营养缺陷相关基因可以是微生物体产生生存或生长所必需的营养素所必需的,或者可以是检测环境中调节转录因子活性的信号所必需的,其中不存在所述信号将引起细胞死亡。
在一些实施例中,营养缺陷型修饰意图使微生物体在缺乏外源添加的生存或生长所必需的营养素时死亡,因为所述微生物体缺乏产生必需营养素所必需的一种或多种基因。在一些实施例中,本文所述的经基因工程改造的细菌中的任一种还包含细胞生存和/或生长所需基因的缺失或突变。
细菌中的各种营养缺陷相关基因是所属领域中众所周知的。示范性营养缺陷相关基因包含但不限于:thyA、cysE、glnA、ilvD、leuB、lysA、serA、metA、glyA、hisB、ilvA、pheA、proA、thrC、trpC、tyrA、uraA、dapF、flhD、metB、metC、proAB、yhbV、yagG、hemB、secD、secF、ribD、ribE、thiL、dxs、ispA、dnaX、adk、hemH、IpxH、cysS、fold、rplT、infC、thrS、nadE、gapA、yeaZ、aspS、argS、pgsA、yeflA、metG、folE、yejM、gyrA、nrdA、nrdB、folC、accD、fabB、gltX、ligA、zipA、dapE、dapA、der、hisS、ispG、suhB、tadA、acpS、era、rnc、fisB、eno、pyrG、chpR、Igt、ft>aA、pgk、yqgD、metK、yqgF、plsC、ygiT、pare、ribB、cca、ygjD、tdcF、yraL、yihA、ftsN、murl、murB、birA、secE、nusG、rplJ、rplL、rpoB、rpoC、ubiA、plsB、lexA、dnaB、ssb、alsK、groS、psd、orn、yjeE、rpsR、chpS、ppa、valS、yjgP、yjgQ、dnaC、ribF、IspA、ispH、dapB、folA、imp、yabQ、flsL、flsl、murE、murF、mraY、murD、ftsW、murG、murC、ftsQ、ftsA、ftsZ、IpxC、secM、secA、can、folK、hemL、yadR、dapD、map、rpsB、in/B、nusA、ftsH、obgE、rpmA、rplU、ispB、murA、yrbB、yrbK、yhbN、rpsl、rplM、degS、mreD、mreC、mreB、accB、accC、yrdC、def、fint、rplQ、rpoA、rpsD、rpsK、rpsM、entD、mrdB、mrdA、nadD、hlepB、rpoE、pssA、yfiO、rplS、trmD、rpsP、ffh、grpE、yfjB、csrA、ispF、ispD、rplW、rplD、rplC、rpsJ、fusA、rpsG、rpsL、trpS、yr/F、asd、rpoH、ftsX、ftsE、ftsY、frr、dxr、ispU、rfaK、kdtA、coaD、rpmB、djp、dut、gmk、spot、gyrB、dnaN、dnaA、rpmH、rnpA、yidC、tnaB、glmS、glmU、wzyE、hemD、hemC、yigP、ubiB、ubiD、hemG、secY、rplO、rpmD、rpsE、rplR、rplF、rpsH、rpsN、rplE、rplX、rplN、rpsQ、rpmC、rplP、rpsC、rplV、rpsS、rplB、cdsA、yaeL、yaeT、lpxD、fabZ、IpxA、IpxB、dnaE、accA、tilS、proS、yafF、tsf、pyrH、olA、rlpB、leuS、Int、glnS、fldA、cydA、in/A、cydC、ftsK、lolA、serS、rpsA、msbA、IpxK、kdsB、mukF、mukE、mukB、asnS、fabA、mviN、rne、yceQ、fabD、fabG、acpP、tmk、holB、lolC、lolD、lolE、purB、ymflC、minE、mind、pth、rsA、ispE、lolB、hemA、prfA、prmC、kdsA、topA、ribA、fabi、racR、dicA、yd B、tyrS、ribC、ydiL、pheT、pheS、yhhQ、bcsB、glyQ、yibJ和gpsA。
在一种修饰中,必需基因thyA被删除或被另一基因替代,使得经基因工程改造的细菌依赖于外源胸腺嘧啶而生长或生存。向生长培养基添加胸腺嘧啶或天然具有高胸腺嘧啶含量的人体肠道可以支持thyA营养缺陷型细菌的生长和生存。这种修饰是为了确保经基因工程改造的细菌不能在肠道外或在缺乏营养缺陷基因产物的环境中生长和生存。
在一些实施例中,宿主细胞是一种或多种选自由以下组成的群组的物质的营养缺陷体:尿嘧啶、亮氨酸、组氨酸、色氨酸、赖氨酸、甲硫氨酸、腺嘌呤和非天然存在的氨基酸。在一些实施例中,非天然存在的氨基酸选自由以下组成的群组:l-4,4′-联苯丙氨酸、对乙酰基-l-苯丙氨酸、对碘-l-苯丙氨酸和对叠氮基-l-苯丙氨酸。
在一些实施例中,宿主细胞包含变构调节的转录因子,所述转录因子能够检测调节所述转录因子的活性的环境中的信号,其中不存在所述信号将引起细胞死亡。所述“信号传导分子-转录因子”对可以包含选自由以下组成的群组的任一种或多种:色氨酸-TrpR、IPTG-LacI、苯甲酸酯衍生物-XylS、ATc-TetR、半乳糖-GalR、雌二醇-雌激素受体杂合蛋白、纤维二糖-CelR和高丝氨酸内酯-luxR。
v.内源质粒的去除
在一些实施例中,经基因工程改造的细菌具有一种或多种内源质粒的去除。
一些底盘细菌宿主包含一种或多种内源质粒,其为其转录消耗相当多的资源。不受任何理论束缚,认为删除这些内源质粒以释放可以更好地用于异源基因表达的资源。
一种或多种内源质粒可以通过所属领域中已知的方法从目标宿主细胞去除。举例来说,EcN包含两种内源质粒pMUT1和pMUT2,其可以通过以下方法从EcN去除。将表达pMUT1的sgRNA的pCBT003_pMUT1_sgRNA和pCBT001质粒转化到EcN中以去除pMUT1(EcNΔpMUT1)。为了删除pMUT2,模拟、制造耐卡那霉素(kanamycin)性质粒pMUT2(pMUT2-kana)且转移到EcNΔpMUT1中。随后,在补充有卡那霉素的LB琼脂板上选择经转化菌株。EcNΔpMUT1/pMUT2-kana在补充有增加浓度的卡那霉素的LB液体中生长若干代直至pMUT2完全被pMUT2-kana替换。EcNΔpMUT1/pMUT2-kana的pMUT2替换克隆用pCBT001转化,且随后用表达卡那霉素基因的sgRNA的质粒pCBT003_Kana-sgRNA转化以切割和去除pMUT2-kana。耗乏两种内源质粒的EcN菌株可以通过PCR检测检验。
vi.分泌产率增强的调节元件和整合位点的组合
经基因工程改造的宿主细胞(如细菌)中的异源蛋白质的分泌产率通常较低。为了治疗疾病,宿主细胞必须能够产生和分泌足够量的Amuc_1100,并且还应维持宿主细胞的健康。本发明人测试了多种基因组整合位点、调节元件和分泌系统,并且鉴别出可以在Amuc_1100表达和分泌中提供出人意料的效果的组合。
在某些实施例中,经基因工程改造的宿主细胞(例如细菌)的特征在于:(1)具有可以增加Amuc_1100转录的调节元件的组合;(2)具有整合到多个基因组位点中的Amuc_1100基因的多于一个拷贝;(3)具有经修饰以得到更好的蛋白分泌的信号肽;(4)具有经修饰的分泌系统,其产生“漏”的外膜;(5)具有额外伴侣基因;(6)具有减少的内源质粒或去除内源质粒;或其任何组合。
本发明人已经个别地或组合地集中测试基因组整合位点、启动子、RBS和顺反子,以增加Amuc_1100基因转录。
在某些实施例中,经基因工程改造的宿主细胞包含提供经基因工程改造的宿主细胞中Amuc_1100基因的转录增加的启动子、RBS和顺反子的组合。启动子、RBS和顺反子可以选自表2中的列表,以及本文中涵盖组合中的任一种。
在一些实施例中,启动子选自由以下组成的群组:BBa_J23119、BBa_J23101、BBa_J23102、BBa_J23103、BBa_J23109、BBa_J23110、BBa_J23114、BBa_J23117、USP45_启动子、Amuc_1102_启动子、OmpA_启动子、BBa_J23100、BBa_J23104、BBa_J23105、BBa_I14018、BBa_J45992、BBa_J23118、BBa_J23116、BBa_J23115、BBa_J23113、BBa_J23112、BBa_J23111、BBa_J23108、BBa_J23107、BBa_J23106、BBa_I14033、BBa_K256002、BBa_K1330002、BBa_J44002、BBa_J23150、BBa_I14034、Oxb19、oxb20、BBa_K088007、Ptet、Ptrc、PlacUV5、BBa_K292001、BBa_K292000、BBa_K137031、BBa_K137029、PLteto1、galP1、PLlacO1和Pfnrs。在一些实施例中,启动子包含BBa_J23101。在一些实施例中,启动子为BBa_J23101。在一些实施例中,RBS选自由以下组成的群组:USP45、合成型、Amuc_1102和OmpA。在一些实施例中,顺反子选自由以下组成的群组:GFP、BCD2、荧光素酶和MBP。
在一些实施例中,启动子包含BBa_J23101,RBS包含选自由以下组成的群组的至少一种(例如1、2、3、4种):USP45、合成型、Amuc_1102和OmpA,和/或顺反子包含选自由以下组成的群组的至少一种(例如1、2、3、4种):GFP、BCD2、荧光素酶和MBP。
在一些实施例中,启动子包含Pfnrs,RBS包含选自由以下组成的群组的至少一种(例如1、2、3、4种):USP45、合成型、Amuc_1102和OmpA,和/或顺反子包含选自由以下组成的群组的至少一种(例如1、2、3、4种):GFP、BCD2、荧光素酶和MBP。
在以上提供的实施例中的任一个中,终止子选自表2中列出的任何终止子。在一些实施例中,终止子是T7终止子。
在一些实施例中,启动子包含BBa_J23101或Pfnrs,RBS包含SEQ ID NO:71,信号肽包含USP45,并且终止子包含终止子1。在一些实施例中,启动子包含BBa_J23101或Pfnrs,RBS包含SEQ ID NO:71,信号肽包含USP45,并且终止子包含终止子2。在一些实施例中,启动子包含SEQ ID NO:15,RBS包含SEQ ID NO:71,信号肽的核苷酸序列包含SEQ ID NO:59,并且终止子包含SEQ ID NO:74。在一些实施例中,启动子包含SEQ ID NO:15,RBS包含SEQ IDNO:71,信号肽的核苷酸序列包含SEQ ID NO:59,并且终止子包含SEQ ID NO:75。
示范性优化Amuc_1100基因盒展示于以下SEQ ID NO:108和SEQ ID NO:148中。
SEQ ID NO:108
SEQ ID NO:148
在某些实施例中,经基因工程改造的宿主细胞包含或进一步包含整合到经基因工程改造的宿主细胞的多个基因组位点中的多于一个Amuc_1100基因的拷贝。
在一些实施例中,整合位点可以是选自由以下组成的群组的任一个或多个位点:agaI/rsmI、araBC、cadA、cadA、dapA、kefB、lacZ、maeB、malE/K、malP/T、rhtB/C、yicS/nepI、adhE、galK、glk、ldhA、lldD、maeA、nth、pflB、rnC、tkrA(ghrB)、yieN(ravA)、yjcS和LPP。在一些实施例中,整合位点可以选自表4中列出的任何位点。在一些实施例中,整合位点为整合位点agaI/rsmI。
在某些实施例中,经基因工程改造的宿主细胞包含或进一步包含伴侣基因的一个或多个额外拷贝,或伴侣基因的表达增加。伴侣基因可以选自由以下组成的群组:dsbA、dsbC、dnaK、dnaJ、grpE、groES、groEL、tig、fkpA、surA、skp、PpiD、DegP和其任何组合。在某些实施例中,伴侣基因选自由以下组成的群组:dsbA、dsbC、dnaK、dnaJ、grpE、groES、groEL、tig、fkpA、surA或其任何组合。在某些实施例中,经基因工程改造的宿主细胞包含一种或多种选自由以下组成的群组的伴侣基因:dsbA、dsbC、dnaK、dnaJ、grpE和其任何组合。在某些实施例中,经基因工程改造的宿主细胞包含一种或多种选自由以下组成的群组的伴侣基因:groES、groEL、tig、fkpA、surA和其任何组合。伴侣基因中的一些或全部可为宿主细胞(例如细菌)基因组中的插入的一个或多个拷贝,或其原生启动子可以被更强的启动子替换,以便增加其表达量。
在某些实施例中,经基因工程改造的宿主细胞在编码外膜蛋白的基因中包含或进一步包含失活或缺失。外膜蛋白为LPP。
图16展示一个编码Amuc_1100基因盒的经基因工程改造和优化的EcN的实例。在此经工程改造的菌株中,调整包含启动子、RBS、顺反子和编码信号肽的元件的Amuc_1100基因调节元件。将三个Amuc_1100基因盒拷贝插入到不同位点(agaI/rsmI、malPT和pflB)中,通过插入包含dsbA、dsbC、dnaK、dnaJ、grpE、groES、groEL、tig、fkpA和surA的伴侣基因,并且还通过删除lpp和thyA基因来修饰宿主EcN。
C.
免疫检查点是免疫系统的调节因子,并且属于免疫抑制路径或免疫刺激路径,负责T细胞反应的协同刺激性或抑制性的相互作用,并且调节和维持自身耐受性和生理免疫反应。
如本文所用,术语“免疫刺激路径”一般是指对宿主中免疫系统具有刺激作用的信号传导路径,其可以积极地调节细胞因子分泌、NK细胞活化、T细胞增殖和抗体产生等。本文所使用的免疫刺激检查点分子可以是任何已知或稍后发现的免疫刺激检查点分子。免疫刺激路径中发现的非限制性免疫刺激检查点分子可以尤其包含:CD2、CD3、CD7、CD16、CD27、CD30、CD70、CD83、CD28、CD80(B7-1)、CD86(B7-2)、CD40、CD40L(CD154)、CD47、CD122、CD137、CD137L、OX40(CD134)、OX40L(CD252)、NKG2C、4-1BB、LIGHT、PVRIG、SLAMF7、HVEM、BAFFR、ICAM-1、2B4、LFA-1、GITR、ICOS(CD278)或ICOSLG(CD275)。
如本文所用,术语“免疫抑制路径”一般是指对宿主中免疫系统具有抑制作用的宿主中的信号传导路径,其可以负面地调节细胞因子分泌、NK细胞活化、T细胞增殖和抗体产生等。本文所使用的免疫抑制检查点分子可以是任何已知或稍后发现的免疫抑制检查点分子。免疫抑制路径中发现的非限制性免疫抑制检查点分子可以尤其包含:LAG3(CD223)、A2AR、B7-H3(CD276)、B7-H4(VTCN1)、BTLA(CD272)、BTLA、CD160、CTLA-4(CD152)、IDO1、IDO2、TDO、KIR、LAIR-1、NOX2、PD-1、PD-L1、PD-L2、TIM-3、VISTA、SIGLEC-7(CD328)、TIGIT、PVR(CD155)、TGFβ或SIGLEC9(CD329)。
如本文所用,术语“免疫检查点调节剂”是指调节(例如,完全或部分地减少、抑制、干扰、活化、刺激、增加、加强或支持)免疫刺激或免疫抑制路径内的一个或多个免疫检查点的功能的分子。
在一些实施例中,本文所提供的免疫检查点调节剂包含免疫活化剂,其可完全或部分地活化、刺激、增加、加强、支持或正调节免疫刺激路径内的一个或多个免疫检查点的功能,或可完全或部分地降低、抑制、干扰或负调节免疫抑制路径内的一个或多个免疫检查点的功能。
免疫检查点调节剂通常能够调节(i)自身耐受性和/或(ii)免疫反应的幅度和/或持续时间。优选地,在本公开中使用的免疫检查点调节剂调节一个或多个人类检查点分子的功能,并且因此为“人类免疫检查点调节剂”。
在一些实施例中,所述免疫检查点调节剂包含一个或多个选自由以下组成的群组的免疫刺激检查点的活化剂:CD2、CD3、CD7、CD16、CD27、CD30、CD70、CD83、CD28、CD80(B7-1)、CD86(B7-2)、CD40、CD40L(CD154)、CD47、CD122、CD137、CD137L、OX40(CD134)、OX40L(CD252)、NKG2C、4-1BB、LIGHT、PVRIG、SLAMF7、HVEM、BAFFR、ICAM-1、2B4、LFA-1、GITR、ICOS(CD278)、ICOSLG(CD275)和其任何组合。
在一些实施例中,所述免疫检查点调节剂包含一个或多个选自由以下组成的群组的免疫抑制检查点的抑制剂:LAG3(CD223)、A2AR、B7-H3(CD276)、B7-H4(VTCN1)、BTLA(CD272)、BTLA、CD160、CTLA-4(CD152)、IDO1、IDO2、TDO、KIR、LAIR-1、NOX2、PD-1、PD-L1、PD-L2、TIM-3、VISTA、SIGLEC-7(CD328)、TIGIT、PVR(CD155)、TGFβ、SIGLEC9(CD329)和其任何组合。
这些上文所提及的免疫刺激和免疫抑制路径中的调节剂的细节在所属领域中已知,参见例如WO 97/28259;WO 98/16247;WO 99/11275;Krieg AM等人,1995;Yamamoto S等人,1992;Ballas ZK等人,1996;Klinman DM等人,1997;Sato Y等人,1996;Pisetsky DS,1996;Shimada S等人,1986;Cowdery JS等人,1996;Roman H等人,1997;Lipford GB等人,1997;WO 98/55495和WO 00/61151等。
在一些实施例中,本公开的免疫检查点调节剂为针对本公开的免疫检查点的抗体或其抗原结合片段或化合物。针对免疫检查点的抗体的非限制性实例包含抗PD-1抗体(包含但不限于纳武单抗(Nivolumab)、匹立珠单抗(Pidilizumab)、派立珠单抗(Pembrolizumab)(MK-3475/SCH900475、拉立珠单抗(lambrolizumab)、REGN2810、PD-1(Agenus))、抗PD-L1抗体(包含但不限于度伐单抗(durvalumab)(MEDI4736)、阿维鲁单抗(avelumab)(MSB0010718C)和阿特珠单抗(atezolizumab)(MPDL3280A、RG7446、R05541267))、抗PDL-2抗体(包含但不限于AMP-224(描述于WO2010027827和WO2011066342中))、CTLA-4抗体(包含但不限于伊匹单抗(Ipilimumab)和曲美单抗(Tremelimumab)(CP675206))、抗4-1BB抗体(包含但不限于PF-05082566和乌瑞鲁单抗(Urelumab))、LAG3抗体(包含但不限于BMS-986016)、抗CD27抗体(包含但不限于瓦力单抗(Varlilumab))。
在一些实施例中,在本文所提供的方法中,使用超过一种免疫检查点调节剂(例如抗PD-1抗体和抗PD-L1抗体)。
在一些实施例中,免疫检查点调节剂调节(或抑制)免疫抑制检查点。在一些实施例中,免疫抑制检查点为PD-L1、PD-L2或PD-1。在一些实施例中,免疫检查点调节剂抑制PD-1与其配体中的任一种之间的相互作用。在一些实施例中,免疫检查点调节剂包含抑制或减少PD-1与其配体中的任一种之间的相互作用或信号传导(例如PD-L1/PD-L2或PD-1之间的相互作用)的抗体(例如人类抗体、人源化抗体或嵌合抗体)或其抗原结合片段或化合物。在一些实施例中,免疫检查点调节剂包含抗PD-1抗体、抗PD-L1抗体、抗PD-L2抗体、小分子PD-1抑制剂、小分子PD-L1抑制剂或小分子PD-L2抑制剂。
在一些实施例中,本文所述的抗体(如抗PD-1抗体、抗PD-L1抗体或抗PD-L2抗体)进一步包含人类或鼠类恒定区。在一些实施例中,人类恒定区选自由以下组成的群组:IgG1、IgG2、IgG2、IgG3、IgG4。在一些实施例中,人类恒定区为IgG1。在一些实施例中,鼠类恒定区选自由以下组成的群组:IgG1、IgG2A、IgG2B、IgG3。在一些实施例中,抗体具有减少或极小的效应功能。在一些实施例中,极小效应功能由原核细胞中的产生造成。在一些实施例中,极小效应功能由“无效应子Fc突变”或非糖基化造成。
因此,本文所使用的抗体可以经糖基化。抗体的糖基化通常是N-连接型或O-连接型。N-连接型是指碳水化合物部分连接于天冬酰胺残基的侧链。三肽序列天冬酰胺-X-丝氨酸和天冬酰胺-X-苏氨酸(其中X是除脯氨酸以外的任何氨基酸)是碳水化合物部分与天冬酰胺侧链的酶连接的识别序列。因此,多肽中这些三肽序列中任一种的存在产生了潜在的糖基化位点。O-连接型糖基化是指糖N-乙酰基半乳糖胺、半乳糖或木糖中的一种连接于羟基氨基酸,最通常丝氨酸或苏氨酸,但也可以使用5-羟基脯氨酸或5-羟基赖氨酸。通过改变氨基酸序列以使得上文所描述的三肽序列中的一种(用于N-连接型糖基化位点)被去除来方便地实现形成抗体的糖基化位点的去除。改变可以通过用另一氨基酸残基(例如甘氨酸、丙氨酸或保守取代)取代糖基化位点内的天冬酰胺、丝氨酸或苏氨酸残基而进行。
抗体或其抗原结合片段可以使用所属领域中已知的方法,例如通过包含以下的工艺制造:在适于产生所述抗体或片段的条件下,以适合于表达的形式培养含有编码先前所描述的抗PD-1、抗PD-L1或抗PDL2抗体或抗原结合片段中的任一种的核酸的宿主细胞,并且回收抗体或片段。
针对本公开的免疫检查点的化合物可以是小分子化合物。如本文所用,术语“小分子化合物”意指可充当生物过程的酶底物或调节因子的低分子量化合物。一般来说,“小分子化合物”为大小小于约5千道尔顿(kD)的分子。在一些实施例中,小分子小于约4kD、3kD、约2kD或约1kD。在一些实施例中,小分子小于约800道尔顿(D)、约600D、约500D、约400D、约300D、约200D或约100D。在一些实施例中,小分子小于约2000g/mol、小于约1500g/mol、小于约1000g/mol、小于约800g/mol或小于约500g/mol。在一些实施例中,小分子是非聚合物。在一些实施例中,根据本公开,小分子不是蛋白质、多肽、寡肽、肽、多核苷酸、寡核苷酸、多糖、糖蛋白、蛋白多糖等。在一些实施例中,小分子为治疗性的。在一些实施例中,小分子是佐剂。在一些实施例中,小分子是药物。
在一些实施例中,个体已表现出对免疫检查点调节剂的不良反应或耐药性。对免疫检查点调节剂的“不良反应”或“耐药性”是指接受免疫检查点调节剂的治疗的个体对治疗无反应或对治疗反应不良,并且由此个体的疾病、病症或病状未得到治疗。如本文所用,术语“耐药性”是指对于治疗剂(如免疫检查点调节剂)具有难治性或复发或非反应性。可以通过现有技术中已知的方式来确定个体对免疫检查点调节剂的治疗的反应。
在一些实施例中,个体经测定对免疫检查点调节剂具有原发性耐药或获得性耐药。如本文所用,“原发性耐药”是指在用给定药剂治疗之前存在的耐药性。因此,“对免疫检查点调节剂的原发性耐药”意味着个体在免疫检查点调节剂治疗之前对免疫检查点调节剂具有耐药性或非反应性。如本文所用,“获得性耐药”是指在用给定药剂进行至少一次治疗之后所获得的耐药性。在至少一次治疗之前,疾病不对药剂具有耐药性(并且因此,疾病如同无耐药性病症那样对第一次治疗有反应)。举例来说,对免疫检查点调节剂具有获得性耐药的个体为最初对免疫检查点调节剂的至少一次治疗有反应,且此后对免疫检查点调节剂的后续治疗产生耐药性的个体。
D.
如上文所提及,本公开提供一种治疗或预防有需要的个体的癌症的方法,本公开还提供一种改善接受免疫检查点调节剂的治疗的个体中癌症的治疗反应的方法。术语“癌症”和“肿瘤”在本文中可互换地使用,并且是指其中细胞展现相对异常、不受控和/或自主生长,使得其呈现异常升高的增殖速率和/或特征在于细胞增殖的显著失控的异常生长表型的疾病、病症或病状。在一些实施例中,由于致癌基因的表达和活性或肿瘤抑制基因(如成视网膜细胞瘤蛋白质(Rb))的有缺陷的表达和/或活性,所述细胞部分或完全展现所述特征。癌细胞通常呈肿瘤形式,但所述细胞可单独存在于动物体内,或可为非致瘤性癌细胞,如白血病细胞。如本文所用,术语“癌症”包含恶变前癌症以及恶性癌症。
短语“改善治疗反应”可以包含例如延迟疾病进展或减少或抑制癌症复发。
如本文所用,“延迟疾病进展”意味着推迟、阻碍、减缓、延缓、稳定化和/或延迟疾病(如癌症)的发展。这一延迟可以具有不同时间长度,取决于疾病病史和/或所治疗的个体。如所属领域的技术人员显而易见,足够或显著延迟可以实际上涵盖预防,从而个体不罹患疾病。举例来说,可延迟晚期癌症,例如转移的发展。
如本文所用,“减少或抑制癌症复发”意思是减少或抑制肿瘤或癌症复发或肿瘤或癌症进展。如本文所公开,癌症复发和/或癌症进展包含但不限于癌症转移。
在一些实施例中,本公开的癌症选自由以下组成的群组:结肠直肠癌、乳癌、卵巢癌、胰腺癌、胃癌、前列腺癌、胰腺癌、肾癌、子宫颈癌、骨髓瘤、淋巴瘤、白血病、甲状腺癌、子宫内膜癌、子宫癌、膀胱癌、神经内分泌癌、头颈癌、肝癌、鼻咽癌、睾丸癌、肺癌(例如小细胞肺癌、非小细胞肺癌)、黑色素瘤、基细胞癌、皮肤癌、鳞状细胞皮肤癌、隆凸性皮肤纤维肉瘤、梅克尔细胞癌、成胶质细胞瘤、神经胶质瘤、肉瘤和间皮瘤。在一些实施例中,癌症为乳癌。
如上文所提及,本公开还提供一种在个体(例如人类)中诱导或改善肠道免疫性的方法。肠道微生物群居于人类已有数百万年。细菌和人类已经确立了相对稳定且共生的进化关系。此关系使得肠道细菌具有调节和塑造肠道免疫系统和生理机能的功能。肠道微生物群失调已与多种疾病相关,所述疾病包含癌症、代谢疾病、自身免疫疾病、心血管疾病、神经退化性疾病、肝病等。近期的证据表明,肠道微生物群组合物,尤其某些细菌菌株可以预测或增加癌症免疫疗法的功效。尽管许多控制肠道免疫性与肠道微生物群之间的复杂相互作用的详细分子机制仍未被破解,但本发明人出乎意料地发现Amuc_1100(或表达Amuc_1100的经基因工程改造的宿主细胞)与免疫检查点调节剂的组合可协同诱导或改善个体的肠道免疫性。
E.
本文提供的Amuc_1100或表达Amuc_1100的经基因工程改造的宿主细胞可以通过所属领域中已知的任何途径施用,例如肠胃外(例如,皮下、腹膜内、静脉内(包含静脉内输注)、肌肉内或皮内注射)或非肠胃外(例如,口服、鼻内、眼内、舌下、经直肠或局部)途径。在一些实施例中,施用是通过口服施用。
在一些实施例中,与免疫检查点调节剂组合施用的本文所提供的Amuc_1100或表达Amuc_1100的经基因工程改造的宿主细胞可与免疫检查点调节剂同时施用,并且在这些实施例中的某些中,Amuc_1100或表达Amuc_1100的经基因工程改造的宿主细胞和免疫检查点调节剂可以作为同一药物组合物的一部分施用。然而,与免疫检查点调节剂“组合”施用的Amuc_1100或表达Amuc_1100的经基因工程改造的宿主细胞不必与药剂同时施用或以与药剂相同的组合物施用。在免疫检查点调节剂之前或之后施用的Amuc_1100或表达Amuc_1100的经基因工程改造的宿主细胞被视为如本文中所使用的与免疫检查点调节剂“组合”施用的短语,即使Amuc_1100或表达Amuc_1100的经基因工程改造的宿主细胞和免疫检查点调节剂通过不同途径施用(例如Amuc_1100或表达Amuc_1100的经基因工程改造的宿主细胞口服施用,而免疫检查点调节剂通过注射施用)。举例来说,Amuc_1100(或表达Amuc_1100的经基因工程改造的宿主细胞)可在免疫检查点调节剂之前施用(例如5小时、10小时、12小时、24小时、48小时、72小时、96小时、1周、2周、3周等),施用要么准时(punctually)要么在施用免疫检查点调节剂之前进行若干次,只要其在重叠时间范围期间在个体中发挥作用即可。对于另一实例,Amuc_1100(或表达Amuc_1100的经基因工程改造的宿主细胞)可在免疫检查点调节剂之前施用某一时间段(例如5小时、10小时、12小时、24小时、48小时、72小时、96小时、1周、2周、3周等),且随后同时施用Amuc_1100(或表达Amuc_1100的经基因工程改造的宿主细胞)和免疫检查点调节剂。当可能时,根据额外治疗剂的产品信息表单中列出的时间表或根据《医师案头参考2003(Physicians'Desk Reference 2003)》(《医师案头参考》第57版;Medical Economics Company;ISBN:1563634457;第57版(2002年11月))或所属领域中众所周知的方案,施用与本文中所公开的Amuc_1100或表达Amuc_1100的经基因工程改造的宿主细胞组合施用的一种或多种免疫检查点调节剂。
在一些实施例中,本公开还提供包含本公开的重组表达盒的核酸载体,其用于与免疫检查点调节剂组合使用。
在另一方面,本公开还提供本公开的经基因工程改造的宿主细胞,其用于与免疫检查点调节剂组合使用。
III.
在另一方面,本公开提供一种试剂盒,其包含:(a)包含本公开的经基因工程改造的宿主细胞或包含分离的Amuc_1100的第一组合物;和(b)包含免疫检查点抑制剂的第二组合物。
在一些实施例中,第一组合物是可食用组合物,和/或第二组合物进一步包含药学上可接受的载剂。在一些实施例中,第一组合物是食品增补剂。在一些实施例中,第二组合物适于口服施用或肠胃外施用。
必要时,所述试剂盒可进一步包含各种常规药物试剂盒组分中的一种或多种,例如具有一种或多种药学上可接受的载剂的容器、额外容器等,如对所属领域的技术人员将显而易见。试剂盒中还可以包含作为插页或标签的说明书,指示应施用的组分的量、施用指南和/或混合组分的指南。
IV.
A.
在一个方面,本公开提供一种组合物,其包含本公开的Amuc_1100或其功能等效物和生理学上可接受的载剂。
在一些实施例中,Amuc_1100包含Amuc_1100多肽。在一些实施例中,Amuc_1100多肽为重组Amuc_1100多肽。在一些实施例中,Amuc_1100多肽为经纯化的重组Amuc_1100多肽。在一些实施例中,经纯化的重组Amuc_1100多肽的纯度为至少80重量%、85重量%、90重量%、91重量%、92重量%、93重量%、94重量%、95重量%、96重量%、97重量%、98重量%、99重量%、99.5重量%或99.9重量%。
用于本文所公开的组合物的生理学上可接受的载剂可包含例如生理学上可接受的液体、凝胶或固体载剂、水性媒剂、非水性媒剂、抗微生物剂、等渗剂、缓冲剂、抗氧化剂、悬浮剂/分散剂、螯合剂(sequestering/chelating agent)、稀释剂、佐剂、赋形剂或无毒辅助物质、所属领域中已知的其它组分或其各种组合。
为了进一步说明,水性媒剂可包含如氯化钠注射液、林格注射液、等渗右旋糖注射液、无菌水注射液或右旋糖和乳酸林格注射液;非水性媒剂可包含如植物来源的不挥发性油、棉籽油、玉米油、芝麻油或花生油;抗微生物剂可为抑细菌或抑真菌浓度,和/或可以添加到在多剂量容器中的组合物中,所述容器包含苯酚或甲酚、汞剂、苯甲醇、氯丁醇、甲基和丙基对羟基苯甲酸酯、硫柳汞、苯扎氯铵和苄索氯铵。等渗剂可以包含如氯化钠或右旋糖;缓冲剂可以包含如磷酸盐或柠檬酸盐缓冲剂;抗氧化剂可以包含如硫酸氢钠;悬浮剂和分散剂可以包含如羧甲基纤维素钠、羟丙基甲基纤维素或聚乙烯吡咯烷酮;螯合剂可以包含如乙二胺四乙酸(EDTA)或乙二醇四乙酸(EGTA)、乙醇、聚乙二醇、丙二醇、氢氧化钠、盐酸、柠檬酸或乳酸。适合的赋形剂可包含例如水、生理盐水、右旋糖、甘油或乙醇。适合的无毒辅助物质可包含例如润湿剂或乳化剂、pH缓冲剂、稳定剂、溶解性增强剂或如乙酸钠、脱水山梨糖醇单月桂酸酯、三乙醇胺油酸酯或环糊精的试剂。
在一些实施例中,本文所提供的组合物可为药物组合物。在一些实施例中,本文所提供的组合物可以是食品增补剂。本文所提供的组合物除本文所提供的Amuc_1100之外可以包含药学上、营养学上(nutritionally/alimentarily)或生理学上可接受的载剂。优选形式将取决于预期施用模式和(治疗性)应用。载剂可以是适合于将本文提供的Amuc_1100递送到哺乳动物(例如人类)的胃肠道,优选地哺乳动物的肠粘膜屏障(更优选地结肠粘膜屏障)附近或内的任何相容性生理学上可接受的无毒物质。
组合物可为液体溶液、悬浮液、乳液、丸剂、胶囊、片剂、持续释放制剂或散剂。口服制剂可包含标准载剂,如药物级甘露糖醇、乳糖、淀粉、硬脂酸镁、聚乙烯吡咯烷酮、糖精钠、纤维素、碳酸镁等。
B.
在另一方面,本公开还提供一种组合物,其包含表达Amuc_1100或其功能等效物的经基因工程改造的宿主细胞和生理学上可接受的载剂。宿主细胞可以约10
在一些实施例中,本文所公开的组合物可以经配制用于口服施用,并且可以是营养或滋养组合物,例如食品、食品增补剂、饲料或饲料增补剂,如乳制品,例如发酵乳制品,如酸奶或酸奶饮料。在此情况下,组合物可包含营养学上可接受的载剂,其可为适合的食物基。所属领域的技术人员知道可涵盖活或死微生物体且可呈现为食品增补剂(例如,丸剂、片剂等)或功能性食品(例如饮料、发酵酸奶等)的多种配方。本文所公开的组合物还可以在胶囊、丸剂、液体溶液中配制为药剂,例如配制为囊封的冻干细菌等。
本文所公开的组合物可以被配制成单次施用或多次施用而对给定个体有效。举例来说,单次施用实质上有效降低施用组合物的哺乳动物个体的靶向疾病病状的监测症状。
在一些实施例中,配制组合物以使得单一口服剂量含有至少或至少约1×10
在一些配方中,按质量计,组合物含有至少或至少约0.5%、1%、2%、5%、10%、20%、30%、40%、50%、60%、70%、80%、90%或大于90%本公开的宿主细胞。在一些配方中,按质量计,所施用的剂量不超过200、300、400、500、600、700、800、900毫克或1、1.1、1.2、1.3、1.4、1.5、1.6、1.7、1.8或1.9克的本公开的宿主细胞。
C.
在另一方面,本公开提供一种组合物,其包含:(a)本公开的经基因工程改造的宿主细胞或本公开的分离的Amuc_1100;和(b)本公开的免疫检查点抑制剂。在一些实施例中,组合物是可食用的。
在另一方面,本公开提供一种包含本公开的经基因工程改造的宿主细胞或分离的Amuc_1100的组合物,其用于与包含本公开的免疫检查点调节剂的制剂组合使用。在一些实施例中,组合物可以通过所属领域中已知的任何途径向个体施用,例如非肠胃外(例如,口服、鼻内、眼内、舌下、经直肠或局部)途径。
在一些实施例中,组合物是口服组合物。在一些实施例中,包含免疫检查点调节剂的制剂为肠胃外(例如皮下、腹膜内、静脉内(包含静脉内输注)、肌肉内或皮内注射)制剂。在一些实施例中,包含免疫检查点调节剂的制剂是口服制剂。
在另一方面,本公开提供包含Amuc_1100(或表达Amuc_1100的经基因工程改造的宿主细胞)的组合物用于制造用于治疗或预防癌症的药剂的用途。
在另一方面,本公开提供包含Amuc_1100(或表达Amuc_1100的经基因工程改造的宿主细胞)的组合物用于制造用于改善接受免疫检查点调节剂的治疗的个体中癌症的治疗反应的药剂的用途。
在另一方面,本公开提供包含Amuc_1100(或表达Amuc_1100的经基因工程改造的宿主细胞)的组合物用于制造用于在个体中诱导或改善肠道免疫性的药剂的用途。
在另一方面,本公开提供包含Amuc_1100(或表达Amuc_1100的经基因工程改造的宿主细胞)的组合物用于制造用于在接受抗癌治疗并且表现出肠道免疫性降低的个体中改善癌症治疗反应的药剂的用途。
提供以下实施例是为了更好地说明所要求的发明,而不应理解为限制本发明的范围。下述所有特定组合物、材料和方法完全或部分地落入本发明的范围内。这些特定组合物、材料和方法并不意图限制本发明,而是仅说明落入本发明范围内的特定实施例。在不脱离本发明范围的情况下,所属领域的技术人员无需履行发明能力即可开发出等效组合物、材料和方法。应理解,可对本文所述的程序作出许多变更,但仍在本发明的范围内。本发明人打算将所述变化形式包含在本发明的范围内。
V.Amuc_1100突变体
本公开也提供了非天然存在的Amuc_1100蛋白。在一些实施例中,Amuc_1100多肽在选自由以下组成的群组的位置处包含一个或多个(例如2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25个或更多个)突变:R36、S37、L39、D40、K41、K42、I43、K48、E49、K51、S52、R62、S63、K70、E71、L72、N73、R74、Y75、A76、K77、A78、Y86、K87、P88、F89、L90、A91、F103、Q104、K108、T109、F110、R111、D112、K119、K120、K121、N122、L124、I125、W131、L132、G133、F134、Q135、Y137、S138、L150、G151、F152、E153、L154、K155、A156、L160、V161、K163、L164、A165、L169、S170、K171、F172、I173、K174、V175、Y176、R177、W200、T201、L205、E206、F209、Q210、R213、E214、L217、K218、A219、M220、N221、Y229、L230、R237、I238、R242、M243、M244、P245、K255、P256、L268、T269、K287、P288、Y289、M290、K292、E293、F296、V297、F306、N307、K310和A311,其中编号相对于SEQ ID NO:1。
在一些实施例中,Amuc_1100多肽在选自由以下组成的群组的位置处包含一个或多个突变:S37、V175、Y289和F296,其中编号相对于SEQ ID NO:1。在一些实施例中,Amuc_1100多肽包含Y289A突变,其中编号相对于SEQ ID NO:1。在一些实施例中,Amuc_1100包含如SEQ ID NO:4或SEQ ID NO:5所示的氨基酸序列。
本文提供的非天然存在的Amuc_1100蛋白,例如,上文提及的包含Y289A突变的Amuc_1100突变体,相比其天然存在形式,表现出对酶消化更强的抵抗性,这对于将表达Amuc-1100的工程菌应用于存在能够降解从工程菌中分泌释放到环境中的Amuc_1100的酶的环境中将具有很大优势。
在另一方面,本公开还提供了编码上述非天然存在的Amuc_1100蛋白的核酸。本领域技术人员能够利用各种可公开获得的工具例如tblastn(可通过美国国立生物技术信息中心(NCBI)网站获取)获取或者优化所述编码非天然存在的Amuc_1100蛋白的核酸。本领域技术人员进行blast时可以使用工具提供的默认参数或者根据需要定制参数,例如选择一个合适的算法。
VI.Usp45信号肽
本公开还提供了利用Usp45信号肽构建包含编码目标多肽的多核苷酸的表达载体,其中所述表达载体适合于在大肠杆菌中表达从而允许在在大肠杆菌中表达并分泌目标多肽。
本公开还提供了Usp45信号肽,用于构建包含编码目标多肽的多核苷酸的表达载体,其中所述表达载体适合于在大肠杆菌中表达从而允许在在大肠杆菌中表达并分泌目标多肽。
本公开还提供了一种包含编码与Usp45信号肽可操作连接的目标多肽的多核苷酸的表达载体,其中所述表达载体适合于在大肠杆菌中表达从而允许在在大肠杆菌中表达并分泌目标多肽。
在另一方面中,本公开还提供了一种包含外源表达盒的基因工程大肠杆菌,所述外源表达盒包含编码与Usp45信号肽可操作连接的目标多肽的多核苷酸。所述基因工程大肠杆菌能够表达和分泌所述目标多肽。
在一些实施例中,前述Usp45信号肽包含如SEQ ID NO:59所示的序列。在某些实施例中,所述大肠杆菌是大肠杆菌的益生菌株,例如大肠杆菌菌株Nissle 1917。
所述目标多肽可以包括Amuc_1100。目标多肽也可以包括除了Amuc_1100外的其它多肽。
Usp45信号肽是来自乳酸乳球菌(Lactococcus lactis)主要分泌蛋白的信号肽,也是在乳酸乳球菌中用于蛋白分泌的最常用信号肽(例如,参见Jhonatan et al.,ApplEnviron Microbiol.2020Aug;86(16):e00903-20.)。总体而言,Usp45信号肽被认为仅在乳酸乳球菌中正常工作,从未考虑或研究其在大肠杆菌用于指导蛋白分泌的能力。因此将Usp45信号肽应用于大肠杆菌,例如大肠杆菌菌株Nissle 1917,以及利用Usp45作为信号肽以分泌目标多肽的基因工程大肠杆菌,例如大肠杆菌Nissle1917,均具有新颖性而且还使得目标蛋白达到了预料不到的高效分泌。
实施例
实施例1.重组Amuc_1100和其突变体的产生
针对Amuc_1100(31-317)的密码子优化的DNA序列在限制酶切位点NdeI和XhoI构建于质粒pET-22b(+)(命名为“pET-22b(+)_Amuc_1100”,如图1)中所示。随后将质粒转化到大肠杆菌BL21(DE3)中。在37℃下将经转化细菌在LB培养基中培养到0.8的OD600值的细胞密度。Amuc_1100(31-317)的表达由乳糖类似物IPTG(0.1mM)在16℃下诱导过夜,或由IPTG(1mM)在37℃下诱导3小时。蛋白质表达通过SDS-PAGE凝胶和考马斯蓝(Coomassie blue)染色检测。
收集Amuc_1100(31-317)表达诱导细菌,并且将其再悬浮于缓冲液(20mM PB,500mM NaCl,pH 7.4)中并且通过超声波处理溶解。细胞溶解物的上清液通过螯合SFF(Ni)柱纯化且通过含咪唑的缓冲液洗脱。从螯合SFF(Ni)柱收集的Amuc_1100通过Q-HP柱富集并且通过含有降低浓度的NaCl的缓冲液(20mM PB,500mM NaCl,pH 7.4)洗脱。经纯化的重组Amuc_1100(31-317)通过胰蛋白酶和胰凝乳蛋白酶的混合物在非还原条件下在PBS中在37℃下消化30分钟。样品通过SDS-PAGE凝胶和考马斯蓝染色分析。
随后使胰蛋白酶和胰凝乳蛋白酶消化的Amuc_1100(31-317)经历MS分析以检测蛋白质的消化位点。如图2的SEQ ID NO:145中所示,紧邻标记“/”的氨基酸为对于酶消化敏感的位点。确切地说,Ser37、Val175、Tyr289和Phe296(编号相对于SEQ ID NO:1)突出显示为对于酶消化敏感的位点。
Amuc_1100(31-317)的一些突变体可以减少蛋白酶的蛋白质降解。由上文所描述的质粒(图1)表达的C端具有6×His标签的Amuc_1100(31-317,Y289A)的氨基酸序列(SEQID NO:146)和核苷酸序列(SEQ ID NO:147)展示于图2中。重组Amuc_1100(31-317)蛋白质和突变蛋白质(31-317,Y289A)在37℃下通过胰蛋白酶(0.01mg/ml)和α-胰凝乳蛋白酶(0.01mg/ml)消化30分钟。随后使样品经历SDS-PAGE凝胶电泳和考马斯蓝染色。如图3A所示,Amuc_1100(31-317,Y289A)蛋白相比于野生型Amuc_1100蛋白能够更好的抵抗蛋白酶消化,这表明单一的点突变Y289A能够给Amuc_1100带来更高的稳定性。
将表达人类TLR2受体和其报告基因的HEK293细胞(InvivoGen,hkb-htlr2)接种于96孔板中并且通过Amuc_1100(31-317)或突变重组Amuc_1100(31-317,Y289A)处理。报告基因活性通过QUANTI-Blue
实施例2.Amuc_1100(31-317)或其突变体在癌症治疗中的用途
MMTV-PyVT转基因C57BL/6雌性小鼠用于产生自发乳癌。将9.5周龄的小鼠随机分成每组4-5只小鼠的6组,并且进行药剂处理。对照组中的小鼠每日通过经口管饲用PBS处理持续4周,加经口管饲一周后每日静脉内注射PBS持续3周。五个测试组中的小鼠用以下处理:(1)Amuc_1100(31-317)蛋白的PBS溶液(3μg/kg),每日经口管饲持续4周,(2)Amuc_1100(31-317,Y289A)蛋白的PBS溶液(3μg/kg),每日经口管饲持续4周,(3)抗PD-1抗体(InVivoMAb抗小鼠PD-1(CD279)(克隆RMP1-14)(BioXcell,目录号BE0146))(经口管饲PBS一周后),每三天静脉内注射(10mg/kg)持续3周,(4)如上文所描述的Amuc_1100(31-317)蛋白和抗PD-1抗体的组合,即Amuc_1100(31-317)(3μg/kg)每日经口管饲持续一周,且随后抗PD-1抗体(10mg/kg,每三天静脉内注射)与Amuc_1100(31-317)(每日经口管饲)同时,再持续3周,(5)如上文所描述的Amuc_1100(31-317,Y289A)蛋白和抗PD-1抗体的组合,即Amuc_1100(31-317,Y289A)(3μg/kg)每日经口管饲持续一周,且随后抗PD-1抗体(10mg/kg,每三天静脉内注射)与Amuc_1100(31-317,Y289A)(3μg/kg)(每日经口管饲)同时,再持续3周。监测所有自发产生的肿瘤。每周两次测量肿瘤大小。密切地监测小鼠的健康状况。分析在药剂处理之后小鼠中的每个肿瘤的肿瘤生长速率。在研究结束时,收集肿瘤组织并且将其固定于福尔马林中。进行H&E染色以测定组织中的肿瘤细胞(深色染色)。染色结果展示于图4中,且每一组中的每只小鼠中的所有肿瘤的平均肿瘤生长速率展示于图5中。
如图4和图5中所示,Amuc_1100(31-317)蛋白质、Amuc_1100(31-317,Y289A)蛋白质和抗PD-1抗体部分地抑制肿瘤生长,而Amuc_1100(31-317)蛋白质和抗PD-1抗体的组合,和Amuc_1100(31-317,Y289A)蛋白质和抗PD-1抗体的组合协同地抑制肿瘤生长,即,比单独任一处理显著更有效。
还测试了在结直肠癌模型小鼠中(通过皮下注射MC-38细胞到C57BL/6小鼠)单独使用Amuc_1100(31-317)或Amuc_1100(Y289A),或者与anti-PD-1抗体联用进行治疗的方案,也能够观察到Amuc_1100蛋白和anti-PD-1抗体取得了相似的协同效果。
实施例3.Amuc_1100或其突变体对模型小鼠的体重增加的影响
将9.5周龄的小鼠随机分成每组4-5只小鼠的3组,并且进行药剂处理。对照组和测试组中的小鼠分别通过每日经口管饲,用PBS、Amuc_1100(31-317)或Amuc_1100(31-317,Y289A)(3μg/kg)处理4周。每组中的每只小鼠的体重每周测量两次。结果展示,与对照组相比,测试组(即用Amuc_1100(31-317)或Amuc_1100(31-317,Y289A)处理)小鼠中未观测到体重的显著变化。
实施例4.Amuc_1100或其突变体对Treg浸润的影响
MMTV-PyVT转基因C57BL/6雌性小鼠用于产生自发乳癌。将9.5周龄的小鼠随机分成每组4-5只小鼠的3组,并且进行药剂处理。对照组和测试组中的小鼠分别通过每日经口管饲,用PBS、Amuc_1100(31-317)或Amuc_1100(31-317,Y289A)处理4周。在研究结束时,收集肿瘤组织并且将其固定于福尔马林中。进行H&E染色或免疫荧光分析以测定肿瘤组织中免疫细胞(包含巨噬细胞、Treg、CD4+T细胞、CD8+T细胞等)的分布。
实施例5.细菌基因组编辑方案
5.1sgRNA设计
在目标整合位点序列的两股上寻找连同NGG PAM序列的20bp序列(N20NGG)并且针对EnN基因组进行blast处理。选择独特的20bp序列作为目标整合位点的sgRNA。选择sgRNA上游和下游的300-500bp序列作为左同源臂(LHA)和右同源臂(RHA)。
5.2sgRNA质粒构建
将sgRNA序列添加到pCBT003质粒上gRNA骨架的反向引物的5'端,随后将gRNA骨架与设计的sgRNA序列一起从pCBT003扩增,PCR产物和pCBT003质粒都用PstI和SpeI进行消化,将消化的pCBT003质粒去磷酸化,且随后与消化的PCR片段接合,来产生pCBT003_sgRNA质粒(图6)。
如图19中显示的,pCBT003的核酸序列如SEQ ID NO:150所示。
5.3供体基因盒设计
待整合到EcN基因组的目标基因由金斯瑞(GeneScript)在克隆质粒(例如pUC57)上合成(pUC57_Amuc_1100,如图7中所示)。从质粒pUC57_Amuc_1100扩增目标基因,从EcN的基因组扩增所选整合位点的LHA和RHA。用于这些片段的PCR的引物彼此具有15-20bp的同源序列,因此它们可以通过重叠PCR与由LHA和RHA侧接的目标基因接合。线性PCR产物用作供体基因盒。
5.4质粒电穿孔
5.4.1制造EcN的电转感受态细胞
将LB琼脂板上的单一EcN菌落接种到试管中的3mL LB培养基且在37℃下在震荡(225rpm)下培育过夜,将300μL此过夜培养物接种到125烧瓶中的30mL LB培养基,在37℃下在震荡(225rpm)下培育直到OD
5.4.2pCBT001质粒的电穿孔
将100-200ng pCBT001质粒(即表达Cas9蛋白的质粒,参见如SEQ ID NO:149所示的核苷酸序列)添加到100μL EcN电转感受态细胞中,将混合物转移到2mm电穿孔比色皿,并且在2.5kV、25μF、200Ω下电穿孔。随后通过将细胞再悬浮于1mL SOC中并且在30℃下在震荡(225rpm)下培育两小时来回收细胞。在回收之后,将所有细胞涂铺于补充有50μg/mL壮观霉素(spectinomycin)和链霉素的LB琼脂板上并且在30℃下培育过夜。板上生长的菌落为具有pCBT001质粒的EcN(EcN/pCBT001,如图8中所示)。
如图20中显示的,pCBT001的核酸序列如SEQ ID NO:149所示。
5.4.3制造EcN/pCBT001的电转感受态细胞
将补充有50μg/mL壮观霉素和链霉素的LB琼脂板上的单一EcN/pCBT001菌落接种到试管中的补充有50μg/mL壮观霉素和链霉素的3mL LB液体培养基,且在30℃下在震荡(225rpm)下培育过夜,将300μL此过夜培养物接种到125烧瓶中的补充有50μg/mL壮观霉素和链霉素的30mL LB液体培养基,在30℃下在震荡(225rpm)下培育。在培育1小时之后,将IPTG以1mM的浓度添加到培养物中。当OD600达到约0.6时,将细胞用20mL、10mL和5mL 10%冰冷甘油洗涤3次,并且最终再悬浮于300μL 10%冰冷甘油中,并且以每管100μL分离到1.5mL离心管。
5.4.4pCBT003_sgRNA质粒和供体基因盒的电穿孔
含有由整合位点的LHA和RHA侧接的目标基因的供体盒的约2μg PCR产物,和表达整合位点的sgRNA的100到200ng pCBT003_sgRNA被转化到如章节5.4.2中所描述的EcN/pCBT001的电转感受态细胞中。在30℃下在1mL SOC中2小时回收之后,将所有细胞涂铺于补充有50μg/mL壮观霉素、链霉素和100μg/mL氨苄西林(ampicillin)的LB琼脂板。
5.5目标基因整合的验证
用于验证的引物设计在LHA的上游和RHA的下游。选取章节5.4.4的板上生长的单一菌落,且使用验证引物基于PCR产物的大小选择具有目标基因整合的恰当克隆。
5.6pCBT003_sgRNA质粒的去除
所选择的恰当克隆在30℃下在震荡(220rpm)下在补充有50μg/mL壮观霉素、链霉素和10mM阿拉伯糖的LB液体培养基中培养过夜,随后将培养物稀释106倍并且将100μL涂铺于补充有50μg/mL壮观霉素和链霉素的LB琼脂板上。随后在补充有50μg/mL壮观霉素、链霉素和100μg/mL氨苄西林的LB琼脂板和补充有50μg/mL壮观霉素和链霉素的LB琼脂板上挑选并点样单一菌落。仅生长在补充有50μg/mL壮观霉素和链霉素的LB琼脂板上的菌落是pCBT003_sgRNA质粒消除的菌落。
5.7pCBT001质粒的去除
将来自章节5.6的pCBT003_sgRNA消除的克隆在LB液体培养基中在42℃下培育过夜,随后将培养物稀释10
5.8膜蛋白的敲除方案
设计靶向敲除目标膜蛋白sgRNA的方法以及sgRNA质粒的构建方法同前所述。选择目标膜蛋白编码基因上游和下游300-500bp的序列作为左同源臂(LHA)和右同源臂(RHA)。所选敲除位点的LHA和RHA以EcN基因组为模板进行扩增。用于扩增这些片段的PCR引物彼此具有15-20bp的同源序列,以便通过重叠PCR相互连接,所得到的包含LHA-RHA的PCR产物用作供体片段。将获得的供体片段PCR产物以及表达敲除位点sgRNA的pCBT003_sgRNA质粒同时转化入感受态细胞中,得到的单菌落利用敲除位点的验证引物进行扩增,通过扩增条带的大小筛选目标蛋白成功从基因组敲除的克隆。
实施例中所敲除的膜蛋白包括tolQ、tolR、tolA、pal、lpp、mrcA和ompT。tolQ敲除位点的sgRNA序列如SEQ ID NO:166所示,tolQ敲除位点两侧同源臂的序列分别如SEQ IDNO:167和168所示;tolR敲除位点的sgRNA序列如SEQ ID NO:169所示,tolR敲除位点两侧同源臂的序列分别如SEQ ID NO:170和171所示;tolA敲除位点的sgRNA的序列如SEQ ID NO:172所示,tolA敲除位点两侧同源臂的序列分别如SEQ ID NO:173和174所示;pal敲除位点的sgRNA的序列如SEQ ID NO:175所示,pal敲除位点两侧同源臂的序列分别如SEQ ID NO:176和177所示;lpp敲除位点的sgRNA序列如SEQ ID NO:178所示,lpp敲除位点两侧同源臂的序列分别如SEQ ID NO:179和180所示;mrcA敲除位点的sgRNA序列如SEQ ID NO:181所示,mrcA敲除位点两侧同源臂的序列分别如SEQ ID NO:182和183所示;ompT敲除位点的sgRNA序列如SEQ ID NO:184所示,ompT敲除位点两侧同源臂的序列分别如SEQ ID NO:185和186所示。
靶向tolQ位点的sgRNA序列(SEQ ID NO:166):
gggaaacgggataatctgag
tolQ位点的左侧同源臂(LHA)序列(SEQ ID NO:167):
gtgaatacaacgctgtttcgatggccggttcgtgtctactatgaagataccgatgccggtggtgtggtgtaccacgccagttacgtcgctttttatgaaagagcacgcacagagatgctgcgtcatcatcatttcagtcaacaggcgctgatggctgaacgcgttgcctttgtggtacgtaaaatgacggtggaatattacgcacctgcgcggctcgacgatatgctcgaaatacagactgaaataacatcaatgcgtggcacctctttggttttcacgcaacgtattgtcaacgccgagaatactttgttgaatgaagcagaggttctggttgtttgcgttgacccactcaaaatgaagcctcgtgcgcttcccaagtctattgtcgcggagtttaagcagtga
tolQ位点的右侧同源臂(RHA)序列(SEQ ID NO:168):
gccatggccagagcgcgtggacgaggtcgtcgcgatctcaagtccgaaatcaacattgtaccgttgctggacgtactgctggtgctgttgctgatctttatggcgacagcgcccatcatcacccagagcgtggaggtcgatctgccagacgctactgaatcacaggcggtgagcagtaacgataatccgccagtgattgttgaagtgtctggtattggtcagtacaccgtggtggttgagaaagatcgtctggagcgtttaccaccagagcaggtggtggcggaagtgtccagccgtttcaaggccaacccgaaaacggtctttctgatcggtggcgcaaaagatgtgccttacgatgaaataattaaagcactgaacttgttacatagtgcgggtgtgaaatcggttggtttaatgacgcagcctatctaaacatctgcgtttcccttgcttgaaagagagcgggtaacaggcgaacagtttttggaaaccgaga
靶向tolR位点的sgRNA序列(SEQ ID NO:169):
tggtattggtcagtacaccg
tolR位点的左侧同源臂(LHA)序列(SEQ ID NO:170):
gcatcgtgccaatagccatgcgccggaagccgtagtggaaggggcgtcgcgtgcgatgcgtatttccatgaatcgtgaacttgaaaatctggaaacgcacattcctttcctcggtacggttggctccatcagcccgtatattggtctgtttggtacggtctgggggatcatgcacgcctttatcgccctcggggcggtaaaacaagcaacactgcaaatggttgcgcccggtatcgcagaagcgttgattgcgacggcaattggtttgttcgccgcaattccggcggtaatggcttataaccgcctcaaccagcgcgtaaacaaactggaactgaattacgacaactttatggaagagtttaccgcgattctgcaccgccaggcgtttaccgttagcgagagcaacaaggggtaa
tolR位点的右侧同源臂(RHA)序列(SEQ ID NO:171):
acatctgcgtttcccttgcttgaaagagagcgggtaacaggcgaacagtttttggaaaccgagagtgtcaaaggcaaccgaacaaaacgacaagctcaagcgggcgataattatttcagcagtgctgcatgtcatcttatttgcggcgctgatctggagttcgttcgatgagaatatagaagcttcagccggaggcggcggtggttcgtccatcgacgctgtcatggttgattcaggtgcggtagttgaacagtacaaacgtatgcaaagccaggaatcaagcgcgaagcgttctgatgagcagcgcaagatgaaggagcagcaggctgctgaagaactgcgtgagaaacaagcggctgaacaggaacgcctgaagcaacttgagaaagagcggttagcggctcaggagcagaaaaagcaggctgaagaagccgcaaaacaggccgagttaaagcagaagcaagcggaagaggc
靶向tolA位点的sgRNA序列(SEQ ID NO:172):
agaactgcgtgagaaacaag
tolA位点的左侧同源臂(LHA)序列(SEQ ID NO:173):
gccatggccagagcgcgtggacgaggtcgtcgcgatctcaagtccgaaatcaacattgtaccgttgctggacgtactgctggtgctgttgctgatctttatggcgacagcgcccatcatcacccagagcgtggaggtcgatctgccagacgctactgaatcacaggcggtgagcagtaacgataatccgccagtgattgttgaagtgtctggtattggtcagtacaccgtggtggttgagaaagatcgtctggagcgtttaccaccagagcaggtggtggcggaagtgtccagccgtttcaaggccaacccgaaaacggtctttctgatcggtggcgcaaaagatgtgccttacgatgaaataattaaagcactgaacttgttacatagtgcgggtgtgaaatcggttggtttaatgacgcagcctatctaaacatctgcgtttcccttgcttgaaagagagcgggtaacaggcgaacagtttttggaaaccgaga
tolA位点的右侧同源臂(RHA)序列(SEQ ID NO:174):
tcgcgatgttgactgttccgacggtcaacatcaggcaccggttgccacggggttctggtagttttgtgtattttagtttgttaacattctgctaaattatcgtgggccatcggtccagataagggagatatgatgaagcaggcattacgagtagcatttggttttctcatactgtgggcatcagttctgcatgctgaagtccgcattgtgatcgacagcggtgtagattccggtcgtcctattggtgttgttcctttccagtgggcggggcctggtgcggcacctgaagatattggcggcatcgttgctgctgacttgcgtaacagcggtaaatttaatccgttagatcgtgctcgtttgccacagcagccgggtagtgcgcaggaagtacaaccagctgcatggtccgcgctgggcattgacgctgtggttgtcggtcaggtcactccgaatccggatggttcttacaatgttgcttatcaacttgttgacactggcgg
靶向pal位点的sgRNA序列(SEQ ID NO:175):
agtcaccgtagaaggtcacg
pal位点的左侧同源臂(LHA)序列(SEQ ID NO:176):
tggtcgcagtaacaataccgaaccgacctggttcccggacagccagaacctggcatttacttctgaccaggccggtcgtccgcaggtttataaagtgaatatcaacggcggtgcgccacaacgtattacctgggaaggttcgcagaaccaggatgcggatgtcagcagcgacggtaaatttatggtaatggtcagctccaacggtgggcagcagcacattgccaaacaagatctggcaacgggaggcgtacaagttctgtcgtccacgttcctggatgaaacgccaagtctggcacctaacggcactatggtaatctacagctcttctcaggggatgggatccgtgctgaatttggtttctacagatgggcgtttcaaagcgcgtcttccggcaactgatggacaggtcaaattccctgcctggtcgccgtatctgtgataataattaattgaatagtaaaggaatcattgaa
pal位点的右侧同源臂(RHA)序列(SEQ ID NO:177):
gagaattgcatgagcagtaacttcagacatcaactattgagtctgtcgttactggttggtatagcggccccctgggccgcttttgctcaggcaccaatcagtagtgtcggctcaggctcggtcgaagaccgcgtcactcaacttgagcgtatttctaatgctcacagccagcttttaacccaactccagcaacaactctctgataatcaatccgatattgattccctgcgtggtcagattcaggaaaatcagtatcaactgaatcaggtcgtggagcggcaaaagcagatcctgttgcagatcgacagcctcagcagcggtggtgcagcggcgcaatcaaccagcggcgatcaaagcggtgtggcggcatcaacgacgccgacagctgatgctggtactgcgaatgctggcgcgccggtgaaaagcggtgatgcaaacacggattacaatgcagctattgcgctggtgcag
靶向lpp位点的sgRNA序列(SEQ ID NO:178):
acgttcaggctgctaaagat
lpp位点的左侧同源臂(LHA)序列(SEQ ID NO:179):
aatacttcccggatgccaccatcctggcactgaccaccaacgaaaaaacggctcaccagttagtactgagcaaaggtgttgtgccgcagctggttaaagagatcacatctactgatgacttctaccgtctgggtaaagaactggctctgcagagcggtctggcacacaaaggtgatgtcgtagttatggtttctggtgcactggtaccgagcggcactactaacaccgcatctgttcacgtcctgtaatattgcttttgtgaattaatttgtatatcgaagcgccctgatgggcgctttttttatttaatcgataaccagaagcaataaaaaatcaaatcggatttcactatataatctcactttatctaagatgaatccgatggaagcatcctgttttctctcaatttttttatctaaaacccagcgttcgatgcttctttgagcgaacgatcaaaaataagtgccttcccatcaaaaaaa
lpp位点的右侧同源臂(RHA)序列(SEQ ID NO:180):
cctgtgaagtgaaaaatggcgcacattgtgcgccatttttttgcctgctatttaccgctactgcgtcgcgcgtaacatattcccttgctctggttccccattctgcgctgactctactgaaggcgcattgctggctgcgggagttgctccactgctcaccgcaaccggataccctgcccgacgatacaacgctttatcgactaacttctgatctacagccttattgtctttaaattgcgtaaagcctgctggcagcgtgtacggcattgtctgaacgttctgctgttcttctgccgatagtggtcgatgtacttcaacataacgcatcccgttaggttccacggaatatttcaccggttcgttgatcactttcaccggtgttcccgtccgcacgctggagaataaagctttaatatccggtgcattcatgcgaataca
靶向mrcA位点的sgRNA序列(SEQ ID NO:181):
caaaccgttcctctacaccg
mrcA位点的左侧同源臂(LHA)序列(SEQ ID NO:182):
gacagccaggccatttgctcccgctcaccgagggacatcgacgggcgtggaaatgaccgctgtaatgtgcgactggcgggaaacgccaacataatgtgatgacgctgcggcagttcacgactccacggtaacaacgtgttagccagccgctgcacatcaacaatccgcccatctttgataatgtcgttctccagtggcaaccgccaccagcggtgcaataagcattcttttgcgctccgcacgatagcaaccgctaccgcttcttgctgttgtaaatgcaaaccaatttgccagttcttaaatgccattgtgatgatctccttatcacccgtcactctgacgggtatatcaatgcgtctggcttgcctttatactaccgcgcgtttgtttataaactgcccaaatgaaactaaatgg
mrcA位点的右侧同源臂(RHA)序列(SEQ ID NO:183):
ttaaaaaggcgcttcggcgcctttttcagtttgctgacaaagtgcacttgtttatgccagatgcgggtgaacgcgttatccggccaacaaaaaattgaaaattcaataaattgcaggaacttcgtaagcctgataagcgttgtgcatcaggcaaacttcacgcatttacactcgcccctgccctttcaaccattcgcgcacgaggaacagcgcgctgacattacgcgcttcgttgaagtcaggggcttccagcaaatccatcatatgcgccagcggccagcgcacctgtggtagcggctctggctcatcgccttccagtgattccgggtagagatcttgcgctaccacgatattcattttgctggaaaagtaagacggtgccatgctgagcttcttcaaaaaagtcagatcgttcgcaccaaatccaacctcttcttttagctcgcggttagcggcttca
靶向ompT位点的sgRNA序列(SEQ ID NO:184):
cctgggactcatggccggat
ompT位点的左侧同源臂(LHA)序列(SEQ ID NO:185):
cccagcccttgtcgatgccgcgttcaggctagcttcgttcaaatccattaaagatatcagactactctcaggtacgtgagtaaggtaaaacccagttccaacgccaatatccagatggttgttacctaaatgttccagaaagtgtggaagaaggtgttcctttgtaggacatccccatgcaagccgatttgatactcccaaaacccaccagtcataaagctttagggtaagtggtgtgtaaattttagccccatcatctgtgttttttttcattagtttcaccgtgttatagttttatttgtgaattaaatcaattatagagatgaattacaaggggttaaatgatgccacggcataacgaatttgaatatattgggtcttgtgttgcggtaagaggttacacgccaggacgcaggttaaatgcttacaatattaggggatattgtttgcttttaacggtaaacaaattccccggggctatacaataccaccggggagaaaatctatttaacgttg
ompT位点的右侧同源臂(RHA)序列(SEQ ID NO:186):
aaaaggtctccattcaatcgttttaatgattgagtatgtattttttatatctaacttaatgagtcaattgtatattgccccactgtttatattttgtttaacattgaatttttgtcacaatgcgctgtcagttttttagtttaattgtctgtttgtgttttgtattgtggttttatgggctaattatttttttctttgtggagtttttatctattcaagtgccatgcctttagatgcatattagcgataatagcatgaggtttatcctcaattgctgtgttttttagtataaaaaagaggaacaaaactgagacacataaggcctcgcaatggcttgcaaggttttacatattttgaggtggtggaacgtgtgaacgcagagatggcgcggtaatttgttgatttaaaatgtcgttcttggagtttctaagtcgtgggctacaggttcgaatcctgcagggcgagccatttcctcgccatttatttgttcccttttacaaattacaccatcactttgttgtcgcggttttgctaaataattggaacacgtcagacgaataaaatgaaaacagaaattctgctaaacaattcatcttgccaggattttgattaggaaagtgaaa
实施例6.编码和分泌Amuc_1100(31-317)的经基因工程改造的细菌的产生
Amuc_1100(31-317)基因盒通过CRISPR-Cas9整合到EcN染色体中的位点agaI/rsmI中。
6.1构建Amuc_1100表达盒
(1)Amuc_1100表达盒的PCR扩增
Amuc_1100编码序列、信号肽、启动子、核糖体结合位点(RBS)以及顺反子等由金斯瑞(GeneScript)在克隆质粒pUC57上合成。Amuc_1100编码序列、信号肽、启动子、核糖体结合位点(RBS)以及顺反子等以合成质粒为模板进行扩增(pUC57_Amuc_1100如图7所示),插入位点的LHA和RHA以EcN基因组为模板扩增,转录终止子以质粒pCBT003为模板扩增。用于扩增这些片段的PCR引物彼此具有15-20bp的同源序列,因此它们可以通过重叠PCR连接起来,从而得到两侧分别与LHA和RHA序列连接的供体基因片段表达盒,表达盒上各元件从5’到3’方向按如下顺序排列:5’-启动子-核糖体结合位点(RBS)-顺反子-信号肽-Amuc_1100编码序列-终止子。Amuc_1100编码序列为编码野生型Amuc_1100(SEQ ID NO:1,在表5中标注为WT)或Amuc_1100的Y289A突变体(第289位的编号是基于SEQ ID NO:1所示序列的编号,在表5中标注为Y289A)的序列。
利用PCR扩增目标基因,随后利用在5.4.1部分中记载的类似方法制备电转感受态细胞。随后,利用在5.4.2部分中记载的类似方法对电转感受态细胞进行电转。电转后,利用如下步骤进行回收。电转后立即向细胞中加入1mL SOC,在30℃,225rpm的条件下下培育1-2小时,随后将细胞接种到添加抗生素的LB琼脂平板上,30℃过夜培养。
(2)Amuc_1100表达盒的基因组整合
利用CRISPR-Cas9技术(图9)将Amuc_1100表达盒的PCR片段整合到EcN的选定位点。简要地,将Amuc_1100表达盒的PCR片段以及pCBT003_agaI/rsmI_sgRNA、pCBT003_malP/T-sgRNA、pCBT003_pflB-sgRNA或者pCBT003_lldD-sgRNA质粒(pCBT003_agaI/rsmI_sgRNA作为代表展示在图10中,表示为ZL-003_agaI/rsmI_sgRNA)电转入包含了pCBT001(ZL-001)的EcN菌株中。利用PCR验证各Amuc_1100表达盒的成功整合。
为了获得表达多拷贝Amuc_1100的菌株,两个、三个或者更多表达盒被整合入EcN基因组。当插入单拷贝的Amuc_1100时,其整合位点为EcN基因组的agaI/rsmI位点;当插入双拷贝的Amuc_1100时,其第一和第二整合位点分别为EcN基因组的agaI/rsmI位点和malP/T位点;当插入三个拷贝的Amuc_1100时,其第一、第二和第三整合位点分别为EcN基因组的agaI/rsmI、malP/T和pflB位点。
靶向agaI/rsmI位点的sgRNA的序列如SEQ ID NO:200所示,该agaI/rsmI位点两侧同源臂的序列分别如SEQ ID NO:201和202所示。靶向malP/T位点的sgRNA序列如SEQ IDNO:203所示,该malP/T位点两侧同源臂的序列分别如SEQ ID NO:204和205所示。靶向pflB位点的sgRNA序列如SEQ ID NO:206所示,该pflB位点两侧同源臂的序列分别如SEQ ID NO:207和208所示。靶向lldD位点的sgRNA序列如SEQ ID NO:209所示,该lldD位点两侧同源臂的序列分别如SEQ ID NO:210和211所示。靶向maeA位点的sgRNA序列如SEQ ID NO:212所示,该maeA位点两侧同源臂的序列分别如SEQ ID NO:213和214所示。
靶向agaI/rsmI位点的sgRNA序列(SEQ ID NO:200):
tgtgtcgtcgttaactcgcc
agaI/rsmI位点的左侧同源臂(LHA)序列(SEQ ID NO:201):
gccgcgcacgctgattttttatgaatctacccaccgtctgttagatagcctggaagatatcgttgcggtattaggcgaatcccgttacgtggttctggcgcgtgagctaaccaaaacctgggaaaccattcacggcgcgcccgttggcgagctgctggcgtgggtaaaagaagatgaaaaccgtcgcaaaggcgaaatggtgctgattgtcgaaggccataaagcgcaggaaaatgacttgcctgctgatgccctgcgcacgctggcgctgctacaggcagaactgccgctgaaaaaagcggcggcgctggccgcagaaattcacggcgtgaagaaaaatgcgctgtataagtatgcgctggagcagcagggagagtgattacaggcgtcgcagcaggaatagcgctac
agaI/rsmI位点的右侧同源臂(RHA)序列(SEQ ID NO:202):
tcacgcatgtgaaatggttatgcagccataaaaatgaagccgggatagcggtggagactttagccgtgagaaaacgttctgtcgcatcctgttttccttcgccagtcaccagtaacaaaacttcgcgggcattgagaatatccttcaggcctaaggtgatcccacgagtcacgggacggtccgcggtttttaacatctcatgttgctgtgttctggcatcaagttgactgatatggcaggctggttgcaggctttctcccggttcgttcagcccaagatgaccgtttttccccaatccgagaacgcataaatccagaccgcctttgcgcgcaatcaggttcgttacccgttcgcactctgtctcatttatctcttcggagcgaaagctgatgagctggtcttcac
靶向malP/T位点的sgRNA序列(SEQ ID NO:203):
cgctttttgaaaatacgcaa
malP/T位点的左侧同源臂(LHA)序列(SEQ ID NO:204):
accagccgagattcaacaggttgttgcccgtcaggcgaccaatcaaaaactccattgagatgtagttaacatgtcgctgattcgccactggcttggcgaatggctgagcacgcagcatttcggccagtgcttcgctcactgccagccaccactggcgaggagtcatttcagccgcagaatttaagccataacgctgccactgacgtgaaagcgcttcctgaaattgcttatcgttaaaaataggttgtgacataggagttccacttttcttagattttcaacacaacgttatcgctagtttgccaggctcgatgttgaccttcctcatcctgcgggggattaggcagggaggagttgcggggatgagcaaggaaatgtgatctcgaccacttaaagctagtgcaatccacaggattagcagcaaatcaatgccataccgcgcagaaaatctgtatctaagtgcaaaaaatggccgtt
malP/T位点的右侧同源臂(RHA)序列(SEQ ID NO:205):
cgtattttcaaaaagcggaaggtaactctataaattaagtaaaggagtgaaacagtttcacaagtaaaatatcaagtgtgctccatctcattcttaatagatttattaagatcatctttttagatggcactttcatcagaaatgaagaagaaacccttgcttaaatgaaactgatgaacataaggaaaataccgtacaacccagtattcacgctggatcagcgtcgttttaggtgagttgttaataaacatttggaattgtgacacagtgcaaattcagacacataaaaaaacgtcatcgcttgcattagaaaggtttctggccgaccttataaccattaattacgaagcgcaaaaaaaataatatttcctcattttccacagtgaagtgattaactatgctgattccgtcaaaattaagtcgtccggttcgactcgaccataccgtggttcgtga
靶向pflB位点的sgRNA序列(SEQ ID NO:206):
ccgtgacgttatccgcacca
pflB位点的左侧同源臂(LHA)序列(SEQ ID NO:207):
acgatgacctgatgcgtccggacttcaacaacgatgactacgctattgcttgctgcgtaagcccgatgatcgttggtaaacaaatgcagttcttcggtgcgcgtgcaaacctggcgaaaaccatgctgtacgcaatcaacggcggcgttgacgaaaaactgaaaatgcaggttggtccgaagtctgaaccgatcaaaggcgatgtcctgaactatgatgaagtgatggagcgcatggatcacttcatggactggctggctaaacagtacatcactgcactgaacatcatccactacatgcacgacaagtacagctacgaagcctctctgatggcgctgcacgaccgtgacgttatcc
pflB位点的右侧同源臂(RHA)序列(SEQ ID NO:208):
gtgacgctatcccgactcagtctgttctgaccatcacttctaacgttgtgtatggtaagaaaactggtaacaccccagacggtcgtcgtgctggcgcgccgttcggaccgggtgctaacccgatgcacggtcgtgaccagaaaggtgctgtagcgtctctgacttccgttgctaaactgccgtttgcttacgctaaagatggtatctcctacaccttctctatcgttccgaacgcactgggtaaagacgacgaagttcgtaagaccaacctggctggtctgatggatggttacttccaccacgaagcatccatcgaaggtggtcagcacctgaacgttaacgtgatgaaccgtgaaatgctgctcgacgcgatggaaaacccggaaaaatatccgcagctgaccatccgtgtatctggctacgcagtacgtttcaactcgct
靶向lldD位点的sgRNA序列(SEQ ID NO:209):
gttatcgtgatgcgcattct
lldD位点的左侧同源臂(LHA)序列(SEQ ID NO:210):
ttccgcagccagcgattatcgcgccgcagcgcaacgcattctgccgccgttcctgttccactatatggatgggggggcatattctgaatacacgctgcgccgcaacgtggaagatttgtcagaagtggcgctgcgccagcgtattctgaaaaacatgtctgacttaagcctggaaacgacgctgtttaatgagaaattgtcgatgccggtggcgctaggtccggtaggtttgtgtggcatgtatgcgcgacgcggcgaagttcaggctgccaaagcagcagatgcgcatggcattccgtttactctctcgacggtttccgtttgcccgattgaagaagtggctccggctatcaaacgtccgatgtggttccagctttatgtgctgcgcgatcgcggctttatgcgtaacgccctggagcgagcaaaagccgcgggttgttcgacgctggttttcacc
lldD位点的右侧同源臂(RHA)序列(SEQ ID NO:211):
atccgcgatttctgggatggcccgatggtgatcaaagggatcctcgatccggaagatgcgcgcgatgcagtacgttttggtgctgatggaattgtggtttctaaccacggtggccgccagttagatggcgtactctcttctgctcgtgcactgcctgctattgcggatgcggtgaaaggtgatatcgccattctggcggatagcggaatacgtaacgggcttgatgtcgtgcgtatgattgcgctcggtgccgacaccgtactgctgggtcgtgctttcctgtatgcactggcaacagcgggccaggcgggtgtagctaatctgctaaatctgatcgaaaaagagatgaaagtggcgatgacgctgactggcgcgaaatcgattagcgaaattacgcaagattcgctggtgcaggggctgggtaaagagttgcctgcggca
靶向maeA位点的sgRNA序列(SEQ ID NO:212):
taacacccagcccgatgccc
maeA位点的左侧同源臂(LHA)序列(SEQ ID NO:213):
ttctgcatagcaggtgaggcaaatgagatttattcgccactacccagtatggatgagatctgaaaaagggagaggaaaattgcccggtagccttcactaccgggcgcaggcttagatggaggtacggcggtagtcgcggtattcggcgtgccagaaattatcatcaatggcctgttgcagagcttcggcagacgttttcaccgccacgccttgctgctgcgccattttgccaaccgcaaaggcaattgcgcgggagactttctgaatatctttcagttccggcagtaccagaccttcgccgttcaggaccagcggcgaatactgagcaagcatttcacttgccgacatcagcatctc
maeA位点的右侧同源臂(RHA)序列(SEQ ID NO:214):
gagattttcgcgcttctgcaccagtttggtctggaaaggcagcaggttcggcatcttgtcggtcagcaggccaaagcgatcgaccataaagactttctgccgcgccgcttcctcgcttaatccttcgcgctgggtctgggcgatgatcatttcggcaatgccgcatcccgctgaacctgcgccaaggaagacgatttttttctcgcttaactgaccacctgccgcgcggctggctgcgatcagtgtgccgactgttaccgccgcggtgccctgaatgtcatcgttaaaagaacaaatttcattgcgatagcggttaagtaacggcatcgcatttttttgtgcgaagtcttcaaactgcaacagtacgtccggccagcgttgtttcacagcctggataaattcatcaacaaattcatagtattcgtcgtcagtgatacgcggattacgccagcccatatacagcggatcgttaagcagctgttggttgttggtgccgacatccagcaccaccggaagggtataggccggactgatgccgccacag
表5总结了实施例中的基因工程菌株。
表5表达Amuc1100的菌株
/>
/>
6.2Amuc_1100(31-317)表达盒的优化
本实施例所构建的工程菌株进入在受试者体内,例如肠道后,不仅需要保持菌株活力,还要有效的表达Amuc_1100并且将Amuc_1100蛋白分泌到胞外。这需要对底盘菌在基因工程方面进行大量的优化试验,从而获得能够在不影响细菌自身活力的情况下表达并分泌Amuc_1100蛋白的工程菌。然而目前对于工程菌特别是基因工程大肠杆菌治疗蛋白的表达和分泌的研究和报道非常有限,因此本实施例进一步开展了Amuc_1100蛋白表达和分泌的测试。
6.2.1信号肽
(1)选取了pelB(来自胡萝卜软腐果胶杆菌)、dsbA(来自大肠杆菌)、ompA(来自大肠杆菌)、Cel-CD(芽孢杆菌纤维素酶的催化结构域)、Amuc1100(来自阿克曼氏菌)以及usp45(来自乳酸乳球菌)的信号肽,构建能够表达分泌Amuc_1100蛋白的表达盒:Pfnrs-信号肽-Amuc_1100蛋白-rrnB_T1_T7Te_终止子,将表达盒连接入质粒载体pCBT010(SEQ IDNO:151)中。(2)将构建的质粒电转入EcN△lpp中。(3)将过夜培养菌液以1%的比例接种于50mL LB培养基,37℃220rpm培养6h,7500rpm离心分别收集培养液上清和沉淀。(4)WesternBlot测定包含不同信号肽的菌株的Amuc_1100表达分泌量。Western Blot制样方法为:在40μL上清中加入10μL上样缓冲液,上样量10μL。
pCBT010的核苷酸序列(SEQ ID NO:151):
gaattccggatgagcattcatcaggcgggcaagaatgtgaataaaggccggataaaacttgtgcttatttttctttacggtctttaaaaaggccgtaatatccagctgaacggtctggttataggtacattgagcaactgactgaaatgcctcaaaatgttctttacgatgccattgggatatatcaacggtggtatatccagtgatttttttctccattttagcttccttagctcctgaaaatctcgataactcaaaaaatacgcccggtagtgatcttatttcattatggtgaaagttggaacctcttacgtgccgatcaacgtctcattttcgccaaaagttggcccagggcttcccggtatcaacagggacaccaggatttatttattctgcgaagtgatcttccgtcacaggtatttattcgggatcctcgctcactgactcgctacgctcggtcgttcgactgcggcgagcggaaatggcttacgaacggggcggagatttcctggaagatgccaggaagatacttaacagggaagtgagagggccgcggcaaagccgtttttccataggctccgcccccctgacaagcatcacgaaatctgacgctcaaatcagtggtggcgaaacccgacaggactataaagataccaggcgtttccccctggcggctccctcgtgcgctctcctgttcctgcctttcggtttaccggtgtcattccgctgttatggccgcgtttgtctcattccacgcctgacactcagttccgggtaggcagttcgctccaagctggactgtatgcacgaaccccccgttcagtccgaccgctgcgccttatccggtaactatcgtcttgagtccaacccggaaagacatgcaaaagcaccactggcagcagccactggtaattgatttagaggagttagtcttgaagtcatgcgccggttaaggctaaactgaaaggacaagttttggtgactgcgctcctccaagccagttacctcggttcaaagagttggtagctcagagaaccttcgaaaaaccgccctgcaaggcggttttttcgttttcagagcaagagattacgcgcagaccaaaacgatctcaagaagatcatctgtaggtcgttcgctccaagctggaaggatcttcacctagatccttttaaattaaaaatgaagttttaaatcaatctaaagtatatatgagtaaacttggtctgacagaatttctgccattcatccgcttattatcacttattcaggcgtagcaaccaggcgtttaagggcaccaataactgccttaaaaaaattacgccccgccctgccactcatcgcagtactgttgtaattcattaagcattctgccgacatggaagccatcacaaacggcatgatgaacctgaatcgccagcggcatcagcaccttgtcgccttgcgtataatatttgcccatggtgaaaacgggggcgaagaagttgtccatattggccacgtttaaatcaaaactggtgaaactcacccagggattggctgagacgaaaaacatattctcaataaaccctttagggaaataggccaggttttcaccgtaacacgccacatcttgcgaatatatgtgtagaaactgccggaaatcgtcgtggtattcactccagagcgatgaaaacgtttcagtttgctcatggaaaacggtgtaacaagggtgaacactatcccatatcaccagctcaccgtctttcattgccatacg
结果如图12B和表6所示,相比Amuc_1100原有的信号肽、大肠杆菌信号肽DsbA和ompA、芽孢杆菌的Cel-CD信号肽以及胡萝卜软腐果胶杆菌的pelB,使用Usp45信号肽能够显著增加Amuc_1100的分泌量,这个结果是难以预料的,因为Usp45信号肽一直被认为仅仅在乳酸乳球菌中发挥作用,从未考虑和研究过其在大肠杆菌中用于指导蛋白分泌的能力。
为了进一步测试不同的信号肽在基因工程菌中(而非上述转染了表达载体的细菌中)的效率,选择了DsbA、OmpA、PelB和YebF,连同Usp45一起构建了基因组中整合了携带不同信号肽并表达不同于Amuc_1100蛋白的测试蛋白的基因工程菌。在LB中培养24小时后表达的测试蛋白在培养基上清及胞内的产量如表6所示。其中,测试菌株1中使用了dsbA信号肽;测试菌株2中使用了ompA信号肽;测试菌株3中使用了pelB信号肽;测试菌株4中使用了yebF信号肽,测试菌株5中使用了USP45信号肽。在同样的稀释度下,使用OmpA、PelB、YebF信号肽的工程菌样品中测试蛋白的浓度低于检测下限,仅使用DsbA信号肽的工程菌样品中测试蛋白的浓度能够高于检测下限。当使用USP45信号肽时,测试蛋白的产量显著高于使用DsbA信号肽的基因工程菌。
表6使用不同信号肽的基因工程菌以及测试蛋白的产量
*“-”表示低于检测下限
以上结果表明,Usp45信号肽是最适合工程大肠杆菌表达并分泌外源蛋白的信号肽。
6.2.2启动子和拷贝数
首先,测试了组成型启动子BBa_J23101和BBa_J23110及其两拷贝的组合对Amuc_1100(Y289A)蛋白的影响,为此构建菌株如下:(1)CBT1101,包含单拷贝的由BBA_J23101启动子驱动的Amuc_1100(Y289A)表达盒;(2)CBT1102,包含单拷贝的由BBA_J23110启动子驱动的Amuc_1100(Y289A)表达盒;(3)CBT1103,包含两拷贝的由BBA_J23110启动子驱动的Amuc_1100(Y289A)表达盒;(4)CBT1104,包含三拷贝的由BBA_J23101启动子驱动的Amuc_1100(Y289A)表达盒,(5)CBT1105,包含单拷贝的由BBA_J23101启动子驱动的Amuc_1100(Y289A)表达盒和两拷贝的由BBA_J23110启动子驱动的Amuc_1100(Y289A)表达盒;(6)CBT1106,包含三个拷贝的由BBA_J23110启动子驱动的Amuc_1100(Y289A)表达盒。
实验方法:将过夜培养的菌液以1%的比例接种于50mL LB培养基,37℃220rpm培养6h,12000rpm离心分别收集培养液上清和沉淀。Western Blot样品处理方式:取40μL上清,加10μL上样缓冲液,使用WB(上样量10μL)测定Amuc_1100含量,结果如图13A所示,所有菌株均能够成功分泌Amuc_1100蛋白,而包含两拷贝和三拷贝表达盒的菌株的表达量明显更高,此外前述两种启动子的表达强度类似,均可以用于Amuc_1100蛋白的表达。结论:BBA_J23110和BBA_J23101启动子都可用于Amuc_1100表达,并且增加表达盒的拷贝数能够增加Amuc_1100蛋白的表达和分泌量。
考虑到工程菌进入肠道环境后,会处于缺氧的环境,因此进一步测试了使用厌氧型启动子的表达和分泌效果,于是构建了使用单拷贝厌氧型启动子Pfnrs的菌株CBT1107和使用双拷贝厌氧型启动子Pfnrs的CBT1108(具体菌株结构信息见表5)。
实验方法:过夜培养的菌液按1%比例接种两瓶50mL培养基,37℃220rpm培养至OD值0.8。然后一瓶转移至厌氧培养箱。此后4瓶菌液每隔1或2小时取样,12000rpm离心并保存上清和沉淀样品,用于WB检测Amuc_1100含量。Western Blot样品处理方法:取40μL上清,加入10μL上样缓冲液,WB(上样量15μL)测定Amuc_1100含量,结果如图13B和13C所示,对比CBT1107和CBT1108菌株在5h时有氧和无氧条件下的产量(分别与标品20ng荧光强度相对值比较)可以看出,CBT1108菌株的蛋白分泌量大约是CBT1107的两倍。这就说明,厌氧型启动子Pfnrs可用于大肠杆菌中Amuc_1100蛋白的表达,并且增加拷贝数能够提高表达和分泌效果。
接下来,构建了更多包含了不同启动子以及拷贝数组合的菌株,并测试了有氧发酵条件下(模拟体外生产环境)对Amuc_1100蛋白分泌的影响,构建和测试的菌株如下:CBT1117(包含两个组成型启动子,分别为BBa_J23110和BBa_J23101),CBT1108(包含两拷贝Pfnrs启动子),CBT1109(包含两拷贝BBa_J23110启动子+单拷贝Pfnrs启动子)和CBT1110(包含两拷贝BBa_J23110启动子+两拷贝Pfnrs启动子)。
实验过程:分别挑取CBT1117、CBT1108、CBT1109和CBT1110的单菌落至50mL LB培养基中,37℃220rpm培养7.5h,12000rpm离心取上清和沉淀并制备Western Blot样品。Western Blot样品处理方式:取40μL上清,加10μL上样缓冲液,使用WB(上样量5μL)测定Amuc_1100含量,结果如图13D所示。
我们意外的发现,在有氧发酵的情况下(模拟了工业上生产菌株的条件),使用厌氧型启动子Pfnrs在保持菌株分泌Amuc_1100蛋白的活性方面更具有优势(比较CBT1108和CBT1117)。同时,在组成型启动子菌株中再添加一个拷贝的厌氧型启动子(CBT1109),可增加Amuc_1100蛋白的分泌量。然而,如果进一步将厌氧和有氧启动子的拷贝数增至四个(见菌株CBT1110),并未带来更多益处,相反导致表达量急剧降低。
接下来又研究了不同拷贝数的Pfnrs启动子(例如两拷贝、三拷贝和四拷贝)的效果,构建和/或测试的菌株如下:CBT1108(两拷贝),CBT1111(三拷贝)和CBT1112(四拷贝)。实验方法:将CBT1108、CBT1111和CBT1112单菌落添加至50mL已补充1% Glu的LB培养基,37℃220rpm有氧培养2h后,转移到厌氧培养箱继续培养。总培养时间为8h,期间分别在6h和8h取菌液,12000rpm离心分别取上清和沉淀并制备Western Blot样品。Western Blot样品处理方式为:取20μL上清,加入10μL上样缓冲液和20μL PBS缓冲液,WB(上样量5μL)测定Amuc_1100含量。结果如图13E所示,在有氧转厌氧条件下,与包含两拷贝Pfnrs启动子的菌株相比,包含三拷贝和四拷贝Pfnrs启动子的菌株培养上清含有更多的Amuc_1100蛋白。但是进一步分析菌株的生长状况显示,包含三拷贝和四拷贝厌氧启动子的两个菌株在转厌氧培养后不久,其菌液的OD
6.2.3敲除外膜蛋白的影响
1)敲除LPP蛋白
按照实施例部分5.8的方案敲除工程菌中的LPP蛋白。将过夜培养的CBT1103和CBT1113菌液按1%比例接种到50mL LB培养基,37℃220rpm培养7h后12000rpm离心取上清和沉淀并制备Western Blot样品。Western Blot样品处理方式:取40μL上清,加10μL上样缓冲液,WB(上样量10μL)测定Amuc_1100含量。结果如图13G所示,敲除LPP能够有效促进Amuc_1100蛋白的分泌。包含两个拷贝Amuc_1100表达盒同时敲除LPP的菌株CBT1103,甚至比包含三拷贝Amuc_1100表达盒但是LPP完整的菌株CBT1113分泌更多的Amuc_1100蛋白,表明敲除LPP能够有效促进Amuc_1100蛋白的分泌。
2)ompT基因敲除对Amuc_1100蛋白分泌的影响
实验方法:将过夜培养的CBT1114和CBT1115菌液按1%比例接种到50mL LB培养基,37℃220rpm培养7h,随后12000rpm离心取上清和沉淀制备Western Blot样品。WesternBlot样品处理方式:取40μL上清,加10μL上样缓冲液,WB(上样量10μL)测定Amuc_1100含量。
结果如图13H所示,未敲除ompT基因的CBT1114有明显的双带,而敲除了ompT基因的CBT1115双带明显减弱,由此可见,敲除ompT基因能够有效阻止Amuc1100蛋白的降解。
6.2.4分子伴侣
分子伴侣对Amuc_1100蛋白表达的影响。实验方法:将过夜培养的CBT1103和CBT1116菌液按1%比例接种到50mL LB培养基,37℃220rpm培养7h后12000rpm离心取上清和沉淀并制备Western Blot样品。Western Blot样品处理方式:取40μL上清,加15μL上样缓冲液,WB(上样量10μL)测定Amuc_1100含量。
结果如图13I所示,是否使用分子伴侣对Amuc_1100蛋白的分泌不具有显著的影响。
顺反子
(1)构建EcN_agaI/rsmI_J23101_MBP_usp45_Amuc_1100
利用顺反子增加Amuc_1100(31-317)的表达,表2中列出的任何顺反子均可被插入Amuc_1100(31-317)表达盒,如图11所示。用于构建EcN_agaI/rsmI_J23101_MBP_usp45_Amuc_1100的引物列于下面的表7
表7.agaI/rsmI_J23101_MBP_usp45_Amuc_1100引物
Sat分泌系统
构建EcN_agaI/rsmI_J23101_sat_Amuc_1100_βdomain
利用基于如图12A中所示的自转运蛋白结构域(β结构域)的Sat分泌系统增加Amuc_1100(31-317)的表达。用于构建EcN_agaI/rsmI_J23101_sat_Amuc_1100_βdomain的引物列于下面的表8。
表8.agaI/rsmI_J23101_sat_Amuc_1100_βdomain引物
实施例7.从经基因工程改造的EcN分泌的Amuc_1100(31-317)刺激TLR2信号。
培养在实施例6中获得的经基因工程改造的细菌EcN菌株CBT1108,并且在后期指数生长阶段收集培养基。Amuc_1100(31-317)蛋白质通过含his标签抗体的树脂柱从培养基中分离。从分离的Amuc_1100(31-317)蛋白质去除内毒素。
通过TLR2报告基因分析测试Amuc_1100(31-317)活性。具体来说,HEK293/TLR2报告细胞在具有一定量的蛋白质的96孔板中刺激24小时,将报告酶的底物添加到细胞培养基中并且通过比色方法测量活性。
如图17中所示,从经基因工程改造的EcN分泌的Amuc_1100(31-317)刺激TLR2信号。本申请提供的经工程改造的EcN可以显著提高Amuc_1100的活性,例如,刺激TLR2信号的活性。
实施例8.编码Amuc_1100(31-317)(或其突变体)的经基因工程改造的EcN和抗PD-1抗体的组合对抑制乳癌小鼠模型中的肿瘤生长的影响。
本实施例测试的经基因工程改造的EcN菌株是CBT1108。MMTV-PyVT转基因C57BL/6雌性小鼠以及原位EMT-6小鼠用作乳癌动物模型。9.5周的小鼠被随机分成六组,每组4-5只,用于接受药物处理。当肿瘤体积增长至100mm
实施例9.编码Amuc_1100(31-317)(或其突变体)的经基因工程改造的EcN和抗PD-1抗体的组合对抑制乳癌小鼠模型中的肿瘤生长的影响。
本实施例测试的经基因工程菌改造的EcN菌株是CBT1117。C57BL/6雌性小鼠乳腺脂肪垫内移植MMTV-PyMT肿瘤块,建立MMTV-PyMT原位乳腺瘤模型。接种后第13天,肿瘤平均体积约为100mm
实施例10.编码Amuc_1100(31-317)(或其突变体)的经基因工程改造的EcN和抗PD-1抗体的组合对体重变化以及肿瘤中免疫细胞浸润的影响
利用实施例9的相近方法对小鼠进行分组和处理。每只小鼠的体重每周测量两次。在本研究终点,收集肿瘤组织用于接下来分析免疫细胞的分布情况(包括巨噬细胞、Treg细胞、CD4
实施例11.编码Amuc_1100(31-317)(或其突变体)的经基因工程改造的EcN和其它免疫检查点抑制剂(ICI)的组合对抑制乳癌小鼠模型中的肿瘤生长的影响
C57BL/6雌性小鼠乳腺脂肪垫内移植MMTV-PyMT肿瘤块,建立MMTV-PyMT原位乳腺瘤模型。接种后第13天,肿瘤平均体积约为100mm
SEQUENCE LISTING
<110>CommBio Therapeutics Co., Ltd.
<120>COMBINATION THERAPIES OF AMUC_1100 AND IMMUNE CHECKPOINT
MODULATORS FOR USE IN TREATING CANCERS
<130>079065-8001WO02
<150>PCT/CN2020/136426
<151>2020-12-15
<150>202110613387.8
<151>2021-06-02
<160>214
<170>PatentIn version 3.5
<210>1
<211>317
<212>PRT
<213>Artificial Sequence
<220>
<223>synthetic
<400>1
Met Ser Asn Trp Ile Thr Asp Asn Lys Pro Ala Ala Met Val Ala Gly
1 5 1015
Val Gly Leu Leu Leu Phe Leu Gly Leu Ser Ala Thr Gly Tyr Ile Val
202530
Asn Ser Lys Arg Ser Glu Leu Asp Lys Lys Ile Ser Ile Ala Ala Lys
354045
Glu Ile Lys Ser Ala Asn Ala Ala Glu Ile Thr Pro Ser Arg Ser Ser
505560
Asn Glu Glu Leu Glu Lys Glu Leu Asn Arg Tyr Ala Lys Ala Val Gly
65707580
Ser Leu Glu Thr Ala Tyr Lys Pro Phe Leu Ala Ser Ser Ala Leu Val
859095
Pro Thr Thr Pro Thr Ala Phe Gln Asn Glu Leu Lys Thr Phe Arg Asp
100 105 110
Ser Leu Ile Ser Ser Cys Lys Lys Lys Asn Ile Leu Ile Thr Asp Thr
115 120 125
Ser Ser Trp Leu Gly Phe Gln Val Tyr Ser Thr Gln Ala Pro Ser Val
130 135 140
Gln Ala Ala Ser Thr Leu Gly Phe Glu Leu Lys Ala Ile Asn Ser Leu
145 150 155 160
Val Asn Lys Leu Ala Glu Cys Gly Leu Ser Lys Phe Ile Lys Val Tyr
165 170 175
Arg Pro Gln Leu Pro Ile Glu Thr Pro Ala Asn Asn Pro Glu Glu Ser
180 185 190
Asp Glu Ala Asp Gln Ala Pro Trp Thr Pro Met Pro Leu Glu Ile Ala
195 200 205
Phe Gln Gly Asp Arg Glu Ser Val Leu Lys Ala Met Asn Ala Ile Thr
210 215 220
Gly Met Gln Asp Tyr Leu Phe Thr Val Asn Ser Ile Arg Ile Arg Asn
225 230 235 240
Glu Arg Met Met Pro Pro Pro Ile Ala Asn Pro Ala Ala Ala Lys Pro
245 250 255
Ala Ala Ala Gln Pro Ala Thr Gly Ala Ala Ser Leu Thr Pro Ala Asp
260 265 270
Glu Ala Ala Ala Pro Ala Ala Pro Ala Ile Gln Gln Val Ile Lys Pro
275 280 285
Tyr Met Gly Lys Glu Gln Val Phe Val Gln Val Ser Leu Asn Leu Val
290 295 300
His Phe Asn Gln Pro Lys Ala Gln Glu Pro Ser Glu Asp
305 310 315
<210>2
<211>288
<212>PRT
<213>Artificial Sequence
<220>
<223>synthetic
<400>2
Met Ile Val Asn Ser Lys Arg Ser Glu Leu Asp Lys Lys Ile Ser Ile
1 5 1015
Ala Ala Lys Glu Ile Lys Ser Ala Asn Ala Ala Glu Ile Thr Pro Ser
202530
Arg Ser Ser Asn Glu Glu Leu Glu Lys Glu Leu Asn Arg Tyr Ala Lys
354045
Ala Val Gly Ser Leu Glu Thr Ala Tyr Lys Pro Phe Leu Ala Ser Ser
505560
Ala Leu Val Pro Thr Thr Pro Thr Ala Phe Gln Asn Glu Leu Lys Thr
65707580
Phe Arg Asp Ser Leu Ile Ser Ser Cys Lys Lys Lys Asn Ile Leu Ile
859095
Thr Asp Thr Ser Ser Trp Leu Gly Phe Gln Val Tyr Ser Thr Gln Ala
100 105 110
Pro Ser Val Gln Ala Ala Ser Thr Leu Gly Phe Glu Leu Lys Ala Ile
115 120 125
Asn Ser Leu Val Asn Lys Leu Ala Glu Cys Gly Leu Ser Lys Phe Ile
130 135 140
Lys Val Tyr Arg Pro Gln Leu Pro Ile Glu Thr Pro Ala Asn Asn Pro
145 150 155 160
Glu Glu Ser Asp Glu Ala Asp Gln Ala Pro Trp Thr Pro Met Pro Leu
165 170 175
Glu Ile Ala Phe Gln Gly Asp Arg Glu Ser Val Leu Lys Ala Met Asn
180 185 190
Ala Ile Thr Gly Met Gln Asp Tyr Leu Phe Thr Val Asn Ser Ile Arg
195 200 205
Ile Arg Asn Glu Arg Met Met Pro Pro Pro Ile Ala Asn Pro Ala Ala
210 215 220
Ala Lys Pro Ala Ala Ala Gln Pro Ala Thr Gly Ala Ala Ser Leu Thr
225 230 235 240
Pro Ala Asp Glu Ala Ala Ala Pro Ala Ala Pro Ala Ile Gln Gln Val
245 250 255
Ile Lys Pro Tyr Met Gly Lys Glu Gln Val Phe Val Gln Val Ser Leu
260 265 270
Asn Leu Val His Phe Asn Gln Pro Lys Ala Gln Glu Pro Ser Glu Asp
275 280 285
<210>3
<211>238
<212>PRT
<213>Artificial Sequence
<220>
<223>synthetic
<400>3
Met Ser Leu Glu Thr Ala Tyr Lys Pro Phe Leu Ala Ser Ser Ala Leu
1 5 1015
Val Pro Thr Thr Pro Thr Ala Phe Gln Asn Glu Leu Lys Thr Phe Arg
202530
Asp Ser Leu Ile Ser Ser Cys Lys Lys Lys Asn Ile Leu Ile Thr Asp
354045
Thr Ser Ser Trp Leu Gly Phe Gln Val Tyr Ser Thr Gln Ala Pro Ser
505560
Val Gln Ala Ala Ser Thr Leu Gly Phe Glu Leu Lys Ala Ile Asn Ser
65707580
Leu Val Asn Lys Leu Ala Glu Cys Gly Leu Ser Lys Phe Ile Lys Val
859095
Tyr Arg Pro Gln Leu Pro Ile Glu Thr Pro Ala Asn Asn Pro Glu Glu
100 105 110
Ser Asp Glu Ala Asp Gln Ala Pro Trp Thr Pro Met Pro Leu Glu Ile
115 120 125
Ala Phe Gln Gly Asp Arg Glu Ser Val Leu Lys Ala Met Asn Ala Ile
130 135 140
Thr Gly Met Gln Asp Tyr Leu Phe Thr Val Asn Ser Ile Arg Ile Arg
145 150 155 160
Asn Glu Arg Met Met Pro Pro Pro Ile Ala Asn Pro Ala Ala Ala Lys
165 170 175
Pro Ala Ala Ala Gln Pro Ala Thr Gly Ala Ala Ser Leu Thr Pro Ala
180 185 190
Asp Glu Ala Ala Ala Pro Ala Ala Pro Ala Ile Gln Gln Val Ile Lys
195 200 205
Pro Tyr Met Gly Lys Glu Gln Val Phe Val Gln Val Ser Leu Asn Leu
210 215 220
Val His Phe Asn Gln Pro Lys Ala Gln Glu Pro Ser Glu Asp
225 230 235
<210>4
<211>288
<212>PRT
<213>Artificial Sequence
<220>
<223>synthetic
<400>4
Met Ile Val Asn Ser Lys Arg Ser Glu Leu Asp Lys Lys Ile Ser Ile
1 5 1015
Ala Ala Lys Glu Ile Lys Ser Ala Asn Ala Ala Glu Ile Thr Pro Ser
202530
Arg Ser Ser Asn Glu Glu Leu Glu Lys Glu Leu Asn Arg Tyr Ala Lys
354045
Ala Val Gly Ser Leu Glu Thr Ala Tyr Lys Pro Phe Leu Ala Ser Ser
505560
Ala Leu Val Pro Thr Thr Pro Thr Ala Phe Gln Asn Glu Leu Lys Thr
65707580
Phe Arg Asp Ser Leu Ile Ser Ser Cys Lys Lys Lys Asn Ile Leu Ile
859095
Thr Asp Thr Ser Ser Trp Leu Gly Phe Gln Val Tyr Ser Thr Gln Ala
100 105 110
Pro Ser Val Gln Ala Ala Ser Thr Leu Gly Phe Glu Leu Lys Ala Ile
115 120 125
Asn Ser Leu Val Asn Lys Leu Ala Glu Cys Gly Leu Ser Lys Phe Ile
130 135 140
Lys Val Tyr Arg Pro Gln Leu Pro Ile Glu Thr Pro Ala Asn Asn Pro
145 150 155 160
Glu Glu Ser Asp Glu Ala Asp Gln Ala Pro Trp Thr Pro Met Pro Leu
165 170 175
Glu Ile Ala Phe Gln Gly Asp Arg Glu Ser Val Leu Lys Ala Met Asn
180 185 190
Ala Ile Thr Gly Met Gln Asp Tyr Leu Phe Thr Val Asn Ser Ile Arg
195 200 205
Ile Arg Asn Glu Arg Met Met Pro Pro Pro Ile Ala Asn Pro Ala Ala
210 215 220
Ala Lys Pro Ala Ala Ala Gln Pro Ala Thr Gly Ala Ala Ser Leu Thr
225 230 235 240
Pro Ala Asp Glu Ala Ala Ala Pro Ala Ala Pro Ala Ile Gln Gln Val
245 250 255
Ile Lys Pro Ala Met Gly Lys Glu Gln Val Phe Val Gln Val Ser Leu
260 265 270
Asn Leu Val His Phe Asn Gln Pro Lys Ala Gln Glu Pro Ser Glu Asp
275 280 285
<210>5
<211>238
<212>PRT
<213>Artificial Sequence
<220>
<223>synthetic
<400>5
Met Ser Leu Glu Thr Ala Tyr Lys Pro Phe Leu Ala Ser Ser Ala Leu
1 5 1015
Val Pro Thr Thr Pro Thr Ala Phe Gln Asn Glu Leu Lys Thr Phe Arg
202530
Asp Ser Leu Ile Ser Ser Cys Lys Lys Lys Asn Ile Leu Ile Thr Asp
354045
Thr Ser Ser Trp Leu Gly Phe Gln Val Tyr Ser Thr Gln Ala Pro Ser
505560
Val Gln Ala Ala Ser Thr Leu Gly Phe Glu Leu Lys Ala Ile Asn Ser
65707580
Leu Val Asn Lys Leu Ala Glu Cys Gly Leu Ser Lys Phe Ile Lys Val
859095
Tyr Arg Pro Gln Leu Pro Ile Glu Thr Pro Ala Asn Asn Pro Glu Glu
100 105 110
Ser Asp Glu Ala Asp Gln Ala Pro Trp Thr Pro Met Pro Leu Glu Ile
115 120 125
Ala Phe Gln Gly Asp Arg Glu Ser Val Leu Lys Ala Met Asn Ala Ile
130 135 140
Thr Gly Met Gln Asp Tyr Leu Phe Thr Val Asn Ser Ile Arg Ile Arg
145 150 155 160
Asn Glu Arg Met Met Pro Pro Pro Ile Ala Asn Pro Ala Ala Ala Lys
165 170 175
Pro Ala Ala Ala Gln Pro Ala Thr Gly Ala Ala Ser Leu Thr Pro Ala
180 185 190
Asp Glu Ala Ala Ala Pro Ala Ala Pro Ala Ile Gln Gln Val Ile Lys
195 200 205
Pro Ala Met Gly Lys Glu Gln Val Phe Val Gln Val Ser Leu Asn Leu
210 215 220
Val His Phe Asn Gln Pro Lys Ala Gln Glu Pro Ser Glu Asp
225 230 235
<210>6
<211>294
<212>PRT
<213>Artificial Sequence
<220>
<223>synthetic
<400>6
Met Ile Val Asn Ser Lys Arg Ser Glu Leu Asp Lys Lys Ile Ser Ile
1 5 1015
Ala Ala Lys Glu Ile Lys Ser Ala Asn Ala Ala Glu Ile Thr Pro Ser
202530
Arg Ser Ser Asn Glu Glu Leu Glu Lys Glu Leu Asn Arg Tyr Ala Lys
354045
Ala Val Gly Ser Leu Glu Thr Ala Tyr Lys Pro Phe Leu Ala Ser Ser
505560
Ala Leu Val Pro Thr Thr Pro Thr Ala Phe Gln Asn Glu Leu Lys Thr
65707580
Phe Arg Asp Ser Leu Ile Ser Ser Cys Lys Lys Lys Asn Ile Leu Ile
859095
Thr Asp Thr Ser Ser Trp Leu Gly Phe Gln Val Tyr Ser Thr Gln Ala
100 105 110
Pro Ser Val Gln Ala Ala Ser Thr Leu Gly Phe Glu Leu Lys Ala Ile
115 120 125
Asn Ser Leu Val Asn Lys Leu Ala Glu Cys Gly Leu Ser Lys Phe Ile
130 135 140
Lys Val Tyr Arg Pro Gln Leu Pro Ile Glu Thr Pro Ala Asn Asn Pro
145 150 155 160
Glu Glu Ser Asp Glu Ala Asp Gln Ala Pro Trp Thr Pro Met Pro Leu
165 170 175
Glu Ile Ala Phe Gln Gly Asp Arg Glu Ser Val Leu Lys Ala Met Asn
180 185 190
Ala Ile Thr Gly Met Gln Asp Tyr Leu Phe Thr Val Asn Ser Ile Arg
195 200 205
Ile Arg Asn Glu Arg Met Met Pro Pro Pro Ile Ala Asn Pro Ala Ala
210 215 220
Ala Lys Pro Ala Ala Ala Gln Pro Ala Thr Gly Ala Ala Ser Leu Thr
225 230 235 240
Pro Ala Asp Glu Ala Ala Ala Pro Ala Ala Pro Ala Ile Gln Gln Val
245 250 255
Ile Lys Pro Tyr Met Gly Lys Glu Gln Val Phe Val Gln Val Ser Leu
260 265 270
Asn Leu Val His Phe Asn Gln Pro Lys Ala Gln Glu Pro Ser Glu Asp
275 280 285
His His His His His His
290
<210>7
<211>244
<212>PRT
<213>Artificial Sequence
<220>
<223>synthetic
<400>7
Met Ser Leu Glu Thr Ala Tyr Lys Pro Phe Leu Ala Ser Ser Ala Leu
1 5 1015
Val Pro Thr Thr Pro Thr Ala Phe Gln Asn Glu Leu Lys Thr Phe Arg
202530
Asp Ser Leu Ile Ser Ser Cys Lys Lys Lys Asn Ile Leu Ile Thr Asp
354045
Thr Ser Ser Trp Leu Gly Phe Gln Val Tyr Ser Thr Gln Ala Pro Ser
505560
Val Gln Ala Ala Ser Thr Leu Gly Phe Glu Leu Lys Ala Ile Asn Ser
65707580
Leu Val Asn Lys Leu Ala Glu Cys Gly Leu Ser Lys Phe Ile Lys Val
859095
Tyr Arg Pro Gln Leu Pro Ile Glu Thr Pro Ala Asn Asn Pro Glu Glu
100 105 110
Ser Asp Glu Ala Asp Gln Ala Pro Trp Thr Pro Met Pro Leu Glu Ile
115 120 125
Ala Phe Gln Gly Asp Arg Glu Ser Val Leu Lys Ala Met Asn Ala Ile
130 135 140
Thr Gly Met Gln Asp Tyr Leu Phe Thr Val Asn Ser Ile Arg Ile Arg
145 150 155 160
Asn Glu Arg Met Met Pro Pro Pro Ile Ala Asn Pro Ala Ala Ala Lys
165 170 175
Pro Ala Ala Ala Gln Pro Ala Thr Gly Ala Ala Ser Leu Thr Pro Ala
180 185 190
Asp Glu Ala Ala Ala Pro Ala Ala Pro Ala Ile Gln Gln Val Ile Lys
195 200 205
Pro Tyr Met Gly Lys Glu Gln Val Phe Val Gln Val Ser Leu Asn Leu
210 215 220
Val His Phe Asn Gln Pro Lys Ala Gln Glu Pro Ser Glu Asp His His
225 230 235 240
His His His His
<210>8
<211>294
<212>PRT
<213>Artificial Sequence
<220>
<223>synthetic
<400>8
Met Ile Val Asn Ser Lys Arg Ser Glu Leu Asp Lys Lys Ile Ser Ile
1 5 1015
Ala Ala Lys Glu Ile Lys Ser Ala Asn Ala Ala Glu Ile Thr Pro Ser
202530
Arg Ser Ser Asn Glu Glu Leu Glu Lys Glu Leu Asn Arg Tyr Ala Lys
354045
Ala Val Gly Ser Leu Glu Thr Ala Tyr Lys Pro Phe Leu Ala Ser Ser
505560
Ala Leu Val Pro Thr Thr Pro Thr Ala Phe Gln Asn Glu Leu Lys Thr
65707580
Phe Arg Asp Ser Leu Ile Ser Ser Cys Lys Lys Lys Asn Ile Leu Ile
859095
Thr Asp Thr Ser Ser Trp Leu Gly Phe Gln Val Tyr Ser Thr Gln Ala
100 105 110
Pro Ser Val Gln Ala Ala Ser Thr Leu Gly Phe Glu Leu Lys Ala Ile
115 120 125
Asn Ser Leu Val Asn Lys Leu Ala Glu Cys Gly Leu Ser Lys Phe Ile
130 135 140
Lys Val Tyr Arg Pro Gln Leu Pro Ile Glu Thr Pro Ala Asn Asn Pro
145 150 155 160
Glu Glu Ser Asp Glu Ala Asp Gln Ala Pro Trp Thr Pro Met Pro Leu
165 170 175
Glu Ile Ala Phe Gln Gly Asp Arg Glu Ser Val Leu Lys Ala Met Asn
180 185 190
Ala Ile Thr Gly Met Gln Asp Tyr Leu Phe Thr Val Asn Ser Ile Arg
195 200 205
Ile Arg Asn Glu Arg Met Met Pro Pro Pro Ile Ala Asn Pro Ala Ala
210 215 220
Ala Lys Pro Ala Ala Ala Gln Pro Ala Thr Gly Ala Ala Ser Leu Thr
225 230 235 240
Pro Ala Asp Glu Ala Ala Ala Pro Ala Ala Pro Ala Ile Gln Gln Val
245 250 255
Ile Lys Pro Ala Met Gly Lys Glu Gln Val Phe Val Gln Val Ser Leu
260 265 270
Asn Leu Val His Phe Asn Gln Pro Lys Ala Gln Glu Pro Ser Glu Asp
275 280 285
His His His His His His
290
<210>9
<211>244
<212>PRT
<213>Artificial Sequence
<220>
<223>synthetic
<400>9
Met Ser Leu Glu Thr Ala Tyr Lys Pro Phe Leu Ala Ser Ser Ala Leu
1 5 1015
Val Pro Thr Thr Pro Thr Ala Phe Gln Asn Glu Leu Lys Thr Phe Arg
202530
Asp Ser Leu Ile Ser Ser Cys Lys Lys Lys Asn Ile Leu Ile Thr Asp
354045
Thr Ser Ser Trp Leu Gly Phe Gln Val Tyr Ser Thr Gln Ala Pro Ser
505560
Val Gln Ala Ala Ser Thr Leu Gly Phe Glu Leu Lys Ala Ile Asn Ser
65707580
Leu Val Asn Lys Leu Ala Glu Cys Gly Leu Ser Lys Phe Ile Lys Val
859095
Tyr Arg Pro Gln Leu Pro Ile Glu Thr Pro Ala Asn Asn Pro Glu Glu
100 105 110
Ser Asp Glu Ala Asp Gln Ala Pro Trp Thr Pro Met Pro Leu Glu Ile
115 120 125
Ala Phe Gln Gly Asp Arg Glu Ser Val Leu Lys Ala Met Asn Ala Ile
130 135 140
Thr Gly Met Gln Asp Tyr Leu Phe Thr Val Asn Ser Ile Arg Ile Arg
145 150 155 160
Asn Glu Arg Met Met Pro Pro Pro Ile Ala Asn Pro Ala Ala Ala Lys
165 170 175
Pro Ala Ala Ala Gln Pro Ala Thr Gly Ala Ala Ser Leu Thr Pro Ala
180 185 190
Asp Glu Ala Ala Ala Pro Ala Ala Pro Ala Ile Gln Gln Val Ile Lys
195 200 205
Pro Ala Met Gly Lys Glu Gln Val Phe Val Gln Val Ser Leu Asn Leu
210 215 220
Val His Phe Asn Gln Pro Lys Ala Gln Glu Pro Ser Glu Asp His His
225 230 235 240
His His His His
<210>10
<211>299
<212>PRT
<213>Artificial Sequence
<220>
<223>synthetic
<400>10
Met His His His His His His Asp Asp Asp Asp Lys Ile Val Asn Ser
1 5 1015
Lys Arg Ser Glu Leu Asp Lys Lys Ile Ser Ile Ala Ala Lys Glu Ile
202530
Lys Ser Ala Asn Ala Ala Glu Ile Thr Pro Ser Arg Ser Ser Asn Glu
354045
Glu Leu Glu Lys Glu Leu Asn Arg Tyr Ala Lys Ala Val Gly Ser Leu
505560
Glu Thr Ala Tyr Lys Pro Phe Leu Ala Ser Ser Ala Leu Val Pro Thr
65707580
Thr Pro Thr Ala Phe Gln Asn Glu Leu Lys Thr Phe Arg Asp Ser Leu
859095
Ile Ser Ser Cys Lys Lys Lys Asn Ile Leu Ile Thr Asp Thr Ser Ser
100 105 110
Trp Leu Gly Phe Gln Val Tyr Ser Thr Gln Ala Pro Ser Val Gln Ala
115 120 125
Ala Ser Thr Leu Gly Phe Glu Leu Lys Ala Ile Asn Ser Leu Val Asn
130 135 140
Lys Leu Ala Glu Cys Gly Leu Ser Lys Phe Ile Lys Val Tyr Arg Pro
145 150 155 160
Gln Leu Pro Ile Glu Thr Pro Ala Asn Asn Pro Glu Glu Ser Asp Glu
165 170 175
Ala Asp Gln Ala Pro Trp Thr Pro Met Pro Leu Glu Ile Ala Phe Gln
180 185 190
Gly Asp Arg Glu Ser Val Leu Lys Ala Met Asn Ala Ile Thr Gly Met
195 200 205
Gln Asp Tyr Leu Phe Thr Val Asn Ser Ile Arg Ile Arg Asn Glu Arg
210 215 220
Met Met Pro Pro Pro Ile Ala Asn Pro Ala Ala Ala Lys Pro Ala Ala
225 230 235 240
Ala Gln Pro Ala Thr Gly Ala Ala Ser Leu Thr Pro Ala Asp Glu Ala
245 250 255
Ala Ala Pro Ala Ala Pro Ala Ile Gln Gln Val Ile Lys Pro Tyr Met
260 265 270
Gly Lys Glu Gln Val Phe Val Gln Val Ser Leu Asn Leu Val His Phe
275 280 285
Asn Gln Pro Lys Ala Gln Glu Pro Ser Glu Asp
290 295
<210>11
<211>249
<212>PRT
<213>Artificial Sequence
<220>
<223>synthetic
<400>11
Met His His His His His His Asp Asp Asp Asp Lys Ser Leu Glu Thr
1 5 1015
Ala Tyr Lys Pro Phe Leu Ala Ser Ser Ala Leu Val Pro Thr Thr Pro
202530
Thr Ala Phe Gln Asn Glu Leu Lys Thr Phe Arg Asp Ser Leu Ile Ser
354045
Ser Cys Lys Lys Lys Asn Ile Leu Ile Thr Asp Thr Ser Ser Trp Leu
505560
Gly Phe Gln Val Tyr Ser Thr Gln Ala Pro Ser Val Gln Ala Ala Ser
65707580
Thr Leu Gly Phe Glu Leu Lys Ala Ile Asn Ser Leu Val Asn Lys Leu
859095
Ala Glu Cys Gly Leu Ser Lys Phe Ile Lys Val Tyr Arg Pro Gln Leu
100 105 110
Pro Ile Glu Thr Pro Ala Asn Asn Pro Glu Glu Ser Asp Glu Ala Asp
115 120 125
Gln Ala Pro Trp Thr Pro Met Pro Leu Glu Ile Ala Phe Gln Gly Asp
130 135 140
Arg Glu Ser Val Leu Lys Ala Met Asn Ala Ile Thr Gly Met Gln Asp
145 150 155 160
Tyr Leu Phe Thr Val Asn Ser Ile Arg Ile Arg Asn Glu Arg Met Met
165 170 175
Pro Pro Pro Ile Ala Asn Pro Ala Ala Ala Lys Pro Ala Ala Ala Gln
180 185 190
Pro Ala Thr Gly Ala Ala Ser Leu Thr Pro Ala Asp Glu Ala Ala Ala
195 200 205
Pro Ala Ala Pro Ala Ile Gln Gln Val Ile Lys Pro Tyr Met Gly Lys
210 215 220
Glu Gln Val Phe Val Gln Val Ser Leu Asn Leu Val His Phe Asn Gln
225 230 235 240
Pro Lys Ala Gln Glu Pro Ser Glu Asp
245
<210>12
<211>299
<212>PRT
<213>Artificial Sequence
<220>
<223>synthetic
<400>12
Met His His His His His His Asp Asp Asp Asp Lys Ile Val Asn Ser
1 5 1015
Lys Arg Ser Glu Leu Asp Lys Lys Ile Ser Ile Ala Ala Lys Glu Ile
202530
Lys Ser Ala Asn Ala Ala Glu Ile Thr Pro Ser Arg Ser Ser Asn Glu
354045
Glu Leu Glu Lys Glu Leu Asn Arg Tyr Ala Lys Ala Val Gly Ser Leu
505560
Glu Thr Ala Tyr Lys Pro Phe Leu Ala Ser Ser Ala Leu Val Pro Thr
65707580
Thr Pro Thr Ala Phe Gln Asn Glu Leu Lys Thr Phe Arg Asp Ser Leu
859095
Ile Ser Ser Cys Lys Lys Lys Asn Ile Leu Ile Thr Asp Thr Ser Ser
100 105 110
Trp Leu Gly Phe Gln Val Tyr Ser Thr Gln Ala Pro Ser Val Gln Ala
115 120 125
Ala Ser Thr Leu Gly Phe Glu Leu Lys Ala Ile Asn Ser Leu Val Asn
130 135 140
Lys Leu Ala Glu Cys Gly Leu Ser Lys Phe Ile Lys Val Tyr Arg Pro
145 150 155 160
Gln Leu Pro Ile Glu Thr Pro Ala Asn Asn Pro Glu Glu Ser Asp Glu
165 170 175
Ala Asp Gln Ala Pro Trp Thr Pro Met Pro Leu Glu Ile Ala Phe Gln
180 185 190
Gly Asp Arg Glu Ser Val Leu Lys Ala Met Asn Ala Ile Thr Gly Met
195 200 205
Gln Asp Tyr Leu Phe Thr Val Asn Ser Ile Arg Ile Arg Asn Glu Arg
210 215 220
Met Met Pro Pro Pro Ile Ala Asn Pro Ala Ala Ala Lys Pro Ala Ala
225 230 235 240
Ala Gln Pro Ala Thr Gly Ala Ala Ser Leu Thr Pro Ala Asp Glu Ala
245 250 255
Ala Ala Pro Ala Ala Pro Ala Ile Gln Gln Val Ile Lys Pro Ala Met
260 265 270
Gly Lys Glu Gln Val Phe Val Gln Val Ser Leu Asn Leu Val His Phe
275 280 285
Asn Gln Pro Lys Ala Gln Glu Pro Ser Glu Asp
290 295
<210>13
<211>249
<212>PRT
<213>Artificial Sequence
<220>
<223>synthetic
<400>13
Met His His His His His His Asp Asp Asp Asp Lys Ser Leu Glu Thr
1 5 1015
Ala Tyr Lys Pro Phe Leu Ala Ser Ser Ala Leu Val Pro Thr Thr Pro
202530
Thr Ala Phe Gln Asn Glu Leu Lys Thr Phe Arg Asp Ser Leu Ile Ser
354045
Ser Cys Lys Lys Lys Asn Ile Leu Ile Thr Asp Thr Ser Ser Trp Leu
505560
Gly Phe Gln Val Tyr Ser Thr Gln Ala Pro Ser Val Gln Ala Ala Ser
65707580
Thr Leu Gly Phe Glu Leu Lys Ala Ile Asn Ser Leu Val Asn Lys Leu
859095
Ala Glu Cys Gly Leu Ser Lys Phe Ile Lys Val Tyr Arg Pro Gln Leu
100 105 110
Pro Ile Glu Thr Pro Ala Asn Asn Pro Glu Glu Ser Asp Glu Ala Asp
115 120 125
Gln Ala Pro Trp Thr Pro Met Pro Leu Glu Ile Ala Phe Gln Gly Asp
130 135 140
Arg Glu Ser Val Leu Lys Ala Met Asn Ala Ile Thr Gly Met Gln Asp
145 150 155 160
Tyr Leu Phe Thr Val Asn Ser Ile Arg Ile Arg Asn Glu Arg Met Met
165 170 175
Pro Pro Pro Ile Ala Asn Pro Ala Ala Ala Lys Pro Ala Ala Ala Gln
180 185 190
Pro Ala Thr Gly Ala Ala Ser Leu Thr Pro Ala Asp Glu Ala Ala Ala
195 200 205
Pro Ala Ala Pro Ala Ile Gln Gln Val Ile Lys Pro Ala Met Gly Lys
210 215 220
Glu Gln Val Phe Val Gln Val Ser Leu Asn Leu Val His Phe Asn Gln
225 230 235 240
Pro Lys Ala Gln Glu Pro Ser Glu Asp
245
<210>14
<211>35
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>14
ttgacagcta gctcagtcct aggtataatg ctagc 35
<210>15
<211>35
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>15
tttacagcta gctcagtcct aggtattatg ctagc 35
<210>16
<211>35
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>16
ttgacagcta gctcagtcct aggtactgtg ctagc 35
<210>17
<211>35
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>17
ctgatagcta gctcagtcct agggattatg ctagc 35
<210>18
<211>35
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>18
tttacagcta gctcagtcct agggactgtg ctagc 35
<210>19
<211>35
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>19
tttacggcta gctcagtcct aggtacaatg ctagc 35
<210>20
<211>35
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>20
tttatggcta gctcagtcct aggtacaatg ctagc 35
<210>21
<211>35
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>21
ttgacagcta gctcagtcct agggattgtg ctagc 35
<210>22
<211>77
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>22
aaagtgtttt gtaatcataa agaaatatta aggtggggta ggaatagtat aatatgttta60
ttcaaccgaa cttaatg 77
<210>23
<211>176
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>23
gggccgagca tgtggaaaca cctctgccag gtcaagcccg cagccatgcc gcatgttttt60
ttcttgaccg cccgggcaga accgtcgggc ggagccgtgt catgctccac acacgctaca 120
tggaaagaaa caagagattt ttgctagcca cagttcggtt tttggtgtaa ctaatc 176
<210>24
<211>339
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>24
gtaaatttag gattaatcct ggaacttttt ttgtcgccca gccaatgctt tcagtcgtga60
ctaattttcc ttgcggaggc ttgtctgaag cggtttccgc gattttcttc tgtaaattgt 120
cgctgacaaa aaagattaaa cgtaccttat acaagacttt tttttcatat gcctgacgga 180
gttcacactt gtaagttttc aactacgttg tagactttac atcgccaggg gtgctcggca 240
taagccgaag atatcggtag agttaatatt gagcagatcc ccggtgaagg atttaaccgt 300
gttatctcgt tggagatatt catggcgtat tttggatga339
<210>25
<211>35
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>25
ttgacggcta gctcagtcct aggtacagtg ctagc 35
<210>26
<211>35
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>26
ttgacagcta gctcagtcct aggtattgtg ctagc 35
<210>27
<211>35
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>27
tttacggcta gctcagtcct aggtactatg ctagc 35
<210>28
<211>30
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>28
gtttatacat aggcgagtac tctgttatgg 30
<210>29
<211>30
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>29
ggtttcaaaa ttgtgatcta tatttaacaa 30
<210>30
<211>35
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>30
ttgacggcta gctcagtcct aggtattgtg ctagc 35
<210>31
<211>35
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>31
ttgacagcta gctcagtcct agggactatg ctagc 35
<210>32
<211>35
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>32
tttatagcta gctcagccct tggtacaatg ctagc 35
<210>33
<211>35
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>33
ctgatggcta gctcagtcct agggattatg ctagc 35
<210>34
<211>35
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>34
ctgatagcta gctcagtcct agggattatg ctagc 35
<210>35
<211>35
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>35
ttgacggcta gctcagtcct aggtatagtg ctagc 35
<210>36
<211>35
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>36
ctgacagcta gctcagtcct aggtataatg ctagc 35
<210>37
<211>35
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>37
tttacggcta gctcagccct aggtattatg ctagc 35
<210>38
<211>35
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>38
tttacggcta gctcagtcct aggtatagtg ctagc 35
<210>39
<211>30
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>39
agaggttcca actttcacca taatgaaaca 30
<210>40
<211>30
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>40
caccttcggg tgggcctttc tgcgtttata 30
<210>41
<211>30
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>41
ggctagctca gtcctaggta ctatgctagc 30
<210>42
<211>30
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>42
aaagtgtgac gccgtgcaaa taatcaatgt 30
<210>43
<211>30
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>43
ggctagctca gtcctaggta ttatgctagc 30
<210>44
<211>30
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>44
taaacaacta acggacaatt ctacctaaca 30
<210>45
<211>178
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>45
aagctgttgt gaccgcttgc tctagccagc tatcgagttg tgaaccgatc catctagcaa60
ttggtctcga tctagcgata ggcttcgatc tagctatgta tcactcatta ggcaccccag 120
gctttacact ttatgcttcc ggctcgtata atgtgtggtg ctggttagcg cttgctat 178
<210>46
<211>190
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>46
aagctgttgt gaccgcttgc tctagccagc tatcgagttg tgaaccgatc catctagcaa60
ttggtctcga tctagcgata ggcttcgatc tagctatgta gaaacgccgt gtgctcgatc 120
gcttgataag gtccacgtag ctgctataat tgcttcaaca gaacatattg actatccggt 180
attacccggc190
<210>47
<211>30
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>47
catacgccgt tatacgttgt ttacgctttg 30
<210>48
<211>73
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>48
taattcctaa tttttgttga cactctatcg ttgatagagt tattttacca ctccctatca60
gtgatagaga aaa 73
<210>49
<211>44
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>49
ttgacaatta atcatccggc tcgtataatg tgtggaattg tgag 44
<210>50
<211>52
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>50
cccaggcttt acactttatg cttccggctc gtataatgtg tggaattgtg ag52
<210>51
<211>30
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>51
tgctagctac tagagattaa agaggagaaa 30
<210>52
<211>30
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>52
ggctagctca gtcctaggta cagtgctagc 30
<210>53
<211>30
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>53
ccccgaaagc ttaagaatat aattgtaagc 30
<210>54
<211>30
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>54
atatatatat atatataatg gaagcgtttt 30
<210>55
<211>74
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>55
tccctatcag tgatagagat tgacatccct atcagtgata gagatactga gcacatcagc60
aggacgcact gacc74
<210>56
<211>68
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>56
attccactaa tttattccat gtcacacttt tcgcatcttt gttatgctat ggttatttca60
taccataa 68
<210>57
<211>76
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>57
ataaatgtga gcggataaca attgacattg tgagcggata acaagatact gagcacatca60
gcaggacgca ctgacc76
<210>58
<211>65
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>58
aaaaacgccg caaagtttga gcgaagtcaa taaactctct acccattcag ggcaatatct60
ctctt65
<210>59
<211>81
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>59
atgaagaaaa agatcattag cgcgatcctg atgagcaccg tgattctgag cgcggcggcg60
ccgctgagcg gtgtttatgc g81
<210>60
<211>63
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>60
atgaagaaaa ccgcgattgc gattgcggtg gcgctggcgg gtttcgcgac cgttgcgcag60
gcg63
<210>61
<211>57
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>61
atgaagaaaa tctggctggc gctggcgggt ctggtgctgg cgttcagcgc gagcgcg 57
<210>62
<211>66
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>62
atgaagtacc tgctgccgac cgcggcggcg ggtctgctgc tgctggcggc gcagccggcg60
atggcg 66
<210>63
<211>63
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>63
atggaaggaa acactcgtga agacaatttt aaacatttat taggtaatga caatgttaaa60
cgc63
<210>64
<211>147
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>64
atgaataaaa tatactccct taaatatagt gctgccactg gcggactcat tgctgtttct60
gaattagcga aaagagtttc tggtaaaaca aaccgaaaac ttgtagcaac aatgttgtct 120
ctggctgttg ccggtacagt aaatgca 147
<210>65
<211>90
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>65
atgagcaact ggatcaccga caacaaaccg gctgcgatgg ttgcgggtgt gggcctgctg60
ctgttcctgg gtctgagcgc gaccggctac 90
<210>66
<211>33
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>66
atgcgtaaag gcgaagagaa ggaggttaac tga 33
<210>67
<211>54
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>67
atgaaagcaa ttttcgtact gaaacatctt aatcatgcta aggaggtttt ctaa54
<210>68
<211>33
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>68
atgattatgt ccggttataa ggaggttaac tga 33
<210>69
<211>45
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>69
atgaaaatcg aagcaggtaa actggtacag aaggaggtta actga45
<210>70
<211>22
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>70
ggaggaaaaa ttaaaaaaga ac 22
<210>71
<211>12
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>71
aaagaggaga aa12
<210>72
<400>72
000
<210>73
<400>73
000
<210>74
<211>186
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>74
cgagctcgat agtgctagtg tagatcgcta ctagagccag gcatcaaata aaacgaaagg60
ctcagtcgaa agactgggcc tttcgtttta tctgttgttt gtcggtgaac gctctctact 120
agagtcacac tggctcacct tcgggtgggc ctttctgcgt ttatatacta gaagcggccg 180
ctgcag186
<210>75
<211>173
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>75
cgagctcgat agtgctagtg tagatcgcta ctagagccag gcatcaaata aaacgaaaga60
ctgggccttt cgttttatct gttgtttgtc ggtgaacgct ctctactaga gtcacactgg 120
ctcaccttcg ggtgggcctt tctgcgttta tatactagaa gcggccgctg cag173
<210>76
<211>27
<212>PRT
<213>Artificial Sequence
<220>
<223>synthetic
<400>76
Met Lys Lys Lys Ile Ile Ser Ala Ile Leu Met Ser Thr Val Ile Leu
1 5 1015
Ser Ala Ala Ala Pro Leu Ser Gly Val Tyr Ala
2025
<210>77
<211>21
<212>PRT
<213>Artificial Sequence
<220>
<223>synthetic
<400>77
Met Lys Lys Thr Ala Ile Ala Ile Ala Val Ala Leu Ala Gly Phe Ala
1 5 1015
Thr Val Ala Gln Ala
20
<210>78
<211>19
<212>PRT
<213>Artificial Sequence
<220>
<223>synthetic
<400>78
Met Lys Lys Ile Trp Leu Ala Leu Ala Gly Leu Val Leu Ala Phe Ser
1 5 1015
Ala Ser Ala
<210>79
<211>22
<212>PRT
<213>Artificial Sequence
<220>
<223>synthetic
<400>79
Met Lys Tyr Leu Leu Pro Thr Ala Ala Ala Gly Leu Leu Leu Leu Ala
1 5 1015
Ala Gln Pro Ala Met Ala
20
<210>80
<211>21
<212>PRT
<213>Artificial Sequence
<220>
<223>synthetic
<400>80
Met Glu Gly Asn Thr Arg Glu Asp Asn Phe Lys His Leu Leu Gly Asn
1 5 1015
Asp Asn Val Lys Arg
20
<210>81
<211>49
<212>PRT
<213>Artificial Sequence
<220>
<223>synthetic
<400>81
Met Asn Lys Ile Tyr Ser Leu Lys Tyr Ser Ala Ala Thr Gly Gly Leu
1 5 1015
Ile Ala Val Ser Glu Leu Ala Lys Arg Val Ser Gly Lys Thr Asn Arg
202530
Lys Leu Val Ala Thr Met Leu Ser Leu Ala Val Ala Gly Thr Val Asn
354045
Ala
<210>82
<211>30
<212>PRT
<213>Artificial Sequence
<220>
<223>synthetic
<400>82
Met Ser Asn Trp Ile Thr Asp Asn Lys Pro Ala Ala Met Val Ala Gly
1 5 1015
Val Gly Leu Leu Leu Phe Leu Gly Leu Ser Ala Thr Gly Tyr
202530
<210>83
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>83
ggcgagttaa cgacgacaca20
<210>84
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>84
cgttgaactg ggtgtggaat20
<210>85
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>85
ctgaaaccgc tgcggcgatg20
<210>86
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>86
gttcgcagtg gaagtaccgt20
<210>87
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>87
ctgtgcaaac aagtgtctca20
<210>88
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>88
gccggaagac actatgaagc20
<210>89
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>89
tcgcacagcg tgtaccacag20
<210>90
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>90
gaaggggaag aggcgcgcgt20
<210>91
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>91
cggtttagtt cacagaagcc20
<210>92
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>92
ttgcgtattt tcaaaaagcg20
<210>93
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>93
tcatcagagt aagtcggata20
<210>94
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>94
ctgaccaacg cttctttacc20
<210>95
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>95
ccgaagtccc tgtgtgcttt20
<210>96
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>96
ccctgccact cacaccattc20
<210>97
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>97
ccttctcctg gcaagcttac20
<210>98
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>98
cgacaagaag tacctgcaac20
<210>99
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>99
gttatcgtga tgcgcattct20
<210>100
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>100
taacacccag cccgatgccc20
<210>101
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>101
atattactgg aacaacataa20
<210>102
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>102
ccgtgacgtt atccgcacca20
<210>103
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>103
tatcgccttc atccacacga20
<210>104
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>104
ccgggctgga tgtcttcgaa20
<210>105
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>105
gagggtgagc cataatgaag20
<210>106
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>106
ggatatgtgg ggtaacgacg20
<210>107
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>107
acgttcaggc tgctaaagat20
<210>108
<211>1258
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>108
tctagagctt gtcgacgtct cgagtttaca gctagctcag tcctaggtat tatgctagcc60
tcgaggaaag aggagaaaga agcttatgaa gaaaaagatc attagcgcga tcctgatgag 120
caccgtgatt ctgagcgcgg cggcgccgct gagcggtgtt tatgcgggta gcagccatca 180
tcaccaccac cacgacgacg acgacaaaat tgttaacagc aagcgtagcg agctggataa 240
gaaaatcagc attgcggcga aagagatcaa gagcgcgaac gcggcggaaa ttaccccgag 300
ccgtagcagc aacgaggaac tggagaaaga actgaaccgt tacgcgaagg cggttggtag 360
cctggaaacc gcgtataaac cgtttctggc gagcagcgcg ctggtgccga ccaccccgac 420
cgcgttccaa aacgagctga aaacctttcg tgacagcctg atcagcagct gcaagaaaaa 480
gaacatcctg attaccgata ccagcagctg gctgggcttc caggtttaca gcacccaagc 540
gccgagcgtg caggcggcga gcaccctggg ttttgagctg aaagcgatta acagcctggt 600
taacaagctg gcggaatgcg gcctgagcaa attcatcaag gtgtatcgtc cgcagctgcc 660
gattgaaacc ccggcgaaca acccggagga aagcgacgaa gcggatcagg cgccgtggac 720
cccgatgccg ctggagatcg cgttccaggg tgaccgtgaa agcgttctga aagcgatgaa 780
cgcgattacc ggcatgcaag attacctgtt taccgtgaac agcatccgta ttcgtaacga 840
acgtatgatg ccgccgccaa tcgcgaaccc ggctgcggcg aagccggctg cggcgcagcc 900
ggcgaccggt gcggcgagcc tgaccccggc ggacgaggct gcggcgccgg ctgcgccggc 960
gatccagcaa gttattaaac cgtatatggg caaggaacag gtgttcgttc aagtgagcct1020
gaacctggtg cactttaacc agccgaaagc gcaagagccg agcgaagatt gacgagctcg1080
atagtgctag tgtagatcgc tactagagcc aggcatcaaa taaaacgaaa ggctcagtcg1140
aaagactggg cctttcgttt tatctgttgt ttgtcggtga acgctctcta ctagagtcac1200
actggctcac cttcgggtgg gcctttctgc gtttatatac tagaagcggc cgctgcag1258
<210>109
<400>109
000
<210>110
<400>110
000
<210>111
<400>111
000
<210>112
<400>112
000
<210>113
<400>113
000
<210>114
<400>114
000
<210>115
<400>115
000
<210>116
<400>116
000
<210>117
<400>117
000
<210>118
<400>118
000
<210>119
<211>27
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>119
gccgcgcacg ctgatttttt atgaatc27
<210>120
<211>45
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>120
ctcgagacgt cgacaagctc tagagtagcg ctattcctgc tgcga45
<210>121
<211>27
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>121
cgctactcta gagcttgtcg acgtctc27
<210>122
<211>59
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>122
cctccttctg taccagttta cctgcttcga ttttcataag cttctttctc ctctttcct 59
<210>123
<211>54
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>123
agcaggtaaa ctggtacaga aggaggttaa ctgatgaaga aaaagatcat tagc54
<210>124
<211>40
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>124
ctacgcttgc tgttaacaat cgcataaaca ccgctcagcg40
<210>125
<211>40
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>125
ctgagcggtg tttatgcgat tgttaacagc aagcgtagcg40
<210>126
<211>40
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>126
cgatctacac tagcactatc gagctcgtta ttagtggtgg40
<210>127
<211>40
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>127
ccaccactaa taacgagctc gatagtgcta gtgtagatcg40
<210>128
<211>22
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>128
ctgcagcggc cgcttctagt at 22
<210>129
<211>44
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>129
tactagaagc ggccgctgca gtcacgcatg tgaaatggtt atgc 44
<210>130
<211>26
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>130
gtgaagacca gctcatcagc tttcgc 26
<210>131
<211>27
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>131
gccgcgcacg ctgatttttt atgaatc27
<210>132
<211>45
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>132
ctcgagacgt cgacaagctc tagagtagcg ctattcctgc tgcga45
<210>133
<211>27
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>133
cgctactcta gagcttgtcg acgtctc27
<210>134
<211>29
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>134
attcataagc ttctttctcc tctttcctc29
<210>135
<211>58
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>135
ctcgaggaaa gaggagaaag aagcttatga ataaaatata ctcccttaaa tatagtgc58
<210>136
<211>22
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>136
tgcatttact gtaccggcaa ca 22
<210>137
<211>52
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>137
tctctggctg ttgccggtac agtaaatgca attgttaaca gcaagcgtag cg52
<210>138
<211>54
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>138
cgcagatcac ccatacgttt gttaaggttg ttgtggtggt ggtgatgatg atct54
<210>139
<211>24
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>139
aaccttaaca aacgtatggg tgat 24
<210>140
<211>42
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>140
cactatcgag ctcgttatca gaaagagtaa cggaagttgg cg 42
<210>141
<211>43
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>141
acttccgtta ctctttctga taacgagctc gatagtgcta gtg43
<210>142
<211>22
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>142
ctgcagcggc cgcttctagt at 22
<210>143
<211>44
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>143
tactagaagc ggccgctgca gtcacgcatg tgaaatggtt atgc 44
<210>144
<211>26
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>144
gtgaagacca gctcatcagc tttcgc 26
<210>145
<211>296
<212>PRT
<213>Artificial Sequence
<220>
<223>synthetic
<400>145
Met Ile Val Asn Ser Lys Arg Ser Glu Leu Asp Lys Lys Ile Ser Ile
1 5 1015
Ala Ala Lys Glu Ile Lys Ser Ala Asn Ala Ala Glu Ile Thr Pro Ser
202530
Arg Ser Ser Asn Glu Glu Leu Glu Lys Glu Leu Asn Arg Tyr Ala Lys
354045
Ala Val Gly Ser Leu Glu Thr Ala Tyr Lys Pro Phe Leu Ala Ser Ser
505560
Ala Leu Val Pro Thr Thr Pro Thr Ala Phe Gln Asn Glu Leu Lys Thr
65707580
Phe Arg Asp Ser Leu Ile Ser Ser Cys Lys Lys Lys Asn Ile Leu Ile
859095
Thr Asp Thr Ser Ser Trp Leu Gly Phe Gln Val Tyr Ser Thr Gln Ala
100 105 110
Pro Ser Val Gln Ala Ala Ser Thr Leu Gly Phe Glu Leu Lys Ala Ile
115 120 125
Asn Ser Leu Val Asn Lys Leu Ala Glu Cys Gly Leu Ser Lys Phe Ile
130 135 140
Lys Val Tyr Arg Pro Gln Leu Pro Ile Glu Thr Pro Ala Asn Asn Pro
145 150 155 160
Glu Glu Ser Asp Glu Ala Asp Gln Ala Pro Trp Thr Pro Met Pro Leu
165 170 175
Glu Ile Ala Phe Gln Gly Asp Arg Glu Ser Val Leu Lys Ala Met Asn
180 185 190
Ala Ile Thr Gly Met Gln Asp Tyr Leu Phe Thr Val Asn Ser Ile Arg
195 200 205
Ile Arg Asn Glu Arg Met Met Pro Pro Pro Ile Ala Asn Pro Ala Ala
210 215 220
Ala Lys Pro Ala Ala Ala Gln Pro Ala Thr Gly Ala Ala Ser Leu Thr
225 230 235 240
Pro Ala Asp Glu Ala Ala Ala Pro Ala Ala Pro Ala Ile Gln Gln Val
245 250 255
Ile Lys Pro Tyr Met Gly Lys Glu Gln Val Phe Val Gln Val Ser Leu
260 265 270
Asn Leu Val His Phe Asn Gln Pro Lys Ala Gln Glu Pro Ser Glu Asp
275 280 285
Leu Glu His His His His His His
290 295
<210>146
<211>296
<212>PRT
<213>Artificial Sequence
<220>
<223>synthetic
<400>146
Met Ile Val Asn Ser Lys Arg Ser Glu Leu Asp Lys Lys Ile Ser Ile
1 5 1015
Ala Ala Lys Glu Ile Lys Ser Ala Asn Ala Ala Glu Ile Thr Pro Ser
202530
Arg Ser Ser Asn Glu Glu Leu Glu Lys Glu Leu Asn Arg Tyr Ala Lys
354045
Ala Val Gly Ser Leu Glu Thr Ala Tyr Lys Pro Phe Leu Ala Ser Ser
505560
Ala Leu Val Pro Thr Thr Pro Thr Ala Phe Gln Asn Glu Leu Lys Thr
65707580
Phe Arg Asp Ser Leu Ile Ser Ser Cys Lys Lys Lys Asn Ile Leu Ile
859095
Thr Asp Thr Ser Ser Trp Leu Gly Phe Gln Val Tyr Ser Thr Gln Ala
100 105 110
Pro Ser Val Gln Ala Ala Ser Thr Leu Gly Phe Glu Leu Lys Ala Ile
115 120 125
Asn Ser Leu Val Asn Lys Leu Ala Glu Cys Gly Leu Ser Lys Phe Ile
130 135 140
Lys Val Tyr Arg Pro Gln Leu Pro Ile Glu Thr Pro Ala Asn Asn Pro
145 150 155 160
Glu Glu Ser Asp Glu Ala Asp Gln Ala Pro Trp Thr Pro Met Pro Leu
165 170 175
Glu Ile Ala Phe Gln Gly Asp Arg Glu Ser Val Leu Lys Ala Met Asn
180 185 190
Ala Ile Thr Gly Met Gln Asp Tyr Leu Phe Thr Val Asn Ser Ile Arg
195 200 205
Ile Arg Asn Glu Arg Met Met Pro Pro Pro Ile Ala Asn Pro Ala Ala
210 215 220
Ala Lys Pro Ala Ala Ala Gln Pro Ala Thr Gly Ala Ala Ser Leu Thr
225 230 235 240
Pro Ala Asp Glu Ala Ala Ala Pro Ala Ala Pro Ala Ile Gln Gln Val
245 250 255
Ile Lys Pro Ala Met Gly Lys Glu Gln Val Phe Val Gln Val Ser Leu
260 265 270
Asn Leu Val His Phe Asn Gln Pro Lys Ala Gln Glu Pro Ser Glu Asp
275 280 285
Leu Glu His His His His His His
290 295
<210>147
<211>891
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>147
atgatcgtga atagtaagcg tagcgaactg gataagaaga ttagcattgc cgccaaagaa60
attaaatctg caaatgcagc agaaattaca cctagtcgca gcagcaatga agaactggag 120
aaagaactga atcgttatgc aaaggcagtt ggcagcctgg aaaccgccta taaaccgttt 180
ctggcaagca gtgcactggt tccgaccaca cctaccgcat ttcagaatga actgaagact 240
ttccgtgata gtctgattag tagctgcaag aagaagaata ttctgattac cgataccagt 300
agttggctgg gtttccaggt ttatagcacc caggcaccga gcgtgcaggc cgcaagtacc 360
ctgggtttcg aactgaaagc cattaatagt ctggtgaata aactggccga atgtggcctg 420
agcaaattta ttaaagttta tcgtccgcag ctgccgattg aaacacctgc caataatccg 480
gaagaaagcg atgaagcaga tcaggcaccg tggacaccta tgccgctgga aattgcattt 540
cagggcgatc gtgaaagcgt gctgaaagcc atgaatgcca ttaccggcat gcaggattat 600
ctgtttaccg tgaatagtat tcgcattcgc aatgaacgca tgatgccgcc gccgattgcc 660
aatccggcag ccgcaaagcc ggcagccgcc cagcctgcaa ccggtgcagc aagcctgaca 720
cctgcagatg aagcagcagc accggcagca cctgcaattc agcaggttat taaaccggcc 780
atgggcaaag aacaggtgtt tgttcaggtt agcctgaatc tggtgcattt caatcagccg 840
aaagcacagg aaccgagcga agatctcgag caccaccacc accaccactg a891
<210>148
<211>1245
<212>DNA
<213>Artificial Sequence
<220>
<223>synthetic
<400>148
tctagagctt gtcgacgtct cgagtttaca gctagctcag tcctaggtat tatgctagcc60
tcgaggaaag aggagaaaga agcttatgaa gaaaaagatc attagcgcga tcctgatgag 120
caccgtgatt ctgagcgcgg cggcgccgct gagcggtgtt tatgcgggta gcagccatca 180
tcaccaccac cacgacgacg acgacaaaat tgttaacagc aagcgtagcg agctggataa 240
gaaaatcagc attgcggcga aagagatcaa gagcgcgaac gcggcggaaa ttaccccgag 300
ccgtagcagc aacgaggaac tggagaaaga actgaaccgt tacgcgaagg cggttggtag 360
cctggaaacc gcgtataaac cgtttctggc gagcagcgcg ctggtgccga ccaccccgac 420
cgcgttccaa aacgagctga aaacctttcg tgacagcctg atcagcagct gcaagaaaaa 480
gaacatcctg attaccgata ccagcagctg gctgggcttc caggtttaca gcacccaagc 540
gccgagcgtg caggcggcga gcaccctggg ttttgagctg aaagcgatta acagcctggt 600
taacaagctg gcggaatgcg gcctgagcaa attcatcaag gtgtatcgtc cgcagctgcc 660
gattgaaacc ccggcgaaca acccggagga aagcgacgaa gcggatcagg cgccgtggac 720
cccgatgccg ctggagatcg cgttccaggg tgaccgtgaa agcgttctga aagcgatgaa 780
cgcgattacc ggcatgcaag attacctgtt taccgtgaac agcatccgta ttcgtaacga 840
acgtatgatg ccgccgccaa tcgcgaaccc ggctgcggcg aagccggctg cggcgcagcc 900
ggcgaccggt gcggcgagcc tgaccccggc ggacgaggct gcggcgccgg ctgcgccggc 960
gatccagcaa gttattaaac cgtatatggg caaggaacag gtgttcgttc aagtgagcct1020
gaacctggtg cactttaacc agccgaaagc gcaagagccg agcgaagatt gacgagctcg1080
atagtgctag tgtagatcgc tactagagcc aggcatcaaa taaaacgaaa gactgggcct1140
ttcgttttat ctgttgtttg tcggtgaacg ctctctacta gagtcacact ggctcacctt1200
cgggtgggcc tttctgcgtt tatatactag aagcggccgc tgcag1245
<210>149
<211>15460
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>149
ctgctcgcgc aggctgggtg ccaagctctc gggtaacatc aaggcccgat ccttggagcc60
cttgccctcc cgcacgatga tcgtgccgtg atcgaaatcc agatccttga cccgcagttg 120
caaaccctca ctgatccgca tgcggatctt gctgtaggca taggcttggt tatgccggta 180
ctgccgggcc tcttgcggga ttacgaaatc atcctgtgga gcttagtagg tttagcaaga 240
tggcagcgcc taaatgtaga atgataaaag gattaagaga ttaatttccc taaaaatgat 300
aaaacaagcg ttttgaaagc gcttgttttt ttggtttgca gtcagagtag aatagaagta 360
tcaaaaaaag caccgactcg gtgccacttt ttcaagttga taacggacta gccttatttt 420
aacttgctat gctgttttga atggttccaa caagattatt ttataacttt tataacaaat 480
aatcaaggag aaattcaaag aaatttatca gccataaaac aatacttaat actatagaat 540
gataacaaaa taaactactt tttaaaagaa ttttgtgtta taatctattt attattaagt 600
attgggtaat attttttgaa gagatatttt gaaaaagaaa aattaaagca tattaaacta 660
atttcggagg tcattaaaac tattattgaa atcatcaaac tcattatgga tttaatttaa 720
actttttatt ttaggaggca aaaatggata agaaatactc aataggctta gatatcggca 780
caaatagcgt cggatgggcg gtgatcactg atgaatataa ggttccgtct aaaaagttca 840
aggttctggg aaatacagac cgccacagta tcaaaaaaaa tcttataggg gctcttttat 900
ttgacagtgg agagacagcg gaagcgactc gtctcaaacg gacagctcgt agaaggtata 960
cacgtcggaa gaatcgtatt tgttatctac aggagatttt ttcaaatgag atggcgaaag1020
tagatgatag tttctttcat cgacttgaag agtctttttt ggtggaagaa gacaagaagc1080
atgaacgtca tcctattttt ggaaatatag tagatgaagt tgcttatcat gagaaatatc1140
caactatcta tcatctgcga aaaaaattgg tagattctac tgataaagcg gatttgcgct1200
taatctattt ggccttagcg catatgatta agtttcgtgg tcattttttg attgagggag1260
atttaaatcc tgataatagt gatgtggaca aactatttat ccagttggta caaacctaca1320
atcaattatt tgaagaaaac cctattaacg caagtggagt agatgctaaa gcgattcttt1380
ctgcacgatt gagtaaatca agacgattag aaaatctcat tgctcagctc cccggtgaga1440
agaaaaatgg cttatttggg aatctcattg ctttgtcatt gggtttgacc cctaatttta1500
aatcaaattt tgatttggca gaagatgcta aattacagct ttcaaaagat acttacgatg1560
atgatttaga taatttattg gcgcaaattg gagatcaata tgctgatttg tttttggcag1620
ctaagaattt atcagatgct attttacttt cagatatcct aagagtaaat actgaaataa1680
ctaaggctcc cctatcagct tcaatgatta aacgctacga tgaacatcat caagacttga1740
ctcttttaaa agctttagtt cgacaacaac ttccagaaaa gtataaagaa atcttttttg1800
atcaatcaaa aaacggatat gcaggttata ttgatggggg agctagccaa gaagaatttt1860
ataaatttat caaaccaatt ttagaaaaaa tggatggtac tgaggaatta ttggtgaaac1920
taaatcgtga agatttgctg cgcaagcaac ggacctttga caacggctct attccccatc1980
aaattcactt gggtgagctg catgctattt tgagaagaca agaagacttt tatccatttt2040
taaaagacaa tcgtgagaag attgaaaaaa tcttgacttt tcgaattcct tattatgttg2100
gtccattggc gcgtggcaat agtcgttttg catggatgac tcggaagtct gaagaaacaa2160
ttaccccatg gaattttgaa gaagttgtcg ataaaggtgc ttcagctcaa tcatttattg2220
aacgcatgac aaactttgat aaaaatcttc caaatgaaaa agtactacca aaacatagtt2280
tgctttatga gtattttacg gtttataacg aattgacaaa ggtcaaatat gttactgaag2340
gaatgcgaaa accagcattt ctttcaggtg aacagaagaa agccattgtt gatttactct2400
tcaaaacaaa tcgaaaagta accgttaagc aattaaaaga agattatttc aaaaaaatag2460
aatgttttga tagtgttgaa atttcaggag ttgaagatag atttaatgct tcattaggta2520
cctaccatga tttgctaaaa attattaaag ataaagattt tttggataat gaagaaaatg2580
aagatatctt agaggatatt gttttaacat tgaccttatt tgaagatagg gagatgattg2640
aggaaagact taaaacatat gctcacctct ttgatgataa ggtgatgaaa cagcttaaac2700
gtcgccgtta tactggttgg ggacgtttgt ctcgaaaatt gattaatggt attagggata2760
agcaatctgg caaaacaata ttagattttt tgaaatcaga tggttttgcc aatcgcaatt2820
ttatgcagct gatccatgat gatagtttga catttaaaga agacattcaa aaagcacaag2880
tgtctggaca aggcgatagt ttacatgaac atattgcaaa tttagctggt agccctgcta2940
ttaaaaaagg tattttacag actgtaaaag ttgttgatga attggtcaaa gtaatggggc3000
ggcataagcc agaaaatatc gttattgaaa tggcacgtga aaatcagaca actcaaaagg3060
gccagaaaaa ttcgcgagag cgtatgaaac gaatcgaaga aggtatcaaa gaattaggaa3120
gtcagattct taaagagcat cctgttgaaa atactcaatt gcaaaatgaa aagctctatc3180
tctattatct ccaaaatgga agagacatgt atgtggacca agaattagat attaatcgtt3240
taagtgatta tgatgtcgat cacattgttc cacaaagttt ccttaaagac gattcaatag3300
acaataaggt cttaacgcgt tctgataaaa atcgtggtaa atcggataac gttccaagtg3360
aagaagtagt caaaaagatg aaaaactatt ggagacaact tctaaacgcc aagttaatca3420
ctcaacgtaa gtttgataat ttaacgaaag ctgaacgtgg aggtttgagt gaacttgata3480
aagctggttt tatcaaacgc caattggttg aaactcgcca aatcactaag catgtggcac3540
aaattttgga tagtcgcatg aatactaaat acgatgaaaa tgataaactt attcgagagg3600
ttaaagtgat taccttaaaa tctaaattag tttctgactt ccgaaaagat ttccaattct3660
ataaagtacg tgagattaac aattaccatc atgcccatga tgcgtatcta aatgccgtcg3720
ttggaactgc tttgattaag aaatatccaa aacttgaatc ggagtttgtc tatggtgatt3780
ataaagttta tgatgttcgt aaaatgattg ctaagtctga gcaagaaata ggcaaagcaa3840
ccgcaaaata tttcttttac tctaatatca tgaacttctt caaaacagaa attacacttg3900
caaatggaga gattcgcaaa cgccctctaa tcgaaactaa tggggaaact ggagaaattg3960
tctgggataa agggcgagat tttgccacag tgcgcaaagt attgtccatg ccccaagtca4020
atattgtcaa gaaaacagaa gtacagacag gcggattctc caaggagtca attttaccaa4080
aaagaaattc ggacaagctt attgctcgta aaaaagactg ggatccaaaa aaatatggtg4140
gttttgatag tccaacggta gcttattcag tcctagtggt tgctaaggtg gaaaaaggga4200
aatcgaagaa gttaaaatcc gttaaagagt tactagggat cacaattatg gaaagaagtt4260
cctttgaaaa aaatccgatt gactttttag aagctaaagg atataaggaa gttaaaaaag4320
acttaatcat taaactacct aaatatagtc tttttgagtt agaaaacggt cgtaaacgga4380
tgctggctag tgccggagaa ttacaaaaag gaaatgagct ggctctgcca agcaaatatg4440
tgaatttttt atatttagct agtcattatg aaaagttgaa gggtagtcca gaagataacg4500
aacaaaaaca attgtttgtg gagcagcata agcattattt agatgagatt attgagcaaa4560
tcagtgaatt ttctaagcgt gttattttag cagatgccaa tttagataaa gttcttagtg4620
catataacaa acatagagac aaaccaatac gtgaacaagc agaaaatatt attcatttat4680
ttacgttgac gaatcttgga gctcccgctg cttttaaata ttttgataca acaattgatc4740
gtaaacgata tacgtctaca aaagaagttt tagatgccac tcttatccat caatccatca4800
ctggtcttta tgaaacacgc attgatttga gtcagctagg aggtgactga agtatatttt4860
agatgaagat tatttcttaa taactaaaaa tatggtataa tactcttaat aaatgcagta4920
atacaggggc ttttcaagac tgaagtctag ctgagacaaa tagtgcgatt acgaaatttt4980
ttagacaaaa atagtctacg aggttttaga gctatgctgt tttgaatggt cccaaaactg5040
cagcgcaata gttggcgaag taatcgcaac atccgcatta aaatctagcg agggctttac5100
taagctccgc aaaaaacccc gcccctgaca gggcggggtt ttttcgcgcc cgatccttgg5160
agcccttgcc ctcccgcacg atgatcgtgc cgtgatcgaa atccagatcc ttgacccgca5220
gttgcaaacc ctcactgatc cgcatgctta tgacaacttg acggctacat cattcacttt5280
ttcttcacaa ccggcacgga actcgctcgg gctggccccg gtgcattttt taaatacccg5340
cgagaaatag agttgatcgt caaaaccaac attgcgaccg acggtggcga taggcatccg5400
ggtggtgctc aaaagcagct tcgcctggct gatacgttgg tcctcgcgcc agcttaagac5460
gctaatccct aactgctggc ggaaaagatg tgacagacgc gacggcgaca agcaaacatg5520
ctgtgcgacg ctggcgatat caaaattgct gtctgccagg tgatcgctga tgtactgaca5580
agcctcgcgt acccgattat ccatcggtgg atggagcgac tcgttaatcg cttccatgcg5640
ccgcagtaac aattgctcaa gcagatttat cgccagcagc tccgaatagc gcccttcccc5700
ttgcccggcg ttaatgattt gcccaaacag gtcgctgaaa tgcggctggt gcgcttcatc5760
cgggcgaaag aaccccgtat tggcaaatat tgacggccag ttaagccatt catgccagta5820
ggcgcgcgga cgaaagtaaa cccactggtg ataccattcg cgagcctccg gatgacgacc5880
gtagtgatga atctctcctg gcgggaacag caaaatatca cccggtcggc aaacaaattc5940
tcgtccctga tttttcacca ccccctgacc gcgaatggtg agattgagaa tataaccttt6000
cattcccagc ggtcggtcga taaaaaaatc gagataaccg ttggcctcaa tcggcgttaa6060
acccgccacc agatgggcat taaacgagta tcccggcagc aggggatcat tttgcgcttc6120
agccatactt ttcatactcc cgccattcag agaagaaacc aattgtccat attgcatcag6180
acattgccgt cactgcgtct tttactggct cttctcgcta accaaaccgg taaccccgct6240
tattaaaagc attctgtaac aaagcgggac caaagccatg acaaaaacgc gtaacaaaag6300
tgtctataat cacggcagaa aagtccacat tgattatttg cacggcgtca cactttgcta6360
tgccatagca tttttatcca taagattagc ggatcctacc tgacgctttt tatcgcaact6420
ctctactgtt tctccatgta ggtcgttcgc tccaagcgtt ttagagctag aaatagcaag6480
ttaaaataag gctagtccgt tatcaacttg aaaaagtggc accagacccc gttgatgata6540
ccgctgcctt actgggtgca ttagccagtc tgaatgacct cgagttttag agctatgctg6600
ttttgaatgg tcccaaaact tcagcacact gagacttgtt gagttccatg ttttagagct6660
atgctgtttt gaatggactc cattcaacat tgccgatgat aacttgagaa agagggttaa6720
taccagcagt cggatacctt cctattcttt ctgttaaagc gttttcatgt tataataggc6780
aaaagaagag tagtgtgatc gtccattccg acgtcgagcc gttccataca gaagctgggc6840
gaacaaacga tgctcgcctt ccagaaaacc gaggatgcga accacttcat ccggggtcag6900
caccaccggc aagcgccgcg acggccgagg tcttccgatc tcctgaagcc agggcagatc6960
cgtgcacagc accttgccgt agaagaacag caaggccgcc aatgcctgac gatgcgtgga7020
gaccgaaacc ttgcgctcgt tcgccagcca ggacagaaat gcctcgactt cgctgctgcc7080
caaggttgcc gggtgacgca caccgtggaa acggatgaag gcacgaaccc agtggacata7140
agcctgttcg gttcgtaagc tgtaatgcaa gtagcgtatg cgctcacgca actggtccag7200
aaccttgacc gaacgcagcg gtggtaacgg cgcagtggcg gttttcatgg cttgttatga7260
ctgttttttt ggggtacagt ctatgcctcg ggcatccaag cagcaagcgc gttacgccgt7320
gggtcgatgt ttgatgttat ggagcagcaa cgatgttacg cagcagggca gtcgccctaa7380
aacaaagtta aacatcatga gggaagcggt gatcgccgaa gtatcgactc aactatcaga7440
ggtagttggc gtcatcgagc gccatctcga accgacgttg ctggccgtac atttgtacgg7500
ctccgcagtg gatggcggcc tgaagccaca cagtgatatt gatttgctgg ttacggtgac7560
cgtaaggctt gatgaaacaa cgcggcgagc tttgatcaac gaccttttgg aaacttcggc7620
ttcccctgga gagagcgaga ttctccgcgc tgtagaagtc accattgttg tgcacgacga7680
catcattccg tggcgttatc cagctaagcg cgaactgcaa tttggagaat ggcagcgcaa7740
tgacattctt gcaggtatct tcgagccagc cacgatcgac attgatctgg ctatcttgct7800
gacaaaagca agagaacata gcgttgcctt ggtaggtcca gcggcggagg aactctttga7860
tccggttcct gaacaggatc tatttgaggc gctaaatgaa accttaacgc tatggaactc7920
gccgcccgac tgggctggcg atgagcgaaa tgtagtgctt acgttgtccc gcatttggta7980
cagcgcagta accggcaaaa tcgcgccgaa ggatgtcgct gcctactggg caatggagcg8040
cctgccggcc cagtatcagc ccgtcatact tgaagctaga caggcttatc ttggacaaga8100
agaagatcgc ttggcctcgc gcgcagatca gttggaagaa tttgtccact acgtgaaagg8160
cgagatcacc aaggtagtcg gcaaataatg tctaacaatt cgttcaagcc gacgccgctt8220
cgcggcgcgg cttaactcaa gcgttagatg cactaagcac ataattgctc acagccaaac8280
tatcaggtca agtctgcttt tattattttt aagcgtgcat aataagccct acacaaattg8340
ggagatatat catgaaaggc tggctttttc ttgttatcgc aatagttggc gaagtaatcg8400
caacatccgc attaaaatct agcgagggct ttactaagct cctgcagaga tctgaattcc8460
ctagagagac gaaagtgatt gcgcctaccc ggatattatc gtgaggatgc gtcatcgcca8520
ttgctcccca aatacaaaac caatttcagc cagtgcctcg tccatttttt cgatgaactc8580
cggcacgatc tcgtcaaaac tcgccatgta cttttcatcc cgctcaatca cgacataatg8640
caggccttca cgcttcatac gcgggtcata gttggcaaag taccaggcat tttttcgcgt8700
cacccacatg ctgtactgca cctgggccat gtaagctgac tttatggcct cgaaaccacc8760
gagccggaac ttcatgaaat cccgggaggt aaacgggcat ttcagttcaa ggccgttgcc8820
gtcactgcat aaaccatcgg gagagcaggc ggtacgcata ctttcgtcgc gatagatgat8880
cggggattca gtaacattca cgccggaagt gaattcaaac agggttctgg cgtcgttctc8940
gtactgtttt ccccaggcca gtgctttagc gttaacttcc ggagccacac cggtgcaaac9000
ctcagcaagc agggtgtgga agtaggacat tttcatgtca ggccacttct ttccggagcg9060
gggttttgct atcacgttgt gaacttctga agcggtgatg acgccgagcc gtaatttgtg9120
ccacgcatct tccccctgtt cgacagctct cacatcgatc ccggtacgct gcaggataat9180
gtccggtgtc atgctgccac cttctgctct gcggctttct gtttcaggaa tccaagagct9240
tttactgctt cggcctgtgt cagttctgac gatgcacgaa tgtcgcggcg aaatatctgg9300
gaacagagcg gcaataagtc gtcatcccat gttttatcca gggcgatcag cagagtgtta9360
atctcctgca tggtttcatc gttaaccgga gtgatgtcgc gttccggctg acgttctgca9420
gtgtatgcag tattttcgac aatgcgctcg gcttcatcct tgtcatagat accagcaaat9480
ccgaaggcca gacgggcaca ctgaatcatg gctttatgac gtaacatccg tttgggatgc9540
gactgccacg gccccgtgat ttctctgcct tcgcgagttt tgaatggttc gcggcggcat9600
tcatccatcc attcggtaac gcagatcgga tgattacggt ccttgcggta aatccggcat9660
gtacaggatt cattgtcctg ctcaaagtcc atgccatcaa actgctggtt ttcattgatg9720
atgcgggacc agccatcaac gcccaccacc ggaacgatgc cattctgctt atcaggaaag9780
gcgtaaattt ctttcgtcca cggattaagg ccgtactggt tggcaacgat cagtaatgcg9840
atgaactgcg catcgctggc atcaccttta aatgccgtct ggcgaagagt ggtgatcagt9900
tcctgtgggt cgacagaatc catgccgaca cgttcagcca gcttcccagc cagcgttgcg9960
agtgcagtac tcattcgttt tatacctctg aatcaatatc aacctggtgg tgagcaatgg 10020
tttcaaccat gtaccggatg tgttctgcca tgcgctcctg aaactcaaca tcgtcatcaa 10080
acgcacgggt aatggatttt ttgctggccc cgtggcgttg caaatgatcg atgcatagcg 10140
attcaaacag gtgctggggc aggccttttt ccatgtcgtc tgccagttct gcctctttct 10200
cttcacgggc gagctgctgg tagtgacgcg cccagctctg agcctcaaga cgatcctgaa 10260
tgtaataagc gttcatggct gaactcctga aatagctgtg aaaatatcgc ccgcgaaatg 10320
ccgggctgat taggaaaaca ggaaaggggg ttagtgaatg cttttgcttg atctcagttt 10380
cagtattaat atccattttt tataagcgtc gactgtttcc tgtgtgaaat tgttatccgc 10440
tcacaattcc acacattata cgagccggaa gcataaagtg taaagcctgg ggtgcctaat 10500
gagtgagaat tcggatctcg acgggatgtt gattctgtca tggcatatcc ttacaactta 10560
aaaaagcaaa agggccgcag atgcgaccct tgtgtatcaa acaagacgat taaaaatctt 10620
cgttagtttc tgctacgcct tcgctatcat ctacagagaa atccggcgtt gagttcgggt 10680
tgctcagcag caactcacgt actttcttct cgatctcttt cgcggtttcc gggttatctt 10740
tcagccaggc agtcgcattc gctttaccct gaccgatctt ctcacctttg tagctgtacc 10800
acgcgcctgc tttctcgatc agcttctctt ttacgcccag gtcaaccagt tcgccgtaga 10860
agttgatacc ttcgccgtag aggatctgga attcagcctg tttaaacggc gcagcgattt 10920
tgttcttcac cactttcacg cgggtttcgc tacccaccac gttttcgccc tctttcaccg 10980
cgccgatacg acggatgtcg agacgaacag aggcgtagaa tttcagcgcg ttaccaccgg 11040
tagtggtttc cgggttaccg aacatcacac caattttcat acggatctgg ttgatgaaga 11100
tcagcagcgt gttggactgc ttcaggttac ccgccagctt acgcatcgcc tggctcatca 11160
tacgtgccgc aaggcccatg tgagagtcgc cgatttcgcc ttcgatttcc gctttcggcg 11220
tcagtgccgc cacggagtca acgacgataa cgtctactgc gccagaacgc gccagggcgt 11280
cacagatttc cagtgcctgc tcgccggtgt ccggctggga gcacagcagg ttgtcgatat 11340
cgacgcccag tttacgtgcg tagattgggt ccagcgcgtg ttcagcatcg ataaacgcac 11400
aggttttacc ttcacgctgc gctgcggcga tcacctgcag cgtcagcgtg gttttaccgg 11460
aagattccgg tccgtagatt tcgacgatac ggcccatcgg cagaccacct gccccaagcg 11520
cgatatccag tgaaagcgaa ccggtagaga tggtttccac atccatggaa cggtcttcac 11580
ccaggcgcat gatggagcct ttaccaaatt gtttctcaat ctggcccagt gctgccgcca 11640
acgctttctg tttgttttcg tcgatagcca tttttactcc tgtcatgccg ggtaataccg 11700
gatagtcaat atgttctgtt gaagcaatta tactgtatgc tcatacagta tcaagtgttt 11760
tgtagaaatt gttgccacaa ggtctgcaat gcatacgcag tagcctgacg acgcaccgca 11820
tcacggtcgc cgctgaagca tcccgccggg taatgccttc accgcgggca gtggcaaaag 11880
caaaccagac ggtgccgaca ggcttctcta gatccgccta cctttcacga gttgcgcagt 11940
ttgtctgcaa gactctatga gaagcagata agcgataagt ttgctcaaca tcttctcggg 12000
cataagtcgg acaccatggc atcacagtat cgtgatgaca gaggcaggga gtgggacaaa 12060
attgaaatca aataatgatt ttattttgac tgatagtgac ctgttcgttg caacaaattg 12120
ataagcaatg ccaagcttgg cactggctga tcagctagct cactgcccgc tttccagtcg 12180
ggaaacctgt cgtgccagct gcattaatga atcggccaac gcgcggggag aggcggtttg 12240
cgtattgggc gccagggtgg tttttctttt caccagtgag acgggcaaca gctgattgcc 12300
cttcaccgcc tggccctgag agagttgcag caagcggtcc acgctggttt gccccagcag 12360
gcgaaaatcc tgtttgatgg tggttaacgg cgggatataa catgagctgt cttcggtatc 12420
gtcgtatccc actaccgaga tatccgcacc aacgcgcagc ccggactcgg taatggcgcg 12480
cattgcgccc agcgccatct gatcgttggc aaccagcatc gcagtgggaa cgatgccctc 12540
attcagcatt tgcatggttt gttgaaaacc ggacatggca ctccagtcgc cttcccgttc 12600
cgctatcggc tgaatttgat tgcgagtgag atatttatgc cagccagcca gacgcagacg 12660
cgccgagaca gaacttaatg ggcccgctaa cagcgcgatt tgctggtgac ccaatgcgac 12720
cagatgctcc acgcccagtc gcgtaccgtc ttcatgggag aaaataatac tgttgatggg 12780
tgtctggtca gagacatcaa gaaataacgc cggaacatta gtgcaggcag cttccacagc 12840
aatggcatcc tggtcatcca gcggatagtt aatgatcagc ccactgacgc gttgcgcgag 12900
aagattgtgc accgccgctt tacaggcttc gacgccgctt cgttctacca tcgacaccac 12960
cacgctggca cccagttgat cggcgcgaga tttaatcgcc gcgacaattt gcgacggcgc 13020
gtgcagggcc agactggagg tggcaacgcc aatcagcaac gactgtttgc ccgccagttg 13080
ttgtgccacg cggttgggaa tgtaattcag ctccgccatc gccgcttcca ctttttcccg 13140
cgttttcgca gaaacgtggc tggcctggtt caccacgcgg gaaacggtct gataagagac 13200
accggcatac tctgcgacat cgtataacgt tactggtttc acattcacca ccctgaattg 13260
actctcttcc gggcgctatc atgccatacc gcgaaaggtt ttgcaccatt cgatgctagc 13320
ccatgggtat ggacagtttt ccctttgata tgtaacggtg aacagttgtt ctacttttgt 13380
ttgttagtct tgatgcttca ctgatagata caagagccat aagaacctca gatccttccg 13440
tatttagcca gtatgttctc tagtgtggtt cgttgttttt gcgtgagcca tgagaacgaa 13500
ccattgagat catacttact ttgcatgtca ctcaaaaatt ttgcctcaaa actggtgagc 13560
tgaatttttg cagttaaagc atcgtgtagt gtttttctta gtccgttacg taggtaggaa 13620
tctgatgtaa tggttgttgg tattttgtca ccattcattt ttatctggtt gttctcaagt 13680
tcggttacga gatccatttg tctatctagt tcaacttgga aaatcaacgt atcagtcggg 13740
cggcctcgct tatcaaccac caatttcata ttgctgtaag tgtttaaatc tttacttatt 13800
ggtttcaaaa cccattggtt aagcctttta aactcatggt agttattttc aagcattaac 13860
atgaacttaa attcatcaag gctaatctct atatttgcct tgtgagtttt cttttgtgtt 13920
agttctttta ataaccactc ataaatcctc atagagtatt tgttttcaaa agacttaaca 13980
tgttccagat tatattttat gaattttttt aactggaaaa gataaggcaa tatctcttca 14040
ctaaaaacta attctaattt ttcgcttgag aacttggcat agtttgtcca ctggaaaatc 14100
tcaaagcctt taaccaaagg attcctgatt tccacagttc tcgtcatcag ctctctggtt 14160
gctttagcta atacaccata agcattttcc ctactgatgt tcatcatctg agcgtattgg 14220
ttataagtga acgataccgt ccgttctttc cttgtagggt tttcaatcgt ggggttgagt 14280
agtgccacac agcataaaat tagcttggtt tcatgctccg ttaagtcata gcgactaatc 14340
gctagttcat ttgctttgaa aacaactaat tcagacatac atctcaattg gtctaggtga 14400
ttttaatcac tataccaatt gagatgggct agtcaatgat aattactagt ccttttcctt 14460
tgagttgtgg gtatctgtaa attctgctag acctttgctg gaaaacttgt aaattctgct 14520
agaccctctg taaattccgc tagacctttg tgtgtttttt ttgtttatat tcaagtggtt 14580
ataatttata gaataaagaa agaataaaaa aagataaaaa gaatagatcc cagccctgtg 14640
tataactcac tactttagtc agttccgcag tattacaaaa ggatgtcgca aacgctgttt 14700
gctcctctac aaaacagacc ttaaaaccct aaaggcttaa gtagcaccct cgcaagctcg 14760
gttgcggccg caatcgggca aatcgctgaa tattcctttt gtctccgacc atcaggcacc 14820
tgagtcgctg tctttttcgt gacattcagt tcgctgcgct cacggctctg gcagtgaatg 14880
ggggtaaatg gcactacagg cgccttttat ggattcatgc aaggaaacta cccataatac 14940
aagaaaagcc cgtcacgggc ttctcagggc gttttatggc gggtctgcta tgtggtgcta 15000
tctgactttt tgctgttcag cagttcctgc cctctgattt tccagtctga ccacttcgga 15060
ttatcccgtg acaggtcatt cagactggct aatgcaccca gtaaggcagc ggtatcatca 15120
acggggtctg acgctcagtg gaacgaaaac tcacgttaag ggattttggt catgagatta 15180
tcaaaaagga tcttcaccta gatcctttta aattaaaaat gaagttttaa atcaatctaa 15240
agtatatatg agtaaacttg gtctgacagt taccaatgct taatcagtga ggcacctatc 15300
tcagcgatct gtctatttcg ttcatccata gttgcctgac tccccgtcgt gtagataact 15360
acgatacggg agggcttacc atctggcccc agtgctgcaa tgataccgcg agacccacgc 15420
tcaccggctc cagatttatc agcaataaac cagccagccg 15460
<210>150
<211>3240
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>150
ccacctgacg tctaagaaac cattattatc atgacattaa cctataaaaa taggcgtatc60
acgaggcaga atttcagata aaaaaaatcc ttagctttcg ctaaggatga tttctggaat 120
tcgcggccgc atctagagtc tcgagttgac agctagctca gtcctaggta taatgctagc 180
ctcgaggaaa gaggagaaag gtctcaatgc gtaaaggcga agagctgttc actggtgtcg 240
tccctattct ggtggaactg gatggtgatg tcaacggtca taagttttcc gtgcgtggcg 300
agggtgaagg tgacgcaact aatggtaaac tgacgctgaa gttcatctgt actactggta 360
aactgccggt accttggccg actctggtaa cgacgctgac ttatggtgtt cagtgctttg 420
ctcgttatcc ggaccatatg aagcagcatg acttcttcaa gtccgccatg ccggaaggct 480
atgtgcagga acgcacgatt tcctttaagg atgacggcac gtacaaaacg cgtgcggaag 540
tgaaatttga aggcgatacc ctggtaaacc gcattgagct gaaaggcatt gactttaaag 600
aagacggcaa tatcctgggc cataagctgg aatacaattt taacagccac aatgtttaca 660
tcaccgccga taaacaaaaa aatggcatta aagcgaattt taaaattcgc cacaacgtgg 720
aggatggcag cgtgcagctg gctgatcact accagcaaaa cactccaatc ggtgatggtc 780
ctgttctgct gccagacaat cactatctga gcacgcaaag cgttctgtct aaagatccga 840
acgagaaacg cgatcatatg gttctgctgg agttcgtaac cgcagcgggc atcgcgcatg 900
gtatggatga actgtacaga tgataacgct gatagtgcta gtgtagatcg ctactagagc 960
caggcatcaa ataaaacgaa aggctcagtc gaaagactgg gcctttcgtt ttatctgttg1020
tttgtcggtg aacgctctct actagagtca cactggctca ccttcgggtg ggcctttctg1080
cgtttatata ctagaagcgg ccgctgcagg aattcaaaaa aagcaccgac tcggtgccac1140
tttttcaagt tgataacgga ctagccttat tttaacttgc tatttctagc tctaaaacgg1200
agacgagcgt ctccactagt attataccta ggactgagct agctgtcaag gatccagcat1260
atgcggtgtg aaataccgca cagatgcgta aggagaaaat accgcatcag gcgctcttcc1320
gcttcctcgc tcactgactc gctgcgctcg gtcgttcggc tgcggcgagc ggtatcagct1380
cactcaaagg cggtaatacg gttatccaca gaatcagggg ataacgcagg aaagaacatg1440
tgagcaaaag gccagcaaaa ggccaggaac cgtaaaaagg ccgcgttgct ggcgtttttc1500
cataggctcc gcccccctga cgagcatcac aaaaatcgac gctcaagtca gaggtggcga1560
aacccgacag gactataaag ataccaggcg tttccccctg gaagctccct cgtgcgctct1620
cctgttccga ccctgccgct taccggatac ctgtccgcct ttctcccttc gggaagcgtg1680
gcgctttctc atagctcacg ctgtaggtat ctcagttcgg tgtaggtcgt tcgctccaag1740
ctgggctgtg tgcacgaacc ccccgttcag cccgaccgct gcgccttatc cggtaactat1800
cgtcttgagt ccaacccggt aagacacgac ttatcgccac tggcagcagc cactggtaac1860
aggattagca gagcgaggta tgtaggcggt gctacagagt tcttgaagtg gtggcctaac1920
tacggctaca ctagaaggac agtatttggt atctgcgctc tgctgaagcc agttaccttc1980
ggaaaaagag ttggtagctc ttgatccggc aaacaaacca ccgctggtag cggtggtttt2040
tttgtttgca agcagcagat tacgcgcaga aaaaaaggat ctcaagaaga tcctttgatc2100
ttttctacgg ggtctgacgc tcagtggaac gaaaactcac gttaagggat tttggtcatg2160
agattatcaa aaaggatctt cacctagatc cttttaaatt aaaaatgaag ttttaaatca2220
atctaaagta tatatgagta aacttggtct gacagttacc aatgcttaat cagtgaggca2280
cctatctcag cgatctgtct atttcgttca tccatagttg cctgactccc cgtcgtgtag2340
ataactacga tacgggaggg cttaccatct ggccccagtg ctgcaatgat accgcgtgac2400
ccacgctcac cggctccaga tttatcagca ataaaccagc cagccggaag ggccgagcgc2460
agaagtggtc ctgcaacttt atccgcctcc atccagtcta ttaattgttg ccgggaagct2520
agagtaagta gttcgccagt taatagtttg cgcaacgttg ttgccattgc tacaggcatc2580
gtggtgtcac gctcgtcgtt tggtatggct tcattcagct ccggttccca acgatcaagg2640
cgagttacat gatcccccat gttgtgcaaa aaagcggtta gctccttcgg tcctccgatc2700
gttgtcagaa gtaagttggc cgcagtgtta tcactcatgg ttatggcagc actgcataat2760
tctcttactg tcatgccatc cgtaagatgc ttttctgtga ctggtgagta ctcaaccaag2820
tcattctgag aatagtgtat gcggcgaccg agttgctctt gcccggcgtc aatacgggat2880
aataccgcgc cacatagcag aactttaaaa gtgctcatca ttggaaaacg ttcttcgggg2940
cgaaaactct caaggatctt accgctgttg agatccagtt cgatgtaacc cactcgtgca3000
cccaactgat cttcagcatc ttttactttc accagcgttt ctgggtgagc aaaaacagga3060
aggcaaaatg ccgcaaaaaa gggaataagg gcgacacgga aatgttgaat actcatactc3120
ttcctttttc aatattattg aagcatttat cagggttatt gtctcatgag cggatacata3180
tttgaatgta tttagaaaaa taaacaaata ggggttccgc gcacatttcc ccgaaaagtg3240
<210>151
<211>1756
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>151
gaattccgga tgagcattca tcaggcgggc aagaatgtga ataaaggccg gataaaactt60
gtgcttattt ttctttacgg tctttaaaaa ggccgtaata tccagctgaa cggtctggtt 120
ataggtacat tgagcaactg actgaaatgc ctcaaaatgt tctttacgat gccattggga 180
tatatcaacg gtggtatatc cagtgatttt tttctccatt ttagcttcct tagctcctga 240
aaatctcgat aactcaaaaa atacgcccgg tagtgatctt atttcattat ggtgaaagtt 300
ggaacctctt acgtgccgat caacgtctca ttttcgccaa aagttggccc agggcttccc 360
ggtatcaaca gggacaccag gatttattta ttctgcgaag tgatcttccg tcacaggtat 420
ttattcggga tcctcgctca ctgactcgct acgctcggtc gttcgactgc ggcgagcgga 480
aatggcttac gaacggggcg gagatttcct ggaagatgcc aggaagatac ttaacaggga 540
agtgagaggg ccgcggcaaa gccgtttttc cataggctcc gcccccctga caagcatcac 600
gaaatctgac gctcaaatca gtggtggcga aacccgacag gactataaag ataccaggcg 660
tttccccctg gcggctccct cgtgcgctct cctgttcctg cctttcggtt taccggtgtc 720
attccgctgt tatggccgcg tttgtctcat tccacgcctg acactcagtt ccgggtaggc 780
agttcgctcc aagctggact gtatgcacga accccccgtt cagtccgacc gctgcgcctt 840
atccggtaac tatcgtcttg agtccaaccc ggaaagacat gcaaaagcac cactggcagc 900
agccactggt aattgattta gaggagttag tcttgaagtc atgcgccggt taaggctaaa 960
ctgaaaggac aagttttggt gactgcgctc ctccaagcca gttacctcgg ttcaaagagt1020
tggtagctca gagaaccttc gaaaaaccgc cctgcaaggc ggttttttcg ttttcagagc1080
aagagattac gcgcagacca aaacgatctc aagaagatca tctgtaggtc gttcgctcca1140
agctggaagg atcttcacct agatcctttt aaattaaaaa tgaagtttta aatcaatcta1200
aagtatatat gagtaaactt ggtctgacag aatttctgcc attcatccgc ttattatcac1260
ttattcaggc gtagcaacca ggcgtttaag ggcaccaata actgccttaa aaaaattacg1320
ccccgccctg ccactcatcg cagtactgtt gtaattcatt aagcattctg ccgacatgga1380
agccatcaca aacggcatga tgaacctgaa tcgccagcgg catcagcacc ttgtcgcctt1440
gcgtataata tttgcccatg gtgaaaacgg gggcgaagaa gttgtccata ttggccacgt1500
ttaaatcaaa actggtgaaa ctcacccagg gattggctga gacgaaaaac atattctcaa1560
taaacccttt agggaaatag gccaggtttt caccgtaaca cgccacatct tgcgaatata1620
tgtgtagaaa ctgccggaaa tcgtcgtggt attcactcca gagcgatgaa aacgtttcag1680
tttgctcatg gaaaacggtg taacaagggt gaacactatc ccatatcacc agctcaccgt1740
ctttcattgc catacg1756
<210>152
<400>152
000
<210>153
<400>153
000
<210>154
<400>154
000
<210>155
<400>155
000
<210>156
<400>156
000
<210>157
<400>157
000
<210>158
<400>158
000
<210>159
<400>159
000
<210>160
<400>160
000
<210>161
<400>161
000
<210>162
<400>162
000
<210>163
<400>163
000
<210>164
<400>164
000
<210>165
<400>165
000
<210>166
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>166
gggaaacggg ataatctgag20
<210>167
<211>405
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>167
gtgaatacaa cgctgtttcg atggccggtt cgtgtctact atgaagatac cgatgccggt60
ggtgtggtgt accacgccag ttacgtcgct ttttatgaaa gagcacgcac agagatgctg 120
cgtcatcatc atttcagtca acaggcgctg atggctgaac gcgttgcctt tgtggtacgt 180
aaaatgacgg tggaatatta cgcacctgcg cggctcgacg atatgctcga aatacagact 240
gaaataacat caatgcgtgg cacctctttg gttttcacgc aacgtattgt caacgccgag 300
aatactttgt tgaatgaagc agaggttctg gttgtttgcg ttgacccact caaaatgaag 360
cctcgtgcgc ttcccaagtc tattgtcgcg gagtttaagc agtga 405
<210>168
<211>496
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>168
gccatggcca gagcgcgtgg acgaggtcgt cgcgatctca agtccgaaat caacattgta60
ccgttgctgg acgtactgct ggtgctgttg ctgatcttta tggcgacagc gcccatcatc 120
acccagagcg tggaggtcga tctgccagac gctactgaat cacaggcggt gagcagtaac 180
gataatccgc cagtgattgt tgaagtgtct ggtattggtc agtacaccgt ggtggttgag 240
aaagatcgtc tggagcgttt accaccagag caggtggtgg cggaagtgtc cagccgtttc 300
aaggccaacc cgaaaacggt ctttctgatc ggtggcgcaa aagatgtgcc ttacgatgaa 360
ataattaaag cactgaactt gttacatagt gcgggtgtga aatcggttgg tttaatgacg 420
cagcctatct aaacatctgc gtttcccttg cttgaaagag agcgggtaac aggcgaacag 480
tttttggaaa ccgaga 496
<210>169
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>169
tggtattggt cagtacaccg20
<210>170
<211>415
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>170
gcatcgtgcc aatagccatg cgccggaagc cgtagtggaa ggggcgtcgc gtgcgatgcg60
tatttccatg aatcgtgaac ttgaaaatct ggaaacgcac attcctttcc tcggtacggt 120
tggctccatc agcccgtata ttggtctgtt tggtacggtc tgggggatca tgcacgcctt 180
tatcgccctc ggggcggtaa aacaagcaac actgcaaatg gttgcgcccg gtatcgcaga 240
agcgttgatt gcgacggcaa ttggtttgtt cgccgcaatt ccggcggtaa tggcttataa 300
ccgcctcaac cagcgcgtaa acaaactgga actgaattac gacaacttta tggaagagtt 360
taccgcgatt ctgcaccgcc aggcgtttac cgttagcgag agcaacaagg ggtaa415
<210>171
<211>474
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>171
acatctgcgt ttcccttgct tgaaagagag cgggtaacag gcgaacagtt tttggaaacc60
gagagtgtca aaggcaaccg aacaaaacga caagctcaag cgggcgataa ttatttcagc 120
agtgctgcat gtcatcttat ttgcggcgct gatctggagt tcgttcgatg agaatataga 180
agcttcagcc ggaggcggcg gtggttcgtc catcgacgct gtcatggttg attcaggtgc 240
ggtagttgaa cagtacaaac gtatgcaaag ccaggaatca agcgcgaagc gttctgatga 300
gcagcgcaag atgaaggagc agcaggctgc tgaagaactg cgtgagaaac aagcggctga 360
acaggaacgc ctgaagcaac ttgagaaaga gcggttagcg gctcaggagc agaaaaagca 420
ggctgaagaa gccgcaaaac aggccgagtt aaagcagaag caagcggaag aggc 474
<210>172
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>172
agaactgcgt gagaaacaag20
<210>173
<211>496
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>173
gccatggcca gagcgcgtgg acgaggtcgt cgcgatctca agtccgaaat caacattgta60
ccgttgctgg acgtactgct ggtgctgttg ctgatcttta tggcgacagc gcccatcatc 120
acccagagcg tggaggtcga tctgccagac gctactgaat cacaggcggt gagcagtaac 180
gataatccgc cagtgattgt tgaagtgtct ggtattggtc agtacaccgt ggtggttgag 240
aaagatcgtc tggagcgttt accaccagag caggtggtgg cggaagtgtc cagccgtttc 300
aaggccaacc cgaaaacggt ctttctgatc ggtggcgcaa aagatgtgcc ttacgatgaa 360
ataattaaag cactgaactt gttacatagt gcgggtgtga aatcggttgg tttaatgacg 420
cagcctatct aaacatctgc gtttcccttg cttgaaagag agcgggtaac aggcgaacag 480
tttttggaaa ccgaga 496
<210>174
<211>500
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>174
tcgcgatgtt gactgttccg acggtcaaca tcaggcaccg gttgccacgg ggttctggta60
gttttgtgta ttttagtttg ttaacattct gctaaattat cgtgggccat cggtccagat 120
aagggagata tgatgaagca ggcattacga gtagcatttg gttttctcat actgtgggca 180
tcagttctgc atgctgaagt ccgcattgtg atcgacagcg gtgtagattc cggtcgtcct 240
attggtgttg ttcctttcca gtgggcgggg cctggtgcgg cacctgaaga tattggcggc 300
atcgttgctg ctgacttgcg taacagcggt aaatttaatc cgttagatcg tgctcgtttg 360
ccacagcagc cgggtagtgc gcaggaagta caaccagctg catggtccgc gctgggcatt 420
gacgctgtgg ttgtcggtca ggtcactccg aatccggatg gttcttacaa tgttgcttat 480
caacttgttg acactggcgg 500
<210>175
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>175
agtcaccgta gaaggtcacg20
<210>176
<211>473
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>176
tggtcgcagt aacaataccg aaccgacctg gttcccggac agccagaacc tggcatttac60
ttctgaccag gccggtcgtc cgcaggttta taaagtgaat atcaacggcg gtgcgccaca 120
acgtattacc tgggaaggtt cgcagaacca ggatgcggat gtcagcagcg acggtaaatt 180
tatggtaatg gtcagctcca acggtgggca gcagcacatt gccaaacaag atctggcaac 240
gggaggcgta caagttctgt cgtccacgtt cctggatgaa acgccaagtc tggcacctaa 300
cggcactatg gtaatctaca gctcttctca ggggatggga tccgtgctga atttggtttc 360
tacagatggg cgtttcaaag cgcgtcttcc ggcaactgat ggacaggtca aattccctgc 420
ctggtcgccg tatctgtgat aataattaat tgaatagtaa aggaatcatt gaa473
<210>177
<211>471
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>177
gagaattgca tgagcagtaa cttcagacat caactattga gtctgtcgtt actggttggt60
atagcggccc cctgggccgc ttttgctcag gcaccaatca gtagtgtcgg ctcaggctcg 120
gtcgaagacc gcgtcactca acttgagcgt atttctaatg ctcacagcca gcttttaacc 180
caactccagc aacaactctc tgataatcaa tccgatattg attccctgcg tggtcagatt 240
caggaaaatc agtatcaact gaatcaggtc gtggagcggc aaaagcagat cctgttgcag 300
atcgacagcc tcagcagcgg tggtgcagcg gcgcaatcaa ccagcggcga tcaaagcggt 360
gtggcggcat caacgacgcc gacagctgat gctggtactg cgaatgctgg cgcgccggtg 420
aaaagcggtg atgcaaacac ggattacaat gcagctattg cgctggtgca g471
<210>178
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>178
acgttcaggc tgctaaagat20
<210>179
<211>482
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>179
aatacttccc ggatgccacc atcctggcac tgaccaccaa cgaaaaaacg gctcaccagt60
tagtactgag caaaggtgtt gtgccgcagc tggttaaaga gatcacatct actgatgact 120
tctaccgtct gggtaaagaa ctggctctgc agagcggtct ggcacacaaa ggtgatgtcg 180
tagttatggt ttctggtgca ctggtaccga gcggcactac taacaccgca tctgttcacg 240
tcctgtaata ttgcttttgt gaattaattt gtatatcgaa gcgccctgat gggcgctttt 300
tttatttaat cgataaccag aagcaataaa aaatcaaatc ggatttcact atataatctc 360
actttatcta agatgaatcc gatggaagca tcctgttttc tctcaatttt tttatctaaa 420
acccagcgtt cgatgcttct ttgagcgaac gatcaaaaat aagtgccttc ccatcaaaaa 480
aa482
<210>180
<211>438
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>180
cctgtgaagt gaaaaatggc gcacattgtg cgccattttt ttgcctgcta tttaccgcta60
ctgcgtcgcg cgtaacatat tcccttgctc tggttcccca ttctgcgctg actctactga 120
aggcgcattg ctggctgcgg gagttgctcc actgctcacc gcaaccggat accctgcccg 180
acgatacaac gctttatcga ctaacttctg atctacagcc ttattgtctt taaattgcgt 240
aaagcctgct ggcagcgtgt acggcattgt ctgaacgttc tgctgttctt ctgccgatag 300
tggtcgatgt acttcaacat aacgcatccc gttaggttcc acggaatatt tcaccggttc 360
gttgatcact ttcaccggtg ttcccgtccg cacgctggag aataaagctt taatatccgg 420
tgcattcatg cgaataca 438
<210>181
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>181
caaaccgttc ctctacaccg20
<210>182
<211>417
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>182
gacagccagg ccatttgctc ccgctcaccg agggacatcg acgggcgtgg aaatgaccgc60
tgtaatgtgc gactggcggg aaacgccaac ataatgtgat gacgctgcgg cagttcacga 120
ctccacggta acaacgtgtt agccagccgc tgcacatcaa caatccgccc atctttgata 180
atgtcgttct ccagtggcaa ccgccaccag cggtgcaata agcattcttt tgcgctccgc 240
acgatagcaa ccgctaccgc ttcttgctgt tgtaaatgca aaccaatttg ccagttctta 300
aatgccattg tgatgatctc cttatcaccc gtcactctga cgggtatatc aatgcgtctg 360
gcttgccttt atactaccgc gcgtttgttt ataaactgcc caaatgaaac taaatgg417
<210>183
<211>460
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>183
ttaaaaaggc gcttcggcgc ctttttcagt ttgctgacaa agtgcacttg tttatgccag60
atgcgggtga acgcgttatc cggccaacaa aaaattgaaa attcaataaa ttgcaggaac 120
ttcgtaagcc tgataagcgt tgtgcatcag gcaaacttca cgcatttaca ctcgcccctg 180
ccctttcaac cattcgcgca cgaggaacag cgcgctgaca ttacgcgctt cgttgaagtc 240
aggggcttcc agcaaatcca tcatatgcgc cagcggccag cgcacctgtg gtagcggctc 300
tggctcatcg ccttccagtg attccgggta gagatcttgc gctaccacga tattcatttt 360
gctggaaaag taagacggtg ccatgctgag cttcttcaaa aaagtcagat cgttcgcacc 420
aaatccaacc tcttctttta gctcgcggtt agcggcttca 460
<210>184
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>184
cctgggactc atggccggat20
<210>185
<211>516
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>185
cccagccctt gtcgatgccg cgttcaggct agcttcgttc aaatccatta aagatatcag60
actactctca ggtacgtgag taaggtaaaa cccagttcca acgccaatat ccagatggtt 120
gttacctaaa tgttccagaa agtgtggaag aaggtgttcc tttgtaggac atccccatgc 180
aagccgattt gatactccca aaacccacca gtcataaagc tttagggtaa gtggtgtgta 240
aattttagcc ccatcatctg tgtttttttt cattagtttc accgtgttat agttttattt 300
gtgaattaaa tcaattatag agatgaatta caaggggtta aatgatgcca cggcataacg 360
aatttgaata tattgggtct tgtgttgcgg taagaggtta cacgccagga cgcaggttaa 420
atgcttacaa tattagggga tattgtttgc ttttaacggt aaacaaattc cccggggcta 480
tacaatacca ccggggagaa aatctattta acgttg 516
<210>186
<211>623
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>186
aaaaggtctc cattcaatcg ttttaatgat tgagtatgta ttttttatat ctaacttaat60
gagtcaattg tatattgccc cactgtttat attttgttta acattgaatt tttgtcacaa 120
tgcgctgtca gttttttagt ttaattgtct gtttgtgttt tgtattgtgg ttttatgggc 180
taattatttt tttctttgtg gagtttttat ctattcaagt gccatgcctt tagatgcata 240
ttagcgataa tagcatgagg tttatcctca attgctgtgt tttttagtat aaaaaagagg 300
aacaaaactg agacacataa ggcctcgcaa tggcttgcaa ggttttacat attttgaggt 360
ggtggaacgt gtgaacgcag agatggcgcg gtaatttgtt gatttaaaat gtcgttcttg 420
gagtttctaa gtcgtgggct acaggttcga atcctgcagg gcgagccatt tcctcgccat 480
ttatttgttc ccttttacaa attacaccat cactttgttg tcgcggtttt gctaaataat 540
tggaacacgt cagacgaata aaatgaaaac agaaattctg ctaaacaatt catcttgcca 600
ggattttgat taggaaagtg aaa 623
<210>187
<400>187
000
<210>188
<400>188
000
<210>189
<400>189
000
<210>190
<400>190
000
<210>191
<400>191
000
<210>192
<400>192
000
<210>193
<400>193
000
<210>194
<400>194
000
<210>195
<400>195
000
<210>196
<400>196
000
<210>197
<400>197
000
<210>198
<400>198
000
<210>199
<400>199
000
<210>200
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>200
tgtgtcgtcg ttaactcgcc20
<210>201
<211>409
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>201
gccgcgcacg ctgatttttt atgaatctac ccaccgtctg ttagatagcc tggaagatat60
cgttgcggta ttaggcgaat cccgttacgt ggttctggcg cgtgagctaa ccaaaacctg 120
ggaaaccatt cacggcgcgc ccgttggcga gctgctggcg tgggtaaaag aagatgaaaa 180
ccgtcgcaaa ggcgaaatgg tgctgattgt cgaaggccat aaagcgcagg aaaatgactt 240
gcctgctgat gccctgcgca cgctggcgct gctacaggca gaactgccgc tgaaaaaagc 300
ggcggcgctg gccgcagaaa ttcacggcgt gaagaaaaat gcgctgtata agtatgcgct 360
ggagcagcag ggagagtgat tacaggcgtc gcagcaggaa tagcgctac 409
<210>202
<211>405
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>202
tcacgcatgt gaaatggtta tgcagccata aaaatgaagc cgggatagcg gtggagactt60
tagccgtgag aaaacgttct gtcgcatcct gttttccttc gccagtcacc agtaacaaaa 120
cttcgcgggc attgagaata tccttcaggc ctaaggtgat cccacgagtc acgggacggt 180
ccgcggtttt taacatctca tgttgctgtg ttctggcatc aagttgactg atatggcagg 240
ctggttgcag gctttctccc ggttcgttca gcccaagatg accgtttttc cccaatccga 300
gaacgcataa atccagaccg cctttgcgcg caatcaggtt cgttacccgt tcgcactctg 360
tctcatttat ctcttcggag cgaaagctga tgagctggtc ttcac 405
<210>203
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>203
cgctttttga aaatacgcaa20
<210>204
<211>477
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>204
accagccgag attcaacagg ttgttgcccg tcaggcgacc aatcaaaaac tccattgaga60
tgtagttaac atgtcgctga ttcgccactg gcttggcgaa tggctgagca cgcagcattt 120
cggccagtgc ttcgctcact gccagccacc actggcgagg agtcatttca gccgcagaat 180
ttaagccata acgctgccac tgacgtgaaa gcgcttcctg aaattgctta tcgttaaaaa 240
taggttgtga cataggagtt ccacttttct tagattttca acacaacgtt atcgctagtt 300
tgccaggctc gatgttgacc ttcctcatcc tgcgggggat taggcaggga ggagttgcgg 360
ggatgagcaa ggaaatgtga tctcgaccac ttaaagctag tgcaatccac aggattagca 420
gcaaatcaat gccataccgc gcagaaaatc tgtatctaag tgcaaaaaat ggccgtt477
<210>205
<211>456
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>205
cgtattttca aaaagcggaa ggtaactcta taaattaagt aaaggagtga aacagtttca60
caagtaaaat atcaagtgtg ctccatctca ttcttaatag atttattaag atcatctttt 120
tagatggcac tttcatcaga aatgaagaag aaacccttgc ttaaatgaaa ctgatgaaca 180
taaggaaaat accgtacaac ccagtattca cgctggatca gcgtcgtttt aggtgagttg 240
ttaataaaca tttggaattg tgacacagtg caaattcaga cacataaaaa aacgtcatcg 300
cttgcattag aaaggtttct ggccgacctt ataaccatta attacgaagc gcaaaaaaaa 360
taatatttcc tcattttcca cagtgaagtg attaactatg ctgattccgt caaaattaag 420
tcgtccggtt cgactcgacc ataccgtggt tcgtga 456
<210>206
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>206
ccgtgacgtt atccgcacca20
<210>207
<211>357
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>207
acgatgacct gatgcgtccg gacttcaaca acgatgacta cgctattgct tgctgcgtaa60
gcccgatgat cgttggtaaa caaatgcagt tcttcggtgc gcgtgcaaac ctggcgaaaa 120
ccatgctgta cgcaatcaac ggcggcgttg acgaaaaact gaaaatgcag gttggtccga 180
agtctgaacc gatcaaaggc gatgtcctga actatgatga agtgatggag cgcatggatc 240
acttcatgga ctggctggct aaacagtaca tcactgcact gaacatcatc cactacatgc 300
acgacaagta cagctacgaa gcctctctga tggcgctgca cgaccgtgac gttatcc357
<210>208
<211>442
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>208
gtgacgctat cccgactcag tctgttctga ccatcacttc taacgttgtg tatggtaaga60
aaactggtaa caccccagac ggtcgtcgtg ctggcgcgcc gttcggaccg ggtgctaacc 120
cgatgcacgg tcgtgaccag aaaggtgctg tagcgtctct gacttccgtt gctaaactgc 180
cgtttgctta cgctaaagat ggtatctcct acaccttctc tatcgttccg aacgcactgg 240
gtaaagacga cgaagttcgt aagaccaacc tggctggtct gatggatggt tacttccacc 300
acgaagcatc catcgaaggt ggtcagcacc tgaacgttaa cgtgatgaac cgtgaaatgc 360
tgctcgacgc gatggaaaac ccggaaaaat atccgcagct gaccatccgt gtatctggct 420
acgcagtacg tttcaactcg ct442
<210>209
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>209
gttatcgtga tgcgcattct20
<210>210
<211>457
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>210
ttccgcagcc agcgattatc gcgccgcagc gcaacgcatt ctgccgccgt tcctgttcca60
ctatatggat gggggggcat attctgaata cacgctgcgc cgcaacgtgg aagatttgtc 120
agaagtggcg ctgcgccagc gtattctgaa aaacatgtct gacttaagcc tggaaacgac 180
gctgtttaat gagaaattgt cgatgccggt ggcgctaggt ccggtaggtt tgtgtggcat 240
gtatgcgcga cgcggcgaag ttcaggctgc caaagcagca gatgcgcatg gcattccgtt 300
tactctctcg acggtttccg tttgcccgat tgaagaagtg gctccggcta tcaaacgtcc 360
gatgtggttc cagctttatg tgctgcgcga tcgcggcttt atgcgtaacg ccctggagcg 420
agcaaaagcc gcgggttgtt cgacgctggt tttcacc457
<210>211
<211>441
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>211
atccgcgatt tctgggatgg cccgatggtg atcaaaggga tcctcgatcc ggaagatgcg60
cgcgatgcag tacgttttgg tgctgatgga attgtggttt ctaaccacgg tggccgccag 120
ttagatggcg tactctcttc tgctcgtgca ctgcctgcta ttgcggatgc ggtgaaaggt 180
gatatcgcca ttctggcgga tagcggaata cgtaacgggc ttgatgtcgt gcgtatgatt 240
gcgctcggtg ccgacaccgt actgctgggt cgtgctttcc tgtatgcact ggcaacagcg 300
ggccaggcgg gtgtagctaa tctgctaaat ctgatcgaaa aagagatgaa agtggcgatg 360
acgctgactg gcgcgaaatc gattagcgaa attacgcaag attcgctggt gcaggggctg 420
ggtaaagagt tgcctgcggc a 441
<210>212
<211>20
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>212
taacacccag cccgatgccc20
<210>213
<211>357
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>213
ttctgcatag caggtgaggc aaatgagatt tattcgccac tacccagtat ggatgagatc60
tgaaaaaggg agaggaaaat tgcccggtag ccttcactac cgggcgcagg cttagatgga 120
ggtacggcgg tagtcgcggt attcggcgtg ccagaaatta tcatcaatgg cctgttgcag 180
agcttcggca gacgttttca ccgccacgcc ttgctgctgc gccattttgc caaccgcaaa 240
ggcaattgcg cgggagactt tctgaatatc tttcagttcc ggcagtacca gaccttcgcc 300
gttcaggacc agcggcgaat actgagcaag catttcactt gccgacatca gcatctc357
<210>214
<211>544
<212>DNA
<213>Artificial Sequence
<220>
<223>Synthetic
<400>214
gagattttcg cgcttctgca ccagtttggt ctggaaaggc agcaggttcg gcatcttgtc60
ggtcagcagg ccaaagcgat cgaccataaa gactttctgc cgcgccgctt cctcgcttaa 120
tccttcgcgc tgggtctggg cgatgatcat ttcggcaatg ccgcatcccg ctgaacctgc 180
gccaaggaag acgatttttt tctcgcttaa ctgaccacct gccgcgcggc tggctgcgat 240
cagtgtgccg actgttaccg ccgcggtgcc ctgaatgtca tcgttaaaag aacaaatttc 300
attgcgatag cggttaagta acggcatcgc atttttttgt gcgaagtctt caaactgcaa 360
cagtacgtcc ggccagcgtt gtttcacagc ctggataaat tcatcaacaa attcatagta 420
ttcgtcgtca gtgatacgcg gattacgcca gcccatatac agcggatcgt taagcagctg 480
ttggttgttg gtgccgacat ccagcaccac cggaagggta taggccggac tgatgccgcc 540
acag544
- 用于癌症免疫疗法的受体选择性类视黄醇和rexinoid化合物和免疫调节剂
- 硝羟喹啉和其类似物与化学疗法和免疫疗法在癌症治疗中的组合用途
- 用于癌症免疫疗法的CRISPR-CPF1相关方法、组合物和组分
- 用于治疗癌症的NOTCH和CDK4/6抑制剂的组合疗法
- 用于治疗癌症的AMUC_1100和免疫检查点调节剂的组合疗法
- 用于治疗癌症的AMUC_1100和免疫检查点调节剂的组合疗法
