浙江楠核基因组SSR分子标记的多态性引物及其应用
文献发布时间:2023-06-19 13:45:04
技术领域
本发明属于林业分子生物学技术领域,具体涉及浙江楠核基因组SSR分子标记的多态性引物以及其在浙江楠群体遗传结构和遗传多样性分析中的应用。
背景技术
浙江楠(Pheobe chekiangensis C.B.Shang),浙江楠(Phoebe chekiangensisC.B.Shahg)为樟科(Lauraceae)楠属(Phobe)植物,是中国著名植物分类学家向其柏教授于20世纪60年代在浙江天目山、龙塘山研究发现的新种。浙江楠主要分布于我国南方的浙江、江西、安徽、福建等地。浙江楠以伴生种零星分布于我国中亚热带地区的以壳斗科树种为建群种的天然常绿阔叶林中,由于长期人为破坏,仅在人为干扰少的深山峡谷旁有以浙江楠为建群种的群落存在。在1999年8月,浙江楠被国务院列为国家II级重点保护野生植物(《国家重点保护野生植物名录》第一批)。天然浙江楠资源蕴涵宝贵地基因资源,但其遗传变异水平及其亲缘地理关系鲜有报道。鉴于此,亟需开展浙江楠天然种群遗传多样性和遗传结构研究,为遗传保护策略的制定奠定基础。
简单重复序列标记(simple sequence repeats,SSR),又称为微卫星标记,是一类由几个核苷酸(1~6个)为重复单位组成的长达几十个核苷酸的重复序列,长度较短,且广泛均匀分布于真核生物基因组中。研究发现,微卫星中重复单位的数目存在高度变异,这些变异表现为微卫星数目的整倍性变异或重复单位序列中的序列有可能不完全相同,因而造成多个位点的多态性。目前,该技术广泛用于群体遗传多样性研究、品种鉴定等领域。
目前有关浙江楠遗传多样性地研究鲜有报道,要了解浙江楠真实的遗传水平多样性必须有足够的有代表性的群体样本。在现有研究中只是针对局部地区的群体,即浙江和江西的6个群体,难以全面的揭示浙江楠遗传水平的多样性。
发明内容
针对现有技术存在的上述问题,本发明所要解决的技术问题在于提供浙江楠核基因组SSR分子标记的多态性引物;本发明所要解决的另一技术问题在于提供浙江楠核基因组SSR分子标记的多态性引物在浙江楠群体遗传结构和遗传多样性分析以及分子标记辅助育种中的应用。
为了解决上述技术问题,本发明所采用的技术方案如下:
本申请基于现有的浙江楠转录组测序数据,采用Primer 3.0设计了96对SSR引物,引物设计原则如下:引物长度18~25bp之间,最适为20bp;退火温度(Tm)50~60℃,正、反向引物退火温度差小于5℃;GC含量为40%~60%,最适50%;经8%聚丙烯酰胺凝胶进行电泳筛选和毛细管电泳技术,从中挑选了15对浙江楠核基因组SSR分子标记的多态性引物,其引物核苷酸序列依次如SEQ ID NO.1-SEQ ID NO.30所示;
具体如下:
Pc06_p6_5:F:5′-AGCCCCTTAACCCTAACCCT-3′;
R:5′-CGGGTAGTTGTTGTGCCTCT-3′;
Pc10_p6_5:F:5′-CGAGATATTTGAGATTTGTAGGCCA-3′;
R:5′-ATGCCAATGCGATCGAAAGC-3′;
Pc13_p6_5:F:5′-TGCAAAAACCCCCTCCACTT-3′;
R:5′-TCGTAGACGAAAACCCTGCC-3′;
Pc14_p6_5:F:5′-GGGAGGCATCTCTTACTCGC-3′;
R:5′-CCACGGGAGATACCACTTGG-3′;
Pc16_p6_5:F:5′-CTAGTCGGAAGCAGAGCTCG-3′;
R:5′-ACCATTCCCCAAACAGTACCC-3′;
Pc17_p6_5:F:5′-TCTGAGGCCATGTTTCGGAC-3′;
R:5′-GCGTTTGTTTAGGTCTCGCC-3′;
Pcl8_p6_7:F:5′-TGCAAAAACCCCCTCCACTT-3′;
R:5′-TCGTAGACGAAAACCCTGCC-3′;
Pc19_p6_5:F:5′-GGACCTCTCTACGGAGTTCCA-3′;
R:5′-GCCATTCATTTCTGGTGGGC-3′;
Pc24_p6_7:F:5′-GCTTCTTCTTCGCATTCCCC-3′;
R:5′-GCCGCCGCCTATTCATTTCT-3′;
Pc28_p6_5:F:5′-AGTGGTCTGAATCACCAAACCA-3′;
R:5′-TGGGTTGGAACTTGGAAGCA-3′;
Pc45_p5_5:F:5′-GGGAAAAGACAGACAGGGCA-3′;
R:5′-CAGGCCCTTTCACCTTTTGC-3′;
Pc71_p5_5:F:5′-TCTTATGGTGAGGTCTCCAATGT-3′;
R:5′-GCAAGATGCGCAGATTTCTCT-3′;
Pc73_p5_5:F:5′-ATCATGGTCTTTGTCGCCGT-3′;
R:5′-AGTCGGATGATTGTCAATCCG-3′;
Pc81_p4_8:F:5′-GCTCCAACATGGGATTCACG-3′;
R:5′-GGATCAGCTTTGTGAGTTCTGC-3′;
Pc96_p3_8:F:5′-CCCTCAACAAGGCCTCATGT-3′;
R:5′-GTGGGATCGAGAGGCTGATG-3′。
所述的浙江楠核基因组SSR分子标记的多态性引物在浙江楠群体遗传结构和遗传多样性分析以及浙江楠分子标记辅助育种中的应用,包括以下步骤:
1)提取浙江楠基因组DNA;
本申请所选取的浙江楠来源于浙江楠自然分布区的10个群体,分别为江西德兴、江西婺源、江西石城、江西庐山、浙江杭州、浙江宁波、浙江松阳、湖北武汉、安徽祁门、湖南武冈;
2)用SEQ ID NO.1-SEQ ID NO.30所示的引物对步骤1)提取的基因组DNA进行PCR扩增,得到扩增产物;
其中,PCR扩增的体系为:2×TSINGKE Master Mix,7.5μl;上下游引物10μl,各1μl;gDNA 30ng/L,1μl;ddH2O 4.5μl;总反应体系为15μl;
PCR扩增的程序为:94℃预变性5min;94℃变性30s,57℃退火30s,72℃延伸30s,35个循环;72℃延伸5min;
3)对步骤2)得到的扩增产物进行聚丙烯酰胺凝胶电泳和/或毛细管电泳;
4)数据分析核基因组SSR位点多态性,进行浙江楠群体遗传结构和遗传多样性分析以及分子标记辅助育种。
相比于现有技术,本发明的有益效果为:
1)SRR分子标记数量丰富、覆盖整个基因组,多态性高;
2)本申请采用了毛细血管电泳,与传统的凝胶电泳相比,分辨率高、准确、高效快速,能够实现自动化操作;
3)本申请参试的10个浙江楠群体来自我国浙江楠主要分布的5个省份,覆盖度广,能够更全面的揭示我国浙江楠的遗传多样性和遗传结构。
附图说明
图1为部分SSR引物筛选的聚丙烯凝胶电泳图;
图2为SSR多态性引物Pc06的部分群体的毛细血管电泳图;
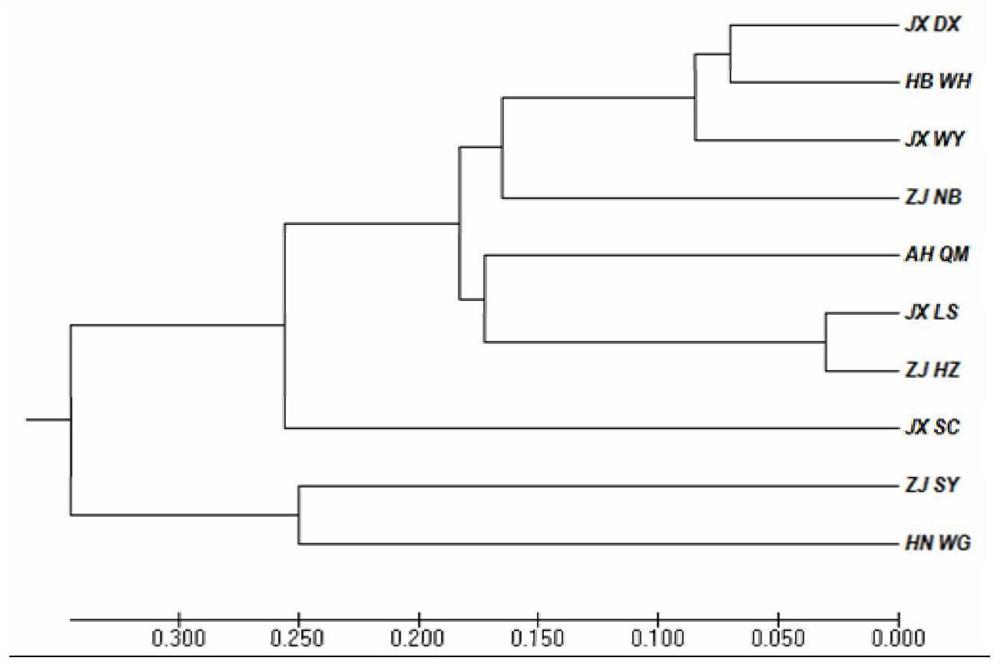
图3为10个种群的UPGMA聚类结果图;
图4为10个种群的175个个体的UPGMA聚类结果图。
具体实施方式
下面结合具体实施例对本发明进一步进行描述。
实施例1
1、供试材料
供试材料来源于浙江楠自然分布区的10个群体,分别为江西德兴、江西婺源、江西石城、江西庐山、浙江杭州、浙江宁波、浙江松阳、湖北武汉、安徽祁门、湖南武冈。选择古浙江楠,每个个体采集8-10片成熟叶片,硅胶干燥保存。每个群体样本量见表1。
表1 10个浙江楠群体采样信息
2、浙江楠总DNA的提取与检测
浙江楠叶组织基因组DNA的提取采用植物基因组DNA快速抽提试剂盒(B518231,上海生工),具体步骤详见说明书。
取少量提取好的浙江楠叶总DNA,用EPPENDORF核酸蛋白测定仪检测其含量及纯度;同时利用1%琼脂糖凝胶电泳检测DNA质量,通过观察条带是否弥散来判断DNA是否降解。得到的DNA用TE Buffer稀释至30ng/L,-20℃冰箱保存。
3、SSR引物的扩展与筛选
基于现有的浙江楠转录组测序数据,采用Primer 3.0(http://primer3.ut.ee/)设计了96对SSR引物,引物设计原则如下:引物长度18~25bp之间,最适为20bp;退火温度(Tm)50~60℃,正、反向引物退火温度差小于5℃;GC含量为40%~60%,最适50%;预计PCR产物长度范围在150~300bp之间。所有引物均由生工生物工程(上海)股份有限公司合成。经8%聚丙烯酰胺凝胶进行电泳筛选(图1),共获得了多对特异性较好且多态性较高的SSR引物,从中挑选了15对多态性较好的SSR引物,见表2。
表2浙江楠核基因组SSR引物序列信息
PCR反应体系:2×TSINGKE Master Mix(blue)(背景擎科生物科技有限公司),7.5μl;上下游引物10μl,各1μl;gDNA 30ng/L,1μl;ddH2O 4.5μl。总反应体系为15μl。
PCR扩增程序:94℃预变性5min;94℃变性30s,57℃退火30s,72℃延伸30s,35个循环;72℃延伸5min。
4、毛细管电泳
用于毛细血管电泳单位15对荧光引物信息见表3。将HiDi与GS500的内标按130∶1混合,配成mix。用国产96孔反应板分装mix,每个孔中加入10ul mix。对应着在96孔板中加入0.5ul样品模板,离心到4000rpm即停。用金属浴加热器将混合板95℃加热预变性5分钟,拿出后立即放入-20℃。冷却后拿出,4000rpm离心,解冻、混匀。上3730测序仪进行毛细管电泳。
表3 15对荧光引物信息
5、数据分析
利用GenAlEx version6.501软件计算SSR位点和群体的各项遗传多样性指标,包括观测等位基因(Na)、有效等位基因(Ne)、香农指数(I)、多态性信息指数(PIC)、观测杂合度(Ho)、期望杂合度(He)、群体间和群体内的变异、分化并进行显著性检验等。
基于贝叶斯模型的群体聚类方法,本实施例的群体遗传结构在SSRUCTUREversion 2.3.3软件中进行。采用的马尔科夫链(Markov Chain Monte Carlo,MCMC)方法可以预设群体分组(K)同时根据等位基因频率对个体进行计算、抽样和分组。参数设置:设K值范围为1-10,每个K值进行20次独立的runs,每个循环的重复抽样次数设置为100,000次。最终,在STRUCTURE HARVESTER(http://taylor0.biology.ucla.edu/STRuctureHarvester/)网站中,基于Evanno et al(2005)的方法,计算最适K值。
基于该标准遗传距离采用非加权组平均法(unweighted 3ir-group method witharithmetic means,UPGMA)建立个体的聚类树。具体方法:在populations-1_2_30软件中构建UPGMA树,数值设置为1000。
(一)核基因组SSR引物多态性分析
利用15对荧光SSR引物,通过毛细管电泳对175个浙江楠进行了检测,共检测出45.7个等位基因,每对引物扩增的等位基因数为2(PC13)~5(PC19),平均每各位点等位基因数目为3.047。有效等位基因总数为35.563,数值变化范围为1.310(PC18)~3.678(PC19),平均每个位点有效等位基因数目为2.371。香农指数(I)的数值范围为0.333(PC18)-1.377(PC19),平均值0.828。多态信息含量(PIC)的数值范围为0.323(PC18)-0.844(PC19),平均值0.6285,15对引物均具有较高的多态信息(PIC>0.25),见表4。
表4 SSR位点多样性检测
注:Na:观测等位基因,Ne:有效等位基因,I:香农指数,PIC:多态信息指数,Ho:观测杂合度,He:期望杂合度,PHWE:哈迪温伯格平衡。*:P<0.05,**:P<0.01,***:P<0.001,NS:P>0.05;Frequency of null alleles:无效等位基因频率。
(二)基于核基因组的浙江楠群体多样性分析
利用15对SSR引物对10个浙江楠群体进行多样性分析,结果表明不同的群体遗传多样性具有较大的差异(表5)。各参试群体的香农系数(I)变化在0.449(ZJ_NB)-1.164(JX_DX)之间,平均为0.828。观测杂合度(Ho)最小的是0.410(HN_WG),最大的是(AH_QM),平均0.621。期望杂合度最小的是0.297(ZJ_NB),最大的是0.616(JX_DX),平均0.478。两个指标均显示遗传多样性最高的群体是江西德兴(JX_DX)。从分子方差分析(AMOVA)结果可知(表5),种群内遗传变异较大,占变异总量的76%,种群间的遗传变异仅占24%,表明浙江楠种质的遗传变异主要来自种群内。
表5浙江楠种质群体的遗传多样性
注:Na:观测等位基因,Ne:有效等位基因,I:香农指数,PIC:多态信息指数,Ho:观测杂合度,He:期望杂合度。
(三)基于核基因组SSR标记的浙江楠群体聚类分析
为了解群体间遗传关系,在Popgen32中对群体间的遗传距离(Nei,1983)进行计算。10个野生群体的遗传距离差异很大,介于0.056-0.831之间。其中JS_LS和ZJ_HZ间的遗传距离最小,为0.056;ZJ_SY和JX_SC间的遗传距离最大,为0.831。基于Nei的遗传距离,我们分别构建了群体(图3)和个体(图4)的UPGMA树,进行聚类分析。种群聚类中,HN_WG、ZJ_SY和JX_SC 3个种源与其它种源间遗传距离均较大,分别单独聚为一组,其余7个种源全部聚类一组。分为基于个体的UPGMA图显示出组间和群体间的个体存在着较多的交叉混合,表明浙江楠群体内存在较多的遗传变异。
序列表
<110> 江西省林业科学院
<120> 浙江楠核基因组SSR分子标记的多态性引物及其应用
<130> 1
<160> 30
<170> SIPOSequenceListing 1.0
<210> 1
<211> 20
<212> DNA
<213> Pc06_p6_5_F(Artificial)
<400> 1
agccccttaa ccctaaccct 20
<210> 2
<211> 20
<212> DNA
<213> Pc06_p6_5_R(Artificial)
<400> 2
cgggtagttg ttgtgcctct 20
<210> 3
<211> 25
<212> DNA
<213> Pc10_p6_5_F(Artificial)
<400> 3
cgagatattt gagatttgta ggcca 25
<210> 4
<211> 20
<212> DNA
<213> Pc10_p6_5_R(Artificial)
<400> 4
atgccaatgc gatcgaaagc 20
<210> 5
<211> 20
<212> DNA
<213> Pc13_p6_5_F(Artificial)
<400> 5
tgcaaaaacc ccctccactt 20
<210> 6
<211> 20
<212> DNA
<213> Pc13_p6_5_R(Artificial)
<400> 6
tcgtagacga aaaccctgcc 20
<210> 7
<211> 20
<212> DNA
<213> Pc14_p6_5_F(Artificial)
<400> 7
gggaggcatc tcttactcgc 20
<210> 8
<211> 20
<212> DNA
<213> Pc14_p6_5_R(Artificial)
<400> 8
ccacgggaga taccacttgg 20
<210> 9
<211> 20
<212> DNA
<213> Pc16_p6_5_F(Artificial)
<400> 9
ctagtcggaa gcagagctcg 20
<210> 10
<211> 21
<212> DNA
<213> Pc16_p6_5_R(Artificial)
<400> 10
accattcccc aaacagtacc c 21
<210> 11
<211> 20
<212> DNA
<213> Pc17_p6_5_F(Artificial)
<400> 11
tctgaggcca tgtttcggac 20
<210> 12
<211> 20
<212> DNA
<213> Pc17_p6_5_R(Artificial)
<400> 12
gcgtttgttt aggtctcgcc 20
<210> 13
<211> 20
<212> DNA
<213> Pc18_p6_7_F(Artificial)
<400> 13
tgcaaaaacc ccctccactt 20
<210> 14
<211> 20
<212> DNA
<213> Pc18_p6_7_R(Artificial)
<400> 14
tcgtagacga aaaccctgcc 20
<210> 15
<211> 21
<212> DNA
<213> Pc19_p6_5_F(Artificial)
<400> 15
ggacctctct acggagttcc a 21
<210> 16
<211> 20
<212> DNA
<213> Pc19_p6_5_R(Artificial)
<400> 16
gccattcatt tctggtgggc 20
<210> 17
<211> 20
<212> DNA
<213> Pc24_p6_7_F(Artificial)
<400> 17
gcttcttctt cgcattcccc 20
<210> 18
<211> 20
<212> DNA
<213> Pc24_p6_7_R(Artificial)
<400> 18
gccgccgcct attcatttct 20
<210> 19
<211> 22
<212> DNA
<213> Pc28_p6_5_F(Artificial)
<400> 19
agtggtctga atcaccaaac ca 22
<210> 20
<211> 20
<212> DNA
<213> Pc28_p6_5_R(Artificial)
<400> 20
tgggttggaa cttggaagca 20
<210> 21
<211> 20
<212> DNA
<213> Pc45_p5_5_F(Artificial)
<400> 21
gggaaaagac agacagggca 20
<210> 22
<211> 20
<212> DNA
<213> Pc45_p5_5_R(Artificial)
<400> 22
caggcccttt caccttttgc 20
<210> 23
<211> 23
<212> DNA
<213> Pc71_p5_5_F(Artificial)
<400> 23
tcttatggtg aggtctccaa tgt 23
<210> 24
<211> 21
<212> DNA
<213> Pc71_p5_5_R(Artificial)
<400> 24
gcaagatgcg cagatttctc t 21
<210> 25
<211> 20
<212> DNA
<213> Pc73_p5_5_F(Artificial)
<400> 25
atcatggtct ttgtcgccgt 20
<210> 26
<211> 21
<212> DNA
<213> Pc73_p5_5_R(Artificial)
<400> 26
agtcggatga ttgtcaatcc g 21
<210> 27
<211> 20
<212> DNA
<213> Pc81_p4_8_F(Artificial)
<400> 27
gctccaacat gggattcacg 20
<210> 28
<211> 22
<212> DNA
<213> Pc81_p4_8_R(Artificial)
<400> 28
ggatcagctt tgtgagttct gc 22
<210> 29
<211> 20
<212> DNA
<213> Pc96_p3_8_F(Artificial)
<400> 29
ccctcaacaa ggcctcatgt 20
<210> 30
<211> 20
<212> DNA
<213> Pc96_p3_8_R(Artificial)
<400> 30
gtgggatcga gaggctgatg 20
- 浙江楠核基因组SSR分子标记的多态性引物及其应用
- 香樟核基因组SSR分子标记的多态性引物及其应用
