一种基于张量分解子空间融合的多模态医学图像分类方法
文献发布时间:2024-04-18 19:58:53
技术领域
本发明涉及一种基于张量分解子空间融合的多模态医学图像分类方法,属于计算机视觉和医学图像处理领域。
背景技术
多模态融合广泛应用于计算机视觉领域,其主要目的是从不同的模态中提取有用并且互补的信息用于后续任务。在医学领域,基于深度学习的方法在疾病的诊断和预后方面起到了不可替代的作用。在深度学习广泛普及之前,图像融合就已经得到了深入的研究,早期实现图像融合的方法采用相关的数学变换来手工分析,并设计基于空间域或者变换域的融合规则,称为传统的融合方法。典型的传统融合方法包括基于多尺度变换的方法、基于稀疏表示的方法、基于子空间的方法、基于显著性的方法等。然而,这些方法也有一定的局限性,传统的方法对于不同的源图像采用相同的变换方法,没有考虑到图像的特征差异,这导致提取的特征表现能力较差。并且这些传统方法的特征融合策略过于粗糙,使得融合性能有限。因此,为了克服传统方法的局限性,将深度学习引入图像融合算法中。基于深度学习的方法可以利用不同的网络分支来实现差异化的特征提取,从而获得更有针对性的特征。其次,基于深度学习的方法可以在精心设计的损失函数的指导下,学习更加合理的特征融合策略,从而实现自适应的特征融合。得益于这些优势,深度学习促进了图像融合的巨大进步,其图像融合的性能远远超过了传统方法。
原始的细胞图像大多呈现透明状态,因此细胞学分析通常需要借助不同的染色试剂使细胞进行显色反应,使得不同细胞或细胞的不同结构呈现不同的颜色,细胞组织内各种物质成分产生不同的折光率,从而便于观察和鉴定。将不同染色的细胞图像作为细胞的不同模态,与单一模态相比,多模态图像有助于从不同的视图中提取特征,并带来互补的信息,有助于更好的数据表示和网络鉴别能力,应用多模态图像也可以降低细胞分类的不确定性,提高临床诊断准确率。但是,随着网络提取的特征维度的增加,多个模态的数据往往会引入大量噪声,给网络训练带来一定的干扰性。大多数的融合方法往往是将这样高维的特征进行简单的拼接或者相加,这种粗略的融合方式并没有考虑到各个模态之间的互补性,并且会增加融合特征的维度,使得网络训练更加困难。因此,我们对网络提取到的高维特征进行分解,将冗余的噪声和真实有用的信息分解,得到更低维度并且干净的特征子空间。我们在子空间的层面上进行融合,创新性的将子空间学习和自注意力机制结合,将Transformer中的自注意力机制引入融合模块,充分利用子空间中各个部分的相关性,从而改变原始特征的空间结构。最后通过Tucker重建特征子空间得到最终用于分类的充分融合后的特征。
发明内容
本发明目的在于提供了一种基于张量分解子空间融合的多模态医学图像分类方法,通过融合两种模态的细胞图像对四种细胞进行分类。
为了实现上述目的,本发明的方案是:
以一般的图像分类网络作为基础框架,设计了一种多模态融合分类框架,能够去除高维特征的冗余噪声,并将自注意力机制引入融合模块。目前具体的步骤如下:
(1)构建多模态医学细胞图像数据集,获得细胞原始图像以及相应的HAL试剂染色图像,并对癌细胞(A549)、间皮细胞(Met 5A)、红细胞(RBC)以及白细胞(WBC)四个类别的细胞进行标记;
(2)设计神经网络,实现多模态图像融合分类功能。使用卷积神经网络对不同模态的图像数据提取高维特征;
(3)使用Tucker分解的思想,将两个模态的高维特征分解为核心张量以及各个维度的因子矩阵,将高维特征去噪并且降维;
(4)将Transformer中的自注意力机制引入融合模块,交换各个维度上的特征信息并且探索子空间中各个成分的相关性。最终通过Tucker重建将子空间中的各个成分重建,得到充分融合后的特征。
(5)进行多次实验,记录每次实验结果的平均准确率、F1-score,以及每一类的准确度、精确度和F1-score;
(6)设计消融实验,探究每个模块对分类的性能的贡献程度。
本发明的有益效果是:本发明提出了一种基于张量分解子空间融合的多模态医学图像分类方法。首先,构建了一个多模态医学图像细胞分类数据集,然后使用卷积神经网络进行特征提取,使用Tucker分解的思想将高维特征分解到低维的特征子空间中,通过调整各个维度上的秩,达到去噪的目的。在融合模块中引入Transformer中的自注意力机制,交换各个维度上的特征信息,使得不同模态的子空间充分融合,消除不同模态的语义差距。最终将子空间中的核心张量以及三个维度上的特征矩阵重建,得到用于分类的特征张量。
附图说明
图1:是构建多模态细胞数据集流程图。
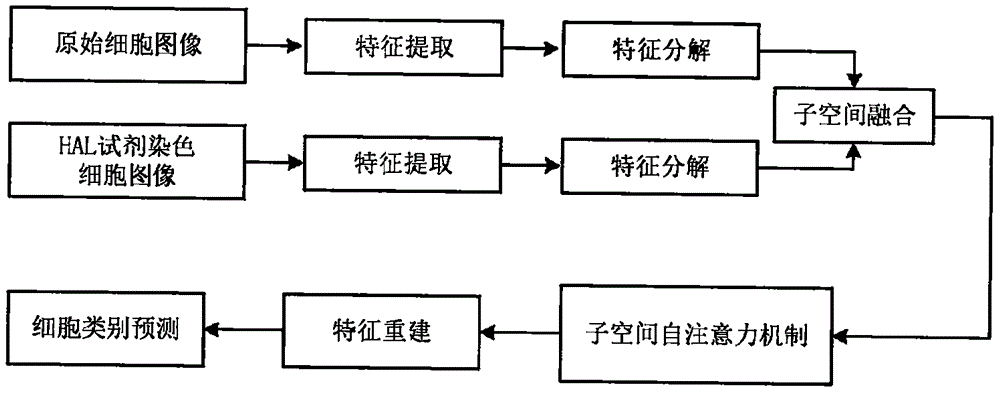
图2:是提出的多模态融合方法流程图。
具体实施方式
获得细胞原始图像以及HAL试剂染色两个模态的细胞图像,两个模态的细胞图像是一一对应的关系,一共分为4个类别,分别是癌细胞(A549)、间皮细胞(Met 5A)、红细胞(RBC)以及白细胞(WBC),数据集收集方法如图1所示。每个类别有100对配对的样本,整个数据集共有400个样本。其中60%用于训练,20%用于验证,20%用于测试。
使用细胞原始图像以及染色后的细胞图像,利用卷积神经网络(ConvolutionalNeural Networks,CNN)和深度学习方法实现多模态融合的分类算法,对两个模态的细胞进行特征提取,并设计多模态特征融合算法,损失函数等。
如图2所示,该分类网络主要包含图中几个部分。先通过两个独立的特征提取网络,提取到两个模态的高维特征,再使用我们提出的Tucker分解模块将高维特征分解为特征子空间,在子空间层面使用子注意力机制充分学习各个维度的相关性,最终重建特征,再进行后续的分类任务。最后根据分类结果计算准确率(Accuracy)、灵敏度(Sensitive)、精确度(Precision)、F1-score以及接受者操作特征曲线下面积(Area Under the ReceiverOperating Characteristic curve,AUROC),评价模型的性能。
在我们的分类算法中,我们先利用两个独立的残差网络ResNet50对于两个模态的细胞图像进行特征提取,使得网络能够学习到不同模态的图像特征。
在提取到不同模态的图像特征后,使用Tucker分解模块将高维特征分解,得到一个核心张量g,以及在通道维度,长度维度,宽度维度上的因子矩阵A、B、C。其中,核心张量g代表了三个维度的相关程度,三个因子矩阵代表了三个维度上的主要成分:
X=((g×
在特征分解之后,使用Transformer中的自注意力机制将子空间中的各个维度上的主要信息进行交换,使得子空间中的低维特征相关性更强,学习到不同模态的互补信息并且将不同模态的特征对齐。在我们的基于自注意力机制的子空间特征融合模块中,每个维度上的因子矩阵分别作为Quary,三个因子矩阵拼接作为Key和Value:
其中,Q
核心张量g既代表了各个维度上因子矩阵的关联程度,也是原始高维特征张量的主要成分。因此我们仍然使用自注意力机制将核心张量中的高频信息和三个因子矩阵融合,我们通过卷积操作将核心张量g的各个维度的通道数减少为1,得到相应维度的注意力权重作为Key和Value,三个维度的因子矩阵作为Query,计算公式如公式(2)所示。
最终,我们通过Tucker分解去除了噪声,保留了高维特征的有用信息,又使用Transformer中的自注意力机制使得两个模态的特征子空间融合,得到最终的干净的充分融合后的特征子空间,我们将特征子空间重建,重建过程即为公式(1)的逆过程。得到最终的用于分类的融合特征,通过全局平均池化操作以及线性层操作,得到最终的预测结果,使用交叉熵损失作为损失函数,公式如下:
其中,N表示样本数,M表示类别数,y
基于多模态融合的医学图像分类算法能够应用于现实医学研究,可以扩展到其他各种医学图像模态,为医学研究贡献微薄之力。
需要说明的是,以上所述仅为本发明实施例,仅仅是解释本发明,并非因此限制本发明专利范围。对属于本发明技术构思而仅仅显而易见的改动,同样在本发明保护范围之内。
- 一种基于子空间投影和字典学习的图像分类方法
- 一种基于多视子空间聚类的多模态医学数据融合系统
- 一种基于多视子空间聚类的多模态医学数据融合系统
