重组聚合酶、编码基因、制备方法、载体、试剂盒及应用
文献发布时间:2023-06-19 10:40:10
技术领域
本发明属于分子生物学和蛋白质工程技术领域,尤其涉及一种重组聚合酶、编码基因、制备方法、载体、试剂盒及应用。
背景技术
目前,随着大规模测序及基因诊断的快速发展,目前,应用最为广泛的核酸扩增技术是聚合酶链式反应(Polymerase Chain Reaction,PCR)。PCR技术广泛应用于分子克隆、基因组测序以及临床诊断等生命科学的各个领域,推动了精准医疗领域的发展,加速了基因与遗传病相关性的研究。在生物学研究、医学检验、法医鉴证及农业等相关领域,核酸的扩增都是一种非常重要的技术。PCR技术利用反应温度的循环变化来实现目的序列的变性、复性和延伸,能够在2-3小时之内实现目的序列的指数级扩增。然而,PCR技术也面临很多方面的局限性,血液和组织样本不能直接用于PCR反应,需要进一步提取DNA,限制了PCR技术的快速应用。
近些年,在DNA、RNA合成的分子生物学领域有许多的研究进展,许多在体内核酸扩增过程中发挥重要辅助作用的蛋白被相继发现。有研究表明,将Sso7d与来源于DNA聚合酶I家族和II家族的高温DNA聚合酶共价连接,能够显著提高高温DNA聚合酶的持续合成能力。在这些研究进展的基础上,研究人员应用q-PCR、RT-PCR、数字PCR,以及ARMS-PCR技术来检测癌症基因、以及流感病毒的诊断与筛查等。因此,Taq DNA聚合酶在PCR诊断、原位PCR和司法鉴定中有着广泛的应用前景。
在PCR核酸扩增过程中,常常使用Taq DNA聚合酶促进扩增。Taq DNA聚合酶是来源于Thermus aquaticus YT-1菌的提取酶。天然的Taq DNA聚合酶的持续合成能力不高,也就是说该聚合酶与模板结合一次能催化的聚合反应数目较少。如何改进Taq DNA聚合酶,提高持续合成能力,加快PCR核酸扩增速率是亟待解决的问题。
Cl7是一种来源于大肠杆菌的非特异性DNA结合蛋白。因此,将Cl7与DNA聚合酶Taq共价连接或许能够为DNA聚合酶的改造提供一种新的思路。
通过上述分析,现有技术存在的问题及缺陷为:
(1)PCR技术也面临很多方面的局限性,血液和组织样本不能直接用于PCR反应,需要进一步提取DNA,限制了PCR技术的快速应用。
(2)天然的Taq DNA聚合酶的持续合成能力不高,该聚合酶与模板结合一次能催化的聚合反应数目较少。
解决以上问题及缺陷的难度为:现有的Taq DNA聚合酶在对底物的灵敏性不高,对应的灵敏度高的突变体现在还没找到,目前市面上的Taq DNA聚合酶大多达不到对体液或者样本直接进行检测的目标。
解决以上问题及缺陷的意义为:Cl7是一种DNA结合蛋白,本发明将该蛋白融合在Taq DNA聚合酶的N端后,提高了Taq DNA聚合酶对底物的结合能力,使得原本的Taq DNA聚合酶的活性能发挥出来,从而达到用Cl7-Taq重组DNA聚合酶能够直接检测体液或者血液样本中的目标基因。从而实现利用该酶进行疾病检测和诊断。
发明内容
针对现有技术存在的问题,本发明提供了一种重组聚合酶、编码基因、制备方法、载体、试剂盒及应用,具体涉及一种Cl7-Taq重组DNA聚合酶及其制备方法与应用。
本发明是这样实现的,一种Cl7-Taq重组DNA聚合酶(基因序列号:P19821.1),所述Cl7-Taq重组DNA聚合酶中引入一种非特异性双链DNA结合蛋白Cl7,所述Cl7序列如SEQ IDNO.1所示。
进一步,所述Cl7-Taq重组DNA聚合酶来源于Thermus aquaticus YT-1菌株中的Taq DNA聚合酶蛋白序列N端以柔性连接序列共价连接蛋白Cl7而得到的杂合DNA聚合酶,氨基酸序列如SEQ ID No.4所示,或该序列经替换、缺失或添加一个或几个氨基酸形成的具有同等功能的氨基酸序列。
本发明的另一目的在于提供一种编码所述Cl7-Taq重组DNA聚合酶的基因,所述基因的核苷酸序列如SEQ ID No.3所示。
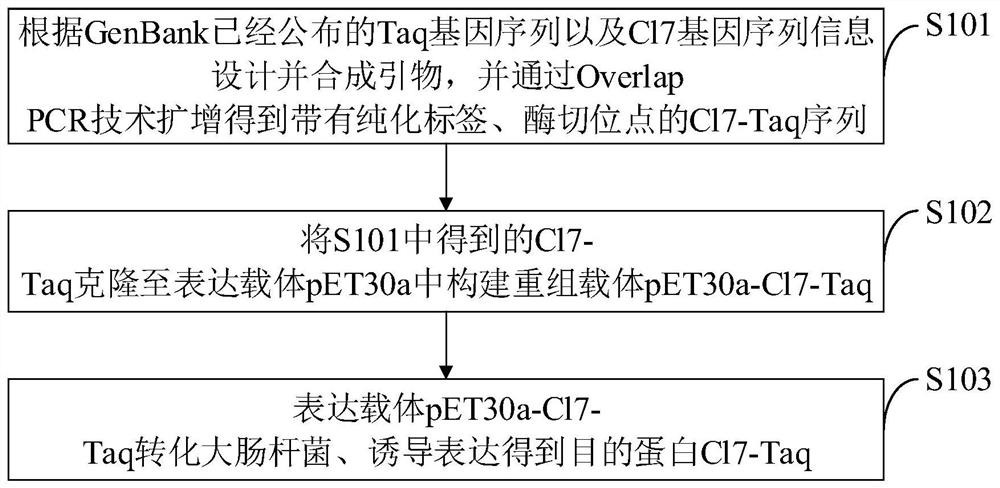
本发明的另一目的在于提供一种应用所述Cl7-Taq重组DNA聚合酶的Cl7-Taq重组DNA聚合酶的制备方法,所述Cl7-Taq重组DNA聚合酶的制备方法包括以下步骤:
步骤一,根据GenBank已经公布的Taq基因序列以及Cl7基因序列信息设计并合成引物,并通过Overlap PCR技术扩增得到带有纯化标签、酶切位点的Cl7-Taq序列;
步骤二,将步骤一中得到的Cl7-Taq克隆至表达载体pET30a中构建重组载体pET30a-Cl7-Taq;
步骤三,表达载体pET30a-Cl7-Taq转化大肠杆菌、诱导表达得到目的蛋白Cl7-Taq。
进一步,步骤二中,所述重组载体pET30a-Cl7-Taq的构建方法,包括:
在合成的Taq基因序列C末端附加6个组氨酸作为标签,目标基因利用引物Taq-Linker和Taq-his进行PCR扩增,包含有T7启动子、核糖结合位点、T7终止子以及卡拉抗性的pET30a载体作为目标基因的高拷贝克隆载体,利用同源重组的方式将Taq基因克隆在质粒pET30a重组载体pET30a-Taq;重组后的pET30a-Taq基因被转化进入感受态细胞并进行DNA测序验证;
根据发布的Cl7氨基酸序列合成其基因序列,根据无酶克隆的方法利用柔性linker将Cl7连接到Taq的N末端,利用引物Cl7-F、Cl7-Linker、Taq-Linker、Taq-his进行PCR扩增从而将Taq、linker、Cl7三个片段的基因组合在一起,提取完整的重组质粒pET30a-Cl7-Taq并用DNA测序进行验证;其中,编码所述柔性linker的核苷酸序列如SEQ ID NO.2所示。
进一步,步骤三中,所述表达载体pET30a-Cl7-Taq转化大肠杆菌、诱导表达得到目的蛋白Cl7-Taq,包括:
(1)将经过测序验证的阳性重组表达质粒pET30a-Cl7-Taq转化入大肠杆菌表达菌株BL21(DE3)中,挑取单个菌落在含有卡那霉素的LB液体培养基中37℃摇床过夜培养;
(2)将过夜菌液以1/100的比例接种至500ml含有卡那霉素的LB液体培养基中,37℃摇床培养至OD600值达到0.6时,加入IPTG至终浓度为0.5mmol/L进行诱导;同时设置表达载体对照组,18℃低温诱导表达12h后取菌液,12000r/min离心10min,去上清;
(3)Ni-NTA裂解缓冲液重悬菌沉淀,超声破碎后,10000r/min离心20min,收集上清和沉淀进行SDS-PAGE电泳;将上清置于75℃热水浴20min后,10000r/min离心20min,去掉已变性的蛋白;
(4)利用AKTA蛋白纯化系统,Ni Sepharose 6Fast Flow亲和层析柱,对目的蛋白进行亲和纯化,上样缓冲液20mM Tris-HCl,0.5M NaCl,10mM咪唑,pH 9.0;洗脱缓冲液20mMTris-HCl,0.5M NaCl,200mM咪唑,pH 9.0;
(5)收集蛋白洗脱峰样品并对蛋白样品进行4℃超滤,10mM Tris-HCl,100mM KCl,1mM DTT,0.2mM EDTA,pH 8.0;蛋白样品保存于-80℃冰箱中。
进一步,所述Ni-NTA裂解缓冲液包括20mM Tris-HCl,0.5M NaCl,10mM咪唑,pH9.0。
本发明的另一目的在于提供一种应用所述Cl7-Taq重组DNA聚合酶的载体或重组细胞。
本发明的另一目的在于提供一种应用所述Cl7-Taq重组DNA聚合酶制备得到的试剂盒。
本发明的另一目的在于提供一种所述Cl7-Taq重组DNA聚合酶在DNA复制中的应用。
结合上述的所有技术方案,本发明所具备的优点及积极效果为:本发明提供的Cl7-Taq重组DNA聚合酶的制备方法,利用基因工程的方法,在来源于Thermus aquaticusYT-1菌的Taq DNA聚合酶N端共价连接来源于大肠杆菌的DNA结合蛋白Cl7进行重组而成,大大提高TaqDNA聚合酶在PCR核酸扩增的持续合成能力。
本发明利用柔性linker在DNA聚合酶Taq的N末端连接蛋白Cl7组合成Cl7-Taq蛋白来增强Taq酶对聚合底物的亲和力,提高Taq酶的持续性合成能力。发明首先对DNA聚合酶Taq基因进行克隆并构建Cl7-Taq融合蛋白,然后通过E.coli BL21菌株表达Cl7-Taq,并对纯化的Cl7-Taq进行聚合酶活性、扩增速率、模板敏感度,热稳定性一系列的对比实验,最终证实Cl7能够改善Taq的酶学性质。本发明还提供了编码该蛋白的基因以及该蛋白的制备方法和用途。Cl7-TaqDNA聚合酶在PCR诊断、原位PCR和司法鉴定中有着广阔的应用前景。
本发明对核酸扩增技术中DNA聚合酶进行了分子生物学改造,改造后Cl7-Taq聚合酶的持续合成能力得到有效提高,并且在重组酶介导的核酸扩增反应中对模板的敏感性大幅提高,同时,扩增速率也提高了一倍;在检测血液或组织样品中,常常因为样品DNA模板浓度太低而很难直接用于PCR检测,因此,需要抽提纯的DNA样品;改造后的DNA聚合酶,由于对模板具有更高的敏感性,因此能够显著提高重组酶介导的核酸扩增技术的DNA扩增效果,具有广泛的应用前景。
对比的技术效果或者实验效果。融合Cl7-Taq DNA聚合酶对比Taq DNA聚合酶的效率比较过程中,发现Cl7-Taq DNA聚合酶在60秒内扩增4000bp产物,而Taq DNA聚合酶需要120秒扩增2000bp的产物。由此可以得出结论,蛋白Cl7与Taq DNA聚合酶的融合使其延伸速率提高了一倍。
附图说明
为了更清楚地说明本发明实施例的技术方案,下面将对本发明实施例中所需要使用的附图做简单的介绍,显而易见地,下面所描述的附图仅仅是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下还可以根据这些附图获得其他的附图。
图1是本发明实施例提供的pET30a-Cl7-Taq重组质粒构建示意图。
图2是本发明实施例提供的Cl7-Taq蛋白SDS-PAGE分析结果示意图;
图中:M为Marker,1为破菌上清,2为热处理后的破菌上清,3为目的蛋白(约108KD)。
图3是本发明实施例提供的PCR反应程序设置图。
图4是本发明实施例提供的Taq和Cl7-Taq扩增速率比较的电泳图;
图中:M为DNA Marker,1为1kb片段,2为2kb片段,3为3kb片段,4为4kb片段。
图5是本发明实施例提供的Taq和Cl7-Taq对模板敏感度比较的电泳图;
图中:M为DNA Marker,1为10ng,2为1ng,3为10
图6是本发明实施例提供的Taq和Cl7-Taq在95℃热稳定性比较的电泳图;
图中,M为DNA Marker,1为1h,2为2h,3为3h,4为4h,5为5h;Cl7、Taq蛋白三维结构预测结果及共价连接位置指示。
图7是本发明实施例提供的Cl7-Taq重组DNA聚合酶的制备方法流程图。
具体实施方式
为了使本发明的目的、技术方案及优点更加清楚明白,以下结合实施例,对本发明进行进一步详细说明。应当理解,此处所描述的具体实施例仅仅用以解释本发明,并不用于限定本发明。
针对现有技术存在的问题,本发明提供了一种Cl7-Taq重组DNA聚合酶及其制备方法与应用,下面结合附图对本发明作详细的描述。
如图7所示,本发明实施例提供的Cl7-Taq重组DNA聚合酶的制备方法包括以下步骤:
S101,根据GenBank已经公布的Taq基因序列以及Cl7基因序列信息设计并合成引物,并通过Overlap PCR技术扩增得到带有纯化标签、酶切位点的Cl7-Taq序列;
S102,将S101中得到的Cl7-Taq克隆至表达载体pET30a中构建重组载体pET30a-Cl7-Taq;
S103,表达载体pET30a-Cl7-Taq转化大肠杆菌、诱导表达得到目的蛋白Cl7-Taq。
下面结合实施例对本发明作进一步描述。
本发明中的生物材料来源:
1、大肠杆菌BL21(DE3)购自Novagen公司;
2、所用引物委托生工生物工程(上海)股份有限公司合成、含有Cl7-linker基因的质粒pUC57-Cl7-linker委托武汉金开瑞生物工程有限公司合成;
3、所用载体pET30a购自Novagen公司。
实施例1
本实施例用于说明重组质粒pET30a-Cl7-Taq的构建(见图1)。
通过武汉金开瑞生物工程有限公司合成Taq基因序列(SEQ ID NO.1),并在其C末端附加6个组氨酸作为标签,目标基因利用引物Taq-Linker和Taq-his进行PCR扩增,包含有T7启动子、核糖结合位点、T7终止子以及卡拉抗性的pET30a载体作为目标基因的高拷贝克隆载体,利用同源重组的方式将Taq基因克隆在质粒pET30a重组载体pET30a-Taq;重组后的pET30a-Taq基因被转化进入感受态细胞并进行DNA测序验证。
根据发布的Cl7氨基酸序列合成其基因序列,根据无酶克隆的方法利用柔性linker(SEQ ID NO.2)将Cl7(SEQ ID NO.1)连接到Taq的N末端,利用引物Cl7-F(SEQ IDNo.14)、Cl7-Linker(SEQ ID No.15)、Taq-Linker(SEQ ID No.16)、Taq-his(SEQ IDNo.17)进行PCR扩增从而将Taq、linker、Cl7三个片段的基因组合在一起,提取完整的重组质粒pET30a-Cl7-Taq并用DNA测序进行验证。
表1引物序列
实施例2:Cl7-Taq蛋白的表达和纯化
本实施例说明重组蛋白的诱导表达和纯化。
1、将经过测序验证的阳性重组表达质粒pET30a-Cl7-Taq转化入大肠杆菌表达菌株BL21(DE3)中,挑取单个菌落在含有卡那霉素的LB液体培养基中37℃摇床过夜培养。
2、将过夜菌液以1/100的比例接种至500ml含有卡那霉素的LB液体培养基中,37℃摇床培养至OD600值达到0.6时,加入IPTG至终浓度为0.5mmol/L进行诱导。同时设置表达载体对照组,18℃低温诱导表达12h后取菌液,12000r/min离心10min,去上清。
3、Ni-NTA裂解缓冲液(20mM Tris-HCl,0.5M NaCl,10mM咪唑,pH 9.0)重悬菌沉淀,超声破碎后,10000r/min离心20min,收集上清和沉淀进行SDS-PAGE电泳。
4、将上清置于75℃热水浴20min后,10000r/min离心20min,去掉已变性的蛋白。
5、利用AKTA蛋白纯化系统,Ni Sepharose 6Fast Flow亲和层析柱,对目的蛋白进行亲和纯化,上样缓冲液(20mM Tris-HCl,0.5M NaCl,10mM咪唑,pH9.0),洗脱缓冲液(20mMTris-HCl,0.5M NaCl,200mM咪唑,pH 9.0)。收集蛋白洗脱峰样品并对蛋白样品进行4℃超滤(10mM Tris-HCl,100mM KCl,1mM DTT,0.2mM EDTA,pH 8.0)。蛋白样品保存于-80℃冰箱中。蛋白纯化过程各部分样品电泳结果如图2所示。
对比例1
对蛋白改造之前的聚合酶Taq和改造之后的聚合酶Cl7-Taq的扩增速率进行比较:
1、以pET23a/dcas9质粒DNA为模板,分别用1U的Cl7-Taq和Taq DNA聚合酶扩增PCR片段(1、2、3和4kb)。所用引物为:
dcas9-F:5’-GCAGCCCAGTAGTAGGTTGAG-3’(SEQ ID No.5)
dcas9-R1:5’-CATAATTTCGGCATGTGCGGT-3’(SEQ ID No.6)
dcas9-R2:5’-CGAACAGCTGGTTGTAGGTCT-3’(SEQ ID No.7)
dcas9-R3:5’-CGGTCACGTATTTCACTTTGGT-3’(SEQ ID No.8)
dcas9-R4:5’-CGGTTCTTGTCGCTTCTGGT-3’(SEQ ID No.9)
2、按照图3所示设置PCR反应程序。取5μL样品在浓度为0.8%琼脂糖凝胶中进行电泳,图4为电泳结果。
根据图4结果显示融合Cl7-Taq DNA聚合酶比Taq DNA聚合酶有更快的延伸速率,即Cl7-Taq DNA聚合酶在60秒内扩增4000bp产物,而Taq DNA聚合酶需要120秒扩增2000bp的产物。由此可以得出结论,蛋白Cl7与Taq DNA聚合酶的融合使其延伸速率提高了一倍。
对比例2
对蛋白改造之前的聚合酶Taq和改造之后的聚合酶Cl7-Taq的模板敏感度进行比较:
以pET23a/dcas9质粒为模板,PCR反应产物为2.7kb。该反应是在降低模板剂浓度(连续10倍稀释梯度)的条件下进行的,PCR反应程序:95℃预变性3min,98℃变性30s,60℃退火30s,72℃延伸1.5min,循环35次,最后72℃延伸5min,4℃保存。
所用引物为:
DA-F:5’-CAGCATCGGCCTGGCCATCGGCACCAACTCTGTGGG-3’(SEQ ID No.10)
HA-R:5’-AAAGCTCTGAGGCACGATGGCGTCCACATCGTAGTCGG-3’(SEQ ID No.11)
2.取5μL样品在浓度为0.8%琼脂糖凝胶中进行电泳,图5为电泳结果。
3.根据图5结果显示融合Cl7的Taq聚合酶仅用0.1pg的质粒DNA即可扩增得到目的片段,而Taq聚合酶至少需要10pg的质粒DNA才能扩增得到目的片段。由此可以得出结论,融合Cl7的Taq聚合酶比天然的Taq聚合酶对模板的敏感度更强。
对比例3
对蛋白改造之前的聚合酶Taq和改造之后的聚合酶Cl7-Taq的热稳定性进行比较:
1.以pET30a-GFP质粒DNA作为PCR模板,将1U纯化的Cl7-Taq或Taq DNA聚合酶分别在95℃加热1、2、3、4、5h后,进行PCR反应,PCR反应程序:95℃预变性3min,95℃变性15s,58℃退火15s,72℃延伸30s,循环28次,最后72℃延伸5min,4℃保存。所用引物为:
GFP-F:5’-TGGTCTTCAATGCTTTGCGAGATAA-3’(SEQ ID No.12)
GFP-R:5’-CTTTTCGTTGGGATCTTTCG-3’(SEQ ID No.13)
2.取5μL样品在浓度为1.2%琼脂糖凝胶中进行电泳,图6为电泳结果。
3.根据图6结果显示天然的Taq DNA聚合酶在95℃的热稳定性为2h,而Cl7-TaqDNA聚合酶在95℃的热稳定性为3h。由此可以得出结论,融合Cl7的Taq聚合酶比天然的Taq聚合酶热稳定性更强。
证明部分(具体实施例/实验/仿真/药理学分析/能够证明本发明创造性的正面实验数据等)具体实施案例1:ARMS-PCR检测方法建立
利用Cl7-Taq DNA没有3'→5'核酸外切酶活性,故3'端错配的碱基不能得到校正,因此不能继续延伸。首先设计四条引物,其中一对引物OUTER-F/OUTER-R用于扩增含有SNP位点的一段基因序列,大小为560bp,另外一对引物SNPrs12516-F/SNPrs12516-R用来检测SNP位点的基因,引物SNPrs12516-F的3'端为C与G碱基互补配对,引物SNPrs12516-R的3'端为A与T碱基互补配对,若BRCA1基因在SNPrs12516位点的基因型为C/C,则出现两条带,大小分别是560bp和90bp;若BRCA1基因在SNPrs12516位点的基因型为T/C,则会出现三条带,大小分别为560bp、230bp和90bp;BRCA1基因在SNPrs12516位点的基因型为T/T,则会出现两条带,大小分别为230bp和90bp。在20μL的PCR反应体系中同时加入四条引物和1μL血液作为模板,扩增35个循环后用琼脂糖凝胶电泳检测,SNP位点出现三种不同大小的条带,分别对应着不同的基因型。
具体实施案例2:ARMS-PCR检测BRCA1(SNPrs12516)基因
首先按照ARMS-PCR检测原理图所示的方法对10个血液样本进行PCR反应来检测乳腺癌易感基因BRCA1(SNPrs12516)的基因型,该基因位于13号染色体上,有相关文献证实该基因的突变与癌症有关,因此,对10例患者的血样进行了ARMS-PCR检测,2,3号血液样本的基因型为T/C,分别将血液样本抽提基因组DNA,送至测序公司进行测序,测序结果显示,2号、3号血液的SNPrs12516基因型均为T/C,检验结果与预期的结果一致。说明ARMS-PCR能够快速准确地检测癌症基因的单核苷酸突变(SNPrs12516)。
具体实施案例3:CL7-Taq DNA聚合酶在小鼠基因分型中的应用
小鼠的基因分型试剂盒需要从小鼠尾巴、耳朵以及脚趾等组织中提取基因组DNA,然后进行PCR扩增。与传统的基因分型的方法相比,本文将CL7-Taq DNA聚合酶应用于小鼠的基因分型中,利用CL7-Taq DNA聚合酶较高的模板敏感度,可以快速有效地检测出小鼠的基因型,无需匀浆、破碎、过夜消化、酚氯仿抽提、DNA沉淀或柱式纯化等操作步骤来提取小鼠的基因组DNA,极大地缩短了检验时间,同时也节省了成本。将小鼠的尾尖组织浸泡在裂解缓冲液中,100℃分别加热2、4、6、8、10min,分别把加热不同时间的裂解液直接作为模板,同时用1U的CL7-Taq DNA聚合酶和Taq DNA聚合酶这两种酶做对比实验,检测结果如图3.12所示,CL7-Taq DNA聚合酶只需要加热2min就可以成功扩增出目的条带,而Taq DNA聚合酶至少需要加热4min才能扩增出目标产物。
以上所述,仅为本发明的具体实施方式,但本发明的保护范围并不局限于此,任何熟悉本技术领域的技术人员在本发明揭露的技术范围内,凡在本发明的精神和原则之内所作的任何修改、等同替换和改进等,都应涵盖在本发明的保护范围之内。
序列表
<110> 湖北大学
<120> 重组聚合酶、编码基因、制备方法、载体、试剂盒及应用
<160> 17
<170> SIPOSequenceListing 1.0
<210> 1
<211> 390
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 1
agcaaaagca atgaaccggg taaagcaacc ggtgaaggta aaccggttaa taacaaatgg 60
ctgaacaatg ccggtaaaga tctgggtagt ccggttccgg atcgtattgc aaataaactg 120
cgtgataaag aattcgagag cttcgatgat tttcgtgaaa ccttttggga agaagttagc 180
aaagatcctg aactgagcaa acagtttagc cgcaataaca atgatcgtat gaaagttggt 240
aaagcaccga aaacacgtac ccaggatgtt agcggtaaac gtacctcatt tgaactgaat 300
catcagaaac cgattgaaca gaatggtggc gtttatgata tggataacat tagcgttgtt 360
accccgaaac gcaacattga tattgaaggc 390
<210> 2
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 2
gaaaacctgt attttcaggg t 21
<210> 3
<211> 2904
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 3
agcaaaagca atgaaccggg taaagcaacc ggtgaaggta aaccggttaa taacaaatgg 60
ctgaacaatg ccggtaaaga tctgggtagt ccggttccgg atcgtattgc aaataaactg 120
cgtgataaag aattcgagag cttcgatgat tttcgtgaaa ccttttggga agaagttagc 180
aaagatcctg aactgagcaa acagtttagc cgcaataaca atgatcgtat gaaagttggt 240
aaagcaccga aaacacgtac ccaggatgtt agcggtaaac gtacctcatt tgaactgaat 300
catcagaaac cgattgaaca gaatggtggc gtttatgata tggataacat tagcgttgtt 360
accccgaaac gcaacattga tattgaaggc gaaaacctgt attttcaggg tcgtggtatg 420
ctgccgctgt ttgaacctaa aggtcgtgtt ctgctggtgg acggtcacca cctggcgtac 480
cgtaccttcc acgcgctgaa aggtctgacc acctctcgtg gtgaacctgt tcaggcggtt 540
tacggttttg cgaaatctct gctgaaggcc ctgaaagaag acggtgacgc tgttatcgtg 600
gttttcgacg cgaaagcgcc gtctttccgt cacgaagcct acggtggtta caaagcgggt 660
cgtgcaccaa ccccggaaga ctttccgcgt cagctggcgc tgattaaaga actggttgac 720
ctgctgggtc tggcgcgtct ggaagttccg ggttacgaag cggacgacgt tctggcgtct 780
ctggcgaaaa aagcggaaaa agaaggttat gaagttcgta tcctgaccgc ggacaaagac 840
ctgtaccagc tgctctctga ccgtatccac gttctccatc cggagggtta cctgattacc 900
cctgcgtggc tgtgggaaaa gtacggtctg cgccctgacc agtgggctga ctatcgtgcg 960
ctgacgggtg acgaatctga caacctgccg ggtgttaaag gtatcggtga aaaaaccgcg 1020
cgtaaactgc tcgaggaatg gggttccctg gaagcgctgc tgaaaaacct ggaccgtctg 1080
aagccggcga tccgcgaaaa gatcctggcg cacatggacg acctgaaact gtcttgggac 1140
ctggcgaaag ttcgtaccga cctgccactg gaagttgact tcgccaagcg tcgtgaaccg 1200
gaccgtgaac gcctgcgtgc gttcctggaa cgtctggaat tcggttctct gctgcacgag 1260
ttcggcctgc tggaatctcc gaaagcgctc gaagaggcgc cgtggcctcc gccggaaggt 1320
gccttcgttg gtttcgttct gtctcgtaaa gaaccaatgt gggcggacct gctcgcgctg 1380
gctgcggctc gtggtggtcg tgttcaccgt gcaccggagc cgtacaaagc tctgcgcgac 1440
ctgaaagagg cgcgtggtct gctggcgaaa gacctgtctg ttctcgcgct gcgtgagggt 1500
ctgggtctgc cacctggtga cgacccgatg ctcctggcct acctgctgga cccgtctaac 1560
accaccccgg aaggtgttgc gcgtcgttac ggtggtgaat ggaccgaaga agcgggtgaa 1620
cgtgcggcac tgagcgaacg cctgttcgca aacctgtggg gtcgcctcga aggtgaagaa 1680
cgtctgctct ggctgtaccg tgaagttgaa cgtcctctgt ctgcggttct ggcgcacatg 1740
gaagcgaccg gtgttcgtct ggacgttgcg tacctgcgtg cgctgtctct ggaagttgcg 1800
gaagaaatcg cgcgtctcga agcggaagtt ttccgtctgg cgggtcatcc gtttaacctg 1860
aactcccgtg accaactgga acgtgtgctg ttcgacgaac tgggtctgcc tgcgatcggt 1920
aaaaccgaaa aaaccggtaa acgttctacc tctgcggcgg ttctggaagc cctgcgcgaa 1980
gcgcacccga tcgttgaaaa aatcctgcag taccgtgaac tgaccaaact gaaatctacc 2040
tacatcgacc ctctgccgga cctcatccac ccgcgtaccg gtcgtctgca cacccgtttc 2100
aaccagaccg ctaccgcgac tggtcgcctg tcttcttctg acccgaacct gcagaacatc 2160
ccggttcgta ccccgctggg tcagcgtatc cgtcgtgcgt tcatcgcgga agaaggttgg 2220
ctgctggttg cgctggacta ctctcagatc gaactgcgtg ttctggcgca cctgtctggt 2280
gacgaaaacc tgatccgtgt tttccaggaa ggtcgtgaca tccacaccga aaccgcttct 2340
tggatgtttg gtgttccgcg tgaggcggtt gacccgctga tgcgtcgtgc ggcgaaaacc 2400
atcaacttcg gtgttctgta cggtatgtct gcgcaccgtc tgtctcagga actggcgatc 2460
ccgtacgaag aagcgcaggc gttcatcgaa cgttacttcc aatctttccc taaagttcgt 2520
gcgtggatcg aaaagaccct ggaagaaggc cgtcgtcgcg gttatgttga aaccctgttc 2580
ggtcgccgtc gctacgttcc ggacctcgaa gcgcgtgtta aatctgttcg tgaagcggcg 2640
gagcgcatgg ccttcaatat gccggttcag ggtaccgctg cggacctgat gaagctggcc 2700
atggtcaaac tgttcccgcg tctggaggaa atgggtgcgc gtatgctgct gcaggttcac 2760
gacgaactgg tactggaagc tccgaaggaa cgtgcggaag cggttgcacg tctggcgaaa 2820
gaagttatgg aaggtgttta cccactggcg gttccgctgg aagtggaagt tggtatcggt 2880
gaagactggc tgtctgcaaa agaa 2904
<210> 4
<211> 968
<212> PRT
<213> 人工序列(Artificial Sequence)
<400> 4
Ser Lys Ser Asn Glu Pro Gly Lys Ala Thr Gly Glu Gly Lys Pro Val
1 5 10 15
Asn Asn Lys Trp Leu Asn Asn Ala Gly Lys Asp Leu Gly Ser Pro Val
20 25 30
Pro Asp Arg Ile Ala Asn Lys Leu Arg Asp Lys Glu Phe Glu Ser Phe
35 40 45
Asp Asp Phe Arg Glu Thr Phe Trp Glu Glu Val Ser Lys Asp Pro Glu
50 55 60
Leu Ser Lys Gln Phe Ser Arg Asn Asn Asn Asp Arg Met Lys Val Gly
65 70 75 80
Lys Ala Pro Lys Thr Arg Thr Gln Asp Val Ser Gly Lys Arg Thr Ser
85 90 95
Phe Glu Leu Asn His Gln Lys Pro Ile Glu Gln Asn Gly Gly Val Tyr
100 105 110
Asp Met Asp Asn Ile Ser Val Val Thr Pro Lys Arg Asn Ile Asp Ile
115 120 125
Glu Gly Glu Asn Leu Tyr Phe Gln Gly Arg Gly Met Leu Pro Leu Phe
130 135 140
Glu Pro Lys Gly Arg Val Leu Leu Val Asp Gly His His Leu Ala Tyr
145 150 155 160
Arg Thr Phe His Ala Leu Lys Gly Leu Thr Thr Ser Arg Gly Glu Pro
165 170 175
Val Gln Ala Val Tyr Gly Phe Ala Lys Ser Leu Leu Lys Ala Leu Lys
180 185 190
Glu Asp Gly Asp Ala Val Ile Val Val Phe Asp Ala Lys Ala Pro Ser
195 200 205
Phe Arg His Glu Ala Tyr Gly Gly Tyr Lys Ala Gly Arg Ala Pro Thr
210 215 220
Pro Glu Asp Phe Pro Arg Gln Leu Ala Leu Ile Lys Glu Leu Val Asp
225 230 235 240
Leu Leu Gly Leu Ala Arg Leu Glu Val Pro Gly Tyr Glu Ala Asp Asp
245 250 255
Val Leu Ala Ser Leu Ala Lys Lys Ala Glu Lys Glu Gly Tyr Glu Val
260 265 270
Arg Ile Leu Thr Ala Asp Lys Asp Leu Tyr Gln Leu Leu Ser Asp Arg
275 280 285
Ile His Val Leu His Pro Glu Gly Tyr Leu Ile Thr Pro Ala Trp Leu
290 295 300
Trp Glu Lys Tyr Gly Leu Arg Pro Asp Gln Trp Ala Asp Tyr Arg Ala
305 310 315 320
Leu Thr Gly Asp Glu Ser Asp Asn Leu Pro Gly Val Lys Gly Ile Gly
325 330 335
Glu Lys Thr Ala Arg Lys Leu Leu Glu Glu Trp Gly Ser Leu Glu Ala
340 345 350
Leu Leu Lys Asn Leu Asp Arg Leu Lys Pro Ala Ile Arg Glu Lys Ile
355 360 365
Leu Ala His Met Asp Asp Leu Lys Leu Ser Trp Asp Leu Ala Lys Val
370 375 380
Arg Thr Asp Leu Pro Leu Glu Val Asp Phe Ala Lys Arg Arg Glu Pro
385 390 395 400
Asp Arg Glu Arg Leu Arg Ala Phe Leu Glu Arg Leu Glu Phe Gly Ser
405 410 415
Leu Leu His Glu Phe Gly Leu Leu Glu Ser Pro Lys Ala Leu Glu Glu
420 425 430
Ala Pro Trp Pro Pro Pro Glu Gly Ala Phe Val Gly Phe Val Leu Ser
435 440 445
Arg Lys Glu Pro Met Trp Ala Asp Leu Leu Ala Leu Ala Ala Ala Arg
450 455 460
Gly Gly Arg Val His Arg Ala Pro Glu Pro Tyr Lys Ala Leu Arg Asp
465 470 475 480
Leu Lys Glu Ala Arg Gly Leu Leu Ala Lys Asp Leu Ser Val Leu Ala
485 490 495
Leu Arg Glu Gly Leu Gly Leu Pro Pro Gly Asp Asp Pro Met Leu Leu
500 505 510
Ala Tyr Leu Leu Asp Pro Ser Asn Thr Thr Pro Glu Gly Val Ala Arg
515 520 525
Arg Tyr Gly Gly Glu Trp Thr Glu Glu Ala Gly Glu Arg Ala Ala Leu
530 535 540
Ser Glu Arg Leu Phe Ala Asn Leu Trp Gly Arg Leu Glu Gly Glu Glu
545 550 555 560
Arg Leu Leu Trp Leu Tyr Arg Glu Val Glu Arg Pro Leu Ser Ala Val
565 570 575
Leu Ala His Met Glu Ala Thr Gly Val Arg Leu Asp Val Ala Tyr Leu
580 585 590
Arg Ala Leu Ser Leu Glu Val Ala Glu Glu Ile Ala Arg Leu Glu Ala
595 600 605
Glu Val Phe Arg Leu Ala Gly His Pro Phe Asn Leu Asn Ser Arg Asp
610 615 620
Gln Leu Glu Arg Val Leu Phe Asp Glu Leu Gly Leu Pro Ala Ile Gly
625 630 635 640
Lys Thr Glu Lys Thr Gly Lys Arg Ser Thr Ser Ala Ala Val Leu Glu
645 650 655
Ala Leu Arg Glu Ala His Pro Ile Val Glu Lys Ile Leu Gln Tyr Arg
660 665 670
Glu Leu Thr Lys Leu Lys Ser Thr Tyr Ile Asp Pro Leu Pro Asp Leu
675 680 685
Ile His Pro Arg Thr Gly Arg Leu His Thr Arg Phe Asn Gln Thr Ala
690 695 700
Thr Ala Thr Gly Arg Leu Ser Ser Ser Asp Pro Asn Leu Gln Asn Ile
705 710 715 720
Pro Val Arg Thr Pro Leu Gly Gln Arg Ile Arg Arg Ala Phe Ile Ala
725 730 735
Glu Glu Gly Trp Leu Leu Val Ala Leu Asp Tyr Ser Gln Ile Glu Leu
740 745 750
Arg Val Leu Ala His Leu Ser Gly Asp Glu Asn Leu Ile Arg Val Phe
755 760 765
Gln Glu Gly Arg Asp Ile His Thr Glu Thr Ala Ser Trp Met Phe Gly
770 775 780
Val Pro Arg Glu Ala Val Asp Pro Leu Met Arg Arg Ala Ala Lys Thr
785 790 795 800
Ile Asn Phe Gly Val Leu Tyr Gly Met Ser Ala His Arg Leu Ser Gln
805 810 815
Glu Leu Ala Ile Pro Tyr Glu Glu Ala Gln Ala Phe Ile Glu Arg Tyr
820 825 830
Phe Gln Ser Phe Pro Lys Val Arg Ala Trp Ile Glu Lys Thr Leu Glu
835 840 845
Glu Gly Arg Arg Arg Gly Tyr Val Glu Thr Leu Phe Gly Arg Arg Arg
850 855 860
Tyr Val Pro Asp Leu Glu Ala Arg Val Lys Ser Val Arg Glu Ala Ala
865 870 875 880
Glu Arg Met Ala Phe Asn Met Pro Val Gln Gly Thr Ala Ala Asp Leu
885 890 895
Met Lys Leu Ala Met Val Lys Leu Phe Pro Arg Leu Glu Glu Met Gly
900 905 910
Ala Arg Met Leu Leu Gln Val His Asp Glu Leu Val Leu Glu Ala Pro
915 920 925
Lys Glu Arg Ala Glu Ala Val Ala Arg Leu Ala Lys Glu Val Met Glu
930 935 940
Gly Val Tyr Pro Leu Ala Val Pro Leu Glu Val Glu Val Gly Ile Gly
945 950 955 960
Glu Asp Trp Leu Ser Ala Lys Glu
965
<210> 5
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 5
gcagcccagt agtaggttga g 21
<210> 6
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 6
cataatttcg gcatgtgcgg t 21
<210> 7
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 7
cgaacagctg gttgtaggtc t 21
<210> 8
<211> 22
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 8
cggtcacgta tttcactttg gt 22
<210> 9
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 9
cggttcttgt cgcttctggt 20
<210> 10
<211> 36
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 10
cagcatcggc ctggccatcg gcaccaactc tgtggg 36
<210> 11
<211> 38
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 11
aaagctctga ggcacgatgg cgtccacatc gtagtcgg 38
<210> 12
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 12
tggtcttcaa tgctttgcga gataa 25
<210> 13
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 13
cttttcgttg ggatctttcg 20
<210> 14
<211> 29
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 14
aagcaatgaa ccgggtaaag caaccggtg 29
<210> 15
<211> 24
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 15
accctgaaaa tacaggtttt cgcc 24
<210> 16
<211> 35
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 16
aacctgtatt ttcagggtcg tggtatgctg ccgct 35
<210> 17
<211> 38
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 17
gtgatggtga tggtgatgtt cttttgcaga cagccagt 38
- 重组聚合酶、编码基因、制备方法、载体、试剂盒及应用
- DNA聚合酶及其应用、重组载体及其制备方法和应用、重组工程菌及其应用
