细胞裂解液、试剂盒及应用
文献发布时间:2023-06-19 11:37:30
技术领域
本发明涉及分子生物学领域。具体而言,本发明涉及一种细胞裂解液、细胞裂解方法、建库方法及应用。
背景介绍
目前,测序文库的构建是测序实现少量细胞染色体数据异常检测的必经步骤,其中细胞裂解获得gDNA是影响测序文库构建成功与否的关键因素。
常用的细胞裂解方法有碱裂解法(主要成分为NaOH)、蛋白酶K裂解法等。
碱裂解法主要采用NaOH裂解细胞,获得的gDNA对后续文库构建体系中缓冲液的要求高,如后续需加入含有能中和碱性的试剂,容易增加污染风险。
蛋白酶K裂解法利用蛋白酶K降解蛋白以实现细胞裂解。蛋白酶K在较广的pH范围(pH 4-12.5)、温度范围(37-60℃)、较高的盐浓度(如3M GuHCl)和变性剂(如4M尿素)中均有活性;一些去垢剂例如Tween20(5%)、Triton X-100(1%)或者SDS(1%)以及金属螯合剂EDTA等均不能使蛋白酶K失活。因为Ca
发明内容
基于单细胞和少量细胞(1~200个细胞)测序的需求及利用已知的细胞裂解液裂解细胞后通常需要更换一个反应体系例如对裂解产物进行分离或纯化才能进行下一个反应的情况,发明人提供了一种细胞裂解液、细胞裂解方法及基于此细胞裂解方法建立的文库制备方法。
本发明第一方面提供了一种细胞裂解液,该细胞裂解液的有效成分由蛋白酶K和NP-40组成。这里所称的有效成分包括任何的有助于细胞裂解的物质,例如利于提高消化酶活性的物质,例如能够与细胞的细胞膜、核膜或基因组上的蛋白、脂类等物质作用的物质。
该细胞裂解液的组成简单,能够有效地去除基因组上的蛋白,获得细胞中的基因组DNA。而且,裂解后的溶液体系不用经过更换或处理,可直接用于对细胞基因组DNA的进一步检测,例如测序等。
在一些实施例中,该细胞裂解液还可以具有以下至少一个附加技术特征。
根据本发明的实施例,上述细胞裂解液由蛋白酶K、NP-40和H
根据本发明的实施例,细胞裂解液中的蛋白酶K的浓度为2ng~200μg/mL,和/或,NP-40体积百分比浓度为1%~10%。分别对蛋白酶K和NP-40不同浓度组合的细胞裂解液进行测试,当细胞裂解液中蛋白酶K和NP-40的浓度组合分别为2ng/mL和1%、2ng/mL和10%、200μg/mL和10%、200μg/mL和1%、1μg/mL和5%、100ng/mL和1%、100μg/mL和10%时,细胞裂解液均能有效裂解细胞。基于已测试的多个不同浓度的蛋白酶K和NP-40的组合,有理由相信,为2ng~200μg/mL范围中的任意浓度的蛋白酶K和1%~10%范围中的任意浓度NP-40的组合,都能对细胞进行有效裂解。
根据本发明的实施例,细胞裂解液中的蛋白酶K浓度0.002~1μg/mL或者蛋白酶K浓度0.002~0.1μg/mL,也能保证细胞被有效裂解,而且能够进一步降低细胞裂解液成本。
上述任一实施例中的细胞裂解液组成简单,能有效的裂解细胞,特别适合少量动物细胞(1~200个细胞)的裂解,且裂解液中的成分不影响后续反应,如核酸片段化反应、扩增反应。从本发明的一个示例中采用上述任一实施例中细胞裂解液裂解细胞后进行建库测序的结果中的基因组覆盖度达99.54%可知,利用上述任一细胞裂解液能有效去除染色体上的蛋白质,从而去除染色体上的蛋白质对测序的影响。
本发明第二方面提供了一种细胞裂解方法,该方法包括利用上述第一方面或者任一实施例中细胞裂解液裂解细胞。上述对本发明第一方面或者任一实施例中的细胞裂解液的技术特征和优点的描述,也适用于该细胞裂解方法。
根据本发明的实施例,所述细胞裂解方法中的细胞为动物细胞,细胞的数量为1-200个,在一示例细胞的数量为1-10个。
根据本发明的实施例,所述细胞裂解方法于55℃~65℃进行裂解,和/或裂解时间为30min~60min。
根据本发明的实施例,所述细胞裂解方法还包括加入终止反应液终止所述裂解。终止反应液的种类包括蛋白酶抑制剂,用于终止蛋白酶K的活性,如PMSF(苯甲基磺酰氟),antipain(抗蛋白酶肽)等,在本发明的示例中,添加的终止反应液为PMSF溶液,为了避免蛋白酶抑制剂对后续实验的影响,作为终止反应液的PMSF浓度不易太高,选用的终止反应液的终浓度为0.5mM~1mM,所述的终浓度为终止反应液终止裂解反应时的反应浓度。
上述细胞裂解方法,反应条件简单、操作方便,能有效裂解少量动物细胞获得基因组DNA,且细胞裂解液和终止反应液中的成分不影响DNA片段化和扩增,裂解后的细胞溶液可直接用于后续实验。
本发明第三方面提供了核酸制备方法,该方法包括采用本发明第二方面或者任一示例中的细胞裂解方法获得细胞核酸。所述的核酸为细胞的DNA和/或RNA。在示例中,所述的核酸为DNA。利用此方法制备的DNA上无蛋白质,可直接用于制备DNA文库,且无需经过纯化去除细胞裂解液和终止反应液。上述对本发明第二方面或者任一实施例中的细胞裂解方法的技术特征和优点的描述,也适用于该核酸制备方法。
本发明第四方面提供了核酸文库制备方法,该方法包括如下步骤:利用本发明第三方面或者任一示例中的核酸制备方法获得核酸;以及片段化所述核酸。
在示例中,所述核酸文库制备方法还包括扩增片段化后的核酸,获得所述核酸文库。
根据本发明的实施例,该方法的步骤之间无纯化或分离操作,即分别在获得核酸和片段化所述核酸步骤后都无纯化操作或分离操作,获得核酸后加入片段试剂进行片段化处理,片段化后的产物直接用于扩增。在核酸文库制备方法中利用片段化试剂进行所述片段化,当核酸为DNA时,利用片段化试剂对DNA进行片段化。可以理解,有多种片段化DNA的方法,如超声打断、DNA片段化试剂打断,其中DNA片段化试剂包括转座酶复合体、DNA打断酶等。对于少量细胞获得的DNA优选DNA片段化试剂对DNA片段化。DNA打断酶可选自天根DNA片段化试剂盒NG305-02、Qiagen CM0162片段化酶试剂盒、NEB片段化酶等具有使DNA片段化的核酸内切酶或内切酶组合等;转座酶复合体包括Tn转座复合体、Mu转座复合体等具有转座功能的复合体,在示例中,所述的转座酶复合体为Tn转座复合体,具体为Tn5转座酶复合体。Tn5转座酶复合体是由Tn5转座酶和转座接头形成的复合体,转座接头由Tn5转座酶识别序列组成或由Tn5转座酶识别序列和一段已知的用于PCR引物识别的序列组成。
上述核酸文库制备方法是利用本发明第三方面或任一示例中提供的核酸制备方法获得核酸,上述对本发明第三方面或者任一示例中的核酸制备方法的技术特征和优点的描述,也适用该核酸文库制备方法。
在示例中,细胞液裂解细胞后,在原反应管中加入终止反应液终止裂解反应,获得核酸;在同一反应管中,加入片段化试剂片段化所述核酸;在同一反应管中,加入扩增反应试剂进行扩增,获得核酸文库。该方法的步骤之间无纯化操作或分离操作,各步骤中的反应在同一反应管中进行。从实验结果可知,细胞裂解液和终止反应液不影响片段化试剂作用效果和扩增效果。利用此核酸文库制备方法,从细胞液裂解细胞到获得DNA文库耗时约3.5h;利用包括两次扩增和多次纯化步骤的核酸文库制备方法获得核酸文库,(裂解细胞获得核酸;MDA(多重置换扩增)技术扩增细胞裂解获得的核酸;片段化核酸;纯化片段化产物;PCR扩增纯化产物;纯化PCR扩增产物,获得核酸文库。)耗时约16h,所用时间约为本发明第四方面或任一实施例中提供的核酸制备方法所用时间的5倍。本发明第四方面或任一实施例中提供的核酸制备方法操作简单、快速,并且利用此方法制备的文库可用于测序。
本发明第五方面提供了一种测序方法,该方法包括将本发明第四方面或任一示例中制备的核酸文库用于测序,获得测序结果。利用上述任一示例中的细胞裂解方法获得的核酸可用于构建不同测序平台的文库,如Illumia公司MiSeq和HiSeq测序平台(HiSeq-2500、Hiseq-2000,NextSeq 500、HiseqX10等测序仪)、PacBio测序平台(PacBio RS系列、Sequel系列测序仪)、华大基因测序平台(MGISEQ-2000、MGISEQ-200、BGISEQ-500、DNBSEQ-T7等测序仪)等。
在一些实施例中,利用上述任一实施例中的细胞裂解方法获得核酸,或者任一示例的构建核酸文库利用发明人搭建的测序平台测序,可检测出不小于1M的CNV突变,且测序结果中的GC bias,MAD(中位数绝对偏差)相较于采用包括两次扩增和多次纯化步骤的核酸文库制备方法获得核酸文库并利用高通量测序平台测序的测序结果中的低。其中,包括两次扩增和多次纯化步骤的核酸文库制备方法包括:裂解细胞获得核酸;MDA技术扩增细胞裂解获得的核酸;片段化核酸;纯化片段化产物;PCR扩增纯化产物;纯化PCR扩增产物,获得核酸文库。在实施例中,采用Illumina HiSeqX10高通量测序平台进行高通量测序。发明人搭建的测序平台是参考CN201610209150.2、CN201710607295.2、CN201610958047.8、CN201710607306.7、CN201620278363.6、CN201520614342.2、CN201520614143.1、CN201520614143.1、CN201711105069.0和/或CN201621419140.3公开的一个或多个方案进行的,操作流程例如可参考https://doi.org/10.1371/journal.pone.0188181(Singlemolecule sequencing of the M13 virus genome without amplification)进行。
本发明第六方面提供了一种试剂盒,试剂盒包含本发明第一方面或任一示例中的的细胞裂解液。试剂盒中的细胞裂解液的有效成分可选择以不同形式储存,如蛋白酶K和NP-40分别单独以高浓度形式储存,或者以两种成分混合后的高浓度形式储存,亦或者以混合后合适浓度形式储存。根据两种试剂在试剂盒中不同的储存形式,使用细胞裂解液时选择不同的处理方法,最终将两种物质配置成合适浓度的混合物,其中合适浓度指的是蛋白酶K的浓度为2ng~200μg/mL,NP-40的体积百分比浓度为1%~10%;高浓度指的是两种物质中的一种或两种的浓度高于各自合适浓度的最高值。
根据本发明的实施例,上述试剂盒还包括终止反应液,所述的终止反应液用以终止细胞裂解反应。终止反应液可为蛋白酶抑制剂,如PMSF(苯甲基磺酰氟),antipain(抗蛋白酶肽)等,用于终止蛋白酶K的活性。在示例中,试剂盒提供的终止反应液为PMSF。试剂盒中提供的PMSF的浓度不低于5mM,或着,试剂盒中提供的PMSF为固体,使用时配置成浓度不低于5mM的PMSF的水溶液。
根据本发明的实施例,上述试剂盒还包括核酸片段化试剂,核酸片段化试剂用以使核酸片段化或者使核酸片段化的同时使片段化的核酸两端或一端添加一段已知序列。核酸片段化试剂的种类包括转座酶复合体、DNA打断酶等。对于微量DNA(<50ng)而言,优选DNA片段化试剂对DNA片段化。DNA打断酶可选自天根DNA片段化试剂盒NG305-02、QiagenCM0162片段化酶试剂盒、NEB片段化酶等具有使DNA片段化的核酸内切酶或内切酶组合等;转座酶复合体选自Tn转座复合体、Mu转座复合体等具有转座功能的复合体。在示例中,DNA片段化试剂为Tn5转座酶复合体,Tn5转座酶复合体是由Tn5转座酶和转座接头形成的复合体,转座接头由Tn5转座酶识别序列组成或由Tn5转座酶识别序列和一段已知的用于PCR引物识别的序列组成。
利用上述试剂盒构建核酸文库时,细胞裂解液和终止反应液不影响片段化试剂作用效果和扩增效果,细胞裂解后得到的核酸无需纯化,可直接用于核酸文库构建。利用此试剂盒构建文库,可以做到从细胞裂解到扩增,反应在一个反应管中进行,不需要更换反应管。利用此试剂盒构建核酸文库一方面简化操作步骤、减少成本,另一方面减少DNA的损失,适用少量样本的文库构建。上述任一实施例中的细胞裂解液、细胞裂解方法、核酸制备方法和核酸文库制备方法的优点的描述也适用该试剂盒。
本发明第七方面提供了试剂盒在涉及细胞裂解的方法或装置中的应用包括应用于胚胎植入前遗传学筛查。利用此试剂盒进行胚胎植入前遗传学筛查的结果和利用高通量测序进行胚胎植入前遗传学筛查的结果一致,能检测到不小于1M的CNV(拷贝数变异)的染色体突变。
上述本发明任一方面或任一实施例中提供的细胞裂解液的组成简单,且能够有效去除基因组上的蛋白,且裂解后的细胞溶液不用纯化,直接用于DNA文库构建。
利用该细胞裂解液的细胞裂解方法能够快速地获得基因组DNA。
利用该细胞裂解液的文库制备方法能快速的制备出DNA文库,且制备的文库能满足不同测序平台的要求。
利用上述任一实施例提供的细胞裂解液进行细胞裂解的方法、文库构建方法、测序方法等,由于操作简单,特别适合于临床。
本申请的实施方式的附加方面和优点将在下面的描述中部分给出,部分将从下面的描述中变得明显,或通过本申请的实施方式的实践了解到。
附图说明
图1实施例中文库经LabChip GX Touch(PerkinElmer)微流控毛细管电泳检测结果;
图2实施例中文库经LabChip GX Touch(PerkinElmer)微流控毛细管电泳检测结果;
图3对比实施例中文库经LabChip GX Touch(PerkinElmer)微流控毛细管电泳检测结果;
图4对比实施例中扩增产物1琼脂糖电泳检测图;
图5Illumina HiseqX10和发明人搭建的单分子测序平台对chES497样本的测序分析结果;
图6Illumina HiseqX10和发明人搭建的单分子测序平台对chES497样本的测序分析结果。
具体实施方式
下面参考具体实施例和附图,对本发明进行说明。需要说明的是,这些实施例仅仅是说明性的,而不能理解为对本发明的限制。实施例中未注明具体技术或条件的,按照本领域内的文献所描述的技术或条件或者按照产品说明书进行。所用试剂或仪器未注明生产厂商者,均为可以通过市场购买获得的常规产品。实施例中采用的细胞样品用于验证细胞裂解方法和文库制备方法的可行性,所用的细胞不局限于实施例中所选用的细胞系,实施例2、3和4所用的细胞系90-P50和chES497(46,XX,del(1p36.33-1p36.32)1.29M,dup(16p13.3-16.13.11)16.72M)均来源于湖南光琇医院。
本文所称的的“高通量测序”指的是能一次并行对几十万到数百万条核酸分子进行序列测定。测序数据中GC bias(GC偏好性)数值是判断测序效果的重要指标,GC bias越低表明由于文库构建和测序时引入的GC偏差越低,对不同GC组成的DNA分子和富含GC或少含GC的染色体测序数据的偏差降低。测序数据中的MAD(中位数绝对偏差)与MAD常规定义相同,对于测序数据而言,指用原数据减去中位数后得到的新数据的绝对值的中位数,用来衡量数据的离散程度,MAD数值越小,表明测得数据离散程度越低,无异常的数据分布越集中,离散的数据更易分辨。
实施例1
以下示例制备试剂盒的方法:
试剂盒包含管1,管1为细胞裂解液,该细胞裂解液的有效成分由蛋白酶K和NP-40组成,两者的浓度分别为2ng~200μg/mL和1%~10%,其中1%~10%为体积百分比浓度。分别对蛋白酶K和NP-40不同浓度组合的细胞裂解液进行测试,当细胞裂解液中蛋白酶K和NP-40的浓度组合分别为2ng/mL和1%、2ng/mL和10%、200μg/mL和10%、200μg/mL和1%、1μg/mL和5%、100ng/mL和1%、100μg/mL和10%时,细胞裂解液均能有效裂解细胞。试剂盒中的蛋白酶K和NP-40为市售常规试剂,如Sigma公司货号为74385的NP-40,Merck Millipore公司货号为539480-100mg的蛋白酶K,两种试剂不含DNA酶活性且无其它影响酶活的物质存在,如SDS等。
在一种情况下,细胞裂解液含有的两种有效成分蛋白酶K和NP-40分别以高浓度形式存放,使用细胞裂解液时,将两种物质混合配置成合适浓度的混合物;另一种情况下,细胞裂解液以高浓度混合物的形式存放,使用细胞裂解液时,用去离子水将高浓度混合物稀释成合适浓度的混合物。合适浓度指的是蛋白酶K的浓度为2ng~200μg/mL,NP-40的体积百分比浓度为1%~10%。高浓度的混合物,指的是两种物质中的一种或两种的浓度高于合适浓度。
在一种情况下,试剂盒还包含管2,管2为终止反应液,终止反应液用于终止蛋白酶K的活性。终止反应液可为蛋白酶抑制剂,如PMSF(苯甲基磺酰氟),antipain(抗蛋白酶肽)等。管2中的PMSF溶液的浓度为不低于5mM,使用时的终浓度为0.5mM~1mM,即将PMSF溶液加入到细胞裂解液终止细胞裂解反应时的浓度为0.5mM~1mM。
利用含有管1和管2的试剂盒裂解动物细胞,获得细胞中的DNA。该试剂盒成分简单且用于理解细胞操作方便,可有效裂解细胞获得DNA。获得的DNA不用纯化,直接用于文库构建,这样的操作避免了少量细胞的核酸经纯化后核酸量减少的问题。该试剂盒适用于单细胞/微量细胞样品或胚胎植入前遗传学筛查样品的DNA提取。
在一种情况下,试剂盒还包含管3,管3为DNA片段化试剂,片段化试剂的种类转座酶复合体、DNA打断酶等,DNA打断酶可选自天根DNA片段化试剂盒NG305-02、Qiagen CM0162片段化酶试剂盒或者NEB片段化酶;转座酶复合体可选择Tn转座复合体、Mu转座复合体等。在一种情况下,管3的DNA片段化试剂为Tn5转座酶复合体,Tn5转座酶复合体是由Tn5转座酶和转座接头形成的复合体,转座接头的序列由Tn5识别序列组成或由Tn5识别序列和一段已知的用于PCR引物识别的序列组成。
利用包含管1、2和3的试剂盒构建测序文库时,细胞裂解后得到的DNA无需纯化即可进行文库构建,细胞裂解液和终止反应液对片段化试剂尤其对Tn5转座酶复合体无影响。利用此试剂盒构建DNA文库,从细胞裂解到扩增,反应在一个反应管中进行,这样一方面可简化操作步骤、减少成本,另一方面减少DNA的损失。
在一种情况下,试剂盒还包含扩增试剂和纯化试剂。
实施例2
本实施例提供了实施例1中的试剂盒在文库构建和Illumina公司高通量测序平台测序中的应用。
1.细胞裂解
1)少量细胞样本的分离
可通过显微操作仪、口吸管等技术进行少量细胞(1~200个细胞)样本的分离,获得的少量90-P50的正常细胞(1~200个细胞)样本,样本中含PBS(pH7.2~7.4),总体积不超过4μL。
2)细胞裂解
取6μL实施例1中制备细胞裂解液加入细胞样本中进行混合,55℃~65℃孵育30min~60min,本实施例中采用的细胞裂解液的蛋白酶K和NP-40浓度分别为1μg/mL和5%、100ng/mL和1%、2ng/mL和1%,选用的三种组合的细胞裂解液都可对细胞进行有效裂解,为了保证实验条件的统一性,实施例2和实施例3采用的细胞裂解液的蛋白酶K和NP-40浓度分别为100ng/mL和1%。
3)终止细胞裂解
向步骤2)中溶液中加入终止反应液PMSF(苯甲基磺酰氟),PMSF终浓度为0.5mM~1mM,置室温反应15min~35min,得到裂解产物。
2.文库构建
采用南京诺唯赞试剂盒(Cat NO.S111-C3,Vazyme)的试剂进行文库构建,具体步骤如下:
1)基因组DNA片段化
采用南京诺唯赞的试剂盒(Cat NO.S111-C3,Vazyme)的试剂对细胞裂解步骤3)中的裂解产物片段化,向裂解产物中加入5×片段化缓冲液4μL,片段化复合体C3 1μL,补水至20μL,混匀,放置PCR仪中,37℃~55℃孵育10~30min,反应结束后加入5×终止反应缓冲液5μL,混匀,室温反应5~7min。
2)PCR扩增
向文库构建步骤1)中的片段化产物中加入PCR反应体系,PCR试剂为南京诺唯赞试剂盒(CatNO.S111-C3,Vazyme)中的试剂,PCR反应体系包含5×TAB扩增缓冲液10μL,TAE 1μL,引物1 2μL,引物2 2μL,补水至50uL,混匀,置于预热的PCR仪中。PCR扩增流程设置如表1所示。
表1
3)纯化
采用南京诺唯赞的纯化磁珠试剂盒(Cat NO.N411-03,Vazyme)对PCR产物进行纯化。PCR产物体系若不足50μL应加水补齐至50μL,随后进行两轮纯化,第一轮纯化采用35μL0.7×VAHTS DNA CleanBeads(0.7×纯化磁珠),第二轮纯化采用7.5μL 0.15×VAHTS DNAClean Beads(0.15x纯化磁珠)。纯化按照说明书操作说明或按照磁珠纯化常规纯化步骤进行,最终洗脱体积为20μL,得到DNA文库。
3.测序
1)文库检测
将获得的DNA文库用Qubit
2)Illumina HiseqX10测序
测序结果显示以该方法构建的DNA文库在测序深度为30.75×的情况下,所得unqiue数据总量((唯一比对的数据总量))约为614.98M,基因组的覆盖度(coverage)高达99.54%,GC bias为0.06。从测序结果中基因组的覆盖度(coverage)高达99.54%可判断利用实施例中的细胞裂解液裂解细胞可有效去除基因组上的蛋白,从而避免了基因组上的蛋白对片段化的影响及对测序结果覆盖度的影响,具体测序结果见表2所示。
表2
实施例3
本实施例为实施例1中的试剂盒在细胞裂解、文库构建和单分子测序平台中的应用。可参考CN201610209150.2、CN201710607295.2、CN201610958047.8、CN201710607306.7、CN201620278363.6、CN201520614342.2、CN201520614143.1、CN201520614143.1、CN201711105069.0和/或CN201621419140.3公开的一个或多个方案进行单分子测序平台的搭建,操作流程例如可参考https://doi.org/10.1371/journal.pone.0188181(Singlemolecule sequencing of the M13virus genome without amplification)。
1.细胞裂解
1)少量细胞样本的分离
可通过显微操作仪、口吸管等技术进行少量细胞样本的分离,获得5~10个CNV(拷贝数变异)细胞系chES497(46,XX,del(1p36.33-1p36.32)1.29M,dup(16p13.3-16.13.11)16.72M)的细胞,并将细胞置于1-4μL PBS(pH7.2~7.4)溶液中获得细胞样本。
2)细胞裂解
取6μL实施例1中制备细胞裂解液加入细胞样本中进行混合,55℃~65℃孵育30min~60min,本实施例中采用的细胞裂解液蛋白酶K和NP-40浓度分别为100ng/mL和1%。
3)终止细胞裂解
向步骤2)中溶液中加入终止反应液(PMSF,苯甲基磺酰氟)PMSF终浓度为0.5mM~1mM,置室温反应15min~35min,得到裂解产物。
2.文库构建
采用真迈生物(全称:深圳市真迈生物科技有限公司)单细胞全基因组扩增试剂盒(CatNO.C0BD01-01AA,GeneMind Biotech)进行文库构建,具体步骤如下:
1)基因组DNA片段化
采用真迈生物(全称:深圳市真迈生物科技有限公司)单细胞全基因组扩增试剂盒(CatNO.C0BD01-01AA,GeneMind Biotech)对本实施例细胞裂解步骤3)中的裂解产物片段化,向裂解产物中加入5×片段化缓冲液4μL,片段化复合体1μL,补水至20μL,混匀,放置PCR仪中,37℃~55℃孵育10~30min,反应结束后加入5×终止反应缓冲液5μL,混匀,室温反应5~7min。
2)PCR扩增
向本实施例文库构建步骤1)中的片段化产物中加入PCR反应体系,PCR反应体系包含5×PCR扩增缓冲液10μL,1U/μL PCR扩增酶1μL,引物1 2μL,引物2 0.2μL,补水至50uL,混匀,置于预热的PCR仪中。PCR扩增流程设置如表3所示。
表3
3)纯化
采用南京诺唯赞的纯化磁珠试剂盒(Cat NO.N411-03,Vazyme)对PCR产物进行纯化。PCR产物体系若不足50μL应加水补齐至50μL,随后进行两轮纯化,第一轮纯化采用35μL0.7×VAHTS DNA CleanBeads(0.7×纯化磁珠),第二轮纯化采用7.5μL 0.15×VAHTS DNAClean Beads(0.15x纯化磁珠)。纯化按照说明书操作说明或按照磁珠纯化常规纯化步骤进行,最终洗脱体积为20μL,得到DNA文库。
3.测序
1)文库检测
将获得的DNA文库用Qubit
2)单分子测序仪测序
采用发明人搭建的单分子测序平台进行测序,测序结果显示以该方法构建的DNA文库测序获得unique数据总量(唯一比对的数据总量)为5.5M,coverage(基因组覆盖度)为7.7%,GC bias为0.05及MAD(median absolute deviation,中位数绝对偏差)为0.21,具体结果见表4。
表4
注:chES497-GC:实施例3中的细胞样本构建的DNA文库,利用发明人搭建的单分子测序平台进行测序;chES497-NGS:对比实施例中细胞样本构建的DNA文库,利用IlluminaHiseqX10测序平台进行测序。
对比实施例
本实施例为实施例3的对比实验,本实施例和实施例3采用相同的细胞样本,采用不同的细胞裂解方法、文库构建方法和测序平台对细胞样本进行文库构建和测序。
对比实施例是目前PGS常使用的方法。目前胚胎植入前筛查方法是采用囊胚滋养层3~5个细胞为活检材料,然后通过各种基因筛查方法,如荧光原位杂交(Fish),聚合酶链式反应(PCR)进行筛查染色体异常检测。基于高通量测序平台的PGS(胚胎植入前遗传学筛查),其数据要求单个样本数据覆盖度不低于4%,单个样本数据量的要求是不低于1M(根据各个平台的测序读长而定)。检测性能需要达到异常片段大小大于4M的阳性参考品对应的染色体异常要求检出率达到100%,异常片段大小小于等于4M的阳性参考品对应的染色体异常要求检出率达到30%以上。
对比实施例中细胞裂解、文库构建和测序步骤如下:
1细胞裂解
1)少量细胞样本的分离
可通过显微操作仪、口吸管等技术进行少量细胞样本的分离,获得5~10个CNV(拷贝数变异)细胞系chES497(46,XX,del(1p36.33-1p36.32)1.29M,dup(16p13.3-16.13.11)16.72M)的细胞,并将细胞置于1-4μL PBS(pH7.2~7.4)溶液中获得细胞样本,随后采用Discover-sc single-cell kit(Cat no.N601-02,Vazyme)进行细胞裂解和文库构建。
2)细胞裂解
取3μLbufferD2(4μL1MDTT加入至36μL的buffer D)加入至细胞样本内,轻弹管壁混匀并短暂离心,65℃孵育10min;
加入3μL的buffer N,混匀终止裂解反应,并将反应管至于冰上,得到细胞裂解产物。
2.文库构建
1)MDA(多重置换扩增)技术扩增
将提前准备的反应混合液(如表5所示)40μL加入到步骤2)10μL的细胞裂解产物中,混匀并短暂离心,30℃孵育6小时。
表5
扩增结束后,65℃孵育3min,使DNA扩增酶失活,得到扩增产物1。
2)扩增产物1检测
将扩增产物1稀释10倍分别进行Qubit
3)DNA片段化
利用试剂盒TruePrep DNA Library Prep Kit V2for Illumina(Cat NO.TD501,Vazyme)对扩增产物1进行片段化,具体操作为:取50ng扩增产物1加入5×TTBL 4μL,TTEMixV50 1.5μL,补水至20μL,混匀,放置PCR仪中,55℃孵育10min,得到片段化产物。
4)纯化
向片段化产物中键入20μL VAHTS DNA Clean Beads(Cat NO.N411-03,Vazyme)进行纯化,纯化步骤按照说明书操作说明或按磁珠常规纯化步骤进行,最终洗脱体积为24μL,得到纯化产物1。
5)PCR扩增
向纯化产物1中加入PCR反应体系,PCR反应体系包含5×TAB10μL,TAE 1μL,N5-XX5μL(XX表示标签位置编号),N7-XX(XX表示标签位置编号)5μL,PPM 5μL,补水至50μL,混匀,置于预热的PCR仪中,PCR扩增流程设置如表6所示。
表6
6)纯化
采用南京诺唯赞的纯化磁珠试剂盒(Cat NO.N411-03,Vazyme)对PCR产物进行纯化。步骤5)PCR产物体系不足50μL应补齐至50μL,随后进行两轮纯化,第一轮纯化采用0.7×VAHTS DNA Clean Beads(0.7×纯化磁珠),第二轮纯化采用7.5μL 0.15×VAHTS DNACleanBeads(0.15x纯化磁珠)。纯化步骤按照说明书操作说明或按磁珠常规纯化步骤进行,最终洗脱体积为20μL,得到DNA文库。
3.测序
1)文库检测
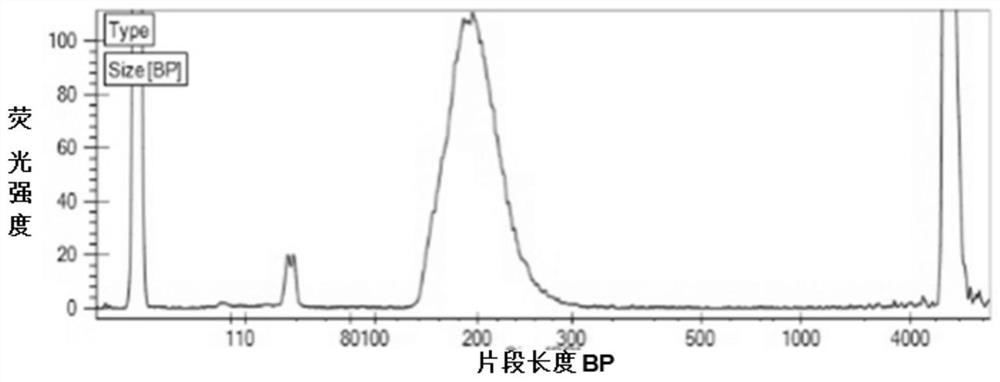
将获得的DNA文库用Qubit dsDNA BR Assay Kit(Cat No.Q32853,InvitrogenBiotech.)进行浓度检测和LabChip GX Touch(PerkinElmer)进行微流控毛细管电泳检测,检测的DNA文库核酸片段主带大小在250~450bp左右。图3为文库经生物分析仪LabChip GXTouch检测结果,横坐标为片段长度,单位为BP,纵坐标为荧光强度,检测文库浓度。
2)Illumina HiseqX10测序
测序结果显示以该方法构建的DNA文库测序获得unique数据总量(唯一比对的数据总量)为4.9M,coverage(基因组覆盖度)为13.31%,利用该方法所得的数据质量及分析结果,具体结果见表4。
测序结果以hg19(正常人染色体,可在
据上面测序结果可知,利用实施例1中试剂盒进行细胞裂解并利用发明人搭建的单分子测序平台对少量细胞如囊胚期细胞进行裂解、建库和测序,测序结果可用于分析细胞的CNV,检测的CNV的大小有大于10M,也有约1M,检测结果与HiseqX10检测的结果一致,符合PGS检测标准。所以,利用本发明实施例1中的试剂盒进行细胞裂解并利用发明人搭建的单分子测序平台进行测序可用于PGS(胚胎植入前遗传学筛查)。同时,利用两种不同的平台进行PGS时,传统的PGS建库流程建库耗时~16h时间,而采用本发明实施例1中的试剂盒进行细胞裂解建库耗时约3.5h,所以利用本发明实施例中裂解建库方法可以大大缩短检测时间。从表4可看出,两种方法测序结果的GC bias(GC偏好)不同,利用本发明实施例1中的试剂盒进行细胞裂解建库,发明人搭建的单分子测序平台测序结果GC bias明显低于HiseqX10建库测序结果;同时,两种方法测序结果MAD(中位数绝对偏差)不同,利用本发明实施例1中的试剂盒进行细胞裂解建库测序的MAD明显小于HiseqX10建库测序结果,该值越小,从检测结果中越容易分析染色体的片段缺失情况,如在5中,箭头所指位置为CNV的位置,对比实施例中的测序结果(chES497-NGS)CNV的位置周边还有其它位点显示,影响CNV的判断,相反,实施例3中的测序结果(chES497-GC)CNV的位置很明显,易判断。
在本说明书的描述中,参考术语“一个实施例”、“一些实施例”、“示例”、“具体示例”、或“一些示例”等的描述意指结合该实施例或示例描述的具体特征、结构、材料或者特点包含于本发明的至少一个实施例或示例中。在本说明书中,对上述术语的示意性表述不必针对的是相同的实施例或示例。而且,描述的具体特征、结构、材料或者特点可以在任一个或多个实施例或示例中以合适的方式结合。此外,在不相互矛盾的情况下,本领域的技术人员可以将本说明书中描述的不同实施例或示例以及不同实施例或示例的特征进行结合和组合。
尽管上面已经示出和描述了本发明的实施例,可以理解的是,上述实施例是示例性的,不能理解为对本发明的限制,本领域的普通技术人员在本发明的范围内可以对上述实施例进行变化、修改、替换和变型。
- 一种细胞裂解液、试剂盒及在制备肿瘤全细胞抗原负载DC肿瘤疫苗中的应用
- 一种用于将拭子上的细胞洗脱、裂解释放核酸的洗脱裂解液、试剂盒和方法
