COVID-19的检测、诊断和治疗方法
文献发布时间:2023-06-19 19:28:50
技术领域
本发明属于生物医药技术领域,具体而言属于免疫细胞治疗领域。
背景技术
严重急性呼吸系统综合征冠状病毒2(SARS-CoV-2),已被世界卫生组织正式命名为2019冠状病毒病(COVID-19)。SARS-CoV-2的基因组序列与SARS-CoV的基因组序列具有79.5%的同源性。与SARS-CoV和MERS-CoV一样,SARS-CoV-2属于冠状病毒科的β属冠状病毒。
T细胞介导的免疫应答是适应性免疫系统抵抗病毒病原体的主要防御机制之一。T细胞根据克隆型T细胞受体(TCR)指示的信号协调适应性免疫,TCR识别组织相容性复合体(MHC)所呈现的短肽(8–15个氨基酸)。TCR识别短肽-MHC复合体(pMHC)后在感染急性期诱导原始T细胞活化和分化为各种功能亚群,从而消除入侵病原体。
TCR的两条链(α和β)分别由可变(V)区、连接(J)区和常数(C)区组成。多样性(D)区域连接V和J区域并形成完整的β链。TCR重组过程产生高度多样的互补决定区(CDR),定位于 TCR的α和β链,形成功能性和高度多样的TCR序列。CDR序列决定了TCR与pMHCs结合的特异性,其中第三个CDR(CDR3)可变性最高,有助于肽直接识别。在病毒感染期间,CD8
尽管在阐明SARS-CoV-2感染过程中的免疫反应方面已经做了相当大的努力,但对于具有不同 TCR库的抗原特异性T细胞在人体内针对病毒的免疫应答知之甚少。在刚感染COVID-19患者的免疫系统中发现针对该病毒的反应性T细胞,并且T细胞随着时间的推移而增加。然而,严重感染 COVID-19患者临床检查的初步数据显示,随着患者病情的恶化,CD4
本发明提供一种结合scTCR-seq和scRNA-seq分析来测量COVID-19康复期个体的TCR克隆动态和功能基因表达,以表明抗原特异性T细胞在抗SARS-CoV-2感染中发挥决定性作用。系统分析显示了三个关键发现:(1)从COVID-19中康复后,一周内TCR基因库多样性迅速下降,但VDJ基因的使用的频率不变;(2)T细胞的基因表达特征随着康复后的时间推移,发生从抗病毒免疫反应到代谢相关通路的转变;(3)占总T细胞~10%的高扩增T细胞克隆代表了CD8
发明内容
本发明提供一种结合scTCR-seq和scRNA-seq分析来测量COVID-19康复期个体的TCR克隆动态和功能基因表达,以表明抗原特异性T细胞在抗SARS-CoV-2感染中发挥决定性作用。鉴于此,本申请提供以下技术方案:一方面,一种COVID-19患者治疗效果的检测和/或评估方法,所述的方法包括检测患者中T细胞受体组库和/或T细胞功能。
进一步的,所述的评估方法包括分离患者外周血单个核细胞(PBMC)以构建cDNA文库,并进行TCR-α/-β转录测序。
进一步的,所述的TCR-α/-β转录测序使用基于10X基因组的单细胞测序平台。
进一步的,所述T细胞受体组库包括具有如下一种或多种序列的多肽:
CVVSAWEDNARLMF(SEQ ID NO:1);
CAVRVQGGKLIF(SEQ ID NO:2);
CAEGGGGSQGNLIF(SEQ ID NO:3);
CATDAYNTDKLIF(SEQ ID NO:4);
CALRDNKFYF(SEQ ID NO:5);
CAVNTGNQFYF(SEQ ID NO:6);
CATDPEPDNDMRF(SEQ ID NO:7);
CAGGSGGGADGLTF(SEQ ID NO:8);
CAVTGQAGTALIF(SEQ ID NO:9);
CATDSFFILYSGAGSYQLTF(SEQ ID NO:10);
CAEVLDGQKLLF(SEQ ID NO:11);
CAVPFAGGYQKVTF(SEQ ID NO:12);
CAASDVVGNKLVF(SEQ ID NO:13);
CAVSEPNNAGNMLTF(SEQ ID NO:14);
CAVSQAGTALIF(SEQ ID NO:15);
CAVPPTSGTYKYIF(SEQ ID NO:16);
CAGIDYGQNFVF(SEQ ID NO:17);
CAEQYSSASKIIF(SEQ ID NO:18);
CAVSGGGADGLTF(SEQ ID NO:19);
CAEIRVSGGSNYKLTF(SEQ ID NO:20);
CASLYSGAGSYQLTF(SEQ ID NO:21);
CVVNGRSSNTGKLIF(SEQ ID NO:22);
CALNTSGTYKYIF(SEQ ID NO:23);
CVVNGRSSNTGKLIF(SEQ ID NO:24);
CAASPANARLMF(SEQ ID NO:25);
CAASVTGNQFYF(SEQ ID NO:26);
CAYRSGAGSYQLTF(SEQ ID NO:27);
CAASLLTGANNLFF(SEQ ID NO:28);
CALNTSGTYKYIF(SEQ ID NO:29);
CAENGPGGGKLIF(SEQ ID NO:30);
CALSGSFSDGQKLLF(SEQ ID NO:31);
CAVNSYNTDKLIF(SEQ ID NO:32);
CATLDNYGQNFVF(SEQ ID NO:33);
CALSGGYSSASKIIF(SEQ ID NO:34);
CVVFRPNTGNQFYF(SEQ ID NO:35);
CAAPAQDTGRRALTF(SEQ ID NO:36);
CAMTAGGGNKLTF(SEQ ID NO:37);
CAESIRDYKLSF(SEQ ID NO:38);
CAVETSYDKVIF(SEQ ID NO:39);
CADSSGDTGRRALTF(SEQ ID NO:40);
CVVSENTGGFKTIF(SEQ ID NO:41);
CALNTGERGNQFYF(SEQ ID NO:42);
CAVEDSNYQLIW(SEQ ID NO:43);
CAASADFNKFYF(SEQ ID NO:44);
CAASYFGNEKLTF(SEQ ID NO:45);
CAVNVGGAALIF(SEQ ID NO:46);
CAVEPPSGTYKYIF(SEQ ID NO:47);
CAWQAGTALIF(SEQ ID NO:48);
CALSEASNYGQNFVF(SEQ ID NO:49);
CATSLIQGAQKLVF(SEQ ID NO:50);
CATDEGGSYIPTF(SEQ ID NO:51);
CAGDTGRRALTF(SEQ ID NO:52);
CAVGHNTDKLIF(SEQ ID NO:53);
CAVRTSSGTYKYIF(SEQ ID NO:54);
CATNSGGYQKVTF(SEQ ID NO:55);
CAVGHNTDKLIF(SEQ ID NO:56);
CAALPPFGNEKLTF(SEQ ID NO:57);
CVVTVYFGNEKLTF(SEQ ID NO:58);
CGTERLTGGGNKLTF(SEQ ID NO:59);
CAVNAVDNNNDMRF(SEQ ID NO:60);
CAVREDSGAGSYQLTF(SEQ ID NO:61);
CAVRTDYGGSQGNLIF(SEQ ID NO:62);
CAVRLGGATNKLIF(SEQ ID NO:63);
CATDPGNKLVF(SEQ ID NO:64);
CAVSVWGSARQLTF(SEQ ID NO:65);
CAGHPGGSEKLVF(SEQ ID NO:66);
CAVSAGAKTSYDKVIF(SEQ ID NO:67);
CAVRDGLNRDDKIIF(SEQ ID NO:68);
CAARPGSGYSTLTF(SEQ ID NO:69);
CAVKGGATNKLIF(SEQ ID NO:70);
CATDEGQGAQKLVF(SEQ ID NO:71);
CAMRESPAGGGNKLTF(SEQ ID NO:72);
CAARPGSGYSTLTF(SEQ ID NO:73);
CAMRITGGGNKLTF(SEQ ID NO:74);
CAGISGGYNKLIF(SEQ ID NO:75);
CALLGQAGTALIF(SEQ ID NO:76);
CAAPRDGANNLFF(SEQ ID NO:77);
CALSDVPSGSARQLTF(SEQ ID NO:78);
CIVYFGNEKLTF(SEQ ID NO:79);
CAVRSGSNYKLTF(SEQ ID NO:80);
CAVRDRGNNRLAF(SEQ ID NO:81);
CAVFRNYKLSF(SEQ ID NO:82);
CVVSEGSGYSTLTF(SEQ ID NO:83);
CALRSGGSNYKLTF(SEQ ID NO:84);
CIVRSPTGDSWGKLQF(SEQ ID NO:85);
CAVGAKGYQKVTF(SEQ ID NO:86);
CAGHNAGNNRKLIW(SEQ ID NO:87);
CALSRGSNYKLTF(SEQ ID NO:88);
CAGAKGNNDMRF(SEQ ID NO:89);
CAVRDITAAGNKLTF(SEQ ID NO:90);
CADYYGQNFVF(SEQ ID NO:91);
CAVWDSNYQLIW(SEQ ID NO:92);
CAAFLRGGRGSTLGRLYF(SEQ ID NO:93);
CAESIRRDKIIF(SEQ ID NO:94);
CAVLSGAGSYQLTF(SEQ ID NO:95);
CVVSGGGADGLTF(SEQ ID NO:96);
CVVSGGGADGLTF(SEQ ID NO:97);
CAERGMGSNTGKLIF(SEQ ID NO:98);
CAERRDGGQLTF(SEQ ID NO:99);
CAVGDSNYQLIW(SEQ ID NO:100);
CAVLNSNYQLIW(SEQ ID NO:101);
CVVNIPPTGGFKTIF(SEQ ID NO:102);
CAVTAKNDMRF(SEQ ID NO:103);
CIVPNAGNMLTF(SEQ ID NO:104);
CAVMDSNYQLIW(SEQ ID NO:105);
CAVGALNNNDMRF(SEQ ID NO:106);
CAVEERNQGGKLIF(SEQ ID NO:107);
CAVLYGNNRLAF(SEQ ID NO:108);
CAESIGLTGGGNKLTF(SEQ ID NO:109);
CVAGGYQKVTF(SEQ ID NO:110);
CAVIGGSYIPTF(SEQ ID NO:111);
CAMRENYGQNFVF(SEQ ID NO:112);
CVAGGYQKVTF(SEQ ID NO:113);
CAGPETSYDKVIF(SEQ ID NO:114);
CAVSNDYKLSF(SEQ ID NO:115);
CAGPETSYDKVIF(SEQ ID NO:116);
CAVGALNNNDMRF(SEQ ID NO:117);
CATDAGYGGATNKLIF(SEQ ID NO:118);
CAPGGNQAGTALIF(SEQ ID NO:119);
CAVMDSNYQLIW(SEQ ID NO:120);
CAVMDSNYQLIW(SEQ ID NO:121);
CAMKRSGGSNYKLTF(SEQ ID NO:122);
CAVRVPPGQKLLF(SEQ ID NO:123);
CAVGSPGNFGNEKLTF(SEQ ID NO:124);
CAVMDSNYQLIW(SEQ ID NO:125);
CATEGNFGNEKLTF(SEQ ID NO:126);
CAVVSYNTDKLIF(SEQ ID NO:127)。
另一方面,COVID-19康复患者中T细胞受体组库和T细胞功能的动态变化机制,包括:对健康组、出院组和随访组进行取样,分离外周血单个核细胞(PBMC)以构建cDNA文库,并使用基于10X 基因组的单细胞平台进行TCR-α/-β转录测序、数据分析。
进一步,健康组、出院组和随访组中,CDR3长度和TCRV/J使用频率相似。
进一步,从COVID-19中康复后,一周内TCR基因库多样性迅速下降,但VDJ基因的使用的频率不变。
进一步,T细胞的基因表达特征随着康复后的时间推移,发生从抗病毒免疫反应到代谢相关通路的转变。
进一步,占总T细胞~10%的高扩增T细胞克隆代表了CD8+T细胞的主要特征。
还一方面,所述的T细胞受体组库和T细胞功能的动态变化机制在抗原特异性T细胞抗 SARS-CoV-2感染中的应用。
还一方面,所述的T细胞受体组库和T细胞功能的动态变化机制在开发对抗SARS-CoV-2感染新型T细胞疫苗中的应用。
还一方面,包括和/或具有如下一种或多种序列的多肽在COVID-19诊断、预防或治疗中的应用:
CVVSAWEDNARLMF(SEQ ID NO:1);
CAVRVQGGKLIF(SEQ ID NO:2);
CAEGGGGSQGNLIF(SEQ ID NO:3);
CATDAYNTDKLIF(SEQ ID NO:4);
CALRDNKFYF(SEQ ID NO:5);
CAVNTGNQFYF(SEQ ID NO:6);
CATDPEPDNDMRF(SEQ ID NO:7);
CAGGSGGGADGLTF(SEQ ID NO:8);
CAVTGQAGTALIF(SEQ ID NO:9);
CATDSFFILYSGAGSYQLTF(SEQ ID NO:10);
CAEVLDGQKLLF(SEQ ID NO:11);
CAVPFAGGYQKVTF(SEQ ID NO:12);
CAASDVVGNKLVF(SEQ ID NO:13);
CAVSEPNNAGNMLTF(SEQ ID NO:14);
CAVSQAGTALIF(SEQ ID NO:15);
CAVPPTSGTYKYIF(SEQ ID NO:16);
CAGIDYGQNFVF(SEQ ID NO:17);
CAEQYSSASKIIF(SEQ ID NO:18);
CAVSGGGADGLTF(SEQ ID NO:19);
CAEIRVSGGSNYKLTF(SEQ ID NO:20);
CASLYSGAGSYQLTF(SEQ ID NO:21);
CVVNGRSSNTGKLIF(SEQ ID NO:22);
CALNTSGTYKYIF(SEQ ID NO:23);
CVVNGRSSNTGKLIF(SEQ ID NO:24);
CAASPANARLMF(SEQ ID NO:25);
CAASVTGNQFYF(SEQ ID NO:26);
CAYRSGAGSYQLTF(SEQ ID NO:27);
CAASLLTGANNLFF(SEQ ID NO:28);
CALNTSGTYKYIF(SEQ ID NO:29);
CAENGPGGGKLIF(SEQ ID NO:30);
CALSGSFSDGQKLLF(SEQ ID NO:31);
CAVNSYNTDKLIF(SEQ ID NO:32);
CATLDNYGQNFVF(SEQ ID NO:33);
CALSGGYSSASKIIF(SEQ ID NO:34);
CVVFRPNTGNQFYF(SEQ ID NO:35);
CAAPAQDTGRRALTF(SEQ ID NO:36);
CAMTAGGGNKLTF(SEQ ID NO:37);
CAESIRDYKLSF(SEQ ID NO:38);
CAVETSYDKVIF(SEQ ID NO:39);
CADSSGDTGRRALTF(SEQ ID NO:40);
CVVSENTGGFKTIF(SEQ ID NO:41);
CALNTGERGNQFYF(SEQ ID NO:42);
CAVEDSNYQLIW(SEQ ID NO:43);
CAASADFNKFYF(SEQ ID NO:44);
CAASYFGNEKLTF(SEQ ID NO:45);
CAVNVGGAALIF(SEQ ID NO:46);
CAVEPPSGTYKYIF(SEQ ID NO:47);
CAWQAGTALIF(SEQ ID NO:48);
CALSEASNYGQNFVF(SEQ ID NO:49);
CATSLIQGAQKLVF(SEQ ID NO:50);
CATDEGGSYIPTF(SEQ ID NO:51);
CAGDTGRRALTF(SEQ ID NO:52);
CAVGHNTDKLIF(SEQ ID NO:53);
CAVRTSSGTYKYIF(SEQ ID NO:54);
CATNSGGYQKVTF(SEQ ID NO:55);
CAVGHNTDKLIF(SEQ ID NO:56);
CAALPPFGNEKLTF(SEQ ID NO:57);
CVVTVYFGNEKLTF(SEQ ID NO:58);
CGTERLTGGGNKLTF(SEQ ID NO:59);
CAVNAVDNNNDMRF(SEQ ID NO:60);
CAVREDSGAGSYQLTF(SEQ ID NO:61);
CAVRTDYGGSQGNLIF(SEQ ID NO:62);
CAVRLGGATNKLIF(SEQ ID NO:63);
CATDPGNKLVF(SEQ ID NO:64);
CAVSVWGSARQLTF(SEQ ID NO:65);
CAGHPGGSEKLVF(SEQ ID NO:66);
CAVSAGAKTSYDKVIF(SEQ ID NO:67);
CAVRDGLNRDDKIIF(SEQ ID NO:68);
CAARPGSGYSTLTF(SEQ ID NO:69);
CAVKGGATNKLIF(SEQ ID NO:70);
CATDEGQGAQKLVF(SEQ ID NO:71);
CAMRESPAGGGNKLTF(SEQ ID NO:72);
CAARPGSGYSTLTF(SEQ ID NO:73);
CAMRITGGGNKLTF(SEQ ID NO:74);
CAGISGGYNKLIF(SEQ ID NO:75);
CALLGQAGTALIF(SEQ ID NO:76);
CAAPRDGANNLFF(SEQ ID NO:77);
CALSDVPSGSARQLTF(SEQ ID NO:78);
CIVYFGNEKLTF(SEQ ID NO:79);
CAVRSGSNYKLTF(SEQ ID NO:80);
CAVRDRGNNRLAF(SEQ ID NO:81);
CAVFRNYKLSF(SEQ ID NO:82);
CVVSEGSGYSTLTF(SEQ ID NO:83);
CALRSGGSNYKLTF(SEQ ID NO:84);
CIVRSPTGDSWGKLQF(SEQ ID NO:85);
CAVGAKGYQKVTF(SEQ ID NO:86);
CAGHNAGNNRKLIW(SEQ ID NO:87);
CALSRGSNYKLTF(SEQ ID NO:88);
CAGAKGNNDMRF(SEQ ID NO:89);
CAVRDITAAGNKLTF(SEQ ID NO:90);
CADYYGQNFVF(SEQ ID NO:91);
CAVWDSNYQLIW(SEQ ID NO:92);
CAAFLRGGRGSTLGRLYF(SEQ ID NO:93);
CAESIRRDKIIF(SEQ ID NO:94);
CAVLSGAGSYQLTF(SEQ ID NO:95);
CVVSGGGADGLTF(SEQ ID NO:96);
CVVSGGGADGLTF(SEQ ID NO:97);
CAERGMGSNTGKLIF(SEQ ID NO:98);
CAERRDGGQLTF(SEQ ID NO:99);
CAVGDSNYQLIW(SEQ ID NO:100);
CAVLNSNYQLIW(SEQ ID NO:101);
CVVNIPPTGGFKTIF(SEQ ID NO:102);
CAVTAKNDMRF(SEQ ID NO:103);
CIVPNAGNMLTF(SEQ ID NO:104);
CAVMDSNYQLIW(SEQ ID NO:105);
CAVGALNNNDMRF(SEQ ID NO:106);
CAVEERNQGGKLIF(SEQ ID NO:107);
CAVLYGNNRLAF(SEQ ID NO:108);
CAESIGLTGGGNKLTF(SEQ ID NO:109);
CVAGGYQKVTF(SEQ ID NO:110);
CAVIGGSYIPTF(SEQ ID NO:111);
CAMRENYGQNFVF(SEQ ID NO:112);
CVAGGYQKVTF(SEQ ID NO:113);
CAGPETSYDKVIF(SEQ ID NO:114);
CAVSNDYKLSF(SEQ ID NO:115);
CAGPETSYDKVIF(SEQ ID NO:116);
CAVGALNNNDMRF(SEQ ID NO:117);
CATDAGYGGATNKLIF(SEQ ID NO:118);
CAPGGNQAGTALIF(SEQ ID NO:119);
CAVMDSNYQLIW(SEQ ID NO:120);
CAVMDSNYQLIW(SEQ ID NO:121);
CAMKRSGGSNYKLTF(SEQ ID NO:122);
CAVRVPPGQKLLF(SEQ ID NO:123);
CAVGSPGNFGNEKLTF(SEQ ID NO:124);
CAVMDSNYQLIW(SEQ ID NO:125);
CATEGNFGNEKLTF(SEQ ID NO:126);
CAVVSYNTDKLIF(SEQ ID NO:127)。
本发明具有的技术效果为:
(1)从COVID-19中康复后,一周内TCR基因库多样性迅速下降,但VDJ基因的使用的频率不变;
(2)T细胞的基因表达特征随着康复后时间推移,发生从抗病毒免疫反应到代谢相关通路的转变;
(3)占总T细胞~10%的高扩增T细胞克隆代表了CD8
附图说明
图1研究流程。(a)研究设计的流程图。收集并分离四组外周血单个核细胞(PBMC):出院组(DC)、早期随访组(早期FU)、晚期随访组(晚期FU)和健康组(HD)。随访组分为早期FU组和晚期FU组。使用基于10x的基因组学平台,将PBMC用于scRNA和scTCR测序。长框表示研究中不同阶段的相同患者。(b)10名SARS-CoV-2感染者的病程时间表。
图2scRNA-seq数据的批次间效应校正。通过Seurat软件包(V3.6.0)的“FindIntergrationAnchors”和“IntegratedData”功能,合并的无批次效应(a)和具有批次效应(b)的样本的scRNA序列数据(出院组和HD组n=6,随访组n=7)。
图3总体CDR3长度和样本中的V/J分布。(a)(b)分别是HD组、出院组和随访组的TCRα链氨基酸长度和TCRβ链氨基酸长度(出院组和HD组n=6,随访组n=7)。(c)(d)分别是HD组、出院组和随访组TCRα链V区、J区使用率和TCRβ链V区、J区使用率(出院组和HD组n=6,随访组n=7)。
图4样品的克隆多样性。克隆多样性通过TCR多样性显示,每个样本的克隆多样性百分比= (TCR分类计数/总细胞数)*100%。环形图(a)和柱形图(b)显示了各组代表性样本的T细胞扩增克隆和克隆多样性百分比。
图5克隆多样性和扩增。(a)扩增克隆的比例,≥100次、50-99次、10-49次、5-9次、2-4次和唯一扩增克隆(unique)(出院组和HD组n=6,随访组n=7)。(b)3组的克隆多样性。(c)从代表性样品中top 10TCRs的总细胞数和每种TCR分类的细胞数比例,比率=每个TCR分类的细胞计数/ 细胞总数。(d)top 10TCRs细胞数与所有样本总细胞数的比率,比率=top 10TCRs细胞数/总细胞数。
图6 V-J基因配对。来自HD组(HD#3)(a)、出院组(出院#2)(b)和随访组(随访#3)(c) 代表性样本的TCRsα链V-J配对。来自HD组(HD#3)(上面板)、出院组(出院#2)(中面板)和随访组(随访#3)(下面板)代表性样本的TCRβ链V-J配对(d)。
图7在总TCR配对scRNA-seq数据中CD3E、CD4和CD8A的表达。通过小提琴图(a)和特征图(b)显示总TCRs配对scRNA-seq整合数据中CD3E、CD4和CD8A的表达(出院组和HD组n=6,随访组n=7)。Teff:效应器T;Tex:枯竭T细胞,Tcm:中央记忆T细胞;Tem:效应记忆T细胞;Tm:记忆T细胞,Tn:原始T细胞;Treg:常规T细胞。
图8top 20克隆在总TCRs配对scRNA-seq数据中的投影。UMAP显示集群中top 20个克隆的分布 (左)和集群的分布(右)。Teff:效应器T;Tex:枯竭T细胞,Tcm:中央记忆T细胞;Tem:效应记忆T细胞;Tm:记忆T细胞,Tn:原始T细胞;Treg:常规T细胞。
图9总TCRs配对scRNA-seq数据的每个簇中的基因表达。气泡图显示集群中标记基因的表达水平。用表达的基因鉴定簇:0:CD4
图10top 20名TCRs配对scRNA-seq数据中每个簇中的基因表达。气泡图显示集群中标记基因的表达水平。用表达基因对簇进行注释:0:CD8A
图11top 20TCRs克隆型配对的scRNA-seq数据中差异表达CD8+亚群的免疫相关基因。(a) 出院组与HD组的对比,top 20TCR克隆型中CD3E+CD8A+CD4-簇的相关基因中显著改变基因的火山图和选定颗粒酶和干扰素相关基因的热图,(b)为随访组与HD组(随访组n=7,HD组n=6),(c) 为出院组和随访组。(d)top 20TCR克隆型中CD3E
图12top 20TCRs配对scRNA-seq数据中CD3E、CD4和CD8A的表达。
通过小提琴图(a)和特征图(b)显示top 20TCR克隆型配对scRNA-seq整合数据中CD3E、 CD4和CD8A的表达(出院组和HD组n=6,随访组n=7)。Teff:效应器T;Tex:枯竭T细胞,Tcm:中央记忆T细胞;Tem:效应记忆T细胞;Tm:记忆T细胞,Tn:原始T细胞;Treg:常规T细胞。
图13top 20TCRs配对scRNA-seq数据中的样本分布。(a)UMAP显示样本在集群中的分布。(b) 条形图显示3组中每个簇的组成。Teff:效应器T;Tex:枯竭T细胞,Tcm:中央记忆T细胞;Tem:效应记忆T细胞;Tm:记忆T细胞,Tn:原始T细胞;Treg:常规T细胞。
图14对患者组与HD组top 20TCRs克隆型配对scRNA-seq数据的CD3E
图15出院组与HD组的top 20TCRs配对scRNA-seq数据中CD3E
图16出院组与HD组总CD3E
图17CD3E
图18随访组与HD组总CD3E
图19早期随访组与HD组top 20TCRs CD3E
图20通过IRIS3网站上的IRIS3对晚期随访组的CD8A
图21康复COIVD-19患者CD8
具体实施方式
本发明揭示COVID-19康复患者中T细胞受体组库和T细胞功能的动态变化机制,本领域技术人员可以借鉴本文内容,拓展研究T细胞受体组库和T细胞功能的动态变化机制。特别需要指出的是,所有类似的替换和改动对本领域技术人员来说是显而易见的,它们都被视为包括在本发明。本发明的方法及应用已经通过较佳实施例进行了描述,相关人员明显能在不脱离本发明内容、精神和范围内对本文所述的方法和应用进行改动或适当变更与组合,来实现和应用本发明技术。
实施例1 CDR3长度和TCR V/J分布的全局概况
样本采集
采集10名患者和6名健康献血者(HD)的外周血样本。6名患者在出院当天或出院前1周内取样为出院组(DC)。随访组(FU)在出院后1周至6周内采集样本。随访组按出院天数分为早期随访组(EFU)和晚期随访组(LFU)。其中,早期随访组的3名患者在出院后7天取样;晚期随访组包括4名患者,其中包括出院组的3名患者在出院后19至40天期间重新取样,1名患者在出院后30天取样(图1,表1)。这项研究是根据赫尔辛基宣言的原则进行的,并得到了中国上海交通大学医学院医学伦理委员会的批准。分离外周血单个核细胞(PBMC)以构建cDNA文库,并使用基于10X基因组的单细胞平台进行TCR-α/-β转录测序分析。
表1.COVID-19患者和健康献血者的临床和病理特征
PBMC分离,单细胞RNA测序及分析
使用Ficoll-Paque(GE Healthcare,Uppsala,Sweden)通过标准密度梯度离心制备分离PBMC,然后将制备的PBMCs用于构建scRNA-seq文库。按照铬单细胞5’v3试剂盒(10XGenomics,Pleasanton, USA)的说明操作。Cell Ranger软件(版本3.1.0)用于处理单细胞表达数据,并将数据与GRCH38 参考基因组对齐,其中相应的vdj包用于TCR v/d/j分析(表2)。通过Seurat(v3.6.0,R软件包)将样本的基因表达矩阵转换为Seurat对象。根据样本的变化,将每个样本应用以下标准:线粒体基因百分比<5%;基因数大于200但小于2200(对于#DC3),2500(对于#HD1/2/3),3000(对于#DC2/4/5/6 和来自随访组的样本),4000(对于#DC1和#HD4/5)和4500(对于#HD6)。此外,保留所有表达TCR α链、TCRβ链和转录RNA数据的细胞。过滤后,66352个细胞(HD组样本为 2740/2323/2239/3852/2744/3476;出院组样本为5237/5446/4483/2034/3412/3428;随访组样本为4824/2419/3419/4689/2859/3795/2933)被保留并用于后续分析(表3和表12-23)。通过Seurat软件包中的“NormalizeData”和“FindVariableFeatures”函数,使用前4000个可变基因对基因表达矩阵进行归一化和计算。使用“FindIntegrationAnchors”和“IntegrateData”功能将基因表达矩阵整合在一起,从CCA中获得50个维度,以纠正样本的批次效应(图2)。保存集成数据以供后续分析。并使用“ScaleData”函数中的默认参数缩放集成数据。主成分分析中使用了50个主成分,然后是UMAP。将“FindNeighbors”的top 20个主成分和以下“FindClusters”函数用于聚类。数据保存下来用于差异表达基因(DEG)分析。为了可视化CD3E、CD4和CD8A基因的表达,进行了“特征图”、“VlnPlot”和“CombinePlot”。通过CD3E、CD4和CD8A基因表达鉴定CD4
表2.由Cell Ranger分析得出的scRNA-seq/scTCR-seq协议和数据特征
表3.由Seurat软件包研究scRNA-seq/scTCR-seq协议和数据特征
首先评估了HD组、出院组和随访组的TCRα和β链的CDR3序列的长度分布,以绘制T细胞多样性图谱。HD组CDR3长度在5~28个氨基酸之间,在TCRα链13aa处峰值为19.76%;出院组显示 TCRα链CDR3长度在4~27个氨基酸之间,在TCRα链14aa处的峰值为22.03%;在随访组中TCRα链具有5~28个氨基酸之间,峰值为13aa(21.62%的细胞)(图3a)。HD组的TCRβ链CDR3长度在 5~26aa之间,随访组为5~27aa,出院组为5~28aa。三组峰值出现在15aa,HD组为24.71%、出院组为23.99%、随访组为25.21%(图3b)。
然后,评估了健康捐赠者和康复患者TCR V/J序列的使用情况。数据显示HD组中TRAV1-2和 TRAJ23表达水平较高。两个康复组在TCRα链结构中表现出TRAV1-2和TRAJ33的表达增加(图 3c)。TRBV20-1是康复患者中使用最多的TRBV基因,与TRBJ2-7具有相同的高表达模式(图3d) (表4)。HD组和康复组中使用最多的TRBV和TRBJ分别为TRBV9/TRBV20-1和TRBJ2-1/TRBJ2-7。综上所述,结果显示在健康供体和康复患者组中,CDR3长度和TCRV/J使用频率相似。
表4各组中TRAV/J使用频率最高
实施例2出院组表现出较高的TCR基因多样性和较低的TCR克隆扩增水平
为了进一步了解SARS-CoV-2感染后T细胞库的动态变化,分析了3组患者T细胞克隆扩增的特点。TCR分类计数(样本中不同TCR序列的数量)占总细胞数的百分比用于测量TCR多样性,即(TCR 分类计数/细胞总数)*100%。出院组(#2)代表性样本中的这一百分比(94.51%)远高于其他两个样本(HD#3为53.75%,随访组#3为61.21%)(图4a)。此外,高扩增克隆(≥100)、中度扩增克隆 (10-49)、小型扩增克隆(5-9)、很少扩增克隆(2-4)比HD组的比例降低,唯一扩增克隆(unique) 的比例增加,表明出院组的克隆型多样性较高(图4b,图5a)。此外,与HD组和随访组相比出院组未扩增的独特T细胞占82.95%。
接下来,计算3组中的TCRs多样性,发现出院组的TCRs多样性水平较高,在83.80%,而HD组的TCRs多样性水平在69.83%,随访组的TCRs多样性水平在66.33%(图5b)。同时还发现HD组 (32.87%)和随访组(35.98%)的扩增T细胞数量显著高于出院组(表5)的17.05%。然后计算每个TCR克隆型中的细胞数与代表性健康供者和康复患者组的总细胞数之比。HD组和随访组中top 10 TCRs克隆型的比例表现出可比的趋势,而出院组与top10TCRs克隆型的比例持续较低(图5c)。与随访组的0.1709相比,出院组TCRs总量top10TCRs的比例更低(0.0803)(图5d)。这些数据表明, SARS-CoV-2感染后,出院组患者血液中T细胞的多样性较高,克隆扩增水平较低。
表5.每组中的唯一克隆和多克隆百分比
实施例3top 10TCRs克隆型中的V/CDR3/D/J片段使用情况和全局V-J配对分布分析
代表性样本在3组中使用率最高的top 10TCRs克隆型中,TRAV1-2、TRAJ23、TRAJ33、 TRBV20-1、TRBJ2-1和TRBJ2-7的高表达率相似(表6-11)。与HD组和随访组相比,出院组top 10s 克隆型的频率显著降低。接下来,使用全局V-J配对分布分析来显示3组的TCR特征(图6)。在HD 和随访组中观察到几种主要连接,包括HD中的TRAV1-2、TRAJ16、TRBV20-1和TRBJ2-1,随访组中的TRAV1-2 TRAJ33、TRBV20-1和TRBJ2-7(图6a、c)。出院组在不同的V-J组合之间具有相对一致的连接,这与该组TCR多样性的增加水平一致(图6b)。其中来源于6个新冠痊愈病人的Top 20TCR 序列信息见表12-23所示。
表6 HD组(HD#3)代表性top 10TCR克隆型信息
/>
表7 HD组(HD#3)代表性top 10TCR克隆型信息
表8出院组(Discharged#2)代表性top 10的TCR克隆型信息
表9出院组(Discharged#2)代表性top 10的TCR克隆型信息
表10随访组(Discharged#2)代表性top 10的TCR克隆型信息
表11随访组(Discharged#2)代表性top 10的TCR克隆型信息
表12来源于新冠痊愈病人(PGW1)的Top 20TCR序列
/>
表13来源于新冠痊愈病人(PGW1)的Top 20TCR序列
/>
/>
表14来源于新冠痊愈病人(PGW4)的Top 20TCR序列
/>
/>
表15来源于新冠痊愈病人(PGW4)的Top 20TCR序列
/>
表16来源于新冠痊愈病人(PGW8)的Top 20TCR序列
/>
/>
表17来源于新冠痊愈病人(PGW8)的Top 20TCR序列
/>
/>
表18来源于新冠痊愈病人(P2)的Top 20TCR序列
/>
表19来源于新冠痊愈病人(P2)的Top 20TCR序列
/>
/>
表20来源于新冠痊愈病人(P5)的Top 20TCR序列
/>
/>
表21来源于新冠痊愈病人(P5)的Top 20TCR序列
/>
表22来源于新冠痊愈病人(P9)的Top 20TCR序列
/>
/>
表23来源于新冠痊愈病人(P9)的Top 20TCR序列
/>
/>
实施例4康复患者中免疫功能与差异表达基因的关系
接下来,重点研究扩展的top T细胞克隆,这些克隆代表了对抗SARS-CoV-2感染最活跃的T细胞。将来自HD组(17374个细胞)、出院组(24040个细胞)和随访组(24938个细胞)的scRNA-seq 和成对TCR数据连接起来(表24)。在每组中,top 20TCR克隆型的细胞数占总细胞数的~10%至20% (表24)。首先研究了TCR配对scRNA序列数据中CD3E、CD4和CD8A的表达,以确定 CD3E
表24.各组均进行scRNA-seq和配对TCR细胞计数
top 20TCRs克隆型CD3E
细胞类型注释是根据已知基因标记手动注释该簇对应的细胞类型(图9,10)
通过比较健康捐赠者和康复患者CD3E
差异表达分析与可视化是在上述数据基础上,对CD4
TCR序列分析是利用包含CDR3序列的TCR测序数据,用于TCRα链和β链分析。在TCRα和β链的CDR3长度和V/J分布分析中使用'as.character','nchar','unlist','table'和'round'函数。通过对top 10TCR克隆型中的V/CDR3/J用法以及每个样本的V-J配对分析,对同时表达TCRα链和β链的具有CDR3序列和C基因的细胞进行克隆扩增。简单地说,分离TCRα和β链,并去除每条链的重复条形码。克隆扩展分析中使用了'as.data.frame','unlist','table','round','order'和'cumsum'函数。此外,使用ggplot2软件包中的‘ggplot’,‘geom_rect’,‘coord_polar’,‘labs’,‘xlim’,‘theme_light’,‘theme’和‘scale_fill_manual’函数将分析结果可视化。对于top 10个TCR克隆型中的V/CDR3/J用法,在stringr(v1.4.0,R包)中应用了‘str_split_fixed’函数、‘paste’,‘as.data.frame’,‘table’和‘order’函数;通过ggplot2和ggalluvial(v0.12.0,R包)中的‘ggplot’,‘scale_x_discrete’,‘theme_minimal’,‘theme’, ‘ggtitle’,‘scale_fill_discrete’,‘geom-alluvium’和‘geom_stratum’函数,可以直观地在top 10TCR克隆型中看出V/CDR3/J的使用情况。此外,stringr中的‘str_split_fixed’函数、‘as.data.frame’,‘paste’和‘table’函数用于V-J配对分析;在Circize(v0.4.10,R软件包)中为V/J配对可视化应用了‘chordDiagram’和‘circos.trackPlotRegion’函数。
总之与随访组和HD组相比出院组高表达IFITM家族和LY6E基因,这说明出院组通过靶细胞对病毒融合抵抗可以更好地抵抗SARS-CoV-2感染。且出院组和随访组均保持强大的病毒清除能力,具有强大的杀灭功能。这些数据表明,随着SARS-CoV-2感染的恢复,抗病毒融合效力将迅速恢复到基础水平,而CD8
实施例5在出院患者中IFN相关抗病毒特征分析
来自top 20克隆T细胞的CD8
通过每个簇中的基因表达确定top 20克隆型T细胞的亚群(图12,13,10)。出院组和随访组出现了CD8A
此外,差异表达基因的基因概念网络分析也支持出院组的路径分析(图15)。基于GSEA的HD 组与出院组相比,总CD8
生物过程富集与基因概念网络分析是用“FindMarkers”功能分析上调的差异表达基因,用clusterProfiler(v3.14.3,R软件包)的“bitr”功能和“enrichGO”函数,org.Hs.eg.db(v3.11.4,R 软件包)的“OrgDb”参数。对于设置“enrichGO”函数,对于“pvalueCutoff”和“qvalueCutoff”, BP为0.05。在enrichplot(v1.6.1,R软件包)中,使用‘dotplot’函数显示BP路径,其中‘showCategory’参数为20。此外,enrichplot(v1.6.1,R软件包)中的“cnetplot”显示了top 5BP路径的基因概念网络。
总之,尽管top 20TCR克隆型占总T细胞的比例不到20%,但是CD8
实施例6随访患者的代谢改变
通过GESA鉴定,HD组相比于随访组中CD8
据采样时间点将随访组分为早期随访组和晚期随访组。通过与HD组在top 20个CD8
对top 20TCRs配对scRNA-seq数据中对出院组和晚期随访组的单核分析,结果发现晚期随访组在CD8A
总体而言,患者在康复期具有较高的抗SARS-CoV-2感染能力,而在生命过程中从抗病毒免疫反应转变为出院后的代谢适应。
其中单细胞发育轨迹重建具体步骤为:Monocle2(v2.14.0,R软件包)用于构建单细胞轨迹,以发现发育过渡。对出院患者和晚期随访组配对样本的top 20TCR克隆型scRNA-seq数据进行聚类分析,并进行伪时间分析。简而言之,表达细胞数大于或等于50的基因用于定义细胞在“DifferentingGenetest”和“setOrderingFolter”函数中的进展和聚集在‘reduceDimension’函数中使用‘DDRTree’来减少维度,并使用‘plot_cell_trajectory’函数进行可视化。
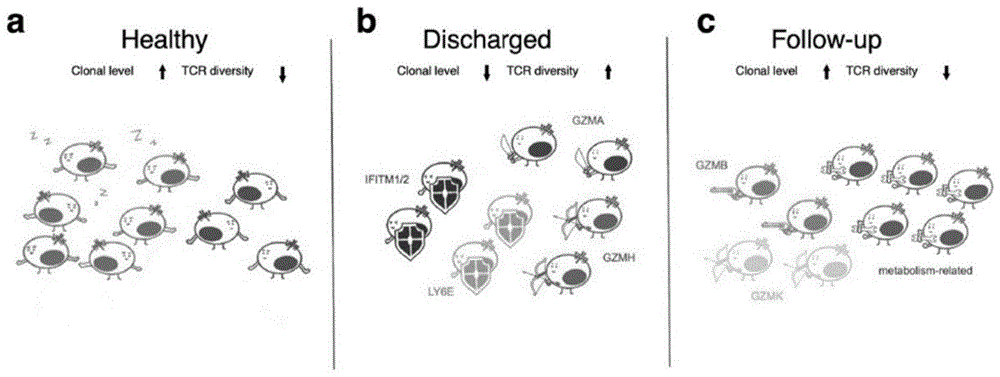
本申请对COVID-19康复患者中TCR克隆动态变化和功能基因分析进行研究,得出结论:在健康组中,只需要很少的克隆免疫监视细胞就可以维持免疫反应的稳态(图21a);在出院组中,大多数低扩增的少量克隆可抵抗病毒感染(IFN相关)和细胞毒性(颗粒酶)(图21b);在随访组中,拥有GZMB细胞毒性克隆和主要代谢克隆(图21c)。
我们相信,研究TCR序列的动态可以提供一个有价值的工具来监测T细胞介导的对潜在SARS- CoV-2疫苗的免疫应答。这一研究领域需要扩大患者数据,可以在不同的时间点取样,以便在不久的将来提供更详细的信息,以设计增加T细胞数量和增强其抗病毒功能的最佳方法。此外,我们提出的研究可以为开发一种新型的T细胞疫苗以对抗SARS-CoV-2感染奠定基础。
以上所述仅是本发明的优选实施方式,应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。
