一种无抗性标记自主发光肺炎克雷伯菌的构建方法及应用
文献发布时间:2023-06-19 09:52:39
技术领域
本发明涉及基因工程技术领域,尤指一种无抗性标记自主发光肺炎克雷伯菌的构建方法及应用。
背景技术
Kp为革兰阴性杆菌,是一种重要的条件致病菌,常引发社区获得性感染和医院内感染;当患者因长期住院、手术、导管留置等引起免疫力下降,或者长期大量使用抗生素导致菌群失调时可引起感染,也可通过患者之间、医护人员与患者接触、医疗器械等造成感染;14%-20%的医院内泌尿系统感染、呼吸道感染和败血症是由Kp造成的。近年来,Kp已成为仅次于大肠杆菌的重要条件致病菌,通常情况下,Kp感染的治疗首选氨基糖苷类抗生素,如庆大霉素、KAN、妥布霉素、丁胺卡那霉素,可肌注、静滴或管腔内用药;重症宜加用头孢菌素如头孢西丁等;哌拉西林,美洛西林与氨基糖苷类联用疗效亦佳。但是随着β-内酰胺类及氨基糖胺类等广谱抗生素的广泛使用,细菌易产生超广谱β-内酰胺酶(Extended Spectrumβ-Lactamases,简称ESBLs)和氨基糖苷类修饰酶(Aminoglycoside Modifying Enzymes,简称AMEs),对常用药物包括第三代头孢菌素和氨基糖苷类呈现出严重的多重耐药性。Kp引起的医院感染率近期逐年增高,且多耐药性菌株的不断增加常导致临床抗菌药物治疗的失败和病程迁延,因此开发治疗耐药的Kp的抗生素十分有必要。目前筛选、评价抗Kp新药及新药物组合,费时、费力、费用高等,因此我们需要构建一个可以实现快速、经济研发抗Kp新药、新药组合的系统。
转座子(Transposon,Tn)又称转座因子或跳跃因子,它可以从遗传物质的一部分跳跃到另一部分,从而引起遗传变异,Tn7转座子是一种定点插入型转座子,固定地插在细菌的glmS基因之后;glmS基因与细胞壁的合成有关,是一个必需基因。Tn7转座子因其具有插入染色体后对细菌基因的表达没有任何影响、不影响细菌的生长状态、插入位点明确、携带大基因片段等特点,已经作为一种基因操作工具应用于多种细菌。
传统遗传操作要求携带抗性标记基因,用于筛选阳性转化结果,在利用转座子系统将目的基因序列整合到细菌基因组上亦需要抗性筛选标记,但是重组菌株含有抗性基因,可能会给后续的遗传操作及应用带来不便,如在药物筛选/评价中可能存在交叉抗性。目前常见的用于切除抗性标记的序列重组系统包括来源于细菌噬菌体P1的Cre/loxP系统,转座子γδ的TnpR/res系统,酿酒酵母的Flp/FRT系统等;但是这些系统都需要将含有解离酶基因的质粒转入宿主菌,在诱导表达并识别抗性基因两端的特定位点消除抗性基因后,还需去除该质粒,在实际应用上非常的不便。Xer-cise特异性内源重组系统主要在细菌染色体复制过程中起作用,Xer解离酶的作用是在细胞分裂的染色体分离阶段识别复制末端的dif序列,催化染色体二聚体解离,从而形成染色体单体。大肠杆菌、霍乱弧菌等细菌都存在内源Xer-cise系统。然而Xer-cise系统用于切除外源抗性基因最近几年才被报道,目前已被用于分枝杆菌属、鲍曼不动杆菌和铜绿假单胞菌等的遗传操作,尚未见用于Kp。与上述所述系统相比,Xer-cise序列特异性重组系统的优势在于,利用菌株本身编码的Xer重组酶,不需要人工表达外源蛋白来实现抗性基因的去除,节省了繁琐工作、节约了大量时间。
发明内容
为解决上述问题,本发明旨在公开基因工程技术领域,尤指一种无抗性标记自主发光肺炎克雷伯菌的构建方法及应用。
为实现上述目的,本发明采用的技术方案是:本发明提供一种无抗性标记自主发光肺炎克雷伯菌的构建方法,其特征在于,所述肺炎克雷伯菌由转移质粒pTXR和辅助质粒pUCTns构建;所述的构建方法包括以下步骤:
S1、准备转移质粒pTXR和辅助质粒pUCTns;
S2、将表达转座酶基因的辅助质粒pUCTns转入肺炎克雷伯菌感受态细胞中,涂于卡那霉素抗性的LB培养基平板,得到含辅助质粒pUCTns的肺炎克雷伯菌;
S3、将含辅助质粒pUCTns的肺炎克雷伯菌制备成感受态,然后将转移质粒pTXR转入含辅助质粒pUCTns的肺炎克雷伯菌感受态细胞中,涂于安普霉素抗性的LB培养基平板,得到自主发光的肺炎克雷伯菌。
优选地,所述的构建方法还包括步骤S4、将步骤S3获得的自主发光的肺炎克雷伯菌在无抗性LB液体培养基中传代培养,筛选出抗性基因apr和pUCTns质粒均丢失的发光菌,获得无抗性标记的自主发光肺炎克雷伯菌。
优选地,所述的转移质粒pTXR按照顺时针依次含有氨苄青霉素抗性基因bla、复制子ori R6kγ、接合转移起始位点ori T、转座子序列Tn7R至Tn7L携带的片段。
优选地,所述转座子序列Tn7R至Tn7L携带的片段中,包括使肺炎克雷伯菌自主发光的一套luxCDABE基因以及用于筛选重组耐药菌的抗性基因apr,apr的两端带有difR和difL序列,其中,difR和difL序列选自Xer-cise特异性重组系统,该系统利用宿主菌内源表达的解离酶即可实现抗性基因的切除;肺炎克雷伯菌通过自身的Xer解离酶系统自动将difR和difL序列之间的apr片段解离下来。
优选地,所述转座子序列Tn7R至Tn7L携带的片段按顺时针依次包含反向重复序列Tn7R、抗性基因apr、hsp60启动子、luxCDABE基因和反向重复序列Tn7L;其中,Tn7R的基因序列优选如SEQ ID NO:9所示,Tn7L的基因序列优选如SEQ ID NO:10所示。
优选地,所述转移质粒pTXR的构建方法,包括以下步骤:
(A1)将起始质粒pUC18T-mini-Tn7T-lux-Tp用限制性内切酶Xba I和BamH I消化后,进行琼脂糖凝胶电泳将不同大小DNA片段区分开,回收大片段产物;
(A2)以质粒pMABH1为模板,PCR扩增出kp-dif-apr基因片段后用限制性内切酶XbaI和BamH I消化并回收,然后将(A1)中的回收产物和回收的kp-dif-apr基因片段进行连接、转化,得到质粒pAPR;PCR扩增使用的引物对如SEQ ID NO:1和SEQ ID NO:2所示;
(A3)将质粒pAPR用限制性内切酶KpnI和NotI消化后,进行琼脂糖凝胶电泳将不同大小DNA片段区分开,回收小片段产物;
(A4)以质粒pP3-lux为模板,PCR扩增hsp60-luxCDABE基因片段,然后将(A3)中的回收产物和hsp60-luxCDABE基因片段进行同源重组、转化,得到质粒pHSP;PCR扩增使用的引物对如SEQ ID NO:3和SEQ ID NO:4所示;
(A5)将质粒pHSP用限制性内切酶ApaL I消化后,进行琼脂糖凝胶电泳将不同大小DNA片段区分开,回收大片段产物;
(A6)以质粒pTNS3为模板,PCR扩增bla-ori R6kγ基因片段,然后将(A5)中的回收产物和bla-ori R6kγ基因片段进行同源重组、转化,得到质粒pTXR;PCR扩增使用的引物对如SEQ ID NO:5和SEQ ID NO:6所示。
优选地,所述辅助质粒pUCTns的构建方法,包括以下步骤:
(B1)将起始质粒pTNS3用限制性内切酶EcoR I和Nde I消化后,进行琼脂糖凝胶电泳将不同大小DNA片段区分开,回收大片段产物;
(B2)以质粒pUCOK为模板,PCR扩增kan-ori基因片段,然后将(B1)中的回收产物和kan-ori基因片段进行同源重组、转化,得到质粒pUCTns;PCR扩增使用的引物对如SEQ IDNO:7和SEQ ID NO:8。
本发明提供一种如无抗性标记自主发光肺炎克雷伯菌的构建方法所构建的无抗性标记的自主发光肺炎克雷伯菌。
本发明提供一种如无抗性标记自主发光肺炎克雷伯菌的构建方法在制备无抗性标记的自主发光肺炎克雷伯菌试剂盒的应用,其特征在于,无抗性标记的自主发光肺炎克雷伯菌试剂盒包括转移质粒,以及表达转座酶基因的辅助质粒。
本发明提供一种如无抗性标记自主发光肺炎克雷伯菌的构建方法在制备或筛选抗肺炎克雷伯菌感染药物中的应用。
本发明的有益效果体现在:
1)本发明将Tn7转座系统与肺炎克雷伯菌(Kp)的dif序列结合,作为特异性重组位点应用到Kp中,并验证了Kp的dif序列可以被内源性Xer解离酶高效解离,为一步法构建自主发光菌,仅需将dif序列连接在抗性基因apr两端,利用宿主菌内源表达的Xer解离酶即可实现抗性切除;相较于其他特异性重组系统如TnpR/res、Flp/FRT系统,该方法无需人工表达外源解离酶,操作简便快捷。
2)本发明中由转移质粒pTXR和辅助质粒pUCTns参与构建的无抗性标记的自主发光肺炎克雷伯菌(SfAlKp),不需要添加任何底物即可发出强光,在黑暗环境中通过肉眼便能看到发光菌落。
3)相对于现有的带抗性标记的自主发光Kp,本发明的SfAlKp没有抗性基因,其生理状态、药物敏感性更接近于野生型,可避免由于抗性基因导致的交叉耐药对药物筛选、评价可能带来的潜在副作用。
4)目前暂未发现luxCDABE基因整合到Kp基因组从而得到可以稳定发光的SfAlKp的技术,游离的luxCDABE基因很容易在细菌传代过程丢失,故本发明制备得到的SfAlKp稳定性强;本发明制备得到的SfAlKp发光强度高,在活体动物中可以被快速检测到;CFU(Colony Forming Unit,菌落形成单位,简称CFU)和RLU(Relative Light Unit,相对光单位,简称RLU)之间存在对应关系,用RLU来代替CFU作为分析细菌生长情况。
5)鉴于SfAlKp发光强度高、稳定性强、与其亲本对不同类型药物敏感性相同,RLU可以替代CFU作为检测对象,因而SfAlKp可用于快速体内外筛选、评价药效;通过对光强度的检测就可对该微生物在环境中的生长、活性和分布等进行实时在线监测,对于其它生物报告基因是显然进步的。
6)本发明SfAlKp的构建方法也适用于其它带有Xer-cise重组系统的细菌,特别是有关去除Kp基因组中特定基因序列,极大地方便了遗传操作。
7)本发明可应用于构建小鼠感染模型,能够简便快速地实时检测化合物体内活性,为体内筛选有效化合物节省了大量的时间、空间、动物和人力等。
附图说明
图1为本发明pTXR质粒构建流程示意图。
图2为本发明pUC18T-mini-Tn7T-lux-Tp质粒图谱。
图3为本发明pMABH1质粒图谱。
图4为本发明pAPR质粒图谱。
图5为本发明pP3-lux质粒图谱。
图6为本发明pHSP质粒图谱。
图7为本发明pTNS3质粒图谱。
图8为本发明pTXR质粒图谱。
图9为本发明pUCTns质粒构建流程示意图。
图10为本发明pUCOK质粒图谱。
图11为本发明pUCTns质粒图谱。
图12为本发明SfAlKp在正常光照和暗室条件下的对比图。
图13为本发明将在APR抗性平板上挑到的发光单菌落AlKp进行PCR扩增验证是否转座成功的电泳结果图:其中泳道M:2Kb plus marker;泳道1:Kp;泳道2:AlKp。
图14为本发明将第六次转接后获得的发光菌分别涂于含有APR抗性平板(左)和无抗生素平板(右)的结果图。
图15为本发明PCR筛选抗性基因apr丢失的AlKp的电泳结果图;其中泳道M:2Kbplus marker;泳道1-3:抗性基因apr丢失的AlKp;泳道4-6:抗性基因apr未丢失的AlKp。
图16为本发明将转接后获得的抗性基因apr丢失的AlKp分别涂于含有KAN抗性平板(左)和无抗生素平板(右)的结果图。
图17为本发明PCR筛选SfAlKp的电泳结果图;其中泳道M:2Kb plus marker;泳道1:抗性基因apr和质粒pUCTns均丢失的AlKp;泳道2:抗性基因apr丢失但质粒pUCTns未丢失的AlKp。
图18为本发明中SfAlKp和Kp的生长曲线,其中,时间为横坐标,OD
图19为本发明中Kp和SfAlKp的CFU与时间关系图和SfAlKp的RLU与时间关系图,其中,时间为横坐标,Log
图20为本发明中SfAlKp中CFU与RLU的线性关系图(Y=0.8005X-0.4467,R
图21为本发明中不同抗生素对SfAlKp的时间-抗菌曲线,其中时间为横坐标,Log
图22为本发明中利用小鼠感染模型实时成像技术检测不同化合物体内活性;浅色箭头部分代表感染时注射位点。
具体实施方式
下面结合附图详细说明本发明的具体实施方式:
本发明提供一种无抗性标记自主发光肺炎克雷伯菌的构建方法,所述肺炎克雷伯菌由转移质粒pTXR和辅助质粒pUCTns构建;所述的构建方法包括以下步骤:
S1、准备转移质粒pTXR和辅助质粒pUCTns;
S2、将表达转座酶基因的辅助质粒pUCTns转入肺炎克雷伯菌感(Kp)受态细胞中,涂于卡那霉素(Kanamycin,简称KAN)抗性的LB培养基(Luria-Bertani培养基,简称LB)平板,得到含辅助质粒pUCTns的肺炎克雷伯菌;
S3、将含辅助质粒pUCTns的肺炎克雷伯菌制备成感受态,然后将转移质粒pTXR转入含辅助质粒pUCTns的肺炎克雷伯菌感受态细胞中,涂于安普霉素(APR)抗性的LB培养基平板,得到自主发光的肺炎克雷伯菌(Autoluminescent Klebsiella pneumoniae,AlKp);
S4、将步骤S3获得的自主发光的肺炎克雷伯菌在无抗性LB液体培养基中传代培养,筛选出抗性基因apr和pUCTns质粒均丢失的发光菌,获得无抗性标记的自主发光肺炎克雷伯菌(SfAlKp,所述SfAlKp的基因组中含有与自主发光反应直接相关的基因luxCDABE基因,且SfAlKp的基因组中没有抗性筛选基因)。
本发明利用Tn7转座子系统将与自主发光直接相关的基因luxCDABE整合到Kp的基因组中,实现luxCDABE基因在Kp中高效稳定的表达;所构建的SfAlKp不存在基因工程改造过程中的抗性筛选标记,无需添加底物即可实现自主发光;所述的转移质粒pTXR按照顺时针依次含有氨苄青霉素(AMP)抗性基因bla、复制子ori R6kγ、接合转移起始位点ori T、转座子序列Tn7R至Tn7L携带的片段;所述转座子序列Tn7R至Tn7L携带的片段中,包括使肺炎克雷伯菌自主发光的一套luxCDABE基因以及用于筛选重组耐药菌的抗性基因apr,apr的两端带有difR和difL序列,肺炎克雷伯菌通过自身的Xer解离酶系统自动将difR和difL序列之间的apr片段解离下来;所述转座子序列Tn7R至Tn7L携带的片段按顺时针依次包含反向重复序列Tn7R、抗性基因apr、hsp60启动子、luxCDABE基因和反向重复序列Tn7L;转移质粒含有以反向重复系列Tn7L和Tn7R为侧翼的转座子序列,转座子序列中间含有抗性基因apr,与自主发光有关的基因luxCDABE和位于apr两端的同向重复序列difR和difL,difR和difL序列为Xer-cise特异性重组系统的识别位点,无需导入表达外源解离酶的基因序列即可实现抗性基因的切除;辅助质粒包括有编码转座酶的基因TnsABCD,TnsABCD基因序列如SEQID NO:11所示;在一些实施例中,本发明的构建方法先转化辅助质粒pUCTns,然后再转化转移质粒pTXR;在一些实施例中,反向重复序列和编码转座酶的基因选自Tn7转座子系统。
所述转移质粒pTXR的构建方法,包括以下步骤(构建步骤如图1):
(A1)将起始质粒pUC18T-mini-Tn7T-lux-Tp(质粒图谱如图2,由弗吉尼亚医护大学Joanna B.Goldberg实验室提供)用限制性内切酶Xba I和BamH I消化后,进行琼脂糖凝胶电泳将不同大小DNA片段区分开,回收大片段产物;
(A2)以质粒pMABH1(质粒图谱图3,为本实验室保有质粒)为模板,PCR扩增出kp-dif-apr基因片段后用限制性内切酶Xba I和BamH I消化并回收,然后将(A1)中的回收产物和回收的kp-dif-apr基因片段进行连接、转化,得到质粒pAPR(质粒图谱如图4);PCR扩增使用的引物对如SEQ ID NO:1和SEQ ID NO:2所示;
(A3)将质粒pAPR用限制性内切酶KpnI和NotI消化后,进行琼脂糖凝胶电泳将不同大小DNA片段区分开,回收小片段产物;
(A4)以质粒pP3-lux(质粒图谱如图5,本实验室保有质粒)为模板,PCR扩增hsp60-luxCDABE基因片段,然后将(A3)中的回收产物和hsp60-luxCDABE基因片段进行同源重组、转化,得到质粒pHSP(质粒图谱如图6);PCR扩增使用的引物对如SEQ ID NO:3和SEQ ID NO:4所示;
(A5)将质粒pHSP用限制性内切酶ApaL I消化后,进行琼脂糖凝胶电泳将不同大小DNA片段区分开,回收大片段产物;
(A6)以质粒pTNS3(质粒图谱如图7,弗吉尼亚医护大学Joanna B.Goldberg实验室提供)为模板,PCR扩增bla-ori R6kγ基因片段,然后将(A5)中的回收产物和bla-ori R6kγ基因片段进行同源重组、转化,得到质粒pTXR(质粒图谱如图8);PCR扩增使用的引物对如SEQ ID NO:5和SEQ ID NO:6所示。
所述辅助质粒pUCTns的构建方法,包括以下步骤:
(B1)将起始质粒pTNS3(质粒图谱如图7,弗吉尼亚医护大学Joanna B.Goldberg实验室提供)用限制性内切酶EcoR I和Nde I消化后,进行琼脂糖凝胶电泳将不同大小DNA片段区分开,回收大片段产物;
(B2)以质粒pUCOK(质粒图谱如图10,弗吉尼亚医护大学Joanna B.Goldberg实验室提供,)为模板,PCR扩增kan-ori基因片段,然后将(B1)中的回收产物和kan-ori基因片段进行同源重组、转化,得到质粒pUCTns(质粒图谱如图11);PCR扩增使用的引物对如SEQ IDNO:7和SEQ ID NO:8。
同时本发明还提供一种如无抗性标记自主发光肺炎克雷伯菌的构建方法所构建的无抗性标记的自主发光肺炎克雷伯菌;提供一种如无抗性标记自主发光肺炎克雷伯菌的构建方法在制备无抗性标记的自主发光肺炎克雷伯菌试剂盒的应用,所述试剂盒包括转移质粒,以及表达转座酶基因的辅助质粒;提供一种如无抗性标记自主发光肺炎克雷伯菌的构建方法在制备或筛选抗肺炎克雷伯菌感染药物中的应用。
下述实施例中采用分子生物学实验技术包括PCR扩增、质粒提取、质粒转化、DNA片段连接或者同源重组、酶切、凝胶电泳等,以进一步验证本发明的技术方案:
以下实施例中重组酶、大肠杆菌感受态Trans T1、PCR反应所用的EasyTaq Mix以及相关试剂均购自北京全式金生物技术有限公司;抗生素:AMK、TGC、APR、PMB和KAN购自美仑生物技术有限公司;实验室自制JM109λpir感受态细胞;DNA连接反应所需的Solution I连接试剂盒均购自宝生物公司;质粒DNA提取试剂盒、DNA回收试剂盒购自美基生物公司;Bio Rad电转化仪(BioradGenePulser Xcel1)以及电转杯购自伯乐生命医学产品有限公司;发光检测仪(GLOMAX2020)购自普洛麦格公司。
实施例1、转移质粒pTXR和辅助质粒pUCTns的构建:
(1)按照流程图1构建转移质粒pTXR,如图1所示,质粒pTXR按顺时针方向依次含有AMP抗性基因(bla),复制起始位点(ori R6kγ),APR抗性基因(apr),luxCDABE基因,位于apr两端的同向重复序列difR和difL;各元件的功能如下:
luxCDABE:即与自主发光相关的基因,是发光所需酶基因,该基因的表达使得宿主菌可以自主发光;
APR抗性基因(apr):apr是一种抗性选择标记,用于筛选获得目的菌株;apr在Kp和大肠杆菌中表达后可使宿主菌获得对APR的抗性,即可在含有APR的培养基中生长;APR是一种抗性筛选药物,用于Kp的APR浓度为30μg/mL,用于大肠杆菌的APR浓度为50μg/mL;
同向重复序列dif:该序列可以被宿主菌自身的Xer解离酶识别,并高效切除dif序列间的抗性基因,因而,可以在后续操作中去掉apr;
AMP抗性基因(bla):是一种筛选标记,用于筛选获得目的菌株;在大肠杆菌中表达后,可使宿主菌获得对AMP的抗性,即可在含有AMP的培养基中生长,用于大肠杆菌的AMP浓度为50μg/mL;
ori R6kγ:含有该复制子的质粒只能在可以表达λpir蛋白的菌株中复制,如大肠杆菌JM109λpir。
(2)转移质粒pTXR的具体构建方法如下:
S1、将起始质粒pUC18T-mini-Tn7T-lux-Tp(图2,弗吉尼亚医护大学JoannaB.Goldberg实验室提供,)用限制性内切酶Xba I和BamH I消化后,进行琼脂糖凝胶电泳将不同大小DNA片段区分开,回收长度较大的功能片段产物A;
S2、以质粒pMABH1(图3)为模板,用引物P
5'-TGCTCTAGAATTTAACATAATATACATTATGCGCACTAAGCTTAGCTCAGCCAATCGAC-3'(SEQID NO:2)扩增出kp-dif-apr基因序列,然后利用限制性内切酶Xba I和BamH I消化PCR产物得到功能片段B;
S3、将功能片段A和功能片段B混合后,加入连接酶Solution I进行连接,得到质粒pAPR(图4),转化大肠杆菌感受态Trans T1,利用含APR的LB固体平板筛选出阳性克隆,挑取单克隆到LB液体培养基中培养,提取质粒pAPR;利用引物P
5'-CGGGATCCAGTGCGCATAATGTATATTATGTTAAATCCATGGCACCACCGACTATTTG-3'(SEQID NO:1)和引物P
5'-TGCTCTAGAATTTAACATAATATACATTATGCGCACTAAGCTTAGCTCAGCCAATCGAC-3'(SEQID NO:2)PCR扩增鉴定质粒pAPR,正确的PCR产物琼脂糖凝胶电泳后的结果是在约900bp处有一明显条带;再将PCR验证正确的质粒送去测序,以选取出kp-dif-apr基因片段不存在突变的质粒,以进行后续实验;
S4、如图1所示,将质粒pAPR用限制性内切酶KpnI和NotI消化后,进行琼脂糖凝胶电泳将不同大小DNA片段区分开,回收约4.8kb的功能片段C;
S4、以质粒pP3-lux(图5)为模板,用引物P
5'-ATATCCATCACACTGGCGGCCGCGGTGACCACAACGACGCG-3'(SEQ ID NO:3)和引物P
5'-GCAAGGCCTTCGCGAGGTACCTCAACTATCAAACGCTTCGGTTAA-3'(SEQ ID NO:4)扩增出hsp60-lux基因序列,获得约6.2kb的功能片段D;
S5、将功能片段C和功能片段D混合后,加入重组酶进行重组,获得重组质粒pHSP(图6),转化大肠杆菌感受态Trans T1,利用含APR的LB固体平板筛选出阳性克隆,挑取单克隆到LB液体培养基中培养,提取质粒pHSP;用引物P
S6、将质粒pHSP用限制性内切酶ApaL I消化后,进行琼脂糖凝胶电泳将不同大小DNA片段区分开,回收约9.3kb的功能片段E;
S7、以质粒pTNS3(图7,弗吉尼亚医护大学Joanna B.Goldberg实验室提供)为模板,用引物P
5'-TCCAAGCTGGGCTGTGTGCACTTTCGGGGAAATGTGCGC-3'(SEQ ID NO:5)和引物P
S8、将功能片段E和功能片段F混合,加入重组酶进行同源重组,获得重组质粒pTXR(图8),转化大肠杆菌JM109λpir感受态,利用含AMP的LB固体平板筛选出阳性克隆,挑取单克隆到LB液体培养基中培养,提取质粒pTXR。用引物P
(3)辅助质粒pUCTns的具体构建方法如下:
S1、将起始质粒pTNS3(图7,弗吉尼亚医护大学Joanna B.Goldberg实验室提供)用限制性内切酶EcoR I和Nde I消化后,进行琼脂糖凝胶电泳将不同大小DNA片段区分开,回收约7.3kb的功能片段G;
S2、以质粒pUCOK(图10,弗吉尼亚医护大学Joanna B.Goldberg实验室提供)为模板,用引物P
5'-GTGCGGTATTTCACACCGCATATGGAGCAAAAGGCCAGCAAAAG-3'(SEQ ID NO:7)和引物P
S3、功能片段G和功能片段H混合后,加入重组酶进行同源重组,得到质粒pUCTns(图11),转化大肠杆菌感受态Trans T1,利用含KAN的LB固体平板筛选出阳性克隆,挑取单克隆到LB液体培养基中培养,提取质粒pUCTns;用引物P
5'-GTCAACTGGGTTCGTGAATTCGATAATAATGGTTTCTTAGACGTCAGGT-3'(SEQ ID NO:8)PCR扩增鉴定质粒pUCTns,正确的PCR产物琼脂糖凝胶电泳后的结果是约1.8kb处有一明显条带;再将PCR验证正确的质粒送去测序,以选取出kan-ori基因片段不存在突变的质粒,以进行后续实验。
实施例2、一种SfAlKp的构建方法:
1.电转化
(1)制备Kp感受态细胞:从-80℃冰箱取出冻存的Kp,放在碎冰上融化,采用划线法接到LB固体培养基上,过夜培养。挑取单菌落接到10mL的LB液体培养基中,37℃摇床培养。OD
(2)将100μL的Kp感受态细胞和10μL(浓度约为300ng/μL)的质粒pUCTns混合后转移至2mm的电转杯中。冰上放置30min后进行电转,加入电转仪前擦尽电转杯上的水分;
(3)用Bio Rad电转仪以25μF,200Ω,2.5kV脉冲波进行电转化;
(4)迅速加入1mL LB培养基,混匀后于37℃摇床中振荡(200rpm)1h,充分复活细菌使其抗性表达;
(5)将菌液在13000g下离心1min,弃去上清液后用200μL LB培养基重悬,将菌液涂布于含KAN 50μg/mL的LB培养基上,于37℃培养直到产生明显菌落。使用引物P
(6)将含有pUCTns质粒的Kp制备成感受态,制备方法同(1);
(7)将100μL的含有质粒pUCTns的Kp感受态细胞和20μL(浓度约为170ng/μL)的转移质粒pTXR混合后转移至2mm的电转杯中。冰上放置30min后进行电转,加入电转仪前擦尽电转杯上的水分;
(8)用Bio Rad电转仪以25μF,200Ω,2.5kV脉冲波进行电转化;
(9)迅速加入1mL LB培养基,混匀后于37℃摇床中振荡(200rpm)1h,充分复活细菌使其抗性表达;
(10)将菌液在13000g下离心1min,弃去上清液后用200μL LB培养基重悬,将菌液涂布于APR 30μg/mL的LB培养基上,于37℃培养直到产生明显菌落。
2.检测是否发生转座
(1)挑取APR 30μg/mL LB平板上生长的单菌落到含有20μL无菌水的1.5mL离心管中,混匀,将离心管放入发光检测仪器,检测发光值大小。根据经验,一般发光值在100以下的可认定为不发光,发光值在10
(2)以上述得到的发光菌为模板,用引物P
3.初步筛选抗性基因apr丢失的AlKp
(1)取在APR 30μg/mL的LB平板上转化出的AlKp以1:100000接种10mL到LB液体培养基中,并置于37℃,200rpm恒温摇床培养;
(2)当培养到12h时即达到指数生长末期后,再将该菌液以1:100000接种10mL到LB液体培养基中,并置于37℃,200rpm恒温摇床培养;
(3)步骤2中每次进行转接时,将菌液适当稀释后涂布到无抗性的LB固体平板上,37℃培养箱中培养得到单菌落,共进行6次转接培养;
(4)从每次转接培养时涂布的平板上随机挑取60个单菌落同时转接到无抗的LB平板和APR 30μg/mL的LB固体平板上,37℃培养箱中培养24h,结果如图14所示,统计可以在无抗性的平板上生长而不能在APR抗性平板上生长的菌落数,计算切除效率,切除效率=不能在APR抗性平板上生长的菌落数/60,结果如表1所示。
表1:抗性基因apr切除的效率
根据表1可知,随着SfAlKp转接次数的增加,抗性基因apr的切除效率也越高,说明dif序列在Kp增殖过程中可以被Xer重组酶识别,从而高效的切除dif序列间的抗性基因。
4.PCR验证筛选的抗性基因apr丢失的AlKp
使用下列引物P
5.初步筛选抗性基因apr且质粒pUCTns均丢失的SfAlKp
(1)取在APR 30μg/mL的LB平板上不能生长的AlKp以1:100000接种10mL到LB液体培养基中,并置于37℃,200rpm恒温摇床培养;
(2)当培养到12h时即达到指数生长末期,再将该菌液以1:100000接种10mL到LB液体培养基中,并置于37℃,200rpm恒温摇床培养;
(3)步骤2中每次进行转接时,将菌液适当稀释后涂布到无抗性的LB固体平板上;
(4)从每次转接培养时涂布的平板上随机挑取60个单菌落同时转接到无抗的LB平板和KAN 50μg/mL的LB固体平板上,37℃培养箱中培养24h,结果如图16所示。
6.PCR验证筛选的抗性基因APR和质粒pUCTns均丢失的SfAlKp
(1)使用下列引物P
(2)PCR验证筛选的抗性基因apr和质粒pUCTns均丢失的AlKp即为SfAlKp。取700μLSfAlKp加300μL 80%甘油,混匀后,保存于-80℃冰箱。
实施例3、验证SfAlKp的发光稳定性:
1.传代培养实施例2制得的SfAlKp
a.取SfAlKp以1:100000接种10mL到LB液体培养基中,并置于37℃,200rpm恒温摇床培养;
b.当菌液OD
c.在重复步骤b数次后,将该菌液稀释后铺于无抗性的LB平板,并置于37℃恒温培养箱培养,24h后观察。
2.统计SfAlKp所占比例
当平板上菌落长出后,随机挑取100个单菌落并用发光检测仪检测RLUs。如果被检测单菌落的RLUs是Kp单菌落RLUs的5倍以上即大于200RLUs,则判定该单菌落为发光菌落。SfAlKp所占的比例为发光菌落数/100。
3.准备待测菌液
1)获取SfAlKp和Kp单菌落:将SfAlKp和Kp以划线接种的方式接种于无抗性LB平板培养,待37℃培养12h即可得到单菌落;
2)接种单菌落培养:从平板上挑取单菌落,接种到10mL的LB液体培养基,37℃,200rpm恒温摇床培养;
3)待液体培养SfAlKp和Kp的OD
4.测定不同时间段的OD
以接种时刻记做0h,开始测定时,每间隔1h便从培养基中取200μL菌液,检测OD
5.绘制生长曲线
通过分析步骤4所得数据,以时间为横坐标,OD
根据图18所示的结果可知,本发明的SfAlKp的生长曲线与野生型的Kp一致,故本发明的SfAlKp的生长不受插入基因的影响,其生长情况可以用检测RLU替代观察。
实施例4、探究SfAlKp的RLU和CFU的对应关系:
1.准备待测菌液
1)自-80℃冰箱取出冻存的SfAlKp和Kp,放在碎冰上融化,采用划线法接种到无抗的LB固体平板上,37℃培养箱中培养12h即可获得单菌落;
2)接种单菌落培养:从平板上挑取单菌落,接种到10mL的LB液体培养基,37℃,200rpm恒温摇床培养;
3)待液体培养SfAlKp和Kp的OD
2.测定不同时间段的RLUs和CFUs并作记录
以接种时刻记做0h,开始测定时,每间隔1h从培养基中取200μL SfAlKp和Kp菌液,检测SfAlKp的RLUs并作记录;将取出的菌液逐次10倍进行稀释,取400μL的稀释菌液涂布到无抗性的LB平板上,放置在37℃培养箱中培养至长出单菌落,计数并作记录;RLUs的测定一直持续到菌液RLUs开始降低为止。
3.绘制CFU和RLU的相关性曲线
通过分析步骤2所得数据,以时间为横坐标,Log
根据图19和图20的所示的结果可知,本发明制备得到的SfAlKp发光强度高、稳定性强,CFU和RLU之间存在对应关系,所以可以用RLU来代替CFU作为分析细菌生长情况的依据;只需通过对光线的检测就可对该微生物在环境中的生长、活性和分布等进行实时在线监测。
实施例5、SfAlKp的药物敏感性的测定:
1.获得SfAlKp和Kp单菌落
自-80℃冰箱取出冻存的SfAlKp和Kp,放在碎冰上融化,采用划线法接种到无抗的LB固体平板上,37℃培养箱中培养12h即可获得单菌落。
2.配置药液:
分别称取25.6mg的替加环素(Tigecycline,简称TGC)、阿米卡星(Amikacin,简称AMK)和多粘菌素B(Polymyxin B,简称PMB)溶于10mL无菌水中,配成2560μg/mL的抗生素母液,将母液与培养基按照1:9比例进行稀释,获得待测药液①。
3.制备待测菌液:
从步骤1中挑取1个野生型的Kp,接到5mL LB培养基中,37℃,200rpm培养4~5h,调整菌液OD
从步骤1中挑取1个SfAlKp单菌落,接到5mL LB培养基中,37℃,200rpm培养4~5h,将菌液用LB培养基稀释到RLU在3000-5000,为待测菌液。
4.最小抑菌浓度(minimal inhibitory concentration,简称MIC)的测定
(1)使用96孔板,在第一列孔中加入200μL待测药液①,逐次2倍稀释至11孔,每孔液体为100μL。往96孔板中加入100μL Kp待测菌悬液。每个浓度做三个重复。其中需要设置空白对照和不含抗生素加细菌的对照。37℃培养16~20h;
(2)根据Kp的MIC,制备检测浓度为4×MIC,2×MIC,1×MIC,1/2×MIC,1/4×MIC的待测溶液,将待测溶液加入1.5mL的Ep管中,往Ep管中加入100μL SfAlKp待测菌悬液,每个浓度做三个平行。其中需要设置空白对照和不含抗生素加细菌的对照。37℃培养,每隔两个小时检测一次RLU。
5.MIC的判定
(1)Kp的MIC的判定:通过肉眼判定MIC,若相邻两孔中,一孔出现菌落或浑浊,一孔澄清,澄清孔对应的抗生素浓度即为MIC,同时可以辅助酶标仪测定OD
(2)SfAlKp的MIC的判定:通过检测RLUs,以相比于对照组发光值90%不发光的最小浓度为MIC;
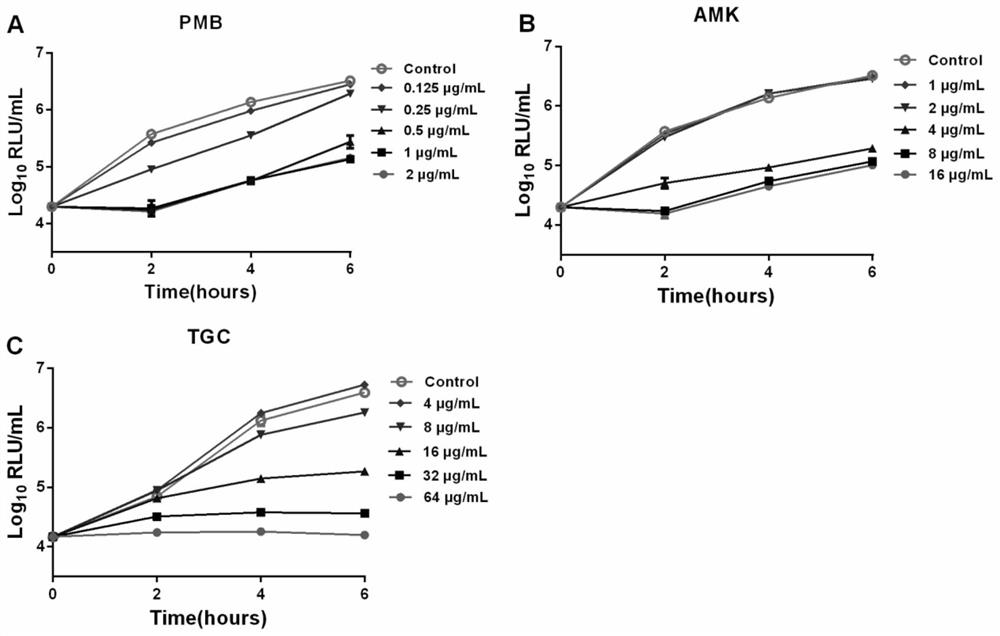
(3)SfAlKp在不同抗生素不同浓度条件下的生长曲线如图21所示,SfAlKp和Kp对抗生素的敏感性检测结果如表2所示。
表2:抗生素敏感性检测结果
Kp和SfAlKp对抗生素AMK和TGC的敏感性结果均一致,但是PMB组的结果却相差8倍,抗生素PMB影响细菌的呼吸从而对于本发明中的SfAlKp的发光值具有一定的影响;经验证,SfAlKp与其亲本Kp对不同类型药物具有相同的敏感性。
实施例6、构建小鼠感染SfAlKp模型并进行体内药物活性实时检测:
1.获得SfAlKp单菌落
自-80℃冰箱取出冻存的SfAlKp,放在碎冰上融化,采用划线法接种到无抗的LB固体平板上,37℃培养箱中培养12h即可获得单菌落。
2.制备感染菌液
(1)从步骤1中挑取1个SfAlKp单菌落,接到5mL LB培养基中,37℃,200rpm培养4~5h后,以1:10000比例转接到50mL LB液体培养基中,放在37℃,200rpm的摇床中培养至OD
(2)将50mL菌液均分到两个50mL离心管中,在25℃,6000rpm条件下离心5min后弃掉上清液,每管加入10mL磷酸缓冲液(Phosphate Buffer Solution,简称PBS)重悬SfAlKp,然后在相同的条件下离心,弃掉上清液后每管加入2.5mL PBS重悬SfAlKp后转移到一个离心管中,同时加入灭菌的玻璃珠打散菌块。
3.配置药液
称取AMK、PMB、TGC和麻醉剂乌拉坦溶于PBS中,使其终浓度分别为26.4mg/mL、1mg/mL、1.4mg/mL和100mg/mL。
4.利用浓缩10倍后的SfAlKp菌液感染小鼠
本发明中用到均为大约六周龄的BALB/c小鼠,小鼠重量大约为20g,将菌液混匀后,向每只小鼠腹腔注射500μL菌液,然后每只小鼠皮下注射200μL麻醉剂乌拉坦,乌拉坦的注射剂量为1g/kg,利用剪刀将小鼠腹部毛剪净尽量减少对发光亮度的影响,所以在之后的拍照间隙应该做好小鼠腹部的保暖工作。
5.不同抗生素治疗小鼠
在感染30min后,皮下注射200μL不同类型抗生素,AMK的治疗剂量为264mg/kg,PMB的治疗剂量为10mg/kg,TGC的治疗剂量为14mg/kg。
6.暗室拍照
在感染后立即进行第一次暗室拍照,感染30min后开始治疗,然后分别在治疗后1h和2h时进行第二次和第三次暗室拍照,数码相机设置参数为:感光度为1600,光圈为2,曝光时间为20min;所有的照片整理后如图22所示,PBS组在感染后随着时间的延长,其发光的强度和亮度越来越强,发光的范围也越来越大,AMK组和PMB组随着治疗时间的增加其发光位点仅分布于注射位点附近,且其发光亮度相对比较微弱,但TGC组随着治疗时间的增加其发光范围和强度相较于感染0h时明显增强,所以我们可以得出AMK和PMB具有体内抗SfAlKp活性而TGC没有体内抗SfAlKp活性,因此可以利用本实验构建的SfAlKp感染小鼠模型,实时有效地检测药物体内活性。
以上所述,仅是本发明的较佳实施例,并非对本发明的技术范围作任何限制,本行业的技术人员,在本技术方案的启迪下,可以做出一些变形与修改,凡是依据本发明的技术实质对以上的实施例所作的任何修改、等同变化与修饰,均仍属于本发明技术方案的范围内。
序 列 表
SEQUENCE LISTING
<110> 中国科学院广州生物医药与健康研究院
<120> 一种无抗性标记自主发光肺炎克雷伯菌的构建方法及应用
<160> 19
<170> PatentIn version 3.3
<210> 1
<211> 58
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 1
CGGGATCCAGTGCGCATAATGTATATTATGTTAAATCCATGGCACCACCGACTATTTG 58
<210> 2
<211> 59
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 2
TGCTCTAGAATTTAACATAATATACATTATGCGCACTAAGCTTAGCTCAGCCAATCGAC 59
<210> 3
<211> 41
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 3
ATATCCATCACACTGGCGGCCGCGGTGACCACAACGACGCG 41
<210> 4
<211> 45
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 4
GCAAGGCCTTCGCGAGGTACCTCAACTATCAAACGCTTCGGTTAA 45
<210> 5
<211> 39
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 5
TCCAAGCTGGGCTGTGTGCACTTTCGGGGAAATGTGCGC 39
<210> 6
<211> 39
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 6
TTCACACCGCATATGGTGCACCCAACGGCGTCAGCCGGG 39
<210> 7
<211> 44
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 7
GTGCGGTATTTCACACCGCATATGGAGCAAAAGGCCAGCAAAAG 44
<210> 8
<211> 49
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 8
GTCAACTGGGTTCGTGAATTCGATAATAATGGTTTCTTAGACGTCAGGT 49
<210> 9
<211> 200
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 9
TGTGGGCGGACAATAAAGTCTTAAACTGAACAAAATAGATCTAAACTATGACAATAAAGTCTTAAACTAGACAGAATAGTTGTAAACTGAAATCAGTCCAGTTATGCTGTGAAAAAGCATACTGGACTTTTGTTATGGCTAAAGCAAACTCTTCATTTTCTGAAGTGCAAATTGCCCGTCGTATTAAAGAGGGGCGTGGG 200
<210> 10
<211> 166
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 10
AACCAGATAAGTGAAATCTAGTTCCAAACTATTTTGTCATTTTTAATTTTCGTATTAGCTTACGACGCTACACCCAGTTCCCATCTATTTTGTCACTCTTCCCTAAATAATCCTTAAAAACTCCATTTCCACCCCTCCCAGTTCCCAACTATTTTGTCCGCCCACA 166
<210> 11
<211> 6110
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 11
ATGGCTAAAGCAAACTCTTCATTTTCTGAAGTGCAAATTGCCCGTCGTATTAAAGAGGGGCGTGGCCAAGGGCATGGTAAAGACTATATTCCATGGCTAACAGTACAAGAAGTTCCTTCTTCAGGTCGTTCCCACCGTATTTATTCTCATAAGACGGGACGAGTCCATCATTTGCTATCTGACTTAGAGCTTGCTGTTTTTCTCAGTCTTGAGTGGGAGAGCAGCGTGCTAGATATACGCGAGCAGTTCCCCTTATTACCTAGTGATACCAGGCAGATTGCAATAGATAGTGGTATTAAGCATCCTGTTATTCGTGGTGTAGATCAGGTTATGTCTACTGATTTTTTAGTGGACTGCAAAGATGGTCCTTTTGAGCAGTTTGCTATTCAAGTCAAACCTGCAGCAGCCTTACAAGACGAGCGTACCTTAGAAAAACTAGAACTAGAGCGTCGCTATTGGCAGCAAAAGCAAATTCCTTGGTTCATTTTTACTGATAAAGAAATAAATCCCGTAGTAAAAGAAAATATTGAATGGCTTTATTCAGTGAAAACAGAAGAAGTTTCTGCGGAGCTTTTAGCACAACTATCCCCATTGGCCCATATCCTGCAAGAAAAAGGAGATGAAAACATTATCAATGTCTGTAAGCAGGTTGATATTGCTTATGATTTGGAGTTAGGCAAAACATTGAGTGAGATACGAGCCTTAACCGCAAATGGTTTTATTAAGTTCAATATTTATAAGTCTTTCAGGGCAAATAAGTGTGCAGATCTCTGTATTAGCCAAGTAGTGAATATGGAGGAGTTGCGCTATGTGGCAAATTAATGAGGTTGTGCTATTTGATAATGATCCGTATCGCATTTTGGCTATAGAGGATGGCCAAGTTGTCTGGATGCAAATAAGCGCTGATAAAGGAGTTCCACAAGCTAGGGCTGAGTTGTTGCTAATGCAGTATTTAGATGAAGGCCGCTTAGTTAGAACTGATGACCCTTATGTACATCTTGATTTAGAAGAGCCGTCTGTAGATTCTGTCAGCTTCCAGAAGCGCGAGGAGGATTATCGAAAAATTCTTCCTATTATTAATAGTAAGGATCGTTTCGACCCTAAAGTCAGAAGCGAACTCGTTGAGCATGTGGTCCAAGAACATAAGGTTACTAAGGCTACAGTTTATAAGTTGTTACGCCGTTACTGGCAGCGTGGTCAAACGCCTAATGCATTAATTCCTGACTACAAAAACAGCGGTGCACCAGGGGAAAGACGTTCAGCGACAGGAACAGCAAAGATTGGCCGAGCCAGAGAATATGGTAAGGGTGAAGGAACCAAGGTAACGCCCGAGATTGAACGCCTTTTTAGGTTGACCATAGAAAAGCACCTGTTAAATCAAAAAGGTACAAAGACCACCGTTGCCTATAGACGATTTGTGGACTTGTTTGCTCAGTATTTTCCTCGCATTCCCCAAGAGGATTACCCAACACTACGTCAGTTTCGTTATTTTTATGATCGAGAATACCCTAAAGCTCAGCGCTTAAAGTCTAGAGTTAAAGCAGGGGTATATAAAAAAGACGTACGACCCTTAAGTAGTACAGCCACTTCTCAGGCGTTAGGCCCTGGGAGTCGTTATGAGATTGATGCCACGATTGCTGATATTTATTTAGTGGATCATCATGATCGCCAAAAAATCATAGGAAGACCAACGCTTTACATTGTGATTGATGTGTTTAGTCGGATGATCACGGGCTTTTATATCGGCTTTGAAAATCCGTCTTATGTGGTGGCGATGCAGGCTTTTGTAAATGCTTGCTCTGACAAAACGGCCATTTGTGCCCAGCATGATATTGAGATTAGTAGCTCAGACTGGCCGTGTGTAGGTTTGCCAGATGTGTTGCTAGCGGACCGTGGCGAATTAATGAGTCATCAGGTCGAAGCCTTAGTTTCTAGTTTTAATGTGCGAGTGGAAAGTGCTCCACCTAGACGTGGCGATGCTAAAGGCATAGTGGAAAGCACTTTTAGAACACTACAAGCCGAGTTTAAGTCCTTTGCACCTGGCATTGTAGAGGGCAGTCGGATCAAAAGCCATGGTGAAACAGACTATAGGTTAGATGCATCTCTGTCGGTATTTGAGTTCACACAAATTATTTTGCGTACGATCTTATTCAGAAATAACCATCTGGTGATGGATAAATACGATCGAGATGCTGATTTTCCTACAGATTTACCGTCTATTCCTGTCCAGCTATGGCAATGGGGTATGCAGCATCGTACAGGTAGTTTAAGGGCTGTGGAGCAAGAGCAGTTGCGAGTAGCGTTACTGCCTCGCCGAAAGGTCTCTATTTCTTCATTTGGCGTTAATTTGTGGGGTTTGTATTACTCGGGGTCAGAGATTCTGCGTGAGGGTTGGTTGCAGCGGAGCACTGATATAGCTAGACCTCAACATTTAGAAGCGGCTTATGACCCAGTGCTGGTTGATACGATTTATTTGTTTCCGCAAGTTGGCAGCCGTGTATTTTGGCGCTGTAATCTGACGGAACGTAGTCGGCAGTTTAAAGGTCTCTCATTTTGGGAGGTTTGGGATATACAAGCACAAGAAAAACACAATAAAGCCAATGCGAAGCAGGATGAGTTAACTAAACGCAGGGAGCTTGAGGCGTTTATTCAGCAAACCATTCAGAAAGCGAATAAGTTAACGCCCAGTACTACTGAGCCCAAATCAACACGCATTAAGCAGATTAAAACTAATAAAAAAGAAGCCGTGACCTCGGAGCGTAAAAAACGTGCGGAGCATTTGAAGCCAAGCTCTTCAGGTGATGAGGCTAAAGTTATTCCTTTCAACGCAGTGGAAGCGGATGATCAAGAAGATTACAGCCTACCCACATACGTGCCTGAATTATTTCAGGATCCACCAGAAAAGGATGAGTCATGAGTGCTACCCGGATTCAAGCAGTTTATCGTGATACGGGGGTAGAGGCTTATCGTGATAATCCTTTTATCGAGGCCTTACCACCATTACAAGAGTCAGTGAATAGTGCTGCATCACTGAAATCCTCTTTACAGCTTACTTCCTCTGACTTGCAAAAGTCCCGTGTTATCAGAGCTCATACCATTTGTCGTATTCCAGATGACTATTTTCAGCCATTAGGTACGCATTTGCTACTAAGTGAGCGTATTTCGGTCATGATTCGAGGTGGCTACGTAGGCAGAAATCCTAAAACAGGAGATTTACAAAAGCATTTACAAAATGGTTATGAGCGTGTTCAAACGGGAGAGTTGGAGACATTTCGCTTTGAGGAGGCACGATCTACGGCACAAAGCTTATTGTTAATTGGTTGTTCTGGTAGTGGGAAGACGACCTCTCTTCATCGTATTCTAGCCACGTATCCTCAGGTGATTTACCATCGTGAACTCAATGTAGAGCAGGTGGTGTATTTGAAAATAGACTGCTCGCATAATGGTTCGCTAAAAGAAATCTGCTTGAATTTTTTCAGAGCGTTGGATCGAGCCTTGGGCTCGAACTATGAGCGTCGTTATGGCTTAAAACGTCATGGTATAGAAACCATGTTGGCTTTGATGTCGCAAATAGCCAATGCACATGCTTTAGGGTTGTTGGTTATTGATGAAATTCAGCATTTAAGCCGCTCTCGTTCGGGTGGATCTCAAGAGATGCTGAACTTTTTTGTGACGATGGTGAATATTATTGGCGTACCAGTGATGTTGATTGGTACCCCTAAAGCACGAGAGATTTTTGAGGCTGATTTGCGGTCTGCACGTAGAGGGGCAGGGTTTGGAGCTATATTCTGGGATCCTATACAACAAACGCAACGTGGAAAGCCCAATCAAGAGTGGATCGCTTTTACGGATAATCTTTGGCAATTACAGCTTTTACAACGCAAAGATGCGCTGTTATCGGATGAGGTCCGTGATGTGTGGTATGAGCTAAGCCAAGGAGTGATGGACATTGTAGTAAAACTTTTTGTACTCGCTCAGCTCCGTGCGCTAGCTTTAGGCAATGAGCGTATTACCGCTGGTTTATTGCGGCAAGTGTATCAAGATGAGTTAAAGCCTGTGCACCCCATGCTAGAGGCATTACGCTCGGGTATCCCAGAACGCATTGCTCGTTATTCTGATCTAGTCGTTCCCGAGATTGATAAACGGTTAATCCAACTTCAGCTAGATATCGCAGCGATACAAGAACAAACACCAGAAGAAAAAGCCCTTCAAGAGTTAGATACCGAAGATCAGCGTCATTTATATCTGATGCTGAAAGAGGATTACGATTCAAGCCTGTTAATTCCCACTATTAAAAAAGCGTTTAGCCAGAATCCAACGATGACAAGACAAAAGTTACTGCCTCTTGTTTTGCAGTGGTTGATGGAAGGCGAAACGGTAGTGTCAGAACTAGAAAAGCCCTCCAAGAGTAAAAAGGTTTCGGCTATAAAGGTAGTCAAGCCCAGCGACTGGGATAGCTTGCCTGATACGGATTTACGTTATATCTATTCACAACGCCAACCTGAAAAAACCATGCATGAACGGTTAAAAGGGAAAGGGGTAATAGTGGATATGGCGAGCTTATTTAAACAAGCAGGTTAGCCATGAGAAACTTTCCTGTTCCGTACTCGAATGAGCTGATTTATAGCACTATTGCACGGGCAGGCGTTTATCAAGGGATTGTTAGTCCTAAGCAGCTGTTGGATGAGGTGTATGGCAACCGCAAGGTGGTCGCTACCTTAGGTCTGCCCTCGCATTTAGGTGTGATAGCAAGACATCTACATCAAACAGGACGTTACGCTGTTCAGCAGCTTATTTATGAGCATACCTTATTCCCTTTATATGCTCCGTTTGTAGGCAAGGAGCGCCGAGACGAAGCTATTCGGTTAATGGAGTACCAAGCGCAAGGTGCGGTGCATTTAATGCTAGGAGTCGCTGCTTCTAGAGTTAAGAGCGATAACCGCTTTAGATACTGCCCTGATTGCGTTGCTCTTCAGCTAAATAGGTATGGGGAAGCCTTTTGGCAACGAGATTGGTATTTGCCCGCTTTGCCATATTGTCCAAAACACGGTGCTTTAGTCTTCTTTGATAGAGCTGTAGATGATCACCGACATCAATTTTGGGCTTTGGGTCATACTGAGCTGCTTTCAGACTACCCCAAAGACTCCCTATCTCAATTAACAGCACTAGCTGCTTATATAGCCCCTCTGTTAGATGCTCCACGAGCGCAAGAGCTTTCCCCAAGCCTTGAGCAGTGGACGCTGTTTTATCAGCGCTTAGCGCAGGATCTAGGGCTAACCAAAAGCAAGCACATTCGTCATGACTTGGTGGCGGAGAGAGTGAGGCAGACTTTTAGTGATGAGGCACTAGAGAAACTGGATTTAAAGTTGGCAGAGAACAAGGACACGTGTTGGCTGAAAAGTATATTCCGTAAGCATAGAAAAGCCTTTAGTTATTTACAGCATAGTATTGTGTGGCAAGCCTTATTGCCAAAACTAACGGTTATAGAAGCGCTACAGCAGGCAAGTGCTCTTACTGAGCACTCTATAACGACAAGACCTGTTAGCCAGTCTGTGCAACCTAACTCTGAAGATTTATCTGTTAAGCATAAAGACTGGCAGCAACTAGTGCATAAATACCAAGGAATTAAGGCGGCAAGACAGTCTTTAGAGGGTGGGGTGCTATACGCTTGGCTTTACCGACATGACAGGGATTGGCTAGTTCACTGGAATCAACAGCATCAACAAGAGCGTCTGGCACCCGCCCCTAGAGTTGATTGGAACCAAAGAGATCGAATTGCTGTACGACAACTATTAAGAATCATAAAGCGTCTAGATAGTAGCCTTGATCACCCAAGAGCGACATCGAGCTGGCTGTTAAAGCAAACTCCTAACGGAACCTCTCTTGCAAAAAATCTACAGAAACTGCCTTTGGTAGCGCTTTGCTTAAAGCGTTACTCAGAGAGTGTGGAAGATTATCAAATTAGACGGATTAGCCAAGCTTTTATTAAGCTTAAACAGGAAGATGTTGAGCTTAGGCGCTGGCGATTATTAAGAAGTGCAACGTTATCTAAAGAGCGGATAACTGAGGAAGCACAAAGATTCTTGGAAATGGTTTATGGGGAAGAGTGA 6110
<210> 12
<211> 18
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 12
GGTGACCACAACGACGCG 18
<210> 13
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 13
GCAATTGTCTTGGCCATTGC 20
<210> 14
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 14
TATAGTCCTGTCGGGTTTCG 20
<210> 15
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 15
AGACGGATTAGCCAAGCTTT 20
<210> 16
<211> 23
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 16
GGTATCAGCCCATTTGGCATACA 23
<210> 17
<211> 22
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 17
ATTAGCTTACGACGCTACACCC 22
<210> 18
<211> 18
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 18
CCATGGAGCTCAGCCAAT 18
<210> 19
<211> 19
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 19
CCACCGACTATTTGCAACA 19
- 一种无抗性标记自主发光肺炎克雷伯菌的构建方法及应用
- 整合质粒pOPHI及无抗性筛选标记的自主发光分枝杆菌
