肿瘤驱动基因识别方法及装置
文献发布时间:2023-06-19 10:29:05
技术领域
本申请涉及肿瘤基因的计算机分析技术领域,特别的涉及一种肿瘤驱动基因识别方法及装置。
背景技术
肿瘤是一种复杂的疾病,其发病过程是一个阶段性发展的过程,其初期表现为肿瘤细胞自身的异常增长,在发展过程中表现为对邻近正常组织的侵犯以及经血管、淋巴管和体腔转移到其他部位,并经过一系列病理生理的变化最终导致个体死亡。
由于大多数技术目前还无法找出驱动肿瘤发生发展过程的关键基因,因此现在研究的重点倾向于获取肿瘤发展的驱动基因。我们发现,现有肿瘤驱动基因的挖掘多基于差异表达分析,但由于基因的差异表达往往在相同阶段的肿瘤样本中稳定性不高,且不同阶段间特异性不强,因此基于差异表达分析的分类性能并不令人满意,也很难实际应用于分析肿瘤的发展。
发明内容
本申请实施例提供了一种肿瘤驱动基因识别方法及装置,可更全面性的反应基因间的相互作用以及基因参与的生物过程,通过连通分量分析以减少零散网络的干扰,并结合中心性度量指标获取肿瘤驱动基因,以提高识别肿瘤驱动基因的精准性。
第一方面,本申请实施例提供了一种肿瘤驱动基因识别方法,包括:
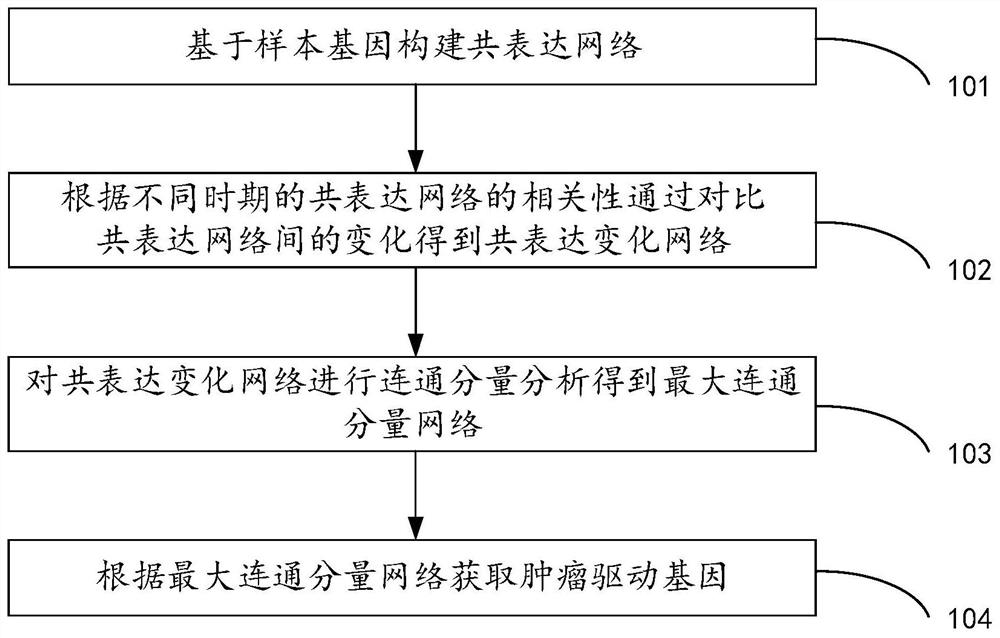
基于样本基因构建共表达网络;样本基因包括正常时期的样本基因及肿瘤四个时期的样本基因;
根据不同时期的共表达网络的相关性通过对比共表达网络间的变化得到共表达变化网络;共表达变化网络用于表征与癌症发展密切相关的样本基因的相关性变化趋势;
对共表达变化网络进行连通分量分析得到最大连通分量网络;
根据最大连通分量网络获取肿瘤驱动基因。
在本申请实施例中,通过构建共表达网络可更全面的反应基因间的相互作用以及基因参与的生物过程,并基于共表达变化模式划分得到共表达变化网络,对共表达变化网络进行连通分量分析以减少零散网络的干扰,并结合中心性度量指标获取肿瘤驱动基因,以提高识别肿瘤驱动基因的精准性。
在第一方面的一种可选方案中,基于样本基因构建共表达网络具体包括:
获取不同时期的样本基因的样本数;
对不同时期的样本基因的样本数进行采样,采样的个数基于不同时期的样本基因的样本数中最小的样本数确定;
基于采样得到的不同时期的样本基因构建对应不同时期的共表达网络。
在第一方面的又一种可选方案中,基于采样得到的不同时期的样本基因构建对应不同时期的共表达网络具体包括:
基于采样得到的不同时期的样本基因构建对应不同时期的初始共表达网络;
基于不同时期的初始共表达网络得到对应于不同时期的P值网络;
根据不同时期的初始共表达网络及对应于不同时期的P值网络得到不同时期的共表达网络。
在第一方面的又一种可选方案中,根据不同时期的共表达网络的相关性通过对比共表达网络间的变化得到共表达变化网络具体包括:
对相邻时期的所述共表达网络进行相关性比较,得到所述肿瘤四个时期的四个共表达变化网络;
基于所述肿瘤四个时期的四个共表达变化网络得到两个共表达变化网络;
其中,共表达变化网络包括呈现递增的共表达变化网络及呈现递减的共表达变化网络。
在第一方面的又一种可选方案中,对共表达变化网络进行连通分量分析得到最大连通分量网络具体包括:
基于共表达变化网络得到每个样本基因所在连通分量中连接的样本基因数量;
将样本基因数量最大的样本基因所在的所述连通分量作为最大连通分量;
根据最大连通分量得到最大连通分量网络。
在第一方面的又一种可选方案中,根据最大连通分量网络获取肿瘤驱动基因具体包括:
计算最大连通分量网络中各个样本基因的中心性指数;
基于各个样本基因的中心性指数确定肿瘤驱动基因。
在第一方面的又一种可选方案中,中心性指数包括度中心性指数、紧密中心性指数及中介中心性指数。
在第一方面的又一种可选方案中,基于超几何分布对肿瘤驱动基因进行富集分析。
第二方面,本申请实施例提供了一种肿瘤驱动基因识别装置,包括:
建立模块,用于基于样本基因构建共表达网络;样本基因包括正常时期的样本基因及肿瘤四个时期的样本基因;
划分模块,用于根据不同时期的共表达网络的相关性通过对比共表达网络间的变化得到共表达变化网络;共表达变化网络用于表征与癌症发展密切相关的样本基因的相关性变化趋势;
分析模块,用于对共表达变化网络进行连通分量分析得到最大连通分量网络;
获取模块,用于根据最大连通分量网络获取肿瘤驱动基因。
第三方面,本申请实施例还提供了一种肿瘤驱动基因识别装置,包括处理器、存储器以及通信接口;
所述处理器与所述存储器、所述通信接口相连;
所述存储器,用于存储可执行程序代码;
所述处理器通过读取所述存储器中存储的可执行程序代码来运行与所述可执行程序代码对应的程序,以用于执行第一方面或第一方面的任意一种实现方式提供的肿瘤驱动基因识别方法。
第四方面,本申请实施例还提供了一种计算机存储介质,计算机存储介质存储有计算机程序,计算机程序包括程序指令,程序指令当被处理器执行时,可实现本申请实施例第一方面或第一方面的任意一种实现方式提供的肿瘤驱动基因识别方法。
可以理解的,上述提供的第四方面提供的计算机存储介质,用于执行第一方面或第一方面的任意一种实现方式所提供的肿瘤驱动基因识别方法,因此,其所能达到的有益效果可参考第一方面或第一方面的任意一种实现方式所提供的肿瘤驱动基因识别方法中的有益效果,此处不再赘述。
附图说明
为了更清楚地说明本申请实施例中的技术方案,下面将对实施例中所需使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本申请的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
图1为本申请实施例提供的一种肿瘤驱动基因识别方法的流程示意图;
图2为本申请实施例提供的一种构建共表达网络的流程示意图;
图3为本申请实施例提供的一种共表达网络的示意图;
图4为本申请实施例提供的一种共表达变化网络的示意图;
图5为本申请实施例提供的一种反应共表达关系的变化趋势的示意图;
图6为本申请实施例提供的又一种反应共表达关系的变化趋势的示意图;
图7为本申请实施例提供的一种最大连通分量的矩阵示意图;
图8为本申请实施例提供的一种肿瘤驱动基因识别装置的结构示意图;
图9为本申请实施例提供的又一种肿瘤驱动基因识别装置的结构示意图。
具体实施方式
下面将结合本申请实施例中的附图,对本申请实施例中的技术方案进行清楚、完整地描述。
本申请的说明书和权利要求书及上述附图中的术语“第一”、“第二”、“第三”等是用于区别不同对象,而不是用于描述特定顺序。此外,术语“包括”和“具有”以及它们任何变形,意图在于覆盖不排他的包含。例如包含了一系列步骤或单元的过程、方法、系统、产品或设备没有限定于已列出的步骤或单元,而是可选地还包括没有列出的步骤或单元,或可选地还包括对于这些过程、方法、产品或设备固有的其他步骤或单元。
请参阅图1,图1示出了本申请实施例提供的一种肿瘤驱动基因识别方法的流程示意图。
如图1所示,该肿瘤驱动基因识别方法包括:
步骤101、基于样本基因构建共表达网络。
具体地,样本基因包括肿瘤四个时期的样本基因及正常时期的样本基因。其中,肿瘤四个时期的样本基因可用于表征患有肿瘤的人群在各阶段变化过程中的样本基因,具体的四个时期可通过S1、S2、S3及S4四个时期进行表示,例如S1时期可表征为肿瘤发展初期,S2时期可表征为肿瘤发展中前期,S3时期可表征为肿瘤发展中后期,S4时期可表征为肿瘤发展末期。正常时期的样本基因可为未患有肿瘤的人群的样本基因,此处引入正常时期的样本基因可与肿瘤四个时期的样本基因做对比,有利于获取肿瘤驱动基因。
具体地,可参阅图2示出的本申请实施例提供的一种构建共表达网络的流程示意图。
如图2所示,该构建共表达网络的方法包括:
步骤1011、获取不同时期的样本基因的样本数。
具体地,可获取多个不同时期的样本基因的样本数量。可能的,不同时期的样本基因的样本数量相同,例如获取10个正常时期的人的样本基因以及各10个分别处于肿瘤四个时期的人的样本基因。可能的,不同时期的样本基因的样本数量不相同,例如获取10个正常时期的人的样本基因以及各100个分别处于肿瘤四个时期的人的样本基因。需要注意的是,此处的样本基因包括人体内的两万多个基因。
步骤1012、对不同时期的样本基因的样本数进行采样。
具体地,不同时期的样本基因的样本数存在一定差异,会在一定程度上降低不同时期的基因样本构建的共表达关系的可比较性,可通过多次采样的方式减小此影响。
具体地,可对不同时期的样本基因进行若干次采样,其采样的样本数基于不同时期的样本基因的样本数确定。具体的,采样的样本数可取决于获取的不同时期的样本基因的样本数中数量最小的样本数,例如以采样的样本数用s
例如,以获取10个正常时期的人的样本基因以及各100个分别处于肿瘤四个时期的人的样本基因为例,当采样次数为10次时,分别对每个时期的样本基因进行采样且采样的样本数为9个,即可以理解为采样得到9个正常时期的人的样本基因以及各9个处于肿瘤四个时期的人的样本基因,其中每一次采样得到的9个正常时期的人的样本基因不完全重叠,每一次采样得到的各9个处于肿瘤四个时期的人的样本基因不完全重叠。
需要说明的是,若基于公式(1)得到采样的样本数不为整数时,可将与该采样的样本数最接近的且数值小于该采样的样本数的整数作为实际采样的样本数。
步骤1013、基于采样得到的不同时期的样本基因构建对应不同时期的共表达网络。
具体地,对采样得到的不同时期的样本基因,可通过斯皮尔曼相关性(Spearman相关性)构建共表达网络。
具体的,可通过公式(2)构建每一次采样的共表达网络
在上述公式(2)中,
进一步的,在采样结束后,可通过下述公式(3)将K次采样得到的结果进行整合,得到不同时期的样本基因的共表达网络。
具体地,在得到的不同时期的样本基因的共表达网络中会发现存在不显著的关联关系的边,为避免混淆,可在构建每一次采样的共表达网络
此处的P值网路与共表达网络属于对应关系,通过显著性分析去除共表达网络中不显著的关联关系,即可先通过删除P值网络中P值大于0.01的所有边,再对应删除共表达网络中与P值网络中被删除的所有边所对应的边,并删除共表达网络中的孤立点(即没有与任何节点相连的节点),最终得到准确的共表达网络。
需要说明的是,本申请实施例中节点可理解为样本基因,构建的共表达网络可用n*n的矩阵进行表示,在矩阵中第i行第j列表示的数值为基因i和j间的共表达值且与第j行第i列的共表达值相同。其中,n、i以及j均为正整数。
具体的,可参阅图3示出的本申请实施例提供的一种共表达网络的示意图,图3中正常对应为正常时期的样本基因的共表达网络,S1、S2、S3及S4分别对应代表肿瘤不同时期的样本基因的共表达网络。
步骤102、根据不同时期的共表达网络的相关性通过对比共表达网络间的变化得到共表达变化网络。
具体地,可将相邻时期的共表达网络中各个节点的相关性进行比较,得出在肿瘤四个时期的共表达网络中呈现不同变化趋势的共表达网络。具体的,可参阅图4示出的本申请实施例提供的一种共表达变化网络的示意图。如图4所示,例如可将正常时期对应的共表达网络与S1时期对应的共表达网络进行比较,其中对于任意一对共表达关系(可以理解为任意两个节点的连接边)在正常时期对应的共表达网络与S1时期对应的共表达网络中均存在一个共表达值,可能的,从正常时期到S1时期若任意一对共表达关系的共表达值呈现显著性增强,记该共表达关系的共表达趋势用1表示。可能的从正常时期到S1时期若任意一对共表达关系的共表达值呈现显著性减小,记该共表达关系的共表达趋势用-1表示。可能的从正常时期到S1时期若任意一对共表达关系的共表达值未发生变化,记该共表达关系的共表达趋势用0表示。进一步的,将共表达关系变化趋势呈现为递增(即用1表示的共表达趋势)或递减(即用-1表示的共表达趋势)的基因关系定义为肿瘤发展相关的基因关系(如图4所示,5个共表达网络中上半部分为共表达关系呈现递增趋势的共表达网络,下半部分为共表达关系呈现递减趋势的共表达网络)。
需要说明的是,上述共表达关系的共表达值在[-1,1]之间,其中共表达值在[-1,0]之间时可表征为负相关,共表达值在[0,1]之间时可表征为正相关,共表达值在0时可表征为不相关,因此可通过观察共表达值的绝对值的变化来判断共表达关系的变化趋势。例如相邻时期的共表达网络中某一对共表达关系的共表达值从0到-1,可理解为共表达值从0到1,即该共表达关系在这相邻时期内呈现递增趋势。又例如相邻时期的共表达网络中某一对共表达关系的共表达值从-1到-0.5,可理解为共表达值从1到0.5,即该共表达关系在这相邻时期内呈现递减趋势。但当出现共表达值从负数到正数或从正数到负数时,可直接通过观察共表达值的变化来判断共表达关系的变化趋势。例如相邻时期的共表达网络中某一对共表达关系的共表达值从0.5到-1,即该共表达关系在这相邻时期内呈现递减趋势。
示例性的,可参阅图5示出的一种反应共表达关系的变化趋势的示意图。如图5所示,正常时期对应的3*3矩阵中的各个数值对应为各个共表达关系的共表达值,S1时期对应的3*3矩阵中的各个数值对应为各个共表达关系的共表达值,正常时期与S1时期为相邻时期,根据正常时期与S1时期对应位置的共表达值的变化趋势确定共表达变化网络的矩阵(如图5中的下半部分),基于共表达趋势为-1的共表达关系在对应的共表达网络中呈现为共表达关系呈现递减趋势的共表达网络,基于共表达趋势为1的共表达关系在对应的共表达网络中呈现为共表达关系呈现递增趋势的共表达网络。
同样的方式,可将S1时期对应的共表达网络与S2时期、S2时期对应的共表达网络与S3时期、S3时期对应的共表达网络与S4时期对应的共表达网络进行比较。可以理解的是,在肿瘤四个时期的共表达网络中均存在一个共表达关系变化趋势呈现为递增的共表达网络及一个共表达关系变化趋势呈现为递减的共表达网络。
进一步的,在相应得到四个共表达关系变化趋势呈现为递增的共表达网络后,分别对该四个共表达关系变化趋势呈现为递增的共表达网络进行归整。具体的,对该四个共表达关系变化趋势呈现为递增的共表达网络中处于同一位置的共表达关系根据共表达趋势判断是否为呈现递增趋势的共表达关系,且需要注意的是允许在四个共表达趋势中存在一个0,即可以理解为当四个共表达关系变化趋势呈现为递增的共表达网络中处于同一位置的共表达关系所对应的共表达趋势满足(1,1,1,0)或(1,0,1,1)或(1,1,0,1)或(1,1,1,1)时,确定将该位置的共表达关系作为归整的共表达关系变化趋势呈现为递增的共表达网络中的同一位置的共表达关系,并根据此方式归整得到一个共表达关系变化趋势呈现为递增的共表达网络。此处还需要注意的是,在正常时期与肿瘤S1时期对应的共表达网络进行比较时需满足共表达趋势为1。
同样的方式,在相应得到四个共表达关系变化趋势呈现为递减的共表达网络后,分别对该四个共表达关系变化趋势呈现为递减的共表达网络进行归整。具体的,对该四个共表达关系变化趋势呈现为递减的共表达网络中处于同一位置的共表达关系根据共表达趋势判断是否为呈现递减趋势的共表达关系,且需要注意的是允许在四个共表达趋势中存在一个0,即可以理解为当四个共表达关系变化趋势呈现为递减的共表达网络中处于同一位置的共表达关系所对应的共表达趋势满足(-1,-1,-1,0)或(-1,0,-1,-1)或(-1,-1,0,-1)或(-1,-1,-1,-1)时,确定将该位置的共表达关系作为归整的共表达关系变化趋势呈现为递减的共表达网络中的同一位置的共表达关系,并根据此方式归整得到一个共表达关系变化趋势呈现为递减的共表达网络。此处还需要注意的是,在正常时期与肿瘤S1时期对应的共表达网络进行比较时需满足共表达趋势为-1。
具体地,我们会发现虽然将不同时期的共表达网络归整划分为共表达关系呈现递增趋势的共表达网络和共表达关系呈现递减趋势的共表达网络,但两种共表达关系呈现趋势的共表达网络存在共同节点,为提高获取肿瘤驱动基因的准确性,可将同时作用于两种共表达关系呈现趋势的共表达网络的节点删除,优选的将两种共表达关系呈现趋势的共表达网络中连接节点数量大于5的节点删除,以得到区分更加明确的共表达关系呈现递增趋势的共表达网络和共表达关系呈现递减趋势的共表达网络,具体的可参阅图6示出的本申请实施例提供的又一种反应共表达关系的变化趋势的示意图。
步骤103、对共表达变化网络进行连通分量分析得到最大连通分量网络。
具体地,为更好的评价与肿瘤发展密切相关的基因间的作用关系,在规整得到一个共表达关系变化趋势呈现为递增的共表达网络以及一个共表达关系变化趋势呈现为递减的共表达网络后,分别对这两个共表达关系变化趋势呈现不同的共表达网络计算连通性,以分别获得对应的连通分量。
具体地,可将共表达关系变化趋势呈现为递增的共表达网络或共表达关系变化趋势呈现为递减的共表达网络以邻接矩阵A表示,此处根据邻接矩阵A相乘的物理意义,可知在A的m次幂中,如果存在
在上述公式(6)中,I为单位矩阵,B
需要说明的是,此处的邻接矩阵A以及矩阵B
具体地,为了便于判断共表达变化网络的节点是否处于同一连通分量,通过公式(7)将B
其中B′
其中,需要说明的是,上述公式(6)中的
示例性的,可参阅图7示出的本申请实施例提供的一种最大连通分量的矩阵示意图。如图7所示,在矩阵中可以得到两个连通分量(A、B、C与D、E)节点A或节点B或节点C连接的节点数量最多,为3个,即可理解为A、B及C组成最大的连通分量。
步骤104、根据最大连通分量网络获取肿瘤驱动基因。
具体地,在得到最大连通分量网络后,可计算在该最大连通分量网络的各个样本基因的中心性指数。其中,中心性指数具体包括度中心性(DC)指数、紧密中心性(CC)指数和中介中心性(BC)指数,并基于公式(8)计算最大连通分量网络的各个样本基因的度中心性指数,基于公式(9)计算最大连通分量网络的各个样本基因的紧密中心性指数以及基于公式(10)计算最大连通分量网络的各个样本基因的中介中心性指数。
其中,V为最大连通分量网络的节点;degree(v)对应节V的链接数;n对应最大连通分量网络中节点的总数;δ
进一步的,通过上述公式(8)得到最大连通分量网络中各个节点的度中心性指数,并进行排列,优选的其排列顺序可为从大到小。通过上述公式(9)得到最大连通分量网络中各个节点的紧密中心性指数,并进行排列,优选的其排列顺序可为从大到小。通过上述公式(10)得到最大连通分量网络中各个节点的中介中心性指数,并进行排列,优选的其排列顺序可为从大到小。
接着,可按照从大到小的顺序在各个中心性指数中获取预设数量的节点,并获取各个中心性指数中的节点的交集,将交集节点作为肿瘤驱动基因对应的节点。其中,预设数量可选取为50、100、200等。
在本申请实施例中,可通过构建共表达网络可更全面性的反应基因间的相互作用以及基因参与的生物过程,并基于共表达变化模式划分得到共表达变化网络,对共表达变化网络进行连通分量分析以减少零散网络的干扰,并结合中心性度量指标获取肿瘤驱动基因,以提高识别肿瘤驱动基因的精准性。
具体地,在获取肿瘤驱动基因后,还可对该肿瘤驱动基因进行富集分析。
具体的,可通过现有的生物过程或通路富集分析,即通过超几何检验(统计显着性临界值=0.005),以评估该肿瘤驱动基因富集在每个生物过程或通路的统计显着性,计算方式可参阅公式(11)。
在上述公式(11)中,m-1表示肿瘤驱动基因在基因总列表中并且在通路中基因的数量;M表示肿瘤驱动基因在基因总列表中的数量;N表示基因总列表中基因的数量;n表示每个通路中基因与基因基因总列表中基因交集的数量。
其中,每一个通路或生物进程可表征对应的基因在肿瘤发展过程中可执行的功能,例如细胞增殖等,每一个通路或生物进程都有对应的基因及数目。
通过比较不同通路或生物进程对应P值的大小来判断该肿瘤驱动基因执行相应通路或生物进程的功能的概率,例如P值越大表明该肿瘤基因执行对应通路或生物进程的功能的概率越高。
请参阅图8,图8示出了本申请实施例提供的一种肿瘤驱动基因识别装置的结构示意图。
如图8所示,该肿瘤驱动基因识别装置800包括建立模块801、划分模块802、分析模块803以及获取模块804,其中各个模块的详细描述如下:
建立模块801,用于基于样本基因构建共表达网络;样本基因包括正常时期的样本基因及肿瘤四个时期的样本基因;
划分模块802,用于根据不同时期的共表达网络的相关性通过对比共表达网络间的变化得到共表达变化网络;共表达变化网络用于表征样本基因的相关性变化趋势;
分析模块803,用于对共表达变化网络进行连通分量分析得到最大连通分量网络;
获取模块804,用于根据最大连通分量网络获取肿瘤驱动基因。
请参阅图9,图9示出了本申请实施例提供的又一种肿瘤驱动基因识别装置的结构示意图。如图9所示,该肿瘤驱动基因识别装置900可以包括:至少一个处理器901、至少一个网络接口905、用户接口904、存储器902以及至少一个通信总线903。
其中,通信总线903可用于实现上述各个组件的连接通信。
其中,用户接口904可以包括按键,可选用户接口还可以包括标准的有线接口、无线接口。
其中,网络接口905可选的可以蓝牙模块、NFC模块、Wi-Fi模块等。
其中,处理器901可以包括一个或者多个处理核心。处理器901利用各种接口和线路连接整个肿瘤驱动基因识别装置900内的各个部分,通过运行或执行存储在存储器902内的指令、程序、代码集或指令集,以及调用存储在存储器902内的数据,执行肿瘤驱动基因识别装置900的各种功能和处理数据。可选的,处理器901可以采用DSP、FPGA、PLA中的至少一种硬件形式来实现。处理器901可集成CPU、GPU和调制解调器等中的一种或几种的组合。其中,CPU主要处理操作系统、用户界面和应用程序等;GPU用于负责显示屏所需要显示的内容的渲染和绘制;调制解调器用于处理无线通信。可以理解的是,上述调制解调器也可以不集成到处理器901中,单独通过一块芯片进行实现。
其中,存储器902可以包括RAM,也可以包括ROM。可选的,该存储器902包括非瞬时性计算机可读介质。存储器902可用于存储指令、程序、代码、代码集或指令集。存储器902可包括存储程序区和存储数据区,其中,存储程序区可存储用于实现操作系统的指令、用于至少一个功能的指令(比如触控功能、声音播放功能、图像播放功能等)、用于实现上述各个方法实施例的指令等;存储数据区可存储上面各个方法实施例中涉及到的数据等。存储器902可选的还可以是至少一个位于远离前述处理器901的存储装置。如图9所示,作为一种计算机存储介质的存储器902中可以包括操作系统、网络通信模块、用户接口模块以及肿瘤驱动基因识别应用程序。
具体地,处理器901可以用于调用存储器902中存储的肿瘤驱动基因识别应用程序,并具体执行以下操作:
基于样本基因构建共表达网络;样本基因包括正常时期的样本基因及肿瘤四个时期的样本基因;
根据不同时期的共表达网络的相关性通过对比共表达网络间的变化得到共表达变化网络,并通过共表达变化网络提取共表达值递增和递减的共表达趋势网络;共表达变化网络用于表征样本基因的相关性变化趋势;
对共表达变化网络进行连通分量分析得到最大连通分量网络;
根据最大连通分量网络获取肿瘤驱动基因。
本申请实施例还提供了一种计算机可读存储介质,该计算机可读存储介质中存储有指令,当其在计算机或处理器上运行时,使得计算机或处理器执行上述图1或图2所示实施例中的一个或多个步骤。上述肿瘤驱动基因识别装置的各组成模块如果以软件功能单元的形式实现并作为独立的产品销售或使用时,可以存储在所述计算机可读取存储介质中。
在上述实施例中,可以全部或部分地通过软件、硬件、固件或者其任意组合来实现。当使用软件实现时,可以全部或部分地以计算机程序产品的形式实现。所述计算机程序产品包括一个或多个计算机指令。在计算机上加载和执行所述计算机程序指令时,全部或部分地产生按照本申请实施例所述的流程或功能。所述计算机可以是通用计算机、专用计算机、计算机网络、或者其他可编程装置。所述计算机指令可以存储在计算机可读存储介质中,或者通过所述计算机可读存储介质进行传输。所述计算机指令可以从一个网站站点、计算机、服务器或数据中心通过有线(例如同轴电缆、光纤、数字用户线(Digital SubscriberLine,DSL))或无线(例如红外、无线、微波等)方式向另一个网站站点、计算机、服务器或数据中心进行传输。所述计算机可读存储介质可以是计算机能够存取的任何可用介质或者是包含一个或多个可用介质集成的服务器、数据中心等数据存储设备。所述可用介质可以是磁性介质,(例如,软盘、硬盘、磁带)、光介质(例如,数字多功能光盘(Digital VersatileDisc,DVD))、或者半导体介质(例如,固态硬盘(Solid State Disk,SSD))等。
本领域普通技术人员可以理解实现上述实施例方法中的全部或部分流程,可以通过计算机程序来指令相关的硬件来完成,该程序可存储于计算机可读取存储介质中,该程序在执行时,可包括如上述各方法的实施例的流程。而前述的存储介质包括:ROM、RAM、磁碟或者光盘等各种可存储程序代码的介质。在不冲突的情况下,本实施例和实施方案中的技术特征可以任意组合。
以上所述的实施例仅仅是本申请的优选实施例方式进行描述,并非对本申请的范围进行限定,在不脱离本申请的设计精神的前提下,本领域普通技术人员对本申请的技术方案作出的各种变形及改进,均应落入本申请的权利要求书确定的保护范围内。
- 肿瘤驱动基因识别方法及装置
- 基于NIR-Ib窗口荧光的肿瘤切除识别方法和装置
