适体选择方法
文献发布时间:2023-06-19 12:16:29
本申请要求在2018年8月3日提交的标题为“Methods for Aptamer Selection”的美国临时申请62/714,102的优先权;其内容通过引用以其全文并入本文。
随本申请一起提交的有电子格式的序列表文本文件。序列表以标题为2066_1011PCT_SL.txt的文件提供,该文件创建于2019年8月1日,大小为14,790,207字节。所述序列表的内容通过引用以其全文并入本文。
技术领域
本公开涉及鉴定针对感兴趣的靶标(例如过敏原)的适体的方法。本公开还提供了适体、信号传导多核苷酸(SPN)、DNA芯片、检测传感器和试剂盒,以及用于检测样品中靶标的测定法(assay)。
技术背景
核酸适体是能够以高亲和力和高特异性与靶分子结合的单链寡核苷酸(DNA、RNA或DNA/RNA杂交体)。通常通过吸附、回收和再扩增的重复过程,例如通过常规SELEX(Systematic Evolution of Ligands by Exponential Enrichment,指数富集的配体系统进化技术)和其他密切相关的方法(参见例如美国专利号:5,270,163、5,567,588;5,637,459、5,670,637、5,705,337和5,723,592),来从具有随机序列的寡核苷酸文库中选择核酸适体。适体可以采取独特的二级和三级结构,并以高亲和力和高特异性识别靶标。
由于不需要在动物或细胞系中进行适体选择,故适体作为抗体的替代物性价比高,它们具有多年的货架期,并且它们可以容易地被修饰以减少与不希望的分子的交叉反应性。适体与抗体相比具有显著的优势,例如更好的特异性和亲和力、更广泛的靶标种类、更容易的合成和修饰、更高的稳定性和更低的成本。这些特性有利于使适体成为广泛用于生物传感器开发以及其他领域的新的检测剂,用于检测样品中靶分子的存在、不存在和/或数量。例如,除许多其他有用的应用(例如诊断性测试和治疗)外,适体和基于适体的测定法还显示出是食品安全控制中有前景的替代物,例如对食物中病原体、毒素、过敏原和其他禁用污染物的检测和控制(Amaya-Gonzalez等人,Sensors,2013,13:16292-16311;和Amaya-Gonzalez等人,Anal.Chem.2014,86(5),2733-2739)。基于适体的测定法替代了许多使用抗体的免疫印迹方法(例如ELISA)。
过敏(例如食物过敏)是常见的医学病况。据估计,在美国有多达2%的成人和多达8%的儿童(尤其是那些三岁以下的儿童)患有食物过敏(约1500万人),并且认为这个患病率在持续增加。无论是在临床背景下还是从消费者的角度,过敏原检测对于对某些类型的食物(例如麸质和花生)过敏的人都是重要的。针对过敏原的灵敏且特异的检测剂是开发能够在食用前高效且快速地对可疑食品进行检测的检测测定法的关键。在许多过敏原检测传感器和测定法中均采用了选择性结合过敏原的适体(Weng和Neethirajan,BiosensBioelectron,2016,85:649-656;Svobodova等人,Food Chem.,2014,165:419-423;Tran等人,Biosens.Bioelectron,2013,43,245-251;和Nadal等人,Plos One,2012,7(4):e35253)。研究已表明,基于适体的测定法相比基于抗体的免疫测定法(例如ELISA)具有显著优势。
本公开开发了鉴定针对特定过敏原靶标的适体序列的改进的选择方法;所述适体和/或衍生自适体的信号多核苷酸可以直接用于检测测定法中,具有增加的特异性和灵敏度。具体而言,所述改进的选择方法联合了多个正选择(positive selection)、负选择(negative selection)和反向选择(counter selection)过程,以鉴定可以特异性识别靶分子(例如,过敏原蛋白)的适体,而此类适体的特征(例如,一级结构和二级结构)阻断了与靶标结合的同一适体与包含与所述同一适体互补的序列的短寡核苷酸杂交。因此,靶标和短互补序列不同时与同一适体结合。
发明内容
本公开提供了为选择针对靶分子的适体而定制的筛选方法,所述适体可以直接用于基于竞争的靶标检测测定法,例如过敏原检测测定法;所述方法包括多个正、负(反向)选择过程(process),以鉴定具有特定一级和二级结构特征的适体,从而当所述适体与靶分子结合形成适体:靶标复合物时,其不会同时与互补于适体序列的短寡核苷酸杂交。所鉴定的适体适合于开发用于靶标检测的芯片传感器,其中在与所述适体的序列互补的寡核苷酸(即锚定序列)存在下,所述适体或源自所述适体的信号多核苷酸竞争结合其靶分子。
在一些实施方案中,所述筛选方法包括:(a)制备包含多个单链DNA(ssDNA)分子的输入DNA文库(input DNA library),每个单链DNA包含一个中央随机核酸序列,两侧是5'端的恒定序列和3'端的恒定序列,所述恒定的5'端和恒定的3'端充当引物;(b)从(a)的输入DNA文库中选择基本上(substantially)与靶标物结合的ssDNA分子的池;(c)从获得自(b)的ssDNA分子靶标结合池中,选择在靶标物存在下不与互补序列杂交(即不同时与靶标和互补序列结合)的ssDNA分子的池;(d)从获得自(c)的正结合ssDNA分子池中,反向选择在靶标物不存在下不与互补序列杂交的ssDNA分子,或者基本上与反向靶标物(couter targetmaterial)结合的ssDNA分子;和(e)从(c)中的正结合ssDNA分子池中减去获得自(d)的ssDNA分子池,并鉴定与感兴趣的靶标特异性结合的候选ssDNA分子。ssDNA分子的每个子池均可通过SELEX和/或芯片上正选择过程进行鉴定,并且每个过程可在相同条件下重复数轮。
因此,通过本发明的筛选方法鉴定的适体和衍生自所述适体的信号传导多核苷酸(SPN)以高亲和力和高特异性与其靶分子结合。在一些实施方案中,所述适体和SPN可在靶分子存在下不与短互补序列杂交,但它们能在靶分子不存在下与短互补序列结合。
在一些实施方案中,本发明的筛选方法进一步包括在每个选择过程之后对每个池中的ssDNA分子进行扩增。可以通过PCR并使用标记有荧光团探针的引物对对所述ssDNA分子进行扩增。由此,所扩增和再生的ssDNA分子标记有荧光团探针。
在一些实施方案中,可以使用输入ssDNA文库和靶标物,通过改进的氧化石墨烯(GO)-SELEX流程来选择基本上与靶标结合的ssDNA分子池。这个正选择可以包括以下步骤:(i)使输入ssDNA文库与靶标物接触,其中在靶标和输入文库中存在的多个ssDNA分子之间形成复合物;(ii)使用氧化石墨烯(GO)溶液分隔步骤(i)中形成的复合物,并分离复合物中的ssDNA分子以产生针对靶标物的ssDNA分子的子集;(iii)使(ii)中的ssDNA分子的子集与相同的靶标物接触,其中在靶标和ssDNA分子子集中存在的第二群ssDNA分子之间形成复合物,以产生ssDNA分子的第二子集;和(iv)任选地将步骤(ii)至(iii)重复一轮、两轮、三轮或更多轮以产生各自的ssDNA分子的第三、第四、第五或更多子集,从而产生基本上与靶标结合的ssDNA分子的富集池。
在一些实施方案中,使用ssDNA分子靶标结合池(即通过GO-SELEX流程选择的ssDNA分子池)、相同的靶标物和包被有包含与ssDNA分子的序列(例如ssDNA的5’末端的恒定序列)互补的序列的短寡核苷酸的固体支持物,通过芯片上正结合选择过程,来选择在靶标物存在下不与互补序列结合的正ssDNA分子池。
在一些实施方案中,可以进一步完善通过芯片上正选择过程选择的正结合ssDNA分子池,以减去非特异性ssDNA分子。反向选择可以包括:(i)从正ssDNA分子池(例如,来自芯片上正选择的池)中反向选择即使在靶标物不存在下也不与互补序列结合的ssDNA分子池(即非结合ssDNA分子);这个选择包括芯片上非结合反向过程,其使用了正结合ssDNA分子池作为输入以及包被有包含ssDNA分子互补序列的短寡核苷酸的芯片;和(ii)从正结合ssDNA分子池中反向选择基本上与反向靶分子结合的ssDNA分子池;这个选择包括芯片上反向结合过程,该过程使用了正结合ssDNA分子池作为输入、一种或多种反向靶标物以及包被有包含与ssDNA分子的序列互补的序列的短寡核苷酸的芯片。
在一些实施方案中,靶标物可以是常见的过敏原,例如常见的食物过敏原。在一个实施方案中,靶标物是花生(peanut)、杏仁(almond)、巴西坚果(brazil nut)、腰果(cashew)、榛子(hazelnut)、山核桃(pecan)、开心果(pistachio)、核桃(walnut)、麸质(gluten)、乳清(whey)和/或酪蛋白(casein)。
在另一方面,本公开提供了用于检测样品中靶标(例如过敏原)的存在、不存在和/或数量的适体、信号传导多核苷酸(SPN)、DNA芯片、基于适体的检测传感器以及试剂盒。
在一些实施方案中,通过本发明的选择过程来选择特异性结合过敏原的适体序列,其中所述过敏原是常见的食物过敏原,例如花生、杏仁、巴西坚果、腰果、榛子、山核桃、开心果、核桃、麸质、乳清和酪蛋白。也可以通过例如多个SELEX方法选择可以与所有坚果结合的适体,所述坚果包括花生、杏仁、巴西坚果、腰果、榛子、山核桃、开心果和核桃。
在一些实施方案中,选择与花生特异性结合的适体序列,其可以包含选自由SEQID NO.3至1002组成的组的独特核酸序列。在一些实例中,针对花生的适体可以包含选自由SEQ ID NO.1003至4002组成的组的核酸序列。
在一些实施方案中,选择与杏仁特异性结合的适体序列,其可以包含选自由SEQID No.4003至5002组成的组的独特核酸序列。在一些实例中,针对杏仁的适体可以包含选自由SEQ ID NO.5003至8002组成的组的核酸序列。
在一些实施方案中,选择与巴西坚果特异性结合的适体序列,其可以包含选自由SEQ ID NO.8003至9002组成的组的独特核酸序列。在一些实例中,针对巴西坚果的适体可以包含选自由SEQ ID NO.9003至12002组成的组的核酸序列。
在一些实施方案中,选择与腰果特异性结合的适体序列,其可以包含选自由SEQID NO.12003至13002组成的组的独特核酸序列。在一些实例中,针对腰果的适体可以包含选自由SEQ ID NO.13003至16002组成的组的核酸序列。
在一些实施方案中,选择与榛子特异性结合的适体序列,其可以包含选自由SEQID NO.16003至17002组成的组的独特核酸序列。在一些实例中,针对榛子的适体可以包含选自由SEQ ID NO.17003至20002组成的组的核酸序列。
在一些实施方案中,选择与山核桃特异性结合的适体序列,其可以包含选自由SEQID NO.20003至21002组成的组的独特核酸序列。在一些实例中,针对山核桃的适体可以包含选自由SEQ ID NO.21003至24002组成的组的核酸序列。
在一些实施方案中,选择与开心果特异性结合的适体序列,其可以包含选自由SEQID NO.24003至25002组成的组的独特核酸序列。在一些实例中,针对开心果的适体可以包含选自由SEQ ID NO.25003至28002组成的组的核酸序列。
在一些实施方案中,选择与核桃特异性结合的适体序列,其可以包含选自由SEQID NO.28003至29002组成的组的独特核酸序列。在一些实例中,针对核桃的适体可以包含选自由SEQ ID NO.29003至32002组成的组的核酸序列。
在一些实施方案中,选择可以与所有坚果结合的适体序列,其可以包含选自由SEQID NO.32003至33002组成的组的独特核酸序列。在一些实例中,针对所有坚果的适体可以包含选自由SEQ ID NO.33003至36002组成的组的核酸序列。
在一些实施方案中,选择与麸质特异性结合的适体序列,其可以包含选自由SEQID NO.40003至41002组成的组的独特核酸序列。在一些实例中,针对麸质的适体可以包含选自由SEQ ID NO.41003至44002组成的组的核酸序列。
在一些实施方案中,选择与乳清特异性结合的适体序列,其可以包含选自由SEQID NO.44003至45002组成的组的独特核酸序列。在一些实例中,针对乳清的适体可以包含选自由SEQ ID NO.45003至48002组成的组的核酸序列。
在一些实施方案中,选择与酪蛋白特异性结合的适体序列,其可以包含选自由SEQID NO.48003至49002组成的组的独特核酸序列。在一些实例中,针对酪蛋白的适体可以包含选自由SEQ ID NO.49003至52002组成的组的核酸序列。
在一些实施方案中,可以选择与靶标对照物特异性结合的适体序列。此类对照序列可以和与靶标结合的适体序列一起用在检测测定中。对照适体序列对样品(例如食物)的应答与靶标特异性适体相似。然而,对照适体将不对靶标(例如靶过敏原)作出应答,并且对靶标特异性适体或与靶标特异性适体互补的短锚定序列不具有结合亲和力。例如,与花生对照物结合的适体序列可以和针对花生的适体序列一起用于检测食物样品中花生的存在/不存在。花生对照序列和对花生特异的适体可以显示出对待测食物类型的相似应答。因此,来自花生对照序列的信号可用作内部样品对照。
在一些实例中,选择与花生对照物结合的适体序列,其可以包含选自由SEQ IDNO.36003至37002组成的组的独特核酸序列。在一些实例中,针对花生对照的适体序列可以包含选自由SEQ ID NO.37003至40002组成的组的核酸序列。
根据本公开,SPN可以包含通过本发明的方法选择的与感兴趣的靶标特异性结合的适体,以及与所述适体序列互补的短核酸序列。可以将所述短互补序列印制在固体表面上用于检测测定。在一些实施方案中,所述短互补序列可以包含选自由SEQ ID NO.52003至52042组成的组的核酸序列。
在另一个方面,本公开提供了使用通过本发明的筛选方法鉴定的适体和SPN来检测样品中靶标的存在、不存在和/或数量的方法。在一些实施方案中,靶标是食物过敏原,且待测样品是食物样品。食物过敏原可以是花生、杏仁、巴西坚果、腰果、榛子、山核桃、开心果、核桃、麸质、乳清和酪蛋白。
附图简要说明
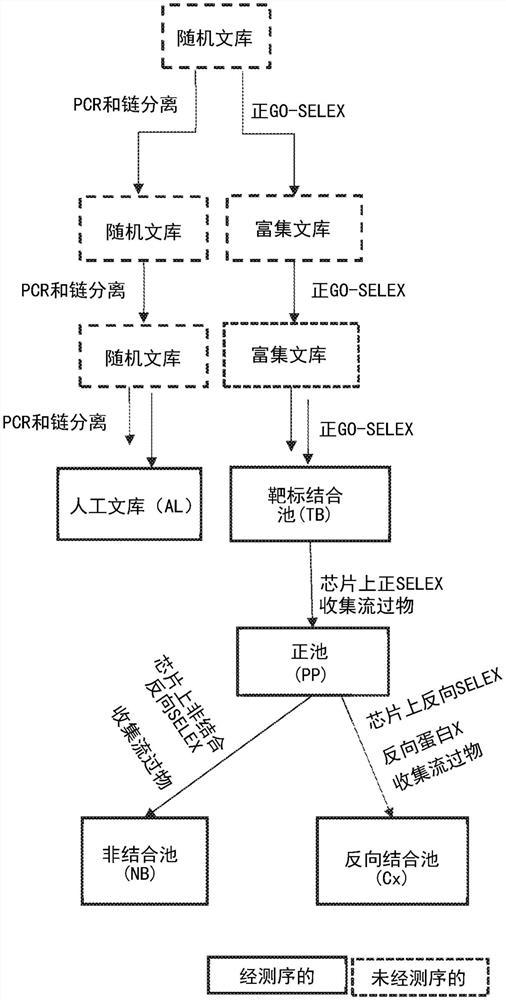
图1是展示本公开的适体筛选方法的实施方案的流程图。
发明详述
前述内容已经相当广泛地概述了本公开的特征和技术优势,以便可以更好地理解以下对本公开的详细描述。在下文中将描述形成本公开的权利要求的主题的本公开的其他特征和优势。本领域技术人员应当理解,所公开的概念和具体实施方案可以容易地用作改动或设计用于实现与本公开相同目的的其他结构的基础。本领域技术人员还应该认识到,这样的等同构造不脱离所附权利要求书中阐述的本公开的精神和范围。当结合附图考虑时,将从以下描述中更好地理解被认为是本公开的特征的新颖特征(就其操作组构和方法而言)以及进一步的目的和优势。然而,应当清楚地理解,每个附图仅出于例示和描述的目的,而并不旨在作为对本公开的限制。除非另有定义,否则本文所用的所有技术和科学术语具有的含义均与本公开所属领域的普通技术人员通常所理解的含义相同。在有冲突的情况下,以本说明书为准。
本发明的筛选方法改进了常规的适体选择方法,其组合了多个正和负(反向)选择,以鉴定与感兴趣的靶标特异性结合的适体。这些选择模拟了基于竞争的检测测定的条件,其中使用适体(或衍生自适体的SPN)捕获其靶标,并使用包含与适体互补的序列的短寡核苷酸来检测适体:靶标复合物的存在或不存在。所述竞争尤其是在适体可以以高水平的特异性和亲和力与其结合的靶标与适体的互补序列之间。所选适体序列能够与其靶标特异性结合,但仅在靶标不存在下与短互补序列杂交。所选适体序列在其靶标存在下不能与短互补序列结合。本发明的筛选方法还选择了针对特定靶标物的对照适体序列。所述对照适体序列可与靶标特异性适体同时使用并用作内部对照。筛选方法的详细说明也涵盖在内。
为了使本公开更容易理解,以下定义了某些术语和短语。其他术语和短语也在说明书中有定义和阐述。
如本文所用,术语“适体(aptamer)”是指能与特定靶分子结合的核酸分子或肽。核酸适体是具有至少一个针对靶分子的结合位点的核酸分子,所述靶分子例如另一个核酸序列、蛋白、肽、抗体、有机小分子、矿物质、细胞和组织。核酸适体可以是单链或双链脱氧核糖核酸(ssDNA或dsDNA),或核糖核酸(RNA),或DNA/RNA杂交体。核酸适体的长度范围通常为10-150个核苷酸,例如长度范围为15-120个核苷酸,或长度范围为20-100个核苷酸,或长度范围为20-80个核苷酸,或长度范围为30-90个核苷酸,或长度范围为50-90个核苷酸。适体的核酸序列可以任选地具有以下最小长度:10个、11个、12个、13个、14个、15个、16个、17个、18个、19个、20个、21个、22个、23个、24个、25个、26个、27个、28个、29个、30个、31个、32个、33个、34个、35个、36个、37个、38个、39个、40个、41个、42个、43个、44个、45个、46个、47个、48个、49个、50个、51个、52个、53个、54个、55个、56个、57个、58个、59个、60个、61个、62个、63个、64个、65个、66个、67个、68个、69个、70个、71个、72个、73个、74个、75个、76个、77个、78个、79个、80个、81个、82个、83个、84个、85个、86个、87个、88个、89个、90个、91个、92个、93个、94个、95个、96个、97个、98个、99个或100个核苷酸。在本公开的上下文中,术语“适体”是指核酸适体。术语“单链DNA(ssDNA)分子”和“适体”可互换使用。
适体可以折叠成特异和稳定的二级、三级或四级构象结构,使其能够以高特异性和高亲和力与靶标结合。所述结构可以包括但不限于发夹环、凸环(bulge loop)、内部环、多分支环、假结(pseudoknot)或其组合。例如,适体的结合位点可以包含茎环构象或G-四联体。
针对靶标的适体可以天然存在或通过合成或重组手段制备。可以通过重复轮次的核酸分子体外分隔(partition)、选择和扩增(例如常规SELEX),从随机寡核苷酸文库中选择适体。如本文所用,术语“SELEX”是指本领域中被称为“指数富集的配体系统进化技术(Systematic Evolution of Ligands by Exponential Enrichment,SELEX)”的方法。SELEX,或等同地,体外选择,是一种功能强大并广泛使用的方法,用于选择以特异性和亲和力与靶标(例如蛋白)结合的核酸序列(即适体)(Ellington AD等人,Nature,1990,346:818-822;Tuerk C等人,Science,1990,249:505-510;和Gold L等人,Anmi Rev Biochem,1995,64:763-797)。SELEX的过程和各种改动在本领域中已有描述,例如,美国专利号5,270,163;5,567,588;5,696,249;5,853,984;6,083,696;6,376190;6,262,774;6,569,620;6,706,482;6,730,482;6,933,116;8,975,388;8,975026;和9,382,533;其内容通过引用以其全文并入本文。SELEX方法基于以下独特理解:核酸具有足够的能力形成各种二维和三维结构,并且其单体具有足够的化学多功能性,可以作为几乎任何化学化合物(无论是单体还是聚合物)的配体(即形成特异性结合复合物)。任何大小或组成的分子都可以作为靶标。SELEX依赖于包含随机序列的单链寡核苷酸的大型文库作为起点。寡核苷酸可以是修饰的或未修饰的DNA、RNA或DNA/RNA杂交体。在一些实例中,文库包含100%随机或部分随机的寡核苷酸。
核酸适体显示出对其靶标稳健的结合亲和力,优选以小于10
适体可以包含天然存在的核苷酸和/或修饰的核苷酸,包括但不限于化学修饰的核碱基、非天然碱基(例如2-氨基嘌呤)、核苷酸类似物、添加标记(例如荧光团)、添加缀合物或以上任意几种的混合物。可以在保持功能方面(例如与靶标的结合)的前提下根据需要对适体的核酸序列进行修饰。
如本文所用,术语“核酸”、“寡核苷酸”和“多核苷酸”可互换使用,是指任何长度的核苷酸的聚合物,并且此类核苷酸可包括脱氧核糖核苷酸(DNA)、核糖核苷酸(RNA)和/或类似物或化学修饰的脱氧核糖核苷酸或核糖核苷酸以及RNA/DNA杂交体。术语“核酸”、“寡核苷酸”和“多核苷酸”包括双链或单链分子以及三螺旋分子。核酸分子可包含至少一个化学修饰。
如本文所用,术语核酸分子的“一级结构”是指其核苷酸序列。核酸分子的“二级结构”包括但不限于发夹环、凸环、内部环、多分支环、假结或其组合。“预选的二级结构”是指被选择并通过设计被工程化到适体中的那些二级结构。
如本文所用,术语“互补的”是指通过碱基配对例如A-T(U)和C-G配对的多核苷酸的天然结合。两个单链分子可以是部分互补的,使得仅有其中一些核酸结合,或者互补可以是“完全的”,以致单链分子之间存在完全的互补性。核酸链之间的互补程度对核酸链之间杂交的效率和强度具有重要影响。如本文所用,术语“杂交”或“与……杂交”是指多核苷酸链在限定的杂交条件下通过碱基配对与互补链退火的过程。特异性杂交表明了两个核酸序列具有高水平的同一性。特定的杂交复合物在允许的退火条件下形成。
如本文所用,术语“高亲和力”是指候选适体与靶标以小于100nM的结合解离常数K
如本文所用,术语“扩增”意指增加一个分子或一个类别的分子的量或拷贝数的任何过程或步骤组合。通常使用但不限于聚合酶链反应(PCR)(例如美国专利号4,683,195和4,683,202;其内容通过引用以其全文并入本文)来进行核酸分子的扩增。
如本文所用,术语“文库(library)”或“池(pool)”或“子集(subset)”是指一群化合物,例如单链DNA(ssDNA)分子。
如本文所用,术语“靶分子”,“靶标物”和“靶标”可互换使用,是指可以由适体结合的任何分子。“靶分子”或“靶标”可以是例如蛋白、多肽、核酸、碳水化合物、脂质、多糖、糖蛋白、激素、受体、抗原、抗体、亲和体(affybody)、抗体模拟物、病毒、病原体、有毒物质、底物、代谢物、过渡态类似物、辅因子、抑制物、药物、小分子、染料、营养物、污染物、生长因子、细胞、组织或微生物,以及上述任何物质的任何片段或部分。在一个实施方案中,靶标可以是过敏蛋白。
如本文所用,术语“反向靶标(counter target)”是指具有与靶标或靶标物相似的结构、相似的活性位点或相似的活性的家族中的分子。在本公开的上下文中,反向靶标可以是与针对感兴趣靶标的所选适体没有交叉特异性的任何分子。可以在反向选择过程中使用反向靶标来完善(refine)候选适体,以分离交叉识别其他紧密相关分子的序列。
如本文所用,术语“过敏原”意指在受试者中引起、引发或触发免疫反应的化合物、物质或组合物。因此,过敏原通常被称为抗原。过敏原通常是蛋白或多肽。
如本文所用,术语“多肽”、“肽”和“蛋白”在本文中互换使用,是指任何长度的氨基酸聚合物。
如本文所用,术语“样品”意指含有或认为含有一个或多个待测靶标的组合物。样品可以是但不限于获得自受试者(包括人和动物)的生物样品,获得自环境的样品(例如土壤样品、水样品、农业样品如植物和农作物样品),化学样品和食物样品。
根据本公开,对选择方法进行改进以鉴定候选适体,所述候选适体能够以高特异性和高亲和力识别靶分子并以较低的交叉反应性识别反向靶标,并且在靶标存在下不与和适体序列互补的寡核苷酸杂交。所选适体和衍生自这些适体的SPN可用作基于竞争的检测测定中的检测剂,在该测定中测试样品中的靶分子和互补寡核苷酸与适体(或SPN)竞争结合。
根据本公开,适体筛选方法可以包括(a)制备包含多个单链DNA(ssDNA)分子的输入DNA文库,每个单链DNA包含一个中央随机核酸序列,两侧是5'端的恒定序列和3'端的恒定序列,所述恒定的5'端和恒定的3'端充当引物;(b)从(a)的输入DNA文库中选择基本上与靶标物结合的ssDNA分子的池;(c)从获得自(b)的ssDNA分子靶标结合池中,选择在靶标物存在下不与互补序列杂交(即不同时与靶标和互补序列结合)的ssDNA分子的池;(d)从获得自(c)的正结合ssDNA分子池中,反向选择在靶标物不存在下不与互补序列杂交的ssDNA分子(称为非结合ssDNA分子),或基本上与反向靶标物结合(交叉特异性)的ssDNA分子;和(e)从(c)中的正结合ssDNA分子池中减去获得自(d)的ssDNA分子池,并鉴定与感兴趣的靶标特异性结合的候选ssDNA分子。
本发明的筛选方法联合了多个正靶标结合选择(positive target bindingselection)(例如,正SELEX和芯片上SELEX)、非结合反向选择(non-binding counterselection)、互补杂交选择(complementary hybridization selection)和反向靶标结合选择(counter target binding selection)。通过重复的正选择和负选择、序列扩增和测序分析来鉴定候选适体。与常规SELEX和本领域中其他已知方法相比,改进的筛选方法提高了适体选择效率,并且确保了选择相比于短互补核酸序列优选结合靶分子的适体。图1中的流程图展示了本发明的筛选方法的示例性实施方案,其用于鉴定对靶标特异并可用于基于竞争的检测测定法中的适体序列。
在一些实施方案中,可以通过正靶标结合选择过程来选择基本上与靶分子结合的ssDNA分子池,所述正靶标结合选择过程包括使用包含随机ssDNA(单链DNA)分子的输入文库和靶标物来重复核酸序列的靶标结合、分隔、分离和扩增。可以使用常规的适体选择过程,例如通过指数富集的配体系统进化技术(SELEX)、选择和扩增的结合位点(SAAB)、靶标的循环扩增和选择(CASTing)。作为非限制性实例,可通过使用包含随机单链DNA序列的输入ssDNA文库和靶标物进行数轮正氧化石墨烯(GO)-SELEX选择来鉴定形成ssDNA:靶标复合物的多个序列(图1)。
SELEX程序通常包括通过重复进行靶标分隔和扩增,从双链或单链核酸(DNA、RNA或DNA/RNA杂交体)的大型文库中逐步选择以高亲和力和高特异性与感兴趣的靶标结合的不同核酸序列。
每轮SELEX过程均由数个步骤组成,包括制备核酸文库,形成核酸-靶标复合物,分离结合的序列和未结合的序列,洗脱适体,PCR扩增以及鉴定对靶标特异的适体。每轮选择均富集了来自核酸文库的候选适体。
输入核酸文库可以包含多个具有随机序列的单链DNA(ssDNA)分子。ssDNA的长度可以是50至150个核苷酸,例如,文库中的ssDNA的长度约50至140个核苷酸,或长度约50至130个核苷酸,或长度约50至120个核苷酸,或长度约50至100个核苷酸,或长度约60至80个核苷酸,或长度约70至90个核苷酸,或长度约70至80个核苷酸。在一些实施方案中,文库中ssDNA的长度可以是60个核苷酸,或长度为61个核苷酸,或长度为62个核苷酸,或长度为63个核苷酸,或长度为64个核苷酸,或长度为65个核苷酸,或长度为66个核苷酸,或长度为67个核苷酸,或长度为68个核苷酸,或长度为69个核苷酸,或长度为70个核苷酸,或长度为71个核苷酸,或长度为72个核苷酸,或长度为73个核苷酸,或长度为74个核苷酸,或长度为75个核苷酸,或长度为76个核苷酸,或长度为77个核苷酸,或长度为78个核苷酸,或长度为79个核苷酸,或长度为80个核苷酸,或长度为81个核苷酸,或长度为82个核苷酸,或长度为83个核苷酸,或长度为84个核苷酸,或长度为85个核苷酸,或长度为86个核苷酸,或长度为87个核苷酸,或长度为88个核苷酸,或长度为89个核苷酸,或长度为90个核苷酸,或长度为91个核苷酸,或长度为92个核苷酸,或长度为93个核苷酸,或长度为94个核苷酸,或长度为95个核苷酸,或长度为96个核苷酸,或长度为97个核苷酸,或长度为98个核苷酸,或长度为99个核苷酸,或长度为100个核苷酸。文库中的每个ssDNA分子均在中心包含一个随机核酸序列,两侧是充当PCR引物的5'端的恒定序列和3'端的恒定序列,其中引物的序列是已知的,并且中央随机序列的长度可以是30至50个核苷酸。可以以多种方式产生随机序列,所述方式包括化学合成和从随机切割的细胞核酸中按大小进行选择。还可以在重复选择/扩增之前或期间通过诱变向测试核酸中引入或增加序列变异。
作为非限制性实例,可以通过在DNA合成仪上进行自动化化学合成来产生输入ssDNA分子文库。
如本文所用,ssDNA内的“中央随机核酸序列”也可以称为ssDNA的“内部序列(inner sequence)”。
在一个优选的实施方案中,输入文库中的ssDNA分子的长度为76个核苷酸,其中长度为30个核苷酸的中央随机核酸序列在每个ssDNA的5'端和3'端侧接两个长度为23个核苷酸的引物。作为非限制性实例,5'端引物可包含5’TAGGGAAGAGAAGGACATATGAT 3’(SEQ IDNO.1)的核酸序列,且3'端引物可包含5’TTGACTAGTACATGACCACTTGA 3’(SEQ ID NO.2)的核酸序列。
如本文所用,术语“引物”是指短核酸,当被置于诱导合成与核酸链互补的引物延伸产物的条件下(即,在核苷酸和诱导剂如DNA聚合酶的存在下,并在合适的温度和pH下),其能够作为合成(例如PCR)的启动点。为使扩增效率最大化,引物优选是单链的,但也可以是双链的。
可以将输入DNA文库与靶标混合,其中在靶标与文库中存在的多个ssDNA分子之间形成复合物。靶标可以是任何分子(例如核酸、蛋白、小分子、糖类、毒素、生物标志物、细胞和病原体)。在一些实施方案中,靶标是蛋白,例如过敏原蛋白或过敏原的混合过敏原组分。过敏原可以包括但不限于食物过敏原,来自环境的过敏原(例如植物、动物、微生物、空气或水)以及医学过敏原(即,在医学或医疗设备中发现的任何过敏原)。
食物过敏原包括但不限于以下之中的蛋白:豆类(legume)例如花生、豌豆(peas)、扁豆(lentil)和菜豆(bean),以及豆类相关植物羽扇豆(lupin),树坚果(tree nut)例如杏仁、腰果、核桃、巴西坚果、榛果(filbert)/榛子(hazelnut)、山核桃、开心果、核桃、山毛榉坚果(beechnut)、灰胡桃(butternut)、栗子(chestnut)、毛枝栗(chinquapin nut)、椰子(coconut)、银杏果(ginkgo nut)、荔枝(lychee nut)、澳洲坚果(macadamia nut)、爪哇橄榄果(nangai nut)和松果(pine nut);蛋(egg);鱼(fish);贝壳类动物(shellfish),例如蟹(crab)、小龙虾(crawfish)、龙虾(lobster)、虾(shrimp)和对虾(prawn);软体动物(mollusk),例如蛤蜊(clam)、牡蛎(oyster)、贻贝(mussel)和扇贝(scallop);奶(milk);豆(soy);小麦(wheat);谷蛋白(gluten);玉米(corn);肉(meat),例如牛肉(beef)、猪肉(pork)、羔羊肉(lamb)、羊肉(mutton)和鸡肉(chicken);明胶(gelatin);亚硫酸盐(sulphite);种子(seed),例如芝麻(sesame)、向日葵(sunflower)和罂粟籽(poppy seed);和香料(spice),诸如芫荽(coriander)、大蒜(garlic)和芥末(mustard);水果(fruit);蔬菜(vegetable),例如芹菜(celery);和稻米(rice)。来自食物过敏原的一些示例性过敏蛋白可以包括鳕鱼(codfish)中的小白蛋白(parvalbumin),甲壳类动物(crustacean)中的原肌球蛋白(tropomyosin),精氨酸激酶(arginine kinase)和肌球蛋白轻链(myosin lightchain),酪蛋白(casein),牛奶中的α-乳白蛋白(α-lactalbumin)和β乳球蛋白(βlactoglobulin),以及球蛋白或豌豆球蛋白种子贮藏蛋白(vicilin seed storageprotein)。
其他靶分子包括但不限于来自样品中的病原微生物的病原体,例如细菌、酵母、真菌、孢子、病毒和朊病毒;疾病蛋白(例如,用于疾病诊断和预后的生物标志物);残留在环境中的杀虫剂和肥料;和毒素。靶标可以包括非蛋白化合物,例如矿物质和小分子(例如抗生素)。
在一些实施方案中,用于选择基本上与靶标物结合的ssDNA分子的富集池的步骤可包括(i)使输入ssDNA文库与靶标物接触,其中在靶标和输入文库中存在的多个ssDNA分子之间形成复合物;(ii)将步骤(i)中形成的复合物与未结合的ssDNA分子分隔,并分离复合物中的ssDNA分子以产生针对靶标物的ssDNA分子的子集,并扩增分离的ssDNA分子的子集;(iii)使来自步骤(ii)的ssDNA分子的富集子集与相同的靶标物接触,其中在靶标和富集文库中存在的第二群ssDNA分子之间形成复合物,以产生ssDNA分子的第二富集子集组;和(iv)任选地根据需要将结合、分隔、分离和扩增的步骤(步骤(i)至(iii))重复一轮、两轮、三轮、四轮或更多轮,以产生对靶标具有高特异性和高亲和力的ssDNA分子,从而产生基本上与靶标物结合的ssDNA分子的富集池(例如,表1中的池1)。
在一个实施方案中,进行由常规SELEX方法改进的氧化石墨烯(GO)-SELEX过程以选择靶标结合池。如本文所用,术语“石墨烯”、“氧化石墨烯(GO)”、“氧化石墨烯纳米片”和“石墨烯纳米片”意指二维碳结构,并且在整个本说明书中可互换使用。ssDNA分子中暴露的核碱基可被吸附到氧化石墨烯表面(Chen等人,J.Agric.Food Chem.2014;62,10368-10374)。因此,当将氧化石墨烯(GO)溶液添加到ssDN文库和靶标物的混合物中时,GO可以将未与特定靶标结合的ssDNA序列吸附到其表面,并使与靶标结合的序列自由(free)。然后可以通过例如离心除去未结合的序列和GO,而与特定靶标结合的ssDNA分子不被吸附到GO表面,然后经过回收且用于随后的选择过程。这个过程可以不需要像在常规SELEX中所用那样固定靶标物。
GO-SELEX过程便宜、快速且简单。在常规SELEX中,使用了许多昂贵、效率较低且耗时的方法,例如色谱法、亲和柱等,以将结合靶标物的核酸分子与不结合靶标物的核酸分子分离。GO-SELEX过程的特征在于,即使靶标物或反向靶标物没有被特异性地固定在特定载体上,也可以通过离心简单地将结合的ssDNA分子与未结合的ssDNA分子分离开(Nguyen等人,Chem.Commun.2014,50,10513-10516;其内容通过引用以其全文并入本文。)
在除去GO吸附的ssDNA分子(例如通过离心)后,可以从收集的DNA:靶标复合物中除去靶标物。可以使用在本领域熟知的溶液沉淀蛋白的方法,例如乙醇沉淀和strataclean树脂。作为非限制性实例,可将strataclean树脂添加到离心后回收的上清液中。可以通过离心除去结合到Strataclean树脂上的靶标物。所述靶标除去步骤可以重复两次、三次、四次或更多次。含有富集的与靶标物结合的ssDNA分子的上清液可用于下一轮的靶标结合选择(图1)。
在一些实施方案中,可以测量与靶标物结合的ssDNA分子的最终浓度,并将其与输入ssDNA文库的初始浓度进行比较。浓度比率将用于确定是否需要进行下一轮GO-SELEX选择。如果比率低于50%,则在相同条件下进行下一轮正GO-SELEX过程。重复相同的过程,直到与靶标物结合的ssDNA分子的回收达到令人满意的比率,例如回收率高于50%。重复进行分隔和分离,直到达到期望的目标为止,例如在相同条件下进行两次、三次、四次、五次、六次、七次、八次或更多次。在最通常的情况下,继续进行选择直到在重复选择中结合强度没有显著提高。
通过使用标记的引物,例如生物素化的反向引物和标记有荧光团的正向引物进行PCR,可以进一步扩增选自正GO-SELEX过程的ssDNA分子靶标结合池。在其他方面,可以通过任何其他已知方法扩增ssDNA分子,例如对所选序列进行测序,并使用寡核苷酸合成仪人工合成它们,以进行下一轮结合和选择。
与正向引物结合的荧光团可以是但不限于Cy5、Alexa Fluor 350、Alexa Fluor430、Alexa Fluor 488、Alexa Fluor 532、Alexa Fluor 546、Alexa Fluor 594、AlexaFluor 647、Alexa Fluor 658、Cyanine-3、Cyanine-5、荧光素、德克萨斯红、FITC(异硫氰酸荧光素)、若丹明等。在一个优选的实施方案中,正向引物结合有Cy5。在另一个实施方案中,正向引物结合有Alexa Fluor 647。
PCR扩增后,可以对所得的双链DNA(dsDNA)分子进行清洁(cleaned),并使其进一步变性以再生单链DNA(ssDNA)分子。生物素化的反向引物可以去除互补链,以从在PCR扩增过程中产生的dsDNA分子中再生ssDNA分子。作为非限制性实例,可将链霉亲和素包被的磁珠添加到PCR产物中。生物素化的互补链与链霉亲和素包被的磁珠结合。ssDNA分子变性(例如添加碱)后,使用磁力将结合的生物素化的互补链分离并去除。收集具有荧光标记的目标ssDNA分子。基本上与靶标结合的标记有荧光团(例如Cy5和Alexa Fluor 647)的ssDNA分子用于下一轮选择过程。
标记有荧光团(例如Cy5)的ssDNA分子在用于基于竞争的检测测定中的显色检测剂时具有若干优点。在适体选择开始时向ssDNA序列中添加Cy5或其他荧光标记,可以确保当所有候选适体作为用于检测测定中的信号传导多核苷酸(SPN)而进一步显色时,它们均具有合适的二级和三级结构。在后期添加Cy5或其他荧光标记可能会影响候选适体的二级和三级结构。这个改进能显著减少选择期间的错误命中。
作为非限制性实例,鉴定基本上与靶标结合的序列池的GO-SELEX过程可包括以下步骤:(i)将输入ssDNA文库(例如表1中的池0)与过敏原组合物在缓冲溶液中混合;并且在常温下诱导它们彼此结合;(ii)向步骤(i)的混合物中添加氧化石墨烯溶液,以除去未与靶标结合的ssDNA分子;(iii)从收集的ssDNA分子除去靶标,并使用ssDNA分子末端的PCR引物通过PCR扩增ssDNA分子;和(iv)使双链PCT产物变性并收集标记有荧光团的ssDNA分子。任选地,可以将正GO-SELEX选择重复2轮、3轮、4轮、5轮、6轮、7轮、8轮、9轮、10轮或更多轮。输入文库中的ssDNA分子包含大约76个核苷酸长度,包括在每个末端用于PCR扩增的引物和在其中央(即适体的内部序列)的约30个核苷酸(结合位点)。靶标物是过敏原物质,尤其是食物过敏原,其包含一种过敏组分,或来自单一过敏原的过敏组分的混合物。食物过敏原可以包括但不限于以下之中的蛋白:豆类例如花生、豌豆、扁豆和菜豆,树坚果(例如杏仁、巴西坚果、腰果、榛子、山核桃、开心果和核桃),小麦,奶,鱼,蛋清和海鲜。
在一些实施方案中,5’恒定序列(即5’引物)包含SEQ ID NO.1的核酸序列,且3’恒定序列(即3’引物)包含SEQ ID NO.2的核酸序列。
可以将ssDNA分子靶标结合池(例如表1中的池1)进一步分隔以选择基本上与靶标结合并与具有互补于ssDNA分子的序列的短寡核苷酸竞争的序列子集。在一些实施方案中,可以使用包被有包含与ssDNA分子互补的序列的短寡核苷酸的固体支持物(例如玻璃或塑料芯片),来进行这种正靶标结合选择。通过这个过程,可以从池中减去可以同时结合靶分子及其互补序列的核酸序列家族。定制这个额外的正选择过程,以区分与靶标结合的ssDNA分子和与结合在固体支持物(例如玻璃或塑料芯片)上的互补序列结合的ssDNA分子。
短寡核苷酸锚可以包含与ssDNA分子末端的恒定序列互补的序列。寡核苷酸锚可以包含与适体的5’末端或3’末端序列互补的序列。互补序列含有约5至25个核苷酸,或5至18个核苷酸,或6至20个核苷酸,或8至20个核苷酸。例如,它可以包含5个核苷酸、6个核苷酸、7个核苷酸、8个核苷酸、9个核苷酸、10个核苷酸、11个核苷酸、12个核苷酸、13个核苷酸、14个核苷酸、15个核苷酸、16个核苷酸、17个核苷酸、18个核苷酸、19个核苷酸、20个核苷酸、21个核苷酸、22个核苷酸、23个核苷酸、24个核苷酸或25个核苷酸。在一个优选实例中,互补锚定序列含有5至15个核苷酸。寡核苷酸锚可以与ssDNA的序列100%、或99%、或98%、或97%、或96%、或95%、或94%、或93%、或92%、或91%、或90%互补。短互补序列直接或通过接头共价连接至固体支持物,例如玻璃芯片。
其上共价结合寡核苷酸的固体支持物可以包括但不限于玻璃、聚合物支持物(例如,参见美国专利号5,919,525)、聚丙烯酰胺凝胶或塑料(例如微孔板)或尼龙膜。玻璃可以是聚合物玻璃(例如,丙烯酸玻璃、聚碳酸酯和聚对苯二甲酸乙二醇酯),或硅酸盐玻璃(例如,派热克斯(Pyrex)玻璃,石英和氧化锗玻璃),或多孔玻璃等。聚合物可以包括但不限于聚酰亚胺、光致抗蚀剂、SU-8负性光致抗蚀剂、聚二甲基硅氧烷(PDMS)、有机硅弹性体PDMS和COC。在一个优选的实施方案中,固体支持物是玻璃芯片。
可以使用不同的技术使短互补序列结合到固体支持物的确定位置。这些方法在相关领域中是公知的。例如,可以通过喷墨、压电或其他类似方法将寡核苷酸以微滴形式沉积在固体支持物的特定位置上。可以对固体支持物进行预处理来为寡核苷酸提供活性结合表面。另外,可以在固体支持物上对所结合的寡核苷酸的密度进行测量和控制。
在一个实施方案中,芯片上靶标结合选择可以包括以下步骤:(i)将ssDNA分子靶标结合池(即池1)与相同的靶标物在缓冲溶液中混合;并且在常温下诱导它们彼此结合;(ii)使步骤(i)的混合物与固体支持物接触,所述固体支持物的表面共价包被有包含与ssDNA分子的序列互补的序列的短寡核苷酸;(iii)收集未与固体支持物结合的ssDNA:靶标复合物(例如流过物)(图1);和(iv)从收集的ssDNA:靶标复合物中除去靶标物,并收集ssDNA分子的富集子集。任选地,使(iii)中收集的混合物再次与包被有互补寡核苷酸的固体支持物接触两次、三次、四次、五次、六次、七次、八次或更多次,并对最终孵育后的流过物进行处理以从复合物中回收ssDNA分子。
通过这个选择,池1中未与靶标物结合的ssDNA分子的子集将与共价结合至固体支持物的互补序列杂交,并从池中被除去。另外,与靶标物结合的ssDNA分子的子集也可与结合至固体支持物的互补序列杂交。这些ssDNA分子留在固体支持物上,并从收集的ssDNA池中被减去。
如本文所述,将收集自最终孵育的ssDNA分子清洗,并与靶标物分离。与正GO-SELEX选择类似,测量回收的ssDNA分子的浓度,并将其与输入池(池1)进行比较。在一些实施方案中,可以将芯片上正选择重复两次、三次、四次、五次、六次、七次、八次或更多次,直到ssDNA分子的回收率达到期望的回收率(例如,大于输入池的50%)。
然后通过进行PCR来扩增ssDNA分子,并如正GO-SELEX过程中所述地回收单链DNA分子。通过这个选择过程,选择了与靶标物结合但在靶标物存在下不与互补序列杂交的ssDNA分子的池(即表1中的正结合池(池2))。这个选择过程模拟了基于竞争的检测测定中所用的条件。常规SELEX(例如GO-SELEX)和芯片上正靶标结合过程的联合增加了适体的特异性和亲和力。
可以对含有在互补序列存在下与靶标物结合的候选适体的ssDNA分子正池(池2)作进一步筛选,以分离即使在序列自由时亦不与互补序列结合的ssDNA分子,以及除感兴趣的靶标外还基本上与反向靶标物结合的序列。在一些实施方案中,可使用预先包被有包含与ssDNA分子互补的序列的短寡核苷酸的固体支持物(例如玻璃芯片),通过反向选择过程来分离这些非特异性ssDNA序列。
在一些实施方案中,进行芯片上非结合反向选择以鉴定池中即使在自由时亦不与其互补序列杂交的ssDNA分子。在未添加靶标物下将包含ssDNA分子正池(池2)的DNA溶液直接与预先包被有包含与池中适体互补的序列的短寡核苷酸的固体支持物(例如玻璃芯片)一起孵育。孵育后,收集包含未结合的ssDNA分子的DNA溶液(即流过物)(图1)。将流过的DNA溶液再次与预先包被有短互补寡核苷酸的固体支持物(例如玻璃芯片)一起孵育,并收集第二流过物。可以将孵育步骤重复两次、三次、四次、五次、六次、七次、八次或更多次,优选八次。
在一个优选的实施方案中,芯片上非结合反向过程可以包括以下步骤:(i)制备包含ssDNA分子正结合池(池2)的DNA溶液;(ii)使所述DNA溶液与包被有包含与ssDNA分子互补的序列的短寡核苷酸的固体支持物接触;(iii)在孵育后收集ssDNA溶液;和(iv)使收集的溶液再次与新的包被有互补序列的固体支持物接触。可以将这些步骤重复两轮、三轮、四轮、五轮、六轮、七轮或八轮,并清洗从最终孵育中收集的ssDNA溶液,并将其扩增以进行测序。在一个实施方案中,将这些步骤重复八轮,清洁从最终孵育中收集的ssDNA溶液,并将其扩增以进行测序。
这个芯片上非结合反向选择产生了ssDNA分子的非结合池(即表1中的池3),其包含即使在靶标物不存在下也不能与互补序列杂交的ssDNA分子。
在其他实施方案中,进行芯片上反向结合选择(on-chip counter bindingselection)以从ssDNA分子正结合池(表1中的池2)中分离出能与非特异性反向靶标结合的任何序列。这个反向选择过程通过消除具有与一个或多个非靶标分子(例如反向靶标)的交叉反应性的核酸序列而提高了所选ssDNA分子的靶标特异性。
在一个优选的实施方案中,芯片上反向选择过程可以包括以下步骤:(i)制备包含ssDNA分子正结合池(池2)的ssDNA溶液,并将所述ssDNA溶液与反向靶标或反向靶标的混合物一起孵育;(ii)使步骤(i)的混合物与包被有包含与所述ssDNA分子互补的序列的短寡核苷酸的固体支持物接触;(iii)在步骤(ii)之后收集ssDNA/反向靶标复合物(即流过物)(图1);和(iv)使步骤(iii)中收集的溶液与包被有互补寡核苷酸的固体支持物接触。可以将孵育和收集步骤重复两轮、三轮、四轮、五轮、六轮、七轮、八轮或更多轮,优选八轮。将在最终孵育步骤后收集的溶液进行清洗和/或扩增以进行测序。
在一些实施方案中,可以根据反向靶标所需的数量,重复进行芯片上反向结合选择,每次都从来自正结合池(即表1中的池2)的同一ssDNA分子池开始。在一些替代的实施方案中,可以在同一轮中同时运行多个反向靶标。
通过这个芯片上反向选择过程,从正结合池中除去了对不期望的相关蛋白(反向靶标物)具有交叉特异性的ssDNA序列。这个反向选择过程产生了包含能与一个或多个反向靶标交叉反应的ssDNA分子的ssDNA分子池(即表1中的池4)。
作为非限制性实例,反向靶标可以是同一家族中的过敏原蛋白,包括可归到这些结构相关过敏原家族中的来自不同来源的过敏原蛋白,例如包括种子贮藏蛋白的醇溶谷蛋白家族(例如,黑麦中的Sec c 20;小麦中的Tri a 19和小麦中的Tri a 36),非特异性脂质转移蛋白家族(例如,猕猴桃中的Act d 10,芹菜中的Api g 2,花生中的Ara h 9,栗子中的Cas s 8,榛子中的Cor a 8,核桃中的Jug r 3,西红柿中的Lyc e 3,香蕉中的Mus a 3和杏仁中的Pru du 3),包括种子贮藏蛋白的2S白蛋白家族(例如,腰果中的Ana o 3,花生中的Ara h 2,巴西坚果中的Ber e 1,荞麦(buckwheat)中的Fag e 2,大豆中的Gly m 8,核桃中的Jug r 1,芝麻中的Ses i 1和芥末中的Sin a 1),包括病程相关蛋白的Bet V1家族(例如,Api g 1/芹菜,Ara h 8/花生,Cor a 1/榛子,Dau c 1/胡萝卜,Gly m 4/大豆,Mal d1/苹果和Pru p 1/桃),7S(豌豆球蛋白样)球蛋白家族(例如,Ana o 1/腰果;Ara h 1/花生;Gly m 5/大豆;Jug r 2/核桃;Pis v 3/开心果),11S(豆球蛋白样)球蛋白家族(例如,Ana o 2/腰果;Ara h 3/花生;Ber e 2/巴西坚果;Cor a 9/榛子;Gly m 6/大豆;Jug r 4/核桃;Pru du 6/杏仁),半胱氨酸蛋白酶C1家族(例如Act d 1/猕猴桃;Gly m Bd 30K/大豆),包括肌动蛋白结合蛋白的组装抑制蛋白(Profilin)家族(例如Act d 9/猕猴桃;Api g4/芹菜;Ara h 5/花生;Cuc m 2/甜瓜;Dau c 4/胡萝卜;Gly m 3/大豆;Lyc e 1/西红柿;Mus a 1/香蕉;Ory s 12/稻米;Pru av 4/樱桃;Pru du 4/杏仁;Pru p 4/桃和Tri a 12/小麦),包括肌肉中的肌动蛋白结合蛋白的原肌球蛋白家族(例如Pen m 1/虾),包括肌肉蛋白的小白蛋白家族(例如Cyp c 1/鲤鱼(carp);Gad c 1/鳕鱼(cod);Ran e 2/青蛙;Sal s1/鲑鱼(salmon);Seb m 1/红鱼(redfish);Xip g 1/箭鱼(swordfish)),包括哺乳动物奶蛋白(milk proteins)的酪蛋白家族(例如Bos d 8-Bos d 12/牛奶),包括来自奶和母鸡卵清的富含硫的离子结合糖蛋白的转铁蛋白家族(例如Bos d乳铁蛋白/牛奶;Gal d 3/鸡蛋),包括丝氨酸蛋白酶抑制剂的serpin家族(例如Gal d 2/鸡蛋),包括三磷酸腺苷:胍基磷酸转移酶的精氨酸激酶家族(例如Pen m 2/虾),包括载体蛋白的脂质运载蛋白家族(例如Bos d 5/牛奶),包括Kazal抑制剂的卵类粘蛋白家族(例如,Gal d 1/鸡蛋),溶菌酶家族(例如,Bos d 4/牛奶;Gal d 4/鸡蛋),和包括血清白蛋白的白蛋白家族(例如,Bos d 6/牛奶;Gal d 5/鸡蛋)。
深度测序(Deep Sequencing)
根据本发明的筛选方法,可以将来自每个选择的ssDNA池(例如,表1中的池1,池2,池3和池4)进行清洗、扩增和测序。在一个实施方案中,所述方法包括使用聚合酶链反应(PCR)扩增单个ssDNA分子。使用深度测序鉴定每个池中的序列。在一些实施方案中,将靶标结合池(即池1)中的ssDNA分子进行扩增并测序。与此同时,可以通过扩增输入ssDNA文库来制备人工文库(artifact library),并且对这个人工文库中的序列进行测序(参见例如图1的流程图)。可以通过将PCR扩增和链分离步骤重复进行与正GO-SELEX选择相同的轮数来制备人工文库(图1)。将这些产生自PCR的过度扩增的序列从靶标结合池(表1中的池2)中除去。
可以对来自最后一轮芯片上靶标结合选择的正结合池(例如,表1中的池2)中的ssDNA分子进行测序。这个池中的ssDNA分子含有在其互补序列存在下优选与其靶标结合的ssDNA序列。
可以对来自最后一轮芯片上非结合反向选择以及来自最后一轮芯片上反向选择的非特异性ssDNA分子(例如,表1中的池3和4)进行测序。这些池中的ssDNA分子含有即使在不存在靶标物和具有与其他反向靶标的交叉特异性的序列的情况下也无法与互补序列杂交的ssDNA序列。
可以将每个池中的ssDNA序列进行条形码化,以用于鉴定。在条形码化后,可以将每个池中的ssDNA分子合并在一起,并在Illumina MiSeq系统上的单泳道中进行深度测序。
表1:选择轮数的综述
在对每个池中的ssDNA序列进行测序和条形码化并运行深度测序后,使用任何可用的生物信息学工具对数据进行分析。在一些实施方案中,通过使用开放来源的生物信息学工具Galaxy的局部再现(Thiel和Giangrande,Methods 2016,97,3-10;其内容通过引用以其全文并入本文),为每个单独的池生成热图,其代表每个池中ssDNA序列的频率。
通过使用每个池中序列的热图分析每个池中的过表达序列,来选择潜在的适体命中(aptamer hit)。本质上,从正结合池(池2)的ssDNA分子的热图中减去非结合池(池3)和反向结合池(池4)的ssDNA分子的热图。最终数据代表了潜在适体命中池,其特征包括:(i)以高特异性和高亲和力与靶蛋白结合,(ii)仅在靶标不存在下与其短互补序列杂交,但在靶标存在下不与短互补序列结合;和(iii)不具有对非特异性反向靶标的交叉反应性。候选适体的这些特征使它们适用于检测样品中的靶标,例如在基于竞争的测定中。
从最终的潜在适体命中池(例如表1中的池5)中,可以构建序列家族树以显示不同适体序列之间的相似性。可以使用预期的测定条件选择并折叠来自家族树结构的各个分支的多个序列。将对二级和三级结构进行评估,且选择显示出多个明确定义的结构的那些序列,以进行合成和进一步评估。这些结构或基序可以包括发夹环、对称和不对称的凸起、假结以及它们的种种组合。测量所选适体的平衡解离常数(K
根据本公开,所述选择方法可进一步包括以下步骤:(i)扩增第一、第二、第三和第四子池中的所有序列,并对每个池中的每个序列进行条形码化;(ii)将每个子池中的序列合并到一起,并一起进行测序;(iii)分析来自(ii)的数据,并根据条形码信息将每个序列数据分到原始子池(original sub-pool)中;(iv)为每个单独的子池生成热图,其代表了池中每个序列的频率;和(v)从第二子池的热图中减去第三子池和第四子池的热图中的序列,其中最终的序列池是候选适体,所述候选适体与感兴趣的靶标特异性结合,并在与各种短互补序列的结合的竞争中优选与感兴趣的靶标结合。
根据本公开,选择与以下特异性结合的序列:花生、树坚果(包括杏仁、巴西坚果、腰果、榛子、山核桃、开心果和核桃)、麸质、奶过敏原(乳清和酪蛋白)。还选择了与所有坚果结合的适体序列。如本文所用,术语“所有坚果”是指花生和树坚果(包括杏仁、巴西坚果、腰果、榛子、山核桃、开心果和核桃)。对“所有坚果”特异的所选适体序列可以与任何坚果(即花生、杏仁、巴西坚果、腰果、榛子、山核桃、开心果和核桃)结合,例如样品中存在的一种、两种、三种、四种、五种、六种、七种或八种坚果。
在一些实施方案中,与花生特异性结合的序列包含选自由SEQ ID NO.3至1002的核酸序列组成的组的内部序列。
在一些实施方案中,与杏仁特异性结合的序列包含选自由SEQ ID NO.4003至5002的核酸序列组成的组的内部序列。
在一些实施方案中,与巴西坚果特异性结合的序列包含选自由SEQ ID NO.8003至9002的核酸序列组成的组的内部序列。
在一些实施方案中,与腰果特异性结合的序列包含选自由SEQ ID NO.12003至13002的核酸序列组成的组的内部序列。
在一些实施方案中,与榛子特异性结合的序列包含选自由SEQ ID NO.16003至17002的核酸序列组成的组的内部序列。
在一些实施方案中,与山核桃特异性结合的序列包含选自由SEQ ID NO.20003至21002的核酸序列组成的组的内部序列。
在一些实施方案中,与开心果特异性结合的序列包含选自由SEQ ID NO.24003至25002的核酸序列组成的组的内部序列。
在一些实施方案中,与核桃特异性结合的序列包含选自由SEQ ID NO.28003至29002的核酸序列组成的组的内部序列。
在一些实施方案中,与所有坚果特异性结合的序列包含选自由SEQ ID NO.32003至33002的核酸序列组成的组的内部序列。
在一些实施方案中,与麸质特异性结合的序列包含选自由SEQ ID NO.40003至41002的核酸序列组成的组的内部序列。
在一些实施方案中,与乳清特异性结合的序列包含选自由SEQ ID NO.44003至45002的核酸序列组成的组的内部序列。
在一些实施方案中,与酪蛋白特异性结合的序列包含选自由SEQ ID NO.48003至49002的核酸序列组成的组的内部序列。
在一些实施方案中,可以改进本发明的选择方法以鉴定与多个靶标结合的适体序列。多靶标选择过程(multiple target selection process)提供了一种鉴定对于一组靶标的最佳结合适体的有效方法。
许多过敏原,尤其是食物过敏原,由多种过敏组分组成。这些组分可以在人体内诱发组分特异性IgE。一些人仅对一种特定组分过敏,而不对相同过敏原中其他组分过敏。一些人对过敏原的所有组分均过敏。例如,奶包括两个主要的过敏组分:乳清蛋白(α-乳白蛋白和β-乳球蛋白)和酪蛋白。对奶过敏的人,可以仅对乳清或酪蛋白过敏,或对乳清和酪蛋白均过敏。在这种情况下,仅与乳清蛋白结合或仅与酪蛋白结合,或与乳清蛋白和酪蛋白均结合的适体配体对于奶过敏来说是期望的。
在一些实施方案中,可以改进本发明的筛选方法以选择可以与过敏原的多个组分结合的适体。
作为非限制性实例,使用完整的过敏原物质作为靶标物(例如,包括酪蛋白和乳清蛋白的完整的奶)进行两轮、三轮、四轮或更多轮正GO-SELEX选择。从这个过程中选出的ssDNA分子(池1)包括与任意的奶组分(例如酪蛋白和乳清蛋白)结合的ssDNA序列的混合物。这些与奶结合的序列用于运行两个平行的选择过程:仅针对酪蛋白的选择过程和仅针对乳清蛋白的选择过程。如先前针对单靶标所述执行这两个选择过程。重要的是,在反向选择过程中,仅使用另一组分作为反向靶标来执行分离选择。也就是说,在选择与酪蛋白特异性结合的适体序列时,在反向选择过程中使用乳清蛋白作为反向靶标,而使用酪蛋白作为反向靶标用于选择与乳清蛋白特异性结合的适体序列。
按照类似的程序,对ssDNA分子的各个子池进行条形码化,并进行深度测序。酪蛋白和乳清样品将分别进行合并,并在深度测序过程中在不同的泳道中运行。测序数据的生物信息学分析将揭示仅针对靶标的适体命中池。为了找到与酪蛋白和乳清均结合的适体序列,可以收集特定反向轮次(special counter round)之间的重叠。
在另一个实例中,所有坚果的混合物可以用作靶标物,可以通过本方法来选择可以识别所有坚果的序列。所选序列可以与测试样品中存在的任何坚果以及任何坚果的组合结合。
在本公开的另一个方面,提供了与过敏原靶标特异性结合的适体,衍生自所选适体的信号传导多核苷酸(SPN),以及包含这些适体和SPN的检测传感器。以高特异性和高亲和力与靶标过敏原结合的适体可以在靶标过敏原存在下不与短互补序列杂交,并且对任何反向靶标都没有或几乎没有交叉特异性。
SPN可以衍生自通过本方法选择的适体序列。SPN可在适体序列的一端或两端进一步包含额外的核苷酸。可以进一步修饰序列,以改变其二级和/或三级结构使其更稳定,增加结合亲和力和/或特异性,或添加荧光标记,或经修饰以包含一个或多个缀合物。
提供了包含所选适体和SPN的检测传感器。在一些实施方案中,检测传感器可以包含SPN、固体支持物和包含与SPN互补的核酸序列的短寡核苷酸,其中所述寡核苷酸通过末端之一直接地或通过接头(例如6个碳原子的臂)共价锚定到固体支持物上。SPN包含与感兴趣的靶标特异性结合的内部序列,并且其在不与感兴趣的靶标结合时与互补寡核苷酸杂交。在一个实例中,短互补序列和感兴趣的靶标将竞争结合SPN。在这个竞争性测定中,例如,SPN可以与结合在固体支持物上的短互补序列结合,或与样品中的感兴趣的靶标结合。在足以使样品中感兴趣的靶标与结合在固体支持物上的短互补序列竞争的条件下,可以检测和测量SPN:靶标复合物。
根据本公开,选择与以下特异性结合的适体序列:花生,树坚果(包括杏仁、巴西坚果、腰果、榛子、山核桃、开心果和核桃),麸质,奶过敏原(乳清和酪蛋白)。还选择了与所有坚果结合的适体序列。
在一些实施方案中,与花生特异性结合的序列包含选自由SEQ ID NO.3至1002的核酸序列组成的组的内部序列。在一些实例中,可以将短核酸序列附接在内部序列的5-末端。短核酸序列可以是用于随机ssDNA文库中的5'引物序列,即SEQ ID NO.1的核酸序列。因此,与花生特异性结合的适体可以包含选自由SEQ ID NO.1003至2002组成的组的核酸序列。在其他实例中,可以将短核酸序列附接在内部序列的3-末端。短核酸序列可以是用于随机ssDNA文库中的3'引物序列,即SEQ ID NO.2的核酸序列。因此,与花生特异性结合的适体可以包含选自由SEQ ID NO.2003至3002组成的组的核酸序列。在其他实例中,内部序列可以包含5’末端短序列(即SEQ ID NO.1)和3’末端短序列(即SEQ ID NO.2)。因此,与花生特异性结合的适体可以包含选自由SEQ ID NO.3003至4002组成的组的核酸序列。在一个实施方案中,与花生特异性结合的本公开的适体可以包含选自由SEQ ID NO.3至4002(列于表2中)组成的组的核酸序列或其变体。
表2:针对花生的适体序列
在一些实施方案中,与杏仁特异性结合的序列包含选自由SEQ ID NO.4003至5002的核酸序列组成的组的内部序列。在一些实例中,可以将短核酸序列附接在内部序列的5-末端。短核酸序列可以是用于随机ssDNA文库中的5’引物序列,即SEQ ID NO.1的核酸序列。因此,与杏仁特异性结合的适体可以包含选自由SEQ ID NO.5003至6002组成的组的核酸序列。在其他实例中,可以将短核酸序列附接在内部序列的3-末端。短核酸序列可以是用于随机ssDNA文库中的3’引物序列,即SEQ ID NO.2的核酸序列。因此,与杏仁特异性结合的适体可以包含选自由SEQ ID NO.6003至7002组成的组的核酸序列。在其他实例中,内部序列可以包含5'末端短序列(即SEQ ID NO.1)和3'末端短序列(即SEQ ID NO.2)。因此,与杏仁特异性结合的适体可以包含选自由SEQ ID NO.7003至8002组成的组的核酸序列。在一个实施方案中,与杏仁特异性结合的本公开的适体可以包含选自由SEQ ID NO.4003至8002(列于表3中)组成的组的核酸序列或其变体。
表3:针对杏仁的适体序列
在一些实施方案中,与巴西坚果特异性结合的序列包含选自由SEQ ID NO.8003至9002的核酸序列组成的组的内部序列。在一些实例中,可以将短核酸序列附接在内部序列的5-末端。短核酸序列可以是用于随机ssDNA文库中的5’引物序列,即SEQ ID NO.1的核酸序列。因此,与巴西坚果特异性结合的适体可以包含选自由SEQ ID NO.9003至10002组成的组的核酸序列。在其他实例中,可以将短核酸序列附接在内部序列的3-末端。短核酸序列可以是用于随机ssDNA文库中的3’引物序列,即SEQ ID NO.2的核酸序列。因此,与巴西坚果特异性结合的适体可以包含选自由SEQ ID NO.10003至11002组成的组的核酸序列。在其他实例中,内部序列可以包含5'末端短序列(即SEQ ID NO.1)和3'末端短序列(即SEQ IDNO.2)。因此,与巴西坚果特异性结合的适体可以包含选自由SEQ ID NO.11003至12002组成的组的核酸序列。在一个实施方案中,与巴西坚果特异性结合的本公开的适体可以包含选自由SEQ ID NO.8003至12002(列于表4中)组成的组的核酸序列或其变体。
表4:针对巴西坚果的适体序列
在一些实施方案中,与腰果特异性结合的序列包含选自由SEQ ID NO.12003至13002的核酸序列组成的组的内部序列。在一些实例中,可以将短核酸序列附接在内部序列的5-末端。短核酸序列可以是用于随机ssDNA文库中的5’引物序列,即SEQ ID NO.1的核酸序列。因此,与腰果特异性结合的适体可以包含选自由SEQ ID NO.13003至14002组成的组的核酸序列。在其他实例中,可以将短核酸序列附接在内部序列的3-末端。短核酸序列可以是用于随机ssDNA文库中的3’引物序列,即SEQ ID NO.2的核酸序列。因此,与腰果特异性结合的适体可以包含选自由SEQ ID NO.14003至15002组成的组的核酸序列。在其他实例中,内部序列可以包含5'末端短序列(即SEQ ID NO.1)和3'末端短序列(即SEQ ID NO.2)。因此,与腰果特异性结合的适体可以包含选自由SEQ ID NO.15003至16002组成的组的核酸序列。在一个实施方案中,与腰果结合的本公开的适体可以包含选自由SEQ ID NO.12003至16002(列于表5中)组成的组的核酸序列或其变体。
表5:针对腰果的适体序列
在一些实施方案中,与榛子特异性结合的序列包含选自由SEQ ID NO.16003至17002的核酸序列组成的组的内部序列。在一些实例中,可以将短核酸序列附接在内部序列的5-末端。短核酸序列可以是用于随机ssDNA文库中的5’引物序列,即SEQ ID NO.1的核酸序列。因此,与榛子特异性结合的适体可以包含选自由SEQ ID NO.17003至18002组成的组的核酸序列。在其他实例中,可以将短核酸序列附接在内部序列的3-末端。短核酸序列可以是用于随机ssDNA文库中的3’引物序列,即SEQ ID NO.2的核酸序列。因此,与榛子特异性结合的适体可以包含选自由SEQ ID NO.18003至19002组成的组的核酸序列。在其他实例中,内部序列可以包含5'末端短序列(即SEQ ID NO.1)和3'末端短序列(即SEQ ID NO.2)。因此,与榛子特异性结合的适体可以包含选自由SEQ ID NO.19003至20002组成的组的核酸序列。在一个实施方案中,与榛子特异性结合的本公开的适体可以包含选自由SEQ IDNO.16003至20002(列于表6中)组成的组的核酸序列或其变体。
表6:针对榛子的适体序列
在一些实施方案中,与山核桃特异性结合的序列包含选自由SEQ ID NO.20003至21002的核酸序列组成的组的内部序列。在一些实例中,可以将短核酸序列附接在内部序列的5-末端。短核酸序列可以是用于随机ssDNA文库中的5’引物序列,即SEQ ID NO.1的核酸序列。因此,与山核桃特异性结合的适体可以包含选自由SEQ ID NO.21003至22002组成的组的核酸序列。在其他实例中,可以将短核酸序列附接在内部序列的3-末端。短核酸序列可以是用于随机ssDNA文库中的3’引物序列,即SEQ ID NO.2的核酸序列。因此,与山核桃特异性结合的适体可以包含选自由SEQ ID NO.22003至23002组成的组的核酸序列。在其他实例中,内部序列可以包含5’末端短序列(即SEQ ID NO.1)和3’末端短序列(即SEQ ID NO.2)。因此,与山核桃特异性结合的适体可以包含选自由SEQ ID NO.23003至24002组成的组的核酸序列。在一个实施方案中,与山核桃特异性结合的本公开的适体可以包含选自由SEQ IDNO.20003至24002(列于表7中)组成的组的核酸序列或其变体。
表7:针对山核桃的适体序列
在一些实施方案中,与开心果特异性结合的序列包含选自由SEQ ID NO.24003至25002的核酸序列组成的组的内部序列。在一些实例中,可以将短核酸序列附接在内部序列的5-末端。短核酸序列可以是用于随机ssDNA文库中的5’引物序列,即SEQ ID NO.1的核酸序列。因此,与开心果特异性结合的适体可以包含选自由SEQ ID NO.25003至26002组成的组的核酸序列。在其他实例中,可以将短核酸序列附接在内部序列的3-末端。短核酸序列可以是用于随机ssDNA文库中的3’引物序列,即SEQ ID NO.2的核酸序列。因此,与开心果特异性结合的适体可以包含选自由SEQ ID NO.26003至27002组成的组的核酸序列。在其他实例中,内部序列可以包含5’末端短序列(即SEQ ID NO.1)和3’末端短序列(即SEQ ID NO.2)。因此,与开心果特异性结合的适体可以包含选自由SEQ ID NO.27003至28002组成的组的核酸序列。在一个实施方案中,与开心果特异性结合的本公开的适体可以包含选自由SEQ IDNO.24003至28002(列于表8中)组成的组的核酸序列或其变体。
表8:针对开心果的适体序列
在一些实施方案中,与核桃特异性结合的序列包含选自由SEQ ID NO.28003至29002的核酸序列组成的组的内部序列。在一些实例中,可以将短核酸序列附接在内部序列的5-末端。短核酸序列可以是用于随机ssDNA文库中的5’引物序列,即SEQ ID NO.1的核酸序列。因此,与核桃特异性结合的适体可以包含选自由SEQ ID NO.29003至30002组成的组的核酸序列。在其他实例中,可以将短核酸序列附接在内部序列的3-末端。短核酸序列可以是用于随机ssDNA文库中的3’引物序列,即SEQ ID NO.2的核酸序列。因此,与核桃特异性结合的适体可以包含选自由SEQ ID NO.30003至31002组成的组的核酸序列。在其他实例中,内部序列可以包含5’末端短序列(即SEQ ID NO.1)和3’末端短序列(即SEQ ID NO.2)。因此,与核桃特异性结合的适体可以包含选自由SEQ ID NO.31003至32002组成的组的核酸序列。在一个实施方案中,与核桃特异性结合的本公开的适体可以包含选自由SEQ IDNO.28003至32002(列于表9中)组成的组的核酸序列或其变体。
表9:针对核桃的适体序列
在一些实施方案中,与所有坚果特异性结合的序列包含选自由SEQ ID NO.32003至33002的核酸序列组成的组的内部序列。在一些实例中,可以将短核酸序列附接在内部序列的5-末端。短核酸序列可以是用于随机ssDNA文库中的5’引物序列,即SEQ ID NO.1的核酸序列。因此,与所有坚果特异性结合的适体可以包含选自由SEQ ID NO.33003至34002组成的组的核酸序列。在其他实例中,可以将短核酸序列附接在内部序列的3-末端。短核酸序列可以是用于随机ssDNA文库中的3’引物序列,即SEQ ID NO.2的核酸序列。因此,与所有坚果特异性结合的适体可以包含选自由SEQ ID NO.34003至35002组成的组的核酸序列。在其他实例中,内部序列可以包含5’末端短序列(即SEQ ID NO.1)和3’末端短序列(即SEQ IDNO.2)。因此,与所有坚果特异性结合的适体可以包含选自由SEQ ID NO.35003至36002组成的组的核酸序列。在一个实施方案中,可以与所有坚果结合的本公开的适体可以包含选自由SEQ ID NO.32003至36002(列于表10中)组成的组的核酸序列或其变体。
表10:针对所有坚果的适体序列
在一些实施方案中,可以通过本公开选择在检测测定过程中能够与对照物结合的序列。作为非限制性序列,通过本发明的方法选择与花生对照物结合的序列,其包含选自由SEQ ID NO.36003至37002的核酸序列组成的组的内部序列。在一些实例中,可以将短核酸序列附接在内部序列的5-末端。短核酸序列可以是用于随机ssDNA文库中的5’引物序列,即SEQ ID NO.1的核酸序列。因此,与花生对照物特异性结合的适体可以包含选自由SEQ IDNO.37003至38002组成的组的核酸序列。在其他实例中,可以将短核酸序列附接在内部序列的3-末端。短核酸序列可以是用于随机ssDNA文库中的3’引物序列,即SEQ ID NO.2的核酸序列。因此,与花生对照物特异性结合的适体可以包含选自由SEQ ID NO.38003至39002组成的组的核酸序列。在其他实例中,内部序列可以包含5’末端短序列(即SEQ ID NO.1)和3’末端短序列(即SEQ ID NO.2)。因此,与花生对照物特异性结合的适体可以包含选自由SEQID NO.39003至40002组成的组的核酸序列。在一个实施方案中,可用于检测花生对照物的本公开的适体可以包含选自由SEQ ID NO.36003至40002(列于表11中)组成的组的核酸序列或其变体。
表11:适体花生对照序列
在一些实施方案中,与麸质特异性结合的序列包含选自由SEQ ID NO.40003至41002的核酸序列组成的组的内部序列。在一些实例中,可以将短核酸序列附接在内部序列的5-末端。短核酸序列可以是用于随机ssDNA文库中的5’引物序列,即SEQ ID NO.1的核酸序列。因此,与麸质特异性结合的适体可以包含选自由SEQ ID NO.41003至42002组成的组的核酸序列。在其他实例中,可以将短核酸序列附接在内部序列的3-末端。短核酸序列可以是用于随机ssDNA文库中的3’引物序列,即SEQ ID NO.2的核酸序列。因此,与麸质特异性结合的适体可以包含选自由SEQ ID NO.42003至43002组成的组的核酸序列。在其他实例中,内部序列可以包含5’末端短序列(即SEQ ID NO.1)和3’末端短序列(即SEQ ID NO.2)。因此,与麸质特异性结合的适体可以包含选自由SEQ ID NO.43003至44002组成的组的核酸序列。在一个实施方案中,与麸质特异性结合的本公开的适体可以包含选自由SEQ IDNO40003至44002(列于表12中)组成的组的核酸序列或其变体。
表12:针对麸质的适体序列
在一些实施方案中,与乳清特异性结合的序列包含选自由SEQ ID NO.44003至45002的核酸序列组成的组的内部序列。在一些实例中,可以将短核酸序列附接在内部序列的5-末端。短核酸序列可以是用于随机ssDNA文库中的5’引物序列,即SEQ ID NO.1的核酸序列。因此,与乳清特异性结合的适体可以包含选自由SEQ ID NO.45003至46002组成的组的核酸序列。在其他实例中,可以将短核酸序列附接在内部序列的3-末端。短核酸序列可以是用于随机ssDNA文库中的3’引物序列,即SEQ ID NO.2的核酸序列。因此,与乳清特异性结合的适体可以包含选自由SEQ ID NO.46003至47002组成的组的核酸序列。在其他实例中,内部序列可以包含5’末端短序列(即SEQ ID NO.1)和3’末端短序列(即SEQ ID NO.2)。因此,与乳清特异性结合的适体可以包含选自由SEQ ID NO.47003至48002组成的组的核酸序列。在一个实施方案中,与乳清特异性结合的本公开的适体可以包含选自由SEQ IDNO.44003至48002(列于表13中)组成的组的核酸序列或其变体。
表13:针对乳清的适体序列
在一些实施方案中,与酪蛋白特异性结合的序列包含选自由SEQ ID NO.48003至49002的核酸序列组成的组的内部序列。在一些实例中,可以将短核酸序列附接在内部序列的5-末端。短核酸序列可以是用于随机ssDNA文库中的5’引物序列,即SEQ ID NO.1的核酸序列。因此,与酪蛋白特异性结合的适体可以包含选自由SEQ ID NO.49003至50002组成的核酸序列。在其他实例中,可以将短核酸序列附接在内部序列的3-末端。短核酸序列可以是用于随机ssDNA文库中的3’引物序列,即SEQ ID NO.2的核酸序列。因此,与酪蛋白特异性结合的适体可以包含选自由SEQ ID NO.50003至51002组成的组的核酸序列。在其他实例中,内部序列可以包含5’末端短序列(即SEQ ID NO.1)和3’末端短序列(即SEQ ID NO.2)。因此,与酪蛋白特异性结合的适体可以包含选自由SEQ ID NO.51003至52002组成的组的核酸序列。在一个实施方案中,与酪蛋白特异性结合的本公开的适体可以包含选自由SEQ IDNO.48003至52002(列于表14中)组成的组的核酸序列或其变体。
表14:针对酪蛋白的适体序列
在一些实施方案中,本公开的SPN包含通过本发明的方法选择的适体和可包被到固体支持物上的短寡核苷酸锚定序列。所述短锚定寡核苷酸包含与相同适体序列的一部分互补的核酸序列。在一些实施方案中,用于检测花生过敏原的SPN包含选自由SEQ ID No.3至4002组成的组的适体序列和与所述适体序列互补的一个或多个短锚定序列。作为非限制性实例,互补序列可以包含选自SEQ ID NO.52003至52042(如表15所示)的核酸序列。在一些实施方案中,可以对锚定寡核苷酸进行修饰以在序列的一端包含间隔子。作为非限制性实例,对锚定序列进行修饰以在序列的5’末端包含12个碳原子的间隔子或6个碳原子的间隔子(表15),或在序列的一端包含polyA尾。短互补序列可以直接地或通过接头共价连接到固体支持物(例如,玻璃或塑料芯片)上。因此,固体表面上的接头(碳原子或polyA尾)的长度可以防止位阻,并降低由于基质的自发荧光而产生干扰的可能性。
表15:短互补锚定序列
在一些实施方案中,本公开提供了用于检测过敏原的检测试剂盒。试剂盒包含(a)包含与感兴趣的靶标特异性结合的适体序列的SPN,其中适体在感兴趣的靶标存在下不与其互补序列结合;(b)固体支持物,其表面包被有与适体的序列互补的短核酸序列。检测试剂盒可进一步包含一种或多种缓冲溶液和其他试剂。缓冲液适用于制备样品溶液、SPN溶液和/或进行检测测定所需的其他溶液(例如洗涤缓冲液)。可以将这些试剂盒组分中的一种或多种分到单独的容器中,或者它们可以以集合状态提供。在一些实施方案中,试剂盒可包含对多个过敏原靶标特异的多个SPN。例如,试剂盒可以包含对花生和常见的树坚果(包括杏仁、巴西坚果、腰果、榛子、山核桃、开心果和核桃)特异的SPN组。
在一些实施方案中,检测试剂盒可以进一步包含一个或多个对照适体序列;所述对照序列可用于测量总蛋白并将基线标准化。例如,包含对花生特异的SPN以用于花生检测的检测试剂盒可以包含花生对照序列,其能测量总蛋白并在花生检测过程中将基线标准化。作为非限制性实例,花生检测试剂盒可以包含一个或多个包含选自SEQ ID NO.3至4002的核酸序列的花生特异性适体,以及一个或多个包含选自SEQ ID NO.36003至40002的核酸序列的花生对照适体。
在一些实施方案中,本公开提供了用于检测食物样品中过敏原的存在和/或不存在的方法,所述方法包括以下步骤:(i)制备待测样品溶液和SPN溶液;(ii)将样品和SPN溶液混合,并孵育混合物以诱导靶标与SPN的结合;(iii)使混合物与包被有包含与SPN互补的序列的短寡核苷酸的固体支持物接触;和(iv)测量信号并检测感兴趣的过敏原的存在和/或不存在。可以在序列的一端用荧光团(例如Cy5和Alexa Fluor 647)标记SPN。
在一些实施方案中,固体支持物是玻璃芯片(例如,硼硅酸盐玻璃芯片),其中玻璃芯片的表面被分成包括至少一个反应组和至少两个对照组的多个组。玻璃芯片的反应性组共价包被有包含与SPN互补的序列的短寡核苷酸,当SPN不与感兴趣的靶标结合时,SPN可以与所述序列杂交以形成双链核酸。反应组可以在每侧有两个对照组。对照组可以包被有与SPN或靶标均不结合的随机序列。
芯片可以是适用于检测设备/系统中的任何尺寸,例如10x10mm。在一些实施方案中,检测芯片可以是塑料芯片。
在一些实施方案中,可以用均质缓冲液处理食物样品,所述均质缓冲液含有对感兴趣的过敏原(例如花生)特异的SPN。食物浆液流在玻璃芯片上的反应组,所述芯片嵌入在设计成使芯片面对激光和光学传感器的盒中。洗涤缓冲液流在反应组,从而从组中除去任何非特异性结合相互作用。通过光学传感器读取测定法的多个步骤,并通过算法进行分析,以提供“检测到过敏原”或“未检测到过敏原”的应答。SPN可以在靶标变应原不存在下自由结合至反应组上的互补寡核苷酸,从而产生高荧光信号。在靶标过敏原存在下,SPN:补体结合界面被阻塞,从而导致反应组上荧光信号的减少。
仅使用常规实验,本领域技术人员就将认识到或能够确定根据本文所述的公开内容的具体实施方案的许多等价物。本公开的范围不旨在限于以上说明书,而是如所附权利要求书中所述。
在权利要求中,诸如“a”,“an”和“the”的冠词可以意指一个或多个,除非另有相反向指示或从上下文中显而易见。如果一个或多个或所有组成员均存在于、用于给定产品或过程或与之相关,则认为在一组中的一个或多个成员之间包含“或”的权利要求或描述即可满足,除非另有相反指示或从上下文中显而易见。本公开包括其中恰好组中的一个成员存在于、用于给定的产品或过程或与之相关的实施方案。本公开包括其中超过一个或全部组成员均存在于、用于给定产品或过程或与之相关的实施方案。
还应注意,术语“包含”旨在是开放的并且允许但不要求包括其他元素或步骤。当在本文中使用术语“包含”时,术语“由……组成”也由此涵盖在内和得到公开。
当给出范围时,包括端点。此外,应理解,除非另有指出或从上下文和本领域普通技术人员的理解中显而易见,否则在本公开的不同实施方案中,表示为范围的值可以认为是所述范围内的任何具体值或子范围,至所述范围的下限单位的十分之一,除非上下文另有明确指示。
另外,应当理解,落入现有技术范围内的本公开的任何具体实施方案均可以明确地从任意一个或多个权利要求中排除。由于这样的实施例被认为是本领域普通技术人员已知的,因此即使在本文中未明确提出排除,也可以将其排除。出于任何原因(不管是否与现有技术的存在有关或无关),本公开的组合物的任何具体实施方案(例如,任何抗生素、治疗成分或活性成分;任何生产方法;任何使用方法;等等)都可以从任意一项或多项权利要求中排除。
应当理解,所使用的词语是描述性的词语而不是限制性的词语,并且在不背离本公开在其更广泛方面的真实范围和精神的前提下,可以在所附权利要求书的范围内进行改变。
尽管已经以一定长度以关于多个所述实施方案的一些特殊性描述了本公开,但并不旨在应该将本公开限制在任何这样的具体情节或实施例或任何具体实施方案中,而是按照所附权利要求来解释,以鉴于现有技术提供对这些权利要求的尽可能广泛的解释,并因此有效地涵盖本公开的预期范围。
实施例
如图1所示,为开始一轮SELEX,将来自随机DNA文库(第1轮)或来自前一轮(富集文库)中的ssDNA分子以一定浓度稀释在水中(例如20ng/μL)。在合适的提取缓冲液中制备靶蛋白溶液,并根据轮次稀释至所需浓度。混合一定体积的稀释的ssDNA分子溶液(例如100μL)和一定体积的靶蛋白溶液(例如300μL),并将所得混合物在室温下根据轮次振荡孵育一段时间。将以规定量稀释在提取缓冲液(例如600μL)中的氧化石墨烯(GO)溶液添加到ssDNA分子和靶标混合物中。氧化石墨烯(GO)能吸附未结合的序列,并使与靶标结合的序列自由。然后通过离心除去未结合的序列和GO。将ssDNA/靶标/GO混合物在室温下振荡孵育20分钟,在此期间,GO表面会吸附任何未与靶标物结合的ssDNA。20分钟后,将混合物以10,000g离心3分钟,并收集含有与靶蛋白结合的ssDNA和过量靶蛋白的上清液。丢弃含有GO和GO表面上吸附的ssDNA的沉淀。
为了从靶标中分离结合的ssDNA,将10%Strataclean树脂添加到含有靶蛋白和ssDNA复合物的收集的上清液中。将所得混合物加热至80℃3分钟,然后以10,000g离心3分钟。丢弃含有与树脂结合的靶蛋白的沉淀,并收集上清液。再重复至少一轮strataclean步骤。测量最终上清液中ssDNA的浓度,并将其与添加靶蛋白和GO之前的初始浓度进行比较。每轮选择后的ssDNA比率用于确定是否需要进一轮的选择。例如,如果比率低于50%,则以相同条件重复下一轮,直到回收率提高为止。
然后使用生物素化的反向引物和标记有Cy5的正向引物通过PCR对收集的ssDNA最终池进行扩增。清洁经PCR扩增的DNA以去除任何残留试剂(例如PCR Clean Up Kit),并测量DNA分子的浓度。将清洁的PCR产物添加到包被有链霉亲和素的磁珠中。生物素化的互补链与包被有链霉亲和素的珠子结合,然后添加碱以使dsDNA分子变性。使用磁体将仍结合有生物素化的互补链的珠子从溶液中拉出,并收集具有Cy5标记的目标ssDNA链。对分离出的ssDNA池进行浓缩、测量,并准备用于下一轮选择。
将实施例1中的ssDNA池在提取缓冲液中稀释至0.2ng/μL。制备相同的靶标溶液,并稀释至严谨条件。将50μL ssDNA溶液与50μL靶蛋白混合,并在室温下振荡孵育1分钟。然后将这个ssDNA/蛋白混合物添加到16孔玻片的两个孔中,其中含有与ssDNA分子的引物区域互补的短锚定物。在室温下振荡孵育1分钟后,将ssDNA/蛋白混合物转移到同一玻片的另两个孔中。重复这个过程以进行共八次孵育。最终孵育后,收集ssDNA/蛋白混合物。清洁、扩增和链分离步骤与正GO-SELEX选择(参见实施例1)中的相同。可以将这个玻璃上选择重复多次,直到回收率可接受为止。
将来自最后一轮正玻璃上SELEX(实施例2)的ssDNA分子在提取缓冲液中稀释至0.1ng/μL的浓度。如上所述,将50μL ssDNA溶液添加到新16孔玻片的两个孔中。如果不存在任何蛋白,则池中所有能够结合互补序列的序列都应结合。在室温下振荡孵育1分钟后,将ssDNA溶液转移到同一玻片的另两个孔中,并再孵育1分钟。重复这个过程以进行共八次孵育。最终孵育后,收集ssDNA并保存用于测序。
将来自最后一轮正玻璃上SELEX(实施例2)的ssDNA分子在提取缓冲液中稀释至0.1ng/μL的浓度。将感兴趣的反向蛋白在提取缓冲液中解离并稀释至10000ppm。将50μLssDNA溶液与50μL反向靶标混合物混合,并将混合物在室温下振荡孵育1分钟。然后如前所述地将这个ssDNA/蛋白混合物添加到新16孔玻片的两个孔中。混合物中任何也具有与这些不希望的反向靶标结合的能力的ssDNA都将与所述蛋白结合,而不与玻璃上的短互补序列结合。在室温下振荡孵育1分钟后,将ssDNA/蛋白混合物转移到同一载玻片的另两个孔中。重复这个过程以进行共八次孵育。最终孵育后,如前所述地收集并清洁ssDNA,然后保存以进行测序。
麸质发现于小麦、荞麦、大麦和黑麦中,其包含两个主要部分:水和醇溶性麦醇溶蛋白(gliadin),以及不溶性麦谷蛋白(glutenin)(Journal of AOAC International2013;96,1-8)。由于这些溶解度差异,使用联合SELEX方法来选择识别麦醇溶蛋白部分的适体。
从食物中提取麸质
使用并比较了几种不同的提取方法(Fallahbaghery等人,J.Agric.FoodChem.2017;65,2857-2866;和Ito等人,Anal Bioanal Chem.2016;408,5973-5984)。测试了表面活性剂、盐类和还原剂。所测试的表面活性剂包括0.1%Tween 20或1%SDS。所测试的盐类包括25mM NaCl和5mM MgCl
为了选择理想的提取缓冲液,测试了24种不同的缓冲液,并通过ELISA测定了它们的提取效率。第一轮测试仅在小麦上进行,而进一步的测试则使用了四种不同的小麦食品:燕麦片、葡萄酒、猪肉末和冰淇淋。从这个测试中得出,最好的麸质提取缓冲液包含20mMHEPES、30%EtOH、0.1%Tween20、2mM盐酸胍、25mM NaCl和5mM MgCl
确定GO和ssDNA分子的比例
确定提取缓冲液中GO与ssDNA分子的最佳比例,以在选择过程中实现ssDNA分子的最大回收率。最佳比例为GO是ssDNA的10倍过量,但是ssDNA对GO的亲和力取决于缓冲液的盐含量。使用相同量ssDNA的GO稀释曲线表明,麸质提取缓冲液中GO相对于ssDNA的所需质量比是2000:1。
每个靶蛋白都具有各自的交叉反应性问题。对于麸质,测试了多个反向蛋白种类,包括树坚果、常用的小麦替代品(竹芋、米粉、荞麦)和其他主要过敏原(蛋、奶、大豆)。
对照序列可以用于检测测定中,例如用于过敏原检测测定中以测量总蛋白。为代替基准(fiducial),或除基准之外,可以将来自控制序列的信号并入测定算法中。在这个实施例中,使用本文所述的SELEX方法从ssDNA文库中选择用于花生检测的对照序列(即花生对照序列)。对照序列的标准包括:1)对相应基质(例如食物类型)具有与靶标例如AraH1(花生过敏原)相似的应答;2)对靶标物例如花生无应答或几乎无应答;3)不与针对靶标(AraH1)的适体或其锚定序列结合。
为了选择花生对照序列,使用不同的结合材料进行重复选择。在每一轮选择之前,进行针对10,000ppm花生的反向选择,并使用从每一轮反向选择中收集的序列。表16列出了使用不同结合材料的重复选择。
表16:用于花生对照序列的材料和选择
对从第16、17和18轮收集的序列进行测序和检测。对以下进行分析:每个序列的热图,所预测的对花生过敏原蛋白AraH1(AraH1探针)特异的适体和锚定序列的结合,以及折叠结构。表11列出了这个选择中的前1000个命中。挑选了13个对照序列(表17)并对它们进行了进一步表征。
这些花生对照序列在高达100nM浓度不与对AraH1(AraH1探针)特异的适体结合。
评估了对照序列与锚定序列(AAAAATCAAGTGGTC;SEQ ID NO.52003)的结合,每个序列对于AraH1探针与花生的结合的干扰,以及每个序列对花生的亲和力。数据显示,当在100nM的浓度下进行检测时,三个序列PC36、PC60和PC87对5000ppm花生的应答最小。
比较了包括无糖华夫饼和草莓果塔饼干的两种食物类型对AraH1适体和花生对照序列PC36、PC60和PC87(每个浓度为100nM)的应答。在食物中掺入0ppm或5000ppm花生。数据表明,AraH1适体对华夫饼产生高信号,而对果塔饼干(poptart)产生较低信号。
表18:AraH1适体和花生对照序列的信号
- 适体选择方法
- 适体传感器用电极材料、适体传感器及其制备方法
