用于预测早产状况的方法、系统和试剂盒
文献发布时间:2023-06-19 12:25:57
优先权声明
本申请要求在2018年10月31日提交的PCT申请PCT/CN2018/112965的优先权,其全部内容通过引用并入本文。
背景技术
早产是全世界5岁以下儿童中死亡的主要原因,并且是围产期发病率和死亡率的主要原因。2015年,早产和低出生体重占婴儿死亡的约17%。在美国,每年有10%的婴儿早产。所有早产儿或早产的三分之一是由未足月胎膜早破(PPROM)引起的。自发的胎膜破裂(ROM)(即羊膜囊破裂)是分娩和胎儿产出的正常组成部分。胎膜早破(PROM)是指在分娩开始之前胎膜的破裂,而与胎龄无关。当PROM发生在足月时,通常会自发分娩或在12-24小时内引产。未足月胎膜早破(PPROM)是指发生在妊娠37周前的胎膜早破(PROM)。PPROM所带来的复杂妊娠的管理更具挑战性。PPROM使所有分娩的约2%至20%复杂化,并与约18%至20%的围产期死亡相关。管理选择包括入院,羊膜穿刺术以排除羊膜内感染,以及施用产前皮质类固醇和广谱抗生素(如果需要的话)。
目前用于诊断PROM和/或PPROM的金标准包括回顾患者的病史、体检和羊水池(pooling)、硝嗪(一种pH指示剂染料)和/或羊齿状结晶(ferning)(即检测宫颈干燥粘液中的“羊齿状”模式以检查羊水的存在)的临床评估。其它诊断方法包括鉴定存在于宫颈阴道分泌物中的生物标记,如甲胎蛋白(AFP)、胎儿纤维连接蛋白(fFN)、胰岛素样生长因子结合蛋白1(IGFBP1)、催乳素、人绒毛膜促性腺激素β亚基(β-hCG)、肌酸酐、尿素、乳酸和胎盘α巨球蛋白1(PAMG-1)。然而,这种检查主要是在潜在的出生条件(如PPROM)发生时进行,但在具有完整胎膜的女性中可能无法进行。换句话说,目前的诊断测试可能无法预测潜在的早产,如PPROM。PROM和PPROM的早期和准确诊断将有助于针对胎龄的产科干预措施,以优化围产期结果并使严重并发症最小化,所述并发症例如脐带脱垂和感染性病症(如绒毛膜羊膜炎和新生儿败血症)。因此,需要快速、准确的早产筛查方法,其是非侵入性的、经济有效的,并且可以应用于孕妇。
发明概述
本公开提供了用于通过处理指示不同类型的多个微生物群体的分布的生物样品来预测早产状况的方法、系统和试剂盒。可分析从受试者获得的生物样品(例如阴道流体样品)以测量微生物组分布。这样的受试者可以包括具有早产状况的受试者和没有早产状况的受试者。
在一个方面,本文公开了用于预测具有未出生胎儿的受试者的早产状况的方法。该方法可包括(a)处理从所述受试者获得的生物样品以产生指示所述生物样品中不同类型的多个微生物群体分布的数据,其中所述多个微生物群体的单个群体的存在、不存在或相对量指示所述受试者中的所述早产状况;(b)使用训练算法处理指示所述多个微生物群体的所述分布的所述数据,以确定所述生物样品中所述多个微生物群体的所述单个群体的存在、不存在或相对量,其中训练算法被配置成以针对独立样品至少90%的准确度预测所述早产状况;(c)基于在(b)中确定的所述多个微生物群体的所述单个群体的所述存在、不存在或相对量,以至少约90%的准确度将所述受试者预测为在所述受试者中具有所述早产状况;和(d)以电子方式输出识别或提供所述受试者中所述早产状况的指示的报告。
在一些实施方案中,训练算法可以用与早产状况的存在相关的第一数量的独立训练样品和与早产状况的不存在相关的第二数量的独立训练样品来训练,并且第一数量不大于第二数量。在一些实施方案中,过程(a)可包括(i)使所述生物样品经受足以分离所述多个微生物群体的条件,和(ii)识别所述多个微生物群体的所述单个群体的所述存在、不存在或相对量。
在一些实施方案中,多个微生物群体中的多个群体可以包括至少5个不同的微生物群体。所述至少5个不同种类的微生物包括选自惰性乳杆菌(Lactobacillus iners)、阴道阿托波氏菌(Atopobium vagie)、大肠杆菌(Escherichia coli)、二路普雷沃氏菌(Prevotella bivia)、卷曲乳杆菌(Lactobacillus crispatus)、解脲支原体(Ureaplasmaurealyticum)、加氏乳杆菌(Lactobacillus gasseri)、BVAB2、粪肠球菌(Enterococcusfaecalis)、詹氏乳杆菌(Lactobacillus jensenii)、巨球型菌2(Megasphaera 2)、羞怯动弯杆菌(Mobiluncus mulieris)、金黄色葡萄球菌(Staphylococcus aureus)、阴道加德纳菌(Gardnerella vagilis)、巨球型菌1(Megasphaera 1)、光滑念珠菌(Candidaglabrata)、克柔念珠菌(Candida krusei)、无乳链球菌(Streptococcus agalactiae)、白色念珠菌(Candida albicans)、沙眼衣原体(Chlamydia trachomatis)、近平滑念珠菌(Candida parapsilosis)、梅毒螺旋体(Treponema pallidum)、人型支原体(Mycoplasmahominis)、克氏动弯杆菌(Mobiluncus curtisii)、淋病奈瑟菌(Neisseria gonorrhoeae)、单纯疱疹病毒I型(Herpes simplex 1)、Trichomos vagilis、杜氏嗜血杆菌(Haemophilusducreyi)、生殖支原体(Mycoplasma genitalium)、葡萄牙念珠菌(Candida lusitaniae)、脆弱拟杆菌(Bacteroides fragilis)、单纯疱疹病毒II型(Herpes simplex 2)、热带念珠菌(Candida tropicalis)和都柏林念珠菌(Candida dubliniensis)中的一个或多个成员。
在一些实施方案中,所述方法可进一步包括监测用于治疗受试者中的早产状况的治疗过程,其中所述监测包括在两个或更多个时间点评估受试者中的早产状况,其中所述评估至少基于在所述两个或更多个时间点中的每一个时间点(b)中确定的所述多个微生物群体的单个群体的存在、不存在或相对量。
在另一方面,本文公开了一种用于预测具有未出生胎儿的受试者的早产状况的计算机系统。在一些实施方案中,计算机系统被编程或配置为实现本公开的方法,例如如上所述的方法。所述计算机系统可以包括数据库和可操作地耦合到数据库的一个或多个计算机处理器,所述数据库被配置成存储指示所述受试者的生物样品中不同类型的多个微生物群体的分布的数据,其中所述多个微生物群体中的单个群体的存在、不存在或相对量指示所述受试者中的早产状况。所述一个或多个计算机处理器单独地共同编程以:(i)使用训练算法处理指示所述多个微生物群体的所述分布的所述数据,以确定所述生物样品中所述多个微生物群体的所述单个群体的存在、不存在或相对量,其中训练算法被配置成以针对独立样品至少90%的准确度预测所述早产状况;(ii)基于(b)中确定的所述多个微生物群体的所述单个群体的所述存在、不存在或相对量,以至少约90%的准确度将所述受试者预测为在所述受试者中具有所述早产状况;和(iii)以电子方式输出识别或提供所述受试者中所述早产状况的指示的报告。
在另一个方面,本文公开了一种非暂时性计算机可读介质,其包括机器可执行代码,所述机器可执行代码在由一个或多个计算机处理器执行时实现用于预测具有未出生胎儿的受试者的早产状况的方法。在一些实施方案中,所述非暂时性计算机可读介质包括机器可执行代码,所述机器可执行代码在由一个或多个计算机处理器执行时实现本公开的方法,例如如上所述的方法。该方法可包括(a)处理从所述受试者获得的生物样品以产生指示所述生物样品中不同类型的多个微生物群体分布的数据,其中所述多个微生物群体的单个群体的存在、不存在或相对量指示所述受试者中的所述早产状况;(b)使用训练算法处理指示所述多个微生物群体的所述分布的所述数据,以确定所述生物样品中所述多个微生物群体的所述单个群体的存在、不存在或相对量,其中训练算法被配置成以针对独立样品至少90%的准确度预测所述早产状况;(c)基于在(b)中确定的所述多个微生物群体的所述单个群体的所述存在、不存在或相对量,以至少约90%的准确度将所述受试者预测为在所述受试者中具有所述早产状况;和(d)以电子方式输出识别或提供所述受试者中所述早产状况的指示的报告。
在另一个方面,本文公开了一种用于预测具有未出生胎儿的受试者的早产的试剂盒。所述试剂盒可包含:用于识别所述受试者的生物样品中不同类型的多个微生物群体中的单个群体的存在、不存在或相对量的探针,其中所述生物样品中所述多个微生物群体中的所述单个群体的存在、不存在或相对量指示具有所述未出生胎儿的所述受试者的早产,其中所述探针相对于所述生物样品中的其他微生物群体对所述多个微生物群体具有选择性;和使用所述探针处理所述生物样品以产生指示所述生物样品中所述不同类型的多个微生物群体的分布的数据,从而以针对独立样品至少90%的准确度预测所述早产的说明书。在一些实施方案中,所述试剂盒用于本公开的方法,例如如上所述的方法。
在另一个方面,本文公开了探针在制备用于预测具有未出生婴儿的受试者的早产的试剂盒中的用途。所述探针用于识别所述受试者的生物样品中不同类型的多个微生物群体的单个群体的存在、不存在或相对量,其中所述生物样品中所述多个微生物群体的所述单个群体的存在、不存在或相对量指示具有未出生胎儿的所述受试者的早产,其中所述探针相对于所述生物样品中的其他微生物群体对所述多个微生物群体具有选择性。所述预测包括:(a)处理从所述受试者获得的生物样品以产生指示所述生物样品中不同类型的多个微生物群体分布的数据,其中所述多个微生物群体的单个群体的存在、不存在或相对量指示所述受试者中的所述早产状况;(b)使用训练算法处理指示所述多个微生物群体的所述分布的所述数据,以确定所述生物样品中所述多个微生物群体的所述单个群体的存在、不存在或相对量,其中训练算法被配置成以针对独立样品至少90%的准确度预测所述早产状况;(c)基于在(b)中确定的所述多个微生物群体的所述单个群体的所述存在、不存在或相对量,以至少约90%的准确度将所述受试者预测为在所述受试者中具有所述早产状况;和任选地(d)以电子方式输出识别或提供所述受试者中所述早产状况的指示的报告。
在一些实施方案中,所述试剂盒用于本公开的方法,例如如上所述的方法。
从以下详细描述中,本公开的另外的方面和优点对于本领域技术人员将变得显而易见,其中仅示出和描述了本公开的说明性实施实施方案。如将认识到的,本公开能够具有其它和不同的实施例,并且其若干细节能够在各种明显方面进行修改,所有这些都不脱离本公开。因此,附图和说明书应被认为是说明性的,而不是限制性的。
援引并入
说明书中提及的所有出版物、专利和专利申请均通过引用并入本文,其程度如同每个单独的出版物、专利或专利申请被具体地和单独地指明通过引用并入本文。在通过引用并入的出版物和专利或专利申请与说明书中包含的公开内容矛盾的程度上,说明书旨在取代和/或优先于任何这种矛盾的材料。
附图简要说明
本发明的新颖特征在所附权利要求中具体阐述。通过参考以下详细描述和附图(在此也称为“图”)将获得对本发明的特征和优点的更好理解,以下详细描述阐述了利用本发明的原理的说明性实施方案,在附图中:
图1示出了根据一些实施方案的随机森林分类器的受试者工作特征曲线(ROC)的示例,所述随机森林分类器被配置成基于对阴道样品中的微生物群体的分析来预测早产状况,其中C
图2A-2G示出了根据图1实施方案的原始试验数据的示例。
图3示出了根据一些实施方案的随机森林分类器的受试者工作特征曲线(ROC)的示例,所述随机森林分类器被配置成基于对阴道样品中的微生物群体的分析来预测早产状况,其中各个微生物的百分比、先前流产的次数和孕妇的年龄用作变量。
图4A-4F示出了根据图3实施方案的原始试验数据的示例。
图5示出了被编程或以其他方式配置以实现本文提供的方法的计算机控制系统。
发明详述
虽然本文已经示出和描述了本发明的各种实施方案,但是对于本领域技术人员来说显而易见的是,这些实施方案仅以示例的方式提供。在不背离本发明的情况下,本领域技术人员可以想到许多变化、改变和替换。应当理解,可以采用这里描述的本发明的实施方案的各种替代方案。
如在说明书和权利要求中所使用的,单数形式“一”、“一个”和“所述”包括复数参考,除非上下文另有明确指示。例如,术语“细胞”包括多个细胞,包括其混合物。
如本文所用,术语“核酸”通常指任何长度的核苷酸,脱氧核糖核苷酸(dNTPs)或核糖核苷酸(rNTPs)或其类似物的聚合形式。核酸可以具有任何三维结构,并且可以执行任何已知或未知的功能。核酸的非限制性实例包括DNA、RNA、基因或基因片段的编码或非编码区、由连锁分析限定的基因座(位点)、外显子、内含子、信使RNA(mRNA)、转运RNA、核糖体RNA、小干扰RNA(siRNA)、短发夹RNA(shRNA)、微小RNA(miRNA)、核酶、cDNA、重组核酸、支链核酸、质粒、载体、任何序列的分离DNA、任何序列的分离RNA、核酸探针和引物。核酸可包含一种或多种修饰的核苷酸,例如甲基化核苷酸和核苷酸类似物。如果存在,可在核酸组装之前或之后对核苷酸结构进行修饰。核酸的核苷酸序列可以被非核苷酸组分中断。核酸可以在聚合后进一步修饰,例如通过与报道试剂缀合或结合。
如本文所用,术语“扩增的”和“扩增”可互换使用,且通常指产生核酸的一个或多个拷贝或“扩增产物”。术语“DNA扩增”通常指产生DNA分子的一个或多个拷贝或“扩增的DNA产物”。术语“逆转录扩增”通常指通过逆转录酶的作用从核糖核酸(RNA)模板产生脱氧核糖核酸(DNA)。
如本文所用,术语“靶核酸”通常指起始核酸分子群中的核酸分子,该起始核酸分子群具有需要确定其存在、量和/或序列或这些中的一种或多种的变化的核苷酸序列。靶核酸可以是任何类型的核酸,包括DNA、RNA及其类似物。如本文所用,“靶核糖核酸(RNA)”通常指为RNA的靶核酸。如本文所用,“靶脱氧核糖核酸(DNA)”通常指为DNA的靶核酸。
如本文所用,术语“受试者”通常指具有可测试或可检测遗传信息的实体或介质。受试者可以是人或个体。受试者可以是脊椎动物,例如哺乳动物。哺乳动物的非限制性实例包括鼠、猿、人、农场动物、运动动物和宠物。受试者的其它实例包括食物、植物、土壤和水。
如本文所用,术语“约”或“大约”是指在所述量附近约10%、5%或1%的量,包括其中的增量。例如,“约”或“大约”可以表示包括特定值的范围,并且范围从低于该特定值的10%到高于该特定值的10%。
如本文所用,术语“早产”通常是指在婴儿预产期前三周以上发生的分娩。换句话说,早产是指在妊娠第37周开始之前发生的早产。早产可由未足月胎膜早破(PPROM)引起。也就是说,未足月胎膜早破(PPROM)是导致早产的原因之一。早产状况可以是未足月胎膜早破(PPROM)。术语“早产(premature birth)”可与术语“先兆早产(premature labor)”互换。
可以分析从受试者获得的生物样品(例如,阴道流体样品、羊水样品)以测量微生物组分布,例如,生物样品中的不同类型的多个微生物群体。这样的受试者可以包括女性受试者、育龄女性受试者、怀孕受试者、具有流产病史的怀孕受试者、具有早产病史的怀孕受试者和/或具有没有任何并发症的分娩病史的怀孕受试者。提供了通过处理指示不同类型的多个微生物群体的分布的生物样品来预测早产的方法、系统和试剂盒。早产可以包括早产状况、早产和/或先兆早产。胎膜早破可引起绒毛膜羊膜炎、新生儿败血症或两者。
对于一些微生物物种,早产样品(例如,从早产的受试者获得的生物样品)中的群体测量值可能大于正常样品(例如,从分娩时未早产的受试者获得的生物样品)中的群体测量值。对于其他微生物物种,早产样品(例如,从早产的受试者获得的生物样品)中的群体测量值可能小于正常样品(例如,从分娩时未早产的受试者获得的生物样品)中的群体测量值。
这些微生物物种可以是用于预测早产的生物标记的候选物,因为它们在早产样品和正常生物样品中存在差异。特别地,由于收集阴道流体样品可能已经是孕妇常规临床检查的一部分,并且第二代测序相对廉价,因此微生物组分布可以用作早产(例如,早产状况)的早期检测,作为传统临床测试(诸如相关生物标志物鉴定和/或身体检查,诸如但不限于无菌窥镜检查)的替代方案或与其结合。微生物组分布可用于监测患者(例如,怀孕或怀孕且处于早产风险的受试者)。在这种情况下,患者的微生物组分布可在监测阶段改变。例如,具有早产风险的患者的微生物组分布可向健康受试者(即,不处于早产风险的受试者)的微生物组分布转变。相反,例如,处于早产风险的患者的微生物组分布可保持不变。
在一方面,本文公开了一种用于预测具有未出生胎儿的受试者的早产的方法。该方法可包括处理从受试者获得的生物样品以生成指示生物样品中不同类型的多个微生物群体的分布的数据。多个微生物群体中的单个群体的存在、不存在或相对量可指示受试者的早产状况。接下来,可以使用训练算法来处理指示多个微生物群体的分布的数据,以确定生物样品中多个微生物群体的单个群体的存在、不存在或相对量。训练算法可被配置为对于至少约10、20、30、40、50、60、70、80、90、100、150、200、250或300个独立样品以至少约50%、60%、70%、80%、90%、95%或更高的准确度预测早产状况。接下来,基于多个微生物群体中的单个群体的存在、不存在或相对量,可以以至少约50%、60%、70%、80%、90%、95%或更高的准确度将受试者识别为具有早产状况。然后可以以电子方式输出识别或提供所述受试者中所述早产状况的指示的报告。该方法可在受试者怀孕期间的不同时间进行,从而可获得早产状况的进展或消退。
生物样品可包括来自人类受试者的阴道流体样品。阴道流体样品可在处理前储存在各种储存条件下,例如不同的温度(例如,在室温、冷藏或冷冻条件下、在4℃、在-18℃、-20℃或在-80℃)或不同的防腐剂(例如,乙醇、甲醛或重铬酸钾)。生物样品可包括来自人受试者的阴道微生物组的另一来源,例如羊水样品。在某些情况下,可以在进行羊膜穿刺术时获取羊水样品。
生物样品可以从患有疾病或病症的受试者、从怀疑患有疾病或病症的受试者、或者从未患有或未怀疑患有疾病或病症的受试者获得。疾病或病症可以是早产状况、早产的情况、堕胎、早期早产、妊娠期糖尿病、先兆子痫、流产、高血压、提前分娩、脐带脱垂、脐带压迫、羊水栓塞、子宫出血、胎盘前置、胎盘早剥、侵入性胎盘、胎盘功能不全、感染性疾病、免疫紊乱或疾病、癌症、遗传病、退行性疾病、生活方式疾病、损伤、罕见疾病和/或年龄相关疾病。感染性疾病可由细菌、病毒、真菌和/或寄生虫引起。癌症可以是子宫癌、子宫内膜癌、宫颈癌或卵巢癌。可以在患有疾病或病症的受试者的治疗之前和/或之后取样。可在疾病和病症发生之前和/或之后取样。可在治疗或治疗方案期间取样。可以从受试者获取多个样品以监测治疗随时间的效果。可以在怀孕期间取样。可以从怀孕受试者身上提取多个样品,以监测胎儿和/或胎盘膜随时间的发育。样品可取自已知或怀疑具有早产状况的受试者,对于该早产状况,通过临床试验(如羊水池试验(pooling test)、硝嗪试验、羊齿状结晶试验(ferntest)和/或纤连蛋白和甲胎蛋白试验)无法获得明确的阳性或阴性诊断。
样品可取自怀疑患有疾病或病症的受试者。样品可以取自经历诸如羊水从阴道渗漏的症状的受试者。样品可取自具有解释过的症状的受试者。样品可取自由于诸如病史、年龄、环境暴露、生活方式风险因素或存在其它已知风险因素的因素而处于发展疾病或病症风险的受试者。PROM的风险因素的非限制性实例包括感染、妊娠期间吸烟、妊娠期间使用违禁药物、在先前的妊娠中已经具有PROM和/或早产、羊水过多、多胎妊娠、妊娠期间任何时间出血、侵入性操作(如羊膜穿刺术)、营养缺陷、宫颈机能不全、社会经济地位低和体重不足。可能是PROM风险因素的感染包括尿路感染、性传播疾病、下生殖器感染(如细菌性阴道病)和羊膜囊内感染。
在从受试者获得生物样品之后,可以处理从受试者获得的生物样品以产生指示生物样品中不同类型的多个微生物群体的分布的数据。多个微生物群体中的单个群体的存在、不存在或相对量可以指示早产状况,例如早产的情况。处理从受试者获得的生物样品可包括(i)使生物样品经受足以分离多个微生物群体的条件,和(ii)鉴定多个微生物群体中的单个群体的存在、不存在或相对量。
可以通过从生物样品中提取核酸分子,并对核酸分子进行测序以识别多个微生物群体中单个微生物群体的存在、不存在或相对量来分离多个微生物群体。核酸分子可以包括脱氧核糖核酸(DNA)或核糖核酸(RNA)。核酸分子可以包括一个或多个微生物群体的DNA或RNA分子。核酸分子(例如,DNA或RNA)可通过多种方法从生物样品中提取,所述方法例如来自MP Biomedicals的FastDNA试剂盒方案、来自Qiagen的QIAamp DNA stool mini试剂盒、或来自Norgen Biotek的stool DNA分离试剂盒方案。提取方法可从样品中提取所有的DNA分子。或者,提取方法可以从样品中选择性地提取一部分DNA分子,例如通过靶向DNA分子中的某些基因,例如一个或多个微生物物种的16S核糖体RNA(rRNA)。从样品中提取的RNA分子可以通过逆转录(RT)转化为DNA分子。
测序可以通过任何合适的测序方法进行,例如大规模并行测序(MPS)、双端测序、高通量测序、下一代测序(NGS)、鸟枪测序、单分子测序、纳米孔测序、半导体测序、焦磷酸测序、边合成边测序(SBS)、连接测序和杂交测序、RNA-Seq(Illumina)。
测序可包括核酸扩增(例如DNA或RNA分子的核酸扩增)。在一些实施方案中,核酸扩增是聚合酶链式反应(PCR)。可以进行适当轮数的PCR(例如,PCR、qPCR、逆转录酶PCR、数字PCR等),以将初始量的核酸(例如,DNA)充分扩增到用于后续测序的所需上样量。在某些情况下,PCR可用于核酸的整体扩增。这可以包括使用衔接子序列,该衔接子序列可以首先连接到不同分子,随后使用通用引物进行PCR扩增。可以使用多种商业试剂盒中的任何一种进行PCR,例如由Life Technologies、Affymetrix、Promega、Qiagen等提供的试剂盒。在其他情况下,可以只扩增核酸群体中的某些靶核酸。特异性引物(可能与衔接子连接结合)可用于选择性地扩增某些靶点以进行下游测序。PCR可包括一个或多个基因组位点的靶向扩增,例如对应于一个或多个16S核糖体RNA(rRNA)基因的基因组位点的靶向扩增。
测序可包括使用同时逆转录(RT)和聚合酶链式反应(PCR),例如Qiagen、NEB、Thermo Fisher Scientific或Bio-Rad的OneStrep RT-PCR试剂盒方案。
DNA或RNA分子可以用例如可鉴定的标签来标记,以允许多个样品的多重反应。任何数量的DNA或RNA样品都可以进行多重反应。例如,多重反应可包含来自至少约2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、25、30、35、40、45、50、55、60、65、70、75、80、85、90、95、100或超过100个初始样品的DNA或RNA。例如,可以用样品条形码标记多个样品,使得每个DNA分子可以追溯到DNA分子所来源的样品(和受试者)。这样的标签可以通过连接或通过使用引物的PCR扩增连接到DNA或RNA分子上。
在对核酸分子进行测序之后,可以对序列读取(sequence read)进行适当的生物信息学处理,以产生指示生物样品中不同类型的多个微生物群体分布的数据。例如,序列读取可以与一个或多个参考基因组(例如,一个或多个细菌物种的基因组)比对。可以在一个或多个基因组位点处定量比对的序列读取,以生成指示生物样品中不同类型的多个微生物群体的分布的数据。例如,对应于多个保守和/或非保守基因组位点的序列的定量可产生指示生物样品中不同类型的多个微生物群体的分布的数据。序列的定量可以表示为或转换为一个或多个微生物群体的操作分类单元(OTU)的单元。OTU测量值可包括未归一化或归一化的值。可以在微生物(例如细菌)属水平或微生物种水平测量OTU。与生物样品中的多个细菌属和/或种相对应的OTU数据的集合可以指示生物样品中不同类型的多个微生物群体的分布。可以从OTU数据的集合推断多个微生物群体中单个微生物群体的存在、不存在或相对量。这种从OTU数据的集合推断的多个微生物群体中单个微生物群体的存在、不存在或相对量可以指示生物样品中不同类型的多个微生物群体的分布。
通过使用探针可以识别受试者的早产状况,或者可以监测受试者中早产状况(例如PPROM)的进展或消退,所述探针被配置成选择性地富集与单个微生物群体对应的核酸(例如DNA或RNA)分子。探针可以是核酸引物。探针可以与来自一个或多个微生物单个群体的核酸序列具有序列互补性。
多个微生物群体可包括至少2个、至少3个、至少4个、至少5个、至少6个、至少7个、至少8个、至少9个、至少10个、至少11个、至少12个、至少13个、至少14个、至少15个、至少16个、至少17个、至少18个、至少19个或至少20个或更多的不同的微生物群体。多个微生物群体可以包括不同种类的微生物。多个微生物群体可包括选自惰性乳杆菌、阴道阿托波氏菌、大肠杆菌、二路普雷沃氏菌、卷曲乳杆菌、解脲支原体、加氏乳杆菌、BVAB2、粪肠球菌、詹氏乳杆菌、巨球型菌2、羞怯动弯杆菌、金黄色葡萄球菌、阴道加德纳菌、巨球型菌1、光滑念珠菌、克柔念珠菌、无乳链球菌、白色念珠菌、沙眼衣原体、近平滑念珠菌、梅毒螺旋体、人型支原体、克氏动弯杆菌、淋病奈瑟菌、单纯疱疹病毒I型、Trichomos vagilis、杜氏嗜血杆菌、生殖支原体、葡萄牙念珠菌、脆弱拟杆菌、单纯疱疹病毒II型、热带念珠菌和都柏林念珠菌中的一个或多个成员。多个微生物群体可包括选自乳杆菌属、埃希氏菌属、普氏菌属、肠球菌属、念珠菌属、葡萄球菌属和疱疹病毒中的一个或多个成员。
可以在没有任何核酸提取的情况下处理生物样品以识别生物样品中的多个微生物群体的分布。例如,所述处理可以包括使用探针来测定生物样品,所述探针针对多个微生物群体具有选择性。多个微生物群体可包括至少2个、至少3个、至少4个、至少5个、至少6个、至少7个、至少8个、至少9个、至少10个、至少11个、至少12个、至少13个、至少14个、至少15个、至少16个、至少17个、至少18个、至少19个或至少20个或更多的不同的微生物群体。多个微生物群体可以包括不同种类的微生物。多个微生物群体可包括选自惰性乳杆菌、阴道阿托波氏菌、大肠杆菌、二路普雷沃氏菌、卷曲乳杆菌、解脲支原体、加氏乳杆菌、BVAB2、粪肠球菌、詹氏乳杆菌、巨球型菌2、羞怯动弯杆菌、金黄色葡萄球菌、阴道加德纳菌、巨球型菌1、光滑念珠菌、克柔念珠菌、无乳链球菌、白色念珠菌、沙眼衣原体、近平滑念珠菌、梅毒螺旋体、人型支原体、克氏动弯杆菌、淋病奈瑟菌、单纯疱疹病毒I型、Trichomos vagilis、杜氏嗜血杆菌、生殖支原体、葡萄牙念珠菌、脆弱拟杆菌、单纯疱疹病毒II型、热带念珠菌和都柏林念珠菌中的一个或多个成员。多个微生物群体包括选自加氏乳杆菌、阴道加德纳菌、阴道阿托波氏菌、解脲支原体和惰性乳杆菌中的一个或多个成员。
探针可以是与多个微生物群体的核酸序列(例如,DNA或RNA)具有序列互补性的核酸分子(例如,DNA或RNA)。这些核酸分子可以是引物或富集序列。使用针对多个微生物群体具有选择性的探针测定生物样品可包括使用阵列杂交、聚合酶链式反应(PCR)或核酸测序(例如,DNA测序或RNA测序)。
处理可包括使用探针测定生物样品,其中探针相对于生物样品中的其它微生物群体对所述多个微生物群体具有选择性。这些探针可以是与多个微生物群体的核酸序列(例如,DNA或RNA)具有序列互补性的核酸分子(例如,DNA或RNA)。这些核酸分子可以是引物或富集序列。测定可包括使用阵列杂交、聚合酶链式反应(PCR)或核酸测序(例如,DNA测序或RNA测序)。
可以在一个或多个基因组位点处定量测定读数(assay readout),以生成指示生物样品中不同类型的多个微生物群体的分布的数据。例如,对应于多个保守和/或非保守基因组位点的阵列杂交或聚合酶链式反应(PCR)的定量可产生指示生物样品中不同类型的多个微生物群体的分布的数据。测定读数可包括定量PCR(qPCR)值、数字PCR(dPCR)值、数字液滴PCR(ddPCR)值、荧光值等。阵列杂交或聚合酶链式反应(PCR)的定量可表示为或转换为一个或多个微生物群体的操作分类学单元(OTU)单元。OTU测量值可包括未归一化或归一化的值。可以在微生物(例如细菌)属水平或微生物种水平测量OTU。与生物样品中的多个细菌属和/或种相对应的OTU数据的集合可以指示生物样品中不同类型的多个微生物群体的分布。可以从OTU数据的集合推断多个微生物群体中单个微生物群体的存在、不存在或相对量。这种从OTU数据的集合推断的多个微生物群体中单个微生物群体的存在、不存在或相对量可以指示生物样品中不同类型的多个微生物群体的分布。
本文提供了用于预测或预测怀孕受试者中的早产状况的试剂盒。试剂盒可包括用于鉴定受试者的生物样品中不同类型的多个微生物群体的单个群体的存在、不存在或相对量的探针。生物体中多个微生物群体的单个群体的存在、不存在或相对量可以指示早产状况。探针可以相对于生物样品中的其他微生物群体对所述多个微生物群体具有选择性。试剂盒可包括使用探针处理生物样品以产生指示生物样品中不同类型的多个微生物群体分布的数据的说明书。
试剂盒中的探针可以相对于生物样品中的其它微生物群体对所述多个微生物群体具有选择性。试剂盒中的探针可配置成选择性富集与单个微生物群体相对应的核酸(例如,DNA或RNA)分子。试剂盒中的探针可以是核酸引物。试剂盒中的探针可与来自一个或多个微生物单个群体的核酸序列具有序列互补性。多个微生物群体可包括至少2个、至少3个、至少4个、至少5个、至少6个、至少7个、至少8个、至少9个、至少10个、至少11个、至少12个、至少13个、至少14个、至少15个、至少16个、至少17个、至少18个、至少19个或至少20个或更多的不同的微生物群体。多个微生物群体可以包括不同种类的微生物。多个微生物群体可包括选自惰性乳杆菌、阴道阿托波氏菌、大肠杆菌、二路普雷沃氏菌、卷曲乳杆菌、解脲支原体、加氏乳杆菌、BVAB2、粪肠球菌、詹氏乳杆菌、巨球型菌2、羞怯动弯杆菌、金黄色葡萄球菌、阴道加德纳菌、巨球型菌1、光滑念珠菌、克柔念珠菌、无乳链球菌、白色念珠菌、沙眼衣原体、近平滑念珠菌、梅毒螺旋体、人型支原体、克氏动弯杆菌、淋病奈瑟菌、单纯疱疹病毒I型、Trichomos vagilis、杜氏嗜血杆菌、生殖支原体、葡萄牙念珠菌、脆弱拟杆菌、单纯疱疹病毒II型、热带念珠菌和都柏林念珠菌中的一个或多个成员。所述多个微生物群体可包括选自加氏乳杆菌、阴道加德纳菌、阴道阿托波氏菌、解脲支原体和惰性乳杆菌中的一个或多个成员。
试剂盒中的说明书可以包括使用探针测定生物样品的说明,所述探针相对于生物样品中的其它微生物群体对所述多个微生物群体具有选择性。这些探针可以是与多个微生物群体的核酸序列(例如,DNA或RNA)具有序列互补性的核酸分子(例如,DNA或RNA)。这些核酸分子可以是引物或富集序列。测定生物样品的说明书可以包括进行阵列杂交、聚合酶链式反应(PCR)或核酸测序(例如,DNA测序或RNA测序)以处理生物样品,从而产生指示生物样品中不同类型的多个微生物群体的分布的数据的说明。多个微生物群体中的单个微生物群体的存在、不存在或相对量可以指示早产状况。
试剂盒中的说明书可包括测量和解释测定读数的说明,所述测定读数可在一个或多个基因组位点处进行定量,以生成指示生物样品中不同类型的多个微生物群体的分布的数据。例如,对应于多个保守和/或非保守基因组位点的阵列杂交或聚合酶链式反应(PCR)的定量可产生指示生物样品中不同类型的多个微生物群体的分布的数据。测定读数可包括定量PCR(qPCR)值、数字PCR(dPCR)值、数字液滴PCR(ddPCR)值、荧光值等。阵列杂交或聚合酶链式反应(PCR)的定量可表示为或转换为一个或多个微生物群体的操作分类学单元(OTU)单元。OTU测量值可包括未归一化或归一化的值。可以在微生物(例如细菌)属水平或微生物种水平测量OTU。与生物样品中的多个细菌属和/或种相对应的OTU数据的集合可以指示生物样品中不同类型的多个微生物群体的分布。可以从OTU数据的集合推断多个微生物群体中单个微生物群体的存在、不存在或相对量。这种从OTU数据的集合推断的多个微生物群体中单个微生物群体的存在、不存在或相对量可以指示生物样品中不同类型的多个微生物群体的分布。
在处理来自受试者的生物样品之后,可以使用训练算法来处理指示多个微生物群体的分布的数据(例如,微生物组数据),以确定生物样品中多个微生物群体的单个群体的存在、不存在或相对量。在一些实施方案中,训练算法可被配置成以针对独立样品至少86.67%的准确度识别或预测早产状况。在一些实施方案中,训练算法可被配置成以针对独立样品至少93.33%的准确度识别或预测早产状况。随着更多的样品数据可用于训练算法,准确度可以提高。
训练算法可以包括监督机器学习算法。训练算法可以包括分类和回归树(CART)算法。监督机器学习算法可以包括,例如,随机森林、支持向量机(SVM)、神经网络或深度学习算法。训练算法可以包括无监督的机器学习算法。
训练算法可以被配置为接受多个输入变量并基于多个输入变量产生一个或多个输出值。多个输入变量可包括指示多个微生物群体的分布的数据(例如,微生物组数据)。例如,输入变量可以包括指示受试者阴道样品中微生物群体(例如,细菌属或细菌种)分布的数据。
除了微生物组数据外,受试者的相关个人基本信息、临床信息等其他因素都可以作为输入变量来训练算法。在一些实施方案中,受试者的基本个人信息包括年龄、孕周等中的一个或多个。在一些实施方案中,受试者的临床信息包括流产病史、疾病病史等中的一个或多个。
训练算法可以包括分类器,使得一个或多个输出值中的每一个包括指示分类器对生物样品进行分类的固定数目的可能值(例如,线性分类器、逻辑回归分类器等)中的一个。训练算法可以包括二元分类器,使得一个或多个输出值中的每一个包括指示分类器对生物样品进行分类的两个值(例如,{0,1}、{阳性、阴性}或{早产、非早产})中的一个。训练算法可以是另一类型的分类器,使得一个或多个输出值中的每一个包括指示分类器对生物样品进行分类的两个以上的值(例如,{0、1、2}、{阳性、阴性或不确定}、或{早产、非早产或不确定})中的一个。输出值可包括描述性标签、数值或其组合。一些输出值可以包括描述性标签。此类描述性标签可以提供受试者的疾病或病症状态的识别或指示,并且可以包括例如阳性、阴性、早产、非早产或不确定。此类描述性标签可以提供针对受试者的疾病或病症状态的治疗的识别,并且可以包括例如治疗干预、治疗干预的持续时间和/或治疗干预的剂量。此类描述性标签可提供对可能适合在受试者上进行的二次临床试验的识别,并且可以包括例如血液试验、超声扫描、羊齿状结晶试验、靛红染色试验、免疫层析试验、硝嗪试验、羊水池试验、通过B超检测宫颈长度、胎儿蛋白的Elisa检测和/或通过Elisa或蛋白质芯片检测7种母体血浆蛋白。一些描述性标签可映射到数值,例如,将“阳性”映射到1和“阴性”映射到0。
一些输出值可以包括数值,例如二进制、整数或连续值。此类二进制输出值可以包括,例如,{0,1}。此类整数输出值可以包括,例如,{0,1,2}。此类连续输出值可以包括,例如,至少0且不大于1的概率值。此类连续输出值可以包括,例如,至少0的未归一化概率值。此类连续输出值可以指示治疗受试者的疾病或病症状态的治疗过程的预测,并且可以包括例如治疗过程的预期疗效的持续时间的指示。一些数值可映射到描述性标签,例如,将1映射到“阳性”和0映射到“阴性”。
可以基于一个或多个截止值来分配输出值中的一些。例如,如果样品指示受试者具有至少50%的早产概率,则样品的二元分类可以分配“阳性”或1的输出值。例如,如果样品指示受试者具有小于50%的早产概率,则样品的二元分类可以分配“阴性”或0的输出值。在这种情况下,50%的单个截止值用于将样品分类为两个可能的二进制输出值中的一个。单个截止值的实例可以包括1%、2%、5%、10%、15%、20%、25%、30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、98%和99%。
作为另一个实例,如果样品指示受试者具有至少50%、至少55%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少98%或至少99%的早产概率,则样品分类可以分配“阳性”或1的输出值。如果样品表明受试者具有大于50%、大于55%、大于60%、大于65%、大于70%、大于75%、大于80%、大于85%、大于90%、大于95%、大于98%或大于99%的早产概率,则样品的分类可以分配“阳性”或1输出值。如果样品指示受试者具有小于50%、小于45%、小于40%、小于35%、小于30%、小于25%、小于20%、小于10%、小于5%、小于2%或小于1%的早产概率,则样品的分类可以分配“阴性”或0的输出值。如果样品指示受试者具有不大于50%、不大于45%、不大于40%、不大于35%、不大于30%、不大于25%、不大于20%、不大于10%、不大于5%、不大于2%或不大于1%的早产概率,则样品分类可分配“阴性”或0的输出值。如果样品没有被分类为“阳性”、“阴性”、1或0,则样品的分类可以分配“不确定”或2的输出值。在这种情况下,使用两个截止值的集合来将样品分类到三个可能输出值中的一个。截止值集合的实例可以包括{1%,99%}、{2%,98%}、{5%,95%}、{10%,90%}、{15%,85%}、{20%,80%}、{25%,75%}、{30%,70%}、{35%,65%}、{40%,60%}、{45%,55%}。类似地,n个截止值的集合可用于将样品分类为n+1个可能输出值中的一个,其中n是任何正整数。
可以用多个独立的训练样品来训练所述训练算法。每个独立训练样品可包括来自受试者的生物样品、通过处理生物样品(如本文别处所述)获得的相关数据、以及对应于生物样品的一个或多个已知输出值(例如,早产或足月妊娠分娩)。独立训练样品可以包括从多个不同受试者获得的生物样品和相关数据和输出。独立训练样品可以与早产的存在相关联(例如,包括从已知具有早产的多个受试者获得的生物样品和相关数据和输出的训练样品)。独立训练样品可与早产的不存在相关(例如,包含从已知未早产的多个受试者获得的生物样品和相关数据和输出的训练样品)。
训练算法可以用至少20、至少40、至少50、至少100、至少150、至少200、至少250、至少300、至少350、至少400、至少450或至少500个独立训练样品来训练。独立训练样品可以包括与早产状况的存在相关联的样品和/或与早产状况的不存在相关联的样品。用与早产状况的存在相关联的不超过500、不超过450、不超过400、不超过350、不超过300、不超过250、不超过200、不超过150、不超过100、不超过50或不超过20个独立训练样品训练所述训练算法。在一些实施方案中,生物样品独立于用于训练所述训练算法的样品。
可以用与早产状况的存在相关联的第一数量的独立训练样品和与早产条件的不存在相关联的第二数量的独立训练样品来训练所述训练算法。与早产状况的存在相关联的独立训练样品的第一数量可不大于与早产状况的不存在相关联的独立训练样品的第二数量。与早产状况的存在相关联的独立训练样品的第一数量可以等于与早产状况的不存在相关联的独立训练样品的第二数量。与早产状况的存在相关联的独立训练样品的第一数量可大于与早产状况的不存在相关联的独立训练样品的第二数量。
训练算法可被配置成以针对独立样品至少约80%、至少约81%、至少约82%、至少约83%、至少约84%、至少约85%、至少约86%、至少约87%、至少约88%、至少约89%、至少约90%、至少约91%、至少约92%、至少约93%、至少约94%、至少约95%、至少约96%、至少约97%、至少约98%或至少约99%的准确度预测早产状况。在一个实施方案中,训练算法可被配置成以至少86.67%的准确度预测早产状况。在一个实施方案中,训练算法可被配置成以至少93.33%的准确度预测早产状况。通过训练算法预测早产状况的准确度可以计算为(1)正确预测为具有早产状况的独立测试样品和(2)正确预测为不具有早产状况的独立测试样品在所有独立测试样品中的比例。
训练算法可以被配置成以针对至少100个独立样品至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%的灵敏度预测早产状况。在一个实施方案中,训练算法可被配置为以至少83.33%的灵敏度预测早产状况。通过训练算法预测早产状况的灵敏度可计算为(1)正确预测为具有早产状况的独立测试样品和(2)错误预测为不具有早产状况的独立测试样品的总和中正确预测为具有早产状况的独立测试样品的比例。
训练算法可以被配置成以针对至少100个独立样品至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、至少99%或100%的特异性预测早产状况。在一个实施方案中,训练算法可被配置为以至少88.89%的特异性预测早产状况。在另一实施方案中,训练算法可被配置为以100%的特异性预测早产状况。通过训练算法预测早产状况的特异性可计算为(1)正确预测为不具有早产状况的独立测试样品和(2)错误预测为具有早产状况的独立测试样品的总和中正确预测为不具有早产状况的独立测试样品的比例。
训练算法可以被配置成以针对至少100个独立样品至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%的阳性预测值(PPV)预测早产状况。在一个实施方案中,训练算法可被配置以83.33%的PPV预测早产状况。在另一实施方案中,训练算法可被配置以100%的PPV预测早产状况。通过训练算法预测早产状况的PPV可计算为(1)正确预测为具有早产状况的独立测试样品和(2)错误预测为具有早产状况的独立测试样品的总和中正确预测为具有早产状况的独立测试样品的比例。PPV也可称为精密度。
训练算法可被配置为以至少约0.05、至少约0.10,至少约0.15,至少约0.20,至少约0.25,至少约0.30,至少约0.35,至少约0.40,至少约0.50,至少约0.65,至少约0.60、至少约0.65、至少约0.70、至少约0.75、至少约0.80、至少约0.81、至少约0.82、至少约0.83、至少约0.84、至少约0.85、至少约0.86、至少约0.87、至少约0.88、至少约0.89、至少约0.90、至少约0.91、至少约0.92、至少约0.93、至少约0.94、至少约0.95、至少约0.96、至少约0.97、至少约0.98、至少约0.99的F分数预测早产状况。在一个实施方案中,训练算法可被配置为以0.8333的F分数预测早产状况。在另一实施方案中,训练算法可被配置为以0.9091%的F分数预测早产状况。用训练算法预测早产状况的F分数可以计算为识别的精密度和召回率(recall)的调和平均值。
训练算法可被配置为以至少约0.80、至少约0.81、至少约0.82、至少约0.83、至少约0.84、至少约0.85、至少约0.86、至少约0.87、至少约0.88、至少约0.89、至少约0.90、至少约0.91、至少约0.92、至少约0.93、至少约0.94、至少约0.95、至少约0.96、至少约0.97、至少约0.98或至少约0.99的曲线下面积(AUC)预测早产状况。在一个实施方案中,训练算法可被配置为以94.44%的AUC预测早产状况。在另一实施方案中,训练算法可被配置为以98.15%的AUC预测早产状况。AUC可以计算为与预测生物样品具有或不具有早产状况的训练算法相关的受试者工作特征(ROC)曲线的积分(例如ROC曲线下面积)。
可以调整或调谐训练算法以提高预测早产状况的准确度、PPV、灵敏度、特异性、AUC或F分数。可以通过调整训练算法的参数(例如,如本文别处所述用于对样品进行分类的一组截止值,或神经网络的权重)来调整或调谐训练算法。可以在训练过程期间或在训练过程完成之后连续地调整或调谐训练算法。
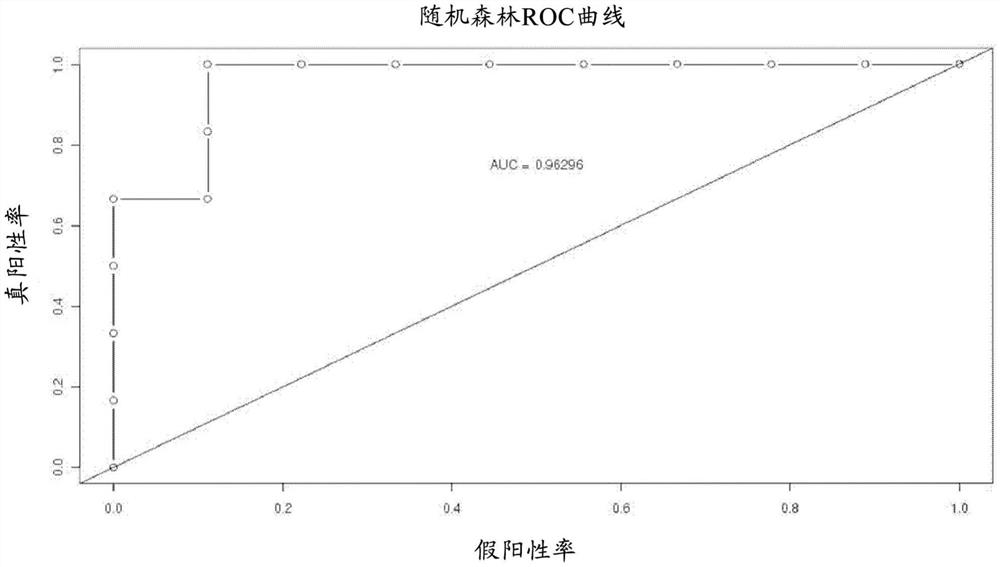
图1示出了根据一些实施方案的随机森林(RF)分类器的受试者工作特征(ROC)曲线的示例,所述随机森林(RF)分类器被配置为基于对阴道样品中微生物群体的分析来预测早产状况。在该实施例中,受试者的年龄、受试者流产的病史和平均C
训练算法包括用于预测早产状况的随机森林分类器,该分类器通过执行多个连续运行来训练。对于多个连续运行中的每一个,执行训练划分,其中随机选择至少200、250或300个生物样品作为随机森林算法的训练集(例如,独立的训练样品集),并且指定至少20个生物样品(例如,先前未被选择用于训练集)作为测试集(例如,独立测试样品集)。在一个实施例中,使用44个生物样品作为测试集。
该随机森林分类器的平均性能指标是:
平均灵敏度~83.33%
平均特异性~88.89%
平均准确度~86.67%
平均精密度~83.33%
平均F分数~0.8333
ROC曲线下平均面积(AUC)~0.963
作为随机森林分类器的有效性的进一步验证,将盲测数据集输入到经训练的随机森林分类器中,并且观察到86.67%的预测准确度。特别地,在基于F分数曲线仔细地调整概率截止值之后(例如,通过调整概率截止值以使F分数值尽可能接近1),可以针对该盲测数据实现甚至更高的准确度。
在一个实施例中,盲测数据集可以包括44个样品,并且受试者的年龄、受试者流产的病史和平均C
图2A-2G示出了原始测定数据的示例,其显示了在对应于上文表1的44个测试样品的每一个中发现的34种微生物的不同量。在该实施例中,图2A-2G中所示的原始测定数据提供了受试者的年龄、受试者流产的病史和平均C
图3示出了根据一些实施方案的随机森林(RF)分类器的受试者工作特征(ROC)曲线的示例,所述随机森林(RF)分类器被配置为基于对阴道样品中微生物群体的分析来预测早产状况。在该实施例中,受试者的年龄、受试者流产的病史和各微生物的百分比被用作训练算法的变量。
训练算法包括用于预测早产状况的随机森林分类器,该分类器通过执行多个连续运行来训练。对于多个连续运行中的每一个,执行训练划分,其中随机选择至少200、250或300个生物样品作为随机森林算法的训练集(例如,独立的训练样品集),并且指定至少20个生物样品(例如,先前未被选择用于训练集)作为测试集(例如,独立测试样品集)。在一个实施例中,使用44个生物样品作为测试集。
该随机森林分类器的平均性能指标是:
平均灵敏度~83.33%
平均特异性~100.00%
平均准确度~93.33%
平均精密度~100.00%
平均F分数~0.9091
ROC曲线下平均面积(AUC)~0.9815
作为随机森林分类器的有效性的进一步验证,将盲测数据集输入到经训练的随机森林分类器中,并且观察到93.33%的预测准确度。特别地,在基于F分数曲线仔细地调整概率截止值之后(例如,通过调整概率截止值以使F分数值尽可能接近1),可以针对该盲测数据实现甚至更高的准确度。
在一个实施例中,盲测数据集可以包括44个样品,并且受试者的年龄、受试者流产的病史和各微生物的百分比被用作训练算法的变量。表2中示出了44个测试样品的数据,包括基于对阴道样品中微生物群体的分析预测的早产状况概率(PBC)和预测的正常出生概率(正常)以及每个测试样品的实际出生结果。
图4A-4F示出了原始测定数据的示例,其显示在对应于上文表2的44个测试样品的每一个中发现的34种微生物的不同量。在该实施例中,图4A-4F中所示的原始测定数据提供了受试者的年龄、受试者流产的病史和各微生物的百分比。
在使用训练算法处理指示多个微生物群体分布的数据之后,可以以至少约86.67%的准确度预测受试者的早产。所述预测可基于所确定的多个微生物群体的单个群体的存在、不存在或相对量。
可以以至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%的准确度预测受试者中的早产。通过训练算法预测早产的准确度可以计算为(1)被正确预测为具有早产的独立测试样品和(2)被正确预测为不具有早产状况的独立测试样品在所有独立测试样品中的比例。
可以以至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%的阳性预测值(PPV)预测受试者中的早产。通过训练算法预测早产的PPV可计算为(1)正确预测为具有早产的独立测试样品和(2)错误预测为具有早产的独立测试样品的总和中正确预测为具有早产的独立测试样品的比例。PPV也可称为精密度。
可以以至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%的灵敏度预测受试者中的早产。通过训练算法预测早产的灵敏度可被计算为(1)被正确预测为具有早产的独立测试样品和(2)被错误预测为不具有早产的独立测试样品的总和中被正确预测为具有早产的独立测试样品的比例。
可以以至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、至少99%或100%的临床特异性预测受试者中的早产。通过训练算法预测早产的特异性可计算为(1)正确预测为不具有早产的独立测试样品和(2)错误预测为具有早产的独立测试样品的总和中正确预测为不具有早产的独立测试样品的比例。
可以以至少约0.05、至少约0.10、至少约0.15、至少约0.20、至少约0.25、至少约0.30、至少约0.35、至少约0.40、至少约0.50、至少约0.65、至少约0.60、至少约0.65、至少约0.70、至少约0.75、至少约0.80、至少约0.81、至少约0.82、至少约0.83、至少约0.84、至少约0.85、至少约0.86、至少约0.87、至少约0.88、至少约0.89、至少约0.90、至少约0.91、至少约0.92、至少约0.93、至少约0.94、至少约0.95、至少约0.96、至少约0.97、至少约0.98或至少约0.99的F分数预测受试者中的早产。用训练算法预测早产的F分数可以计算为识别的精密度和召回率的调和平均值。
预测早产的方法可在妊娠过程中对受试者进行多次。例如,受试者可在怀孕10-12周、20-24周和28-32周接受该方法。可以比较指示阴道样品中不同类型的多个微生物群体分布的数据,所述阴道样品随时间取样,以确定患者早产可能性的变化和/或受试者早产状况的进展或消退。
在预测受试者将具有早产时,可向受试者提供治疗性干预(例如,开出适当的疗程以预防早产)。治疗干预可包括开收缩抑制剂处方、硫酸镁处方和糖皮质激素处方。
生物样品中的微生物组分布可用于监测患者(例如,怀孕且有早产状况风险的受试者)。在这种情况下,患者的微生物组分布在治疗过程中可能改变。例如,处于PROM风险的患者的微生物组分布可能向健康受试者(即,不处于PROM风险的受试者)的微生物组分布转变。相反,例如,处于PROM风险的患者的微生物组分布可以保持不变。
受试者中早产状况的进展或消退可通过监测用于治疗受试者中早产状况的治疗过程来监测。监测可包括在两个或更多时间点评估受试者中的早产状况。评估可至少基于在两个或多个时间点中的每一个时间点处确定的多个微生物群体中的微生物单个群体的存在、不存在或相对量。
在两个或更多个时间点之间确定的多个微生物群体的微生物单个群体的存在、不存在或相对量之间的差异可指示一种或多种临床适应症,例如(i)受试者中早产状况的诊断,(ii)受试者中早产状况的预后,(iii)受试者中早产状况的进展,(iv)受试者中早产状况的消退,(v)用于治疗受试者中早产状况的治疗过程的功效,和(vi)早产状况对用于治疗受试者中早产状况的治疗过程的抗性。
在两个或多个时间点之间确定的多个微生物群体中的微生物单个群体的存在、不存在或相对量之间的差异可以指示受试者的早产状况的诊断。例如,如果在较早的时间点在受试者中未检测到早产状况,但在较晚的时间点在受试者中检测到早产状况,则该差异指示受试者中早产状况的诊断。基于受试者中早产状况的诊断的这种指示,可以做出临床行动或决定,例如,为受试者开出新的治疗干预。
在两个或多个时间点之间确定的多个微生物群体中的微生物单个群体的存在、不存在或相对量之间的差异可以指示受试者中早产状况的预后。
在两个或多个时间点之间确定的多个微生物群体中的微生物单个群体的存在、不存在或相对量之间的差异可以指示受试者中早产状况的进展。例如,如果在较早时间点和较晚时间点两者处检测到受试者中的早产状况,并且如果差异是负差异(例如,多个微生物群体的单个微生物群体的存在、不存在或相对量从较早时间点到较晚时间点增加),则该差异可指示受试者中的早产状况的进展(例如,增加的肿瘤负担、肿瘤负荷或肿瘤大小)。可基于进展的这种指示做出临床动作或决定,例如,为受试者开出新的治疗干预或切换治疗干预(例如,结束当前治疗并开出新的治疗)。
在两个或多个时间点之间确定的多个微生物群体中的单个微生物群体的存在、不存在或相对量之间的差异可以指示受试者中早产状况的消退。例如,如果在较早时间点和较晚时间点两者处检测到受试者中的早产状况,并且如果差异是正差异(例如,多个微生物群体的单个微生物群体的存在、不存在或相对量从较早时间点到较晚时间点减少),则该差异可指示受试者中早产状况的消退(例如,减少的肿瘤负担、肿瘤负荷或肿瘤大小)。可基于这种消退的指示做出临床动作或决定,例如,继续或结束针对受试者的当前治疗干预。
在两个或多个时间点之间确定的多个微生物群体中的单个微生物群体的存在、不存在或相对量之间的差异可以指示用于治疗受试者中的早产状况的治疗过程的功效。例如,如果在较早的时间点在受试者中检测到早产状况,但在较晚的时间点在受试者中检测不到早产状况,则差异可指示用于治疗受试者的早产状况的治疗过程的功效。可基于治疗受试者早产状况的治疗过程的功效的这种指征做出临床行动或决定,例如继续或结束对受试者的当前治疗干预。
在两个或多个时间点之间确定的多个微生物群体中的单个微生物群体的存在、不存在或相对量之间的差异可以指示早产状况对用于治疗受试者中早产状况的治疗过程的抗性。例如,如果在较早的时间点和较晚的时间点都在受试者中检测到早产状况,并且如果差异是负差异或零差异(例如,多个微生物群体的单个微生物群体的存在、不存在或相对量从较早的时间点到较晚的时间点增加或保持在恒定水平),并且如果在较早的时间点指示有效的治疗,则该差异可以指示用于治疗受试者中的早产状况的治疗过程的抗性(例如,增加的或恒定的肿瘤负担、肿瘤负荷或肿瘤大小)。可基于治疗受试者中早产状况的治疗过程的抗性的这种指示来出临床动作或决定,例如,结束当前治疗干预和/或切换至(例如,开处方)用于受试者的不同的新治疗干预。
在预测受试者中的早产状况之后,可以电子输出指示具有早产状况的风险或可能性的报告。报告可以在用户的电子设备的图形用户界面(GUI)上呈现。用户可以是受试者、看护者、医生、护士或其他健康护理工作者。
本公开提供被编程以实现本公开的方法的计算机控制系统。图5示出了计算机系统301,其被编程或以其他方式配置成例如(i)训练和测试训练算法,(ii)使用训练算法处理指示多个微生物群体分布的数据,(iii)确定生物样品中多个微生物群体的单个微生物群体的存在、不存在或相对量,(iv)将受试者识别为具有早产状况,或(v)以电子方式输出识别或提供受试者中早产状况的指示的报告。
计算机系统301可以调节本公开的分析、计算和生成的各个方面,例如,(i)训练和测试训练算法,(ii)使用训练算法来处理指示多个微生物群体的分布的数据,(iii)确定生物样品中多个微生物群体中的单个微生物群体的存在、不存在或相对量,(iv)识别受试者具有早产状况,或(v)以电子方式输出识别或提供所述受试者中所述早产状况的进展或消退的指示的报告。计算机系统301可以是用户的电子设备或相对于该电子设备位于远程的计算机系统。电子设备可以是移动电子设备。
计算机系统301包括中央处理单元(CPU,这里也称为“处理器”和“计算机处理器”)305,其可以是单核或多核处理器,或者用于并行处理的多个处理器。计算机系统301还包括存储器或存储器位置310(例如,随机存取存储器、只读存储器、闪存)、电子存储单元315(例如,硬盘)、用于与一个或多个其它系统通信的通信接口320(例如,网络适配器)、以及外围设备325,如高速缓存、其它存储器、数据存储器和/或电子显示适配器。存储器310、存储单元315、接口320和外围设备325通过诸如主板的通信总线(实线)与CPU305通信。存储单元315可以是用于存储数据的数据存储单元(或数据存储库)。计算机系统301可以借助通信接口320可操作地耦合到计算机网络(“网络”)330。网络330可以是互联网、因特网和/或外联网、或与因特网通信的内联网和/或外联网。
在一些情况下,网络330是电信和/或数据网络。网络330可以包括一个或多个计算机服务器,其可以启用分布式计算,例如云计算。例如,一个或多个计算机服务器可使得通过网络330(“云”)的云计算能够执行本公开的分析、计算和生成的各个方面,例如,(i)训练和测试训练算法,(ii)使用训练算法处理指示多个微生物群体的分布的数据,(iii)确定生物样品中多个微生物群体的单个微生物群体的存在、不存在或相对量,(iv)将受试者识别为具有早产状况,或(v)以电子方式输出识别或提供所述受试者中所述早产状况的进展或消退的指示的报告。这样的云计算可以由云计算平台提供,例如,Amazon Web服务(AWS)、Microsoft Azure、Google云平台和IBM云。在一些情况下,网络330借助于计算机系统301可实现对等网络,其可使耦合到计算机系统301的设备表现为客户端或服务器。
CPU 305可以包括一个或多个计算机处理器和/或一个或多个图形处理单元(GPU)。CPU 305可以执行机器可读指令序列,其可以包含在程序或软件中。指令可以存储在诸如存储器310的存储器位置中。指令可被引导到CPU 305,其可随后编程或以其他方式配置CPU 305以实施本公开的方法。由CPU 305执行的操作的实例可以包括提取、解码、执行和回写。
CPU 305可以是电路的一部分,例如集成电路。系统301的一个或多个其它组件可以包括在电路中。在一些情况下,电路是专用集成电路(ASIC)。
存储单元315可以存储文件,例如驱动程序、库和保存的程序。存储单元315可以存储用户数据,例如用户偏好和用户程序。在一些情况下,计算机系统301可以包括计算机系统301外部的一个或多个附加数据存储单元,例如位于通过内联网或因特网与计算机系统301通信的远程服务器上。
计算机系统301可以通过网络330与一个或多个远程计算机系统通信。例如,计算机系统301可以与用户的远程计算机系统通信。远程计算机系统的实例包括个人计算机(例如,便携式电脑)、平板或平板PC(例如,
本文所述的方法可以通过存储在计算机系统301的电子存储位置(例如,存储器310或电子存储单元315)上的机器(例如,计算机处理器)可执行代码来实现。机器可执行或机器可读代码可以以软件的形式提供。在使用期间,该代码可由处理器305执行。在一些情况下,代码可以从存储单元315检索并存储在存储器310上,以供处理器305随时访问。在一些情况下,可以排除电子存储单元315,并且将机器可执行指令存储在存储器310上。
代码可以被预编译并被配置成与具有适于执行代码的处理器的机器一起使用,或者可以在运行时期间被编译。代码可以以编程语言提供,所述编程语言可以被选择以使代码能够以预编译或按编译方式执行。
在此提供的系统和方法的各方面,例如计算机系统301,可以在编程中体现。本技术的各个方面可以被认为是“产品”或“制品”,其通常具有机器(或处理器)可执行代码和/或相关数据的形式,其被承载在一种机器可读介质上或被包含在一种机器可读介质中。机器可执行代码可以存储在电子存储单元上,例如存储器(例如,只读存储器、随机存取存储器、闪存)或硬盘。“存储”类型介质可包括计算机、处理器等的任何或所有有形存储器或其相关模块,例如各种半导体存储器、磁带驱动器、磁盘驱动器等,其可以在任何时间为软件编程提供非暂时性存储。软件的全部或部分有时可通过因特网或各种其它电信网络进行通信。这样的通信例如可以使得能够将软件从一台计算机或处理器加载到另一台计算机或处理器中,例如从管理服务器或主机加载到应用服务器的计算机平台中。因此,可承载软件单元的另一类型的介质包括光、电和电磁波,诸如通过有线和光陆线网络以及通过各种空中链路跨本地设备之间的物理接口使用的。承载这种波的物理元件,例如有线或无线链路、光链路等,也可以被认为是承载软件的介质。如本文所使用,除非限于非暂时性的、有形的“存储”介质,否则诸如计算机或机器“可读介质”的术语是指参与向处理器提供指令以供执行的任何介质。
因此,诸如计算机可执行代码的机器可读介质可以采取多种形式,包括但不限于有形存储介质、载波介质或物理传输介质。非易失性存储介质包括例如光学或磁盘,例如任何计算机等中的任何存储设备,例如可用于实现附图中所示的数据库等。易失性存储介质包括动态存储器,例如这种计算机平台的主存储器。有形传输介质包括同轴电缆;铜线和光纤,包括构成计算机系统内总线的导线。载波传输介质可以采取电信号或电磁信号、或声波或光波的形式,例如在射频(RF)和红外(IR)数据通信期间产生的那些。因此,计算机可读介质的常见形式例如包括:软盘、软磁盘、硬盘、磁带、任何其它磁介质、CD-ROM、DVD或DVD-ROM、任何其它光学介质、穿孔卡纸带、具有孔图案的任何其它物理存储介质、RAM、ROM、PROM和EPROM、FLASH-EPROM、任何其它存储芯片或盒、传输数据或指令的载波、传输这种载波的电缆或链路、或计算机可从中读取编程代码和/或数据的任何其它介质。许多这些形式的计算机可读介质可以参与将一个或多个指令的一个或多个序列承载到处理器以供执行。
计算机系统301可以包括电子显示器335或与电子显示器335通信,所述电子显示器包括用户界面(UI)340,用于提供例如,(i)指示训练算法的训练和测试的可视显示,(ii)指示多个微生物群体的分布的数据的可视显示,(iii)生物样品中多个微生物群体中单个微生物群体的确定的存在、不存在或相对量,(iv)具有早产状况的受试者的识别,或(v)识别或提供受试者中早产状况的进展或消退的指示的电子报告。UI的实例包括但不限于图形用户界面(GUI)和基于Web的用户界面。
本公开的方法和系统可以通过一个或多个算法来实现。算法可以在由中央处理单元305执行时通过软件来实现。该算法可以,例如,(i)训练和测试训练算法,(ii)使用训练算法来处理指示多个微生物群体的分布的数据,(iii)确定生物样品中多个微生物群体中的单个微生物群体的存在、不存在或相对量,(iv)将受试者识别为具有早产状况,或(v)以电子方式输出识别或提供所述受试者中所述早产状况的进展或消退的指示的报告。
实施例
实施例1-早产状况的预测
在实施例中,怀孕6个月的患者呈现出以下风险因素:社会经济状况低,过去在她怀孕期间出血的历史,以及在以前的怀孕期间早产的历史。医生需要识别患者早产的可能性,并建议使用本文提供的方法和系统来预测早产的可能性。获取患者阴道流体样品,以分析阴道微生物组。对阴道样品进行处理,以产生指示阴道样品中不同类型的多个微生物群体的分布的数据。训练算法识别不同类型的微生物,并识别单个微生物群体的存在、不存在或相对量,所述微生物例如为乳杆菌属、埃希氏菌属、普雷沃氏菌属、肠球菌属、念珠菌属、葡萄球菌属和疱疹病毒。训练算法预测受试者具有大约88%的早产风险。训练算法基于阴道样品中各微生物群体的存在、不存在或相对量,以98.15%的准确度预测该风险百分比。系统输出电子报告,指示受试者中存在88%的早产状况风险。医生收到电子报告并开出补充孕酮给患者作为预防措施,以预防在妊娠后期发生的早产状况。
实施例2-早产风险的预测
在本实施例中,通过本发明方法评估在妊娠的不同时间点显示威胁性早产迹象的四个孕妇(即受试者#1-4)的早产风险。具体地,如实施例1所示,从每个受试者获得阴道流体样品并进行处理。如实施例1所示,使用准确度为98.15%的训练算法预测受试者早产状况的风险。表3示出了预测的早产状况概率(PBC)和基于对阴道样品中微生物群体的分析预测的出生结果以及每个受试者的实际出生结果的数据。
表3
虽然在此已经示出和描述了本发明的优选实施例,但是对于本领域技术人员来说显而易见的是,这些实施例仅作为示例提供。本发明不受说明书中提供的具体实施例的限制。尽管已经参照上述说明书描述了本发明,但是本文实施例的描述和说明不意味着以限制的意义来解释。在不背离本发明的情况下,本领域技术人员可以想到许多变化、改变和替换。此外,应当理解,本发明的所有方面不限于本文所述的具体描述、配置或相对比例,其取决于各种条件和变量。应当理解,在实施本发明时可以采用本文所述的本发明实施例的各种替代方案。因此,可以预期本发明还将包括任何这样的替代、修改、变化或等同物。下面的权利要求旨在限定本发明的范围,并且这些权利要求范围内的方法和结构及其等同物包括在内。
- 用于预测早产状况的方法、系统和试剂盒
- 用于预测天气预报状况、环境预报状况和/或地质预报状况的影响的方法和系统
