一种脂质纳米颗粒组合物以及由其制备的药物递送系统
文献发布时间:2023-06-19 19:28:50
技术领域
本发明属于生物医药制剂技术领域,尤其涉及一种含有新的脂质化合物的脂质纳米颗粒, 以及由其制备得到的携带活性成分的药物组合物或药物递送系统,比如mRNA疫苗。
背景技术
不同类型的核酸制剂正被研发用于治疗各种重大疾病如传染性疾病、癌症、罕见病等。 该类核酸制剂包括:DNA、反义核酸(ASO)、小干扰RNA(siRNA)、微小RNA(miRNA)、 小激活RNA(saRNA)、信使RNA(mRNA)、适配体(aptamer)、核酶(ribozyme)等。 其中,mRNA疫苗在安全性、快速制备和免疫原性方面具有颠覆性的优势。基于mRNA修饰 和递送工具的发展,一旦获得病毒抗原序列,可在数周内迅速设计和制造具有临床规模的 mRNA疫苗,可以实现标准化生产,使其在应对大流行暴发方面非常具有吸引力。且mRNA 疫苗不存在减毒疫苗潜在的逆转危险;不存在灭活疫苗的恢复突变问题。在免疫原性上, mRNA疫苗能够诱导B细胞和T细胞免疫应答,能引起免疫记忆效果,传递更多有效抗原,并 且能一次表达多个抗原。此外,mRNA只需要穿过细胞膜在细胞质中就可以高效表达抗原蛋 白;mRNA没有基因整合到基因组中的风险。再次,mRNA被翻译成蛋白质后易被降解,其 瞬时表达的特性不仅确保了mRNA药物的安全性,而且使其剂量可控,避免了疫苗药物长期 暴露引起的抗原免疫耐受(对特定抗原无反应的状态)。
但是,一方面核酸制剂带负电而且多数分子量较大很难直接进入细胞内,另一方面,RNA 不稳定,在引入体内的过程中极易被核酸酶降解,从而失去生物功能。因此,开发高效安全 并且通用的核酸递送系统,是核酸药物转化过程中亟待解决的问题。
目前,核酸的递送方法包括化学修饰、生物偶联技术、纳米载体技术、基于脂质的制剂、 外泌体、球形核酸、DNA纳米结构、刺激-响应聚合物纳米材料等。其中,较为成熟的核酸递 送载体是脂质纳米颗粒(LNP),并且2018年LNP包裹的siRNA药物Onpattro(patisiran)获批上 市,2021年LNP包裹的mRNA疫苗已经正式获得FDA审批应用于新冠疫情防控,临床结果显 示出较高的有效性,且目前无严重不良反应。
该类脂质制剂的主要成分包括:阳离子/可电离脂质、辅助型脂质、胆固醇和聚乙二醇- 脂质结合物。在这四种脂质成分中,阳离子脂质带电荷的头部能够与带负电的核酸结合,同 时也能与细胞膜上的磷脂分子结合,在核酸包裹和膜融合过程中都发挥着关键的作用。考虑 到永久性的阳离子脂质的潜在毒性,可电离阳离子脂质的脂质纳米颗粒存在更大的应用价值。
可电离的阳离子脂质包括三个重要的结构组成部分:含胺基的亲水极性头部;疏水脂质 链;负责连接极性头部和非极性尾部的连接链。目前,商业化的可电离阳离子脂质主要有MC3 系列和用于新冠mRNA疫苗的ALC-0315、SM-102。其中,MC3具有很强的肝靶向,对于可能 具有潜在肝毒性的核酸制剂其应用将会受限,而且MC3是针对分子量较小的siRNA递送开发 的,对于分子量较大的核酸制剂其负载量可能存在限制。ALC-0315和SM-102的递送效率需要 进一步提高。因此,为了进一步推进我国脂质制剂的发展及其在核酸药物递送等方面的应用, 我们需要开发出新的可电离的阳离子脂质,并筛选及优化新的纳米递送系统实现核酸的安全、 高效递送,例如实现mRNA疫苗成分安全高效的递送。
发明内容
为解决活性成分,例如核酸(例如mRNA分子),生物应用时存在的结构不稳定,易被核酸酶降解以及入胞困难的问题,需要开发新的递送技术。此外,现有递送技术中普遍存在溶酶体逃逸差,递送效率较低的问题,本发明基于新合成的可电离阳离子脂质分子,形成多种功能多样性的以脂质为基础的递送系统。
本发明的第一方面,提供一种脂质纳米颗粒组合物,所述脂质纳米颗粒组合物中含有脂 质纳米颗粒,脂质纳米颗粒中包含:式I的可电离阳离子脂质分子。
根据本发明,所述脂质纳米颗粒组合物,进一步包含其他脂质分子。所述其他脂质分子 可以是本领域中用于构建脂质纳米颗粒已知或常规使用的脂质分子,包括但不限于中性脂质 分子、胆固醇类脂质分子、PEG化的脂质分子。
根据本发明,所述脂质纳米颗粒组合物,进一步包含活性成分,所述活性成分可以是小 分子化合物、核酸、蛋白质、多肽等。所述活性成分位于脂质纳米颗粒中。所述核酸包括但 不限于DNA、反义核酸(ASO)、小干扰RNA(siRNA)、微小RNA(miRNA)、小激活RNA(saRNA)、信使RNA(mRNA)、适配体(aptamer)等。
根据本发明,所述脂质纳米颗粒组合物中,脂质纳米颗粒中含有占其总体脂质分子 30-60mol%的式I的脂质分子,优选32-55mol%,进一步优选34-46mol%。
根据本发明,所述脂质纳米颗粒组合物中,脂质纳米颗粒中可以含有占其总体脂质分子 5-30mol%的中性脂质分子,优选8-20mol%,进一步优选9-16mol%。
根据本发明,所述脂质纳米颗粒组合物中,脂质纳米颗粒中可以含有占其总体脂质分子 30-50mol%的胆固醇类脂质分子,优选35-50mol%,进一步优选37-49mol%。
根据本发明,所述脂质纳米颗粒组合物中,脂质纳米颗粒中可以含有占其总体脂质分子 0.4-10mol%的PEG化的脂质分子,优选0.5-5mol%,进一步优选1.3-2.7mol%。
根据本发明,当活性成分为核酸时,所述脂质纳米颗粒组合物中脂质分子的总质量与核 酸质量之比为5-20:1。
根据本发明,式I的可电离阳离子脂质分子的结构式为
Q为经取代或未取代的直链C2-20亚烷基,所述亚烷基的1个或1个以上C原子任选被独立 地选自O、S和N的杂原子所替换;或,Q为经取代或未取代、饱和或不饱和的4-6元环,所述 4-6元环的环原子任选含有1个或1个以上独立地选自O、S、N的杂原子;所述取代的取代基团 选自卤素、-OH、直链或支链的C1-20烷基、直链或支链的C1-20烷氧基、直链或支链的C2-20 烯基、直链或支链的C2-20炔基、-CH
R
条件是,R
R
R
n选自1~8的整数,m选自0~8的整数,n和m彼此独立,可以相同也可以不同;
当R
在本发明的优选实施方式中,Q为经取代或未取代的直链C2-20亚烷基,所述亚烷基的1 个或1个以上C原子任选被独立地选自O、S和N的杂原子所替换;
优选,Q为
优选,Q为
在本发明的一些实施例中,所述饱和或不饱和的4-6元环为哌嗪基或环己基。
在本发明的优选实施方式中,R
在本发明的优选实施方式中,n选自4~8的整数,m选自4~8的整数。
在本发明的优选实施方式中,所述式I化合物为下式A、B、C或D:
其中每个n
其中每个n
其中每个n
其中每个n
在本发明的一些具体实施方式中,所述式I化合物选自表1所示的以下化合物:
表1
/>
/>
/>
/>
/>
/>
/>
/>
/>
根据本发明,式I的脂质分子在脂质纳米颗粒的脂质中的摩尔百分比为30-60mol%,例 如32-55mol%,例如可以为30mol%,31mol%,32mol%,33mol%,34mol%,35mol%,36mol%, 37mol%,38mol%,39mol%,40mol%,41mol%,42mol%,43mol%,44mol%,45mol%,46mol%, 47mol%,48mol%,49mol%,50mol%,51mol%,52mol%,53mol%,54mol%,55mol%等。
根据本发明,所述中性脂质分子是不带电脂质分子或两性离子型脂质分子,例如磷脂酰 胆碱类化合物,或/和磷脂酰乙醇胺类化合物。
磷脂酰胆碱类化合物的结构如式E所示:
中性脂质分子的实例包括但不限于5-十七基苯-1,3-二醇(间苯二酚)、二棕榈酰基磷脂酰 胆碱(DPPC)、二硬脂酰基磷脂酰胆碱(DSPC)、磷酸胆碱(DOPC)、二肉豆蔻酰基磷脂酰胆碱 (DMPC)、磷脂酰胆碱(PLPC)、1,2-二硬脂酰基-sn-甘油-3-磷酸胆碱(DAPC)、磷脂酰乙醇胺(PE)、 卵磷脂酰胆碱(EPC)、二月桂酰基磷脂酰胆碱(DLPC)、二肉豆蔻酰基磷脂酰胆碱(DMPC)、1- 肉豆蔻酰基-2-棕榈酰基磷脂酰胆碱(MPPC)、1-棕榈酰基-2-肉豆蔻酰基磷脂酰胆碱(PMPC)、 1-棕榈酰基-2-硬脂酰基磷脂酰胆碱(PSPC)、1,2-二花生酰基-sn-甘油-3-磷酸胆碱(DBPC)、1- 硬脂酰基-2-棕榈酰基磷脂酰胆碱(SPPC)、1,2-二十碳烯酰基-sn-甘油-3-磷酸胆碱(DEPC)、棕 榈酰油酰基磷脂酰胆碱(POPC)、溶血磷脂酰胆碱、二油酰基磷脂酰乙醇胺(DOPE)、二硬脂酰 基磷脂酰乙醇胺(DSPE)、二肉豆蔻酰基磷脂酰乙醇胺(DMPE)、二棕榈酰基磷脂酰乙醇胺 (DPPE)、棕榈酰油酰基磷脂酰乙醇胺(POPE)、溶血磷脂酰乙醇胺以及它们的组合。
在一个实施方案中,中性脂质分子可选自由以下组成的组:二硬脂酰基磷脂酰胆碱 (DSPC)、二油酰基磷脂酰乙醇胺(DOPE)和二硬脂酰基磷脂酰乙醇胺(DSPE)。在另一个实施方 案中,中性脂质分子可为二肉豆蔻酰基磷脂酰乙醇胺(DMPE)。在另一个实施方案中,中性脂 质分子可为二棕榈酰基磷脂酰胆碱(DPPC)。
根据本发明,中性脂质分子在脂质纳米颗粒的脂质中的摩尔百分比为5-30mol%,例如 8-20mol%,例如可以为8mol%,9mol%,10mol%,11mol%,12mol%,13mol%,14mol%, 15mol%,16mol%,17mol%,18mol%,19mol%,20mol%。
根据本发明,胆固醇类脂质分子是指甾醇以及含有甾醇部分的脂质,包括但不限于胆固 醇,5-十七基间苯二酚,粪甾醇,谷甾醇,麦角甾醇,菜油甾醇,豆甾醇,菜子甾醇,番茄 次碱,番茄碱,熊果酸,α-生育酚及其混合物、胆固醇半琥珀酸酯。在一个实施方案中,胆固醇类脂质分子为胆固醇(CHOL)。在一个实施方案中,胆固醇类脂质分子为胆固醇半琥 珀酸酯。
根据本发明,胆固醇类脂质分子在脂质纳米颗粒的脂质中的摩尔百分比为30-50mol%, 例如可以为30mol%,31mol%,32mol%,33mol%,34mol%,35mol%,36mol%,37mol%,38mol%,39mol%,40mol%,41mol%,42mol%,43mol%,44mol%,45mol%,46mol%,47mol%, 48mol%,49mol%,50mol%等。
根据本发明,PEG化的脂质分子包含脂质部分和基于PEG的聚合物部分。在一些实施方 案中,脂质部分可衍生自二酰基甘油或二酰基甘油酰胺(diacylglycamide)、包括包含具有独立 地包含约C4至约C30饱和或不饱和碳原子的烷基链长度的二烷基甘油或二烷基甘油酰胺基 团的那些,其中该链可包含一个或多个官能团,诸如酰胺或酯。在一些实施方案中,烷基链 长度包含约C10至C20。二烷基甘油或二烷基甘油酰胺基团可还包含一个或多个取代的烷基。 链长可为对称或不对称的。除非另外指明,否则如本文所用,术语“PEG”意指任何聚乙二 醇或其他聚亚烷醚聚合物。在一个实施方案中,PEG部分为乙二醇或环氧乙烷的任选地取代 的直链或支链聚合物。在某些实施方案中,PEG部分可被例如一个或多个烷基、烷氧基、酰 基、羟基或芳基取代。在一个实施方案中,PEG部分包括PEG共聚物,诸如PEG-聚氨酯或 PEG-聚丙烯(参见例如J.Milton Harris,Poly(ethylene glycol)chemistry:biotechnical and biomedical applications(1992));或者,PEG部分不包括PEG共聚物,例如其可为PEG单聚物。 在一个实施方案中,PEG的分子量为约130至约50,000,在子实施方案中,为约150至约30,000, 在子实施方案中,为约150至约20,000,在子实施方案中,为约150至约15,000,在子实施 方案中,为约150至约10,000,在子实施方案中,为约150至约6,000,在子实施方案中,为 约150至约5,000,在子实施方案中,为约150至约4,000,在子实施方案中,为约150至约 3,000,在子实施方案中,为约300至约3,000,在子实施方案中,为约1,000至约3,000,并 且在子实施方案中,为约1,500至约2,500。在某些实施方案中,PEG为“PEG 2000”,其平 均分子量为约2,000道尔顿。在本发明的一些实施方式中,PEG在本文中由下式
在一些实施方案中,PEG化的脂质分子可表示为“脂质部分-PEG-数均分子量”或者“PEG- 脂质部分”或者“PEG-数均分子量-脂质部分”,所述脂质部分是二酰基甘油或二酰基甘油酰 胺,选自二月桂酰甘油、二肉豆蔻酰甘油、二棕榈酰甘油、二硬脂酰甘油、二月桂基甘油酰 胺、二肉豆蔻基甘油酰胺、二棕榈酰甘油酰胺、二硬脂酰甘油酰胺、1,2-二硬脂酰基-sn-甘油 -3-磷酸乙醇胺、1,2-二肉豆蔻酰基-sn-甘油-3-磷酸乙醇胺;PEG的数均分子量为约130至约 50,000,例如约150至约30,000,约150至约20,000,为约150至约15,000,为约150至约 10,000,为约150至约6,000,为约150至约5,000,为约150至约4,000,为约150至约3,000, 为约300至约3,000,为约1,000至约3,000,为约1,500至约2,500,例如约2000。
在一些实施方案中,PEG化的脂质分子可选自PEG-二月桂酰甘油、PEG-二肉豆蔻酰甘 油(PEG-DMG)、PEG-二棕榈酰甘油、PEG-二硬脂酰甘油(PEG-DSPE)、PEG-二月桂基甘油酰胺、PEG-二肉豆蔻基甘油酰胺、PEG-二棕榈酰甘油酰胺和PEG-二硬脂酰甘油酰胺、PEG-胆固醇(1-[8'-(胆甾-5-烯-3[β]-氧基)甲酰胺基-3',6'-二氧杂辛基]氨基甲酰基-[ω]-甲基-聚(乙二醇)、 PEG-DMB(3,4-二-十四氧基苯甲基-[ω]-甲基-聚(乙二醇)醚)、1,2-二肉豆蔻酰基-sn-甘油-3-磷酸 乙醇胺-N-[甲氧基(聚乙二醇)-2000](DMG-PEG2000)、1,2-二硬脂酰基-sn-甘油-3-磷酸乙醇胺 -N-[甲氧基(聚乙二醇)-2000](DSPE-PEG2000)、1,2-二硬脂酰基-sn-甘油-甲氧基聚乙二醇 (DSG-PEG2000)、聚(乙二醇)-2000-二甲基丙烯酸酯(DMA-PEG2000)和1,2-二硬脂酰氧基丙基 -3-胺-N-[甲氧基(聚乙二醇)-2000](DSA-PEG2000)。在一个实施方案中,PEG化的脂质分子可 为DMG-PEG2000。在一些实施方案中,PEG化的脂质分子可为DSG-PEG2000。在一个实施 方案中,PEG化的脂质分子可为DSPE-PEG2000。在一个实施方案中,PEG化的脂质分子可 为DMA-PEG2000。在一个实施方案中,PEG化的脂质分子可为C-DMA-PEG2000。在一个 实施方案中,PEG化的脂质分子可为DSA-PEG2000。在一个实施方案中,PEG化的脂质分 子可为PEG2000-C11。在一些实施方案中,PEG化的脂质分子可为PEG2000-C14。在一些实 施方案中,PEG化的脂质分子可为PEG2000-C16。在一些实施方案中,PEG化的脂质分子可 为PEG2000-C18。
根据本发明,PEG化的脂质分子在脂质纳米颗粒的脂质中的摩尔百分比为0.4-10mol%, 例如0.5-5mol%,例如可以为0.4mol%,0.5mol%,0.6mol%,0.7mol%,0.8mol%,0.9mol%, 1.0mol%,1.1mol%,1.2mol%,1.3mol%,1.4mol%,1.5mol%,1.6mol%,1.7mol%,1.8mol%, 1.9mol%,2.0mol%,2.1mol%,2.2mol%,2.3mol%,2.4mol%,2.5mol%,2.6mol%,2.7mol%, 2.8mol%,2.9mol%,3.0mol%,3.1mol%,3.2mol%,3.3mol%,3.4mol%,3.5mol%,3.6mol%, 3.7mol%,3.8mol%,3.9mol%,4.0mol%,4.1mol%,4.2mol%,4.3mol%,4.4mol%,4.5mol%, 4.6mol%,4.7mol%,4.8mol%,4.9mol%,5.0mol%等。
在本发明的一些实施方式中,所述脂质纳米颗粒中含有式C所示的脂质分子,中性脂质 分子、胆固醇类脂质分子、PEG化的脂质分子,其中:
式C
中性脂质分子选自式E所示的磷脂酰胆碱类化合物
胆固醇类脂质分子选自胆固醇、胆固醇半琥珀酸酯。胆固醇类脂质分子占脂质纳米颗粒 中脂质的摩尔百分比为30-50mol%,优选为35-50mol%,更优选为37-49mol%。
PEG化的脂质分子表示为“脂质部分-PEG-数均分子量”,所述脂质部分是二酰基甘油 或二酰基甘油酰胺,选自二月桂酰甘油、二肉豆蔻酰甘油、二棕榈酰甘油、二硬脂酰甘油、 二月桂基甘油酰胺、二肉豆蔻基甘油酰胺、二棕榈酰甘油酰胺、二硬脂酰甘油酰胺、1,2-二硬 脂酰基-sn-甘油-3-磷酸乙醇胺、1,2-二肉豆蔻酰基-sn-甘油-3-磷酸乙醇胺;PEG的数均分子量 为130~50,000,例如150~30,000,150~20,000,150~15,000,150~10,000,150~6,000,150~5,000, 150~4,000,150~3,000,300~3,000,1,000~3,000,1,500~2,500,约2000。PEG化的脂质分子 占脂质纳米颗粒中脂质的摩尔百分比为0.5-5mol%,优选为1.3-2.7mol%。
在本发明的一个实施方式中,所述核酸是mRNA。
根据本发明,所述mRNA从5’端至3’端可以包含5’帽结构、5’UTR,开放阅读框(ORF),3’UTR和poly-A尾。
根据本发明,所述帽结构可以是Cap1结构,Cap2结构或Cap3结构。在本发明的一个实施 方式中,所述帽结构是Cap1结构。
根据本发明,所述5’UTR可以包含β-珠蛋白或α-珠蛋白的5’UTR或其同源物、片段。在本 发明的一些实施方式中,所述5’UTR包含与SEQ ID NO:6所示的β-珠蛋白的5’UTR核苷酸序列 至少60%,至少70%,至少75%,至少80%,至少85%,至少90%,至少95%,至少96%, 至少97%,至少98%,至少99%或约100%同源性的核苷酸序列。在本发明的一个具体实施方 式中,所述5’UTR包含SEQ ID NO:6所示的β-珠蛋白的5’UTR核苷酸序列。
在本发明的一些实施方案中,所述5'UTR还包含Kozak序列。在本发明的一个实施方式中, 所述Kozak序列为GCCACC。
根据本发明,所述3’UTR可以包含β-珠蛋白或α-珠蛋白的3’UTR或其同源物、片段,或片 段的组合。在本发明的一些实施方式中,所述3’UTR包含与SEQ ID NO:7所示α2-珠蛋白3’UTR 的片段至少60%,至少70%,至少75%,至少80%,至少85%,至少90%,至少95%,至少 96%,至少97%,至少98%,至少99%或约100%同源性的核苷酸序列。在本发明的另一些实 施方式中,所述3’UTR包含2个首尾相连的与SEQ ID NO:7所示的α2-珠蛋白3’UTR的片段至少 60%,至少70%,至少75%,至少80%,至少85%,至少90%,至少95%,至少96%,至少 97%,至少98%,至少99%或约100%同源性的核苷酸序列。在本发明的一个具体实施方式中, 所述3’UTR包含2个首尾相连的SEQ ID NO:7所示的核苷酸序列。
根据本发明,所述poly-A尾的长度可以为50-200个核苷酸,优选为100-150个核苷酸,例 如110-120个核苷酸,例如约110个核苷酸,约120个核苷酸,约130个核苷酸,约140个核苷酸, 约150个核苷酸。
在本发明的一个实施方案中,所述开放阅读框(ORF)是编码2019-nCov的S蛋白突变体 的开放阅读框(ORF),其核酸序列是与SEQ ID NO:8所示核苷酸序列至少60%,至少70%, 至少75%,至少80%,至少85%,至少90%,至少95%,至少96%,至少97%,至少98%, 至少99%或约100%同源性的核苷酸序列。该ORF翻译后的S蛋白突变体的氨基酸序列是由从N 端向C端直接连接的SEQ ID NO:2所示氨基酸序列和SEQ ID NO:3所示氨基酸序列组成。在本 发明的一个具体实施方式中,所述S蛋白突变体的开放阅读框(ORF)的核苷酸序列如SEQ ID NO:8所示。
在本发明的一个实施方案中,所述mRNA包含与SEQ ID NO:9所示的核苷酸序列至少 60%,至少70%,至少75%,至少80%,至少85%,至少90%,至少95%,至少96%,至 少97%,至少98%,至少99%或约100%同源性的核苷酸序列。在本发明的一个具体实施方 式中,所述mRNA包含SEQ ID NO:9所示的核苷酸序列。
可采用本领域已知的方法制备本发明的mRNA。在本发明的一些实施方式中,可以人工 合成编码mRNA的核酸序列,将该序列克隆到载体中,构建体外转录用质粒。将构建的质粒 转化到宿主菌中培养扩增,提取质粒。将提取的质粒使用紧邻着polyA尾后面的限制性内切 酶酶切消化成线性分子。以制备的线性化质粒分子为模板,使用体外转录法制备mRNA。可 以在体外转录过程中添加帽结构类似物,直接得到具有帽结构的mRNA;也可以在体外转录 结束后,使用加帽酶和二甲基转移酶给mRNA添加上帽结构。可采用本领域常规的方法纯化 得到的mRNA,例如化学沉淀法、磁珠法、亲和层析法等。
根据本发明,所述mRNA中的一个或多个核苷酸可以是经修饰的。例如,所述mRNA中的一个或多个核苷酸(例如所有核苷酸)可以各自独立替换为天然存在的核苷酸类似物或人工 合成的核苷酸类似物,例如选自假尿苷(pseudouridine)、2-硫尿苷(2-thiouridine)、5-甲基尿苷 (5-methyluridine)、5-甲基胞苷(5-methylcytidine)、N6-甲基腺苷(N6-methyladenosine)、N1-甲基 假尿苷(N1-methylpseudouridine)等。
本发明还提供一种mRNA疫苗,所述疫苗中含有脂质纳米颗粒,所述脂质纳米颗粒中含 有式C所示的脂质分子,中性脂质分子、胆固醇类脂质分子、PEG化的脂质分子和编码2019-nCoV S蛋白突变体的mRNA,所述mRNA包含与SEQ ID NO:9所示的核苷酸序列至少60%,至少70%,至少75%,至少80%,至少85%,至少90%,至少95%,至少96%,至 少97%,至少98%,至少99%或约100%同源性的核苷酸序列;脂质分子的总质量与mRNA 的质量比为5-20:1;其中:
式C
中性脂质分子选自式E所示的磷脂酰胆碱类化合物
胆固醇类脂质分子选自胆固醇、胆固醇半琥珀酸酯。胆固醇类脂质分子占脂质纳米颗粒 中脂质的摩尔百分比为37-49mol%;
PEG化的脂质分子表示为“脂质部分-PEG-数均分子量”,所述脂质部分是二酰基甘油或 二酰基甘油酰胺,选自二月桂酰甘油、二肉豆蔻酰甘油、二棕榈酰甘油、二硬脂酰甘油、二 月桂基甘油酰胺、二肉豆蔻基甘油酰胺、二棕榈酰甘油酰胺、二硬脂酰甘油酰胺、1,2-二硬脂 酰基-sn-甘油-3-磷酸乙醇胺、1,2-二肉豆蔻酰基-sn-甘油-3-磷酸乙醇胺;PEG的数均分子量为 约130至约50,000,例如约150至约30,000,约150至约20,000,为约150至约15,000,为约150 至约10,000,为约150至约6,000,为约150至约5,000,为约150至约4,000,为约150至约3,000, 为约300至约3,000,为约1,000至约3,000,为约1,500至约2,500,例如约2000。PEG化的脂质分 子占脂质纳米颗粒中脂质的摩尔百分比为1.3-2.7mol%。
在本发明的一些实施方式中,式C所示的脂质分子、中性脂质分子、胆固醇和PEG化的 脂质分子摩尔比为35:15:48.5:1.5。
在本发明的一些实施方式中,式C所示的脂质分子、中性脂质分子、胆固醇和PEG化的 脂质分子摩尔比为40:10:48.5:1.5。
在本发明的一些实施方式中,式C所示的脂质分子、中性脂质分子、胆固醇和PEG化的 脂质分子摩尔比为45:15:38.5:1.5。
在本发明的一个实施方式中,式C脂质分子为化合物II-37。
在本发明的一个实施方式中,所述中性脂质分子是DOPE和/或DSPC。
在本发明的一个实施方式中,所述PEG化的脂质分子是DMG-PEG2000和/或 DSPE-PEG2000。
本发明还提供式I的可电离阳离子脂质分子的制备方法。
本发明的可电离脂质化合物可以采用本领域已有的方法进行合成,例如,使一当量或一 当量以上的胺与一当量或一当量以上的环氧基封端化合物在合适的条件下反应形成。可电离 脂质化合物的合成是在有或无溶剂的情况下进行,并且所述合成可在25-100℃范围内的较高 温度下进行。可任选纯化制得的可电离脂质化合物。举例来说,可纯化可电离脂质化合物的 混合物,得到特定的可电离脂质化合物。或可纯化混合物,得到特定立体异构体或区域异构 体。所述环氧化物可以商业购买,或者合成制备。
在本发明的一些实施方式中,本发明的可电离脂质化合物可以采用如下的一般制备方法 进行制备。
式A、B、C或D
步骤1:还原
在还原剂的存在下,将化合物A1的羧基还原成羟基以获得化合物A2。还原剂的实例包括 但不限于氢化铝锂、二异丁基氢化铝等。反应所用溶剂的实例包括但不限于醚类(如乙醚、 四氢呋喃和二氧六环等),卤代烃(如氯仿、二氯甲烷和二氯乙烷等),烃类(如正戊烷、 正己烷、苯和甲苯等),及这些溶剂的两种或以上形成的混合溶剂。
步骤2:氧化
在氧化剂的存在下,将化合物A2的羟基氧化成醛基以获得化合物A3。氧化剂的实例包括 但不限于2-碘酰基苯甲酸(IBX)、氯铬酸吡啶(PCC)、二氯铬酸吡啶盐(PDC)、戴斯-马丁氧化 剂、二氧化锰等。反应所用溶剂的实例包括但不限于卤代烃(如氯仿、二氯甲烷和二氯乙烷 等),烃类(如正戊烷、正己烷、苯和甲苯等),腈类(例如乙腈等),及这些溶剂的两种 或以上形成的混合溶剂。
步骤3:卤代-还原
首先在酸性条件下,将化合物A3的醛α-氢与卤代试剂发生卤代反应以获得α-卤代醛中间 体,然后在还原剂存在下,将α-卤代醛的醛基还原成羟基以获得化合物A4。提供酸性条件的 实例包括但不限于DL-脯氨酸。卤代试剂的实例包括但不限于N-氯代丁二酰亚胺(NCS)和N- 溴代丁二酰亚胺(NBS)。还原剂的实例包括但不限于硼氢化钠、氰基硼氢化钠和三乙酰氧基硼 氢化钠。
步骤4:环氧化
将化合物A4在碱的存在下进行分子内的亲核取代反应以获得环氧化合物A5。碱的实例包 括但不限于碱金属的氢氧化物或氢化物,例如氢氧化钠、氢氧化钾和氢化钠。反应所用溶剂 的实例包括但不限于二氧六环和水的混合物。
步骤5:开环反应
使化合物A5与胺(例如N,N-二(2-氨基乙基)甲胺)发生开环反应以获得最终化合物。反 应用溶剂的实例包括但不限于乙醇、甲醇、异丙醇、四氢呋喃、三氯甲烷、己烷、甲苯、乙 醚等。
所述制备方法中的原料A1可以商购也可以采用常规方法合成。
本发明还提供制备脂质纳米颗粒组合物的方法。
根据本发明,所述制备方法,包括:将各脂质分子按摩尔比用有机溶剂溶解制成混合脂 质的溶液,以混合脂质的溶液为有机相,以被递送物(例如核酸)的水溶液为水相,混合有 机相和水相,制备脂质纳米颗粒。可以采用包括但不限于喷雾干燥、单一和双重乳液溶剂蒸 发、溶剂萃取、相分离、纳米沉淀、微流控、简单和复杂凝聚以及所属领域的普通技术人员 熟知的其它方法来制备脂质纳米颗粒。
在一些实施方式中,所述有机溶剂是醇,例如乙醇。
在一些实施方式中,所述有机相和水相的体积比为(2~4):1。
在一些实施方案中,采用微流控平台制备纳米颗粒。
根据本发明,所述制备方法还进一步包括,分离和纯化得到所述脂质纳米颗粒的步骤。
根据本发明,所述制备方法还进一步包括,冻干所述脂质纳米颗粒的步骤。
本发明式I的可电离脂质分子结构中含有两个相邻的顺式双键,使其在后续应用于递送系 统包裹活性物质(例如核酸如mRNA)时,具有较高的包封率,较好的细胞转染率;此外, 在制备脂质纳米颗粒时,得到的脂质纳米颗粒粒径更均匀。本发明的可电离脂质化合物尤其 适合于制备实心结构的纳米颗粒。
此外,对于本发明的mRNA疫苗,由于本发明的mRNA具有高的翻译效率和稳定性,其所编码的S蛋白突变体具有高的稳定性,加之本发明脂质纳米颗粒包封率高、粒径更均匀,具 有较好的细胞转染率,本发明的mRNA疫苗具有高的mRNA包封率、载药量,细胞转染效率,高效和稳定的体内翻译,抗原稳定性,免疫效果好。
本发明中脂质纳米颗粒的粒径范围在1nm到1000nm范围,例如10~500nm,10~200nm等。
本发明的脂质纳米颗粒还可修饰上靶向分子,使其成为靶向剂而能靶向特定细胞、组织 或器官。靶向分子可位于颗粒表面上。靶向分子可为蛋白质、肽、糖蛋白、脂质、小分子、 核酸等,其实例包括(但不限于)抗体、抗体片段、低密度脂蛋白(LDL)、转铁蛋白(transferrin)、 脱唾液酸糖蛋白(asialycoprotein)、受体配体、唾液酸、适体等。靶向分子可以连接在脂质纳 米颗粒的胆固醇类脂质分子或者PEG化的脂质分子上。
本发明的脂质纳米颗粒组合物和疫苗中,可以进一步含有一种或一种以上医药赋形剂。 术语“医药赋形剂”的意思是任何类型的无毒、惰性固体、半固体或液体填充剂、稀释剂等, 包括但不限于糖,诸如乳糖、海藻糖、葡萄糖和蔗糖;淀粉,诸如玉米淀粉和马铃薯淀粉; 纤维素和其衍生物,诸如羧甲基纤维素钠、乙基纤维素和乙酸纤维素;明胶;滑石;油,诸 如花生油、棉籽油、红花油、橄榄油、玉米油和大豆油;二醇,诸如丙二醇;酯,诸如油酸乙酯和月桂酸乙酯;表面活性剂,诸如吐温80(Tween 80);缓冲剂,诸如磷酸盐缓冲溶液、乙酸盐缓冲液和柠檬酸盐缓冲液;着色剂、甜味剂、调味剂和芳香剂、防腐剂和抗氧化剂等。
在本发明的一个实施方式中,所述脂质纳米颗粒组合物和疫苗是液体制剂,进一步含有 蔗糖,蔗糖的质量百分比浓度为5-20%,优选为8-10%。
本发明的脂质纳米颗粒组合物和疫苗,可经口、经直肠、静脉内、肌注、阴道内、鼻内、 腹膜内、经颊或以口服或鼻用喷雾等形式投予人类和/或动物。
本发明中的S蛋白突变体是亲本S蛋白发生氨基酸突变产生的。在本发明的一个实施方式 中,所述亲本S蛋白是2019-nCoV B1.351突变毒株的S蛋白,2019-nCoV B1.351突变毒株的S 蛋白与2019-nCoV野生株的S蛋白相比具有以下突变:L18F,D80A,D215G,L242_244L del, R246I,K417N,E484K,N501Y,D614G,A701V(所述位点以SEQ ID NO:1所示氨基酸序 列的位置来定位描述)。
在本发明中,S蛋白突变体和亲本S蛋白的氨基酸位置均以野生型S蛋白的氨基酸序列为描 述依据,野生型S蛋白的氨基酸序列可以在NCBI GeneID:43740568获得,共有1273个氨基酸, 其序列如下所示,在本发明中标示为SEQ ID NO:1。
本发明所述S蛋白突变体至少包含胞外域,其胞外域相对于亲本S蛋白的胞外域包含如下 位置的氨基酸突变:F817P、A892P、A899P、A942P、和KV986_987PP以及将682-685位氨基 酸RRAR突变为GSAS;以及不包含S蛋白的跨膜域和胞质尾;在胞外区的C端直接融合辅助形 成三聚体的结构域T4 Fibritin Foldon Trimerization Motif。所述S蛋白突变体包含从N端向C端 直接连接的SEQ ID NO:2的氨基酸序列和SEQ ID NO:3的氨基酸序列。
本发明上述序列的列表:
/>
/>
/>
/>
/>
/>
/>
术语说明:
术语“烷基”是指从含有1到30个碳原子的烃部分通过去除单个氢原子得到的饱和烃基。 烷基的实例包括(但不限于)甲基、乙基、丙基、异丙基、正丁基、仲丁基、叔丁基、正戊基、 异戊基、新戊基、正己基、正庚基、正辛基、正癸基、正十一烷基和正十二烷基。
术语“烯基”表示从具有至少一个碳-碳双键的烃部分通过去除单个氢原子得到的单价基 团。烯基包括例如例如乙烯基、丙烯基、丁烯基、1-甲基-2-丁烯-1-基等。
术语“炔基”是指从具有至少一个碳-碳三键的烃通过去除单个氢原子得到的单价基团。 代表性炔基包括乙炔基、2-丙炔基(炔丙基)、1-丙炔基等。
术语“烷氧基”是指如前文所定义的烷基通过氧原子连接于母体分子。烷氧基的实例包 括(但不限于)甲氧基、乙氧基、丙氧基、异丙氧基、正丁氧基、叔丁氧基、新戊氧基和正己 氧基。
术语“卤基”和“卤素”是指选自氟、氯、溴和碘的原子。
术语“饱和或不饱和的4-6元环”是指具有4-6个环原子的环,所述环原子可以是C、N、S、 O,其实例包括但不限于4-6元饱和的环烷基,例如环丙基、环丁基、环戊基、环己基;4-6元 芳基,例如苯基;4-6元杂环基,例如吡咯烷基、哌啶基、哌嗪基、吗啉基等;4-6元杂芳基, 例如三唑基、唑基、异唑基、噻唑基等。在本发明的一些实施例中,所述饱和或不饱和的4-6 元环优选为哌嗪基、环己基。
术语“经取代”(无论前面是否存在术语“任选地”)和“取代基”是指将一个官能团变 为另一个官能团的能力,条件是维持所有原子的价数。当任何特定结构中的一个以上位置可 经一个以上选自指定群组的取代基取代时,所述取代基可在每一位置相同或不同。
“和/或”将被视为具有或不具有另一个的两个指定特征或组件中的每一个的具体公开。 因此,在诸如“A和/或B”之类的短语中使用的术语“和/或”旨在包括“A和B”、“A或B”、“A”(单独)和“B”(单独)。同样地,在诸如“A、B和/或C”的短语中使用的术语“和/或” 旨在涵盖以下方面中的每一个:A、B和C;A、B或C;A或C;A或B;B或C;A和C;A和B; B和C;A(单独);B(单独);和C(单独)。
“包括”和“包含”具有相同的含义,旨在是开放的并且允许但不要求包括额外的元件 或步骤。当在本文中使用术语“包括”或“包含”时,因此也包括和公开了术语“由......组成” 和/或“基本上由……组成”。
在本说明书和权利要求书中,核苷酸通过其通常接受的单字母代码来指代。除非另有说 明,否则核苷酸序列以5'至3'方向从左向右书写。核碱基在本文中由IUPAC-IUB生物化学命 名委员会推荐的通常已知的单字母符号表示。技术人员将理解,本文公开的密码子中的T碱基 存在于DNA中,而T碱基在相应RNA中将被U碱基取代。例如,本文公开的DNA形式的密码 子-核苷酸序列,例如载体或体外翻译(IVT)模板,其T碱基在其相应的转录mRNA中转录为U 碱基。在这一方面,密码子优化的DNA序列(包含T)和它们相应的mRNA序列(包含U)都被认 为是本公开的密码子优化的核苷酸序列。本领域技术人员还将理解,可以通过用非天然碱基 替换一个或多个碱基来产生等同的密码子图谱。
术语“核酸序列”、“核苷酸序列”或“多核苷酸序列”可互换使用,并且是指连续的核酸序列。序列可以是单链或双链的DNA或RNA,例如mRNA。
“编码…的核苷酸序列”是指编码多肽的核酸(例如,mRNA或DNA分子)编码序列。编码序列可以进一步包括与调控元件可操作地连接的起始和终止信号,所述调控元件包括能够 指导在施用核酸的个体或哺乳动物的细胞中的表达的启动子和多腺苷酸化信号。
在本说明书和权利要求书中,使用氨基酸残基的常规单字母或三字母代码。除非另有说 明,氨基酸序列以氨基至羧基取向从左至右书写。
“约”:在整个说明书和权利要求中与数值结合使用的术语“约”表示本领域技术人员 熟悉和可接受的准确度区间。通常,这种精确度的区间为±10%。
为了便于参照,本发明的S蛋白突变体使用如下命名规则描述:原始氨基酸:位置:取代氨 基酸。根据该命名规则,例如在第30位天冬酰胺被丙氨酸取代表示为:Asn30Ala或N30A;在 相同位置缺失天冬酰胺表示为:Asn30*或N30*;插入另一氨基酸残基,例如赖氨酸,表示为: Asn30AsnLys或N30NK;缺失连续的一段氨基酸残基,例如缺失氨基酸残基242-244,表示为 (242-244)*或Δ(242-244)或242_244del;如果与其它S蛋白亲本相比,S蛋白突变体含有“缺 失”且在该位置含有插入,则表示为:*36Asp或*36D,表示在第36位缺失同时插入天冬氨酸。 当在给定位置可插入一个或多个可选择的氨基酸残基时,表示为:N30A,E,或N30A或N30E。
同源性:如本文所用,术语“同源性”是指聚合物分子之间的总体相关性,例如,在核 酸分子(例如DNA分子和/或RNA分子)之间和/或在多肽分子之间。通常,术语“同源性”意味 着两个分子之间的进化关系。因此,两个同源的分子将具有共同的进化祖先。在本公开的背 景下,术语同源性包括同一性和相似性。
在一些实施方案中,如果分子中至少25%,30%,35%,40%,45%,50%,55%,60%,65%,70%,75%,80%,85%,90%,95%,96%,97%,98%,99%或100%的单体 是相同的(完全相同的单体)或相似(保守置换),聚合物分子被认为是彼此“同源的”。术语“同 源的”必然是指至少两个序列(多核苷酸或多肽序列)之间的比较。
同一性:如本文所用,术语“同一性”是指聚合物分子之间,例如多核苷酸分子(例如DNA 分子和/或RNA分子)之间和/或多肽分子之间的整体单体保守性。例如,可以通过以进行最佳 比较目的比对两个序列来进行两个多核苷酸序列的百分同一性的计算(例如,可以在第一和第 二核酸序列之一或两个中引入空位用于最佳比对和非相同的序列可以对于比较目的放弃。当 比较DNA和RNA时,胸腺嘧啶(T)和尿嘧啶(U)可被认为是等同的。
合适的软件程序可从各种来源获得,并用于蛋白质和核苷酸序列两者的比对。例如, Bl2seq,Needle,Stretcher,Water或Matcher等。
术语“编码区”和“编码区域”是指多核苷酸中的开放阅读框(ORF),其在表达时产生多 肽或蛋白质。
“可操作地连接”是指两个或更多个分子,构建体,转录物,实体,部分等之间的功能 性连接。
结构域:如本文所用,当提及多肽时,术语“结构域”是指具有一个或多个可识别的结 构或功能特征或性质(例如,结合能力,用作蛋白质-蛋白质相互作用的位点)的多肽的基序。
表达:如本文所用,核酸序列的“表达”是指一个或多个以下事件:(1)从DNA序列产生 mRNA模板(例如,通过转录);(2)mRNA转录物的加工(例如,通过剪接,编辑,5'帽形成和/或3'末端加工);(3)将mRNA翻译成多肽或蛋白质;和(4)多肽或蛋白质的翻译后修饰。
术语“蛋白突变体”或“多肽突变体”是指其氨基酸序列与天然或参考序列不同的分子。 与天然或参考序列相比,氨基酸序列突变体可在氨基酸序列内的某些位置具有置换、缺失和/ 或插入等。通常,突变体与天然或参考序列将具有至少约50%的同一性,至少约60%的同一 性,至少约70%的同一性,至少约80%的同一性,至少约90%的同一性,至少约95%的同一 性,至少约99%的同一性。
附图说明
图1:使用RNA 6000nano chip在2100生物分析仪上分析B1.351mRNA完整性结果。
图2:ELISA法检测核酸转染CHO-K1细胞后上清中S蛋白表达水平。
图3:S蛋白突变体的3D结构图。
图4:ELISA法检测用II-37制成的纳米颗粒包封本发明mRNA转染细胞后上清中S蛋白表 达水平。
图5:II-37和MC3制备的LNP包封本发明mRNA转染细胞后上清中S蛋白表达水平。
图6:以Firefly Luc为报告蛋白,不同的体外转录载体制备的mRNA在细胞内蛋白表达量 的统计图。
图7:本发明S蛋白突变体免疫BALB/c鼠后体内结合抗体的产生情况统计图。图中a为野 生型S蛋白三聚体结果,b为B1.351 mRNA翻译出的S蛋白的三聚体结果,c为空白对照。
图8:编码所述S蛋白突变体的mRNA的脂质纳米颗粒免疫BALB/c鼠后,体内结合抗体和 中和抗体的产生情况统计图。图中d、e、f分别为5μg、1μg、0.2μg mRNA的脂质纳米颗粒(LNP) 免疫后的结合抗体检测结果,图中纵坐标为浓度(μg/ml);图中g、h、i分别为5μg、1μg、 0.2μg mRNA的脂质纳米颗粒(LNP)免疫后的中和抗体检测结果,图中横坐标为血清稀释倍 数的log转换值,纵坐标为抑制率%。
具体实施方式
下文将结合具体实施例对本发明的技术方案做更进一步的详细说明。应当理解,下列实 施例仅为示例性地说明和解释本发明,而不应被解释为对本发明保护范围的限制。凡基于本 发明上述内容所实现的技术均涵盖在本发明旨在保护的范围内。
除非另有说明,以下实施例中使用的原料和试剂均为市售商品,或者可以通过已知方法 制备。所述实验方法是本领域常规的分子生物学方法,可以参照本领域的分子生物学实验手 册或试剂盒产品说明书的指引进行操作。
实施例1脂质II-37的合成
亚麻油醇(a2)的合成:在0℃下,向950mL四氢呋喃中加入LiAlH
1
(9Z,12Z)-十八碳-9,12-二烯醛(a3)的合成:在室温下,向170mL乙腈中加入亚麻油醇(25.0 g,a2)和2-碘酰基苯甲酸(39.4g),之后混合物在85℃下搅拌4h。反应液通过布氏漏斗过滤并 用二氯甲烷洗涤滤饼,收集滤液并蒸发浓缩,得到目标产物(9Z,12Z)-十八碳-9,12-二烯醛(a3) 24.0g。
1
(9Z,12Z)-2-氯代-十八碳-9,12-二烯-1-醇(a4)的合成:在0℃下,向246mL乙腈中加入 (9Z,12Z)-十八碳-9,12-二烯醛(43.0g,a3),DL-脯氨酸(5.62g)和N-氯代丁二酰亚胺,然后在0 ℃下搅拌2h。反应完成后,用无水乙醇(246mL)稀释反应液,再加入硼氢化钠(8.8g),之后 在0℃下搅拌4h。向反应混合物中加水(120mL)淬灭,并用甲基叔丁基醚萃取,合并有机相后 用饱和食盐水洗涤,硫酸钠干燥之后过滤、蒸发浓缩,得到目标产物(9Z,12Z)-2-氯代-十八碳 -9,12-二烯-1-醇(a4,46g),直接用于下一步。
1
2-[(7Z,10Z)-十六碳烷-7,10-二烯]环氧乙烷(a5)的合成:在室温下,向450mL1,4-二氧六环 中加入(9Z,12Z)-2-氯代-十八碳-9,12-二烯-1-醇(45g,a4)和氢氧化钠水溶液(120g氢氧化钠溶 于585mL水),滴加完毕后混合物在35℃下搅拌2h。TLC显示反应完成后,反应液通过分液 漏斗分离,并用饱和食盐水洗涤,硫酸钠干燥后过滤并蒸发浓缩,然后通过快速过柱法用石 油醚/乙酸乙酯洗脱来纯化残余物,得到目标产物2-[(7Z,10Z)-十六碳烷-7,10-二烯]环氧乙烷 (a5)29.11g。
1
II-37的合成:在室温下,向10mL乙醇中加入2-[(7Z,10Z)-十六碳烷-7,10-二烯]环氧乙烷(5 g)和N,N-二(2-氨基乙基)甲胺(739mg),之后混合物在90℃下搅拌36h。反应液蒸发浓缩,然 后通过快速过柱法用二氯甲烷/甲醇洗脱来纯化残余物,得到粗产物II-37(4g)。再次通过快速 过柱法用二氯甲烷/甲醇纯化目标产物,得到II-37(2.2g)。
1
ESI-MS:m/z 910.8[M+H]
实施例2B1.351 mRNA的制备及其翻译
1.人工合成能够编码前述SEQ ID No.8所示mRNA的核酸序列,将该序列克隆到pUC57-kana载体的T7启动子后面,所述载体在之前经过改造,已含有能够编码SEQ ID NO:6、 Kozak序列、2个首尾相连的SEQ ID NO:7、polyA尾的序列。编码前述SEQ ID NO:8所示mRNA 的核酸序列克隆在Kozak序列和2个首尾相连SEQ ID NO:7之间的多克隆位点上,构建体外转录 用质粒。
2.将构建的质粒转化到大肠杆菌Dh5a中,培养扩增,提取质粒。
3.将提取的质粒使用紧邻着polyA尾后面的限制性内切酶SpeI酶切消化成线性分子。
4.以制备的线性化质粒分子为模板,使用体外转录法(Thermo公司的体外转录试剂盒 A45975)制备mRNA,所述mRNA的序列如SEQ ID NO:9所示,以下将所述mRNA简称为B1.351 mRNA,由该mRNA翻译后获得的是本发明S蛋白突变体,其氨基酸序列从N端向C端为直接 连接的SEQ ID NO:2的氨基酸序列和SEQ ID NO:3的氨基酸序列。在体外转录结束后,使用加 帽酶和二甲基转移酶给mRNA添加上CAP1的帽结构。
5.mRNA的纯化:得到的mRNA原液使用亲和层析法纯化。
6.mRNA的质控:将制备出来的mRNA,使用RNA 6000nano chip在2100生物分析仪上分析mRNA完整性,结果如图1所示,转录mRNA条带单一,无明显降解。
另外,通过限制性内切酶HindIII和EcoRI从商品化的质粒pCMV3-spike上切下Spike片段, 插入到实施例5的IVT1载体的HindIII和EcoRI位点之间,得到IVT1-spike质粒。再对该质粒进 行点突变,得到IVT1-spike-D614G质粒,以该质粒为模板,体外转录得到spike-D614G mRNA, 表达包含D614G突变的全长S蛋白。
7.B1.351 mRNA细胞水平表达检测:以CHO-K1细胞系为表达体系,使用Lipofectamine Messenger MAX Reagent(Invitrogen,Cat#1168-027)将mRNA转染,培养48h后,收集细胞培 养上清,采用检测S蛋白的酶联免疫法检测试剂盒,检测S蛋白表达水平,以评判mRNA可否 翻译成蛋白。结果如图2所示。图2中,“spike DNA”为商品化的质粒pCMV3-spike(采购自 义翘神州公司),表达全长野生型S蛋白;“spike-D614G mRNA”为前述表达包含D614G突 变的全长S蛋白的mRNA,“spike B1.351 mRNA”为前述B1.351 mRNA,表达的是本发明所 述的S蛋白突变体,结果表明本发明的mRNA可以在细胞内高表达出S蛋白突变体。
将得到的S蛋白突变体纯化后,采用冷冻电镜进行结构解析,该S蛋白的3D结构如图3所 示,S蛋白突变体为预融合(prefusion spike structure)的稳定结构。B1.351突变毒株的序列和 野生毒株的序列有9个突变位点的差异,其中3个在RBD区域。已经报道的野生毒株的预融合S 蛋白的RBD区域状态主要为1个OPEN、2个CLOSE的结构。本发明的S蛋白突变体的结构主要 为2个OPEN和1个CLOSE的柔性状态。这种结构差异,是病毒与受体ACE2结合能力增强和传 染性增强的结构基础,并且该结构差异,也会导致S蛋白的免疫原性表位的显著差异,从而基 于不同结构诱导的抗体尤其是中和抗体的显著不同。
实施例3包含核酸的脂质纳米颗粒组合物的制备
准确称取化合物II-37、DOPE、CHOL、DSPE-PEG2000、DSPC、DMG-PEG2000等,将 每种脂质于适当容器中,无水乙醇充分溶解备用。
将各脂质按下表中的摩尔比例混合均匀,作为有机相,将核酸(mRNA或DNA)配置成水溶液(以纯水为溶剂)作为水相pH=4。
有机相与水相体积比例为3:1混合,在微流控平台(如,PNI Ignite)上制备得到脂质纳米颗 粒混悬液。将得到的脂质纳米颗粒悬浮液经100KDa超滤离心管离心过滤,纯化浓缩,将浓缩 后的液体进行分装。
将制备所得脂质纳米颗粒用激光纳米粒度仪测粒径、PDI、电位,用紫外分光光度计结合 RiboGreen RNA试剂盒测包封率(%),示例性结果如下。
在优化制备工艺后,还能获得理化质控数据更好的脂质纳米颗粒,以II-37:DSPC:CHOL:DMG-PEG2000的配方结果示例如下表,其中tri-009-BJ-LNP-21040601的脂质摩尔配比为:40:10:48.5:1.5;tri-009-BJ-LNP-21040602的脂质摩尔配比为:35:15:48.5:1.5;tri-009-BJ-LNP-21040603的脂质摩尔配比为:45:15:38.5:1.5。将其中一部分样本按照实施例2 的方式转染细胞CHO,并通过Elisa检测蛋白表达量以评估细胞转染率。
细胞转染结果见图4,图4中,“tri-009-BJ-LNP-21040601”、“tri-009-BJ-LNP-21040602”、 “tri-009-BJ-LNP-21040603”是上述对应配方包载实施例2的B1.351 mRNA,“lipoMax-spike mRNA”是用lipoMax
实施例4II-37和商业化可电离阳离子脂质分子MC3的效果对比
MC3为:4-(N,N-二甲基氨基)丁酸(6Z,9Z,28Z,31Z)-庚三十碳-6,9,28,31-四稀-19-基脂。
按照实施例3所述的方法,分别使用II37和MC3制备脂质纳米颗粒,具体摩尔配比为: II-37:DSPC:CHOL:DMG-PEG2000=45:15:38.5:1.5;MC3:DSPC:CHOL: DMG-PEG2000=45:15:38.5:1.5;包载实施例2中B1.351 mRNA。
制备得到的脂质纳米颗粒理化质控数据如下表所示:
由上表可见,在相同的制备工艺下,II-37制备的脂质纳米颗粒包封率高达90.5%,远高于 MC3的脂质纳米颗粒,并且粒径更小更均一,电位更高。
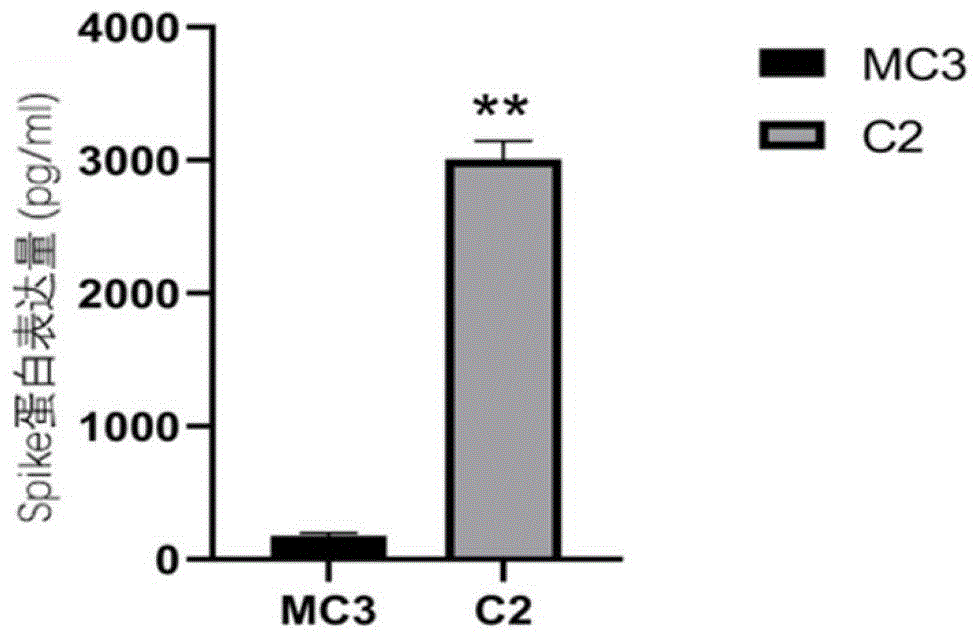
采用实施例2相同的转染方式,将制备的脂质纳米颗粒转染细胞,了解蛋白的表达情况, 结果如图5所示,II-37(图中以C2表示)制备的脂质纳米颗粒携带mRNA转染细胞之后,细胞 中蛋白表达量远远高于MC3的,说明II-37制成的脂质纳米颗粒的细胞转染效率很高。
实施例5本发明IVT载体的效率对比实验
本实施例以Firefly Luc为报告蛋白,构建了不同的IVT载体用于在体外转录合成能够翻译 Firefly Luc的mRNA,对比合成的不同序列特征的mRNA的翻译效率。
采用本领域常规的质粒载体构建技术,将Firefly Luc的编码序列克隆到相应载体的多克隆 位点上,得到编号分别为IVT1,IVT2,IVT3和IVT4的载体,之后使用AM1344试剂盒依据前 述载体体外转录制备得到对应的Firefly Luc mRNA样品。
载体IVT1~IVT4均为在商业化载体psp73的基础上改造而来,以下序列在载体psp73酶切位 点XhoI/NdeI处插入,其中IVT1中没有添加UTR序列,polyA尾长度为64个A;IVT2中使用了 SEQ ID NO:6所示的5’UTR和 GCTCGCTTTCTTGCTGTCCAATTTCTATTAAAGGTTCCTTTGTTCCCTAAGTCCAACTAC TAAACTGGGGGATATTATGAAGGGCCTTGAGCATCTGGATTCTGCCTAATAAAAAACA TTTATTTTCATTGC的3’UTR序列(β珠蛋白的3’UTR序列),polyA长度为120个A;IVT3 中使用了SEQ ID NO:6所示的5’UTR和SEQ ID NO:7所示的3’UTR序列,polyA长度为120个A;IVT4中使用了SEQ ID NO:6所示的5’UTR和2个串联重复的SEQ ID NO:7所示的3’UTR序列,polyA长度为120个A。在上述5’UTR和3’UTR的序列中间插入包含常见酶切位点HindIII和EcoRI的多克隆位点,再将Firefly Luc的编码序列克隆到HindIII和EcoRI的多克隆位点中。所有 载体均为金斯瑞公司使用基因合成的方法构建得到。
将每种Firefly Luc mRNA样品以Lipofectamine2000(cat#11668030,购自赛默飞世尔公司) 为转染试剂,转染到CHO细胞中,使用Dual-Lumi
结果如图6所示。图中“DNA”是阳性对照(携带有Firefly Luc的DNA的psicheck2质粒), “IVT1-Luc”、“IVT2-Luc”、“IVT3-Luc”、“IVT4-Luc”分别代表由IVT1,IVT2,IVT3 和IVT4的载体体外转录得到的对应Firefly Luc mRNA,“Nagative control”为阴性对照。由图 6可见,在相同mRNA的转染量下,IVT4-Luc的蛋白表达量远远高于其他三个mRNA,为其2-3 倍,说明IVT4-Luc的稳定性好且翻译效率高。
实施例6S蛋白突变体免疫原性的测定
使用BALB/c鼠评价S蛋白突变体诱导结合抗体和中和抗体的产生:6周龄雌性BALB/c鼠, 初次免疫及二次免疫间隔2周;免疫14天后采血。ELISA法检测针对S蛋白突变体的结合抗体 的表达,化学发光检测针对S蛋白突变体的中和抗体滴度。
ELISA方法检测结合抗体:通过在酶标板上包被商业化S蛋白捕获免疫小鼠血浆中针对S 蛋白突变体的结合抗体,再用生物素标记的检测抗体进行吸光度检测。化学发光检测针对S 蛋白突变体的中和抗体滴度:免疫后小鼠血浆与携带荧光素酶报告基因的SPIKE慢病毒(中 吉当康;商品名称:SRAS-CoV-2假病毒(B.1.351)-LUC;商品编号:DZPSC-L-0;批次: K05202102)中和后去感染高表达ACE-2的293T细胞(中吉当康;商品名称:“YJ1B09”hACE2-293T cell lines;商品编号:YJ293T-01;批次:A23202001),用化学发光(Bright-Lumi II萤火虫萤光素酶报告基因检测试剂盒,品牌:碧云天;商品编号:RG052M)评定血浆的中 和抗体滴度。
对照组共9只小鼠,每只小鼠皮下注射2ug蛋白。其中,3只注射的蛋白为实施例2的B1.351 mRNA翻译出的S蛋白的三聚体纯化物,3只注射的蛋白为野生型的S蛋白的三聚体,3只注射 的是空白脂质纳米颗粒,脂质纳米颗粒的脂质配方是实施例3中tri-009-BJ-LNP-21040602。
实验组共18只小鼠,皮下注射mRNA的脂质纳米颗粒;其中1-6号注射0.2μg mRNA的脂质 纳米颗粒(LNP),7-12号注射1μg mRNA的脂质纳米颗粒(LNP),13-18号注射5μg mRNA的脂质纳米颗粒(LNP),其中的mRNA是实施例2的B1.351 mRNA,脂质纳米颗粒的配方是 实施例3中tri-009-BJ-LNP-21040602。
初次免疫及二次免疫后小鼠结合抗体表达水平以及中和抗体水平如图7-8所示。
由图7中的a、b、c可见,实施例2的B1.351 mRNA翻译出的S蛋白三聚体及野生型的S蛋白 三聚体在小鼠体内均可以诱导出抗S蛋白的结合抗体:在实验小鼠中第二次免疫时已经能产生 较高浓度的结合抗体,在二次免疫后8周,所述结合抗体的浓度仍保持较高水平,经计算,二 次免疫后结合抗体浓度在2.2μg/ml左右,二次免疫后8周,仍能维持1.6μg/ml左右。
由图8中的d、e、f可见,二次免疫LNP包裹的mRNA制剂后的小鼠,即使是低剂量(0.2μg) 注射组也可在小鼠体内诱导出抗S蛋白的结合抗体,经计算,结合抗体水平约为0.1-0.3μg/ml。 由图8中的g、h、i可见,二次免疫LNP包裹的mRNA制剂后的小鼠体内诱导出了较好的中和抗 体,中和抗体的GMT值分别为78.69,21.9和72.19。
以上,对本发明的实施方式进行了说明。但是,本发明不限定于上述实施方式。凡在本 发明的精神和原则之内,所做的任何修改、等同替换、改进等,均应包含在本发明的保护范 围之内。
SEQUENCE LISTING
<110> 北京启辰生生物科技有限公司
<120> 一种脂质纳米颗粒组合物以及由其制备的药物递送系统
<130> CPCN21411053
<160> 9
<170> PatentIn version 3.5
<210> 1
<211> 1273
<212> PRT
<213> Unknown
<220>
<223> 2019-nCoV野生型S蛋白
<400> 1
Met Phe Val Phe Leu Val Leu Leu Pro Leu Val Ser Ser Gln Cys Val
1 5 1015
Asn Leu Thr Thr Arg Thr Gln Leu Pro Pro Ala Tyr Thr Asn Ser Phe
202530
Thr Arg Gly Val Tyr Tyr Pro Asp Lys Val Phe Arg Ser Ser Val Leu
354045
His Ser Thr Gln Asp Leu Phe Leu Pro Phe Phe Ser Asn Val Thr Trp
505560
Phe His Ala Ile His Val Ser Gly Thr Asn Gly Thr Lys Arg Phe Asp
65707580
Asn Pro Val Leu Pro Phe Asn Asp Gly Val Tyr Phe Ala Ser Thr Glu
859095
Lys Ser Asn Ile Ile Arg Gly Trp Ile Phe Gly Thr Thr Leu Asp Ser
100 105 110
Lys Thr Gln Ser Leu Leu Ile Val Asn Asn Ala Thr Asn Val Val Ile
115 120 125
Lys Val Cys Glu Phe Gln Phe Cys Asn Asp Pro Phe Leu Gly Val Tyr
130 135 140
Tyr His Lys Asn Asn Lys Ser Trp Met Glu Ser Glu Phe Arg Val Tyr
145 150 155 160
Ser Ser Ala Asn Asn Cys Thr Phe Glu Tyr Val Ser Gln Pro Phe Leu
165 170 175
Met Asp Leu Glu Gly Lys Gln Gly Asn Phe Lys Asn Leu Arg Glu Phe
180 185 190
Val Phe Lys Asn Ile Asp Gly Tyr Phe Lys Ile Tyr Ser Lys His Thr
195 200 205
Pro Ile Asn Leu Val Arg Asp Leu Pro Gln Gly Phe Ser Ala Leu Glu
210 215 220
Pro Leu Val Asp Leu Pro Ile Gly Ile Asn Ile Thr Arg Phe Gln Thr
225 230 235 240
Leu Leu Ala Leu His Arg Ser Tyr Leu Thr Pro Gly Asp Ser Ser Ser
245 250 255
Gly Trp Thr Ala Gly Ala Ala Ala Tyr Tyr Val Gly Tyr Leu Gln Pro
260 265 270
Arg Thr Phe Leu Leu Lys Tyr Asn Glu Asn Gly Thr Ile Thr Asp Ala
275 280 285
Val Asp Cys Ala Leu Asp Pro Leu Ser Glu Thr Lys Cys Thr Leu Lys
290 295 300
Ser Phe Thr Val Glu Lys Gly Ile Tyr Gln Thr Ser Asn Phe Arg Val
305 310 315 320
Gln Pro Thr Glu Ser Ile Val Arg Phe Pro Asn Ile Thr Asn Leu Cys
325 330 335
Pro Phe Gly Glu Val Phe Asn Ala Thr Arg Phe Ala Ser Val Tyr Ala
340 345 350
Trp Asn Arg Lys Arg Ile Ser Asn Cys Val Ala Asp Tyr Ser Val Leu
355 360 365
Tyr Asn Ser Ala Ser Phe Ser Thr Phe Lys Cys Tyr Gly Val Ser Pro
370 375 380
Thr Lys Leu Asn Asp Leu Cys Phe Thr Asn Val Tyr Ala Asp Ser Phe
385 390 395 400
Val Ile Arg Gly Asp Glu Val Arg Gln Ile Ala Pro Gly Gln Thr Gly
405 410 415
Lys Ile Ala Asp Tyr Asn Tyr Lys Leu Pro Asp Asp Phe Thr Gly Cys
420 425 430
Val Ile Ala Trp Asn Ser Asn Asn Leu Asp Ser Lys Val Gly Gly Asn
435 440 445
Tyr Asn Tyr Leu Tyr Arg Leu Phe Arg Lys Ser Asn Leu Lys Pro Phe
450 455 460
Glu Arg Asp Ile Ser Thr Glu Ile Tyr Gln Ala Gly Ser Thr Pro Cys
465 470 475 480
Asn Gly Val Glu Gly Phe Asn Cys Tyr Phe Pro Leu Gln Ser Tyr Gly
485 490 495
Phe Gln Pro Thr Asn Gly Val Gly Tyr Gln Pro Tyr Arg Val Val Val
500 505 510
Leu Ser Phe Glu Leu Leu His Ala Pro Ala Thr Val Cys Gly Pro Lys
515 520 525
Lys Ser Thr Asn Leu Val Lys Asn Lys Cys Val Asn Phe Asn Phe Asn
530 535 540
Gly Leu Thr Gly Thr Gly Val Leu Thr Glu Ser Asn Lys Lys Phe Leu
545 550 555 560
Pro Phe Gln Gln Phe Gly Arg Asp Ile Ala Asp Thr Thr Asp Ala Val
565 570 575
Arg Asp Pro Gln Thr Leu Glu Ile Leu Asp Ile Thr Pro Cys Ser Phe
580 585 590
Gly Gly Val Ser Val Ile Thr Pro Gly Thr Asn Thr Ser Asn Gln Val
595 600 605
Ala Val Leu Tyr Gln Asp Val Asn Cys Thr Glu Val Pro Val Ala Ile
610 615 620
His Ala Asp Gln Leu Thr Pro Thr Trp Arg Val Tyr Ser Thr Gly Ser
625 630 635 640
Asn Val Phe Gln Thr Arg Ala Gly Cys Leu Ile Gly Ala Glu His Val
645 650 655
Asn Asn Ser Tyr Glu Cys Asp Ile Pro Ile Gly Ala Gly Ile Cys Ala
660 665 670
Ser Tyr Gln Thr Gln Thr Asn Ser Pro Arg Arg Ala Arg Ser Val Ala
675 680 685
Ser Gln Ser Ile Ile Ala Tyr Thr Met Ser Leu Gly Ala Glu Asn Ser
690 695 700
Val Ala Tyr Ser Asn Asn Ser Ile Ala Ile Pro Thr Asn Phe Thr Ile
705 710 715 720
Ser Val Thr Thr Glu Ile Leu Pro Val Ser Met Thr Lys Thr Ser Val
725 730 735
Asp Cys Thr Met Tyr Ile Cys Gly Asp Ser Thr Glu Cys Ser Asn Leu
740 745 750
Leu Leu Gln Tyr Gly Ser Phe Cys Thr Gln Leu Asn Arg Ala Leu Thr
755 760 765
Gly Ile Ala Val Glu Gln Asp Lys Asn Thr Gln Glu Val Phe Ala Gln
770 775 780
Val Lys Gln Ile Tyr Lys Thr Pro Pro Ile Lys Asp Phe Gly Gly Phe
785 790 795 800
Asn Phe Ser Gln Ile Leu Pro Asp Pro Ser Lys Pro Ser Lys Arg Ser
805 810 815
Phe Ile Glu Asp Leu Leu Phe Asn Lys Val Thr Leu Ala Asp Ala Gly
820 825 830
Phe Ile Lys Gln Tyr Gly Asp Cys Leu Gly Asp Ile Ala Ala Arg Asp
835 840 845
Leu Ile Cys Ala Gln Lys Phe Asn Gly Leu Thr Val Leu Pro Pro Leu
850 855 860
Leu Thr Asp Glu Met Ile Ala Gln Tyr Thr Ser Ala Leu Leu Ala Gly
865 870 875 880
Thr Ile Thr Ser Gly Trp Thr Phe Gly Ala Gly Ala Ala Leu Gln Ile
885 890 895
Pro Phe Ala Met Gln Met Ala Tyr Arg Phe Asn Gly Ile Gly Val Thr
900 905 910
Gln Asn Val Leu Tyr Glu Asn Gln Lys Leu Ile Ala Asn Gln Phe Asn
915 920 925
Ser Ala Ile Gly Lys Ile Gln Asp Ser Leu Ser Ser Thr Ala Ser Ala
930 935 940
Leu Gly Lys Leu Gln Asp Val Val Asn Gln Asn Ala Gln Ala Leu Asn
945 950 955 960
Thr Leu Val Lys Gln Leu Ser Ser Asn Phe Gly Ala Ile Ser Ser Val
965 970 975
Leu Asn Asp Ile Leu Ser Arg Leu Asp Lys Val Glu Ala Glu Val Gln
980 985 990
Ile Asp Arg Leu Ile Thr Gly Arg Leu Gln Ser Leu Gln Thr Tyr Val
995 10001005
Thr Gln Gln Leu Ile Arg Ala Ala Glu Ile Arg Ala Ser Ala Asn
101010151020
Leu Ala Ala Thr Lys Met Ser Glu Cys Val Leu Gly Gln Ser Lys
102510301035
Arg Val Asp Phe Cys Gly Lys Gly Tyr His Leu Met Ser Phe Pro
104010451050
Gln Ser Ala Pro His Gly Val Val Phe Leu His Val Thr Tyr Val
105510601065
Pro Ala Gln Glu Lys Asn Phe Thr Thr Ala Pro Ala Ile Cys His
107010751080
Asp Gly Lys Ala His Phe Pro Arg Glu Gly Val Phe Val Ser Asn
108510901095
Gly Thr His Trp Phe Val Thr Gln Arg Asn Phe Tyr Glu Pro Gln
110011051110
Ile Ile Thr Thr Asp Asn Thr Phe Val Ser Gly Asn Cys Asp Val
111511201125
Val Ile Gly Ile Val Asn Asn Thr Val Tyr Asp Pro Leu Gln Pro
113011351140
Glu Leu Asp Ser Phe Lys Glu Glu Leu Asp Lys Tyr Phe Lys Asn
114511501155
His Thr Ser Pro Asp Val Asp Leu Gly Asp Ile Ser Gly Ile Asn
116011651170
Ala Ser Val Val Asn Ile Gln Lys Glu Ile Asp Arg Leu Asn Glu
117511801185
Val Ala Lys Asn Leu Asn Glu Ser Leu Ile Asp Leu Gln Glu Leu
119011951200
Gly Lys Tyr Glu Gln Tyr Ile Lys Trp Pro Trp Tyr Ile Trp Leu
120512101215
Gly Phe Ile Ala Gly Leu Ile Ala Ile Val Met Val Thr Ile Met
122012251230
Leu Cys Cys Met Thr Ser Cys Cys Ser Cys Leu Lys Gly Cys Cys
123512401245
Ser Cys Gly Ser Cys Cys Lys Phe Asp Glu Asp Asp Ser Glu Pro
125012551260
Val Leu Lys Gly Val Lys Leu His Tyr Thr
12651270
<210> 2
<211> 1205
<212> PRT
<213> Artificial Sequence
<220>
<223> 2019-nCoV S蛋白突变体
<400> 2
Met Phe Val Phe Leu Val Leu Leu Pro Leu Val Ser Ser Gln Cys Val
1 5 1015
Asn Phe Thr Thr Arg Thr Gln Leu Pro Pro Ala Tyr Thr Asn Ser Phe
202530
Thr Arg Gly Val Tyr Tyr Pro Asp Lys Val Phe Arg Ser Ser Val Leu
354045
His Ser Thr Gln Asp Leu Phe Leu Pro Phe Phe Ser Asn Val Thr Trp
505560
Phe His Ala Ile His Val Ser Gly Thr Asn Gly Thr Lys Arg Phe Ala
65707580
Asn Pro Val Leu Pro Phe Asn Asp Gly Val Tyr Phe Ala Ser Thr Glu
859095
Lys Ser Asn Ile Ile Arg Gly Trp Ile Phe Gly Thr Thr Leu Asp Ser
100 105 110
Lys Thr Gln Ser Leu Leu Ile Val Asn Asn Ala Thr Asn Val Val Ile
115 120 125
Lys Val Cys Glu Phe Gln Phe Cys Asn Asp Pro Phe Leu Gly Val Tyr
130 135 140
Tyr His Lys Asn Asn Lys Ser Trp Met Glu Ser Glu Phe Arg Val Tyr
145 150 155 160
Ser Ser Ala Asn Asn Cys Thr Phe Glu Tyr Val Ser Gln Pro Phe Leu
165 170 175
Met Asp Leu Glu Gly Lys Gln Gly Asn Phe Lys Asn Leu Arg Glu Phe
180 185 190
Val Phe Lys Asn Ile Asp Gly Tyr Phe Lys Ile Tyr Ser Lys His Thr
195 200 205
Pro Ile Asn Leu Val Arg Gly Leu Pro Gln Gly Phe Ser Ala Leu Glu
210 215 220
Pro Leu Val Asp Leu Pro Ile Gly Ile Asn Ile Thr Arg Phe Gln Thr
225 230 235 240
Leu His Ile Ser Tyr Leu Thr Pro Gly Asp Ser Ser Ser Gly Trp Thr
245 250 255
Ala Gly Ala Ala Ala Tyr Tyr Val Gly Tyr Leu Gln Pro Arg Thr Phe
260 265 270
Leu Leu Lys Tyr Asn Glu Asn Gly Thr Ile Thr Asp Ala Val Asp Cys
275 280 285
Ala Leu Asp Pro Leu Ser Glu Thr Lys Cys Thr Leu Lys Ser Phe Thr
290 295 300
Val Glu Lys Gly Ile Tyr Gln Thr Ser Asn Phe Arg Val Gln Pro Thr
305 310 315 320
Glu Ser Ile Val Arg Phe Pro Asn Ile Thr Asn Leu Cys Pro Phe Gly
325 330 335
Glu Val Phe Asn Ala Thr Arg Phe Ala Ser Val Tyr Ala Trp Asn Arg
340 345 350
Lys Arg Ile Ser Asn Cys Val Ala Asp Tyr Ser Val Leu Tyr Asn Ser
355 360 365
Ala Ser Phe Ser Thr Phe Lys Cys Tyr Gly Val Ser Pro Thr Lys Leu
370 375 380
Asn Asp Leu Cys Phe Thr Asn Val Tyr Ala Asp Ser Phe Val Ile Arg
385 390 395 400
Gly Asp Glu Val Arg Gln Ile Ala Pro Gly Gln Thr Gly Asn Ile Ala
405 410 415
Asp Tyr Asn Tyr Lys Leu Pro Asp Asp Phe Thr Gly Cys Val Ile Ala
420 425 430
Trp Asn Ser Asn Asn Leu Asp Ser Lys Val Gly Gly Asn Tyr Asn Tyr
435 440 445
Leu Tyr Arg Leu Phe Arg Lys Ser Asn Leu Lys Pro Phe Glu Arg Asp
450 455 460
Ile Ser Thr Glu Ile Tyr Gln Ala Gly Ser Thr Pro Cys Asn Gly Val
465 470 475 480
Lys Gly Phe Asn Cys Tyr Phe Pro Leu Gln Ser Tyr Gly Phe Gln Pro
485 490 495
Thr Tyr Gly Val Gly Tyr Gln Pro Tyr Arg Val Val Val Leu Ser Phe
500 505 510
Glu Leu Leu His Ala Pro Ala Thr Val Cys Gly Pro Lys Lys Ser Thr
515 520 525
Asn Leu Val Lys Asn Lys Cys Val Asn Phe Asn Phe Asn Gly Leu Thr
530 535 540
Gly Thr Gly Val Leu Thr Glu Ser Asn Lys Lys Phe Leu Pro Phe Gln
545 550 555 560
Gln Phe Gly Arg Asp Ile Ala Asp Thr Thr Asp Ala Val Arg Asp Pro
565 570 575
Gln Thr Leu Glu Ile Leu Asp Ile Thr Pro Cys Ser Phe Gly Gly Val
580 585 590
Ser Val Ile Thr Pro Gly Thr Asn Thr Ser Asn Gln Val Ala Val Leu
595 600 605
Tyr Gln Gly Val Asn Cys Thr Glu Val Pro Val Ala Ile His Ala Asp
610 615 620
Gln Leu Thr Pro Thr Trp Arg Val Tyr Ser Thr Gly Ser Asn Val Phe
625 630 635 640
Gln Thr Arg Ala Gly Cys Leu Ile Gly Ala Glu His Val Asn Asn Ser
645 650 655
Tyr Glu Cys Asp Ile Pro Ile Gly Ala Gly Ile Cys Ala Ser Tyr Gln
660 665 670
Thr Gln Thr Asn Ser Pro Gly Ser Ala Ser Ser Val Ala Ser Gln Ser
675 680 685
Ile Ile Ala Tyr Thr Met Ser Leu Gly Val Glu Asn Ser Val Ala Tyr
690 695 700
Ser Asn Asn Ser Ile Ala Ile Pro Thr Asn Phe Thr Ile Ser Val Thr
705 710 715 720
Thr Glu Ile Leu Pro Val Ser Met Thr Lys Thr Ser Val Asp Cys Thr
725 730 735
Met Tyr Ile Cys Gly Asp Ser Thr Glu Cys Ser Asn Leu Leu Leu Gln
740 745 750
Tyr Gly Ser Phe Cys Thr Gln Leu Asn Arg Ala Leu Thr Gly Ile Ala
755 760 765
Val Glu Gln Asp Lys Asn Thr Gln Glu Val Phe Ala Gln Val Lys Gln
770 775 780
Ile Tyr Lys Thr Pro Pro Ile Lys Asp Phe Gly Gly Phe Asn Phe Ser
785 790 795 800
Gln Ile Leu Pro Asp Pro Ser Lys Pro Ser Lys Arg Ser Pro Ile Glu
805 810 815
Asp Leu Leu Phe Asn Lys Val Thr Leu Ala Asp Ala Gly Phe Ile Lys
820 825 830
Gln Tyr Gly Asp Cys Leu Gly Asp Ile Ala Ala Arg Asp Leu Ile Cys
835 840 845
Ala Gln Lys Phe Asn Gly Leu Thr Val Leu Pro Pro Leu Leu Thr Asp
850 855 860
Glu Met Ile Ala Gln Tyr Thr Ser Ala Leu Leu Ala Gly Thr Ile Thr
865 870 875 880
Ser Gly Trp Thr Phe Gly Ala Gly Pro Ala Leu Gln Ile Pro Phe Pro
885 890 895
Met Gln Met Ala Tyr Arg Phe Asn Gly Ile Gly Val Thr Gln Asn Val
900 905 910
Leu Tyr Glu Asn Gln Lys Leu Ile Ala Asn Gln Phe Asn Ser Ala Ile
915 920 925
Gly Lys Ile Gln Asp Ser Leu Ser Ser Thr Pro Ser Ala Leu Gly Lys
930 935 940
Leu Gln Asp Val Val Asn Gln Asn Ala Gln Ala Leu Asn Thr Leu Val
945 950 955 960
Lys Gln Leu Ser Ser Asn Phe Gly Ala Ile Ser Ser Val Leu Asn Asp
965 970 975
Ile Leu Ser Arg Leu Asp Pro Pro Glu Ala Glu Val Gln Ile Asp Arg
980 985 990
Leu Ile Thr Gly Arg Leu Gln Ser Leu Gln Thr Tyr Val Thr Gln Gln
995 10001005
Leu Ile Arg Ala Ala Glu Ile Arg Ala Ser Ala Asn Leu Ala Ala
101010151020
Thr Lys Met Ser Glu Cys Val Leu Gly Gln Ser Lys Arg Val Asp
102510301035
Phe Cys Gly Lys Gly Tyr His Leu Met Ser Phe Pro Gln Ser Ala
104010451050
Pro His Gly Val Val Phe Leu His Val Thr Tyr Val Pro Ala Gln
105510601065
Glu Lys Asn Phe Thr Thr Ala Pro Ala Ile Cys His Asp Gly Lys
107010751080
Ala His Phe Pro Arg Glu Gly Val Phe Val Ser Asn Gly Thr His
108510901095
Trp Phe Val Thr Gln Arg Asn Phe Tyr Glu Pro Gln Ile Ile Thr
110011051110
Thr Asp Asn Thr Phe Val Ser Gly Asn Cys Asp Val Val Ile Gly
111511201125
Ile Val Asn Asn Thr Val Tyr Asp Pro Leu Gln Pro Glu Leu Asp
113011351140
Ser Phe Lys Glu Glu Leu Asp Lys Tyr Phe Lys Asn His Thr Ser
114511501155
Pro Asp Val Asp Leu Gly Asp Ile Ser Gly Ile Asn Ala Ser Val
116011651170
Val Asn Ile Gln Lys Glu Ile Asp Arg Leu Asn Glu Val Ala Lys
117511801185
Asn Leu Asn Glu Ser Leu Ile Asp Leu Gln Glu Leu Gly Lys Tyr
119011951200
Glu Gln
1205
<210> 3
<211> 28
<212> PRT
<213> Artificial Sequence
<220>
<223> 辅助形成三聚体的结构域
<400> 3
Gly Tyr Ile Pro Glu Ala Pro Arg Asp Gly Gln Ala Tyr Val Arg Lys
1 5 1015
Asp Gly Glu Trp Val Leu Leu Ser Thr Phe Leu Gly
2025
<210> 4
<211> 3615
<212> DNA
<213> Artificial Sequence
<220>
<223> 人工序列
<400> 4
atgttcgtgt tcctggtgct gcttcccctg gtctctagcc agtgcgtgaa cttcacgacc 60
cggacccaac tgccccccgc gtacacaaac tccttcacca gaggcgtgta ctaccctgac 120
aaggtgttcc gcagcagcgt gctgcacagc acccaggacc tgttcctccc attcttcagc 180
aacgtgacct ggttccacgc catccacgtg tccggcacca atggaacaaa gagatttgcg 240
aaccccgtgc tacctttcaa cgacggcgtg tacttcgcct ccaccgagaa gagcaacatc 300
atccggggct ggatcttcgg caccaccctg gactctaaaa cccagagcct gctgatcgtg 360
aataatgcca ccaacgtggt gatcaaggtg tgcgagttcc agttctgcaa cgaccctttc 420
ctgggcgtct actaccacaa gaacaacaag agttggatgg aaagcgagtt cagagtgtac 480
tcttctgcta acaactgcac cttcgagtac gtgtcccagc ctttcctgat ggacctggaa 540
ggcaagcagg ggaacttcaa gaacctgcgg gagttcgtgt tcaagaacat cgacgggtat 600
ttcaagatct actccaagca cacacctatc aatctggtga gaggcctgcc ccagggcttc 660
agcgccctgg aacctctggt cgacctgcca atcggcatca acatcacccg gttccaaaca 720
ctgcatatca gctacctgac acctggcgat agctcctccg gctggaccgc cggcgctgcc 780
gcttattacg tcggctacct gcagcctaga acgttcctgc tgaagtacaa cgagaacggc 840
accatcaccg acgccgtcga ctgcgccctg gaccccctct ccgagacaaa atgcaccctg 900
aagagcttca ctgttgaaaa gggcatctac cagaccagca actttagagt gcagcctaca 960
gagtctatcg tgagattccc taacattacc aacctgtgtc cttttggaga agtgttcaac 1020
gccacaagat tcgcttctgt gtatgcctgg aaccggaaga gaatctcgaa ctgcgtggct 1080
gattacagcg tgctgtacaa cagcgctagc tttagcacat ttaagtgcta cggcgtgagc 1140
cccaccaagc tgaatgattt gtgcttcaca aatgtgtacg ccgactcttt cgtgataaga 1200
ggggacgagg tgcggcagat agctccaggc cagaccggca acatcgccga ttacaattac 1260
aagctgcctg acgactttac cggatgtgtg atcgcctgga acagcaacaa cctggatagc 1320
aaggtgggcg gaaactacaa ctacctgtac agactgttcc ggaaatctaa ccttaagcct 1380
tttgagcggg atatcagcac cgagatctac caagctggct ctacaccctg caacggcgtg 1440
aaggggttta attgttactt ccccctgcag agctacggct tccaaccgac ctacggagtg 1500
ggctaccagc cctaccgggt cgtggtgctg agctttgagc tgctgcacgc ccctgctaca 1560
gtgtgcggcc ccaagaagtc tacgaacctg gtgaagaaca agtgtgtgaa ttttaatttc 1620
aacggactga ccggcacagg cgtcctgacc gaatctaaca agaaattcct ccctttccag 1680
cagttcggga gagatatcgc cgacaccacc gacgccgtgc gggaccctca aacactggaa 1740
atcctggata tcaccccttg ttctttcgga ggcgtgtccg tgatcacccc aggtacgaac 1800
acatctaacc aggtggctgt gctgtaccag ggcgtgaact gcaccgaggt gcctgtggcc 1860
attcacgccg accagctgac tcctacctgg cgggtgtaca gcacgggctc caacgtgttt 1920
cagaccagag ctggctgtct gatcggagcc gagcacgtga acaactctta tgagtgcgat 1980
atccccatcg gcgctggaat ctgtgcctcc taccagactc aaaccaacag ccctggcagc 2040
gctagcagcg tggccagcca gagcatcatc gcctacacca tgagcctggg agtcgaaaac 2100
agcgtggcct actcaaacaa ctccatcgct atccctacca acttcaccat cagcgtaacg 2160
accgaaatcc tgcccgtgag catgaccaag accagcgtgg actgcacaat gtacatctgc 2220
ggcgatagca cagaatgcag caatctgcta ctgcagtacg gtagcttttg cacccaactg 2280
aatagagccc tgaccggcat cgccgtggaa caggataaaa acacccaaga ggtcttcgct 2340
caggtgaagc agatctacaa gacacctccc atcaaggact tcggaggatt caactttagc 2400
cagatcctgc ctgatccaag caaacctagc aagcggagtc ctatcgagga cctgctgttt 2460
aacaaggtga cactggccga cgccggcttc atcaagcagt atggcgactg tctgggcgac 2520
atcgccgcca gggatctgat ctgtgcccaa aaattcaacg gcctgacagt gctgccacct 2580
ctgctgaccg acgagatgat cgctcaatac accagcgccc tcctcgccgg cacgatcacc 2640
agcggctgga cattcggcgc cggccctgcc ctccagatcc ctttccctat gcagatggcc 2700
tacagattca acggcatcgg cgtgacacaa aacgtgctgt acgaaaacca gaagctgatc 2760
gccaatcagt ttaatagcgc catcgggaag atccaggata gcctgtcatc taccccttct 2820
gccctgggaa agctgcagga cgtggtgaac cagaacgccc aggccctgaa caccctggtg 2880
aaacagctgt ctagcaactt cggcgctatc agcagcgtgc tgaatgatat cctgagcaga 2940
ctggatcctc ctgaggccga ggtgcagatc gacagattga tcaccggccg gctgcagagc 3000
ctgcaaacct acgttacaca gcagctgatc agagccgctg aaatcagagc ctctgccaac 3060
ctggccgcca ccaaaatgag cgagtgcgtg ctgggacaga gcaaaagggt ggacttctgc 3120
gggaagggct accacctcat gagttttccc cagagcgccc cccacggcgt ggtgttcctg 3180
cacgtgacat atgtcccggc ccaggagaaa aactttacaa cagcccctgc catttgccat 3240
gacggaaagg cccacttccc tcgggaaggt gtgttcgtga gcaacggcac acactggttc 3300
gtgacccaga gaaacttcta cgagcctcaa atcatcacca cagacaacac cttcgttagt 3360
ggaaattgcg acgtggttat cggcatcgtg aacaacaccg tctacgaccc actgcagcct 3420
gaactggata gcttcaagga ggaactggat aagtatttca agaaccacac ctcccccgac 3480
gtggatctgg gcgacattag cggcatcaac gccagcgtgg tgaacatcca gaaagagatc 3540
gatagactta atgaggtggc caagaacctg aacgagagcc tgatcgacct gcaggagctc 3600
ggcaaatacg agcag 3615
<210> 5
<211> 84
<212> DNA
<213> Artificial Sequence
<220>
<223> 人工序列
<400> 5
ggctatatcc cagaggcccc tagagatggc caggcctacg ttagaaagga cggcgagtgg 60
gtcctgctga gcacattcct gggc 84
<210> 6
<211> 50
<212> RNA
<213> Artificial Sequence
<220>
<223> 人工序列
<400> 6
acauuugcuu cugacacaac uguguucacu agcaaccuca aacagacacc 50
<210> 7
<211> 88
<212> RNA
<213> Artificial Sequence
<220>
<223> 人工序列
<400> 7
gcuggagccu cgguagccgu uccuccugcc cgcugggccu cccaacgggc ccuccucccc 60
uccuugcacc ggcccuuccu ggucuuug 88
<210> 8
<211> 3702
<212> RNA
<213> Artificial Sequence
<220>
<223> 人工序列
<400> 8
auguucgugu uccuggugcu gcuuccccug gucucuagcc agugcgugaa cuucacgacc 60
cggacccaac ugccccccgc guacacaaac uccuucacca gaggcgugua cuacccugac 120
aagguguucc gcagcagcgu gcugcacagc acccaggacc uguuccuccc auucuucagc 180
aacgugaccu gguuccacgc cauccacgug uccggcacca auggaacaaa gagauuugcg 240
aaccccgugc uaccuuucaa cgacggcgug uacuucgccu ccaccgagaa gagcaacauc 300
auccggggcu ggaucuucgg caccacccug gacucuaaaa cccagagccu gcugaucgug 360
aauaaugcca ccaacguggu gaucaaggug ugcgaguucc aguucugcaa cgacccuuuc 420
cugggcgucu acuaccacaa gaacaacaag aguuggaugg aaagcgaguu cagaguguac 480
ucuucugcua acaacugcac cuucgaguac gugucccagc cuuuccugau ggaccuggaa 540
ggcaagcagg ggaacuucaa gaaccugcgg gaguucgugu ucaagaacau cgacggguau 600
uucaagaucu acuccaagca cacaccuauc aaucugguga gaggccugcc ccagggcuuc 660
agcgcccugg aaccucuggu cgaccugcca aucggcauca acaucacccg guuccaaaca 720
cugcauauca gcuaccugac accuggcgau agcuccuccg gcuggaccgc cggcgcugcc 780
gcuuauuacg ucggcuaccu gcagccuaga acguuccugc ugaaguacaa cgagaacggc 840
accaucaccg acgccgucga cugcgcccug gacccccucu ccgagacaaa augcacccug 900
aagagcuuca cuguugaaaa gggcaucuac cagaccagca acuuuagagu gcagccuaca 960
gagucuaucg ugagauuccc uaacauuacc aaccuguguc cuuuuggaga aguguucaac 1020
gccacaagau ucgcuucugu guaugccugg aaccggaaga gaaucucgaa cugcguggcu 1080
gauuacagcg ugcuguacaa cagcgcuagc uuuagcacau uuaagugcua cggcgugagc 1140
cccaccaagc ugaaugauuu gugcuucaca aauguguacg ccgacucuuu cgugauaaga 1200
ggggacgagg ugcggcagau agcuccaggc cagaccggca acaucgccga uuacaauuac 1260
aagcugccug acgacuuuac cggaugugug aucgccugga acagcaacaa ccuggauagc 1320
aaggugggcg gaaacuacaa cuaccuguac agacuguucc ggaaaucuaa ccuuaagccu 1380
uuugagcggg auaucagcac cgagaucuac caagcuggcu cuacacccug caacggcgug 1440
aagggguuua auuguuacuu cccccugcag agcuacggcu uccaaccgac cuacggagug 1500
ggcuaccagc ccuaccgggu cguggugcug agcuuugagc ugcugcacgc cccugcuaca 1560
gugugcggcc ccaagaaguc uacgaaccug gugaagaaca agugugugaa uuuuaauuuc 1620
aacggacuga ccggcacagg cguccugacc gaaucuaaca agaaauuccu cccuuuccag 1680
caguucggga gagauaucgc cgacaccacc gacgccgugc gggacccuca aacacuggaa 1740
auccuggaua ucaccccuug uucuuucgga ggcguguccg ugaucacccc agguacgaac 1800
acaucuaacc agguggcugu gcuguaccag ggcgugaacu gcaccgaggu gccuguggcc 1860
auucacgccg accagcugac uccuaccugg cggguguaca gcacgggcuc caacguguuu 1920
cagaccagag cuggcugucu gaucggagcc gagcacguga acaacucuua ugagugcgau 1980
auccccaucg gcgcuggaau cugugccucc uaccagacuc aaaccaacag cccuggcagc 2040
gcuagcagcg uggccagcca gagcaucauc gccuacacca ugagccuggg agucgaaaac 2100
agcguggccu acucaaacaa cuccaucgcu aucccuacca acuucaccau cagcguaacg 2160
accgaaaucc ugcccgugag caugaccaag accagcgugg acugcacaau guacaucugc 2220
ggcgauagca cagaaugcag caaucugcua cugcaguacg guagcuuuug cacccaacug 2280
aauagagccc ugaccggcau cgccguggaa caggauaaaa acacccaaga ggucuucgcu 2340
caggugaagc agaucuacaa gacaccuccc aucaaggacu ucggaggauu caacuuuagc 2400
cagauccugc cugauccaag caaaccuagc aagcggaguc cuaucgagga ccugcuguuu 2460
aacaagguga cacuggccga cgccggcuuc aucaagcagu auggcgacug ucugggcgac 2520
aucgccgcca gggaucugau cugugcccaa aaauucaacg gccugacagu gcugccaccu 2580
cugcugaccg acgagaugau cgcucaauac accagcgccc uccucgccgg cacgaucacc 2640
agcggcugga cauucggcgc cggcccugcc cuccagaucc cuuucccuau gcagauggcc 2700
uacagauuca acggcaucgg cgugacacaa aacgugcugu acgaaaacca gaagcugauc 2760
gccaaucagu uuaauagcgc caucgggaag auccaggaua gccugucauc uaccccuucu 2820
gcccugggaa agcugcagga cguggugaac cagaacgccc aggcccugaa cacccuggug 2880
aaacagcugu cuagcaacuu cggcgcuauc agcagcgugc ugaaugauau ccugagcaga 2940
cuggauccuc cugaggccga ggugcagauc gacagauuga ucaccggccg gcugcagagc 3000
cugcaaaccu acguuacaca gcagcugauc agagccgcug aaaucagagc cucugccaac 3060
cuggccgcca ccaaaaugag cgagugcgug cugggacaga gcaaaagggu ggacuucugc 3120
gggaagggcu accaccucau gaguuuuccc cagagcgccc cccacggcgu gguguuccug 3180
cacgugacau augucccggc ccaggagaaa aacuuuacaa cagccccugc cauuugccau 3240
gacggaaagg cccacuuccc ucgggaaggu guguucguga gcaacggcac acacugguuc 3300
gugacccaga gaaacuucua cgagccucaa aucaucacca cagacaacac cuucguuagu 3360
ggaaauugcg acgugguuau cggcaucgug aacaacaccg ucuacgaccc acugcagccu 3420
gaacuggaua gcuucaagga ggaacuggau aaguauuuca agaaccacac cucccccgac 3480
guggaucugg gcgacauuag cggcaucaac gccagcgugg ugaacaucca gaaagagauc 3540
gauagacuua augagguggc caagaaccug aacgagagcc ugaucgaccu gcaggagcuc 3600
ggcaaauacg agcagggcua uaucccagag gccccuagag auggccaggc cuacguuaga 3660
aaggacggcg aguggguccu gcugagcaca uuccugggcu ga 3702
<210> 9
<211> 4093
<212> RNA
<213> Artificial Sequence
<220>
<223> 人工序列
<400> 9
gggagaccgg ccucgagaca uuugcuucug acacaacugu guucacuagc aaccucaaac 60
agacaccaag cuugccacca uguucguguu ccuggugcug cuuccccugg ucucuagcca 120
gugcgugaac uucacgaccc ggacccaacu gccccccgcg uacacaaacu ccuucaccag 180
aggcguguac uacccugaca agguguuccg cagcagcgug cugcacagca cccaggaccu 240
guuccuccca uucuucagca acgugaccug guuccacgcc auccacgugu ccggcaccaa 300
uggaacaaag agauuugcga accccgugcu accuuucaac gacggcgugu acuucgccuc 360
caccgagaag agcaacauca uccggggcug gaucuucggc accacccugg acucuaaaac 420
ccagagccug cugaucguga auaaugccac caacguggug aucaaggugu gcgaguucca 480
guucugcaac gacccuuucc ugggcgucua cuaccacaag aacaacaaga guuggaugga 540
aagcgaguuc agaguguacu cuucugcuaa caacugcacc uucgaguacg ugucccagcc 600
uuuccugaug gaccuggaag gcaagcaggg gaacuucaag aaccugcggg aguucguguu 660
caagaacauc gacggguauu ucaagaucua cuccaagcac acaccuauca aucuggugag 720
aggccugccc cagggcuuca gcgcccugga accucugguc gaccugccaa ucggcaucaa 780
caucacccgg uuccaaacac ugcauaucag cuaccugaca ccuggcgaua gcuccuccgg 840
cuggaccgcc ggcgcugccg cuuauuacgu cggcuaccug cagccuagaa cguuccugcu 900
gaaguacaac gagaacggca ccaucaccga cgccgucgac ugcgcccugg acccccucuc 960
cgagacaaaa ugcacccuga agagcuucac uguugaaaag ggcaucuacc agaccagcaa 1020
cuuuagagug cagccuacag agucuaucgu gagauucccu aacauuacca accugugucc 1080
uuuuggagaa guguucaacg ccacaagauu cgcuucugug uaugccugga accggaagag 1140
aaucucgaac ugcguggcug auuacagcgu gcuguacaac agcgcuagcu uuagcacauu 1200
uaagugcuac ggcgugagcc ccaccaagcu gaaugauuug ugcuucacaa auguguacgc 1260
cgacucuuuc gugauaagag gggacgaggu gcggcagaua gcuccaggcc agaccggcaa 1320
caucgccgau uacaauuaca agcugccuga cgacuuuacc ggauguguga ucgccuggaa 1380
cagcaacaac cuggauagca aggugggcgg aaacuacaac uaccuguaca gacuguuccg 1440
gaaaucuaac cuuaagccuu uugagcggga uaucagcacc gagaucuacc aagcuggcuc 1500
uacacccugc aacggcguga agggguuuaa uuguuacuuc ccccugcaga gcuacggcuu 1560
ccaaccgacc uacggagugg gcuaccagcc cuaccggguc guggugcuga gcuuugagcu 1620
gcugcacgcc ccugcuacag ugugcggccc caagaagucu acgaaccugg ugaagaacaa 1680
gugugugaau uuuaauuuca acggacugac cggcacaggc guccugaccg aaucuaacaa 1740
gaaauuccuc ccuuuccagc aguucgggag agauaucgcc gacaccaccg acgccgugcg 1800
ggacccucaa acacuggaaa uccuggauau caccccuugu ucuuucggag gcguguccgu 1860
gaucacccca gguacgaaca caucuaacca gguggcugug cuguaccagg gcgugaacug 1920
caccgaggug ccuguggcca uucacgccga ccagcugacu ccuaccuggc ggguguacag 1980
cacgggcucc aacguguuuc agaccagagc uggcugucug aucggagccg agcacgugaa 2040
caacucuuau gagugcgaua uccccaucgg cgcuggaauc ugugccuccu accagacuca 2100
aaccaacagc ccuggcagcg cuagcagcgu ggccagccag agcaucaucg ccuacaccau 2160
gagccuggga gucgaaaaca gcguggccua cucaaacaac uccaucgcua ucccuaccaa 2220
cuucaccauc agcguaacga ccgaaauccu gcccgugagc augaccaaga ccagcgugga 2280
cugcacaaug uacaucugcg gcgauagcac agaaugcagc aaucugcuac ugcaguacgg 2340
uagcuuuugc acccaacuga auagagcccu gaccggcauc gccguggaac aggauaaaaa 2400
cacccaagag gucuucgcuc aggugaagca gaucuacaag acaccuccca ucaaggacuu 2460
cggaggauuc aacuuuagcc agauccugcc ugauccaagc aaaccuagca agcggagucc 2520
uaucgaggac cugcuguuua acaaggugac acuggccgac gccggcuuca ucaagcagua 2580
uggcgacugu cugggcgaca ucgccgccag ggaucugauc ugugcccaaa aauucaacgg 2640
ccugacagug cugccaccuc ugcugaccga cgagaugauc gcucaauaca ccagcgcccu 2700
ccucgccggc acgaucacca gcggcuggac auucggcgcc ggcccugccc uccagauccc 2760
uuucccuaug cagauggccu acagauucaa cggcaucggc gugacacaaa acgugcugua 2820
cgaaaaccag aagcugaucg ccaaucaguu uaauagcgcc aucgggaaga uccaggauag 2880
ccugucaucu accccuucug cccugggaaa gcugcaggac guggugaacc agaacgccca 2940
ggcccugaac acccugguga aacagcuguc uagcaacuuc ggcgcuauca gcagcgugcu 3000
gaaugauauc cugagcagac uggauccucc ugaggccgag gugcagaucg acagauugau 3060
caccggccgg cugcagagcc ugcaaaccua cguuacacag cagcugauca gagccgcuga 3120
aaucagagcc ucugccaacc uggccgccac caaaaugagc gagugcgugc ugggacagag 3180
caaaagggug gacuucugcg ggaagggcua ccaccucaug aguuuucccc agagcgcccc 3240
ccacggcgug guguuccugc acgugacaua ugucccggcc caggagaaaa acuuuacaac 3300
agccccugcc auuugccaug acggaaaggc ccacuucccu cgggaaggug uguucgugag 3360
caacggcaca cacugguucg ugacccagag aaacuucuac gagccucaaa ucaucaccac 3420
agacaacacc uucguuagug gaaauugcga cgugguuauc ggcaucguga acaacaccgu 3480
cuacgaccca cugcagccug aacuggauag cuucaaggag gaacuggaua aguauuucaa 3540
gaaccacacc ucccccgacg uggaucuggg cgacauuagc ggcaucaacg ccagcguggu 3600
gaacauccag aaagagaucg auagacuuaa ugagguggcc aagaaccuga acgagagccu 3660
gaucgaccug caggagcucg gcaaauacga gcagggcuau aucccagagg ccccuagaga 3720
uggccaggcc uacguuagaa aggacggcga guggguccug cugagcacau uccugggcug 3780
agaauucgcu ggagccucgg uagccguucc uccugcccgc ugggccuccc aacgggcccu 3840
ccuccccucc uugcaccggc ccuuccuggu cuuuggcugg agccucggua gccguuccuc 3900
cugcccgcug ggccucccaa cgggcccucc uccccuccuu gcaccggccc uuccuggucu 3960
uuguuaauua aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4020
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 4080
aaaaaaaaac uag 4093
- 使用脂质纳米颗粒用于递送编码VEGF-A多肽的经修饰的RNA的方法和包含其的药物组合物
- 一种mRNA脂质纳米颗粒递送系统及其制备方法和应用
