医学文本中对基因名称的标准化的方法和系统
文献发布时间:2023-06-19 11:21:00
技术领域
本公开内容总体上涉及用于对基因名称进行识别和标准化的方法和系统。
背景技术
鉴于对精确医学的日益关注(包括以具有特定遗传变体的患者的治疗性处置为目标的临床试验的数量不断增加),从患者病历和临床试验描述中提取相关信息必须包括对特定基因测试和变体的提及内容的标识和提取。
基因名称通常是对医学文本中旨在标识对特定基因的提及内容。这样的提及内容的功能类似于文本中的名词短语,并且其内部构成尊重英语或其他自然语言的敏感性。但是它的命名通常由高科技的分子和临床词汇、子专业符号、其他术语以及类似首字母缩写的符号组成。因此,基因名称通常是自然语言与特殊符号之间的混合体。识别基因名称涉及定位这种提及内容,将其分类为基因名称,并且识别特定的引用基因。
除了识别基因名称之外,对基因名称进行标准化也有益处。对基因名称进行标准化通常需要理解标识同一基因的可能提及内容的范围,了解或确定这些提及内容中的一个提及内容是该范围的首选代表(“规范名称”),并且将观察到的提及内容与其首选代表相关联。
现有的系统试图处理对基因名称的识别和标准化。例如,具有基因命名的多种规范和标准,基因名称及其之间的关系的多个数据库,以及结合文本语料库中的命名规则、数据库条目和实际命名模式的多种机器学习系统。然而,由于基因命名模式和上下文大量产生并且持续演变,因此这些方法的查全率和查准率通常在实践中最多只能达到70的最高水平。因此,基因名称识别器/标准化器需要依赖于外部资源和工具,并且需要复杂的机制和数据,但性能却不高。
发明内容
持续需要以更高的准确度和效率来识别与基因名称相关联的各种各样的命名并将这些基因名称变体转换为规范形式的方法和系统。本文的各种实施例和实施方式涉及被配置为标识在各种可能源中找到的基因标识符的规范基因名称的方法和系统。所述系统将来自源的文本标记化为标记流,并且将来自所述标记流的第一标记与根据基因标识符和针对多个基因中的每个基因的对应规范基因名称的数据库生成的数据结构进行比较。所述数据结构包括具有根节点、多条边和多个叶节点的前缀树结构。为了在所述标记流中标识基因标识符以及与所标识的基因标识符相关联的规范名称,所述系统确定所述第一标记匹配从所述根节点延伸到相关联的第一叶节点的多条边中的哪条边。如果所述标记匹配所述多条边中的任一条边,则利用与匹配的边相关联的所述第一叶节点的位置来更新以促进对规范基因名称的标识为任务的标识指针。所述标记然后遍历树结构,更新所述标识指针。例如,如果有的话,所述系统确定来自所述标记流的后续的第二标记匹配从所述第一叶节点延伸到第二叶节点的多条边中的哪条边。如果所述第二标记匹配从所述第一叶节点延伸的边,则利用与匹配的边相关联的所述第二叶节点的位置来更新所述标识指针。使用延伸的边和节点对来自所述标记流的后续标记重复执行该操作,直到后续标记无法匹配从叶节点延伸的边为止。当后续标记无法匹配从叶节点延伸的边时,基于所述标识指针的最新位置来提供对所述规范基因名称的标识。
通常,一方面,提供了一种用于对基因命名进行标准化的方法。所述方法包括:(i)接收包括一个或多个基因标识符的源;(ii)将来自所述源的文本标记化为标记流;(iii)将来自所述标记流的第一标记与根据基因标识符和针对多个基因中的每个基因的对应规范基因名称的数据库生成的数据结构进行比较,所述数据结构包括具有根节点、多条边和多个叶节点的前缀树结构;(iv)确定所述第一标记匹配从所述根节点延伸到相关联的第一叶节点的一条或多条边中的哪条边;(v)利用与匹配的边相关联的所述第一叶节点的位置来更新标识指针;(vi)如果有的话,确定来自所述标记流的后续的第二标记匹配从所述第一叶节点延伸到第二叶节点的一条或多条边中的哪条边;(vii)如果所述第二标记匹配从所述第一叶节点延伸的边,则利用与匹配的边相关联的所述第二叶节点的位置来更新所述标识指针;(viii)使用任何额外的延伸的边和叶节点对来自所述标记流的后续标记重复执行所述确定步骤和所述更新步骤,直到后续标记无法匹配从叶节点延伸的边或者没有从所述叶节点延伸的边为止;并且(ix)当后续标记无法匹配从叶节点延伸的边时或者如果没有从所述叶节点延伸的边,则基于来自所述标识指针的最新位置来提供对规范基因名称的标识。
根据实施例,所述方法还包括:生成基因标识符和相关联的规范基因名称的策展表(curated table);将所述策展表标记化为标记流;并且使用所述标记流来生成所述前缀树结构。
根据实施例,将所述策展表标记化为标记流以及将来自所述源的文本标记化为标记流利用了相同的标记化逻辑。
根据实施例,所述方法还包括:生成语言中的最常用词的列表;将所述最常用词的列表与所述策展表中的所述基因标识符进行比较;并且识别在所述最常用词的列表中找到的任何基因标识符。
根据实施例,基于来自所述标识指针的最新位置来提供对规范基因名称的标识包括:利用最新的匹配的叶节点上游的叶节点的位置来更新所述标识指针。
根据实施例,所述方法还包括确定所标识的规范基因名称是否也是常用自然语言词的步骤。
根据实施例,标记化包括:为文档中的每一个大写短语生成首字母缩略词;并且防止生成的首字母缩略词的每次出现或者从所述标记流中去除生成的首字母缩略词的每次出现。
根据实施例,标记化包括:生成每个罗马字母字符串的单个标记;生成每个数字序列的单个标记;并且利用每个希腊字母生成单个标记。
根据实施例,提供对规范基因名称的标识包括关于所述源中的基因标识符的位置的信息。
通常,在第二方面是一种用于对基因命名进行标准化的系统。所述系统包括:源,其包括一个或多个基因标识符;数据结构,其是根据基因标识符和针对多个基因中的每个基因的对应规范基因名称的数据库生成的,所述数据结构包括具有根节点、多条边和多个叶节点的前缀树结构;以及处理器,其被配置为:(i)将来自所述源的文本标记化为标记流;(ii)将来自所述标记流的第一标记与所述数据结构进行比较;(iii)确定所述第一标记匹配从所述根节点延伸到相关联的第一叶节点的一条或多条边中的哪条边;(iv)利用与匹配的边相关联的所述第一叶节点的位置来更新标识指针;(v)如果有的话,确定来自所述标记流的后续的第二标记匹配从所述第一叶节点延伸到第二叶节点的一条或多条边中的哪条边;(vi)如果所述第二标记匹配从所述第一叶节点延伸的边,则利用与匹配的边相关联的所述第二叶节点的位置来更新所述标识指针;(vii)使用任何额外的延伸的边和叶节点对来自所述标记流的后续标记重复执行所述确定和所述更新,直到后续标记无法匹配从叶节点延伸的边或者没有从所述叶节点延伸的边为止;并且(viii)当后续标记无法匹配从叶节点延伸的边时或者如果没有从所述叶节点延伸的边,则基于来自所述标识指针的最新位置来提供对规范基因名称的标识。
将意识到,前述概念和下面更加详细讨论的额外概念的所有组合(假设这样的概念并不相互矛盾)都被认为是本文公开的发明主题的部分。特别地,出现在本公开内容的结尾处的所要求保护的主题的所有组合都被认为是本文公开的发明主题的部分。还将意识到,本文明确采用的,也可能出现在通过引用并入的任何公开内容中的术语应当被赋予与本文公开的特定概念最一致的含义。
参考下文描述的(一个或多个)实施例,各种实施例的这些方面和其他方面将变得显而易见并且得到阐明。
附图说明
在附图中,贯穿不同的视图,相似的附图标记通常指代相同的部分。而且,附图并不一定是按比例绘制的,而是通常将重点放在说明各种实施例的原理上。
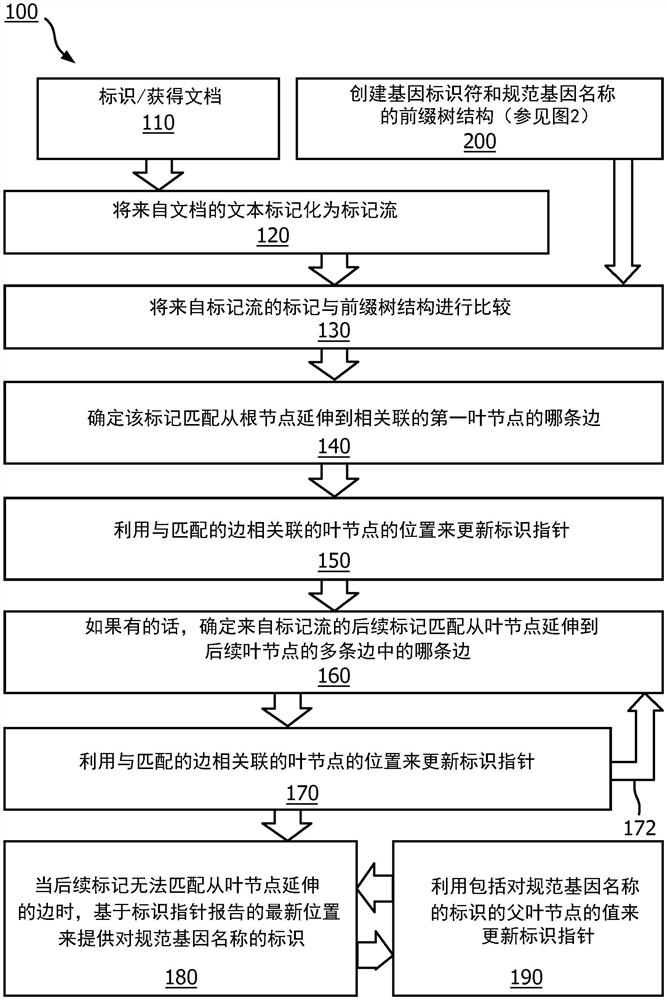
图1是根据实施例的用于标识规范基因名称的方法的流程图。
图2是根据实施例的用于创建基因标识符树结构的方法的流程图。
图3是根据实施例的基因标识符树结构。
图4是根据实施例的用于标识规范基因名称的方法的流程图。
图5是根据实施例的用于标识规范基因名称的系统的示意图。
具体实施方式
本公开内容描述了被配置为对在各种可能源中找到的各种各样的基因标识符定位规范基因名称的系统和方法的各种实施例。更一般而言,申请人已经认识并意识到:提供更准确且更有效地标识规范基因名称的系统将是有益的。该系统对来自文档或其他源的信息进行标记化,并且将来自标记流的第一标记与根据基因标识符和针对多个基因中的每个基因的对应规范基因名称的数据库生成的数据结构进行比较。数据结构是具有根节点、多条边和多个叶节点的前缀树结构。为了在标记流中标识基因标识符以及与所标识的基因标识符相关联的规范名称,该系统确定第一标记匹配从根节点延伸到相关联的第一叶节点的多条边中的哪条边。如果该标记匹配多条边中的任一条边,则使用与匹配的边相关联的第一叶节点的位置来更新以促进对规范基因名称的标识为任务的标识指针。该标记然后遍历树结构,更新标识指针。例如,如果有的话,该系统确定来自标记流的后续的第二标记匹配从第一叶节点延伸到第二叶节点的多条边中的哪条边。如果第二标记匹配从第一叶节点延伸的边,则利用与匹配的边相关联的第二叶节点的位置来更新标识指针。使用延伸的边和节点对来自标记流的后续标记重复执行该操作,直到后续标记无法匹配从叶节点延伸的边为止。当后续标记无法匹配从叶节点延伸的边时,将基于标识指针的最新位置来提供对规范基因名称的标识。
识别基因标识符并将这些基因标识符标准化为规范名称提出了各种各样的挑战。例如,通常用于自然语言的匹配方法并不适用于带有特殊标记的命名。保持等效性的可用变体种类较少,并且在非词代码中,即使是单个字母或数字的改变也可能会改变含义。另外,短语基因名称可能包含内部词,可以在不改变基因标识符的情况下用等效的同义词替换该内部词(例如,将“……铁硫……”替换为“……Fe-S……”)。在某些情况下,基因标识符中的通用内部词可能是任选的,并且可能会被完全丢弃。
基因标识符可能包含内部非词符号,可以在不改变标识符的身份的情况下用等效的同义词替换该内部非词符号。例如,在特定上下文中,小写希腊字母β可以被替换为罗马字母B或罗马字母名称beta。例如,同一基因可以被称为“TGFβGF”、“TGFB1”、“TGFbeta”和“转化生长因子,beta1”。
基因标识符可能包含标点符号,可以在不改变基因标识符的情况下用不同的标点符号或用空格替换该标点符号,或者甚至完全丢弃该标点符号。最后的“+”或“-”(例如,“HER2+”)指示行为或存在,因此不是基因名称的部分。相比之下,带括号的子表达式可能完全是内部的,也可能是名称初始的或名称最后的,并且是基因标识符的部分。例如,诸如“P138(TOX)”和“C-P4Halpha(II)”之类的基因标识符不容易被自动化系统标记化或识别。较小的基因标识符可以与非名称词进行字符连接(例如,“BRAF相关”、“p53缺陷型”、“非CCND1族群”、“HER2过表达”、“HER2阳性”和“孕激素受体调节”)或者与单独的基因名称进行字符连接(例如在其他示例中描述融合)。作为另一示例,罗马字母大小写的变化可能会偏离适当且易用的标准,而不会改变引用对象的身份。
另外,可以经由在多片基因标识符上的分散式表达来联合提及多个基因。该表达指示必须重建其单独名称的基因的成对或列表或分离或结合。示例包括“CCR2/5”、“HLA-A、B或C”和“HLA-A、B、C、DRB1和DQB1”。
识别基因标识符时面临的另一问题是:类似于首字母缩写词的基因符号也可以是英语或其他自然语言中的词,或者也可以是指代某些非基因/非蛋白质引用对象的首字母缩写词。通常,为了消除歧义和正确标识,可能需要在基因标识符本身之外的上下文。例如,句子“肺对一氧化碳的弥散功能(DLCO)<40%(校正后),肺总量(TLC)<40%,一秒钟的用力呼气量(FEV1)<40%和/或接收补充连续氧气”并不包括任何由缩写词的上下文示出的基因标识符,但是许多自动化系统会将DLCO、TLC和/或FEV1标识为基因标识符。
除了基因标识符识别之外,基因名称标准化或规范化还存在若干问题。例如,基因名称标准化需要主要知识源,而不是基因名称的许多不同源。具有多个源会导致不同的基因标识符,这些不同的基因标识符与任何规则绑定的转换都不相关,并且无法从上下文统计数据中得到充分学习。
另外,非规范基因名称可能对于其所指代的基因而言是有歧义的。也就是说,所提及的名称本身可能具有一个以上的候选规范名称,这取决于它所指代的是哪个基因。为了消除歧义,需要在提及内容之外的上下文。因此,虽然一个基因具有唯一的规范名称,但是一个基因名称可能会映射到多个规范名称。即使将基因名称的范围限制为排除对明确的非人类基因的引用,这种歧义仍然存在。
因此,本文描述的或以其他方式设想的基因名称标识系统和方法专门解决了与对基因标识符的识别和标准化有关的这些已知问题和其他已知问题。
参考图1,在一个实施例中,图1是使用基因名称标识系统在经标记化的源中标识规范基因名称的方法100的流程图。结合附图描述的方法仅被提供作为示例,并且应当被理解为不限制本公开内容的范围。基因名称标识系统能够是本文描述的或以其他方式设想的任何系统。
在该方法的步骤110处,由基因名称标识系统获得或接收一个或多个文档或其他源。这些文档或其他源能够是任何文本、文档或其他包括文本或图像的记录或源。根据优选实施例,源是数字源或数字化源。例如,源可以是期刊、数据库、临床信息、实验室报告和/或可以包含或可能包含基因标识符的任何其他信息。这些仅是示例,并不意味着详尽无遗。源能够由个人或另一系统提供给基因名称标识系统。额外地和/或替代地,能够由基因名称标识系统来检索源。例如,基因名称标识系统可以连续地或周期性地访问数据库、网站或包括或提供源的任何其他资源。仅作为一个示例,基因名称标识系统可以访问研究文章的私有或公共储存库,例如,PubMed或任何其他数据库。
所接收或获得的源可以被存储在本地或远程数据库中,以供基因名称标识系统使用。例如,基因名称标识系统可以包括用于存储源的数据库,并且/或者可以与存储源的数据库通信。这些数据库可以与基因名称标识系统定位在一起,或者可以被定位为远离基因名称标识系统,例如被定位在云存储装置和/或其他远程存储装置中。
在该方法的步骤120处,基因名称标识系统对来自所获得或接收的文档或其他源的信息进行标记化。基因名称标识系统可以使用促进对下游基于文本的分析的任何用于处理的方法或系统来准备用于标记化的源。该处理可以包括例如从源中标识和/或提取文本,特别是在源包括除了文本之外的内容(例如,图像、表或其他非文本内容)的情况下。该处理还可以包括对所提取的文本的标准化,对提取文本的翻译以及许多其他形式或种类的处理。可以将经处理的基于文本的源或其中的经处理的内容存储在本地或远程存储装置中,以用于该过程的后续步骤。基因名称标识系统可以在接收每个基于文本的源时对其进行处理,或者可以批量处理基于文本的源,或者可以在该方法的后续步骤中对基于文本的源进行分析之前就处理它。
如本文描述的或以其他方式认识到的,基因标识符能够以无限数量的格式和变体出现,因此合适的标记化是对基因标识符的识别和标准化中的关键步骤。因此,基因名称标识标记器配置为以优化基因标识符识别的方式对来自所获得或接收的文档或其他源的数据进行标记化。可以利用各种标记器和/或标记化方法。
根据实施例,标记器包括以下设置、配置或阈值中的一个或多个。标记器可以被配置为构造非基因组特殊符号(例如,时间、日期和数字)的每次出现的单个标记。标记器可以被配置为生成每串罗马字母的单个标记,并且单独地生成每串数字的单个标记。例如,短语MEKK4可以被标记化以生成标记“MEKK”和“4”。
根据实施例,标记器可以被配置为生成每个希腊字母或其他特殊符号的标记。因此,基因标识符p38α可以被标记化为“p”、“38”和“α”。标记器可以被配置为生成所有其他剩余字符的标记,同时生成空白的每一个流中断运行的标记,从而生成段落标记。根据实施例,从标记流中去除所有其他空白。在一些实施例或变体中,空格可以是任选的。
根据实施例,其他剩余字符可以被进一步处理。例如,标记器可以被配置为过滤掉一些标点符号,例如,字符连接符。标记器可以被配置为将所有罗马字母的大小写都折叠为小写。例如,“MEKK”可以被标记化为“mekk”。标记器还可以被配置为将希腊字母的每个罗马名称折叠为希腊字母本身。例如,p38alpha可以被标记化为“p”、“38”和“α”。
根据实施例,标记器可以包括轻量级解析器,该轻量级解析器用于经由诸如“Xs<--X”和“Xs<--Xs(,)?(and)?X”等迭代操作来构成连接词或分离词的列表。
标记器或标记化指令可以利用各种各样的机制来促进对基因标识符的改善的标记化,这随后改善了规范基因名称标识。标记化可能会遇到困难,例如当遇到任选的内部词、同义词替换,罗马字母被替换为希腊字母以及与非基因首字母缩写的歧义时。
根据实施例,标记器可以通过为相同文本中的每个大写短语生成缩写词(就像它是专有名称一样)并征集那些被误认为基因名称的词的出现来过滤掉非基因缩写词。根据实施例,标记器可以通过简单的标记级别的过滤和折叠来管理任选词和同义词,这里在词级别上具有小的词汇量。根据另一实施例,标记器可以利用试探法来用罗马字母代替希腊字母,例如仅尝试一个罗马字母而不后跟另一字母。对于其他情况,该系统可以将适当的条目添加到策展表中。
在该方法的步骤130处,基因名称标识器系统将来自标记流的标记与被配置为促进基因名称标识的数据结构进行比较。根据实施例,数据结构是包括基因标识符和相关联的规范基因名称的前缀树结构。可以使用任何方法或系统来构造或以其他方式生成诸如前缀树结构之类的数据结构。
参考图2,在一个实施例中,图2是一种用于生成被配置为促进基因名称识别的数据结构,并且特别用于创建基因标识符和规范基因名称的前缀树结构的方法200。
在该方法的步骤210处,由基因名称标识系统获得或接收一个或多个文档或其他源,并且利用这一个或多个文档或其他源来标识基因标识符和规范名称。这些文档或其他源能够是任何文本、文档或其他包括文本或图像的记录或源,这些文本或图像促进标识基因标识符和针对数据结构的规范名称。例如,这可以是来自期刊、数据库、临床信息、实验室报告和/或可以包含或可能包含基因标识符的任何其他信息的语料库。源能够由个人或另一系统提供给基因名称标识系统。额外地和/或替代地,能够由基因名称标识系统来检索源。所接收或获得的源可以被存储在本地或远程数据库中以供在该方法中使用。
在该方法的步骤220处,构建基因标识符的策展表,每个基因标识符都被映射到其规范基因名称。该表能够由审阅者或以关联基因标识符与规范基因名称为任务的其他人员手动地部分或完全生成。某些基因标识符可能会映射到多个规范基因名称,而某些规范基因名称也可能会映射到多个基因标识符。许多其他变体是可能的。
根据实施例,在映射的任一侧上的基因标识符(即,基因标识符或规范名称)可以是短语,这意味着它们由序列中的多个术语或词或符号组成。该表可以是适合于该方法的任何大小,并且可以取决于其基因名称正被规范化的生物和针对该生物生成的多种多样的基因名称以及许多其他特征。例如,表中的基因标识符和规范基因名称的总数可以是成百上千或成千上万的。虽然该信息被标识和描述为“表”,但是应当认识到,该信息能够以各种不同的格式和数据结构来组织和/或存储。
参考表1,表1是包括基因标识符和规范基因名称的列表的数据结构的部分的示例。在该示例中,四个不同的基因标识符“ajuba LIM蛋白”、“JUB”、“jub,ajuba同源物(非洲爪蟾)”和“MGC15563”均指代规范基因名称“AJUBA”。因此,表中的四个不同的基因标识符中的每个基因标识符都与规范基因名称“AJUBA”相关联。由于该表或其他数据结构中的数据的这种关联,对这四个不同的基因标识符中任一个基因标识符的标识将随后标识相关联的规范基因名称“AJUBA”。
表1.基因标识符和相关联的规范基因名称的表
该表或数据结构可以被存储在本地或远程数据库中以供基因名称标识系统使用。例如,基因名称标识系统可以包括数据结构,并且/或者可以与包括该数据结构的存储器通信。因此,数据结构可以与基因名称标识系统定位在一起,或者可以被定位为远离基因名称标识系统,例如被定位在云存储装置和/或其他远程存储装置中。
在该方法的步骤240处,对表中的基因标识符和规范名称进行标记化。根据实施例,用于生成数据结构的标记器与在该方法的步骤120中用于生成标记流以用于基因名称标准化的标记器是相同的标记器,或者利用相同的逻辑。根据另一实施例,用于生成数据结构的标记器与用于生成标记流的标记器具有一个或多个差异。
在该方法的步骤250处,根据来自基因标识符和规范基因名称的表的经标记化的信息来生成数据结构。根据实施例,该数据结构是前缀树结构,并且前缀树结构的键是标记序列。根据实施例,当正在生成数据结构时,当将经标记化的基因标识符添加到数据结构但表中已经存在规范基因名称时,将经标记化的基因标识符的值添加到该键的现有值。因此,每个基因标识符将映射到一组规范名称,而不是每个基因标识符映射到单个规范名称,如表1所示。根据实施例,在将所有键/值对加载到数据结构中之后,通过将每个规范名称映射到其自身来执行闭合。数据结构的构造的这些变化和其他变化是可能的。
参考图3,在一个实施例中,图3是使用来自表1的经标记化的信息的生成或加载的树结构的部分300。该生成或加载的树结构仅是示例性的,因此不限制本公开内容的范围。在整个树结构的该隔离部分中,节点310能够代表根节点。边312a、312b和312c中的每条均被加载有或包括由系统基于表1识别(并且,如本文所讨论的,由系统生成)的三个可能标记之一,即,“ajuba”、“jub”(因为在所有罗马字母在标记化期间都被转换为小写的实施例中,“JUB”和“jub”是等效的)和“MGC”。
边312a和312b分别通向叶节点314a和314b,每个叶节点在相应的基因标识符中包括词或符号。例如,由于“ajuba”和“jub”都可以有效地肯定地标识规范基因名称AJUBA,因此叶节点314a和314b的值就是规范基因名称AJUBA。然而,如果该树结构包括针对AJUBA的可能的基因标识符的额外部件,则该树结构被配置为处理后续标记。例如,标记“jub”可以是标识规范基因名称AJUBA的孤立基因标识符,而标记“jub”也可以是从“jub,ajuba同源物(非洲爪蟾)”提取的标记字符串中的第一标记。因此,如果该字符串中的下一个标记是“,”(逗号),则该标记匹配从叶节点314b延伸到叶节点316的边,该边是占位符,其指示在短语“jub,ajuba同源物(非洲爪蟾)”中的“,”与“ajuba”之间的空格。该占位符还指示树结构是由来自基因标识符的额外的可能信息构造的,但是由于叶节点314b,已经能够肯定地标识为规范基因名称AJUBA。每个字符串终止于包括规范基因名称的叶节点,例如,包括规范基因名称AJUBA的叶节点318。
在该方法的步骤230处,该系统生成包括一些最常用的英语(或其他语言)词的列表的表或其他数据结构。例如,这能够通过基于使用情况标识或检索最常用的英语(或其他语言)词的公共或私人排名或其他列表来实现。根据实施例,除了许多其他可能性之外,该列表还可以基于例如科学文献内的特定用途。可以通过包括一个或多个用户设置、信息源和/或其他因素在内的多种因素来确定在表或其他数据结构中包括的词的数量。例如,在表或其他数据结构中包括的词的数量可以是成百上千或成千上万的。
在该方法的步骤260处,将来自表的基因标识符与包括最常用的英语(或其他语言)词的列表的表或其他数据结构进行比较。例如,这能够使用比较两个数据源的任何方法来完成。在该方法的步骤270处,该系统对被找到既是基因标识符又是最常用的英语(或其他语言)词的任何词进行标识和/或加标记。如本文描述的或以其他方式设想的,当执行基因标识和规范化时,能够使用该列表。
在该方法的步骤280处,如本文描述的或以其他方式设想的,能够使用所生成的前缀树结构来标识针对基因标识符的规范名称。数据结构可以被存储在本地或远程数据库中以供基因名称标识系统使用。例如,基因名称标识系统可以包括数据结构,并且/或者可以与包括该数据结构的存储器通信。因此,数据结构可以与基因名称标识系统定位在一起,或者可以被定位为远离基因名称标识系统,例如被定位在云存储装置和/或其他远程存储装置中。
返回图1,在该方法的步骤140处,基因名称标识系统基于在步骤130的比较来确定被比较的第一标记匹配从根节点延伸到相关联的第一叶节点的多条边中的哪条边。这种确定可以使用用于比较两个数据点并标识匹配的任何方法或算法来执行。根据实施例,可能需要精确匹配,并且/或者可能在匹配中允许模糊性,这能够基于用户设置、系统配置和/或任何其他源。参考图3中的树结构的部分300,仅作为实施例的一个示例,该系统确定第一标记匹配从该代表性根节点310延伸的多条边312a、312b和312c中的哪条边。如果该标记匹配边312a、312b和312c之一,则基因名称标识系统确定存在匹配,并且该方法能够进行到下一步骤。
参考图3中的树结构的部分300,在一个实施例中,标记“mgc”(来自基因标识符MGC15563)是正在与前缀数据结构进行比较的标记流中的部分。“mgc”标记匹配边312c,因此该系统确定该标记与该边之间存在匹配。
在该方法的步骤150处,利用由该系统使用标记流标识的最新的叶节点的位置、值或其他指示符来更新标识指针。标识指针能够是任何指针、数据点或用于记录或以其他方式通知或定位沿着数据结构300的位置的其他机制。例如,标识指针能够是缓存数据点,该缓存数据点会随着每个新标识或遍历的叶节点而更新。标识指针的默认值可以是数据结构的根节点,并且仅当标记与从根节点延伸到叶节点的边之间存在匹配时才可以更新。根据实施例,标识指针用于标识与基因标识符相关联的规范基因名称。
例如,参考图3,其中,标记“mgc”匹配边312c,利用叶节点320的值、位置或其他指示符来更新标识指针。由于叶节点320并不包括规范基因名称,因此标识指针的该值尚未标识规范基因名称。
在该方法的步骤160处,该系统将来自标记流的下一个标记与从当前叶节点延伸的一条或多条边进行比较,以确定下一个标记匹配这一条或多条边中的哪条边(如果有的话)。这种确定可以使用用于比较两个数据点并标识匹配的任何方法或算法来执行。根据实施例,可能需要精确匹配,并且/或者可能在匹配中允许模糊性,这能过基于用户设置、系统配置和/或任何其他源。再次参考图3中的树结构的部分300,在一个实施例中,第一标记(“mgc”)标识叶节点320。将下一个标记(“15563”)与从叶节点320延伸的一条边322进行比较,以确定边322与标记“15563”之间是否存在匹配。在该示例中,该系统确定存在匹配。
在该方法的步骤170处,利用由该系统使用标记流标识的新的叶节点的位置、值或其他指示符来更新标识指针。例如,如果标识指针是利用每个新标识或遍历的叶节点更新的缓存数据点,则利用新的叶节点信息来更新该数据点。再次参考图3中的树结构的部分300,在一个实施例中,其中,标记“15563”匹配边322,利用叶节点324的值、位置或其他指示符来更新标识指针。在该示例中,叶节点324的值或位置是jujub,它是针对基因标识符MGC15563的规范基因名称,基因标识符MGC15563被标记化为“mgc”和“15563”。
在图1的172处,任选地,使用任何额外的延伸的边和叶节点对来自标记流的后续标记重复执行步骤160和170,直到后续标记无法匹配从叶节点延伸的边或者没有从叶节点延伸的边为止。例如,被最新标记遍历的最新的叶节点——因此是标识指针的最新位置或值——可能是树的分支的末端,在这种情况下,没有延伸的边。替代地,标记流中的下一个标记可能并不匹配从叶节点延伸的一条或多条边中的任一条边,因为针对规范基因名称的基因标识符家族的某些成员包括任选的额外用语、符号、数字或并非在所有基因标识符中都找到的其他术语,或者是因为标记可能是该基因标识符家族中以前未知或未分类的基因标识符,或者是因为标记可能是与先前的基因标识符没有关联的单独的词或后续基因标识符,或者还有许多其他原因。
再次参考图3中的树结构的部分300,在一个实施例中,最近利用叶节点324的值、位置或其他指示符更新了标识指针,标识指针的值是ajuba,即,针对基因标识符MGC15563的规范基因名称。在该示例中,叶节点324是树300的该分支的末端,因此没有从叶节点324延伸的边。因此,没有更多的边或叶节点要遍历,并且标识指针包括该分支的末端值或规范基因名称。
在该方法的步骤180处,当后续标记无法匹配从匹配的叶节点延伸的边时或者如果没有从该匹配的叶节点延伸的边,如果标识是可能的,则该系统基于标识指针的最新位置来提供或以其他方式标识或生成所确定的规范基因名称。例如,如果在一个实施例中图3中的树结构的部分300是完整的树并且第一标记是与从根节点延伸的边中的任一条边都不匹配的“mekk”,则将不会更新标识指针并且无法标识所确定的规范基因名称。标识指针的值可能保留默认值,也可能为空(其指示未标识规范基因名称)。
根据实施例,能够提供、记录、报告规范基因名称的确定结果或值或者以其他方式使之可供使用或利用。例如,该系统能够将标识指针的值或位置(其可以是所确定的规范基因名称)提供给另一数据点、表、报告或其他位置。例如,该系统可以创建在源中标识的所有规范基因名称的表或列表。这可以在存储器或数据库中创建,在屏幕或其他用户接口上显示或者以其他方式提供。报告或列表还可以包括用于标识规范基因名称的一个或多个基因标识符以及关于标记或标记在被分析的源中的位置的信息。报告可以是视觉显示、印刷文本、电子邮件、可听报告、传输内容和/或任何其他传送信息的方法。报告可以在本地或远程提供,因此该系统或用户接口可以包括通信系统或者以其他方式连接到通信系统。例如,该系统可以通过诸如互联网或其他网络之类的通信系统来传达报告。许多提供、记录、报告规范基因名称或者以其他方式使之可供使用或者使用规范基因名称的许多其他方法也是可能的。根据实施例,在提供了规范基因名称的确定结果或值之后,能够将标识指针返回到默认值或空值。
如果叶节点包括规范基因名称,则只能利用该叶节点的值来更新标识指针。替代地,可以利用每一个遍历的叶节点的值来更新标识指针,在这种情况下,标识指针可以包括不是规范基因名称的值或位置。例如,参考图3,如果已经使用标记“ajuba”和“lim”来利用叶节点326的值或位置更新标识指针,则该值或位置不是规范基因名称。如果标记流中的下一个标记是“alpha”而不是预期的“蛋白质”,则没有与边328的匹配,并且不会利用任何其他叶节点的值来更新标识指针。然而,标识指针包括不是规范基因名称的值或位置。因此,在该方法的步骤190处,该系统利用包括对规范基因名称的标识的父叶节点或上游叶节点的值来更新标识指针,或者以其他方式报告该父叶节点或上游叶节点的值或位置。再次参考图3中的示例,其中,标识指针的最后的值或位置是叶节点326(其与规范基因名称没有关联),该系统可以指示利用包括对规范基因名称的标识的最接近的父叶节点或上游叶节点来更新标识指针,该包括对规范基因名称的标识的最接近的父叶节点或上游叶节点将是具有规范基因名称“ajuba”的叶节点314a。
根据实施例,如果标记并不匹配从根延伸的任一条边,则该系统可以确定是否尝试任何其他注释器或分析。如果不是,则该系统可以丢弃该标记并移动到下一个标记,从而利用该下一个标记重复执行文本描述的或以其他方式设想的方法。
根据实施例,如果所标识的规范基因名称也是在常用词列表中找到的词(如根据图2中的方法200的步骤270从加标记的列表找到的词),则所标识的规范基因名称能够被加标记、标注或者以其他方式指示。例如,这可以指示所标识的规范基因名称是基因与英语(或其他语言)有歧义的,这能够使标识和/或标准化变得复杂。
根据实施例,该系统可以在该系统已经暂停或者似乎在基因标识符的末尾的任何地方利用轻量级解析器来组成或标识连接词或分离词。该轻量级解析器可以利用诸如“Xs<--X”和“Xs<--Xs(,)?(and)?X”等迭代操作。
参考图4,在一个实施例中,图4是一种用于使用如本文描述的或以其他方式设想的基因名称标识系统来标识经标记化的源中的规范基因名称的决策流程图400。该过程以根据源来生成的标记流而开始于步骤410。该源能够是包含或可能包含一个或多个基因标识符的任何源,并且能够使用本文描述的或者以其他方式设想的任何方法或协议对该源进行标记化。
在412处,在基于树的数据结构的根节点处对标识指针进行初始化。这可以包括将标识指针设置为零、默认值或根节点的值。
在步骤414处,将来自标记流的标记一次一个地馈送到该系统中。如果这是来自源的第一标记,则它将是该流程图的第一次前进。如果这是来自标记流中的第二标记或后续标记,则该系统将已经前进通过了该图并返回到步骤412或414以分析下一个标记。
在步骤416处,如果存在从当前节点延伸的任一条边,则该系统确定当前标记是否匹配从当前节点(其至少将是针对第一标记的根节点)延伸的一条或多条边中的任一条边。这种确定可以使用用于比较标记与边以标识匹配的任何方法或算法来执行。根据实施例,可能需要精确匹配,并且/或者可能在匹配中允许模糊性,这能够基于用户设置、系统配置和/或任何其他源。
在418处,如果当前标记匹配从当前节点延伸的一条或多条边之一,则将利用在匹配的边的端部的叶节点的值或位置来更新标识指针。然后,该系统将返回到步骤414(如“A”所示)以处理标记流中的下一个标记。
在420处,如果当前标记并不匹配从当前节点延伸的一条或多条边中的任一条边或者如果没有任何从当前节点延伸的边,则该系统任选地确定标识指针是否包括根节点值。如果是,则标记不包括或者被认为不包括基因标识符,并且该系统返回到步骤412(如“B”所示)以处理标记流中的下一个标记并确保标识指针为默认值或初始值。
如果标识指针不包括根节点值,这意味着已经使用一个或多个标记,利用叶节点的值或位置对标识指针进行了一次或多次更新,则在422处,该系统确定标识指针是否包括填充的叶节点值的值或位置。例如,如图3所示,某些叶节点(例如,316、320和326)不包括规范基因名称值或位置,其可能表示占位符或其他信息。如果标识指针包括填充的叶节点(例如,图3中的叶节点314a、314b或318)的值的值或位置,则在424处,该系统提供来自标识指针的叶节点值,该叶节点值可以是规范基因名称或与之相关联的值或位置,从而标识规范基因,如426处所示。如果标记流已经完成,则该系统能够结束,或者该系统能够返回到步骤414,如图4中的“A”所示。
如果标识指针不包括填充的叶节点值的值或位置,则该系统前进到430,在这种情况下,标识指针被更新为父节点或上游节点的值,并且该系统返回到步骤420,如图4中的“C”所示。例如,参考图3,如果标识指针包括叶节点320的值或位置并且无法进一步前进,则该系统将利用作为根节点310的父节点的值来更新标识指针。然后,该系统将返回到步骤412以对后续标记再次开始执行操作。作为图3的另一示例,如果标识指针包括叶节点326的值或位置并且无法进一步前进,则该系统将在步骤422处利用父节点(即,叶节点314a)的值来更新标识指针。将在步骤424/426中提供该根节点的值,并且如果流中没有更多标记,则该系统能够在428处结束,或者能够前进到后续标记。
参考图5,在一个实施例中,图5是用于生成基因组参考的基因名称标识系统500的示意图。系统500可以是本文描述的或以其他方式设想的任何系统,并且可以包括本文描述的或以其他方式设想的任何部件。
根据实施例,系统500包括经由一条或多条系统总线512互连的处理器520、存储器530、用户接口540、通信接口550和存储装置560中的一项或多项。应当理解,在某些方面,图5构成了一种抽象,并且系统500的部件的实际组织方式可以与所图示的方式不同并且更加复杂。
根据实施例,系统500包括处理器520,该处理器520能够执行在存储器530或存储装置560中存储的指令或以其他方式处理数据,以便例如执行该方法的一个或多个步骤。处理器520可以由一个或多个模块形成。处理器520可以采取任何合适的形式,包括但不限于微处理器、微控制器、多个微控制器、电路、现场可编程门阵列(FPGA)、专用集成电路(ASIC)、单个处理器或多个处理器。
存储器530能够采用任何合适的形式,包括非易失性存储器和/或RAM。存储器530可以包括各种存储器,例如,L1、L2或L3高速缓冲存储器或系统存储器。正因如此,存储器530可以包括静态随机存取存储器(SRAM)、动态RAM(DRAM)、闪存存储器、只读存储器(ROM)或其他类似的存储器设备。存储器还能够存储操作系统等。RAM由处理器用于临时存储数据。根据实施例,操作系统可以包含在由处理器执行时控制系统500的一个或多个部件的操作的代码。显而易见的是,在处理器以硬件实施本文描述的功能中的一种或多种功能的实施例中,可以省去在其他实施例中被描述为与这种功能相对应的软件。
用户接口540可以包括用于使得能够与用户通信的一个或多个设备。用户接口能够是允许传送和/或接收信息的任何设备或系统,并且可以包括用于接收用户命令的显示器、鼠标和/或键盘。在一些实施例中,用户接口540可以包括可以经由通信接口450被呈现给远程终端的命令行接口或图形用户接口。用户接口可以与系统的一个或多个其他部件定位在一起,或者可以被定位为远离系统并且经由有线和/或无线通信网络进行通信。
通信接口550可以包括用于使得能够与其他硬件设备进行通信的一个或多个设备。例如,通信接口550可以包括被配置为根据以太网协议进行通信的网络接口卡(NIC)。另外,通信接口550可以实施用于根据TCP/IP协议进行通信的TCP/IP栈。针对通信接口550的各种替代或额外的硬件或配置将是显而易见的。
存储装置560可以包括一种或多种机器可读存储介质,例如,只读存储器(ROM)、随机存取存储器(RAM)、磁盘存储介质、光学存储介质、闪存存储器设备或类似的存储介质。在各种实施例中,存储装置560可以存储由处理器520执行的指令或处理器520可以对其进行操作的数据。例如,存储装置560可以存储用于控制系统500的各种操作的操作系统561。存储装置560还可以存储一个或多个文档或源562、基因名称表563、前缀树或数据结构564和/或标记器565。
显而易见的是,被描述为被存储在存储装置560中的各种信息可以额外地或替代地被存储在存储器530中。在这方面,存储器530也可以被认为构成存储设备,并且存储装置560可以被认为是存储器。各种其他布置也将是显而易见的。另外,存储器530和存储装置560都可以被认为是非瞬态机器可读介质。如本文所使用的,术语“非瞬态”将被理解为排除瞬态信号,而是包括所有形式的存储,包括易失性存储器和非易失性存储器。
虽然基因名称标识系统500被示为包括每个描述的部件中的一个部件,但是在各种实施例中可以复制各种部件。例如,处理器520可以包括多个微处理器,这多个微处理器被配置为独立执行本文描述的方法,或者被配置为执行本文描述的方法的步骤或子例程,使得多个处理器协作以实现本文描述的功能。另外,在云计算系统中实施系统500的一个或多个部件的情况下,各种硬件部件可能属于单独的物理系统。例如,处理器520可以包括第一服务器中的第一处理器和第二服务器中的第二处理器。许多其他变型和配置也是可能的。
根据实施例,基因名称标识系统500的存储装置560可以存储一种或多种算法和/或指令,以执行本文描述的或以其他方式设想的方法的一种或多种功能或一个或多个步骤。例如,除了其他指令之外,处理器520还可以包括标记化指令566、比较指令567和/或报告指令568。
根据实施例,标记化指令566指导系统对基因标识符和规范基因名称的列表或其他聚集集合563进行标记化,如关于图2所描述的那样以及如本文描述的或以其他方式设想的其他方法那样。例如,标记化指令指导系统根据标记化指令中阐述的标记化逻辑,根据基因标识符和规范基因名称的列表或其他聚集集合来生成标记流,该标记流能够用于生成前缀树或类似的数据结构564以用于分析。
根据实施例,标记化指令566还指导系统对来自获得或接收的文档或其他源562的信息进行标记化,以用于标识源内的基因名称。例如,标记化指令指导系统根据标记化指令中阐述的标记化逻辑,根据文档或其他源562来生成标记流,该标记流能够用于标识源内的基因名称。
如本文描述的或以其他方式认识到的,基因标识符能够以无限数量的格式和变体出现,因此合适的标记化是对基因标识符的识别和标准化中的关键步骤。因此,标记化指令被配置或设计为以优化基因标识符识别的方式对数据进行标记化。可以使用多种标记化方法。
根据实施例,比较指令567指导系统将标记与前缀树或类似的数据结构564进行比较。比较指令567指导系统一次一个地将标记与数据结构564内的适当位置进行比较。例如,比较指令指导系统根据标记的当前位置将标记与从根节点或叶节点引出的一条或多条边进行比较。比较指令还指导系统利用关于根节点或叶节点的信息来更新标识指针的值或位置,因为新的节点是根据标记与一条或多条边的比较的标识。根据实施例,如果不存在与叶节点相关联的规范基因名称并且没有从该叶节点延伸的另外的边或匹配的边,则比较指令能够指导系统利用关于最新上游或父节点的信息来更新标识指针的值或位置,直到到达与规范基因名称相关联的叶节点(或根节点)为止。
根据实施例,报告指令568指导系统提供、记录和/或报告由标记流中的一个或多个标记标识的规范基因名称。例如,报告指令能够指导系统将标识指针的值或位置(其可以是所确定的规范基因名称)提供给另一数据点、表、报告或其他位置。例如,该系统可以创建在源中标识的所有规范基因名称的表或列表。这可以在存储器或数据库中创建,在屏幕或其他用户接口上显示或者以其他方式提供。报告或列表还可以包括用于标识规范基因名称的一个或多个基因标识符以及关于标记或标记在被分析的源中的位置的信息。报告可以是视觉显示、印刷文本、电子邮件、可听报告、传输内容和/或任何其他传送信息的方法。报告可以在本地或远程提供,因此该系统或用户接口可以包括通信系统或者以其他方式连接到通信系统。
由系统提供、记录和/或报告的一个或多个规范基因名称具有许多应用。实际上,本文描述的或以其他方式设想的轻量级方法和系统为许多临床信息学应用提供了容易获得的可访问性、可用性、可延伸性、控制性和可移植性。
例如,患者与他们可能有资格参加的临床试验的匹配通常取决于在每个临床试验描述的文本中搜索遗传纳入和排除准则的能力。患者的遗传状况可能还需要挖掘其文本病历,以获取基因测试、基因型诊断和先前的遗传学特异性处置的结果。患者遗传状况与试验准则的匹配需要准确有效的基因名称规范化以及匹配各种表达的遗传变体。
另外,一个或多个规范基因名称可能在医学或其他临床环境中有用。该系统能够用于将生物医学语言转换为医院和其他与健康相关的软件系统中的商定标准。对于任何试图现代化历史数据并使其可采取行动的机构,这样的方法都是有价值的工具,例如查询系统以返回具有特定标准化基因变体的患者。
更一般地,基因名称识别和标准化使得人们能够理解其中提及和讨论基因的文本。例如,基因提及内容会参与对遗传和染色体不规则现象的描述,这反过来又有助于记录医疗保健患者的基因型、诊断和处置。当这样的文本记录中的知识不仅是人类可读的,而且还是机器可读的时候,这些知识也更有价值。
本文定义和使用的所有定义应当被理解为控制字典定义、通过引用并入的文献中的定义和/或定义术语的普通含义。
除非明确指出相反情况,否则本说明书和权利要求书中使用的词语“一”和“一个”应当被理解为表示“至少一个”。
本说明书和权利要求书中使用的短语“和/或”应当被理解为表示如此结合的元件中的“任一个或两者”,即,在某些情况下结合存在而在其他情况下分离存在的元件。用“和/或”列出的多个元件应当以相同的方式进行解释,即,如此结合的元件中的“一个或多个”元件。除了用“和/或”分句具体标识的元件之外,还可能任选地存在其他元件,无论是与具体标识的那些元件相关还是不相关都可以。
本文在说明书和权利要求书中使用的“或”应当被理解为具有与上文所定义的“和/或”相同的含义。例如,在分隔列表中的项目时,“或”或“和/或”应当被解读为包含性的,即,包含至少一个,但也包含多个元件或元件列表的多于一个的元件以及(任选的)额外的未列出的项目。只有明确指示相反情况的术语(例如,“中的仅一个”或“中的恰好一个”或者在权利要求中使用的“由……组成”)将指代包括多个元件或元件列表中的恰好一个元件。一般而言,当在本文使用的术语“或”前面有排他性术语(例如,“任一个”、“中的一个”、“中的任一个”或“中的恰好一个”)时,术语“或”应当仅被解读为表示排他性替代方案(即,“一个或另一个但不是两者”)。
本文在说明书和权利要求书中使用的引用一个或多个元件的列表的短语“至少一个”应当被理解为表示选自元件列表中的元件中的任何一个或多个元件中的至少一个元件,但不一定包括元件列表中具体列出的每个元件中的至少一个元件,并且不排除元件列表中元件的任何组合。该定义还允许除了在短语“至少一个”所指的元件列表中具体标识的元件之外的任选存在的元件,无论是与具体标识的那些元件相关还是不相关都可以。
还应当理解,除非明确指出相反情况,否则在本文要求保护的包括多于一个步骤或动作的任何方法中,该方法的步骤或动作的顺序不一定限于记载该方法的步骤或动作的顺序。
在权利要求以及上面的说明书中,所有过渡短语(例如,“包括”、“包含”、“携带”、“具有”、“含有”、“涉及”、“持有”、“带有”等)应当被理解为开放式的,即,意指包括但不限于。只有过渡短语“由……组成”和“基本上由……组成”应当分别为闭合或半闭合过渡短语。
虽然本文已经描述和说明了若干发明实施例,但是本领域普通技术人员将容易想到用于执行功能和/或获得结果和/或本文描述的优点中的一个或多个优点的各种其他单元和/或结构。这样的变化和/或修改中的每个变化和/或修改都被认为是在本文描述的发明实施例的范围内。更一般地,本领域技术人员将容易理解,本文描述的所有参数、尺寸、材料和配置均旨在是示例性的,并且实际参数、尺寸、材料和/或配置将取决于一个或多个使用本发明教导的具体应用。本领域技术人员将认识到或者能够使用不超过常规的实验手段来确定本文描述的具体发明实施例的许多等同物。因此,应当理解,前述实施例仅作为示例呈现,并且在权利要求及其等同物的范围内,本发明的实施例可以以不同于具体描述和要求保护的方式来实践。本公开内容的发明实施例涉及本文描述的每个单独的特征、系统、物品、材料、套件和/或方法。另外,如果这样的特征、系统、物品、材料、套件和/或方法并不相互矛盾,则两个或更多个这样的特征、系统、物品、材料、套件和/或方法的任何组合能够被包括在本公开内容的发明范围内。
- 医学文本中对基因名称的标准化的方法和系统
- 标准化数据的生成方法、医学文本数据的处理方法和装置
