一种Hsf1基因敲除小鼠模型的建立及应用
文献发布时间:2023-06-19 11:52:33
技术领域
本发明涉及生物医药技术领域,具体地说,涉及一种Hsf1基因敲除小鼠模型的建立及其应用。
背景技术
骨肉瘤(osteosarcoma,OS)是青少年儿童中最常见的恶性骨肿瘤类型,特点为恶性程度高、易转移且预后不良。因此,越来越多的学者着手于骨肉瘤不良预后相关基因的研究,以期寻找骨肉瘤关键的治疗靶点。其中,已经报道的骨肉瘤预后基因大多都与热休克因子1(Heat shock factor 1,HSF1)的调控密切相关。热休克因子1由Hsf1基因编码而成,是热休克反应(Heat shock response,HSR)的主要调节因子,可以调节细胞内多种蛋白质的正确折叠及分布。HSF1作为重要的调节因子,在各种环境应激反应中可以快速诱导和调控多种热休克蛋白(HSP)和应激蛋白的表达。例如,HSF1常常在物理因素、化学因素及病理生理的刺激下被激活,进而结合热休克基因相应的启动子,启动、转录基因,调节热休克蛋白的表达。越来越多的证据表明,HSF1在骨肉瘤等多种恶性肿瘤中均高表达,且与肿瘤侵袭性及不良预后相关,可作为独立的诊断或预后生物标志物。因此,建立HSF1基因敲除的动物模型为研究骨肉瘤的发生发展等生物学行为具有重要的临床意义。
发明内容
在本发明中,我们提供了一种HSF1基因敲除的动物模型的建立方法,该HSF1基因敲除的动物模型可以用于研究骨肉瘤的有用分子工具,可以更好的了解HSF1基因与骨肉瘤起始和发展过程中的相关分子机制。为研究病人体内骨肉瘤的发生、耐药机制及治疗提供良好的体外模型,进而促进骨肉瘤的基础研究、预防及临床诊治。
具体而言,本发明的目的在于提供以下技术方案:
一种Hsf1基因敲除小鼠模型的构建方法,包括如下步骤:
(1)根据Hsf1基因结构的第9外显子选择相应的gRNA(guide RNA)并进行筛选;
(2)通过PCR扩增出sgRNA(small guide RNA)转录模版,得到四个上游引物和一个下游引物;
(3)将sgRNA进行体外转录并通过TubeScreen平台筛选,筛选切割有效的sgRNA,从筛选结果中选取切割效率最高的sgRNA用于后续实验;
(4)运用PCR扩增出转录spCas9 mRNA的模板,接着体外转录Cas9mRNA;
(5)将体外转录得到的sgRNA与Cas9 mRNA一起注射入健康的C57BL/6小鼠受精卵内,剪取出生小鼠尾巴提取组织并进行PCR测序鉴定,代孕小鼠分娩得到的小鼠记为F0代基因敲除小鼠;
(6)选取F0代杂合子母鼠与野生型公鼠交配得到F1代子鼠;
(7)选取F1代杂合子公鼠与F0代母鼠回交得到F2代子鼠,剪取鼠尾组织并测序,最终得到F2代纯合子敲基因小鼠。
进一步优选的,步骤(2)中四个上游引物序列如SEQ ID NO:1,SEQ ID NO:2,SEQID NO:3,SEQ ID NO:4所示,下游引物序列如SEQ ID NO:5所示。
进一步优选的,步骤(2)中四个上游引物序列为5’-GCCTCTGTCCAGCAGCACTCTGG-3’、5’-GGGTACTGGAGGCGAGCCCTGGG-3’、5’-GTGTTTGAGGCAGCAGGGGTAGG-3’、5’-TCTCAGGGGTGGAGGGGGTCGGG-3’,下游引物序列为5’-AAAAGCACCGACTCGGTGCC-3’。
进一步优选的,步骤(6)中还包括结合琼脂凝胶电泳和基因测序检测F1代鼠基因碱基的突变情况。
进一步优选的,步骤(7)之后还包括步骤(8),运用PCR观察sgRNA切割效率与鼠尾组织的测序,通过观察凝胶电泳结果来判断小鼠是否为杂合,同时进行基因序列比对判断碱基片段缺失。
本发明还提供另一技术方案,上述任一项所述方法在骨肉瘤研究中的应用。
本发明还提供另一技术方案,上述任一项所述方法获得的小鼠模型的敲除Hsf1基团的细胞。
本发明还提供另一技术方案,一种基于CRISPR/Cas9基因敲除技术构建Hsf1基因敲除小鼠模型的sgRNA,其特征在于,sgRNA序列如SEQ ID NO:1,SEQ ID NO:2,SEQ ID NO:3,SEQ ID NO:4,SEQ ID NO:5所示。
本发明还提供另一技术方案,SEQ ID NO:1,SEQ ID NO:2,SEQ ID NO:3,SEQ IDNO:4,SEQ ID NO:5所示序列在建立Hsf1基因敲除小鼠中的应用。
本发明的有益效果,本发明获得的Hsf1基因敲除小鼠为Hsf1在胚胎发育中的功能、对组织和个体放射敏感性的影响及其在包括骨肉瘤等肿瘤发生发展中的作用等方面的研究提供可靠经济的动物模型。目前没有CRISPR/Cas9基因敲除技术构建Hsf1基因敲除小鼠的报道,本发明设计了特定序列的引物提高了基因敲除的效率,获得的Hsf1基因敲除小鼠为Hsf1在胚胎发育中的功能、对组织和个体放射敏感性的影响及其在包括骨肉瘤等肿瘤发生发展中的作用等方面的研究提供可靠经济的动物模型。
附图说明:
图1.sgRNA体外切割验证结果;
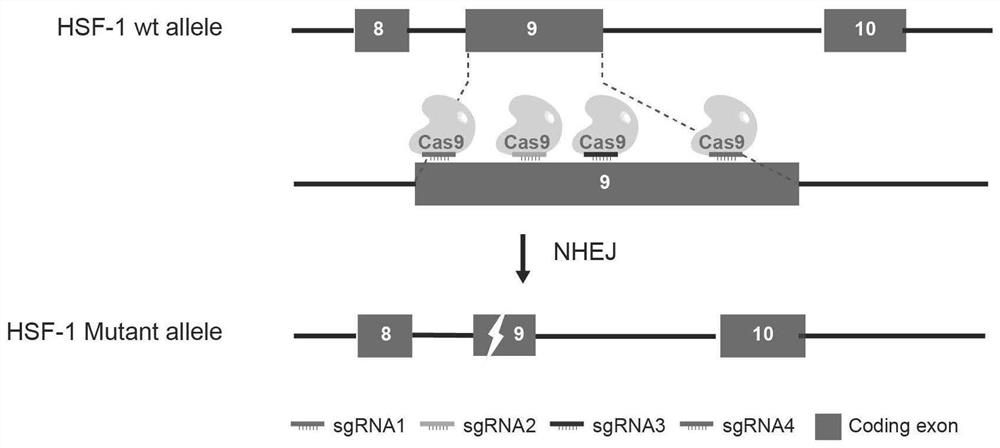
图2.Hsf1基因敲除小鼠构建原理示意图;
图3.F0代出生小鼠PCR鉴定结果;
图4.F0代出生小鼠测序鉴定结果;
图5.F1代出生小鼠PCR鉴定结果;
图6.F2代出生小鼠测序鉴定结果;
图7.纯合子小鼠的繁育情况。
具体实施方式
下面结合具体实施例来进一步描述本发明,但实施例仅是范例性的,并不对本发明的范围构成任何限制。本领域技术人员应该理解的是,在不偏离本发明的精神和范围下可以对本发明技术方案的细节和形式进行修改或替换,但这些修改和替换均落入本发明的保护范围内。
实施例1
F0代小鼠的构建
根据Hsf1(NCBI Gene ID:15499)基因的第9个外显子设计sgRNA,得到4条sgRNA。再根据sgRNA结构通过PCR扩增出sgRNA转录模版,得到上游引物sgRNA-1primer-F、sgRNA-2primer-F、sgRNA-3primer-F、sgRNA-4primer-F与下游引物sgRNA primer-R。引物序列见表1。
表1 Hsf1-sgRNA引物序列
以四个sgRNA质粒为模板,选择相应的引物作PCR扩增,扩增条件为:95℃预变性5min,95℃30s,55℃退火30s,72℃延伸30s,共30个循环。通过体外转录试剂盒(MEGAshortscriptT7 Kit,Life Technologies)转录RNA,随后使用RNA回收试剂盒(MEGA clearKit,Life Technologies)回收得到的sgRNA并分装,随后冻存于-80℃冰箱中。
通过TubeScreen平台进行体外筛选,筛选切割有效的sgRNA,检测结果如图1所示。4个sgRNA对目的片段均可进行有效切割,则选取切割效率最高的sgRNA-1,sgRNA-2和sgRNA-4用于后续实验。
通过引物将T7 promoter添加到Cas9 mRNA的5’端,运用PCR从PX330上扩增出转录spCas9 mRNA的模板;通过体外转录试剂盒(mMESSAGE mMACHINE T7 ULTRA Kit,LifeTechnologies)转录Cas9 mRNA,使用RNA回收试剂盒(MEGA clear Kit,LifeTechnologies)回收mRNA,分装冻存于-80℃冰箱。
将转录好的sgRNA和Cas9 mRNA的终浓度分别定为50ng/ul和100ng/ul。将两者混合一起后用RNase free的水稀释成终浓度之后,在4℃条件下离心20分钟。取上清5ul,分装后一部分用于注射,一部分用于-80℃冻存。混合好的Cas9-sgRNA溶液采用胞浆注射法注入到小鼠受精卵中,注射后的胚胎培养在KSOM培养基中,在37℃,5%二氧化碳条件下培养。选取状态好的受精卵(二细胞)注射到假孕母鼠的壶腹部,详细操作程序按照标准的胚胎移植手术的程序进行。手术缝合后,放入编号的干净的鼠笼,待苏醒后,先断水断粮5小时,后正常饲喂待产。
实验结果
共收集并注射32个小鼠受精卵,其中形成胚胎的27个。随后将27个胚胎进行sgRNA活性检测,得到25个合格的胚胎,将所有合格的胚胎移植到3只受孕小鼠体内。3只受孕小鼠经三周左右分娩出21只小鼠,其中1只死亡,将剩余所有存活小鼠进行基因型测序。
实施例2
鉴定F0小鼠的基因型
F0代小鼠出生后一周龄剪取小鼠尾巴并编号,通过鼠尾鉴定试剂盒(MouseGenotypingKit,Vazyme)进行PCR测序鉴定。根据被敲除的序列位置设计PCR扩增引物,基因敲除示意及引物信息分别如图2、表2所示,对PCR扩增产物进行T-A克隆并进行DNA测序。PCR扩增条件:首先94℃条件下变性30s,随后58℃条件下退火30s,72℃条件下延伸30s,共35个循环。扩增后进行琼脂糖凝胶电泳,电泳结果良好的PCR产物进行DNA测序。
实验结果
如图3琼脂糖凝胶电泳结果所示,F0代中3,5,7,8,9,10,13,14号小鼠的电泳结果为两个条带,而其余小鼠均为一个条带。这表明这8只小鼠存在DNA其中一条单链的碱基片段缺失,为杂合子基因突变型小鼠。
结合测序及电泳结果显示F0代共产生8只杂合子基因突变小鼠,突变起始位点在315p处。其中3,5,7,8,9,10,13,14对应的小鼠为Hsf1基因敲除杂合子小鼠。其余突变小鼠由于脱靶效应导致靶序列未敲除,所以只出现单条带。三次重复试验的电泳结果与测序结果均保持一致。
实施例3
HSF1小鼠纯合品系的构建
将代孕小鼠分娩得到的小鼠记为F0代基因敲除小鼠,对F0代小鼠进行基因测序鉴定小鼠基因型。选择突变的F0代杂合雌鼠和雄鼠交配获得后代F1,对F1代小鼠再次进行基因测序鉴定小鼠基因型。最终使F1代中纯合小鼠互相交配,得到HSF1小鼠纯合品系。
实验结果
选取F0代中的3号母鼠与一只健康的野生型公鼠交配,三周后生育10只F1代小鼠,将得到的F1代小鼠进行基因型测序。电泳结果显示,经过PCR扩增的野生型为单条带,而杂合子小鼠组均为双条带(见图5)。三次重复试验的电泳结果与测序结果均保持一致。结合电泳和测序结果,确定F1代有3只杂合小鼠,同时也证明了3号母鼠的基因能够稳定遗传。
将得到的3只F1代杂合子公鼠与4号母鼠进行回交。在所有出生的F2代小鼠中,其中7只小鼠出现清晰突变型条带且未出现相应的野生型条带。此外,PCR扩增产物测序结果中存在同样的碱基缺失。结合电泳、测序的结果可以证明我们成功地利用CRISPR/Cas9技术获得了F2代Hsf1基因敲除纯合子小鼠。F2代的测序比对结果如图6所示。
建立Hsf1基因敲除纯合子小鼠模型后,我们将得到的纯合子小鼠在海军军医大学实验动物中心小鼠繁育室中进行繁育。目前纯合子Hsf1基因敲除小鼠已经稳定繁育4代,纯合子小鼠繁育情况如图7所示。
综上所述,Hsf1基因敲除小鼠模型的可作为骨肉瘤发病机制及个体化治疗体外研究的有用工具,进而为建立骨肉瘤发生机理模型及制备、筛选、评价抗肿瘤药物提供基础。
以上仅是本发明的优选实施方式,本发明的保护范围并不仅限制于本文所示的实施例,凡属于本发明思路下的技术方案均属于本发明的保护范围。应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明原理前提下的若干修改和润饰也应视为本发明的保护范围。
序列表
<110> 上海长征医院
<120> 一种Hsf1基因敲除小鼠模型的建立及应用
<160> 5
<170> SIPOSequenceListing 1.0
<210> 1
<211> 23
<212> DNA
<213> 人工序列(人工合成)
<400> 1
gcctctgtcc agcagcactc tgg 23
<210> 2
<211> 23
<212> DNA
<213> 人工序列(人工合成)
<400> 2
gggtactgga ggcgagccct ggg 23
<210> 3
<211> 23
<212> DNA
<213> 人工序列(人工合成)
<400> 3
gtgtttgagg cagcaggggt agg 23
<210> 4
<211> 23
<212> DNA
<213> 人工序列(人工合成)
<400> 4
tctcaggggt ggagggggtc ggg 23
<210> 5
<211> 20
<212> DNA
<213> 人工序列(人工合成)
<400> 5
aaaagcaccg actcggtgcc 20
- 一种Hsf1基因敲除小鼠模型的建立及应用
- 一种基于MYPT1基因敲除建立新型高血压小鼠模型的方法及其用途
