用于区分体细胞基因组序列与种系基因组序列的方法和系统
文献发布时间:2023-06-19 18:32:25
相关专利申请的交叉引用
本申请要求2020年6月5日提交的美国临时专利申请号63/035,572和2020年6月19日提交的美国临时专利申请号63/041,437的权益,这些临时申请以全文引用的方式并入本文。
技术领域
本公开涉及用于区分体细胞基因组序列与种系基因组序列的系统和方法。
背景技术
种系基因组序列是指生物体从其亲本继承的那些序列。特别地,如果生物体的亲本中的一者或两者都具有某些基因组突变(或如果该生物体在其非常早期的发育中经历了某些突变),那么这些突变可能是该生物体的种系,并将传递给该生物体的后代(如果有的话)。
相比之下,体细胞基因组序列是那些不会从亲本传递给子代的序列。例如,生物体可能由于外部因素(例如,污染、辐射、饮食、吸烟等)而发生基因组突变,其中这些基因组突变仅限于某些组织、流体或其他解剖材料。在一些情况下,这些突变会导致不期望的医学状况,包括但不限于癌症。
精准医学是这样的领域,其中用针对患者的个体特征或其状况的疗法来治疗该患者。对于许多患者(包括癌症患者),这可能包括:确定关于患者的“正常”基因组状态以及患者的“异常”组织、流体或其他解剖材料的基因组状态的基因组信息。该信息可以源自来自患者的样品,诸如肿瘤活检、抽血或具有正常和异常组织、流体或其他解剖材料的一些其他类型的样品。
可以测定这些样品以(至少部分地)确定其中所含材料的基因组序列。然而,鉴定特定基因组序列是否来自患者的正常解剖材料或其是否来自异常解剖材料有时是具有挑战性的;即,确实特定基因组序列是种系或体细胞有时是具有挑战性的。
了解在癌症患者的DNA中观察到的遗传变体是种系起源还是体细胞起源在临床实践和癌症研究两者中是至关重要的。可以例如通过对来自同一患者的匹配的肿瘤和正常组织进行测序来进行体细胞/种系区分。存在于肿瘤中但不存在于正常组织中的变体被分类为体细胞,而同时存在于两者中的变体被分类为种系。然而,这种双样品方法受到成本和标本可用性的限制。通常在临床实践中没有获得匹配的正常标本。例如,在组织活检的情况下,收集包含肿瘤及其邻近正常组织两者的单个标本。因此,需要开发能够可靠地将检测到的变体在起源上分类为体细胞或种系的方法。
发明内容
本文描述了用于区分体细胞基因组序列与种系基因组序列的方法、设备和计算机可读介质。
本文公开了将感兴趣基因组序列鉴定为种系或体细胞的方法,该方法包括:提供从来自受试者的样品获得的多个核酸分子,其中该多个核酸分子包括肿瘤核酸分子和非肿瘤核酸分子的混合物;任选地,将一个或多个衔接子连接到来自该多个核酸分子的一个或多个核酸分子上;扩增来自该多个核酸分子的核酸分子;从所扩增的核酸分子捕获核酸分子,其中所捕获的核酸分子是通过与一个或多个诱饵分子杂交而从扩增的核酸分子捕获的;通过测序仪对该所捕获的核酸分子进行测序,以获得对应于一个或多个基因组基因座的多个序列读段;通过一个或多个处理器在来自该一个或多个基因组基因座的基因组基因座处选择感兴趣基因组序列;通过该一个或多个处理器,选择针对该感兴趣基因组序列的一个或多个代理基因组序列;通过该一个或多个处理器,使用该感兴趣基因组序列的观察到的等位基因频率和指示该一个或多个代理基因组序列的观察到的等位基因频率的汇总统计量或分布来确定等位基因频率距离;以及通过该一个或多个处理器,使用该等位基因频率距离来将该感兴趣基因组序列鉴定为种系或体细胞。
在一些实施例中,受试者为癌症患者。在一些实施例中,样品包括组织活检样品、液体活检样品、循环肿瘤细胞(CTC)样品、无细胞DNA(cfDNA)样品或正常对照。在一些实施例中,样品为液体活检样品并且包括血液、血浆、脑脊髓液、痰、粪便、尿液或唾液。在一些实施例中,肿瘤核酸分子源自异质组织活检样品的肿瘤部分,并且非肿瘤核酸分子源自异质组织活检样品的正常部分。在一些实施例中,肿瘤核酸分子源自无细胞DNA样品的循环肿瘤DNA(ctDNA)分数,并且非肿瘤核酸分子源自无细胞DNA样品的非肿瘤分数。在一些实施例中,一个或多个衔接子包括扩增引物或测序衔接子。在一些实施例中,一个或多个诱饵分子包括一个或多个核酸分子,每个核酸分子包括与所捕获的核酸分子的区域互补的区域。在一些实施例中,扩增核酸分子包括执行聚合酶链式反应(PCR)或等温扩增技术。在一些实施例中,测序包括使用下一代测序(NGS)技术。在一些实施例中,测序仪包括下一代测序仪。在一些实施例中,一个或多个代理基因组序列位于受试者的基因组序列的限定片段内,并且所选择的感兴趣基因组序列位于同一限定片段内。在一些实施例中,基于每个片段内的拷贝数均匀性将受试者的基因组序列分割为多个片段。在一些实施例中,汇总统计量是平均等位基因频率或中位等位基因频率。在一些实施例中,等位基因频率距离是使用感兴趣基因组序列的观察到的等位基因频率和指示多个代理基因组序列的观察到的频率的分布来确定的,并且其中基于感兴趣基因组序列的观察到的等位基因频率拟合或不拟合在分布内的概率,将感兴趣基因组序列鉴定为种系或体细胞。
在一些实施例中,将感兴趣基因组序列鉴定为种系或体细胞的方法包括:通过一个或多个处理器从针对患者样品获得的患者基因组序列内在基因组基因座处选择感兴趣基因组序列,该患者样品包括肿瘤核酸分子和非肿瘤核酸分子的混合物;通过该一个或多个处理器,选择针对该感兴趣基因组序列的一个或多个代理基因组序列;通过该一个或多个处理器,使用该感兴趣基因组序列的观察到的等位基因频率和指示该一个或多个代理基因组序列的观察到的等位基因频率的汇总统计量或分布来确定等位基因频率距离;以及通过该一个或多个处理器,使用该等位基因频率距离来将该感兴趣基因组序列鉴定(例如,分类)为种系或体细胞。
在该方法的一些实施例中,汇总统计量是平均等位基因频率或中位等位基因频率。在一些实施例中,等位基因频率距离是使用感兴趣基因组序列的观察到的等位基因频率和指示多个代理基因组序列的观察到的频率的分布来确定的,并且其中基于感兴趣基因组序列的观察到的等位基因频率拟合或不拟合在分布内的概率,将感兴趣基因组序列鉴定为种系或体细胞。
在一些实施例中,肿瘤核酸分子和非肿瘤核酸分子包括DNA分子。在一些实施例中,肿瘤核酸分子和非肿瘤核酸分子包括RNA分子。
在一些实施例中,该方法进一步包括对来自患者样品的肿瘤核酸分子和非肿瘤核酸分子进行测序,以确定患者基因组序列。在一些实施例中,使用下一代测序技术获得或确定患者基因组序列。在一些实施例中,测序仪是下一代测序仪。
在该方法的一些实施例中,一个或多个代理基因组序列位于患者基因组序列的限定片段内,并且所选择的感兴趣基因组序列位于同一限定片段内。在一些实施例中,患者基因组序列基于每个片段内的拷贝数均匀性被分割为多个片段。在一些实施例中,该方法包括将患者基因组序列分割为多个片段。
在该方法的一些实施例中,使用靶向测序确定患者基因组序列。在一些实施例中,靶向测序包括对与癌症相关的一个或多个基因或其部分的靶向测序。在一些实施例中,靶向测序包括对一个或多个外显子区域的靶向测序。
在一些实施例中,该方法包括:通过一个或多个处理器,鉴定患者样品中在基因组基因座处的感兴趣基因组序列;通过该一个或多个处理器鉴定针对感兴趣序列的一个或多个代理基因组序列;通过该一个或多个处理器将感兴趣序列的观察到的频率与该一个或多个代理基因组序列的观察到的频率的中心性量度进行比较;以及基于比较,将该感兴趣基因组序列鉴定(例如,分类或表征)为种系或体细胞。
在该方法的一些实施例中,一个或多个代理基因组序列包括单核苷酸多态性(SNP)。
在该方法的一些实施例中,一个或多个代理基因组序列包括等位基因。
在一些实施例中,该方法进一步包括通过一个或多个处理器鉴定患者的基因组的片段,该片段中包括基因组基因座。在一些实施例中,通过一个或多个处理器鉴定片段包括对患者的基因组的连续部分执行分割过程。在一些实施例中,患者的基因组的部分大到足以鉴定三个不同的片段。在一些实施例中,代理被一个或多个处理器鉴定为与基因组基因座位于相同的片段上。在一些实施例中,分割过程根据基因组参数跨每个单独片段的整体是否相等来鉴定片段。在一些实施例中,基因组参数是拷贝数。
在将感兴趣基因组序列鉴定为种系或体细胞的上述方法中的任何上述方法的一些实施例中,通过一个或多个处理器,将感兴趣基因组序列鉴定为种系或体细胞的步骤包括:将等位基因频率距离输入到经训练的统计模型中;以及从该经训练的统计模型输出指示该感兴趣基因组序列为种系的可能性的值或指示该感兴趣基因组序列为体细胞的可能性的值。在一些实施例中,调整等位基因频率距离,以校正患者样品中的污染水平、低测序读段深度、等位基因频率的噪声估计、低片段种系单核苷酸多态性(SNP)计数或片段种系SNP等位基因频率中的高变异性。在一些实施例中,经训练的统计模型包括函数,该函数将等位基因频率距离与指示感兴趣基因组序列为种系的可能性的值或指示感兴趣基因组序列为体细胞的可能性的值相关联。
在一些实施例中,经训练的统计模型是逻辑回归模型。在一些实施例中,经训练的统计模型是使用具有已知种系序列的肿瘤样品来训练的。在一些实施例中,经训练的统计模型是使用针对具有已知种系序列和已知体细胞序列的肿瘤样品的数据来训练的。在一些实施例中,该方法进一步包括使用针对具有已知种系序列的肿瘤样品的数据来训练统计模型。在一些实施例中,该方法进一步包括使用针对具有已知种系序列和已知体细胞序列的肿瘤样品的数据来训练统计模型。
在一些实施例中,经训练的统计模型是使用针对变体等位基因频率的数据来训练的,该数据排除位于已知具有偏离预期值的等位基因频率的基因组区域中的变体。在一些实施例中,该方法进一步包括使用针对变体等位基因频率的数据来训练统计模型,该数据排除位于已知具有偏离预期值的等位基因频率的基因组区域中的变体。
在一些实施例中,使用结合以下的数据来训练所述统计模型:基于历史数据或数据库,变体为种系变体、体细胞变体或不确定潜能的克隆性造血(CHIP)变体的可能性的先验知识。在一些实施例中,该方法进一步包括使用结合以下的数据来训练所述统计模型:基于历史数据或数据库,变体为种系变体、体细胞变体或不确定潜能的克隆性造血(CHIP)变体的可能性的先验知识。
在一些实施例中,经训练的统计模型是使用考虑到针对给定变体调用及其基因组情景的噪声水平的数据来训练的。在一些实施例中,该方法进一步包括使用考虑到针对给定变体调用及其基因组情景的噪声水平的数据来训练统计模型。
在一些实施例中,一个或多个代理基因组序列包括单核苷酸多态性(SNP)。在一些实施例中,一个或多个代理基因组序列包括等位基因。在该方法的一些实施例中,感兴趣基因组序列包括基因组变体。
在该方法的一些实施例中,该方法进一步包括通过一个或多个处理器,生成指示感兴趣基因组序列为种系或体细胞的报告。在一些实施例中,该方法包括向例如医疗保健提供者传输报告。在一些实施例中,经由计算机网络或对等连接传输报告。
在上述方法中的任何上述方法的一些实施例中,患者样品源自包括肿瘤组织和非肿瘤组织的组织活检。在一些实施例中,组织活检是固体组织活检或液体活检。在一些实施例中,组织活检为包括血液、血浆、脑脊髓液、痰、粪便、尿液或唾液的液体活检。在一些实施例中,患者样品包括从受试者获得的无细胞DNA(cdDNA)。在一些实施例中,患者样品包括从受试者获得的循环肿瘤DNA(ctDNA)。
本文还描述了一种治疗患者癌症的方法,该方法包括通过一个或多个处理器,使用上述任何方法中的任何方法将一个或多个感兴趣基因组序列鉴定为体细胞;基于一个或多个所鉴定的体细胞序列选择癌症治疗方式;使用所选择的癌症治疗方式来治疗癌症。在一些实施例中,将一个或多个所鉴定的体细胞序列与使用所选择的治疗方式的成功癌症治疗相关联。在一些实施例中,该方法包括通过一个或多个处理器,使用一个或多个所鉴定的体细胞序列来确定癌症的微卫星不稳定性状态;以及基于癌症的微卫星不稳定性状态来选择癌症治疗方式。在一些实施例中,该方法包括通过一个或多个处理器,使用一个或多个所鉴定的体细胞序列来确定癌症的肿瘤突变负荷;以及基于该肿瘤突变负荷高于预定肿瘤突变负荷阈值来选择癌症治疗方式。在一些实施例中,癌症治疗方式包括在肿瘤突变负荷高于预定阈值的情况下,向患者施用有效量的一种或多种抗癌剂。在一些实施例中,一种或多种抗癌剂包括免疫肿瘤剂。在一些实施例中,免疫肿瘤剂是免疫检查点抑制剂。
本文还描述了一种监测患者癌症进展或复发的方法,该方法包括:通过一个或多个处理器,使用上述方法中的任何上述方法将一个或多个感兴趣基因组序列鉴定为体细胞;以及在癌症已经被治疗之后,通过一个或多个处理器,检测从患者获得的第二患者样品内被鉴定为体细胞的一个或多个感兴趣基因组序列的存在或不存在。在一些实施例中,该方法包括从患者获得第二患者样品。在一些实施例中,该方法包括在从患者获得第一患者样品之后和在从患者获得第二患者样品之前治疗患者的癌症。在一些实施例中,第二患者样品包括无细胞DNA。在一些实施例中,检测第二患者样品内被鉴定为体细胞的一个或多个感兴趣基因组序列在的存在或不存在包括:对第二患者样品中的核酸分子进行测序。
本文还描述了针对为患有癌症的受试者个性化的癌症疫苗选择新抗原的方法,其包括:通过一个或多个处理器,使用上述任何方法中的任何方法将一个或多个感兴趣基因组序列鉴定为体细胞,其中被鉴定为体细胞的该一个或多个感兴趣基因组序列位于基因的外显子区域内;以及通过该一个或多个处理器从被鉴定为体细胞的该一个或多个感兴趣基因组序列选择基因组序列,该基因组序列编码适合作为针对受试者的癌症疫苗的新抗原。在一些实施例中,该方法包括制备包括新抗原的疫苗。
本文还描述了一种存储一个或多个程序的非暂态计算机可读存储介质,该一个或多个程序包括指令,该指令在由电子设备的一个或多个处理器执行时使该电子设备:从针对患者样品获得的患者基因组序列内在基因组基因座处选择感兴趣基因组序列,该患者样品包括肿瘤核酸分子和非肿瘤核酸分子的混合物;选择针对该感兴趣基因组序列的一个或多个代理基因序列;使用该感兴趣基因组序列的观察到的等位基因频率和指示该一个或多个代理基因组序列的观察到的等位基因频率的汇总统计量或分布来确定等位基因频率距离;以及使用该等位基因频率距离来将该感兴趣基因组序列鉴定为种系或体细胞。在一些实施例中,汇总统计量是平均等位基因频率或中位等位基因频率。在一些实施例中,等位基因频率距离是使用感兴趣基因组序列的观察到的等位基因频率和指示多个代理基因组序列的观察到的频率的分布来确定的,并且其中基于感兴趣基因组序列的观察到的等位基因频率拟合或不拟合在分布内的概率,将感兴趣基因组序列鉴定为种系或体细胞。在一些实施例中,肿瘤核酸分子和非肿瘤核酸分子包括DNA分子。在一些实施例中,肿瘤核酸分子和非肿瘤核酸分子包括RNA分子。
在非暂态计算机可读存储介质的一些实施例中,一个或多个代理基因组序列位于患者基因组序列的限定片段内,并且所选择的感兴趣基因组序列位于同一限定片段内。在一些实施例中,患者基因组序列基于每个片段内的拷贝数均匀性被分割为多个片段。
在非暂态计算机可读存储介质的一些实施例中,一个或多个程序进一步包括指令,该指令当由电子设备的一个或多个处理器执行时,使该电子设备将患者基因组序列分割为多个片段。
在非暂态计算机可读存储介质的一些实施例中,使用靶向测序来确定患者基因组序列。在一些实施例中,使用下一代测序确定患者基因组序列。在一些实施例中,靶向测序包括对与癌症相关的一个或多个基因或其部分的靶向测序。在一些实施例中,靶向测序包括对一个或多个外显子区域的靶向测序。
在一些实施例中,非暂态计算机可读存储介质存储一个或多个程序,该一个或多个程序包括指令,该指令在由电子设备的一个或多个处理器执行时使该电子设备:鉴定患者样品中在基因组基因座处的感兴趣基因组序列;针对该感兴趣序列鉴定一个或多个代理基因组序列;相对于一个或多个代理基因组序列的观察到的频率的中心性量度,鉴定感兴趣序列的观察到的频率;以及基于比较,将该感兴趣基因组序列表征为种系或体细胞。
在非暂态计算机可读存储介质的一些实施例中,一个或多个程序进一步包括指令,该指令在由电子设备的一个或多个处理器执行时使该电子设备生成指示感兴趣基因组序列为种系或体细胞的报告。在一些实施例中,电子设备包括显示器,并且一个或多个程序进一步包括指令,该指令在由电子设备的一个或多个处理器执行时使该电子设备显示报告。
在非暂态计算机可读存储介质的一些实施例中,一个或多个代理基因组序列包括单核苷酸多态性(SNP)。
在非暂态计算机可读存储介质的一些实施例中,一个或多个代理基因组序列包括等位基因。
在非暂态计算机可读存储介质的一些实施例中,一个或多个程序进一步包括指令,该指令在由电子设备的一个或多个处理器执行时使该电子设备鉴定患者的基因组的片段,该片段中包括基因组基因座。在一些实施例中,鉴定片段包括对患者的基因组的连续部分执行分割过程。在一些实施例中,患者的基因组的部分大到足以鉴定三个不同的片段。在一些实施例中,一个或多个代理基因组序列被鉴定为与基因组基因座位于相同的片段上。在一些实施例中,分割过程根据基因组参数跨每个单独片段的整体是否相等来鉴定片段。在一些实施例中,基因组参数是拷贝数。
在非暂态计算机可读存储介质的一些实施例中,感兴趣基因组序列包括基因组变体。
在非暂态计算机可读存储介质的一些实施例中,一个或多个程序进一步包括指令,该指令在由电子设备的一个或多个处理器执行时使该电子设备接收与患者基因组序列相关联的测序数据。在一些实施例中,一个或多个程序进一步包括指令,该指令在由电子设备的一个或多个处理器执行时使该电子设备使用测序数据来组装患者基因组序列。在一些实施例中,一个或多个程序进一步包括指令,该指令在由电子设备的一个或多个处理器执行时使该电子设备操作测序仪来对源自患者样品的核酸分子进行测序,由此获得测序数据。
在非暂态计算机可读存储介质的一些实施例中,一个或多个程序进一步包括指令,该指令在由电子设备的一个或多个处理器执行时使该电子设备生成指示感兴趣基因组序列为种系或体细胞的报告。在一些实施例中,一个或多个程序进一步包括指令,该指令在由电子设备的一个或多个处理器执行时使该电子设备使用计算机网络来传输报告。
在非暂态计算机可读存储介质的一些实施例中,电子设备包括显示器,并且一个或多个程序进一步包括指令,该指令在由电子设备的一个或多个处理器执行时使该电子设备显示报告。
在非暂态计算机可读存储介质的一些实施例中,一个或多个代理基因组序列包括单核苷酸多态性(SNP)。
在非暂态计算机可读存储介质的一些实施例中,一个或多个代理基因组序列包括等位基因。
在非暂态计算机可读存储介质的一些实施例中,感兴趣基因组序列包括基因组变体。
本文还描述了电子设备,其包括:一个或多个处理器;以及存储器,该存储器存储被配置为由该一个或多个处理器执行的一个或多个程序,该一个或多个程序包括用于以下项的指令:从针对患者样品获得的患者基因组序列内在基因组基因座处选择感兴趣基因组序列,该患者包括肿瘤核酸分子和非肿瘤核酸分子的混合物;选择针对该感兴趣基因组序列的一个或多个代理基因序列;使用该感兴趣基因组序列的观察到的等位基因频率和指示该一个或多个代理基因组序列的观察到的等位基因频率的汇总统计量或分布来确定等位基因频率距离;以及使用该等位基因频率距离来将该感兴趣基因组序列鉴定为种系或体细胞。在一些实施例中,汇总统计量是平均等位基因频率或中位等位基因频率。在一些实施例中,等位基因频率距离是使用感兴趣基因组序列的观察到的等位基因频率和指示多个代理基因组序列的观察到的频率的分布来确定的,并且其中基于感兴趣基因组序列的观察到的等位基因频率拟合或不拟合在分布内的概率,将感兴趣基因组序列鉴定为种系或体细胞。在一些实施例中,肿瘤核酸分子和非肿瘤核酸分子包括DNA分子。在一些实施例中,肿瘤核酸分子和非肿瘤核酸分子包括RNA分子。在一些实施例中,使用下一代测序确定患者基因组序列。
在电子设备的一些实施例中,一个或多个代理基因组序列位于患者基因组序列的限定片段内,并且所选择的感兴趣基因组序列位于同一限定片段内。在一些实施例中,患者基因组序列基于每个片段内的拷贝数均匀性被分割为多个片段。在一些实施例中,一个或多个程序进一步包括用于将患者基因组序列分割为多个片段的指令。
在电子设备的一些实施例中,使用靶向测序确定患者基因组序列。在一些实施例中,靶向测序包括对与癌症相关的一个或多个基因或其部分的靶向测序。在一些实施例中,靶向测序包括对一个或多个外显子区域的靶向测序。
在一些实施例中,一种电子设备,其包括:一个或多个处理器;以及存储器,该存储器存储被配置为由该一个或多个处理器执行的一个或多个程序,该一个或多个程序包括用于以下项的指令:鉴定患者样品中在基因组基因座处的感兴趣基因组序列;针对该感兴趣序列鉴定一个或多个代理基因组序列;将该感兴趣序列的观察到的频率鉴定为该一个或多个代理基因组序列的观察到的频率的中心性量度;以及基于比较,将该感兴趣基因组序列表征为种系或体细胞。
在电子设备的一些实施例中,一个或多个代理基因组序列包括单核苷酸多态性(SNP)。
在电子设备的一些实施例中,一个或多个代理基因组序列包括等位基因。
在电子设备的一些实施例中,一个或多个程序进一步包括用于鉴定患者的基因组的片段的指令,该片段中包括基因组基因座。在一些实施例中,鉴定片段包括对患者的基因组的连续部分执行分割过程。在一些实施例中,患者的基因组的部分大到足以鉴定三个不同的片段。在一些实施例中,代理被鉴定为与基因组基因座位于相同的片段上。在一些实施例中,分割过程根据基因组参数跨每个单独片段的整体是否相等来鉴定片段。在一些实施例中,基因组参数是拷贝数。
在电子设备的一些实施例中,感兴趣基因组序列包括基因组变体。
在电子设备的一些实施例中,一个或多个程序进一步包括用于接收与患者基因组序列相关联的测序数据的指令。在一些实施例中,一个或多个程序进一步包括用于使用测序数据来组装患者基因组序列的指令。在一些实施例中,一个或多个程序进一步包括用于使测序仪对源自患者样品的核酸分子进行测序由此获得测序数据的指令。
在电子设备的一些实施例中,一个或多个代理基因组序列包括单核苷酸多态性(SNP)。
在电子设备的一些实施例中,一个或多个代理基因组序列包括等位基因。
在电子设备的一些实施例中,感兴趣基因组序列包括基因组变体。
在电子设备的一些实施例中,一个或多个程序进一步包括用于生成指示感兴趣基因组序列为种系或体细胞的报告的指令。在一些实施例中,一个或多个程序进一步包括用于经由计算机网络或对等连接来传输报告的指令。在一些实施例中,设备进一步包括显示器,并且一个或多个程序进一步包括用于显示报告的指令。
在电子设备的一些实施例中,患者样品源自包括肿瘤组织和非肿瘤组织的组织活检。在一些实施例中,组织活检是固体组织活检或液体活检。在一些情况下,组织样品为包括血液、血浆、脑脊髓液、痰、粪便、尿液或唾液的液体活检。在一些实施例中,患者样品包括从受试者获得的无细胞DNA(cfDNA)。在一些实施例中,患者样品包括从受试者获得的循环肿瘤DNA(ctDNA)。
本文还描述了一种系统,该系统包括本文所述的任何电子设备中的任何电子设备和被配置为对源自患者样品的核酸分子进行测序的测序仪。在一些实施例中,测序仪是下一代测序仪。
本文公开了将感兴趣基因组序列鉴定为种系或体细胞的方法,该方法包括:通过一个或多个处理器,鉴定患者样品中在基因组基因座处的感兴趣基因组序列;通过该一个或多个处理器鉴定针对感兴趣基因组序列的代理基因组序列;通过该一个或多个处理器将该感兴趣基因组序列的观察到的等位基因分数与该代理基因组序列的观察到的等位基因分数进行比较;以及通过该一个或多个处理器基于比较将感兴趣基因组序列鉴定为种系或体细胞。在一些实施例中,代理基因组序列具有与感兴趣基因组序列相同的拷贝数。在一些实施例中,通过一个或多个处理器,将感兴趣基因组序列鉴定为种系或体细胞包括:将等位基因频率距离输入到经训练的统计模型中;以及从该经训练的统计模型输出指示该感兴趣基因组序列为种系的可能性的值或指示该感兴趣基因组序列为体细胞的可能性的值。在一些实施例中,使用下一代测序技术确定基因组序列的等位基因分数和代理基因组序列的等位基因分数。在一些实施例中,使用微阵列技术确定基因组序列的等位基因分数和代理基因组序列的等位基因分数。在一些实施例中,患者样品包括固体组织活检或液体活检。在一些实施例中,患者样品为包括血液、血浆、脑脊髓液、痰、粪便、尿液或唾液的液体活检。在一些实施例中,患者样品包括从受试者获得的无细胞DNA(cfDNA)。在一些实施例中,患者样品包括从受试者获得的循环肿瘤DNA(ctDNA)。在一些实施例中,患者是癌症患者。
以引用方式并入
本说明书中提到的所有出版物、专利和专利申请都以全文引用的方式并入本文,所达到的程度如同每个单独的出版物、专利或专利申请都被具体地和单独地指出以全文引用的方式并入。如果文本的术语与并入的参考文献中的术语发生冲突,则以本文的术语为准。
附图说明
图1是患者的基因组的部分的示意图。
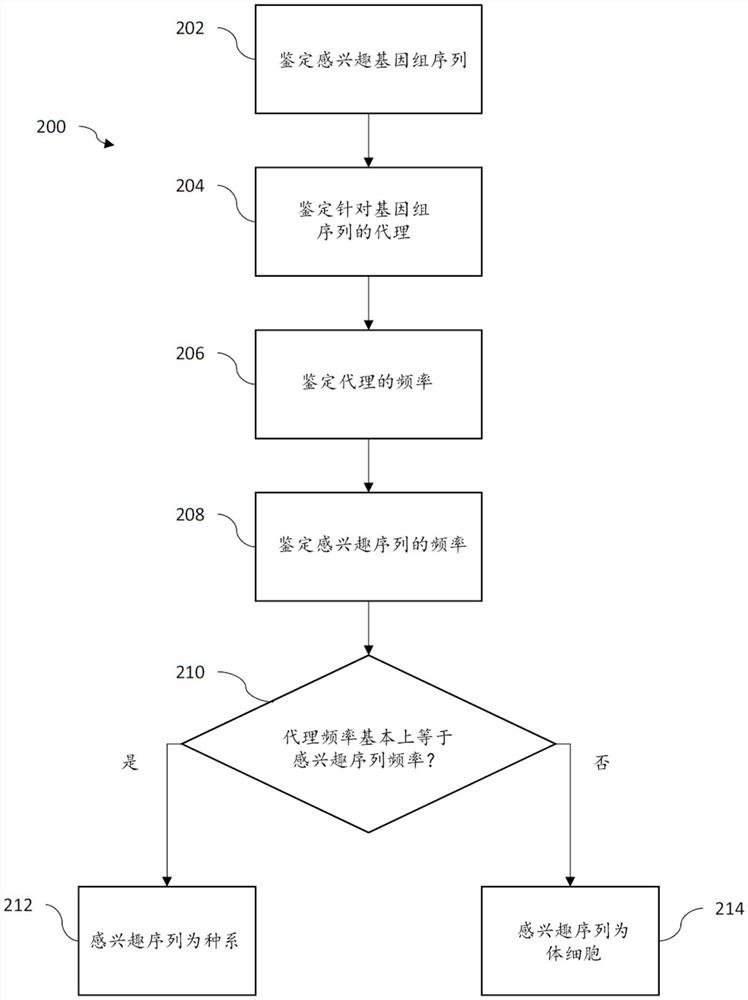
图2是针对用于区分种系基因组序列和体细胞基因组序列的过程的流程图。
图3是基因组划分的示意图。
图4示出了包括电子设备的示例性系统,该电子设备可以用于执行本文所述的方法。
图5A示出了用于在给定相同的肿瘤分数、倍性和拷贝数的情况下确定针对体细胞变体和种系变体的预期变体等位基因分数的差异的示例性过程。
图5B示出了用于从预期种系等位基因频率(AFDIS)确定等位基因频率的距离的示例性方法,以及AFDIS的示例性密度分布,从其可以建立经验累积分布函数(ECDF)。
图5C示出了根据肿瘤样品的计算纯度绘制的AFDIS的示例图。
图5D示出了根据本文公开的方法针对对肿瘤样品中的体细胞变体和种系变体进行分类的ROC曲线的非限制性示例。
图5E示出了针对可以与一些实施例一起使用的示例性逻辑回归模型的概率图的非限制性示例。
图5F示出了使用示例性逻辑回归模型确定的不同变体的体细胞概率的图。
图5G示出了所要求保护的方法相对于现有SGZ方法的改进。
图5H示出了根据本文公开的示例性方法的针对用于训练和测试逻辑回归模型的训练数据和测试数据的灵敏度图的非限制性示例。
图5I示出了根据本文公开的示例性方法的针对用于训练和测试逻辑回归模型的训练数据和测试数据的阳性预测值(PPV)图的非限制性示例。
图5J示出了针对使用所述方法的示例性实施例对BRCA1和BRCA2基因中的变体进行分类的数据的非限制性示例。
图5K示出了针对使用所述方法的示例性实施例对STH11基因中的变体进行分类的数据的非限制性示例。
图6A示出了针对肿瘤样品中的已知种系变体的变体等位基因频率(AF)与片段次要等位基因频率(MAF)的图的非限制性示例。
图6B示出了分别对应于片段MAF值为0.1、0.2和0.3的密度与变体AF图的非限制性示例,如源自图6A中绘制的数据。
具体实施方式
本文描述了用于区分体细胞基因组序列与种系基因组序列的方法、设备和计算机可读介质。可以鉴定患者样品中在基因组基因座处的感兴趣基因组序列。然后,对于感兴趣序列,可以鉴定一个或多个代理基因组序列。可以将感兴趣序列的观察到的频率与一个或多个代理基因组序列的观察到的频率的中心性量度进行比较;并且,基于比较,可以将感兴趣基因组序列表征为种系序列或体细胞序列。
过去已经开发了几种方法以确定单个样品设置中的变体的体细胞/种系状态,其包括:与公共种系数据库(诸如dbSNP)进行匹配或使用从大量正常个体构建的替代物来代替匹配的正常个体。参见例如Hiltemann等人,Discriminating somatic and germlinemutations in tumor DNA samples without matching normal,Genome Res.vol.25,no.9,pp.1382–1390(2015)。然而,在处理仅限于家族或小群体的罕见种系变体时,此类方法是无效的。也存在一种所谓的“基本方法”,其中等位基因频率(或等位基因分数)接近50%或100%的变体被认为是种系变体,而那些不满足这一标准的则被分类为体细胞变体。参见Jones等人,Personalized genomic analyses for cancer mutation discovery andinterpretation,Sci.Transl.Med.,vol.7,no.283,p.283ra53(2015)。这种基本方法无法解释这样的事实,即非整倍性可以使种系变体的等位基因频率显着偏离50%或100%的预期。术语“等位基因频率”和“等位基因分数”在本文中可互换使用,并且是指对应于特定等位基因的序列读段相对于针对基因组基因座的序列读段的总数量的分数。
SGZ(体细胞-种系-接合性)算法于2018年初发布,旨在通过计算肿瘤含量、肿瘤倍性和局部拷贝数来提供对单样品体细胞/种系分类问题的解决方案。SGZ被证明在验证数据集中在体细胞/种系调用准确性方面大大优于“基本方法”(Sun等人,A computationalapproach to distinguish somatic vs.germline origin of genomic alterationsfrom deep sequencing of cancer specimens without a matched normal,PLoS ComputBiol.,vol.14,no.2,p.e1005965(2018),其以全文引用的方式并入本文)。SGZ算法在FMI的基于深度大规模并行测序(MPS)的诊断产品中的应用已经实现了针对短变体(替代和插入缺失)的有效体细胞/种系状态确定,并成为针对诸如肿瘤突变负荷(TMB)估计等应用的不可或缺的工具。
本文所述的用于体细胞/种系分类的方法代表了对SGZ方法的进一步改进。新方法基于相同的基本原理而构建,即,在肿瘤/正常混合物中,体细胞变体和种系变体通常具有不同的预期等位基因频率,该等位基因频率由肿瘤分数、肿瘤倍性和局部拷贝数决定。然而,与通过肿瘤分数、肿瘤倍性和局部拷贝数的计算模型来估计预期种系等位基因频率的SGZ相比,本文公开的新方法直接从与所讨论变体位于相同拷贝数片段上的已知种系SNP推断预期种系等位基因频率。因此,使用本文所述方法,无需确定拷贝数或肿瘤纯度或对其建模来获得针对体细胞变体和种系变体的准确调用。
在一些实施例中,基于观察到的变体等位基因频率和推断出的预期种系变体等位基因频率之间的差异,使用经训练模型(诸如逻辑回归模型)来预测变体是体细胞的概率。在一些实施例中,使用针对匹配的肿瘤/正常对的数据来训练模型,并使用独立的数据集来验证模型。在一些实施例中,使用针对具有已知种系序列(以及任选地具有已知体细胞序列)的肿瘤样品的数据来训练模型。在一些实施例中,使用针对具有已知种系(以及任选地具有已知体细胞)序列的混合肿瘤/正常样品的数据来训练模型。验证表明,新分类器在针对体细胞变体分类的敏感性和阳性预测值(PPV)方面优于SGZ。
确定的基因组序列可以是体细胞变体序列或种系序列。存在已知种系序列的可公开访问数据库(参见例如dbSNP(可在www.ncbi.nlm.nih.gov/snp/获得)或gnomAD(可在gnomad.broadinstitute.org获得)),并且已知种系序列与通过对从受试者的样品获得中的核酸进行测序来确定的序列之间的匹配表明与该样品相关联的该序列可能是种系序列。然而,与已知种系序列不匹配并不能证明该序列是体细胞变体序列,因为它可能是受试者的先前未知(或未记录)种系序列。本文所述方法允许将序列分类为种系序列或体细胞变体序列。
调用体细胞或种系序列的方法
本文所述方法允许将感兴趣基因组序列鉴定为种系序列或体细胞序列。在一些实施例中,体细胞序列与患者的癌症相关联。例如,患者样品可以包括肿瘤核酸分子(即,直接(诸如在肿瘤活检的情况下)或间接(诸如在液体活检或体液样品的情况下,包括循环肿瘤DNA(ctDNA)以及无细胞DNA(cfDNA))源自肿瘤的核酸分子)和非肿瘤核酸分子(即,源自非肿瘤并且优选健康的组织、细胞、液体活检样品或体液样品的核酸分子)的混合物。该方法可以包括从患者基因组序列内选择感兴趣基因组序列的步骤(即,针对患者获得的基因组序列,该基因组序列可以是整个基因组或其部分(例如,整个基因组内的外显子组或靶向区域)),以及选择针对该感兴趣基因组序列的一个或多个代理基因组序列的步骤。患者基因组序列可以包括在任何给定基因座处的一个或多个等位基因(例如,在任何给定基因座处的体细胞序列和/或种系序列)。
可以对来自样品(例如,混合肿瘤/正常组织样品或含有ctDNA和非肿瘤cfDNA混合物的无细胞DNA(cfDNA)样品)的核酸分子进行测序以确定患者基因组序列。可以在基因组基因座处从患者基因组序列鉴定或选择感兴趣基因组序列。所选择的基因组序列是被表征为种系或体细胞的测试序列。在一些实施例中,感兴趣基因组序列不同于参考序列。在一些实施例中,感兴趣基因组序列不同于所选择的种系序列数据库中的序列。
图1是样品基因组区域的示意图。区域100可以包括生物体的整个基因组或可以仅包括整个基因组的分数。尽管区域100在图1中被示出为实线,但通常区域100可能包括在生物体的染色体上物理分离的几种成分。在一些实施方式中,从其确定区域100的样品可以包括正常患者组织、包括正常细胞或无细胞DNA的流体或其他解剖材料。在一些实施方式中,样品可以包括异常(例如,癌性或基因突变)组织、包括异常细胞或循环肿瘤DNA的流体或其他解剖材料。在一些实施方式中,样品可以包括正常和异常组织、流体或其他解剖材料的组合。
图1所示的基因组区域100可以对应于DNA的单链或链片段或RNA的链或链片段。虽然未在图1示出,但区域100包括由各种碱基(即,胞嘧啶(“C”)、鸟嘌呤(“G”)、腺嘌呤(“A”)、胸腺嘧啶(“T”)或尿嘧啶(“U”))组成的序列。碱基的特定序列通常可以确定解剖材料或患者的重要特征,例如,患者是否患有癌症,并且如果是,什么疗法可能对治疗癌症有效或无效。
下面描述的技术包括将基因组区域100内的感兴趣序列102表征为种系或体细胞。通过使用参考序列104来辅助表征。参考序列104是表示“正常”(例如,非癌性)患者的示例性基因组序列。在一些实施方式中,参考序列104可以包括由人类基因组计划确定的序列,例如,hg19。
在参考序列104中,存在已知多态性区域106a、106b。多态性区域106a、106b是这样的区域(包括从单个碱基到数百个或更多个碱基的任何数量的碱基),其中预期特定生物体的基因组序列在生物体群体中发生变异,而没有对应于该变异的不利后果。例如,在人类中存在对应于各种头发颜色、眼睛颜色或其他个性化特征的多态性区域。对应于实际患者样品的基因组区域100将在区域100中在对应于参考序列104中的多态性区域106a、106b的位置处具有特定的碱基值108a、108b。换句话说,参考序列104的多态性区域106a、106b是确定人的某些特征(例如,头发颜色)的位置;基值108a、108b是对描述特定患者的那些特征(例如,红头发)的个体化确定。
在一些情况下,多态性区域106a、106b包括一个或多个单核苷酸多态性(或“SNP”)。在一些情况下,多态性区域可以包括整个等位基因或其部分。
图2是针对用于区分种系基因组序列和体细胞基因组序列的过程的流程图。过程200开始于鉴定(即,选择或分类)感兴趣基因组区域(步骤202)。在一些实施方式中,步骤202包括从更大的基因组区域100内鉴定感兴趣区域(即,感兴趣序列)102。
从物理样品确定基因组序列(例如,基因组区域100)可以以多种方式完成。在美国专利号9,340,830中描述了一种这样的方式,以及在美国专利发布2017/0356053中描述了另一种,两者的全部内容均以引用方式并入本文。更一般地,存在可用于确定输入样品的遗传序列的一类机器,其被称为基因组测序仪。在一些情况下,所公开的方法和系统可以使用各种下一代测序(NGS)技术和测序仪中的任何一者来实现,包括被配置用于大规模并行测序的循环阵列测序仪和单分子测序仪。此外,存在人类和其他生物体的基因组的各种已知子区域,这些子区域已知与各种医学状况相关。
本文描述的技术不依赖于使用特定测序平台或特定测序技术,并且这些机器和伴随技术中的任何一者都可以在步骤202中使用。在一些情况下,所公开的方法可以使用另选的核酸序列分析技术来实现,例如,微阵列、荧光原位杂交(FISH)等。
在一些实施方式中,感兴趣区域(即,序列)102被鉴定为对应于参考基因组104内的已知遗传基因座。在一些实施方式中,感兴趣区域102对应于相对于参考序列104的突变(即,除了具有与参考序列104的对应部分不同的遗传序列的多态性区域之外的基因组区域100的子部分)。在一些实施方式中,感兴趣序列对应于与患者所患的医学疾病相关的基因。在一些实施方式中,感兴趣区域102是致癌基因或其部分。
在步骤204中,鉴定针对基因组序列的一个或多个代理基因组序列(步骤204)。所选择的一个或多个代理基因组序列可以是已知种系序列(例如,基于与来自已知种系序列数据库的已知种系序列匹配或通过对来自受试者或另一健康个体的健康组织、细胞或无细胞DNA进行测序)。参考图1,代理110的一个表征是遗传基因座处的序列,(a)已知该序列编码种系遗传信息,以及(b)已知该序列与感兴趣序列102具有相同的拷贝数(例如,因为在物理上靠近感兴趣序列102或被确认位于与该感兴趣序列相同的拷贝数片段内)。另选的表征是要求已知代理110编码体细胞遗传信息。为了方便起见,除非另有规定外,本文档将假定代理110编码种系信息,但本领域技术人员将理解这两种方法的等效性。
特定候选代理序列的种系状态可从研究文献、公开可用的数据库(例如,dbSNP(可在www.ncbi.nlm.nih.gov/snp/获得)或gnomAD(可在gnomad.broadinstitute.org获得))获知或者可以通过其他从头开始的方式发现。另一方面,可以从匹配的肿瘤/正常样品鉴定体细胞变体;即,来自同一患者的同时包括肿瘤DNA和非肿瘤(“正常”)DNA两者的样品。特别地,在肿瘤DNA中看到但在相应的正常DNA中看不到的变体必然是体细胞变体。也可以通过其他从头开始的方式发现已知体细胞变体。
参考图3,在一些实施方式中,通过采用划分过程来执行步骤204。在这样的过程中,基于遗传参数将患者的基因组的部分100分割为片段(在图3中由垂直虚线描绘)。定义片段,使得特定片段中的参数值全部相等(在期望范围内或在期望阈值内)。例如,片段可以是具有大致相同(即,在期望范围内或在期望阈值内)的测序深度或拷贝数的连续序列。在一些实施方式中,用于划分输入的遗传参数包括拷贝数、感兴趣等位基因或亚等位基因片段的频率或其他。一个或多个代理序列可以位于与感兴趣基因组序列相同的片段内,从而使得一个或多个代理基因组序列和感兴趣基因组序列很可能具有相同的拷贝数。
各种分割过程是本领域中已知的。例如,iSeg(在Girimurugan等人,iSeg:anEfficient Algorithm for Segmentation of Genomic and Epigenomic Data,BMCBioinformatics 19:131(2018)中描述,其全部内容并入本文)、CBS(在Olshen等人,Circular Binary Segmentation for the Analysis of Array-Based DNA Copy NumberData,Biostatistics 2004Oct;5(4):557-72中描述,其全部内容以引用的方式并入本文)、SLMSuite(在Orlandini等人,SLMSuite:A Suite of Algorithms for SegmentingGenomic Profiles,BMC Bioinformatics 18:321(2017)中描述)、Pelt(在Killick等人,Optimal detection of changepoints with a linear computational cost,Journal ofthe American Statistical Association,107:500(2012)中描述,其全部内容以引用的方式并入本文)是许多此类算法中的四种。在一些实施例中,患者基因组序列基于每个片段内的拷贝数均匀性被分割为多个片段。
返回参考图2,在一些实施方式中,仅鉴定与感兴趣区域102位于同一片段上的代理110。在一些实施方式中,代理110包括位于与感兴趣区域102相同的片段上的所有已知种系SNP。在一些实施方式中,代理110包括位于与感兴趣区域102相同的片段上的所有已知种系等位基因。在一些实施方式中,例如,如果难以将基因组序列正确分割为对应于不同拷贝数的片段,则仅鉴定与感兴趣区域102相距不超过预定碱基数的代理110。例如,在一些情况下,将感兴趣区域与代理序列分开的最大碱基数可以在约10个碱基至约1,000个碱基的范围内。在一些情况下,将感兴趣区域与代理序列分开的最大碱基数可以是约10个碱基、20个碱基、30个碱基、40个碱基、50个碱基、60个碱基、70个碱基、80个碱基、90个碱基、100个碱基、200个碱基、300个碱基、400个碱基、500个碱基、600个碱基、700个碱基、800个碱基、900个碱基或1,000个碱基。在一些情况下,将感兴趣区域与代理序列分开的最大碱基数可以具有本段所述的值范围内的任何值。
在步骤206中,鉴定代理110的频率。在步骤208中,鉴定来自感兴趣区域(即,感兴趣基因组序列)102的序列的等位基因频率(等位基因分数)。此处,“频率”是指归一化的统计频率,例如,样品内的序列或代理的出现次数除以同一基因组基因座处的任何序列的出现总次数。在一些实施方式中,可以进行多个频率测量。感兴趣基因组序列和一个或多个代理基因组序列的等位基因频率可以通过对来自受试者的样品中的核酸分子进行测序来确定。在某些情况下,可以使用其他方法确定等位基因频率,例如,微阵列或荧光原位杂交(FISH)技术。当使用多个代理时,异常代理频率可能被丢弃,并且其余频率可能被组合为单个统计中心性量度(例如,汇总统计量(诸如平均值,中位数,众数或其他)或代理序列的等位基因频率的分布(诸如概率分布),以便步骤210包括单个数值比较。例如,在一些实施例中,中心性量度(汇总统计量)是针对一个或多个代理序列的平均等位基因频率。在一些实施例中,中心性量度(汇总统计量)是针对一个或多个代理序列的中位等位基因频率。当使用单个代理基因组序列时,代理基因组序列的观察到的频率的中心量度是该代理序列的频率。在一些实施例中,中心性量度可以是针对代理序列的观察到的等位基因频率的分布。
在决策210中,将一个或多个代理频率(例如,一个或多个代理序列的观察到的频率的中心性量度)与感兴趣区域的一个或多个频率进行比较,以确定它们是否相等。此处以及整个本申请,术语“相等”包括“等于在期望范围内”或“等于在期望阈值内”,其可以基于过程200的期望选择性和特异性而常规地确定。可以例如使用本领域技术人员选择的统计阈值或统计检验来设置范围或阈值。如果使用多个代理110并进行单独比较而不是对如上所述的代理频率进行组合,则如果一定比例的比较(例如,大于50%、大于55%、大于60%、大于65%、大于70%、大于75%、大于80%、大于85%、大于90%、大于95%)相等,决策210会导致“是”。
如果代理频率等于感兴趣序列的频率,则感兴趣序列被分类为种系(步骤212)。否则,感兴趣序列被分类为体细胞(步骤214)。另选地,如果代理110被选择为已知编码体细胞信息(而不是种系信息),则相等频率被解释为感兴趣序列为体细胞,并且不相等频率被解释为感兴趣序列为种系。
在一些实施方式中,决策210中的比较也可以用于消除潜在的错误分类。特别地,真正的体细胞变体的频率必然低于真正的种系变体的频率,因为肿瘤和非肿瘤DNA都对种系变体的频率计数有贡献,而只有肿瘤DNA对体细胞变体的频率计数有贡献。因此,在一些实施方式中,如果感兴趣序列的频率大于代理频率,则感兴趣序列被分类为种系。
举例来说,在一些实施例中,将感兴趣基因组序列的观察到的频率与一个或多个代理基因组序列的观察到的频率的中心性量度进行比较可以包括:从预期等位基因频率确定感兴趣基因组序列的“等位基因频率距离”(AFDIS)。如果感兴趣基因组序列为种系序列,则基于一个或多个代理序列的频率(或指示一个或多个代理序列的观察到的频率的汇总统计量)来确定预期等位基因频率,基于对该一个或多个代理序列的选择将该一个或多个代理序列假设为种系。在一些实施例中,根据下式,AFDIS可以用数字表示
AFDIS=AF
其中如果感兴趣基因组序列为种系,则AF
在一些实施例中,等位基因频率距离可以使用代理基因组序列的观察到的频率的分布来确定。分布可以用于确定感兴趣基因组序列为种系或体细胞的概率。在一些实施例中,等位基因频率距离是感兴趣基因组序列的观察到的频率拟合(或不拟合)在多个代理序列的观察到的频率的分布内的概率。例如,如果感兴趣基因组序列的等位基因频率拟合在分布内,则感兴趣基因组序列可以被鉴定为种系序列。如果感兴趣基因组序列的等位基因频率不符合分布,则感兴趣基因组序列可以被鉴定为体细胞序列。本领域技术人员可以选择统计检验或预定阈值来确定感兴趣基因组序列的等位基因频率是否拟合在分布内。
在一些实施例中,等位基因频率距离可以用于对感兴趣基因组序列进行分类。例如,在一些实施例中,如果等位基因频率距离高于所选择的阈值,则感兴趣基因组序列被分类为体细胞。在一些实施例中,如果等位基因频率距离低于所选择的阈值,则感兴趣基因组序列被分类为种系。阈值可以基于所需的准确性或特异性公差来设置。
在一些实施例中,将感兴趣基因组序列分类为种系或体细胞可以包括使用统计模型。统计模型可以接收例如针对给定感兴趣基因组序列的等位基因频率距离,并输出对感兴趣基因组序列的分类作为体细胞(或可能是体细胞)或种系(或可能是种系)。分类可以是基于感兴趣基因组序列为体细胞或种系的概率。在一些实施方式中,感兴趣基因组序列可以被分类为不明确的,例如,如果序列为体细胞或种系的概率不够高。进行调用的概率阈值可以是基于调用的所需特异性和/或准确性。例如,在一些实施例中,如果感兴趣基因组序列为体细胞的概率高于0.8、0.85、0.9、0.95、0.96、0.97、0.98或0.99(或其间的任何所选择的值)中的任何一者,则感兴趣基因组序列被分类为体细胞,并且如果感兴趣基因组序列为体细胞的概率低于0.2、0.15、0.1、0.05、0.04、0.03、0.02或0.01(或其间的任何所选择的值)中的任何一者,则感兴趣基因组序列被分类为种系。未被分类为体细胞或种系的感兴趣基因组序列基于统计模型可能被标记为不明确的。
在一些实施例中,使用来自一个或多个匹配的肿瘤/正常样品对的数据来训练统计模型。可以对匹配的肿瘤/正常样品对中的正常样品进行测序,以建立针对种系序列的基本真值,并且可以对肿瘤样品进行测序,以建立针对体细胞变体序列(即,根据匹配的正常样品不是种系的那些序列)的基本真值。来自其可以包括混合正常和肿瘤核酸分子的肿瘤样品的测序数据可以用于确定针对所选择的感兴趣基因组序列的等位基因频率距离,然后该所选择的感兴趣基因组序列被标记为体细胞(为体细胞的概率p
可以使用训练统计模型的其他方法。例如,在一些实施例中,仅使用针对种系序列的数据或仅使用针对体细胞序列的数据来训练模型。
在一些实施方式中,步骤210的比较可以通过统计模型的方式来间接地执行。例如,如果代理集合的中位等位基因频率被用作步骤206的中心量度,则可以构建逻辑回归模型,该逻辑回归模型描述感兴趣序列的等位基因频率与代理的中位等位基因频率的差异。在一些实施方式中,可以从针对一组匹配的肿瘤/正常样品的数据构建此逻辑回归模型,使得上一句所述的差异与
这种表征的基本原理是每个代理在物理上都靠近患者的基因组中的感兴趣序列。因此,代理和感兴趣序列很可能经历相同或相似的基因组动力学或突变,诸如重复事件或缺失。这种方法不是试图对感兴趣序列的特定动力学进行建模以将观察到的频率与种系/体细胞状态相关联,而是用直接经验测量来代替这种模型。就现有技术模型历来在某种程度上不敏感或不准确而言,这种方法提供了优势。
本文所述的方法还可以包括:生成报告,该报告将一个或多个感兴趣基因组序列指示为种系或体细胞。可以将所生成的报告传输到患者、医疗保健提供者或其他(例如,使用计算机网络)。该报告对于评估癌症治疗疗法、制定治疗决策、监测癌症进展或复发、设计个性化癌症疫苗以及其他有益用途特别有益。
电子设备和系统
图4示出了根据一个实施例的系统的示例。设备400可以是连接到网络的主机计算机。设备400可以是客户端计算机或服务器。如图4所示,设备400可以是任何合适类型的基于微处理器的设备,诸如个人计算机、工作站、服务器或手持计算设备(便携式电子设备),诸如电话或平板电脑。设备可以包括例如一个或多个处理器410、输入设备420、输出设备430、存储器440和/或通信设备460。输入设备420和输出设备430可以与计算机连接或集成。在一些实施例中,设备被配置为操作测序仪470,该测序仪可以对患者样品中的核酸分子进行测序以获得测序数据。
输入设备420可以是提供输入的任何合适的设备,诸如触摸屏、键盘或小键盘、鼠标或语音识别设备。输出设备430可以是提供输出的任何合适的设备,诸如显示器、触摸屏、触觉设备或扬声器。
存储器440可以是提供存储的任何合适的设备,诸如电存储器、磁存储器或光学存储器,包括RAM、高速缓存、硬盘驱动器或可移动存储盘。通信设备460可以包括能够通过网络发送和接收信号的任何合适的设备,诸如网络接口芯片或设备。计算机的组件可以以任何合适的方式连接,诸如经由物理总线或无线方式。
可以被存储在存储器440中并由处理器410执行的软件(诸如SGZ模块450和其他序列分析和变体调用程序模块)可以包括例如针对基于AFDIS的逻辑回归模型的代码以及体现本公开的功能的其他程序(例如,如在如上所述设备中体现)。
软件(诸如SGZ模块450和其他序列分析和变体调用程序模块)也可以在任何非暂态计算机可读存储介质中存储和/或传输,以供指令执行系统、装置或设备(诸如上述那些)使用或与其结合使用,该软件可以从指令执行系统、装置或设备获取与该软件相关联的指令并执行该指令。在本公开的上下文中,计算机可读存储介质可以是任何介质,诸如存储器440,其可以包括或存储供指令执行系统、设备或装置使用或与其结合使用的程序。
软件(诸如SGZ模块450和其他序列分析和变体调用程序模块)也可以在任何传输介质中传播,以供指令执行系统、装置或设备(诸如上述那些)使用或与其结合使用,该软件可以从指令执行系统、装置或设备获取与该软件相关联的指令并执行该指令。在本公开的上下文中,传输介质可以是可以传送、传播或传输程序以供指令执行系统、设备或装置使用或与其结合使用的任何介质。传输可读介质可以包括但不限于电子、磁、光学、电磁或红外有线或无线传播介质。
设备400可以连接到网络,该网络可以是任何合适类型的互连通信系统。网络可以实现任何合适的通信协议并且可以由任何合适的安全协议保护。网络可以包括可以实现网络信号的传输和接收的任何合适布置的网络链路,诸如无线网络连接、T1或T3线路、有线网络、DSL或电话线。
设备400可以实现适合在网络上运行的任何操作系统。软件(诸如SGZ模块450和其他序列分析和变体调用程序模块)可以用任何合适的编程语言编写,诸如C、C++、Java或Python。例如,在各种实施例中,体现本公开的功能的应用软件可以以不同的配置(诸如以客户端/服务器布置或通过网络浏览器)来部署作为基于网络的应用或网络服务。
受试者、样品和测序
与本文所述的方法一起使用的受试者样品(例如,患者样品)可以包括肿瘤和非肿瘤核酸分子的混合物。肿瘤核酸分子可以直接或间接地获自肿瘤。例如,肿瘤核酸分子可以从肿瘤的组织活检获得。肿瘤活检通常包括肿瘤和非肿瘤组织,由此提供肿瘤和非肿瘤核酸分子的混合物。在一些实施例中,肿瘤和非肿瘤核酸分子从体液或液体活检样品获得(例如,血液、血浆、脊髓液等),其可以包括无细胞(或循环无细胞)DNA,该无细胞DNA包括肿瘤(例如,循环肿瘤DNA或ctDNA)和非肿瘤无细胞核酸分子。
患者样品可以取自例如患有癌症的受试者、疑似患有癌症的受试者或先前已经接受针对癌症的治疗的受试者。在某些实施方案中,样品获自患有实体瘤、血液癌或其转移形式的受试者。在某些实施例中,样品从患有或高风险患有癌症的受试者获得。在某些实施方案中,样品从未接受治疗癌症的疗法、正在接受治疗癌症的疗法或已经接受治疗癌症的疗法的受试者获得,如本文所述。
各种组织可以是本方法中使用的样品来源。基因组或亚基因组核酸(例如,DNA或RNA)可以与受试者的样品(例如,包括肿瘤细胞的样品、血液样品、血液成分样品,包括无细胞DNA(cfDNA)的样品、包括循环肿瘤DNA(ctDNA)的样品、包括循环肿瘤细胞(CTC)的样品或任何正常对照(例如,正常相邻组织(NAT))分离。
在一些实施例中,样品获自液体活检。液体活检患者样品可以源自例如血液、血浆、脑脊髓液、痰、粪便、尿液或唾液。
在一些实施例中,患者样品源自固体组织样品,诸如实体瘤活检。实体瘤活检通常包括肿瘤和非-肿瘤组织的混合物。在一些实施例中,固体组织活检样品为新鲜样品。在一些实施例中,固体组织活检样品为冷冻样品或先前冷冻样品。在一些实施例中,固体组织活检样品为新鲜样品。在一些实施例中,固体组织活检样品为保存的样品(例如,化学保存的样品)。在某些实施例中,样品为福尔马林固定的、石蜡包埋的(FFPE)样品。
在一些实施例中,对于本文公开的样品类型中的任何样品类型,患者样品的肿瘤纯度(即,其为肿瘤核酸分子的样品部分与总核酸分子相比)为约1%或更多、约5%或更多、约10%或更多、约15%或更多、约20%或更多、约25%或更多、约30%或更多、约40%或更多、约50%或更多,约60%或更多,约70%或更多或约80%或更多。在一些实施例中,患者样品的肿瘤纯度为约99%或更少、约95%或更少、约90%或更少、约85%或更少、约80%或更少、约75%或更少、约70%或更少、约60%或更少、约50%或更少、约40%或更少、约30%或更少、约25%或更少、或约20%或更少。
在一个实施例中,该方法进一步包括获得样品,例如,本文所述的患者样品。可以直接或间接地获取样品。在实施例中,例如通过分离或纯化从包括cfDNA的样品获取样品。在实施例中,例如通过分离或纯化从包括ctDNA的样品获取样品。在实施例中,例如通过分离或纯化从包括恶性细胞和非恶性细胞(例如,肿瘤浸润淋巴细胞)的样品获取样品。在实施例中,例如通过分离或纯化从包括CTC的样品获取样品。在一些实施例中,通过固体组织活检获得样品。
可以使用已知方法从患者样品制备测序文库。可以从患者样品纯化或分离核酸分子。在一些实施例中,使用已知方法将分离的核酸进行片段化或剪切。例如,核酸分子可以通过物理剪切方法(例如,超声处理)、酶促裂解方法、化学裂解方法和本领域技术人员熟知的其他方法进行片段化。可以将核酸连接到用于测序的衔接子序列。在一些情况下,衔接子可以包括扩增引物和/或测序衔接子。在一些情况下,可以例如使用本领域技术人员熟知的聚合酶链式反应(PCR)或等温扩增方法来扩增从患者样品中纯化或分离的核酸分子或由其制备的测序文库。
在一些实施例中,对来自患者样品并用于制备测序文库(或其所选择的(例如,所捕获的)子集)的核酸分子进行测序,以生成患者基因组序列。测序方法是本领域众所周知的,并且可以使用多重(例如,下一代)或单分子测序来执行。通过测序确定的患者基因组序列不必是患者的全基因组。例如,在一些实施例中,靶向测序方法(例如,使用特定探针(或诱饵)分子进行基于杂交的捕获)用于对患者的基因组的部分进行测序(即,少于全基因组)。参见,例如美国专利号9,340,830B2。靶向测序可以用于靶向例如一个或多个外显子区域、一个或多个内含子区域、一个或多个基因内区域、一个或多个3′-UTR(未翻译区)和/或一个或多个5′-UTR。
在一些实施例中,靶向测序可用于对与癌症相关联的一个或多个基因或一个或多个基因的部分进行测序。可以使用靶向测序进行测序的与癌症相关联的示例性基因包括但不限于ABL2、AKT2、AKT3、ARAF、ARFRP1、ARID1A、ATM、ATR、AURKA、AURKB、BCL2、BCL2A1、BCL2L1、BCL2L2、BCL6、BRCA1、BRCA2、CARD11、CBL、CCND1、CCND2、CCND3、CCNE1、CDH1、CDH2、CDH20、CDH5、CDK4、CDK6、CDK8、CDKN2B、CDKN2C、CHEK1、CHEK2、CRKL、CRLF2、DNMT3A、DOT1L、EPHA3、EPHA5、EPHA6、EPHA7、EPHB1、EPHB4、EPHB6、ERBB3、ERBB4、ERG、ETV1、ETV4、ETV5、ETV6、EWSR1、EZH2、FANCA、FBXW7、FGFR4、FLT1、FLT4、FOXP4、GATA1、GNA11、GNAQ、GNAS、GPR124、GUCY1A2、HOXA3、HSP90AA1、IDH1、IDH2、IGF1R、IGF2R、IKBKE、IKZF1、INHBA、IRS2、JAK1、JAK3、JUN、KDR、LRP1B、LTK、MAP2K1、MAP2K2、MAP2K4、MCL1、MDM2、MDM4、MEN1、MITF、MLH1、MPL、MRE11A、MSH2、MSH6、MTOR、MUTYH、MYCL1、MYCN、NF2、NKX2-1、NTRK1、NTRK3、PAK3、PAX5、PDGFRB、PIK3R1、PKHD1、PLCG1、PRKDC、PTCH1、PTPN11、PTPRD、RAF1、RARA、RICTOR、RPTOR、RUNX1、SMAD2、SMAD3、SMAD4、SMARCA4、SMARCB1、SMO、SOX10、SOX2、SRC、STK11、TBX22、TET2、TGFBR2、TMPRSS2、TOP1、TSC1、TSC2、USP9X、VHL、WT1、ABL1、AKT1、ALK、APC、AR、BRAF、CDKN2A、CEBPA、CTNNB1、EGFR、ERBB2、ESR1、FGFR1、FGFR2、FGFR3、FLT3、HRAS、JAK2、KIT、KRAS、MET、MLL、MYC、NF1、NOTCH1、NPM1、NRAS、PDGFRA、PIK3CA、PTEN、RB1、RET和TP53。
在一些实施例中,从患有癌症的受试者获取样品。示例性癌症包括但不限于B细胞癌,例如,多发性骨髓瘤、黑色素瘤、乳腺癌、肺癌(诸如非小细胞肺癌或NSCLC)、支气管癌、结直肠癌、前列腺癌、胰腺癌、胃癌、卵巢癌、膀胱癌、脑或中枢神经系统癌、周围神经系统癌、食道癌、宫颈癌、子宫或子宫内膜癌、口腔癌或咽癌、肝癌、肾癌、睾丸癌、胆道癌、小肠或阑尾癌、唾液腺癌、甲状腺癌、肾上腺癌、骨肉瘤、软骨肉瘤、血液组织癌、腺癌、炎症性肌纤维母细胞瘤、胃肠道间质瘤(GIST)、结肠癌、多发性骨髓瘤(MM)、骨髓增生异常综合征(MDS)、骨髓增殖性疾病(MPD)、急性淋巴细胞白血病(ALL)、急性髓细胞白血病(AML)、慢性粒细胞性白血病(CML)、慢性淋巴细胞白血病(CLL)、真性红细胞增多症、霍奇金淋巴瘤、非霍奇金淋巴瘤(NHL)、软组织肉瘤、纤维肉瘤、粘液肉瘤、脂肪肉瘤、成骨肉瘤、脊索瘤、血管肉瘤、内皮肉瘤、淋巴管肉瘤、淋巴管内皮肉瘤、滑膜瘤、间皮瘤、尤文氏瘤、平滑肌肉瘤、横纹肌肉瘤、鳞状细胞癌、基底细胞癌、腺癌、汗腺癌、皮脂腺癌、乳头状癌、乳头状腺癌、髓样癌、支气管癌、肾细胞癌、肝癌、胆汁癌导管癌、绒毛膜癌、精原细胞瘤、胚胎癌、维尔姆斯瘤、膀胱癌、上皮癌、神经胶质瘤、星形细胞瘤、髓母细胞瘤、颅咽管瘤、室管膜瘤、松果体瘤、血管母细胞瘤、听神经瘤、少突胶质细胞瘤、脑膜瘤、神经母细胞瘤、视网膜母细胞瘤、滤泡性淋巴瘤、弥漫性大B细胞淋巴瘤、套细胞淋巴瘤、肝细胞癌、甲状腺癌、胃癌、头颈癌、小细胞癌、原发性血小板增多症、特发性骨髓化生、嗜酸性粒细胞增多综合征、系统性肥大细胞增多症、常见嗜酸性粒细胞增多症、慢性嗜酸性粒细胞白血病、神经内分泌癌、类癌瘤等。
在实施例中,癌症是血液恶性肿瘤(或癌前病变)。如本文所用,血液恶性肿瘤是指造血或淋巴组织的肿瘤,例如,影响血液、骨髓或淋巴结的肿瘤。示例性血液恶性肿瘤包括但不限于白血病(例如,急性淋巴细胞白血病(ALL)、急性髓性白血病(AML)、慢性淋巴细胞性白血病(CLL)、慢性粒细胞性白血病(CML)、多毛细胞白血病、急性单核细胞白血病(AMoL)、慢性粒单核细胞白血病(CMML)、幼年粒单核细胞白血病(JMML)或大颗粒淋巴细胞白血病)、淋巴瘤(例如,艾滋病相关淋巴瘤、皮肤T细胞淋巴瘤、霍奇金淋巴瘤(例如,经典霍奇金淋巴瘤或结节性淋巴细胞为主的霍奇金淋巴瘤)、蕈样肉芽肿、非霍奇金淋巴瘤(例如,B细胞非霍奇金淋巴瘤(例如,伯基特淋巴瘤、小淋巴细胞淋巴瘤(CLL/SLL)、弥漫性大B细胞淋巴瘤、滤泡性淋巴瘤、免疫母细胞性大细胞淋巴瘤、前体B淋巴母细胞淋巴瘤或套细胞淋巴瘤)或非T细胞淋巴瘤-霍奇金淋巴瘤(蕈样肉芽肿、间变性大细胞淋巴瘤淋巴瘤或前体T淋巴母细胞淋巴瘤)、原发性中枢神经系统淋巴瘤、Sézary综合征、
在一些实施例中,从受试者(例如,患有症状或疾病(例如,过度增殖性疾病(例如,如本文所述))的患者)获取(例如,收集)样品。在一些实施例中,疾病是过渡增殖性疾病。在一些实施例中,过度增殖性疾病是癌症,例如,实体瘤或血液癌症。在一些实施例中,该癌症为实体瘤。在一些实施例中,癌症是血液癌症,例如,白血病或淋巴瘤。
在一些实施例中,受试者患有癌症。在一些实施例中,受试者已经接受或正在接收针对癌症的治疗。在一些实施例中,受试者需要针对癌症进展或消退被监测,例如,在接受癌症治疗之后。在一些实施例中,受试者需要针对癌症复发被监测。在一些实施例中,受试者有患癌症的风险。在一些实施例中,受试者尚未接受癌症治疗。在一些实施例中,受试者具有癌症的遗传倾向(例如,具有增加他或她患癌症的基线风险的突变)。在一些实施例中,受试者已经暴露于增加他或她患癌症风险的环境(例如,辐射或化学物质)。在一些实施例中,受试者需要针对患癌症被监测。
在一些实施例中,患者先前已经接受用靶向疗法治疗,例如,一种或多种靶向疗法。在一些实施例中,对于先前已经接受用靶向疗法治疗的患者,获得(例如,收集)靶向疗法后样品,例如,标本。在一些实施例中,靶向治疗后样品为在完成靶向治疗之后获得(例如,收集)的样品。
在一些实施例中,患者先前未接受用靶向疗法治疗。在一些实施例中,对于先前未接受用靶向疗法治疗的患者,样品包括切除物,例如,原始切除物或复发肿瘤,例如,疾病复发后疗法,例如,非靶向疗法。在一些实施例中,样品为原发性肿瘤或转移瘤或是其部分,例如,转移活检。在一些实施例中,样品从位点,例如,肿瘤位点获得,与相邻位点(例如,具有肿瘤细胞的相邻位点)相比具有最高百分比的肿瘤,例如,肿瘤细胞。在一些实施例中,样品从位点,例如,肿瘤位点获得,与相邻位点(例如,具有肿瘤细胞的相邻位点)相比具有最大肿瘤焦点。
在一些实施例中,受试者是人。
治疗癌症的方法
癌症的基因组特征通常会影响各种癌症治疗方式的成功率。例如,给定的抗癌剂可能相对于另一种癌症更有可能成功治疗具有一种基因组特征的特定癌症。本文所述的方法可以用于通过区分可以归因于癌症的体细胞序列与种系序列来表征癌症的基因组特征。
举例来说,治疗患者癌症的方法可以包括:使用本文所述的方法将一个或多个感兴趣基因组序列鉴定(例如,分类)为体细胞,并基于一个或多个所鉴定的体细胞序列来选择癌症治疗方式。然后,可以使用有效量的所选择的癌症治疗方式来治疗癌症。这允许基于特定于该患者的癌症的体细胞序列对患者进行个性化的癌症治疗。相反,如果治疗选择是基于种系变体而不是体细胞变体,则存在所选择的治疗方式可能对患者的癌症无效的一定风险。
示例性癌症治疗方式可以包括例如所选择的化学治疗剂、所选择的免疫肿瘤剂(诸如免疫检查点抑制剂)、切除手术、放射疗法、靶向疗法、基因表达调节剂、血管生成抑制剂和激素疗法等。
可以例如基于一个或多个所鉴定的体细胞序列与使用所选择的治疗方式的成功癌症治疗之间的关联来选择癌症治疗。表1中列出了癌症类型、体细胞序列和治疗方式之间的示例性关联。
表1
癌症的微卫星不稳定性(MSI)状态可以用于选择癌症的治疗方式。微卫星不稳定性可以由癌细胞中的缺陷DNA错配修复(MMR)通路引起,这导致异常高频率的基因突变。参见Kim等人,The Landscape of Microsatellite Instability in Colorectal andEndometrial Cancer Genomes,Cell,vol.,155,no.4,pp.858-868(2013)。MSI状态通常基于MSI特征被表征为高(MSI-H)、低(MSI-L)或稳定(MSS)(或另选地,MSI-H或非MSI-H;或MSI-H或MSI未确定的)。MSI-H状态已经针对多种类型的实体瘤被检测到,并且可能是使用某些癌症治疗方式成功治疗癌症的指标。参见Cortes-Ciriano等人,A molecularportrait of microsatellite instability across multiple cancers,NatureCommunications,vol.8,no.15180(2017)。微卫星中的突变(即,MSI事件)可以通过使用本文所述的方法区分体细胞序列和种系序列来检测。
某些癌症治疗方式的成功已经与癌症的MSI-H状态有关联。例如,已发现PD-1抑制剂(即,派姆单抗)在治疗MSI-H实体瘤(例如,不可切除或转移性实体瘤)方面特别有效。在一些实施例中,被确定具有MSI-H状态的癌症用有效量的免疫肿瘤剂来进行治疗。在一些实施例中,被确定具有MSI-H状态的癌症用有效量的免疫检查点抑制剂来进行治疗。在一些实施例中,免疫检查点抑制剂为AMP-224、AMP-514、阿特朱单抗、AUNP12、阿维单抗、BGB-A317、BMS-986189、CA-170、卡瑞利珠单抗、西米普利单抗、CK-301、多塔利单抗,德瓦鲁单抗、伊匹木单抗、INCMGA00012、KN035、纳武单抗、派姆单抗、信迪利单抗、斯巴达珠单抗、替雷利珠单抗或特瑞普利单抗。在一些实施例中,被确定具有MSI-H状态的癌症用有效量的PD-1抑制剂、PD-L1抑制剂或CTLA-4抑制剂来进行治疗。在一些实施例中,被确定具有MSI-H状态的癌症用有效量的派姆单抗来进行治疗。
在一些实施例中,治疗癌症的方法包括:使用本文所述的方法将一个或多个感兴趣基因组序列鉴定(例如,分类)为体细胞;使用所鉴定的体细胞序列确定癌症的微卫星不稳定性状态;以及基于癌症的微卫星不稳定性状态来选择癌症治疗方式。然后,可以使用有效量的所选择的癌症治疗方式来治疗癌症。在一些实施例中,癌症是结肠直肠癌、子宫内膜癌、胆道癌、膀胱癌、乳腺癌、食道癌、胃癌、胃食管连接部癌、胰腺癌、前列腺癌、肾细胞癌、腹膜后腺癌、肉瘤、小细胞肺癌、小肠癌或甲状腺癌。
在一些实施例中,癌症的肿瘤突变负荷(TMB)是使用一个或多个体细胞序列来确定的,该体细胞序列使用本文所述的方法被鉴定为选择治疗方式。TMB是针对癌症的基因组生物标志物,该基因组生物标志物量化患者的肿瘤中体细胞突变的频率。TMB-high与更高的新抗原表达相关,这有助于免疫系统识别肿瘤。它已经在许多肿瘤类型中被检测到,并且与针对接受免疫疗法的患者的缓解率提高和无进展生存期延长有关联。参见Goodman等人,Tumor Mutational Burden as an Independent Predictor of Response toImmunotherapy in Diverse Cancers,Mol.Cancer Ther.,vol.16,no.11,pp.2598-2608(2017)。
可以通过使用本文所述的方法鉴定与癌症相关的体细胞序列来针对癌症确定肿瘤突变负荷。
TMB可以提供定量值,从而可以基于肿瘤突变负荷高于或低于预定肿瘤突变负荷阈值来选择癌症治疗方式。在一些实施例中,预定阈值是约5个突变/Mb、约10个突变/Mb、约15个突变/Mb、约20个突变/Mb、约25个突变/Mb、约30个突变/MB、约40个突变/Mb,约50个突变/Mb或更高或其间的任何数量(例如,预定阈值可以在5个突变/Mb与约50个突变/Mb之间)。举例来说,已经发现某些免疫肿瘤剂在用于治疗具有高肿瘤突变负荷的肿瘤时特别有效。参见例如Fabrizio等人,Beyond microsatellite testing:assessment of tumormutational burden identifies subsets of colorectal cancer who may respond toimmune checkpoint inhibition,J.Gastrointestinal Oncology,vol.9,no.4,pp.610-617(2018)。
在一些实施例中,用有效量的免疫肿瘤剂来治疗被确定具有高于预定阈值的TMB的癌症。在一些实施例中,用有效量的免疫检查点抑制剂来治疗被确定具有高于预定阈值的TMB的癌症。在一些实施例中,免疫检查点抑制剂为AMP-224、AMP-514、阿特朱单抗、AUNP12、阿维单抗、BGB-A317、BMS-986189、CA-170、卡瑞利珠单抗、西米普利单抗、CK-301、多塔利单抗,德瓦鲁单抗、伊匹木单抗、INCMGA00012、KN035、纳武单抗、派姆单抗、信迪利单抗、斯巴达珠单抗、替雷利珠单抗或特瑞普利单抗。在一些实施例中,用有效量的PD-1抑制剂、PD-L1抑制剂或CTLA-4抑制剂来治疗被确定具有高于预定阈值的TMB的癌症。在一些实施例中,用有效量的派姆单抗来治疗被确定具有高于预定阈值的TMB的癌症。在一些实施例中,用有效量的派姆单抗来治疗被确定具有高于预定阈值的TMB的癌症,其中预定阈值为约10个突变/Mb。
在一些实施例中,治疗癌症的方法包括:使用本文所述的方法将一个或多个感兴趣基因组序列鉴定为体细胞;使用一个或多个所鉴定的体细胞序列来确定针对癌症的肿瘤突变负荷;以及基于肿瘤突变负荷高于预定肿瘤突变负荷阈值来选择癌症治疗方式。然后,可以使用有效量的所选择的癌症治疗方式来治疗癌症。在一些实施例中,癌症是结肠直肠癌、子宫内膜癌、胆道癌、膀胱癌、乳腺癌、食道癌、胃癌、胃食管连接部癌、胰腺癌、前列腺癌、肾细胞癌、腹膜后腺癌、肉瘤、小细胞肺癌、小肠癌或甲状腺癌。
监测癌症进展
癌症进展监测和/或微小残留病症检测有利于评估癌症治疗计划和/或针对癌症复发监测患者。癌症患者可以接受癌症治疗,直至无法再检测到癌症。然而,患者可能仍然容易出现复发。可以通过检测源自复发肿瘤的核酸分子(例如,ctDNA分子)来针对癌症复发监测患者。在其他实施例中,癌症患者可以接受疾病治疗,并且可以通过量化患者的所检测到的肿瘤核酸分子的量(例如,ctDNA水平)来监测癌症的进展(例如,癌症量的增加或减少)。
对体细胞序列的鉴定在监测癌症进展或检测癌症的微小残留病灶方面可能特别有用。体细胞序列提供针对癌症的基因组特征,并且它们可以用于区分肿瘤核酸分子和非肿瘤核酸分子。
可以在两个或多个时间点处获得和分析患者样品,以监测癌症进展或癌症复发。根据本文所述的方法分析第一样品以鉴定一个或多个体细胞序列。第一样品可以在癌症治疗之前、期间或之后获得,尽管患者通常具有一定量的可检测癌症。
第二样品可以在患者接受癌症治疗之后的稍后时间点处获得,并且可以被分析以确定样品中是否存在一个或多个所鉴定的体细胞序列。体细胞序列的存在指示患者仍然患有癌症或癌症已经复发。未能检测到体细胞序列并不能明确证明患者没有癌症,但指示癌症水平可能很低。
第二患者样品可以是与第一患者样品类型相同的样品类型或者可以是不同的样品类型。在一些实施例中,第二患者样品从液体活检获得。例如,液体活检患者样品可以是血液、血浆、脑脊髓液、痰、粪便、尿液或唾液。在一些实施例中,患者样品从固体组织样品,诸如实体瘤活检获得。在一些实施例中,固体组织活检样品为新鲜样品。在一些实施例中,固体组织活检样品为冷冻样品或先前冷冻样品。在一些实施例中,固体组织活检样品为新鲜样品。在一些实施例中,固体组织活检样品为保存的样品(例如,化学保存的样品)。在某些实施例中,样品为福尔马林固定的、石蜡包埋的(FFPE)样品。
可以在来自第二样品的DNA或RNA(或两者)中检测体细胞序列。可以通过测序、定量PCR(qPCR)、逆转录PCR(RT-PCR)、荧光原位杂交(FISH)或一个或多个体细胞序列的特异性检测的任何其他合适方法来检测体细胞序列在第二样品中的存在或不存在。在某些实施例中,核酸分子是从第二样品中分离出来的。在一些实施例中,直接从第二样品检测核酸分子。
在一些实施例中,在第二样品中鉴定了一个或多个体细胞序列的存在,患者可以使用对于先前治疗癌症相同的治疗方式或不同的治疗方式来接受癌症治疗。
在一些实施例中,监测患者癌症进展或复发的方法包括:使用本文所述的方法将一个或多个感兴趣基因组序列鉴定为体细胞,其中患者样品从患有癌症的患者获得;在已经治疗癌症之后从患者获取第二患者样品;以及检测第二患者样品内被鉴定为体细胞的一个或多个感兴趣基因组序列的存在或不存在。例如,可以通过从针对患者样品获得的患者基因组序列内在基因组基因座处选择感兴趣基因组序列来将一个或多个感兴趣基因组序列鉴定为体细胞,该患者包括肿瘤核酸分子和非肿瘤核酸分子的混合物;选择针对该感兴趣基因组序列的一个或多个代理基因序列;使用该感兴趣基因组序列的观察到的等位基因频率和指示该一个或多个代理基因组序列的观察到的等位基因频率的汇总统计量来确定等位基因频率距离;以及使用该等位基因频率距离来将该感兴趣基因组序列鉴定为种系或体细胞。在一些实施例中,该方法包括在从患者获得第一患者样品之后和在从患者获得第二患者样品之前治疗患者的癌症。在一些实施例中,该方法包括:如果在第二患者样品中检测到被鉴定为体细胞的一个或多个感兴趣基因组序列的存在,则治疗患者的癌症。
新抗原选择和癌症疫苗生产
在各种基因的外显子区域检测到的体细胞序列可能适合作为新抗原,例如在个性化癌症疫苗的开发中。可以基于由体细胞变体序列编码的核酸序列生成多肽,其可以刺激免疫系统以杀死癌细胞。参见例如Richters等人,Best practices for bioinformaticscharacterization of neoantigens for clinical utility,Genome Medicine,vol.,11no.56(2019)。
在一些实施例中,为针对患有癌症的受试者个性化的癌症疫苗选择新抗原的方法包括:使用本文所述的方法将一个或多个感兴趣基因组序列鉴定为体细胞,其中被鉴定为体细胞的该一个或多个感兴趣基因组序列位于基因的外显子区域内;以及从被鉴定为体细胞的该一个或多个感兴趣基因组序列选择基因组序列,该基因组序列编码适合作为针对受试者的癌症疫苗的新抗原。例如,可以通过从针对患者样品获得的患者基因组序列内在基因组基因座处选择感兴趣基因组序列来将一个或多个感兴趣基因组序列鉴定为体细胞,该患者包括肿瘤核酸分子和非肿瘤核酸分子的混合物;选择针对该感兴趣基因组序列的一个或多个代理基因序列;使用该感兴趣基因组序列的观察到的等位基因频率和指示该一个或多个代理基因组序列的观察到的等位基因频率的汇总统计量来确定等位基因频率距离;以及使用该等位基因频率距离来将该感兴趣基因组序列鉴定为种系或体细胞。
在一些实施例中,该方法进一步包括制备包括新抗原的疫苗。
实例
示例1–基于等位基因频率距离(AFDIS)区分体细胞变体和种系变体
提供以下示例以说明本文所述的本发明的示例性实施例,并且以下示例不旨在限制本发明的范围。
先前描述的SGZ算法(参见例如Sun等人(2018),同上)可以用于确定针对体细胞变体和种系变体(例如,用T替换C的突变)的预期变体等位基因频率的差异,前提是针对样品的肿瘤分数、变体的等位基因计数和基因组基因座的拷贝数被确定,如图5A中绘制的数据。针对体细胞变体和种系变体的预期变体等位基因频率(VAF)可以确定如下:
其中p是肿瘤纯度,V是变体等位基因计数,并且C是等位基因的拷贝数。例如,假设样品的肿瘤纯度(p)为0.25,变体等位基因计数(V)为3,并且拷贝数(C)为4,如果变体是体细胞,则预期等位基因频率为0.3;如果是种系,则预期的等位基因频率为0.6。参见例如Sun等人,A computational approach to distinguish somatic vs.germline origin ofgenomic alterations from deep sequencing of cancer specimens without amatched normal,PLoS Comput Biol.,vol.14,no.2,p.e1005965(2018)。
此示例提供了先前所述的SGZ算法的另选方法,该方法不需要对肿瘤纯度、变体等位基因计数或拷贝数值进行建模。与预期种系等位基因频率(AFDIS)的等位基因频率距离被确定为:
AFDIS=AF
AF
表2
0.1AFDIS的阈值(对应于基于上述ECDF的0.993的累积分布)根据经验被确定能够有效地将体细胞变体与种系变体分离。如表2所示,范围从约0.05到0.1的AFDIS阈值都提供了体细胞变体和种系变体之间的良好区分。然而,如下所述,建立了经训练的统计模型来理解任何给定序列为种系或体细胞的概率。
然后,针对92个基因型匹配的高纯度/低纯度肿瘤样品确定了等位基因频率距离,这些样品具有已知种系序列、体细胞序列和肿瘤纯度。低纯度样品用于建立针对所选择的序列的体细胞/种系状态的基本真值,因为通常低纯度样品被认为是正常样品的近似值,并且允许可靠地确定变体的体细胞状态与种系状态。图5C示出了来自92个肿瘤样品的种系和体细胞序列的变体AFDIS,针对样品计算纯度作图。灰色圆圈表示基本真值体细胞序列,并且黑色圆圈表示基本真值种系序列。
示例2-基于AFDIS的体细胞/种系状态的逻辑回归
使用来自21个匹配的肿瘤/正常对(肺鳞状细胞癌(n=5)、卵巢浆液性癌(n=4)、肺腺癌(n=3)、乳腺浸润性导管癌(n=2)、肛门癌(n=1)、膀胱尿路上皮癌(n=1)、CRC(n=1)、肾透明细胞癌(n=1)、卵巢高级别浆液性癌(n=1)、皮肤肉瘤(n=1)、子宫内膜腺癌(n=1))的可用数据来生成逻辑回归模型。匹配的肿瘤/正常对允许可靠地确定体细胞序列和种系序列。图5D显示了针对此方法的接受者操作特性(ROC)曲线,即,在体细胞变体与种系变体之间进行区分时分类模型的真阳性(TP)和假阳性(FP)性能图。针对模型的“留一法交叉验证”(LOOCV)结果表明准确度为0.97(95%置信区间=[0.95,0.99]),并且科恩(未加权)Kappa统计量为0.93。使用匹配的肿瘤/正常对数据训练模型,以输出给定序列是体细胞序列的概率。对于训练数据中的已知种系序列,该序列为体细胞的概率为0。对于训练数据中的已知体细胞序列,该序列为体细胞的概率为1。根据以下函数,使用训练数据集训练逻辑回归模型:
其中p
将针对三个不同测试集中的总共188个肿瘤样品中的变体按上述方法计算的AFDIS数据输入到训练模型中,以确定每个所选择的序列为体细胞或种系的概率。基于体细胞变体概率,变体序列被标记为体细胞(如果高于体细胞概率阈值)、种系(如果低于种细胞概率阈值)或不明确(即,在体细胞概率阈值与种系概率阈值之间)。参见图5F。
AFDIS分类器对一组93个肿瘤样品的分类结果与在现有SGZ方法验证中使用的匹配正常样品相比,证明了对现有SGZ方法的改进,如图5G所示。93个肿瘤样品的基因组序列是使用混合捕获诱饵集(不同于训练数据集中使用的那些)获得的,表明AFDIS分类器是稳健的,适用于以不同方式收集的基因组数据。表3中总结了该方法的变体级性能(#真阳性(True)、#假阳性(FP)和阳性预测值(PPV))的非限制性示例。
表3
针对该方法的样品级灵敏度性能的数据的非限制性示例在图5H中示出,并且针对阳性预测值(PPV)性能的数据的非限制性示例在图5I中示出。在图5H和图5I所示的“小提琴图”中,图的形状表示纵轴上的值的概率密度。嵌套在小提琴图内的箱线图表示针对纵轴上绘制的参数的中值、第一和第三四分位数、最小值、最大值和离群值。对于该示例的PPV图,大多数样品的PPV为100%,因此压缩了中值、最大值以及第一和第三四分位数指标。
针对对BRCA1和BRCA 2基因中的变体进行分类的数据的非限制性示例在图5J中示出。针对对STK11基因中的变体进行分类的数据的非限制性示例在图5K中示出。如预期,与其他癌症类型相比,发现BRCA1和BRCA2突变在乳腺癌中富含种系起源变体(p=0.025卡方检验),并且与其他癌症类型相比,发现STK11突变在肺癌中富含体细胞起源变体(p=0.0026卡方检验)。
示例3-基于AFDIS的体细胞/种系状态的逻辑回归
所公开的用于区分体细胞变体与种系变体的方法是基于所讨论变体的等位基因频率(AF)与紧邻其基因组位置的已知变体的等位基因频率的比较。在一些情况下,如上所述,种系数据库(例如,公共数据库)中已知种系变体可以用于比较。如果所讨论变体的AF与位于紧邻处的已知种系变体的AF非常相似或非常不同,则可以得出结论,所讨论变体分别很可能或不太可能为种系。
通常,给定变体的AF主要由其拷贝数以及样品的肿瘤分数决定。肿瘤分数对于特定样品是一个常数,因此给定样品中的给定变体的AF主要由其拷贝数决定。这意味着,为了推断变体的体细胞/种系状态,可以将AF与相同拷贝数的种系变体的AF进行比较。下面并在示例4中描述了实现此类比较的两个非限制性示例。
在一种实施方式中,计算“等位基因频率距离”(AFDIS),其表示所讨论变体的AF与位于同一拷贝数片段上(例如,位于同一物理连续的基因组片段上或位于不连续的基因组片段上,只要该片段与所讨论变体具有相同的拷贝数即可)的种系变体的中值AF之间的距离。最初,AFDIS计算如下:
AFDIS=|MAF
其中MAF=次要等位基因频率,即,针对感兴趣变体的次要等位基因频率和针对片段种系变体的次要等位基因频率的中位数用于计算它们的绝对距离。然后,使用由已知体细胞变体和种系变体组成的训练数据集来训练逻辑回归模型,以捕获“体细胞概率”与AFDIS之间的关系。模型随后通过使用带方向的距离进行了改进,即,将AFDIS重新定义为AFDIS=AF
基于AFDIS的方法具有简单和易于计算的优点,因此可以很容易地修改该方法以在给定实施方式中包括其他考虑因素。具体地,由于AFDIS是逻辑回归模型中的单一预测变量,因此可以很容易地调整AFDIS值以修改结果,从而说明其他潜在技术问题。例如,为了说明由核酸样品的轻度污染带来的不确定性增加,可以根据污染水平对AFDIS值进行调整,以将AFDIS值移动到对应于模型对体细胞/种系变体的更准确的分类的范围内。可以进行类似的调整以说明由诸如低读取深度、噪声AF估计、低片段种系SNP计数、片段种系SNP AF的高变异性等因素带来的另外的不确定性。可以使用包括已知体细胞变体和种系变体的训练数据集来设计和调整实现这些调整的程度和方式。
示例4-基于种系等位基因频率概率分布的种系排除
在该特定实施方式中,构建了已知种系变体的大数据集,每个种系变体具有其自己的AF和对应片段MAF,该对应片段MAF为位于相同拷贝数片段中的其他已经种系变体的中位MAF。图6A示出了变体AF与片段MAF的图。对于待分类的未知变体,确定其AF和对应片段MAF。为了对未知变体进行分类,数据取自已知种系数据集,其包括具有与未知变体相似的片段MAF的已知种系变体的子集(例如,图6B所示三种密度与变体AF图中的一者,其分别对应于0.1、0.2和0.3附近的片段MAF处的变体等位基因频率分布,如图6A所示)。使用该数据,可以针对给定片段MAF(即,给定拷贝数,因为片段MAF本质上由片段的拷贝数决定)建立种系AF值的分布。将未知变体的AF与该种系AF分布进行比较,以推断未知变体为种系变体的概率。例如,AF为0.1或0.9且片段MAF为0.1的未知变体可能为种系变体,而AF为0.4且片段MAF为0.1的未知变体可能为体细胞变体。
示例5-性能验证
所公开的方法提供了用于从基线组织或液体活检样品中选择体细胞变体以用于血浆监测的示例性技术。已经设计了一些另外的措施来进一步提高针对该特定目的的性能,包括:i)选择表现良好的变体(例如,通过排除位于已知具有或预期具有偏离期望值的等位基因频率的基因组区域中的变体(诸如位于具有重复序列的区域或与基因组的其他区域共享同源性的区域中的变体))用于构建逻辑回归模型;ii)基于历史数据和公共数据库,结合变体为种系变体、体细胞变体或不确定潜能的克隆性造血(CHIP)变体的可能性的先验知识;以及iii)考虑变体调用及其基因组情景的噪音水平。发现这些措施提高了体细胞变体分类的性能。
使用例如来自匹配的肿瘤/正常对的数据验证了所公开的基于AFDIS的逻辑回归模型区分样品中的体细胞变体与种系变体的能力。表4和表5分别总结了用于开发逻辑回归模型的初始训练和测试数据集以及针对变体级和样品级性能的所得性能指标(#误报(FP)、灵敏度和阳性预测值(PPV))的非限制性示例。
表4.变体级性能数据的非限制性示例
表5.样品级性能数据的非限制性示例
变体调用管道验证研究中使用的数据集包括来自86个匹配的组织/外周血单核细胞(PBMC)样品对的数据。表6和表7分别总结了变体级和样品级的性能度量。
表6.变体级性能的非限制性示例
表7.样品级性能的非限制性示例
在另外的变体调用管道验证研究中使用的数据集包括来自746个匹配的组织/外周血单核细胞(PBMC)样品对的数据。表8和表9分别总结了变体级和样品级的性能度量。
表8.变体级性能的非限制性示例
表9.样品级性能的非限制性示例
应当理解,上述方法和系统是通过示例而非限制的方式要求保护的。许多变化、添加、省略和其他修改对于本领域的普通技术人员将是显而易见的。另外,以上描述和附图中的方法步骤的顺序或呈现并不旨在要求执行所列举步骤的这种顺序,除非明确要求特定顺序或以其他方式从上下文中清楚。
本文所述本发明的方法步骤旨在包括使一个或多个其他方或实体执行这些步骤的任何合适的方法,除非明确提供了不同的含义或以其他方式从上下文中清楚。在一些方面,此类各方或实体无需在任何其他方或实体的指导或控制下,并且无需位于特定管辖范围内。因此,例如,“将第一数加到第二数”的描述或叙述包括使一个或多个方或实体将两个数加在一起。例如,如果人X与人Y进行公平交易,将两个数相加,并且人Y确实将两个数相加,则人X和人Y都执行所述步骤:人Y是因为他实际上将数相加的事实,且人X是因为他让人Y将数相加的事实。此外,如果人X位于美国境内并且人Y位于美国境外,则由于人X参与使该步骤被执行,因此该方法在美国被执行。
本文各种所述实施例的描述中所使用的术语仅出于描述特定实施例的目的,并非旨在进行限制。如在各种所述实施例和附加权利要求的描述中所用,单数形式“一个”、“一种”“该”和“所述”也旨在包括复数形式,除非上下文另外明确地指出。还将理解,本文所用的术语“和/或”是指并涵盖相关联列出的一项或多项的任何和所有可能组合。将进一步理解,当在本说明书中使用术语“包括”和/或“包含”(includes/including/comprises/comprising)时,其指定了所规定的整数、步骤、操作、要素和/或组分的存在,但并不排除存在或添加一个或多个其他整数、步骤、操作、要素、组分和/或它们的组。
本文所引用的所有出版物、专利和专利申请的公开内容均以全文引用的方式并入本文。如果以引用方式并入的任何参考文献与即时公开内容冲突,则以即时公开内容为准。
虽然已经示出并描述了本发明的特定实施例,但在不脱离由以下权利要求书限定的本发明的精神和范围的情况下在形式和细节上做出各种改变和修改对于本领域的技术人员来说显而易见的。以下权利要求书旨在包括可能属于其范围的所有此类变更和修改,并且应在法律允许的最广泛意义上进行解释。
- 一种多鱗鯔鰕虎魚线粒体基因组全序列引物及设计、系统进化分析全序列扩增方法
- 植物基因组改造中常用的在核苷酸序列上修饰植物基因组的方法和工具
- 用于从压缩的基因组序列读段重建基因组参考序列的方法和系统
- 基因组叠阵、基因组架构、基因组序列组装方法及系统
