方法
文献发布时间:2023-06-19 09:24:30
技术领域
本文提供了使用如纳米孔和酶等检测器表征多核苷酸分析物的方法。还提供了可以在所述方法中使用的组合物和系统,所述组合物和系统包含例如用于连接到双链多核苷酸和标签修饰的纳米孔的衔接子。在一些实施例中,本文提供了使用跨膜孔对一种或多种靶多核苷酸进行测序的方法。
背景技术
当前在广泛的应用范围内需要快速且便宜的多核苷酸(例如,DNA或RNA)测序和鉴定技术。
跨膜孔(例如,纳米孔)已用于鉴定小分子或折叠的蛋白质并用于监测单分子水平的化学或酶促反应。跨膜孔(例如,纳米孔)作为聚合物和各种小分子的直接、电生物传感器具有巨大的潜力。具体来说,最近正在关注纳米孔作为潜在的DNA测序技术和生物标志物识别。
可以在跨纳米孔施加的电位差下测量通过纳米孔的离子流。分析物与纳米孔的相互作用可以引起离子流的特征改变,并且对所产生的信号的测量可以用于表征分析物。例如,所测量的信号可以是当前的,并且可以例如用于确定多核苷酸的序列。可以使多核苷酸链通过孔易位,并且可以从所测量的信号得出核苷酸的如序列的同一性。此类测序方法公开于例如 WO0142782、WO2016034591、WO2013041878、WO2014064443和WO2013153359。
已经开发了用于为双链多核苷酸测序的方法,例如涉及通过发夹连接的模板和补体链的易位。链测序通常涉及使用多核苷酸结合蛋白(如解旋酶)来控制多核苷酸通过纳米孔的运动。此类方法公开于例如WO2013057495中。纳米孔的尺寸可以使其仅允许单链多核苷酸易位。双链多核苷酸可以通过在易位通过纳米孔之前将链分离以提供单链多核苷酸来确定。多核苷酸结合蛋白如解旋酶可用于同时分离双链多核苷酸并控制所得单链易位通过纳米孔的速率。双链多核苷酸的两条链可以通过桥接部分如发夹环连接,并且制备此类构建体的方法描述于例如WO2013057495中。这确保在正向(模板)链易位之后反向(互补)链易位。以这种方式测量两条链是有利的,因为来自两条相连补体链的信息可以组合并用于提供比仅测量模板链可以获得的置信度更高的观察结果。然而,此类发夹连接的多核苷酸的制备会增加样品制备时间并导致有价值的分析物的损失。此外,发夹连接的模板和互补多核苷酸链易位通过纳米孔可以引起纳米孔的另一(反)侧上的链的再杂交。这可以改变易位速率,从而降低测序精确度。此外,由于模板链和补体链的电流-时间数据的差异,使用两种算法进行计算,这使得计算更加复杂和密集。
因此,需要改进的表征分析物(例如双链多核苷酸)的方法,其具有提高的精确度和更高的效率/通量。
发明内容
在其最广泛的方面,本公开涉及使用酶来处理传感器/检测器附近的多核苷酸分析物的任何感测方法。更具体地,本公开涉及双链多核苷酸分析物的两条链的顺序处理,而不需要通过桥接部分(如发夹环)将两条链共价连接。在所描述的方法中,双链多核苷酸分析物的第一链紧邻检测器进行测序,使得处理酶可以起到处理分析物的作用。当第一链被测序时,双链多核苷酸的两条链被部分分离,并且双链多核苷酸分析物的第二链将定位于检测器。具体地,双链多核苷酸的第二链通过杂交标签定位于检测器。在第一链紧邻检测器进行测序之后,然后第二链紧邻检测器进行测序。可以通过任何方法,例如,涉及与检测器直接相互作用的方法,如链测序方法或核酸外切酶方法,来实现分析物的两条链的顺序处理。替代性地或另外地,分析物的顺序处理可以涉及检测聚合酶反应的副产物。
本公开总体上涉及用于使用检测器(例如,纳米孔)和可以在本文所描述的方法中使用的组合物(例如,衔接子和纳米孔)表征多核苷酸分析物的方法。本公开部分基于出乎意料的发现,即,双链多核苷酸的两条链都可以通过例如聚合酶等酶依次处理,并且检测到这种处理的副产物,例如,通过纳米孔易位以提供序列信息,而无需通过桥接部分(如发夹环) 共价连接两条链。例如,在一些实施例中,可以向双链多核苷酸的每个端提供具有双链体茎的衔接子,所述双链体茎包括与孔标签互补的捕获序列,所述孔标签与检测器(例如,纳米孔)缀合,其中捕获序列仅在解链时才显露。因此,在处理双链多核苷酸的第一链时,将衔接子的双链体茎解链,以使捕获序列暴露在双链多核苷酸的第二链上,然后由检测器的孔标签捕获。这种方法不仅使第二链保持靠近检测器,还缩短了处理第一链和第二链之间的时间延迟,由此提高了测序方法的总体准确度和效率。还发现随后处理的在纳米孔处捕获的多种分析物可以增强表征这些分析物的灵敏度和/或通量。
本发明人还发现,当使用例如聚合酶等酶来处理双链多核苷酸的两条链中的一条链时,第二链可以保留在孔附近,并且在通过孔处理第一链之后,第二链可以通过孔捕获,并且聚合酶可以用于处理第二链。
因此,本发明的一个方面提供了一种测序靶多核苷酸的方法,所述方法包括:
(a)使跨膜孔与以下接触:
(i)双链多核苷酸,所述双链多核苷酸包括所述靶多核苷酸和与所述靶多核苷酸互补的多核苷酸;以及
(ii)至少一种能够处理所述双链多核苷酸的链的聚合酶;
其中与所述双链多核苷酸的一部分结合的至少一个标签与所述跨膜孔缀合;
(b)检测与通过所述孔的离子流相对应的信号,以检测处理反应的副产物通过所述孔的易位;
(c)鉴定与通过所述聚合酶处理所述靶多核苷酸所得的副产物的易位相对应的信号和与通过所述聚合酶处理同所述靶多核苷酸互补的所述多核苷酸所得的副产物的单独转译相对应的顺序信号;
(d)分析(c)中鉴定的信号,
由此对所述靶多核苷酸进行测序。
在此方面,双链条形码序列可以连接到靶双链多核苷酸的一个或两个端,前导序列可以包括在衔接子中,衔接子可以包括双链区和至少一个单链区,衔接子可以包括双链条形码序列,衔接子可以包含膜-拴或孔-拴,连接到靶双链多核苷酸的两个端的前导序列可以不同,双链多核苷酸在其每个端处可以具有不同的衔接子和/或聚合酶可以结合到前导序列。在聚合酶与前导序列结合的情况下,聚合酶的活性可能会停滞,直到多核苷酸接触跨膜孔。在双链条形码序列连接到靶双链多核苷酸的一个或两个端时,独特的条形码序列可以连接到样品中的每个双链多核苷酸。在这方面,双链多核苷酸可以连接到微粒和/或可以修饰孔以增强多核苷酸的捕获。例如,吸引或结合多核苷酸或衔接子的一个或多个分子可以与孔连接。此类分子可选自,例如,PNA标签、PEG接头、短寡核苷酸、带正电荷的氨基酸和适体。在这方面,跨膜孔可以是,例如,蛋白质孔,如源自或基于Msp、α-溶血素(α-HL)、胞溶素、CsgG、 ClyA、Sp1或FraC的孔,或固态孔和/或膜可以是两亲性层或固态层。
该方法优于为双链多核苷酸测序的已知方法,其中两条链使用桥接部分如发夹环连接。该方法也优于仅测量模板多核苷酸链的已知方法。具体而言,本发明的方法结合了仅模板链的方法和发夹环方法的优点,而没有所提到的发夹环方法的缺点。
例如,WO2013/014451中公开的方法使用多个衔接子,并且样品中仅一些双链多核苷酸将在一端添加Y衔接子,在另一端添加包含桥接部分的衔接子,丢弃样品中的其它多核苷酸。本发明的方法可以使用可以添加到双链多核苷酸的两端的单个前导序列或衔接子来进行。当使用此类单个前导序列/衔接子的系统时,需要丢弃较少的样品(如果有的话)。
在该方法中,双链靶多核苷酸的任一末端均可被孔捕获。与WO2013/014451中公开的方法相比,这提高了灵敏度,其中只有不包括桥接部分的双链多核苷酸的末端可以被孔捕获。
本文还描述了衔接子群体,其包括双链条形码序列、单链前导序列和能够分离双链多核苷酸的链并控制多核苷酸通过跨膜孔的移动的多核苷酸结合蛋白,其中群体中每个衔接子的条形码序列是唯一的。
本文提供的另一方面涉及一种表征多核苷酸的方法。所述多核苷酸可以包括DNA或 RNA。所述方法包括:
(i)将以下组合在溶液中:
(a)构建体,所述构建体包括具有模板链和补体链的双链多核苷酸,其中所述模板链和所述补体链不共价连接,与
(b)检测器,其中与所述构建体的一部分结合的至少一个标签与所述检测器缀合;以及
(c)聚合酶和核苷酸;
以及
(ii)提供一种条件,以允许所述构建体的所述模板链通过所述聚合酶进行处理并且处理反应的产物和/或副产物由所述检测器检测,由此检测通过所述聚合酶向多核苷酸链添加核苷酸;
其中在所述双链多核苷酸的所述模板链被处理时,所述补体链将通过与所述检测器缀合的所述至少一个标签与所述检测器结合。
在一些实施例中,检测所述处理反应的所述产物和/或所述副产物涉及测量指示所述处理反应的所述产物和/或所述副产物的性质;并且所述多核苷酸基于所述处理反应的所述产物和/ 或所述副产物的所测量性质来表征。
在一些实施例中,在所述构建体的所述模板链通过所述聚合酶处理之后,所述聚合酶从所述模板链中解离。在一些实施例中,在所述模板链通过所述聚合酶处理之后,所述构建体的所述补体链通过聚合酶处理,并且所述处理反应的所述产物和/或所述副产物由纳米孔检测。
在一些实施例中,表征所述多核苷酸包括检测所述多核苷酸的核苷酸序列。在一些实施例中,所述多核苷酸的所述核苷酸序列基于由所述检测器检测所述处理反应的所述产物和/或所述副产物的顺序确定。在一些实施例中,所述溶液中的所述核苷酸被标记。在一些实施例中,所述溶液中的每种类型的核苷酸根据核苷酸的类型进行可区分地标记。在一些实施例中,所述溶液中的所述核苷酸用光学标记物和/或聚合物标签标记。在一些实施例中,所述聚合物标签是带电聚合物标签。
在一些实施例中,所述方法包括:
(i)将以下组合在溶液中:
a)构建体,所述构建体包括具有模板链和补体链的双链多核苷酸,其中所述模板链和所述补体链不共价连接,与
b)检测器,其中与所述构建体的一部分结合的至少一个标签与所述检测器缀合;并且
c)聚合酶和核苷酸;
并且
(ii)提供一种条件,以允许所述构建体的所述模板链通过所述聚合酶进行处理并且处理反应的副产物由所述检测器检测,由此检测通过所述聚合酶向多核苷酸链添加核苷酸;
其中在所述双链多核苷酸的所述模板链被处理时,所述补体链将通过与所述检测器缀合的所述至少一个标签与所述检测器结合。
在一些实施例中,当核苷酸通过聚合酶依次添加到所述多核苷酸链中时,所述一个或多个处理反应的所述产物和/或所述副产物依次释放。在一些实施例中,所述一个或多个处理反应的所述副产物是标记的磷酸盐物质。在一些实施例中,所述一个或多个处理反应的所述副产物根据通过所述聚合酶添加到所述多核苷酸链的核苷酸的类型进行可区分地标记。在一些实施例中,所述一个或多个处理反应的所述副产物用光学标记物和/或聚合物标签标记。在一些实施例中,所述聚合物标签是带电聚合物标签。
在一些实施例中,所述方法包括检测通过所述聚合酶向核酸链依次添加多核苷酸所得的产物。在一些实施例中,通过所述聚合酶向所述核酸链依次添加多核苷酸所得的所述产物是所述聚合酶的一种或多种性质的改变。在一些实施例中,通过所述聚合酶向所述核酸链依次添加多核苷酸所得的所述产物是所述聚合酶的构象的改变。在一些实施例中,所述方法的步骤(ii)包括通过所述聚合酶处理所述构建体的所述模板链以及由所述检测器检测所述处理反应的产物,由此检测通过所述聚合酶向所述多核苷酸链添加核苷酸;其中通过所述聚合酶向所述多核苷酸链依次添加一种或多种核苷酸导致所述聚合酶的构象的改变,并且其中所述处理反应的检测到的产物是构象改变的聚合酶。
在一些实施例中,所述检测器选自以下:(i)零模波导;(ii)场效应晶体管,任选地纳米线场效应晶体管;(iii)AFM尖端;(iv)纳米管,任选地碳纳米管以及(v)纳米孔。在一些实施例中,所述检测器是纳米孔。在一些实施例中,所述纳米孔也起到对多核苷酸进行解链的作用。在一些实施例中,所述纳米孔是马达蛋白纳米孔,任选地是phi29。
在一些实施例中,所述聚合酶也起到对多核苷酸进行解链的作用。换句话说,所述聚合酶可以具有链置换活性。
在一些实施例中,在所述构建体通过所述聚合酶进行处理之前,将衔接子连接到所述双链多核苷酸的两个端中的一个或两个。在一些实施例中,在所述构建体通过所述聚合酶进行处理之前,将衔接子连接到所述双链多核苷酸的两个端中的每个端。
在一些实施例中,每个衔接子包括双链体茎和从所述双链体茎延伸的第一单链,其中一个衔接子的第一单链与所述模板链邻接,并且另一个衔接子的第一单链与所述补体链邻接。在一些实施例中,每个衔接子包括从所述双链体茎延伸的第二单链,其中所述一个衔接子的第二单链与所述补体链邻接和/或所述另一个衔接子的第二单链与所述模板链邻接。
在一些实施例中,所述衔接子或每个衔接子包括聚合酶。在一些实施例中,所述聚合酶预先结合到所述衔接子或每个衔接子。
在一些实施例中,所述检测器是纳米孔,并且所述聚合酶定位在紧邻所述纳米孔的桶或通道的开口的位点处。所述聚合酶提供在所述纳米孔的内腔内。在一些实施例中,所述聚合酶的活性位点朝向所述纳米孔的所述开口取向。
在一些实施例中,所述检测器是纳米孔,并且与所述构建体的一部分结合的至少一个标签与所述纳米孔的外缘缀合。在一些实施例中,与所述衔接子的一部分结合的一个或多个标签与所述纳米孔缀合。在一些实施例中,与所述衔接子的一部分结合的一个或多个标签与所述纳米孔的所述外缘缀合。
在一些实施例中,与所述构建体的一部分结合的所述一个或多个标签中的至少一个标签是与所述构建体的所述部分具有序列互补性的核酸。在一些实施例中,与所述衔接子的一部分结合的所述一个或多个标签中的至少一个标签是与所述衔接子的所述部分具有序列互补性的核酸。核酸可以是不带电荷的,包含例如但不限于PNA或吗啉代。
在一些实施例中,通过所述聚合酶对所述模板链的处理显露了所述补体链的用于与标签杂交的一部分。
在一些实施例中,包括双链体茎和从所述双链体茎延伸的第一单链的衔接子连接到所述双链多核苷酸的至少一个端,使得所述衔接子的所述第一单链与所述补体链邻接;并且所述条件维持足够的时间以允许将所述构建体的所述模板链处理到一定程度,以使所述衔接子的具有与所述补体链邻接的其第一单链的所述一部分可用于与标签杂交。
在一些实施例中,所述方法包括使所述条件维持足够的时间以允许所述补体链通过聚合酶进行处理,并且由此在处理和表征所述模板链后进行表征。
在一些实施例中,所述检测器是纳米孔,并且所述纳米孔包括第一标签和第二标签,并且所述第一标签和所述第二标签分别结合到所述衔接子的所述第一单链的与所述模板链邻接的部分以及所述衔接子的所述第一单链的与所述补体链邻接的部分。
在一些实施例中,所述检测器是纳米孔,并且所述条件是跨所述纳米孔的电位差。在一些实施例中,步骤(ii)包括跨所述纳米孔施加电位差,以允许所述处理反应的所述副产物进入到所述纳米孔;并且所述电位差跨所述纳米孔维持足够的时间段,以允许所述处理反应的所述副产物的至少一部分通过所述纳米孔易位。
在一些实施例中,所述检测器是纳米孔,并且所述溶液是离子的,并且所测量性质是流过所述纳米孔的离子电流。所述方法因此可以包括在所述处理反应的所述副产物通过所述纳米孔易位时,测量流过所述纳米孔的离子电流的改变。所述多核苷酸可以基于在所述处理反应的所述副产物通过所述纳米孔易位时而测量的流过所述纳米孔的所述离子电流的改变来表征。可以获得指示所测量性质的数据,所测量性质指示处理所述双链多核苷酸的所述模板链和所述补体链所得的产物和/或副产物,并将所述数据用于表征所述多核苷酸。可以将模板链数据与补体链数据进行比较或组合,以表征所述多核苷酸。
在一些实施例中,所述多核苷酸包括RNA和/或DNA。
在一些实施例中,所述方法进一步包括:
基于测量的指示通过所述聚合酶处理所述模板链所得的所述副产物的性质改变,确定所述模板链的序列,
基于测量的指示通过聚合酶处理所述补体链所得的所述副产物的性质改变,确定所述补体链的序列,以及
将所述模板链的所述序列与所述补体链的所述序列进行比较以建立所述多核苷酸的序列。
还提供了一种用于表征多核苷酸的系统,例如可以在本文所描述的方法的任何方面中使用的系统。所述系统包括:(i)构建体,所述构建体包括具有模板链和补体链的多核苷酸,其中所述模板链和所述补体链不共价连接;(ii)检测器,其中与所述构建体的一部分结合的至少一个标签与所述检测器缀合;以及(iii)聚合酶和核苷酸。所述检测器可以是安置于膜中的纳米孔,其中与所述构建体的一部分结合的至少一个标签与所述纳米孔缀合。
在一些实施例中,衔接子连接到所述多核苷酸的两个端,每个衔接子包括双链体茎和从所述双链体茎延伸的第一单链。一个衔接子的第一单链可以与所述模板链邻接,并且另一个衔接子的第一单链可以与所述补体链邻接。对于每个衔接子,聚合酶可以与从所述双链体茎延伸的所述第一单链结合。
在一些实施例中,每个衔接子可以包括从所述双链体茎延伸的第二单链,其中一个衔接子的第二单链与所述补体链邻接并且另一个衔接子的第二单链与所述模板链邻接。
在一些实施例中,与所述纳米孔缀合的所述至少一个标签(a)与所述衔接子的在所述双链体茎内位于与所述第一单链邻接的链上的一部分具有序列互补性,并且(b)与所述衔接子的位于所述第二单链内的一部分具有另外的序列互补性。
在一些实施例中,所述检测器是纳米孔,并且至少两个标签与所述纳米孔缀合,其中所述至少两个标签中的一个标签与所述衔接子的在所述双链体茎内位于与所述第一单链邻接的链上的一部分具有序列互补性,并且其中所述至少两个标签中的另一个标签与所述衔接子的位于所述第二单链内的一部分具有序列互补性。在一些实施例中,所述至少两个标签可以与所述纳米孔的外缘缀合。
另外的方面涉及一种用于制备用于表征多核苷酸的系统的方法。所述方法包括:(i)获得构建体,所述构建体包括具有模板链和补体链的多核苷酸,其中所述模板链和所述补体链不共价连接;以及(ii)将所述构建体与(a)安置于膜中的纳米孔和(b)聚合酶和核苷酸组合;在将所述构建体暴露于所述纳米孔的外缘的条件下,其中将与所述构建体的一部分具有序列互补性的至少一个标签与所述纳米孔的所述外缘缀合。
在一些实施例中,衔接子可以连接到所述多核苷酸的两个端中的每个端,每个衔接子包括双链体茎和从所述双链体茎延伸的第一单链,其中一个衔接子的第一单链与所述模板链邻接,并且另一个衔接子的第一单链与所述补体链邻接。对于每个衔接子,聚合酶可以与从所述双链体茎延伸的所述第一单链结合。
在本文所描述的方法的任何方面中形成的包括两种或更多种组分的复合物也在本公开的范围内。在一些实施例中,复合物包括:(i)纳米孔,所述纳米孔具有标签,(ii)补体多核苷酸链,所述补体多核苷酸链通过所述标签与所述纳米孔结合,以及(iii)模板多核苷酸链,所述模板多核苷酸链与所述补体多核苷酸链部分杂交,其中通过聚合酶处理所述模板多核苷酸所得的至少一种副产物安置于所述纳米孔的内腔内。在本文所描述的复合物的任何实施例中,所述标签在其内腔外部的外缘处。
在另一方面,一种用于使用纳米孔确定多核苷酸的特性的方法,所述方法包括:(i)提供所述多核苷酸;(ii)使所述多核苷酸与标签结合,所述标签与所述纳米孔的外缘缀合,所述外缘位于所述纳米孔的内腔外部,以及(iii)在使副产物相对于所述纳米孔移动的同时,获得通过聚合酶处理所述多核苷酸所得的副产物的测量结果,其中所述测量结果指示所述多核苷酸的一种或多种特性;以及(iv)基于在步骤(iii)中获得的所述测量结果表征所述多核苷酸。
还提供了一种用于通过纳米孔依次检测两个非共价结合的分子的方法。所述方法包括:在促进通过聚合酶处理一对非共价结合的分子的第一成员并通过纳米孔检测处理反应的副产物的条件下,使所述一对非共价结合的分子与所述纳米孔接触,其中在通过所述聚合酶处理所述一对的所述第一成员期间暴露所述一对的第二成员上的结合位点,并且其中所述结合位点与存在于所述纳米孔上的标签可逆地结合。
在一些实施例中,所述非共价结合的分子是互补核酸链。在一些实施例中,所述一对非共价结合的分子可以包括连接到衔接子的靶核酸,并且所述结合位点可以存在于所述衔接子上。
在一些实施例中,所述纳米孔上的所述标签可以是寡核苷酸,并且所述第二成员上的所述结合位点可以是核酸的具有与所述标签互补的序列的一部分。
本文还提供了一种表征多核苷酸的方法,所述方法包括:在以下条件下,使一对非共价结合的分子与纳米孔接触:促进通过聚合酶处理所述一对非共价结合的分子的第一成员以及通过所述纳米孔下检测处理反应的副产物,然后依次促进通过聚合酶处理所述一对非共价结合的分子中的第二成员以及通过所述纳米孔检测处理反应的副产物;测量指示所述处理反应的所述副产物的性质,以及获得指示所测量性质的数据;以及基于获得的所述第一成员和所述第二成员两者的数据确定特性。
本文还提供了一种表征多核苷酸的方法,所述方法包括:
(i)将以下组合在溶液中:
a)构建体,所述构建体包括具有模板链和补体链的双链多核苷酸,其中所述模板链和所述补体链不共价连接,与
b)检测器,其中与所述构建体的一部分结合的至少一个标签与所述检测器缀合;并且
(c)核酸外切酶;
以及
(ii)提供条件以允许所述构建体的所述模板链通过所述核酸外切酶处理,使得所述核酸外切酶从所述构建体的一端消化单个核苷酸,并且所述单个核苷酸与所述检测器相互作用;
(iii)在所述构建体的相同端处重复步骤(ii),由此确定所述构建体的序列;
其中在所述双链多核苷酸的所述模板链被处理时,所述补体链将通过与所述检测器缀合的所述至少一个标签与所述检测器结合。
附图说明
以下附图形成本说明书的一部分,并且被包含以进一步说明本公开的某些方面,通过参考这些附图中的一个或多个并结合本文呈现的具体实施例的具体实施方式,可以更好地理解所述本公开的某些方面。
仅出于说明目的,本文所述的图中的链根据其捕获末端标记为“模板”和“互补序列”。穿过纳米孔的第一链标记为模板,在第一链之后的补体链标记为互补序列。在分析从第一链和第二链获得的序列信息后,确定双链多核苷酸的实际模板和互补序列。
图1A-1B说明了使用跨膜孔为双链多核苷酸(例如,DNA)构建体测序的现有技术方法,其中模板和补体链通过发夹环连接并且模板链包含5'前导序列。图1A是多核苷酸(例如, DNA)构建体在酶的控制下易位通过纳米孔的示意图。模板进入纳米孔并且相同的酶在发夹周围前进以控制模板后面的互补序列的运动。一旦发夹区易位通过纳米孔,发夹就可在纳米孔的反侧上重新形成。图1B示出了表示从模板的易位、从补体的易位获得的序列信息以及在从模板的易位和补体的易位获得的序列信息通过算法组合时的准确度。
图2A-2B示出了根据本文所公开的一种方法在不使用发夹的情况下对双链多核苷酸(例如,DNA)构建体进行“后续”测序的方法。模板多核苷酸链和补体多核苷酸(例如,DNA)链两者在每个端均包括衔接子,所述衔接子包含前导序列。图2A是双链多核苷酸(例如,DNA)构建体在酶控制下易位通过纳米孔的示意图。双链多核苷酸的模板和互补序列不共价连接,并且每条链具有负载在衔接子上的酶。在模板链穿过纳米孔(并且酶解离)后,通过孔分别捕获补体链并测序。在不存在将模板连接到互补序列的发夹的情况下,在纳米孔的反侧上几乎没有或没有形成二级发夹结构。图2B显示了表示从模板易位,从互补序列易位以及将从模板易位和互补序列易位获得的序列信息在算法上组合时获得的序列信息的准确度的峰。
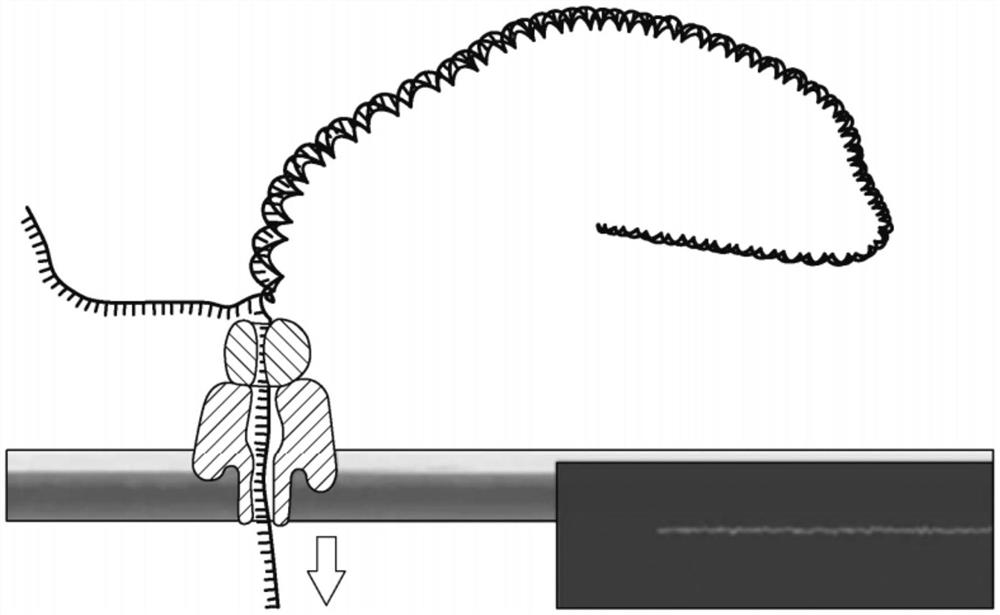
图3A-3B说明了根据本文所述的一个实施例的载酶衔接子的结构。图3A是负载酶的衔接子的示意图。标记物表示以下:(1)间隔子(例如,前导序列);(2)多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶);(3)间隔子;以及(4)锚,如胆固醇锚。其它实线表示多核苷酸序列。图3B示出了连接到双链多核苷酸(如基因组DNA的片段)的每个端的衔接子,其中多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶)负载在每个衔接子上。
图4是在多核苷酸链通过纳米孔易位期间随时间测量的电流信号的示意性图示。
图5是双链多核苷酸在纳米孔界面处分离和随后单链多核苷酸易位通过纳米孔的图示。
图6是在为多核苷酸测序期间,图4的电流时间信号的一部分的事件检测的图示。
图7是使用递归神经网络(RNN)模型分析信号测量的示意图。
图8是如何采用维特比算法(Viterbi algorithm)来确定通过具有最高似然性的可能转变的路径的图示。
图9A-9B说明了根据本文所述的一个实施例的载酶衔接子的结构。图9A是负载酶的衔接子的示意图。标记物表示以下:(1)间隔子(例如,前导序列);(2)多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶);(3)间隔子;(4)锚,如胆固醇锚,其是任选的;以及(5)定位在(1)间隔子(例如,前导序列)的相反端上的双链体茎,双链体茎在与(1) 间隔子(例如,前导序列)对准的链上包括捕获序列,其中所述捕获序列与标签(例如,捕获多核苷酸)互补,所述标签与纳米孔的外缘缀合。其它实线表示多核苷酸序列。图9B示出了当衔接子连接到双链多核苷酸的每个端时的构建体,其中多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶)负载在每个衔接子上。
图10(图A-D)展示了根据本文所公开的一种方法使用纳米孔对双链多核苷酸进行测序的方法的示意图。所述方法涉及提供(i)双链多核苷酸,所述双链多核苷酸的每个端连接到衔接子(例如,如图9A所展示的,没有锚(4))以及负载在衔接子上的多核苷酸结合蛋白(例如,多核苷酸解链酶),以及(ii)纳米孔,所述纳米孔具有与纳米孔的外缘缀合的捕获多核苷酸。通过将衔接子的连接到第二链的捕获序列结合到与纳米孔的外缘缀合的标签(例如,捕获多核苷酸),将双链多核苷酸的第二链(补体)偶联到纳米孔。
图11A-11B示出了根据本文所公开的一种方法使用方法获取的链数据的实例部分。链数据示出了单通道的电数据的电流(pA)对时间(秒)的关系。图11A显示了链数据的实例部分,其显示没有阻断电流的链的开孔水平为约200pA。当链被捕获时,电流降低至50-100pA 范围,这取决于序列组成。当链完全穿过孔时,电流恢复到200pA的开孔水平。对于成对的模板链和补体链而言,将单独的链标记为T
图12A-12D示出了使用根据本文所公开的一种方法获取的链数据的实例部分。链数据示出了MinION芯片上单通道的电数据的电流(pA)与时间(秒)的关系。图12A显示了跟随模板-互补序列对的另一示例电迹线。图12B、图12C和图12D显示了图12A中的迹线的放大图,其中星形标记双链体茎中的sp18间隔区(例如,互补标签部分),其被添加到链中以使得与孔-标签(例如,与纳米孔的外缘缀合的捕获多核苷酸)的偶联成为可能。图12B显示了模板链起始处的sp18。图12C显示了模板末端和互补序列起始处的sp18,并且图12D显示了互补序列末端的sp18。这些标志可用于证明dsDNA底物具有连接到dsDNA两端的酶-衔接子,并测量连接效率。
图13显示了当连续链穿过孔(从MinION芯片上的所有通道聚集)时,随后的链(x轴) 之间的开孔时间分布的直方图。上图,对照衔接子(如实例2中所述)显示,在可以与孔标签(例如,捕获核苷酸)偶联的多核苷酸构建体中没有捕获序列的情况下进行测序时,链间的时间分布平均大约为3秒。下图,跟随衔接子2(如实例4中所述)显示,当链在互补序列中含有可以与孔标签偶联的捕获序列(例如,捕获多核苷酸)时,观察到大约50毫秒的新群体。50ms的短群体来自其模板对后不久快速捕获的补体链。捕获很快,因为补体链通过互补序列与孔标签的结合保持与孔非常靠近,因此不允许其扩散。
图14显示了从模板易位,从互补序列易位以及将从模板易位和互补序列易位获得的序列信息在算法上组合时获得的序列信息(随机片段化大肠杆菌)的碱基识别(basecall)的准确度分布的直方图。使用如图10所示的方法获得序列信息。
图15显示了具有两种或更多种类型标签的纳米孔的示例性实施例。例如,可以提供一种标签以增加表征分析物的方法的灵敏度(“灵敏度标签”),同时可以提供另一种标签以增加对双链多核苷酸的模板链之后的补体链测序的可能性(“跟随标签”)。孔标签可以以多种方式构造。例如,寡聚孔的每个单体可以具有相同类型的标签构型(例如,具有多个结合位点,如Tag-A和Tag-B所示)。Tag-A和Tag-B可以组合形成单个标签,每个单体包含Tag-A/Tag-B组合标签。可替代地,寡聚孔可包含有不同标签连接的混合单体,使得至少一种单体具有与其它单体不同的标签构型。在另一个实例中,Tag-A和Tag-B可以保持为单独的标签,并且每个单体可以包括两种单独的标签。如果灵敏度标签和跟随标签与衔接子中使用的独特序列互补,则可以单独组合,如衔接子原理图设计的下图所示。
图16是可如何使用具有两种不同标签类型的纳米孔从溶液中捕获链(以便提高灵敏度) 的示意图。连接到双链多核苷酸末端的衔接子包含可用于偶联至第一孔标签的捕获序列(例如,形成Y-衔接子的非互补臂),同时双链体茎内只有在被拉开时才显露的单独捕获序列允许互补序列结合第二孔标签,因此使互补序列捕获成为可能以进行跟随测序。
图17A是可以如何在衔接子的两个位置使用相同捕获序列的示意图,一个显露允许链从溶液中结合纳米孔的孔标签以提高灵敏度,另一个最初未显露并且是在模板拉开通过孔隙时显露,变得可以与孔(仅具有一种类型的标签的孔)上的多个标签中的另一个结合,以实现跟随测序。图17B提供了可用于此目的一些示例序列。顶部构建体显示了示例Y衔接子的一部分。“FO001/FO002”和“FO003/FO004”序列是双链体茎的实例,其可以连接到示例Y衔接子上产生单个衔接子构建体,其可以实现根据本文所述的一个或多个实施例的方法。“FO001/FO002”和“FO003/FO004”序列中的浅蓝色序列具有与紫色序列相同的序列,紫色序列是纳米孔的结合序列位点。纳米孔的相同结合序列位点可以在衔接子的双链体茎内使用不止一次(例如,两次),其中浅蓝色序列未暴露。
图18显示了衔接子设计的示意图,该设计可实现跟随测序和增加灵敏度。孔结合序列(在图18中标记为“吗啉代孔标签的Hyb夹板”)暴露于周围溶液中,并且最初可用于与孔标签结合,因此提高灵敏度。当连接时,孔结合位点也与补体链邻接,使得当模板链已经通过孔或已经处理时,补体链保持结合到孔。此过程如图19示意性示出。
图19是显示双链多核苷酸的示意图,其中图18的衔接子连接到每个末端。链通过暴露的孔结合位点从溶液中偶联到纳米孔,从而提高随后捕获附近模板链的灵敏度。结合位点也与补体链邻接,以致当模板已经穿过纳米孔时,互补序列保持与纳米孔结合。互补序列可以在最终捕获和测序之进入许多可能的构象,如下所示,以实现跟随测序。在图19中,分别连接到待检测链的两端的绿色和黄色互补区段可以结合在一起形成包含所述链的发夹结构,通过使链更靠近纳米孔来促进测序过程以增加测序效率。当要检测的链是长链时,这将特别有益处。
图20示出了衔接子设计的示意性图示,其中双链体茎内的相同序列(绿色)在衔接子的不同位置处重复,如所示出的,以启用后续方法,例如,如图21所示。
图21是示出了具有连接到每个端的图20的衔接子的双链多核苷酸的示意性图示。如图 20所述,dsDNA链通过侧臂上连接到衔接子的结合位点与孔结合。当模板被捕获在孔中时,侧臂序列被拉开并保持与孔标签结合,如图所示。后来进入模板拉开,具有相同序列的第二位点显露以与侧臂结合(本身仍然与孔标签结合)。以这种方式,孔上的单个标签可以用于提高捕获灵敏度,并且可以重复用于稍后实现底物的互补序列的跟随。最后,孔标签保留了侧臂序列,但是侧臂本身被孔捕获并从孔标签中剥离以释放孔标签用于另一个循环。
图22显示当模板-酶接近模板链的末端时,显露的序列如何暴露以与孔标签偶联。跟随过程的效率可以增加,例如,通过包括间隔区(例如,所示序列中的4个sp18间隔区,例如六乙二醇)或使酶短暂停留的类似特征(这允许更多时间进行偶联),或具有优化几何特性或灵活性的特征。显露部分中的双重结合位点也提高了与孔标签偶联的机会。
图23提供了可以实现图21中公开的方法的示例衔接子/序列。
图24提供了可以实现图21中公开的方法,并且在使酶暂停方面更加优化的其它衔接子/ 序列。
图25提供了可以实现图19中公开的方法的示例衔接子/序列。
图26提供了构成上图中描述的衔接子的组分的示例序列。
图27A显示SYPRO Ruby蛋白凝胶,其显示出经或未经吗啉代孔标签修饰的CsgG的单体和寡聚纳米孔。图27B显示经吡啶基-二硫代吗啉基修饰的纳米孔的示意图。
图28显示Cy3荧光凝胶,其显示分析物与吡啶基-二硫代吗啉基修饰的孔的杂交。
图29显示SYBR金核酸凝胶染剂,其显示分析物与吡啶基-二硫代吗啉基修饰的孔的杂交。
图30显示SYPRO Ruby蛋白凝胶,其显示分析物与吡啶基-二硫代吗啉基修饰的孔的杂交。
图31说明了显示纳米孔(例如,CsgG纳米孔)的计算机渲染的图,突出显示了可以添加半胱氨酸以与孔标签缀合的位置。当纳米孔置于膜中时,孔标签可以与纳米孔的外表面缀合,例如,在膜的顺侧或反侧。
图32A显示了Y衔接子设计的一个实施例,其包括两个杂交位点,一个用于孔系链(红色),另一个用于膜或珠粒系链(蓝色)。在此设计中,孔系链紧挨着前导序列。图32B示出了连接的分析物,例如,双链多核苷酸,其两端均具有Y衔接子。
图33是示出了图32A中所展示的Y衔接子设计的示例序列的示意图。
图34A显示了Y衔接子设计的不同实施例,其包括两个杂交位点,一个用于孔系链(红色),另一个用于膜或珠粒系链(蓝色)。在该设计中,膜系链紧邻前导序列。图34B显示连接的分析物,例如在任一端具有Y衔接子的双链多核苷酸。
图35是显示图34A中说明的Y衔接子设计的示例序列的示意图。
图36是显示Y衔接子设计的一个替代实施例的示意图,其包括两个杂交位点,一个用于珠粒系链,另一个用于膜系链。在该设计中,珠粒具有两条不同的系链,一条连接到分析物 (蓝色),另一条连接到孔(红色)。
图37是显示图36中说明的Y型衔接子设计的示例序列并显示分析物与孔的间接连接的示意图。
图38显示了易位通过纳米孔的连续链的示例迹线,所述纳米孔没有可以与链结合以允许跟随测序的孔标签。两条链之间的时间由红色条指示,在这些实例中,两条链之间的时间范围为2-5秒。
图39示出了根据本文所公开的一种方法使修饰的孔易位的顺序链的示例迹线。两条链之间的时间由红色条指示,在这些实例中,两条链之间的时间范围为0.02-3秒。
图40显示了以对数标度说明连续链之间的间隔时间的直方图。左图显示具有单一分布的纳米孔(例如,CsgG孔),其中链间隔时间大于1秒。右图显示了易位通过拴系孔的链间隔时间。这显示了两个群体,即快速捕获群体,并且链间隔时间低于0.1秒。
图41描绘了显示从输入20ng DNA起的6小时内每个芯片测序的碱基数量的图表。红色线条表示拴系孔,蓝色线条表示非拴系纳米孔。
图42显示来自大肠杆菌运行的数据表,其显示具有拴系孔的跟随链的数量增加。图43 显示了可用于连接单链和双链核酸的方法。
图44显示了表征和串接许多双链靶多核苷酸的方法,其中第一双链靶多核苷酸的补体链募集许多其它双链靶多核苷酸并使其达到孔的局部浓度。这样在孔周围提供比在一般本体溶液中更高的局部浓度,因此双链靶多核苷酸彼此跟随通过开孔,链间隔时间最短。当双链靶多核苷酸的浓度较低时,这尤其有用。使用由与单链结合蛋白偶联的寡核苷酸组成的系链。当对第一双链靶多核苷酸的模板链进行测序时,补体链作为ssDNA释放到溶液中。其它双链靶多核苷酸的单链结合蛋白能够与ssDNA结合。当对补体链进行测序时,补体链的3'被拉回到孔中。ssDNA补体链上的单链结合蛋白在遇到马达蛋白时从补体链中移位,并且因此沉积在孔周围,增加局部浓度。
图45展示了根据本文提供的方法对核酸构建体进行测序的一种方法,其中检测到测序反应的副产物。图45A是在聚合酶控制下处理的DNA模板的示意性图示,作为通过合成反应测序的一部分,使得当发生掺入与DNA构建体的碱基互补的核苷酸时,磷酸盐标记的物质被释放并由纳米孔检测。图45B示出了DNA模板的顺序处理和随后通过掺入DNA模板的下一个互补核苷酸而检测到的释放的磷酸盐标记的物质。
图46和47展示了根据本文提供的方法的一个实施例的在不使用发夹的情况下对双链核酸构建体进行“后续”测序的方法。双链核酸构建体的有义链和反义链两者在每个端处均包括衔接子。
图46是在酶控制下处理的双链核酸的示意性图示,使得确定了双链核酸的两条链的序列。双链核酸的有义链和反义链不是共价连接的。在双链核酸模板的第一链被处理时,由检测器获得指示第一链的序列的信息,并且反义链将通过与检测器缀合的捕获标签定位于检测器。在双链核酸的第一链被处理后,第二链被分别处理,并且由检测器获得指示第二链的序列的信息。
图47表示图46中所示方法的实例。图47是在聚合酶控制下处理的双链DNA模板的示意性图示,使得检测到测序反应的副产物。双链模板的有义链和反义链没有共价连接,并且每条链具有负载在衔接子上的聚合酶。在处理双链DNA模板的有义链并通过纳米孔检测通过合成反应测序的副产物时,反义链将通过与纳米孔的外缘缀合的捕获多核苷酸定位于纳米孔。在处理了DNA模板的有义链后(并且酶已经解离),分别对反义链进行测序,并通过纳米孔检测通过合成反应测序的副产物。
图48是本文提供的另外的方法的示意性图示。在核酸外切酶控制下处理双链DNA模板,从而检测处理反应的产物。双链模板的有义链和反义链不是共价连接的。核酸外切酶(灰色) 处理有义链,其中核酸外切酶反应的产物由例如纳米孔(例如,跨膜蛋白纳米孔)等检测器检测。当有义链通过核酸外切酶处理时,反义链将通过与检测器缀合的捕获标签而定位于检测器。DNA模板的有义链已经通过核酸外切酶处理后,反义链通过核酸外切酶(黑色)分别处理,其与处理有义链的外切核酸酶(灰色)可以是相同或不同的核酸外切酶。核酸外切酶反应的产物由检测器检测,从而允许由检测器获得指示DNA模板的序列的信息。
具体实施方式
尽管例如跨膜孔(例如,蛋白纳米孔或固态纳米孔)等检测器可用作检测或表征生物聚合物的传感器,但是仍然存在提高使用检测器(如跨膜孔)的检测方法的准确度和/或效率的挑战。例如,通过发夹连接的双链多核苷酸的模板链和补体链两者通过纳米孔的易位均存在各种缺点。虽然以这种方式测量两条链是有利的,因为来自两条相连补体链的信息可以组合并用于提供比仅测量模板链可以获得的更高的准确度,这种发夹连接的多核苷酸的制备更复杂且更耗时并且可导致有价值的分析物的损失。此外,发夹连接的模板和互补多核苷酸链易位通过纳米孔可以引起纳米孔的另一(反)侧上的链的再杂交。这可以改变易位速率,从而降低测序精确度。具有发夹结构的链也更难以像线性单链那样快速易位。另外,由于模板链和补体链当前数据的差异,使用了两种算法进行计算,这使得计算更加复杂和密集。因此,需要改进表征分析物(例如,多核苷酸)的方法。
对于分析物检测,通常在一种分析物的易位与下一种分析物的易位之间存在时间延迟。这种延迟可以是几秒到几分钟的量级,这可以导致表征更慢,孔开放电流更高(更快地耗尽参比电极),和/或当孔打开时纳米孔被阻塞的可能性增加。因此,需要开发提高使用纳米孔表征分析物的准确度和/或效率或通量的方法和组合物。
本公开部分地基于出乎意料的发现,即,双链多核苷酸的两条链都可以例如通过酶(如聚合酶)顺序地处理,以提供序列信息,而无需通过桥接部分(如发夹环)共价连接两条链。例如,一方面,本发明人发现,当使用多核苷酸结合蛋白(例如,聚合酶)来处理双链多核苷酸的两条链中的第一条时,第二链可以保留在检测器(例如,孔)附近,并且在处理第一链之后,第二链可以由检测器捕获并且多核苷酸结合蛋白(例如,聚合酶)可以用于处理第二链。
在另一方面,本发明人发现,可以向双链多核苷酸的每个端提供具有双链体茎的衔接子,所述双链体茎包括与标签互补的捕获序列,所述标签与检测器(例如,纳米孔)缀合,其中捕获序列仅在处理时才显示。因此,在例如通过多核苷酸结合蛋白(例如,聚合酶)处理双链多核苷酸的第一链时,将衔接子的双链体茎解链,以使捕获序列暴露在双链多核苷酸的第二链上,然后由纳米孔的孔标签捕获。这种方法将第二链(否则通常会扩散开)保持在检测器(例如,纳米孔)附近,以便在测序模板后进行测序。具体地,与通常在通常纳米孔测序中观察到的时间的0.1%-1%相比,本文所描述的方法可以显著增加模板处理后的补体后续处理达到时间的至少约60%的可能性。
还发现修饰纳米孔以包括用于多种分析物的多个结合位点,使得一种或多种分析物可以通过结合位点结合到纳米孔,而通过纳米孔表征的分析物可以提高表征分析物的灵敏度和/或通量。在不希望受理论束缚的情况下,偶联或捕获在纳米孔的外缘处的分析物可以提高在孔处的分析物的局部浓度。进一步,在纳米孔附近的至少一种或多种分析物可以容易地一个接一个地进入用于表征的纳米孔,因此减少了时间延迟,并且因此减少了每个分析物表征之间的开孔电流时间。
因此,本文的各个方面涉及使用检测器(例如,纳米孔)表征一种或多种分析物的方法,以及在本文所描述的方法中可以使用的包含例如衔接子和纳米孔的组合物和系统。一些方面的特征在于使用纳米孔来表征双链多核苷酸的方法和组合物,例如,不使用连接双链多核苷酸的模板和补体的发夹。其它方面的特征在于使用标签修饰的纳米孔以增加的灵敏度和/或更高的通量表征分析物的方法和组合物。
因此,本文提供了一种方法,所述方法提供了一种通过检测测序(如通过合成反应进行的测序)反应的产物和/或副产物对核酸链进行测序的装置。可以通过使用紧邻检测器的聚合酶检测和分析信号来检测和/或表征副产物。可以标记核苷磷酸盐(核苷酸),以便在将核苷酸添加到与模板链互补的合成核酸链中时释放磷酸盐标记的物质,并检测磷酸盐标记的物质。合适的标记物可以是使用纳米孔或零模波导或通过拉曼光谱(Ramanspectroscopy)或其它检测器检测的光学标记物。合适的标记物可以是使用纳米孔或其它检测器检测的非光学标记物,例如,聚合物标签(例如,带电聚合物标签)。在另一种方法中,不标记核苷磷酸盐(核苷酸),并且在向与模板链互补的合成核酸链中添加核苷酸后,检测到天然副产物物质。合适的检测器可以是离子敏感的场效应晶体管或其它检测器。这些方法在本文中更详细地描述。
本领域的技术人员可以容易地实现检测通过酶处理双链多核苷酸所得的副产物。某些酶处理的副产物的检测先前描述于Stranges等人,“通过电极阵列上的合成用于单分子DNA测序的纳米孔偶联的聚合酶的设计和表征(Design and characterization of ananopore-coupled polymerase for single-molecule DNA sequencing by synthesison an electrode array)”,《美国国家科学院院刊(Proc Natl Acad Sci U S A.)》2016年11月1日;113(44):E6749–E6756,并且此类方法可以应用于本文所提供的方法中。
用于表征分析物(例如,双链多核苷酸)的方法
一方面,本公开提供了一种测序靶多核苷酸的方法,所述方法包括:
(a)使跨膜孔与以下接触:
(i)双链多核苷酸,所述双链多核苷酸包括所述靶多核苷酸和与所述靶多核苷酸互补的多核苷酸;以及
(ii)至少一种能够处理所述双链多核苷酸的链的聚合酶;
其中与所述双链多核苷酸的一部分结合的至少一个标签与所述跨膜孔缀合;
(b)检测与通过所述孔的离子流相对应的信号,以检测处理反应的副产物通过所述孔的易位;
(c)鉴定与通过所述聚合酶处理所述靶多核苷酸所得的副产物的易位相对应的信号和与通过所述聚合酶处理同所述靶多核苷酸互补的所述多核苷酸所得的副产物的单独转译相对应的顺序信号;以及
(d)分析(c)中鉴定的信号,
由此对所述靶多核苷酸进行测序。
所述方法可以在步骤(a)之前进一步包括将单链前导序列连接到靶多核苷酸和互补多核苷酸的步骤。所述方法在步骤(a)之前还可包括消化靶多核苷酸的一端以在补体链上产生前导序列和/或消化互补多核苷酸的一端以在靶链上产生前导序列的步骤。所述方法还可以进一步包括将多核苷酸结合蛋白(例如,多核苷酸解链酶)结合到前导序列。
在此方面,多核苷酸结合蛋白(例如,多核苷酸解链酶)通常是聚合酶。在此方面,第一多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶)分离双链多核苷酸的靶链和补体链,并处理靶多核苷酸或互补多核苷酸。第二多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶)可以是与第一多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶)相同类型的另一种蛋白质,或者可以是不同类型的多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶),处理其中互补多核苷酸已经被处理的靶多核苷酸,或者其中靶多核苷酸已经被处理的互补多核苷酸。第二多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶)不需要分离靶多核苷酸和互补多核苷酸,这是因为双链多核苷酸的两条链已经发生分离(第一多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶)将分离两条链,同时处理其中一条链)。第一多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶)通常是处理双链多核苷酸的蛋白质。第二多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶)通常是处理单链多核苷酸的蛋白质。第一和/或第二多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶)可以能够处理双链多核苷酸和单链多核苷酸。
本文还公开了一种通过纳米孔顺序易位两个非共价结合的分子的方法。所述方法包括:在促进一对非共价结合的分子对中的第一成员通过纳米孔易位的条件下,使所述一对非共价结合的分子与所述纳米孔接触,其中所述一对中的第二成员上的结合位点在第一成员通过纳米孔易位期间暴露,并且其中暴露的结合位点与存在于纳米孔上的标签或系链结合。在第一成员通过纳米孔易位之前,第二成员上的结合位点没有暴露(或被屏蔽)。
还提供了一种通过纳米孔顺序检测两个非共价结合的分子的方法。所述方法包括:在促进通过聚合酶处理一对非共价结合的分子的第一成员并通过纳米孔检测处理反应的副产物的条件下,使所述一对非共价结合的分子与所述纳米孔接触。一对中的第二成员上的结合位点在通过聚合酶处理一对中的第一成员期间暴露,并且所述结合位点与存在于纳米孔上的标签可逆地结合。
如本文所使用的,术语“非共价结合的分子”是指包括第一成员和第二成员的分子,其中第一成员和第二成员通过非共价连接彼此结合并且可以作为单独的实体彼此分离。第一成员和第二成员之间的分离和结合过程是可逆的。非共价连接方式的实例包括但不限于互补碱基配对、离子相互作用、疏水性相互作用和/或范德华(Van der Waals)相互作用。
在一些实施例中,非共价结合的分子包含互补多核苷酸链。
在一些实施例中,纳米孔上的标签是寡核苷酸,并且第二成员上的所述结合位点是具有与所述标签互补的序列的核酸的一部分。
在一些实施例中,该对非共价结合的分子包含与衔接子核酸连接的靶核酸(例如,靶双链多核苷酸),并且其中所述结合位点存在于所述衔接子上。
仅作为实例,图9B显示了非共价结合的分子,其包含连接到每个末端的互补多核苷酸链 (例如,模板链和补体链)和衔接子(例如,衔接子核酸)。如图9A所示,衔接子包含双链体茎(5)和从双链体茎的模板链延伸的第一单链多核苷酸(1)。双链体茎(5)包含与第一单链多核苷酸(1)对准的链上的捕获序列,其中捕获序列与标签(例如,捕获多核苷酸)互补,所述标签与纳米孔外缘缀合。在一些实施例中,第一单链多核苷酸(1)还可包含前导序列。虽然图9A显示了从双链体茎的补体链(例如,互补序列链)延伸的第二单链多核苷酸,但不是必需的。然而,在一些实施例中,可能需要具有一个或多个第二单链多核苷酸,其包含一条或多条用于固体基质,例如膜或珠粒和/或纳米孔的系链。当第二单链多核苷酸不与第一单链多核苷酸互补时,形成Y-衔接子,例如,如图9A中所示。
图9A显示了包含至少一个用于固体基质(例如,膜或珠粒)的锚的示例衔接子,而图 15显示了包含至少两个锚的示例衔接子,其中第一锚能够拴系到固体基质,例如,膜或珠粒,并且第二锚能够拴系到纳米孔。纳米孔的第二锚可以被配置成结合到与纳米孔缀合的标签。在一些实施例中,纳米孔的第二锚可以被配置成直接结合到与纳米孔缀合的标签。例如,纳米孔的第二锚可以包含捕获多核苷酸标签互补的序列,所述标签与纳米孔缀合。在替代实施例中,用于纳米孔的第二系链可以被配置成间接结合与纳米孔缀合的标签。例如,图36显示连接到分析物的衔接子可以通过微粒与纳米孔上的标签偶联,这将在下面的“微粒”部分中进一步详细描述。
应注意,本文描述的衔接子可以连接到双链多核苷酸的任一端或两端。在一些实施例中,相同的衔接子连接到双链多核苷酸的两端。在一些实施例中,不同的衔接子可以连接到双链多核苷酸的末端。例如,通过将两个或更多个不同衔接子群体与双链多核苷酸混合在一起,可以实现不同衔接子与双链多核苷酸末端的连接。通常,形成与不同衔接子连接的双链多核苷酸的混合物,但也存在获得所需异衔接子混合物的方法(例如,通过纯化或通过控制衔接子与双链多核苷酸末端的连接)。
在一些实施例中,双链多核苷酸可在其3'端或5'端具有衔接子。
平端双链多核苷酸可以由检测器(如纳米孔)捕获,并解链或以其它方式处理,例如,通过聚合酶。因此,在一些实施例中,不具有衔接子(例如,本文所描述的衔接子)的平端构建体可以用于本文所描述的方法的任何方面。尽管不是必需的,但是在一些实施例中,期望使前导序列与双链多核苷酸的至少一个端偶联,例如以提高通过聚合酶处理的效率。
在衔接子连接到双链多核苷酸的两端的一些实施例中,本领域的普通技术人员将容易认识到,当衔接子的第一单链多核苷酸在双链多核苷酸的一端处与模板链偶联时,另一个衔接子的第一单链多核苷酸在双链多核苷酸的相反端处与补体链偶联。
在一些实施例中,衔接子可以在靶多核苷酸的每个端处具有预结合的多核苷酸结合蛋白 (例如,多核苷酸解链酶,例如,聚合酶)。在一些实施例中,所述方法可以进一步包括在溶液中添加多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶),使得其在靶多核苷酸的每个端处结合到衔接子。
在图10中所展示的公开方法中,双链多核苷酸的每个端连接到如本文所描述的衔接子,并且多核苷酸结合蛋白(例如,多核苷酸解链酶)被负载在衔接子上,其中衔接子包括具有与标签互补的捕获序列的双链体茎,所述标签与纳米孔缀合(参见例如图9A作为示例衔接子)。从包括双链多核苷酸的构建体延伸的单链多核苷酸(其可以任选地包括前导序列)进入纳米孔(图A)。进入纳米孔的第一链标记为模板(T),捕获的第一链的反向互补序列标记为互补序列(C)。当第一模板链在多核苷酸结合蛋白(例如,多核苷酸解链酶,例如解旋酶马达) 的控制下穿过孔时,互补序列逐渐被拉开。图B显示,在模板链的末端,互补序列上在双链体茎内的捕获序列通过拉开而暴露,并因此与纳米孔上的标签(例如,捕获多核苷酸)偶联。在图C中,当模板链最终穿过纳米孔并且酶解离时,补体链通过与纳米孔上的标签(例如,捕获多核苷酸)结合而保持与纳米孔偶联。在一定时间后,补体链被其前导序列捕获。在图D 中,补体链在负载的第二多核苷酸结合蛋白,例如多核苷酸解链酶如解旋酶马达的控制下穿过纳米孔。在补体链穿过纳米孔时,捕获序列将在某些点上从纳米孔的标签(例如,捕获多核苷酸)解链,因此释放出纳米孔的标签(例如,捕获多核苷酸),使得可用于下一条链。在此公开的方法中,两种多核苷酸结合蛋白(例如,多核苷酸解链酶)都能够处理双链多核苷酸。在一些方法中,在两端处负载的多核苷酸结合蛋白(例如,多核苷酸解链酶)可以相同或不同。
虽然图10(图B)展示了在双链体中暴露捕获序列(用于结合到纳米孔上的标签)是在模板链易位结束时发生的(例如,捕获远端衔接子,因此使其局部定位于孔中),更一般地,可以设计链,使得双链多核苷酸(例如,通过端或尾部)与纳米孔的结合可以恰好在解链过程开始时发生。在这种情况下,捕获序列可以定位在衔接子中(例如,作为Y衔接子部分的非互补臂),使得在整个解链过程阶段中或者甚至在解链过程之前,都将所述捕获序列暴露用于与纳米孔结合。
在一些实施例中,代替使多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶) 结合在连接到靶多核苷酸的衔接子上,可以将多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶)固定在纳米孔的腔内,使得可以使用单个多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶)来处理两条链。
在一些实施例中,当处理一条链时,解链或分离链是由多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶)控制的。链的解链或分离可以在不存在多核苷酸结合蛋白(例如,多核苷酸解链酶)的情况下发生。控制多核苷酸的两条链的移动和/或分离的这种无酶方法是本领域已知的。例如,某些纳米孔本身可以提供使多核苷酸如马达蛋白纳米孔解链的力,包括例如phi29马达蛋白纳米孔,例如,如Wendell等人“Translocation ofdouble-stranded DNA through membrane-adapted phi29 motor protein nanopores”Nat Nanotechnol,4(2009),第765–772 页所述,和/或美国专利号8,986,528中所述的纳米孔,其各自的内容是通过引用整体并入本文。
如本文所用,术语“易位(translocate或translocation)”是指沿着纳米孔的至少一部分运动。在一些实施例中,易位从纳米孔的顺侧移动到纳米孔的反侧。
靶多核苷酸通常存在于包含靶多核苷酸的多个拷贝的样品中和/或存在于包含多种不同多核苷酸的样品中。在一些实施例中,本文所述任何方面的方法均可包括确定样品中一个或多个靶多核苷酸的序列。所述方法可包括使孔与两个或更多个双链多核苷酸接触。例如,所述方法可包括使孔与样品接触,其中基本上所有双链多核苷酸在其两条链的每一条上都具有单链前导序列。在一些实施例中,双链多核苷酸仅通过互补碱基配对彼此偶联。在这些实施例中,双链多核苷酸可具有四个游离末端,其中游离末端是多核苷酸链的末端。多核苷酸链的末端可为单链,例如单链突出端,或与另一条多核苷酸链碱基配对。在一些实施例中,测序的双链多核苷酸的两条链不共价连接(例如,无发夹或其它共价连接)。然而,不桥接模板多核苷酸和补体多核苷酸的部分可以添加到一个或多个自由端。
在本文所描述的各个方面的一些实施例中,所述方法可以进一步包括在与检测器(如纳米孔)接触之前,将单链前导序列生成或连接到样品中基本上所有双链多核苷酸的两条链上的步骤。添加的前导序列可以具有连接到其上的一个或多个多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶),使得双链多核苷酸群体各自包括具有在其两条链的每条链的一端处连接到其的多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶)的前导序列。
包括靶多核苷酸(例如,模板)和与靶多核苷酸(例如,补体)互补的多核苷酸的双链多核苷酸可以具有包括在其每个端处连接的单链前导序列的衔接子。在涉及多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶)的各个方面的方法的一些实施例中,所述方法可以包括使孔与可以相同或不同的两种或更多种多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶)接触。可以将不同的多核苷酸结合蛋白(例如,多核苷酸解链酶)结合到可以相同或不同的单独的前导序列。例如,在5'至3'方向起作用的多核苷酸结合蛋白(例如,多核苷酸解链酶)可以与靶多核苷酸的5'末端和/或互补多核苷酸的5'末端的前导序列结合。在3'至5'方向起作用的多核苷酸结合蛋白(例如,多核苷酸解链酶)可以与靶多核苷酸的3'末端和/或互补多核苷酸的3'末端的前导序列结合。
本文所述的各个方面的方法中使用的衔接子可以被进一步配置成允许分析物(例如,靶多核苷酸或非共价结合的分子)与纳米孔结合,以提高所述表征方法的灵敏度和/或通量。这在目的上不同于其中衔接子的双链体茎中的捕获序列被暴露以允许与非共价结合的分子的第二成员(例如,双链多核苷酸的补体链)结合的实施例的目的,例如如图10所展示的用于增加模板易位后补体易位的可能性,从而提高测序信息的准确度。如图16所展示的,衔接子可以被进一步配置成包含用于结合到纳米孔的系链,例如以促进分析物的捕获。因此,在分析物的第一成员(例如,双链多核苷酸的模板链)进入纳米孔之前,分析物结合到与纳米孔缀合的第一标签。当第一成员易位通过孔并拉开双链体茎以暴露第二成员上的捕获序列时,第二个成员结合到与纳米孔缀合的第二标签,使得第二成员保持靠近纳米孔以在第一成员后进行后续表征。根据衔接子的设计,纳米孔上的第一标签和第二标签可以是不同的(例如,如图16所示),或者可以是相同的(例如,如图17A所示)。
因此,本文所公开的另外的方法涉及使用纳米孔确定分析物的特性,所述方法包括:(a) 提供被修饰为包括至少两个或更多个在纳米孔的内腔外部的标签的纳米孔,其中标签提供至少两种或更多种分析物的结合位点;以及(b)在使得至少一种或多种(例如,至少一种、至少两种、至少三种、至少四种、至少五种、至少六种、至少七种、至少八种、至少九种或更多种)分析物结合到纳米孔上的标签的条件下,使多种分析物与纳米孔接触,而来自多种中的分析物通过纳米孔易位。
与例如通过改变纳米孔内腔内的氨基酸的电荷和/或疏水性而被修饰以改善纳米孔与靶分析物的相互作用的纳米孔不同,本文所描述的纳米孔被修饰以提供多个标签以在处理分析物时捕获用于表征的多种分析物。这减少了每个分析物表征之间的开孔时间,还增加了分析物的局部浓度,由此提高了方法的灵敏度。在一些实施例中,如下文“标签或系链修饰的纳米孔 (例如,用于增强分析物捕获,如多核苷酸捕获)”部分中所描述的标签修饰的纳米孔可以用于实现这种目的。
在一些实施例中,可以修饰分析物以结合到检测器上的标签(例如,纳米孔)。在一些实施例中,分析物包括本文所描述的衔接子,例如,包括用于纳米孔的锚的衔接子。
检测器(例如,纳米孔)上的标签与分析物上的结合位点(例如,存在于连接到分析物的衔接子中的结合位点,其中结合位点可以由衔接子的锚或前导序列或由衔接子的双链体茎内的捕获序列提供)之间的相互作用可以是可逆的。例如,分析物可以例如通过其衔接子结合到纳米孔上的标签,并且例如在通过纳米孔表征分析物期间在某些点处释放。强的非共价结合(例如,生物素/抗生物素蛋白)仍然是可逆的,并且可以用于本文所描述的方法的一些实施例中。例如,为了确保在处理模板后处理双链多核苷酸的补体,可以期望设计一对孔标签和分析物衔接子,以在双链多核苷酸的补体(或衔接子的与补体连接的一部分)与纳米孔之间提供足够的相互作用,使得补体保持靠近纳米孔(在模板易位期间不会与纳米孔分离并扩散),但能够在处理时从纳米孔中释放出来。
因此,在一些实施例中,本文所描述的方法中使用的一对孔标签和分析物衔接子可以被配置成使得分析物上的结合位点(例如,存在于连接到分析物的衔接子中的结合位点,其中结合位点可以由衔接子的锚或前导序列或由衔接子的双链体茎内的捕获序列提供)与纳米孔上的标签的结合强度或亲和力足以维持纳米孔与分析物之间的偶联持续一段时间,直到施加的力放置于其上以从纳米孔释放结合的分析物。在分析物是双链多核苷酸的一些实施例中,施加的力可以是例如通过聚合酶处理补体链。
在本文所描述的各个方面的一些实施例中,所述方法可以进一步包括,在跨膜施加电位时,检测响应于通过纳米孔的分析物(例如,通过聚合酶处理多核苷酸所得的副产物)的信号。在一些实施例中,电位差可以由提供离子流的渗透性不平衡来驱动。在一些实施例中,可以在位于纳米孔两侧的两个电极之间跨纳米孔施加电位差。信号可以是电测量和/或光学测量。可能的电测量包含:电流测量、阻抗测量或隧穿测量(Ivanov AP等人,《纳米快报(Nano Lett.)》2011年1月12日;11(1):279-85)和FET测量(国际申请号WO 2005/124888),例如,电压FET测量。在一些实施例中,信号可以是跨固态纳米孔的电子隧穿或跨固态纳米孔的电压FET测量。光学测量可以与电测量结合(Soni GV等人,《科学仪器综述(Rev Sci Instrum.)》 2010年1月;81(1):014301)。测量可以是跨膜电流测量,如对流过孔的离子电流的测量。图 11A-11B示出了使用本文所公开的方法在多核苷酸结合蛋白(例如,多核苷酸解链酶)的控制下通过纳米孔为双链多核苷酸测序期间,随时间推移测量的典型电流信号。
替代性地,测量可以是指示通过通道的离子流的荧光测量,如通过Heron等人,《美国化学协会期刊(J.Am.Chem.Soc.)》2009,131(5),1652-1653所公开的或使用FET测量跨膜的电压。在一些实施例中,所述方法可以进一步包括,在施加跨膜电位时,检测处理多核苷酸时流过纳米孔的离子电流。在一些实施例中,可以使用膜片钳或电压钳来进行所述方法。在一些实施例中,可以使用电压钳来进行所述方法。电测量可以使用标准信号通道记录设备来进行,如下所述:Stoddart D等人,《美国国家科学院院刊(Proc Natl Acad Sci)》,12;106(19):7702-7; Lieberman KR等人,《美国化学会志(J Am Chem Soc.)》2010;132(50):17961-72;以及国际申请WO 2000/28312。可替代地,可以使用多通道系统进行电测量,例如如国际申请WO 2009/077734和国际申请WO 2011/067559中所述。
可以提供纳米孔阵列以增加通量并因此增加对多核苷酸链进行测量,如国际申请WO2014/064443中公开的,其内容通过引用并入本文。
从以上讨论将显而易见的是,本文提供的方法可以包括提供一种条件,以允许构建体的模板链通过聚合酶进行处理并且处理反应的副产物由检测器检测,由此检测通过聚合酶向多核苷酸链添加核苷酸。
探针可以包括适合于与单个双链多核苷酸如DNA或RNA靶分子相互作用的酶如聚合酶或逆转录酶。响应于依次遇到的模板链核酸碱基和/或掺入模板指定的天然或类似物碱基(即,掺入事件),催化将核苷酸碱基模板依赖性掺入生长的寡核苷酸链中的寡核苷酸链中的酶发生构象变化。这种构象变化可以调节流过与探针偶联的桥接分子的电流,由此以依赖于模板分子的方式提供序列特异性信号模式。
因此,所述方法可以涉及检测通过如聚合酶等酶向核酸链依次添加多核苷酸所得的产物,其中产物是酶的一种或多种性质的改变,如酶的构象中的改变。这种方法因此可以包括在以下条件下使如聚合酶或逆转录酶等酶经受双链多核苷酸:响应于依次遇到的模板链核酸碱基和/或掺入模板指定的天然或类似物碱基(即,掺入事件),使得将核苷酸碱基模板依赖性掺入生长中的寡核苷酸链中会引起酶的构象变化,响应于这种掺入事件检测酶的构象变化,并且由此检测模板链的序列。这种方法可以涉及使用本领域的技术人员已知的方法,如在US 2017/0044605中描述的方法,检测和/或测量掺入事件。
信号测量分析
根据本文提供的方法,任何合适的信号可以用于检测酶对多核苷酸的处理。适用于本文提供的方法的各种检测方法是本领域的技术人员已知的。通过非限制性实例,处理反应的产物和/或副产物可以使用纳米孔或通过光谱法(例如,拉曼光谱法,如表面增强拉曼光谱法) 或显微技术(例如,原子力显微镜)检测。使用表面增强拉曼光谱法检测核碱基已经描述于 Chen,Li,Kerman,Neutens,Willems,Carnelissen,Lagae,Stakenborg和VanDorpe(“用于单分子核碱基感测的高空间分辨率纳米狭缝SERS(High spatial resolutionnanoslit SERS for single-molecule nucleobase sensing)”,《自然通讯(NatureCommunications)》,(2018)9:1733) 中,并且适用于本文提供的方法。因此,在一些实施例中,使用光谱或显微镜技术,优选地 SERS来检测处理反应的产物和/或副产物。
在一些其它实施例中,所述方法包括检测与通过孔的离子流相对应的信号,所述信号指示相互作用,例如,通过聚合酶处理多核苷酸。在一些实施例中,电位差可以由提供离子流的渗透性不平衡来驱动。在一些实施例中,可以在定位在孔的任一侧上的两个电极之间的跨膜孔之间施加电位差。替代性地,测量可以是指示通过通道的离子流的荧光测量,如通过Heron 等人,《美国化学协会期刊(J.Am.Chem.Soc.)》2009,131(5),1652-1653所公开的。可以提供纳米孔阵列以增加通量并因此增加对多核苷酸链进行测量,如通过WO2014064443所公开的。
图4示出了在公开的方法中在酶控制下在多核苷酸通过纳米孔易位的期间随时间测量的典型电流信号。当要易位的多核苷酸通过发夹连接时,可以在发夹中提供非核苷酸或修饰的核苷酸以提供指示发夹的信号。电流信号反映了多核苷酸在易位通过纳米孔时的序列。因此,可以确定信号的哪些部分指示模板和互补序列。通常,酶使多核苷酸松脱通过纳米孔,产生特征电流水平。信号随时间变化的幅度取决于纳米孔的性质,并且多于一个核苷酸可以在任何特定时间影响电流。
在一些公开的方法中,在任何特定时间影响电流的核苷酸的数量可以取决于一组k个核苷酸单元,其中k是复数整数,以下称为“k聚体”。这可以在概念上被认为是具有比测量的聚合物单元大的“平端读取头”的纳米孔。在这种情况下,要求解的不同k聚体的数量增加到k 的幂。例如,如果存在n个可能的聚合物单元,则要求解的不同k聚体的数量为n
通过执行已知分析技术如运行‘t-检验’,可以将电流与时间采样数据点关联成连续组,运行‘t-检验’试图找到信号的局部均值的变化。这些组称为事件。指示特定k聚体的事件可以如图6中所示进行确定。事件用一些概括特征(相关联的组内的数据点的平均电流和关于平均电流的标准偏差)表示。
为了确定多核苷酸序列,在一些公开的方法中,可以参考模型,所述模型考虑了k聚体之间可能的转变的数目,并且还考虑了电流水平。在WO2013041878中公开了这种分析技术,在此通过引用将其并入,其中参考如隐马尔可夫模型(HMM)等概率分析技术用于确定可能的转变的总数,并且其中最可能的转变随后通过如维特比算法等分析技术来确定。可以采用递归神经网络(RNN)作为HMM的替代,并且在例如描述事件与产生事件的核苷酸数量之间的潜在关系方面,提供比HMM更大的数学表达自由度。此类采用RNN的方法在图7中通过实例进行了说明,其中由事件推导出包含来自其它相邻事件的信息的特征。这为RNN提供了额外信息,RNN是一种数学模型,其输出取决于先前对数据序列的计算。图8中示意性地和简单地示出了如何采用维特比算法来确定通过具有最高似然性的可能转变的路径的实例。
k聚体之间最可能的转变可用于确定k聚体序列,从而确定核苷酸链的核苷酸序列。由于所采用的数学方法的性质,经常以准确度%来表示核苷酸序列。
在仅涉及模板链的测量的方法中,上述方法可以用于确定模板序列。然而,在测量模板及其反向补体的情况下,模板及其补体之间的配对关系可以为测序测量提供额外的功率。利用这种关系的特定技术的实例公开于WO2013041878中,由此将模板事件t
原则上,2D碱基识别器必须检查(t
可替代地,可以通过比较模板和互补序列事件数据或核苷酸序列并确定模板和互补序列之间的最佳关联来进行核苷酸序列的确定。然而,这种方法不能提供更高的2D模型准确度,因为它未考虑模板和补体链之间组合的最高可能性。公开了比较模板碱基调用和补体碱基调用的共识方法的实例。
与通过发夹连接模板链和补体链的情况不同,补体链可能并不总是在模板链之后按顺序进行处理。例如,在处理双链构建体的模板链之后,存在可以处理来自第二双链构建体的模板链的可能性。此外,在处理模板(第一)链之后,存在补体(第二)链可能不被检测器(例如,纳米孔)的结合位点捕获的可能性。这可能是由于例如检测器(例如,纳米孔)的一个或多个结合位点已经被一个或多个补体链占据,并且因此结合位点不可用于补体链。没有被检测器(例如,纳米孔)捕获的任何补体链可能会扩散离开纳米孔,并且不会被纳米孔捕获。因此,为了利用模板链和补体链的增加的功率,首先必须确定信号测量结果是否对应于模板及其对应的补体。
在本文描述的各个方面的一些实施例中,所述方法可以进一步包括鉴定与靶多核苷酸的处理相对应的信号和对应于与靶多核苷酸互补的多核苷酸的单独处理的顺序信号,以及由此鉴定的信号的分析。对于如此鉴定的那些信号,以上数学方法可以用于确定靶标的核苷酸序列,其中所述方法利用靶标(模板)和补体两者的信息及其相关优点。
为了鉴定信号(例如,顺序信号)是否对应于靶标及其补体,可以将事件彼此对准以确定对准程度。根据对准程度,可以确定实际上信号是否对应于靶标及其互补序列。用于产生正相关的对准%可以任意选择,并且可以例如大于95%。可以采用已知的成对对准方法,例如 Smith-Waterman或Needleman-Wunsch算法。可以使用的对准方法的合适实例在WO2015/140535或WO 2016/059427中公开。
已经观察到,一般而言,补体链以其连续顺序跟随其模板链,或者从检测器(例如,纳米孔)扩散。在与该特定补体链不相关的另外的链之后,补体链跟随其模板链的机会要低得多。模板和补体对具有独特的特性。例如,模板/补体对通常倾向于共享相同长度的核苷酸(事件数)。另外,一对的后续链可以比新链更快地处理,和/或补体的测序倾向于更快,等等。这些特性中的一个或多个可以用于鉴定模板/补体对而无需复杂的计算分析。
在一些实施例中,为了减少计算需求,可以将对准限制于链的相邻测量。一旦确立了链间的模板-互补序列关系,序列的确定就可以利用对模板或补体链的测量或对模板和补体链的测量。例如,可以确定模板链的序列,其中可以认为测序准确度不够高。在这种情况下,该方法可以选择通过考虑模板和互补序列数据来确定序列,以提供高于仅通过确定模板序列而获得的序列准确度。可替代地,可以认为模板链的序列准确度足够好,以致不需要考虑模板和互补序列数据两者。确定是否使用模板序列数据或者是否使用模板和互补序列数据两者的因素可以是例如基础序列是否具有难以精确识别的碱基或碱基组,或例如特定碱基是否是单个核苷酸多态性变体。
在没有确定序列关系的情况下,可以按照对于仅模板链的测量所进行的相同方式确定该特定链的序列。在确定序列关系的情况下,可以按照对于模板和补体链的测量所进行的相同方式确定该特定链的序列。此信息可以被组合以提供整体序列确定。
一些公开的方法进一步包括分析当第一链(例如,靶多核苷酸)通过跨膜孔易位时产生的信号,以及与第一链互补的第二链通过相同的纳米孔易位时产生的信号。第一链(例如,靶多核苷酸)及其补体(第二链)通过碱基配对连接。因此,一旦第一多核苷酸结合蛋白(例如,多核苷酸解链酶)沿着双链多核苷酸的长度移动,第一链和第二链就不再连接。参见,例如,图10。因此,第二链易位通过纳米孔是与第一链易位通过纳米孔分开的事件,例如,如图11所示,其中在第一链和第二链易位间期观察到约200pA的开孔电流(没有阻断电流的链)。虽然不是必需的,但是期望第二链的易位在第一链易位后尽快,例如立即(例如,小于 1秒)发生。参见例如,图13。
所公开的方法包括鉴定与靶多核苷酸和与靶多核苷酸互补的多核苷酸的顺序易位相对应的信号的步骤。顺序易位包含互补多核苷酸通过与靶多核苷酸相同的孔易位的情况。靶多核苷酸和互补多核苷酸可以以任何顺序易位通过该孔。其它多核苷酸,例如1、2、3、4或5个至约10个多核苷酸可以穿过靶多核苷酸和互补多核苷酸之间的孔。优选地,靶多核苷酸和互补多核苷酸以任何顺序连续地穿过该孔。孔优选地返回到第一靶多核苷酸和互补多核苷酸通过孔的易位与第二靶多核苷酸和互补多核苷酸通过孔的易位之间的开放状态。
在一些实施例中,可以通过将靶多核苷酸和/或互补多核苷酸系链到膜和/或孔上来促进靶多核苷酸和互补多核苷酸的连续处理。促进双链多核苷酸的两条链的连续处理的其它方法包含将靶多核苷酸和/或互补多核苷酸连接到微粒和/或修饰孔以增加/增强多核苷酸捕获。
可以使用条形码来促进与靶多核苷酸的处理相对应的信号的鉴定和对应于与靶多核苷酸互补的多核苷酸的单独处理的顺序信号。通常,双链条形码包含在或连接到双链多核苷酸。当靶多核苷酸和互补多核苷酸分离时(通过多核苷酸结合蛋白(例如,多核苷酸解链酶)),条形码既保留在靶多核苷酸中又保留在互补多核苷酸中。跨膜孔对条形码的表征将创建该条形码的信号特性。孔对条形码的第二次和后续检测可以用于确定靶多核苷酸及其补体已经被孔顺序确定。由此可以鉴定对应于靶多核苷酸以及分别与靶多核苷酸互补的多核苷酸的信号。
标签或系链修饰的纳米孔(例如,用于增强分析物捕获,如多核苷酸捕获)
用于本文所描述的方法的检测器被修饰以包括一个或多个结合位点,以与一种或多种分析物结合。
当检测器是纳米孔时,用于本文所描述的方法的纳米孔被修饰以包括一个或多个结合位点,以与一种或多种分析物结合。在一些实施例中,纳米孔可以被修饰以包括一个或多个结合位点,以与连接到分析物的衔接子结合。例如,在一些实施例中,纳米孔可以与连接到分析物的衔接子的前导序列结合。在一些实施例中,纳米孔可以与连接到分析物的衔接子中的单链序列结合。在一些实施例中,纳米孔可以与连接到分析物的衔接子的双链体茎内的捕获序列结合,其中仅在对双链体茎进行解链时才显露捕获序列。
在一些实施例中,可以修饰纳米孔以包括一个或多个结合位点,以与连接到双链寡核苷酸的第一链或第二链的衔接子结合,例如,以促进通过聚合酶对第一链和第二链的顺序处理。
纳米孔可以被修饰以包括一个或多个标签或系链,每个标签或系链包括分析物的结合位点。
在一些实施例中,纳米孔被修饰以包括两个或更多个标签或系链。例如,可以提供一个标签或系链以增加表征如多核苷酸等分析物的方法的灵敏度(“灵敏度标签”),而可以提供另一个标签或系链以增加跟随多核苷酸的模板链的补体链测序的可能性(“后续标签”)。如图15 所示,孔标签可以以多种方式构造。仅举例来说,在一些实施例中,寡聚孔的每个单体可以具有相同类型的标签构型(例如,具有多个结合位点,如Tag-A和Tag-B所示)。Tag-A和Tag-B 可以组合形成单个标签,至少一个或多个单体包含Tag-A/Tag-B组合标签。可替代地,寡聚孔可包含有不同标签连接的混合单体,使得至少一种单体具有与其它单体不同的标签构型。在另一个实例中,Tag-A和Tag-B可以保持为单独的标签,并且至少一个或多个单体可以包括两种单独的标签。如果灵敏度标签和跟随标签与如本文所述的衔接子中使用的独特序列互补,则可以单独组合。
图16是可如何使用具有两种不同标签类型的纳米孔从溶液中捕获链(以便提高灵敏度) 的示意图。连接到双链多核苷酸末端的衔接子包含可用于偶联至第一孔标签的捕获序列(例如,形成Y-衔接子的非互补臂),同时双链体茎内只有在被拉开时才显露的单独捕获序列允许互补序列结合第二孔标签,因此使互补序列捕获成为可能以进行跟随测序。
纳米孔上的标签与分析物上的结合位点(例如,存在于连接到分析物的衔接子中的结合位点,其中结合位点可以由衔接子的锚或前导序列或由衔接子的双链体茎内的捕获序列提供) 之间的相互作用可以是可逆的。例如,分析物可以例如通过其衔接子结合到纳米孔上的标签,并且例如在通过纳米孔表征分析物期间和/或在聚合酶处理期间在某些点处释放。强的非共价结合(例如,生物素/抗生物素蛋白)仍然是可逆的,并且可以用于本文所描述的方法的一些实施例中。例如,为了确保在处理模板后处理双链多核苷酸的补体,可以期望设计一对孔标签和分析物衔接子,以在双链多核苷酸的补体(或衔接子的与补体连接的一部分)与纳米孔之间提供足够的相互作用,使得补体保持靠近纳米孔(不会与纳米孔分离并扩散),但能够在处理时从纳米孔中释放出来。
因此,在本文所描述的各个方面的一些实施例中,孔标签和分析物衔接子对可以被配置成使得分析物上的结合位点(例如,存在于连接到分析物的衔接子中的结合位点,其中结合位点可以由衔接子的锚或前导序列或由衔接子的双链体茎内的捕获序列提供)与纳米孔上的标签的结合强度或亲和力足以维持纳米孔与分析物之间的偶联,直到施加的力放置于其上以从纳米孔释放结合的分析物。在分析物是双链多核苷酸的一些实施例中,施加的力可以是通过聚合酶处理补体链。
在一些实施例中,标签或系链不带电。这样可以确保在电位差的影响下,标签或系链不会被拉入纳米孔中。
吸引或结合多核苷酸或衔接子的一个或多个分子可以与检测器(例如,孔)连接。可以使用与衔接子和/或靶多核苷酸杂交的任何分子。连接到孔的分子可选自PNA标签、PEG接头、短寡核苷酸、带正电荷的氨基酸和适体。具有与它们连接的此类分子的孔是本领域已知的。例如,具有连接到其的短寡核苷酸的孔公开于Howarka等人(2001)《自然生物技术(Nature Biotech.)》19:636-639和WO 2010/086620,并且包括连接在孔的内腔内PEG的孔公开于 Howarka等人(2000)《美国化学协会期刊》122(11):2411-2416。
连接到检测器(例如,跨膜孔)的短寡核苷酸,所述寡核苷酸包括与前导序列中的序列或衔接子中另一个单链序列互补的序列,可以用于在本文描述的任何方面的方法中增强靶多核苷酸和/或互补多核苷酸的捕获。
在一些实施例中,标签或系链可以包括或可以是寡核苷酸(例如,DNA、RNA、LNA、BNA、PNA或吗啉代)。寡核苷酸(例如,DNA、RNA、LNA、BNA、PNA或吗啉基)可具有约10-30个核苷酸的长度或约10-20个核苷酸的长度。示例性寡核苷酸(例如,DNA、RNA、 LNA、BNA、PNA或吗啉基)可包含SEQ ID NO:8中所示的序列。在一些实施例中,用于标签或系链中的寡核苷酸(例如,DNA、RNA、LNA、BNA、PNA或吗啉基)可具有至少一个被修饰用于与其它修饰或固体基质表面(包括,例如珠粒)的末端(例如,3'-或5'-末端)缀合。末端改性剂可以添加可以用于缀合的反应性官能团。可添加的官能团的实例包括但不限于氨基、羧基、硫醇、马来酰亚胺、氨氧基及其任何组合。官能团可以与不同长度的间隔区 (例如,C3、C9、C12、间隔区9和18)组合以增加官能团与寡核苷酸序列末端的物理距离。在一些实施例中,标签或系链可以是寡核苷酸(例如,DNA、RNA、LNA、BNA、PNA或吗啉基),其具有SEQ ID NO:8所示的具有5'-马来酰胺修饰的序列。在一些实施例中,标签或系链可以是寡核苷酸(例如,DNA、RNA、LNA、BNA、PNA或吗啉基),其具有SEQ ID NO: 8所示的具有3'-马来酰胺修饰的序列。在一些实施例中,标签或系链可以是寡核苷酸(例如, DNA、RNA或PNA),其具有SEQ ID NO:8所示的具有5'-C9-硫醇修饰的序列。在一些实施例中,标签或系链可以是寡核苷酸(例如,DNA、RNA、LNA、BNA、PNA或吗啉基),其具有SEQ ID NO:8所示的具有3'-C9-硫醇修饰的序列。在一些实施例中,标签或系链可以是寡核苷酸(例如,DNA、RNA、LNA、BNA、PNA或吗啉基),其具有SEQID NO:8所示的具有5'-硫醇修饰的序列。在一些实施例中,标签或系链可以是寡核苷酸(例如,DNA、RNA、 LNA、BNA、PNA或吗啉基),其具有SEQ ID NO:8所示的具有3'-硫醇修饰的序列。
在一些实施例中,标签或系链可包含或是吗啉基寡核苷酸。吗啉基寡核苷酸可具有约 10-30个核苷酸的长度或约10-20个核苷酸的长度。示例性吗啉基寡核苷酸可包含SEQID NO: 8中所示的序列。吗啉基寡核苷酸可以是修饰的或未修饰的。例如,在一些实施例中,吗啉基寡核苷酸可以在寡核苷酸的3'和/或5'末端被修饰。吗啉基寡核苷酸的3'和/或5'末端上的修饰的实例包括但不限于3'亲和标签和用于化学连接的官能团(包括,例如3'-生物素、3'-伯胺、 3'-二硫化物酰胺、3'-吡啶基二硫基及其任何组合);5'末端修饰(包括,例如5'-伯胺和/或 5'-dabcyl),用于点击化学的修饰(包括,例如3'-叠氮化物、3'-炔烃、5'-叠氮化物、5'-炔烃) 及其任何组合。在一些实施例中,标签或系链可以是吗啉基寡核苷酸,其具有SEQ ID NO:8 所示的具有5'-叠氮化物修饰的序列。在一些实施例中,标签或系链可以是吗啉基寡核苷酸,其具有SEQ ID NO:8所示的具有3'-叠氮化物修饰的序列。在一些实施例中,标签或系链可以是吗啉基寡核苷酸,其具有SEQ ID NO:8所示的具有5'-炔烃修饰的序列。在一些实施例中,标签或系链可以是吗啉基寡核苷酸,其具有SEQ ID NO:8所示的具有3'-炔烃修饰的序列。在一些实施例中,标签或系链可以是吗啉基寡核苷酸,其具有SEQ ID NO:8所示的具有3'-吡啶基二硫修饰的序列。
在一些实施例中,标签或系链可以进一步包括聚合物接头,例如,以刺进偶联到检测器,例如,纳米孔。示例性聚合物接头包含但不限于聚乙二醇(PEG)。聚合物接头可具有约500Da 至约10kDa(包括端值),或约1kDa至约5kDa(包括端值)的分子量。聚合物接头(例如,PEG)可以用不同的官能团官能化,包括例如但不限于马来酰亚胺、NHS酯、二苯并环辛炔(DBCO)、叠氮化物、生物素、胺、炔烃、醛及其任何组合。在一些实施例中,标签或系链还可包含具有5'-马来酰亚胺基团和3'-DBCO基团的1kDa PEG。在一些实施例中,标签或系链还可包含具有5'-马来酰亚胺基团和3'-DBCO基团的2kDa PEG。在一些实施例中,标签或系链还可包含具有5'-马来酰亚胺基团和3'-DBCO基团的3kDa PEG。在一些实施例中,标签或系链还可包含具有5'-马来酰亚胺基团和3'-DBCO基团的5kDa PEG。
标签或系链的其它实例包含但不限于His标签、生物素或链霉亲和素、与分析物结合的抗体、与分析物结合的适体、分析物结合结构域,如DNA结合结构域(包含例如肽拉链,如亮氨酸拉链、单链DNA结合蛋白(SSB))及其任何组合。
可以使用本领域已知的任何方法,将标签或系链连接到纳米孔的外表面,例如,在膜的顺式侧。例如,一种或多种标签或系链可以通过一种或多种半胱氨酸(半胱氨酸键)、一种或多种伯胺(如赖氨酸)、一种或多种非天然氨基酸、一种或多种组氨酸(His标签)、一种或多种生物素或链霉亲和素、一种或多种基于抗体的标签、表位的一种或多种酶修饰(包含例如乙酰转移酶)及其任意组合连接到纳米孔。用于进行此类修饰的合适方法在所属领域中是众所周知的。合适的非天然氨基酸包含但不限于4-叠氮基-L-苯丙氨酸(Faz),以及LiuC.C.和 Schultz P.G.,《生物化学年鉴(Annu.Rev.Biochem.)》,2010,79,413-444的图1中编号为1-71 的氨基酸中的任一种。
在一个或多个标签或系链通过半胱氨酸键连接到纳米孔的一些实施例中,可以将一种或多种半胱氨酸引入到通过取代形成纳米孔的一种或多种单体中。在一些实施例中,可以通过连接如下来对纳米孔进行化学修饰:(i)马来酰亚胺,包括二溴马来酰亚胺,如:4-苯氮霉素、 1.N-(2-羟乙基)马来酰亚胺、N-环己基马来酰亚胺、1.3-马来酰亚胺基丙酸、1.1-4-氨基苯基-1H- 吡咯,2,5,二酮、1.1-4-羟基苯基-1H-吡咯,2,5,二酮、N-乙基马来酰亚胺、N-甲氧基羰基马来酰亚胺、N-叔丁基马来酰亚胺、N-(2-氨基乙基)马来酰亚胺、3-马来酰亚胺基-PROXYL、N-(4-氯苯基)马来酰亚胺、1-[4-(二甲基氨基)-3,5-二硝基苯基]-1H-吡咯-2,5-二酮、N-[4-(2-苯并咪唑基) 苯基]马来酰亚胺、N-[4-(2-苯并恶唑基)苯基]马来酰亚胺、N-(1-萘基)马来酰亚胺、N-(2,4-二甲苯基)马来酰亚胺、N-(2,4-二氟苯基)马来酰亚胺、N-(3-氯-对-甲苯基)-马来酰亚胺、1-(2-氨基- 乙基)-吡咯-2,5-二酮盐酸盐、1-环戊基-3-甲基-2,5-二氢-1H-吡咯-2,5-二酮、1-(3-氨基丙基)-2,5- 二氢-1H-吡咯-2,5-二酮盐酸盐、3-甲基-1-[2-氧代-2-(哌嗪-1-基)乙基]-2,5-二氢-1H-吡咯-2,5-二酮盐酸盐、1-苄基-2,5-二氢-1H-吡咯-2,5-二酮、3-甲基-1-(3,3,3-三氟丙基)-2,5-二氢-1H-吡咯-2,5- 二酮、1-[4-(甲基氨基)环己基]-2,5-二氢-1H-吡咯-2,5-二酮三氟乙酸、SMILES O=C1C=CC(=O)N1CC=2C=CN=CC2、SMILES O=C1C=CC(=O)N1CN2CCNCC2、1-苄基-3-甲基-2,5-二氢-1H-吡咯-2,5-二酮、1-(2-氟苯基)-3-甲基-2,5-二氢1H-吡咯-2,5-二酮、N-(4-苯氧基苯基)马来酰亚胺、N-(4-硝基苯基)马来酰亚胺,(ii)碘代乙酰胺,如3-(2-碘乙酰氨基)-PROXYL、N-(环丙基甲基)-2-碘乙酰胺、2-碘-N-(2-苯乙基)乙酰胺、2-碘-N-(2,2,2-三氟乙基)乙酰胺、N-(4-乙酰基苯基)-2-碘代乙酰胺、N-(4-(氨基磺酰基)苯基)-2-碘代乙酰胺、N-(1,3- 苯并噻唑-2-基)-2-碘代乙酰胺、N-(2,6-二乙基苯基)-2-碘代乙酰胺、N-(2-苯甲酰基-4-氯苯基)-2- 碘代乙酰胺,(iii)溴代乙酰胺:如N-(4-(乙酰氨基)苯基)-2-溴代乙酰胺、N-(2-乙酰基苯基)-2- 溴代乙酰胺、2-溴-N-(2-氰基苯基)乙酰胺、2-溴-N-(3-(三氟甲基)苯基)乙酰胺、N-(2-苯甲酰基苯基)-2-溴代乙酰胺、2-溴-N-(4-氟苯基)-3-甲基丁酰胺、N-苄基-2-溴-N-苯基丙酰胺、N-(2-溴- 丁酰基)-4-氯-苯磺酰胺、2-溴-N-甲基-N苯基乙酰胺、2-溴-N-苯乙基-乙酰胺、2-金刚烷-1-基-2- 溴-N-环己基-乙酰胺、2-溴-N-(2-甲基苯基)丁酰胺、乙酰替对溴苯胺,(iv)二硫化物,如: ALDRITHIOL-2、ALDRITHIOL-4、异丙基二硫化物、1-(异丁基二硫烷基)-2-甲基丙烷、二苄基二硫化物、4-氨基苯基二硫化物、3-(2-吡啶基二硫代)丙酸酸、3-(2-吡啶基二硫代)丙酸酰肼、 3-(2-吡啶基二硫代)丙酸N-琥珀酰亚胺酯、am6amPDP1-βCD;以及(v)硫醇,如:4-苯基噻唑-2-硫醇、Purpald、5,6,7,8-四氢-喹唑啉-2-硫醇。
在一些实施例中,标签或系链可以直接或通过一个或多个接头连接到纳米孔。可以使用 WO 2010/086602中描述的杂交接头将标签或系链连接到纳米孔。可替代地,可以使用肽接头。肽接头是氨基酸序列。肽接头的长度、柔性和亲水性通常被设计为使得其不干扰单体和孔的功能。优选的柔性肽接头是2个到20个,如4个、6个、8个、10个或16个丝氨酸和/或甘氨酸的延伸段。更优选的柔性接头包含(SG)
跨膜孔可以被修饰以增强对通过聚合酶的多核苷酸和/或处理反应的副产物的捕获。例如,孔可以被修饰以增加孔入口内和/或孔的筒内的正电荷。此类修饰是所属领域中已知的。例如, WO 2010/055307公开了α-溶血素中的突变,其增加孔的筒体内的正电荷。
WO 2012/107778、WO 2013/153359和WO 2016/034591中分别公开了包含增强多核苷酸捕获的突变的经修饰的MspA、胞溶素和CsgG孔。这些出版物中公开的任何经修饰的孔均可以在本文中使用。
在一些实施例中,可以将CsgG纳米孔修饰为包含如本文所述的一个或多个标签或系链。可以在以下位置通过氨基酸修饰将一个或多个标签或系链连接到CsgG纳米孔的一个或多个单体(例如,1、2、3、4、5、6、7、8、9个或更多个):SEQ ID NO:7的T3、K7、R11、Q19、 K22、A29、T31、R76、N102、G103、N108、R110、Q114、E170、C215、L216、D238、A243、 D248和H255。在一些实施例中,可以在以下位置通过氨基酸取代将一个或多个标签或系链连接到CsgG纳米孔的一个或多个单体(例如,1、2、3、4、5、6、7、8、9个或更多个): SEQ ID NO:7的T3C、K7C、R11C、Q19C、K22C、A29C、T31C、R76C,E170C、D238C、 A243C、D248C、H255C、C215A/T/S/M/G/I/L、L216V。
在一些实施例中,CsgG纳米孔可以进一步被修饰以改善通过纳米孔的分析物(例如,通过聚合酶处理反应的副产物)的捕获和/或易位,以改善分析物的识别或辨别力,以改善与多核苷酸解链酶的相互作用和/或提高信噪比。例如,在一些实施例中,形成CsgG纳米孔的单体中的至少一种单体可以包括如WO 2016/034591中所公开的一种或多种突变。
在一些实施例中,CsgG纳米孔可包含以下氨基酸取代的组合之一(相对于SEQ IDNO: 7):(T3C);(K7C);(R11C);(Q19C);(K22C);(A29C);(T31C);(R76C);(E170C);(D238C);(A243C);(D248C);(H255C);(C215A);(C215T);(C215S);(C215M);(C215G);(C215I);(C215L);(C215A、L216V);(A29C、C215T);(T31C、C215T);(R76C、C215T);(T3C、C215A);(K7C、C215A);(R11C、C215A);(Q19C、C215A);(K22C、C215A);(A29C、C215A);(T31C、C215A);(R76C、C215A);(E170C、C215A);(C215A、D238C);(C215A、A243C);(C215A、 D248C);(C215A、H255C);(R76C、N91R、C215A);(R76C、N91R、C215A);(R76C、C215A);和(R76C、C215T)。
多核苷酸多核苷酸
多核苷酸可以是核酸,如脱氧核糖核酸(DNA)或核糖核酸(RNA)。多核苷酸可包含与一个DNA链杂交的一个RNA链。多核苷酸可以是本领域中已知的任何合成核酸,如肽核酸(PNA)、甘油核酸(GNA)、苏糖核酸(TNA)、锁核酸(LNA)或具有核苷酸侧链的其它合成聚合物。PNA主链由通过肽键连接的重复N-(2-氨基乙基)-甘氨酸单元构成。GNA主链由通过磷酸二酯键连接的重复二醇单元构成。TNA主链由通过磷酸二酯键连接在一起的重复苏糖构成。LNA由如上文所论述的具有额外的连接核糖部分中的2'氧和4'碳的桥的核糖核苷酸形成。
多核苷酸优选是DNA、RNA或DNA或RNA杂交体,最优选是DNA。靶多核苷酸可以是双链。靶多核苷酸可以包含单链区和具有其它结构的区域,例如发夹环、三链体和/或四链体。DNA/RNA杂交体可以在同一条链上包含DNA和RNA。优选地,DNA/RNA杂交体包含与RNA链杂交的一条DNA链。
在一些实施例中,靶多核苷酸不包含连接模板和互补序列的发夹结构或任何共价连接。在一些实施例中,靶多核苷酸(例如,模板)和与靶多核苷酸互补的多核苷酸(例如,补体) 不通过桥接部分(如发夹环)连接。
在一些公开的方法中,单链(例如,模板或补体)通过纳米孔易位,并且由于其两端的衔接子的相互作用,链本身可以形成发夹结构。参见例如,图19。这种衔接子设计可以有益于表征长多核苷酸,例如,通过维持链的另一端靠近纳米孔。
在一些实施例中,靶多核苷酸可以是任何长度。例如,多核苷酸的长度可以是至少10个、至少50个、至少100个、至少150个、至少200个、至少250个、至少300个、至少400个或至少500个核苷酸或核苷酸对。靶多核苷酸可以是1000个或更多个核苷酸或核苷酸对,长度为5000个或更多个核苷酸或核苷酸对或长度为100000个或更多个核苷酸或核苷酸对或长度为500,000个或更多个核苷酸或核苷酸对,或长度为1,000,000个或更多个核苷酸或核苷酸对,长度为10,000,000或更多个核苷酸或核苷酸对,或长度为100,000,000或更多个核苷酸或核苷酸对,或长度为200,000,000或更多个核苷酸或核苷酸对,或染色体的整个长度。靶多核苷酸可以是寡核苷酸。寡核苷酸是短核苷酸聚合物,其通常具有50个或更少核苷酸,如40 个或更少、30个或更少、20个或更少、10个或更少或5个或更少核苷酸。靶寡核苷酸的长度优选是约15至约30个核苷酸,例如长度是约20至约25个核苷酸。例如,寡核苷酸的长度可以是约15、约16、约17、约18、约19、约20、约21、约22、约23、约24、约25、约 26、约27、约28、约29或约30个核苷酸。
靶多核苷酸可以是较长的靶标多核苷酸的片段。在该实施例中,较长的靶多核苷酸通常片段化为多个,例如两个或更多个较短的靶多核苷酸。本发明的方法可用于对那些较短靶多核苷酸中的一个或多个,例如2、3、4、5个或更多个测序。
在一些实施例中,本文所述各个方面的方法可用于对样品内的多个靶多核苷酸,例如2、 3、4或5个至10、15、20个或更多个多核苷酸取样。
在一些实施例中,本文所述各个方面的方法可用于为样品中以双链形式存在的多核苷酸测序。
在一些实施例中,本文所述各个方面的方法可用于通过首先合成单链多核苷酸的互补序列以产生双链多核苷酸来为单链多核苷酸测序。例如,单链多核苷酸可以是RNA,如mRNA,并且可以合成互补cDNA链以产生双链多核苷酸,用于在本发明的方法中进行测序。例如,单链多核苷酸可以是DNA,并且可以合成补体链以产生双链DNA多核苷酸,用于在本发明的方法中进行测序。
在一些实施例中,多核苷酸可以是串接的多核苷酸。串接多核苷酸的方法在 PCT/GB2017/051493中有描述。在一个实施例中,用于将多核苷酸连接在一起的连接方法是点击化学。在该实施例中,当模板和互补序列不共价连接时,使用纳米孔表征第一双链多核苷酸的模板(捕获的第一链)和互补序列(第一链的反向互补序列)。当模板和互补序列分离时,与连接到第一双链多核苷酸的跟随衔接子中的孔系链互补的序列在互补序列中暴露,并且互补序列结合连接到纳米孔的孔系链。在一些实施例中,串接衔接子也连接到第一双链多核苷酸,使得补体链可以串接到第二双链多核苷酸。
在一些公开的方面,可以制备含有马达蛋白和释放蛋白的串接衔接子复合物。此串接衔接子可以连接到靶多核苷酸的两端。马达蛋白和释放蛋白都可以在连接的衔接子复合物上停滞,直到多核苷酸被孔捕获。一旦捕获了第一多核苷酸,阻断化学物质就被两种蛋白质克服,并且马达蛋白如前所述控制多核苷酸与孔的相互作用。释放蛋白,可以比马达蛋白更快地易位,到达第一多核苷酸的3'以释放与串接衔接子复合物的前导链的5'核酸序列互补的杂交位点。通过显露该杂交位点,第二多核苷酸然后可以与显露的位点杂交,并且第一多核苷酸的 3'末端与第二多核苷酸的5'的共价偶联可以发生(图43)。然后重复该过程以进一步连接靶多核苷酸。
在一个实施例中,提供了表征和串接双链靶多核苷酸的方法,其中连接方法是非共价的。在该实施例中,第一双链靶多核苷酸的补体链募集第二双链靶多核苷酸并使其达到孔的局部浓度。反过来,当对第一补体链测序时,募集的第二双链靶多核苷酸变得从补体链去杂交并替代地与孔系链杂交。这使得第一和第二(以及随后的,第三、第四、第五等)双链靶多核苷酸能够依次被处理,链间隔时间最短。当双链靶多核苷酸的浓度较低时,这特别有用,因为第二靶多核苷酸可以在为第一靶多核苷酸测序时募集。
在另一个实施例中,表征和串接双链靶多核苷酸的方法(例如其中连接方法是非共价的),可以使用双组分捕捞系链进行,所述双组分捕捞系链为跟随序列和孔系链提供第二杂交位点,以增加所见事件的比例。
在一个实施例中,表征和串接许多双链靶多核苷酸的方法(例如其中连接方法是非共价的),可用于将多个,例如2至20个,例如4、5、6、8、10、12或15个双链靶多核苷酸带到孔处。第一双链靶多核苷酸的补体链可以募集许多其它双链靶多核苷酸并使其集中在孔附近。这样在孔周围提供比在一般本体溶液中更高的局部浓度,因此双链靶多核苷酸依次被处理,链间隔时间最短。当双链靶多核苷酸的浓度较低时,这尤其有用。在该实施例中,可以使用由与单链结合蛋白偶联的寡核苷酸组成的系链。当对第一双链靶多核苷酸的模板链进行测序时,补体链作为ssDNA释放到溶液中。其它双链靶多核苷酸的单链结合蛋白能够与ssDNA 结合。作为跟随过程的一部分,当对补体链进行测序时,补体链的3'被拉回到孔中。ssDNA 补体链上的单链结合蛋白在遇到控制补体的处理的蛋白时从补体链中移位,并且因此沉积在孔周围,增加局部浓度。这在图44中描绘。如果靶多核苷酸的序列是已知的,则可以进行这种分析物拖网,但也可以将互补序列添加到孔系链的3',其可以用于平铺补体链的区段。
样品
分析物(包括,例如蛋白质、肽、分子、多肽、多核苷酸)可以存在于样品中。样品可以是任何合适的样品。样品可以是生物样品。可以在体外对从任何生物体或微生物获得或提取的样品进行本文所述方法的任何实施例。生物体或微生物通常是太古细菌、原核或真核微生物,且通常属于以下五界中的一个:植物界、动物界、真菌界、原核生物界和原生生物界。在一些实施例中,可以对从任何病毒中获得或提取的样品在体外执行本文所述各个方面的方法。
样品优选是流体样品。样品通常包含体液。体液可获自人类或动物。人类或动物可能患有、被怀疑患有或处于疾病风险中。样品可以是尿液、淋巴液、唾液、粘液、精液或羊水,但优选全血、血浆或血清。通常,样品是人类来源的,但是可替代地,它可以来自另一种哺乳动物,例如来自商业养殖的动物,例如马、牛、绵羊或猪,或者可替代地可以为宠物,例如猫或狗。
可替代地,植物来源的样品通常获自商业作物,例如谷物、豆类、水果或蔬菜,例如小麦、大麦、燕麦、油菜、玉米、大豆、大米、香蕉、苹果、番茄、马铃薯、葡萄、烟草、豆、扁豆、甘蔗、可可、棉花、茶或咖啡。
样品可以是非生物样品。非生物样品优选是流体样品。非生物样品的实例包括手术液;水,如饮用水、海水或河水;以及实验室测试用试剂。
可以在分析前处理样品,例如通过离心或通过膜,所述膜过滤掉不需要的分子或细胞,如红血细胞。样品可以在获取之后立即测量。通常还可以在分析之前,优选在低于-70℃下储存样品。
在一些实施例中,样品可以包括基因组DNA。基因组DNA可以被片段化,或者本文所述任何方法还可包括使基因组DNA片段化。可以通过任何合适的方法使DNA片段化。例如,DNA的片段化方法是本领域中已知的。此类方法可以使用转座酶,例如MuA转座酶或可商购获得的G-管。
前导序列
前导序列通常包含聚合物。聚合物优选带负电荷。聚合物优选是多核苷酸,例如DNA或 RNA、修饰的多核苷酸(例如脱碱基DNA)、PNA、LNA、聚乙二醇(PEG)或多肽。前导优选包含多核苷酸,并且更优选包含单链多核苷酸。单链前导序列最优选包含DNA的单链,例如poly dT区段。前导序列优选地包含一个或多个间隔子。
前导序列可以是任何长度,但其长度通常是10到150个核苷酸,例如20到150个核苷酸。前导的长度通常取决于方法中使用的跨膜孔。
前导序列优先旋入跨膜孔中,并且从而促进多核苷酸通过孔的移动。前导序列还可以用于将多核苷酸连接到如本文所论述的一个或多个锚。
通常,前导序列存在于靶多核苷酸的一端和与靶多核苷酸互补的多核苷酸的一端。前导序列可以存在于靶多核苷酸的5'末端和靶多核苷酸的互补序列的5'末端。可替代地,前导序列可以存在于靶多核苷酸的3'末端和靶多核苷酸的互补序列的3'末端。
前导序列可以存在于靶多核苷酸的5'末端和互补多核苷酸的3'末端,或反之亦然。在这些公开的实施例中,通常使用两种不同的多核苷酸结合蛋白(例如,多核苷酸解链酶),其中第一多核苷酸结合蛋白(例如,多核苷酸解链酶)沿着多核苷酸在5'到3'方向上移动,并且第二多核苷酸结合蛋白(例如,多核苷酸解链酶)沿着多核苷酸在3'到5'方向上移动。
前导序列可以通过任何合适的方法连接到双链多核苷酸。例如,前导序列可以与靶多核苷酸和/或其互补序列连接。替代性地,可以通过消化双链多核苷酸的一条链以在另一条链上产生单链突出端来产生前导序列。
多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶)可以在其与靶多核苷酸或其补体连接之前与前导序列结合。多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶)可以与双链多核苷酸中存在的前导序列结合。与前导序列结合的多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶)的活性可以停滞,直到多核苷酸接触跨膜孔。使多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶)停滞的方法是本领域已知的,例如在WO2014/135838中。
衔接子
前导序列可以存在于衔接子中,其中所述衔接子包含双链区(例如,双链体茎)和至少一个单链区。单链区中的至少一个可以是前导序列。衔接子可以包含至少一个非多核苷酸区域。连接到靶双链多核苷酸的两个末端的衔接子可以相同或不同。优选地,该对中的衔接子是相同的。
前导序列优选存在于衔接子的一条链的5'末端(或3'末端)的第一单链区中。第二单链区可以存在于衔接子另一条链的3'末端(或5'末端)。第一和第二单链区不互补。在该实施例中,衔接子可以称为Y衔接子。
Y衔接子通常包含(a)双链区(例如,双链体茎)和(b)单链区或在另一端不互补的区域。如果Y衔接子包含单链区,则可以将其描述为具有突出端。Y衔接子中非互补区的存在赋予了衔接子Y形状,因为两条链通常不彼此杂交,不同于双链部分。Y衔接子可以包含一个或多个锚。
在一些实施例中,所述衔接子可包含一个或多个(例如,至少一个、至少两个、至少三个或更多个)用于纳米孔上的一个或多个(例如,至少一个、至少两个、至少三个或更多个) 标签的结合位点。在一些实施例中,用于纳米孔上的标签的结合位点可以是在双链区(例如,双链体茎)内,使得在双链区的两条链分离时结合位点被暴露。参见,例如,图10。另外或可替代地,用于纳米孔上的标签的结合位点可以是在衔接子的单链部分上。仅作为实例,图 9A显示了包含至少一个用于固体基质(例如,膜或珠粒)的锚的示例衔接子,而图15显示了包含至少两个锚的示例衔接子,其中第一锚能够偶联到固体基质,例如,膜或珠粒,并且第二锚能够偶联到纳米孔。纳米孔的第二锚可以被配置成结合到与纳米孔缀合的标签。
Y衔接子包括可以螺旋孔中的前导序列。
Y衔接子可以使用本领域已知的任何方法连接到多核苷酸。例如,可以使用连接酶,如T4 DNA连接酶、大肠杆菌DNA连接酶、Taq DNA连接酶、Tma DNA连接酶以及9°N DNA 连接酶,来连接衔接子中的一个或两个。
在一个优选实施例中,修饰双链多核苷酸,例如样品中的双链多核苷酸,使得它们在两端包含Y衔接子。可以使用任何修饰方式。该方法可包括通过添加衔接子来修饰双链靶多核苷酸。
通过使多核苷酸与MuA转座酶和一群双链MuA底物接触,可以向双链多核苷酸提供衔接子,如Y衔接子或锚。转座酶将双链多核苷酸片段化并且将MuA底物连接到片段的一端或两端。这样就产生了多个修饰后的双链多核苷酸,其包含衔接子或锚。然后可以使用本发明的方法研究修饰后的双链多核苷酸。
这些基于MuA的方法公开于WO 2015/022544和WO 2016/059363中。还在 WO2015/150786中详细论述了这些方法。
所述衔接子还可包含锚,以将包含靶多核苷酸和/或其互补序列的双链多核苷酸拴系到跨膜孔或包含孔的膜上,即衔接子还可包含膜-系链或孔-系链。锚优选地连接到不是前导序列的单链区。
可以将多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶)结合到衔接子中的前导序列,或者可以在衔接子已经连接到双链多核苷酸之后添加多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶)。与前导序列结合的多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶)的活性可以停滞,直到多核苷酸接触跨膜孔。
前导序列或衔接子可以通过任何合适的方法连接到双链多核苷酸。例如,前导序列可以与靶多核苷酸和/或其互补序列连接,或者衔接子可以与双链多核苷酸连接。
在一些实施例中,双链条形码序列可以连接到靶双链多核苷酸的一端或两端。可以在添加前导序列或衔接子之前将条形码序列添加至双链多核苷酸。例如,条形码序列可位于靶双链多核苷酸的末端和衔接子之间。优选地,条形码序列包含在衔接子中。
可以将唯一的条形码序列连接,例如,连接到样品中的每个双链多核苷酸。条形码序列可以用于鉴定与通过聚合酶处理靶多核苷酸和与靶多核苷酸互补的多核苷酸所得的副产物通过孔的顺序易位相对应的信号。
衔接子可以包括一个或多个间隔子,以防止预结合的多核苷酸结合蛋白(例如,多核苷酸解链酶)处理双链多核苷酸。这些间隔子防止多核苷酸结合蛋白(例如,多核苷酸解链酶) 的移动,直到多核苷酸结合蛋白(例如,多核苷酸解链酶)定位在孔处并且在孔上施加电位差。由电位差提供的附加力将多核苷酸结合蛋白(例如,多核苷酸解链酶)推向间隔子,并允许其处理多核苷酸。因此,仅当多核苷酸在纳米孔中而不是之前时,多核苷酸结合蛋白(例如,多核苷酸解链酶)的移动才可能发生。用于防止预结合的多核苷酸结合蛋白(例如,多核苷酸解链酶)处理双链多核苷酸直到多核苷酸在纳米孔中的间隔子和方法的实例描述于例如WO2015/110813中,其内容通过引用以其全文并入本文。
条形码
多核苷酸条形码是本领域中众所周知的(Kozarewa,I.等人,(2011),《分子生物学方法 (Methods Mol.Biol.)》733,第279-298页)。条形码是多核苷酸的特定序列,其以特定且已知的方式影响流过孔的电流。
条形码可包含核苷酸序列。核苷酸通常含有核碱基、糖以及至少一个磷酸基。核碱基通常是杂环的。核碱基包括但不限于嘌呤和嘧啶,以及更具体地,腺嘌呤、鸟嘌呤、胸腺嘧啶、尿嘧啶以及胞嘧啶。糖通常是戊糖。核苷酸糖包括但不限于核糖和脱氧核糖。核苷酸通常是核糖核苷酸或脱氧核糖核苷酸。核苷酸通常含有单磷酸、二磷酸或三磷酸。磷酸可以连接在核苷酸的5'或3'侧上。
核苷酸包括但不限于:单磷酸腺苷(AMP)、二磷酸腺苷(ADP)、三磷酸腺苷(ATP)、单磷酸鸟苷(GMP)、二磷酸鸟苷(GDP)、三磷酸鸟苷(GTP)、单磷酸胸苷(TMP)、二磷酸胸苷(TDP)、三磷酸胸苷(TTP)、单磷酸尿苷(UMP)、二磷酸尿苷(UDP)、三磷酸尿苷 (UTP)、单磷酸胞苷(CMP)、二磷酸胞苷(CDP)、三磷酸胞苷(CTP)、5-甲基胞苷单磷酸、 5-甲基胞苷二磷酸、5-甲基胞苷三磷酸、5-羟甲基胞苷单磷酸、5-羟甲基胞苷二磷酸、5-羟甲基胞苷三磷酸、环单磷酸腺苷(cAMP)、环单磷酸鸟苷(cGMP)、单磷酸脱氧腺苷(dAMP)、二磷酸脱氧腺苷(dADP)、三磷酸脱氧腺苷(dATP)、单磷酸脱氧鸟苷(dGMP)、二磷酸脱氧鸟苷(dGDP)、三磷酸脱氧鸟苷(dGTP)、单磷酸脱氧胸苷(dTMP)、二磷酸脱氧胸苷(dTDP)、三磷酸脱氧胸苷(dTTP)、单磷酸脱氧尿苷(dUMP)、二磷酸脱氧尿苷(dUDP)、三磷酸脱氧尿苷(dUTP)、单磷酸脱氧胞苷(dCMP)、二磷酸脱氧胞苷(dCDP)和三磷酸脱氧胞苷(dCTP)、 5-甲基-2'-脱氧胞苷单磷酸、5-甲基-2'-脱氧胞苷二磷酸、5-甲基-2'-脱氧胞苷三磷酸、5-羟甲基 -2'-脱氧胞苷单磷酸、5-羟甲基-2'-脱氧胞苷二磷酸以及5-羟甲基-2'-脱氧胞苷三磷酸。衔接子中的核苷酸优选地选自AMP、TMP、GMP、UMP、dAMP、dTMP、dGMP或dCMP。核苷酸可以是无碱基的(即缺乏核碱基)。核苷酸可以含有额外修饰。具体地说,适当的经修饰核苷酸包含但不限于2'氨基嘧啶(如2'-氨基胞苷和2'-氨基尿苷)、2'-羟基嘌呤(如2'-氟嘧啶)(如 2'-氟胞苷和2'-氟尿苷)、羟基嘧啶(如5'-α-P-硼烷尿苷)、2'-O-甲基核苷酸(如2'-O-甲基腺苷、 2'-O-甲基鸟苷、2'-O-甲基胞苷和2'-O-甲基尿苷)、4'-硫代嘧啶(如4'-硫代尿苷和4'-硫代胞苷),并且核苷酸具有对核碱基的修饰(如5-戊炔基-2'-脱氧尿苷、5-(3-氨基丙基)-尿苷和1,6-二氨基己基-N-5-氨基甲酰基甲基尿苷)。
条形码可以包括一种或多种不同的核苷酸种类。例如,T k聚体(即其中中心核苷酸是基于胸腺嘧啶的k聚体,如TTA、GTC、GTG和CTA)通常具有最低的电流状态。在一些公开的方法中,可以将T核苷酸的修饰版本引入经修饰的多核苷酸中以进一步降低电流状态,并且由此增加当条形码移动通过孔时看到的总电流范围。
G k聚体(即其中中心核苷酸是基于鸟嘌呤的k聚体,如TGA、GGC、TGT和CGA)趋于受到k聚体中其它核苷酸的强烈影响,并且因此修饰了经修饰的多核苷酸中的G核苷酸,可以帮助它们具有更多独立的电流位置。
包括同一核苷酸种类而不是三个不同种类的三个拷贝可以促进表征,因为那样仅需要定位修饰后的多核苷酸中的例如3-核苷酸k聚体。但是,这种修饰确实减少了条形码提供的信息。
条形码中可包括一个或多个无碱基核苷酸。使用一个或多个无碱基核苷酸导致特征性电流尖峰。这允许清楚地突出条形码中一个或多个核苷酸种类的位置。
条形码中的核苷酸种类可以包含化学原子或基团,例如丙炔基、硫基、氧代基、甲基、羟甲基、甲酰基、羧基、羰基、苯甲基、炔丙基或炔丙胺基。化学基团或原子可以是或可以包含荧光分子、生物素、地高辛(digoxigenin)、二硝基苯酚(DNP)、光不稳定基团、炔烃、DBCO、叠氮化物、游离氨基、氧化还原染料、汞原子或硒原子。
条形码可以包含含有卤素原子的核苷酸种类。卤素原子可以连接到不同核苷酸种类,例如核碱基和/或糖上的任何位置。卤素原子优选地是氟(F)、氯(Cl)、溴(Br)或碘(I)。卤素原子最优选是F或I。
锚
靶多核苷酸可以使用锚(膜-系链)与膜偶联。可以使用一个或多个锚来将靶多核苷酸与膜偶联。通常,一个或多个锚连接到靶多核苷酸的每条链。锚可以是衔接子的一部分。
如果膜是如三嵌段共聚物膜等两亲层,则所述一个或多个锚优选包含可以被插入膜中的多肽锚和/或疏水性锚。疏水性锚优选是脂质、脂肪酸、固醇、碳纳米管、多肽、蛋白质或氨基酸,例如胆固醇、棕榈酸盐或生育酚。在优选实施例中,所述一个或多个锚不是孔。
膜组分,如两亲分子、共聚物或脂质,可以经过化学修饰或功能化而形成所述一个或多个锚。下文更详细地论述了合适的化学修饰和使膜组分功能化的合适方式的实例。可以对任何比例的膜组分进行功能化,例如至少0.01%、至少0.1%、至少1%、至少10%、至少25%、至少50%或100%。
一个或多个锚优选包含接头。一个或多个锚可以包含一个或多个,例如2、3、4个或更多个接头。
优选的接头包括但不限于聚合物,如多核苷酸、聚乙二醇(PEG)、多糖以及多肽。这些接头可以是线性、支链或环状的。例如,接头可以是环状多核苷酸。靶多核苷酸可以与环状多核苷酸接头上的互补序列杂交。
一个或多个锚或一个或多个接头可以包含能被切割或分解的组分,如限制位点或光不稳定基团。
功能化的接头和其与分子的偶联方式是本领域中已知的。例如,用马来酰亚胺基团功能化的接头将与蛋白质中的半胱氨酸残基反应并且与之连接。
可以使用“锁和钥”布置来避免多核苷酸的交联。每个接头只有一端可以一起反应形成更长的接头,并且接头的另一端各自分别与多核苷酸或膜反应。这种接头描述于WO2010/086602中。
在本发明的测序方法中,接头的使用是优选的。如果在与孔和/或与聚合酶相互作用时,多核苷酸是永久性地直接偶联到膜上,从某种意义上说所述多核苷酸不会解耦,则由于膜与孔之间的距离,测序运行不能继续到多核苷酸的端,因此一些序列数据将丢失。如果使用接头,则多核苷酸可以得到完全处理。
偶联可以是永久的或稳定的。换句话说,偶联可以使得多核苷酸在与孔和/或与聚合酶相互作用时可以与膜解耦。
偶联可以是暂时的。换句话说,偶联可以使得多核苷酸在与孔相互作用时可以与膜解耦。对于多核苷酸测序,暂时性质的偶联是优选的。如果永久性或稳定接头直接连接到多核苷酸的5'或3'端并且接头比膜与跨膜孔的通道之间的距离短,则因为该轮测序不能持续到多核苷酸的末端,所以一些序列数据将丢失。如果偶联是暂时的,则当偶联的末端随机摆脱膜时,多核苷酸就可以得到完全处理。形成永久性/稳定或暂时连接的化学基团在下文更详细地论述。可以使用胆固醇或脂肪酰基链,将靶多核苷酸和/或其互补序列暂时与膜,如两亲层,例如三嵌段共聚物膜或脂质膜偶联。可以使用长度为6至30个碳原子的任何脂肪酰基链,如十六烷酸。
在优选实施例中,锚将靶多核苷酸和/或其互补序列与两亲层,如三嵌段共聚物膜或脂质双层偶联。先前已用各种不同的系链策略进行了核酸与合成脂质双层的偶联。这些汇总在下文表1中。
表1
合成多核苷酸和/或接头可以在合成反应中使用修饰后的亚磷酰胺来功能化,所述修饰后的亚磷酰胺容易与直接加入的合适的锚定基团相容,所述锚定基团例如胆固醇、生育酚、棕榈酸盐、硫醇、脂质以及生物素基团。这些不同的连接化学为多核苷酸的连接提供了一套选择。每个不同的修饰基团以稍微不同的方式偶联多核苷酸,并且偶联未必总是永久性的,因此赋予多核苷酸在膜上不同的停留时间。
多核苷酸与接头或与功能化膜的偶联还可以通过多种其它手段来实现,条件是可以向多核苷酸中加入互补反应性基团或锚定基团。先前已经报道了向多核苷酸的任一末端加入反应性基团。可使用T4多核苷酸激酶和ATPγS向ssDNA或dsDNA的5'中添加巯基(Grant,G.P. 和P.Z.Qin(2007).“一种用于连接在核酸的5'端处的氮氧化物自旋标记的便捷方法(A facile method for attaching nitroxide spin labels at the 5'terminusof nucleic acids)”.《
所述一个或多个锚可以通过杂交将靶多核苷酸和/或其互补序列与膜偶联。杂交可以是在一个或多个锚与靶多核苷酸和/或其互补序列之间,在一个或多个锚内或在一个或多个锚与膜之间。如上文所论述的,在一个或多个锚中的杂交允许以暂时方式进行偶联。举例来说,接头可以包含两个或更多个杂交在一起的多核苷酸,如3、4或5个多核苷酸。所述一个或多个锚可以与靶多核苷酸或与靶多核苷酸互补的多核苷酸杂交。所述一个或多个锚可以直接与连接到靶多核苷酸和/或其互补序列的Y衔接子和/或前导序列杂交。可替代地,所述一个或多个锚可以与一个或多个,如2或3个中间多核苷酸(或“夹板”)杂交,所述中间多核苷酸与多核苷酸,与连接到靶多核苷酸和/或其互补序列的Y衔接子和/或前导序列杂交。
所述一个或多个锚可以包含单链或双链多核苷酸。锚的一部分可以连接到单链或双链多核苷酸分析物上。已经报道过使用T4 RNA连接酶I来连接ssDNA的短片(Troutt,A.B.,M.G. McHeyzer-Williams等人(1992).“接合锚定的PCR:具有单侧特异性的简单扩增技术 (Ligation-anchored PCR:a simple amplification technique with single-sided specificity)”.《美国国家科学院院刊》89(20):9823-5)。替代地,单链或双链多核苷酸可与双链多核苷酸接合,且接着两个链通过热或化学变性分开。对于双链多核苷酸,可以将一片单链多核苷酸加入到双链体的一端或两端,或将双链多核苷酸加入到一端或两端。为了将单链多核苷酸加入到双链多核苷酸中,可以使用T4 RNA连接酶I来实现与单链多核苷酸的其它区域的连接。为了将双链多核苷酸加入到双链多核苷酸中,则连接可以是“平端的”,其中互补的3'dA/dT尾分别在多核苷酸和所加入的多核苷酸上(这是许多样品制备应用的常规做法,用以防止多联体或二聚体形成),或使用通过限制性消化多核苷酸和连接相容的衔接子生成的“粘末端”。接着,当双链体解链时,如果使用单链多核苷酸进行连接,则每条单链将具有5'或3'端修饰;或者如果使用双链多核苷酸进行连接,则每条单链将在5'端、3'端或两端具有修饰。
如果衔接子或靶多核苷酸的互补序列是合成链,则可以在衔接子或互补序列化学合成期间掺入所述一个或多个锚。例如,可以使用有反应性基团连接到其上的引物来合成衔接子或互补序列。
腺苷酸化多核苷酸是连接反应中的中间物,其中单磷酸腺苷连接至多核苷酸的5'-磷酸。可获得各种用于生成这种中间物的试剂盒,例如来自NEB的5'DNA腺苷酰化试剂盒。通过用反应中的ATP取代修饰后的三磷酸核苷酸,就可以向多核苷酸的5'加入反应性基团(如硫醇、胺、生物素、叠氮化物等)。还可以使用具有适当修饰的核苷酸(例如胆固醇或棕榈酸盐)的 5'DNA腺苷酰化试剂盒将锚直接加入到多核苷酸中。
用于扩增基因组DNA区段的常见技术是使用聚合酶链式反应(PCR)。此处,使用两个合成寡核苷酸引物,可产生大量相同DNA片段的拷贝,其中对于每个拷贝,双螺旋中的每个链的5'将是合成多核苷酸。可以通过采用聚合酶将单个或多个核苷酸加入到单链或双链DNA 的3'端。可以使用的聚合酶的实例包括但不限于末端转移酶、Klenow以及大肠杆菌Poly(A) 聚合酶。通过用反应中的ATP取代修饰后的三磷酸核苷酸,就可以将锚,如胆固醇、硫醇、胺、叠氮化物、生物素或脂质,掺入到双链多核苷酸中。因此,所扩增多核苷酸的每个拷贝都将含有锚。
理想地,多核苷酸与膜偶联而不必使多核苷酸功能化。这可以通过将一个或多个锚,如多核苷酸结合蛋白(例如,多核苷酸解链酶)或化学基团,与膜偶联并且使所述一个或多个锚与多核苷酸相互作用或通过使膜功能化来实现。所述一个或多个锚可以通过本文所述的任何方法与膜偶联。具体来说,所述一个或多个锚可以包含一个或多个接头,如马来酰亚胺功能化的接头。在这个实施例中,多核苷酸通常是RNA、DNA、PNA、TNA或LNA,并且可以是双链的或单链的。这个实施例尤其适合于基因组DNA多核苷酸。
一个或多个锚可包含,与单链或双链多核苷酸、多核苷酸内的特异性核苷酸序列或多核苷酸内的修饰核苷酸的图案或存在于多核苷酸上的任何其它配体偶联、结合或与相互作用的任何基团。
适合用于锚中的结合蛋白包括但不限于:大肠杆菌单链结合蛋白、P5单链结合蛋白、T4 gp32单链结合蛋白、TOPO V dsDNA结合区、人类组蛋白蛋白质、大肠杆菌HU DNA结合蛋白,以及其它古细菌的、原核生物的或真核生物的单链或双链多核苷酸(或核酸)结合蛋白,包括下文所列的那些。
特异性核苷酸序列可以是由转录因子、核糖体、核酸内切酶、拓扑异构酶(topoisomerase) 或复制起始因子识别的序列。修饰后的核苷酸的模式可以是甲基化模式或损伤模式。
一个或多个锚可包含与多核苷酸偶联、结合、插入或相互作用的任何基团。基团可以通过静电、氢键或范德华(Van der Waals)相互作用而插入多核苷酸中或与多核苷酸相互作用。此类基团包括赖氨酸单体、聚赖氨酸(其将与ssDNA或dsDNA相互作用)、溴化乙锭(其插入dsDNA中)、通用碱基或通用核苷酸(其可以与任何多核苷酸杂交)以及锇络合物(其可以与甲基化碱基反应)。因此,多核苷酸可以使用连接至膜的一个或多个通用核苷酸而与膜偶联。每个通用核苷酸可使用一个或多个接头而与膜偶联。通用核苷酸优选地包含以下核碱基中的一个:次黄嘌呤、4-硝基吲哚、5-硝基吲哚、6-硝基吲哚、甲酰基吲哚、3-硝基吡咯、硝基咪唑、4-硝基吡唑、4-硝基苯并咪唑、5-硝基吲唑、4-氨基苯并咪唑或苯基(C6-芳环)。通用核苷酸更优选地包含以下核苷中的一个:2'-脱氧肌苷、肌苷、7-脱氮-2'-脱氧肌苷、7-脱氮- 肌苷、2-氮杂-脱氧肌苷、2-氮杂-肌苷、2-O'-甲基肌苷、4-硝基吲哚2'-脱氧核苷、4-硝基吲哚核苷、5-硝基吲哚2'-脱氧核苷、5-硝基吲哚核苷、6-硝基吲哚2'-脱氧核苷、6-硝基吲哚核苷、 3-硝基吡咯2'-脱氧核苷、3-硝基吡咯核苷、次黄嘌呤的非环糖类似物、硝基咪唑2'-脱氧核苷、硝基咪唑核苷、4-硝基吡唑2'-脱氧核苷、4-硝基吡唑核苷、4-硝基苯并咪唑2'-脱氧核苷、4- 硝基苯并咪唑核苷、5-硝基吲唑2'-脱氧核苷、5-硝基吲唑核苷、4-氨基苯并咪唑2'-脱氧核苷、 4-氨基苯并咪唑核苷、苯基C-核苷、苯基C-2'-脱氧核糖基核苷、2'-脱氧烟云杯伞素、2'-脱氧异鸟苷、K-2'-脱氧核苷、P-2'-脱氧核苷和吡咯烷。通用核苷酸更优选地包含2'-脱氧肌苷。通用核苷酸更优选地是IMP或dIMP。通用核苷酸最优选地是dPMP(2'-脱氧-P-核苷单磷酸)或 dKMP(N6-甲氧基-2,6-二氨基嘌呤单磷酸)。
一个或多个锚可通过Hoogsteen氢键(其中两个核碱基通过氢键保持在一起)或反向 Hoogsteen氢键(其中一个核碱基相对于其它核碱基旋转180°)与多核苷酸偶联(或结合)。举例来说,一个或多个锚可包含与多核苷酸形成Hoogsteen氢键或反向Hoogsteen氢键的一个或多个核苷酸、一个或多个寡核苷酸或一个或多个多核苷酸。这些氢键类型允许第三多核苷酸链卷绕双链螺旋于周围,且形成三链螺旋。一个或多个锚可以通过与双链双链体形成三链体而与双链多核苷酸偶联(或结合)。
在此实施例中,可以使至少1%、至少10%、至少25%、至少50%或100%的膜组分功能化。
当一个或多个锚包含蛋白质时,所述一个或多个锚可能能够直接锚定至膜而无需进一步功能化,例如当其已具有与膜相容的外部疏水区时。此类蛋白质的实例包括但不限于跨膜蛋白、膜内蛋白以及膜蛋白。替代地,可表达具有与膜相容的基因融合疏水区的蛋白质。这种疏水性蛋白质区域是本领域中已知的。
一个或多个锚优选在递送到膜之前与多核苷酸混合,但是一个或多个锚可以与膜接触并且随后与多核苷酸接触。
另一方面,可以使用上文所述的方法使多核苷酸功能化,使得其可以被特异性结合基团识别。具体地说,多核苷酸可以用配体进行官能化,所述配体如,生物素(用于结合于抗生蛋白链菌素)、直链淀粉(用于结合于麦芽糖结合蛋白或融合蛋白)、Ni-NTA(用于结合于聚组氨酸或聚组氨酸标签的蛋白质)或肽(如抗原)。
根据优选的实施例,一个或多个锚可以用于当多核苷酸连接到前导序列时将多核苷酸与膜偶联,所述前导序列可以螺旋到孔中。优选地,多核苷酸与前导序列连接(如连接),所述前导序列可以螺旋到孔中。这种前导序列可以包括同聚多核苷酸或无碱基区。前导序列通常设计成直接与一个或多个锚杂交,或通过一个或多个中间多核苷酸(或夹板)与所述一个或多个锚杂交。在这些情况下,一个或多个锚通常包含与前导序列中的序列或一个或多个中间多核苷酸(或夹板)中的序列互补的多核苷酸序列。在这些情况下,一个或多个夹板通常包含与前导序列中的序列互补的多核苷酸序列。
上文论述的任何用于使多核苷酸与例如两亲层的膜偶联的方法当然可以应用于其它多核苷酸和膜组合。在一些实施例中,氨基酸、肽、多肽或蛋白质与例如三嵌段共聚物层或脂质双层的两亲层偶联。可以获得各种用于这类多核苷酸的化学连接的方法。化学连接中使用的分子的实例是EDC(1-乙基-3-[3-二甲氨基丙基]碳化二亚胺盐酸盐)。还可以使用市售试剂盒 (Thermo Pierce,产品号22980)向多核苷酸的5'中加入反应性基团。合适的方法包括但不限于使用组氨酸残基和Ni-NTA的暂时亲和力连接,以及通过反应性半胱氨酸、赖氨酸或非天然氨基酸的更稳健的共价连接。
微颗粒
微粒(通常为珠粒)可用于将分析物(例如,多核苷酸或多肽)递送至跨膜孔。这在WO 2016/059375中有描述,其内容通过引用整体并入本文。可以在本发明的方法中使用任何数量的微粒。例如,本发明的方法可以使用单个微粒或2个、3个、4个、5个、6个、7个、8 个、9个、10个、20个、30个、50个、100个、1,000个、5,000个、10,000个、100,000个、500,000个或1,000,000个或更多个微粒。如果使用两个或更多个微粒,则微粒可以是相同的。或者,可以使用不同微粒的混合物。
每个微粒可以连接有一个分析物(例如,多核苷酸或多肽)。可替代地,每个微粒可以连接有两个或更多个分析物(例如,多核苷酸或多肽),如3个或更多个、4个或更多个、5个或更多个、6个或更多个、7个或更多个、8个或更多个、9个或更多个、10个或更多个、20 个或更多个、30个或更多个、50个或更多个、100个或更多个、500个或更多个、1,000个或更多个、5,000个或更多个、10,000个或更多个、100,000个或更多个、1000,000个或更多个、或5000,000个或更多个分析物(例如,多核苷酸或多肽)。微粒可以基本上或完全被分析物(例如,多核苷酸或多肽)涂布或覆盖。微粒可以在其基本上全部或全部表面上连接有分析物(例如,多核苷酸或多肽)。微粒可以通过衔接子连接到分析物(例如,多核苷酸或多肽)。衔接子可以是Y衔接子,例如,如图36所示。
合适的结合部分的实例包括:蛋白质结合标签(链霉抗生物素蛋白标签、flag标签等)、缀合连接(多核苷酸、聚合物、生物素、肽)以及氨基酸(半胱氨酸、Faz等)。
在一些实施例中,多核苷酸可以连接到两个或更多个微粒。
微粒是微观颗粒,其大小通常以微米(μm)为单位测量。微粒也可以被称为微球或微珠。微粒可以是纳米颗粒。纳米颗粒是微观颗粒,其大小通常以纳米(nm)为单位测量。
微粒通常具有约0.001μm至约500μm的粒径。例如,纳米颗粒可以具有约0.01μm至约 200μm或约0.1μm至约100μm的粒径。更常见的是,微粒具有约0.5μm至约100μm,或例如约1μm至约50μm的粒径。微粒可以具有约1nm至约1000nm,如约10nm至约500nm、约20nm至约200nm或约30nm至约100nm的粒径。
微粒可以是球形或非球形。球形微粒可以被称为微球。非球形颗粒可以是例如板状、针状、不规则状或管状。如本文所使用的术语“粒径”意指颗粒的直径,如果颗粒是球形的,或者如果颗粒是非球形的,则意指基于体积的粒径。基于体积的粒径是与所论述的非球形颗粒具有相同体积的球体的直径。
如果在所述方法中使用两个或更多个微粒,则微粒的平均粒径可以是上文所论述的任何大小,如约0.5μm至约500μm。两个或更多个微粒的群体优选具有10%或更小,如5%或更小或2%或更小的变异系数(标准差与平均值的比率)。
可以使用任何方法来确定微粒的大小。合适的方法包括但不限于流式细胞术(参见例如 Chandler等人,《血栓形成与止血杂志(J Thromb Haemost.)》2011年6月;9(6):1216-24)。
微粒可以由任何材料形成。微粒优选由陶瓷、玻璃、二氧化硅、聚合物或金属形成。聚合物可以是天然聚合物,如聚羟基烷酸酯、葡聚糖、聚丙交酯、琼脂糖、纤维素、淀粉或壳聚糖,或合成聚合物,如聚氨酯、聚苯乙烯、聚(氯乙烯)、硅烷或甲基丙烯酸酯。合适的微粒是本领域中已知的并且是可商购的。陶瓷和玻璃微球可购自
微粒可以是固体。微粒可以是中空的。微粒可以由聚合物纤维形成。
微粒可以源自用于提取和分离多核苷酸的试剂盒(例如,多核苷酸或多肽)。
微粒表面可以与分析物相互作用并连接分析物。所述表面可自然地与分析物如多核苷酸或多肽相互作用,不需要功能化。通常将微粒表面功能化以促进分析物的连接。合适的功能化是本领域已知的。例如,微粒表面可以用以下各者进行功能化:多组氨酸标签(六组氨酸标签、6xHis-标签、His6标签或
微粒可以用与多核苷酸特异性地结合的分子或基团进行功能化。在这种情况下,将要与微粒连接并被递送到跨膜孔的多核苷酸可以被称为靶多核苷酸。这允许微粒从含有其它多核苷酸的样品中选择或捕获靶多核苷酸。如果分子或基团优先地或以高亲和力与靶多核苷酸结合,但不与其它多核苷酸或不同的多核苷酸结合或仅以低亲和力结合,则所述分子或基团与靶多核苷酸特异性地结合。如果分子或基团以1×10
优选地,所述分子或基团与靶多核苷酸结合的亲和力是其对其它多核苷酸的亲和力的至少10倍,如至少50倍、至少100倍、至少200倍、至少300倍、至少400倍、至少500倍、至少1000倍或至少10,000倍。亲和力可以使用已知的结合分析来测量,如利用荧光和放射性同位素的结合分析。竞争性结合分析也是本领域中已知的。肽或蛋白质与多核苷酸之间的结合强度可以使用纳米孔力光谱来测量,如Hornblower等人,《自然·方法(NatureMethods.)》4: 315-317。(2007)中所述。
微粒可以用与靶多核苷酸或衔接子特异性地杂交或包含与靶多核苷酸或衔接子的一部分或区域互补的部分或区域的寡核苷酸或多核苷酸进行功能化。这允许微粒从含有其它多核苷酸的样品中选择或捕获靶多核苷酸。
当寡核苷酸或多核苷酸优先地或以高亲和力与靶多核苷酸杂交,但与其它多核苷酸基本上不杂交、不杂交或仅以低亲和力杂交时,所述寡核苷酸或多核苷酸与靶多核苷酸特异性地杂交。如果寡核苷酸或多核苷酸与靶多核苷酸以解链温度(T
允许杂交的条件是本领域中众所周知的(例如,Sambrook等人,2001,《分子克隆:实验室手册(Molecular Cloning:a laboratory manual)》,第3版,冷泉港实验室出版社(Cold Spring Harbor Laboratory Press);和《当代分子生物学实验手册(CurrentProtocols in Molecular Biology)》,第2章,Ausubel等人编,格林出版与威利交叉科学出版社(Greene Publishing and Wiley-lnterscience),纽约(1995))。杂交可以在低严格度条件下进行,例如在37℃下在30%至35%甲酰胺、1M NaCl以及1%十二烷基硫酸钠(SDS)的缓冲溶液存在下,然后在50℃下在1X(0.1650M Na
多核苷酸可以包括与靶多核苷酸的部分或区域基本上互补的部分或区域。因此,与靶多核苷酸中的部分或区域相比,多核苷酸的区域或部分可以在5个、10个、15个、20个、21 个、22个、30个、40个或50个核苷酸的区上具有1个、2个、3个、4个、5个、6个、7个、 8个、9个、10个或更多个错配。
区域的一部分通常为50个核苷酸或更少,如40个核苷酸或更少、30个核苷酸或更少、 20个核苷酸或更少、10个核苷酸或更少或5个核苷酸或更少。
微粒优选是顺磁性或磁性的。微粒优选包含顺磁性或超顺磁性材料或顺磁性或超顺磁性金属,如铁。可以使用任何合适的磁性微粒。例如,可以使用可购自例如Clontech、Promega、 Invitrogen ThermoFisher Scientific以及NEB的磁珠。在一些实施例中,微粒包含连接着有机基团的磁性颗粒,有机基团例如金属螯合基团,如次氮基三乙酸(NTA)。有机组分可以例如包含选自以下的基团:-C(=O)O-、-C-O-C-、-C(=O)-、-NH-、-C(=O)-NH、-C(=O)-CH
微粒最优选是可购自Life Technologies的His-Tag
多核苷酸结合蛋白(例如,多核苷酸解链酶)
本文提供的方法使用聚合酶来处理多核苷酸,使得通过纳米孔检测处理反应的副产物,由此检测通过聚合酶向多核苷酸链添加核苷酸。聚合酶是多核苷酸结合蛋白(例如,多核苷酸解链酶)的实例。
多核苷酸结合蛋白(例如,多核苷酸解链酶)是能够结合到多核苷酸的蛋白质。多核苷酸结合蛋白可以能够控制多核苷酸通过孔的移动。确定蛋白是否结合到多核苷酸在本领域中是简单的。蛋白质通常与多核苷酸的至少一种性质相互作用并且修饰其至少一种性质。蛋白质可以通过裂解多核苷酸以形成单个核苷酸或如单核、二核苷酸或三核苷酸等更短的核苷酸链来修饰多核苷酸。所述部分可以通过使多核苷酸定向或将其移动到特定位置,即控制其移动来修饰多核苷酸。
多核苷酸结合蛋白(例如,多核苷酸解链酶)可以源自多核苷酸处理酶。多核苷酸操作酶是能够与多核苷酸相互作用并且修饰其的至少一个性质的多肽。酶可以通过裂解多核苷酸以形成单个核苷酸或如二核苷酸或三核苷酸等更短的核苷酸链来修饰多核苷酸。酶可以通过将多核苷酸定向或将其移动到特定位置来修饰多核苷酸。在一些公开的方法中,多核苷酸操作酶并不需要显示酶促活性,只要其能够结合多核苷酸并且控制其移动通过孔即可。例如,在这种方法中,可以对酶进行修饰以移除其酶促活性或可以在防止其充当酶的条件下使用。下文更详细地论述了这种条件。在本文提供的方法中,多核苷酸结合蛋白通过向多核苷酸链添加核苷酸来处理多核苷酸,并且通过纳米孔检测处理反应的副产物,由此检测向多核苷酸链添加核苷酸。
多核苷酸处理酶可以源自溶核酶。多核苷酸处理酶可以源自任何酶分类(EC)组的成员: 3.1.11、3.1.13、3.1.14、3.1.15、3.1.16、3.1.21、3.1.22、3.1.25、3.1.26、3.1.27、3.1.30和3.1.31。所述酶可以是WO 2010/086603中公开的酶。
优选地,多核苷酸处理酶是聚合酶。其它多核苷酸处理酶包含核酸外切酶、解旋酶、易位酶和拓扑异构酶,如旋转酶。聚合酶可以是
多核苷酸处理酶可以源自解旋酶。解旋酶可以是或源自Hel308解旋酶、如TraI解旋酶或 TrwC解旋酶等RecD解旋酶、XPD解旋酶或Dda解旋酶。解旋酶可以是或源自Hel308Mbu、 Hel308 Csy Hel308 Tga、Hel308 Mhu、TraI Eco、XPD Mbu或其变体。
解旋酶可以是在WO 2013/057495、WO 2013/098562、WO2013098561、WO 2014/013260、 WO 2014/013259、WO 2014/013262和WO/2015/055981中公开的任何解旋酶、经修饰的解旋酶或解旋酶构建体。
Dda解旋酶优选地包括WO/2015/055981和WO 2016/055777中公开的任何修饰。
在本文公开的一些方法中,可以使用任何数量的解旋酶。例如,可以使用1个、2个、3 个、4个、5个、6个、7个、8个、9个、10个或更多个解旋酶。在公开的一些方法中,可以使用不同数量的解旋酶。可以使用上述两个或更多个解旋酶的任何组合。两个或更多个解旋酶可以是两个或更多个Dda解旋酶。两个或更多个解螺旋酶可以是一个或多个Dda解螺旋酶和一个或多个TrwC解螺旋酶。两个或更多个解旋酶可以是相同解旋酶的不同变体。两个或更多个解旋酶可以优选地彼此连接。两个或更多个解旋酶可以更优选地彼此共价连接。解旋酶可以按任何次序和使用任何方法来连接。用于这种方法的优选的解旋酶构建体描述于WO2014/013260、WO 2014/013259、WO 2014/013262和WO2015/055981中。
在本文提供的一些实施例中,多核苷酸结合蛋白是多核苷酸解链酶。多核苷酸解链酶是能够将双链多核苷酸解链成单链的酶。如本文所使用的,多核苷酸解链酶可以是聚合酶。在一些实施例中,多核苷酸解链酶能够将双链DNA解链成单链。因此,多核苷酸解链酶可以具有解旋酶活性。多核苷酸解链酶的实例包含例如本文所描述的解旋酶。
可以使用本领域已知的任何方法来测量多核苷酸结合能力。例如,可以使蛋白质与多核苷酸接触,并且可以测量蛋白质与多核苷酸结合并沿所述多核苷酸移动的能力。蛋白质可以包括有助于多核苷酸结合和/或有助于其在高盐浓度和/或室温下的活性的修饰。蛋白质可进行修饰,使得其结合多核苷酸(即保持多核苷酸结合能力)但不充当解链酶(即当具备所有便于移动的必需组分(例如ATP和Mg
酶可以共价连接到孔。任何方法可以用于将酶共价连接到孔。
在链测序中,如所公开的,通过所施加的电位或抵抗所施加的电位,多核苷酸通过纳米孔易位。在双链多核苷酸上逐渐或逐步起作用的外切核酸酶可以在孔的顺式侧使用,以在施加的电位下供给剩余的单链或在反向电势下供给反式侧。同样,使双链DNA解旋的解螺旋酶也可以类似的方式使用。还可使用聚合酶。需要链抵抗施加的电位而易位的测序应用也有可能,但DNA必须首先在逆转或无电位下由酶“捕获”。随着电位接着切换回后续结合,链应以顺式到反式的方式穿过孔,且通过电流保持为延长的构形。单链DNA核酸外切酶或单链 DNA依赖性聚合酶可以充当分子马达,所述分子马达抵抗施加的电位将最近易位的单链以逐步受控方式(反式到顺式)牵拉回反式通过孔。
这种方法中可以使用任何解旋酶。解旋酶可以相对于孔的两种模式来起作用。首先,解旋酶可以使多核苷酸通过孔移动,其场由施加的电压引起。在这种模式中,多核苷酸的5'端首先在孔中被捕获,并且解旋酶使多核苷酸移动到孔中,使得其在场的情况下通过孔,直到其最终易位通过到膜的反式侧为止。替代性地,解旋酶可以使多核苷酸通过孔抵抗由施加的电压引起的场而移动。在这种模式中,多核苷酸的3'端首先在孔中被捕获,并且解旋酶使多核苷酸移动通过孔,使得其抵抗所施加场的情况下牵拉出孔,直到其最终推回到膜的顺式侧为止。
还可以在相反的方向上进行这种方法。多核苷酸的3'端可以首先在孔中被捕获,并且解旋酶可以使多核苷酸移动到孔中,使得其在场的情况下通过孔,直到其最终易位通过到膜的反式侧为止。
当解旋酶不具备便于移动的必需组分或进行修饰以阻止或防止其移动时,所述解旋酶可以结合多核苷酸并且当多核苷酸被施加的场拉入孔中时起到减慢多核苷酸移动的作用。在非活性模式中,多核苷酸是否被3'或5'向下捕获无关紧要,其是通过充当制动器的酶朝向反式侧将多核苷酸拉入孔中的所施加的场。当在非活动模式中时,通过解螺旋酶对多核苷酸的移动控制可以多种方式(包括棘轮、滑动和制动)描述。还可以以这种方式来使用缺乏解旋酶活性的解旋酶变体。
在本文提供的方法中,多核苷酸可以以任何顺序与多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶)和孔接触。优选的是,当多核苷酸与多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶)和孔接触时,多核苷酸首先与多核苷酸结合蛋白(例如,多核苷酸解链酶)形成复合物。当跨孔施加电压时,多核苷酸/多核苷酸结合蛋白(例如,多核苷酸解链酶)复合物然后与孔形成复合物并且控制多核苷酸通过孔的移动。
本文提供的使用多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶)的方法在存在游离核苷酸或游离核苷酸类似物和促进多核苷酸结合蛋白(例如,多核苷酸解链酶)作用的酶辅因子的情况下执行。游离核苷酸可以是上文论述的任何单个核苷酸中的一种或多种。游离核苷酸包括(但不限于):单磷酸腺苷(AMP)、二磷酸腺苷(ADP)、三磷酸腺苷(ATP)、单磷酸鸟苷(GMP)、二磷酸鸟苷(GDP)、三磷酸鸟苷(GTP)、单磷酸胸苷(TMP)、二磷酸胸苷(TDP)、三磷酸胸苷(TTP)、单磷酸尿苷(UMP)、二磷酸尿苷(UDP)、三磷酸尿苷 (UTP)、单磷酸胞苷(CMP)、二磷酸胞苷(CDP)、三磷酸胞苷(CTP)、环单磷酸腺苷(cAMP)、环单磷酸鸟苷(cGMP)、单磷酸脱氧腺苷(dAMP)、二磷酸脱氧腺苷(dADP)、三磷酸脱氧腺苷(dATP)、单磷酸脱氧鸟苷(dGMP)、二磷酸脱氧鸟苷(dGDP)、三磷酸脱氧鸟苷(dGTP)、单磷酸脱氧胸苷(dTMP)、二磷酸脱氧胸苷(dTDP)、三磷酸脱氧胸苷(dTTP)、单磷酸脱氧尿苷(dUMP)、二磷酸脱氧尿苷(dUDP)、三磷酸脱氧尿苷(dUTP)、单磷酸脱氧胞苷(dCMP)、二磷酸脱氧胞苷(dCDP)和三磷酸脱氧胞苷(dCTP)。游离核苷酸优选地选自AMP、TMP、 GMP、CMP、UMP、dAMP、dTMP、dGMP或dCMP。游离核苷酸优选地是三磷酸腺苷(ATP)。酶辅因子是一种允许构筑体起作用的因子。酶辅因子优选地是二价金属阳离子。二价金属阳离子优选为Mg
核苷酸优选地被标记。核苷酸优选地用光学标记物标记。样品中的核苷酸根据样品中核苷酸的类型优选地进行可区分地标记。例如,如果样品包括两种、三种、四种或更多种类型的核苷酸,则两种、三种、四种或更多种类型的核苷酸可以各自包括不同的标记物,以使得样品中的核苷酸的两种、三种、四种或更多种类型的核苷酸进行可区分地标记。例如,如果样品包括ATP、CTP、GTP、TTP、UTP、dAPT、dCTP、dGTP、dTTP和/或dUTP,则ATP、 CTP、GTP、TTP、UTP、dAPT、dCTP、dGTP、dTTP和/或dUTP优选地被可区分地标记。标记优选地使得通过聚合酶的处理反应的副产物被标记,优选地具有光学标记物和/或聚合物标签。聚合物标签优选地带电。使用聚合物标签检测酶处理反应的副产物先前描述于Stranges 等人,“通过电极阵列上的合成用于单分子DNA测序的纳米孔偶联的聚合酶的设计和表征”, 《美国国家科学院院刊》,2016年11月1日;113(44):E6749–E6756。
优选地,因此一个或多个处理反应的副产物是标记的磷酸盐物质。因此一个或多个处理反应的副产物根据通过聚合酶添加到多核苷酸链的核苷酸的类型优选地进行可区分地标记。
分子制动器可以是结合多核苷酸并且减缓多核苷酸通过孔的移动的任何化合物或分子。分子制动器可以是上文讨论的任何一种。分子制动器优选地包括与多核苷酸结合的化合物。所述化合物优选地是大环化合物。合适的大环包括但不限于环糊精、杯芳烃、环肽、冠醚、葫芦脲、柱芳烃、其衍生物或其组合。环糊精或其衍生物可以是在Eliseev,A.V.和Schneider, H-J.(1994)《美国化学协会期刊(J.Am.Chem.Soc)》116:6081-6088。环糊精更优选是七-6- 氨基-β-环糊精(am
膜
可根据本文所述的各个方面使用任何膜。合适的膜是本领域熟知的。膜优选是两亲层或固态层。
两亲层是由两亲分子形成的层,所述两亲分子如磷脂,其具有亲水性和亲脂性两种特性。两亲分子可以是合成的或天然存在的。非天然存在的两亲物和形成单层的两亲物在所属领域中是已知的,且包括例如嵌段共聚物(Gonzalez-Perez等人,《朗缪尔(Langmuir)》,2009,25, 10447-10450)。嵌段共聚物是聚合在一起的两个或更多个单体亚基产生单一聚合物链的聚合材料。嵌段共聚物通常具有通过每个单体亚基贡献的性质。然而,嵌段共聚物可以具有由单独的亚单元形成的聚合物所不具备的独特性质。嵌段共聚物可以被工程化为使得单体亚基之一在水性介质中是疏水性的(即亲脂性),而一个或多个其它亚基是亲水性的。在这种情况下,嵌段共聚物可以具备两亲性质,并且可以形成模拟生物膜的结构。嵌段共聚物可以是二嵌段 (由两个单体亚单元组成),但也可以由多于两个单体亚单元构建以形成表现为两亲物的更复杂布置。共聚物可以是三嵌段、四嵌段或五嵌段共聚物。膜优选是三嵌段共聚物膜。
古细菌双极性四醚脂质是天然存在的脂质,其被构建成使得脂质形成单层膜。这些脂质一般发现于在苛刻生物环境中存活的嗜极生物、嗜热生物、嗜盐生物和嗜酸生物中。认为其稳定性是源于最终双层的融合性质。直接了当的做法是,通过产生具有一般基序亲水性-疏水性-亲水性的三嵌段聚合物来构建模拟这些生物实体的嵌段共聚物材料。这种材料可形成表现类似于脂质双层并且涵盖从囊泡到层状膜的一系列阶段表现的单体膜。由这些三嵌段共聚物形成的膜相对于生物脂质膜保持若干优势。因为三嵌段共聚物是合成的,所以可小心地控制准确的构建,以提供形成膜并与孔和其它蛋白质相互作用所需的正确链长度和特性。
还可以由不被归类为脂质亚材料的亚单元来构建嵌段共聚物,例如可由硅氧烷或其它非基于烃的单体来制成疏水性聚合物。嵌段共聚物的亲水性亚区段还可以具备低蛋白质结合特性,这允许产生当暴露于原始生物样品时具有高度抗性的膜。这种头基单元还可来源于非经典的脂质头基。
与生物脂质膜进行比较,三嵌段共聚物膜还具有增加的机械和环境稳定性,例如高许多的操作温度或pH范围。嵌段共聚物的合成性质提供定制用于广泛范围应用的基于聚合物的膜的平台。
膜最优选是WO2014/064443或WO2014/064444中所公开的膜中的一种。
两亲分子可进行化学修饰或官能化,以便于偶联多核苷酸。
两亲性层可以是单层或双层。两亲层通常是平面的。两亲层可以是弯曲的。两亲层可以是支撑式的。两亲层可以是凹入的。两亲层可以从凸起的柱子上悬挂下来,使得两亲层的周边区域(其与柱子连接)高于两亲层区域。这可以允许微粒如上文所描述的沿着膜行进、移动、滑动或滚动。
两亲膜通常天然地是流动的,基本上以大致10
膜可以是脂质双层。脂质双层是细胞膜的模型,且用作一系列实验研究的极佳平台。举例来说,脂质双层可用于通过单通道记录对膜蛋白的活体外研究。替代地,脂质双层可用作检测一系列物质的存在的生物传感器。脂质双层可以是任何脂质双层。合适的脂质双层包含但不限于平面脂质双层、支持双层或脂质体。脂质双层优选地是平坦脂质双层。合适脂质双层公开于WO 2008/102121、WO 2009/077734和WO 2006/100484中。
用于形成脂质双层的方法在所属领域中是已知的。脂质双层通常通过Montal和Mueller 的方法(《美国国家科学学院院报(Proc.Natl.Acad.Sci.USA.)》,1972;69:3561-3566)来形成,其中脂质单层携载于通过开孔两侧的水溶液/空气界面上,所述开孔垂直于所述界面。通常通过首先将脂质溶解在有机溶剂中,且接着使在开孔两侧上的水溶液的表面上蒸发一滴溶剂,来将脂质添加到水性电解质溶液的表面。一旦有机溶剂已蒸发,那么开孔两侧上的溶液/ 空气界面来回物理地移动通过开孔,直到形成双层为止。可跨越膜中的开孔或跨越凹槽中的开口形成平坦脂质双层。
Montal和Mueller的方法是常用的,这是因为是节约成本的,且是形成适合于蛋白孔插入的良好品质脂质双层的相对直接了当的方法。双层形成的其它常见方法包括脂质体双层的尖端浸没、双层涂刷和贴片夹持。
尖端浸没双层形成需要使开孔表面(例如移液管尖端)接触到携载脂质单层的测试溶液的表面。同样,通过将溶解于有机溶剂中的一滴脂质在溶液表面处蒸发来首先在溶液/空气界面处产生脂质单层。接着,通过朗缪尔-沙佛(Langmuir-Schaefer)过程形成双层,且需要机械自动以使开孔相对于溶液表面移动。
对于涂刷的双层,将溶解于有机溶剂中的一滴脂质直接应用于开孔,所述开孔浸没在水性测试溶液中。使用画刷或等同物将脂质溶液薄薄地铺展在孔上。溶剂的薄化导致脂质双层的形成。然而,难以从双层完全去除溶剂,并且因此通过这种方法形成的双层在电化学测量期间不太稳定并且更容易产生噪声。
贴片夹持是在生物细胞膜研究中常用的。通过抽汲将细胞膜夹持到移液管的末端,且膜贴片变为连接在开孔内。所述方法适用于通过夹持接着爆裂以离开密封在移液管的开孔内的脂质双层的脂质体来产生脂质双层。所述方法需要稳定的、巨大的且单层脂质体和在具有玻璃表面的材料中制造小开孔。
脂质体可以通过超声处理、挤出或Mozafari方法(Colas等人(2007)《微米(Micron)》 38:841-847)来形成。
在一个优选的实施例中,脂质双层如WO 2009/077734中所述形成。在此方法中有利的是,脂质双层是由干燥的脂质形成的。在一最优选实施例中,跨越开口形成脂质双层,如 WO2009/077734中所描述。
由脂质的两个相对层形成脂质双层。两个脂质层被布置成使得其疏水尾部基团面朝彼此,形成疏水性的内部。脂质的亲水性头基朝外面向双层每侧上的水性环境。双层可存在于多种脂质阶段中,所述阶段包括(但不限于)液体无序阶段(液体片层)、液体有序阶段、固体有序阶段(片层凝胶阶段、交错结合的凝胶阶段)和平坦双层晶体(片层亚凝胶阶段、片层结晶阶段)。
可以使用形成脂质双层的任何脂质组合物。对脂质组合物加以选择,以便形成具有所需特性的脂质双层,所述特性例如表面电荷、支撑膜蛋白的能力、堆积密度或机械特性。脂质组合物可以包含一种或多种不同脂质。举例来说,脂质组合物可含有最多100种脂质。脂质组合物优选地含有1到10种脂质。脂质组合物可包含天然存在的脂质和/或人工脂质。
脂质通常包含头基、界面部分和可相同或不同的两个疏水尾部基团。合适的头基包括(但不限于):中性头基,例如二酰基甘油酯(DG)和脑酰胺(CM);两性离子头基,例如磷脂酰胆碱(PC)、磷脂酰乙醇胺(PE)和鞘磷脂(SM);带负电头基,例如磷脂酰甘油(PG)、磷脂酰丝氨酸(PS)、磷脂酰肌醇(PI)、磷脂酸(PA)和心磷脂(CA);和带正电头基,例如三甲基铵丙烷(TAP)。合适界面部分包括(但不限于)天然存在的界面部分,例如基于甘油或基于脑酰胺的部分。合适的疏水尾部基团包括(但不限于):饱和烃链,例如月桂酸(正十二烷酸)、肉豆蔻酸(正十四烷酸)、棕榈酸(正十六烷酸)、硬脂酸(正十八烷酸)和花生酸(正二十烷酸);不饱和烃链,例如油酸(顺-9-十八烷酸);和分支链烃链,例如植烷酰基。链的长度和不饱和烃链中的双键的位置与数量可变化。链的长度和分支链烃链中的分支(如甲基)的位置和数量可变化。疏水尾部基团可作为醚或酯连接到界面部分。脂质可以是分枝菌酸。
脂质还可以进行化学修饰。脂质的头基或尾基可以进行化学修饰。头基已进行化学修饰的合适脂质包括但不限于:PEG修饰的脂质,如1,2-二酰基-sn-甘油-3-磷酸乙醇胺-N-[甲氧基 (聚乙二醇)-2000];功能化的PEG脂质,如1,2-二硬脂酰基-sn-甘油-3磷酸乙醇胺-N-[生物素基 (聚乙二醇)2000];以及针对缀合修饰的脂质,如1,2-二油酰基-sn-甘油-3-磷酸乙醇胺-N-(琥珀酰基)和1,2-二棕榈酰基-sn-甘油-3-磷酸乙醇胺-N-(生物素基)。尾基已进行化学修饰的合适脂质包括但不限于:可聚合脂质,如1,2-双(10,12-二十三碳二炔基)-sn-甘油-3-磷酸胆碱;氟化脂质,如1-软脂酰基-2-(16-氟软脂酰基)-sn-甘油-3-磷酸胆碱;氘化脂质,如1,2-二棕榈酰基 -D62-sn-甘油-3-磷酸胆碱;以及醚连接的脂质,如1,2-二-O-植烷基-sn-甘油-3-磷酸胆碱。脂质可以进行化学修饰或官能化,以便于偶联多核苷酸。
两亲层,例如脂质组合物,通常包含将影响层的特性的一种或多种添加剂。合适的添加剂包括但不限于:脂肪酸,如棕榈酸、肉豆蔻酸和油酸;脂肪醇,如棕榈醇、肉豆蔻醇和油醇;甾醇,如胆固醇、麦角固醇、羊毛甾醇、谷甾醇和豆甾醇;溶血磷脂,如1-酰基-2-羟基-sn-甘油-3-磷酸胆碱;以及神经酰胺。
固态层可以由有机材料和无机材料形成,所述材料包括但不限于:微电子材料;绝缘材料,如Si
通常使用以下来进行所述方法:(i)包含孔的人工两亲层;(ii)包含孔的分离的天然存在的脂质双层;或(iii)插入有孔的细胞。通常使用人工两亲层(例如人工三嵌段共聚物层)来实行方法。层可包含其它跨膜和/或膜内蛋白质以及除孔以外的其它分子。下文论述了合适的设备和条件。通常在体外进行本发明的方法。
根据本发明的方法将多核苷酸递送到其上的膜包含在液体中。液体使膜保持“湿润”并防止其变干。液体通常是水溶液。水溶液通常具有与水相同的密度。水溶液的密度通常是约1g/cm
膜通常将两个体积的水溶液隔开。膜抵挡所述体积之间的电流流动。插入膜中的跨膜孔选择性地允许离子通过膜,这可以记录为由两个体积的水溶液中的电极检测到的电信号。包含靶多核苷酸的复合物的存在可调节离子的流动,并通过观察所产生的电信号变化来检测。
阵列
膜通常是膜阵列的一部分,其中每个膜优选包含跨膜孔。因此,本发明提供了一种使用膜阵列检测靶多核苷酸的方法。
膜可以被包含在具有电隔离膜阵列的设备中,每个膜使用其自己的电极单独寻址,使得阵列等同于从测试样品平行测量的许多单独传感器。膜可以相对密集地填充,允许大量的膜用于给定体积的测试样品。在本领域中,例如在WO 2009/077734和WO2012/042226中描述了合适的膜阵列和设备。例如,WO 2009/077734公开了在微孔孔口阵列上形成的多个可单独寻址的脂质双层,每个微孔含有电极和与脂质双层接触的水性介质。
所述设备通常以‘即用’状态提供给最终用户,其中膜和跨膜孔是预先插入的。以‘即用’状态提供的典型设备包含两亲膜阵列,每个膜包含跨膜孔并且跨含有液体的孔设置。 WO2014/064443公开了这样的设备和其制造方法。将待分析的测试液体施加到两亲膜的上表面。
然而,以‘即用’状态提供的设备还需要考虑另外的因素,即传感器不会变干,即液体不会通过两亲膜而从孔中损失,那样的话会导致性能损失或损坏传感器。解决传感器变干问题的一种解决方案是在两亲膜的表面上设置含缓冲液的装置,使得通过膜表面的任何蒸发最小化,并且在膜的任一侧提供的液体可以具有相同的离子强度以减少任何渗透效应。在使用中,可以从两亲膜表面去除缓冲液,并引入待分析的测试液体以接触表面。
一些应用可以使用跨膜电特性的测量,例如离子电流。为了提供这样的测量,所述设备可以进一步在每个隔室中包含相应的电极,使其与包含极性介质的体积电接触。可以进行其它类型的测量,例如光学测量,例如荧光测量和FET测量。光学测量和电测量可以同时进行 (Heron AJ等人,《美国化学会志》2009;131(5):1652-3)。
所述设备可以进一步包含公共电极。所述设备可以进一步包含连接在公共电极与每个隔室中的相应电极之间的电路,布置所述电路是为了进行电测量。这种电测量可以取决于在膜处或穿过膜发生的过程。
设备可以包括用于测量纳米孔阵列的FET阵列。
检测器:
在本文提供的方法中,检测器可以选自以下:(i)零模波导、(ii)场效应晶体管,任选地纳米线场效应晶体管;(iii)AFM尖端;(iv)纳米管,任选地碳纳米管;以及(v)纳米孔。优选地,所述检测器是纳米孔。
跨膜孔
跨膜孔是在某种程度上穿过膜的结构。它允许由施加的电位驱动的水合离子流过膜或膜内。跨膜孔通常穿过整个膜,使得水合离子可以从膜的一侧流到膜的另一侧。然而,跨膜孔不是必须得穿过膜。它的一端可以封闭。例如,孔可以是膜中的阱、间隙、通道、沟槽或狭缝,水合离子可以沿着它流动或流入其中。
本发明中可以使用任何跨膜孔。孔可以是生物的或人造的。合适的孔包括但不限于蛋白质孔、多核苷酸孔和固态孔。孔可以是DNA折纸孔(Langecker等人,《科学(Science)》,2012; 338:932-936)。孔可以是马达蛋白纳米孔,例如,允许双链多核苷酸易位的纳米孔。在一些实施例中,马达蛋白纳米孔能够使双链多核苷酸解链。示例性马达蛋白纳米孔包括但不限于 phi29马达蛋白纳米孔,例如,如Wendell等人“双链DNA易位通过膜适应的phi29马达蛋白纳米孔(Translocation of double-stranded DNA throughmembrane-adapted phi29 motor protein nanopores)”《自然纳米技术(NatNanotechnol)》,4(2009),第765-772页所述。在一些实施例中,如Feng等人“基于纳米孔的第四代DNA测序技术(Nanopore-based fourth-generation DNA sequencingtechnology)”《基因组学、蛋白质组学和生物信息学(Genomics,Proteomics&Bioinformatics)》(2015)第13卷,第1期,第4-16页所述或引用的任何纳米孔,均可用于本文所述的各个方面。
跨膜孔优选地是跨膜蛋白质孔。跨膜蛋白质孔是多肽或多肽的集合,其允许如用聚合酶处理多核苷酸所得的副产物等水合离子从膜的一侧流到膜的另一侧。在本发明中,跨膜蛋白质孔能够形成孔,所述孔允许由施加的电位驱动的水合离子从膜的一侧流到另一侧。跨膜蛋白质孔优选地允许多核苷酸从膜的一侧,如三嵌段共聚物膜流到另一侧。跨膜蛋白质孔允许如DNA或RNA等多核苷酸移动通过孔。
跨膜蛋白质孔可以是单体或寡聚体。孔优选由几个重复亚单元组成,如至少6个、至少7 个、至少8个、至少9个、至少10个、至少11个、至少12个、至少13个、至少14个、至少15个或至少16个亚单元。孔优选是六聚体、七聚体、八聚体或九聚体孔。孔可以是同型寡聚体或异型低聚物。
跨膜蛋白孔通常包括离子可以流过的桶或通道。孔的亚单元通常围绕中心轴线并且为跨膜β筒体或通道或跨膜α-螺旋束或通道提供链。
跨膜蛋白质孔的桶或通道通常包括促进与核苷酸、多核苷酸或核酸相互作用的氨基酸。这些氨基酸优选地位于筒体或通道的收缩部附近。跨膜蛋白质孔通常包含一个或多个带正电荷的氨基酸,如精氨酸、赖氨酸或组氨酸,或芳香族氨基酸,如酪氨酸或色氨酸。这些氨基酸通常促进孔与核苷酸、多核苷酸或核酸之间的相互作用。
根据本发明使用的跨膜蛋白孔可以源自β-筒形孔或α-螺旋束孔。β-桶孔包括由β-链形成的桶或通道。合适的β-筒形孔包括但不限于β-毒素,如α-溶血素、炭疽毒素和杀白细胞素,以及细菌的外膜蛋白/孔蛋白,如耻垢分枝杆菌(Mycobacterium smegmatis)孔蛋白(Msp),例如MspA、MspB、MspC或MspD、CsgG,外膜孔蛋白F(OmpF)、外膜孔蛋白G(OmpG)、外膜磷脂酶A以及奈瑟氏球菌(Neisseria)自主转运蛋白脂蛋白(NalP)以及其它孔,如胞溶素(lysenin)。α-螺旋束孔包括由α-螺旋形成的桶或通道。合适的α-螺旋束孔包括但不限于内膜蛋白和α外膜蛋白,如WZA和ClyA毒素。
跨膜孔可以源自或基于Msp、α-溶血素(α-HL)、胞溶素、CsgG、ClyA、Sp1以及溶血蛋白溶血毒素(fragaceatoxin)C(FraC)。跨膜蛋白孔优选源自CsgG,更优选源自大肠杆菌菌株 K-12亚株MC4100的CsgG。WO 2016/034591中公开了源自CsgG的合适的孔。跨膜孔可以源自胞溶素。WO 2013/153359中公开了源自胞溶素的合适的孔。
野生型α-溶血素孔由7个相同的单体或亚单元形成(即,它是七聚体)。α-溶血素-NN的一个单体或亚单元的序列公开在例如WO2016/059375中。
跨膜蛋白孔优选地源自Msp,更优选源自MspA。WO 2012/107778中公开了源自MspA的合适的孔。
可以例如通过加入组氨酸残基(his标签)、天冬氨酸残基(asp标签)、链霉亲和素标签、 flag标签、SUMO标签、GST标签或MBP标签,或在多肽天然地不含信号序列的情况下通过加入这样的序列以促进其从细胞分泌,来修饰本文所述的任何蛋白质,如跨膜蛋白孔,从而帮助其鉴定或纯化。引入基因标签的替代方案是将标签化学反应到孔或构建体上的天然的或工程化的位置上。此的实例将是使凝胶迁移试剂与孔外部上的工程化半胱氨酸反应。这已被证明是一种用于分离溶血素异型低聚物的方法(《生物化学(Chem Biol).》1997年7月; 4(7):497-505)。
孔可以用显露标记来进行标记。显露标记可以是允许孔被检测到的任何合适的标记。合适的标记包括但不限于荧光分子;放射性同位素,例如
本文所描述的任何蛋白质,如跨膜蛋白孔,可以合成方式或通过重组手段制得。例如,可以通过体外翻译和转录(IVTT)来合成孔。孔的氨基酸序列可以经过修饰而包括非天然存在的氨基酸或增加蛋白质的稳定性。当通过合成手段制造蛋白质时,可以在制造期间引入此类氨基酸。还可以在合成或重组制造后改变孔。
本文所述的任何蛋白质,如跨膜蛋白孔,可以使用本领域中已知的标准方法制造。编码孔或构建体的多核苷酸序列可以使用本领域中的标准方法衍生和复制。编码孔或构建体的多核苷酸序列可以使用本领域中的标准技术在细菌宿主细胞中表达。可以通过从重组表达载体原位表达多肽而在细胞中产生孔。表达载体任选地携带诱导型启动子以控制多肽的表达。这些方法描述于Sambrook,J.和Russell,D.(2001)中所述。《分子克隆:实验室手册》,第3版. 纽约冷泉港的冷泉港实验室出版社中。
可以在通过任何蛋白质液相色谱系统纯化之后或在重组表达之后,从产蛋白质生物体大规模产生孔。典型的蛋白质液相色谱系统包括FPLC、AKTA系统、Bio-Cad系统、Bio-Rad BioLogic系统以及Gilson HPLC系统。
测量离子流
可以使用电测量和/或光学测量来监测通过跨膜孔的离子流。
电测量可以是电流测量、阻抗测量、隧穿测量或场效应晶体管(FET)测量。
当通过聚合酶对多肽进行处理的多肽的副产物穿过孔时,通过跨膜孔的离子流的改变可以被检测为电流、电阻或光学性质的改变。所测量的效果可以是跨膜孔的电子隧穿。所测量的效应可以是由于多核苷酸与跨膜孔的相互作用引起的电位改变,其中在FET测量中使用局部电位传感器监测效应。
可以进行各种不同类型的测量。这包括但不限于:电测量和光学测量。一种涉及荧光测量的合适的光学方法公开于《美国化学会志》2009,131 1652-1653中。可能的电测量包含:电流测量、阻抗测量、隧穿测量(Ivanov AP等人,《纳米快报》2011年1月12日;11(1):279-85) 以及FET测量(国际申请WO 2005/124888)。光学测量可以与电测量结合(Soni GV等人,《科学仪器综述(Rev Sci Instrum.)》2010年1月;81(1):014301)。测量可以是跨膜电流测量,如对流过孔的离子电流的测量。
可以使用标准单通道记录设备来进行电测量,如以下文献中所述:
Stoddart D等人,《美国国家科学院院刊》,12;106(19):7702-7;Lieberman KR等人,《美国化学会志》2010;132(50):17961-72;以及国际申请WO 2000/28312。或者,可以使用多通道系统来进行电测量,所述系统例如,如WO 2009/077734和WO 2011/067559中所述。
所述方法优选在跨膜施加电位的情况下进行。外加电位可以是电压电位。或者,外加电位可以是化学电位。在一些实施例中,施加的电位可以由渗透失衡驱动。化学电位的实例是跨膜,如跨两亲层使用盐梯度。盐梯度公开于Holden等人《美国化学学会杂志》2007年7月 11日;129(27):8650-5在一些情况下,使用在多核苷酸相对于孔移动时通过孔的电流来评估或确定多核苷酸的序列。
多核苷酸表征
在本文描述的各个方面的一些实施例中,所述方法可以包括进一步表征靶多核苷酸。在靶多核苷酸与孔接触时,当多核苷酸或通过聚合酶处理这种多核苷酸所得的副产物相对于孔移动时,采用指示靶多核苷酸的一种或多种特性的一种或多种测量。
该方法可以涉及确定多核苷酸是否被修饰。可以测量任何修饰的存在或不存在。所述方法优选包含:用一个或多个蛋白质或用一个或多个标记、标签或间隔区确定多核苷酸是否通过甲基化、通过氧化、通过损坏来修饰。特定的修饰将引起与孔的特定的相互作用,这可以使用下文所描述的方法测量。例如,可以在孔与每个核苷酸相互作用期间通过孔的离子流的基础上区别甲基胞嘧啶和胞嘧啶。
设备
可以使用适合于研究膜/孔系统的任何设备来进行所述方法,在所述系统中,孔存在于膜中。可以使用适合于跨膜孔感测的任何设备来进行所述方法。例如,设备包含腔室,所述腔室包含水溶液和将腔室分成两个部分的屏障。屏障通常具有孔口,在其中形成含有孔的膜。或者,屏障形成其中存在孔的膜。
可以使用在WO 2008/102120中所述的设备来进行所述方法。
可以进行各种不同类型的测量。这包括但不限于:电测量和光学测量。一种涉及荧光测量的合适的光学方法公开于《美国化学学会杂志》2009,131 1652-1653中。可能的电测量包含:电流测量、阻抗测量、隧穿测量(Ivanov AP等人,《纳米快报》2011年1月12日; 11(1):279-85)以及FET测量(国际申请WO 2005/124888)。光学测量可以与电测量结合(SoniGV等人,《科学仪器综述(Rev Sci Instrum.)》2010年1月;81(1):014301)。测量可以是跨膜电流测量,如对流过孔的离子电流的测量。
可以使用标准单通道记录设备来进行电测量,如以下文献中所述:Stoddart D等人,《美国国家科学院院刊》,12;106(19):7702-7;Lieberman KR等人,《美国化学会志》2010; 132(50):17961-72;以及国际申请WO 2000/28312。可替代地,可以使用多通道系统进行电测量,例如如国际申请WO 2009/077734和国际申请WO 2011/067559中所述。
所述方法优选在跨膜施加电位的情况下进行。外加电位可以是电压电位。或者,外加电位可以是化学电位。化学电位的实例是跨膜,如跨两亲层使用盐梯度。盐梯度公开于Holden 等人《美国化学学会杂志》2007年7月11日;129(27):8650-5在一些情况下,使用在通过聚合酶处理多核苷酸所得的副产物相对于孔移动时通过孔的电流来评估或确定多核苷酸的序列。
所述方法可以涉及在通过聚合酶处理多核苷酸所得的副产物相对于孔移动时测量穿过孔的电流。因此,设备还可以包括能够施加电位并且测量跨膜和孔的电信号的电路。可以使用膜片钳或电压钳来进行所述方法。方法优选地涉及使用电压钳。
本发明的方法可以涉及在通过聚合酶处理多核苷酸所得的副产物相对于孔移动时测量穿过孔的电流。用于测量通过跨膜蛋白孔的离子电流的适当条件在本领域中是已知的并且在实例中公开。通常通过跨膜和孔施加的电压来实行所述方法。所用电压通常是+5V到-5V,例如+4V到-4V、+3V到-3V,或+2V到-2V。所用电压通常是-600mV到+600mV或-400mV到+400mV。所用电压优选地在具有下限和上限的范围内,下限选自:-400mV、-300mV、 -200mV、-150mV、-100mV、-50mV、-20mV和0mV,上限独立地选自:+10mV、+20 mV、+50mV、+100mV、+150mV、+200mV、+300mV和+400mV。所用电压更优选地在 100mV到240mV的范围内,并且最优选在120mV到220mV的范围内。通过使用增加的外加电位,可以增加孔对不同核苷酸的区分。
通常在存在任何电荷载流子的情况下进行所述方法,所述电荷载流子如金属盐,例如碱金属盐;卤盐,例如氯化物盐,如碱金属氯化物盐。电荷载流子可以包括离子液体或有机盐,例如四甲基氯化铵、三甲基苯基氯化铵、苯基三甲基氯化铵或1-乙基-3-甲基氯化咪唑鎓。在下文论述的示范性设备中,盐在腔室中的水溶液中存在。通常使用氯化钾(KCl)、氯化钠 (NaCl)、氯化铯(CsCl),或亚铁氰化钾和铁氰化钾的混合物。优选的是KCl、NaCl,以及亚铁氰化钾和铁氰化钾的混合物。电荷载流子可以是跨膜不对称的。举例来说,电荷载流子的类型和/或浓度可在膜的每侧上不同。
盐浓度可以是饱和的。盐浓度可以是3M或更低,并且通常是0.1至2.5M、0.3至1.9M、 0.5至1.8M、0.7至1.7M、0.9至1.6M或1M至1.4M。盐浓度优选是150mM至1M。所述方法优选使用至少0.3M的盐浓度进行,例如至少0.4M、至少0.5M、至少0.6M、至少 0.8M、至少1.0M、至少1.5M、至少2.0M、至少2.5M或至少3.0M。高盐浓度提供高信噪比并允许相对于正常电流波动的背景鉴定指示核苷酸的存在的电流。
通常在存在缓冲液的情况下进行所述方法。在上文论述的示例性设备中,缓冲液存在于腔室中的水溶液中。在本发明的方法中可以使用任何缓冲液。通常,缓冲液是磷酸盐缓冲液。其它合适的缓冲液是HEPES和Tris-HCl缓冲液。通常在以下的pH下来实行方法:4.0到12.0、 4.5到10.0、5.0到9.0、5.5到8.8、6.0到8.7或7.0到8.8或7.5到8.5。所用pH优选是约7.5。
可以在以下温度下进行所述方法:0℃至100℃、15℃至95℃、16℃至90℃、17℃至85℃、 18℃至80℃、19℃至70℃或20℃至60℃。通常在室温下进行所述方法。任选地在支持酶功能的温度下进行所述方法,如在约37℃下进行。
游离核苷酸和辅因子
在游离核苷酸或游离核苷酸类似物和/或促进多核苷酸结合蛋白(例如,多核苷酸解链酶) 作用的酶辅因子存在下执行方法。游离核苷酸可以是上文论述的任何单个核苷酸中的一种或多种。游离核苷酸包括(但不限于):单磷酸腺苷(AMP)、二磷酸腺苷(ADP)、三磷酸腺苷 (ATP)、单磷酸鸟苷(GMP)、二磷酸鸟苷(GDP)、三磷酸鸟苷(GTP)、单磷酸胸苷(TMP)、二磷酸胸苷(TDP)、三磷酸胸苷(TTP)、单磷酸尿苷(UMP)、二磷酸尿苷(UDP)、三磷酸尿苷(UTP)、单磷酸胞苷(CMP)、二磷酸胞苷(CDP)、三磷酸胞苷(CTP)、环单磷酸腺苷 (cAMP)、环单磷酸鸟苷(cGMP)、单磷酸脱氧腺苷(dAMP)、二磷酸脱氧腺苷(dADP)、三磷酸脱氧腺苷(dATP)、单磷酸脱氧鸟苷(dGMP)、二磷酸脱氧鸟苷(dGDP)、三磷酸脱氧鸟苷(dGTP)、单磷酸脱氧胸苷(dTMP)、二磷酸脱氧胸苷(dTDP)、三磷酸脱氧胸苷(dTTP)、单磷酸脱氧尿苷(dUMP)、二磷酸脱氧尿苷(dUDP)、三磷酸脱氧尿苷(dUTP)、单磷酸脱氧胞苷(dCMP)、二磷酸脱氧胞苷(dCDP)和三磷酸脱氧胞苷(dCTP)。游离核苷酸优选地选自AMP、TMP、GMP、CMP、UMP、dAMP、dTMP、dGMP或dCMP。游离核苷酸优选地是三磷酸腺苷(ATP)。酶辅因子是允许多核苷酸结合蛋白(例如,多核苷酸解链酶)起作用的因子。酶辅因子优选地是二价金属阳离子。二价金属阳离子优选为Mg
如上文所描述的,核苷酸优选地被标记,优选地用光学标记物和/或聚合物标签标记。
试剂盒
本发明还提供了衔接子群体,所述衔接子群体包括双链条形码序列、单链前导序列和能够处理双链多核苷酸的链的多核苷酸结合蛋白(例如,多核苷酸解链酶,例如,聚合酶),其中群体中的每个衔接子的条形码序列是唯一的。
还公开了在本文所描述的方法中使用的试剂盒。试剂盒通常包括本文所描述的衔接子群体。试剂盒可以另外包括一个或多个膜锚、多核苷酸结合蛋白(例如,多核苷酸解链酶)(其可以预先结合到衔接子)、连接酶、聚合酶和/或游离核苷酸或辅因子。
试剂盒可以包含上文所公开的任何膜的组分,所述膜例如两亲层或三嵌段共聚物膜。试剂盒可以进一步包含跨膜孔。上文参考本发明方法论述的实施例中任一个同样适用于试剂盒。
试剂盒可以额外包括使上文提到的实施例中的任何实施例能够被进行的一种或多种其它试剂或仪器。此类试剂或仪器包括以下各项中的一个或多个:合适的缓冲液(水溶液)、用于从受试者获得样品的装置(如包含针的容器或仪器)、用于扩增和/或表达多核苷酸的装置、如上文所定义的膜或电压钳或膜片钳设备。试剂可以按干燥状态存在于试剂盒中,使得流体样品用于再悬浮试剂。试剂盒还可以任选地包括使试剂盒能够在本发明的方法中使用的说明书。所述试剂盒可以包含磁铁或电磁铁。试剂盒可以任选地包含核苷酸。
以下实施例说明了本申请的非限制性方面。
该实例显示当具有连接到模板链和补体链的单链前导序列的双链基因组DNA与CsgG纳米孔接触时,模板链和补体链在Dda解旋酶的控制下依次但分开地易位通过纳米孔(模板未通过发夹与互补序列连接)。与通过发夹连接在一起时相同模板/互补序列的易位相比,观察到测序准确度增加。
材料与方法
如下将基因组DNA片段化。将46μl的1μg基因组DNA转移至Covaris g-TUBE。然后将g-TUBE在室温下以根据制造商的方案建议所需的片段大小的速度旋转1分钟。然后将 g-TUBE倒置并再旋转1分钟以收集片段化DNA。将片段化DNA转移到干净的1.5ml EppendorfDNA LoBind管中。根据制造商的方案,通过使用Agilent Bioanalyzer 12000DNA芯片分析1μl样品来评估片段化过程的成功。
根据制造商的方案,用NEB的FFPE修复试剂盒以62μl体积处理回收的DNA,并使用1x Agencourt AMPure XP珠粒纯化DNA,之后在46μl无核酸酶的水中洗脱。
然后用NEB的Ultra II End-prep模块处理经FFPE修复的DNA,向片段化DNA的每个末端添加5'磷酸和单个dA-核苷酸。根据制造商的方案,使用45μl经FFPE修复的DNA在60μl体积中进行反应,之后使用1x Agencourt AMPure XP珠粒纯化并在31μl无核酸酶的水中洗脱。使用QuBit荧光计定量1μl回收的准备完成的DNA。
接下来,将30μl准备完成的DNA添加到干净的1.5ml Eppendorf DNA LoBind管中。然后添加含有如图3A所示的其上连接有Dda解旋酶的衔接子的溶液(20μl的OxfordNanopore 的SQK-LSK108衔接子混合物,其具有连接到每个衔接子的Dda解旋酶,如图3A所示(连接测序试剂盒1D(R9.4)的组分可从牛津纳米孔技术公司(Oxford NanoporeTechnologies))购得的(该体积的衔接子混合物优化用于~350ng DNA,其片段大小大于或等于8kb),然后添加50μl NEB的平端/TA连接主混合物,之后通过倒置5次将其混合。然后将反应物在室温下放置10分钟。
为了纯化DNA,将40μl AMPure XP珠粒添加到来自前一步骤的衔接子连接反应中,并通过倒置5次将其混合。然后将管在旋转混合器(Hula混合器)上在室温下温育5分钟。然后将管置于磁架上,使珠粒沉淀,然后通过移液移除上清液。在添加衔接子珠粒结合缓冲液(140μl Oxford Nanopore的SQK-LSK108衔接子珠粒结合缓冲液(可从Oxford NanoporeTechnologies商购获得的连接测序试剂盒1D(R9.4))的组分之前,将管从架上移除。然后通过轻弹管使珠粒重新悬浮。在这之后将管放回磁架上,使珠粒沉淀,然后通过移液移除上清液。将该步骤重复第二次。洗涤步骤后,将管从磁架上移除,使沉淀重新悬浮在洗脱缓冲液(25μl 的Oxford Nanopore的SQK-LSK108洗脱缓冲液(可从Oxford NanoporeTechnologies商购获得的连接测序试剂盒1D(R9.4)的组分))中。然后将管在室温下温育10分钟并放回到磁架上以使珠粒沉淀。将洗脱液移至干净的1.5ml Eppendorf DNA LoBind管中。
然后制备测序反应混合物以产生图3B中描绘的基因组DNA构建体。通过向12.0μl回收文库中添加均来自于Oxford Nanopore的SQK-LSK108试剂盒的37.5μl的RBF和25.5μl的 LLB(可从Oxford Nanopore Technologies商购获得的连接测序试剂盒1D(R9.4)的组分)来制备测序反应混合物。
为了产生具有将模板连接到互补序列的发夹的双链基因组DNA(参见图1A中构建体的示意图),按照与上述类似的程序。在类似的程序中,使用Oxford Nanopore SQK-LSK102衔接子混合物和SQK-LSK208 HP衔接子(可从Oxford Nanopore Technologies商购获得的连接测序试剂盒2D(R9.4)的组分),而不是上面提到的衔接子混合物,并且使用的所有其它步骤和组分与上面提到的相同。
使用Oxford Nanopore MinION R9.4流通池获得电测量值。将双链基因组DNA(有或没有将模板连接到互补序列的发夹)添加到纳米孔系统中。进行实验,并且监测解链酶控制的DNA 移动。
事件的比对
使用WO2016059427中公开的方法进行信号的比对。95%或更高的比对值表示各个事件指示模板及其各自的互补序列
比对信号的分析
使用如上所述的2D方法进行对比信号的后续分析,以确定核苷酸序列。
图1包括DNA构建体的示意图,所述DNA构建体包含与Y-衔接子和发夹连接的模板和互补DNA序列,在酶的控制下易位通过纳米孔(图1A),并且显示获得的测序准确度的图表(图1B)。图2A和2B包括DNA构建体的卡通图示,所述DNA构建体包含均与前导序列连接的模板和互补DNA序列,在酶的控制下易位通过纳米孔(图2A),并且包括显示获得的测序精确度的图表(图2B)。图1B和2B显示了说明使用单独的模板、单独的互补序列和来自模板和互补序列的组合信息获得的测序精确度的峰。通过比较图1B和2B,可以清楚地看出,当模板和互补序列未通过发夹环连接时,单独的互补序列的准确度增加(从~75%增加到 >85%),并且当模板和互补序列未连接时,将来自模板和互补序列的信息组合时,准确度也增加。
该实例描述了当模板和互补序列不共价连接时表征双链多核苷酸的模板(捕获的第一链) 和互补序列(第一链的反向互补)的方法。如下所述在数据分析后对所确定的模板和互补序列的鉴定在实施例中称为“跟随配对”。在一些实施例中,当该配对在彼此1分钟内出现>80%重叠时,则鉴定潜在的跟随配对。在一些实施例中,当跟随配对立即出现时,并且具有95-100%的重叠,则鉴定潜在的跟随配对。
仅模板链(即那些未被分类为属于跟随配对的链)在本文中称为“T”。属于跟随配对的模板链在本文中称为“T
在该实例中,使用含有与连接到经修饰的纳米孔的孔标签(例如,捕获多核苷酸)不互补的多核苷酸序列的对照衔接子来说明使用经修饰的纳米孔检测跟随配对的频率。
对照衔接子与基因组DNA的连接
对照衔接子是NB01(天然条形码1),包括条形码上链(SEQ ID NO:1)和条形码下链(SEQ ID NO:2)。
SEQ ID NO:1
/5Phos/AAGGTTAACACAAAGACACCGACAACTTTCTTCAGCACCT
SEQ ID NO:2
/5Phos/GGTGCTGAAGAAAGTTGTCGGTGTCTTTGTGTTAACCTTAGCAAT
使用Oxford Nanopore Technologies测序试剂盒按照制造商指导进行对照衔接子与基因组 DNA的连接。将1000ng末端修复的和dA尾的大肠杆菌基因组DNA在室温下以100μl与来自上面的5.5μl的640nM对照衔接子在1x Blunt/TA主混合物(NEB M0367L)中连接20分钟。如下进行样品的SPRI纯化:添加40μL的Agencourt AMPure珠粒(Beckman Coulter),用移液管混合样品,并在室温下温育5分钟。用不含核酸酶的水(Ambion
使珠粒在磁力架上沉淀并去除上清液。将沉淀的珠粒用500μL来自以上的70%乙醇溶液洗涤,而不破坏沉淀。去除上清液,再次用500μL的70%乙醇溶液洗涤沉淀的珠粒。去除70%乙醇溶液,并将沉淀物在离心机中短暂脉冲,然后放回磁力架,然后去除最后残留的70%乙醇溶液。
通过移液管混合使沉淀重新悬浮在50μL无核酸酶的水(Ambion
酶-衔接子复合物与对照-衔接子-基因组DNA的连接
将可从Oxford Nanopore Technologies测序试剂盒商购获得的等分试样的BAM(条形码衔接子混合物)在冰上解冻。将20μL的BAM在室温下以100μL与50μL对照-衔接子-基因组 -DNA、20μL的NEBNext Quick连接反应缓冲液和10μL的QuickT4 DNA连接酶(E6056L)连接10分钟。如下进行样品的SPRI纯化2:然后添加40μL的Agencourt AMPure珠粒(BeckmanCoulter),用移液管混合样品,并在室温下温育5分钟。使珠粒在磁力架上沉淀并去除上清液。用140μL衔接子珠粒结合缓冲液洗涤沉淀的珠粒,通过连续两次180
通过移液管混合使沉淀重新悬浮在25μL无核酸酶的水(Ambion
标签修饰的纳米孔的制备
制备经修饰的CsgG纳米孔以允许孔标签的缀合。例如,修饰CsgG单体(例如,通过氨基酸取代),例如提供半胱氨酸、非天然碱基等,用于孔标签缀合。使用含有编码氨基酸序列 SEQ ID NO:7,具有如本文所述的一个或多个氨基酸取代的质粒的PT7载体制备经修饰的 CsgG单体。
SEQ ID NO:7:无信号序列的野生型大肠杆菌CsgG的氨基酸序列(Uniprot登录号P0AEA2)
CLTAPPKEAARPTLMPRAQSYKDLTHLPAPTGKIFVSVYNIQDETGQFKPYPASNFSTAV PQSATAMLVTALKDSRWFIPLERQGLQNLLNERKIIRAAQENGTVAINNRIPLQSLTAANIMV EGSIIGYESNVKSGGVGARYFGIGADTQYQLDQIAVNLRVVNVSTGEILSSVNTSKTILSYEV QAGVFRFIDYQRLLEGEVGYTSNEPVMLCLMSAIETGVIFLINDGIDRGLWDLQNKAERQN DILVKYRHMSVPPES
将质粒转化到BL21衍生细胞系中,使其突变以置换具有卡那霉素抗性的内源CsgG基因。将细胞接种在含有氨苄青霉素(100μg/ml)和卡那霉素(30μg/ml)的琼脂板上并在37℃下温育16小时。使用单菌落接种100ml含有羧苄青霉素(100μg/ml)和卡那霉素(30μg/ml)的LB培养基,并且然后使起始培养物在37℃/250rpm下生长16小时。使用4×25ml起始培养物接种4×500ml含有羧苄青霉素(100μg/ml)、卡那霉素(30μg/ml)3mM ATP、15mM MgSo4和0.5mM鼠李糖的LB。使培养物生长直至达到平稳期,并且然后在37℃/250rpm下再生长2小时。添加葡萄糖达到0.2%,将温度降低到18℃,一旦培养物处于18℃,就可通过添加1%α-乳糖一水合物诱导蛋白质表达。将培养物在18℃/250rpm下温育16小时。
通过离心收获细胞并进行洗涤剂溶解(Bugbuster)。一旦溶解,就将样品进行初始链霉亲和素纯化(5ml HP链霉亲和素阱),将洗脱级分加热到60℃离心并将上清液进行qIEX纯化(1ml Hi trap Q HP)。合并含有正确蛋白质的级分,浓缩并在24ml Superdex上进行最终精制。
如下所述,用吗啉基寡核苷酸(SEQ ID NO:8)修饰上述纳米孔的等分试样:
SEQ ID NO:8:由GeneTools提供的吗啉基寡核苷酸
/5'/-GGAACCTCTCTGACAA/-3'-吡啶基-二硫代/
将1.3μL的1M DTT(二硫苏糖醇)添加到130μL来自以上的纳米孔中,其含有大约9.75 μg的纳米孔,并使其在室温下温育1小时。按照制造商的指导,使用0.5mL 7MWCO Zeba脱盐柱(Thermo Fisher Scientific)将该样品缓冲液交换到反应缓冲液(25mM Tris、150mM NaCl、2mM EDTA、0.1%SDS和0.1%Brij58,pH 7)中。按照制造商的指导,使用7MWCOZeba脱盐柱(Thermo Fisher Scientific)将该样品再次缓冲液交换到反应缓冲液中。通过将由 GeneTools提供的300nmol吗啉基寡核苷酸溶解在150μL无核酸酶的水(Ambion
电测量
从插入在磷酸盐缓冲液(例如,含亚铁氰化钾(II)和铁氰化钾(III),pH 8.0)中的嵌段共聚物中的单个经修饰纳米孔获取电测量。在到达插入嵌段共聚物中的单个经修饰纳米孔后,通过用2mL缓冲液冲洗去除任何过量的经修饰纳米孔。
使引发缓冲液流过纳米孔系统。为了制备测序混合物,将400nM系链(SEQ ID NO:9),回收的珠粒纯化的文库和文库负载珠粒按照制造商的说明在缓冲液中混合。然后将测序混合物添加到纳米孔系统中。在180mV下进行实验,并且监测解链酶控制的DNA移动。
SEQ ID NO:9
/5Chol-TEG/TT/iSp18//iSp18//iSp18//iSp18/TTGACCGCTCGCCTC
数据分析
当DNA链穿过经修饰的纳米孔时,测量并收集通过纳米孔的电流变化。然后使用碱基识别算法(例如,递归神经网络(RNN)算法)确定链的序列,以获得fastq数据。随后使用本领域已知的序列比对工具将fastq序列数据与参考基因组进行比对。
为了鉴定成对的相互互补的链(成对的模板和补体链),计算了链间的重叠分数。重叠分数定义为基因组中两条链共有的碱基的连续区段的长度(以碱基计),通过两条链所跨越(不一定重叠)的基因组部分的长度(以碱基计)标准化。计算每条链的最大重叠分数作为链与在该链的1分钟内(在链之前或之后)通过孔的所有其它链之间的最大重叠。因此,高的最大分数表明,给定的链属于互补对(作为模板或互补序列),而低的重叠分数表明该链不属于互补对。
表2显示了使用如本文所述的未修饰或修饰的纳米孔测量的具有不同最大重叠分数值的链的百分比
解旋酶,例如Dda解旋酶,例如国际PCT公开号WO2015/055981(其内容通过引用整体并入本文)中描述的解旋酶,用于控制多核苷酸移动通过经修饰的纳米孔的,例如,如国际 PCT公开号WO 2016/034591(其内容通过引用整体并入本文)中所述的经修饰的CsgG纳米孔。表3示出了如本文所描述的实例2-5的数据。它列出了映射链的数量(例如,使用本领域已知的映射方法),其被指定属于如上面数据分析部分中描述的T、T
在实例5中,在使用未修饰的纳米孔的情况下,跟随配对的频率较低(表3,第5行),仅为0.6%。这表明即使在分析物不含确切的序列时,在纳米孔上使用标签也会增强跟随事件。不希望受理论束缚,这可能是因为孔-标签可以低效率结合互补序列的暴露ssDNA(相对于确定的位点)。
该实例描述了当模板和互补序列不共价连接时表征双链多核苷酸的模板(捕获的第一链) 和互补序列(第一链的反向互补)的方法。如下所述在数据分析后对所确定的模板和互补序列的鉴定在实施例中称为“跟随配对”。在一些实施例中,当该配对在彼此1分钟内出现>80%重叠时,则鉴定潜在的跟随配对。在一些实施例中,当跟随配对立即出现时,并且具有95-100%的重叠,则鉴定潜在的跟随配对。
仅模板链(即那些未被分类为属于跟随配对的链)在本文中称为“T”。属于跟随配对的模板链在本文中称为“T
在该实例中,根据本文所述的一个实施例,通过跟随衔接子的连接来实现检测跟随配对的频率增加。如图9-10所示,衔接子在双链体茎内含有捕获多核苷酸序列,使得捕获多核苷酸序列仅在链解链时才显露。捕获多核苷酸序列与连接到经修饰的纳米孔的多核苷酸序列互补。在该实例中,捕获多核苷酸序列在双链体茎内不含间隔区,例如sp18,并且此类衔接子产生约10%的跟随效率(即,所有链的跟随%达到约10%)。
跟随衔接子与基因组DNA的连接
跟随衔接子的一个实施例包括条形码上链(SEQ ID NO:3)和条形码下链(SEQ IDNO:4),其分别以10μM和11μM在50mM HEPES pH 8、100mM乙酸钾中以2℃/分钟从95℃到22℃一起退火。杂交的DNA被称为条形码衔接子2。将6.4μL跟随衔接子添加到93.6μL的50mMTris-HCl pH 7.5、20mM氯化钠中以产生跟随衔接子1的640nM稀释液。
SEQ ID NO:3
/5Phos/GGCGTCTGCTTGGGTGTTTAACCTTTTT
SEQ ID NO:4
/5Phos/GGAACCTCTCTGACTTGGAACCTCTCTGACAAAAAGGTTAAACACCCAAGCAGACGCCAGCAAT
使用Oxford Nanopore Technologies测序试剂盒按照制造商指南进行跟随衔接子与基因组 DNA的连接。将1000ng末端修复的和dA尾的大肠杆菌基因组DNA在室温下以100μL与来自上面的5.5μL的640nM跟随衔接子在1x Blunt/TA主混合物(NEB M0367L)中连接20分钟。然后如实例2中所述进行SPRI纯化。该样品将被称为跟随衔接子-基因组DNA。
将酶-衔接子复合物与跟随衔接子-基因组DNA的连接
将可从Oxford Nanopore Technologies测序试剂盒商购获得的等分试样的BAM(条形码衔接子混合物)在冰上解冻。将20μL的BAM在室温下以100μL与50μL跟随衔接子-基因组 DNA、20μL的NEBNext Quick连接反应缓冲液和10μL的QuickT4 DNA连接酶(E6056L)连接10分钟。然后如实例2中所述进行SPRI纯化2。
标签修饰的纳米孔的制备
制备纳米孔的方法以与上述实例2中所述类似的方式进行,所述纳米孔经修饰w为包括与衔接子的捕获多核苷酸序列互补的多核苷酸序列。
电测量
当链穿过纳米孔时测量并获得电测量的方法以与以上实例2中所述类似的方式进行。
数据分析
以与以上实例2中所述类似的方式进行对收集到的电测量值的数据处理和分析。
解旋酶,例如Dda解旋酶,例如国际PCT公开号WO2015/055981(其内容通过引用整体并入本文)中描述的解旋酶,用于控制多核苷酸移动通过经修饰的纳米孔的,例如,如国际 PCT公开号WO 2016/034591(其内容通过引用整体并入本文)中所述的经修饰的CsgG纳米孔。表3的第3行展示了在使用如在此实例中描述的跟随衔接子的实例中,通过数据分析指定的T、T
该实例描述了当模板和互补序列不共价连接时表征双链多核苷酸的模板(捕获的第一链) 和互补序列(第一链的反向互补)的方法。如下所述在数据分析后对所确定的模板和互补序列的鉴定在实施例中称为“跟随配对”。在一些实施例中,当该配对在彼此1分钟内出现>80%重叠时,则鉴定潜在的跟随配对。在一些实施例中,当跟随配对立即出现时,并且具有95-100%的重叠,则鉴定潜在的跟随配对。
仅模板链(即那些未被分类为属于跟随配对的链)在本文中称为“T”。属于跟随配对的模板链在本文中称为“T
在该实例中,根据本文所述的一个实施例,通过跟随衔接子的连接来实现检测跟随配对的频率增加。衔接子在双链体茎内含有捕获多核苷酸序列,使得捕获多核苷酸序列仅在链解链时才显露。捕获多核苷酸序列与连接到经修饰的纳米孔的多核苷酸序列互补。该衔接子还含有增强C
跟随衔接子与基因组DNA的连接
跟随衔接子的一个实施例包括条形码上链(SEQ ID NO:5)和条形码下链(SEQ IDNO:6),其分别以10μM和11μM在50mM HEPES pH 8、100mM乙酸钾中以2℃/分钟从95℃到22℃一起退火。杂交的DNA被称为条形码衔接子2。将6.4μL跟随衔接子添加到93.6μL的50mMTris-HCl pH 7.5、20mM氯化钠中以产生跟随衔接子2的640nM稀释液。
SEQ ID NO:5
/5Phos/GGCGTCTGCTTGGGTGTTTAACC/iSp18//iSp18//iSp18//iSp18/TTTTT
SEQ ID NO:6
/5Phos/GGAACCTCTCTGACTTGGAACCTCTCTGACAAAAA/iSp18//iSp18//iSp18//iSp18/GGTTAAACACCCAAGCAGACGCCAGCAAT
使用Oxford Nanopore Technologies测序试剂盒按照制造商指南进行跟随衔接子与基因组 DNA的连接。将1000ng末端修复的和dA尾的大肠杆菌基因组DNA在室温下以100μL与来自上面的5.5μL的640nM跟随衔接子在1x Blunt/TA主混合物(NEB M0367L)中连接20分钟。然后如实例2中所述进行SPRI纯化。该样品将被称为跟随衔接子-基因组DNA。
将酶-衔接子复合物与跟随衔接子-基因组DNA的连接
将可从Oxford Nanopore Technologies测序试剂盒商购获得的等分试样的BAM(条形码衔接子混合物)在冰上解冻。将20μL的BAM在室温下以100μL与50μL跟随衔接子-基因组 DNA、20μL的NEBNext Quick连接反应缓冲液和10μL的QuickT4 DNA连接酶(E6056L)连接10分钟。然后如实例2中所述进行SPRI纯化2。
标签修饰的纳米孔的制备
制备纳米孔的方法以与上述实例2中所述类似的方式进行,所述纳米孔经修饰w为包括与衔接子的捕获多核苷酸序列互补的多核苷酸序列。
电测量
当链穿过纳米孔时测量并获得电测量的方法以与以上实例2中所述类似的方式进行。
数据分析
以与以上实例2中所述类似的方式进行对收集到的电测量值的数据处理和分析。
解旋酶,例如Dda解旋酶,例如国际PCT公开号WO2015/055981(其内容通过引用整体并入本文)中描述的解旋酶,用于控制多核苷酸移动通过经修饰的纳米孔的,例如,如国际PCT公开号WO 2016/034591(其内容通过引用整体并入本文)中所述的经修饰的CsgG纳米孔。表3的第4行显示了在使用如本实例中所述的跟随衔接子的实例中通过数据分析所指定的T、T
图11A-11B显示了来自该实例中描述的实验的电流迹线。将T、T
图12A-12D显示了跟随配对多核苷酸的模板(T
SEQ ID NO:10和SEQ ID NO:11是由图12A至12D的多核苷酸的Oxford NanooporeTechnologies碱基识别RNN算法推导的序列。这些可以高保真度进行比对以证明通过经修饰的纳米孔为来自随机片段化双链多核苷酸片段的模板和互补序列孔进行测序。
在图12A-12D中,用*标记的位置是在SEQ ID NO:5和SEQ ID NO:6中发现的Sp18间隔区,该基序的存在表明如本实施例中描述的跟随衔接子成功连接到多核苷酸上,并且该跟随衔接子的存在大大增强了跟随配对的百分比(如表3所示)。
图13以对数标度显示了“链间隔时间”,以秒为单位。标记“链”用于将解旋酶控制的多核苷酸序列通过纳米孔的运动的电信号分类。这使用本领域已知的方法进行分类。链容易因返回开孔而分裂。箭头表示来自实例4的数据中的显著群体,其使用如该实例中所述的跟随衔接子。该群体表示跟随配对的比例增加,因为在实例2和4中,T
该实例描述了当模板和互补序列不共价连接时表征双链多核苷酸的模板(捕获的第一链) 和互补序列(第一链的反向互补)的方法。如下所述在数据分析后对所确定的模板和互补序列的鉴定在实施例中称为“跟随配对”。在一些实施例中,当该配对在彼此1分钟内出现>80%重叠时,则鉴定潜在的跟随配对。在一些实施例中,当跟随配对立即出现时,并且具有95-100%的重叠,则鉴定潜在的跟随配对。
仅模板链(即那些未被分类为属于跟随配对的链)在本文中称为“T”。属于跟随配对的模板链在本文中称为“T
在该实例中,来自Oxford Nanopore Technologies测序试剂盒的标准组分与Minion和 Flowcell(包含未经修饰为包括如本文所述的孔标签的纳米孔阵列的SpotON流通池)一起用于证明在没有如本文所述的经修饰的纳米孔和跟随衔接子的情况下,检测跟随配对的频率。
对照衔接子与基因组DNA的连接
使用Oxford Nanopore Technologies测序试剂盒按照制造商指南进行对照衔接子(例如,如实例2中所述)与基因组DNA的连接。将1000ng末端修复的和dA尾的大肠杆菌基因组 DNA在室温下以100μL与来自上面的5.5μL的640nM跟随衔接子在1x Blunt/TA主混合物 (NEB M0367L)中连接20分钟。然后如实例2中所述进行SPRI纯化。该样品将被称为跟随衔接子-基因组DNA。
将酶-衔接子复合物与跟随衔接子-基因组DNA的连接
将可从Oxford Nanopore Technologies测序试剂盒商购获得的等分试样的BAM(条形码衔接子混合物)在冰上解冻。将20μL的BAM在室温下以100μL与50μL对照-衔接子-基因组 -DNA、20μL的NEBNext Quick连接反应缓冲液和10μL的QuickT4 DNA连接酶(E6056L)连接10分钟。然后如实例2中所述进行SPRI纯化2。
电测量
当链穿过纳米孔时测量并获得电测量的方法以与以上实例2中所述类似的方式进行。
数据分析
以与以上实例2中所述类似的方式进行对收集到的电测量值的数据处理和分析。
解旋酶,例如Dda解旋酶,例如国际PCT公开号WO2015/055981(其内容通过引用整体并入本文)中描述的解旋酶,用于控制多核苷酸移动通过经修饰的纳米孔的,例如,如国际PCT公开号WO 2016/034591(其内容通过引用整体并入本文)中所述的经修饰的CsgG纳米孔。下面表3的第5行显示了在实例中,通过数据分析指定的T、T
表3
表3显示了对于本文的实例2-5,确定为仅模板(未跟随其互补序列)、模板-n(跟随配对的第一链)和互补序列-n(跟随配对的反向互补序列)的链数。
表4
表4包含来自MinION跟随运行的单个通道(通道241)的图11中所示的链的分析数据。该表包含以下各列:
Fast5=链的唯一文件名,在MinION运行期间保存为fast5输出
开始=链的起始时间(秒)
结束=链的结束时间(秒)
持续时间=链的持续时间(秒)
基因组_起始_位置=在将碱基识别的链与大肠杆菌参考比对后,基因组参考中比对链的区段的起始位置
基因组_结束_位置=在将碱基识别的链与大肠杆菌参考比对后,基因组参考中比对链的区段的结束位置
间隔时间=对于配对而言,链的间隔时间(秒)
重叠率=对于配对而言,比较对准重叠(基因组_起始_位置和基因组_结束_位置之间)的重叠率
通过链间非常短的间隔时间来识别配对,并且当与参考比对时它们重叠。
该实例描述了当将低浓度DNA加入测序装置时增加测序DNA分子的数量的方法。
在该实例中,这通过产生经修饰的DNA-酶衔接子来实现,所述经修饰的DNA-酶衔接子含有与连接到经修饰的纳米孔的多核苷酸序列互补的DNA序列。在该实例中使用的示例性衔接子在图32A-35中示出。
衔接子制备(衔接子设计A,例如,如图32A所示)
通过以2℃/分钟将温度从95℃调节至22℃,使上链(SEQ ID NO:12)、阻断链(SEQID NO:13)和下链(SEQ ID NO:14)分别以10μM、11μM和11μM在50mM Hepes、100mM KOAc,pH8(总体积40μL)中退火。将退火的链与800μL的2.8μM解旋酶(例如,包括如本领域已知的野生型或其突变体的Dda解旋酶)混合,并在室温下温育5分钟。向该溶液添加10μL的8.1mM四甲基偶氮二甲酰胺,并在35℃下温育1小时。添加NaCl使最终浓度为500mM,添加MgCl
SEQ ID NO:12
/5SpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3// iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3// iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3/GGCGTCTGCT TGGGTGTTTAACC TTTTTTTTTT/iSp18/AATGTACTTCGTTCAGTTACGTA TTGCT
SEQ ID NO:13
/5BNA-G//iBNA-G//iBNA-T//iBNA-T//iBNA-A/AACACCCAAGCAGACGCCTAAGTCAGAGAGGTTCC
SEQ ID NO:14
/5Phos/GCAAT ACGTAACTGAACGAAGT/iBNA-A//iBNA-meC//iBNA-A// iBNA-T//iBNA-T/TTT GAGGCGAGCGGTCAA
衔接子制备(衔接子设计B,例如,如图34A所示)
通过以2℃/分钟将温度从95℃调节至22℃,使上链(SEQ ID NO:12)、阻断链(SEQID NO:15)和下链(SEQ ID NO:16)分别以10μM、11μM和11μM在50mM Hepes、100mM KOAc,pH8(总体积40μL)中退火。将退火的链与800μL的2.8μM解旋酶(例如,包含如本领域已知的野生型或其突变体的Dda解旋酶)混合,并在室温下温育5分钟。向该溶液添加10μL的8.1mM四甲基偶氮二甲酰胺,并在35℃下温育1小时。添加NaCl使最终浓度为500mM,添加MgCl
SEQ ID NO:15
GGTTAAACACCCAAGCAGACGCC TTT GAGGCGAGCGGTCAA
SEQ ID NO:16
/5Phos/GCAAT ACGTAACTGAACGAAGT/iBNA-A//iBNA-meC//iBNA-A// iBNA-T//iBNA-T/TTT TAAGTCAGAGAGGTTCC
连接制备
使用75μL 7k MWCO zeba离心柱,使用2根柱,每柱10μL,并遵循制造商方案,将衔接子设计A缓冲液交换到50mM Tris、20mM NaCl,pH 8中。正如使用
然后添加40μL的Agencourt AMPure珠粒(Beckman Coulter),用移液管混合样品,并在室温下温育5分钟。使珠粒在磁力架上沉淀并去除上清液。用140μL衔接子珠粒结合缓冲液洗涤沉淀的珠粒,通过连续两次180
通过移液管混合使沉淀重新悬浮在25μL含系链(SEQ ID NO:9)的洗脱缓冲液中,并使该文库在冰上从珠粒上洗脱10分钟。
孔修饰
制备经修饰的CsgG纳米孔以允许孔标签的缀合。例如,修饰CsgG单体(例如,通过氨基酸取代),例如提供半胱氨酸、非天然碱基等,用于孔标签缀合。使用含有编码氨基酸序列 SEQ ID NO:7,具有如本文所述的一个或多个氨基酸取代的质粒的PT7载体制备经修饰的 CsgG单体。将质粒转化到BL21衍生细胞系中,使其突变以置换具有卡那霉素抗性的内源CsgG 基因。将细胞接种在含有氨苄青霉素(100μg/ml)和卡那霉素(30μg/ml)的琼脂板上并在 37℃下温育16小时。使用单菌落接种100ml含有羧苄青霉素(100μg/ml)和卡那霉素(30μg/ml)的LB培养基,并且然后使起始培养物在37℃/250rpm下生长16小时。使用4×25ml 起始培养物接种4×500ml含有羧苄青霉素(100μg/ml)、卡那霉素(30μg/ml)3mM ATP、 15mMMgSo4、0.5mM鼠李糖的LB。使培养物生长直至达到平稳期,并且然后在37℃/250rpm 下再生长2小时。添加葡萄糖达到0.2%,将温度降低到18℃,一旦培养物处于18℃,就可通过添加1%α-乳糖一水合物诱导蛋白质表达。将培养物在18℃/250rpm下温育16小时。
通过离心收获细胞并进行洗涤剂溶解(Bugbuster)。一旦溶解,就将样品进行初始链霉亲和素纯化(5ml HP链霉亲和素阱),将洗脱级分加热到60℃离心并将上清液进行qIEX纯化(1ml Hi trap Q HP)。合并含有正确蛋白质的级分,浓缩并在24ml Superdex上进行最终精制。
如下所述,用吗啉基寡核苷酸(SEQ ID NO:8)修饰上述纳米孔的等分试样:将1.3μL 的1M DTT(二硫苏糖醇)添加到130μL来自以上的纳米孔中,其含有大约9.75μg的纳米孔,并使其在室温下温育1小时。按照制造商的指导,使用0.5mL 7MWCO Zeba脱盐柱(ThermoFisher Scientific)将该样品缓冲液交换到反应缓冲液(25mM Tris、150mM NaCl、2mMEDTA、 0.1%SDS和0.1%Brij58,pH 7)中。按照制造商的指导,使用7MWCO Zeba脱盐柱(Thermo Fisher Scientific)将该样品再次缓冲液交换到反应缓冲液中。通过将由GeneTools提供的300 nmol吗啉基寡核苷酸溶解在150μL无核酸酶的水(Ambion
电测量
从插入在缓冲液(25mM磷酸盐缓冲液、150mM亚铁氰化钾(II)、150mM铁氰化钾(III), pH 8.0)中的嵌段共聚物中的单一经修饰的纳米孔获取电测量。在到达插入在嵌段共聚物中的单一经修饰的孔之后,2mL缓冲液(25mM磷酸盐缓冲液、150mM亚铁(II)氰化钾、150mM 铁(III)氰化钾,pH 8.0)流过系统以去除任何过量的经修饰的纳米孔。
使引发缓冲液流过纳米孔系统。为了制备测序混合物,将引发缓冲液、(正如使用
解旋酶(例如,如本领域已知的Dda解旋酶,包括其野生型或突变体)用于控制所述多核苷酸通过经修饰的纳米孔的移动。图38显示了当多核苷酸易位通过未修饰的纳米孔时的电流迹线,即这是在纳米孔和分析物之间不存在寡核苷酸杂交的系统。在该系统中,链间隔时间均匀分布在1秒左右(参见图40)。
图39显示了当多核苷酸易位通过经SEQ ID NO:8修饰的纳米孔时的电流迹线,即这是允许纳米孔和分析物之间杂交的系统。在该系统中,存在两个链间隔时间群:(a)均匀分布在1 秒左右,和(b)快速捕获分析物(<0.1秒)(参见图40)。这表明分析物与孔杂交,而孔正在对另一条链进行测序。
两个系统之间的这种差异(孔和分析物之间没有杂交相对于孔和分析物之间杂交)汇总在图40中,其显示了两个系统之间的链间隔时间的直方图。这证明在没有杂交的情况下,仅观察到一种捕获类型,并且当分析物可以与孔杂交时,存在另外的捕获类型,其中分析物在前一分析物之后被快速捕获(<0.1秒)。这种减少的链间间隔时间增加了测序的链的总数图41。
分析物的两端都能够拴系到孔上。
以下是使用吡啶基二硫代吗啉基修饰纳米孔(例如,CsgG纳米孔)以具有连接到纳米孔外表面的孔标签的示例性方案。
标签修饰的纳米孔的制备
制备经修饰的纳米孔(例如,CsgG纳米孔)以允许孔标签的缀合。例如,修饰CsgG单体(例如,通过氨基酸取代),例如提供半胱氨酸、非天然碱基等,用于孔标签缀合。使用含有编码氨基酸序列SEQ ID NO:7,具有如本文所述的一个或多个氨基酸取代的质粒的PT7载体制备经修饰的CsgG单体。将质粒转化到BL21衍生细胞系中,使其突变以置换具有卡那霉素抗性的内源CsgG基因。将细胞接种在含有氨苄青霉素(100μg/ml)和卡那霉素(30μg/ml) 的琼脂板上并在37℃下温育16小时。使用单菌落接种100ml含有羧苄青霉素(100μg/ml) 和卡那霉素(30μg/ml)的LB培养基,并且然后使起始培养物在37℃/250rpm下生长16小时。使用4×25ml起始培养物接种4×500ml含有羧苄青霉素(100μg/ml)、卡那霉素(30μg/ml) 3mM ATP、15mM MgSo4和0.5mM鼠李糖的LB。使培养物生长直至达到平稳期,并且然后在37℃/250rpm下再生长2小时。添加葡萄糖达到0.2%,将温度降低到18℃,一旦培养物处于18℃,就可通过添加1%α-乳糖一水合物诱导蛋白质表达。将培养物在18℃/250rpm下温育16小时。
通过离心收获细胞并进行洗涤剂溶解(Bugbuster)。一旦溶解,就将样品进行初始链霉亲和素纯化(5ml HP链霉亲和素阱),将洗脱级分加热到60℃离心并将上清液进行qIEX纯化(1ml Hi trap Q HP)。合并含有正确蛋白质的级分,浓缩并在24ml Superdex上进行最终精制。
如下所述,用孔标签如吗啉基寡核苷酸(例如,如SEQ ID NO:8所示)修饰上述纳米孔的等分试样。将1.3μL的1M DTT(二硫苏糖醇)添加到130μL来自以上的纳米孔中,其含有大约9.75μg的纳米孔,并使其在室温下温育1小时。按照制造商的指导,使用0.5mL 7 MWCOZeba脱盐柱(Thermo Fisher Scientific)将该样品缓冲液交换到反应缓冲液(25mM Tris、150mM NaCl、2mM EDTA、0.1%SDS和0.1%Brij58,pH 7)中。按照制造商的指导,使用 7MWCOZeba脱盐柱(Thermo Fisher Scientific)将该样品再次缓冲液交换到反应缓冲液中。在无核酸酶的水(Ambion
分析和质量控制
SDS-PAGE-方案
将2uL经修饰和未经修饰的纳米孔添加到8uL反应缓冲液中。将样品在PCR块中加热4 分钟达到95℃,以使样品由寡聚体分解成单体。向每份样品中添加10uL的2x Laemmli样品缓冲液(pH 6.8的65.8mM Tris-HCL、26.3%(w/v)甘油、2.1%SDS、0.01%溴酚蓝)。将样品在4-20%TGX凝胶上以300mV电泳23分钟并使用SYPRO Ruby蛋白凝胶染色。凝胶的结果示于图27A中。
与吡啶基-二硫代吗啉基修饰的孔的杂交-方案
10uL经修饰的纳米孔具有相应的荧光杂交序列,其比经修饰的纳米孔过量2倍添加。将样品制成总体积为20uL。将每份样品在室温下放置1小时。添加5uL的5x无染料负载(pH 8.0的50mM Tris-HCl、25%甘油、5mM EDTA)。样品在4-20%TBE凝胶上以160mV电泳 80分钟。使凝胶在Cy3荧光的凝胶扫描仪上运行,使用SYBR Gold核酸凝胶染剂染色,并使用SYPRO Ruby蛋白凝胶染色。凝胶的结果示于图28-30中。
该实例描述了表征连接多核苷酸的方法,其中用于将多核苷酸连在一起的连接方法是点击化学。在该实例中,当模板和互补序列不共价连接时,使用纳米孔表征第一双链多核苷酸的模板(捕获的第一链)和互补序列(第一链的反向互补序列)。当模板和互补序列分离时,与连接到第一双链多核苷酸的跟随衔接子中的孔系链互补的序列在互补序列中暴露,并且互补序列结合连接到纳米孔的孔系链。串接衔接子也连接到第一双链多核苷酸,使得补体链可以串接到第二双链多核苷酸。
跟随衔接子与基因组DNA的连接
跟随衔接子包括条形码上链(SEQ ID NO:17)和条形码下链(SEQ ID NO:18),其分别以10μM和11μM在50mM HEPES pH 8、100mM乙酸钾中以2℃/分钟从95℃到22℃一起退火。杂交DNA称为跟随衔接子。将6.4μL跟随衔接子添加到93.6μL的50mM Tris-HCl pH 7.5、20mM氯化钠中以产生跟随衔接子的640nM稀释液。
SEQ ID NO:17:跟随衔接子上链
/5Phos/TAACGAGGTTGTTTCTATCTCGGCGTCTGCTTGGGTGTTTAACC/iSp18//iSp18//iSp18//iSp18/TTTTTGTCAGAGAGGTTCCAAGTCAGAGAGGTTCCT
SEQ ID NO:18:跟随衔接子下链
/5Phos/GGAACCTCTCTGACTTGGAACCTCTCTGACAAAAA/iSp18//iSp18//iSp18//iSp18/ GGTTAAACACCCAAGCAGACGCCGAGATAGAAACAACCCATCAGATTGTGTTTGTTAGT CGCT/iSp18//iSp18//iSp18//iSp18/AGCGACTAACAAACACAATCTGATG/DBCO/
使用Oxford Nanopore Technologies测序试剂盒按照制造商指南进行跟随衔接子与基因组 DNA的连接。将1000ng末端修复的和dA尾的大肠杆菌基因组DNA在室温下以100μL与来自上面的5.5μL的640nM跟随衔接子在1x Blunt/TA主混合物(NEB M0367L)中连接20分钟。如实例2中所述进行样品的SPRI纯化。该样品将被称为跟随衔接子-基因组DNA。
串接酶-衔接子复合物与跟随衔接子-基因组DNA的连接
前导链(SEQ ID NO:19)、阻断链(SEQ ID NO:20)和下链(SEQ ID NO:21)分别以5.5uM、6uM和6uM在50mM HEPES pH 8、100mM乙酸钾中以2℃/分钟从95℃到22℃一起退火。杂交的DNA被称为串接酶-衔接子复合物。
SEQ ID NO:19:串接测序衔接子上链
/叠氮化物/GGTTGTTTCTATCTC/iSpC3//iSpC3//iSpC3//iSpC3//iSpC3// iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3/GGCGTCTGCTTGGGTGTTTAACCTTTTTTTTTT/iSp18/AATGTACTTCGTTCAGTTACGT
SEQ ID NO:20:连接测序衔接子阻断链
GGTTAAACACCCAAGCAGACGCCTTTGAGGCGAGCGGTCAA
SEQ ID NO:21:串接测序衔接子下链
/5Phos/TCGTTAACGTAACTGAACGAAGT/iBNA-A//iBNA-meC//iBNA-A// iBNA-T//iBNA-T/
根据制造商的说明,等分试样的T4 Dda-(E94C/F98W/C109A/C136A/A360C)(SEQID NO:51,具有突变E94C/F98W/C109A/C136A/A360C并且然后(ΔM1)G1G2(其中(ΔM1)G1G2= M1的缺失并且然后添加G1和G2)在冰上解冻,然后通过0.5ml Zeba柱将50μl缓冲液交换到50mM HEPES pH 8,100mM乙酸钾,2mM EDTA中。使用A280 nm值将回收的蛋白质进行定量,并使用相同的缓冲液调节至0.25mg ml-1。
将27μl缓冲液交换蛋白与3μl串接酶-衔接子复合物在DNA低结合eppendorf中混合,并在35℃下温育10分钟。然后添加0.37μl的8.1mM TMAD,将样品在35℃下温育60分钟。然后加入30μl的50mM HEPES pH 8、1M NaCl、2mM MgCl2、2mM rATP,并在室温下再放置20分钟。
加入222μl的Agencourt AMPure珠粒(Beckman Coulter)并将样品在室温下在旋转器上温育5分钟。使珠粒在磁力架上沉淀并去除上清液。在仍然在磁架上时,用500μl的50mM Tris pH 7.5、2.5M NaCl、20%PEG 8,000洗涤珠粒,转360
将20μL的预装串接酶-衔接子复合物在室温下以100μL与50μL跟随衔接子-基因组DNA、20μL的NEBNext Quick连接反应缓冲液和10μL的QuickT4 DNA连接酶(E6056L) 连接10分钟。如下进行SPRI纯化,然后添加40μL的Agencourt AMPure珠粒(Beckman Coulter),通过移液管混合样品,并在室温下温育5分钟。使珠粒在磁力架上沉淀并去除上清液。用140μL衔接子珠粒结合缓冲液洗涤沉淀的珠粒,通过连续两次180
通过移液管混合使沉淀重新悬浮在25μL无核酸酶的水(Ambion
标签修饰的纳米孔的制备
制备纳米孔的方法以与上述实例2中所述类似的方式进行,所述纳米孔经修饰w为包括与衔接子的捕获多核苷酸序列互补的多核苷酸序列。
电测量
当链穿过纳米孔时测量并获得电测量的方法以与以上实例2中所述类似的方式进行。
数据分析
当DNA链穿过经修饰的纳米孔时,测量并收集通过纳米孔的电流变化。然后使用碱基识别算法(例如,递归神经网络(RNN)算法)确定链的序列,以获得fastq数据。随后使用本领域已知的序列比对工具将fastq序列数据与参考基因组进行比对。
该实例描述了表征和串接双链靶多核苷酸的方法,其中连接方法是非共价的。第一双链靶多核苷酸的补体链募集第二双链靶多核苷酸并使其达到孔的局部浓度。反过来,当对第一补体链测序时,募集的第二双链靶多核苷酸变得从补体链去杂交,而是以与实例2中进行的类似方式与孔系链杂交。这使得第一和第二(以及随后的,第三、第四、第五等)双链靶多核苷酸能够彼此跟随通过该孔,链间隔时间最短。当双链靶多核苷酸的浓度较低时,这特别有用,因为第二靶多核苷酸可以在为第一靶多核苷酸测序时募集。
以与实例7中所述相同的方式制备分析物,但使用SEQ ID NO:5、6、22、15和23,而不是SEQ ID NO:17-21。所有其它程序、试剂和条件与实例7中描述的相同。
SEQ ID NO:5:跟随衔接子上链
/5Phos/GGCGTCTGCTTGGGTGTTTAACC/iSp18//iSp18//iSp18//iSp18/TTTTTGTCAGAG AGGTTCCAAGTCAGAGAGGTTCCT
SEQ ID NO:6:跟随衔接子下链
/5Phos/GGAACCTCTCTGACTTGGAACCTCTCTGACAAAAA/iSp18//iSp18//iSp18//iSp18/ GGTTAAACACCCAAGCAGACGCCAGCAAT
SEQ ID NO:22:捕捞衔接子上链
/5SpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3// iSpC3//iSpC3//iSpC3//iSpC3//iSpC3/TTGTCAGAGAGGTTCC/iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3//iSpC3/GGCGTCTGCTTGGGTGTTTAACCTTTTTTTTTT /iSp18/AATGTACTTCGTTCAGTTACGT
SEQ ID NO:15:捕捞衔接子阻断链
GGTTAAACACCCAAGCAGACGCCTTTGAGGCGAGCGGTCAA
SEQ ID NO:23:捕捞衔接子下链
/5Phos/ACGTAACTGAACGAAGT/iBNA-A//iBNA-meC//iBNA-A//iBNA-T//iBNA-T/
这是描述表征和串接双链靶多核苷酸的方法的另一个实施例,其中连接方法是非共价的。这种方法与实例8完全相同,但使用与SEQ ID NO:9不同的系链。双组分捕捞系链为跟随序列和孔系链提供第二杂交位点,以增加所见事件的比例。
当形成测序混合物时,SEQ ID NO:9用400nM退火的SEQ ID NO:24和SEQ ID NO:25替换。所有其它程序、试剂和条件与实例8中描述的相同。
SEQ ID NO:24:捕捞系链上链
TTGTCAGAGAGGTTCCTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTGGTTGT TTCTGTTGGTGCTGATATTGCTTTTTTGACCGCTCGCCTC
SEQ ID NO:25:捕捞系链下链
GCAATATCAGCACCAACAGAAACAACCTT/iSp18//iSp18//iSp18//iSp18// iSp18//iSp18/TT/3CholTEG/
该实例描述了表征和串接许多双链靶多核苷酸的方法,其中连接方法是非共价的。第一双链靶多核苷酸的补体链募集许多其它双链靶多核苷酸并使其集中在孔附近。这样在孔周围提供比在一般本体溶液中更高的局部浓度,因此双链靶多核苷酸彼此跟随通过开孔,链间隔时间最短。当双链靶多核苷酸的浓度较低时,这尤其有用。该实例像实例8那样进行。然而,不是使SEQ ID NO:9退火,而是使用由与单链结合蛋白偶联的寡核苷酸组成的系链。
当对第一双链靶多核苷酸的模板链进行测序时,补体链作为ssDNA释放到溶液中。其它双链靶多核苷酸的单链结合蛋白能够与ssDNA结合。作为跟随过程的一部分,当对补体链进行测序时,补体链的3'被拉回到孔中。ssDNA补体链上的单链结合蛋白在遇到控制互补序列通过孔的运动的马达蛋白时从补体链中移位,因此沉积在孔周围,增加局部浓度。
本说明书中公开的所有特征可以任意组合。本说明书中公开的每个特征可以用用于相同、等效或类似目的替代特征代替。因此,除非另有明确说明,否则所公开的每个特征仅是一系列等效或类似特征的实例。
从以上描述中,本领域技术人员可以容易地确定本公开的基本特征,并且在不脱离其精神和范围的情况下,可以对本公开进行各种改变和修改以使其适应各种用途和条件。因此,其它实施例也在权利要求内。
尽管本文已经描述和说明了本发明的若干个实施例,但本领域普通技术人员将容易想到用于执行本文描述的功能和/或获得这些结果和/或这些优点中的一个或多个优点的各种其它装置和/或结构,并且此类变型和/或修改中的每一个被认为是在本文所述的发明实施例的范围内。更一般来讲,本领域的技术人员将容易认识到,本文中描述的所有参数、尺寸、材料以及构造意味着是示例性的,并且实际参数、尺寸、材料和/或构造将取决于发明传授内容所用于的一种或多种具体应用。本领域的技术人员仅仅使用常规实验将认识到或能够确认本文中描述的本发明的具体实施例的许多等效物。因此,应理解,前述实施例是仅通过实例方式来介绍的,并且在所附权利要求和其等效物的范围内,发明实施例可以按与具体描述和要求不同的方式来实践。本公开的发明实施例涉及本文中描述的每个单独的特征、系统、物品、材料和/或方法。此外,两个或更多个这样的特征、系统、物品、材料、试剂盒和/或方法的任何组合,如果这样的特征、系统、物品、材料、试剂盒和/或方法并不相互矛盾,被包含在本公开的发明范围内。
如本文定义和使用的所有定义应理解为先于字典定义,通过引用并入的文献中的定义,和/或所定义的术语的普通含义。
本文公开的所有参考文献、专利和专利申请均通过引用关于各自所引用的主题而并入本文,在某些情况下可涵盖整个文件。
除非明确相反指出,否则本说明书和权利要求书中使用的不定冠词“一”和“一个”应理解为表示“至少一个”。
如本文在说明书中使用的,短语“和/或”应当理解为是指这样联合的要素中的“任一个或两个”,即,要素在一些情况下共同存在而在其它情况下分开存在。用“和/或”列出的多个要素应以相同的方式解释,即“一个或多个”如此结合的要素。除了用“和/或”短语具体标识的要素,其它要素可以任选地存在,无论是与具体标识的那些要素相关还是不相关。因此,作为非限制性实例,当连同开放式语言,如“包含”使用时,提到“A和/或B”,在一个实施例中,可以指仅有A(任选地包括除了B之外的要素);在另一个实施例中,指仅有B(任选地包括除了 A之外的要素);在又另一个实施例中,指A和B两者(任选地包括其它要素);等等。
如本文中在本说明书和权利要求中所使用的,“或”应被理解为具有与如上所定义的“和/ 或”相同的含义。例如,当将列表中的项目分开时,“或”或“和/或”应被解释为包容性的,即包括多个要素或要素清单中的至少一个要素、而且还包括多于一个要素,以及任选地其它未列出的项。仅仅清楚地指示相反的用语,如“……中的仅一个”或“……中的确切一个”或者在权利要求中使用时“由……组成”将指包括多个要素或要素清单中的恰好一个要素。一般而言,当之前有排他性术语、比如“任一个”、“……之一”、“……中的仅一个”、或“……中的确切一个”时,本文中使用的用语“或”应当仅被解释为指示排他性替代品(即,“一个或另一个、而不是两个”)。当在权利要求中使用时,“基本上由……组成”应当具有如在专利法领域中所使用的普通含义。
如在本说明书和权利要求中所使用的,关于一个或多个要素的清单的短语“至少一个”应被理解为是指选自要素清单中的任一个或多个要素的至少一个要素、但不一定包括要素清单内具体列出的每一个要素的至少一个、并且不排除要素清单中要素的任何组合。这个定义还允许可以任选地存在除了在短语“至少一个”所指的要素清单内具体标识的要素之外的要素,而无论是否与具体标识的那些元素相关还是不相关。因此,作为非限制性实例,“A和B中的至少一个”(或等效地,“A或B中的至少一个”,或等效地“A和/或B中的至少一个”)在一个实施例中可以是指至少一个、任选地包括多于一个A,而不存在B(并且任选地包括除了B的元件);在另一个实施例中,可以是指至少一个、任选地包括多于一个B,而不存在A(并且任选地包括除了A的元件);在又另一个实施例中,可以是指至少一个、任选地包括多于一个A,以及至少一个、任选地包括多于一个B(并且任选地包括其它元件);等等。
还应该理解,除非明确指出相反,否则在本文要求保护的包括一个以上步骤或操作的任何方法中,该方法的步骤或操作的顺序不一定限于叙述的该方法的步骤或步骤的顺序。
序列表
<110> 牛津纳米孔科技公司
<120> 方法
<130> N413849WO
<150> GB 1809323.7
<151> 2018-06-06
<160> 53
<170> PatentIn版本3.5
<210> 1
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 对照衔接子NB01(条形码上链)
<400> 1
aaggttaaca caaagacacc gacaactttc ttcagcacct 40
<210> 2
<211> 45
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 对照衔接子NB01(条形码下链)
<400> 2
ggtgctgaag aaagttgtcg gtgtctttgt gttaacctta gcaat 45
<210> 3
<211> 59
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 跟随衔接子(条形码上链)
<400> 3
ggcgtctgct tgggtgttta acctttttgt cagagaggtt ccaagtcaga gaggttcct 59
<210> 4
<211> 64
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 跟随衔接子(条形码下链)
<400> 4
ggaacctctc tgacttggaa cctctctgac aaaaaggtta aacacccaag cagacgccag 60
caat 64
<210> 5
<211> 59
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 跟随衔接子(条形码上链)
<220>
<221> misc_binding
<222> (23)..(24)
<223> 核苷酸被4 × iSp18分离
<400> 5
ggcgtctgct tgggtgttta acctttttgt cagagaggtt ccaagtcaga gaggttcct 59
<210> 6
<211> 64
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 跟随衔接子(条形码下链)
<220>
<221> misc_binding
<222> (35)..(36)
<223> 核苷酸被4 × iSp18分离
<400> 6
ggaacctctc tgacttggaa cctctctgac aaaaaggtta aacacccaag cagacgccag 60
caat 64
<210> 7
<211> 262
<212> PRT
<213> 大肠杆菌(Escherichia coli)
<400> 7
Cys Leu Thr Ala Pro Pro Lys Glu Ala Ala Arg Pro Thr Leu Met Pro
1 5 10 15
Arg Ala Gln Ser Tyr Lys Asp Leu Thr His Leu Pro Ala Pro Thr Gly
20 25 30
Lys Ile Phe Val Ser Val Tyr Asn Ile Gln Asp Glu Thr Gly Gln Phe
35 40 45
Lys Pro Tyr Pro Ala Ser Asn Phe Ser Thr Ala Val Pro Gln Ser Ala
50 55 60
Thr Ala Met Leu Val Thr Ala Leu Lys Asp Ser Arg Trp Phe Ile Pro
65 70 75 80
Leu Glu Arg Gln Gly Leu Gln Asn Leu Leu Asn Glu Arg Lys Ile Ile
85 90 95
Arg Ala Ala Gln Glu Asn Gly Thr Val Ala Ile Asn Asn Arg Ile Pro
100 105 110
Leu Gln Ser Leu Thr Ala Ala Asn Ile Met Val Glu Gly Ser Ile Ile
115 120 125
Gly Tyr Glu Ser Asn Val Lys Ser Gly Gly Val Gly Ala Arg Tyr Phe
130 135 140
Gly Ile Gly Ala Asp Thr Gln Tyr Gln Leu Asp Gln Ile Ala Val Asn
145 150 155 160
Leu Arg Val Val Asn Val Ser Thr Gly Glu Ile Leu Ser Ser Val Asn
165 170 175
Thr Ser Lys Thr Ile Leu Ser Tyr Glu Val Gln Ala Gly Val Phe Arg
180 185 190
Phe Ile Asp Tyr Gln Arg Leu Leu Glu Gly Glu Val Gly Tyr Thr Ser
195 200 205
Asn Glu Pro Val Met Leu Cys Leu Met Ser Ala Ile Glu Thr Gly Val
210 215 220
Ile Phe Leu Ile Asn Asp Gly Ile Asp Arg Gly Leu Trp Asp Leu Gln
225 230 235 240
Asn Lys Ala Glu Arg Gln Asn Asp Ile Leu Val Lys Tyr Arg His Met
245 250 255
Ser Val Pro Pro Glu Ser
260
<210> 8
<211> 16
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 吗啉代寡核苷酸
<400> 8
ggaacctctc tgacaa 16
<210> 9
<211> 17
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 系链
<220>
<221> misc_binding
<222> (2)..(3)
<223> 核苷酸被4 × iSp18分离
<400> 9
ttttgaccgc tcgcctc 17
<210> 10
<211> 12068
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 碱基识别_比对_模板 90e5fe72_读取_ch2_文件10
<400> 10
tggtttgtca gagaggttca agtccagggt tccttgcctg tcatctatct tcggcgtctg 60
cttgggtgtt aacgcctctg caccggacgg tcccctcgcc cctttgagga gggttaggaa 120
gagaaaaccg cacctgtacc ggatcggtta tttcgcattc ttatcagacc cactgcaacc 180
gctgaatatg caggcataac aggtgcagga tcaccaggca atgcagcagg taaatatcat 240
tgccgtccca gactttggtg gtgaccttat tgtccacgtc cagctcctgc caccgggaac 300
catttcatag tccatccagg tacttatgca gtactctccc accatgtttg ataccaggtt 360
tccttggcga tcgccagtat tgacggtgta aacgcgtagg ccgtacccat tgcttcgacg 420
ataggccagc gtacacgttc gcggaccacc ggtttccttc ccagtcaacg acataaacgg 480
ctccgtccgc gccatcggtg cccaggcatc gcgcacggtg gcgttaaacg aacctttggc 540
atcttctagc aacctgctgg tggttgttcg caacgggctt cccagggcca tatggatgta 600
cggcggcatt aaacggcccc attcgatccg gtggcctggt gcactcgggc gcgcggaagc 660
gatgcgccgg gttatctttg ttgtaatccc ggcagccgga ttccactggg tatcagtgtt 720
cgtgctgccc gataatgatt atttctggcg acatcgtgga tgtcacggaa gccacgcgaa 780
tcgcgcgatc agccattttg tcggtgacgt cataaacaat cgaagcttcc accgcgtgca 840
tattggcatt gccgccgcgg tactcttcga ttttctgaag gcttcgtccc gggattcagg 900
catatcttct tctccagaaa tatttctcga taatttcgta tgggccttaa tcagtagcgc 960
gcttccaggt gaccgtttgg cgacggcgct ggcggcaccc agcggaaaca aagtgttagt 1020
aagccctgtt gcaggcatcc accacgccct ctgatgccgt tcgcacgcgg cgtaccagcc 1080
gccataattt tatccgcacc atcgccgttc atggctttga tgccgtgatc aaccaacgat 1140
ggcgccaggt cagcccatcg ccgcaaccag cgatcgcgtg caacatacga gcggtgatcc 1200
acagatgggt gcccatctct tctttgattt gccctttgtg cctaaccagc catgcagtcg 1260
gcactacgga attttaccaa aatcaaagat gcggtcggtt tcctgttcag ccaacggttg 1320
gcagcagggt gttaaaccat ttcattcata ccatctcatc ttggcggcgt ttagccatca 1380
tttcgtcgac gatatcgcca agttgttgta atttctgatt acaaacgtca cgcagcatca 1440
gctcgttgtc tggtaaaccg acgaccgtct tgaactgcac ggcctgccag gaatccccga 1500
taacccaccg tcgttactgc cacgcgtacg acgcaggaac gatttttcgt cgacaccgaa 1560
gagatctcac ccatggcgcc attaatgctg tcattcagac gttgtgaagc acagaaagct 1620
cttgttgcgg acctttgcca taaagcggca ttttcagctt tgtgaaggtc agcgccactt 1680
tctcgcccgg ctctttgagc acggcatcga tgatcagcaa ccgcgatcga atttatgctc 1740
acggcacaac ggtgggcgga cgctggttcg tgatgcttac cagaccgtga gtggcacagt 1800
tcgttgaact ctttcaccat atcgaagacg ttgctgcgca tcttcatcac tacgccacag 1860
caccagcagt tttaggcttt gccaccgtct tgtttgatct tgtggattga ttttgcggtc 1920
agcaaccacg ctatcgtgga gattcgttct gggaatgaac tttcatcggc ggcgaccgca 1980
atcatggcgc aactttgtaa tcgcattttg ctcaaccacc tggcggtaac cgtgacgatc 2040
taccaagtcc ttgagcataa ggcgaagacg cgccgccggc gttaacacga aatccagtta 2100
aaacgctact cctgagcttc tggaagggcg gtgccccagc cgcggcaaac atcatgcacg 2160
ctaaacttct gcgctgatcg acctgctgcg tagcaaaacc gccgatgcgc gtaatgtcgt 2220
tgatggtgtg cttattcatt catttattcc ttttatccga tcgttacatt ttactttggc 2280
agtcatccca gcgcgacacg gacctgttca agaatggcgg accgatctgg cgacccgcga 2340
cccgctgcgc gcctggctat aaacctccca tcgggcagcc cagcggcatt ggcacatgca 2400
gctggttggc gacatcaagg gcgatgcaag attcttctgg gcaagatcga tcatgaaggc 2460
gggaaaagat ctccgctgag gactttgttt ggccaggaag tggtgaagtg acctttaccg 2520
gcagcatttg ctttcatcgc gacggcaaca tcgaggaagt tagggcttcg cacaaacggc 2580
aacttctgcc gaaagcacgc caggcgcaat gcctcgccag ttgttgatga gcaacgcgga 2640
tccccatgcc cggaccgcca gcgttgatca actcactgcc gctcaccatc ggatcggtgt 2700
ggcacgtcca actgttcaac ggtgccgcca gccagccagt aacagagtac cggtaatggc 2760
atttgcagaa gtacggccta ccggtgggtg tcatcatact gaaactgcgg caccatcggc 2820
aatcaattcg gttttgcagc ggatggatgg tggacatatc aatgaccaac acatggtgat 2880
aaagccttcg caaaacgccg tttcaccgaa caacacgttg cgcaccagat cgccattcgg 2940
caacatggta tgataaattc ggcatcttta gcggcctgcc ggttggcggc ggggtcacgc 3000
tgtctaccag atactacacc ttcggcgttc acatcaaaga cgcgaagttg atgcccttgc 3060
tgcaataaat tgctcaccgg tggcggacca tttgtcctaa accgataaac gcgattgctg 3120
ccgcaaccct cctcctagaa tatggtgtgc tcacttttgt catttatgac atgctttgct 3180
tgtctgtttt gatcgtattt gtaatttatc gtcaagattg acagccgtca ccccaaacaa 3240
ttggtgaggt ggtagacgca tcccaaatca ctgagaatga ccatgattca ttgttgccaa 3300
tggccaagta ccaaccgtga tggatcgcat ctattacgtg gaaggaatac cgacggagag 3360
cgtgaaaccg tggcgaggta tactggagag ttggtggcgg gccagcggcg acggcggcgg 3420
ttgcggcagc aggctgcgag gcgcaaggtc atttattggt cacgtgggtg atggcggctg 3480
gtggcaacct gctggcagag ctggaatcct gggcgttaac acccgttacg caaacgagca 3540
taaccaggcg aaatattgca atcaccatca tggtggatac aaggcgagcg gataatcatt 3600
aactaccccc atgccggatc ctgctgcctg acagagtggt tggagaattg atttctctcc 3660
ggtgggatgt tggctggcag atgtacgctg gcacgacgag cgctaaaagc cttcgccctg 3720
gccgtcaggc gggtgtgatg accgttctgg acggggacga tgcgccgcag gaccatccgg 3780
tgaaactggt ggcattaagc gatcacgcgg cctttcagaa ccggtctggc agcaacgggc 3840
gtgaagatgg cttggtgcgc taaaacaggc acaaacgctc taaatggtca tgtctatgtg 3900
accagggtag ccgcgagctg cgatagctgg aaaatggtgg gcgtcgcatc gtagccttca 3960
aagttgatgt ggtagatacc acaggtgcgg gtgatgtttt cacggcgcgc acagcggtgg 4020
cactggctgg caacaagtgg ggatttagcg agtcggtcca actacgtaac agtgtggcaa 4080
cgttaaaata cgtcggtgga cgaccgggat ctgactgtga tcaaacccga tctttgtcac 4140
ttttgtatga aatgccaggg gtgatggttt cgaggaattt tcgcaggcct taccgaacta 4200
accggtaacc cgcagcgacc aactcctcct gctgatcgcc gagcgggtat atgaatattg 4260
atgagctggc aaatcgctgg atgtctccac acagacggtc cgcgggatat tcgtagtcat 4320
gagcaggctg attacgcact gcagctcacg gtggcgcggt cagctcgcat cagtgtgacg 4380
ttcggcagcg tgaggtttcg caagctcgag aaaaacgttg ccgaagcggt ggcgaactat 4440
attcctgatg gttcaacaat atttatcacc agtggtacgg cttctattcg acgtatttgc 4500
ccaggcgttg ccaaccataa tcatttacgg ataatctacc agcttgaagc ctcgtgtggc 4560
gcatattgct acaacccgcg ctttgaagtg gtgcccggcg gtacgttgcg ctctcgccaa 4620
tggcgggatc attggccttc cggcggcgtc gctgtggccg atttcgtgcg cgattatcgg 4680
taacaagcgt tgccggccgg tgagcgatgg cgcgttgatg gagtttgata agcagaaact 4740
aacggctggg tgaaaacgat gatggcgcac ccgaaatatt ctggtcgccg atcacacaag 4800
tatcatgcct tcggcagcgg ttgaaattga taacgtggca caggtcactg cgctcttacc 4860
gacgagctgc cgcccgctgc gctaaatcac gcttacaaga caataggtaa atcattcttc 4920
cccaggagag acgcgcgtta cgaccgttaa cctggccata ccttgccaca acccaaaccc 4980
atcctttcca ctacagttaa tttcttgtgg cgcgaaagac acaaatactc tatatctttg 5040
atttgagtaa tgtgattatc aatgtcaaca ccaacgtgtg ctggggcctg ggcgattgcg 5100
catgttactt cagcatcgct taagaagagt ttattgctgg gagcgtttca tctggcatgg 5160
gcattgaaat tagcgacaag cgttcacaga gagcgctgta tgtctgaatg gctctaccac 5220
taagctacga cagttctctc acggctggcg acagtgtttg ttgcgctacg ccggaagtga 5280
tcgccgtaat gcataaacta cctgcgtgac aggggcatcg tggtggtgct ttccaatacc 5340
aacctgcata ccaccttcta gcccggaaga atacccgaaa ttcgtgatgc tgctattata 5400
tctatctgtc gcagatctgg ggatgcgcaa acctgaagca cgaatttacc agcatgtttt 5460
gcaggcggaa ggttttcacc cagcgatacg gtctttcgac gataacgccg ataatatgaa 5520
gggcaatcaa ctgggcatta ccggtgttcg gtgaagataa accaccatcc aagctatttc 5580
gcagtgttat gtaaaaacca ttcaggacaa aactaaggca ccatgccatc cactattggg 5640
cctggcacaa aactactctg gcagcacgta ttgaggacaa catgacaacc tggcggtaac 5700
cttgcctatg catttatgct ctcggctaat tcgccgctgg ttgccgttgt tttgcgcttt 5760
tcgccgcttt tcccatgttt tccgacgtca gcattcagtt acgtcacttt attattttgt 5820
aactttctgc ctgctgcagc gatgttattc agcggtatat gaacaatttg ttgccaattc 5880
aacaagatga ccgccgttgg ggctgatggg ctgatcgtca ccgcgttatt gttgatgtac 5940
tccatccgat agcgcgttga ataccatctg gcgcagtaag taagcgcgac caaaatttac 6000
tcgttcaccg tcactggtgg attttaacac taaagacccg cttctggcaa gggccagtct 6060
ggcgatcagt tcctgttgct ctccctgcgc tgggcgaaac gtctcgctct gtcatcgata 6120
acgtgcgtat ttttcccgtt gctgttgtcg tggatctctc cttctggttg ccagctggca 6180
ttgttctacc atccgtacct aaccgcggcg cgattgtcag cgtttgtcgc cacactcctg 6240
ttcgaagcag gaaaggtttc gcgcatatct acatgttccc gtctatcagc tcatttatgg 6300
tgtgcggcgg tgatccccat tctttgtttg ggtctactgg acatggtgta tcgtcttcgc 6360
agcgccgaag tactgtcact ctcgggaata ccgcaaaagc taaaacaagc agcttcgaca 6420
agaagacgac gaaccatgat tgcattaatt caacgcgtaa cccgtgccag cgtcaccgtg 6480
gagagaagtg ggcgagggag tagctgggac tttgcggtgt tattagcggt gctcaaaggt 6540
gtgaacgaaa gcaaaccatc tgtgcgagca ttaacagcta ccgcatcttt atgatgcgag 6600
gcagatgaat ctccagcgat gcaacgggcg gcggcagtgt gctggtggtt tcccagttta 6660
ccctcgccac cagatattct gaacagggga tacttaagtt tctctcaggt gcatcacgga 6720
tcgcgcaggg cgttatatga ctatttcgtc gaacgctgcc gtcggcaaga gatgaacaca 6780
ccgacaggcc gcttcgctgc ggatatgcag gtatctggtc agccggccag ccccatgtga 6840
cattctgtga tttgcagtgc tgagccagct tccggtttgt cacgggaaac gaaagtatcg 6900
ctatgtatct gccaggttcc acaaacagaa gaagaattga gagcgttact atcagtttcg 6960
ctggaaatgt tgcgcccttc atcaacaggt tcggaacacg cgcgtgggat gcgatggcgc 7020
atcgcagatg gtcacgtcga cgacagggta atctcggtaa cggtaaactg cgactgtata 7080
ttgtctaccg acaatgaagc gtccattcaa catggccgtt catcccggcg tgcgggacaa 7140
gggttaggcg ctgatggcga tgaccacagg tcggtggcgc gtcaggaaga gcgttaagcg 7200
catgactaac agcgcccgtg aagacgcggt gcggagtttt cgccagcggg tttgtgtcag 7260
ggagaaatca ccacataacc acgccgattc gccatttttg atgattaagc ccgtcgcctc 7320
gttgacattc accgcatcgc ggcgactggt gcgcctggct gtgggcggta cgaacatatc 7380
gcgggtgaaa aatgggcgtg cgcattcagc aatataccgg gcaaaattta tcactaccat 7440
gccgaaaccg gcaatcaagt ccgcaccata cgctgtttac cgaagacatt tctcggcgac 7500
gctcactgga ctggggactt atctgtgata cgcgtgaacg ccacctcggc gaacgattat 7560
tcttgcggat acgcatatcc acttcaacca gccgattggc ggtaaacctc gtgcggtacg 7620
ccgacctcgg tatacgcaag cggcgatctc gaccgtcgcg cgcggacgaa agcaggtgca 7680
gatgcaggtc gaaatctttg gcaacagggc taccgggtgc agtgtttgaa ggcacgtatg 7740
tcgttctgct tgcccgcgaa agccatttgg cccgtatgaa ggcgggcgag tagctgatgg 7800
aaggcgaaca cctctctctc ttcaggtgca ttatgataga tcgccaagct atttcagttg 7860
ctgtaaataa gaactctata agttacgcat tcttcttcgt tggctccatg cggtgtcttg 7920
ccacatcact gttcaggagc atttaaataa tactttagcg aactggtggc aattatcacg 7980
ccgaatagcg cttattccat catcttagtt catgtttgag cgatttttga tagtcaaaac 8040
cccactctgg tgccatactt ttctcttaaa tactttaaaa tctatagcta acaaacaata 8100
tacaaagata atggatagct cgtcagtatc aaccatttac cctttcttgt gttatgaata 8160
ttatgttgta tgtttgctct taaaccgtgg gaaggaggct ataatggttg ccaatataat 8220
ctatttctgt gaaagttatt ctgttgttaa tttgaaaaca actttcccag actgttacca 8280
acaaaattat atgatgaaca agtaaagacc ggttggtggc agtactacaa ccgatgcgat 8340
gaagattaat ttcaccgcca aatcctggtg ctgttaggat gctggcgttt tcggctacac 8400
cattcaagat tatgcaaata atatctcaga ccaacaatat attcttaaca cccataattc 8460
atctgagcga tactaaacca ttttgagtaa taagtaaaca gctacagcac tcttattatt 8520
tctggaaaac gcctcgcaaa aacaaaacat aaaatccgct ttttaaatga ttttccggcg 8580
ccattttcgc gccacattaa tgagtaatat aatgagtatt agtaatgtgt gtatgcggtt 8640
ttcttcattt acatcgaaga accgttaaac ggctgaacaa gcttcggggc tggcgtaagg 8700
tttctgatca atttacgcga agcggtagag caatatctgg aacgtcagca attcccgaat 8760
acgtaaaaca aaggagagca ggcaaaaggg aggtagttgg tgttgatgat cgttaggagc 8820
ataagggaat gacaagaatt ttgttcttga atataaaaac agatgccctt attctggtat 8880
taatgaggct gtttgcgaac ttataataac tgcaactgtt acatcatatc tggaaacgcc 8940
tcgcaaataa aaatgatgca ataaatagag ctttcttcaa gattcggcaa ggtacttttc 9000
tctccatgtg gattcaaaat gatattggcg gtgttatgaa ctctctggca gtacagatat 9060
gggcgggaat tttactcgat ttatcgaatg gggtgattca aagcaacttg atgatctcga 9120
aggtgcagcg caatgtttcc tcgtgcggac ctattaaggg aagcggtaga tcatacctga 9180
taatcaaatc aaacaagcaa gaaccaatgt tcctggcatc tggcaaggaa ggtgcagaag 9240
atgattatcg aatatcggcg taagctgcgc gagaatggtg tgggaaaatg atgtcaatca 9300
gattgctgac aacgtgcccg ttgttcgaaa tgccggatgc ggcgtgaacg ccttatccgg 9360
cctacaaagt cagtataaat tcaatgtatt gcagtttatt ggtaggcctg atggctggcg 9420
catcaggcaa tcttgctgtc atcggtctca tgcccataac cttattgatg tgggtggtgg 9480
gtaaattggt aaaccggcac tggcggcatg gcggtgttaa cgcggaatac gagatggtgt 9540
cgtcaccacc taacgagctg taatccgggc gacctgcgcc accggaaaac gtcggggtac 9600
tcttcacccg gaaacaggaa cgggttgatg gaatgcgaac tcttcttctt gtattcgtgc 9660
gtccaaagta ccaagaggcg aaggtcatct actacggctt cgttttggtc tttttcctga 9720
taagaaggta ctggtagtgt cgctggtcat cgcagctttc tggctttaat gcccggccta 9780
tttcgtcatc agcagccagt attacggttt gccgctctct gcggattgct gcattcccgc 9840
gcactacgtg ccattcagtt tcacgggtta cggtgcagta acaccttcgg tggtgccaat 9900
ttgcaccatg ctggacttcg gcagcagaac cacaaaccgg gcaatattga cgttgctcgc 9960
caaacattca gcgcgggctt tgccggatca ggtcggccat gcgcccggta ggcgacagtg 10020
cagccagata aacggcgctt tatcgctgct gacggacgag aaaatcagga gcaaacagcg 10080
cgctggccat atcttccagc tcctgagtca ttaccttctc cagattctca atcactgcca 10140
ccagcgccgg gccgctcatt tcaggtttca gctcggcaat cagcgccatc aagcagcttt 10200
ccggtgctta tcgcacggca ggcggaatat ccagcgggct tgccttgtgc gctggcttct 10260
ttaatgcgcc tgcagtcaga tccatctcca gcgcacgatt tgttcgtaca gcaccacttc 10320
ctgggcggta aggcgtcagc tttgtaaagc gcaggtaatc gcagaggatt tattttctgc 10380
cagctcgcgc agacgctcgg cgcggcggtt gtataaattc ttgagccgag gaacaataac 10440
ggcggaatcg catatccacc gtacgtttct cgcgaaccca gctcatcttg cggattatgc 10500
gaatattcag ctctttccgt tgtcttgcag ttccgcattt tctgctgccg ggtgatgttt 10560
cttcgccctg gcattgtatc cttccaccat ggcattgtcg tgcctttcac ccaagggtga 10620
atgccaccat gataaccctg aataacgcca cctgcggcaa atagatgcaa cacgtgctgt 10680
agaatcggat caccggattg aaagcaaggc gcaaaataag gacgccagat aatcacgcgc 10740
tcgcagcagc agaaccggga aaataatcgc accgcccgaa gaaacgcatt tcggccgaag 10800
ttataacgcc cgtatactca cctcctcgtt gacgacgatc ttacgaatat tcttcttcaa 10860
aagatagcat catccccgat tgattggttg tgatgcccag taacggaaaa acggtgatga 10920
accctgagac aaaacataaa ccacgcaccg acaaacgggt gcagaattct acatatacag 10980
ctgcgaatac cgcggttgca tcaaccagtt gaaggacggg gcaaaagccg ctcaccgccg 11040
ccggggatga agcggccacg gtgatccagt ggttgatacg ttccagcgcg gtgcagcgca 11100
cggtggtgtc gtcgtttcat ttgcgctctc gtcttctctt cgtgcagatt attctcttcc 11160
tcatcaccac ggttcaggcc gacaccccat agtagaagat actggccgca agggccagcc 11220
gcaaaataac agccgcgagc ggtttcagat gcctttcaga atttgcggtt tcttcccggt 11280
tcgggttctc cggcaaacca tgatactgaa tttggcttgt caacgtggtg caacacatta 11340
cgtgtactac cgacataccg gatcgtacag acccacattg tcgtaaccac caggttttca 11400
gctcagcaac gcgctcgctc tttccaacgt tttcatcaaa ctcgtacaag tgaatccgcg 11460
cccgttgggc aggtcttcac ctgagctgaa gttctaccaa ccaccgcacg gatcgacgca 11520
gcgtacattg gcgaacacgg ttgtcagcgc aggtttgagc gcggggtaat gaacggacag 11580
cctggtacat gccaccaacc aatgcactgc tcggctgaag tcgacattac cgttgacacc 11640
tgaataattg cacgctacac caggcacgcc ttcaggcagc ctggatggaa cagtacgcca 11700
cagccgtctt tgcggatcaa agccattcag tttgtctggc tgtccacttc gagcgcatta 11760
ccgtccacga tttggcgcta aatcattggg gttgtcgtac accccaatgt tattgccgac 11820
attgtatcgc agtgtcgttc cctctgaaca cgccaccaca accgggcttt acagccgata 11880
caggtggtaa cgtcgatgag tttcgcccac ttcttcctgg aagtcccgcg cctgaggcgc 11940
ggggtcgaac attagtcgcg gaacgacgaa taagttaaac atgacagacg ccgagataga 12000
gcgacgagca aagtaaggaa ccccgcctct ctgacttgga acctcgttcc agcagtacag 12060
ccaacttg 12068
<210> 11
<211> 11605
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 碱基识别_比对_模板 90e5fe72_读取_ch2_文件11
<400> 11
cactttgata agaggttcca agtcattcct actgcctgtc gctctatctt cggcgtctgc 60
ttggtgttta acctattcgt cgttccgcga ctaacggtct gaccccgcgc ctccaggcgc 120
ggaacttcag aaaagtggcg aaactcatcg acgttaccac cagctctggc tgtaaacccc 180
tgtcaaaggt ggcgtgttca agtggaacga cattcgcgac cgtcgtaata acattggggt 240
gtacgacaac cccaatgatt taaacgcacc aaatcgtgga cggtaatgcg cgctatcggt 300
gaacagaacg acaaactgga atggctgatc caccaagacg gcagcgtact gttcgatcag 360
gctgcctggg cgtgccggcg gaagattacc agtgtcagta tggcctcgtc gacttcagtc 420
ccaggcggtg cggtggcatg cggattgtgc gacaccgcgg gctgtccgtt cgacattccg 480
cgcggaagac aaccgcgtct acaatgtgcc gctgtgcgtc gaccgcgtgg tggttgggcg 540
aaccagcctg cgtgaagacc accgcgcggg cgcgattcac ttcggtacga aagagtcggt 600
aaacgctggc gaaacgagcg ttgctgagct gaaaacccgc ggttacgaca atgcgggtct 660
gtacgatcca gcgagaacgt cggtggtacg tcatgtacac gtgccgcacc atgctgaaac 720
tgaccaaatc tgtatcgcga agcacaggag actaatcagc gaaaccgcaa ttctggaagg 780
cctcctggaa gccgctcgcg gctgttggct tgcggctacc cttgcggcca gtatgtcccg 840
ccgtggtgtc ggtcgaaccg tgcggatgag aagaaataat cctgcacgag aaagacgggc 900
gcaaatgaac gacgtgacct gtcgtgcgct actgcgcgcc ggaacgctat ccaaccactg 960
gatcaccgcc ttcttcatcc tggcggcggt gagcgggctg ggctttattt cccgtccctt 1020
caactggttg atccaccaaa tcgccattta ccgcaactgg cgttctgcac ccgtttgtca 1080
gcgtggttca tttgcctcgt tcatcatcat gttttccgtt gccagcatct ggcttcatca 1140
atcgggtaat cttttggcga agaatattcg tagatcgtcg tcaacgaagt aggtgacacc 1200
gggcgttata actggtcaga aatgcgtttt cctgggcggc gattattttt ctggttctgc 1260
tgctggtgag cggcgtgatt atcagcgatc atatttacgc ctgctttctc cgtagtgatc 1320
gattcgcgtt aatgctgcca ttcatttacc agtggcgaca gccagcggat tatcatggtg 1380
catatctgcg ccgccctttg ggtgaaaggc acgatgccgc catgaaggat gtaccagcgc 1440
ctgtgggcga aacatcgcac ccgcgctgat gccgtgaggt ccgcaagaca acggaaagcc 1500
aatcagtatt cgcataatcc tggaatgagc tgggtcaagc ggcttgtacg gcgagatatg 1560
attccgccgt tattgttcct cggctcaaaa ttaaccgcgc cgagctgtct gcgcgagctg 1620
gcgaaataat cctctggtga ttacctgcgc tttgctgcgc ttatcgcccg ccaggaaagt 1680
ggtgctgtac gaccatccac tggagatggt ctgtgcgcgc ataaaagcca gcgcacaagg 1740
caagcccgca gatattcacg ttctgccgcg tgataagcac tggcaaaaac tgctgatggc 1800
gctgattgct ggcgctgaaa ccacaaatag gcccggcgct ggcggtgaag tctggaggca 1860
tcgactcagg gctggaagac cagcccagcg cctcctgatt tctcgtccgc gtccgtgctg 1920
ataaaacgcc gttgtatcct gggctacgca atcgctctac tggctgaatg gccaatctca 1980
gtcaacaaag cccgcgctga gccacgacga gcaacgtcag ccttgcccgg tttgtggttt 2040
atgccggtgt ccaaagcata agttaccgtg gcaccactcg gaactacgac ctgcactgca 2100
actattgtga aactgaatag cacgtggtgc gcagccatca acaactgcga gcaggcgaca 2160
aacccattac cattcgctgg atgacgagca aacgcgataa agccaaagct gcgtgacgac 2220
gcctgaaaat tctctatcgg aaaagtccag aaatcaagcc gtaacgggtg gcctcctctg 2280
gtactggacg cacgaatggg caagaggcta tgcccgcagt tccatcaacc gttcacattt 2340
ccgggtgaag gtaatctccc gacgttttcg ggtaacaggt cgcccggatt acagcgctca 2400
tggggtgtga cgacaccatc tctcgtattc gttaccacct caaactgtgc cgccggtgcc 2460
agtttgactc aatttcacca catcaataag gaccttggcg gggcggtgac aagcgtagat 2520
tgcctgatgc gctgcgcgct tatcaggcct accaataaac tgcaatacga tgaatttata 2580
cgactttggc aaggccagat ggcgttcgcc gcatccggca tgaacaacgc gttgtcagca 2640
actcgattga catcatttta ttgttcctcg cgcaacccca cgctgatatt cgacaccatc 2700
ttcacaccgc ctgccagatg ccgggaacac tggttcttgc tgttgcgttg atttatggtg 2760
atgatccagc ttccttaatg ggtccgcacg gaagattaca tttgcctcga gatcatcgtt 2820
gtaatcacct cattcgtcgt caggtaaaat tctgcccata tctatacctg ccaaagttca 2880
ccaatatcac ttcaatatca ttttgaatct ggaaaagtac cctgccgaat ctttaaaaac 2940
tcatttacac ttatcatttt attttgcgag gcgttttcag atatggtcat ggcagttgcg 3000
gattatattc aagtaaaaca accttgtatt aataccagaa taagggcatc tgtactatgt 3060
tcaaaattga aaattttgtc attccttatg ctcttacact gatcatcata ccaactacct 3120
ccccctttgc ctgctcctta cagcactggg aattactgac gttcagatat tgctctacca 3180
cttcgcatcc attgcagatc gtgaaaccca cgccagcaag cgttccgtgt tagtgcctct 3240
tctgataaat gaaaaaccgt atacatcgcc attaatacct tgtattactc agccaatgct 3300
gtgcgaaagt atggaaaatc atttaagcgg attttgcatg ttgttttgtt gcgaggcgtt 3360
ttccatatgc tctatagagt gttgcgttgt ttcatttatt actcaaatgg ttagttatcg 3420
ctaaagtgga ttatgattac tatgaatata ttatattcag aatgttgttg cataatctga 3480
atggtgtaac tgaaaaacat atcagcatcc taacagcacc ggattttaaa ttgaaattaa 3540
tctttcatct gaaccggtca gtactgccgc aaccggtctt tacttgctct atattctata 3600
tttgtggtaa tgattcagga aggattgttt tctaaattaa caacagaata actttcttgt 3660
ttaatgagat taatgtggca accattataa taccacggtt tagagcaaac atacaacata 3720
ttattcataa catacagagc agtaaatgaa tgatactgac aatgatgcta gtccattatc 3780
tttgcataat atttattgct ataatttaaa aatatttatg agaaagtatg gcacagagaa 3840
cgactatcaa gtatcagcaa actaagatga tggtggcatt atctgcgata aaattgccac 3900
tcagttctac aaataatatt taaatatact gaacagtgat ctgtgaacgt aagacccaac 3960
gagaataaaa atttatagag ttcttgtttt aatataacga aaccgggacc agcaaatgac 4020
ctgaataggg agaacgcttg cctatgccat cagctactct tcttcgttcc tcctcttctt 4080
accgggccaa atagctgcgc caggcgaacg atacgtgcct tcaaacagct gcacccggcg 4140
tctcgtcgcc aagattttcg acctgcatct gccgcggcca gcgccgtcgc gcgccagacg 4200
gtcgagatcg ccgctgagca ccgaggtcgg ctaccgcatg agtttaccgc tatcggcctt 4260
ggcggcggtt aagccgcatc ccatcacgaa caagtcgttc cgccggggtg tggcgaccgc 4320
agcatcggcc agataagtcc caaccagtga gcgtcgccag tgaataaact cccgaaacaa 4380
aaccgccgta tggtgcagtt cctgattacg gttctggcat ggtagtgacc gtacgcccgg 4440
tatgataccc gaatgcgcgc cattttcact ggcgcggaat atgttcgtac cacgcctgtt 4500
cgctgcgcgc accgccgcga tgcggtcatc cagagtggcg acgggcttaa tcgtggcgaa 4560
tcggcgtgtg gttggcgtgg tgatttctcc cagtgctcgg caacggcgaa aaactcaccg 4620
cgtcttcacg ggcgctacag gtcgcacgcc aacgccttcc tgacgccacc gactccaggt 4680
catcgccatc ggcgcccttc atcctgcgtc gggatgaacg gccataaagc aaatggacgc 4740
ttcattgtcg gcattaatat acggcctacc gccagattac cctgctcgtc gacgaccatc 4800
tggtgatgcg ccatcgcatc ccacgcgtcg cgttcagaac cttttggttg atgcgagcac 4860
gacatttcag cagagcctga taacgcatac aattcttcct gcacggaacc gaaggtgata 4920
cgccagcgta ctctcttgtt tcccgtgacg accctgaaac ttcagctcgc ctgcaaccaa 4980
gaatgtcacg ggccatcgtg ataacgatac cctgcatatc ccgcagcgaa gcgtgtttgc 5040
gtgttcatct cttgcggcgg cggcgttcga cgaaatagtc atataacgcc tctgcgcgat 5100
ccggtgatgc acctttgggc ttgggctttg tcccgttcag tatcacggcg agggctgaaa 5160
ccaccagcct gccgccgcct gttgcgttga gattcatctt gcttcggcat cgctaaaatc 5220
cggtatgagc acgtgattct cctttcgaca cccaataaca aggtccgcgc caatttcgcc 5280
cgtcgcctat cctctacatt tgggattacg cgttaattaa tgcaatcatg gttcgtcatt 5340
cttgttcttc ttgctggttg cggtattccc cgagtggtaa tttcgcgcca agcagacgta 5400
caccatgtca gtgttaaaac gaagagaatg gtcaccgcca gcacaccata aatgagctga 5460
tatgagcgga acatgatgta aacgcaaacc tttcttctgc tgattaagtg cggcgacaaa 5520
acgcgccaaa cgataaatcg cggttaggta cgccggatgg taggaacaat gctgtacagc 5580
aaccagagat ccccgacaac aggaaaaata cgcagcacgt tgtcggtaac cgtattgatc 5640
ctcgccagcg caggagaagg caatgaactg atcgccagac tggcccctac cagcattcgc 5700
aaatcatcca gtacgaacag taaattttgg tcgcgctcgt ttaccacgcc agatggtatt 5760
caacgctatc gatggagtac atcagcaaca ataacgcggt gacgtcagcc cgcacgcccc 5820
aacggcggtc atcttgttgg gtggcaacaa attgttcgat ataccgctgg taacatcgcc 5880
agtagcaggc gaagttgctt aaagtagtga cggcttgaat gctgacgtcg aaaacatggg 5940
aaaagcgacg ctttaaaagc aacggcagcc agcggcacta atgaacaacg acccttgagc 6000
aaggttacct gccagggttg tcatgttgtc ctctcaatgc gttggtagtt tgctttcgac 6060
cttggacggg tacggtgcct ggctttgtct gaatggtttt aacaacacta gcgaaacggt 6120
ccgggatcag gtggttgctc gctactgatg ctggtaatgc ccggctgatt ggctccttct 6180
atgacccatc ggcgttatcg tcgaaagacc ttatcgctgg gtgaaaacgc cttccgccgc 6240
aaacatgctg ttaattcagc cttttcggga cgcatctaga tcttgcgaca gatagatatg 6300
attgtagcat cacaattttc cgggtattct ccggcagaag gtggtatgcg agcggttgtg 6360
gaagcaccac ccgcatgata cctgctcacg cagtttgtgc atgatgtggc gatcacttcc 6420
gggcgcagcg cagcaaacac cgcctgccag ccgtgagaga gcgctgctcg tagcttggct 6480
gagccaaatc tcgtgacaca gcgcctctgc gaacgcttcg tcataatttc cccacactca 6540
tgctgatgaa acactaccat atgaaaactc ttcttaagcg atgccagcgg aatacgcgtt 6600
gaaatcatca ggctcccaac acggttaaag tcgatatcga caacattacc taaatcaaga 6660
tatagagcat tttgcctcct ttcgcgccac aagaagtgct gtagcaggaa aggatgggtt 6720
taactatgtg gcaagtgtaa accaggttaa cggtcacaaa atctacgcgt cttcctggga 6780
aaatgatttc gttggctgtc tcccaaccga aaacgtgatt ttagcgccgg cgagcggcaa 6840
ctcgtcggta agagcgcagt gacctgtgcc acgttaccaa tttcaaaaac ctgccaagca 6900
tgatgccgtg tgatcggcga ccagcagggc tgttctcgcg tacgccatca tgtttcacca 6960
cgccgctgaa cacatcaact ccatcaacgc gccatcctct aatcgctaac cttacattca 7020
ataatcggca caatcataca aaggacgcgc tgaagggcca atgatcccgc tattatgagg 7080
cgcaacgtac cgccgggcac tatcgcttta aacgcgggtt gcggtaaaga ataatgcgcc 7140
gcgcaggctt cggtggtgat tatccgcaaa tgattatggt tcgtaacgcg gcaacatgct 7200
caacagtcat gccaatggtg ataatattgt tgaatataag acctggtctg ccaccgctgc 7260
ggcaatcgct ttttctcggt ttgcgaaacc tcgctgctcg aacgccgtat taacgacact 7320
ggaagcccga cccgcgccac cgtgatggcg cgtaatcaga actgccgctc atggtgaata 7380
tccccagcgg accgtctgcg tggagacatc caacgaattt gccaacaata tcataaatat 7440
accacgctcg acacgatcaa cactggtggt cgtgccgcgg gttaccggtt agttcagtgg 7500
gctgtgaaaa attcctcgaa aaccatcacc ctggcatttt gccaaaaggt gacaaaaaga 7560
tcgggtttga tcggtcactg gggatcccga agcaaatccc gccggatgat gtcgtttgcc 7620
cgctacccgc tggcgaagcg gactgactcg cttcatccca ccacttgttg ccagcgccac 7680
cgccaaagcg ccgtgaaaac atcacccgca gtacatcacc accacctcgg ctttgaaggc 7740
cggttgatgc tggcgcccac caccagtagt cgcagcctgc acgctaccct gggtcatagg 7800
catgacaaat ttagccaggc gacatgcctg ttttggcgct gtctcttttc gcccgttaag 7860
cgcgccagac ccggttctga ggccgcgcga tcgcgaaacg ctactacccg gctcacgccg 7920
atatcctgcg gcgtaatccc cgtccagaac aggtcatcac cgcctgacgg tggggtgaag 7980
aacttttagc gccgtctgcc agcgtacgtc tgccagcaca acatcccgca ggaaatcgat 8040
ttcctcaacc gcctctgcat catcaggcag cagatccggg ctggggtggt taatgattat 8100
ccgctctgct agcggtatcc gcatgatggc cggattgccg aagatttcgc ctggttatac 8160
cgtttggtgt aacgggtgtt aacgcccagg attccagctc tgccagcagg ctgttgccga 8220
tgtctgtcat cacctacgta tgcatcgacc ctgcgcccac aacctttacc cgccaccaac 8280
cgctaccgtc gccgctggcc tgcaacttcc gtataatttc tcgccacatg cccaccgctc 8340
tcgtcgtgtc cttccacgta atagatgcga tcctgtccac gtttatacct acacaagcag 8400
atcatggtca ttctccttaa acattgcggg atgcgttcat tttaattcac cagtgtttaa 8460
aagtgacggc tacatcaatt ttgactgtcg atacaaacca gtcaaacgaa caaagcaaag 8520
catgtcataa atgacaaaaa gtgacataac tgtattcagg agggttatgg caacaatcac 8580
cgacctcgtt ttacggacaa tgggtccgcc aatggcgagc aatttattgc agcaagaacg 8640
taacttcgcg tcttgatgtg aacgccgaag ctgccgacat ctggtgaaca aggtgcgact 8700
cgccgccaac cggcgcagac cactaaagat gaatttatca ttactgttct tgaatggcga 8760
tctggtgcgc aacattgttc tgattgaaaa cggcgtttgc gaaggcttat ctgttcgctg 8820
gtcgttgata tgtccacctc atgcaaaccg ataaattcgt tgccgatata cagcaaaggc 8880
ttcagcatga tggatgttcc gacgaaaccg tacttctgca aatgccatta ccggtactct 8940
gttactgctg atggcggcct gaaccgttga acgatgccac gcccgtccca ccggtggcga 9000
tgggcggtgg ttgatcgcgc tggcggtccg ggcatgggga tcacgttagc tcatcagcaa 9060
ctacatgagc atcgcgctca atgcgctctg acggaaaccg ccgtttgtga ccctggatct 9120
tccgtgttgc cgcaaagtga tgagcgcacg ccgcaagtaa gatcttcacc gcttcggcaa 9180
acaagtctcg gcggagatct tctccgccat actcatgatc gatcttgccg cagaatccca 9240
gcatcgcctt gatgtcgcca accaaagctg catcgtgcca atgctggggc cgcctcgcag 9300
ggttgtaact gagcgcgcgg cgatcgcgat cgccaggacg gtccgccatt ctggagcagg 9360
tcgtgtcagg tctgggatgg ctaccagtaa aaatgtaacg actggataaa ggaataaatg 9420
aatgaataag tacaccatca acgacattac gcgcgcatcg ggcggttttg ccatactggc 9480
ggtcgatcag caagccatgc cgcaatgttt accacagctg gggcacctgc tcggtagccg 9540
tgggcacaac gatttcaaag ttaacactgc aaggccctct cgccttgtcc tcggcgattc 9600
tggtagatca gcaattcgcc accgccaggt ggttgagcaa acgcgttata gcaggacgcg 9660
ccatgattgt catgccgatg agttcattct acagcagcag tattccggtc gatagcatgg 9720
tgtcgaccgc aaaatcaatc gctacagatc aaacaagacg gtggcaagcg gcaaaactgc 9780
tggtgctgtg gcgtgatgaa gatgcgcagc agcgtctgga tatggtgaag agttcggcga 9840
actgtgccac tcacacggtg ttgagccagt cattccgccc accgcgtcgt ggcgataaat 9900
tcgatcgcga acaagcgatc atcgataccg ccagagctgg gcgacggtgg cgctgacctc 9960
tcaaagttga aatgccgctt tatggcaggt ccgcaacaga acttctgctt cacaacgtca 10020
gaccgagcca catcaatatg ccatgcgggt gatctctctt ccggtgtctc gtaagctgtt 10080
cccgcattgc cgtacgcgtg gcaatgacgg cgggcgcatc ggacctggca ggccgtgcgg 10140
tctgggcatc ggtcgtcggt ttaccagaca acgagctgat gctgcgtgac gtttgcaccg 10200
aagtacaaca acagcgacat cgtcagcaaa ccaatggcta aacgccgcta tgaatgagga 10260
taaagatggt ttaacaccta agcttacaac cgttggctgg aacggaaacc gaccatcttt 10320
gattttggta aaaattccgt agtgccgact ggtttggctg gttaggcaat aaaggcaaat 10380
caaagagatg ggcaccatct gtggatcacc gctcattgcg tttattccgt tgccacggcg 10440
tgggtcgacc tggcgcttac tcgttggttg atcacggtat caagccatga gcgtgataaa 10500
tatggcggct gagccatgcc tgcacgtgaa tgacggggcg tggtggatgc ctcgagcaga 10560
tatcaacgct aacagcatgc cagcacgggt gcctgccagc gccgtcacgg cagattttcc 10620
aggcgctcgt tacaccattg aaattatcca gaaatatttc tgaacgaaga aggcagatgt 10680
gctggagtcc tgggacgaag ccttccagca aaaccgaaga gtaccgcgac ggcaatgcca 10740
tatgcacgcg gtggaaactt ctcttgattg tttatgacgt cactcacgac aaaaatgagc 10800
tggatcgcgc gattcgcgtg gcttccccgc gatatccggc aatcgccgaa ataatcatta 10860
tcgcgttaac gaacacttcg atacccgtgg aatccgctgc cggattacaa caaagatcgc 10920
cggcacgtcg cttccgcgcg ttcgggtaca ccaggccact ggatcgaatg gggccgttta 10980
atgctgcaca tcatgctgcc tggaagcccg ttgcgaacaa ccagcgtaac gctgaagata 11040
ccaaggtctg tttaacgcca ccgtgcgcga tgcctgggca cccgatggcg cggacgggat 11100
tgcgacatac cgttgactgg gaaggaaaac cggtggtccg cgaacgatgt acgttggcct 11160
atcaagcaat ggtacggcct acgcgctcca ccgtcactgg cgatcgccag tatgaaaccc 11220
tggtatcaaa catggtgggg tgcattgagt acctgatgga ctatgaaaat ggttcctggt 11280
ggcggagctg gatgcagtta atggtcctac aaagtctggg acgctaaaca ggatattatc 11340
acctgctgca ttgcctgagt gatcccacgt atcccgttag cgcccggcat gctccagcag 11400
tttgcagcag attactggat aataatgcga aataactgat ctgaaaatag gtgcgggttt 11460
cctctaaccc tctccccaaa ggggcgaggg gacgtccggt gcagaaggtt aaacgcaagc 11520
agacgccgaa gatagagcat tgagcaagga acactctcgt ttggaacatc tggcaaacgc 11580
gaagcagacg ccagcaatac gtaac 11605
<210> 12
<211> 61
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 衔接子设计A/B(上链)
<220>
<221> misc_binding
<222> (1)..(1)
<223> 核苷酸之前带有1 × 5SpC3和29 × iSpC3
<220>
<221> misc_binding
<222> (33)..(34)
<223> 核苷酸被1 × iSp18分离
<400> 12
ggcgtctgct tgggtgttta accttttttt tttaatgtac ttcgttcagt tacgtattgc 60
t 61
<210> 13
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 衔接子设计A(阻断链)
<220>
<221> modified_base
<222> (1)..(1)
<223> 5BNA-G
<220>
<221> modified_base
<222> (2)..(2)
<223> iBNA-G
<220>
<221> modified_base
<222> (3)..(3)
<223> iBNA-T
<220>
<221> modified_base
<222> (4)..(4)
<223> iBNA-T
<220>
<221> modified_base
<222> (5)..(5)
<223> iBNA-A
<400> 13
ggttaaacac ccaagcagac gcctaagtca gagaggttcc 40
<210> 14
<211> 45
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 衔接子设计A(下链)
<220>
<221> modified_base
<222> (23)..(23)
<223> iBNA-A
<220>
<221> modified_base
<222> (24)..(24)
<223> iBNA-meC
<220>
<221> modified_base
<222> (25)..(25)
<223> iBNA-A
<220>
<221> modified_base
<222> (26)..(26)
<223> iBNA-T
<220>
<221> modified_base
<222> (27)..(27)
<223> iBNA-T
<400> 14
gcaatacgta actgaacgaa gtacattttt gaggcgagcg gtcaa 45
<210> 15
<211> 41
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 衔接子设计B(阻断链)
<400> 15
ggttaaacac ccaagcagac gcctttgagg cgagcggtca a 41
<210> 16
<211> 47
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 衔接子设计B(下链)
<220>
<221> modified_base
<222> (23)..(23)
<223> iBNA-A
<220>
<221> modified_base
<222> (24)..(24)
<223> iBNA-meC
<220>
<221> modified_base
<222> (25)..(25)
<223> iBNA-A
<220>
<221> modified_base
<222> (26)..(26)
<223> iBNA-T
<220>
<221> modified_base
<222> (27)..(27)
<223> iBNA-T
<400> 16
gcaatacgta actgaacgaa gtacattttt taagtcagag aggttcc 47
<210> 17
<211> 80
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 跟随衔接子上链
<220>
<221> misc_binding
<222> (44)..(45)
<223> 核苷酸被4 × iSp18分离
<400> 17
taacgaggtt gtttctatct cggcgtctgc ttgggtgttt aacctttttg tcagagaggt 60
tccaagtcag agaggttcct 80
<210> 18
<211> 123
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 跟随衔接子下链
<220>
<221> misc_binding
<222> (35)..(36)
<223> 核苷酸被4 × iSp18分离
<220>
<221> misc_binding
<222> (98)..(99)
<223> 核苷酸被4 × iSp18分离
<400> 18
ggaacctctc tgacttggaa cctctctgac aaaaaggtta aacacccaag cagacgccga 60
gatagaaaca acccatcaga ttgtgtttgt tagtcgctag cgactaacaa acacaatctg 120
atg 123
<210> 19
<211> 70
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 串联测序衔接子上链
<220>
<221> misc_binding
<222> (15)..(16)
<223> 核苷酸被15 × iSpC3分离
<220>
<221> misc_binding
<222> (48)..(49)
<223> 核苷酸被1 × iSp18分离
<400> 19
ggttgtttct atctcggcgt ctgcttgggt gtttaacctt ttttttttaa tgtacttcgt 60
tcagttacgt 70
<210> 20
<211> 41
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 串联测序衔接子阻断链
<400> 20
ggttaaacac ccaagcagac gcctttgagg cgagcggtca a 41
<210> 21
<211> 28
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 串联测序衔接子下链
<220>
<221> modified_base
<222> (24)..(24)
<223> iBNA-A
<220>
<221> modified_base
<222> (25)..(25)
<223> iBNA-meC
<220>
<221> modified_base
<222> (26)..(26)
<223> iBNA-A
<220>
<221> modified_base
<222> (27)..(27)
<223> iBNA-T
<220>
<221> modified_base
<222> (28)..(28)
<223> iBNA-T
<400> 21
tcgttaacgt aactgaacga agtacatt 28
<210> 22
<211> 71
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 捕捞衔接子上链
<220>
<221> misc_binding
<222> (1)..(1)
<223> 核苷酸被1 × 5SpC3和14 × iSpC3分离
<220>
<221> misc_binding
<222> (16)..(17)
<223> 核苷酸被15 × iSpC3分离
<220>
<221> misc_binding
<222> (49)..(50)
<223> 核苷酸被1 × iSp18分离
<400> 22
ttgtcagaga ggttccggcg tctgcttggg tgtttaacct ttttttttta atgtacttcg 60
ttcagttacg t 71
<210> 23
<211> 22
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 捕捞衔接子下链
<220>
<221> modified_base
<222> (18)..(18)
<223> iBNA-A
<220>
<221> modified_base
<222> (19)..(19)
<223> iBNA-meC
<220>
<221> modified_base
<222> (20)..(20)
<223> iBNA-A
<220>
<221> modified_base
<222> (21)..(21)
<223> iBNA-T
<220>
<221> modified_base
<222> (22)..(22)
<223> iBNA-T
<400> 23
acgtaactga acgaagtaca tt 22
<210> 24
<211> 100
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 捕捞系链上链
<400> 24
ttgtcagaga ggttcctttt tttttttttt tttttttttt tttttttttt ttttggttgt 60
ttctgttggt gctgatattg cttttttgac cgctcgcctc 100
<210> 25
<211> 31
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 捕捞系链下链
<220>
<221> misc_binding
<222> (29)..(30)
<223> 核苷酸被6 × iSp18分离
<400> 25
gcaatatcag caccaacaga aacaaccttt t 31
<210> 26
<211> 3587
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 3.6kb dA尾DNA
<400> 26
gccatcagat tgtgtttgtt agtcgctgcc atcagattgt gtttgttagt cgcttttttt 60
ttttggaatt ttttttttgg aatttttttt ttgcgctaac aacctcctgc cgttttgccc 120
gtgcatatcg gtcacgaaca aatctgatta ctaaacacag tagcctggat ttgttctatc 180
agtaatcgac cttattccta attaaataga gcaaatcccc ttattggggg taagacatga 240
agatgccaga aaaacatgac ctgttggccg ccattctcgc ggcaaaggaa caaggcatcg 300
gggcaatcct tgcgtttgca atggcgtacc ttcgcggcag atataatggc ggtgcgttta 360
caaaaacagt aatcgacgca acgatgtgcg ccattatcgc ctagttcatt cgtgaccttc 420
tcgacttcgc cggactaagt agcaatctcg cttatataac gagcgtgttt atcggctaca 480
tcggtactga ctcgattggt tcgcttatca aacgcttcgc tgctaaaaaa gccggagtag 540
aagatggtag aaatcaataa tcaacgtaag gcgttcctcg atatgctggc gtggtcggag 600
ggaactgata acggacgtca gaaaaccaga aatcatggtt atgacgtcat tgtaggcgga 660
gagctattta ctgattactc cgatcaccct cgcaaacttg tcacgctaaa cccaaaactc 720
aaatcaacag gcgccggacg ctaccagctt ctttcccgtt ggtgggatgc ctaccgcaag 780
cagcttggcc tgaaagactt ctctccgaaa agtcaggacg ctgtggcatt gcagcagatt 840
aaggagcgtg gcgctttacc tatgattgat cgtggtgata tccgtcaggc aatcgaccgt 900
tgcagcaata tctgggcttc actgccgggc gctggttatg gtcagttcga gcataaggct 960
gacagcctga ttgcaaaatt caaagaagcg ggcggaacgg tcagagagat tgatgtatga 1020
gcagagtcac cgcgattatc tccgctctgg ttatctgcat catcgtctgc ctgtcatggg 1080
ctgttaatca ttaccgtgat aacgccatta cctacaaagc ccagcgcgac aaaaatgcca 1140
gagaactgaa gctggcgaac gcggcaatta ctgacatgca gatgcgtcag cgtgatgttg 1200
ctgcgctcga tgcaaaatac acgaaggagt tagctgatgc taaagctgaa aatgatgctc 1260
tgcgtgatga tgttgccgct ggtcgtcgtc ggttgcacat caaagcagtc tgtcagtcag 1320
tgcgtgaagc caccaccgcc tccggcgtgg ataatgcagc ctccccccga ctggcagaca 1380
ccgctgaacg ggattatttc accctcagag agaggctgat cactatgcaa aaacaactgg 1440
aaggaaccca gaagtatatt aatgagcagt gcagatagag ttgcccatat cgatgggcaa 1500
ctcatgcaat tattgtgagc aatacacacg cgcttccagc ggagtataaa tgcctaaagt 1560
aataaaaccg agcaatccat ttacgaatgt ttgctgggtt tctgttttaa caacattttc 1620
tgcgccgcca caaattttgg ctgcatcgac agttttcttc tgcccaattc cagaaacgaa 1680
gaaatgatgg gtgatggttt cctttggtgc tactgctgcc ggtttgtttt gaacagtaaa 1740
cgtctgttga gcacatcctg taataagcag ggccagcgca gtagcgagta gcattttttt 1800
catggtgtta ttcccgatgc tttttgaagt tcgcagaatc gtatgtgtag aaaattaaac 1860
aaaccctaaa caatgagttg aaatttcata ttgttaatat ttattaatgt atgtcaggtg 1920
cgatgaatcg tcattgtatt cccggattaa ctatgtccac agccctgacg gggaacttct 1980
ctgcgggagt gtccgggaat aattaaaacg atgcacacag ggtttagcgc gtacacgtat 2040
tgcattatgc caacgccccg gtgctgacac ggaagaaacc ggacgttatg atttagcgtg 2100
gaaagatttg tgtagtgttc tgaatgctct cagtaaatag taatgaatta tcaaaggtat 2160
agtaatatct tttatgttca tggatatttg taacccatcg gaaaactcct gctttagcaa 2220
gattttccct gtattgctga aatgtgattt ctcttgattt caacctatca taggacgttt 2280
ctataagatg cgtgtttctt gagaatttaa catttacaac ctttttaagt ccttttatta 2340
acacggtgtt atcgttttct aacacgatgt gaatattatc tgtggctaga tagtaaatat 2400
aatgtgagac gttgtgacgt tttagttcag aataaaacaa ttcacagtct aaatcttttc 2460
gcacttgatc gaatatttct ttaaaaatgg caacctgagc cattggtaaa accttccatg 2520
tgatacgagg gcgcgtagtt tgcattatcg tttttatcgt ttcaatctgg tctgacctcc 2580
ttgtgttttg ttgatgattt atgtcaaata ttaggaatgt tttcacttaa tagtattggt 2640
tgcgtaacaa agtgcggtcc tgctggcatt ctggagggaa atacaaccga cagatgtatg 2700
taaggccaac gtgctcaaat cttcatacag aaagatttga agtaatattt taaccgctag 2760
atgaagagca agcgcatgga gcgacaaaat gaataaagaa caatctgctg atgatccctc 2820
cgtggatctg attcgtgtaa aaaatatgct taatagcacc atttctatga gttaccctga 2880
tgttgtaatt gcatgtatag aacataaggt gtctctggaa gcattcagag caattgaggc 2940
agcgttggtg aagcacgata ataatatgaa ggattattcc ctggtggttg actgatcacc 3000
ataactgcta atcattcaaa ctatttagtc tgtgacagag ccaacacgca gtctgtcact 3060
gtcaggaaag tggtaaaact gcaactcaat tactgcaatg ccctcgtaat taagtgaatt 3120
tacaatatcg tcctgttcgg agggaagaac gcgggatgtt cattcttcat cacttttaat 3180
tgatgtatat gctctctttt ctgacgttag tctccgacgg caggcttcaa tgacccaggc 3240
tgagaaattc ccggaccctt tttgctcaag agcgatgtta atttgttcaa tcatttggtt 3300
aggaaagcgg atgttgcggg ttgttgttct gcgggttctg ttcttcgttg acatgaggtt 3360
gccccgtatt cagtgtcgct gatttgtatt gtctgaagtt gtttttacgt taagttgatg 3420
cagatcaatt aatacgatac ctgcgtcata attgattatt tgacgtggtt tgatggcctc 3480
cacgcacgtt gtgatatgta gatgataatc attatcactt tacgggtcct ttccggtgaa 3540
aaaaaaggta ccaaaaaaaa catcgtcgtg agtagtgaac cgtaagc 3587
<210> 27
<211> 41
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 跟随衔接子下链
<400> 27
acgtaactga acgaagtaca tttttgaggc cgagcggtca a 41
<210> 28
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 跟随衔接子阻断链
<400> 28
ggttaaacac ccaagcagac gcctaagtca gagaggttcc 40
<210> 29
<211> 38
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> FO016_FOSEN_尾连接_TOP_A
<400> 29
ggtgctgaag aaagttgtcg gtgtctttgt gttaacct 38
<210> 30
<211> 60
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> FO017_FOSEN_尾连接_BOT_A
<400> 30
ggttaacaca aagacaccga caactttctt cagcaccagc aattaagtca gagaggttcc 60
<210> 31
<211> 77
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> FO018_FOSEN_尾连接_TOP_B
<220>
<221> misc_binding
<222> (23)..(24)
<223> 核苷酸被4 × iSp18分离
<400> 31
ccaatttgtg ggttcgtctg cggtttttgg cgtctgcttg ggtgtttaac caaggcgtct 60
gcttgggtgt ttaacct 77
<210> 32
<211> 99
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> FO019_FOSEN_尾连接_BOT_B
<220>
<221> misc_binding
<222> (53)..(54)
<223> 核苷酸被iSp18分离
<400> 32
ggttaaacac ccaagcagac gccttggtta aacacccaag cagacgccaa aaaccgcaga 60
cgaacccaca aattggagca attaagtcag agaggttcc 99
<210> 33
<211> 82
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> FO020_FOSEN_尾连接_BOT_B_非侧边
<220>
<221> misc_binding
<222> (53)..(54)
<223> 核苷酸被4 × iSp18分离
<400> 33
ggttaaacac ccaagcagac gccttggtta aacacccaag cagacgccaa aaaccgcaga 60
cgaacccaca aattggagca at 82
<210> 34
<211> 66
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> FO021_FOSEN_尾连接_TOP_C
<220>
<221> misc_binding
<222> (23)..(24)
<223> 核苷酸被4 × iSp18分离
<400> 34
ggcgtctgct tgggtgttta acctttttgg tgctgaagaa agttgtcggt gtctttgtgt 60
taacct 66
<210> 35
<211> 88
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> FO022_FOSEN_尾连接_BOT_C
<220>
<221> misc_binding
<222> (42)..(43)
<223> 核苷酸被4 × iSp18分离
<400> 35
ggttaacaca aagacaccga caactttctt cagcaccaaa aaggttaaac acccaagcag 60
acgccagcaa ttaagtcaga gaggttcc 88
<210> 36
<211> 44
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> FO023_FOSEN_夹板再次_TOP_A
<220>
<221> misc_binding
<222> (15)..(16)
<223> 核苷酸被4 × iSp18分离
<400> 36
ccaatttgtg gttccttttt ggcgtctgct tgggtgttta acct 44
<210> 37
<211> 49
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> FO024_FOSEN_夹板再次_BOT_A
<220>
<221> misc_binding
<222> (28)..(29)
<223> 核苷酸被4 × iSp18分离
<400> 37
ggttaaacac ccaagcagac gccaaaaagg aaccacaaat tggagcaat 49
<210> 38
<211> 69
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> FO025_FOSEN_夹板再次_TOP_B
<220>
<221> misc_binding
<222> (15)..(16)
<223> 核苷酸被4 × iSp18分离
<400> 38
ccaatttgtg gttccttttt ggcgtctgct tgggtgttta accaaggcgt ctgcttgggt 60
gtttaacct 69
<210> 39
<211> 74
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> FO026_FOSEN_夹板再次_BOT_B
<220>
<221> misc_binding
<222> (53)..(54)
<223> 核苷酸被4 ×iSp18分离
<400> 39
ggttaaacac ccaagcagac gccttggtta aacacccaag cagacgccaa aaaggaacca 60
caaattggag caat 74
<210> 40
<211> 61
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> FO027_TOP_1sp18
<220>
<221> misc_binding
<222> (23)..(24)
<223> 核苷酸被1 × iSp18分离
<400> 40
ggcgtctgct tgggtgttta acctttttgt cagagaggtt ccaagtcaga gaggttccaa 60
t 61
<210> 41
<211> 66
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> FO028_BOT_1sp18
<220>
<221> misc_binding
<222> (37)..(38)
<223> 核苷酸被1 × iSp18分离
<400> 41
ttggaacctc tctgacttgg aacctctctg acaaaaaggt taaacaccca agcagacgcc 60
agcaat 66
<210> 42
<211> 45
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> FO029_TOP_1杂交位点
<220>
<221> misc_binding
<222> (23)..(24)
<223> 核苷酸被4 × iSp18分离
<400> 42
ggcgtctgct tgggtgttta acctttttgt cagagaggtt ccaat 45
<210> 43
<211> 50
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> FO030_BOT_1杂交位点
<220>
<221> misc_binding
<222> (21)..(22)
<223> 核苷酸被4 × iSp18分离
<400> 43
ttggaacctc tctgacaaaa aggttaaaca cccaagcaga cgccagcaat 50
<210> 44
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 阻断链ID2(TT008)
<400> 44
ggttaaacac ccaagcagac gcctaagtca gagaggttcc 40
<210> 45
<211> 14
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 孔-系链
<400> 45
ggaacctctc tgac 14
<210> 46
<211> 47
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 下链ID5(TT186)
<400> 46
gcaatacgta actgaacgaa gtacattttt taagtcagag aggttcc 47
<210> 47
<211> 59
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> FO003双链体茎示例顶部
<400> 47
ggcgtctgct tgggtgttta acctttttgt cagagaggtt ccaagtcaga gaggttcct 59
<210> 48
<211> 64
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> FO004_双链体茎示例底部
<400> 48
ggaacctctc tgacttggaa cctctctgac aaaaaggtta aacacccaag cagacgccag 60
caat 64
<210> 49
<211> 44
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 条形码1nt突出端
<400> 49
gcaatacgta acgtaacgaa gtacatttaa gtcagagagg ttcc 44
<210> 50
<211> 39
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 条形码6nt突出端
<400> 50
acgtaacgta acgaagtaca tttaagtcag agaggttcc 39
<210> 51
<211> 439
<212> PRT
<213> 肠杆菌噬菌体T4(enterobacteria phage T4)
<400> 51
Met Thr Phe Asp Asp Leu Thr Glu Gly Gln Lys Asn Ala Phe Asn Ile
1 5 10 15
Val Met Lys Ala Ile Lys Glu Lys Lys His His Val Thr Ile Asn Gly
20 25 30
Pro Ala Gly Thr Gly Lys Thr Thr Leu Thr Lys Phe Ile Ile Glu Ala
35 40 45
Leu Ile Ser Thr Gly Glu Thr Gly Ile Ile Leu Ala Ala Pro Thr His
50 55 60
Ala Ala Lys Lys Ile Leu Ser Lys Leu Ser Gly Lys Glu Ala Ser Thr
65 70 75 80
Ile His Ser Ile Leu Lys Ile Asn Pro Val Thr Tyr Glu Glu Asn Val
85 90 95
Leu Phe Glu Gln Lys Glu Val Pro Asp Leu Ala Lys Cys Arg Val Leu
100 105 110
Ile Cys Asp Glu Val Ser Met Tyr Asp Arg Lys Leu Phe Lys Ile Leu
115 120 125
Leu Ser Thr Ile Pro Pro Trp Cys Thr Ile Ile Gly Ile Gly Asp Asn
130 135 140
Lys Gln Ile Arg Pro Val Asp Pro Gly Glu Asn Thr Ala Tyr Ile Ser
145 150 155 160
Pro Phe Phe Thr His Lys Asp Phe Tyr Gln Cys Glu Leu Thr Glu Val
165 170 175
Lys Arg Ser Asn Ala Pro Ile Ile Asp Val Ala Thr Asp Val Arg Asn
180 185 190
Gly Lys Trp Ile Tyr Asp Lys Val Val Asp Gly His Gly Val Arg Gly
195 200 205
Phe Thr Gly Asp Thr Ala Leu Arg Asp Phe Met Val Asn Tyr Phe Ser
210 215 220
Ile Val Lys Ser Leu Asp Asp Leu Phe Glu Asn Arg Val Met Ala Phe
225 230 235 240
Thr Asn Lys Ser Val Asp Lys Leu Asn Ser Ile Ile Arg Lys Lys Ile
245 250 255
Phe Glu Thr Asp Lys Asp Phe Ile Val Gly Glu Ile Ile Val Met Gln
260 265 270
Glu Pro Leu Phe Lys Thr Tyr Lys Ile Asp Gly Lys Pro Val Ser Glu
275 280 285
Ile Ile Phe Asn Asn Gly Gln Leu Val Arg Ile Ile Glu Ala Glu Tyr
290 295 300
Thr Ser Thr Phe Val Lys Ala Arg Gly Val Pro Gly Glu Tyr Leu Ile
305 310 315 320
Arg His Trp Asp Leu Thr Val Glu Thr Tyr Gly Asp Asp Glu Tyr Tyr
325 330 335
Arg Glu Lys Ile Lys Ile Ile Ser Ser Asp Glu Glu Leu Tyr Lys Phe
340 345 350
Asn Leu Phe Leu Gly Lys Thr Ala Glu Thr Tyr Lys Asn Trp Asn Lys
355 360 365
Gly Gly Lys Ala Pro Trp Ser Asp Phe Trp Asp Ala Lys Ser Gln Phe
370 375 380
Ser Lys Val Lys Ala Leu Pro Ala Ser Thr Phe His Lys Ala Gln Gly
385 390 395 400
Met Ser Val Asp Arg Ala Phe Ile Tyr Thr Pro Cys Ile His Tyr Ala
405 410 415
Asp Val Glu Leu Ala Gln Gln Leu Leu Tyr Val Gly Val Thr Arg Gly
420 425 430
Arg Tyr Asp Val Phe Tyr Val
435
<210> 52
<211> 69
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 衔接子上链
<400> 52
ccaatttgtg gttccttttt ggcgtctgct tgggtgttta accaaggcgt ctgcttgggt 60
gtttaacct 69
<210> 53
<211> 74
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 衔接子下链
<400> 53
ggttaaacac ccaagcagac gccttggtta aacacccaag cagacgccaa aaaggaacca 60
caaattggag caat 74
