一种利用FRET探针鉴定绿壳蛋鸡基因型的引物及应用
文献发布时间:2023-06-19 09:27:35
技术领域
本发明属于家禽分子生物技术及育种领域,主要是涉及一种用于鉴定绿壳蛋鸡基因型的引物及其应用与使用方法。
背景技术
鸡蛋的营养品质和外观影响其经济价值。绿壳蛋品质优良,相对于非绿壳蛋,绿壳蛋具有蛋白质含量高、人体必需氨基酸及鲜味氨基酸含量高及不饱和脂肪酸含量高等特点;同时,绿壳蛋中VA、胆固醇、磷、钾含量显著高于非绿壳蛋,是理想的天然保健食品。而且,产绿壳蛋是我国少部分地方鸡品种(如:雪峰乌骨鸡)的特色性状,故绿壳被视为土鸡蛋的标签,具有良好的育种价值。
鸡绿壳蛋性状是由显性单基因控制的质量性状,绿壳蛋形成的分子机制是:内源性逆转录病毒EAV-HP元件以反向插入的方式,整合在溶质载体有机阴离子转运蛋白家族成员SLCO1B3基因的5’—非编码区,从而启动了绿壳基因(SLCO1B3)在卵壳腺中的特异性高表达,蛋壳在卵壳腺中着绿而产生绿壳蛋。因此,EVA-HP插入时产生突变(Mutant type),蛋壳为绿色;无EVA-HP插入为野生型(Wild type),蛋壳不呈现绿色。
产蛋性状为限性性状,故无法根据表型对公鸡进行选择;同时,因绿壳受显性基因控制,难以根据蛋壳颜色进行选择,在母鸡群中也难以完全淘汰绿壳杂合子,这些都制约着绿壳蛋纯系的选育效率和种质资源的开发利用。因此,研发准确、稳定和便捷的绿壳分子标记开展辅助选择,可快速提升我国地方鸡品种绿壳种群或纯系的选育效率,提高育种效率和经济价值。
发明内容
本发明的目的是提供一种一种利用FRET探针鉴定绿壳蛋鸡基因型的引物及应用,5条寡核苷酸DNA序列联用进行PCR后升温熔解,根据熔解峰准确鉴别EAV-HP的插入与否,从而成为筛选绿壳蛋鸡的有效分子遗传标记。
本发明的实现要求:用于鉴定鸡绿壳基因型的引物,包括3条引物,即1条上游引物F,1条下游引物BSR,1条下游引物NSBR;探针Anchor、探针Sensor,上游引物F及下游引物BSR为SLCO1B3基因EAV-HP序列插入引物,上游引物F与下游引物NBSR为SLCO1B3基因非插入序列引物。其中,上游引物F的引物序列为:5’-TCAGGAACACCTCTGTAGTCA-3’,下游引物BSR的引物序列为:5’-GGGAGATGTTGTATGCGTAG-3’;下游引物NBSR的引物序列为:5’-TGTTGGTACTTGGTAGAGGAA-3’。两条探针为,Anchor probe:5’-ACCAGCGTAGATAAACATGTATTTTGGGACCTTCAACA-BlackHQ-3’;Sensor probe:5’-FAM-AGGGCAGAACTGGGAGGAGTGT-PO
所述的引物在鉴别鸡绿壳基因型中的应用的实现步骤:
1、以待检测鸡的DNA基因组为模板,用引物进行PCR扩增,其中F引物与下游引物BSR扩增产物长度为535bp,F引物与下游引物NBSR扩增产物为284bp。
2、得到扩增产物后利用FRET探针技术对扩增产物进行基因分型,其中扩增产物为535bp的片段为绿壳基因片段。
提取雪峰乌骨鸡基因组DNA,按照DNA的提取方法提取鸡的DNA。
用探针及引物,对样品进行扩增并进行FRET探针检测,确定样品的绿壳基因型。
对样品进行的普通PCR扩增引物所依据的序列可从GenBank中获得,编号为KP256532.1。
本发明的主要技术贡献在于:
1、针对EAV-HP基因插入位点设计出了用于探针检测的引物与探针;
2、利用FRET探针技术检测PCR产物中的不同基因型,证实此方法可靠。
需要特别说明的是,引物F、引物BSR、引物NBSR、探针Anchor、探针Sensor和PCR反应液之间的用量比例的选取是一个重要因素,但并不影响该用量比例的选取,扩增试剂最好使用分装的Taq酶、10×PCR Buffer和dNTPs。
为了让使用本发明进行的实验更为简便,本发明推荐采用如下用量比例方案:当以25μL反应体系为基准时,体系组成如下:F引物(10μmol/L)0.5μL,BSR(10μmol/L)和NBSR(10μmol/L)分别为2.5μL,10×PCR Buffer 2.5μL,dNTPs(25μmol/L)1μL,Taq DNAPolymerase(含Mg
此外,FRET技术是早已存在的技术,但迄今为止,国内外尚无公开报道将其用于SLCO1B3基因分型中。
现对FRET探针技术简述如下:
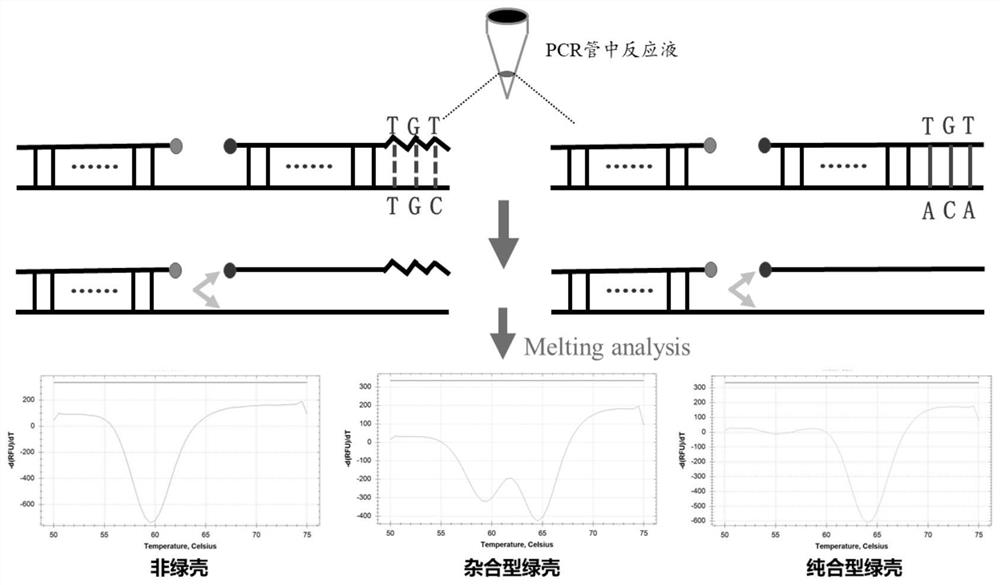
原理:FRET探针依靠荧光能量从一个荧光染料到另一个的传递。两个独立的特异寡核苷酸序列都标记上荧光基团。上游探针Anchor在3’末端提供一个供体基团,下游探针Sensor在5’末端有一受体基因。设计探针是他们在与目标序列结合时互相临近,使供体和受体荧光基团紧密接近。一旦探针杂交到模板上,从供体到受体荧光基团的能力传递产生了一个不同波长的荧光信号。供体荧光信号的减弱和受体信号的加强都能分别检测到。因此,只有当两个探针都结合上去才能检测到荧光信号。
基因点突变的检测基于两条探针,一条探针横跨突变位点,另一条为锚定探针,与无突变位点的靶序列杂交。两条探针用两种不同的发光基团标记。如靶序列中无突变,探针杂交便完全配对,如有突变,则探针与靶序列不完全配对,会降低杂交体的稳定性,从而降低其熔解温度。这样便可对基因的突变和多态性进行分析。
本发明采用改进的FRET探针原理:
本发明所设计的是经过改进的FRET探针,上游探针不标记荧光信号,只携带淬灭基团。
1、采用荧光基团与淬灭基团的组合代替了之前的两个荧光基团(荧光能量共振转移);
2、在退火阶段,55℃检测时(温度较低,时间较短)。两条探针未充分杂交上去,故没有信号变化;
3、熔解曲线体现的是sensor探针的熔解过程,anchor探针是Tm较高的探针,在sensor探针熔解反应前保持杂交与目标序列;
4、在熔解过程中,当两个探针都结合上去时,因为淬灭剂的存在,荧光基团被激发后产生的光子被淬灭,所以产生了一个倒置峰。
这样在所有的荧光定量PCR上只利用一个通道在不到两个小时就能对36、72、96个,甚至多达384个样本(根据所使用仪器的孔道)进行检测。
本发明根据SLCO1B3基因突变型和野生型DNA序列差异,针对性在插入位点设计探针,以准确区分基因型。
附图说明
图1是FRET技术原理图。
图2是实施例中的基因分型结果图。
具体实施方式
实施例1:
本实施例中所用的试剂为分离包装的引物F、引物BSR、引物NBSR、探针Anchor、探针Sensor和PCR反应液,其中
引物F和BSR、NBSR的序列为:
F:5’-TCAGGAACACCTCTGTAGTCA-3’(SEQ ID No.3)
BSR:5’-GGGAGATGTTGTATGCGTAG-3’(SEQ ID No.4)
NBSR:5’-TGTTGGTACTTGGTAGAGGAA-3’(SEQ ID No.5)
探针序列为:
探针Anchor:5’-ACCAGCGTAGATAAACATGTATTTTGGGACCTTCAACA-BlackHQ-3’(SEQID No.6)
探针Sensor:5’-FAM-AGGGCAGAACTGGGAGGAGTGT-PO
在所述探针Anchor、Sensor上,BlackHQ是一种淬灭剂,FAM是绿色荧光标记物。
本实施例中以25μL反应体系为基准制作,体系组成如下:F引物(10μmol/L)0.5μL,BSR(10μmol/L)和NBSR(10μmol/L)分别为2.5μL,10×PCR Buffer 2.5μL,dNTPs(25μmol/L)1μL,Taq DNA Polymerase(含Mg
实验步骤:
1)DNA提取:按照常规的DNA提取方法或试剂盒进行DNA提取。
2)目的基因扩增:
采用上述反应体系对绿壳基因进行PCR扩增。扩增时使用机器为美国伯乐Bio-Rad实时定量PCR仪CFX384。
PCR反应程序:
熔解分析程序:
95℃3min
50℃30min
50℃升温至75℃,0.5℃/5s FAM通道检测荧光。
3)基因型分析
根据熔解峰位置的不同,判断不同的基因型。绿壳纯合型的熔解峰位于非绿壳型之后,杂合型绿壳熔解峰横跨两者。(如图2所示)。
4)测序验证
采用雪峰乌骨鸡绿壳纯合以及杂合型个体对上述结果进行测序验证。
待测鸡基因组DNA提取按照常规酚氯仿抽提法或试剂盒法进行提取。
待测个体基因型测序验证:
引物采用上述F、BSR、NBSR引物组合。
多重PCR反应体系(25μL):2*Taq PCR MasterMix(北京天根)12.5μL、F引物(10μmol/L)1μL、BSR(10μmol/L)和NBSR(10μmol/L)各0.5μL,样本DNA(80ng/μL)1.0μL,ddH
PCR反应程序:95℃预变性3min,95℃变性30s,55℃退火30s,72℃延伸30s,最后72℃延伸5min,12℃保存。
PCR产物检测:120V,2%琼脂糖凝胶电泳30min。
产物测序:测序结果如SEQ ID No.1和Seq ID No.2。
需要说明的是,上述实施例只为说明本发明的技术构思及特点,并不以限制本发明的保护范围。凡根据本发明精神实质所作的等效变化或修饰,都应涵盖在本发明的保护范围之内。
序列表
<110> 湖南农业大学
湖南云飞凤农业有限公司
<120> 一种利用FRET探针鉴定绿壳蛋鸡基因型的引物及应用
<160> 7
<170> SIPOSequenceListing 1.0
<210> 1
<211> 535
<212> DNA
<213> 鸡(Gallus gallus)
<400> 1
tcaggaacac ctctgtagtc aggacacccc atttttaaac atcaggaagg tgtgaatgac 60
ttgaatttat aaactgaata agcttgggaa agcaggcatt tcacaaacgg gtgtacaaat 120
agaacacact tgcacatttt ctacccaatt aatttttgac cagcgtagat aaacatgtat 180
tttgggacct tcaacagagg gcagaactgg gaggagtgtt gcatgtagtc tccgttcgct 240
cgtccggtgt tcgtcctctg tccacatgta gggcttactg ctgggcgaaa ccgacccttt 300
accaggtcgg ggccagatgc tcacccagac cccaggagta agtgaggcaa atggcgttta 360
ttgctatagg ctacgtgttt aaatacaagt gtttcctcca atcacgaagt tacacttggc 420
acacaaaggt ggcataacac acaggtggca taggaacctg cacgcgtcac acctcgtttc 480
cctcgctacg cctacaacac acctcgtttc cctcgctacg catacaacat ctccc 535
<210> 2
<211> 284
<212> DNA
<213> 人工序列(artifical sequence)
<400> 2
tcaggaacac ctctgtagtc aggacacccc atttttaaac atcaggaagg tgtgaatgac 60
ttgaatttat aaactgaata agctcgggaa agcaggcatt tcacaaacgg gtgtacaaat 120
agagcacact tgcacatttt ctacccaatt aatttttgac cagcgtagat aaacatgtat 180
tttgggacct tcaacagagg gcagaactgg gaggagacaa gattaaaact gcttaaacca 240
tagtttgtgt tagaagttaa tgattcctct accaagtacc aaca 284
<210> 3
<211> 21
<212> DNA
<213> 人工序列(artifical sequence)
<400> 3
tcaggaacac ctctgtagtc a 21
<210> 4
<211> 20
<212> DNA
<213> 人工序列(artifical sequence)
<400> 4
gggagatgtt gtatgcgtag 20
<210> 5
<211> 21
<212> DNA
<213> 人工序列(artifical sequence)
<400> 5
tgttggtact tggtagagga a 21
<210> 6
<211> 38
<212> DNA
<213> 人工序列(artifical sequence)
<400> 6
accagcgtag ataaacatgt attttgggac cttcaaca 38
<210> 7
<211> 22
<212> DNA
<213> 人工序列(artifical sequence)
<400> 7
agggcagaac tgggaggagt gt 22
