MTHFD2蛋白作为食管癌转移诊断和/或预后评估的生物标记及其应用
文献发布时间:2023-06-19 09:29:07
技术领域
本发明涉及生物技术领域,特别是涉及MTHFD2蛋白作为食管癌转移诊断和 /或预后评估的生物标记及其应用。
背景技术
食管癌是最常见的恶性肿瘤之一。全世界确诊病例中,中国人占很大比例。尽管在治疗上有所改善,但大多数EC患者在确诊时已进入晚期,5年总生存率低于20%。因此,迫切需要寻找新的治疗靶点和分子生物标记物,用于早期发现和临床治疗。
基因组改变和表观遗传调控在肿瘤发生发展中起着关键作用。DNA甲基化是一种显性的表观遗传修饰,参与调控肿瘤基因组。CpG岛或启动子区域的高甲基化经常导致肿瘤抑制基因沉默,而区域的低甲基化与癌基因的上调相关。大规模和多组学分析为各种癌症的基因组和表观基因组异常提供了全面的分析。在食管鳞状细胞癌(ESCC)中,许多与癌症相关的基因通过调节DNA甲基化而发挥调控功能。例如,在食管癌细胞中发现IGFBPL1完全甲基化,它通过抑制PI3K-AKT信号传导抑制食管癌细胞生长。在ESCC样本中发现TGFBR2启动子高甲基化,慢病毒介导的TGFBR2过表达可抑制ESCC细胞增殖。通过对 DNA甲基化和RNA测序数据的综合分析,Chen等人鉴定出ESCC中的16个关键基因,如SIX4、CRABP2和EHD3,表明DNA甲基化与mRNA表达呈负相关。因此,多组分综合数据可以准确筛选潜在的预后生物标志物,为癌症的精确治疗提供新的视角。
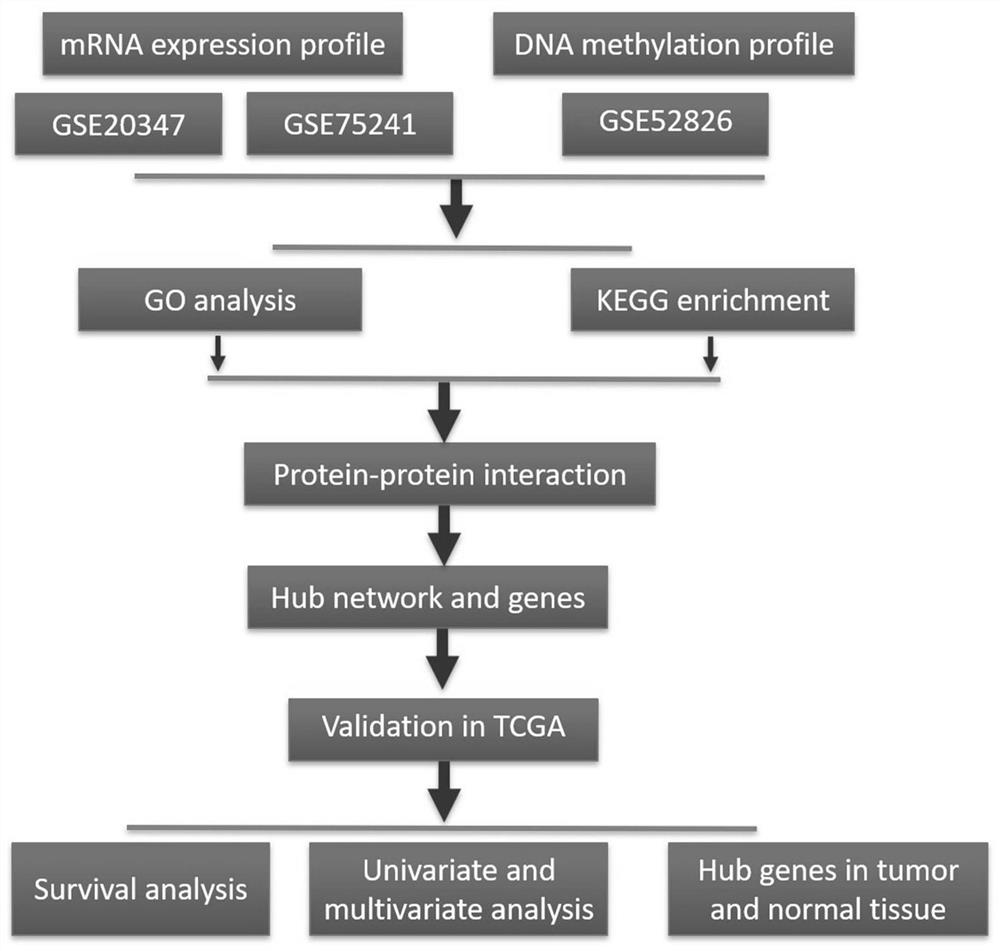
在本发明中,通过整合基因表达综合数据库(GEO)中转录组和甲基化数据的分析,我们筛选了EC样本与正常组织样本相比差异表达和差异甲基化的基因。通过蛋白质相互作用(PPI)网络的构建,对候选基因进行功能富集分析,确定相关的基因本体(GO)术语和路径类别。在进一步的数据验证之后,我们重点研究了三个表达水平和DNA甲基化状态相反的基因(MSN、MTHFD2和 PELI1)。采用生存分析和cox回归分析探讨其对EC患者预后的影响。基于转录组数据和DNA甲基化谱的生物信息学分析,设计概念的示意图如图1所示。我们的系统分析结果为了解EC的病理生物学提供了新的见解,并可能有助于开发新的EC患者预后生物标记物。
发明内容
本发明的目的是针对现有技术的上述不足,提供一种食管癌转移诊断和/或预后评估的生物标记。
本发明的木的可通过以下技术方案实现:
MTHFD2蛋白作为生物标记物在制备食管癌转移诊断和/或预后评估试剂、试剂盒或检测装置中的应用。
本发明确定了8个表达反向和甲基化相关的hub基因为候选基因。EC患者 MSN、PELI1、MTHFD2的异常表达与总生存时间相关(P<0.05)。单因素分析(P=0.037)和多因素分析(P=0.043)显示,只有MTHFD2与病理分期显著相关。我们的研究通过对转录组数据和甲基化谱的综合分析确定了一些具有预后价值的新生物标记物。MTHFD2可作为预测EC预后和病理分期的独立生物标志物。
检测MTHFD2蛋白表达的试剂在制备食管癌转移诊断和/或预后评估试剂、试剂盒或检测装置中的应用。
一种食管癌检测试剂盒,包括与MTHFD2蛋白特异性结合的检测物。
所述检测物优选与MTHFD2蛋白特异性结合的抗体或者与MTHFD2编码基因特异性结合的引物或探针。
本发明的有益效果是:本发明揭示确定了8个表达反向和甲基化相关的 hub基因为候选基因。EC患者MSN、PELI1、MTHFD2的异常表达与总生存时间相关(P<0.05)。单因素分析(P=0.037)和多因素分析(P=0.043)显示,只有MTHFD2与病理分期显著相关。我们的研究通过对转录组数据和甲基化谱的综合分析确定了一些具有预后价值的新生物标记物。MTHFD2可作为预测 EC预后和病理分期的独立生物标志物。
DNA甲基化是食管癌(EC)重要的表观遗传调控机制,与基因组不稳定和癌变密切相关。本研究旨在探讨肿瘤生物标记物对EC患者预后的预测作用。我们从GEO数据库下载了与EC和DNA甲基化相关的mRNA表达谱。筛选肿瘤组织与癌旁正常组织间差异表达和差异甲基化基因。进行GO项和KEGG路径分析,然后进行PPI网络构建。基于TCGA数据库的甲基化谱进行数据验证。通过生存分析和cox回归分析进一步验证了候选基因。我们发现肿瘤样本与正常样本相比,存在多个差异表达或甲基化状态的基因。在对三个差异基因集进行交叉后,我们最终得到了232个重叠基因。功能富集分析表明这些基因与谷胱甘肽代谢途径、p53信号途径、局灶性粘连等有关。MTHFD2可作为预测EC 预后和病理分期的潜在靶点。
附图说明
图1为本发明的基于转录组数据和DNA甲基化谱的生物信息学分析设计概念示意图。
图2为本发明的ECs中差异表达和甲基化基因的鉴定
图2A、图2B、图2C(左)为本发明根据微阵列数据分析鉴定EC样本中的候选基因。在GSE20347、GSE75241和GSE52826数据集的热图中显示了层次聚类分析结果。列和行代表临床样本和差异基因示意图。
图2A、图2B、图2C(右)为本发明火山图显示了基因在转录和甲基化水平上的表达变化图。log2fold≥2和P<0.05的基因分别标记为红色和绿色。左侧的绿点是显著下调或低甲基化的基因,而右侧的红点是指上调或高甲基化的基因的示意图。
图2D为本发明文氏图分析表明图,在3个数据集的基础上,共有232个重叠基因被确定为进一步研究的候选基因示意图。
图3A为本发明根据GO分析确定FDR值小于0.05的前20个功能项,包括BP、CC、MF的综合评价。红色的点表示p值较小。节点大小与基因数相对应,较大的点意味着更多的由GO项富集的基因数示意图。
图3B为本发明通过ClueGO插件丰富性分析的重要路径类别示意图。
图4A为本发明与EC中差异表达基因相关的PPI网络分析结果示意图。
图4B为本发明与EC相关的hub基因调控网络示意图。
图5为本发明在系数小于-0.3和P<0.05的阈值下识别具有逆甲基化和表达状态的hub基因示意图。
图6MTHFD2作为食管癌独立危险因素的研究
图6A为本发明Kaplan-Meier图显示了MSN、MTHFD2和PELI1三个候选基因的生存分析结果示意图。
图6B为本发明将基于TCGA和GSE17351数据集,对MTHFD2在正常组织和肿瘤组织中的表达进行了数据验证示意图。
具体实施方式
为了方便理解本发明的上述技术方案,以下通过具体使用方式上对本发明的技术方案进行详细说明:
下面结合附图对本发明做进一步说明。
实施例1
材料和方法
数据资源与DEGs筛选
分别从GEO数据库下载EC相关的转录组数据(访问号GSE20347、 GSE75241、GSE17351)和DNA甲基化谱(GSE5286]),包括相应的探针注释信息。GSE20347的mRNA微阵列数据集包括34个临床样本,并在 Affymetrix人类基因组U133A 2.0阵列平台上进行检测;GSE75241由30个活检样本组成,并在Affymetrix人类外显子1.0ST阵列平台上进行验证。GSE17351包括5对原发性ESCC肿瘤和在Affymetrix人类基因组U133 Plus 2.0 阵列平台上检测的正常组织。从TCGA数据库中提取的EC甲基化数据作为数据验证。此外,GSE52826的DNA甲基化谱是用450K的核酸甲基化芯片产生的,数据集包括来自ESCC患者和健康人的12个临床样本。
此外,limma软件包被用于筛选肿瘤组织和邻近正常组织之间的差异表达基因(DEGs)。用minfi软件包[16]分析DNA甲基化数据集GSE52826,将差异基因筛选标准设为|倍变|大于2,调整p值小于0.05。筛选差异甲基化基因的标准为β2,调整P<0.05。利用pheatmap软件包,火山图被用来可视化整个上调和下调基因。选择P<0.05和|log2fold(FC)|≥2的基因作为统计差异。
DEGs的功能富集分析
用于注释、可视化和集成发现的数据库(DAVID, http://david.abcc.ncifcrf.gov网站/)生物信息学资源是由批处理基因的功能注释组成,可按GO术语进行分类,如生物过程(BP)、细胞成分(CC)和分子功能 (MF)。
而且,KEGG(http://www.genome.jp/kegg/)进行富集分析以确定这些DEGs 的潜在途径。p值小于0.05为显著性差异。
PPI网络建设
通过在线工具串搜索蛋白质之间的相互作用(http://www.string-db.org/,版本11.0),相关系数得分>0.4作为阈值。使用Cytoscape软件构建PPI网络。利用MCODE插件从大蛋白相互作用网络中筛选出功能模块。P值小于0.05为显著性差异。
生存分析
利用Pearson相关系数(PCC)评价基因转录组水平与甲基化水平的相关性。小于0的系数值被认为具有统计学意义。此外,我们进一步进行了生存分析并构建了cox回归模型,以筛选与EC进展相关的候选基因。根据单变量和多变量分析结果,我们确定了对EC患者有预后价值的关键基因。
结果
ECs中差异表达和甲基化基因的鉴定
用limma包和minfi包进行mRNA表达和DNA甲基化分析。数据标准化后,对GSE20347和GSE75241的层次聚类分析表明,在EC组织样本和正常样本中,多个基因表现出显著的差异表达(图2A,B)。随后,我们从GSE52826数据集中发现了大量差异甲基化基因,并随机选取100个基因进行聚类分析。热图谱结果表明,这些基因在EC样品和正常样品之间表现出显著的甲基化差异。大量位于火山图两侧的点表明许多探针显示出不同的甲基化状态(图2C)。
本发明提取了三个差异基因集的交叉点,最后在三个微阵列数据集中获得了232个具有差异表达或甲基化水平的重叠基因(图2D)。
EC相关基因的功能富集分析
GO分析表明这些蛋白质与BP、CC和MF的许多方面有关。通过评估基因计数和FDR值,我们筛选出前20个生物类别,如“细胞骨架”、“细胞周期”、“生物粘附”、“细胞粘附”、“细胞外区部分”、“M期”和“损伤反应”(图3A)。
KEGG富集分析表明,这些候选基因与多种信号通路相关,如“小细胞肺癌”(hsa05222,计数=5)、“p53信号通路”(hsa04115,计数=7)、“谷胱甘肽代谢”(hsa00480,计数=7)、“局灶性粘附”(hsa04510,计数=9),“脂肪酸代谢” (hsa00071,计数=4)、“ECM受体相互作用”(hsa04512,计数=9)、“DNA 复制”(hsa03030,计数=4)、“细胞周期”(hsa04110,计数=8)和“不饱和脂肪酸的生物合成”(hsa01040,计数=3)。使用clueGO插件在图3中显示了信号通路。
PPI网络分析
在研究了STRING数据库之后,本发明探索了这些蛋白质之间潜在的相互作用。使用Cytoscape软件可视化PPI网络(图4A)。网络由多个边缘和节点组成,代表了基因和相互作用。在这些基因中,本发明进一步从PPI网络中鉴定了几个hub基因,并使用MCODE插件构建了一个调控网络(图4B)。这些 hub基因被认为是与EC发生有关的候选基因,包括MTHFD2、MCM6、 CENPE、NCAPH、RRM2、PELI1、KIF15、CCNG2和MGST2。
经mRNA表达和DNA甲基化鉴定,EC中有8个差异甲基化基因
本发明分析了从TCGA数据库下载的EC的甲基化数据和转录组学数据。在系数小于-0.3,p值小于0.05的阈值下,我们最终筛选出8个基因,它们与 mRNA表达和DNA甲基化之间存在着相反的相关性(图5)。结果表明,8个基因在肿瘤组织中有高甲基化和下调,或低甲基化和上调,是与EC进展相关的关键基因。这些基因包括CCNB1、CENPF、KIF15、MCM7、MSN、MTHFD2、NCAPH和PELI1。
MTHFD2作为食管癌独立危险因素的研究
首先,生存分析显示MSN或PELI1高表达的患者与两个基因低表达的患者相比,总生存时间更长(图6A)。MSN,HR:0.672,95%置信区间0.169- 2.681,P=0.0468;PELI1,HR:0.228,95%置信区间0.037-1.405,P=0.0452。相比之下,MTHFD2表达水平较低的EC患者预后优于高表达组(HR:2.394, 95%CI 0.312-18.345,P=0.0361)。结果表明,MSN和PELI1可能是抑癌基因, MTHFD2可能是癌基因。
此外,本发明还结合年龄、性别、病理分期和肿瘤类型等临床病理特征分析了EC患者的总体生存率见表1。
表1为TCGA食管癌队列临床病理特征及重要生存基因的单因素和多因素分析示意表
HR,hazard ratio.CI,confidence interval.
单因素分析和多因素分析显示,MTHFD2的表达与病理分期相关(HR:1.971, 95%CI1.032-3.764,P=0.037;HR:1.885,95%CI0.965-3.681,P=0.043),提示 MTHFD2可能是EC进展的独立预后因素。
基于TCGA和GSE17351数据集的数据验证,进一步分析了MTHFD2在肿瘤和邻近正常组织中的表达水平(图6B)。与正常组织标本相比,食管癌标本 (GSE17351数据集,n=10,P=0.008;TCGA,n=173,P<0.001)中MTHFD2 显著上调,与我们的预测结果一致。本发明表明,MTHFD2可作为一个独立的生物标记物,与不良预后相关。
结论
在本发明中,我们根据转录数据和甲基化数据集分析,确定了232个表达异常和甲基化状态的基因。对这些基因的功能富集分析显示,它们与ECM受体相互作用、局灶粘附、p53信号通路、细胞周期和谷胱甘肽代谢等信号通路有显著相关性。在PPI网络中有8个基因被鉴定为hub基因,如MTHFD2、 MCM6、CENPE等。根据生存分析,MSN、MTHFD2和PELI1三个基因的失活与ECs的总生存时间相关。Cox回归分析显示MTHFD2的表达与病理分期有显著相关性(P<0.05)。最后,从TCGA数据库得到的数据验证表明, MTHFD2在肿瘤组织中的表达明显高于癌旁正常组织,提示MTHFD2可能是 EC发生发展中具有预后价值的关键基因。
MTHFD2编码一种具有亚甲基四氢叶酸脱氢酶和环水解酶活性的线粒体酶。这种蛋白在发育中的胚胎中表达,而在大多数成人组织中不表达;有趣的是,近年来的研究主要集中在MTHFD2在肿瘤生物学过程中的作用。MTHFD2蛋白在19种肿瘤中高表达,高表达与乳腺癌患者预后不良相关。shRNA或 siRNA靶向MTHFD2可减少乳腺癌细胞系的侵袭和迁移。然而,目前对 MTHFD2在EC中的调控机制还知之甚少。基于代谢酶表达分析,MTHFD2强调了线粒体一碳叶酸途径在各种癌症中的关键作用。由于叶酸代谢是细胞增殖的核心,因此MTHFD2也被报道为肿瘤细胞增殖所需的叶酸偶联酶。它负责增殖癌细胞中线粒体NADPH的产生,并参与生化反应,包括脱氧胞苷、嘌呤核苷酸的产生、氨基酸的相互转化。因此,我们认为MTHFD2是否通过调节一碳代谢和叶酸途径参与EC的发生发展,有待进一步证实。
此外,MTHFD2作为靶基因可被多种miRNAs调控。在结直肠癌中, MTHFD2的表达促进癌细胞生长,miR-33a-5p靶向的MTHFD2在肿瘤发生中起主要作用。另一篇论文显示miR-92a可以通过调节急性髓系白血病中 MTHFD2抑制癌细胞增殖并诱导凋亡。同样,miR-9可以直接靶向MTHFD2在乳腺癌细胞中发挥的抗增殖和促凋亡活性。最近的一项研究表明miRNA-940与 MTHFD2相互作用可导致线粒体叶酸代谢功能障碍,并导致胶质瘤进展的抑制。线粒体同工酶MTHFD2表达明显上调,与肿瘤预后有关。通过阻断线粒体叶酸途径,靶向MTHFD2可能是一种潜在的癌症治疗策略。
以上显示和描述了本发明的基本原理、主要特征和本发明的优点。本行业的技术人员应该了解,本发明不受上述实施例的限制,上述实施例和说明书中描述的仅为本发明的优选例,并不用来限制本发明,在不脱离本发明精神和范围的前提下,本发明还会有各种变化和改进,这些变化和改进都落入要求保护的本发明范围内。本发明要求保护范围由所附的权利要求书及其等效物界定。
- MTHFD2蛋白作为食管癌转移诊断和/或预后评估的生物标记及其应用
- 一种食管癌转移诊断和/或预后评估的生物标记及其应用
