检测胎儿游离DNA含量的数字PCR检测试剂盒及其方法
文献发布时间:2023-06-19 09:30:39
技术领域
本发明涉及生物技术领域,具体涉及一种检测胎儿游离DNA含量的数字PCR检测试剂盒及其方法。
背景技术
无创产前DNA检测技术(NIPT)在临床获得了广泛应用,研究表明,孕妇的外周血中含有胎儿游离DNA(cffDNA),对胎儿游离DNA进行检测可以实现出生缺陷的产前诊断和筛查。目前通过二代测序技术(NGS)已实现对胎儿染色体非整倍体的筛查和检测。但使用该技术时,可能出现由于cffDNA浓度过低而导致假阴性的发生,因此,cffDNA浓度高于4%被认为是一个非常重要的质控指标;另外,在无创单基因病检测中,对cffDNA浓度的准确估算更加重要,cffDNA浓度是分析位点变异的重要参数。
cffDNA来源于胎盘凋亡的滋养细胞,比母体游离DNA更短,长度约在140-145个碱基,而母体cfDNA一般长度约为166bp。cffDNA平均占母体血浆cfDNA总量的10%-20%,该比例因个体而异,一般随孕龄增加而增加。分娩后,cffDNA在数小时内即从母体血浆中清除。
目前检测胎儿游离DNA的比例主要依赖于Y染色体序列(例如SRY,DYS14基因),但该方法仅对男胎有效。研究发现,母体与胎儿游离DNA在甲基化表观修饰方面存在差异,因此开发出基于差异甲基化区域(DMR)进行检测的技术,包括Bisulfite(重亚硫酸盐)处理,通过bisulfite处理,将没有甲基化修饰的胞嘧啶转化为尿嘧啶,后续通过PCR-测序或者二代测序技术可以获得每个胞嘧啶位点上的甲基化修饰的百分比。该方法作为甲基化检测的金标准,准确,但bisulfite处理的转换效率非常重要,目前商业化的bisulfite kit的转化效率可以达到99%以上。Bisulfite处理的问题在于步骤繁琐,处理后DNA损失较高,因此需要的DNA量较高,目前商品化的试剂盒一般需要20ng DNA以上才能进行处理,另外,bisulfite处理后DNA断裂损伤比较严重,对于游离DNA来说,可能较难达到较高的灵敏度。
另外一种方法是限制性内切酶处理(下文简称酶切法),使用甲基化敏感的限制性内切酶消化DNA样本,针对特定酶切位点设计引物,通过PCR检测酶切产物,从而判断该位点是否存在甲基化修饰及修饰的比例,这种方法需要检测区域具有甲基化敏感的限制性内切酶酶切位点,而且要确保酶切反应完全。另外,该方法目前一般处理时间较长,需要分步加入不同的限制性内切酶,以达到完全消化的目的。
数字PCR是适用于对核酸进行定量的技术,该技术通过微流控芯片手段,生成数微米到数百微米的液滴,而微液滴可以看作皮/纳升尺度的试管,包裹单分子进行PCR反应。数字PCR独特的优势如下:(1)灵敏度高,一个液滴可包含单个分子或细胞,在物理层面实现单分子级检测;(2)准确性强,每次微流控芯片可生成数百万个微液滴,对逐个微液滴统计,通过泊松分布计算模板数量,可实现数字化,结论可靠。
本发明针对酶切法反应时间长,多数隔夜消化(16小时),反应不完全等缺点进行优化和改进,发明一种基于酶切法和数字PCR定量检测胎儿游离DNA含量的试剂盒及其方法。
发明内容
为了解决上述问题,本发明提供一种检测胎儿游离DNA含量的数字PCR检测试剂盒,所述试剂盒包括分别用于检测胎儿标志物基因的含CpG岛区域、内参基因含甲基化敏感的限制性内切酶酶切位点区域和所述内参基因不含甲基化敏感的限制性内切酶酶切位点区域的数字PCR检测试剂,所述胎儿标志物基因的含CpG岛区域是胎儿和母体差异甲基化表达的区域,可通过甲基化敏感的限制性内切酶进行区分,去除母体中该区域序列,从而得到胎儿游离DNA的含量;和,所述数字PCR检测试剂包括分别用于检测胎儿标志物基因的CpG岛区域、内参基因含甲基化敏感的限制性内切酶酶切位点区域和内参基因不含甲基化敏感的限制性内切酶酶切位点区域的第一检测探针、第二检测探针和第三检测探针,所述第一检测探针用第一种荧光标记,所述第二检测探针用第一种荧光标记和第二种荧光分别标记,和所述第三检测探针用第二种荧光标记。在本发明中,通过用两种荧光标记可以区分3个检测靶标,根据信号团位置进行区分,其中第一检测探针和第三检测探针分别在以全阴性液滴作为原点的垂直的两个方向,而第二检测探针同时含第一种荧光标记和第二种荧光标记,在微液滴中两个荧光标记均产生信号,从而使第二检测探针可以位于原点的斜上方,与第一和第三探针的位置区分,根据该设计,可以对每个检测靶标进行准确定量。
在一种实施方式中,所述数字PCR检测试剂盒包括甲基化敏感的限制性内切酶试剂,所述限制性内切酶试剂是多种限制性内切酶的组合。
在一种实施方式中,所述限制性内切酶酶切位点是GCGC或CCGG。
在一种实施方式中,所述限制性内切酶试剂是BstUI限制性内切酶、HhaII限制性内切酶和BstY1限制性内切酶的组合。
在一种实施方式中,所述胎儿标志物基因是RASSF1A基因,和/或,所述内参基因是ACTB基因。
在一种实施方式中,数字PCR扩增RASSF1A基因的上游引物是SEQ ID NO:1:AGCCTGAGCTCATTGAGCTG,下游引物是SEQ ID NO:2:AGCCTGAGCTCATTGAGCTG,和检测RASSF1A基因的探针是SEQ ID NO:3:CCAACGCGCTGCGCA。
在一种实施方式中,数字PCR扩增ACTB基因含甲基化敏感的限制性内切酶酶切位点区域的上游引物是SEQ ID NO:4:AGCCTGAGCTCATTGAGCTG,下游引物是SEQ ID NO:5:CGGCGGATCGGCAAA,和其检测探针是SEQ ID NO:6:ACCGCCGAGACCGCGTC。
在一种实施方式中,数字PCR扩增ACTB基因不含甲基化敏感的限制性内切酶酶切位点区域的上游引物是SEQ ID NO:7:GCATCCCCCAAAGTTCAC,下游引物是SEQ ID NO:8:CGCATCTCATATTTGGAATGAC,和其检测探针是SEQ ID NO:9:GTGGCCGAGGACTTTGATTGC。
在一种实施方式中,本发明提供一种检测胎儿游离DNA含量的数字PCR检测方法,所述方法包括检测胎儿标志物基因的含CpG岛区域、内参基因含甲基化敏感的限制性内切酶酶切位点区域和检测所述内参基因不含甲基化敏感的限制性内切酶酶切位点区域,所述胎儿标志物基因的含CpG岛区域是胎儿和母体差异甲基化表达的区域,可通过甲基化敏感的限制性内切酶进行区分,去除母体中该区域序列,从而得到胎儿游离DNA的含量;和,所述数字PCR试剂包括分别用第一检测探针、第二检测探针和第三检测探针检测胎儿标志物基因的CpG岛区域、内参基因含甲基化敏感的限制性内切酶酶切位点区域和内参基因不含甲基化敏感的限制性内切酶酶切位点区域,所述第一检测探针用第一种荧光标记,所述第二检测探针用第一种荧光标记和第二种荧光分别标记,和所述第三检测探针用第二种荧光标记。
本发明检测胎儿游离DNA含量的数字PCR检测试剂盒将数字PCR技术和限制性内切酶方法结合,实现了胎儿游离DNA含量定量的检测。本发明检测胎儿游离DNA含量的数字PCR检测试剂盒检测时,反应时间较普通限制性内切酶方法大大缩短,本发明试剂盒一次性加入所有的反应原材料,大大简化了操作,因为数字PCR在生成液滴时已对反应体系进行了再分配,不再进行后续的加酶操作步骤。因此,本发明一方面进行限制酶完全消化,另一方并可兼容后续的数字PCR反应。通过本发明,可对胎儿标志物基因的甲基化定量,从而定量胎儿游离DNA在母体中含量,母体游离DNA总量通过不含有酶切位点的内参基因片段进行定量,限制酶消化完全是准确定量的前提,否则会造成胎儿游离DNA定量结果偏高,因此,本发明进一步通过含酶切位点的内参基因片段的检测结果来质控酶切效果,当该片段不被检出时,说明酶切完全,通过该方法与男胎特异基因SRY定量结果进行比较,结果表明,本发明定量准确,没有性别选择性,是一个普适的母体血浆中胎儿游离DNA检测方式。
附图说明
为了更清楚地说明本申请实施例中的技术方案,下面将对实施例中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本申请中记载的一些实施例,对于本领域普通技术人员来说,在不付出创造性劳动的前提下,还可以根据这些附图获得其它的附图。
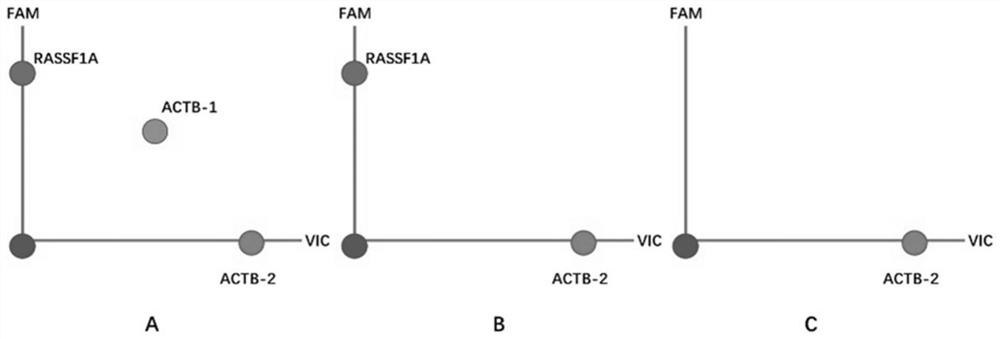
图1是本发明试剂盒检测原理示意图;
图2是RASSF1A甲基化酶切法与SRY定量胎儿游离DNA(cffDNA)比例的相关性结果图。
具体实施方式
为了使本领域技术领域人员更好地理解本申请中的技术方案,下面将结合实施例对本发明作进一步说明,显然,所描述的实施例仅仅是本申请一部分实施例,而不是全部的实施例。基于本申请中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其它实施例,都应当属于本申请保护的范围。下面结合附图及实施例对本发明作进一步描述。
实施例一本发明检测原理
本发明检测原理如图1所示:分别针对RASSF1A的CpG岛区和ACTB基因含酶切位点区域设计引物探针,作为甲基化敏感的限制性内切酶的作用靶标,同时,针对ACTB基因不含甲基化敏感的限制性内切酶酶切位点的区域作为扩增靶标,用于定量DNA总量。使用多个甲基化敏感的限制性内切酶对游离DNA进行消化,然后通过数字PCR定量检测未消化的甲基化RASSF1A(代表胎儿cffDNA含量)与游离cfDNA总量(不含酶切位点的ACTB含量)的比值,即为游离cffDNA的比例。具体描述如下:
(1)RASSF1A基因扩增靶标区域设计在CpG岛区,该区域是胎儿和母体差异甲基化表达的区域,可以通过甲基化敏感的限制性内切酶进行区分,去除母体中该区域序列,从而得到胎儿游离DNA的含量。
(2)针对母体和胎儿均低甲基化的ACTB基因设计两个靶标区域:一个含多个的酶切位点的区域(图中ACTB-1),用于质控酶切效果是否完全;另外一个是不含酶切位点的区域(图中ACTB-2),用于定量游离DNA总量。
图1中,A:cfDNA未进行酶切,所有靶标有信号;B:孕妇cfDNA酶切后,ACTB-1(含多个酶切位点)酶切完全,无信号,RASSF1A胎儿cffDNA甲基化,不被酶切,有信号,ACTB-2无酶切位点,不被酶切,有信号;C:非孕妇cfDNA进行酶切,ACTB-1(含多个酶切位点)酶切完全,无信号,RASSF1A非孕妇cfDNA无甲基化,酶切完全,无信号,ACTB-2无酶切位点,不被酶切,有信号。
实施例二本发明的试剂盒及其应用
1.提取cfDNA
将1-2ml血浆用游离DNA提取试剂盒(天根生物)按说明书操作进行DNA提取,最终得到的DNA洗脱液保存于-20℃备用。
2.数字PCR扩增体系
扩增体系30μL,终浓度含1×
酶切条件根据实验设定,PCR循环条件如下:95℃10min,95℃30秒和60℃60秒,45个循环。
表1.引物探针序列
3.甲基化敏感的酶组合筛选及优化试验
用非孕妇血浆样本提取的cfDNA混合后,进行酶组合消化的筛选和反应时间的优化,扩增反应体系如上,使用的cfDNA的量为15μL,加入不同的酶组合及进行不同反应时间(如表2所示),然后在PCR热循环仪上进行酶切反应和数字PCR反应。以不加酶的样本作为对照组,确定cfDNA是否完全消化。因为使用非孕妇cfDNA,如果没有检测到RASSF1A序列和ACTB-1序列的扩增,则认为是完全消化。检测结果如表2所示,单独使用甲基化敏感的BstUI酶,作用3小时不能达到完全消化,因此采用多个酶组合进行消化,可以看到,联合使用BstUI+HhaII+BstY1比用前两个酶效果更好,选择BstYI的目的是该酶在两个基因的检测区均基本不含该酶酶切位点,但使用该酶可将游离DNA中较大片段进行消化,从而提高其他两个酶的酶切效率,同时对于数字PCR来说,起到降低模板复杂度,使模板易于分散导不同液滴中从而提高检测灵敏度。当BstUI的使用量达到50U时,则基本上完全消化非甲基化的含酶切位点区域,无RASSF1A和ACTB-1的拷贝数检出,ACTB-2正常检出。酶切反应与后续的数字PCR反应结合起来,使用95℃10分钟作为限制性内切酶灭活程序,不影响结果检出。
表2不同酶组合消化试验
4.缩短消化时间试验
酶切法主要的一个问题是反应时间较长,因此本发明进一步考察缩短消化时间是否可行,用非孕妇血浆cfDNA为模板,使用50U BstUI+50U HhaII+10U BstYI组合消化,消化时间如表3所示,在消化时间为30min时,即可达到较好的完全消化的效果,消化15min,仍有微量的未消化的片段存在。因此,确定消化时间为30min。
表3不同消化时间试验
5.缩短灭活时间试验
限制性内切酶消化后需要进行灭活处理,否则可能影响后续数字PCR反应,因为后续合成的PCR产物没有甲基化的保护,可能被限制酶降解,但在数字PCR中,由于模板已在最初液滴生成时进行分割,因此,如果限制性内切酶未完全灭活,则主要影响阳性液滴中PCR产物量,从而影响信号强度,理论上不影响定量结果。用孕妇血浆样本提取的cfDNA混合后,进行酶灭活时间的优化,使用50U BstUI+50U HhaII+10U BstYI组合消化,扩增反应体系如上,使用的cfDNA的量为15μL,进行不同时间(如表4所示)的酶切消化处理,然后在PCR热循环仪上进行酶切反应和数字PCR反应。以不加酶的样本作为对照组,确定灭活时间是否影响检测效果。因为使用孕妇的cfDNA,理论上应检测到一定比例的RASSF1A序列,但ACTB-1序列未甲基化,应被消化而无扩增。检测结果如表4所示,不同的灭活时间不影响检测结果。定量的胎儿cffDNA比例比较一致。
表4.灭活时间试验
6.DNA模板投入量的影响
限制性内切酶是否能完全消化DNA模板也与模板投入量有较大关系,理论上,DNA量越高,则需要的酶量和反应时间越高越长,将非孕妇血浆样本cfDNA混合,然后用真空浓缩仪(Eppendorf)浓缩5倍,进行DNA模板投入量的验证,使用50U BstUI+50U HhaII+10UBstYI组合消化,消化和灭活时间为37℃30min+95℃10min,扩增反应体系如上,使用的cfDNA的量为3、9、18、45ng(如表5所示),然后在PCR热循环仪上进行酶切反应和数字PCR反应。确定在高DNA模板投入量是否可对DNA进行完全消化。检测结果如表5所示,当DNA模板投入量达到45ng(理论值为135000拷贝)时,仍能进行完全消化。
表5.DNA模板投入量
7.临床样本检测
用10个携带男胎的孕妇样品进行测试,提取cfDNA,使用50U BstUI+50U HhaII+10U BstYI组合消化,消化和灭活时间为37℃30min+95℃10min,扩增反应体系如上,与SRY/ACTB-2对照反应进行比较,确定本发明的检测方法的准确性。检测结果如表6,用RASSF1A甲基化酶切法和SRY定量结果基本一致,相关性较高,相关系数R2为0.9974,因此说明,本发明方法具有准确性,而且没有性别选择性,可作为普适的定量胎儿游离DNA比例的方法。
表6.临床样本检测结果
应该理解到披露的本发明不仅仅限于描述的特定的方法、方案和物质,因为这些均可变化。还应理解这里所用的术语仅仅是为了描述特定的实施方式方案的目的,而不是意欲限制本发明的范围,本发明的范围仅受限于所附的权利要求。
本领域的技术人员还将认识到,或者能够确认使用不超过常规实验,在本文中所述的本发明的具体的实施方案的许多等价物。这些等价物也包含在所附的权利要求中。
序列表
<110> 国家卫生健康委科学技术研究所
北京新羿生物科技有限公司
<120> 检测胎儿游离DNA含量的数字PCR检测试剂盒及其方法
<160> 12
<170> SIPOSequenceListing 1.0
<210> 1
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 1
agcctgagct cattgagctg 20
<210> 2
<211> 16
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 2
accagctgcc gtgtgg 16
<210> 3
<211> 15
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 3
ccaacgcgct gcgca 15
<210> 4
<211> 17
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 4
gcgccgttcc gaaagtt 17
<210> 5
<211> 15
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 5
cggcggatcg gcaaa 15
<210> 6
<211> 17
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 6
accgccgaga ccgcgtc 17
<210> 7
<211> 18
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 7
gcatccccca aagttcac 18
<210> 8
<211> 22
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 8
cgcatctcat atttggaatg ac 22
<210> 9
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 9
gtggccgagg actttgattg c 21
<210> 10
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 10
tggcgattaa gtcaaattcg c 21
<210> 11
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 11
ccccctagta ccctgacaat gtatt 25
<210> 12
<211> 26
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 12
agcagtagag cagtcaggga ggcaga 26
