一种高通量靶向建库的方法和应用
文献发布时间:2023-06-19 09:41:38
技术领域
本发明涉及测序检测技术领域,特别涉及一种高通量靶向建库的方法和应用。
背景技术
新一代测序在分子检测中得到越来越多的认同和应用;可以分为靶向测序,全转录组测序和全基因组测序。靶向测序是获取疾病相关基因信息高效可行的方法,但由于关键环节受限于现有的方法,费用仍然较为昂贵,且存在操作繁琐和灵敏度不高等不足。靶向建库是基因靶向测序的关键环节,目前国内外测序靶向建库方法主要采用多重PCR或探针捕获的方法。多重PCR建库实验条件摸索难度较大,且存在检测基因数目较少,无法去除PCR偏倚,检测方法灵敏度不高等缺点。探针捕获建库的方法需要合成特制的探针,成本高昂。这两种技术价格昂贵,也无法根据实际需求而快速灵活地应用。发明人整合上述两种方法,并对诸多技术点进行改进,提出一种反转探针捕获PCR的建库方法。反转探针捕获PCR建库方法利用常规分子试剂即可完成建库,大大降低实验条件摸索的难度,降低成本,提高灵敏度。该方法有望应用于药物代谢基因、疾病相关基因和肿瘤突变基因检测等,具有良好的应用前景。
发明内容
本发明的首要目的在于克服现有技术的缺点与不足,提供一种高通量靶向建库的方法。
本发明的另一目的在于提供上述高通量靶向建库的方法的应用。
本发明的目的通过下述技术方案实现:一种高通量靶向建库的方法,包括如下步骤:
(1)基于检测的基因设计并合成靶向捕获探针;
步骤(1)所述的靶向捕获探针(MIP)的骨架为5'端测序引物ATCCGACGGTAGTGT和3'端测序引物CTTCAGCTTCCCGAT,靶向捕获探针的骨架两端分别为靶向目的基因位点两端的靶向片段,左右各15-30nt,目的基因片段长度为100-200nt。
步骤(1)所述的靶向捕获探针序列为:NNNNNCTTCAGCTTCCCGATATCCGACGGTAGTGTNNNNN;其中NNNNN分别为靶向目的基因位点两端的靶向片段。
步骤(1)所述的靶向捕获探针是用TE buffer(PH 8.0)溶解,将溶解后的探针混合,配置成探针混合液(probe mix),再将探针混合液中的探针5'端加磷酸基团得到的靶向捕获探针。
所述的探针混合液的总浓度为100uM,每个探针的浓度为总浓度除以探针数,探针混合液中每一种探针加入量为1uL。
步骤(1)所述的基因包括肿瘤治疗药物相关基因和药物代谢相关基因。
步骤(1)所述的靶向捕获探针的骨架匹配Illumina测序平台。
(2)将样本基因组DNA和步骤(1)所述的靶向捕获探针杂交过夜,得到捕获的目的片段,环化,酶切,纯化,PCR扩增,得到构建的靶向测序文库;
步骤(2)所述的基因组DNA提取的样本优选为石蜡标本和外周血白细胞。
步骤(2)所述的基因组DNA与靶向捕获探针按500ng:1~100pmol的比例混合;优选为按500ng:3pmol的比例混合。
步骤(2)中所述的杂交的反应体系:2.5uL 10×Ampligase buffer、500ng基因组DNA、终浓度为3pmol的靶向捕获探针,ddH
步骤(2)中所述的杂交的反应条件:95℃、10min,0.1℃/s缓慢降温至60℃后保持24h。
步骤(2)中所述的环化是将0.32uL Hemo KlenTaq酶、0.5uL 0.025mM dNTPs、1uLAmpligase加入杂交后的捕获产物中环化。
步骤(2)中所述的环化的反应条件为60℃、20h。
步骤(2)中所述的酶切是采用核酸外切酶ExoⅠ和ExoⅢ各2uL进行消化。
步骤(2)所述的PCR扩增的引物中,上游引物为:adaptor序列-barcode序列-间隔序列-连接序列-靶向捕获探针骨架5'测序引物的相同序列。
所述的barcode序列相当于样本的“身份证”,一个样本对应一个barcode,在引物合成之后即可获得这段序列。
所述的上游引物优选为:5'-CCATCTCATCCCTGCGTGTCTCCGACTCAG-NNNNN-GAT-ACACGCACG-ATCCGACGGTAGTGT-3';所述NNNNN为barcode序列。
所述的adaptor序列匹配测序平台PGM。
步骤(2)所述的PCR扩增的引物中,下游引物为:测序平台的另一个adaptor序列和连接序列-靶向捕获探针骨架3'测序引物的反向互补序列。
所述的下游引物优选为:5'-TCCGCTTTCCTCTCTATGGGCAGTCGGTGATCATACGAGATCCGTA-ATCGGGAAGCTGAAG-3'。
所述的测序平台的另一个adaptor序列配测序平台PGM。
步骤(2)所述的PCR扩增得到的序列为:上游引物–NNNNN–捕获的目的基因序列–NNNNN–3'端测序引物;其中,捕获的目的基因序列为SNP位点,为100-200nt;NNNNN分别为靶向目的基因位点两端的靶向片段。
步骤(2)中所述的PCR扩增的反应体系:0.5uL Phusion酶、10uL 5×buffer、0.5uL10nM的dNTP、5uL模板、2.5nM的上下游引物各2uL,ddH
步骤(2)中所述的PCR扩增的反应条件:98℃、3min;98℃、20s,60℃、30s,72℃、30s,一共35个循环;72℃、5min。
(3)将步骤(2)所述的靶向测序文库纯化,检测文库DNA片段浓度和大小,进行二代测序,根据测序结果分析判断靶向测序文库的质量和测序信息。
步骤(3)所述的纯化为采用VAHTS DNA Clean Beads磁珠进行纯化,用30uL的ddH
一种用于上述高通量靶向建库的方法的试剂盒,包括上述高通量靶向建库的方法中所用的靶向捕获探针、PCR扩增引物、捕获和酶切目的片段的试剂、PCR扩增和纯化的试剂。
一种宏基因组测序方法,包括上述高通量靶向建库的方法和建库的产物,将其用于直接测序。
所述的测序的基因优选为药物代谢基因、疾病相关基因以及肿瘤突变基因。
本发明相对于现有技术具有如下的优点及效果:
(1)本发明能够在一轮PCR反应中靶向扩增一百多个检测基因,解决了现有多重PCR技术在同一PCR反应体系只能检测少量基因位点、实验条件难优化的问题。
(2)本发明在二代测序前只有一轮PCR反应,且探针骨架上有随机barcode,能够大大减少PCR造成的偏倚,能够识别PCR引入的碱基错误和测序引入的碱基错误。
(3)本发明简化了二代测序文库制备的操作步骤,减少了试剂和时间成本。
附图说明
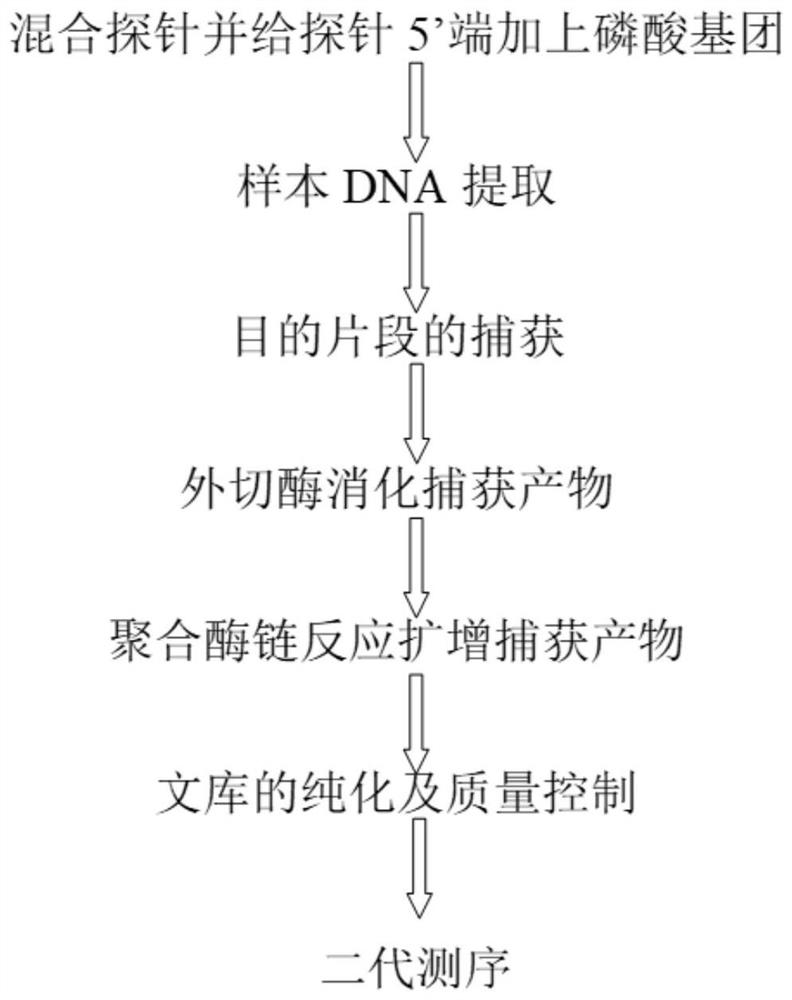
图1是本发明的流程示意图。
图2是本发明的原理示意图。
图3是不同浓度比例探针捕获文库的琼脂糖凝胶图。
图4是KIT探针捕获文库的一代测序验证图。
图5是PIK3CA探针捕获文库的一代测序验证图。
具体实施方式
下面结合实施例及附图对本发明作进一步详细的描述,但本发明的实施方式不限于此。
实施例1
1、探针的设计、合成和加磷酸基团
1.1设计探针
通过NCBI序列(https://www.ncbi.nlm.nih.gov/gene/),基于检测的基因位点设计靶向捕获探针(MIP)。探针骨架采用匹配Illumina测序平台的5’测序引物(ATCCGACGGTAGTGT)和3’端测序引物(CTTCAGCTTCCCGAT)各15nt,共30nt,探针两端分别为靶向目的基因位点两端的靶向片段,左右各15-30nt,目的基因片段长度为100-200nt,探针序列:
NNNNNCTTCAGCTTCCCGATATCCGACGGTAGTGTNNNNN;其中NNNNN为靶向目的基因位点两端的靶向片段。
所述的基因位点包括肿瘤治疗药物相关基因和药物代谢相关基因位点见表1,共设计16种探针,探针序列如SEQ ID NO.6~SEQ ID NO.21所示。
表1检测的目标基因
1.2在上海生工公司订购探针。
1.3合成的探针用TE buffer(PH 8.0)溶解,溶解后的探针浓度为100uM。
充分涡旋混匀配置成探针混合液。其中探针混合液(probe mix)总浓度为100uM,每个探针在混合液中的浓度为总浓度除以探针数,每一种探针加入的量都是1uL。
1.4按表2配置反应体系,在探针5’端加入一个磷酸基团。
表2
反应条件:37℃、45min,80℃、20min。
注意事项:引物溶解过程加样要准确,确保每一管引物的浓度都是100uM,并且充分混匀;配置probe mix的时候加样要准确,确保每一种探针加入的量都是1uL,以免造成引物浓度的不均匀。
1.5反应结束后把步骤1.4的探针混合液用去离子水稀释成不同的浓度。
2、DNA提取
2.1提取外周血白细胞的DNA(外周血白细胞来源于中山大学孙逸仙纪念医院,利用天根公司的血液/细胞/组织基因组DNA提取试剂盒(DP304),-20℃保存。提取具体步骤如下:
2.1.1使用EDTA抗凝的抗凝管采集外周血。
2.1.2取EDTA抗凝的血液,加入蛋白酶K溶液,混匀。
2.1.3再加入裂解液,充分颠倒混匀,70℃加热10分钟。溶液变清亮后简短离心去除EP管内壁的水珠。
2.1.4加无水乙醇,充分震荡混匀使DNA沉淀。
2.1.5将上一步的溶液和沉淀过吸附柱,离心后倒掉废液。
2.1.6向吸附柱内加入漂洗液,离心后倒掉废液。洗涤两次。
2.1.7倒掉废液后,再离心2分钟。将吸附柱打开盖子,室温静置几分钟至吸附材料晾干。
2.1.8将吸附柱转移至一个干净的离心管内,加入50μL TE缓冲液。室温放置3分钟后离心,将溶液收集至离心管内。
3、目的片段的捕获
3.1、将步骤1得到的5’端有磷酸基团的探针混合液与500ng步骤2得到的基因组DNA进行杂交过夜,捕获目的片段。反应体系见表3。设置对照:1、PCR NC:PCR的阴性对照,扩增模板为H
表3
反应条件:95度10min,0.1℃/s缓慢降温至60度后保持24小时。
捕获结束后,根据碱基互补配对原则,利用DNA聚合酶进行探针的环化,填补缺口。将表4的各物质加入捕获产物中。
表4
反应条件:60℃、20h。
注意事项:关于缓慢降温,如果没有实验条件,可以采用普通降温的方法,但是效果没有缓慢降温好。
4、外切酶消化及PCR反应
4.1、捕获产物用核酸外切酶ExoⅠ(M0293)和ExoⅢ(M0206)各2uL进行消化。
4.2、纯化消化产物,利用天根公司的通用型DNA纯化回收试剂盒(DP214)对酶切反应进行纯化。
4.3、PCR反应
4.3.1、PCR反应引入测序需要的adaptor和barcode序列。不同样品需要使用不同barcode的上游引物,可以使用同一条下游引物。
上游引物如下:
PGM-PCR-FW:5'-CCATCTCATCCCTGCGTGTCTCCGACTCAG-CTAAGGTAAC-GAT-ACACGCACG-ATCCGACGGTAGTGT-3'(SEQ ID NO.22);
下游引物如下:
PGM_PCR_RV:5'-TCCGCTTTCCTCTCTATGGGCAGTCGGTGATCATACGAGATCCGTA-ATCGGGAAGCTGAAG-3'(SEQ ID NO.23)。
PCR扩增得到的序列为:
5'-CCATCTCATCCCTGCGTGTCTCCGACTCAGCTAAGGTAACGATACACGCACGATCCGACGGTAGTGTNNNNNCTTCAGCTTCCCGAT-3';
其中,N为目的基因的SNP位点(100-200nt)以及目的基因位点两端的片段(各15-30nt)。
合成引物,将引物用TE buffer(PH 8.0)溶解,溶解后的引物充分涡旋混匀配置成引物混合液,总浓度为100uM。
4.3.2、PCR反应条件及体系如表5所示。
表5
反应条件:98度3min;98度20s,60度30s,72度30s,一共35个循环;72度5min。
5、纯化PCR反应产物
5.1、反应结束后,使用诺唯赞VAHTS DNA Clean Beads磁珠(N411),按照说明书,对建库PCR反应后的文库进行纯化,用30uL的ddH
5.2、用Qubit进行DNA定量及DNA浓度质控。
5.2、安捷伦Agilent 2100生物分析仪检测文库片段的大小,进行DNA片段长度质控。
5.3、取文库进行TA克隆(TAKARA,6011),将克隆的菌落送一代测序验证是否捕获到目的片段。
6、二代测序
文库DNA浓度和片段长度质量控制均合格后,按测序试剂盒的说明书要求将文库混合制备成文库混合物进行二代测序,获取二代测序结果,根据二代测序结果分析判断所述的靶向测序文库的质量。
以上本发明建库的流程和原理见图1和图2。利用不同浓度的探针捕获的建库产物如图3所示。图3表明目的基因的捕获及建库步骤适用于不同的基因组DNA与靶向捕获探针比例,基因组DNA与靶向捕获探针按500ng:1~100pmol的比例混合,进行捕获及PCR扩增之后,PCR产物进行琼脂糖凝胶电泳。文库大小为300bp。PCR NC:PCR的阴性对照,扩增模板为H
上述实施例为本发明较佳的实施方式,但本发明的实施方式并不受上述实施例的限制,其他的任何未背离本发明的精神实质与原理下所作的改变、修饰、替代、组合、简化,均应为等效的置换方式,都包含在本发明的保护范围之内。
序列表
<110> 中山大学孙逸仙纪念医院
<120> 一种高通量靶向建库的方法和应用
<160> 24
<170> SIPOSequenceListing 1.0
<210> 1
<211> 15
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 5’端测序引物
<400> 1
atccgacggt agtgt 15
<210> 2
<211> 15
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 3’端测序引物
<400> 2
cttcagcttc ccgat 15
<210> 3
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 靶向捕获探针序列
<220>
<221> misc_feature
<222> (1)..(1)
<223> n is a, c, g, t or u
<220>
<221> misc_feature
<222> (2)..(2)
<223> n is a, c, g, t or u
<220>
<221> misc_feature
<222> (3)..(3)
<223> n is a, c, g, t or u
<220>
<221> misc_feature
<222> (4)..(4)
<223> n is a, c, g, t or u
<220>
<221> misc_feature
<222> (5)..(5)
<223> n is a, c, g, t or u
<220>
<221> misc_feature
<222> (36)..(36)
<223> n is a, c, g, t or u
<220>
<221> misc_feature
<222> (37)..(37)
<223> n is a, c, g, t or u
<220>
<221> misc_feature
<222> (38)..(38)
<223> n is a, c, g, t or u
<220>
<221> misc_feature
<222> (39)..(39)
<223> n is a, c, g, t or u
<220>
<221> misc_feature
<222> (40)..(40)
<223> n is a, c, g, t or u
<400> 3
nnnnncttca gcttcccgat atccgacggt agtgtnnnnn 40
<210> 4
<211> 62
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 上游引物
<220>
<221> misc_feature
<222> (31)..(31)
<223> n is a, c, g, t or u
<220>
<221> misc_feature
<222> (32)..(32)
<223> n is a, c, g, t or u
<220>
<221> misc_feature
<222> (33)..(33)
<223> n is a, c, g, t or u
<220>
<221> misc_feature
<222> (34)..(34)
<223> n is a, c, g, t or u
<220>
<221> misc_feature
<222> (35)..(35)
<223> n is a, c, g, t or u
<400> 4
ccatctcatc cctgcgtgtc tccgactcag nnnnngatac acgcacgatc cgacggtagt 60
gt 62
<210> 5
<211> 61
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 下游引物
<400> 5
tccgctttcc tctctatggg cagtcggtga tcatacgaga tccgtaatcg ggaagctgaa 60
g 61
<210> 6
<211> 75
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> AKT1_1_0021
<400> 6
gtactcccct acagacgtgc gggtggtgac ttcagcttcc cgatatccga cggtagtgtg 60
agaagttgtt gaggg 75
<210> 7
<211> 75
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> APC_1_0061
<400> 7
cgttctctct ccaaacttct atctttttcc ttcagcttcc cgatatccga cggtagtgtc 60
ttcttccatg acttt 75
<210> 8
<211> 74
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> CYP19A1_1_0023
<400> 8
gccatgggcc actgagtgtt cactgtcttc agcttcccga tatccgacgg tagtgtctca 60
aactcttggc ctct 74
<210> 9
<211> 75
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> CYP2C19_1_0016
<400> 9
tcgatggaca tcaacaaccc tcgggacttc ttcagcttcc cgatatccga cggtagtgtg 60
caataatttt cccac 75
<210> 10
<211> 75
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> CYP2D6_1_0039
<400> 10
cagaacacac catactgctt cgaccaggtc ttcagcttcc cgatatccga cggtagtgtc 60
catcttcctg ctcct 75
<210> 11
<211> 75
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> DPYD_1_0004
<400> 11
gctcaatatt cagaaaggag ctttgtccac ttcagcttcc cgatatccga cggtagtgtc 60
ttatgccaat tctct 75
<210> 12
<211> 75
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> EGFR_1_0208
<400> 12
gaaatttaca gggtgagagg ctgggatgcc ttcagcttcc cgatatccga cggtagtgtt 60
ttggaaaacc tgcag 75
<210> 13
<211> 73
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> KIT_1_0051
<400> 13
gctgttatgc actgatccgg gctttgtctt cagcttcccg atatccgacg gtagtgtttc 60
tgtttttctt ggc 73
<210> 14
<211> 75
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> KRAS_1_0019
<400> 14
caagtttata ttcagtcatt ttcagcaggc ttcagcttcc cgatatccga cggtagtgtg 60
catattaaaa caaga 75
<210> 15
<211> 75
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> MET_1_0217
<400> 15
gtatgattgt ggggaaagac atgtcgctgc ttcagcttcc cgatatccga cggtagtgtt 60
tacagatgaa aggac 75
<210> 16
<211> 73
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> MTHFR_1_0002
<400> 16
gcaccgacat gggcatcact tgccccactt cagcttcccg atatccgacg gtagtgtggg 60
agctttgagg ctg 73
<210> 17
<211> 75
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> PIK3CA_1_0013
<400> 17
cgactttgtg accttcggct ttttcaaccc ttcagcttcc cgatatccga cggtagtgtt 60
taaagaagca agaaa 75
<210> 18
<211> 75
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> PTEN_1_0007
<400> 18
acttagactt gacctgtatc catttctgcc ttcagcttcc cgatatccga cggtagtgtg 60
cttctgccat ctctc 75
<210> 19
<211> 73
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> TP53_4_0004
<400> 19
gcaaaacatc ttgttgaggg caggggactt cagcttcccg atatccgacg gtagtgtctc 60
atggtggggg cag 73
<210> 20
<211> 75
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> UGT1A1_1_0037
<400> 20
gcaaaggcgc catggctgtg gagtcccact tcagcttccc gatatccgac ggtagtgtgt 60
ggactgacag ctttt 75
<210> 21
<211> 74
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> CTNNB1_1_0144
<400> 21
ccattctggt gccactaccc ttcagcttcc cgatatccga cggtagtgtc tactaatgct 60
aatactgttt cgta 74
<210> 22
<211> 67
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> PGM_PCR_FW
<400> 22
ccatctcatc cctgcgtgtc tccgactcag ctaaggtaac gatacacgca cgatccgacg 60
gtagtgt 67
<210> 23
<211> 61
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> PGM_PCR_RV
<400> 23
tccgctttcc tctctatggg cagtcggtga tcatacgaga tccgtaatcg ggaagctgaa 60
g 61
<210> 24
<211> 87
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> PCR扩增得到的序列
<220>
<221> misc_feature
<222> (68)..(68)
<223> n is a, c, g, t or u
<220>
<221> misc_feature
<222> (69)..(69)
<223> n is a, c, g, t or u
<220>
<221> misc_feature
<222> (70)..(70)
<223> n is a, c, g, t or u
<220>
<221> misc_feature
<222> (71)..(71)
<223> n is a, c, g, t or u
<220>
<221> misc_feature
<222> (72)..(72)
<223> n is a, c, g, t or u
<400> 24
ccatctcatc cctgcgtgtc tccgactcag ctaaggtaac gatacacgca cgatccgacg 60
gtagtgtnnn nncttcagct tcccgat 87
