一种联合检测慢性粒细胞白血病检测试剂盒
文献发布时间:2023-06-19 09:41:38
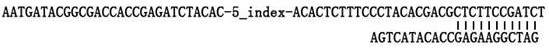
技术领域
本发明属于分子生物检测领域,具体涉及一种联合检测慢性粒细胞白血病检测试剂盒。
背景技术
慢性粒细胞白血病(chronic myeloid leukemia,CML)是基于多能干细胞水平上的恶性疾病,特点是具有获得性的异常的染色体,以骨髓及外周血中粒细胞的无限增殖为主要特征。慢性粒细胞白血病对血液和骨髓的影响主要表现在产生大量不成熟的白细胞,这些白细胞在骨髓内聚集,抑制骨髓的正常造血;并且能够通过血液在全身扩散,导致病人出现贫血、容易出血、感染及器官浸润等。
特异性的细胞遗传学变化在大约95%的慢性粒细胞白血病患者中都有发生,具体表现为9号和22号染色体发生易位,导致形成费城染色体(Ph染色体),并且产生BCR/ABL融合基因,其对应的融合蛋白具有可异常升高酪氨酸蛋白激酶活性的特点,此融合蛋白可催化并且磷酸化多种底物蛋白酪氨酸残基,继而激活多条信号传导途径,促进细胞増殖,减少细胞调亡。
慢性粒细胞白血病主要依靠血常规、骨髓穿刺活检来诊断的,血常规检测难以对疾病的早期进行判断,骨髓穿刺活检对病人造成的损伤较大且操作不便。
研究癌症的遗传谱能够鉴定导致恶性表型的基因。这些基因谱(包括基因表达和突变)提供了关于正常细胞和疾病细胞的生物学过程的有价值的信息。然而,癌症在它们的遗传“特征”中存在广泛的不同,这导致在诊断和治疗、以及开发有效疗法中存在困难。
传统的荧光PCR法检测存在每次只能检测一个位点,需要的样品量较多;检测成本较高;实验操作数量较多,检测程序耗时长等缺陷。
中国专利201710493398.0中公开了一种癌症相关基因突变高灵敏度检测方法和试剂盒。通过针对目的基因设计一组带标签引物组,并分别进行两步扩增,然后再进行二代测序的过程,完成癌症相关基因突变的检测。该方法能够有效排除假阳性的低频突变,检测结果灵敏度和特异性都较高。但其数据分析过程复杂,增加了检测时间。
提供一种能够简易且高效的慢性粒细胞白血病检测试剂盒十分必要。
发明内容
本发明提供了一种检测试剂盒,通过对送检样品的DNA和RNA共同提取,对RNA进行逆转录成cDNA后,与DNA一起进行高通量测序文库构建,并进行高通量测序,针对慢性粒细胞白血病常见的分子遗传学变异,一次性检测多种变异类型。
一方面,本发明提供了一组用于检测多种基因突变、融合、缺失等变异类型的特异性复合引物。
所述的特异性复合引物的序列如下表1。
表1 特异性复合引物表
表1续
所述的特异性引物与通用引物1同时使用,所述的通用引物1的序列为SEQ ID NO:38。
所述的基因突变、融合、缺失变异类型包括:BCR/ABL融合、FGFR1重排、JAK2基因V617F突变、JAK2重排、NUP98重排、PDGFRA重排、PDGFRB重排。
另一方面,本发明提供了一种联合检测慢性粒细胞白血病的试剂盒。
所述的联合检测慢性粒细胞白血病的试剂盒包括本发明上述的检测多种突变、融合、缺失等变异类型的特异性复合引物。
所述的试剂盒还包含一种DNA文库构建的专用接头,所述的专用接头为接头引物1分别和8条不同的i5端引物构成的8个二聚体。所述的接头引物1和8条不同的i5端引物的序列如下表2。
表2 专用接头引物表
所述的专用接头具有附图1所示的结构。其中,所述的5_index的序列为:CTCTCTAT、TATCCTCT、GTAAGGAG、ACTGCATA、AAGGAGTA、CTAAGCCT、CGTCTAAT或TCTCTCCG。
所述的专用接头包括附图2所示的8个接头结构。
所述的试剂盒还包含一组文库扩增复合引物,所述的文库扩增复合引物的序列如下表3。
表3 文库扩增复合引物表
所述的文库扩增复合引物与通用引物2同时使用,所述的通用引物2的序列为SEQ IDNO:47。
本发明提供的试剂盒中,上述(1)检测多种突变、融合、缺失等变异类型的特异性复合引物、(2)DNA文库构建的专用接头、(3)文库扩增复合引物试剂独立包装。
所述的试剂盒中还包括以下任意一个或多个试剂组:逆转录反应试剂组;DNA片段化/末端修复/dA添加试剂组;DNA连接试剂组;PCR扩增试剂组;高保真热启动PCR扩增试剂组。
所述的逆转录反应试剂组包括随机引物、第一链合成缓冲液、第一链合成酶混合液、第二链合成缓冲液、第二链合成酶混合液和超纯水。
所述的DNA片段化/末端修复/dA添加试剂组包括10×DNA片段化缓冲液、5×片段化酶混合液和超纯水。
所述的DNA连接试剂组包括5×连接酶缓冲液、TIANSeq DNA连接酶、专用接头和超纯水。
所述的PCR扩增试剂组包括5×多重PCR反应酶混合液、PCR引物。
所述的高保真热启动PCR扩增试剂组包括高保真热启动酶反应液、PCR引物和超纯水。
又一方面,本发明提供了前述试剂盒的使用方法。
所述的检测方法包括以下步骤:
(1)使用DNA文库构建的专用接头连接检测样本DNA;
(2)使用检测多种突变、融合、缺失等变异类型的特异性复合引物对步骤(1)的连接产物进行复合PCR扩增;
(3)使用文库扩增复合引物对步骤(2)获得的扩增产物进行PCR扩增,获得测序文库;
(4)对步骤(3)获得的测序文库进行测序。
作为一些优选的实施方案,所述的检测方法包括以下步骤:
S1.提取样本中的DNA和RNA;
S2.将步骤S1所得的RNA进行逆转录制备cDNA;
S3.将步骤S2所得的cDNA与步骤S1所得的DNA混合后,进行核酸片段化、末端修复及加A,得cDNA与DNA混合液;
S4.将步骤S3所得的cDNA与DNA混合液进行接头连接并纯化,得纯化后的接头连接产物;
S5.将步骤S4所得的纯化后的接头连接产物进行第一步特异性PCR并纯化,得第一步PCR纯化产物;
S6.将步骤S5所得的第一步PCR纯化产物进行第二步通用PCR并纯化,得测序文库;
S7.上机测序;
S8.生物信息学分析软件对进行数据结果分析。
在一些实施例中,所述的试剂盒的检测流程图如图7所示。
又一方面,本发明还提供了所述用于DNA文库构建的专用接头、所述用于检测基因突变、融合、缺失等变异类型的特异性复合引物、所述用于文库扩增的复合引物在制备联合检测慢性粒细胞白血病的试剂盒中的应用。
本发明的有益效果:
1、就本发明的整体构思而言,本发明实现了基因重排、基因融合、基因突变等变异类型的特异性复合引物的同时检测,且提高了检测的灵敏度。
2、就本发明针对文库构建的设计构思而言:
传统扩增子文库由于使用双引物扩增,生成的文库双端位置固定,导致某个位置的测序序列无法有效去除扩增导致的序列重复,从而无法识别降低扩增过程中引入的随机突变,导致灵敏度降低。如图3所示。
本发明采用随机打断后,在DNA两端连接通用引物,一端采用特异性引物扩增,一端采用通用引物,保留其中一段的随机性,从而可以有效识别重复测序序列。如图4所示。
3、与传统扩增子文库构建检测融合基因比较
融合基因是指将两个或多个基因的编码区首尾相连,置于同一套调控序列(包括启动子、增强子、核糖体结合序列、终止子等)控制之下,构成的嵌合基因。例如BCR/ABL 融合,其中ABL为核心基因,BCR为伴侣基因,融合基因通常有多个伴侣基因,并且融合断点往往不一样。
因此传统扩增子文库构建需要同时知晓伴侣基因和核心基因两个基因,并且断点位置也已知的情况下,进行引物设计检测。容易遗漏断点未知,或者伴侣基因未知的融合类型。
本发明将单引物设置在核心基因上,不必事先知道断点或伴侣基因,在引物扩增过程中,都能够将序列包含在文库中,通过测序识别伴侣基因及断点位置。如图5所示。
4、与利用arms_PCR法检测上述突变比较
利用arms_PCR法突变操作复杂,每管反应只能检测一个至三个位点,本发明试剂盒通过加入多个位点的检测引物,并进行高通量测序可同时检测上述所有位点。
arms_PCR法无法利用mRNA检测,本发明试剂盒可以检测。
5、与利用RT-PCR法比较
RT-PCR法检测效率较低,一次只能检测一种类型的突变,本发明试剂盒通过同时加入多组引物,可实现多种突变同时检测。且RT-PCR只能检测已知种类,限定特定断点的融合基因类型,本发明则克服了这一缺陷。
6、与利用Sanger测序法检测比较
Sanger测序法检测突变位点敏感性及灵敏度较低,只能达到10%,本发明试剂盒利用高通量测序法,每次测序每个突变并行测序10000次以上,灵敏度可达到0.1%。另外,Sanger测序法操作复杂,每管反应只能检测一个位点,本发明试剂盒通过加入多个位点的检测引物,并进行高通量测序,可同时检测上述所有位点。
7、与液相杂交捕获文库构建比较
传统液相杂交捕获文库流程片长,涉及约24个步骤,耗时20-24h,操作过程复杂,本试剂盒发明精简操作流程至11个步骤和耗时缩短至6-8h。如图6所示。
8、本发明整体技术方案利用高通量测序法,每次测序每个突变并行测序10000次以上,结合整体构思、文库构建的设计构思,以及“(1)DNA文库构建的专用接头;(2)检测融合基因和点突变的特异性复合引物;(3)文库扩增复合引物”的设计构思,一方面实现了同时检测上述所有位点(融合基因和多个突变位点);另一方面从多方面多角度提升检测灵敏度,灵敏度可达到0.1%。而常规检测方法(例如利用Sanger测序)敏感性较低,灵敏度较低,只能达到10%。
附图说明
图1为专用接头结构。
图2为专用接头的8个接头结构。
图3为构建传统扩增子文库检测基因位点突变。
图4为构建本发明单引物文库检测基因位点突变。
图5为构建传统扩增子文库与本发明单引物文库检测融合基因的比较。
图6为构建液相杂交捕获文库与本发明单引物文库的比较。
图7为本发明试剂盒使用方法的实验流程图。
图8为本发明单引物扩增文库构建流程图。
图9为BCR-ABL1阳性检测图。
图10为BCR-ABL1阴性检测图。
图11为ZMYM2-FGFR1阳性检测图。
图12为ZMYM2-FGFR1阴性检测图。
图13为JAK c.1849G>T p.V617F阳性检测图。
图14为JAK c.1849G>T p.V617F阴性检测图。
图15为PCM1-JAK2阳性检测图。
图16为PCM1-JAK2阴性检测图。
图17为NUP98-HOXA9阳性检测图。
图18为NUP98-HOXA9阴性检测图。
图19为FIP1L1-PDGFRA阳性检测图。
图20为FIP1L1-PDGFRA阴性检测图。
图21为ETV6-PDGFRB阳性检测图。
图22为ETV6-PDGFRB阴性检测图。
具体实施方式
下面结合具体实施例,对本发明作进一步详细的阐述,下述实施例不用于限制本发明,仅用于说明本发明。以下实施例中所使用的实验方法如无特殊说明,实施例中未注明具体条件的实验方法,通常按照常规条件,下述实施例中所使用的材料、试剂等,如无特殊说明,均可从商业途径得到。
实施例1 一种联合检测慢性粒细胞白血病的试剂盒
该试剂盒中包括:
(1)DNA文库构建的专用接头:专用接头为接头引物1分别和8条不同的i5端引物构成的8个二聚体。
所述的接头引物1和8条不同的i5端引物的序列如下表4。
表4专用接头引物表
表4续
专用接头具有如图1所示的结构。其中,所述的5_index的序列为:CTCTCTAT、TATCCTCT、GTAAGGAG、ACTGCATA、AAGGAGTA、CTAAGCCT、CGTCTAAT或TCTCTCCG。
因此,专用接头包括如图2所示的8个接头结构。
(2)检测基因突变、融合、缺失等变异类型的特异性复合引物和通用引物1,特异性复合引物的序列及其对应的检测位点如下表5。
表5特异性复合引物表
表5续
表5续
特异性引物与通用引物1同时使用。
(3)文库扩增复合引物的序列如下表6。
表6文库扩增复合引物表
文库扩增复合引物与通用引物2同时使用。
(4)逆转录反应试剂组;
(5)DNA片段化/末端修复/dA添加试剂组;
(6)DNA连接试剂组;
(7)PCR扩增试剂组;
(8)高保真热启动PCR扩增试剂组。
其中:
逆转录反应试剂组包括随机引物、第一链合成缓冲液、第一链合成酶混合液、第二链合成缓冲液、第二链合成酶混合液和超纯水;
DNA片段化/末端修复/dA添加试剂组包括10×DNA片段化缓冲液、5×片段化酶混合液和超纯水;
DNA连接试剂组包括5×连接酶缓冲液、TIANSeq DNA连接酶、专用接头和超纯水;
PCR扩增试剂组包括5×多重PCR反应酶混合液、PCR引物;
高保真热启动PCR扩增试剂组包括高保真热启动酶反应液、PCR引物和超纯水。
实施例2
本实施例的试剂盒为实施例1提供的试剂盒,图7为检测方法的实验流程图,图8为单引物扩增文库构建流程图。试剂盒的检测方法包括如下具体步骤:
一、cDNA逆转录
1、第一链cDNA合成
1.1试剂准备:将提取好的样本RNA冰上缓慢解冻后移液器混匀,将随机引物第一链合成酶混合液、第一合成缓冲液(购自天根科技生化科技有限公司,产品类型为TIANSeq cDNA合成模块,目录号为NG308-T1)从-20℃取出,解冻后轻弹混匀。
1.2在PCR管中建立如下反应体系,并用移液器轻轻吹打充分混匀:
RNA XμL(1000ng);
随机引物 1μL;
超纯水 (10-X)μL;
总体积 11μL。
1.3 65℃孵育5分钟,然后置于冰上2分钟。
1.4步骤1.3结束后,在原管中继续加入下表所列的反转录组分,配制如下反应体系:
步骤1.3结束反应液 11μL;
第一链合成缓冲液 7μL;
第一链合成酶混合液 2μL;
总体积 20μL。
1.5用移液器轻轻吹打充分混匀,进行第一链cDNA合成反应,反应程序如下:25℃10min;42℃15min;70℃15min;4℃hold。PCR仪热盖温度设置为105℃。
注意:反应结束后立即进行cDNA第二链的合成反应。
2、第二链cDNA合成
2.1将第二链合成酶缓冲液和第二链合成酶混合液(购自天根科技生化科技有限公司,产品类型为TIANSeq cDNA 合成模块,目录号为NG308-T1)从-20°C取出,轻弹混匀,在PCR管中建立如下反应体系,并用移液器轻轻吹打充分混匀:
合成的第一链cDNA 20μL;
第二链合成酶缓冲液 8.5μL;
第二链合成酶混合液 3.5μL;
超纯水 48μL;
总体积 80μL。
2.2在PCR仪中进行第二链cDNA合成反应,反应程序如下:16℃60min;4℃hold。PCR仪热盖温度设定为≤40℃。
注意:反应结束后,cDNA第二链的合成产物可在4°C暂存1小时,但是建议反应结束后立即进行下步纯化步骤。
2.3 1.8×纯化磁珠(购自天根科技生化科技有限公司,产品类型为TIANSeq DNA片段分选磁珠,目录号为NG306)纯化cDNA第二链的合成产物,37μL纯水洗脱,cDNA定量。
二、cDNA与样品提取的DNA混合
取50ngcDNA和同一份样本中提取的50 ngDNA等量混合,得到cDNA和DNA混合液100ng。
三、文库构建:核酸片段化、末端修复及加A
1、取10×DNA片段化缓冲液、5×片段化酶混合液(购自天根科技生化科技有限公司,产品类型为TIANSeq快速DNA片段化/末端修复/dA添加模块,目录号为NG301)配制如下反应体系(DNA上样量>10ng),冰上操作,各组分加入后,请轻柔吸打混匀,注意不要涡旋:
10×DNA片段化缓冲液 5μL;
5×片段化酶混合液 10μL;
cDNA和DNA混合液 XμL(100ng);
超纯水 (35-X)μL;
总体积 50μL。
2、在PCR仪中进行如下反应程序:4℃ 1min;32℃7min;65℃30min;4℃ hold。PCR仪热盖温度设置为70℃。
其中,32℃片段化时间选择参考如下:
DNA主峰为250bp时,10ng DNA上样量32℃片段化时间为24min,100ng DNA上样量32℃片段化时间为16min,1000ng DNA上样量32℃片段化时间为14min。
DNA主峰为350bp时,10ng DNA上样量32℃片段化时间为16min,100ng DNA上样量32℃片段化时间为10min,1000ng DNA上样量32℃片段化时间为8min。
DNA主峰为450bp时,10ng DNA上样量32℃片段化时间为14min,100ng DNA上样量32℃片段化时间为8min,1000ng DNA上样量32℃片段化时间为6min。
DNA主峰为550bp时,10ng DNA上样量32℃片段化时间为10min,100ng DNA上样量32℃片段化时间为6min,1000ng DNA上样量32℃片段化时间为4min。
3、反应结束后,立即进入接头连接步骤。
四、文库构建:接头连接
1、取5×Ligase Buffer、TIANSeq DNA Ligase(购自天根科技生化科技有限公司,产品类型为TIANSeq快速连接模块,目录号为NG303)在步骤三反应结束管中配制如下反应体系:
步骤三反应产物 50μL;
5×Ligase Buffer 20μL;
TIAN Seq DNA Ligase 10μL;
专用接头(15μM) 2.5μL;
超纯水 17.5μL;
总体积 100μL。
其中,专用接头包括以下8个接头结构:
接头1:
接头2:
接头3:
接头4:
接头5:
接头6:
接头7:
接头8:
2、移液器吸打混匀,放入PCR仪,进行如下反应程序:20℃ 15min;4℃ hold。无热盖。
3、1.6×磁珠(购自天根科技生化科技有限公司,产品类型为TIANSeq DNA片段分选磁珠,目录号为NG306)纯化接头连接产物,16μL超纯水洗脱,取15μL上清液到新的PCR管中,进行第一步PCR扩增反应。
五、第一步多重PCR扩增
1、取5×多重PCR反应酶混合液(购自Takara,产品类型为Multiplex PCR Assay KitVer.2,货号为RR062A)配制如下PCR反应体系:
5×多重PCR反应酶混合液 4μL;
特异性引物混合物 0.56μL;
通用引物1(现取现用) 4.7μL;
步骤四纯化后的接头连接产物 10.74μL;
总体积 20μL。
其中,特异性引物混合物配制方法:先将各引物稀释至100μM,然后按照下表7所示配制混合物体系。
表7 特异性引物混合物体系
表7续
2、反应混合液配好后,移液器吹打10次混匀,放入PCR仪,运行以下反应程序:99℃2min;99℃ 15s,69℃ 4min,18个循环;72℃ 10min;4℃ hold。PCR仪热盖温度设置为105℃。
3、PCR结束后,电泳。1.6×磁珠(购自天根科技生化科技有限公司,产品类型为TIANSeq DNA片段分选磁珠,目录号为NG306)纯化,使用15μL纯水洗脱,得到第一步PCR扩增产物,Qubit定量。
六、第二步PCR扩增
1、取高保真热启动酶反应液(购自天根科技生化科技有限公司,产品类型为高保真PCR反应预混液,目录号为NG219),及第一步PCR纯化产物进行第二轮PCR扩增,扩增体系如下:
高保真热启动酶反应液 25μL;
i7-primer(10μM) 1.5μL;
通用引物2(10μM) 1.5μL;
第一步PCR纯化产物 10μL;
超纯水 12μL;
总体积 50μL。
2、第二步PCR扩增反应混合液配好后,移液器吹打10次混匀,放入PCR仪,运行以下反应程序95℃ 3min;98℃ 20s,58℃ 15s,72℃ 15s,8个循环;72℃ 1min;4℃ hold。PCR仪热盖温度设置为105℃。
3、PCR反应结束后,Qubit定量,电泳。
4、1.3×磁珠(购自天根科技生化科技有限公司,产品类型为TIANSeq DNA片段分选磁珠,目录号为NG306)纯化,10μL超纯水洗脱,Qubit定量。
七、文库稀释到2-3ng/μL,Agilent 4150 TapeStation系统进行检测。
八、测序上机。
九、测序数据反馈。
试验例1
采用本发明实施例2记载的方法针对样本进行检测。
样本包括慢性粒细胞白血病患者和健康人,均已获得许可。
1.样本1:来源为云南省肿瘤医院编号为198272_KTD072的临床样品。
检测结果如图9所示,该样本为BCR-ABL1阳性。
2.样本2:来源为云南省肿瘤医院编号为196547_KTD031的临床样品。
检测结果如图10所示,该样本为BCR-ABL1阴性。
3.样本3:来源为云南省肿瘤医院编号为198276_KTD069的临床样品。
检测结果如图11所示,该样本为ZMYM2-FGFR1阳性。
4.样本4:来源为云南省肿瘤医院编号为191561_NW685的临床样品。
检测结果如图12所示,该样本为ZMYM2-FGFR1阴性。
5.样本5:来源为云南省肿瘤医院编号为191463_NW636的临床样品。
检测结果如图13所示,该样本为JAK c.1849G>T p.V617F阳性。
6.样本6:来源为云南省肿瘤医院编号为198273_KTD071的临床样品。
检测结果如图14所示,该样本为JAK c.1849G>T p.V617F阴性。
7.样本7:来源为云南省肿瘤医院编号为191242_NW608的临床样品。
检测结果如图15所示,该样本为PCM1-JAK2阳性。
8.样本8:来源为云南省肿瘤医院编号为198277_KTD073的临床样品。
检测结果如图16所示,该样本为PCM1-JAK2阴性。
9.样本9:来源为云南省肿瘤医院编号为197548_KTD094的临床样品。
检测结果如图17所示,该样本为NUP98-HOXA9阳性。
10.样本10:来源为云南省肿瘤医院编号为197550_KTD095的临床样品。
检测结果如图18所示,该样本为NUP98-HOXA9阴性。
11.样本11:来源为云南省肿瘤医院编号为200888_KTD029的临床样品。
检测结果如图19所示,该样本为FIP1L1-PDGFRA阳性。
12.样本12:来源为云南省肿瘤医院编号为297013_KTD091的临床样品。
检测结果如图20所示,该样本为FIP1L1-PDGFRA阴性。
13.样本13:来源为云南省肿瘤医院编号为197527_KTD054的临床样品。
检测结果如图21所示,该样本为ETV6-PDGFRB阳性。
14.样本14:来源为云南省肿瘤医院编号为197884_KTD117的临床样品。
检测结果如图22所示,该样本为ETV6-PDGFRB阴性。
试验例2 一种联合检测慢性粒细胞白血病的试剂盒的准确度检测
对试验例1提供的阳性样本分别采用Sanger测序法进行验证,与本发明提供的方法检测结果一致,表明本发明提供的试剂盒具有较好的准确度。
试验例3一种联合检测慢性粒细胞白血病的试剂盒的稳定性检测
对实施例1提供的试剂盒进行稳定性检测,方法如下:
将试剂盒分别置于4℃、20℃条件下7天、14天、21天,然后采用实施例2提供的检测方法对试验例1的样本进行检测,结果如下表8。
表8稳定性检测结果
表8续
其中,Y表示检测结果准确,N表示检测结果不准确。根据以上数据可知,本申请提供的试剂盒的稳定性较好,能够至少在4℃条件下保存21天,20℃条件下保存14天。
以上所述实施例仅表达了本发明的几种实施方式,其描述较为具体和详细,但并不能因此而理解为对本发明专利范围的限制。应当指出的是,对于本领域的普通技术人员来说,在不脱离本发明构思的前提下,还可以做出若干变形和改进,这些都属于本发明的保护范围。因此,本发明专利的保护范围应以所附权利要求为准。
序列表
<110> 苏州科贝生物技术有限公司
<120> 一种联合检测慢性粒细胞白血病检测试剂盒
<130> 20201012
<160> 47
<170> SIPOSequenceListing 1.0
<210> 1
<211> 22
<212> DNA
<213> 人工序列(artificial sequence)
<400> 1
gatcggaaga gccacatact ga 22
<210> 2
<211> 70
<212> DNA
<213> 人工序列(artificial sequence)
<400> 2
aatgatacgg cgaccaccga gatctacacc tctctataca ctctttccct acacgacgct 60
cttccgatct 70
<210> 3
<211> 70
<212> DNA
<213> 人工序列(artificial sequence)
<400> 3
aatgatacgg cgaccaccga gatctacact atcctctaca ctctttccct acacgacgct 60
cttccgatct 70
<210> 4
<211> 70
<212> DNA
<213> 人工序列(artificial sequence)
<400> 4
aatgatacgg cgaccaccga gatctacacg taaggagaca ctctttccct acacgacgct 60
cttccgatct 70
<210> 5
<211> 70
<212> DNA
<213> 人工序列(artificial sequence)
<400> 5
aatgatacgg cgaccaccga gatctacaca ctgcataaca ctctttccct acacgacgct 60
cttccgatct 70
<210> 6
<211> 70
<212> DNA
<213> 人工序列(artificial sequence)
<400> 6
aatgatacgg cgaccaccga gatctacaca aggagtaaca ctctttccct acacgacgct 60
cttccgatct 70
<210> 7
<211> 70
<212> DNA
<213> 人工序列(artificial sequence)
<400> 7
aatgatacgg cgaccaccga gatctacacc taagcctaca ctctttccct acacgacgct 60
cttccgatct 70
<210> 8
<211> 70
<212> DNA
<213> 人工序列(artificial sequence)
<400> 8
aatgatacgg cgaccaccga gatctacacc gtctaataca ctctttccct acacgacgct 60
cttccgatct 70
<210> 9
<211> 70
<212> DNA
<213> 人工序列(artificial sequence)
<400> 9
aatgatacgg cgaccaccga gatctacact ctctccgaca ctctttccct acacgacgct 60
cttccgatct 70
<210> 10
<211> 60
<212> DNA
<213> 人工序列(artificial sequence)
<400> 10
gtgactggag ttcagacgtg tgctcttccg atcccttctt ggatttgcag cccaccagct 60
<210> 11
<211> 61
<212> DNA
<213> 人工序列(artificial sequence)
<400> 11
gtgactggag ttcagacgtg tgctcttccg atcggttttc cttggagttc caacgagcgg 60
c 61
<210> 12
<211> 59
<212> DNA
<213> 人工序列(artificial sequence)
<400> 12
gtgactggag ttcagacgtg tgctcttccg atctcaaagt cagatgctac tggccgctg 59
<210> 13
<211> 62
<212> DNA
<213> 人工序列(artificial sequence)
<400> 13
gtgactggag ttcagacgtg tgctcttccg atcggtttgg gcttcacacc attccccatt 60
gt 62
<210> 14
<211> 62
<212> DNA
<213> 人工序列(artificial sequence)
<400> 14
gtgactggag ttcagacgtg tgctcttccg atccgtcggc caccgttgaa tgatgatgaa 60
cc 62
<210> 15
<211> 62
<212> DNA
<213> 人工序列(artificial sequence)
<400> 15
gtgactggag ttcagacgtg tgctcttccg atcgctgcac caggttaggg tgtttgatct 60
ct 62
<210> 16
<211> 61
<212> DNA
<213> 人工序列(artificial sequence)
<400> 16
gtgactggag ttcagacgtg tgctcttccg atcccgaggc atggagtatc tggcctccaa 60
g 61
<210> 17
<211> 61
<212> DNA
<213> 人工序列(artificial sequence)
<400> 17
gtgactggag ttcagacgtg tgctcttccg atccatggac aagcccagta actgcaccaa 60
c 61
<210> 18
<211> 64
<212> DNA
<213> 人工序列(artificial sequence)
<400> 18
gtgactggag ttcagacgtg tgctcttccg atcgagatga ggaaggcccc tgtgcaatag 60
atga 64
<210> 19
<211> 64
<212> DNA
<213> 人工序列(artificial sequence)
<400> 19
gtgactggag ttcagacgtg tgctcttccg atcccagagt tcatggatgc actggagtca 60
gcag 64
<210> 20
<211> 60
<212> DNA
<213> 人工序列(artificial sequence)
<400> 20
gtgactggag ttcagacgtg tgctcttccg atccagccct ctcccagggg tttgcctaag 60
<210> 21
<211> 65
<212> DNA
<213> 人工序列(artificial sequence)
<400> 21
gtgactggag ttcagacgtg tgctcttccg atcggactgc atcgaagtac atacagtcca 60
gtctg 65
<210> 22
<211> 66
<212> DNA
<213> 人工序列(artificial sequence)
<400> 22
gtgactggag ttcagacgtg tgctcttccg atcgtcccac tgaggttgta ctcttcattc 60
tcattt 66
<210> 23
<211> 67
<212> DNA
<213> 人工序列(artificial sequence)
<400> 23
gtgactggag ttcagacgtg tgctcttccg atcgaaacac cattcgttct gaagactaga 60
aggtttg 67
<210> 24
<211> 67
<212> DNA
<213> 人工序列(artificial sequence)
<400> 24
gtgactggag ttcagacgtg tgctcttccg atcgttgacc gtagtctcct acttctcttc 60
gtacgcc 67
<210> 25
<211> 66
<212> DNA
<213> 人工序列(artificial sequence)
<400> 25
gtgactggag ttcagacgtg tgctcttccg atcgaaagct tgctcatcat acttgctgct 60
tcaaag 66
<210> 26
<211> 61
<212> DNA
<213> 人工序列(artificial sequence)
<400> 26
gtgactggag ttcagacgtg tgctcttccg atcatcacaa tcaccaacag caccaggact 60
g 61
<210> 27
<211> 62
<212> DNA
<213> 人工序列(artificial sequence)
<400> 27
gtgactggag ttcagacgtg tgctcttccg atcagggctg attgattcaa tgaccctcca 60
gc 62
<210> 28
<211> 59
<212> DNA
<213> 人工序列(artificial sequence)
<400> 28
gtgactggag ttcagacgtg tgctcttccg atcttcaacc accttcccaa acgctccag 59
<210> 29
<211> 60
<212> DNA
<213> 人工序列(artificial sequence)
<400> 29
gtgactggag ttcagacgtg tgctcttccg atccgttagt ctccagctgg ctctcctctt 60
<210> 30
<211> 60
<212> DNA
<213> 人工序列(artificial sequence)
<400> 30
gtgactggag ttcagacgtg tgctcttccg atccaggatg gctgagatca ccaccacctt 60
<210> 31
<211> 60
<212> DNA
<213> 人工序列(artificial sequence)
<400> 31
gtgactggag ttcagacgtg tgctcttccg atccaccttc catcggatct cgtaacgtgg 60
<210> 32
<211> 60
<212> DNA
<213> 人工序列(artificial sequence)
<400> 32
gtgactggag ttcagacgtg tgctcttccg atccgacata agggcttgct tctcactgct 60
<210> 33
<211> 60
<212> DNA
<213> 人工序列(artificial sequence)
<400> 33
gtgactggag ttcagacgtg tgctcttccg atcctctcac ttagctccag cactcggaca 60
<210> 34
<211> 58
<212> DNA
<213> 人工序列(artificial sequence)
<400> 34
gtgactggag ttcagacgtg tgctcttccg atccatcgtg gcctgagaat ggctcagg 58
<210> 35
<211> 62
<212> DNA
<213> 人工序列(artificial sequence)
<400> 35
gtgactggag ttcagacgtg tgctcttccg atctgttccc aaacaaagat gcctgtccag 60
ca 62
<210> 36
<211> 61
<212> DNA
<213> 人工序列(artificial sequence)
<400> 36
gtgactggag ttcagacgtg tgctcttccg atctgctgga gaacagcctg ctgggcagca 60
g 61
<210> 37
<211> 62
<212> DNA
<213> 人工序列(artificial sequence)
<400> 37
gtgactggag ttcagacgtg tgctcttccg atcgaagcag caagtatgat gagcaagctt 60
tc 62
<210> 38
<211> 23
<212> DNA
<213> 人工序列(artificial sequence)
<400> 38
aatgatacgg cgaccaccga gat 23
<210> 39
<211> 53
<212> DNA
<213> 人工序列(artificial sequence)
<400> 39
caagcagaag acggcatacg agattcacaa gcgtgactgg agttcagacg tgt 53
<210> 40
<211> 53
<212> DNA
<213> 人工序列(artificial sequence)
<400> 40
caagcagaag acggcatacg agatactaca cggtgactgg agttcagacg tgt 53
<210> 41
<211> 53
<212> DNA
<213> 人工序列(artificial sequence)
<400> 41
caagcagaag acggcatacg agatatcgta cggtgactgg agttcagacg tgt 53
<210> 42
<211> 53
<212> DNA
<213> 人工序列(artificial sequence)
<400> 42
caagcagaag acggcatacg agatagacac aggtgactgg agttcagacg tgt 53
<210> 43
<211> 53
<212> DNA
<213> 人工序列(artificial sequence)
<400> 43
caagcagaag acggcatacg agatttgtcc tggtgactgg agttcagacg tgt 53
<210> 44
<211> 53
<212> DNA
<213> 人工序列(artificial sequence)
<400> 44
caagcagaag acggcatacg agattgtgag aggtgactgg agttcagacg tgt 53
<210> 45
<211> 53
<212> DNA
<213> 人工序列(artificial sequence)
<400> 45
caagcagaag acggcatacg agataaggtt gggtgactgg agttcagacg tgt 53
<210> 46
<211> 53
<212> DNA
<213> 人工序列(artificial sequence)
<400> 46
caagcagaag acggcatacg agatattagc cagtgactgg agttcagacg tgt 53
<210> 47
<211> 19
<212> DNA
<213> 人工序列(artificial sequence)
<400> 47
aatgatacgg cgaccaccg 19
