一种烟草支链淀粉酶基因及其应用
文献发布时间:2023-06-19 12:18:04
技术领域
本发明属于烟草基因工程技术领域,具体涉及一种烟草支链淀粉酶基因及其应用。
背景技术
烟草作为一种重要的经济作物,其品质和安全性一直是研究人员关注的重点。淀粉是烟草生长过程中积累的重要碳水化合物,广泛存在于烟草的茎叶中。成熟的鲜烟叶中淀粉含量高达40%左右,经调制后,大部分淀粉降解为还原糖,但仍有部分留在烟叶中。烟叶中淀粉含量的高低影响着烟叶的外观和内在品质以及卷烟的香吃味。一方面,以淀粉形态存在的糖类在烟支燃吸时会对烟气质量产生不良影响,影响烟丝的燃烧速度及其完全性,也影响阴然持火力;另一方面,淀粉在燃烧时会产生焦糊气味,破坏烟草燃吸时所形成的香味,影响卷烟香气质及吸味口感,同时使安全性下降。近年来,我国烤烟水平不断提高,但与进口烟相比在其内在的化学组成协调性等方面还存在着较大差异。目前,我国烤烟中淀粉含量(质量分数)约为4%~6%,而国外优质烤烟淀粉含量仅为1%~2%。
因此,烟草中影响淀粉含量的基因功能的研究将为烟叶品质改善、烟草品种遗传改良提供理论支持,对提高我国烟草产品品质具有重要的意义。
发明内容
本发明的目的是提供一种烟草支链淀粉酶基因及其应用,以解决现烤烟中淀粉含量过高的问题,从而为烟叶品质调控、烟草新品种培育奠定一定的基础。
为实现上述目的,本发明是通过以下技术方案实现的:
一种烟草支链淀粉酶基因,碱基序列具体如SEQ ID NO.1所示,含有2679个碱基,命名为NtGlgX。
进一步的,烟草支链淀粉酶基因的氨基酸序列如SEQ ID NO.2所示,由952个氨基酸残基组成。
进一步的,烟草支链淀粉酶基因的PCR扩增制备方法,包括如下步骤:
(1)提取基因组,并反转录为cDNA备用;
(2)设计PCR扩增用引物,并进行PCR扩增,具体引物序列设计如下:
NtGlgX-F:5’-CGCAGCATCAGGGAAAGTC-3’,
NtGlgX-R:5’-AGAAGTATCTAATGTTTTGGACTCC-3’。
进一步的,在步骤(1)中提取基因组时,以烟草品种红花大金元叶片为样品。
上述任一项的烟草支链淀粉酶基因的应用,该基因表达的蛋白与植物叶片中淀粉含量相关,降低该蛋白表达后,叶片中淀粉含量明显降低。
进一步的,利用基因沉默技术、或者基因超表达方法,通过调节烟草支链淀粉酶基因的表达量,来调节控制烟叶中淀粉含量。
进一步的,通过转基因技术、瞬时表达技术或基因组编辑技术,构建含有烟草支链淀粉酶基因的病毒诱导沉默载体、RNAi干涉载体、超表达载体,转化烟草,筛选获得淀粉含量变化的烟草新品种。
具体例如:利用病毒诱导的基因沉默(VIGS)的技术,干扰烟草支链淀粉酶基因的表达使其沉默,烟草支链淀粉酶基因沉默植株中淀粉含量显著下降,进而获得淀粉含量下降的植物新品种。
本发明的有益效果是:
本申请中,通过对特定烟草支链淀粉酶基因NtGlgX的初步研究,发现其与烟草淀粉含量高度相关,在将该基因沉默后,烟草中淀粉含量发生了明显降低。基于这一特性,可为烟叶品质调控、烟草新品种培育提供一定的应用基础和参考借鉴。
附图说明
图1为与对照植株相比,NtGlgX基因沉默植株中该基因的相对表达量;
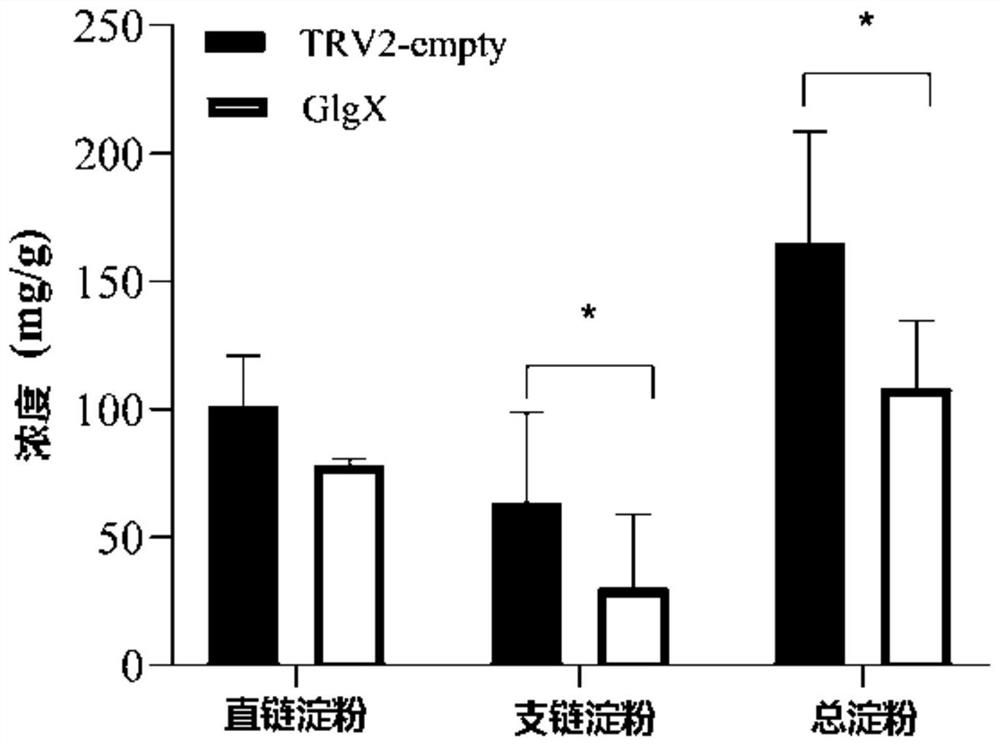
图2为病毒诱导基因沉默的烟叶及对照烟叶中的淀粉含量比较。
具体实施方式
以下通过实施例来详细说明本发明的技术方案,以下的实施例仅是示例性的,仅能用来解释和说明本发明的技术方案,而不能解释为是对本发明技术方案的限制。
生物材料:
本氏烟草,一种现常用烟草材料,育苗钵中育苗,待发芽后两周进行分苗,种于塑料钵(10cm×10cm)中,22℃、16h光/8h暗条件下进行日常肥水管理等栽培管理。
下述实施例中所采用的VIGS载体是一种来自烟草脆裂病毒的病毒载体(tobaccorattle virus,TRV),所具体利用的TRV2(一种常用载体)带有卡那霉素筛选标记和35S启动子,同时TRV2带有EcoR I和BamH I等多克隆位点,可以用来携带和转化外源基因。
实验试剂:
LB液体培养基,1L含量中含有:10g细菌蛋白胨(bacteriological peptone);10g氯化钠(NaCl);5g酵母抽提物(yeast extract),高温高压灭菌。
YEB液体培养基,1L含量中含有:5g牛肉浸膏(beef extract);5g细菌蛋白胨(bacteriological peptone);5g蔗糖(sucrose);1g酵母抽提物(yeast extract);2mL 1M硫酸镁(MgSO4),高温高压灭菌。
1M 2-(N-吗啉)乙磺酸(MES)储备液:ddH
200mM乙酰丁香酮(Acetosyringone,As)储备液:二甲基亚砜(DSMO)溶解,-20℃储存备用。
实施例1
本实施例就烟草NtGlgX基因克隆及沉默载体的构建过程简要介绍如下。
(1)烟草NtGlgX基因克隆
根据前期对于烟草基因组及相关NtGlgX基因研究,选择特异编码序列为目标片段,设计PCR扩增用引物序列如下:
NtGlgX-F:5’-CGCAGCATCAGGGAAAGTC-3’,
NtGlgX-R:5’-AGAAGTATCTAATGTTTTGGACTCC-3’。
以烟草红花大金元叶片(先提取基因组,再反转录为cDNA)的cDNA为模板,进行PCR扩增获得NtGlgX基因。
PCR扩增程序为:95℃预变性3min;95℃变性15s,53℃退火15s,72℃延伸3min,34个循环后,72℃彻底延伸5min。
对PCR扩增产物进行琼脂糖凝胶电泳检测,并回收电泳产物备用。
(2)构建重组TRV2-NtGlgX载体
将步骤(1)中的PCR扩增产物进行EcoRI、BamHI双酶切,同时对空载体TRV2进行EcoRI、BamHI双酶切,分别回收酶切产物,利用T4 DNA连接酶进行连接。
将连接产物转化大肠杆菌感受态DH5α,转化操作结束后将转化产物涂布在含50mg/L Kan的LB固体培养基上,37℃过培养夜。
挑选阳性单菌落扩增后进一步进行PCR鉴定,并结合测序验证,确保获得构建正确的重组载体TRV2-NtGlgX。
需要说明的是,烟草NtGlgX基因,包括2679个碱基,碱基序列如SEQ ID NO.1所示。
烟草支链淀粉酶蛋白NtGlgX,包括952个氨基酸,氨基酸序列如SEQ ID NO.2所示。
实施例2
在实施例1基础上,利用农杆菌介导的VIGS技术,进一步将所构建的重组TRV2-NtGlgX载体转化了烟草植株,并就相关植物表型变化情况做了验证分析,具体实验过程简介如下。
(1)转化农杆菌
需要说明的是,参考实施例1操作及现有技术,同时制备了TRV2-GFP重组载体作为对照,具体转化过程为:
将TRV2-GFP(载体对照)及TRV2-NtGlgX的阳性克隆质粒,分别通过电击转化方式转化进入农杆菌GV3101感受态细胞中,利用含50mg/L Kan和50mg/L Rif的YEB平板进行培养筛选,在28℃倒置培养2d后,利用菌落PCR筛选带有目的基因的农杆菌。
(2)制备转染用菌液
将步骤(1)中筛选所得阳性农杆菌克隆在5mL的YEB液体培养基(含50mg/L Kan和50mg/L Rif)中,28℃、250rpm条件下培养过夜。
取50uL过夜培养物接种至50mL的YEB液体培养基(含50mg/L Kan)中,培养至OD
最后室温放置3h左右后,作为转染用菌液。
(3)瞬时转化
以3-4w(周)苗龄的本氏烟草叶片为实验材料,利用1mL规格注射器,将步骤(2)中所制备转染用菌液注射至烟草叶片中,注射后的烟草继续在人工培养箱内培养,观察表型变化。
进一步通过qRT-PCR对NtGlgX基因表达情况进行了检测,结果如图1所示,可以看出,TRV2-NtGlgX的侵染植株中,NtGlgX的表达量显著降低,qRT-PCR引物如下:
NtGlgX-F:5’-CCTTTAACTCTGCGGTGCTC-3’,
NtGlgX-R:5’-CTCCAGAACCCACATTCCAT-3’。
进一步地,对实验组(TRV2-NtGlgX浸染植株)和对照组(TRV2-GFP浸染植株)中的叶片淀粉含量情况进行了检测(检测方法参照:格锐思生物的支链-直链-总淀粉含量试剂盒(分光光度法)),结果如图2所示。
从图2结果可以看出,实验组中支链淀粉和总淀粉含量与对照组相比均有显著下降,支链淀粉含量降低44.4%,总淀粉含量降低24.4%。这进一步表明,通过沉默NtGlgX基因,可对烟草叶片中植物淀粉的含量进行调控,进而可为烟叶品质调控、烟草新品种培育奠定一定技术基础。
以上显示和描述了本发明的基本原理和本发明的优点。本行业的技术人员应该了解,本发明不受上述实施例的限制,上述实施例和说明书中描述的只是说明本发明的原理,在不脱离本发明精神和范围的前提下,本发明还会有各种变化和改进,这些变化和改进都落入要求保护的本发明范围内。本发明要求保护范围由所附的权利要求书及其等效物界定。
序列表
<110> 云南中烟工业有限责任公司
<120> 一种烟草支链淀粉酶基因及其应用
<130> WPC211441
<141> 2021-05-25
<160> 2
<170> SIPOSequenceListing 1.0
<210> 1
<211> 2679
<212> DNA
<213> 人工序列(NtGlgX)
<400> 1
caccaatctt caaccccttc ttcatgttct caggatagtc tattctatag cagagcattt 60
tgggttagca gatcaataat tgcatggaat gtgggtgctg gagagggtga atgctactta 120
tatgctagta gaaatgctgc attatgtgtt gctgatggtg aaattcaagg ccatgatgtg 180
aaaatcaaac ttgaacagac aaattatgga ctttcacagc aggtagttga aaaatttcct 240
catattcgag attatacagc atttgaagtc cctgcaacta tgaactgcca aagtttagtc 300
aagtgccaat tagctgttgc tatcttcaat tctgatggaa gatgtgcaag tgctactgga 360
ttgcagttac ctggtattct tgatgagtta ttctcctata ccgggcccct cggtgctgtt 420
tttgatactg aagctgtctc tctctacctt tgggcaccaa ctgctcaagt ggtgcgagct 480
ctcatttata agagtccatc agagactgac ccagtagaaa ttgttcagct caaggagcta 540
aaaggcgttt ggagtgctaa ggggccaaga cattgggagg gttgctacta tgtttatgaa 600
gtgtctgttt accatcatag caccttgcgg attgagaaat gtgtgagcaa tgatccgtat 660
gccagaggtc tctctgctga tggcaaacgg acattattag ttaatcttgt ttctgatgat 720
gtaaaacctg aaggatggga taatctacaa gatgagaaac cagatcttct ttccttttcc 780
gatatcagcc tatatgagct gcatgttaga gatttcagtg ccaatgaccc tactgtgcct 840
catgaatttc agggtggtta tcttgccttc acattgcagg actcagctgg tgtcaaacac 900
ttaaaaagat tatcaagtgc tggtatcact catgtccatc tgctaccaac ctttcaattt 960
gccggtgttg aagatgagaa acataaatgg aagcatgtag atatcgagaa gctcaactct 1020
tttccaccag attctgagga gcagcaggcc cttatcacag ccatccaaaa tgaagatggg 1080
tataattggg ggtataatcc tgttatttgg ggagttccaa agggaagcta cgctagtaat 1140
gcaaatggtc cttgtcgtat cattgagttt cggaaaatgg tgcaggcact taatcgtatt 1200
ggtctacgtg ttgtattgga tgttgtttac aatcatatta atgcaagtgg tcactttgat 1260
gacttctctg ttctagacaa gattgttcca ggttactatc taagaagaaa tgctgatggt 1320
ggtattgaac atagcacatg cgtaaacaat actgccagtg agcatttcat ggttgagcgc 1380
ttaattcttg acgacctcaa gtgctgggca gttcatttta agattgatgg atttcgcttt 1440
gacctgatgg gtcatataat gaagcgtaca atggtgaagg caaagtcatt gctgaatagc 1500
ttgtcaaaag ataaaaatgg cgtagatgga ccaagtattt atatatatgg tgaaggatgg 1560
gattttggtg aagtggcaaa caacggacgt gggataaatg catcgcaatt caaccttttt 1620
ggatctggaa ttgggagctt caatgatcgc attcgggatg ctctgctggg tggatcacct 1680
tttggtcatc ctctacacca aggttttgta actggccttt atttggagcc taatggtcat 1740
gatcttggtg ataaagccaa tgtagaacgt atgcttgctg tgtcgaagga tcacattcag 1800
gttggaatgg ctgcaaactt aaaggatttt gtgctaacca actgtgatgg tgaagaggtg 1860
aaaggatctg aagtgttttt gcatgatagg aagccagttg gatatgcttc ttctcccatt 1920
gaaacagtta actatgtgtc tgctcatgac aatgagacac tgttcgacat cattagtctt 1980
aagactccaa aggacatttc tgtggaggag agatgcagga tgaaccactt agcgaccagt 2040
gtaatagctc tgtctcaggg aatacctttt ttccactctg gagatgaaat gctgcgctca 2100
aaatcaatag accgtgattc ttacaactcc ggtgattggt tcaacaggct ggattttagc 2160
tacaactcta acaactgggg tgttggtctc cctccgaaag agaagaatga atggaactgg 2220
ccactaatca aacccagact agcagatcca tcctacaagc ctcagaaaag tcatatcctt 2280
gcagctgttg aaaatttttc gaaccttatg caaataagat actcctcacc actcttccgg 2340
ctaaggacgg ccaattctat ccaggaaaga gtacgatttc ataatactgg tccgtcatgg 2400
attcctggtc ttatagtaat gagcattgaa gatggccatc cgggagttcc agggctttct 2460
caattggatc caatttattc ctatatagtt gtcatcatta atcccgaccc gactgatgtc 2520
tcatttgcca atcctgcact aagagcaaga tctcttcaat tgcatccaat acagatgaac 2580
tcaactgatg atgttgttaa gaactcaaca tatgatgcat cctcgggttg ctttaatgtg 2640
cctgccagga caacttctgt gtttgttgaa cctcggtag 2679
<210> 2
<211> 952
<212> PRT
<213> 人工序列(NtGlgX)
<400> 2
Met Ser Ala Ala Leu Leu Ser Tyr Ala Pro Leu Ser Pro Ile Ser Ala
1 5 10 15
Pro Ser Glu Ser Thr Arg Phe Leu Leu Pro Thr His His Lys Arg Leu
20 25 30
Arg Leu Ser Ser Leu Thr Arg Tyr Arg Pro Gly Leu Lys Leu Gly Thr
35 40 45
Ser Arg Pro Leu Thr Leu Arg Cys Ser Ser Thr Met His Gln Ser Ser
50 55 60
Thr Pro Ser Ser Cys Ser Gln Asp Ser Leu Phe Tyr Ser Arg Ala Phe
65 70 75 80
Trp Val Ser Arg Ser Ile Ile Ala Trp Asn Val Gly Ala Gly Glu Gly
85 90 95
Glu Cys Tyr Leu Tyr Ala Ser Arg Asn Ala Ala Leu Cys Val Ala Asp
100 105 110
Gly Glu Ile Gln Gly His Asp Val Lys Ile Lys Leu Glu Gln Thr Asn
115 120 125
Tyr Gly Leu Ser Gln Gln Val Val Glu Lys Phe Pro His Ile Arg Asp
130 135 140
Tyr Thr Ala Phe Glu Val Pro Ala Thr Met Asn Cys Gln Ser Leu Val
145 150 155 160
Lys Cys Gln Leu Ala Val Ala Ile Phe Asn Ser Asp Gly Arg Cys Ala
165 170 175
Ser Ala Thr Gly Leu Gln Leu Pro Gly Ile Leu Asp Glu Leu Phe Ser
180 185 190
Tyr Thr Gly Pro Leu Gly Ala Val Phe Asp Thr Glu Ala Val Ser Leu
195 200 205
Tyr Leu Trp Ala Pro Thr Ala Gln Val Val Arg Ala Leu Ile Tyr Lys
210 215 220
Ser Pro Ser Glu Thr Asp Pro Val Glu Ile Val Gln Leu Lys Glu Leu
225 230 235 240
Lys Gly Val Trp Ser Ala Lys Gly Pro Arg His Trp Glu Gly Cys Tyr
245 250 255
Tyr Val Tyr Glu Val Ser Val Tyr His His Ser Thr Leu Arg Ile Glu
260 265 270
Lys Cys Val Ser Asn Asp Pro Tyr Ala Arg Gly Leu Ser Ala Asp Gly
275 280 285
Lys Arg Thr Leu Leu Val Asn Leu Val Ser Asp Asp Val Lys Pro Glu
290 295 300
Gly Trp Asp Asn Leu Gln Asp Glu Lys Pro Asp Leu Leu Ser Phe Ser
305 310 315 320
Asp Ile Ser Leu Tyr Glu Leu His Val Arg Asp Phe Ser Ala Asn Asp
325 330 335
Pro Thr Val Pro His Glu Phe Gln Gly Gly Tyr Leu Ala Phe Thr Leu
340 345 350
Gln Asp Ser Ala Gly Val Lys His Leu Lys Arg Leu Ser Ser Ala Gly
355 360 365
Ile Thr His Val His Leu Leu Pro Thr Phe Gln Phe Ala Gly Val Glu
370 375 380
Asp Glu Lys His Lys Trp Lys His Val Asp Ile Glu Lys Leu Asn Ser
385 390 395 400
Phe Pro Pro Asp Ser Glu Glu Gln Gln Ala Leu Ile Thr Ala Ile Gln
405 410 415
Asn Glu Asp Gly Tyr Asn Trp Gly Tyr Asn Pro Val Ile Trp Gly Val
420 425 430
Pro Lys Gly Ser Tyr Ala Ser Asn Ala Asn Gly Pro Cys Arg Ile Ile
435 440 445
Glu Phe Arg Lys Met Val Gln Ala Leu Asn Arg Ile Gly Leu Arg Val
450 455 460
Val Leu Asp Val Val Tyr Asn His Ile Asn Ala Ser Gly His Phe Asp
465 470 475 480
Asp Phe Ser Val Leu Asp Lys Ile Val Pro Gly Tyr Tyr Leu Arg Arg
485 490 495
Asn Ala Asp Gly Gly Ile Glu His Ser Thr Cys Val Asn Asn Thr Ala
500 505 510
Ser Glu His Phe Met Val Glu Arg Leu Ile Leu Asp Asp Leu Lys Cys
515 520 525
Trp Ala Val His Phe Lys Ile Asp Gly Phe Arg Phe Asp Leu Met Gly
530 535 540
His Ile Met Lys Arg Thr Met Val Lys Ala Lys Ser Leu Leu Asn Ser
545 550 555 560
Leu Ser Lys Asp Lys Asn Gly Val Asp Gly Pro Ser Ile Tyr Ile Tyr
565 570 575
Gly Glu Gly Trp Asp Phe Gly Glu Val Ala Asn Asn Gly Arg Gly Ile
580 585 590
Asn Ala Ser Gln Phe Asn Leu Phe Gly Ser Gly Ile Gly Ser Phe Asn
595 600 605
Asp Arg Ile Arg Asp Ala Leu Leu Gly Gly Ser Pro Phe Gly His Pro
610 615 620
Leu His Gln Gly Phe Val Thr Gly Leu Tyr Leu Glu Pro Asn Gly His
625 630 635 640
Asp Leu Gly Asp Lys Ala Asn Val Glu Arg Met Leu Ala Val Ser Lys
645 650 655
Asp His Ile Gln Val Gly Met Ala Ala Asn Leu Lys Asp Phe Val Leu
660 665 670
Thr Asn Cys Asp Gly Glu Glu Val Lys Gly Ser Glu Val Phe Leu His
675 680 685
Asp Arg Lys Pro Val Gly Tyr Ala Ser Ser Pro Ile Glu Thr Val Asn
690 695 700
Tyr Val Ser Ala His Asp Asn Glu Thr Leu Phe Asp Ile Ile Ser Leu
705 710 715 720
Lys Thr Pro Lys Asp Ile Ser Val Glu Glu Arg Cys Arg Met Asn His
725 730 735
Leu Ala Thr Ser Val Ile Ala Leu Ser Gln Gly Ile Pro Phe Phe His
740 745 750
Ser Gly Asp Glu Met Leu Arg Ser Lys Ser Ile Asp Arg Asp Ser Tyr
755 760 765
Asn Ser Gly Asp Trp Phe Asn Arg Leu Asp Phe Ser Tyr Asn Ser Asn
770 775 780
Asn Trp Gly Val Gly Leu Pro Pro Lys Glu Lys Asn Glu Trp Asn Trp
785 790 795 800
Pro Leu Ile Lys Pro Arg Leu Ala Asp Pro Ser Tyr Lys Pro Gln Lys
805 810 815
Ser His Ile Leu Ala Ala Val Glu Asn Phe Ser Asn Leu Met Gln Ile
820 825 830
Arg Tyr Ser Ser Pro Leu Phe Arg Leu Arg Thr Ala Asn Ser Ile Gln
835 840 845
Glu Arg Val Arg Phe His Asn Thr Gly Pro Ser Trp Ile Pro Gly Leu
850 855 860
Ile Val Met Ser Ile Glu Asp Gly His Pro Gly Val Pro Gly Leu Ser
865 870 875 880
Gln Leu Asp Pro Ile Tyr Ser Tyr Ile Val Val Ile Ile Asn Pro Asp
885 890 895
Pro Thr Asp Val Ser Phe Ala Asn Pro Ala Leu Arg Ala Arg Ser Leu
900 905 910
Gln Leu His Pro Ile Gln Met Asn Ser Thr Asp Asp Val Val Lys Asn
915 920 925
Ser Thr Tyr Asp Ala Ser Ser Gly Cys Phe Asn Val Pro Ala Arg Thr
930 935 940
Thr Ser Val Phe Val Glu Pro Arg
945 950
- 一种烟草支链淀粉酶基因及其应用
- 一种支链淀粉酶型淀粉去分支酶基因启动子及其应用
