使用位点特异性核酸酶优化核酸的体外分离
文献发布时间:2023-06-19 12:24:27
技术领域
本发明涉及制备用于分离的核酸靶区域的方法,以及优化核酸分离和富集的方法。事实上,样本分离和富集是核酸研究的至关重要的第一步,影响核酸数量和质量,这又直接影响下游应用中获得的数据质量(例如,灵敏度、覆盖范围、稳健性和重现性)。这对于从更复杂的混合物仅分析某些靶核酸区域的应用中、或在存在少量靶核酸的情况下的应用中特别重要。例如,人类“外显子组”(编码蛋白的区域)仅占总基因组的约1%,然而占已知与遗传疾病有关的DNA变异的85%。因此,在与外显子组相关的临床应用(例如,诊断和遗传风险评估)中,分离和富集特别重要。
虽然只有少数靶区域是感兴趣的,但可以使用全基因组分析,然而由于技术、经济和/或时间约束,对整个基因组进行测序通常是不可行的。此外,全基因组测序需要大大增加计算能力和存储以分析产生的大量数据。因此,希望将核酸分离以便将分析限制在核酸分子的特定子集。
背景技术
最近,已经描述核酸分离和富集的方法,其中通过使用一种或多种位点特异性核酸酶(特别是CRISPR/Cas核酸酶)从样本中分离出核酸分子的靶区域(参见,特别是PCT申请号EP2018/071557,通过引用其整体并入本文)。因为核酸酶与靶位点结合,从而保护它们免于受到外部处理(例如核酸外切酶的消化),因此这些核酸酶能够高度特异性地分离核酸分子内的靶区域。这些方法特别有利于分离核酸靶区域,因为原始核酸分子的表征(例如,化学修饰、如碱基修饰、以及核酸序列信息(如错配或SNP))可以保存下来。特别是,这些方法避免扩增靶的需要。此外,这些方法与多种下游技术兼容,例如核酸测序的方法。这些方法在分离核酸靶区域上表现出极大增加的特异性。
发明人出人意料地进一步鉴定出制备用于下游分离出靶区域的核酸分子的方法,并且已经证明当与下游分离相配合时,效果得到改善。事实上,发明人出人意料地表明,效果可以至少得到10,000倍的改善。
发明内容
在详细描述本发明之前,应理解本发明不限于具体示例的方面以及当然可以改变。还应理解,这里使用的术语仅用于描述本发明的具体实施方案,而不应用于限制性。
本文引用的所有出版物、专利和专利申请,无论是在上文还是下文,通过引用其整体并入本文。然而,引用本文中提及的出版物是为了描述和公开出版物中报道的并且可能与本发明结合使用的操作方案、试剂和蛋白。本文中的任何内容均不应被解释为承认本发明无权因在先发明而先于此类公开。
此外,除非另有说明,否则本发明的实践采用本领域技术范围内的蛋白质化学、分子生物学、微生物学、重组DNA技术和药理学的常规技术。这些技术在文献中充分阐明。参见,例如,Ausubel等人编,Current Protocols in Molecular Biology,John Wiley&Sons,Inc.New York,1995,Remington’s Pharmaceutical Sciences,第17版,Mack PublishingCo.,Easton,Pa.,1985,和Sambrook等人,Molecular cloning:A laboratory manual第2版,Cold Spring Harbor Laboratory Press-Cold Spring Harbor,NY,USA,1989。
在所附的权利要求和之前的描述中,以各种意思使用词语“包含”、“包括”、“含有”和其它的变体,即指定所述特征的存在,但不排除本发明的各种实施方案中其它特征的存在或添加,除非因明文或必要的暗示,上下文另有要求。此外,除非本申请的内容另有清楚地指出,如本文所用的术语“一个(a)”、“一种(an)”和“该(the)”,包括复数形式。例如,“靶区域”因此还包括两个或多个靶区域。
本发明的第一个方面是制备核酸分子的方法,其包含用于从样本中分离的靶区域。该方法有利的是,用该方法制备的包含靶区域的核酸分子之后可以用于各种下游分离方法。此外,发明人出人意料地表明,当所述方法与下游分离方法结合时,改善了核酸靶区域分离的效果。事实上,如上所述,效果可以得到至少10,000倍的改善。在不需要高度富集靶区域的情况下,用该制备方法获得的包含靶区域的核酸分子本身可以被充分地分离或富集以供下游使用。此外,本文描述的方法还保留许多其它优点。有利地,因为当使用该方法时最初的核酸分子保持完整,核酸靶区域(例如,化学修饰、错配)的所有表征得以保留。另外,为了制备多种包含靶区域的核酸分子,可以容易地设计使用多种Cas蛋白的多重测定,而没有引物相互作用或交叉识别的风险。此外,该方法可以有利地用于制备核酸,所述核酸包含来自小尺寸样本的靶区域或具有低水平靶核酸的样本的靶区域。此外,本发明保持快速和廉价、可以直接在样本上进行,并且具有很少的处理步骤。有利地,可以在同一容器中进行所有步骤。因此,在不存在容器之间的材料转移的情况下,该方法简单且使得样本损失最小化。最后,在这种方法中没有任何扩增步骤的情况下,也可以减少下游方法中的偏差。
所述制备包含从样本中分离的靶区域的核酸分子的方法包括以下步骤:
a)使核酸分子的群体与以下接触:
-第一2类V型Cas蛋白-gRNA复合物,其中gRNA包含与核酸分子内的第一序列互补的向导段,所述第一序列毗邻所述靶区域,从而形成2类V型Cas蛋白-gRNA-核酸复合物,以及
-保护剂分子,
b)使核酸分子的群体与至少一种具有核酸外切酶活性的酶接触,
c)在步骤a)中形成的2类V型Cas蛋白-gRNA-核酸复合物中回收所述包含靶区域的核酸分子,以及
d)将步骤c)的核酸分子与进行性聚合酶接触。
在上述方法中,按照所提供的顺序依次进行步骤:首先进行步骤a),接着是步骤b),之后是步骤c),然后是步骤d)。在步骤a中,包含靶区域的核酸分子可以与2类V型Cas蛋白-gRNA复合物同时或顺序接触、以及和保护剂分子以任何顺序接触(即,核酸分子可以首先与2类V型Cas蛋白-gRNA复合物接触,然后是保护剂分子或反之亦然)。步骤b)必然在步骤a)之后,步骤a)的2类V型蛋白-gRNA复合物和保护剂分子保护靶区域免于被根据步骤b)的一种或多种具有核酸外切酶活性的酶进行下游降解。
优选地,制备包含从样本中分离的靶区域的核酸分子的方法包括以下步骤:
a)使核酸分子的群体与以下接触:
-2类V型Cas蛋白-gRNA复合物,其中gRNA包含与核酸分子内的第一序列互补的向导段,所述第一序列毗邻所述靶区域,从而形成2类V型Cas蛋白-gRNA-核酸复合物,以及
-第一保护剂分子,
b)使核酸分子的群体与至少一种具有核酸外切酶活性的酶接触,从而降解未受保护的核酸分子,
c)在步骤a)中形成的2类V型Cas蛋白-gRNA-核酸复合物中回收所述包含靶区域的核酸分子,以及
d)使步骤c)的核酸分子与进行性聚合酶接触,优选嗜温或嗜热聚合酶,甚至更优选为T4 DNA聚合酶,从而在所述核酸分子上进行末端修复。
在一些情况下,可以在方法中包括附加步骤。
作为非限制性示例,上述方法可以在步骤a)之前、与步骤a)同时、或者在步骤a)和b)之间,可以包括将一种或多种核酸分子片段化以获得核酸分子的群体的附加步骤。作为非限制性示例,上述方法中在步骤a)之前、或者在步骤a)和b)之间,可以包括将一种或多种核酸分子线性化的附加步骤。当核酸分子的样本或群体包含非线性分子时,可以显著地包括该步骤。作为非限制性示例,在上述方法的任何步骤之前、之间、或之后还可以进一步包括孵育的附加步骤。作为非限制性示例,可以在步骤c)之后进一步包括储存步骤。这些可选的附加步骤如下进一步详述。
制备包含用于分离的靶区域的核酸分子
如本文所用的表述“制备包含用于分离的靶区域的核酸分子”、“制备包含靶区域的核酸分子”或“包含用于分离的靶区域的核酸分子的制备”是指获得核酸,所述核酸包含靶区域和在所述靶区域一侧或两侧的另外的非靶区域。包含用于分离的靶区域的核酸分子有利地适用于各种下游方法。有利地,鉴于步骤d),使用该制备方法获得的核酸分子可包含平末端或3'单腺嘌呤碱基突出。用该方法获得的核酸分子可以有利地进一步经受核酸末端的修饰(例如,连接平末端衔接子(adaptor)或接头)用于下游方法。
如本文所用的术语“样本”是指包含核酸分子的群体的任何材料或物质,包括例如,生物、环境或合成样本。“生物样本”可以是可含有生物有机体的任何样本,例如细菌、病毒、古生菌、动物、植物和/或真菌。根据本发明的“生物样本”还指的是可以从生物有机体中获得的样本,例如,从细菌、病毒、古生菌、植物、真菌、动物和/或其它真核生物中获得的细胞提取物。可以直接从有机体或从有机体中获得的生物样本(例如,来自血液、尿液、脑脊液、精液、唾液、痰、粪便和组织(例如,细胞组织或植物组织))中获得目的核酸分子。在本发明的上下文中,任何细胞、组织或体液都可以用作核酸来源。也可以从培养的细胞中回收或纯化核酸分子,例如原代细胞培养物或细胞系。可以用病毒或其它细胞内病原体感染从中获得目的核酸的细胞或组织。样本也可以是从生物标本中提取的总核酸。“环境样本”可以是包含不直接取自生物有机体(例如,土壤、海水、空气等)的核酸的任何样本,并且可包含不再存在于生物有机体内的核酸。“合成样本”包括人工或工程化的核酸。可选择地,样本可以是来自怀疑包含靶核酸区域的任何来源。
在某些实施方案中,本发明的方法可包含一个或多个处理样本的步骤,以促进根据本发明方法制备包含靶区域的核酸。作为非限制性示例,可以浓缩、稀释或破坏(例如,通过机械或酶裂解)样本。在本方法的步骤a)之前,可以完全或部分纯化核酸,或者核酸可以是非纯化的形式。
如本文所定义的术语“核酸分子”是指核苷酸单体的聚合物,包括脱氧核糖核苷酸(DNA)、核糖核苷酸(RNA)、或其类似物,以及其组合(例如,DNA/RNA嵌合体)。本文所述的脱氧核苷酸和核糖核苷酸单体是指单体单元,其分别包含三磷酸基团、腺嘌呤(“A”)、胞嘧啶(“C”)、鸟嘌呤(“G”)、胸腺嘧啶(“T”)、或尿嘧啶(U)含氮碱基、以及脱氧核糖或核糖。在本文中还包括修饰的核苷酸碱基,其中核苷酸碱基是,例如次黄嘌呤、黄嘌呤、7-甲基鸟嘌呤、肌苷、黄嘌呤核苷、7-甲基鸟苷、5,6-二氢尿嘧啶、5-甲基胞嘧啶、假尿苷、二氢尿苷、或5-甲基胞苷。在本发明的上下文中,当描述核苷酸时,“N”表示任何核苷酸、“Y”代表任何嘧啶、以及“R”代表任何嘌呤。核苷酸单体通过核苷酸之间的键相连,例如,磷酸二酯键、或其磷酸酯类似物和相关的反离子(counter ions)(例如,H
核酸分子长度可从仅若干单体单位(例如寡核苷酸,其可从例如少于100至多达200单体长)至数千、数万、数十万、或数百万单体单位。优选地,所述核酸分子包含一种或多种cfDNA分子,例如上述那些。在第一方面,核酸分子的长度小于300bp,例如在约125和225bp之间,优选在130和200bp之间。在第二方面,核酸分子的长度等于或优于300bp。在本申请中,除非另有说明,否则应当理解,核酸分子从左到右以5'至3'方向表示。
如本文所用的术语“核酸分子的群体”是指多于一种核酸分子。所述群体可包含如上所定义的任何序列的任何长度的一种或多种不同核酸分子。核酸分子的群体可明显包含多于10
如本文所定义的“核酸靶区域”或“靶区域”是指特异性核酸分子或特异性核酸区域,所述特异性核酸分子存在于更复杂的样本或核酸分子的群体中,所述特异性核酸区域存在于更大的核酸分子中,并且其将被特异性靶向以用于分离或富集。当靶区域存在于更大的核酸分子中时,优选在一侧或两侧为非靶区域。靶区域优选在其第一侧的侧翼为第一序列,所述第一序列至少部分地与以下互补:2类V型Cas蛋白-gRNA复合物所包含的crRNA分子的向导段或所包含的gRNA。此外,核酸靶区域优选地在其第二侧的侧翼为保护剂分子。当所述保护剂分子是2类Cas蛋白-gRNA复合物时,所述核酸靶区域在其第二侧的侧翼为第二序列,所述第二序列至少部分与2类Cas蛋白-gRNA复合物所包含的crRNA分子的向导段互补或与所包含的gRNA互补。因此,所述第一和第二序列位于靶区域的侧翼。
在一些实施方案中,可以制备两种或多种不同的包含靶区域的核酸分子。因此,本发明的“包含靶区域的核酸分子”可以包含两种或多种不同的核酸分子,其包含靶区域,优选至少2、5、10、25、50、100个或更多个区域。核酸靶区域可以是编码的或非编码的、或两者的组合。靶区域可以是基因组或附加体的(episomic)。靶区域可以包括一种或多种重复区域、重排、复制(duplication)、易位、缺失、错配、SNP和/或修饰的碱基,例如表观遗传修饰。在一些情况下,所述核酸靶区域可以是相同的(例如,对应于重复序列)。在其它情况下,核酸靶区域可能是不同的。优选地,所述靶核酸区域将具有至少约44、50、100、250、500、1,000、5,000、10,000、20,000、30,000、40,000、50,000、60,000、70,000、80,000、90,000或100,000个核苷酸长。
这里使用的术语“区域”是指不间断的核苷酸聚合物。虽然给定的gRNA可以允许制备包含靶区域的多个核酸(例如,由于非特异性结合,或识别在核酸分子中存在的多于一个的序列),在本发明的情况下每种gRNA优选识别核酸分子的群体中的单一序列。优选通过使用位于所述靶区域侧翼的靶向第一序列和第二序列的至少两种不同gRNA,来制备包含靶区域的核酸分子。下面描述其它实施方案。
Cas蛋白
本文所用的术语“Cas蛋白”是指特异性识别和结合核酸靶区域的RNA指导的核酸内切酶。为了特异性识别和结合特异性靶区域,Cas蛋白复合物具有“向导RNA”或“gRNA”,以形成“Cas蛋白-gRNA复合物”。Cas蛋白的结合特异性由gRNA确定,其包含“向导段”,其序列必须至少部分地与给定核酸序列的序列互补。Cas蛋白-gRNA复合物内的向导段与所述核酸序列杂交,从而形成Cas蛋白-gRNA-核酸复合物。Cas蛋白-gRNA复合物与核酸序列的成功结合进一步需要在核酸分子中存在短、保守的序列,所述序列位于杂交区域紧邻的核酸分子中。该序列称为原间隔序列相关基序(protospacer adjacent motif)或“PAM”。因此,结合核酸序列的Cas蛋白-gRNA复合物包含通过向导段的核酸杂交以及Cas蛋白自身与PAM的相互作用。在Cas蛋白-gRNA复合物与核酸序列结合后,Cas蛋白通常通过在双链核酸分子的每条链中的两个相邻核苷酸之间断裂磷酸二酯键来切割核酸。具体地,Cas蛋白的一个结构域切割与gRNA杂交的核酸链,而Cas蛋白的第二结构域切割非杂交的核酸链。可以交错地切割双链分子的两条链,产生单链突出、或平末端。如技术人员可以容易地理解,当在步骤a)中,核酸群体与多于一种的Cas蛋白-gRNA复合物接触,可以形成多于一种的Cas蛋白-gRNA核酸复合物。这些方面在下面进一步详述。
至今已经描述三个主要类别的Cas蛋白(1类、2类和3类)。还应注意,在2类Cas蛋白中,迄今为止鉴定了至少五种不同的类型(即I、II、III、IV、V型)。作为非限制性示例,Cas蛋白可以选自2类Cas蛋白,特别是2类V型和2类II型Cas蛋白,更特别是来自于Cas9、Cpf1、C2c1、C2c3和C2c2(Cas13a)蛋白。
作为非限制性示例,2类Cas蛋白可以来自以下物种之一:肺炎链球菌(Streptococcus pneumoniae)、化脓性链球菌(Streptococcus pyogenes)、嗜热链球菌(Streptococcus thermophilus)、犬链球菌(Streptococcus canis)、金黄色葡萄球菌(Staphylococcus aureus)、脑膜炎奈瑟菌(Neisseria meningitidis)、齿垢密螺旋体(Treponema denticola)、土拉弗朗西斯菌(Francisella tularensis)、新生弗朗西斯菌(Francisella novicida)、多杀性巴氏杆菌(Pasteurella multocida)、变异链球菌(Streptococcus mutans)、空肠弯曲杆菌(Campylobacter jejuni)、红嘴鸥弯曲杆菌(Campylobacter lari)、鸡败血支原体(Mycoplasma gallisepticum)、盐渍硝化菌(Nitratifractor salsuginis)、食清洁剂细小棒菌(Parvibaculum lavamentivorans)、小肠罗氏杆菌(Roseburia intestinalis)、灰色奈瑟球菌(Neisseria cinerea)、固氮葡糖醋杆菌(Gluconacetobacter diazotrophicus)、固氮螺菌(Azospirillum)、球状球藻(Sphaerochaeta globosa)、柱状黄杆菌(Flavobacteriumcolumnare)、塔夫嗜黄杆菌(Fluviicola taffensis)、嗜粪拟杆菌(Bacteroides coprophilus)、移动支原体(Mycoplasma mobile)、法氏乳杆菌(Lactobacillus farciminis)、巴氏链球菌(Streptococcus pasteurianus)、约氏乳杆菌(Lactobacillus johnsonii)、巴氏葡萄球菌(Staphylococcus pasteuri)、龈沟产线菌(Filifactor alocis)、韦氏菌属(Veillonellasp)、沃兹沃斯沙特氏菌(Suterella wadsworthensis)、钩端螺旋体(Leptotrichia sp.)、白喉棒状杆菌(Corynebacterium diphtheriae)、酸胺球菌属(Acidaminococcus sp.)、或毛螺菌属(Lachnospiraceae sp.)、阿尔伯普雷沃菌(Prevotella albensis)、异型真杆菌(Eubacterium eligens)、溶纤维丁酸弧菌(Butyrivibrio fibrisolvens)、史密塞拉属(Smithella sp.)、黄杆菌属(Flavobacterium sp.)、白菜卟啉单孢菌(Porphyromonascrevioricanis)、或毛螺细菌(Lachnospiraceae bacterium)ND2006。
作为非限制性示例,2类Cas蛋白可以是J3F2B0、Q0P897、Q6NKI3、A0Q5Y3、Q927P4、A1IQ68、C9X1G5、Q9CLT2、J7RUA5、Q8DTE3、Q99ZW2、G3ECR1、Q73QW6、G1UFN3、Q7NAI2、E6WZS9、A7HP89、D4KTZ0、D0W2Z9、B5ZLK9、F0RSV0、A0A1L6XN42、F2IKJ5、S0FEG1、Q6KIQ7、A0A0H4LAU6、F5X275、F4AF10、U5ULJ7、D6GRK4、D6KPM9、U2SSY7、G4Q6A5、R9MHT9、A0A111NJ61、D3NT09、G4Q6A5、A0Q7Q2、或U2UMQ6。登录编号来自UniProt(www.uniprot.org),版本最新修订在2017年1月10日。作为非限制性示例,编码2类Cas蛋白的基因可以是包含核苷酸序列的任何基因,其中所述序列产生相应Cas蛋白的氨基酸序列,例如上面列出的那些之一。本领域技术人员将容易理解,由于遗传密码的简并化,可以改变基因的核苷酸序列而不改变氨基酸序列。此外,2类Cas蛋白可以是密码子优化的,用于在细菌(例如,大肠杆菌)、昆虫、真菌或哺乳动物细胞中表达。
也已在其它细菌种中鉴定出2类Cas蛋白和直系同源蛋白,并且在PCT申请No.WO2015/071474的实施例1中特别描述,通过引用并入本文。在一些情况下,Cas蛋白可以是同源物或直系同源物,例如,上列的物种之一的2类Cas蛋白。
虽然野生型Cas蛋白具有核酸内切酶活性,但是结合核酸序列的Cas蛋白-gRNA复合物与其催化活性无关。已经描述野生型Cas蛋白的变体和突变体。作为非限制性示例,已经描述突变的Cas蛋白缺乏切割含有靶区域的靶核酸分子的一条或两条链的能力,如Cas变体保留核酸内切酶活性但具有改善的结合特异性(例如,2类Cas蛋白eSpCas9,如Slaymaker等人所述,Science,2015,351(6268):84-86)。
本文所用的术语“Cas切口酶”是指包含一种失活的催化核酸酶结构域和一种活性的催化核酸酶结构域的修饰的Cas蛋白。与gRNA复合的Cas切口酶将与如上所述的特异性核酸序列结合,但是仅在双链核酸的一条链的两个核苷酸之间破坏磷酸二酯键。Cas切口酶可以切割与gRNA杂交的核酸链,或者与gRNA至少部分相同的非杂交核酸链。“切口位点”是指双链核酸分子中经历一条链破裂的位点。在切口位点产生3'羟基和5'磷酸基团。
如本文所用术语“催化死亡的”、“催化失活的”、或“死亡的”是指包含两种催化失活的核酸酶结构域的修饰的Cas蛋白。与gRNA复合的催化失活的Cas蛋白将结合如上所述的特异性序列,但不切割或切开双链核酸的任一链。
修饰的2类Cas蛋白可包含一种或多种修饰,导致如上所述的其核酸酶结构域的一种或两种的特异性失活。优选地,这种修饰不影响2类Cas蛋白-gRNA复合物形成、2类Cas蛋白识别PAM基序、和/或与核酸序列结合的强度和/或稳定性、和/或2类Cas蛋白-gRNA复合物结合至核酸序列靶区域的强度和/或稳定性。作为非限制性示例,2类Cas蛋白的可能修饰包括一种或多种以下氨基酸处的取代:E762、HH983或D986、D10、H840、G12、G17、N854、N863、N982、或A984,其中根据化脓性链球菌的Cas9蛋白的氨基酸序列对氨基酸进行编号(例如,在Uniprot数据库中登录编号为Q99ZW2),或在另一种2类Cas蛋白中的等价氨基酸位置。例如,一种或多种氨基酸可以被丙氨酸(例如,E762A、HH983AA或D986A、D10A、H840A、G12A、G17A、N854A、N863A、N982A或A984A)或者通过另一个氨基酸取代,其导致相应的催化结构域的失活。
作为非限制性示例,2类Cas切口酶可以包含在氨基酸位置取代,其等同于如上所述的化脓性链球菌的Cas9蛋白的H840(例如,H840A)或D10(例如,D10A)位置。优选地,“Cas9n”蛋白包含在等同于为H840(例如,H840A)或在位置D10(例如,D10A)的氨基酸位置的取代。取决于切口酶变体(例如,包含在D10或H840的取代),切口酶将切开gRNA杂交链或非杂交链。特别地,包含在D10的取代的切口酶将切开gRNA杂交链,而包含在H840的取代的切口酶将切开不与gRNA杂交的链。或者,2类Cas切口酶可包含在等同于Cpf1蛋白的R1226(例如,R1226A)氨基酸位置的取代。包含在R1226取代的2类Cas切口酶将切开不与gRNA杂交的链。
作为非限制性示例,催化失活的2类Cas蛋白,例如,催化失活的Cas9蛋白(在本文中也称为“Cas9d”、或者“dCas9”)将包括至少两个在等同于Cas9蛋白中的D10和H840的氨基酸位置上的取代。优选地,Cas9d包括至少在两个氨基酸位置D10和H840处的取代。
2类V型Cas蛋白
在制备用于分离的核酸靶区域的方法的上下文中,使用的Cas蛋白是2类Cas蛋白,特别是2类V型Cas蛋白。事实上,如上所述,制备包含用于分离的靶区域的核酸分子的方法的第一步(步骤a))包括:使得核酸分子的群体与2类V型Cas蛋白接触。当具有催化活性时,与适当的gRNA复合的2类V型蛋白产生交错的切口(例如,3至5个碱基的短5'突出),其位于双链核酸分子的PAM序列远端(例如,位于距PAM至少10个核苷酸远)(参见,例如Zetsche等人,Cell,2015,163(3):759-771)。进一步观察到,2类V型蛋白-gRNA复合物可以保持与核酸分子结合,因此可以有利地保护所述核酸免于降解,例如当所述核酸分子暴露于核酸外切酶时。不受理论束缚,在切割双链核酸分子之后,2类V型蛋白-gRNA复合物可以保持与核酸链结合,所述核酸链包含PAM序列,其中相对链(即,不包含PAM序列的链)从分子复合物上解离。
优选地,2类V型Cas蛋白是催化活性的(即,切割双链分子的两条链)。优选地,本发明的2类V型Cas蛋白选自Cpf1(也称为Cas12a)和C2c1,更优选为Cpf1。Cpf1蛋白优选是上述列出合适物种之一的Cpf1蛋白,更优选以下物种或菌株之一:新生弗朗西斯菌U112(登录号:AJI61006.1)、阿尔伯普雷沃菌(登录号:WP_024988992.1)、酸胺球菌属BV3L6(登录号:WP_021736722.1)、异型真杆菌(登录号:WP_012739647.1)、溶纤维丁酸弧菌(登录号:WP_027216152.1)、史密塞拉属SCADC(登录号:KFO67988)、黄杆菌属316(登录号:WP_045971446.1、白菜卟啉单孢菌(登录号:WP_036890108.1)、拟杆菌口腔菌群(oral taxon)274(登录号:WP_009217842.1)、或者毛螺菌ND2006(登录号:WP_051666128.1)。在优选的实施方案中,所述酸胺球菌属BV3L6Cpf1是包含以下氨基酸取代的变体:S542R/K607R或S542R/K548V/N552R(Gao等人,Nat Biotechnol.2017,35(8):789-792)。
向导RNA
本文所用的术语“向导RNA”或“gRNA”指两种向导RNA分子,其由crRNA分子和tracrRNA分子组成。或者,本文所用的术语gRNA指单一向导RNA分子、或sgRNA,其包括crRNA和tracrRNA序列段。或者,gRNA可以仅由crRNA分子组成。gRNA分子可以进行化学修饰,例如,包含一种或多种核糖核苷酸的碱、糖或磷酸修饰。任选地,可以修饰gRNA分子的5'和/或3'端,例如通过与另一种分子或化学基团共价缀合。
crRNA分子或段优选为20至75个核苷酸长,更优选为30至60个核苷酸,甚至更优选为40至45个核苷酸长。crRNA分子或段优选包含第一区域,在本文中称为“向导段”,其序列至少部分地与核酸分子中存在的序列互补。示例性通用crRNA核苷酸序列显示在SEQ IDNO:2,其中向导段由‘N’核苷酸的延伸表示。优选地,本发明的gRNA的向导段包含与核酸分子中存在的序列至少70%、至少80%、至少85%、至少90%、至少95%、至少96%、至少为97%、至少98%、至少99%、或更优选100%序列互补性。优选地,当互补性小于100%时,错配位于crRNA末端附近,其杂交在距离PAM最远处。例如,当2类II型Cas蛋白是Cas9时,错配优选包含在crRNA分子的5'端或段的5'端(例如,在前7个核苷酸中),因为Cas9识别在crRNA的3'端的PAM。或者,当2类V型Cas蛋白是Cpf1时,错配优选包含在crRNA分子的3'端或段的3'端(例如,在后7个核苷酸中),因为Cpf1识别在crRNA的5'端的PAM。向导段优选为至少10、11、12、13、14、15、16、17、18、19、20、21、22、23、24或25个核苷酸长,更优选为15、16、17,18、19、20、21、22、23、24或25,甚至更优选为17、18、19、20、21、22、23或24个核苷酸长。或者,向导段优选为10至30,更优选为15至25,甚至更优选为17至24个核苷酸长。crRNA分子或段优选包含第二区域,在本文中称为“tracr-匹配段”。tracr-匹配段包含序列,所述序列优选至少部分与tracrRNA分子或段互补,更优选至少部分与tracrRNA的5'端互补。tracr-匹配段优选为至少10、11、12、13、14、15、16、17、18、19、20、25、30、40、50个核苷酸长,更优选至少15个核苷酸长,甚至更优选至少20个核苷酸长。优选地,向导段位于crRNA分子或段的5'端。优选地,tracr-匹配段位于crRNA分子或段的3'端处或其附近。
tracrRNA分子或段优选为10至175个核苷酸长,更优选为40至110,更优选为60至90,甚至更优选为65至80个核苷酸长。tracrRNA分子或段优选包含至少一个二级结构,优选至少两个二级结构,更优选至少三个二级结构,甚至更优选为三个或四个二级结构。优选地,所述至少一个二级结构位于tracrRNA分子或段的3'端处或附近。示例性通用tracrRNA核苷酸序列显示在SEQ ID NO:3中。tracrRNA分子优选包含tracrRNA结合段,所述tracrRNA结合段与crRNA的tracr-匹配段互补。优选地,tracrRNA结合段包含与crRNA的tracr-匹配段至少50%、60%、70%、80%、85%、90%、95%、99%或100%互补的序列。优选地,tracrRNA结合段是至少10个核苷酸长。如本文所用的核酸分子的术语“在5’-端处或附近”指从5’到3'将段或结构放置在分子的前半部分内。类似地,如本文所用的核酸分子的术语“在3’-端处或附近”是指将段或结构放置在分子的后半部分内。
作为非限制性示例,在gRNA中存在的“二级结构”可以是茎环或发夹、凸起、四环和/或假结。术语“发夹”和“茎环”在gRNA的上下文中可互换地使用,并且在下面定义(参见“保护剂分子”部分)。根据优选的实施方案,所述gRNA包含至少一个发夹二级结构。
根据第一优选实施方案,gRNA是包含crRNA和tracrRNA段的sgRNA分子。示例性通用sgRNA核苷酸序列显示在SEQ ID NO:4,其中向导段由‘N’核苷酸的延伸表示。甚至更优选地,sgRNA由crRNA和tracrRNA段组成。优选地,crRNA和tracrRNA段融合在一起。所述段优选通过磷酸二酯键或包含一种或多种核苷酸的核酸接头融合在一起。所述sgRNA优选为30至180个核苷酸长,更优选为40至90个核苷酸长。优选地,crRNA的3'-端融合到tracrRNA的5'-端。优选地,crRNA和tracrRNA通过加入的接头融合。可选择地,crRNA和tracrRNA分子可通过化学连接融合,例如共价键(例如,三唑连接)。
根据第二优选的实施方案,gRNA由两个分开的RNA分子组成,所述分开的RNA分子由crRNA分子和tracrRNA分子组成。
根据第三优选实施方案,当2类V型Cas蛋白是Cpf1时,gRNA仅由crRNA分子组成。因此,“Cpf1-gRNA复合物”在本文中还可互换地称为“Cpf1-crRNA复合物”。当gRNA仅为crRNA分子时,至少必须存在向导段。示例性通用crRNA核苷酸序列显示在SEQ ID NO:1,其中向导段由‘N’核苷酸的延伸表示。优选地,所述crRNA分子还包含二级结构,例如发夹。优选地,所述crRNA分子不包含tracr-匹配段。优选地,向导段位于crRNA分子的3'-端。优选地,二级结构位于crRNA分子的5'-端处或附近。优选地,所述crRNA是40至50个核苷酸长。
如本文所用的术语“互补”指一个核酸序列或分子(例如gRNA)与另一个核酸序列或分子(例如,核酸分子中的序列)进行序列特异性反平行核苷酸碱基配对相互作用,而导致形成双链体或其它更高有序结构的能力。主要相互作用类型是核苷酸碱基特异性,例如,通过Watson-Crick和Hoogsteen型氢键的A:T、A:U和G:C。这也称为“核酸结合”、“杂交”、或“退火”。在本领域中公知核酸与靶核酸的互补区域杂交的条件(参见,例如Nucleic AcidHybridization,A Practical Approach,Hames和Higgins编,IRL Press,Washington,D.C.(1985))。杂交条件取决于具体应用,并且可以由本领域技术人员常规确定。
在本发明的上下文中,互补结合并不意味着两个核酸序列或分子(例如,gRNA和靶区域,或tracr-匹配段和tracrRNA)必须彼此完全互补。此外,crRNA序列段或分子没有必要完全互补核酸分子中的序列。事实上,已知2类Cas蛋白-gRNA复合物可特异性结合与gRNA少至8或9个碱基互补的核酸序列。优选地,在gRNA最接近PAM的10个碱基、以及互补核酸序列最接近PAM的相应10个碱基之间不存在错配,更优选在gRNA最接近PAM的6个碱基和互补核酸序列最接近PAM的相应6个碱基之间不存在错配,甚至更优选在gRNA距离PAM的4、5、和/或6个碱基的碱基和互补核酸序列距离PAM 4、5、和/或6个碱基的相应碱基之间不存在错配。事实上,如果在所述碱基的一个或多个位置存在错配,则结合将是不稳定的,并且通过2类Cas蛋白-gRNA复合物保护靶区域免于核酸外切酶消化将被减少甚至废除。如本文之前所述,通过增加crRNA段的长度,或者通过将错配放置在距离PAM最远的crRNA段的末端处或附近,可以减少脱靶杂交。可选择地,可通过存在一种或多种修饰的碱基或化学修饰来修饰gRNA以增加结合特异性,例如,见于Cromwell等人,Nat Communt.2018,9(1):1448,或OrdenRueda等人,Nat Communce.2017,8:1610的那些描述,通过引用并入本文。此外,核酸可以在一种或多种区域中杂交,使得干扰或相邻区域不参与杂交事件(例如,环或发夹结构)。基于其通用知识并且鉴于以上详述的参数,根据待杂交的核酸序列,本领域技术人员可以容易地设计一种或多种gRNA分子。
之前已经显示出核酸与Cas蛋白与gRNA(核酸:Cas蛋白:gRNA)分子数的比率影响靶区域分离的效果。根据包含待制备的靶区域和/或起点的核酸和/或核酸分子的群体的复杂性在这里可显著优化包含靶区域的核酸:Cas蛋白:gRNA分子数的比率。不受理论的限制,优化可特别依赖于DNA复杂性,更复杂的核酸群体需要较高的Cas蛋白和gRNA的量。作为非限制性示例,较不复杂的核酸群体可以基本上包含重复序列或PCR扩增的片段,而更复杂的核酸群体可包含基因组DNA。作为非限制性示例,当所述核酸分子存在于PCR产生的核酸分子的群体中时,1:10:20的比率可用于制备包含靶区域的核酸。相反,当所述群体包含或由大肠杆菌基因组DNA组成时,优选1:1600:3200的比率,而当所述群体包含或由人类基因组DNA组成时,优选为1:100000:200000的比率。当使用多种gRNA(例如,其中两种gRNA识别两种位于靶区域侧翼的第一序列和第二序列,或者多路复用(multiplexing)以制备包含不同靶区域的多种核酸分子),可以为所有gRNA选择核酸:Cas蛋白:gRNA的单一优化比率。可选择地,可以单独为每种gRNA选择优化的比率。
根据优选的实施方案,该比率为至少1:10:10,更优选为至少1:10:20,甚至更优选为至少1:10:50。当通过PCR产生模板DNA时,特别优选至少1:10:20的比率。优选地,选择向导RNA以有效使用PCR模板,必要时(例如,如果所述模板不同),接着在适当的模板上优化包含靶区域的核酸:Cas蛋白:gRNA的比率。优选地,野生型Cas蛋白-gRNA复合物的切割效率为至少70%,更优选至少80%,甚至更优选至少90%。优选地,Cas蛋白-gRNA复合物对靶区域的保护效率为至少70%,更优选至少80%,甚至更优选至少90%。优选地,当核酸由细菌(例如,革兰氏阴性细菌,例如大肠杆菌)来源的核酸制备时,包含靶区域的核酸:Cas蛋白:gRNA分子数量的比率至少为1:200:400,更优选至少1:400:800,甚至更优选至少1:800:1600,至少1:1600:3200,或至少1:3200:6400。根据可选择的优选实施方案,包含靶区域的核酸:Cas蛋白:gRNA的比率至少为1:10,000:20,000,更优选至少为1:100,000:200,000。根据包含待制备的靶区域和/或起点的核酸和/或核酸分子的复杂性以及鉴于以上提供的比率,技术人员可以容易地调整包含靶区域的核酸:Cas蛋白:gRNA的比率。虽然Cas蛋白相对gRNA的比例可以变化,但是有利地将gRNA提供为Cas蛋白的至少过量两倍,以确保Cas蛋白成功地装载上gRNA。可理所当然使用更高比率的Cas蛋白(例如,1:20:40、1:50:100等,用于PCR靶)以及任选的,更高比率的gRNA(例如1:10:30,1:10:40等用于PCR靶)。上述比例优选用于本发明的方法的步骤a)中,特别是关于包含靶区域的核酸:2类V型Cas蛋白:gRNA的比率。在保护剂分子是2类Cas蛋白的情况下,也可以使用上述比率。
原间隔序列相邻基序(PAM)
如本文所用的术语“原间隔序列相邻基序”或“PAM”是指短核苷酸序列(例如,2至6个核苷酸),其通过Cas蛋白自身直接识别,更具体地通过2类Cas蛋白来识别。PAM序列及其放置(placement)将根据所使用的Cas蛋白而变化,并且本领域技术人员可以根据其一般知识、或者使用诸如在Karvelis等人,Genome Biology,2015,16:253描述的技术容易地确定。例如,新生弗朗西斯菌的Cpf1蛋白识别PAM5'-TTTN-3'或5'-YTN-3',酸胺球菌属的Cpf1蛋白识别PAM 5’-TTTN-3。作为另一个示例,化脓性链球菌的Cas9蛋白识别PAM 5’-NGG-3’。相比之下,金黄色葡萄球菌的Cas9蛋白识别PAM 5’-NNGRRT-3’、脑膜炎奈瑟菌的Cas9识别PAM5’-NNNNGATT-3’、嗜热链球菌的Cas9识别PAM 5’-NNAGAA-3’、齿垢密螺旋体的Cas9识别PAM 5’-NAAAAC-3’、犬链球菌的Cas9识别PAM 5’-NNG-3’、源自新生弗朗西斯菌的工程化的Cas9蛋白识别PAM 5’-YG-3’。PAM基序通常位于与gRNA杂交的核酸序列的5’或3’端紧密相邻位点上的双链靶核酸分子中的非杂交链上。PAM所需的放置位置取决于所用的Cas蛋白(例如,当使用Cas9蛋白时,PAM优选地位于紧邻gRNA的3'-端,而使用Cpf1蛋白时,PAM优选地位于紧邻gRNA的5'-端)。可选择地,PAM基序可以包含在gRNA分子本身中或单独的DNA寡核苷酸中,其可添加到样本中。例如,当使用本方法分离单链RNA分子时,可能必要通过这些方法中的一种将PAM加入样本。认为2类Cas蛋白与PAM的结合是轻微破坏双链核酸,从而允许gRNA与核酸序列杂交。
当Cas蛋白为2类V型时,所述PAM优选位于与gRNA的5'端紧邻的靶区域的非杂交链上。当Cas蛋白为2类II型时,所述PAM优选位于与gRNA的3'端紧邻的靶区域的非杂交链上。然而,在一些情况下,所述PAM优选包含在gRNA分子自身或DNA寡核苷酸内。
保护剂分子
如本文所用的术语“保护剂分子”是指与核酸分子相结合并保护相邻的核酸区域(即,靶区域)免于降解的分子。保护剂分子可以结合核酸分子的游离端或在核酸分子内结合。当在核酸分子内结合时,所述保护剂分子优选结合至特异性序列。与2类V型Cas蛋白-gRNA复合物相比,所述保护剂分子必须与相同的不间断核苷酸聚合物结合。在本发明的上下文中,保护剂分子优选与靶区域相邻的第二序列结合,使得所述靶区域所在的一侧是与2类V型Cas蛋白-gRNA复合物结合的第二序列(由此形成2类V型Cas蛋白-gRNA-核酸复合物)以及在另一侧是保护剂分子。
在本发明的上下文中,保护剂分子优选是发夹衔接子或位点特异性核酸内切酶,更优选是Cas蛋白-gRNA复合物,甚至更优选为2类Cas蛋白-gRNA复合物。然而,在本发明的范围内还包括,在特异性序列处稳定结合核酸的其它位点特异性核酸酶,例如,类转录激活因子效应物核酸酶(TALEN)或锌-指蛋白。
根据优选的实施方案,保护剂分子与2类V型Cas蛋白-gRNA-核酸复合物距离至少50个核苷酸远,优选至少100、250、500、1,000、5,000、10,000、20,000、30,000、40,000、50,000、60,000、70,000、80,000、90,000、100,000、200,000、500,000、750,000或1,000,000个核苷酸远的序列相结合。因此,根据优选的实施方案,靶区域的长度至少为50、100、250、500、1,000、5,000、10,000、20,000、30,000、40,000、50,000、60,000、70,000、80,000、90,000、100,000、200,000、500,000、750,000或1,000,000个核苷酸。
在本发明方法的一个实施方案中,保护剂分子与包含靶区域的核酸分子的游离端结合。因此,根据优选的实施方案,所述保护剂分子是发夹衔接子。
如本文所用的术语“发夹”或“发夹衔接子”是指这样的分子,其碱基对自身形成具有双链茎和环的结构,其中一条链的5’端通过未配对的环物理连接到另一条链的3’端。所述物理连接可以是共价的或非共价的。优选地,所述物理链接是共价键。如本文所用的术语“环”是指不与所述核酸的相同链或另一链的核苷酸通过氢键配对的核酸链的连续核苷酸,因此是单链的。如本文所用的“茎”是指链内配对的区域。优选地,所述茎包含至少3、5、10或20个碱基对,更优选至少5、10或20个碱基对,甚至更优选至少10或20个碱基对。当发夹与双链核酸分子的游离端结合时,发夹的3'和5'端分别与双链核酸分子的5'和3'端连接。优选地,所述发夹衔接子与核酸分子的任何一个游离端(优选仅游离端中的一个)相结合。所述发夹衔接子可以特异性结合核酸分子的一个游离端。作为非限制性示例,可以通过用非回文限制性酶将核酸分子片段化进行特异性结合,从而在核酸分子的每个新的游离端产生不同的突出。当保护剂分子是发夹衔接子时,所述护剂分子结合至核酸分子的游离端的任何一个,优选仅结合至核酸分子的游离端的一个,使得所述靶区域的一侧侧翼是与2类V型Cas蛋白-gRNA复合物结合的第一序列(由此形成2类V型Cas蛋白-gRNA-核酸复合物)以及在另一侧侧翼是发夹衔接子。
如本文所用的术语“游离端”是指核酸分子的端,其可在5'端包含磷酸基团和/或在3'端包含羟基基团。游离端可以是平的或包括单链的突出。所述单链突出可以是3'或5'突出。所述单链突出优选具有小于100、50、25、10、5、4或小于3个核苷酸的长度。
优选地,所述发夹衔接子与核酸分子连接,优选连接到核酸分子上。更优选地,步骤a)包括使核酸群体与发夹衔接子接触、以及将所述发夹衔接子连接到核酸分子的游离端。在优选的实施方案中,如本文所述,如果必要的话(例如,当样本包含环形核酸分子),该方法还包括在使所述群体与发夹衔接子接触之前,将核酸分子的群体线性化的步骤。当所述包含靶区域的核酸分子小于300bp长时,所述保护剂分子优选为发夹衔接子。
可选择地,当保护剂分子是第二位点特异性核酸内切酶时,例如第二2类Cas蛋白-gRNA复合物、TALEN或锌-指蛋白,优选地,所述保护剂分子在核酸分子内结合。
根据一个具体实施方案,所述保护剂分子是2类V型或2类II型Cas蛋白-gRNA复合物。当保护剂分子是Cas蛋白-gRNA复合物时,特别优选与2类V型-gRNA复合物和所述保护剂分子同时接触核酸分子的群体。这是有利的,因为该方法的持续时间降低。优选地,所述保护剂分子是Cpf1-gRNA复合物,其中PAM位点与核酸靶区域相邻。因此,根据制备包含用于分离的靶区域的核酸分子的方法的具体实施方案,步骤a)包括分别使核酸分子的群体与第一和第二Cpf1-gRNA复合物接触,所述第一和第二复合物分别结合到位于所述靶区域侧翼的第一和第二序列。
根据优选的实施方案,当保护剂分子是Cas蛋白-gRNA复合物时,Cas蛋白可以是具有催化活性的Cas蛋白、切口酶或失活的Cas蛋白。因此,根据具体实施方案,当使用两种Cpf1-gRNA复合物时,所述两种Cpf1-gRNA复合物是催化活性的。根据可选择的实施方案,第一Cpf1-gRNA复合物是催化活性的,而与第二Cpf1-gRNA复合物对应的保护剂分子是切口酶或催化活性上失活的。
可以在任何上述实施方案中使用多种Cas蛋白-gRNA复合物和保护剂分子。事实上,根据一个特别优选的实施方案,可以单独使用两种或多种Cas蛋白-gRNA复合物,或者将其与两种或多种保护剂分子组合使用,以制备包含用于分离的靶区域的两种或多种核酸分子。根据另一个实施方案,当方法是用于制备多种核酸(核酸各自包含用于分离的靶区域)时,步骤a)包括使核酸分子的群体与多种2类V型Cas蛋白-gRNA复合物和保护剂分子接触,使得每个靶区域在第一侧为所述复合物将结合的第一序列(从而形成2类V型Cas蛋白-gRNA-核酸复合物),以及在第二侧为所述保护剂分子。优选地,当所述靶区域存在于相同的核酸分子时,所述核酸靶区域彼此分开至少100、200、300、500、750、1000、2000、5000或10000个核苷酸。当包含靶区域的所述核酸分子长度大于300bp时,所述保护剂分子优选为Cas蛋白-gRNA复合物。
根据具体实施方案,当分离多种靶区域时,核酸分子的群体可以与多于一种类型的保护剂分子接触。在一个优选的实施方案中,多于一种类型的保护剂分子包括保护剂分子的组合,优选发夹衔接子和位点特异性核酸内切酶,例如Cas蛋白-gRNA复合物,更优选2类Cas蛋白-gRNA复合物。
如本文所用的术语“接触”是指将两种或多种分子和/或产物放置在相同溶液中,使得所述分子和/或产物可以彼此相互作用。使核酸分子的群体与2类V型Cas蛋白-gRNA复合物接触,例如,允许这些分子的相互作用以及形成复合物,其中2类V型Cas蛋白-gRNA复合物已在特异性位点处结合核酸分子。类似地,使核酸分子的群体与保护剂分子接触从而允许这些分子的相互作用,以及保护剂分子与核酸的结合。“结合”可能更具体地指互补核苷酸通过氢键(即退火)形成、或任何其它共价或非共价相互作用的杂交。作为另一个示例,当分子或产物是酶的底物时,使得所述分子或所述产物与所述酶(例如核酸外切酶)“接触”,将导致酶反应。例如,使核酸分子的群体与具有核酸外切酶活性的酶接触,将导致消化与所述酶的底物相对应并且可被酶所接近的核酸分子。
具有核酸外切酶活性的酶
根据本文所述的方法,将核酸分子的群体与至少一种2类V型Cas蛋白-gRNA复合物和至少一种保护剂分子接触之后,所述方法还包括将核酸分子的群体与至少一种具有核酸外切酶活性的酶接触的步骤。该步骤降解不受所述2类V型Cas蛋白-gRNA-核酸复合物和所述保护剂分子保护的核酸分子,从而增加靶核酸分子相较于非靶核酸分子的比率。本领域技术人员将理解,该步骤必须在步骤a)之后进行,以防止不期望地降解核酸靶区域。
如本文所用的术语“具有核酸外切酶活性的酶”是指具有5'至3'和/或3'至5'核酸外切酶活性的酶。所述具有核酸外切酶活性的酶可以是核糖核酸外切酶、或脱氧核糖核酸外切酶、或两者。所述酶可以识别双链、或单链核酸分子或两者。所述具有核酸外切酶活性的酶可以具有或不具有一种或多种其它酶活性(例如,特异性或非特异性的核酸内切酶活性)。作为非限制性示例,可用于本发明的具有核酸外切酶活性的酶包括λ(λ)核酸外切酶(在本文中也称为λexo)、核酸外切酶I(Exo I)、核酸外切酶III(Exo III)、核酸外切酶VII、S1核酸酶、核酸外切酶T、T5核酸外切酶、T7核酸外切酶、RecBCD核酸酶、RecJf、绿豆核酸外切酶、RNaseD、RNaseR核糖核酸外切酶I、核糖核酸酶外切II等。酶降解可以是部分的(即,即使群体已经与具有核酸外切酶活性的酶接触之后,在该群体中也可以存在未受保护的核酸区域或分子)或完全的。这可能取决于孵育条件、样本组合物、核酸群体本身(例如,核酸结构)、或本领域技术人员已知的其它变量。因此,术语“降解”包括至少部分地降解未受保护的核酸分子。
根据优选的实施方案,具有核酸外切酶活性的所述酶没有核酸内切酶活性。当靶区域包含可以被位点特异性核酸内切酶识别的位点时、或当靶区域对于非特异性核酸内切酶降解易感时,这可能是有利的。根据优选的实施方案,所述至少一种具有核酸外切酶活性的酶是λ核酸外切酶、核酸外切酶I(Exo I)、核酸外切酶III(Exo III)、核酸外切酶VII、核酸外切酶T、T5核酸外切酶、T7核酸外切酶、S1核酸酶、RecBCD核酸酶、RecJf、RNase D、RNaseR核糖核酸外切酶I、核糖核酸外切酶II、优选λ核酸外切酶或Exo I,更优选其一种或多种的组合,甚至更优选为λ核酸外切酶和Exo I两者。
回收包含用于分离的靶区域的核酸
2类V型Cas蛋白-gRNA复合物稳定且紧密地与核酸序列结合,以形成2类V型Cas蛋白-gRNA-核酸分子复合物。由于该结合可以防止包含靶区域的核酸与其它化合物相互作用(例如蛋白、多肽、核酸分子),有利的是从2类V型Cas蛋白-gRNA-核酸分子复合物中分离核酸分子,便于在下游方法中使用。优选从溶液中除去2类V型Cas蛋白和gRNA复合物,例如通过这些元件降解或进一步纯化包含靶区域的核酸分子。作为非限制性示例,通过使核酸分子与至少一种蛋白酶接触,可以从2类V型Cas蛋白-gRNA-核酸复合物中回收包含靶区域的核酸分子。这降解2类V型Cas蛋白和任何其它可以存在的蛋白(即,保护剂分子,例如2类Cas蛋白-gRNA复合物,或从初始样本中保留的污染蛋白)。作为非限制性示例,蛋白酶可以选自丝氨酸蛋白酶、半胱氨酸蛋白酶、苏氨酸蛋白酶、天冬氨酸蛋白酶、谷氨酸蛋白酶、金属蛋白酶和/或天冬酰胺肽裂解酶。
此外、或可选择地,通过使核酸分子与能够螯合二价阳离子(特别是Mg
根据优选的实施方案,通过使2类Cas蛋白-gRNA-核酸分子复合物与二价阳离子的螯合剂接触(优选螯合Mg
此外,或可选择地,通过使核酸分子与至少一种RNase(例如,RNaseA、RNaseH、或RNaseI)接触,可以从2类V型Cas蛋白-gRNA-核酸复合物(以及保护剂分子,如果适用的话)中回收包含靶区域的核酸分子。这将降解gRNA。在另一个实施方案中,因为在升高的温度中RNA不稳定,任选地在二价金属离子和/或碱性pH下存在,可以加热样本(例如,至少65℃)。
在一些实施方案中,例如使用顺磁珠,进一步纯化回收的包含靶区域的核酸分子。
聚合
根据本文提供的方法,在回收包含靶区域的核酸分子后,使所述核酸分子与进行性聚合酶接触。如本文所用的术语“进行性聚合酶”是指能够催化连续核苷酸的掺入而不释放模板链的聚合酶。不受理论的限制,所述进行性聚合酶可以修复包含靶区域的核酸分子末端存在的任何突出(因此,在本文中该步骤还可互换地称为“末端修复”步骤)。优选地,所述进行性聚合酶具有至少3'至5'加工活性,因此能够修复(或“补平”)5'突出,产生双链区域。在没有校对活性的情况下(例如,使用3'至5’核酸外切酶不足的进行性聚合酶),可以获得包含由单一碱基(通常为腺嘌呤)组成的3'突出的核酸分子。根据优选的实施方案,所述进行性聚合酶优选是嗜温或嗜热聚合酶,甚至更优选为T4 DNA聚合酶。
因此,如本文所用的“末端修复”是导致双链核酸分子的两条链在碱基对(也称为平末端)上终止的过程。可以在5'和/或3'突出进行末端修复,优选在5'突出上进行。如上所述,优选使用进行性聚合酶进行末端修复。优选地,通过使包含5'突出的核酸分子与进行性聚合酶和dNTP在适当的缓冲液中接触来进行末端修复。技术人员鉴于其一般知识,其很清楚末端修复对应的是什么。
如本文所用的“嗜温聚合酶”是指明显从嗜温微生物(即,在20℃至45℃的温度中生长最佳)中已经分离的聚合酶或源自所述分离的聚合酶。考虑到核酸底物,所述聚合酶可以是DNA或RNA聚合酶。当核酸分子是DNA时,所述聚合酶优选是嗜温DNA聚合酶。通常,在约37℃的温度之时或之下(例如约12℃至37℃),嗜温聚合酶将具有聚合酶活性。嗜温聚合酶的示例包括,例如PolI、Klenow、Klenow外切、M-MuLV、phi29、T4、T5、以及T7(RNA和DNA聚合酶两者)。
与嗜温聚合酶相反,“嗜热聚合酶”具有较高的聚合酶活性的最佳温度(例如,在45至80℃之间),以及与嗜温聚合酶相比对热失活具有更强的抗性。然而,本文所用的术语热稳定性聚合酶不必然指对热失活具有完全抗性的酶。因此,热处理可以在一定程度上降低嗜热聚合酶的活性。例如,通过将嗜温T5 DNA聚合酶暴露于90℃的温度30秒,所述酶3'-至-5'活性完全失活,而嗜热DNA聚合酶(例如,Taq聚合酶),则在95℃时具有超过6小时的半衰期。在本文中,包含靶区域的核酸分子不变性。优选地,当使包含靶区域的核酸分子与聚合酶接触时,所述接触步骤发生在小于或等于80℃的温度下,优选小于或等于75℃、70℃、60℃,甚至更优选小于或等于50℃。根据一个具体实施方案,所述包含靶区域的核酸分子与聚合酶在50℃下接触20分钟。
如本领域技术人员所理解的,聚合酶活性需要存在核苷酸(例如,对于DNA聚合酶是四种核苷酸dATP、dTTP、dGTP、dCTP,对于RNA聚合酶是ATP、UTP、CTP和GTP)。如技术人员已知的,可以在各种试剂(包括缓冲剂、盐等)存在下进一步进行聚合反应。
在一些实施方案中,例如,使用顺磁珠进一步纯化已经过末端修复的包含靶区域的核酸分子。
任选的补充步骤
片段化
根据本发明的具体实施方案,在上述方法中,核酸分子在与2类V型Cas蛋白-gRNA复合物和保护剂分子接触之前或之后可以被片段化,有利地是在核酸分子与2类V型Cas蛋白-gRNA复合物接触之后。本文所用的术语“片段化”是指通过将核酸分子断裂成至少两个较小分子,增加核酸分子的5'-和3'-游离端的数量。因为可以改善本方法中的核酸外切酶消化的效率,核酸片段化是有利的。事实上,从如上定义,核酸外切酶活性只能从5'-和/或3’-游离端起始。
片段化可以通过剪切来进行,例如通过超声处理、水力剪切、超声、雾化,或可以通过酶促片段化来进行,例如通过使用一种或多种位点特异性核酸内切酶,如限制性酶。可以使用至少2、3、4、5或更多种位点特异性的核酸内切酶。应当理解,数据库中可获得的越来越多数量的序列,使得本领域技术人员能够容易地鉴定出一种或多种限制性酶,其裂解位点位于包含靶区域的核酸之外。有利地,当同时使用2种或更多种酶时,所述酶彼此兼容(例如,相同的缓冲液需求、失活条件)。片段化可以是部分的(例如,核酸分子的群体中存在的所有切割位点并非都被限制性酶切割)或完全的。因此,术语“片段化”包括核酸分子和区域的至少部分片段化,其侧翼不是2类V型Cas蛋白-gRNA-核酸分子复合物和保护剂分子。这些核酸分子或序列在本文中也可称为“未受保护的”分子或区域。
因此,在一些实施方案中,制备包含靶区域的核酸的方法还包括以下步骤:
·优选通过将核酸分子的群体与至少一种位点特异性核酸内切酶接触,更优选与至少一种限制性酶接触,片段化不受保护的区域。
技术人员将理解,在包含靶区域的核酸分子与2类V型Cas蛋白-gRNA复合物和保护剂分子接触后,发生上述片段化的步骤。然而,在某些情况下,在核酸分子的群体与2类V型Cas蛋白-gRNA复合物和/或保护剂分子接触之前或接触同时,可发生片段化的步骤。当在与2类V型Cas蛋白-gRNA复合物或保护剂分子相同的条件(例如,缓冲液、温度)中,所述位点特异性核酸内切酶切割时,情况尤其如此。
如果样本中存在一种或多种环形核酸分子,则片段化将有利地线性化所述核酸分子,从而产生游离端,其之后可以被靶向而降解。当存在环形核酸分子时,优选在如本文所述的制备包含靶区域的核酸分子的方法的步骤a)或b)之前或同时进行片段化。
根据优选的实施方案,通过将核酸分子的群体与至少一种位点特异性的核酸内切酶接触,优选至少2、3、4、5或更多位点特异性核酸内切酶,来片段化核酸分子。优选地,所述位点特异性核酸内切酶是限制性酶,更优选II型、III型或人工限制性酶,甚至更优选为II型限制性酶,和/或2类V型Cas蛋白-gRNA复合物,如Cpf1-gRNA复合物。II型限制性酶包括IIP、IIS、IIC、IIT、IIG、IIE、IIF、IIG、IIM和IIB型类别,如Pingoud和Jeltsch,NucleicAcids Res,2001,29(18):3705–3727所述。优选地,来自这些类别的一种或多种酶用于片段化本发明中的核酸分子。可以由技术人员选择适当的酶。在使用彼此不兼容的多种限制性酶的情况下,片段化可以包含使用不同限制性酶和条件(例如,温度、时间、缓冲液)的多个顺序步骤。优选地,所述至少一种位点特异性核酸内切酶产生非回文突出。优选地,通过位点特异性核酸内切酶切割至少50%、60%、70%、80%、90%、95%或100%的切割位点。在所述位点特异性核酸内切酶是2类V型Cas蛋白-gRNA复合物的情况下,所述蛋白-gRNA复合物结合包含待制备的靶区域的核酸分子并在所述核酸分子外的位点切割。优选地,位点特异性核酸内切酶结合这样的序列,其距包含靶区域的核酸100至5000碱基,更优选距包含靶区域的核酸区域150至1000碱基,甚至更优选距包含靶区域的核酸区域250-750碱基。优选地,所述位点特异性核酸内切酶靶向在核酸分子中多次存在的特异性序列,例如人类基因组中的Alu元件,但是在包含靶区域的核酸分子中不存在。
根据具体实施方案,根据上述任何实施方案中的任何一个,在将核酸分子的群体与2类V型Cas蛋白-gRNA复合物和保护剂分子接触之后,所述群体同时与至少一种具有核酸外切酶活性的酶和至少一种位点特异性核酸内切酶接触,使所述核酸分子片段化。这是特别有利的,因为它降低了该方法的持续时间。根据另一个优选的实施方案,具有核酸外切酶活性的所述酶也可具有位点特异性核酸内切酶活性。根据该实施方案,具有位点特异性核酸内切酶活性的酶的切割位点位于包含靶区域的核酸分子的外部。使用具有核酸外切酶和位点特异性核酸内切酶活性的单一酶是特别有利的,因为其降低所需试剂的数量和花费。
孵育和/或储存
根据本发明的一个具体实施方案,可以在上述方法的步骤c)之后储存核酸分子。根据具体实施方案,核酸分子可以在以下时进行孵育:在与2类V型Cas蛋白-gRNA复合物和保护剂分子接触期间或之后、在与至少一种具有核酸外切酶活性的酶接触期间或之后、在回收包含靶区域的核酸分子期间、和/或在与上述方法中的进行性聚合酶接触期间或之后。优选地,将核酸分子孵育30分钟至2小时。
靶区域的分离
根据上述方法已经制备的包含靶区域的核酸分子可以视为已经过第一轮的分离或富集。然后可以在下游方法中使用包含用上述方法制备的靶区域的核酸分子,例如但不限于进一步的分离或富集方法。事实上,在需要高水平的分离或富集核酸靶区域的情况下,用上述方法获得的核酸分子有利地分离所述靶区域。
因此,在另一个方面,本发明涉及从核酸分子中分离核酸靶区域的方法,其中所述核酸分子已经使用如上所述的任何制备方法进行制备。出人意料的是,发明人发现,当在上述制备方法的下游进行这种分离核酸靶区域的方法时,效果得到改善。更具体地,本发明出人意料地表明,当在根据上述方法(特别是包括将核酸分子与进行性聚合酶接触的步骤)制备的包含靶区域的核酸分子上进行分离时,效果改善至少10,000倍。
此外,本文描述的方法还保留了许多额外的优点。首先,核酸靶区域(例如,化学修饰、错配)的所有表征是保守的。此外,该方法可以有利地用于从小样本量中分离靶区域,或从具有低水平的所述靶区域的样本中分离靶区域。这种分离方法快速且廉价,可以直接在使用上述方法制备的核酸分子上进行,处理步骤少并且相对简单。有利地,可以在同一容器中进行所有步骤。因此,在没有容器之间材料转移的情况下,样本损失最小。在这种分离方法中没有任何扩增步骤,也可以减少下游方法中的偏差。另外,可以容易地设计使用多个2类Cas蛋白用于分离多个靶区域的多重测定,没有引物相互作用或交叉识别的风险。最后,该方法与现有的下游核酸分析平台(包括“第二”和“第三代”测序技术,其中在微结构(例如,纳米孔、零模波导(zero-mode wave guide)或微孔)中分析单核酸分子)兼容,所以该方法是有利的。有利地,本方法提供在一端或两端包括单链核酸突出的分离的核酸靶区域,在其上可以特异性地连接各种衔接子或接头,使靶区域可用在各种下游分析与应用。
所述分离核酸靶区域的方法更具体地包括以下步骤:
a)提供包含核酸靶区域的核酸分子,其中已经使用上述制备方法制备所述核酸分子,
b)将所述核酸分子与2类Cas蛋白-gRNA复合物接触,其中所述gRNA包括与第三序列互补的向导段,所述第三序列在第一序列和第一保护剂分子之间,从而形成一个2类Cas蛋白-gRNA-核酸复合物,
c)任选地,使所述核酸分子与第二保护剂分子接触,所述第二保护剂分子优选是第二2类Cas蛋白-gRNA复合物,其中所述gRNA包括与第四序列互补的向导段,从而形成第二2类Cas蛋白-gRNA-核酸复合物,所述第四序列在第一序列和第一保护剂分子之间,其中所述第三和第四序列优选包含在所述靶区域中,
d)使所述核酸分子与至少一种具有核酸外切酶活性的酶接触,以及
e)从2类Cas蛋白-gRNA-核酸复合物中回收分离的核酸靶区域。
在上述特定分离方法的上下文中,按照提供的顺序依次进行步骤,其中首先进行步骤a)以及最后进行步骤e)。上述方法的步骤b)和c)可以以任何具体顺序进行,只要它们在步骤d)之前进行。事实上,步骤b)可以在步骤c)之前进行,步骤c)可以在步骤b)之前进行,或者可以同时进行两个步骤。步骤d)和e)顺序进行。在一些情况下,如下所示,可以在方法中包括其它任选的步骤。技术人员将进一步理解,可以制备多于一种包含靶区域的核酸分子,因此,本分离方法也可包括分离多于一种核酸靶区域(例如,2、5、10、20、50、100等不同的靶区域)。
如上所述,本发明人出人意料地表明,当本方法与制备包含用于分离的靶区域的核酸的上述方法组合使用时,效果可以得到至少10,000倍的改善,其中所述方法包括使核酸分子与进行性聚合酶接触的步骤(即,末端修复)。不受理论的限制,当上述制备核酸的方法与目前所描述的分离方法组合使用时观察到的改善效果,可能是由于在上述制备包含靶区域的核酸的方法中在步骤a)和/或b)期间,当包含PAM的核酸链从2类V型Cas蛋白-gRNA-核酸复合物中解离时可以产生的5’单链突出得到补平。在这种情况下,当在步骤b)中当Cas蛋白-gRNA-核酸复合物与至少一种具有核酸外切酶活性的酶接触时,包含PAM的链可能不受保护免于核酸外切酶消化,因此导致产生对核酸外切酶处理有抗性的5’突出。在上述制备方法的步骤d)中去除这些突出可有利地制备包含靶区域的核酸分子,以便用于下游的方法中,特别是用于分离靶区域的本方法。
本文所用的术语“分离(isolation)”或“分离的(isolating)”是指样本中的一种或多种靶核酸区域相对于一种或多种其它区域或分子的比例的增加。作为非限制性示例,这些其它分子可包含蛋白、脂质、碳水化合物、代谢物、核酸或其组合。作为非限制性示例,这些其它区域可以对应于这样的核酸区域,其存在于与所述靶区域相同的分子中(或者甚至与之相邻)但不包含在所述靶区域之内。作为具体示例,在上述制备方法中制备的核酸分子的修复后的末端可以不包含在靶区域中。在一些情况下,可以在包含靶区域中的分离的核酸中包含5’或3’突出(即,如果仅使用单一核酸外切酶消化双链核酸的单链)。优选地,所述突出的长度为100至700个核苷酸长。在另一示例中,当靶区域对应于基因时,靶区域不包括相邻的非编码区域。作为可选择的非限制性示例,靶区域还可包括与PAM序列相邻的多达50个碱基。如本文所用,靶核酸区域的“分离”可以更具体地指在样本中的一种或多种靶核酸区域与样本中的一种或多种其它分子相比,或与初始样本(即,在进行制备和分离本发明靶区域的方法之前)中的分子的总数相比的比例增加至少2倍(例如,2、3、4、5、10、20、30、40、50、60、70、80、90、100、250、500、750、1000或10,000倍或更多)。靶核酸区域的分离还可以指,当与样本中的一种或多种其它分子的水平相比时,样本中的靶核酸区域的比例增加至少5%(例如,5%、6%、7%、8%、9%、10%、15%、20%、25%、30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%、或100%)。当靶核酸区域的比例为100%时,样本中不包含其它分子。如本文所用的术语“富集”更具体地指相对于样本中的其它核酸分子分离一种或多种靶核酸区域。作为一个示例,富集靶区域是指与总初始核酸的量相比,分离的靶区域的比例增加,其中分离的靶区域的比例增加至少10%、20%、30%、40%、50%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%。根据优选的实施方案,与总初始核酸的量相比,分离的靶区域的比例增加至少10%,更优选至少20%、至少30%、至少40%、至少50%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少96%、至少97%、至少98%,甚至更优选至少99%或100%。
根据一个实施方案,分离的核酸靶区域富集至少10倍、至少20倍、至少50倍、至少100倍、至少250倍、至少500倍、至少750倍、优选至少1000倍、至少10,000倍、至少100,000倍、甚至更优选至少1,000,000倍、至少2,000,000倍或至少3,000,000倍。作为具体的示例,来自等同于约32亿bp的人类基因组的核酸分子的群体的单一1kb片段的100%富集表示3,000,000倍增加。
根据可选择的实施方案,分离的靶区域是基本上纯的。“基本上纯的”是指在根据本发明的方法分离靶区域后,所述分离的靶区域包含在样本中的总核酸中的至少99%,优选至少99.5%。
根据优选的实施方案,目的靶区域在初始样本(例如,在制备包含靶区域的核酸并分离靶区域两者之前)中包含总核酸的少于10%、优选少于5%、更优选少于2%、少于0.05%、少于0.02%、甚至更优选少于0.01%、少于0.005%、少于0.001%、少于0.0005%、少于0.0001%、少于0.00005%、少于0.00001%、或少于0.0000005%。技术人员将认识到,样本的总核酸内目的靶区域的量或百分比将取决于待分离的靶区域的数量和待分离的靶区域的长度变化而变化。作为非限制性示例,约32亿bp的人类基因组内的1kb目的靶区域占总基因组的少于0.0000005%。
与制备方法相关的上述包含靶区域的核酸分子:Cas蛋白:gRNA的数量的任何比率也可用于分离靶区域的本方法中,考虑到核酸靶区域:Cas蛋白(优选2类Cas蛋白):gRNA的数量比率。优选的比率可以对应之前提供的任何比率。
分离核酸分子的本方法的第一步(步骤a))包括提供至少一种包含核酸靶区域的核酸分子,所述核酸分子已经根据上述制备方法制备。所述核酸分子包括靶区域和侧翼区域,所述侧翼区域包括在制备方法期间2类V型Cas蛋白-gRNA复合物所结合的至少第一序列。所述第一序列至少部分与2类V型Cas蛋白-gRNA复合物包含的gRNA的向导段互补。取决于在制备方法中提供的保护剂分子(即,第一保护剂分子)是否是第二位点特异性核酸内切酶或发夹,所述包含靶区域的核酸分子可包含第二序列,所述第二序列至少部分与2类V型Cas蛋白-gRNA复合物包含的gRNA的向导段互补或不互补。事实上,当保护剂分子是发夹时,所述发夹可以与平末端或突出结合。因此,发夹不一定必须与特异性序列结合。优选地,如前所述在制备方法的上下文中,发夹将与游离端结合。与之相反,当保护剂分子是位点特异性核酸内切酶时,所述位点特异性核酸内切酶优选与第二序列结合,其中所述第一和第二序列位于待分离的靶区域侧翼。
在本方法分离靶区域的上下文中,由制备方法获得的所述包含靶区域的核酸分子,与2类Cas蛋白-gRNA复合物接触,其中所述gRNA包含与第三序列互补的向导段。在本文中,所述第三序列位于制备方法的第一序列和保护剂分子之间。在不存在保护剂分子的情况下(即,保护剂分子不再与通过上述制备方法制备的核酸分子相关),参考在制备方法期间所述保护剂分子之前结合之处描述第三序列的位置。换句话说,所述第三序列位于第一序列和保护剂分子之间,更具体地,当所述保护剂分子本身(即,2类Cas蛋白-gRNA复合物)不再存在时,位于第一序列和所述保护剂分子之前结合的第二序列之间。因此,所述第三序列位于上述制备方法制备的核酸分子内。优选地,所述第三序列与靶区域结合。
如上所述在制备方法的上下文中提供的Cas蛋白、gRNA和PAM的一般描述也适用于本分离方法中。事实上,本发明的分离方法中使用的2类Cas蛋白可以特别是上述任何2类Cas蛋白,优选2类V型或2类II型Cas蛋白。优选地,所述2类Cas蛋白选自以下物种之一:肺炎链球菌、化脓性链球菌、嗜热链球菌、犬链球菌、金黄色葡萄球菌、脑膜炎奈瑟菌、齿垢密螺旋体、土拉弗朗西斯菌、新生弗朗西斯菌、多杀性巴氏杆菌、变异链球菌、空肠弯曲杆菌、红嘴鸥弯曲杆菌、鸡败血支原体、盐渍硝化菌、食清洁剂细小棒菌、小肠罗氏杆菌、灰色奈瑟球菌、固氮葡糖醋杆菌、固氮螺菌、球状球藻、柱状黄杆菌、塔夫嗜黄杆菌、嗜粪拟杆菌、移动支原体、法氏乳杆菌、巴氏链球菌、约氏乳杆菌、巴氏葡萄球菌、龈沟产线菌、韦氏菌属、沃兹沃斯沙特氏菌、钩端螺旋体、白喉棒状杆菌、酸胺球菌属、或毛螺菌属、阿尔伯普雷沃菌、异型真杆菌、溶纤维丁酸弧菌、史密塞拉属、黄杆菌属、白菜卟啉单孢菌、或毛螺细菌ND2006,更优选为化脓性链球菌、脑膜炎奈瑟菌、嗜热链球菌或齿垢密螺旋体,甚至更优选为化脓性链球菌。优选地,所述2类Cas蛋白选自以下蛋白之一:J3F2B0、Q0P897、Q6NKI3、A0Q5Y3、Q927P4、A1IQ68、C9X1G5、Q9CLT2、J7RUA5、Q8DTE3、Q99ZW2、G3ECR1、Q73QW6、G1UFN3、Q7NAI2、E6WZS9、A7HP89、D4KTZ0、D0W2Z9、B5ZLK9、F0RSV0、A0A1L6XN42、F2IKJ5、S0FEG1、Q6KIQ7、A0A0H4LAU6、F5X275、F4AF10、U5ULJ7、D6GRK4、D6KPM9、U2SSY7、G4Q6A5、R9MHT9、A0A111NJ61、D3NT09、G4Q6A5、A0Q7Q2、或U2UMQ6(登录号来自UniProt(www.uniprot.org),版本上次在2017年1月10日修改)。在分离核酸靶区域的特定方法的上下文中使用的术语“2类Cas蛋白”,可以指本文所述的任何2类Cas蛋白,所述2类Cas蛋白与本文所述的gRNA形成复合物。优选地,编码2类Cas蛋白的基因是包含核苷酸序列的任何基因,其中所述序列产生相应的Cas蛋白的氨基酸序列,例如上面列出的那些之一。
优选地,所述2类Cas蛋白是Cas9、Cpf1、C2c1、C2c3、或C2c2,优选Cas9或Cpf1,甚至更优选Cas9。虽然所述2类Cas蛋白可以是具有催化活性的(切口酶)、或催化失活的,所述2类Cas蛋白优选为催化失活的。事实上,不受受理论的限制,使用催化失活的酶特别有利,因为这种Cas蛋白对所述核酸分子的端部的结构没有影响,因为没有进行切割。因此,如下所述,可以通过将所述核酸分子与包含核酸外切酶活性的适当的酶一起孵育,可选择核酸分子末端的结构(即平末端、5’突出、或3’突出)。
在本发明的分离靶区域的方法的上下文中,所述包含靶区域的核酸分子可以与保护剂分子接触,特别是第二保护剂分子(与上述参考制备方法中使用的第一保护剂分子相反)。本发明分离靶区域的方法的第二“保护剂分子”可对应于任何保护剂分子,所述保护剂分子描述涉及之前描述的制备包含靶区域的核酸分子的方法。因此,2类Cas蛋白-gRNA复合物、和任选的第二保护剂分子保护靶区域免受下游通过一种或多种具有核酸外切酶活性的酶降解。
在本分离方法中,第二保护剂分子的用途是任选的。作为非限制性示例,当在制备方法中之前已经使用发夹作为保护剂分子,则包含靶区域的核酸分子不需要与第二保护剂分子接触,因为所述发夹仍将包含在根据上述制备方法制备的包含靶区域的核酸分子中。事实上,发夹不易受到在非变性条件下进行的核酸外切酶活性或聚合的影响,因此将保留作为包含靶区域的核酸分子的部分。作为进一步的非限制性示例,在靶区域可以被单一2类Cas-蛋白-gRNA复合物保护的情况下(例如,所述靶区域与2类Cas蛋白-gRNA复合形成2类Cas蛋白-gRNA-核酸复合物),第二保护剂分子可能不是必需的。在具体实施方案中,所述靶区域优选具有小于44个核苷酸或bp的长度。或者,在制备方法包括使核酸分子的群体与保护剂分子接触的情况下,其中所述保护剂分子是位点特异性核酸内切酶,在分离靶区域的本方法中优选使包含靶区域的核酸分子与第二保护剂分子接触。在分离靶区域的本方法中使用的保护剂分子是第二2类Cas蛋白-gRNA复合物的情况下,所述gRNA优选包括向导段,其与使用上述制备方法制备的核酸分子内的第四序列互补。在本文中,如以上关于制备方法所述,如第三序列,所述第四序列也位于第一序列和保护剂分子之间。在不再存在保护剂分子的情况下(即,保护剂分子不再与通过上述制备方法制备的核酸分子相关),还参考制备方法期间所述保护剂分子之前结合的位置,描述第四序列的位置,并且如以上用于第三序列的描述。优选地,所述第三序列和所述第四序列包含在所述靶区域内。优选地,所述第三和第四序列嵌套在所述第一序列和保护剂分子之间,更具体地当所述保护剂分子是位点特异性核酸内切酶时在所述第一序列和第二序列之间。优选地,所述第三序列位于距离所述第一序列(以及任选的所述保护剂分子或所述第二序列)少于1000、750、600、500、400、300、200,更优选少于100个核苷酸。优选地,所述第四序列距离所述保护剂分子或所述第二序列少于1000、750、600、500、400、300、200,更优选少于100个核苷酸。当然,如本文所述,所述第三和第四序列可以与所述第一序列和所述保护剂分子相邻。事实上,与其它序列(除非特别指出)相比,术语第一、第二、第三和第四仅仅是指特定不同核酸序列,而没有特定朝向或方向。所述第三和第四序列优选位于靶区域的每个端部(extremity)。这里使用的术语“端部”是指靶区域的外边界,其不必须对应于游离端。事实上,在大多数情况下,所述靶区域将包含在较长的核酸分子中(参见图1E或图2A(v),其中由Cas9-gRNA复合物结合的序列对应于靶区域的端部)。在一些情况下,所述第三序列(和任选的,所述第四序列)可以对应于靶区域。优选通过将2类Cas蛋白-gRNA复合物与每个序列结合来保护所述第三和第四序列免于降解。在这种情况下,靶区域有利地以其整体直接得到保护。在其它情况下,如上所述,优选使用两种不同的2类-gRNA复合物分离所述靶区域,所述gRNA分别与第三序列和第四序列至少部分地互补。
在分离靶区域的本方法的上下文中,将所述包含靶区域的核酸分子与至少一种具有核酸外切酶活性的酶接触。所述至少一种具有核酸外切酶活性的酶对应于与制备方法相关的上述限定的任何实施方案。之前关于制备方法描述的任何酶也可用于分离靶区域的本方法中。优选地,所述至少一种具有核酸外切酶活性的酶是λ核酸外切酶、核酸外切酶I(ExoI)、核酸外切酶III(Exo III)、核酸外切酶T、T5核酸外切酶、T7核酸外切酶、S1核酸酶、RecBCD核酸酶、RecJf、RNase D、RNase R核糖核酸外切酶I、核糖核酸外切酶II、优选其两个或更多的组合。使用单一核酸外切酶(例如,λ核酸外切酶或Exo III)是有利的,因为除了未受保护的分子降解之外,也可以同时制备核酸靶区域的末端用于下游应用,其可以例如需要存在5’单链突出(例如,由Exo III产生)或3’单链突出(例如,由λ核酸外切酶产生)。因此,根据具体的实施方案,所述至少一种具有核酸外切酶活性的酶是λ核酸外切酶或ExoIII。在可选择的实施方案中,可以使用特异性降解双链DNA的核酸外切酶,例如选自上面列出的那些。在可选择的实施方案中,可以使用特异性降解双链DNA和单链DNA的核酸外切酶的组合,例如选自上面列出的那些的核酸外切酶组合。
在分离靶区域的本方法的背景下,然后回收所述核酸靶区域。可以根据之前关于制备方法描述的任何实施方案来进行回收。具体地,可以通过将核酸分子与至少一种蛋白酶接触来回收靶区域。优选地,蛋白酶可以选自丝氨酸蛋白酶、半胱氨酸蛋白酶、苏氨酸蛋白酶、天冬氨酸蛋白酶、谷氨酸蛋白酶、金属蛋白酶和/或天冬酰胺肽裂解酶。
此外,或可选择地,通过使核酸分靶区域与能够螯合二价阳离子(特别是Mg
此外,可选择地,通过使核酸分子与至少一种RNase(例如,RNaseA、RNaseH、或RNaseI)接触,可以回收靶区域。在另一个实施方案中,因为RNA在升高的温度中不稳定,可以加热样本(例如,至少65℃),任选地在二价金属离子和/或碱性pH下存在。
在一些实施方案中,例如,使用顺磁珠进一步纯化步骤b)、c)或e)的靶区域。
根据具体实施方案,根据本发明的分离核酸靶区域的方法包括以下步骤:
a)使核酸分子的群体与以下接触:
-第一2类V型Cas蛋白-gRNA复合物,其中gRNA包含与核酸分子内的第一序列互补的向导段,所述第一序列毗邻所述靶区域,从而形成2类V型Cas蛋白-gRNA-核酸复合物,以及
-第一保护剂分子,其中所述保护剂分子优选是发夹衔接子或位点特异性核酸内切酶,所述位点特异性核酸内切酶更优选为TALEN、锌指蛋白或2类Cas蛋白-gRNA复合物,甚至更优选是2类V型或2类II型Cas蛋白-gRNA复合物,其中gRNA包括与核酸分子内的第二序列互补的向导段,所述第二序列毗邻所述靶区域并且其中所述第一序列和所述保护剂分子(或所述第二序列)位于所述靶区域侧翼,
b)使核酸分子的群体与至少一种具有核酸外切酶活性的酶接触,
c)在步骤a)中形成的至少2类V型Cas蛋白-gRNA-核酸复合物中回收所述包含靶区域的核酸分子,
d)将步骤c)的核酸分子与进行性聚合酶接触,
e)将步骤d)的核酸分子与2类Cas蛋白-gRNA复合物接触,其中所述gRNA包括与第三序列互补的向导段,所述第三序列在所述第一序列和所述保护剂分子之间形成,从而形成2类Cas蛋白-gRNA-核酸复合物,
f)任选地,使步骤d)的所述核酸分子与保护剂分子接触,所述保护剂分子优选是2类Cas蛋白-gRNA复合物,其中所述gRNA包括与第四序列互补的向导段,所述第四序列位于第一序列和第二序列之间,从而形成2类Cas蛋白-gRNA-核酸复合物,
g)使核酸分子与至少一种具有核酸外切酶活性的酶接触,以及
h)从至少2类Cas蛋白-gRNA-核酸复合物中回收分离的核酸靶区域。
在上述方法中,所述第三和第四序列包含在靶区域中,更优选地,所述第三和第四序列位于所述靶区域的端部,使得所述第三和第四2类Cas蛋白-gRNA复合物在所述靶区域的端部形成Cas蛋白-gRNA-核酸复合物,从而整体保护所述靶区域。在上述方法中,步骤e)和f)可以以任何顺序有序地或同时进行。
根据具体实施方案,根据本发明的分离核酸靶区域的方法包括以下步骤:
a)使核酸分子的群体与以下接触:
-Cpf1-gRNA复合物,其中gRNA包含与核酸分子内的第一序列互补的向导段,从而形成Cpf1蛋白-gRNA-核酸复合物,所述第一序列毗邻所述靶区域,以及
-第二Cpf1-gRNA复合物,其中gRNA包含与核酸分子内的第二序列互补的向导段,从而形成第二Cpf1蛋白-gRNA-核酸复合物,所述第二序列毗邻所述靶区域,其中所述第一序列和所述第二序列位于所述靶区域侧翼,
b)使核酸分子的群体与至少一种具有核酸外切酶活性的酶接触,
c)在步骤a)中形成的Cpf1蛋白-gRNA-核酸复合物中回收所述包含靶区域的核酸分子,
d)将步骤c)的核酸分子与进行性聚合酶接触,
e)将步骤d)的核酸分子与Cas9-gRNA复合物接触,其中所述gRNA包含与第三序列互补的向导段,从而形成Cas9-gRNA-核酸复合物,所述第三序列位于所述第一序列和所述第二序列之间,以及任选地,使所述核酸分子与第二Cas9-gRNA复合物接触,其中所述gRNA包含与第四序列互补的向导段,从而形成Cas9-gRNA-核酸复合物,所述第四序列位于第一序列和第二序列之间,其中所述第三和所述第四序列位于所述靶区域的端部,
f)使步骤e)中形成的核酸分子与至少一种具有核酸外切酶活性的酶接触,以及
g)从步骤e)中形成的Cas9蛋白-gRNA-核酸复合物中回收分离的核酸靶区域。
在该方法中,Cpf1优选是具有催化活性的。在该方法中,Cas9优选是催化失活的。优选地,步骤a)至g)以上面提供的顺序依序进行。
根据上述方法之一分离的核酸靶区域有利地是高度富集的。分离的核酸在各种应用中特别有用。事实上,根据本发明分离的核酸可以经受进一步的加工、反应或分析(其可以在或不在同一容器中发生)。作为一个示例,根据本发明分离的核酸可用于检测、克隆、测序、扩增、杂交、cDNA合成、诊断和任何技术人员已知的需要核酸的其它方法。在一些情况下,分离的核酸靶区域可以经历所述靶区域的进一步分离、或富集、或纯化的方法。
本发明方法特别适用于在分离一个或多个靶区域之后产生发夹文库,其中每个发夹包含至少一个核酸靶区域。因此,该方法特别方便用于检测或确定从整个核酸分子的群体(例如,在生物样本中)中分离出的目的靶区域的序列(例如,特定等位基因)。
根据本发明的优选方面,本发明的方法可以进一步包括附加步骤。作为非限制性示例,可以使用公知的纯化方法(例如,珠或柱纯化,例如用顺磁珠纯化)进一步纯化分离的核酸以除去蛋白,例如2类Cas蛋白、盐、蛋白酶、EDTA、过量的寡核苷酸等。作为非限制性示例,通过合成互补链可以补平核酸分子中的单链空位,核酸分子可以杂交至和/或连接至靶区域,和/或可以进行链取代(displacement)。这些附加的步骤对于产生发夹文库特别有用,而且在制备用于其它下游应用的分离的核酸时也可以是必要的。在一个特定的示例中,当根据本发明的方法分离一个或多个双链核酸分子,如前所限定,然后可以将发夹分子连接到所述分子的一个或两个游离端。优选地,将发夹连接到分离的靶核酸分子的一个游离端(参见图4)。优选地,所述分离的靶核酸分子的至少一个游离端包含3'或5'突出。优选地,所述发夹包含3'或5'突出,其分别至少部分地与所述分离的靶核酸分子中的5'或3'突出中的至少一个互补。优选地,所述发夹在分离的靶核酸分子的一端连接到3'突出。作为可选择的示例,在切割5'DNA侧翼(flap)的FEN1酶的存在下,所述发夹有利地连接到3'突出。事实上,发明人发现,在具有催化活性的Cpf1用于制备待分离的片段的情况下,在FEN1存在下,将发夹连接到3’突出促进切割在寡核苷酸的5'端存在的凸出的(protruding)核苷酸。事实上,与Cas9相比,Cpf1并不总是在相同位置切割。在连接所述发夹之后,使用本领域熟知的方法进行空位填充和连接反应。
因此,根据第一实施方案,本发明的方法还包括以下步骤:
·将一种或多种单链或双链核酸分子杂交至和/或连接至分离的核酸靶区域。
优选地,所述单链或双链核酸分子杂交至靶区域的5’-或3’-突出。在杂交之后,优选进行连接。在具体实施方案中,该方法可以包括以下步骤:
·将至少一种单链核酸分子杂交到分离的靶区域,以及
·将所述延伸的单链核酸分子连接到双链区域。
然而,当单链核酸分子(例如,寡核苷酸)与直接邻接双链区域的靶的单链区域结合,也可以直接进行连接而不进行杂交。
由于2类Cas蛋白保护的核苷酸的数量可以变化,所以所述单链核酸分子优选与位于距双链区域至少50个核苷酸远的分离的核酸靶区域的单链区域杂交。
根据另一实施方案,该方法包括以下步骤:
·将至少一种单链核酸分子杂交至分离的靶区域,
·将单链核酸分子延伸到双链区域,优选通过使所述分离的靶区域与核酸聚合酶接触,以及
·将所述延伸的单链核酸分子连接到双链区域。
根据优选的实施方案,至少一种单链核酸分子在3’突出上杂交并聚合。优选地,当使用2类Cas蛋白切口酶或催化失活的2类Cas蛋白时,更优选催化失活的2类Cas蛋白,甚至更优选Cas9d,与5’端发生连接。优选地,所述单链核酸分子与距离PAM至少50个核苷酸远的区域杂交。杂交、延伸和连接的方法对于技术人员而言是公知的。
在一些情况下,可以重复任何上述实施方案,例如将第二单链核酸分子添加到分离的靶区域中。所述第二单链核酸分子可以与相同的链或相对的链杂交,并且可以包含或不包含标记。所述单链核酸分子可以仅部分地与分离的靶区域的序列互补。所述单链核酸分子可以优选包含间隔子区域(例如,12-碳间隔子),其不与分离的靶区域结合(例如,不与分离的靶区域的序列互补)。优选地,单链核酸分子包含用于连接的5'磷酸基团。
任选地,然后可以消除过量的试剂,例如未杂交的单链核酸分子。作为一个示例,通过使包含分离的靶区域的样本与具有3'至5-核酸外切酶活性的酶(更优选核酸外切酶I)接触,可以消除未杂交的单链核酸分子。
根据优选的实施方案,在将单链核酸分子与靶区域杂交之后,本发明的方法还包括以下步骤:
·在分离的靶区域进行链取代。
链取代的方法是本领域已知的。这有利地允许回收靶区域,其中所述靶区域包括短的5'突出。优选地,所述5'突出的长度对应于受Cas9保护的序列的长度,更优选地,所述突出是23至25个核苷酸长。然后,可以将具有5'-突出的分离的靶区域用作模板以杂交和连接寡核苷酸,例如,用于构建发夹结构。优选通过将分离的靶区域与寡核苷酸以及任选地聚合酶,优选在室温下孵育进行链取代。根据具体实施方案,可以在RecA的存在下进行链取代。
优选地,当2类Cas蛋白切口酶已经切开靶区域的一条链时,进行链取代。
在链取代后,可以消除过量的单链核酸分子和链取代产物。因此,根据优选的实施方案,该方法还包括以下步骤:
·消除过量的单链核酸分子和链取代产物。
根据优选的实施方案,通过使靶区域与具有3’至5-核酸外切酶活性的酶接触,更优选为核酸外切酶I以消除所述过量的单链核酸分子和链取代产物。有利的是,特定地消除过量的单链核酸分子和链取代产物,而对双链靶区域或对5’突出没有影响。
根据优选的实施方案,然后可以将一种或多种单链核酸分子杂交并连接到靶区域的5'-突出。优选地,在链取代、以及任选地在消除过量的单链核酸分子和链取代产物之后,发生连接到5'-突出。这有利地产生发夹结构,其特别适用于下游应用,例如WO2011/147931、WO2011/147929、WO2013/093005、和WO2014/114687中描述的那些,通过引用整体并入本文。或者,这里产生的发夹结构特别适合用作发夹前体分子(例如,WO2016/177808中描述的HP2分子,通过引用整体并入本文)。
优选地,任何上述实施方案的一种或多种单链核酸分子具有优化的杂交特异性,如Zhang等人,Nat Chem,2012,4(3):208-214所述,通过引用整体并入本文。或者,所述任何上述实施方案的一种或多种单链核酸分子可以是简并的。
优选地,任何上述实施方案的一种或多种单链核酸分子包含标记。作为非限制性示例,标记可以是FITC、地高辛、生物素、或技术人员已知的任何其它标记。使用例如化学偶联和化学交联剂技术,所述标记可以与蛋白缀合。有利地,可以检测所述靶区域,以及任选地在样本中量化所述靶区域,例如通过荧光标记或技术人员已知其它可检测标记。在一些情况下,可以使用所述标记进一步分离或纯化靶区域。在第一方面,根据本领域技术人员已知的方法,例如当用生物素标记寡核苷酸时,在链霉亲和素包被的珠上通过下拉(pull-down)反应可以分离靶区域。在第二方面,靶区域可以通过所述标记附着到支持物上,例如珠或芯片。优选地,所述支持物进行官能化以促进标记的靶区域附着,所述标记与支持物上存在的官能团反应(例如,与适当的标记反应的支持物可以包被有链霉亲和素或COOH基团)。
根据具体实施方案,任何上述实施方案的单链核酸分子中的至少一种包含与结合到表面的寡核苷酸互补的序列。优选地,所述寡核苷酸在其3'端包含修饰以防止延伸。无论有或没有标签,单链核酸分子杂交和连接至3'突出有利地产生了一种发夹结构,其特别适用于下游应用,例如WO2011/147931、WO2011/147929、WO2013/093005和WO2014/114687中描述的那些。优选地,上述任何实施方案产生具有“Y”形状的发夹。
本发明还允许技术人员枚举携带所述序列的核酸分子的数量。根据优选的实施方案,本发明的方法还包括检测和定量如WO2013/093005中所述的核酸分子。
本发明的分离的靶区域特别适用于通过以下进行下游分析:单分子分析方法(例如,WO2011/147931和WO2011/147929中描述的方法)、核酸检测和量化方法(例如,WO2013/093005所描述的方法)、以及检测与核酸结合的蛋白的方法(如WO2014/114687所述)。因此,本方法的进一步实施方案和应用可以在这些应用中找到,其通过引用整体并入本文。
根据本发明优选的实施方案,该方法包括富集在分离的靶区域内包含的SNP或遗传嵌合。SNP或遗传嵌合优选包含在2类Cas蛋白-gRNA复合物中的gRNA所识别的序列中。优选地,gRNA包含与SNP的次要等位基因对应的核苷酸碱基,从而允许保护包含所述次要等位基因的靶区域。当SNP的多个等位基因存在于给定基因座上时,可以提供多个gRNA分子,其对应于每个等位基因,优选对应于每个次要的等位基因。在提供与主要和次要等位基因对应的gRNA分子的情况下,可以量化包含每种等位基因的分离的靶区域的数量,例如以确定受试者是否是SNP基因座的纯合子或杂合子。优选地,与SNP基因座对应的碱基位于gRNA序列内相对于PAM位点的碱基-1至-10,优选-1至-6,优选-4、-5、或-6中的任何一个。事实上,当在这些碱基中的一个或多个中发生错配时,减少或消除保护核酸区域免于核酸外切酶消化。该位置特别有利,因为可以通过减少的误差可能性来确定SNP的存在或不存在。在一些情况下,进一步测序靶核酸区域以确定SNP基因座的等位基因。当使用在SNP基因座上包含简并碱基的gRNA时,或鉴定靶区域内的相邻SNP基因座中可存在的等位基因时,可以特别进行这一点。事实上,如本领域技术人员所公知的,在基因组中位置彼此接近的SNP倾向于一起遗传。
减少或消除对靶区域保护的程度将根据实验条件、使用的2类Cas蛋白、和/或使用的gRNA变化。例如,已知Cpf1具有比Cas9更大的结合特异性(Strohkendl等人,MolecularCell,2018,71:1-9)。因此,与使用Cpf1时相比,当使用Cas9时,对包含错配的区域的保护将更大。根据是否需要分离包含错配的区域,可以使用Cpf1蛋白或其具有优化的结合特异性的变体、或具有增加的结合特异性的突变的Cas9蛋白,例如本文所述的那些。
根据本发明的优选的实施方案,该方法还可包括测序分离的靶区域。本领域可获得有许多测序方法。本发明的方法特别适用于产生用于单分子测序方法的发夹,例如,WO2011/147931或WO2011/147929中描述的那些。分离的核酸可以进一步用作以下的模板:特异性或非特异性聚合酶链式反应、等温扩增、例如环介导的等温扩增、链取代扩增、螺旋酶依赖性扩增、切口酶扩增反应、逆转录、酶促消化、核苷酸掺入、寡核苷酸连接和/或链侵袭。分离的核酸也可用作用于测序的底物,例如桑格双脱氧测序或链终止反应、全基因组测序、基于杂交的测序、焦磷酸测序、毛细管电泳、循环测序、单碱基延伸、固相测序、高通量测序、大规模平行特征测序、纳米孔测序、透射电子显微镜测序、光学测序、质谱、454测序、可逆终止子测序、“配对末端”或“配对”测序、核酸外切酶测序、连接测序(例如,SOLiD技术)、短读取测序、单分子测序、化学降解测序、合成测序、大规模平行测序、实时测序,半导体离子测序(例如,Ion Torrent)、双端双标签的多重测序(MS-PET)、液滴微流体测序、部分测序、片段作图,以及任何这些方法的组合。
根据优选的实施方案,本发明的方法还包括通过单分子测序、下一代测序、部分测序、或片段作图的方式,更优选通过单分子测序的方式(例如,WO2011/147931或WO2011/147929所描述的)来测序靶区域。根据本发明的优选实施方案,该方法还可包括检测蛋白与特异性核酸序列的结合。用于检测蛋白结合的各种方法是技术人员可以获得的。本发明的方法特别适用于产生用于使用单分子的蛋白结合方法的发夹,例如,WO2014/114687中描述的。分离的靶区域可以进一步用作检测蛋白与核酸结合的底物,例如用于检测表观遗传修饰的底物。分离的靶区域可以用在以下:例如,亚硫酸氢盐转化、高分辨率熔解分析、免疫沉淀(例如ChIP、enChIP)、微阵列杂交和技术人员公知的核酸/蛋白相互作用的其它分析。本文所用的术语“表观遗传修饰”,是指在合成核酸分子之后发生的构成所述核酸分子的碱基的修饰。作为非限制性示例,碱基修饰可能是由对所述碱基损坏所产生的。表观遗传修饰包括,例如,尤其在DNA中的3-甲基胞嘧啶(3mC)、4-甲基胞嘧啶(4mC)、5-甲基胞嘧啶(5mC)、5-羟甲基胞嘧啶(5hmC)、5-甲酰胞嘧啶(5fC)和5-羧基胞嘧啶(5caC)、以及6-甲基腺苷(m6A)),RNA中的5-羟甲基尿嘧啶(5hmU)和假尿苷,以及DNA和RNA中的3-甲基胞嘧啶(3mC)和N6-甲基腺苷(m6A)。
同样,该方法可以进一步包括检测由核酸损伤(例如,DNA损伤)所产生的修饰碱基。由于化学物质(即嵌入剂)、辐射和其它诱变剂可能在分离的核酸上进行,DNA损伤常常发生。由这些类型的DNA损伤引起的DNA碱基修饰广泛存在,并在影响生理状态和疾病表型方面发挥重要作用。示例包括8-氧代鸟嘌呤、8-氧代腺嘌呤(氧化损伤;衰老、阿尔茨海默病、帕金森病)、1-甲基腺嘌呤、6-O-甲基鸟嘌呤(烷基化;胶质瘤和结直肠癌)、苯并[a]芘二醇环氧化物(BPDE)、嘧啶二聚体(加合物形成;吸烟、工业化学品暴露、紫外线暴露;肺癌和皮肤癌)和5-羟基胞嘧啶、5-羟基尿嘧啶、5-羟基甲基尿嘧啶和胸腺嘧啶乙二醇(电离辐射损伤;慢性炎症疾病、前列腺癌、乳腺癌和结直肠癌)。
优选地,如WO2014/114687所述,本发明的方法还包括检测蛋白与特异性核酸序列的结合。
本发明的另一主题是试剂盒,其可用于根据本文所述的本发明的任何方法或实施方案的核酸分离和富集。试剂盒将提供用于制备包含靶区域的核酸的材料,以及任选地,根据本文所述的方法分离所述靶区域的材料。根据本文所述的任何方式,可以根据待使用的2类V型Cas蛋白(例如,Cpf1、C2c1)、所用的保护剂分子(例如,发夹或位点特异性核酸内切酶)、靶向的核酸区域等等,内容可有所不同。
根据具体实施方案,本发明的试剂盒包括:
a)2类V型Cas蛋白,优选具有催化活性的Cpf1,
b)2类Cas蛋白,优选Cas9d,
c)至少两种gRNA,更优选至少三、四、五、六、10种gRNA,每种所述gRNA与特异性核酸序列互补,
d)至少一种具有核酸外切酶活性的酶,
e)任选地,一种进行性聚合酶,
f)任选地,至少一种蛋白酶,以及
g)任选地,使用说明。
根据另一实施方案,所述试剂盒包括EDTA,优选EDTA溶液,以取代至少一种蛋白酶或者除此之外还有至少一种蛋白酶。
根据一个具体实施方案,所述试剂盒包含每种靶区域的四种gRNA,其中两种所述gRNA与位于所述靶区域侧翼的序列互补,两种所述gRNA与位于靶区域的端部的序列互补。因此,位于靶区域的端部的序列嵌套在位于所述靶区域侧翼的序列之间。在需要下游多重分析的情况下,试剂盒可包含两种或更多种2类Cas蛋白以及两种或更多种gRNA,从而靶向至少两种不同的靶区域。在一些情况下,在试剂盒中包含的所述2类Cas蛋白的至少一种与gRNA预先装载,形成2类Cas蛋白-gRNA复合物。根据具体实施方案,当试剂盒包含用于制备包含靶区域的核酸分子的多种2类Cas蛋白-gRNA复合物(例如,多种2类V型Cas蛋白-gRNA复合物)时,所述复合物优选在单一容器中混合在一起。类似地,当试剂盒包含用于分离靶区域的多种2类Cas蛋白-gRNA复合物时,所述复合物优选在单一容器中混合在一起。然而,用于制备包含靶区域的核酸分子的2类Cas蛋白-gRNA复合物与用于下游使用以分离靶区域的2类Cas蛋白-gRNA复合物并不混合。优选地,为方便使用,已经预先确定在所述试剂盒中包含的每种2类Cas蛋白-gRNA复合物的比率。
优选地,gRNA的向导段与靶区域自身互补或与靶区域内的序列互补,该靶区域是临床诊断或遗传风险评估所感兴趣的。作为一个示例,所述gRNA与septin9(SEPT9)或表皮生长因子受体(EGFR)的编码区下游的非编码靶区域互补。事实上,已知这些区域的表观遗传状态对癌症结果很重要。作为另一示例,所述gRNA与位于编码脆性X智力低下1(FMR1)的序列(其涉及脆性X综合征)下游的靶区域互补。该基因中5'-CGG-3'重复的拷贝数的突变是疾病的原因。该CpG岛的上游区域的表观遗传状态(例如,甲基化)已知还与疾病的临床严重程度有关。作为另一示例,所述gRNA与DM1蛋白激酶(DMPK)的编码区域中的靶区域互补。事实上,5'CTG-3'重复数量的扩大是肌营养不良1型的特征。作为另一示例,所述gRNA与包含一种或多种cfDNA分子的靶区域互补。事实上,分离特异性cfDNA(例如,cffDNA或ctDNA),对于以下特别有用:包括产前检验的各种下游应用(参见,例如Gahan,Int J WomensHealth.2013,5:177-186)和癌症诊断和/或监测(参见,例如Ghorbian和Ardekani,Avicenna J Med Biotech.2012,4(1):3-13)。可以有利地从生物样本(例如,血浆、血清、或尿液样本)中直接分离cfDNA内包含的一种或多种cfDNA或靶区域。
本文所述的试剂盒优选能够分离至少两种不同的靶区域。事实上,某些表观遗传癌症诊断检验的值已经通过多路复用证明得到改善,其中在单一检验中分析两种或更多种不同靶区域的序列或结构的特性(例如甲基化状态)。作为非限制性示例,本文提供的试剂盒能够分离靶区域,所述靶区域包含或由以下组成:根据本文所述的任一方法,经历DNA甲基化的人GSTP1、APC和/或RASSF1基因或其合适区域。然后,可以对所述分离的靶区域进行甲基化状态的下游分析,例如根据本文提供的方法(例如,如WO2014/114687所提供)。这种试剂盒特别有利于确定受试者发展出前列腺癌的风险((Wojno等人,American health&drug benefits,2014,7(3):129),并且优于在PCR之后显著使用亚硫酸氢盐处理样本DNA的现有试剂盒。与本发明的方法相比,用现有试剂盒分离的核酸可特别容易出现假阳性和假阴性信号,以及由于粗糙和低效的化学处理导致的样本损失。根据具体实施方案,试剂盒优选包含每种靶区域的至少两种gRNA,所述gRNA与位于如本文所定义的人类基因GSTP1、APC和RASSF1的侧翼的位点互补。
作为另一非限制性示例,本发明的试剂盒能够制备和分离至少一个位于以下位置的人类基因组内的以下靶区域:在染色体17上的65676359-65676418、在染色体9上的21958446-21958585、在染色体6上的336844-336903、在染色体21上的33319507-33319636、在染色体6上的166502151-166502220、在染色体18上的896902-897031、在染色体5上的32747873-32748022、在染色体6上的27949195-27949264、在染色体7上的27191603-27191672、在染色体16上的170170302-170170361、在染色体15上的30797737-30797876、在染色体1上的7936767-7936866、在染色体1上的170077565-170077634、在染色体2上的1727592-1727661、在染色体8上的72919092-72919231,优选所有15个靶区域。因为所述靶区域的DNA甲基化状态的下游分析可用于检测膀胱癌,所以分离所述靶区域是有利的。现有试剂盒使用甲基化敏感限制性酶,然后用PCR鉴定甲基化序列,因此可以被通过靶区域中适当的限制性位点的存在、复杂的检验设计和限制的灵敏度进行限制。因此,用于分离和检测膀胱癌的改进的试剂盒可以优选包含两种gRNA和两种附加的gRNA,所述两种gRNA至少部分地与位于所述靶区域侧翼的序列互补,以及所述两种附加的gRNA至少部分地与位于所述靶区域的端部的序列互补,用于根据本文所述的方法分离这些15个靶区域中的每一个,优选用于分离所有15个靶区域。
作为非限制性示例,靶区域可包括或不包括特异性序列、特异性数目的序列重复、一种或多种核苷酸碱基修饰。作为另一非限制性示例,靶向分离的区域可以是特异性长度或与所述特异性长度不同的长度。优选地,本发明的试剂盒还包含至少一种限制性酶和/或RNase。优选地,试剂盒还包含适当的2类V型Cas蛋白反应缓冲液和适当的2类II类Cas蛋白反应缓冲液,例如下面实施例中详述的那些。
试剂盒可以进一步包含附加元件,适用于指定应用。例如,试剂盒可以进一步包含一种或多种保护剂分子,优选发夹衔接子或位点特异性核酸内切酶、连接酶和/或聚合酶、寡核苷酸、dNTP、适当的缓冲液等。
下面的附图和实施例中示出本发明的附加特征和有利方面。
附图说明
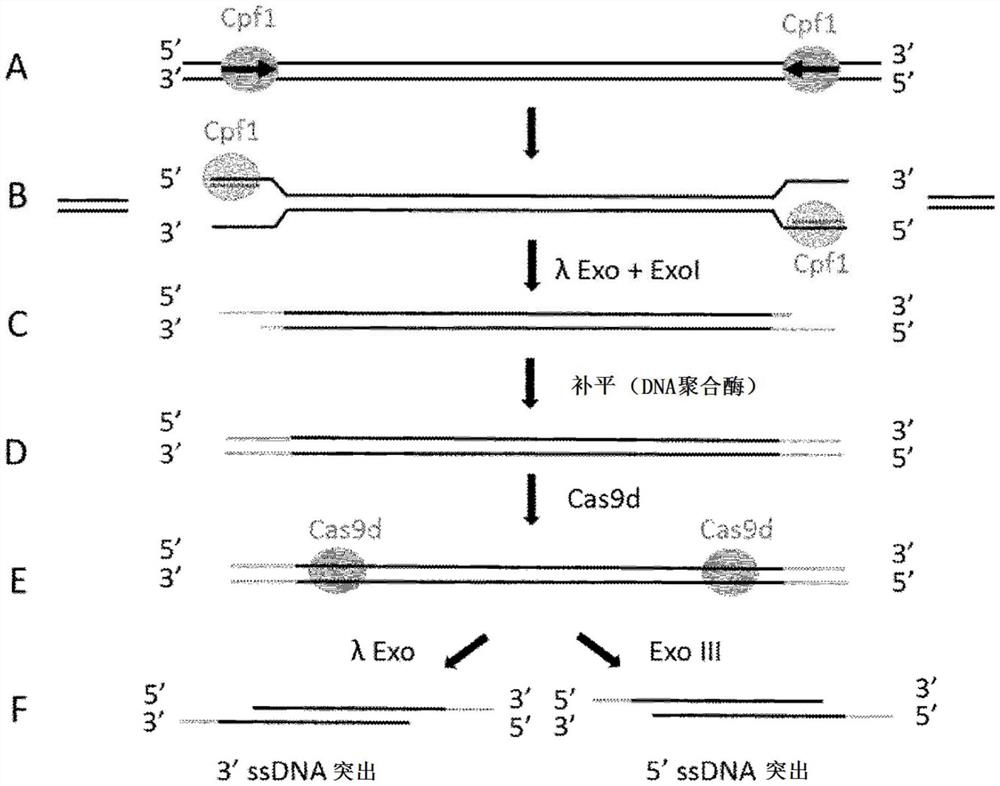
图1.图式说明本发明的方法:使用两种2型V类Cas蛋白-gRNA复合物,之后依次使用两种嵌套的2型II类Cas蛋白-gRNA复合物。(A)包含靶区域的DNA(片段化或不片段化)首先与两种Cpf1-gRNA复合物接触,所述两种Cpf1-gRNA复合物结合位于待分离的靶区域的侧翼的第一和第二序列。设计Cpf1向导,使得Cpf1 PAM序列位于包含靶区域的核酸分子内(如向内指的箭头所示)。(B)不受理论限制,不包含PAM序列的链可以在切割发生后从复合物中解离。(C)向反应管补充核酸外切酶(在该具体情况下,λ核酸外切酶(λexo)以及核酸外切酶I(exoI))的混合物,其将降解不受保护的核酸分子。降解可以是部分或完全的。在该步骤中可以有利地添加核酸内切酶(例如,限制性酶),以产生更多的末端用于核酸外切酶降解。(D)所得片段含有由Cpf1和exoI两者处理产生的5'突出,该5'突出应该被修复(末端优选被补平),以促进5’链的下游λexo降解。这是使用嗜温DNA聚合酶(更优选T4 DNA聚合酶)或嗜热聚合酶来完成的。(E)片段(或片段群体)与每个靶区域的2种Cas9d-gRNA复合物接触,所述Cas9d-gRNA复合物结合位于靶区域的每个末端的第三和第四序列,优选地距离Cpf1-gRNA复合物靶向的起始序列至少100个碱基。(F)将所述复合物装载到片段上后,可以向反应补充λexo以获得含有3'突出的片段(在F中为左侧分子)或核酸外切酶III以在靶区域的两末端产生5'突出(在F中为右侧分子)。
图2.本发明方法的具体示例:使用两种2型V类Cas蛋白-gRNA复合物,使用或不使用末端修复步骤,之后使用两种嵌套的2型II类Cas蛋白-gRNA复合物。(A)为证明末端修复步骤在产生具有用3'突出的适当片段时的效力,我们生成包含靶区域Sept9.2的PCR片段(步骤(i)、(ii);预期的片段尺寸:1972bp)。我们将PCR片段与Cpf1-crRNA复合物(其结合到位于靶区域的侧翼的序列)一起孵育,并使用λexo以及exoI处理反应(步骤(iii);预期片段的尺寸:1050bp)。然后将反应物分成两管,将其用末端修复(End Repair)处理(步骤(iv);预期的片段尺寸:1100bp)或不用末端修复处理(步骤(vii))。然后将该靶相应的Cas9d-gRNA加入两个管中(步骤(v)或(viii))以及加入λexo和exoI(步骤(vi)或(ix))。如果该片段是λexo的合适底物,则所得片段的尺寸将对应于由两个Cas9d-gRNA复合物保护的片段,包括dCas9结合的第三和第四序列(步骤(vi))。如果不是这种情况,则λexo将从长的5'突出无效(ineffectivly)起始消化,并将导致产生多个不同尺寸的片段(步骤(ix))。(B)在(A)中描述的实验的结果。泳道1对应于PCR产物Sept9.2。泳道2对应于Cpf1和核酸外切酶处理后的片段(步骤(iii)的片段)。泳道3表示末端修复(步骤(iv))后的片段。由于末端被修复,因此所述片段比步骤(iii)的片段迁移稍慢,并且目的片段的尺寸增加42bp。泳道4对应于当没有末端修复时在核酸外切酶处理后产生的步骤(ix)的部分片段。泳道5对应于步骤(vi)的想要片段,其中在使用两种Cas9d-gRNA复合物和核酸外切酶处理的分离之后,进行末端修复(分离步骤均被称为“dCas9”;预期的片段尺寸:750bp)。通过与泳道1-5左侧的分子量标记物进行比较确定尺寸。因为没有其它片段存在,末端修复允许将制备的包含靶区域的核酸完全转化成分离的靶区域。
图3.使用大肠杆菌基因组DNA作为起始材料,比较末端修复在产生预期的片段中的效力。(A)在没有末端修复步骤的情况下获得的结果。从大肠杆菌基因组DNA中选择3种不同尺寸的靶(靶#1、#2和oriC)。与这些靶相对应的Cpf1-gRNA首先与基因组DNA一起孵育,然后用λexo以及exoI处理。在此处理之后,将反应使用High Sensitivity NGS FragmentAnalysis Kit上样到Fragment
图4.图式表示从图1所示方法中获得的分离的靶区域用于生成发夹的方法。(i)的包含3'突出的片段与每种靶的3种不同的寡核苷酸杂交,如(ii)所示:1.含有结合部分(例如生物素)的寡核苷酸,其杂交在距Cas9 PAM序列至少50个碱基处。2.表面寡核苷酸在其3’末端含有磷酸以防止聚合,以及其对应于Cpf1靶序列。这两种寡核苷酸均杂交到相同3'突出。3.与相对的3'突出杂交的第三寡核苷酸以产生用于连接环的限制性位点((ii))。在使用DNA聚合酶(优选Bst全长DNA聚合酶)补平后,该片段对应于(iii)中所示。用非回文限制性酶BsaI消化该片段(该位点由表面和环寡核苷酸产生)以允许表面寡核苷酸和环的定向连接((iv)所示的最终产品)。
图5.分析从大肠杆菌基因组DNA产生的“靶#1”片段。(A)使用与靶#1对应的寡核苷酸CAAG的典型鉴定痕迹(左图)。产生对应于实验位阻(blockage)的直方图(右图)。基于该直方图,成功鉴定与该寡核苷酸对应的位阻,如图下表格所总结。预期的碱基数目显示在组织图谱的右轴以及表格(“预期”)中。(B)还在(A)中所述相同分子上检验对表观遗传修饰m
图6.分析从大肠杆菌基因组DNA产生的“靶#2”片段。直方图/表如之前图5所示。
图7.分析从大肠杆菌基因组DNA产生的“DAM”片段。直方图/表如之前图5所示。
图8.分析从大肠杆菌基因组DNA产生的“oriC”片段。直方图/表如之前图5所示。
图9.估计靶向四个大肠杆菌区域的效力改善及其在SIMDEQ
图10.从人类基因组DNA制备含15种不同靶的测序文库。选择靶区域以富集具有相应基因组坐标。选择这些区域要么是由于表观遗传生物标志物的存在,要么是由于与人类疾病相关的具有扩展重复的已知基因座的存在。
图11.如图10中列出的从含有15种不同靶的人类基因组DNA上制备的文库中进行Illumina测序的示例结果。屏幕截图表示Sept9.2富集的区域(由黑色矩形描绘)。以浅灰色显示与该区域比对的读数。该区域显示出高覆盖率(以深灰色显示),而正如预期的那样周围区域显示很少(如果有任何读数的话),表明良好的富集。
图12.分析FMR1基因座的重复长度。使用10种特异性的8碱基寡核苷酸分析STR(短串联重复)尺寸,该特异性的8碱基寡核苷酸特异性结合位于CGG重复位置下游和上游的序列。然后,通过找到理论结合和实验阻断位置之间的最佳拟合来确定估计的重复次数。四种DNA样本(HEK293细胞DNA和两种临床样本、NA06896和NA07537)的FMR1的CGG重复长度(“重复尺寸”)分布的直方图。n代表在以下三个类别中的每种所鉴定的分子数:正常(<50个重复)、前突变(50至200个重复之间)和完全突变(>200个重复)。
图13.制备的各种文库的概述和从这些样本中获得的结果。
图14.FMR1启动子的甲基化状态分析。对于一种DNA样本(NA06896-重复1),分析位于FMR1的启动子区域内的CpG岛区域的甲基化状态。对于相同分子而言,对所有预测的CpG和非CpG位点的CGG重复的尺寸和甲基化状态两者进行表征。用于碱基修饰检测,抗-m
图15.FMR1基因座上的CpG和非CpG位点。NA06896DNA样本的分子上所有位置和甲基化比率的列表。
具体实施方式
实施例
纳入以下实施例以证明本发明优选的实施方案。在以下实施例和附图中列出或所示的所有主题,用于阐释说明,而非限制意义。以下实施例包括可以由本领域技术人员确定的任何替代、等同和修改。
实施例1:选择gRNA的方法
对于以下描述的所有策略,使用可获得的在线工具设计一种或多种向导RNA。然后,可以使用病毒转录系统(例如,T7、SP6或T3RNA聚合酶等)体外合成或使用自动合成仪化学生产RNA向导作为单一向导,或者作为两部分向导,所述两部分向导由crRNA(其含有与靶区域互补的序列)和不变的tracrRNA(其含有与crRNA互补的区域以及引入酶活性所需特异性结构的序列)组成。使用野生型Cas核酸酶(例如,Cas9-gRNA、Cpf1-crRNA等)在标准化的/对照样本(例如,PCR片段)上在体外评估每种gRNA的效率。这是为确保每种Cas蛋白-gRNA复合物将以高效率切割(例如,至少80%的起始PCR片段被切割)。事实上,之前已经证明,虽然使用Cas酶的催化失活版本,然而靶片段的保护水平与野生型(WT)酶(与特异性gRNA结合)在体外切割DNA的能力直接相关。换句话说,即使使用Cas蛋白的催化失活版本,在给定序列上无效率切割的WT Cas蛋白-向导RNA复合物也将不能有效保护所述序列免于核酸外切酶消化。
在本实施例中,使用自动合成仪根据公共通用序列(SEQ ID NO:1)化学制造Cpf1向导RNA。如上所述,使用病毒转录系统根据通用序列(SEQ ID NO:4)体外合成、或使用自动合成仪化学生产Cas9向导RNA。在某些情况下,通用tracrRNA(SEQ ID NO:3)可以与靶特异性crRNA(在SEQ ID NO:2所示的通用序列)进行退火。
将gRNA在相应的缓冲液中在95℃中孵育5分钟,然后通过渐进式斜坡(ramp)在80℃、50℃、37℃和室温下在每个步骤进行10分钟用于退火和/或二级结构形成。
实施例2:一般反应操作方案
2.1
1.向导RNA(特别是当2类Cas蛋白属于V型时是crRNA、或当2类Cas蛋白属于II型是gRNA时),上样到2类Cas蛋白上,通过在室温(例如25℃)在适当的反应缓冲液中孵育10分钟,从而允许形成蛋白-RNA复合物。
对于V型Cas蛋白,反应缓冲液包含50mM NaCl、10mM Tris-HCl、10mM MgCl
2.将步骤1中制备的上样复合物加入到包含核酸分子的样本中,并在37℃孵育1小时,以允许2类V型Cas蛋白-gRNA复合物结合并切割包含靶区域的核酸。
3.加入核酸外切酶(例如,λ核酸外切酶和核酸外切酶I)的混合物,并将混合物在37℃中孵育1小时。然后,在75℃中15分钟使酶失活。
4.通过加入“终止缓冲液”(包含1.2单位的蛋白酶K和20mM EDTA的混合物)以终止反应,从而从靶区域中去除蛋白-gRNA复合物。在一些情况下,可以添加RNase A以消化gRNA。特别可以在37℃下连续进行15分钟的RNase A和蛋白酶K处理。在这种情况下,可以任选地添加EDTA。
5.将样本与DNA聚合酶一起孵育,所述DNA聚合酶修复凹进去的3'端。
2.2
1.如上所述,将催化失活的2类Cas蛋白与gRNA上样,通过在适当的Cas蛋白反应缓冲液中在室温(例如25℃)下孵育10分钟。
2.然后,将上样的复合物加入根据操作方案2.1制备的DNA中,并在37℃下孵育1小时以允许Cas蛋白-gRNA复合物与靶区域结合。
3.向反应中加入一种或多种核酸外切酶(例如,λ核酸外切酶),并将混合物在37℃中孵育1小时。然后,在75℃中15分钟使酶失活。
4.通过添加终止缓冲液(上述)来终止反应,从而从靶区域去除Cas蛋白-gRNA复合物。
然后,由λ核酸外切酶所产生的3'ssDNA突出可用作寡核苷酸的模板,以产生用于下游应用的特异性分子(例如,发夹或测序文库)。
实施例3:在PCR片段上的通用反应操作方案说明
作为上述操作方案的特定示例,根据制造商的说明,从SEPT9.2靶区域(SEQ IDNO:5)使用GXL PrimeStar Polymerase(Takara)与寡核苷酸PS1131和PS1132(分别是SEQID NO:6和7)产生2千碱基(kb)的PCR片段。将该片段分别与两种不同的Cpf1复合物在37℃在V型Cas蛋白反应缓冲液中孵育1小时,所述Cpf1复合物分别预装上Cpf1-SEPT9.2-crRNA#1或#2(分别为SEQ ID NO:8和9),以允许Cpf1蛋白-gRNA复合物结合位于靶区域侧翼的核酸序列并切割。然后,向反应补充λ核酸外切酶和核酸外切酶I,在37℃下达1小时以消化未受保护的DNA。用Cpf1切割后,以及不受理论束缚,可以从复合物中在PAM位点上释放远端3'链DNA,并且所述远端3'链DNA成为核酸外切酶消化的底物。通过添加终止缓冲液终止反应,并使用KAPA Pure珠(Roche Life technology)纯化核酸分子。然后,使用EndRepairReaction Mix(NEB)处理或(不处理)这些两个反应,以补平dsDNA/或修复可能已经产生的任何5'ssDNA末端。
在使用KAPA珠纯化后,将所得核酸分子与两种dCas9复合物在II型反应缓冲液中在37℃中孵育1小时,所述两种dCas9复合物分别预装有Cas9-SEPT9.2-gRNA#1或#2(分别为SEQ ID NO:10和11)。加入λ核酸外切酶和核酸外切酶I的混合物,以确定在末端修复之后靶的末端是否为λ核酸外切酶的更好底物。如果是这种情况,则预期所得到的3’ssDNA被核酸外切酶I消除,所得片段的长度应对应于由两种Cas9d-gRNA复合物保护的片段(如图2A所示,(vi));所述第三和第四序列位于所述靶区域的端部)。在当λ核酸外切酶不能从一个或两个5'突出起始消化的情况中,所得片段的长度将对应于由Cpf1-crRNA复合物所结合的第一和第二序列之间的距离、或对应于由Cpf1-crRNA复合物所结合的第一(或第二)序列和Cas9d-gRNA复合物所结合的第三(或第四)序列(如图2A,(ix)所绘)之间的距离。使用终止缓冲液终止反应,并在用High Sensitivity NGS Fragment Analysis Kit(1bp–6,000bp)在Fragment
实施例4:使用大肠杆菌基因组DNA的通用反应操作方案的说明
使用与实施例3中类似的操作方案,我们评估当使用大肠杆菌基因组DNA作为起始材料时修复DNA片段末端的效果。我们设计6种不同的Cpf1-crRNA复合物,其靶向3种不同区域(对于大肠杆菌靶#1(SEQ ID NO:12)是Cpf1-Ecoli#1-crRNA#1和#2(SEQ ID NO:13和14)、对于大肠杆菌靶#2(SEQ ID NO:15)是Cpf1-Ecoli#2-crRNA#1和#2(SEQ ID NO:16和17)、对于大肠杆菌靶oriC(SEQ ID NO:18)是Cpf1-oriC-crRNA#1和#2(SEQ ID NO:19和20)。将预加载的Cpf1-crRNA复合物与4μg大肠杆菌基因组DNA在37℃中孵育1小时。然后补充λ核酸外切酶和核酸外切酶I到反应中在37℃中1小时,以降解未受保护的DNA。通过加入终止缓冲液来终止反应并使用KAPA Pure珠纯化。为确定末端修复效率,将所得DNA分成2个管,用EndRepair Reaction Mix(NEB)处理或不用其处理。将两个反应与Cas9d复合物一起孵育,所述Cas9d复合物预装载有以下:对于大肠杆菌靶#1为Cas9-Ecoli#1-crRNA#1和#2(SEQ ID NO:21和22);对于大肠杆菌靶#2为Cas9-Ecoli#2-crRNA#1和#2(SEQ ID NO:23和24)、或者对于大肠杆菌靶oriC为Cas9-oriC-crRNA#1和#2(SEQ ID NO:25和26)。将反应在37℃中孵育1小时,然后加入λ核酸外切酶和核酸外切酶I。通过添加终止缓冲液终止反应,并使用KAPA Pure珠进行纯化。使用High Sensitivity NGS Fragment Analysis Kit(1bp–6,000bp)在Fragment
实施例5:来自分离的靶区域的发夹构建
将在实施例3中描述的操作方案使用在大肠杆菌基因组DNA中,其使用Cpf1-crRNA靶向4个不同区域,对于大肠杆菌靶#1(SEQ ID NO:12)是Cpf1-Ecoli#1-crRNA#1和#2(SEQID NO:13和14)、对于大肠杆菌靶#2(SEQ ID NO:15)是Cpf1-Ecoli#2-crRNA#1和#2(SEQ IDNO:16和17),对于大肠杆菌靶oriC(SEQ ID NO:18)是Cpf1-oriC-crRNA#1和#2(SEQ ID NO:19和20)和对于大肠杆菌靶dam(SEQ ID NO:27)是Cpf1-dam-crRNA#1和#2(SEQ ID NO:28和29)。将预加载的Cpf1-crRNA复合物与4μg大肠杆菌基因组DNA在37℃中孵育1小时。然后补充λ核酸外切酶和核酸外切酶I到反应中在37℃中1小时,以降解未受保护的DNA片段。通过加入终止缓冲液终止反应并使用KAPA Pure珠纯化。然后在使用KAPA Pure珠再次纯化样本之前,使用EndRepair reaction Mix(NEB)处理纯化的样本。将剩余的DNA与预加载的Cas9d在37℃中孵育1小时,所述Cas9d与以下复合:对于大肠杆菌靶#1为Cas9-Ecoli#1-crRNA#1和#2(SEQ ID No:21和22)、对于大肠杆菌靶#2为Cas9-Ecoli#2-crRNA#1和#2(SEQ ID NO:23和24)、对于大肠杆菌靶oriC为Cas9-oriC-crRNA#1和#2(SEQ ID NO:25和26)、以及对于大肠杆菌靶dam是Cas9-dam-crRNA#1和#2(SEQ ID NO:30和31)。然后掺入λ核酸外切酶用于在靶的每一侧产生3’-ssDNA突出。通过加入终止缓冲液终止反应并使用KAPA Pure珠纯化。加入与通过λ核酸外切酶消化产生的3’单链突出杂交的寡核苷酸,以产生想要的核酸结构。如图4所示,向反应中补充对每个靶区域的3种不同的寡核苷酸:1)首先,加入5'生物素化的寡核苷酸,所述寡核苷酸的3'端与距离PAM序列约50个碱基的序列互补(对于大肠杆菌靶#1为PS1150(SEQ ID NO:32)、对于大肠杆菌靶#2为PS1219(SEQ ID NO:33)、对于大肠杆菌靶oriC为PS1396(SEQ ID NO:34)、以及对于大肠杆菌靶dam为PS1395(SEQ ID NO:35))。然后,将具有3'磷酸基团的第二组寡核苷酸杂交至3'ssDNA突出,以在其聚合后产生BsaI位点(对于大肠杆菌靶#1为PS1421(SEQ ID NO:36)、对于大肠杆菌靶#2为PS1422(SEQ ID NO:37)、对于大肠杆菌靶oriC为PS1425(SEQ ID NO:38)、以及对于大肠杆菌靶dam为PS1423(SEQ IDNO:39))。最后,第三组寡核苷酸,其3'端与靶的另一端的3'单链突出互补,在聚合后产生BsaI限制性位点(对于大肠杆菌靶#1为PS1268(SEQ ID NO:42)、对于大肠杆菌靶#2为PS1221(SEQ ID NO:43)、对于大肠杆菌靶oriC为PS1378(SEQ ID NO:44)、以及对于大肠杆菌靶dam为PS1424(SEQ ID NO:45))。
使用Bst全长DNA聚合酶进行聚合,使用以下封闭切口:Taq DNA连接酶在补充有200μM dNTP和1mM NAD的ThermoPol缓冲液(20mM Tris-HCl、10mM(NH
为成功识别样本中存在的四种DNA分子,我们使用4碱基的寡核苷酸(5'-CAAG-3'),其产生特定的图案(遗传指纹识别),如图5至图8中A图所示。如这些图所示,成功鉴定出对应于5'-CAAG-3'寡核苷酸的阻断。
如图5至图8中的B图所示,我们还使用来自Diagenode的抗体克隆ICC/IF从这些分子内在CCWGG基序(W可以是A或T)中成功地检测到存在的m
实施例6:当将包含靶区域的核酸与进行性聚合酶接触时,分离效力得到改善
为证明分离效力增加,在制备方法的最后步骤中当将包含靶区域的核酸分子与进行性聚合酶接触时,我们进行与具有和不具有末端修复步骤相同的操作方案,并测量在每种条件下在SIMDEQ平台上观察到多少种功能分子。从2μg大肠杆菌基因组DNA开始,使用与实施例5中所述的相同试剂已经富集四种靶区域,并在SIMDEQ平台上进行检验。基于在我们的平台上观察到的功能性发夹的数量,估计在所有这些步骤之后在制备(包括制备包含靶区域的核酸分子的方法和进一步分离所述靶区域的方法两者)中存在的潜在发夹的总数。出人意料地是,功能性发夹的数量有利地增加超过10,000倍的因数。
实施例7:从人类基因组DNA中富集靶区域
实施例3中描述的操作方案已被用于产生富集的具有3’ssDNA末端的片段,这些片段针对已知为涉及癌症的表观遗传标志物的15种不同的人类靶,或由已知导致人类疾病的STR(短串联重复)组成(参见图10;这些区域的序列对应于SED ID NO:47到60和5)。已经设计Cpf1向导RNA杂交到位于各靶侧翼的序列(SEQ ID NO:61至88、8和9)并设计Cas9向导RNA位于由Cpf1保护的这些区域内(SEQ ID NO:89到116、10和11)。从HEK293细胞培养物中提取人类基因组DNA。操作方案这样进行:将10μg的基因组DNA与390fmol的每种Cpf1-crRNA复合物在37℃孵育1小时,接着加入400U的λ核酸外切酶和核酸外切酶I以消化任何未受到Cpf1复合物保护的DNA。将基因组DNA同时与所有Cpf1-crRNA复合物进行孵育。通过加入终止缓冲液终止反应。使用
然后,将如上述制备的DNA与所有dCas9-gRNA复合物在37℃下孵育1小时,所述dCas9-gRNA复合物结合每个靶的第三和第四序列,然后通过λ核酸外切酶处理在靶区域的两端产生3’ssDNA突出。通过加入终止缓冲液终止反应。为每个分离的靶区域设计寡核苷酸,其中20个碱基对与Cpf1的切割位置互补,以及衔接子序列在其5'端(SEQ ID NO:117到146)。这些寡核苷酸用于起始聚合:通过Bst全长DNA聚合酶去补平ssDNA突出,并且Taq DNA连接酶封闭切口。在补充有200μM dNTP和1mM NAD的ThermoPol Buffer中,在50℃下同时进行两种反应20分钟。在这种补平反应之后,使用核酸外切酶I在37℃下处理30分钟,以除去过量的寡核苷酸。使用KAPA Pure珠(Roche Life technology)纯化反应。使用
使用Nextera XT DNA Library Preparation Kit(Illumina)进行用于Illumina测序的文库制备。在使用双端(pair-end)测序法(每侧150个碱基对)的NextSeq500Illumina测序仪上进行测序反应。获得总计167 966 810个质量评分高于32的簇,其代表序列的50 726Mb。使用Bowtie算法比对人类参考基因组上的读数,并使用Samtools进行覆盖计算。使用IGV软件可视化读数。靶区域(SEPT9.2)之一的代表性覆盖显示在图11中(来自IGV软件的屏幕截图的提取物)。
实施例8:从临床样本中富集四种不同的人类靶,用于磁性镊子平台的分析
实施例3中描述的操作方案已被用于生产具有3'ssDNA末端的四种富集的片段(FMR1、C9ORF72、SEPT9.1和SEPT9.2、SEQ ID NO:5、48、51和58)用于磁性镊子平台的分析。对于每个靶,设计两种Cas9-crRNA位于待保护的目的区域的侧翼(SEQ ID NO:10-11、89-90、101-102和113-114)以及设计两种Cpf1-crRNA(SEQ ID NO:8-9、61-62、81-82和83-84)距离Cas9-crRNA位置至少100个碱基远。使用各种量的起始材料,并且从HEK239培养细胞或临床样本NA06896和NA07537中分离基因组DNA。根据起始材料的量,使用相同的Cpf1与DNA的比率(对于每个DNA分子,加入800,000个Cpf1分子)。例如,在补充有10mM DTT的NEB2.1缓冲液中,3pmol的Cpf1/crRNA复合物用于NA06896的第一次重复(8.5μg的基因组DNA(gDNA)),并在37℃下孵育1小时。加入核酸外切酶的混合物(λ核酸外切酶(20U/μg的gDNA)、核酸外切酶I(20U/μg的gDNA)),并将反应在37℃下再孵育1小时。使用40ng蛋白酶K和20nM的终浓度的EDTA进行失活反应,然后使用KAPA Pure珠(1×)纯化。
使用T4 DNA聚合酶(NEB)和dNTP(200μM)在NEB3.1缓冲液(100mM NaCl、50mMTris-HCl、10mM MgCl
在两端含有长3’ssDNA突出的富集的片段与1pmol的靶特异性生物素、表面和环寡核苷酸(SEQ ID NO:147至158)、以及40单位的Tag DNA连接酶(Enzymatics)和0.25单位的全长Bst DNA聚合酶(NEB)在补充有200μM dNTPs、1mM NAD的
得到的发夹分子在1×Passivation Buffer(PB)(PBS 1×、1mM EDTA、2mg/mLBSA、2mg/mL Pluronic表面活性剂、0.6mg/mL叠氮化钠)中结合到5μl的Dynabeads
对于DNA样本之一(NA06896-重复1),我们还分析位于FMR1的启动子区域内的CpG岛的甲基化状态,其作为重复将其包含在同一分子上。我们根据它们的重复数量测量CpG和非CpG位点的甲基化状态(参见图14和图15)。对于碱基修饰检测,抗-m
如本文所示,使用本文所述的方法成功分离各种核酸片段。此外,所提出的方法是高度特异性,因为在我们的仪器中只观察到靶区域。此外,我们能够在临床样本中观察到预期数量的重复。最后,因为我们的方法不需要任何扩增,所以可以确定CpG岛的甲基化状态位于FMR1的启动子区域内。
序列表
<110> 德皮克斯公司
<120> 使用位点特异性核酸酶优化核酸的富集
<130> B377090PCT D38869
<150> 18306507.7
<151> 2018-11-16
<160> 160
<170> PatentIn version 3.5
<210> 1
<211> 44
<212> RNA
<213> 人工序列
<220>
<223> 用于Cpf1的通用单一向导RNA
<220>
<221> misc_feature
<222> (21)..(44)
<223> n是a、c、g或u
<400> 1
uaauuucuac ucuuguagau nnnnnnnnnn nnnnnnnnnn nnnn 44
<210> 2
<211> 42
<212> RNA
<213> 人工序列
<220>
<223> 用于Cas9的通用crRNA序列
<220>
<221> misc_feature
<222> (1)..(20)
<223> n是a、c、g或u
<400> 2
nnnnnnnnnn nnnnnnnnnn guuuuagagc uaugcuguuu ug 42
<210> 3
<211> 89
<212> RNA
<213> 人工序列
<220>
<223> 用于Cas9的通用tracrRNA序列
<400> 3
guuggaacca uucaaaacag cauagcaagu uaaaauaagg cuaguccguu aucaacuuga 60
aaaaguggca ccgagucggu gcuuuuuuu 89
<210> 4
<211> 94
<212> RNA
<213> 人工序列
<220>
<223> 用于Cas9的通用单一向导RNA
<220>
<221> misc_feature
<222> (1)..(20)
<223> n是a、c、g或u
<400> 4
nnnnnnnnnn nnnnnnnnnn guuuuagagc uagaaaguua aaauaaggcu aguccguuau 60
caacuugaaa aaguggcacc gagucggugc uuuu 94
<210> 5
<211> 2161
<212> DNA
<213> 智人
<400> 5
agaggtcgtg gcactgagat gggtctggca gatcccagcg tccaggccca gcccctatag 60
tgtcagctcc ctcctctggg gacccccttg cttgtgcccc tctgggtccc agcacatccc 120
aggcctgcag ggagggggag aggaagagac tgactcactg gccaggtccc ccaggggctg 180
gagaggctgg agaggcagga gctggatcag atctgaatcc agaggctctc ggaggaagag 240
ctcagggtga gctgcgcccc catcccctgc ccctcctctc ctgctccttc tcccttccat 300
ggtcccagcc agcaagcacc tggggtagag gggacaaacc caggtggctg tgttccagcc 360
ctggctgcag gtctgaatgg ctttctgggg tggctggcca tgctccctga gagcccagct 420
gtggcgatgt ctgagcaggt aggtggggga gcacctagga agcaggggtg tcaggcagag 480
cacaaggaga gagggtgtcc aggtcagttt caggacctgg ctgagaggag ggggctcctc 540
acgggcaccg cctctggcaa gcacagggac aagggcaagg acggcatggc cagaggtccc 600
tgggagcctc ttcccctctc ttcttcctag cagctccccc tcactcttcc cagggaccct 660
gtcactttcc tttagcgtgt ggcagctcct tggcgtccct cccgtgcctt caggttgctt 720
ctgcgccggg cctgccgctg ggcgccccta tctctgcctg ccccctcctc ctgctcccct 780
cgccctgccc ccttggagca attccccacc gagcctccct tcccaggcag tcgaggtccc 840
tccctacctc tgccccgcgc tctgggaggc tccttgttcc gcgaccacaa agcccctttg 900
atcctctgct cggctctgag ccatgtgacc cggtgggcgg gccgcggctc tcggcgcgtc 960
cagcgcagcc cgacgttccg ctgctggggt gagtcctgct cctttgttct tcccagcctt 1020
gcaccactgg ctcgggggct ctcaggtggc gcggccgcga ggcggaccct gatggccatg 1080
gtggcggtgc cgggagccac gctgtccctg ggccccggcc cgaggccggc aggaccgagc 1140
ggggtcccca ggagaggggt ggcggggagc tcgatctcca cgcggggacc agattttcgg 1200
cctcaaaata gaagaatagg gctttgtgtg gtcacagcta tctctttgta aatatttggc 1260
caactaagct gagtggctaa gttctcctgc tgcccggagc ttcttggaac atgtttcctt 1320
ttcgcaaggg gtttccctgg cttccaggag ggccaggaag aaattcgaat tggccaccgc 1380
tttctctaaa atcactccgc tcaagttatc acccctctgg gctcccgaag accggctggc 1440
tggaggctgg agatagtctc aatgctcgaa atgccgtaac cgaagctccc cgcggcgccg 1500
gcactgggat ccagggagct gctgctacag cgcagctctg gattcctgga tgtgttggat 1560
atgtgcaggg cgttcctggg aggagcgggg agggagggtg ctgctggcgg ggctggtctg 1620
cgtgtgcttt gcttctctac aatggcatgc tgcgtgtcgg ccatgcagag gcatgtcagt 1680
gagcaggggc tgagggatct ccctaacgga cctgctttca gagggtcttt tcatgctggg 1740
agaaccccag agactaaatc atgcagccaa cggggtggtc cccggcctca aagcagggag 1800
gggcgaggag ctttgtaggc aatgccatct gctcctgaaa cgccgtccca gtgactctgg 1860
ggactgactc agcctccagc ctgctgcact tcatccctgg cccctctctc tttgcttttt 1920
catgtgaaat ctgctgtgtt ttggtcagag gtttggggaa cagctctctg ctcatctaag 1980
ataagtttgt aattcctttc ctggagaaaa atatgccacc cggaagcagg ttgagcagtg 2040
gttttctggc aggtctgttc ggggctgtgg agacagcacc tgctggatca ggatggagtt 2100
ggaatttggt ttggatccca catgagaaaa cccgttggaa gaggaggaag cagaaaaagg 2160
c 2161
<210> 6
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> 用于PCR扩增的寡核苷酸PS1131(正向SEPT9.2)
<400> 6
agaggtcgtg gcactgagat 20
<210> 7
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> 用于PCR扩增的寡核苷酸PS1132(反向SEPT9.2)
<400> 7
gcctttttct gcttcctcct 20
<210> 8
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-SEPT9.2-crRNA#1的靶特异性序列
<400> 8
uccccucuac cccaggugcu ugcu 24
<210> 9
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-SEPT9.2-crRNA#2的靶特异性序列
<400> 9
ucuaaaauca cuccgcucaa guua 24
<210> 10
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-SEPT9.2-crRNA#1的靶特异性序列
<400> 10
ggcaaggacg gcauggccag 20
<210> 11
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-SEPT9.2-crRNA#2的靶特异性序列
<400> 11
aaacauguuc caagaagcuc 20
<210> 12
<211> 5225
<212> DNA
<213> 大肠杆菌
<400> 12
cagacggtaa ttgttcacct tcgccaaatt cacgacggcg gatcatctgt tccagctctt 60
cttccaccat ttcggagagt tttttacgcg ccagcgggcg gctacgcaag ttgcgaccaa 120
ttgcaggtga agaatcttcg gtttgcgaat caaatgcgtt cataaggccc attctgtaag 180
gtcagtgtga ttaacatcat cagtgacatc ctatcacagg attgaaagta ggggaaaatg 240
gcagggtttt ctctttgtgc ctcatcatta ccataattaa cggaataatt aactattgcg 300
aaaaattaat gtaacgcaga taaaaacatc ccgtttgaat tatttataag actattcacg 360
agcattatga atattatgaa tgtgttctta caaaataatc ataagcgcat attttttaat 420
gaaaaatcac ctcacctaca attaaaaaca cgacatccgc accataaata gccttgcaaa 480
aaatataaca tcgttgtttt caatctgccg tttatgggat tgaccgtttt cttttgacac 540
ggagttcaac aatgttcggc ataattatat ctgtcatcgt attaattacg atgggctatt 600
tgatcctgaa aaactacaaa cctcaggtgg tgctggctgc cgcaggtatc ttcctgatga 660
tgtgcggtgt ctggttaggg ttcggtggtg tactcgatcc caccaaaagc agcggctact 720
tgatcgtcga tatttataat gaaatcctgc gcatgctgtc caaccgcatt gccggattgg 780
ggctgtcgat tatggcggtg ggcggttatg cccgctacat ggagcgcata ggggccagtc 840
gcgcgatggt gagcttgtta agccgcccgt taaaactcat tcgctcgccg tatattattc 900
tgtcggcaac ttacgtcatc ggccaaatca tggcgcagtt tattaccagc gcctccggtc 960
tgggtatgtt gctgatggtc accttatttc cgacgctggt gagtctggga gtaagtcgtc 1020
tctctgcggt ggcagttatc gcaaccacga tgtccattga gtgggggatt ctggaaacga 1080
actccatttt tgctgcccag gtagcgggaa tgaaaattgc cacatacttc ttccactacc 1140
agcttccggt cgcctcttgc gtcattatct cggtggcgat ctcccacttt ttcgtgcaac 1200
gcgcttttga caaaaaagat aaaaatatca atcacgaaca ggcagagcaa aaagctctcg 1260
ataatgtccc gccgctctat tacgccattt tacctgtgat gccgttaatc ctgatgctcg 1320
gctcgctgtt cctcgcccac gtcgggctga tgcagtcaga actgcatctg gtggtggtga 1380
tgttactgag tttgactgtg acgatgtttg ttgagttctt ccgcaagcat aacttgcgcg 1440
aaacaatgga cgatgtgcag gcgttttttg acggcatggg tacgcagttt gccaacgtgg 1500
taacgctggt ggtcgcgggt gaaatatttg cgaaaggctt aacgacgatt ggcactgtcg 1560
atgcggttat caggggggcg gagcattctg gtctgggcgg tattggcgtg atgattatta 1620
tggcgctggt cattgccatt tgtgccattg tgatgggctc tggcaatgcg ccgtttatgt 1680
catttgccag tcttattccg aatatcgcag ccggactaca tgtaccagcg gttgtaatga 1740
ttatgccgat gcattttgcc acgacgctag cgcgcgcggt ttcgccgatt actgcggtgg 1800
tggtcgttac gtcaggaatt gcaggcgttt cgccttttgc ggtggtgaag cggacagcga 1860
tccccatggc agtcggtttc gtggtgaata tgattgccac aatcacgcta ttttattaag 1920
tcattaaaaa gacaaaacag gccgcctggg cctgttttgt attacttcac aacgcgtaat 1980
gccggtcgac caccgcgtgg tggctgcgga ggttcatcgt caggatgagt gtcatcatcg 2040
tgatctggct tgtcgccatc aataaccgac ataacggttt cgttgtctgc cgatgcctct 2100
tcatcattca tgatgctggt atcttcatcg taggcagctt caggctcaaa catcgtgcct 2160
gcgccatttt cacgggcgta gatagccagc acggcagcca gcggcacaga aacctgacgc 2220
ggaatgccac caaagcgcgc gttaaagcgc acctcatcat tcgccagttc cagattgccg 2280
acagcacgcg gcgcaatgtt gagtacgatt tgcccgtcac gcgcatattc cataggaacc 2340
tgcacgccag ggagcgtcac atccaccacc aggtgcggcg tgagctggtt atccagcaac 2400
cactcataga atgcacgcag cagataggga cgacgtggtg ttagctgtga caaatccata 2460
cagattaact ccggcccaga cgcatttcac gttctgcttc agttaaagaa gcaaggaaag 2520
agtcacgctc aaagacgcgg gtcatatagc ctttcagctc tttcgcaccc gggccgctga 2580
actcgatgcc cagttgcggc agacgccaca gcagcggagc aagatagcaa tcgaccaggc 2640
tgaactcatc gctcaggaag tacggcttct gaccgaagac cggcgcaatc gccagcagtt 2700
cttcgcgcag ttgcttacgt gcggcatctg cttcagaagc tgaaccgttg atgatggtgt 2760
tcatcagcgt gtaccagtct ttttcgatgc gatgcatgta cagacggctt tcaccgcgag 2820
ctaccgggta aacaggcatc agtggcggat gcgggaaacg ctcatccaga tattccataa 2880
tgatgcgaga ttcccacagg gtcagctcac gatccaccag ggtcggaacg ctctgattcg 2940
ggttgaggtc aatcagatcc tgaggcggat tgtccttttc cacgtgttcg atctcgaaac 3000
ttacaccttt ctcagccagc acaatgcgga cctgatggct atagatgtca gtaggaccgg 3060
aaaacagcgt cattaccgaa cgtttgttgg cagcgacagc catgaaaacc tccaggtata 3120
gtcagaattt ttactgctac cagccaccag gtggccagtc agaagttgtg ttacccaata 3180
aggaacgact ctctttgttc gaaaatcaaa caaaaaatga gcaatacccg acatttgggc 3240
agaaaattgg atgatagttt accagatttt gcgaccattg tggtgagtcg atgccggaaa 3300
tggggaaaaa gagatgcgct ttagtctgaa atagttgact tagtccctta ttggcgatgt 3360
ggtttttgtt ttacctgtct gtcaggtggc agcaaaaagc aactttccag tttttacgct 3420
gattcagatt ttagctataa aaaaacccgc cgaagcgggt tttttcgaaa attgttttct 3480
gccggagcag aagccaatta acgtttggag aactgcggac gacgacgtgc tttacgcaga 3540
ccgactttct tacgttcaac ctgacgagcg tcacgagtaa cgaagccagc tttacgcagt 3600
tcagaacgca gggactcgtc gtattccatc agagcgcggg tgataccgtg acggatcgca 3660
ccagcctgac cagagatacc accaccttta acggtgatgt acaggtccag tttctcaacc 3720
atgtcgacca gttccagcgg ctgacgaact accatgcggg cagtttcacg accgaagtac 3780
tgttccagag aacgttggtt gattacgatt ttaccgttgc ccggtttgat gaaaacgcga 3840
gctgcggaac ttttgcggcg accagtgccg tagtattgat tttcagccat tgcctataat 3900
cccgattaga tgtcaagaac ttgcggttgc tgtgccgcgt ggttgtgctc gttacccgcg 3960
taaactttca gtttacggaa catagcacga cccagcgggc cttttggcaa catgccttta 4020
accgcgattt caatcacacg ctcaggacgg cgagcaatca tctcttcaaa ggtcgcttgt 4080
ttgataccac cgatgtggcc ggtgtggtga tagtacactt tgtcagtacg cttgttgccg 4140
gttacagcaa ctttgtcagc gttcagaacg atgatgtaat caccggtatc tacgtgcgga 4200
gtgtattccg ctttgtgctt accgcgcagg cgacgagcca gttcagtagc cagacggccc 4260
agagttttac cggtcgcgtc aacaacatac cagtcgcgtt ttacggtttc tggtttagct 4320
gtaaaagttt tcattaaaag cttacccaat aaatagttac acgttggtga acacccaaac 4380
gtcttcaatt gttgaggttc acacgacaaa gtccggcaaa cctacccctt cgaatagcct 4440
atgccagcac acaaaaagtt ttgggaaaaa aactttcttg taacgtgggg tcgcaggatt 4500
atagagaagt cggggtcaaa gatcgacccc tttttgtgat ttgtgacagg ttttaacccg 4560
ccaaatgctc gcgcttcaga tactcttcgc tttgcatctc ttgcagacgt gacaggcaac 4620
gctggaactc aaacttcagc cgatcgccct gataaatttc atacagcggc acttctgcac 4680
tcaccactaa tttgacatgg cgctcgtaaa actcatccac cagcgcaata aagcgccgcg 4740
cttcgctctc catcaaccgc gtcataactg gtacatcaaa caacatgacc gtatgaaaga 4800
gacgtgagag cgcaatatag tcatgctgac tgcgggcgtc gacgcacagc gtagtaaaag 4860
agaccgccag cgtctggttc tcgacgccca ttgttgctaa tggccgatgg ttgatttcta 4920
acgtcggtga attttctcgt ttcccccccg ccagcgccaa ccatagttta tccatttgcg 4980
cccgggtttc atcgtgaagt ggcgaaagcc acagatgcgc ctgagtgagt gtacgcagac 5040
gataatcaac accagcgtcc acgttcatta catcacaatg ctgtttaatg gcatcgattg 5100
caggcagaaa acgcgcacgt tgcaggccat ttcgataaag ttcatccggc ggaatatttg 5160
acgtcgctac cagggtaata ccgcgagcga acagggcttt catcagaccg ccaagtagca 5220
tggca 5225
<210> 13
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-Ecoli#1-crRNA#1(靶#1)的靶特异性序列
<400> 13
gcgaagguga acaauuaccg ucug 24
<210> 14
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-Ecoli#1-crRNA#2(靶#1)的靶特异性序列
<400> 14
aucagaccgc caaguagcau ggca 24
<210> 15
<211> 1643
<212> DNA
<213> 大肠杆菌
<400> 15
ttgctaaaga catacgggtt ctccgaaaat taatatttcc aaatttatca agtgcttaaa 60
taattaaatc tgtgctaaaa accaggtaag gatcagtagg tcagcactgc cgcctggact 120
gagatttcgt tcgatacact ccctgtcgaa ctgccggaga taatcgagat cggcgggggt 180
tcgaatgccc cctttttgca ataatgtttg cgcctcgcgc tgtagccagc gcaggccccc 240
ctcgccaccg cgcgatgcaa cgttggtatc gccgttgatc gccatcagta ggagcaaggt 300
atcgagcaat gccagttcag gatctaaccc ctgatccagc agagtgaggt aatgcggcaa 360
ggcgtgattg atcaccagtg gataacccgc ttcggcttca ccgcgtgcgc cggtaaggcc 420
aagctgttgg tacaaccgtt gacctgccgt cagttgtgaa ttattggtac gcagttcgcg 480
atcggtcagg ccacggcaga aacttgccgc cgtagaacaa acggttgttg gcgttaccgg 540
ttggttgagt tgaagcaaac ggccaattgc cgcacatagc agccctaaag aaaaaatgct 600
gcctttatgc gtgtttacgc ccgcagtggc gcggaacata tcaccttcgc aagccatacc 660
aattgggcgt aatccgtgga gtaccgcttc tggtgccatt tccgcactac aggcaccaaa 720
ttcaatgaaa cggggtagcc agccctgaat cgccagcgcg ctgcggtgga aatcttccag 780
cgccatatct ttgtgcgcac cgcagttaat gcgatccacg aggcctggtt tcggtgacag 840
attgacttca gtcagcatgg cgcgccagcc cagcagggcg tactcatcga ttaatgacgt 900
cgcaagcttt gtggttttag ttgacgttgc aggcatcgac atcgttcagc agtgcctcca 960
tgcggttgag taaatcggtc agttgatggg tttttccacg cgcgcagacg gctgcgcttt 1020
gttcgcacaa caggcagcgg cgaggcggca gtgaatagtc gcggcgggag agaatttcgc 1080
cttcgggcgt caggacatcg atatcccata accgcccgag aggatgacta tgttcaagct 1140
caatggtggc gagcttgagg tcgcgagccg gggcggcaat gctcaacatg ccctccggcc 1200
cgctggcgga aaccagtgca gcctgctcct gaatttgcca gccctgtttt gcggctaagg 1260
cacgcaaggc tgtcacgcca tgattaaaaa ttcggcgtgt gacctcgctg tctttaatcg 1320
gcccaggcgc aaccacggta aaggagacca gtggaacagg atggcgcttg agccagacgt 1380
gttgccgtgc ttgcctttca tcccggctga cgagcagctc gggaattgat accgcatggt 1440
ggctggcgag ttcaggaagc aggtgcatgg cttattcctt cacctgatgc acaacatcga 1500
tcaccgagcc atcgcggtaa cgcacaacgg caacgacgcg gtctgtgaat tcaatcggct 1560
gtggttcacc ggtcagcaga cgcgcacgtt cgcgcagcca ctcaatggaa accactttaa 1620
tgcccgcttc ctgcagacgt tct 1643
<210> 16
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-Ecoli#2-crRNA#1(靶#2)的靶特异性序列
<400> 16
ggagaacccg uaugucuuua gcaa 24
<210> 17
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-Ecoli#2-crRNA#2(靶#2)的靶特异性序列
<400> 17
augcccgcuu ccugcagacg uucu 24
<210> 18
<211> 806
<212> DNA
<213> 大肠杆菌
<400> 18
gtcctttgct tgcgtttagt atcctaaact ggatacccgc ctgatcgatc gctttcgcca 60
tcagaccgcc gagtgcatcc acttctttta ccagatgtcc cttcccaata ccgccgatcg 120
ccgggttgca gctcatctgc cccagagtgt cgatattgtg tgtcaaaagc agagtctgtt 180
gacccatacg cgccgcggcc atcgcggcct cggtgcctgc atgacccccg ccaatgatga 240
tgacgtcaaa aggatccgga taaaacatgg tgattgcctc gcataacgcg gtatgaaaat 300
ggattgaagc ccgggccgtg gattctactc aactttgtcg gcttgagaaa gacctgggat 360
cctgggtatt aaaaagaaga tctatttatt tagagatctg ttctattgtg atctcttatt 420
aggatcgcac tgccctgtgg ataacaagga tccggctttt aagatcaaca acctggaaag 480
gatcattaac tgtgaatgat cggtgatcct ggaccgtata agctgggatc agaatgaggg 540
gttatacaca actcaaaaac tgaacaacag ttgttctttg gataactacc ggttgatcca 600
agcttcctga cagagttatc cacagtagat cgcacgatct gtatacttat ttgagtaaat 660
taacccacga tcccagccat tcttctgccg gatcttccgg aatgtcgtga tcaagaatgt 720
tgatcttcag tgtttcgcct gtctgttttg caccggaatt tttgagttct gcctcgagtt 780
tatcgatagc cccacaaaag gtgtca 806
<210> 19
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-oriC-crRNA#1(靶oriC)的靶特异性序列
<400> 19
ggauacuaaa cgcaagcaaa ggac 24
<210> 20
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-oriC-crRNA#2(靶oriC)的靶特异性序列
<400> 20
ucgauagccc cacaaaaggu guca 24
<210> 21
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-Ecoli#1-crRNA#1(靶#1)的靶特异性序列
<400> 21
caucagugac auccuaucac 20
<210> 22
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-Ecoli#1-crRNA#2(靶#1)的靶特异性序列
<400> 22
ccggaugaac uuuaucgaaa 20
<210> 23
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-Ecoli#2-crRNA#1(靶#2)的靶特异性序列
<400> 23
ccggagauaa ucgagaucgg 20
<210> 24
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-Ecoli#2-crRNA#2(靶#2)的靶特异性序列
<400> 24
gaacgugcgc gucugcugac 20
<210> 25
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cas9-oriC-crRNA#1(靶oriC)的靶特异性序列
<400> 25
ggauacuaaa cgcaagcaaa ggac 24
<210> 26
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cas9-oriC-crRNA#12(靶oriC)的靶特异性序列
<400> 26
ucgauagccc cacaaaaggu guca 24
<210> 27
<211> 1550
<212> DNA
<213> 大肠杆菌
<400> 27
cgacaacctg aacggttggg cgaagaaaga gaatctgaaa aactacgttg tctatgaaac 60
gacgcgtaat ggtcagccgt ggtatgtcct ggtttctggc gtgtacgctt cgaaagaaga 120
ggcgaaaaaa gcggtatcta cattgccagc agatgtccag gccaaaaacc cgtgggcgaa 180
accgctgcgt caggtacagg ccgatctgaa gtaatcaagg ttatctcccg caatggttta 240
tcgttgcggg agttgcctga agcgctggat gctgtcggag ctttctccac agccggagaa 300
ggtgtaatta gttagtcagc atgaagaaaa atcgcgcttt tttgaagtgg gcagggggca 360
agtatcccct gcttgatgat attaaacggc atttgcccaa gggcgaatgt ctggttgagc 420
cttttgtagg tgccgggtcg gtgtttctca acaccgactt ttctcgttat atccttgccg 480
atatcaatag cgacctgatc agtctctata acattgtgaa gatgcgtact gatgagtacg 540
tacaggccgc acgcgagctg tttgttcccg aaacaaattg cgccgaggtt tactatcagt 600
tccgcgaaga gttcaacaaa agccaggatc cgttccgtcg ggcggtactg tttttatatt 660
tgaaccgcta cggttacaac ggcctgtgtc gttacaatct gcgcggtgag tttaacgtgc 720
cgttcggccg ctacaaaaaa ccctatttcc cggaagcaga gttgtatcac ttcgctgaaa 780
aagcgcagaa tgcctttttc tattgtgagt cttacgccga tagcatggcg cgcgcagatg 840
atgcatccgt cgtctattgc gatccgcctt atgcaccgct gtctgcgacc gccaacttta 900
cggcgtatca cacaaacagt tttacgcttg aacaacaagc gcatctggcg gagatcgccg 960
aaggtctggt tgagcgccat attccagtgc tgatctccaa tcacgatacg atgttaacgc 1020
gtgagtggta tcagcgcgca aaattgcatg tcgtcaaagt tcgacgcagt ataagcagca 1080
acggcggcac acgtaaaaag gtggacgaac tgctggcttt gtacaaacca ggagtcgttt 1140
cacccgcgaa aaaataattc tcaaggagaa gcggatgaaa cagtatttga ttgccccctc 1200
aattctgtcg gctgattttg cccgcctggg tgaagatacc gcaaaagccc tggcagctgg 1260
cgctgatgtc gtgcattttg acgtcatgga taaccactat gttcccaatc tgacgattgg 1320
gccaatggtg ctgaaatcct tgcgtaacta tggcattacc gcccctatcg acgtacacct 1380
gatggtgaaa cccgtcgatc gcattgtgcc tgatttcgct gccgctggtg ccagcatcat 1440
tacctttcat ccagaagcct ccgagcatgt tgaccgcacg ctgcaactga ttaaagaaaa 1500
tggctgtaaa gcgggtctgg tatttaaccc ggcgacacct ctgagctatc 1550
<210> 28
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-dam-crRNA#1(靶dam)的靶特异性序列
<400> 28
uucgcccaac cguucagguu gucg 24
<210> 29
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-dam-crRNA#2(靶dam)的靶特异性序列
<400> 29
gcugccgcug gugccagcau cauu 24
<210> 30
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cas9-dam-crRNA#1(靶dam)的靶特异性序列
<400> 30
uucgcccaac cguucagguu gucg 24
<210> 31
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cas9-dam-crRNA#2(靶dam)的靶特异性序列
<400> 31
gcugccgcug gugccagcau cauu 24
<210> 32
<211> 51
<212> DNA
<213> 人工序列
<220>
<223> PS1150(对于大肠杆菌靶#1的5'生物素)
<400> 32
tggtcaagca tgccgctttt cggttcccgg gtgaagaatc ttcggtttgc g 51
<210> 33
<211> 50
<212> DNA
<213> 人工序列
<220>
<223> PS1219(对于大肠杆菌靶#2的5’生物素)
<400> 33
tggtcaagca tgccgctttt cggttcccgg atcagtaggt cagcactgcc 50
<210> 34
<211> 50
<212> DNA
<213> 人工序列
<220>
<223> PS1396(对于大肠杆菌靶oriC的5’生物素)
<400> 34
tggtcaagca tgccgctttt cggttcccgg atacccgcct gatcgatcgc 50
<210> 35
<211> 53
<212> DNA
<213> 人工序列
<220>
<223> PS1395(对于大肠杆菌靶dam的5’生物素)
<400> 35
tggtcaagca tgccgctttt cggttcccgc tacgttgtct atgaaacgac gcg 53
<210> 36
<211> 46
<212> DNA
<213> 人工序列
<220>
<223> PS1421(用于大肠杆菌靶#1的3'磷酸化表面)
<400> 36
tgacgaggtc tccattcgcg ttcagacggt aattgttcac cttcgc 46
<210> 37
<211> 46
<212> DNA
<213> 人工序列
<220>
<223> PS1422(用于大肠杆菌靶#2的3'磷酸化表面)
<400> 37
tgacgaggtc tccattcatc ttttgctaaa gacatacggg ttctcc 46
<210> 38
<211> 46
<212> DNA
<213> 人工序列
<220>
<223> PS1425(用于大肠杆菌靶oriC的3'磷酸化表面)
<400> 38
tgacgaggtc tccattccgc cggtcctttg cttgcgttta gtatcc 46
<210> 39
<211> 46
<212> DNA
<213> 人工序列
<220>
<223> PS1423(用于大肠杆菌靶dam的3'磷酸化表面)
<400> 39
tgacgaggtc tccattcaac tacgacaacc tgaacggttg ggcgaa 46
<210> 40
<211> 29
<212> DNA
<213> 人工序列
<220>
<223> PS1420(用于表面衔接子的具有5'磷酸化的寡核苷酸)
<400> 40
gaatccactt cctaatctgt catcttctg 29
<210> 41
<211> 57
<212> DNA
<213> 人工序列
<220>
<223> PS867(用于表面衔接子的寡核苷酸)
<400> 41
gtgtcttttg gtctttctgg tgctcttcga atcagaagat gacagattag gaagtgg 57
<210> 42
<211> 48
<212> DNA
<213> 人工序列
<220>
<223> PS1268(用于大肠杆菌靶#1的环)
<400> 42
gtattagctg aggaccgata ccgatgccat gctacttggc ggtctgat 48
<210> 43
<211> 48
<212> DNA
<213> 人工序列
<220>
<223> PS1221(用于大肠杆菌靶#2的环)
<400> 43
gtattagctg aggaccgatc tggcagaacg tctgcaggaa gcgggcat 48
<210> 44
<211> 46
<212> DNA
<213> 人工序列
<220>
<223> PS1378(用于大肠杆菌靶oriC的环)
<400> 44
tgacgaggtc tcctttagaa tatgacacct tttgtggggc tatcga 46
<210> 45
<211> 46
<212> DNA
<213> 人工序列
<220>
<223> PS1424(用于大肠杆菌靶dam的环)
<400> 45
tgacgaggtc tcctttaaag gtaatgatgc tggcaccagc ggcagc 46
<210> 46
<211> 26
<212> DNA
<213> 人工序列
<220>
<223> 具有5'磷酸化的PS421环寡核苷酸
<400> 46
taaagcactg agatttttct cagtgc 26
<210> 47
<211> 2250
<212> DNA
<213> 智人
<400> 47
cccagagcag ggcgtcatgc acaagaaagc tttgcacttt gcgaaccaac gataggtggg 60
ggtgcgtgga ggatggaaca cggacggccc ggcttgctgc cttcccaggc ctgcagtttg 120
cccatccacg tcagggcctc agcctggccg aaagaaagaa atggtctgtg atccccccag 180
cagcagcagc agcagcagca gcagcagcag cagcagcagc agcagcagca gcagcagcat 240
tcccggctac aaggaccctt cgagccccgt tcgccggccg cggacccggc ccctccctcc 300
ccggccgcta gggggcgggc ccggatcaca ggactggagc tgggcggaga cccacgctcg 360
gagcggttgt gaactggcag gcggtgggcg cggcttctgt gccgtgcccc gggcactcag 420
tcttccaacg gggccccgga gtcgaagaca gttctagggt tcagggagcg cgggcggctc 480
ctgggcggcg ccagactgcg gtgagttggc cggcgtgggc caccaaccca atgcagccca 540
gggcggcggc acgagacaga acaacggcga acaggagcag ggaaagcgcc tccgataggc 600
caggcctagg gacctgcggg gagagggcga ggtcaacacc cggcatgggc ctctgattgg 660
ctcctgggac tcgccccgcc tacgcccata ggtgggcccg cactcttccc tgcgccccgc 720
ccccgcccca acagcctaca gctgttgtta gtccactcgc acgcctcgaa tcccgtccga 780
actcgtcatt ggctgcttcc tagcggcctg tgttgattgg ctgcccgaag atccgccctc 840
ctgccgtggg cccagccccg caaatgcgca gctaagcggg tggcaagggg cgggtggagc 900
gcggggcgcg acggcggagg ggggcgtggg cagccggacg taccctggca gggagcagca 960
ggtggcggcg gtgcatgggg cctggcccca ccagcgggca ctggcccaca gccacggccg 1020
gggggccatc tagctggaga gagaagggac aggtgacccg atcggagccc agcccagccc 1080
tcagcggtgg ggcgagagac agcgagggga atcgaggttg gggaggttat ctagggagat 1140
cccggaggga atctggtgag gcctgaacgg agggagatct ggggctgaat aaagggcttc 1200
tgccctctaa agtcgcaaag acgtagggtg agccctatat ctggacgggg agaccaggag 1260
ccagggaggg gatctgcaga atgggcagca ggtctgaggc aggggaaaga gaggggtctt 1320
acatgggaag gtggatccgt ggcccgggga ctggggaccc ccgtgacagc tggaaggaga 1380
agaaagaggc atagggcgcg tggaggggcg aaggagggcg gtggcgcggc gtgccccagc 1440
gtgggtccct tccctcctcc aggtgtctat acacgccccg cggagcagac ggcccacctc 1500
ctcccggtcc tccggggaag gggacacatg agggactcac ctgtggctcc ctctgcctgc 1560
agcaactcca tccgctcctg caactgccgg acgtgtgcct ctaggtcccg gttccgagcc 1620
tctgcctcgc gtagttgact gtggggaggt aaggacggtg agtccgtccg ggccggacga 1680
gaggggatgc caagggttgc caccggcccg catcccggcc ccggccccgg ccccgatccc 1740
gacctggcga agttctggtt gtccgtgcgg atggcctcca tctcccggct caggctctgc 1800
cgggtgagca cctcctcctc cagggcttcc tggagctccc gcagcgtcac ctcggcctca 1860
gcctctgccg cagggacagc cgctggaact gccacttcag cctgtgtatg gggaccaggc 1920
ttaaggctgc ctgtggctcc tggaagactc aggacttggg cactggttcc aggctaggaa 1980
tccttgttta tcccctactc ctccgtcccc tcaacatttc tggaatcccc atagctcctg 2040
caatgatcca agccccctcc cttccctacc tccctcagcc ccatccctga gtctggtcct 2100
ctaaatctac acagggacca gagggctggt gctcaaacac taacacaacc tatgtccctc 2160
tgctgctcaa aatccctcca gctccctaat gccctcacga caaaaggcct tgctgggttt 2220
tgtttcctgc tggcctctcc agccttctca 2250
<210> 48
<211> 1487
<212> DNA
<213> 智人
<400> 48
aatgcactta acagtgtctt acggtaaaaa caaaatttca tccaccaatt atgtgttgag 60
cgcccactgc ctaccaagca caaacaaaac cattcaaaac cacgaaatcg tcttcacttt 120
ctccagatcc agcagcctcc cctattaagg ttcgcacacg ctattgcgcc aacgctcctc 180
cagagcgggt cttaagataa aagaacagga caagttgccc cgccccattt cgctagcctc 240
gtgagaaaac gtcatcgcac atagaaaaca gacagacgta acctacggtg tcccgctagg 300
aaagagaggt gcgtcaaaca gcgacaagtt ccgcccacgt aaaagatgac gcttggtgtg 360
tcagccgtcc ctgctgcccg gttgcttctc ttttgggggc ggggtctagc aagagcaggt 420
gtgggtttag gaggtgtgtg tttttgtttt tcccaccctc tctccccact acttgctctc 480
acagtactcg ctgagggtga acaagaaaag acctgataaa gattaaccag aagaaaacaa 540
ggagggaaac aaccgcagcc tgtagcaagc tctggaactc aggagtcgcg cgctaggggc 600
cggggccggg gccggggcgt ggtcggggcg ggcccggggg cgggcccggg gcggggctgc 660
ggttgcggtg cctgcgcccg cggcggcgga ggcgcaggcg gtggcgagtg ggtgagtgag 720
gaggcggcat cctggcgggt ggctgtttgg ggttcggctg ccgggaagag gcgcgggtag 780
aagcgggggc tctcctcaga gctcgacgca tttttacttt ccctctcatt tctctgaccg 840
aagctgggtg tcgggctttc gcctctagcg actggtggaa ttgcctgcat ccgggccccg 900
ggcttcccgg cggcggcggc ggcggcggcg gcgcagggac aagggatggg gatctggcct 960
cttccttgct ttcccgccct cagtacccga gctgtctcct tcccggggac ccgctgggag 1020
cgctgccgct gcgggctcga gaaaagggag cctcgggtac tgagaggcct cgcctggggg 1080
aaggccggag ggtgggcggc gcgcggcttc tgcggaccaa gtcggggttc gctaggaacc 1140
cgagacggtc cctgccggcg aggagatcat gcgggatgag atgggggtgt ggagacgcct 1200
gcacaatttc agcccaagct tctagagagt ggtgatgact tgcatatgag ggcagcaatg 1260
caagtcggtg tgctccccat tctgtgggac atgacctggt tgcttcacag ctccgagatg 1320
acacagactt gcttaaagga agtgactatt gtgacttggg catcacttga ctgatggtaa 1380
tcagttgtct aaagaagtgc acagattaca tgtccgtgtg ctcattgggt ctatctggcc 1440
gcgttgaaca ccaccaggct ttgtattcag aaacaggagg gaggtcc 1487
<210> 49
<211> 1272
<212> DNA
<213> 智人
<400> 49
aagcgagaat ggaggaatac aagccaaagg gaaggaaggg gacgaaggcg gacagggagt 60
gacctcttcc tccaaccccc gggcccgctg ggagcggcgc gaggccagag gcccttgaga 120
ggctcgggct gtcctggggg cctcagtcct ctgcctgtac cccatggggg accctgctgc 180
caccaggcgc cccgcactca ctcgacctgc agcgtgctgg gtttaatctt cacctcaacc 240
ttgtaggagg agccggtgag cagcttgatg gtgcggttct ggccgaagcg ctgcccgtcc 300
accttgtaaa agaccgggcc gtcattaggc tggatgcgca gcgcgatgga gaggcgcacg 360
aggcccggca ggtcccccat gtctgggcga gggtctggcg cggcggctcc ggggggcgga 420
ggacagcgcc ggctgcggcc gagtggctgg agcgcgaggg gcggagagga agcgcgggga 480
gggtgaggga ggtggtggag ctgaggctgc cgctaggaac ccgcgccgtc gccgccgtcc 540
gcccgggctt ttgaggagca gctccttagg ctgtggcccc cctccccact cggcgaggaa 600
gcgggcccaa gagacggctc caaggccgcg cgcttcccca tcccccgctc cagtgctgcg 660
ccctccacgc acccgaaggc tcgctctggc ccgcaggccg ccgcgcagat ccgcgcagct 720
gggggcgagg gagttaatcc tgtttacgca ccacaatccc cttcagctgg ggaagcggac 780
atttaggctc ctcctagaac agccccgggc aggaggagga gaggtttggg aggcactggg 840
aaggcgctgg agttaagcga ccactatgcc aaggagcgag acccccggaa tctggatacc 900
gcctcggcca gctacgtgag gtggacactg ctgctcgcgg atccggcgcc agccaggcgg 960
gaggaggctg agggggggta aagggaggcg ggaagggggg acaggaaacc gctagccggt 1020
gatttaaatt tcaggaaata tgagtctttc caaagcttag gggaaatggc cgaggaaagg 1080
cgcaattcca cgtgatggag ccacgctgga tgaggaatgg atgcaagagg aagaaaataa 1140
ccatattcaa ggagctacat cttcttgtgg gtgtacattt ccattatacg tatgctcgtc 1200
ccaaaaatga cacatacata aatatatgta atgaatcaca tatatttaca cagattttga 1260
agggtgagct at 1272
<210> 50
<211> 2352
<212> DNA
<213> 智人
<400> 50
gggcagtggg acacttagcc tctctaaaag cacctccacg gctgtttgtg tcaagccttt 60
attccaagag cttcactttt gcgaagtaat gtgcttcaca cattggcttc aaagtaccca 120
tggctggttg caataaacat taaggaggcc tgtctctgca cccggagttg ggtgccctca 180
tttcagatga tttcgagggt gcttgacaag atctgaagga ccctcggact ttagagcacc 240
acctcggacg cctggcaccc ctgccgcgcg ggcacggcga cctcctcagc tgccaggcca 300
gcctctgatc cccgagaggg tcccgtagtg ctgcagggga ggtggggacc cgaataaagg 360
agcagtttcc ccgtcggtgc cattatccga cgctggctct aaggctcggc cagtctgtct 420
aaagctggta caagtttgct ttgtaaaaca aaagaaggga aagggggaag gggaccctgg 480
cacagatttg gctcgacctg gacataggct gggcctgcaa gtccgcgggg accgggtcca 540
gaggggcagt gctgggaacg cccctctcgg aaattaactc ctcagggcac ccgctcccct 600
cccatgcgcc gccccactcc cgccggagac taggtcccgc gggggccacc gctgtccacc 660
gcctccggcg gccgctggcc ttgggtcccc gctgctggtt ctcctccctc ctcctcgcat 720
tctcctcctc ctctgctcct cccgatccct cctccgccgc ctggtccctc ctcctcccgc 780
cctgcctccc cgcgcctcgg cccgcgcgag ctagacgtcc gggcagcccc cggcgcagcg 840
cggccgcagc agcctccgcc ccccgcacgg tgtgagcgcc cgacgcggcc gaggcggccg 900
gagtcccgag ctagccccgg cggccgccgc cgcccagacc ggacgacagg ccacctcgtc 960
ggcgtccgcc cgagtccccg cctcgccgcc aacgccacaa ccaccgcgca cggccccctg 1020
actccgtcca gtattgatcg ggagagccgg agcgagctct tcggggagca gcgatgcgac 1080
cctccgggac ggccggggca gcgctcctgg cgctgctggc tgcgctctgc ccggcgagtc 1140
gggctctgga ggaaaagaaa ggtaagggcg tgtctcgccg gctcccgcgc cgcccccgga 1200
tcgcgccccg gaccccgcag cccgcccaac cgcgcaccgg cgcaccggct cggcgcccgc 1260
gcccccgccc gtcctttcct gtttccttga gatcagctgc gccgccgacc gggaccgcgg 1320
gaggaacggg acgtttcgtt cttcggccgg gagagtctgg ggcgggcgga ggaggagacg 1380
cgtgggacac cgggctgcag gccaggcggg gaacggccgc cgggacctcc ggcgccccga 1440
accgctccca actttcttcc ctcactttcc ccgcccagct gcgcaggatc ggcgtcagtg 1500
ggcgaaagcc gggtgctggt gggcgcctgg ggccggggtc ccgcacgtgc gccccgcgct 1560
gtcttcccag ggcgcgacgg ggtcctggcg cgcacccgag gggcgggcgc tgcccacccg 1620
ccgagactgc actgtttagg gaagctgagg aaggaaccca aaaatacagc ctcccctcgg 1680
accccgcggg acaggcggct ttctgagagg acctccccgc ctccgccctc cgcgcaggtc 1740
tcaaactgaa gccggcgccc gccagcctgg ccccggcccc tctccaggtc cccgcgatcc 1800
tcgttcccca gtgtggagtc gcagcctcga cctgggagct gggagaactc gtctaccacc 1860
acctgcggct cccggggagg ggtggtgctg gcggcggtta gtttcctcgt tggcaaaagg 1920
caggtggggt ccgacccgcc ccttgggcgc agaccccggc cgctcgcctc gcccggtgcg 1980
ccctcgtctt gcctatccaa gagtgccccc cacctcccgg ggaccccagc tccctcctgg 2040
gcgcccgcgc cgaaagcccc aggctctcct tcgatggccg cctcgcggag acgtccgggt 2100
ctgctccacc tgcagccctt cggtcgcgcc tgggcttcgc ggtggagcgg gacgcggctg 2160
tccggccact gcaggggggg atcgcgggac tcttgagcgg aagccccgga agcagagctc 2220
atcctggcca acaccatggt gtttcaaaat ggggctcaca gcaaacttct cctcaaaacc 2280
cggagacttt ctttcttgga tgtctctttt tgctgtttga agaatttgag ccaaccaaaa 2340
tattaaacct gt 2352
<210> 51
<211> 1711
<212> DNA
<213> 智人
<400> 51
tgaggtttgt ttcacagtgg aatgtaaagg gttgcaagga ggtgcatcgg cccctgtgga 60
caggacgcat gactgctaca cacgtgttca ccccaccctc tggcacaggg tgcacataca 120
gtaggggcag aaatgaacct caagtgctta acacaatttt taaaaaatat atagtcaagt 180
gaaagtatga aaatgagttg aggaaaggcg agtacgtggg tcaaagctgg gtctgaggaa 240
aggctcacat tttgagatcc cgactcaatc catgtccctt aaagggcaca gggtgtctcc 300
acagggccgc ccaaaatctg gtgagagagg gcgtagacgc ctcaccttct gcctctacgg 360
gtcacaaaag cctgggtcac cctggttgcc actgttccta gttcaaagtc ttcttctgtc 420
taatccttca cccctattct cgccttccac tccacctccc gctcagtcag actgcgctac 480
tttgaaccgg accaaaccaa accaaaccaa accaaaccaa accagaccag acaccccctc 540
ccgcggaatc ccagagaggc cgaactggga taaccggatg catttgattt cccacgccac 600
tgagtgcacc tctgcagaaa tgggcgttct ggccctcgcg aggcagtgcg acctgtcacc 660
gcccttcagc cttcccgccc tccaccaagc ccgcgcacgc ccggcccgcg cgtctgtctt 720
tcgacccggc accccggccg gttcccagca gcgcgcatgc gcgcgctccc aggccacttg 780
aagagagagg gcggggccga ggggctgagc ccgcgggggg agggaacagc gttgatcacg 840
tgacgtggtt tcagtgttta cacccgcagc gggccggggg ttcggcctca gtcaggcgct 900
cagctccgtt tcggtttcac ttccggtgga gggccgcctc tgagcgggcg gcgggccgac 960
ggcgagcgcg ggcggcggcg gtgacggagg cgccgctgcc agggggcgtg cggcagcgcg 1020
gcggcggcgg cggcggcggc ggcggcggag gcggcggcgg cggcggcggc ggcggcggct 1080
gggcctcgag cgcccgcagc ccacctctcg ggggcgggct cccggcgcta gcagggctga 1140
agagaagatg gaggagctgg tggtggaagt gcggggctcc aatggcgctt tctacaaggt 1200
acttggctct agggcaggcc ccatcttcgc ccttccttcc ctcccttttc ttcttggtgt 1260
cggcgggagg caggcccggg gccctcttcc cgagcaccgc gcctgggtgc cagggcacgc 1320
tcggcgggat gttgttggga gggaaggact ggacttgggg cctgttggaa gcccctctcc 1380
gactccgaga ggccctagcg cctatcgaaa tgagagacca gcgaggagag ggttctcttt 1440
cggcgccgag ccccgccggg gtgagctggg gatgggcgag ggccggcggc aggtactaga 1500
gccgggcggg aagggccgaa atcggcgcta agtgacggcg atggcttatt ccccctttcc 1560
taaacatcat ctcccagcgg gatccgggcc tgtcgtgtgg gtagttgtgg aggagcgggg 1620
ggcgcttcag ccgggccgcc tcctgcagcg ccaagagggc ttcaggtctc ctttggcttc 1680
tcttttccgg tctagcattg ggacttcgga g 1711
<210> 52
<211> 2266
<212> DNA
<213> 智人
<400> 52
aaccacgtcc tctcctttgg ggcagaaatc tctgggagta ggggcaaggt agtttaacca 60
cgaacggggt ggggactaaa caaccctagc acctctaagg tgcccccagg aggtcttgcc 120
aaggagtctt ccacagggag agtcatcctg cccgttgttc tggatcttga aacttgggag 180
acccacacca aaggagggaa ccggttcctt taccctttaa gcgcgtcgtg tcctccaccg 240
cctcttctct tggcccgact ggctctggtt tccttcacgt tcccagcctc caaattggcg 300
tccgccccat ggctcgtgta ggacgctaaa agcacggtaa atcccagggc gatctccagc 360
agacgccctc gacgcatgat gccgagccgc caccggctcc cgccgcctct tgccgcgccc 420
ggggctcggt ctgcggccgc cgctgcgccc tgaagcgcac cgcgccgccg gggtcccgct 480
atcccgccgc ccgtcccccg ggccgggctc ctcccgcctt ctccaggcgc tgctcccact 540
tcaggcggcc cctgccagct gcaacacaga gacaaatccc cgaggcgggg agactttcag 600
ggcatcggga tgctgaagcc tcgcggtccc cattcccaag cccaacccgt gcgccgcctg 660
cgcgtggcgc agttaatttg ggtaagggaa gaccgtttgg tccacagctg gctggaaagc 720
ccaggccccc ggcggcagca gccccggccg atccctcggc ctcccgggcc ccgccagaag 780
agcgcggcgc aaagtaccca tatccccagc ggtcctaagc cccgggcgag ctctctgggt 840
ttattttttt tagcggcacg aattgcgcgc gatgcgcgct ctgaacgccc cactcctcag 900
cctgccctct ccgccaggct ggaagccagc cgcgccgtcc ccgccgactc cgggcgatgc 960
caccgccccc cgaacatctg acgcggagcc cggcaccaag agccccgggc caggaagctg 1020
tcaggcaggg gagggccagc gccagctgtg gacgttccgg gacagcccct caccccagtc 1080
cctgtccctg ccctcggcgc ggcgcaaacg caaaagcctc cgtggccgca gctccagccc 1140
cggtggccgc ccgacccccg tcgggacccc ggctgcaccc actggagaag cggcggctcc 1200
gactcccgag gaggcggcgg cggctgctcc cagtcgtggc cgctacagcc actgctcggc 1260
tccgctcgca gctgtcccag ctgtggctcc tgtccgcttg tggcacccac agtctctgcc 1320
gcggctcccg cagccccctc ctttccccac ctcttcaagt atcgtccgcg cagcggtttc 1380
tcgcgagaga aatacttttt ttaaaaaaag aaagaaaaag aaaatgacac cccctccttc 1440
gtcgccctca tcaccacccc accccccggc cccatccatc ctccctttcc actccccttt 1500
gccagcctcc gcctcggtgc ggggcctctc gctcgcagga ttagcgcagt gggaggaggc 1560
agaggtgatc aggtcctgcc cggcctggga ctttttgtct tgaggtgggg aggggagaaa 1620
tgggaagagg tggagtagcg gttttagccc gctctgcggc tgcgaggttt agatccgaga 1680
ttaacctctc ccgcgatagg tgaagcccta ccggagcaga aagctgttcc acctgcacca 1740
agaatgcgcg ctggagacgg ttgccccgga ggccctggcc cgagagaaaa ccagtccccg 1800
ctgcccgcgc ctcccggtag cgcgctccct gcgcctctcc cgccggacac tcagcagacg 1860
ccggaggccg ggaggctaag actgggcgcg tcgcaggccg ggaccgcggc agaggctgct 1920
gtgccgaccg aggagagggc tctgccgccc ccacttgccc tgggtgtcga gagcccactc 1980
cagacgcggc tcttctgagg ctcaattcaa gccacccagg cctgaatcca gggtgctctc 2040
tctaagtcgg tgtccaaccc aggggcctgt aaatgttgga acctaggatg taggatggga 2100
gcggtgaagg gatggtcctc ttgcccagcc cagattaaga ctggggttca tctggaggac 2160
tctacttaca ccctcccggt cccctcgccc tcccccacac acagctgcct ctccccagga 2220
tatgcgcggc acagtgaatt tagtggcttt gaaaggtata gtattt 2266
<210> 53
<211> 1643
<212> DNA
<213> 智人
<400> 53
ggtcagagca ggggtgcact cccataaaga aacgccccca ggtcgggact cattcctgtg 60
ggcggcatct tgtggccata gctgcttctc gctgcactaa tcacagtgcc tctgtgggca 120
gcaggcgctg accacccagg cctgccccag accctctcct cccttccggg gcgctgcgct 180
gggaccgatg gggggcgcca ggcctgtgga caccgccctg caggggcctc tccagctcac 240
tgggggtggg gtgggggtca cacttggggt cctcaggtcg tgccgaccac gcgcattctc 300
tgcgctctgc gcaggagctc gcccaccctc tccccgtgca gagagccccg cagctggctc 360
cccgcagggc tgtccgggtg agtatggctc tggccacggg ccagtgtggc gggagggcaa 420
accccaaggc cacctcggct cagagtccac ggccggctgt cgccccgctc caggcgtcgg 480
cgggggatcc tttccgcatg ggcctgcgcc cgcgctcggc gccccctcca cggccccgcc 540
ccgtccatgg ccccgtcctt catgggcgag cccctccatg gccctgcccc tccgcgcccc 600
acccctccct cgccccacct ctcaccttcc tgccccgccc ccagcctccc cacccctcac 660
cggccagtcc cctcccctat cccgctccgc ccctcagccg ccccgcccct cagccggcct 720
gcctaatgtc cccgtcccca gcatcgcccc gccccgcccc cgtctcgccc cgcccctcag 780
gcggcctccc tgctgtgccc cgccccggcc tcgccacgcc cctacctcac cacgcccccc 840
gcatcgccac gccccccgca tcgccacgcc tcccttacca tgcagtcccg ccccgtccct 900
tcctcgtccc gcctcgccgc gacacttcac acacagcttc gcctcacccc attacagtct 960
caccacgccc cgtcccctct ccgttgagcc ccgcgccttc gcccgggtgg ggcgctgcgc 1020
tgtcagcggc cttgctgtgt gaggcagaac ctgcgggggc aggggcgggc tggttccctg 1080
gccagccatt ggcagagtcc gcaggctagg gctgtcaatc atgctggccg gcgtggcccc 1140
gcctccgccg gcgcggcccc gcctccgccg gcgcagcgtc tgggacgcaa ggcgccgtgg 1200
gggctgccgg gacgggtcca agatggacgg ccgctcaggt tctgctttta cctgcggccc 1260
agagccccat tcattgcccc ggtgctgagc ggcgccgcga gtcggcccga ggcctccggg 1320
gactgccgtg ccgggcggga gaccgccatg gcgaccctgg aaaagctgat gaaggccttc 1380
gagtccctca agtccttcca gcagcagcag cagcagcagc agcagcagca gcagcagcag 1440
cagcagcagc agcagcaaca gccgccaccg ccgccgccgc cgccgccgcc tcctcagctt 1500
cctcagccgc cgccgcaggc acagccgctg ctgcctcagc cgcagccgcc cccgccgccg 1560
cccccgccgc cacccggccc ggctgtggct gaggagccgc tgcaccgacc gtgagtttgg 1620
gcccgctgca gctccctgtc ccg 1643
<210> 54
<211> 1816
<212> DNA
<213> 智人
<400> 54
ctctgcaggt cctcactgta actggaaaaa cacgacctcg ccctcgggaa ggctttctgt 60
gcgcctcacc tcaggatgag ggtgggtgta ggggacacct cccagaaacc cctaacctcc 120
cagtcggtta aagaagaggg gatagggtca agggatgcga cagagctgtg tggtttccgg 180
atgggaaacc tcagtcgttt aggcacccct ccgctcgagt cacttccgaa gcagtcgatt 240
cttggggaga agcgctgcgg aaaggggcga ctccgatgca gatggccctg tcccggcgcc 300
ccaggtcgtc gcgcgcgcag ctgcggtagt cactgcgcct ccccgccccc actcctggat 360
gccccccttc cctctcccgg ccagactctg agcaggagct ccgcccccag cgcgccgccc 420
cagccccggc gccttaaaag ccgggcgcac cgccccgccg cgccctgcct gccgcacctc 480
tcctttcttc tgtagctcgc gttgaagccg cacgtccggc cccgatcccg gcaccatgag 540
cttcggctcg gagcactacc tgtgctcctc ctcctcctac cgcaaggtgt tcggggatgg 600
ctctcgcctg tccgcccgcc tctctggggc cggcggcgcg ggcggcttcc gctcgcagtc 660
gctgtcccgc agcaatgtgg cctcctcggc cgcctgctcc tcggcctcgt cgctcggcct 720
cggcctggcc tatcgccggc cgccggcgtc cgacgggctg gacctgagcc aggcggcggc 780
gcgcaccaac gagtacaaga tcatccgcac caacgagaag gagcagctgc agggcctcaa 840
cgaccgcttc gccgtgttca tcgagaaggt gcatcagctg gagacgcaga accgcgcgtt 900
ggaggccgag ctggccgcgc tgcgacagcg ccacgctgag ccgtcgcgcg tcggcgagct 960
cttccagcgc gagctgcgcg acctgcgcgc gcagctggag gaggccagct cggctcgctc 1020
gcaggccctg ctggagcgcg acgggctggc ggaggaggtg cagcggctgc gggcgcgctg 1080
cgaggaggag agccgcggac gcgaaggcgc cgagcgcgcc ctgaaggcgc agcagcgcga 1140
cgtggacggc gccacgctgg cccgcctgga cctggagaag aaggtggagt cgctgctgga 1200
cgagctggcc ttcgtacgcc aggtgcacga cgaggaggta gccgagctgc tggccacgct 1260
gcaggcgtcg tcgcaggccg cggccgaggt ggacgtgact gtggctaaac cagacctgac 1320
ctcggctctg agggagatcc gcgcccagta tgagtccctg gccgctaaga acctgcagtc 1380
cgcggaagaa tggtacaagt ccaagtttgc caacctgaac gagcaggcgg cgcgcagcac 1440
cgaggccatc cgggccagcc gcgaggagat ccacgagtat cggcgccagc tgcaggcgcg 1500
caccatcgag atcgagggcc tgcgcggggc caacgagtcc ttggagaggc agatcctgga 1560
gctggaggag cggcacagtg ccgaggtagc tggctaccag gtaagggccg gggctgggcg 1620
tggggagggg tgccctgccc tcttccgcgc gtaccctctt cctctggtaa aactgggccc 1680
caggacttaa ggggagggca aaagagagga gagaagagcc gcggctggag gcgctggtta 1740
acaaaaaacc ctggagtctt taatgttaat tttagggaac gcccctcatt tatgtccctg 1800
ccccagccct tcaaac 1816
<210> 55
<211> 1934
<212> DNA
<213> 智人
<400> 55
aaacaccggc agtcaacagg caggcaaaga acctgggggt gggggtagca gcggtcccac 60
cctcaaaagg cccgggctgc ccagaccaag agaaagcgat gaatctcttc tggtaacgtc 120
ccttcctgtc gcatggattc aaggccgacc tgccccagca ccaccaccag cagccttctg 180
ctggggccgg cacagctggg agcaacctcc tactctcagg cagacgcgca gcaccaagca 240
gagaggcccg gtgcaggatc ccagcgccga accagcgccg gctcagtgga cgcggaaggg 300
gccggcggcc gcggccggtc ccatccccca ctgcagaccc ccagcctgtg gcggtggtcc 360
agttccgcca ggaaaccgcc gcctggagct gtgggtcgcg cacattaacg catccagcgg 420
aaaaatgaag gagacccaaa ttcaaagtta aagtaatggt gacccgagag gtgccttgat 480
gagaaggttt ggggtcccgg ttactgatgg ttatcattct tacgagatgc tggtcaccta 540
cgaagggaga aaggcacgag gagcgcctga ccaaagtggt tttgccctgc ttcccgcaag 600
aggtggcacc cacggctgga acgcaggagt cagacccaca gtccccagct ctggacgccc 660
gcagcggggc ctcgaagagg ttcagggcgg tgcccgcggc gctcgggccg ggtctcccgg 720
ggcgtggggc ggggggcggg gttgggcggc ggccggggct cctccctctt ctgccccggg 780
ctcccctgct cttaacccgc gcgcgggggc gcccaggcca ctgggctccg cggagccagc 840
gagaggtctg cgcggagtct gagcggcgct cgtcccgtcc caaggccgac gccagcacgc 900
cgtcatggcc cccgcagcgg cgacgggggg cagcaccctg cccagtggct tctcggtctt 960
caccaccttg cccgacttgc tcttcatctt tgagtttgtg agtggctcct ggccggggaa 1020
gggacggggt gggctgagcc gtgcgctctc tcgggcgccc agcacagctg tcggacggga 1080
tccgctagct gcgcaggttc tgggagcatc ggggcagcag gcgcagggcg gggactaagc 1140
cagggaagtc ccctcccacc tccggtcctt tgtgcccttc tagaccaaca gaatgagggg 1200
aacagtctac aggactatgg aggaaaaact gggttcccaa ctggggtcag atgtaggcag 1260
cggggcaggg ggggacggct cttggttcgc tggtcccaaa gctgcgcgcg gggcccactt 1320
gacgcgcgca gcgccaccga agctcccgcc gcgctttgcg cggttgggta gaagtgcgca 1380
gcttttacaa gggagaaggt ttcgttaaaa aagaaaaaaa aatcagcaag agaaacatta 1440
gtattaccaa ccgagatttg gagatgagag ggagctgaat ccggtttatt ttcttctggc 1500
cttttaaagt ttctggcgag ggaacgtatt tgcgaccaat tcgatctgga aatgaggcca 1560
tcgtttgctt ggccgcagtc cttctgcccc gtgtgcgggg tgggggtgga ggagatgggg 1620
ggtggggggt ggggggtggc ggcgagagcg atccgcgcgc ctcgactgac cttgggcagg 1680
cccggggcct ctgcacctgc ggtcggtccc gccttgcacg cacggtctct gcctgaggct 1740
gcaggaaagc gcttcctact gagaactcct gataagcgct cacggtgtcg cgaagccgaa 1800
gtgacctccc tcagcctcaa ctccccgggg gccgctggcc ttcacctggg aggggtgtgc 1860
cctgtatgtc ctgtgggtgc ggtccgtcac cgcctgaggg acaccttttc cggcacccca 1920
ccctcagaag tgtc 1934
<210> 56
<211> 2574
<212> DNA
<213> 智人
<400> 56
ctggagtggg acttcatctg ggaccaaagg agggctggtg aggggagtgg caggagggag 60
gagtgcctcg gggccccgag caggatgagc ctgaggaaga gacgggtccc catgttccct 120
ttcccgctca gataatggag gtgaattgag gggagcagag acctccccac cttcagggtg 180
ggaccctgag ggaccaggac acctttgcta ggggatgtcc ctcctcactc ctgcacaagt 240
tcctcaagga caccctcggg ctccgaaaac ggggggaggg ggacgacgcc ccagaggccc 300
ctgagcccct ggttcttccc gaccctaagg gcttttctcc ctcggttccc aggcggcgac 360
ggcgggtagc gcgaagcagc aggcgcaggg gcgctgggat ggggatgtct ctgcaggtct 420
aaggttcccc ttgggagtct aaacaaagac tacggcagcg ccgtcccctc ccccgggaac 480
ccgacgccgc gcggccacag ggggcctgga ggggcgggca gggcctcgca gcgcacccag 540
cacagtccgc gcggcggagc gggtgagaag tcggcggggg cgcggatcga ccggggtgtc 600
ccccaggctc cgcgtcgcgg tccccgctcg ccctcccgcc cgcccaccgg gcaccccagc 660
cgcgcagaag gcggaagcca cgcgcgaggg accgcggtcc gtccgggact agccccaggc 720
ccggcaccgc cccgcgggcc gagcgcccac acccgccaaa cccacgcggg cacgcccccg 780
cggcgcaccg cccccagccc ggcctccgcc cctgcagccg cgggcacgcg gaggggctcc 840
tggctgcccg cacctgcacc cgcgcgtcgg cggcgccgaa gccccgctcc ccgcctgcgc 900
gtctgtctcg tccgcatctc cgcggtgagt cggcggcgcc ctcgcccctg agcccagggc 960
cagcttctct cgccgccgcg gctgctgcgc gcgtccccgc ccagcccagc ccagccccga 1020
gcacgacccc agccccacgc acgaccctag ccccgcgagt cccgcaccga ctcgctcccg 1080
ccccatttcg cctccgcggg ggcggcgccc cctcctcccc gcggctcccg ctctccttcc 1140
tcgccttccc ggccgcgctg gggaccccca gccgccgtcc gcgacccccc accgcgacgc 1200
ccggaggcgg cggggtctct ttgttcgggc ggcgggcacg ggggaccacc tcccacggtg 1260
tcaccgcacc caccccgcgc ccttcctccg cctcctggag ttcaccggga ccaggtggcg 1320
gcgggtgcct ttttgggggt gcgcggccat gcaattggtg gattttttta aaccgttttg 1380
gaggggggag cgcggcgttg ggggcgggag agcgctcctg gctgtgagct gctcctgccg 1440
cttcgctccg cgctctcctg ccgctccgct ccgggtctcc cgcgctcctc tccccggctc 1500
ggccgagcgc gctgccccga cgccgccacc cagagccggg ccgcgccggg cgccgagatg 1560
aaggtgctgg gacaccggct ggagctgctc acaggtaccg cccgcctgcc ccgcagccgg 1620
ccgccacttt ccgagttgga gcggactccg ggcgcggcgg ccggggactg gggcggctcg 1680
ggtctgagca ggaaggggtg cggaccccaa ctaagtccta gttttgtgct acctgtttgt 1740
gtgcggagcc cagccccggg agaggacttg aggttgtggc gagtccctgg cgctggcgtc 1800
cgggctgcgg gagcaccggt cagggggtgg ccccatgggg tctctgacca gcggagctcg 1860
gattaggacc ctgaaagcta gctcagggct cctgccctcc aatcagtgtc gcttgtcccc 1920
taagaaagga cccgtgggct tctggcagga cccgcgccat ggacctctta tttctgcgcc 1980
ctgtgacaat ctgagccgtc tttctctggg ggagaagttt cttgctggga gtggaggcga 2040
cgccaagtgg cctgggaagt gggaagccag attggaccct actgactggg gaccctcagc 2100
cttggggctc ctctggagaa gtgatcagtt gccctgctgg aaactcacat ccagggggca 2160
gtggctggag agcaagagcg aacggtcagg aagaggaggt gggaaaggga gcagggacgg 2220
gggggaggat tcgaggagtg acttctgtgt tctccccggt gtggagagac ccagacagga 2280
ggaaaggaaa gcaacccggt ttcctccagc tctgggactt ataggtgctc catccgtgta 2340
tgtcagatga gcacagattc cagtaagtgt cctccgacac ctgggggagg gggctgatca 2400
ctgccttcca ggaccttaat gtccgatgag ggagcagagc cccggagccc tgttacaagg 2460
ctggaagggg cagccgtctg tgggtgcgct caggaaacgg tggaatccga gtcgggggca 2520
gcttttgaag acctcgaaaa acaatttttg ttaatgaaga aggaggtggc atta 2574
<210> 57
<211> 1393
<212> DNA
<213> 智人
<400> 57
ggggggtggg tgacatatct gcaggaaatg gaacgtggaa ggcactgtct gacttggctg 60
cattgcccgt gtggacccgg gcgctagtgc ctgcatgtcc ctctgaccgt gtgccctctg 120
gtgtgcgctt ggatggtgtc tgccttggtg atgcctgggg ccatcaggcc agggggcggg 180
ggtgtccttg gtgagccccc gaggctgtgt gcccagtgtg cgtgccgggt ctgtgcgtgc 240
aggggctgcc gtcccagtgg gcggccgtct agcttgtttg gatggctgcg ccagtgtgat 300
gggaaggaca ggcggcctct tccttaggag ccacctcaca gttcccaggg ctctgctgcg 360
ccagccgggc agaggcaagg aggtctgggc cacacaggaa tgacagcctt tcaggcaggg 420
gtccctgctg gggaggaacg aggaggggtt gcctgcctgc cttattttgt aggtggggga 480
aggcaacgct ggagccgtga agctggcagg gctaggggac cccaggtgga gcccagggca 540
cctcctctcg ccggggcatc aggtaccccg gccccattct tcctcaggag ggagaggcag 600
gggcagactc acccccaccc ccgggcccca cacgcctgcc ctgccggttc cgcagacgat 660
aggtcacccc gcacgcacgg aggtgacgac gtcagcacct gcccgcccat gcagaccctg 720
tcccctgaat tattgatgcg gctgacaggc cgaaccacag caccaccagc accaactccc 780
acaccggcgc aaagcccggc tcaaagcccc actcccctcc agtgctcaag gtcacaggcg 840
aggggagacg gaccgaccga gaggagccgc cacagccccc tcccctgctg ccgcagtgct 900
tcccgcctgc gcctacctgg aggcggggcc ggctctgacg tcacccagag ccaatgggag 960
tgctcggccc tgagggttgg gggcctcaga gtgcaccgcg ctgtggccct tgtggggctg 1020
cccttgcgca gcgctccaag ggacggggat gtttggcttc gatcaggctg aactgagtcc 1080
tgagctctcc gcagagggtg agagaaggcg ttagggaggt tcgcagcagg gttcagcgaa 1140
ggtcactgga cttcgtagga caggtagccc ggtgacgccc aggcccagcc ccagcccttc 1200
ccatcctggg agatgagccc tagtagagcc tgatcacgtc acctcagggg gatggggaca 1260
ggggcggcca caccagggct gggagaagac agtggggcct cctcagcagc agagagcaga 1320
cacccctcac acccctcagg cggacccgca cgctgcgggt ctgggtttgg aaggcaccgc 1380
cctgggctct cga 1393
<210> 58
<211> 2295
<212> DNA
<213> 智人
<400> 58
ctccttcctt cacaccttcc ttcggaaacg tctgctcctg acaaggtcta cttcctgctc 60
tcaggaggcc cttattgtgg aggaagggag gcgtcgcccg tccctggctt ctctgacagc 120
cgtgttccat ccccgccctg tgccccttct cccggacagt gccttctcca gggctcaccc 180
aggagggtgc agcggtggcc cccggggcgg tggtcgtggt gggggtgtta gctgcagggg 240
tgccctcggt gggtgggagt tggtggcctc tcgctggtgc catgggactc gcatgttcgc 300
cctgcgcccc tcggctcttg agcccacagg ccgggatcct gcctgccagc cgcgtgcgct 360
gccgtttaac ccttgcaggc gcagagcgcg cggcggcggt gacagagaac tttgtttggc 420
tgcccaaata cagcctcctg cagaaggacc ctgcgcccgg ggaaggggag gaatctcttc 480
ccctctgggc gcccgccctc ctcgccatgg cccggcctcc acatccgccc acatctggcc 540
gcagcggggc gcccgggggg aggggctgag gccgcgtctc tcgccgtccc ctgggcgcgg 600
gccaggcggg gaggaggggg gcgctccggt cgtgtgccca ggactgtccc ccagcggcca 660
ctcgggcccc agccccccag gcctggcctt gacaggcggg cggagcagcc agtgcgagac 720
agggaggccg gtgcgggtgc gggaacctga tccgcccggg aggcgggggc ggggcggggg 780
cgcagcgcgc ggggaggggc cggcgcccgc cttcctcccc cattcattca gctgagccag 840
ggggcctagg ggctcctccg gcggctagct ctgcactgca ggagcgcggg cgcggcgccc 900
cagccagcgc gcagggcccg ggccccgccg ggggcgcttc ctcgccgctg ccctccgcgc 960
gacccgctgc ccaccagcca tcatgtcgga ccccgcggtc aacgcgcagc tggatgggat 1020
catttcggac ttcgaaggtg ggtgctgggc tggctgctgc ggccgcggac gtgctggaga 1080
ggaccctgcg ggtgggcctg gcgcgggacg ggggtgcgct gaggggagac gggagtgcgc 1140
tgaggggaga cgggacccct aatccaggcg ccctcccgct gagagcgccg cgcgcccccg 1200
gccccgtgcc cgcgccgcct acgtggggga ccctgttagg ggcacccgcg tagaccctgc 1260
gcgccctcac aggaccctgt gctcgttctg cgcactgccg cctgggtttc cttcctttta 1320
ttgttgtttg tgtttgccaa gcgacagcga cctcctcgag ggctcgcgag gctgcctcgg 1380
aactctccag gacgcacagt ttcactctgg gaaatccatc ggtcccctcc ctttggctct 1440
ccccggcggc tctcgggccc cgcttggacc cggcaacggg atagggaggt cgttcctcac 1500
ctccgactga gtggacagcc gcgtcctgct cgggtggaca gccctcccct cccccacgcc 1560
agtttcgggg ccgccaagtt gtgcagcccg tgggccggga gcaccgaacg gacacagccc 1620
aggtcgtggc agggtctaga gtgggatgtc ccatggcccc catccaggcc tggggatatc 1680
ctcatccgcc tcccagaatc gggccgtggg ggacagaagg ggcctgcgtg cgggcaggga 1740
gagtattttg gctctctcct gtcttcgggg tttacaaagt gtgttgggac ttgcggggct 1800
gctctgtcca agcctgggtc tggcgtccgc gtctctgagc ctgtgagtgc gtgcgctttc 1860
ctgcgtcctc ttgactgccg gtgctggggc tctgcgtcct gcgtccgcgg gagtaaatac 1920
agcaggcgaa ggggaagctc acacaatggt ctccagcgct ctggggcagg gcttctgagg 1980
ggcgggcctg cctctgccgg gacctggagc ccccgcccct cggagaggct cctaggctga 2040
cttgggcaga gccctctggt gggccgggag ggggaaaggc tgtgttgaaa tgagcaaact 2100
gtccaggtgt caggccaagc tgggaggtga ccagcctgag gtcctccccg ctccatggcc 2160
agaaccaggg ctgacatctg ggtgtcctga gcccagctgc ccacacggcc cacctggggt 2220
cagccctatc tgagtggggg aggcggggcc tcctggggga ccagaacttt ggctggacgc 2280
caagcagagt gccag 2295
<210> 59
<211> 1404
<212> DNA
<213> 智人
<400> 59
agaagggaat atcagaagca ggacgaaagc caggtcaagt ctctttcctt aggctcccca 60
aagggacaag tactcacctc ccagagacct ggcccagcgg gtcctcatgg cagcaccacc 120
ccctcccggt gcccacgacc attcgtctcc catccggcgt tctccaggat ttccaaagac 180
gcccgtttag atccacaggt acttacctga gtccctctgc ttctccacaa ctccgactcc 240
tccccgcctc tctctttttt taaaaaaaaa atttaaacaa ttcccaaata atatttaata 300
ggaaagaaga aggaaaagga gcgcacagga agggcggagg cggcacccgg gggggactgt 360
ccccagagga ggccgctgtg agccggcgac gcgaggctgg gggagtggga ggcaaacccg 420
ctaacctgtc gtcgaatggc cactcccagt tctccgctca cgagggtgga aaggcagaag 480
gcttgaaggc aaggcgtgag ggagcgccca ggacgctctc ggaggggccg ggccggggtc 540
tgcgctgcag cccgcacgca cctcacttcc gcgtcgcggc gctcggtcgc tcgccccctc 600
tcttgggccc cttctggtcg tcgccgtcct cctcctccta gtcctcctcc ttctccttct 660
cctcggctct ccgcccccac cgctgatttg tcagcgcttc tgcccgcccc tctcccggcg 720
ctcgctgctt tccctgcagc gtgctcctcg tccctatctc ggatggggat ggggcagggg 780
gcgcggggtg agccatcaac tccagccgcc tgcctggccg cgcaaggcgg gaaagtgggg 840
gcgcttttgc gcctcacgtt aagtttaggg tcacgagcac tcttgtggag atcgggagcg 900
gttgggctag ggatgatgcc tcttcctctt tccccttccc ctcccttctt gtccccgcct 960
ttcctctggg tcctcccggg gaccaccccc tacccctcgt cagagtcgcc ctgggagagc 1020
cgagggcctg agggtcgacc accaagggca gagcctatcc cttggggagc tgctggagga 1080
gacaggcagc gccgggaggt ggctcacacc gcaccgccga gagcgctttg ccctcgcatc 1140
gctgaaattt aatcacggtc acaggttaca acgttagggg tcgctttagt tcaaatatct 1200
tcaaggtttt cgtttgctct taatctagga agagccggaa agggtcctga gggtgaaagg 1260
gagactacta aggaagagtc acaggttgag gaggcaggag aaggctcagc aaatcctctt 1320
tccacgccac tatcaccatt ttcctttcca ccaatcagcg cctgccagac gctgattttc 1380
ccatcagtgg aacaaggaat aaat 1404
<210> 60
<211> 1756
<212> DNA
<213> 智人
<400> 60
caaaggctgt aacaacctgg aacgcaaatt cgttaaatta tgatacaata aggccaacct 60
cgaggatcct cggtggacta ctagcgcact tccttacaat tagaaagtga aaaactgggt 120
cagccctaga gtcacgcaat aatgctgcgt cacgaaactg cgcattccaa gagaaacttt 180
tctcaggccc agaattaagt cggagggatc cgccgcgcag ctcaatccct aggaagcatg 240
aaagaatgta ttgtaaagag taaaatcacg cgcaatccat cccaatataa ccgcagttgt 300
tcgtgggcct tcttgcagag aaaggctgcc caaccttgac ggggccatgg gcgctgcggg 360
agggcaaaag accacagaga tctgcgaagc caaagtaaac aacggggttg gggcggcgag 420
aatgatcaat agcgatgctc cgaaaggact ccgcgatagg aatcgagcgg gaaggattcc 480
ctccatccta acagtccatg ggttaagagg ccgaagcttc cctaaatacc acactcccgc 540
gagagaaggg actgagaaca acttctcaaa ctattctctc tcgaaatcgc tcccttcctc 600
gaaaattcca tctctgagac tcggatgagg tccccacccc ctccaccctt gtcccgtgac 660
cgtcgcccgc tcagcctccc agccgagtcc gcggggcctg gggcgcccac cccgcccacc 720
caagggaccg cgcaggggtg aactcccccg cgccccacct gcgccttcca gacctcgggc 780
gccccggcgt tgcctcgaga gctccctgcg cggccgccgc ggcacggacc agctcccact 840
cccttacact gggcgccgct gcgctcgccg ggggccggtc ccgaggttcc caaggcctcg 900
cgcgcgcgct tgccgtggca accaagacgt tccacgacgc gcgctctcga acgcttcgcg 960
tcacgcggcc gcgcggcccc gcccgtcggc ctcgctcccg ccacagagcc cgcagcacgc 1020
cgccgccgca gcctaggtca cgtgagtacc cacgcgcgcg tcttgccagc ggattcatca 1080
ccggcctgct cagactaggt tctgcccact ctgaccttct aaatggtacg tgggaggacg 1140
tccgtcccct tcggacccaa gagtcaccgt aacactctag aaggggagaa aaggagcgag 1200
ggcggcaggc gacagagaac ctcgcgagtc agcggccccg cgcagacccc cccaggcacg 1260
gtcccctgcg gccacgtcgg ctgctcggcg cctgcgcaat ctctttctct ccagcgaaac 1320
cgaggcctcc ggagagccta gtagagagtg tgggcagtga gcgcttgtag ccgctagagg 1380
gagcgctggg cacagtgcac gagagacaat aaaggctgat tatcccctca atgtctctgc 1440
agcctgagct ttgtgacttc tgtatccaga gcacagttaa tttccaaaag cccgcctgca 1500
ccacgtgttt taagtcttgg tgtgtgtgta aagtctatat cactatcata aaactgttag 1560
ttcctgattc cgggatacaa agagtaaaat caagatgaac atactttacc cttcttgaca 1620
agtgttttta aattagtaaa atattgtaga tgacagactc ttcaataggg ctgccttagg 1680
gaaaaaaaaa cagaaaaaag aaaaagcagt atgtacttgc ccggagcatt tagaaccagt 1740
tttgttgttg ttgtgt 1756
<210> 61
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-C9orf72-crRNA#1的靶特异性序列
<400> 61
ccguaagaca cuguuaagug cauu 24
<210> 62
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-C9orf72-crRNA#2的靶特异性序列
<400> 62
uauucagaaa caggagggag gucc 24
<210> 63
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-CNRIP1-crRNA#1的靶特异性序列
<400> 63
gcuuguauuc cuccauucuc gcuu 24
<210> 64
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-CNRIP1-crRNA#2的靶特异性序列
<400> 64
cacagauuuu gaagggugag cuau 24
<210> 65
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-DMPK-crRNA#1的靶特异性序列
<400> 65
uugugcauga cgcccugcuc uggg 24
<210> 66
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-DMPK-crRNA#2的靶特异性序列
<400> 66
cugcuggccu cuccagccuu cuca 24
<210> 67
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-EGFR-crRNA#1的靶特异性序列
<400> 67
gagaggcuaa gugucccacu gccc 24
<210> 68
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-EGFR-crRNA#2的靶特异性序列
<400> 68
agccaaccaa aauauuaaac cugu 24
<210> 69
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-FBN1-crRNA#1的靶特异性序列
<400> 69
ugccccaaag gagaggacgu gguu 24
<210> 70
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-FBN1-crRNA#2的靶特异性序列
<400> 70
guggcuuuga aagguauagu auuu 24
<210> 71
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-HTT-crRNA#1的靶特异性序列
<400> 71
ugggagugca ccccugcucu gacc 24
<210> 72
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-HTT-crRNA#2的靶特异性序列
<400> 72
ggcccgcugc agcucccugu cccg 24
<210> 73
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-INA-crRNA#1的靶特异性序列
<400> 73
caguuacagu gaggaccugc agag 24
<210> 74
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-INA-crRNA#2的靶特异性序列
<400> 74
ugucccugcc ccagcccuuc aaac 24
<210> 75
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-MAL-crRNA#1的靶特异性序列
<400> 75
ccugccuguu gacugccggu guuu 24
<210> 76
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-MAL-crRNA#2的靶特异性序列
<400> 76
cggcacccca cccucagaag uguc 24
<210> 77
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-NDRG4.1-crRNA#1的靶特异性序列
<400> 77
gucccagaug aagucccacu ccag 24
<210> 78
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-NDRG4.1-crRNA#2的靶特异性序列
<400> 78
uuaaugaaga aggagguggc auua 24
<210> 79
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-NDRG4.2-crRNA#1的靶特异性序列
<400> 79
cugcagauau gucacccacc cccc 24
<210> 80
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-NDRG4.2-crRNA#2的靶特异性序列
<400> 80
gaaggcaccg cccugggcuc ucga 24
<210> 81
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-SEPT9.1-crRNA#1的靶特异性序列
<400> 81
cgaaggaagg ugugaaggaa ggag 24
<210> 82
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-SEPT9.1-crRNA#2的靶特异性序列
<400> 82
gcuggacgcc aagcagagug ccag 24
<210> 83
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-FMR1-crRNA#1的靶特异性序列
<400> 83
cauuccacug ugaaacaaac cuca 24
<210> 84
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-FMR1-crRNA#2的靶特异性序列
<400> 84
cggucuagca uugggacuuc ggag 24
<210> 85
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-SNCA-crRNA#1的靶特异性序列
<400> 85
guccugcuuc ugauauuccc uucu 24
<210> 86
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-SNCA-crRNA#2的靶特异性序列
<400> 86
ccaucagugg aacaaggaau aaau 24
<210> 87
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-SPG20-crRNA#1的靶特异性序列
<400> 87
cguuccaggu uguuacagcc uuug 24
<210> 88
<211> 24
<212> RNA
<213> 人工序列
<220>
<223> Cpf1-SPG20-crRNA#2的靶特异性序列
<400> 88
gaaccaguuu uguuguuguu gugu 24
<210> 89
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9 gRNA-FMR1#1(FMR1)的靶特异性序列
<400> 89
cagacugcgc uacuuugaac 20
<210> 90
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9 gRNA-FMR1#2(FMR1)的靶特异性序列
<400> 90
gcgcuagggc cucucggagu 20
<210> 91
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-CNRIP1-crRNA#1的靶特异性序列
<400> 91
cuccaacccc cgggcccgcu 20
<210> 92
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-CNRIP1-crRNA#4的靶特异性序列
<400> 92
aaauuuaaau caccggcuag 20
<210> 93
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-MAL-crRNA#1的靶特异性序列
<400> 93
gggagcaacc uccuacucuc 20
<210> 94
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-MAL-crRNA#4的靶特异性序列
<400> 94
cgacaccgug agcgcuuauc 20
<210> 95
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-HTT-crRNA#2的靶特异性序列
<400> 95
acuaaucaca gugccucugu 20
<210> 96
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-HTT-crRNA#3的靶特异性序列
<400> 96
agcagcggcu gugccugcgg 20
<210> 97
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-SNCA-crRNA#1的靶特异性序列
<400> 97
agacgcccgu uuagauccac 20
<210> 98
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-SNCA-crRNA#3的靶特异性序列
<400> 98
ggugauagug gcguggaaag 20
<210> 99
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-EGFR-crRNA#1的靶特异性序列
<400> 99
cugucucugc acccggaguu 20
<210> 100
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-EGFR-crRNA#3的靶特异性序列
<400> 100
agccccauuu ugaaacacca 20
<210> 101
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-C9orf72-crRNA#1的靶特异性序列
<400> 101
gcgccaacgc uccuccagag 20
<210> 102
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-C9orf72-crRNA#3的靶特异性序列
<400> 102
cucggagcug ugaagcaacc 20
<210> 103
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-INA-crRNA#2的靶特异性序列
<400> 103
agaagagggg auagggucaa 20
<210> 104
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-INA-crRNA#3的靶特异性序列
<400> 104
cuggggccca guuuuaccag 20
<210> 105
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-SPG20-crRNA#1的靶特异性序列
<400> 105
caauuagaaa gugaaaaacu 20
<210> 106
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-SPG20-crRNA#3的靶特异性序列
<400> 106
acaccaagac uuaaaacacg 20
<210> 107
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-FBN1-crRNA#2的靶特异性序列
<400> 107
uaguuuaacc acgaacgggg 20
<210> 108
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-FBN1-crRNA#4的靶特异性序列
<400> 108
augaacccca gucuuaaucu 20
<210> 109
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-NDRG4.1-crRNA#2的靶特异性序列
<400> 109
cuuucccgcu cagauaaugg 20
<210> 110
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-NDRG4.1-crRNA#4的靶特异性序列
<400> 110
cccccucccc caggugucgg 20
<210> 111
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-NDRG4.2-crRNA#2的靶特异性序列
<400> 111
gccaucaggc cagggggcgg 20
<210> 112
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-NDRG4.2-crRNA#4的靶特异性序列
<400> 112
gacgugauca ggcucuacua 20
<210> 113
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-SEPT9.1-crRNA#1的靶特异性序列
<400> 113
ucaggaggcc cuuauugugg 20
<210> 114
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-SEPT9.1-crRNA#4的靶特异性序列
<400> 114
guucuggcca uggagcgggg 20
<210> 115
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-DMPK-crRNA#2的靶特异性序列
<400> 115
uccacgucag ggccucagcc 20
<210> 116
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> Cas9-DMPK-crRNA#3的靶特异性序列
<400> 116
ggaucauugc aggagcuaug 20
<210> 117
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 用于寡核苷酸结合的靶特异性序列(Cpf1-crRNA的20bp + 5bp)(C9orf72-左侧)
<400> 117
gatgcgctga gggttcgtgt gtttttgaat gcacttaaca gtgtctt 47
<210> 118
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 用于寡核苷酸结合的靶特异性序列(Cpf1-crRNA的20bp+ 5bp)(C9orf72-右侧)
<400> 118
ccattgtctt atgaggccca gggtgcagga cctccctcct gtttctg 47
<210> 119
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 用于寡核苷酸结合的靶特异性序列(Cpf1-crRNA的20bp + 5bp)(CNRIP1-左侧)
<400> 119
gatgcgctga gggttcgtgt gtgagggaag cgagaatgga ggaatac 47
<210> 120
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 用于寡核苷酸结合的靶特异性序列(Cpf1-crRNA的20bp + 5bp)(CNRIP1-右侧)
<400> 120
ccattgtctt atgaggccca ggggttaata gctcaccctt caaaatc 47
<210> 121
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 用于寡核苷酸结合的靶特异性序列(Cpf1-crRNA的20bp + 5bp)(DMPK-左侧)
<400> 121
gatgcgctga gggttcgtgt gtcgctcccc agagcagggc gtcatgc 47
<210> 122
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 用于寡核苷酸结合的靶特异性序列(Cpf1-crRNA的20bp + 5bp)(DMPK-右侧)
<400> 122
ccattgtctt atgaggccca ggattcctga gaaggctgga gaggcca 47
<210> 123
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 用于寡核苷酸结合的靶特异性序列(Cpf1-crRNA的20bp + 5bp)(EGFR-左侧)
<400> 123
gatgcgctga gggttcgtgt gttacagggg cagtgggaca cttagcc 47
<210> 124
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 用于寡核苷酸结合的靶特异性序列(Cpf1-crRNA的20bp + 5bp)(EGFR-右侧)
<400> 124
ccattgtctt atgaggccca gggtaagaca ggtttaatat tttggtt 47
<210> 125
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 用于寡核苷酸结合的靶特异性序列(Cpf1-crRNA的20bp + 5bp)(FBN1-左侧)
<400> 125
gatgcgctga gggttcgtgt gtcatttaac cacgtcctct cctttgg 47
<210> 126
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 用于寡核苷酸结合的靶特异性序列(Cpf1-crRNA的20bp + 5bp)(FBN1-右侧)
<400> 126
ccattgtctt atgaggccca gggccacaaa tactatacct ttcaaag 47
<210> 127
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 用于寡核苷酸结合的靶特异性序列(Cpf1-crRNA的20bp + 5bp)(FMR1-左侧)
<400> 127
gatgcgctga gggttcgtgt gtaaagttga ggtttgtttc acagtgg 47
<210> 128
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 用于寡核苷酸结合的靶特异性序列(Cpf1-crRNA的20bp + 5bp)(FMR1-右侧)
<400> 128
ccattgtctt atgaggccca gggagctctc cgaagtccca atgctag 47
<210> 129
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 用于寡核苷酸结合的靶特异性序列(Cpf1-crRNA的20bp + 5bp)(HTT-左侧)
<400> 129
gatgcgctga gggttcgtgt gtcggaaggt cagagcaggg gtgcact 47
<210> 130
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 用于寡核苷酸结合的靶特异性序列(Cpf1-crRNA的20bp + 5bp)(HTT-右侧)
<400> 130
ccattgtctt atgaggccca ggcccgccgg gacagggagc tgcagcg 47
<210> 131
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 用于寡核苷酸结合的靶特异性序列(Cpf1-crRNA的20bp + 5bp)(INA-左侧)
<400> 131
gatgcgctga gggttcgtgt gtgctcgctc tgcaggtcct cactgta 47
<210> 132
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 用于寡核苷酸结合的靶特异性序列(Cpf1-crRNA的20bp + 5bp)(INA-右侧)
<400> 132
ccattgtctt atgaggccca ggtctttgtt tgaagggctg gggcagg 47
<210> 133
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 用于寡核苷酸结合的靶特异性序列(Cpf1-crRNA的20bp + 5bp)(MAL-左侧)
<400> 133
gatgcgctga gggttcgtgt gtacatgaaa caccggcagt caacagg 47
<210> 134
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 用于寡核苷酸结合的靶特异性序列(Cpf1-crRNA的20bp + 5bp)(MAL-右侧)
<400> 134
ccattgtctt atgaggccca ggacgctgac acttctgagg gtggggt 47
<210> 135
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 用于寡核苷酸结合的靶特异性序列(Cpf1-crRNA的20bp + 5bp)(NDRG4.1-左侧)
<400> 135
gatgcgctga gggttcgtgt gtactccctg gagtgggact tcatctg 47
<210> 136
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 用于寡核苷酸结合的靶特异性序列(Cpf1-crRNA的20bp + 5bp)(NDRG4.1-右侧)
<400> 136
ccattgtctt atgaggccca ggacccataa tgccacctcc ttcttca 47
<210> 137
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 用于寡核苷酸结合的靶特异性序列(Cpf1-crRNA的20bp + 5bp)(NDRG4.2-左侧)
<400> 137
gatgcgctga gggttcgtgt gttggttggg gggtgggtga catatct 47
<210> 138
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 用于寡核苷酸结合的靶特异性序列(Cpf1-crRNA的20bp + 5bp)(NDRG4.2-右侧)
<400> 138
ccattgtctt atgaggccca ggagacgtcg agagcccagg gcggtgc 47
<210> 139
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 用于寡核苷酸结合的靶特异性序列(Cpf1-crRNA的20bp + 5bp)(SEPT9.1-左侧)
<400> 139
gatgcgctga gggttcgtgt gtcctagctc cttccttcac accttcc 47
<210> 140
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 用于寡核苷酸结合的靶特异性序列(Cpf1-crRNA的20bp + 5bp)(SEPT9.1-右侧)
<400> 140
ccattgtctt atgaggccca ggagccactg gcactctgct tggcgtc 47
<210> 141
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 用于寡核苷酸结合的靶特异性序列(Cpf1-crRNA的20bp + 5bp)(SEPT9.2-左侧)
<400> 141
gatgcgctga gggttcgtgt gtcagccagc aagcacctgg ggtagag 47
<210> 142
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 用于寡核苷酸结合的靶特异性序列(Cpf1-crRNA的20bp + 5bp)(SEPT9.2-右侧)
<400> 142
ccattgtctt atgaggccca ggggtgataa cttgagcgga gtgattt 47
<210> 143
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 用于寡核苷酸结合的靶特异性序列(Cpf1-crRNA的20bp + 5bp)(SNCA-左侧)
<400> 143
gatgcgctga gggttcgtgt gttgtggaga agggaatatc agaagca 47
<210> 144
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 用于寡核苷酸结合的靶特异性序列(Cpf1-crRNA的20bp + 5bp)(SNCA-右侧)
<400> 144
ccattgtctt atgaggccca ggggaatatt tattccttgt tccactg 47
<210> 145
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 用于寡核苷酸结合的靶特异性序列(Cpf1-crRNA的20bp + 5bp)(SPG20-左侧)
<400> 145
gatgcgctga gggttcgtgt gttatttcaa aggctgtaac aacctgg 47
<210> 146
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 用于寡核苷酸结合的靶特异性序列(Cpf1-crRNA的20bp + 5bp)(SPG20-右侧)
<400> 146
ccattgtctt atgaggccca ggcaaaaaca caacaacaac aaaactg 47
<210> 147
<211> 41
<212> DNA
<213> 人工序列
<220>
<223> 寡核苷酸PS1455(用于SEPT9.1的环)
<400> 147
ggtacggtct cacgctagcc actggcactc tgcttggcgt c 41
<210> 148
<211> 41
<212> DNA
<213> 人工序列
<220>
<223> 寡核苷酸PS1456(用于SEPT9.1的3'磷酸化表面)
<400> 148
ggtacggtct caattcccta gctccttcct tcacaccttc c 41
<210> 149
<211> 35
<212> DNA
<213> 人工序列
<220>
<223> 寡核苷酸PS1457(用于SEPT9.1的5'生物素)
<400> 149
catcgatctg atgcagtcta cttcctgctc tcagg 35
<210> 150
<211> 41
<212> DNA
<213> 人工序列
<220>
<223> 寡核苷酸PS1458(用于SEPT9.2的环)
<400> 150
ggtacggtct cacgctggtg ataacttgag cggagtgatt t 41
<210> 151
<211> 41
<212> DNA
<213> 人工序列
<220>
<223> 寡核苷酸PS1459(用于SEPT9.2的3'磷酸化表面)
<400> 151
ggtacggtct caattccagc cagcaagcac ctggggtaga g 41
<210> 152
<211> 36
<212> DNA
<213> 人工序列
<220>
<223> 寡核苷酸PS1460(用于SEPT9.2的5'生物素)
<400> 152
catcgatctg atgcactggc tgcaggtctg aatggc 36
<210> 153
<211> 41
<212> DNA
<213> 人工序列
<220>
<223> 寡核苷酸PS1711(用于C9orf72的环)
<400> 153
ggtacggtct cacgctgtgc aggacctccc tcctgtttct g 41
<210> 154
<211> 41
<212> DNA
<213> 人工序列
<220>
<223> 寡核苷酸PS1712(用于C9orf72的3'-磷酸化表面)
<400> 154
ggtacggtct caattctttt gaatgcactt aacagtgtct t 41
<210> 155
<211> 35
<212> DNA
<213> 人工序列
<220>
<223> 寡核苷酸PS1713(用于C9orf72的5'生物素)
<400> 155
catcgatctg atgcagccca ctgcctacca agcac 35
<210> 156
<211> 35
<212> DNA
<213> 人工序列
<220>
<223> 寡核苷酸PS1430(用于FMR1的5'生物素)
<400> 156
catcgatctg atgcagcaag gaggtgcatc ggccc 35
<210> 157
<211> 45
<212> DNA
<213> 人工序列
<220>
<223> 寡核苷酸PS145'(用于FMR1的3'磷酸化表面)
<400> 157
ggtacggtct caattcaaag ttgaggtttg tttcacagtg gaatg 45
<210> 158
<211> 41
<212> DNA
<213> 人工序列
<220>
<223> 寡核苷酸PS1393(用于FMR1的环)
<400> 158
ggtaccgtct caaagcgagc tctccgaagt cccaatgcta g 41
<210> 159
<211> 26
<212> DNA
<213> 人工序列
<220>
<223> 寡核苷酸PS1472(用于SEPT9.1和C9orf72的5'磷酸化的环)
<400> 159
agcggcactg agatttttct cagtgc 26
<210> 160
<211> 26
<212> DNA
<213> 人工序列
<220>
<223> 具有5'磷酸化基团的寡核苷酸PS189
<400> 160
aatggcactg agcttttgct cagtgc 26
- 使用位点特异性核酸酶优化核酸的体外分离
- 位点特异性核酸酶活性的DNA检测方法
