一种筛选与目的化合物合成相关的基因的方法与应用
文献发布时间:2023-06-19 12:27:31
技术领域
本发明属于生物基因技术领域,特别涉及一种筛选与目的化合物合成相关的基因的方法与应用。
背景技术
右旋龙脑,又称天然冰片,是一种双环单萜类化合物,具有抗菌、抗炎、止痛、促进药物透过血脑屏障、提高其他药物的吸收利用率、保护心脑等器官。用于预防和治疗疾病已有2000多年历史。
现在市场上的天然右旋龙脑主要来自于龙脑樟(Cinnamomum camphora)以及龙脑型阴香(Cinnamomum burmanni)—梅片树中的提取。但是龙脑樟优良品质极少,且出苗量偏低,管理费时费力,经济和时间成本较高;而相比之下梅片树则具有较强的生命力,成活率较高,是目前提取天然右旋龙脑的主要植物来源。但是直接从植物中提取右旋龙脑,对植物的生长、产量有着很大的要求,同时受到环境、自然资源、大量的时间和经济成本等因素的制约,不能大量的从植物中获得右旋龙脑。为了解决这一问题,研究者运用植物细胞技术来改善现状,通过组织培养来促使优良植株的规模化生产、优良品种的培育、次生代谢产物生产等。而获取阴香中右旋龙脑合成的相关基因,有目的的对阴香的组织培养具有重大的意义。
目前常用的转录组测序技术主要有二代测序和三代全长转录组测序。在之前玉米的研究中,Wang B等利用二代的数据,通过Cufflinks以及Trinity这两款不同的组装软件去评估二代能否组装出三代的效果,评估的标准是以能否重复出三代转录本中的保守的剪切点的位置为基础,最终的结果显示Cufflinks的组装效果略好于Trinity,但是组装出来的转录本均不多,只有三代转录本的三分之一,说明二代数据组装的效果并不好。与二代测序相比,三代测序的读长更长,嵌合体相对较少,能够较为准确的得到完整的转录本信息,增加表达量、可变剪切、基因融合等分析的准确性;但是三代测序不能得到基因的表达量不能进行差异表达基因的分析。
因此,有必要研究一种能克服两种测序方法缺陷的方法。
发明内容
本发明的首要目的在于克服了仅用二代转录组测序和三代转录组测序技术在筛选萜类物质合成相关基因方面的缺点和不足,提供一种筛选与目的化合物合成相关的基因的方法。
本发明的另一目的在于提供上述筛选与目的化合物合成相关的基因的方法的应用。
为达到上述目的,本发明主要提供了如下的技术方案:
一种筛选与目的化合物合成相关的基因的方法,包括如下步骤:
(1)选择目的化合物表达量有差异的不同样本,提取得到不同样本的总RNA;
(2)对步骤(1)得到的不同样本的总RNA分别进行三代全长转录组测序;
(3)对步骤(2)测序得到的原始数据进行分析,去冗余聚类,并用非全长序列对其进行校正,得到全长转录本;
(4)对步骤(1)得到的不同样本的总RNA分别进行二代测序,对下机数据进行数据质控;
(5)将步骤(3)得到的三代转录组的测序数据和步骤(4)得到的二代转录组的测序数据进行比对,以三代测序为参考,将二代的测序结果比对到三代,在二代数据中得到全长序列,降低二代测序中的拼接错误以及降低嵌合体的数量;同时可以根据二代的结果对三代测序中可能存在的碱基错误进行纠正,得到更为准确地测序结果;将三代测序得到的转录本(isoform)作为参考,在不同的蛋白数据库进行比对,得到跟给定isoform具有最高序列相似性的蛋白,对步骤(3)得到的全长转录本进行基因功能注释和基因结构分析,得到目的化合物合成过程中的相关基因;
(6)用RSEM进行序列比对定量,统计每个样本中基因检测的情况;表达量以原始数据量(reads count)和RPKM展示,对二代测序深度进行校正,再对基因或转录本的长度进行校正,获得基因的RPKM值后,再进行后续差异表达分析;
(7)利用软件DESeq2对基因表达水平分析中得到的数据量(reads count)数据进一步分析,筛选FDR<0.05且|log2FC|>1的基因为显著差异基因,得到在合成目的化合物的过程中相对比较重要的基因。
所述的筛选目的化合物合成相关的基因的方法,还包括如下步骤:
(8)选择若干个差异表达基因进行荧光定量分析(qRT-PCR),证明步骤(7)分析得到的结果的可信度。
步骤(1)和步骤(5)中所述的目的化合物优选为樟科阴香中萜类物化合物。
步骤(1)中所述的样本优选通过如下方法确定:通过索氏提取的方法,在无水乙醇中提取不同阴香样本中的有机化合物,并通过气相质谱联用检测成分和含量,确定樟科阴香中萜类物化合物表达量有差异的不同样本。
所述的样本设置平行样本。
所述的平行样本优选为三个。
步骤(1)中所述的总RNA是OD
所述的总RNA优选通过如下方法进行质量检测:通过琼脂糖凝胶电泳判断总RNA的完整性;通过使用
步骤(2)中所述的三代全长转录组测序的测序平台优选为Pacbio Sequel。
所述的三代全长转录组测序的步骤优选如下:
1)对总RNA进行逆转录,得到第一链cDNA;
2)对第一链cDNA进行PCR,扩增得到含双链cDNA的PCR产物;
3)对步骤2)得到的PCR产物进行纯化;
4)SMRTbell文库构建:包括DNA损伤修复、末端修复、连接适配体、文库质量评估;
5)将SMRTbell文库退火结合引物和聚合酶,采用MagBead Loading上机测序。
所述的三代全长转录组测序的文库构建更优选通过Clontech SMARTer PCR cDNASynthesis Kit实现。
步骤(3)中所述的分析优选为通过SMRT Link v6.0进行。
步骤(3)中所述的全长转录本优选通过如下步骤获得:提取CCS(CircularConsensus Sequence)序列,根据CCS序列是否含3’端引物和5’端引物以及是否嵌合对CCS进行分类,对全长序列进行去冗余聚类,并用非全长序列对其进行校正,得到全长转录本。
步骤(4)中所述的二代测序优选包括如下步骤:
A、从总RNA中分离得到具有polyA尾巴的真核mRNA;接着将真核mRNA片段化,以片段化的mRNA为模板,逆转录得到cDNA第一条链;随后用RNaseH降解RNA链,并在DNApolymerase I体系下,以dNTPs为原料合成cDNA第二条链,接着纯化,得到纯化后的双链cDNA;
B、将步骤A最终得到的双链cDNA经过末端修复、加A尾并连接测序接头,筛选150-250bp的cDNA,进行PCR扩增,将得到的PCR产物纯化,获得文库;
C、将步骤B得到的文库进行二代测序。
步骤A中所述的具有polyA尾巴的真核mRNA优选通过使用带有Oligo(dT)的磁珠富集。
步骤A中所述的片段化优选为通过超声波实现。
步骤A中所述的逆转录中的引物为随机寡核苷酸,逆转录酶为M-MuLV逆转录酶。
步骤B中所述的筛选优选为使用AMPure XP beads进行。
步骤B中所述的纯化优选为使用AMPure XP beads进行。
步骤(4)中所述的二代测序的平台优选为Illumina平台。
步骤(4)中所述的数据质控优选包括如下步骤:对下机的原始数据(raw reads)利用fastp进行质控,去除含适配体(adapter)的数据(reads),去除含N比例大于10%的数据(reads),去除全部都是A碱基的数据(reads),去除低质量数据(reads)(质量值Q≤20的碱基数占整条read的50%以上),得到干净数据(clean reads)。
步骤(5)中所述的蛋白数据库包括NR、SwissProt、KEGG和COG/KOG。
步骤(5)中所述的基因功能注释和基因结构分析主要包括NR的富集注释、KEGGPathway富集分析以及Gene Ontology功能注释。
步骤(8)中所述的若干个优选为12个。
上述筛选与目的化合物合成相关的基因的方法在筛选阴香中萜类物质合成相关基因中的应用。
与现有常用的二代转录组测序技术分析阴香中萜类物质合成相关的基因相比,本发明的优点及有益效果是:
(1)本发明提供了一种筛选阴香中萜类物质合成相关酶的方法,将三代转录组测序结果得到的Isoform作为参考转录本,然后将二代测序数据比对到Isoform获得各转录本丰度。再使用RSEM软件对二代转录组测序得到的Isoform进行定量,进行基因间的差异表达分析,获得萜类物质合成过程中的关键基因。
(2)相较于单独的使用二代转录组测序技术和三代全长转录组测序技术,本发明既能充分利用三代转录组无需拼接即可得到全长转录本序列从而准确性高的优点,又能充分考虑二代转录组测序数据对基因定量表达检测的优点,大大提高了基因组注释的准确性,通过KEGG、GO注释等筛选得到阴香中萜类物质合成途径中的相关基因。
(3)通过本发明提供的方法,注释得到阴香中萜类化合物合成酶的相关基因,并且首次得到4条与单萜类物质合成酶相关的基因,对进一步探索右旋龙脑合成路径、后续的组织培养、通过构建细胞工厂实现右旋龙脑的优质高产等提供了一定的理论基础。
附图说明
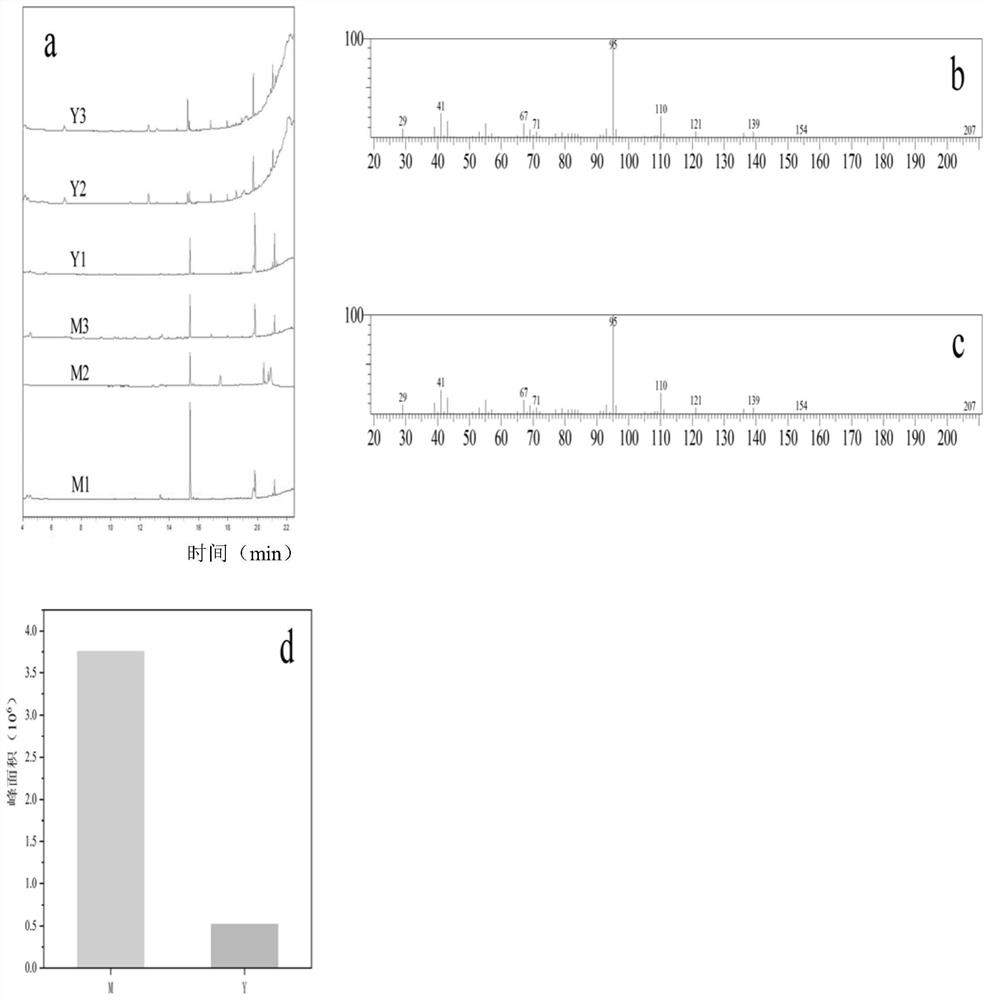
图1为阴香中化学成分的测定结果图;其中,a为6个样品的气相图;b为右旋龙脑标准品的质谱图;c为气相中12.56出峰物质的质谱图;d为两种化学型中右旋龙脑含量。
图2为三代全长转录组测序结果图;a为三代测序结果在四个数据库注释后的韦恩图统计结果图;b为三代测序结果在KEGG中的注释结果图;c为GO分类统计图。
图3为二代转录组中差异表达基因的统计图。
图4为阴香中基因参与萜类物质合成路径示意图及相关基因表达量的示意图。
图5为选取的12对差异表达基因验证转录组测序结果的qPCR分析柱状图;其中,左纵轴为通过qRT-PCR得到的基因的相对表达量,右纵坐标轴为转录组测序得到的基因表达量RPKM,M和Y中的左侧柱子对应左纵轴,右侧柱子对应右纵轴。
具体实施方式
为更进一步阐述本发明为达成预定发明目的所采取的的技术手段及功效,以下以较佳实施例,对依据本发明申请的具体实施方式、技术方案、特征及其结果,详细说明如下。
实施例1
1、阴香中次生代谢产物的提取
称取1g新鲜的阴香叶片去掉叶脉和叶柄剪碎置于60mL无水乙醇中,鲜叶样品与无水乙醇的料液比为1:60(g/mL),利用索氏提取法,在80℃的温度下,提取时间为6h,之后将挥发油定容至100mL。
2、GC-MS分析两种化学型阴香中化合物组成
气相色谱分析采用Agilent 7890A气相色谱仪(色谱柱:Agilent 19091N-113,30m×320μm×0.25μm)。载气为氮气,流速为2mL/min。GC程序:70℃/min,3℃/min升至100℃,15℃/min升至250℃,维持1min。进样量为1.0μL,不溅出。注射口温度220℃,检测温度230℃。
气相色谱-质谱联用仪使用岛津QP2010 PLUS GC/MS仪器(色谱柱:SH-RXI-5SILMS,30m×0.25mm×0.25μm)。气相色谱柱温度程序与上述相同。注射口温度280℃,EI离子源温度200℃,连接线温度250℃。MS扫描范围29~500(m/z)。
结果如图1a所示,M1、M2、M3、Y1、Y2和Y3是六个样本(六个样本是从六棵树上取得的,选择的原则是基因相近但不同)的气相图,图1b是右旋龙脑标品的质谱图,图1c是其中一个样本12.56min出峰成分的质谱图(注:其他五个样本12.56min出峰成分的质谱图与图1c相同),可以看出,12.56min左右出的就是阴香中的主要成分右旋龙脑,其中,Y1、Y2、Y3是低右旋龙脑含量型阴香,M1、M2、M3是高右旋龙脑含量型阴香;图1d是两种化学型中平均右旋龙脑含量的柱形图(M代表M1、M2、M3中右旋龙脑含量的平均值,Y代表Y1、Y2、Y3中右旋龙脑含量的平均值),可以看出,高右旋龙脑脑含量型阴香中右旋龙脑含量约是低右旋龙脑含量型阴香的8倍左右。
实施例2
1、RNA的提取
采用TaKaRa MiniBEST Plant RNA Extraction Kit试剂盒。
(1)将新鲜的或超低温冻存的植物组织样品迅速转移至用液氮预冷的研钵中,用研杵研磨组织,其间不断加入液氮,直至研磨成粉末状(无明显的可见颗粒,如果没有研磨彻底会影响RNA的收率和质量)。将研磨成粉末状的样品(20~50mg)加入到含有450μLBuffer PE的1.5mL灭菌管中,用移液器反复吹打直至裂解液中无明显沉淀。
(2)将裂解液12,000rpm,4℃离心5分钟。
(3)将上清液小心吸取到新的1.5mL灭菌管中。加入上清液1/10体积的Buffer NB(此时会出现沉淀),振荡混匀。
(4)12,000rpm、4℃离心5分钟。
(5)将上清液小心吸取到新的1.5mL灭菌管中。加入450μL的Buffer RL(使用前请确认已加入50×DTT溶液),使用移液枪将溶液混合均匀。
(6)加入样品裂解步骤中上清液或混合液1/2体积的无水乙醇(此时可能会出现沉淀),使用移液枪将溶液混合均匀。
(7)立即将混合液(含沉淀)全部转入到RNA纯化柱(含2mL收集管)中。
(8)12,000rpm,离心1分钟,弃滤液。将RNA纯化柱放回到2ml收集管中。
(9)将500μL的Buffer RWA加入至RNA纯化柱中,12,000rpm离心30秒钟,弃滤液。
(10)将600μL的Buffer RWB加入至RNA纯化柱中,12,000rpm离心30秒钟,弃滤液。
(11)DNase I消化(可选择):利用本试剂盒提取RNA时可以有效地去除植物组织中绝大部分的基因组DNA,提取的RNA很少含基因组DNA。若后续实验对RNA纯度要求比较严格,可以选择性地在RNA纯化柱膜上进行DNase I消化。
①DNase I反应液的配制:取5μL 10×DNase I Buffer,4μL重组DNase I(无RNase,5U/μL),41μL无RNA酶dH
②向RNA纯化柱膜中央加入50μL DNase I反应液,室温静置15分钟。
③向RNA纯化柱膜中央加入350μL的Buffer RWB,12,000rpm离心30s,弃滤液。
(12)重复操作步骤(10)。
(13)将RNA纯化柱重新安置于2mL收集管上,12,000rpm离心2分钟。
(14)将RNA纯化柱安置于1.5mL无RNase收集管(试剂盒提供)上,在RNA纯化柱膜中央处加入50~200μL的无RNA酶dH
(15)12,000rpm离心2分钟洗脱RNA。
2、RNA质量检测
使用1.2%的琼脂糖凝胶电泳检测RNA的完整性及污染程度,使用Nanodrop 2000检测RNA纯度,结果如表1所示。260/280和260/230都在1.8~2.0之间,说明RNA的完整性和纯度都符合要求。
表1:6个样品中RNA在260、280、230nm下的吸光值
实施例3
1、三代全长转录组测序
(1)第一链cDNA合成:检测合格后的RNA(或poly A+RNA)使用Clontech SMARTerPCR cDNA Synthesis Kit反转录为第一链cDNA;PCR扩增合成双链cDNA;PCR产物纯化:使用AMPure PB Bead对PCR扩增产物进行纯化;SMRTbell文库构建:包括DNA损伤修复、末端修复、连接适配体、文库质量评估;上机测序:将SMRTbell文库退火结合引物和聚合酶,采用MagBead Loading上机测序。
(2)利用Pacific Biosciences支持的SMRT Link v5.0.1对cDNA文库的原始测序数据(reads)
进行分类并聚集成一致的转录本。简单地说,CCS(circular consensussequence)是从BAM文件中提取出来的。然后根据cDNA引物和polyA尾信号将CCS数据分为全长非嵌合(full-length non-chimeri,FL)数据、非全长(non-full-length,nFL)数据、嵌合短数据。短读被丢弃。随后,通过迭代聚类纠错(ICE)软件对全长非嵌合(FLNC)读段进行聚类,生成聚类一致性异构体。为了提高PacBio数据的准确性,采用了两种策略。一种是,利用非全长数据通过Quiver软件对上述获得的聚类一致性亚型进行抛光,得到FL抛光的高质量一致性序列(准确度≥99%)。另一种是,使用LoRDEC工具(version 0.8)从相同样品中获得的Illumina短读码进一步校正低质量的异构体。然后使用CD-HIT-v4.6.7软件去除冗余序列,以0.99身份验证阈值过滤最终的转录组亚型序列。结果分别如表2和表3所示。
表2三代测序序列数据统计
表3去除冗余后的Isoform数据统计
2、二代转录组测序
正常物种中,90%以上的RNA为rRNA,因此我们需要在提取样本的总RNA后,通过常规试剂盒去除rRNA,通过带有Oligo(dT)的磁珠富集具有polyA尾巴的真核mRNA后,用超声波把mRNA打断。以片段化的mRNA为模版,随机寡核苷酸为引物,在M-MuLV逆转录酶体系中合成cDNA第一条链,随后用RNaseH降解RNA链,并在DNA polymerase I体系下,以dNTPs为原料合成cDNA第二条链。纯化后的双链cDNA经过末端修复、加A尾并连接测序接头,用AMPure XPbeads筛选200bp左右的cDNA,进行PCR扩增并再次使用AMPure XP beads纯化PCR产物,最终获得文库,并对文库进行质检。
为了保证数据质量,要在信息分析前对原始数据进行数据过滤,以减少无效数据所来带的分析干扰。首先,我们对下机的raw reads利用fastp进行质控,过滤低质量数据,1)去除含适配体的数据;2)去除含N比例大于10%的数据;3)去除全部都是A碱基的数据;4)去除低质量数据(质量值Q≤20的碱基数占整条数据的50%以上),得到干净数据(cleanreads)。
3、KEGG、GO等功能注释
将二代转录组和三代转录组的测序数据进行比对,以三代测序为参考,将二代的测序结果比对到三代,可以在二代数据中得到全长序列,降低二代测序中的拼接错误以及降低嵌合体的数量;同时可以根据二代的结果对三代测序中可能存在的碱基错误进行纠正,即纠正三代测序中存在的错误序列,得到更为准确地测序结果。
之后,通过blastx将转录本(Isoform)序列比对到蛋白数据库NR、SwissProt、KEGG和COG/KOG,得到跟给定Isoform具有最高序列相似性的蛋白,从而得到该Isoform的蛋白功能注释信息。Nr、SwissProt是两个著名的蛋白数据库,其中SwissProt是经过严格筛选去冗余的。COG/KOG是对基因产物进行直系同源分类的数据库,每个COG/KOG蛋白都被假定来自祖先蛋白,COG/KOG数据库是基于细菌、藻类、真核生物具有完整基因组的编码蛋白、系统进化关系进行构建的。KEGG是系统分析基因产物在细胞中的代谢途径以及这些基因产物的功能的数据库,用KEGG可以进一步研究基因在生物学上的复杂行为。结果如图2所示。图2b注释如下:A为环境适应;B为代谢途径,从上到下依次为全局与概述、碳水化合物代谢、能量代谢、氨基酸代谢、脂质代谢、辅助因子和维生素代谢、其他氨基酸代谢、其他次生代谢产物的生物合成、核苷酸代谢、萜类和聚酮化合物的代谢、聚糖的生物合成与代谢;C为遗传信息处理方向,从上到下依次为翻译,折叠,分类和降解,转录,复制和修复;D为环境信息处理,从上到下依次为信号传导、膜运输;细胞过程(E):运输和代谢传导。图2c注释如下:(1)生物过程,从左到右依次为1:生物粘附;2:生物调节;3:细胞杀伤;4:细胞成分的组织或生物发生;5:细胞过程;6:排毒;7:发展过程;8:生长;9:免疫系统过程;10:本土化;11:运动;12:代谢过程;13:多种生物过程;14:多细胞生物过程;15:再生产;16:生殖过程;17:对刺激的反应;18:有节奏的过程;19:发信号;20:单一生物过程。(2)细胞组分从左到右依次为1:细胞;2:细胞连接;3:细胞部分;4:细胞外基质;5:细胞外基质成分;6:细胞外区域;7:细胞外区域部分;8:大分子复合物;9:膜;10:膜部分;11:膜封闭腔;12:核苷;13:细胞器;14:细胞器部分;15:超分子纤维;16:病毒体;17:病毒体部分。(3)分子功能从左到右依次为1:抗氧化活性;2:联合剂;3;催化活性;4:电子载流子活性;5:分子功能调节剂;6:分子换能器活性;7:核酸结合转录因子活性;8:信号传感器活动;9:结构分子活性;10:转录因子活性,蛋白质联合剂;11:翻译调节器活动;12:运输活动。
使用RSEM软件对三代组装出来的Isoform进行定量。RSEM是通过构建RNA-seqreads的生成模型(generative model)和利用最大期望算法(Expectation MaximizationAlgorithm,EM algorithm)估计数据量(read counts),计算基因的丰度。和其他工具相比,RSEM最大的优势在于不需要参考基因组就可以进行定量。软件重复性好,稳定性高,定量准确。
表达量以原始数据量和RPKM展示,原始数据量表示该转录本所包含的数据数目,但受测序量和基因长度的影响,原始数据量不利于样本间的差异基因比较。为保证后续分析准确性,我们先对测序深度进行校正,再对基因或转录本的长度进行校正,获得基因的RPKM值后,再进行后续分析。
基因差异表达分析的输入数据为基因表达水平分析中得到的数据量数据(如表4所示),使用DESeq2软件分析,分析主要分为三部分:
1)对数据量进行标准化(normalization);
2)根据模型进行假设检验概率(pvalue)的计算;
3)最后进行多重假设检验校正,得到FDR值(错误发现率)
基于差异分析结果(如表5所示),我们筛选FDR<0.05且|log2FC|>1的基因为显著差异基因。结果显示有250条显著差异表达的基因,相比于M组,Y组中发现26个上调基因,224个下调基因(图3)。
表4阴香二代测序数据结果
表5二代数据在三代数据中比对率统计表
结果分析:
经过原始数据处理,获得了17116个亚型,这些亚型序列与蛋白质数据库NCBI非冗余蛋白质序列(NR),Swissprot,《京都基因与基因组百科全书》(KEGG)和蛋白质直系同源簇(COG/KOG)进行了比对,在总共1,7116个基因中,有16,516个得到注释,占96.49%。其中,在NR中观察到16481个同工型,在Swissport中观察到14544个,在COG/KOG中观察到11690个,而在COG/KOG中观察到8127个。在这些基因中,有6824条基因在四个数据库中均得到注释,占41.32%。
为了更好地理解每种转录本在全长转录本中的功能,我们通过KEGG数据库对其功能进行了注释,其中8127条基因在19个代谢途径中得到注释。根据KEGG注释结果,有246条基因在有机系统(A)中被得到注释;有6089条基因在属于代谢(B)途径中的9条代谢通路得到注释;1711条基因在4种代谢通路种得到注释,这四条代谢通路又属于遗传信息处理方向的;在环境信息处理(D)的2条代谢通路,391个基因被得到注释;在运输和分解代谢通路中,有412条基因得到注释。在所有基因中发现了132条有关萜类化合物和聚酮化合物代谢的相关基因。
在对所有的序列进行GO注释后,在三个类别(生物过程,细胞成分和分子功能)下获得了49个生物学功能注释。在生物过程中,有20术语的细胞过程被注释,其中代谢过程和单一生物过程所占比例很高。在细胞成分中,17术语被注释到,其中细胞部分和细胞器部分占很高的比例。在分子功能上,12术语得到注释,其中结合和催化活性占很高的比例。
在二代测序基因表达量的分析过程中,我们主要分析了KEGG注释中与萜类物质合成相关的基因,主要包括与萜类物质碳骨架合成相关的通路ko00900;与单萜类物质合成相关的代谢通路ko00902;与二萜物质合成相关的代谢通路ko00904;以及与三萜和倍半萜合成相关的代谢通路ko00909。分别注释到57条、4条、8条、7条基因,如图4所示。图4中的英文缩写对应的中文如下:MVA:甲羟戊酸;Acetyl-CoA:乙酰辅酶A;AACT:乙酰辅酶A乙酰基转移酶;HMGS:乙酰乙酰基辅酶A在3-羟基-3-甲基戊二酰辅酶A合成酶;HMG-CoA:3-羟基-3-甲基戊二酰辅酶A;HMGR:3-羟基-3-甲基戊二酰辅酶A还原酶;MVK:甲羟戊酸激酶;MVAP:5-磷酸-甲羟戊酸;PMK:5-磷酸-甲羟戊酸激酶;MVAPP:甲羟戊酸焦磷酸;MPD:甲羟戊酸焦磷酸脱羧酶;IPP:异戊二烯焦磷酸;DMAPP:二甲基烯丙基焦磷酸;MEP:2-甲基-D赤藻糖醇-4-磷酸;Pyruvate:丙酮酸;GA-3P:3-磷酸甘油醛;DXS:5-磷酸脱氧木酮糖合成酶;DXP:1-脱氧-D-木酮糖-5-磷酸;DXR:5-磷酸酮糖还原异构酶;MCT:2-C-甲基-D-赤藓糖醇-4-磷酸胞苷酰转移酶;CDP:磷酸胞苷;CDP-ME:4-(5-焦磷酸胞苷)-2-C-甲基-D-赤藓醇;CMK:4-(5’-焦磷酸胞苷)-2-C-甲基-D-赤藓糖醇激酶;CDP-ME2P:4-(5’-磷酸胞苷)-2-C-甲基-D-赤藓醇-2-磷酸;MDS:2-C-甲基赤藓醇-2,4-环焦磷酸合成酶;MecDP:2-C-甲基赤藓醇-2,4-环焦磷酸;HDS:羟甲基丁烯基-4-磷酸合成酶;HMBPP:1-羟基-2-甲基-2丁烯基-4-二磷酸;IDI:异戊转移酶;GPP:香叶基焦磷酸;GGPP:香叶基香叶基焦磷酸;GGPPS:香叶基香叶基焦磷酸合成酶;FPPS:法尼基焦磷酸合成酶;FPP:法尼基焦磷酸。
结合二代和三代转录组测序的结果,可以比较清晰地看出与萜类物质合成相关酶的相关基因。且通过差异表达分析,可以得到合成过程中比较重要的酶,为后续的生物代谢产生次生代谢产物提供理论基础。
实施例4
实时荧光定量PCR验证差异表达基因
1、cDNA的合成
反转录试剂盒为Takara Prime ScriptTM RT reagent Kit with gDNA Eraser(Perfect Real Time),具体操作如下表6:
表6反转录操作体系
2、qPCR实时荧光定量分析:基于SYBR法对差异表达基因进行qRT-PCR验证,所用的内参基因为EF1,是植物中常用的内参基因,二代测序结果表明,在六个样本中表达情况相对稳定。选取的12个基因为Isoform 2990、Isoform 5726、Isoform 7560、Isoform 8306、Isoform 8315、Isoform 8424、Isoform 8609、Isoform 8823、Isoform 9335、Isoform9336、Isoform 16328和Isoform 16988,表7为qPCR的反应体系,表8为引物信息和内参基因的序列。
表7 qPCR反应体系
表8引物信息
结果如图5所示,左纵轴为通过qRT-PCR得到的基因的相对表达量,右纵坐标轴为转录组测序得到的基因表达量RPKM,可见qRT-PCR的结果和测序结果基本一致,数据具有可信度。M表示M1、M2、M3的平均值,Y表示Y1、Y2、Y3的平均值。
上述实施例为本发明较佳的实施方式,但本发明的实施方式并不受上述实施例的限制,其他的任何未背离本发明的精神实质与原理下所作的改变、修饰、替代、组合、简化,均应为等效的置换方式,都包含在本发明的保护范围之内。
序列表
<110> 华南理工大学
<120> 一种筛选与目的化合物合成相关的基因的方法与应用
<160> 26
<170> SIPOSequenceListing 1.0
<210> 1
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 内参F
<400> 1
agacggttgc tgttggagtt 20
<210> 2
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 内参R
<400> 2
caccatccac ccctttgtca 20
<210> 3
<211> 18
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> Isoform 2990 F
<400> 3
ggcatcaagc catcaact 18
<210> 4
<211> 19
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> Isoform 2990 R
<400> 4
agaatcgctg gagaatcat 19
<210> 5
<211> 18
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> Isoform 5726 F
<400> 5
gcatccaaac tggcaaga 18
<210> 6
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> Isoform 5726 R
<400> 6
caatcggaga acctcaaata 20
<210> 7
<211> 18
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> Isoform 7560 F
<400> 7
gaaagcaagg gaagaggt 18
<210> 8
<211> 19
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> Isoform 7560 R
<400> 8
gtggatacaa tcggagaac 19
<210> 9
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> Isoform 8306 F
<400> 9
tagttcagac ggcgaaggac 20
<210> 10
<211> 18
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> Isoform 8306 R
<400> 10
gctcagatgc ggcaagtg 18
<210> 11
<211> 18
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> Isoform 8315 F
<400> 11
gtattcccgt tgttgatg 18
<210> 12
<211> 18
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> Isoform 8315 R
<400> 12
atttgaccta accgtgcc 18
<210> 13
<211> 18
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> Isoform 8424 F
<400> 13
cgcccttcat ctcaacct 18
<210> 14
<211> 19
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> Isoform 8424 R
<400> 14
gctcgtccca ccaatactt 19
<210> 15
<211> 18
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> Isoform 8609 F
<400> 15
gaaagcaagg gaagaggt 18
<210> 16
<211> 19
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> Isoform 8609 R
<400> 16
gtggatacaa tcggagaac 19
<210> 17
<211> 18
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> Isoform 8823 F
<400> 17
gaaagcaagg gaagaggt 18
<210> 18
<211> 19
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> Isoform 8823 R
<400> 18
gtggatacaa tcggagaac 19
<210> 19
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> Isoform 9335 F
<400> 19
tcaataatca gggcaacact 20
<210> 20
<211> 19
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> Isoform 9335 R
<400> 20
tctcatccta cgcaacacc 19
<210> 21
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> Isoform 9336 F
<400> 21
actccacctt actttcatct 20
<210> 22
<211> 18
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> Isoform 9336 R
<400> 22
cttattagca cggtttcc 18
<210> 23
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> Isoform 16328 F
<400> 23
gagcgatgac agtgaaagcg 20
<210> 24
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> Isoform 16328 R
<400> 24
ccttcatcgg ccaaatccct 20
<210> 25
<211> 19
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> Isoform 16988 F
<400> 25
tccacccttt tatcccatc 19
<210> 26
<211> 18
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> Isoform 16988 R
<400> 26
tgctctgaca agcccaat 18
- 一种筛选与目的化合物合成相关的基因的方法与应用
- 一种筛选与研究目的相关的关键基因、关键蛋白集的方法
